摘要
背景
聚半乳糖醛酸酶抑制蛋白(PGIPs)是一种富含亮氨酸重复(LRR)的植物细胞壁糖蛋白,参与植物免疫。它们通常由具有少量基因副本的基因家族编码,其进化起源研究很少。在这里,我们报告了完整的特性的充分补充pgip大豆科(大豆(L。] Merr.)和围绕的基因组区域的特征pgip四种豆科植物的家族。
结果
BAC克隆和基因组序列分析表明,大豆基因组中含有两个pgip位点。每个位点由三个聚集的基因组成,这些基因是在真菌病原体感染后诱导的菌核病sclerotiorum的残馀序列pgip基因。分析的同源大豆基因组区(约126 Kb)包括pgip基因座具有很强的保守性,这种保守性也延伸到豆类物种的基因组中菜豆lMedicago truncatulaGaertn。而且中投arietinumL.,每个包含一个单pgip轨迹。基于极大似然的基因树表明pgip在每个物种中,集群都独立地经历了串联复制。
结论
古多倍体大豆基因组包含两个pgip基因座由大而高度保守的重复区域组成,这些重复区域在bean中也是保守的。m . truncatula而且c . arietinum.这些豆科植物的基因组特征pgip家族暗示着驱动进化的力量pgip基因遵循出生-死亡模型,类似于nbs - lrr型抗性(R)基因的进化。
背景
植物细胞壁是微生物病原体在植物组织中定植的主要障碍之一。为了克服这一障碍,大多数真菌病原体产生细胞壁降解酶(CWDEs),其中聚半乳糖醛酸内源性酶(PGs;EC 3.2.1.15)在感染过程的早期阶段分泌[1].pg裂解均半乳糖醛酸中d -半乳糖醛酸残基之间的α-(1-4)键,导致细胞分离和宿主组织浸渍。为了中和PGs的活性,植物拥有细胞壁糖蛋白,称为聚半乳糖醛酸酶抑制蛋白(PGIPs),其在防御中的重要性已在不同的植物物种中得到证明[2] - [12].
与许多抗性基因的产物一样,PGIPs属于含有胞外质型富亮氨酸重复序列(LRRs)的蛋白质亚类(LxxLxLxxNxLT/ sgxipxxlxxllxx) [13].PGIP的LRR结构域通常由10个不完善的LRR组成,每个LRR有24个残基,负责与pg的分子相互作用。LRRs被组织成两张β-片,其中一张(B1片)位于分子内凹侧,含有PG识别的关键残基[14].
为了中和真菌病原体产生的许多pgip,植物进化出了各种不同特异性的pgip。由于PGIPs是由基因家族编码的,在每个植物物种中都有2个成员拟南芥[3.高达16英寸芸苔属植物显著[15].已报道的豆科成员对真菌和昆虫pg抑制特性的种内变异的一个明显例子(菜豆l .)pgip家庭(16].不同家庭成员之间的差异也延伸到他们表达的调节[17].
大豆(大豆(L。)稳定)。pgip科是由至少四个基因组成的两个簇,其中一个包含Gmpgip1而且Gmpgip2,间隔约3 kbp,另含Gmpgip3而且Gmpgip4,间隔最大60 Kbp [18].这些发现特别有趣,因为大豆是一种成熟的古多倍体植物物种。因此,对大豆的全部补体进行了表征pgip家族可以提供有关驱动该基因家族进化的力量的重要信息。
在本研究中,我们描述了pgip结果表明,大豆威廉斯82基因型中存在2个基因pgip位点。该研究扩展到其他豆科植物,通过描述约140 Kb的区域,包括单一的pgip豆荚,还有pgip在最近公布的基因组区域Medicago truncatulaGaertn。[19),中投arietinuml . (20.].对比分析pgip这些物种的区域表明豆科植物pgip家庭遵循生与死的演化模式。
结果
大豆BAC无性系的鉴定及两个新无性系的分离Gmpgip基因
七个BAC克隆,以前从大豆BAC库中分离使用pgip探针,用特异性引物进行分析Gmpgip1,Gmpgip2,Gmpgip3而且Gmpgip4[18].没有一个BAC克隆体包含全部四种pgip序列在一起。其中3个(95O4, 85 M15, 28B18)含有这两种成分Gmpgip1而且Gmpgip22个(26I2, 6 F5)两者都含有Gmpgip3而且Gmpgip4.其余两个BAC克隆(1 F11, 62 K14)没有产生任何Gmpgip引物组合的清晰扩增子;因此,没有进一步分析。每个BAC克隆中包含的插入体的大小,由脉冲场凝胶电泳(PFGE)确定不我消化,变化在大约50 Kb和190 Kb(附加文件1而且2).下面是BAC克隆的指纹后III消化显示重叠的轮廓,其中含有Gmpgip1而且Gmpgip2(95O4, 85 M15, 28B18),以及含有Gmpgip3而且Gmpgip4(26I2, 6 F5)(附加文件1).BAC端测序和霰弹枪亚克隆测序鉴定出两个新的Gmpgip基因,一个在克隆85 M15 (Gmpgip5),另一个位于克隆26I2 (Gmpgip7).Gmpgip5在85 M15末端,呈局部;对其基因组DNA进行PCR,获得其完整序列。的编码区域Gmpgip5而且Gmpgip7分别包含1008 bp和1011 bp的不间断开放阅读帧(orf),包括停止密码子。这两个orf预测的氨基酸序列显示了典型的PGIP结构,包括一个21个氨基酸的外质体分泌信号肽,10个富含亮氨酸的重复序列(LRRs),每个重复序列约有24个氨基酸,8个半胱氨酸残基,在蛋白的N端和c端各有4个(附加文件)3.).
由于指纹分析表明两组BAC克隆之间缺乏重叠部分,我们假设存在两组BAC克隆pgip位点,包括Gmpgip1,Gmpgip2而且Gmpgip5,在BAC克隆85 M15和另一个包括Gmpgip3,Gmpgip4而且Gmpgip7在BAC克隆26I2中鉴定。大豆基因组序列的最新可用性[21让我们确认了两个的存在pgip并定义了位点的空间分布Gmpgip基因(见下文)。
转录本积累在体外抑制试验Gmpgip5而且Gmpgip7
我们之前报道了基因表达模式的变异Gmpgip基因(Gmpgip1,Gmpgip2,Gmpgip3而且Gmpgip4)在大豆下胚轴感染坏死真菌病原体后菌核病sclerotiorum[18].在这项工作中,我们将研究扩展到Gmpgip5而且Gmpgip7.qRT-PCR分析显示Gmpgip5而且Gmpip7在大豆下胚轴表达,并在感染美国sclerotiorum.两者的基础转录水平Gmpgip5而且Gmpgip7比的要低得多Gmpgip3因为它的表达水平最高,所以被用作对照Gmpgip大豆下胚轴的基因[18];此外,基本的表达Gmpgip7的值高于Gmpgip5(表1).的表达Gmpgip5感染后24小时(hpi)下降美国sclerotiorum,在48 hpi(分析的最后一个时间点),当组织几乎完全浸渍时,大大增加了1000倍以上(表2).感染后,表达Gmpgip7显示在前24 hpi中有一个适度的增加,达到48 hpi的高水平(表2).Gmpgip3表现出不同的转录物积累动力学,在24 hpi时最多增加5倍,在48 h时不再增加(表2).
数据库搜索证实存在对应的表达序列标签(ESTs)Gmpgip5而且Gmpgip7并在发育过程中表现出表达的变化Gmpgip5est存在于下胚轴和荚果中Gmpgip7根和茎中存在ESTs(附加文件4).其余的无害环境技术Gmpgip基因也被鉴定Gmpgip3在大豆组织中最常见(附加文件)4).特别是,在下胚轴中发现的该基因的转录本大约多两倍Gmpgip5,证实表达水平较高Gmpgip3qRT-PCR结果显示(表1).
为了验证其抑制活性Gmpgip5而且Gmpgip7抗真菌pg,我们已经表达了这些基因烟草benthamiana利用马铃薯病毒X (PVX;[22])。使用针对豆类PGIP的抗体对总蛋白提取物进行Western blot分析,显示GmPGIP7和GmPGIP3的积累,这被用作阳性对照(附加文件)5).相反,从对照植物(未接种或用空载体接种)制备的提取物中未检测到免疫装饰信号(附加文件)5)或从感染PVX-Gmpgip5结构的植物中提取(数据未显示)。尽管付出了巨大的努力,但所有基于pvx的表达GmPGIP5的尝试都失败了。
总蛋白提取物n benthamiana然后用PVX-Gmpgip7感染的叶片检测GmPGIP7对真菌pg的抑制活性美国sclerotiorum,镰刀菌素graminearum,炭疽菌acutatum而且黑曲霉(数据未列示).未观察到抑制;相反,所有检测的pg均被GmPGIP3抑制,作为对照(附加文件5).
大豆pgip基因由两个不同的位点组成
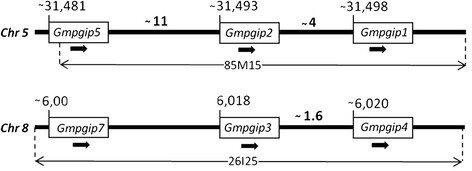
序列比较GmpgipBAC克隆中包含的基因和组装的大豆基因组序列使我们确认了两种基因的存在pgip并确定位点的排列Gmpgip它们各自的基因。每一簇pgip基因在5号染色体上有一个相似长度(约18kb)的区域(依次为Gmpgip5,Gmpgip2而且Gmpgip1)和第8号染色体(Gmpgip7,Gmpgip3而且Gmpgip4).所有基因转录方向相同(图1).除了这些转录pgip在第5号染色体和第8号染色体上分别发现2个和1个残馀序列。在5号染色体的位点上的残馀物之一[Gmpgip*(1)]是严重碎片化的,而另外两个在第5染色体上[Gmpgip*(2)]及8 (Gmpgip *)分别编码65个c端残基和220个N端残基和中间残基(附加文件6).
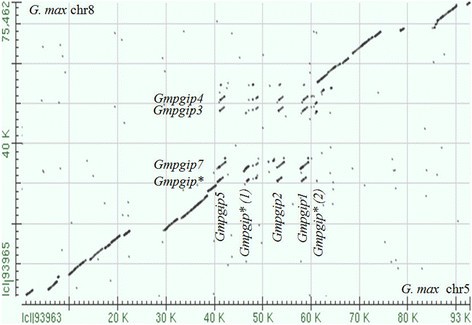
两种大豆区域的核苷酸序列pgip通过Bl2seq分析对基因座进行比较,结果表明pgip基因座在核苷酸序列上很保守,在基因顺序和方向上共线(图2).这种共线性的主要例外是由对齐中的两个主要间隙表示,这是由于LTR逆转录转座子插入在5号染色体的位点上(图2).值得注意的是,该区域包含Gmpgip拷贝显示了序列共线性的大多数例外,因为在这个区域没有清晰的对角线可见,并且对齐仅限于不同的编码区域pgip成员(图2).
在位点之间的比较中,只有Gmpgip1而且Gmpgip3,在一边,和Gmpgip2而且Gmpgip7,在另一边,3′区具有相似性,仅限近200 bp序列。不同的5 '区域Gmpgip基因也具有很强的差异性,这些区域的序列差异也反映在基因的组成上独联体元素。序列分析。限于已知独联体调节参与防御反应的基因的作用元件,表明所有六种Gmpgip基因包含与这些元素相同的序列;然而,在类型和数量上有所不同。例如Gmpgip3包含最多的W-box元素,而它缺乏匹配BIHD1OS2元素的序列(附加文件7).
bean的结构分析pgip轨迹
用于bean的特性描述pgip豆BAC克隆129 F4和10G1跨越豆pgip从BAT93基因型基因组文库中分离得到基因座。129个F4和10G1克隆分别包含约37150 bp和107473 bp的插入片段,重叠部分为5201 bp [16].这些克隆被完全测序,鉴定长度为139420 bppgip面积约50 Kb的区域。这个区域代表唯一pgip该位点存在于豆基因组中,包含4个无内含子位点Pvpgip基因(Pvpgip1蝙蝠Pvpgip2蝙蝠Pvpgip3蝙蝠和Pvpgip4蝙蝠)向同一方向转录[16].
的BAC组装序列p .寻常的BAT93被定位在最近可用的基因组序列的两个不同的基因组区域p .寻常的入世G19833。BAT93序列的主要部分(1..126624)被定位在2号染色体上[补体(36019507..36152120)],并显示包含几个注释基因(从phvul . 002g2008001到Phvul.002G201900.1),而剩余的13 Kb被定位在1号染色体上(49570303..49583535),不包含注释基因。有三种假设可以解释这种差异:1)包含这两个区域的BAC 10G1是嵌合的,即BAT93基因组的两个独立部分克隆在同一个BAC中;2)在整个基因组序列中存在涉及所分析区域的装配错误,3)发生了易位,从而分化了p .寻常的BAT3和G19833的加入。由于较短的区域仅占整个BAC组装序列的10%左右,并且显示出与主BAC序列组装可能不同的起源,因此我们将其排除在后续分析中。
为了分析包含bean的区域的结构pgip基因,Blast2seq分析(序列与自身对齐),仅限于包含四个bean的组装序列的前62 Kbpgip基因,被执行。除了对角线对齐,还有一些短对齐(附加文件8).其中包括两个逆转录转座子元件(位置约为10 Kb和40 Kb),它们显示了它们的长串联重复序列(LTR)的非对角线排列,通常由LTR逆转录转座子末端显示(附加文件)8).这四个pgip基因之间排列,只有在编码区以外的基因排列被观察到PvBpgip1而且PvBpgip2和之间的PvBpgip3而且PvBpgip4(附加文件8).相反,对齐之间pgip双PvBpgip1/PvBpgip3,PvBpgip2/PvBpgip3,PvBpgip1/PvBpgip4,PvBpgip2/PvBpgip4仅限于编码区域。
的比较pgip四种豆科植物的基因座
的集合bean序列(1..126624)pgip基因与大豆基因组和最近释放的基因组序列进行了比较m . truncatula[19),c . arietinum[20.].就像在豆子里,基因组m . truncatula而且c . arietinum只包含一个pgip轨迹。的pgip轨迹的m . truncatulaMTR_119s0023, MTR_119s0023,Mtpgip1;MTR_119s0021,Mtpgip2),一个可能的假基因,用40 bp的内含子注释以恢复正确的开放阅读框(MTR_119s0017,Mtpgip3),一个残基对应编码c端53残基PGIP片段的序列。编码的产物Mtpgip1 Mtpgip2而且Mtpgip3在附加文件中显示9.它们都含有一个向外质体分泌的信号肽,典型的10个lrr和8个半胱氨酸残基,在蛋白的N端和c端各有4个;MtPGIP1在c端区域含有一个额外的半胱氨酸(附加文件9).类似地,pgip轨迹的c . arietinum横跨约30k的区域,包含两个pgip基因(附加文件)10),其中一个(LOC101504619,Capgip2)在编码区中间被一个约17kb的片段打断。编码的假定蛋白质Capgip1(LOC101505245)还包含上面描述的典型PGIP特性(附加文件10).总的来说,这些序列分析强调了典型的PGIP结构在所有这些之间和内部是强烈保守的pgip家庭。正如预期的那样,这种保守伴随着组成每种蛋白质的LRRs和不同蛋白质的LRRs的典型变化。
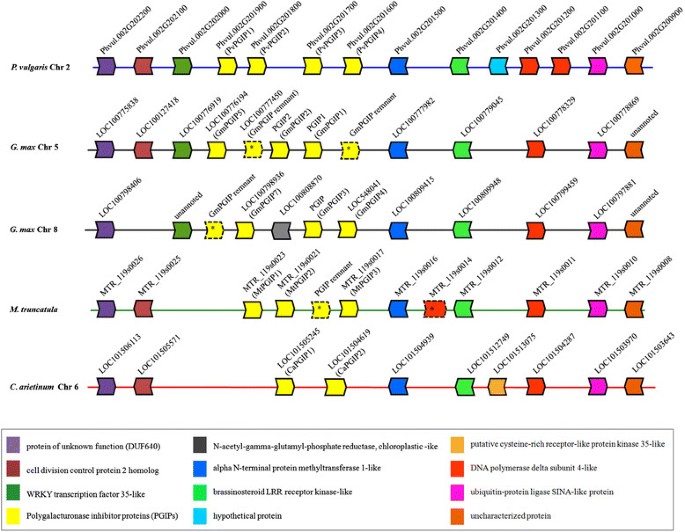
序列比较的侧翼区域pgip在所有四个物种中,聚类显示出很少的基因和非常好的保守顺序,几乎没有例外。十个豆子基因的侧翼pgip在这四种豆科植物中,只有Pv_202000和Pv_201300不保守3.).此外,Pv_201200和Pv_201100仅在大豆基因组中存在重复基因,Pv 202100在大豆8号染色体中丢失(图3.).
包含的基因组区域的示意图表示pgip位点在蝶形花科物种。基因组区域包含pgip分析基因座的共享同步性。每个方柱箭头代表一个预测基因和编码区从ATG到终止密码子的方向。彩色方块箭头是同源基因。灰色方块箭头表示没有同源基因。从左到右的基因组位置:p .寻常的2号染色体(36,203,534至36,041,993 bp),g·马克斯第5染色体(31,445,987至31,542,227 bp),g·马克斯8号染色体(59,75,212至60,52,779 bp),m . truncatula(未放置的基因组支架119)和c . aretinum(14,308,467,与14,227,083 bp互补)。*,遗迹;空空的,染色体。
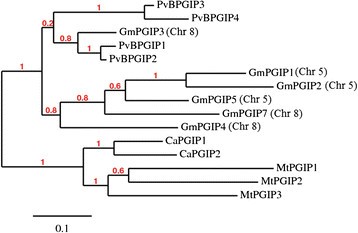
四种豆科植物的PGIP蛋白序列(p .寻常的,g·马克斯,m . truncatula,c . arietinum)用MUSCLE比对,用最大似然法(PhyML)构建系统发育树。如图所示4,无根树显示:i)m . truncatula而且C. arietinum pgip复制呈种特异性簇状,表明复制扩增发生在物种分化之后;二)m . truncatula而且C. arietinum pgip集群彼此靠近,形成一个非常受支持的集群;3)g·马克斯而且P. vulgaris pgip的成员在不同的聚类中,与这些成员的一般分类关系一致蝶形花科.此外,g·马克斯而且p .寻常的基因分布在三个主要簇上:包括PvBPGIP1、PvBPGIP2和GmPGIP3的簇;b)包含PvBPGIP3和PvBPGIP4的集群;c)包含其余集群的集群g·马克斯基因。
讨论
在这项工作中,我们已经证明了大豆的充分补体pgip家族由6个转录基因组成,分别位于染色体5和8的亚末端和末端的两个不同位点。每一个pgip基因座既包含完整的编码区域,也包含中断的编码区域,这表明家族中存在假化(“死亡”)过程。假设为NBS-LRRR基因(23],即最近复制的集群pgip副本应该提供一个遗传变异的储存库pgip基因可以进化。与nbc - lrr的相似之处R-基因的研究结果进一步支持了先前的研究结果pgip一个基因家族内不同种类或拷贝的基因的差异主要是由于LRR结构域内的单一取代,特别是在xxLxLxx溶剂暴露区[15],[16],[18],[24].就像在R-基因、密码子进化模型表明,在PGIP的溶剂暴露区域存在正选择位点[25] - [27],功能分析表明,该区域内的单一取代或短时间缺失可导致PGIP对真菌pg抑制性能的改变[16],[28] - [33].
大豆的一个有趣特征pgip位点,包括在一个更大的复制区域,是周围区域的高序列守恒pgip与基因间区域的变异形成对比的簇pgip基因。差异在区域的外部pgip基因簇仅局限于8号染色体区域内的小indedels和两个转座元件的存在。这种保守的组织是典型的古多倍体大豆基因组,它经历了两轮全基因组复制(WGD) [34],[35].最近对大豆全基因组序列的分析表明,这在很大程度上确实是由复制基因块组成的[21];然而。在获得完整的组装大豆基因组之前,数据表明大豆基因组是一个由保持高序列保守性的交替同源区域和显示极低序列保守性的区域组成的马赛克[35].
的同源区域pgip簇,显著的保护被中断只有显著的序列变异之间的基因间区域pgip基因。这种低序列保守包括近端5 '侧区,表明差异的差异调节pgip基因。事实上,这六个pgip基因显示变异独联体已知的调控防御反应基因的作用元件,它们在病原体感染后的表达模式显示出明显的差异。这两本小说pgip的基因,Gmpgip5而且Gmpgip7,在大豆下胚轴中低表达,但在真菌病原体感染的后期强烈诱导美国sclerotiorum。的表达式模式类似于Gmpgip2和的不同Gmpgip1, Gmpgip3而且Gmpgip4,均表现出较为迅速的归纳跟随美国sclerotiorum感染(18].真菌病原体感染或应激刺激后的基因调控多样化已被报道pgip基因家族,包括拟南芥[3.], bean [16),显著[15,并建议适应压力。然而,在大豆中,这种可能性在蛋白质水平上仍然缺乏支持,因为目前已经显示出对真菌pg的抑制活性在体外仅用于编码的产品Gmpgip3,也是表达量最高的大豆家族成员[18].事实上,尽管GmPGIP1, GmPGIP2和GmPGIP4 [18]和GmPGIP7的表达n benthamiana以PVX为载体,未表现出任何抑制活性。缺乏在体外GmPGIPs的抑制活性不排除它们可以抑制来自不同来源的pg的可能性,或者只有GmPGIPs的抑制活性在足底环境提供了一个合适的上下文来支持与pg的交互。朱伯特等人提出了最后一种可能性。6他们发现由运动引起的症状有所减轻葡萄孢菌与植物组织共渗透时,BcPG2在植物组织中的作用葡萄VvPGIP1,虽然没有检测到VvPGIP1和BcPG2之间的相互作用在体外.此外,如前所述[18], GmPGIP1、GmPGIP2、GmPGIP4和GmPGIP7对真菌pg抑制活性的缺失可能反映了不同的生理作用在足底.这种可能性得到了对不同植物种类的几项观察的支持。例如,具有PG抑制能力的水稻蛋白OsFOR1影响水稻花器官原基的形成和/或维持[36];PGIP1水平决定拟南芥胚根突出的时间[37),而Vvpgip1可影响转基因烟草植株的基因表达和细胞壁结构[38],[39].
基因组区域包括pgip将大豆的基因与大豆相应区域的基因进行比较,发现大豆与大豆具有非常密切的系统发育关系。的pgip豆基因型BAT93的基因家族,由4个聚类旁系组成,以前已被鉴定[16].我们现在已经将分析扩展到豆BAT93序列的侧翼pgip聚类研究发现,与重复大豆区相比,基因分布具有很强的保守性pgip位点。十个豆子基因的侧翼pgip7个在大豆第5和第8染色体上均保守。Pv_202100仅在大豆8号染色体上丢失,重复的大豆基因Pv_201200和Pv_201100在大豆5号和8号染色体上均为单拷贝。
基因组的保护pgip区域性在远亲豆科植物中也很明显m . truncatula而且c . arietinum,其组装的基因组最近已被公布[19],[20.].两个物种都只有一个pgip轨迹,有一个簇状组织的平行线,和区域的侧翼pgip维持在大豆和大豆中观察到的基因的强保守分布的阵列。十个豆子基因中的pgip聚类中,7个在所有4种豆科植物中均保守。发现大多数pgip基因以物种特有的系统发育簇组织,这表明pgip在物种形成后,每个簇内的副本独立形成。GmPGIP3、PvPGIP1和PvPGIP2是这一观察结果的一个例外。事实上,大豆和大豆PGIPs在最大似然基基因树中形成了分离的聚类,在大豆聚类中,四个PvPGIPs形成了两个分离良好的组。然而,如前所述[16],[18],大豆GmPGIP3组与PvPGIP1和PvPGIP2的分离,表明起源于PvPGIP1/PvPGIP2和PvPGIP3/PvPGIP4祖先的复制发生在PvPGIP1和PvPGIP2的分离之前甘氨酸而且菜豆血统。在这种情况下,值得注意的是,PvPGIP2的高序列守恒延伸到一系列的p .寻常的种质和其他菜豆物种,表明适应意义[30.].
结论
古多倍体大豆基因组包含两个pgip基因座由大而高度保守的重复区域组成,可能起源于WGD。该地区包括pgip基因座在豆子中也是保守的,m . truncatula而且c . arietinum.这些豆科植物的基因组特征pgip科,其中包括推断最近的复制和伪化pgip副本,表明驱动进化的力量pgip基因遵循出生-死亡模型,类似于nbs - lrr型的进化R基因(23].
方法
植物材料及侵染实验
大豆籽(g·马克斯(L。)稳定。简历。Williams 82)通过将它们放在事先消毒和浸泡过的纸巾上发芽,然后将纸巾卷起来,在24°C的黑暗环境中培养5天。
B-24分离物美国sclerotiorum(自由。De Bary)在24℃条件下在马铃薯葡萄糖琼脂上培养3天,获得用于大豆幼苗接种的菌丝。
以黄化大豆下胚轴中部接种生长活跃的菌丝体进行侵染实验美国sclerotiorum如Favaron等人所述。[40].大豆幼苗水平放置在塑料托盘上。根上盖了一层潮湿的纸巾。从活跃生长的菌落边缘地带切下琼脂,沿下胚轴中部放置菌丝定植的小栓子(5 × 2 mm)接种植物。对照大豆幼苗用无菌琼脂培养基模拟接种。在24℃孵育后,在接种后0、8、16、24和48 h,用刀片在正下方横向切割下胚轴(约5 mm),在液氮中冷冻,-80℃保存,以备后续分析。根据Favaron等人的报道,组织损伤随着时间的推移而增加,在48 hpi时影响下胚轴的大部分。[40].进行了两个独立的感染实验。
RNA提取及RT-PCR分析
根据制造商的说明,使用RNeasy Plant Mini Kit (Qiagen, Italy)提取总RNA。在琼脂糖凝胶电泳后,用分光光度法和密度法测定rRNA片段的RNA浓度。定量反转录试剂盒(Qiagen)用于去除基因组DNA污染并合成cDNA。基因翻译延伸因子的引物在不同外显子上对齐,通过PCR验证cDNA制备中的基因组DNA的消除(GmELF1A)和葡萄糖-6-磷酸脱氢酶(GmG6PD),正如米兰达等人所描述的。[41].
实时荧光定量PCR实验使用iCycler (Bio-Rad,意大利)和含有SYBR Green I DNA结合染料的主mix iQTMSYBER Green Supermix (BioRad,意大利)进行。每个反应重复三次。引物使用Primer 3软件设计(http://fokker.wi.mit.edu/primer3/, (42])的基础上GmpgipGmPGIP3-3 - F 5 ' -ACCCCAACCCTAATCGGTCA-3 '和GmPGIP3-3R 5 ' -AGGTGATTCCGACGAGATTG-3 'Gmpgip3;GmPGIP5-1 F 5 ' - accggactccttcggctacttcc -3 '和GmPGIP5-1R 5 ' - TGTTTCCCAGATACATGTGCC-3 '用于Gmpgip5;gmpgip7 - 1f 5 ' - taagggtgtcaaagaccttgtt -3 '和GmPGIP7-1R 5 ' - CACTTGTTATGAGCGTACAGC-3 'Gmpgip7;GmELF1A-F 5 ' -GACCTTCTTCGTTTCTCGCA-3 '和GmELF1A-R 5 ' -GAACCTCTCAATCACACGC-3 'GmELF1A[41].总反应量为20 μl,其中主合剂10 μl (2×), cDNA 100 ng,正、反引物各0.5 μl (10 μM),用水调节反应量。PCR反应条件为:50°C 2 min, 94°C 15 min, 95°C 15 sec, 60°C 50 sec, 72°C 50 sec,循环40次。扩增子核苷酸测序(MWG, Germany)证实引物特异性。靶基因Ct值(Gmpgip3, Gmpgip5而且Gmpgip7)和内参基因(GmELF1A)进行进一步的相对表达分析——ΔΔCT法[43].相对诱导水平是相对于每个分析时间点对应的非感染样本。通过Gene Expression Macro™Version 1.1 (Bio-Rad,意大利)进行计算和统计分析。qRT-PCR实验包括为每个样品在两个不同的生物副本中设置三个副本。根据阈值周期与cDNA对数稀释序列的线性回归斜率,按公式ε =(10^(-1/斜率)-1)*100计算每个基因的PCR效率(ε)。
pvx介导的GmPGIP5和GmPGIP7的表达及免疫印迹
的编码区Gmpgip5而且Gmpgip7用引物PCR扩增班我和萨尔我或NruI分别在5 '端和3 '端。放大的片段被双消化班我和萨尔我或Nru并克隆到pPVX201表达载体的相应位点。获得的质粒用于接种n benthamiana如Baulcombe等人所述,每株植物使用30 μg质粒DNA。[22].瞬时表达的GmPGIP7从叶中提取n benthamiana用单一PVX-Pgip结构或空载体感染的植株。叶片在1 M NaCl (2 ml/g)中均质,在4°C下轻轻摇晃1小时,在10,000 g下离心20分钟。上清液经miraccloth (Calchem, USA)过滤,离心去除碎屑,保存于-20°C。用Coomassie Plus™(Bradford)检测试剂盒(Pierce, Rockford, IL, USA)测定蛋白浓度。如前所述进行SDS-PAGE和免疫印迹[18].采用大豆PGIP多克隆抗体进行免疫印迹实验。
真菌生长,PG制备和酶的测定
真菌生长和PG制备进行如前所述答:尼日尔[44),美国sclerotiorum隔离B-24 [45),而c . acutatum分离SHK788 [16].f . graminearum隔离3827 [46].PGIP的抑制活性如D 'Ovidio等人先前所述。[18].PG活性以还原性单位(RU)表示。1 RU定义为在1 μmol min内释放还原基团所需的酶量-1以d -半乳糖醛酸为标准。
基因组文库筛选和测序
细菌人工染色体(BAC)库(从g·马克斯简历。Williams 82)是从克莱姆森大学基因组研究所(CUGI;克莱姆森,SC,美国)。根据D 'Ovidio等人先前的描述,对BAC克隆进行筛选和测序。[18].豆BAC克隆129 F4和10G1跨越豆pgip从BAT93基因型基因组文库的16.603个重组克隆中分离得到基因座。16].
测序反应使用“ABI PRISM染料终止剂周期测序准备反应”试剂盒进行,DNA序列使用半自动ABI PRISM 310测序仪(Applied Biosystem, Monza, Italy)测定。序列也通过MWG-BIOTECH AG (Ebersberg, Germany)和PRIMM Srl确定。测序服务(米兰,意大利)。
核酸操作及氨基酸序列分析
按标准程序进行核酸操作、PCR、克隆[47].DNAMAN软件(Lynnon, BioSoft, Quebec, Canada)用于核苷酸和氨基酸序列分析。GmPGIP5和GmPGIP7信号肽预测http://wolfpsort.org/[48].5 '的侧翼区域Gmpgip基因扫描是否存在独联体-元素使用PLACE (http://www.dna.affrc.go.jp/PLACE/signalscan.html),一个植物资料库独联体-作用调控DNA元件[49].
系统发育分析
使用bean PGIP序列进行BLASTp分析,以识别已标注的PGIP基因,并检测部分PGIP序列(残基)的出现g·马克斯,m . truncatula而且c . arietinum基因组。
豆科四种植物的PGIP蛋白序列(p .寻常的,g·马克斯,m . truncatula,c . arietinum)采用多重序列比对(MUltiple Sequence Comparison by Log- Expectation, MUSCLE)进行比对,用于系统发育树的重建。这是在web界面上执行的www.phylogeny.fr[50],使用基于最大似然原理的PhyML软件。
比较分析
BAC克隆129 F4和10G1(登录号HG964426)的组装序列p .寻常的基因型BAT93被定位于p .寻常的用Bl2seq进行基因组测序www.phytozome.orgweb界面。
以大豆基因组序列phvull . 002g200900.1至phvull . 002g202200.1这段基因的蛋白序列作为Bl2seq的查询,检测出大豆基因组序列中最相似的序列g·马克斯,c . arietinum而且m . truncatula.
附加文件
参考文献
- 1.
10 Have A, Mulder W, Visser J, van Kan JA:内聚半乳糖醛酸酶基因Bcpg1是全毒力所必需的葡萄孢菌.植物与微生物的相互作用,1998,11:1009-1016。10.1094 / MPMI.1998.11.10.1009。
- 2.
Powell AL, van Kan J, ten Have A, Visser J, Greve LC, Bennett AB, Labavitch JM:番茄中梨PGIP转基因表达限制真菌定植。植物与微生物的相互作用,2000,13:942-950。10.1094 / MPMI.2000.13.9.942。
- 3.
法拉利S, Vairo D,奥苏贝尔FM, Cervone F, De Lorenzo G:串联复制拟南芥编码聚半乳糖醛酸酶抑制蛋白的基因在真菌感染时受到不同信号转导途径的协调调控。植物细胞工程学报,2003,15:93-106。10.1105 / tpc.005165。
- 4.
Agüero CB, Uratsu SL, Greve C, Powell ALT, Labavitch JM, Meredith CP, Dandekar AM:皮尔斯病耐受性的评估和葡萄孢属在转基因植物中葡萄L.表达梨PGIP基因。植物病理学杂志,2005,6:43-51。10.1111 / j.1364-3703.2004.00262.x。
- 5.
Manfredini C, Sicilia F, Ferrari S, Pontiggia D, Salvi G, capari C, Lorito M, De Lorenzo G:聚半乳糖醛酸酶抑制蛋白2菜豆抑制BcPG1,一种聚半乳糖醛酸酶葡萄孢菌重要的致病性,并保护转基因植物免受感染。中国生物医学工程学报,2005,29(3):344 - 344。10.1016 / j.pmpp.2005.10.002。
- 6.
Joubert DA, Slaughter AR, Kemp G, Becker JVW, Krooshof GH, Bergmann C, Benen J, Pretorius IS, Vivier MA:葡萄聚半乳糖醛酸酶抑制蛋白(VvPGIP1)减少葡萄孢菌转基因烟草的敏感性和对真菌聚半乳糖醛酸酶的差异抑制。转基因学报,2006,15:687-702。10.1007 / s11248 - 006 - 9019 - 1。
- 7.
Janni M, Sella L, Favaron F, Blechl AE, De Lorenzo G, D 'Ovidio R:转基因小麦中大豆PGIP的表达增强了对真菌病原体的抗性Bipolaris sorokiniana.植物与微生物的相互作用,2008,21:171-177。10.1094 / mpmi - 21 - 2 - 0171。
- 8.
黄波峰,裴浩,林海生,金凯波,金世杰,林明华,朴b - s, Kim DS, Kim J:大白菜聚半乳糖醛酸酶抑制蛋白2 (PGIP2)的过表达(芸苔属植物拉伯ssp。北京种)增强了对细菌病原体的抵抗力Pectobacterium carotovorumssp。carotovorum。中国生物医学工程学报,2010,27(3):344 - 344。10.1007 / s11240 - 010 - 9779 - 4。
- 9.
Pérez-Donoso AG, Sun Q, Roper MC, Greve LC, Kirkpatrick B, Labavitch JM:细胞壁降解酶扩大了健康小鼠血管间孔膜孔径Xylella fastidiosa来华的葡萄藤。中国生物医学工程学报,2010,29(3):344 - 344。10.1104 / pp.109.148791。
- 10.
Volpi C, Janni M, Lionetti V, Bellincampi D, Favaron F, D 'Ovidio R:一种果胶甲酯酶抑制剂的异位表达增加了果胶甲酯化,限制了小麦真菌疾病。植物与微生物相互作用,2011,24:1012-1019。10.1094 / mpmi - 01 - 11 - 0021。
- 11.
费丽娟,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚拟南芥小麦增强了对花病菌的抵抗力镰刀菌素graminearum.植物学报,2012,14:31-38。10.1111 / j.1438-8677.2011.00449.x。
- 12.
Borras-Hidalgo O, capari C, Hernandez-Estevez I, De Lorenzo G, Cervone F:植物保护基因:大豆聚半乳糖醛酸酶抑制剂在烟草中的表达具有很强的抗性辣椒还有两个卵菌。植物学报,2012,3:268-10.3389/fpls.2012.00268。
- 13.
Jones DA, Jones JDG:富含亮氨酸的重复蛋白在植物防御中的作用。中国机械工程学报,1997,27(4):457 - 457。10.1016 / s0065 - 2296(08) 60072 - 5。
- 14.
Di Matteo A, Federici L, Mattei B, Salvi G, Johnson KA, Savino C, De Lorenzo G, Tsernoglou D, Cervone F: polygalacturonase- inhibitory protein (PGIP)的晶体结构,PGIP是一种富含亮氨酸的重复蛋白,参与植物防御。自然科学进展,2003,30(4):344 - 344。10.1073 / pnas.1733690100。
- 15.
Hegedus DD, Li R, Buchwaldt L, Parkin I, Whitwill S, Coutu C, Bekkaoui D, Rimmer SR:芸苔属植物显著拥有一套扩展的聚半乳糖醛酸酶抑制剂蛋白基因,这些基因在对菌核病sclerotiorum感染、损伤及防御激素治疗。植物学报,2008,28(3):344 - 344。10.1007 / s00425 - 008 - 0733 - 1。
- 16.
D 'Ovidio R, Raiola A, Capodicasa C, Devoto A, Pontiggia D, Roberti S, Galletti R, Conti E, O 'Sullivan D, De Lorenzo G:大豆编码多聚半乳糖醛酸酶抑制蛋白的复杂位点的特征揭示了防御真菌和昆虫的亚功能化。植物科学学报,2004,26(3):344 - 344。10.1104 / pp.104.044644。
- 17.
Kalunke RM, Janni M, Sella L, David P, Geffroy V, Favaron F, D 'Ovidio R:对大豆多聚半乳糖醛酸酶抑制蛋白基因家族的转录分析Pvpgip2在全株均有表达,受病原菌侵染有很强的诱导作用。中国植物病理学杂志,2011,29(3):344 - 344。
- 18.
D 'Ovidio R, Roberti S, Giovanni MD, Capodicasa C, Melaragni M, Sella L, Tosi P, Favaron F:大豆多聚半乳糖醛酸酶抑制蛋白(Pgip)基因家族的鉴定表明,在大豆组织中检测到的活性由单一成员负责。植物学报,2006,29(3):344 - 344。10.1007 / s00425 - 006 - 0235 - y。
- 19.
Young ND, Debellé F, Oldroyd GED, Geurts R, Cannon SB, Udvardi MK, Benedito VA, Mayer KFX, Gouzy J, Schoof H, Van de Peer Y, Proost S, Cook DR, Meyers BC, Spannagl M,张F, de Mita S, Krishnakumar V, Gundlach H, Zhou S, Mudge J, Bharti AK, Murray JD, Naoumkina MA, Rosen B, Silverstein KAT, Tang H, Rombauts S, Zhao PX, Zhou P,等Medicago基因组提供了深入了解根瘤菌共生的进化。自然科学学报,2011,38(4):344 - 344。10.1038 / 480162。
- 20.
Varshney RK, Song C, Saxena RK, Azam S, Yu S, Sharpe AG, Cannon S, Baek J, Rosen BD, Tar an B, Millan T, Zhang X, Ramsay LD, Iwata A, Wang Y, Nelson W, Farmer AD, Gaur PM, Soderlund C, Penmetsa RV, Xu C, Bharti AK, He W, Winter P, Zhao S, Hane JK, Carrasquilla-Garcia N, Condie JA, Upadhyaya HD, Luo M-C,等:鹰嘴豆基因组序列图(中投arietinum)为性状改进提供了资源。中国生物工程学报,2013,31:240-246。10.1038 / nbt.2491。
- 21.
Schmutz J, Cannon SB, Schlueter J, Ma J, Mitros T, Nelson W, Hyten DL, Song Q, Thelen JJ, Cheng J, Xu D, Hellsten U, May GD, Yu Y, Sakurai T, Umezawa T, Bhattacharyya MK, Sandhu D, Valliyodan B, Lindquist E, Peto M, Grant D, Shu S, Goodstein D, Barry K, Futrell-Griggs M, Abernathy B, Du J, Tian Z, Zhu L,等:古多倍体大豆基因组序列分析。自然科学进展,2010,38(4):344 - 344。10.1038 / nature08670。
- 22.
鲍科姆特区,查普曼S,圣克鲁斯S:水母绿色荧光蛋白作为病毒感染的报告者。植物学报,2004,19(4):457 - 457。10.1046 / j.1365 - 313 x.1995.07061045.x。
- 23.
Michelmore RW, Meyers BC:植物中抗性基因簇通过发散选择和生死过程进化。基因组学报,1998,8:1113-1130。
- 24.
De Lorenzo G, D 'Ovidio R, Cervone F:聚半乳糖醛酸酶抑制蛋白(pgips)在防御病原真菌中的作用。植物学报,2001,39:313-335。10.1146 / annurev.phyto.39.1.313。
- 25.
Stotz H, Bishop J, Bergmann CW, Koch M, Albersheim P, Darvill AG, Labavich JM:影响真菌聚半乳糖醛酸酶及其植物抑制剂相互作用的目标氨基酸的鉴定。中国生物医学工程学报,2000,29(3):344 - 344。10.1006 / pmpp.2000.0258。
- 26.
Bishop JG:定向突变证实了聚半乳糖醛酸酶抑制剂蛋白阳性选择位点的功能重要性。中国生物医学工程学报,2005,22:1531-1534。10.1093 / molbev / msi146。
- 27.
Casasoli M, Federici L, Spinelli F, Matteo AD, Vella N, Scaloni F, Fernandez-Recio J, Cervone F, De Lorenzo G:植物免疫蛋白功能位点的整合进化和脱溶能量分析自然科学进展,2009,26(3):366 - 366。10.1073 / pnas.0812625106。
- 28.
Leckie F, Mattei B, Capodicasa C, Hemmings A, Nuss L, Aracri B, De Lorenzo G, Cervone F:聚半乳糖醛酸酶抑制蛋白(PGIP)的特异性:在富亮氨酸重复序列(LRRs)的溶剂暴露的β -链/ β -转区,单氨基酸取代赋予了一种新的识别能力。环境科学学报,1999,30(4):344 - 344。10.1093 / emboj / 18.9.2352。
- 29.
Sicilia F, Fernandez-Recio J, capari C, De Lorenzo G, Tsernoglou D, Cervone F, Federici L:聚半乳糖醛酸酶抑制蛋白PGIP2菜豆已经形成了一种抑制内聚半乳糖醛酸酶PG1的混合模式葡萄孢菌.植物科学学报,2005,29(3):344 - 344。10.1104 / pp.105.067546。
- 30.
Farina A, Rocchi V, Janni M, Benedettelli S, De Lorenzo G, D 'Ovidio R:大豆聚半乳糖醛酸酶抑制蛋白2 (PvPGIP2)在普通大豆中高度保守(菜豆l)种质资源和近缘种。应用物理学报,2009,29(3):379 - 379。10.1007 / s00122 - 009 - 0987 - 4。
- 31.
Spinelli F, Mariotti L, Mattei B, Salvi G, Cervone F, capari C:来自中国的聚半乳糖醛酸酶抑制蛋白(PGIP)的三个天冬氨酸残基菜豆对抑制镰刀菌素phyllophilum中国生物工程学报,2009,29(3):738-743。10.1111 / j.1438-8677.2008.00175.x。
- 32.
Benedetti M, Leggio C, Federici L, De Lorenzo G, Pavel NV, Cervone F:真菌聚半乳糖醛酸酶与植物聚半乳糖醛酸酶抑制蛋白复合物的小角度x射线散射结构解析。中国生物医学工程学报,2011,27(3):344 - 344。10.1104 / pp.111.181057。
- 33.
Benedetti M, Andreani F, Leggio C, Galantini L, Di Matteo A, Pavel NV, De Lorenzo G, Cervone F, Federici L, Sicilia F:单氨基酸取代允许内聚半乳糖醛酸酶镰刀菌素verticillioides以获得PGIP2的识别菜豆.公共科学学报,2013,8:e80610-10.1371/journal. psci .0080610。
- 34.
Blanc G, Wolfe KH:从复制基因的年龄分布推断模式植物中广泛的古多倍体。植物细胞工程学报,2004,16(3):366 - 366。10.1105 / tpc.021345。
- 35.
Schlueter JA, Lin J- y, Schlueter SD, Vasylenko-Sanders IF, Deshpande S, Yi J, O 'Bleness M, Roe BA, Nelson RT, Scheffler BE, Jackson SA, Shoemaker RC:大豆基因复制和古多倍体及其对全基因组测序的影响。中国生物医学工程学报,2007,8:330-10.1186/1471-2164-8-330。
- 36.
张松,李波,金春华,金世杰,严俊,韩建杰,李松,金世荣,安庚:水稻OsFOR1基因编码一种调控花器官数量的聚半乳糖醛酸酶抑制蛋白(PGIP)。中国生物医学工程学报,2003,23(3):357-372。10.1023 / B: PLAN.0000006940.89955.f1。
- 37.
Kanai M, Nishimura M, Hayashi M:在ABI5的控制下,过氧化物酶体ABC转运体通过诱导果胶降解促进种子萌发。中国生物医学工程学报,2010,29(4):344 - 344。
- 38.
黄晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明,王晓明。葡萄聚半乳糖醛酸酶抑制蛋白的表达及其对烟草细胞壁特性的影响。BMC Res Notes, 2011, 4: 493-10.1186/1756-0500-4-493。
- 39.
nguemma - ona E, Moore JP, Fagerström AD, Fangel JU, Willats WG, Hugo A, Vivier MA:在没有真菌感染的情况下,葡萄PGIP1在烟草中过表达导致叶片阿拉伯氧葡聚糖网络的组成变化。中国生物医学工程学报,2013,26(4):344 - 344。
- 40.
王晓明,王晓明,王晓明。植物聚半乳糖醛酸酶(PGIP)与草酸盐、pH的相互作用关系菌核病sclerotiorum和大豆。植物与微生物的相互作用,2004,17:1402-1409。10.1094 / MPMI.2004.17.12.1402。
- 41.
Miranda Vde J, Coelho RR, Viana AAB, de Oliveira Neto OB, Carneiro RMDG, Rocha TL, de Sa MFG, Fragoso RR:针对大豆线虫寄生和昆虫攻击的qPCR数据的精确归一化内参基因的验证。BMC Res Notes, 2013, 6: 196-10.1186/1756-0500-6-196。
- 42.
Rozen S, Skaletsky H: WWW上的入门3,适用于普通用户和生物学家程序员。生物信息方法。编辑:Misener S, Krawetz SA。1999, Humana出版社,托托瓦,新泽西州,132:365-386,
- 43.
Livak KJ, Schmittgen TD:利用实时定量PCR和2(-Delta Delta C(T))方法分析相关基因表达数据。方法,2001,25:402-408。10.1006 / meth.2001.1262。
- 44.
cerone F, De Lorenzo G, Degrà L, Salvi G:诱导坏死豇豆属unguiculataWalp。通过均匀黑曲霉内聚半乳糖醛酸酶和α-d-半乳糖醛酸低聚物。中国农业科学,2004,27(3):326 - 326。10.1104 / pp.85.3.626。
- 45.
法龙F, Castiglioni C, D 'Ovidio R, Alghisi P:聚半乳糖醛酸酶抑制蛋白韭葱及其在植物组织中对抗真菌内聚半乳糖醛酸酶的作用。中国生物医学工程学报,1997,26(3):344 - 344。10.1006 / pmpp.1997.0099。
- 46.
Tomassini A, Sella L, Raiola A, D 'Ovidio R, Favaron F:表征和表达镰刀菌素graminearum小麦侵染过程中体外聚半乳糖醛酸酶的研究。植物病理学杂志,2009,38(4):357 - 357。10.1111 / j.1365-3059.2008.02019.x。
- 47.
桑布鲁克J,弗里奇E,曼尼提斯T:分子克隆:实验室手册。1989,冷泉港实验室出版社,Plainview,纽约
- 48.
Horton P, Park K- j, Obayashi T, Fujita N, Harada H, Adams-Collier CJ, Nakai K: WoLF PSORT:蛋白质定位预测器。中国生物医学工程学报,2007,35 (2):W585-W587。10.1093 / nar / gkm259。
- 49.
黄志刚,黄志刚,黄志刚:植物顺式作用调控DNA元件(PLACE)数据库:1999。中国生物医学工程学报,1999,27(4):457 - 457。10.1093 / nar / 27.1.297。
- 50.
Dereeper A, Guignon V, Blanc G, Audic S, Buffet S, Chevenet F, Dufayard J-F, Guindon S, Lefort V, Lescot M, Claverie J-M, Gascuel O:系统发育。fr:非专家的稳健系统发育分析。中国生物医学工程学报,2008,36:W465-W469。10.1093 / nar / gkn180。
致谢
MIUR (Ministero dell ' istruzione, dell ' universita ' e della Ricerca;将PRIN(国家科学研究研究项目)授予RD和GDL。作者感谢Daniëlle Smeitink的技术支持。
豆基因组序列数据(p .寻常的G19833)是由美国能源部联合基因组研究所生产的http://www.jgi.doe.gov/与用户社区合作。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
RMK进行了分子分析,并为论文的初稿做出了贡献;AC进行序列和系统发育分析;CV进行BAC文库筛选、克隆鉴定和异源表达分析;DOS,监督BAC文库筛选和克隆表征;LS进行感染实验;FF、FC和GDL对数据和论文进行了批判性的讨论;RD设计研究,协调研究并撰写论文。所有作者阅读、编辑并批准最终手稿。
电子辅助材料
大豆BAC无性系插入体尺寸估计及指纹图谱分析。
附加文件1:A)利用脉冲场凝胶电泳(PFGE)测定大豆BAC无性系的大小不我消化。1、26 i2;2、95年o4;3、6 f5;4, 28岁的energisk b18;5、85 m15。M1和M2分别表示PFGE分子质量阶梯和Lambda-DNA/后梯,分别。B)指纹分析。BAC克隆用后用1.0%琼脂糖凝胶分离。1、6 f5;2、28的energisk b18;95 o4;4、26 i2;5、85 m15;M, 1 kb DNA阶梯。(doc 1mb)
通过筛选BAC文库获得BAC克隆
附加文件2:g·马克斯简历。威廉姆斯82使用大豆pgip调查。随后用脉冲场凝胶电泳(PFGE)测定镶片大小不我消化。(docx11kb)
推导出的氨基酸序列的比对
附加文件3:g·马克斯PGIPs。编号是指GmPGIP1序列,从成熟蛋白的第一个残基开始。区域A-D是根据豆PvPGIP2的晶体学分析预测的(Di Matteo et al. 2003, Proceedings of the National Academy of Sciences, 100,10124 -10128)。xxLxLxx区域被框起来。为了更好地显示单个蛋白质中LRR序列之间的同一性/相似性,已经添加了空格。预测信号肽区(A区)采用Wolfpsort (http://wolfpsort.org/;Horton等,2007,核酸研究(Web服务器版),35:W585-W587)。圆点表示相同的氨基酸残基;虚线表示缺失的氨基酸。半胱氨酸残基下划线。Gmpgip1, Gmpgip2而且Gmpgip5都在pgip5号染色体上的位点。Gmpgip3,Gmpgip4而且Gmpgip7在8号染色体上。(docx23kb)
大豆的基因表达模式
附加文件4:pgip从公共数据库中发现的表达序列标签(ESTs)计数推断的基因一个.(docx42 kb)
总蛋白提取的Western blot
附加文件5:n benthamiana植物接种PVX-pgip结构和琼脂糖扩散试验的PGIP抑制。A)用总蛋白提取物进行Western blot分析n benthamiana以单个PVX 201为基础的构建物接种的植物,用于表达GmPGIP3或GmPGIP7或空载体,作为对照。1、蛋白质阶梯;2、pvx201(空矢量);3、GmPGIP7 (5 μg);4、GmPGIP7 (10 μg);5、GmPGIP7 (20 μg);6、GmPGIP3 (10 μg)。B)粗蛋白提取物琼脂糖扩散试验n benthamiana以PVX-Gmpgip3或PVX-Gmpgip7载体或空载体接种的植株作为对照。用0.011还原单位的美国sclerotiorumendopolygalacturonase (SsPG)。无晕表示PG活性受到抑制。1、SsPG;2、SsPG + GmPGIP3 (1 μg);3、SsPG加煮沸的GmPGIP (1 μg);4、SsPG + GmPGIP7 (20 μg);5、SsPG加煮沸的GmPGIP7 (20 μg);6、SsPG + PVX 201空载体(20 μg);7、SsPG加煮空pvx201载体(20 μg)。的PG也得到了类似的结果f . graminearum,c . acutatum而且答:尼日尔。GmPGIP3抑制了所有四种pg的完成,而GmPGIP7没有表现出任何抑制活性(数据未显示)。(docx775 kb)
残基推导氨基酸序列的比对
附加文件6:g·马克斯PGIPs。以GmPGIP3为内参基因进行序列比对。编号是指GmPGIP3序列,从成熟蛋白的第一个残基开始。区域A-D是根据豆PvPGIP2的晶体学分析预测的(Di Matteo et al. 2003, Proceedings of the National Academy of Sciences, 100,10124 -10128)。xxLxLxx区域被框起来。预测信号肽区(A区)采用Wolfpsort (http://wolfpsort.org/;Horton等,2007,核酸研究(Web服务器版),35:W585-W587)。的残余GmPGIP * (1)美国严重碎片化,以及GmPGIP * (2)位于第5染色体上。重建的GmPGIP*(1)蛋白序列包含一个分泌信号肽(a区)和一个含有299个氨基酸的成熟蛋白。GmPGIP*(2)对应一个65个氨基酸组成的c端片段。的遗迹GmPGIP *该蛋白位于8号染色体上,与一个PGIP片段相对应,该片段包含一个假定的信号肽和一个成熟蛋白的220个氨基酸部分。圆点表示相同的氨基酸;虚线表示缺失的氨基酸。为了更好地显示单个蛋白质中LRR序列之间的同一性/相似性,已经添加了空格。半胱氨酸残基下划线。*,遗迹;空空的,染色体。(doc 40kb)
作用调控DNA元件相关
附加文件7:独联体作用调控DNA元件相关pathogen-induced表达式。各5 '侧区序列(~ 1kb)Gmpgip基因分析采用PLACE数据库(http://www.dna.affrc.go.jp/PLACE/).(docx19kb)
含豆区域的Blast2seq分析
附加文件8:pgip基因。一种长度为62 Kb的核苷酸序列pgip基因(PvBpgip1,PvBpgip2,PvBpgip3,PvBpgip4)是自对齐的。红色矩形框表示长串联重复序列(LTR)逆转录转座子。蓝色矩形框表示具体的pgip基因。椭圆表示周围保守区域之间的对齐Pgip基因(蓝色)和两个LTR逆转录元件之间(红色)。(docx67 kb)
对推导出的氨基酸序列进行比对
附加文件9:m . truncatulaPGIPs。编号是指MtPGIP1序列,从成熟蛋白的第一个残基开始。区域A-D是根据豆PvPGIP2的晶体学分析预测的(Di Matteo et al. 2003, Proceedings of the National Academy of Sciences, 100,10124 -10128)。xxLxLxx区域被框起来。预测信号肽区(A区)采用Wolfpsort (http://wolfpsort.org/;Horton等,2007,核酸研究(Web服务器版),35:W585-W587)。为了更好地显示单个蛋白质中LRR序列之间的同一性/相似性,已经添加了空格。圆点表示相同的氨基酸残基;虚线表示缺失的氨基酸。半胱氨酸残基下划线。(docx26kb)
对推导出的氨基酸序列进行比对
附加文件10:c . arietinumPGIPs。CaPGIP1序列从成熟蛋白的第一个残基开始编号。区域A-D是根据豆PvPGIP2的晶体学分析预测的(Di Matteo et al. 2003, Proceedings of the National Academy of Sciences, 100,10124 -10128)。xxLxLxx区域被框起来。空格表示间隔,以最大化序列之间的同一性/相似性。预测信号肽区(A区)采用Wolfpsort (http://wolfpsort.org/;Horton等,2007,核酸研究(Web服务器版),35:W585-W587)。为了更好地显示单个蛋白质中LRR序列之间的同一性/相似性,已经添加了空格。圆点表示相同的氨基酸残基;虚线表示缺失的氨基酸。半胱氨酸残基下划线。(docx22 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。
本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。
如欲查看本牌照的副本,请浏览https://creativecommons.org/licenses/by/4.0/.
创作共用公共领域奉献弃权书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
卡伦克,r.m.,岑西,A.,沃尔皮,C.。et al。大豆和其他三种豆科植物中的pgip家族:进化的出生和死亡模型的证据。BMC植物生物学14日,189(2014)。https://doi.org/10.1186/s12870-014-0189-3
收到了:
接受:
发表:
关键字
- ᅟ