摘要
背景
的母系表达基因(梅格)家族是玉米的一个局部复制基因家族,编码富半胱氨酸蛋白(CRPs)。这个家族的创始成员Meg1,是基础胚乳转移细胞层(BETL)正常发育所必需的,并参与向生长中的种子分配母体营养。尽管角色很重要Meg1在玉米种子的发育过程中,进化的历史梅格簇和重复基因的活动尚不清楚。
结果
在玉米中,梅格该基因簇位于一个2.3 mb长的基因组区域,该区域表现出非着丝点异染色质的许多特征。利用系统发育重建和共线比对,我们鉴定了该病毒的谱系梅格这个家族的13个成员中有11个在4.8亿年的异体四倍化后出现在玉米中。系统发育和群体遗传分析确定了可能的特征,表明在最近的积极选择梅格同源染色体。结构分析表明,Meg蛋白在膨胀过程中发生了从α-螺旋到β-链的二级结构的适应性变化。玉米胚乳的转录组分析表明,6梅格BETL的基因被选择性激活,并且更年轻梅格基因比老基因更活跃。在B73的胚乳中,Mo17互交最多梅格基因未表现出亲本特异性表达模式。
结论
Recently-duplicated梅格这些基因具有不同的蛋白质二级结构,并且在BETL中的表达高于较老成员的表达。以及年轻人积极选择的迹象梅格这些结果表明基因的扩张梅格家庭涉及潜在的适应性转变,具有新功能的新成员压倒了老成员。
背景
植物中的转移细胞在外质体和同质体之间介导溶质的转运。植物转移细胞的一个结构特征是广泛的次生细胞壁生长,这增加了质膜的表面积,并被认为促进了溶质在质膜上的快速运输[1]。与它们的溶质交换活性一致,转移细胞通常在维管组织附近的库或源组织中观察到。在玉米胚乳的基部,一层转移细胞面向母体胎盘-合点带[2]。玉米种子的发育依赖于养分通过这个细胞层的转移,称为基础胚乳转移细胞层(BETL)。
富半胱氨酸蛋白(CRPs)是真核生物中大量分泌的小蛋白的一个大型超家族[3.]、[4]。CRPs参与了两种细胞信号传导[5]、[6]和抗菌工艺[7]。在植物中,由分泌的CRPs介导的细胞-细胞通信有助于气孔分化[8],引导花粉管生长[9]在自我不相容中[10],以及胚胎发育模式[11]。玉米胚乳中的BETL也分泌多种类型的CRPs,包括基胚乳转移层1 (bert -1),2 (BETL-2)而且4 (BETL-4)[12],软面包卷[13),而母系表达基因1(Meg1) [14]。研究表明,在BETL发育中起关键作用的myb样转录因子ZmMRP-1参与了BETL的表达BETL-1,BETL-2,Meg1[14] - [17]。鉴于BETL位于母系-子代界面,这些CRPs可能保护发育中的种子免受母系传播病原体的侵害[18]。也有可能一些BETL CRPs作为细胞外信号分子,在种子发育过程中协调母体营养的供应[3.]。
的Meg1基因是BETL正常发育所必需的,其表达升高Meg1增加BETL大小和种子生物量。有趣的是,异位表达Meg1驱动非betl胚乳细胞中betl特异性基因如ZmMRP-1和INCW2的表达。因为Meg1是否是母系表达的印迹基因,其影响如何Meg1有无剂量依赖性,促进营养吸收的途径Meg1提供证据表明种子发育过程中的养分吸收受母体控制[19]、[20.]。增强的营养分配导致Meg1过表达表明Meg1蛋白通过控制BETL来建立发育中种子的汇强。一组被称为胚胎周围因子1 (ESF1)的CRPs在拟南芥中起着类似Meg1的作用。拟南芥胚胎基部的胚柄参与养分运输,中央细胞和胚乳细胞产生的ESF1s促进胚柄发育[11]。
同源染色体的Meg1也在发育中的胚乳中转录[14]。我们已经证明了Meg1同源基因是BETL中表达水平最高的基因之一[21]。主动的存在Meg1同源性提出了这个家族是如何产生的以及是否多样化的问题Meg1同源词起着相似或不同的功能作用。在本研究中,我们确定了泛函和非泛函的全局补梅格玉米和近亲高粱外群中的家族基因;我们结合系统发育和群体遗传技术来描述这些基因的选择压力,并将选择与基因表达和蛋白质结构的变化联系起来。我们发现梅格基因家族在玉米中迅速扩张,一些证据表明正选择可能驱动了蛋白质结构的变化。我们的分析表明,与较老的重复基因相比,最近的重复基因表现出更高的表达水平,更广泛的结构变化,以及更强的适应证据,这表明更新的,功能不同梅格在最近的适应过程中,同源词可能比旧同源词占优势。
结果与讨论
的识别梅格玉米中的基因
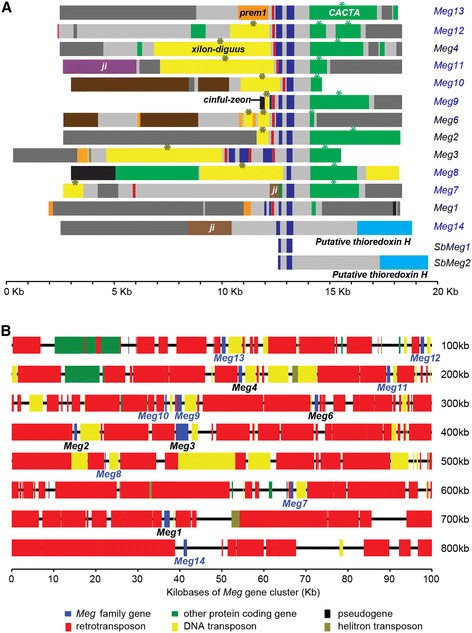
的Meg1玉米中的基因是大型Meg/Ae1超群中的一员,该超群由17个亚群组成,它们共享一个简单的CXCC基序,但几乎没有可检测到的序列相似性[4]。我们将注意力集中在子组CRP5420上,其中包括Meg1和其他含有半胱氨酸基序的成员:CX(6)CX(4)CYCCX(14)CX(3)C,并表现出保守的氨基酸序列。基于序列守恒,我们在B73玉米基因组中鉴定出13个同源位点Meg1,包括Meg2, Meg3, Meg4, Meg6之前已经被确认的Meg1[14]。B76基因组不包含任何匹配的开放阅读框Meg5.我们任命了8名新成员Meg7- - - - - -Meg14根据它们的染色体位置。上游的七个位点Meg1被命名为Meg7- - - - - -Meg13从近端到远端Meg1基因,和下游的位点Meg1被任命为Meg14(附加文件1:表S1)。
的Meg1该基因由两个编码外显子组成,由一个内含子和一个上游启动子分开,在基础胚乳转移细胞(BETCs)中进行特异性表达[14]。我们找到了完整的Meg1基因结构由8共享梅格同源染色体(图1A).例外Meg7,Meg8,Meg3,Meg10而且Meg14。Meg14是否有两个规范外显子,但其启动子与的不同Meg1.第一个编码外显子缺失Meg10而且Meg8。Meg8似乎没有启动子元件,这表明它可能不会被转录。侧翼序列Meg8而且Meg10提示两个基因的破坏是由非同源末端连接引起的。Meg7有两个编码外显子,但其启动子通过转座插入在第一个外显子上游约6.2 KB处脱位。的结构Meg3是异常的,因为它有多个调节元件和额外的外显子是紊乱的。
基因结构和基因组排列的13梅格玉米中的基因。(一个)梅格基因及其侧翼区域排列以说明它们的基因结构。的启动子和外显子梅格基因分别用红色和蓝色矩形表示。请注意,Meg14就是缺少规范梅格启动子。转座子的每个超族显示为一个矩形,其颜色代码如下:xillon-digus -黄色的,prem1- - - - - -橙色,霁-布朗。每个基因模型上游10 kb和下游5 kb内的转座子插入显示。所有的梅格除了基因Meg1,Meg13而且Meg145 '端有xillon-digus序列,3 '端有CACTA序列(星号)。下游的两种假定的h型硫氧还蛋白Meg14而且SbMeg2颜色都是浅蓝色。所有其他区域都是灰色的。该区域的所有组成部分都根据其物理大小按比例绘制。(B)染色体7S中包含13梅格基因是详细的。图中提供了区域内6个主要元素的颜色代码。
玉米的聚类梅格基因
全部13梅格该基因座位于玉米7S染色体上,位于两个分子标记之间p-asg8而且p-asg34.当与基因密度高或局部基因重复集中的染色体区域比较时,这梅格该地区有几个明显的特点。首先,不像其他玉米基因簇那样紧密地聚集在一个基因岛中[22的13个位点梅格家族分布在约800 kb的基因组区域(图1B)基因密度也较低梅格区域比玉米基因组的其他基因区要好;相邻之间的平均距离梅格基因为62 kb,大于相似的局部重复基因之间的平均间隔,如p1,一国,玉米蛋白,kn1,pl1,a1-b,或rp3(附加文件2:表S2)。基因的密度梅格该簇甚至低于整个玉米基因组的平均基因密度(1个基因/52 kb,基于筛选的2066 Mb RefGen_v2全基因组组装的基因含量http://maizesequence.org/).过度分散的性质梅格基因簇是惊人的,考虑到玉米基因集中在紧密整合的基因岛的一般趋势[22]。
大约85%的玉米基因组由转座因子组成吉普赛转座子倾向于在基因差的异色区占主导地位[23),突变转座子倾向于在基因区和开放染色质中占主导地位[24]。相比之下,玉米基因组的这种普遍模式,吉普赛在800-kb中,转座子的长度占所有转座子长度的75%梅格地区,突变转座子在这个区域完全不存在(图1B)。
染色体重组往往发生在常染色质中,但在异染色质中被抑制[25]。与存在相一致的吉普赛异色标记转座子和高度分散的基因,2.3 mb基因组区域包含梅格染色体7S的10.85 ~ 13.86 Mb的聚类显示出< 1厘摩根(cM)的低重组率(Liu et al. [24];http://www.maizegdb.org).上游3.3 Mb区域(7.38 ~ 10.68 Mb)和下游3.7 Mb区域(13.92 ~ 17.05 Mb)位于低重组区两侧梅格区域的遗传距离分别为~15.8 cM和~8.5 cM梅格基因簇代表了重组减少的局部区域。综合来看,这些数据表明梅格基因区表现出玉米非周熵异染色质的特征。
我们发现所有的成员梅格集群中,除了Meg1而且Meg14,被同源的5 '和3 '侧翼序列所包围(图1A).同源侧翼序列的长度从几百个碱基对到超过5 kb不等。9个基因的5'侧序列(Meg2, 3, 4, 6, 8, 9, 10, 11和12)包含xilon-digus逆转录转座子,长度不同。相比之下,Meg13而且Meg1有prem1逆转录转座子插入在他们的5'侧翼序列的开始。3'的夹击序列梅格基因,除了Meg14,是同源的。Meg14两边的侧翼序列与其他12个序列都不同源,这有什么特别之处吗梅格基因,表明它可能有一个独特的起源。的周围序列的一般同源性梅格基因暗示着扩张梅格家族可主要归因于转座元件的不平等交叉和插入,在重复基因的上游和下游留下特征特征。
动物的进化史梅格基因
的梅格基因簇只存在于玉米的7S染色体上。我们搜索了公共数据库找出同源的梅格其他草种的基因。高粱的两个开放阅读框(高粱二色的)与的序列相似性较强Meg1和其他玉米品种梅格基因簇,谷子的一个基因(Setaria italica)被鉴定为潜在同源。我们在水稻或其他近亲物种中没有发现同源物,这表明梅格基因起源于高粱/玉米分裂之前,但在Panicoideae组与其他草种分离之后[PMID: 22580950]。尽管拟南芥中meg1相关肽ESF1s已被鉴定并进行了功能表征[11],两者之间没有可检测到的序列相似性ESF1s在玉米和其他草种中发现的基因,除了它们的半胱氨酸残基的保守模式外。短分泌肽,如梅格通常进化非常迅速,使得在大时间尺度上确定精确的系统发育关系变得困难。因此,我们的分析仅限于这些方面梅格同源体显示可靠的序列相似性,尽管该基因家族的实际进化起源可能要早得多。
使用序列相似性梅格基因和玉米两侧的其他基因梅格聚类,我们在玉米、高粱和水稻基因组中确定了与800 kb同源或同源的区域梅格包含地区。玉米梅格基因及其高粱同源物只存在于草基因组中保守的共链块中(附加文件)3.:图S1)。在玉米、高粱和水稻的共线区中,基因共线性被很好地保留了下来,尽管玉米染色体7S的4mb区域含有基因共线性梅格基因比大米的相应区域大5倍梅格同源染色体。完全缺乏梅格玉米2号染色体同源区基因提示,玉米2号染色体中存在重复事件梅格家族只出现在第7号染色体上,主要是在480万年前同种异体四倍化(mya) [26]、[27],而梅格2号染色体的拷贝丢失了。
以确认膨胀的梅格基因家族不是B73自交系的异常,我们估计了另外6个玉米品种的拷贝数。所有梅格扩增每个品种的基因座,并对扩增子进行测序,以确定每个品种是否具有特定的多态性梅格基因存在于扩增子中(附加文件3.:图S2)。除少数例外,所有6个自交系都有完整的梅格基因,这表明梅格基因家族的扩张可能发生在现代玉米品种建立之前。进一步支持这一假设,我们能够证实所有的梅格大刍草的同源物(Zea mays ssp。parviglumis),表明梅格大约4000 - 10000年前,在玉米从其野生祖先驯化之前,基因簇已经完全扩展3.:图S2)。
我们重建了梅格家族基因采用最大似然法,与谷子有远亲关系梅格用作外部群体的基因由此产生的系统发育确定了一个由12个B73组成的大分支梅格基因和高粱之一梅格同源染色体(SbMeg1),与Meg14和其他高粱同源物(SbMeg2),并有强有力的统计数据支持(图2一个玉米)。Meg14和高粱SbMeg2共享同源的下游侧翼序列和附近推定的硫氧还蛋白H基因(图1A),进一步支持他们的分组。总之,这些数据表明玉米Meg14/SbMeg2可能已经与玉米分道扬镳了Meg1-13/SbMeg1玉米/高粱组从小米分裂后的分枝,但在玉米/高粱分化之前。
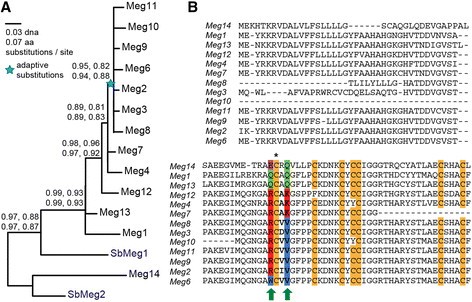
玉米的系统发育分析梅格基因鉴定适应性氨基酸取代。(一)我们从蛋白质和相应的DNA序列数据中重建了最大似然系统发育。类sh的aLRT支持[28]对于有或没有gblock的蛋白质序列数据,在关键节点显示[29]处理,以去除不可靠的对齐位置(上一行)和DNA对齐是否有Gblocks处理(下一行)。在任何分析中具有<0.8 SH-like aLRT支持的节点都将崩溃,并且使用基因-物种树调和来建立树的根,以最大限度地减少重复/丢失事件。蓝色星号表示该分支中适应性替代的显著支持(p多重试验校正后< 0.05),使用基于密码子的分析推断(见方法)。(B)我们绘制氨基酸取代推断为自适应的分支位点分析(Zhang等) [30.]沿着Meg蛋白序列的排列(绿色箭头)。氨基酸的生化性质标记为:粉红色为亲水极性,绿色为亲水极性不带电,红色为亲水极性碱性,蓝色为疏水非极性氨基酸。保守的半胱氨酸残基以橙色突出显示。
另外用谷子外群生根梅格序列,我们使用基因树/物种树调和,通过最小化基因增益/损失事件来估计根系发生[31]。最吝啬的根(图2A)支持两种观点梅格基因存在于玉米和高粱的共同祖先中。这些祖先基因中的一个在两个物种(玉米)中都保留为单一副本Meg14/SbMeg2),而另一个祖先在玉米基因组中经历了至少两次的一系列快速扩张。玉米Meg1落在玉米特定膨胀的底部,并与另一个分离梅格同源性有很强的支持。Meg1也位于下游的其他特定的玉米梅格基因(图1A),认为Meg1基因可能是通过一系列“上游”复制事件发生的玉米扩张的原始祖先。系统发育“年龄”和染色体位置之间的一致性支持这一一般模型,基因在物理位置上更接近Meg1下坠的倾向于向底部坠落的梅格系统发育(见图1B和2一个)。
确定…的时间梅格在基因重复方面,我们使用一个分子钟,以玉米-高粱的分歧时间为约11.9 mya校准,重建了最大似然系统发育树。26]。与不存在相一致梅格玉米2号染色体上的基因分子钟分析表明梅格玉米异体四倍化后发生基因扩增(附加文件)3.:图S3)。根据这一分析,大多数梅格基因(Meg2-11)最近通过一系列快速的复制事件出现,无法在系统发育学上解决(即大约0.90-1.58 mya)。Meg12推测其产生时间约为1.77-2.77 mya,是玉米-高粱分裂后最古老的复制体,Meg1而且Meg13,在玉米异体四倍化后立即产生~ 3.07-4.80 mya。尽管我们对这些复制事件的具体日期的分配很谨慎,因为分子钟的假设很可能被违反,这些结果提出了一个模型梅格在同种异体四倍化后(约4.8 mya),驯化前(约4000- 10000年前),玉米基因簇迅速扩增。这些结果被同步性检查和系统发育分析证实(图2A、附加文件3.:图S1),它们不依赖于分子钟的假设。
积极选择的证据推动了梅格蛋白质二级结构
富半胱氨酸蛋白(CRPs)的功能分化通常与基因复制有关,然后是改变蛋白质功能的正选择[32] - [34]。为了描述适应过程在形成玉米蛋白质功能中的可能作用,我们使用了基于非同义替换与同义替换比值的统计分析梅格同源染色体。这些分析确定了系统发育上的一个分支显示出蛋白质编码适应的强有力证据,分支联合Meg3-9,表示最近特定于玉米的扩展事件(p多次校正后< 0.05;数字2一个)。
分支位点分析进一步鉴定了两种氨基酸的取代Meg3-9似乎是由正向选择驱动的分支(图2B).这些取代用VV基序取代了第一个保守半胱氨酸旁边的保守AK基序,改变了该区域的大小、电荷和疏水性。另外一个不寻常的精氨酸到色氨酸的取代Meg6提示该位置可能是Meg蛋白功能分化的一个“热点”。
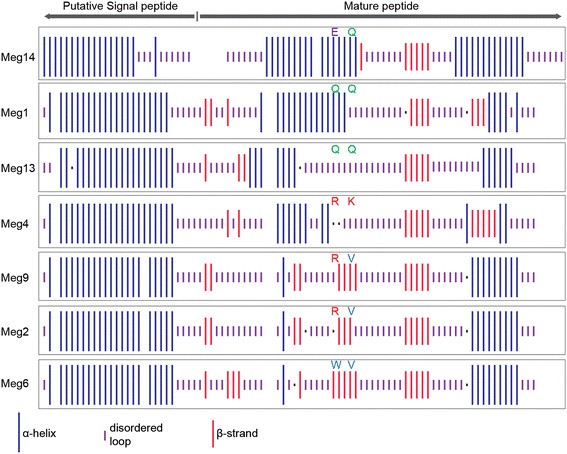
虽然目前还没有支持Meg蛋白同源建模的晶体结构,但我们对Meg蛋白的二级结构进行了表征,以确定氨基酸取代的可能结构后果。我们发现,在玉米特异过程中,α-螺旋的比例普遍减少,而β-链的比例相应增加梅格家庭扩张(表1,图3.).例如,最古老的Meg蛋白Meg1和Meg14被预测分别含有52.81%和45.45%的α-螺旋。相比之下,最年轻的蛋白质Meg9、Meg2和Meg6分别为35.63%、36.36%和36.36%的α -helix(表2)1).进化中间产物Meg13和Meg4的α -螺旋含量介于最古老和最年轻的基因之间(即。分别为38.64%和37.50%)。β-链比例呈现相反的趋势,β-链比例从年龄大到年龄小逐渐增加(表2)1).
Meg蛋白二级结构随着玉米特异基因家族的扩展而发生变化。在网络序列分析服务器(NPS@, Network Protein sequence analysis,http://npsa-pbil.ibcp.fr).α-螺旋区、β-链区和无序环区分别以最长、次长和次短的条形表示。最短的条表示具有模糊状态的残差。积极选择的氨基酸符号显示在相应的条形图上方。根据氨基酸序列比对引入间隙,以便对二级结构元素进行比对以实现可视化。该图显示了氨基酸序列梅格编码序列完整的基因。
我们在解释二级结构预测时很谨慎,因为现代方法只能达到80%的精度[http://ieeexplore.ieee.org/xpls/abs_all.jsp?arnumber=6217208]。然而,值得注意的是,预测的蛋白质二级结构的局部变化与被确定为正选择的特定氨基酸密切相关(图3.).该蛋白区域形成Meg1和Meg14中成熟肽的第一个α-螺旋。在中年Meg4和Meg13中,围绕自适应变化的区域被预测为无序的,导致第一个α-螺旋长度的整体减少。在最近衍生的Meg2、Meg6和Meg9中,第一个α-螺旋被预测完全缺失,并被一个保守的β-链所取代(图3.).总的来说,这些结果表明,玉米Meg蛋白的n端区域经历了一个系统的和定向的结构重组梅格基因家族。尽管三维结构数据的缺乏和二级结构预测的低准确性限制了我们对Meg蛋白序列的变化如何改变蛋白质功能得出强有力结论的能力,但自适应蛋白质编码变化和预测二级结构的改变合流确实表明,这些进化变化在某种程度上改变了Meg蛋白功能。
最近玉米选择性扫荡的证据梅格基因簇
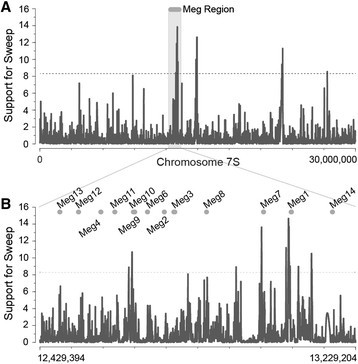
研究近期选择性扫荡在玉米中的作用梅格基因进化,我们分析了玉米多态性数据[35]、[36]采用综合似然方法确定种群水平的适应性[37]。我们发现梅格在玉米染色体7S的整个远端30 Mb区域具有最强的自适应扫描特征(图4A).尽管我们对这些方法识别基因组中选择性扫描的精确位置的能力持谨慎态度[37],我们注意到,最有力的支持种群水平适应本地化到Meg9-10就在上游Meg1而且Meg7(图4B)这些假定的适应性扫掠的功能后果仍然未知,尽管这些结果确实表明玉米梅格基因簇最近可能经历了正选择,进一步支持了玉米适应的一般模式梅格基因家族扩展与多样化。
仅从系统发育和群体遗传分析中不可能得出关于蛋白质功能适应性变化的明确结论,因此我们认为这些结论在这一点上是推测性的。然而,我们注意到,非同义/同义替代比升高的统计证据、非保守的氨基酸替代、预测二级结构的局部变化以及可能的选择性横扫的群体遗传证据都支持适应在玉米中发挥作用的模型梅格基因的扩张。
表达谱梅格基因
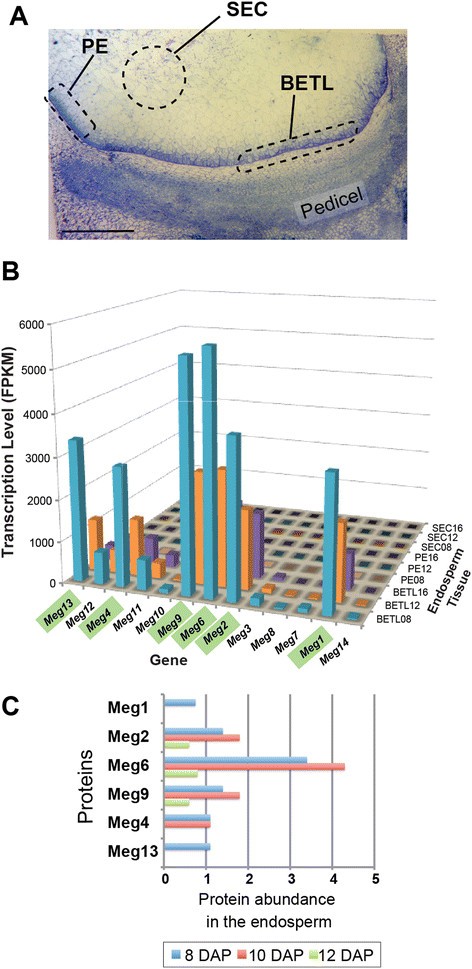
测定…的转录谱梅格我们测量了三个发育阶段基础胚乳转移细胞(BETCs)、淀粉胚乳细胞(SECs)和含糊粉细胞的外周胚乳(PE)的mRNA水平(图5A).我们发现抄本水平为6梅格基因(Meg1,Meg2, Meg4, Meg6, Meg9,Meg13)显著高于其他梅格基因(未配对t测试:双尾p< 0.0001)(图5B).这些基因均在授粉后8、12、16天(DAP)的betc中特异性高表达(FPKM > 4800),且三者连续梅格的基因,Meg2, Meg6而且Meg9是转录水平最高的(图5B).与这些高度表达的梅格同系物,五梅格基因在所有细胞类型和时间点的转录水平均可忽略不计(Meg7, Meg8, Meg3, Meg10而且Meg14, FPKM < 365),其余两个梅格基因具有中间水平的转录,特别是在BETCs (FPKM = 1368和1910)Meg9而且Meg11分别)。
这些转录水平的差异梅格基因与基因完整性的保存有良好的相关性梅格基因。启动子和/或两个典型外显子在五个中被破坏梅格FPKM值低的基因(图1).Meg11而且Meg12表现为中等转录水平,似乎具有规范梅格基因结构。然而,Meg11它的启动子有22个bp的缺失Meg12包含一个帧移突变,可能影响其转录本的稳定性。Meg12已被注释为伪基因(www.maizesequences.org).
尽管转录水平有很大差异,所有梅格基因表现出相似的时空表达模式。它们的转录严格局限于BETCs,转录水平在8dap时最高,但随后下降(图5B).这些结果表明梅格玉米基因家族不包括表达模式的多样化,但包括同源基因之间表达水平的变化,最近衍生的完整基因通常具有更高的表达水平。
为了进一步研究的表达梅格在蛋白质水平上,我们搜索了玉米蛋白质类型图集数据库(http://maizeproteome.ucsd.edu),其中对玉米种子组织的蛋白质组学分析结果进行了编目。从6种中鉴定出多肽梅格基因,对应于胚乳中转录物浓度最高的6个基因(图5C)其他7种多肽梅格数据库中没有基因。此外,蛋白丰度高表达梅格基因在8 - 10dap达到峰值,随后下降,与转录水平一致。
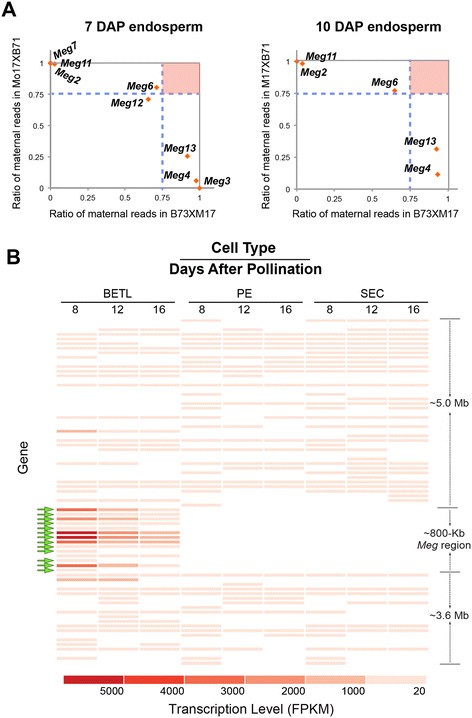
因为Meg1是母系表达的印迹基因,我们检查了其他的印迹状态吗梅格基因来自公开的B73XMo17互交生成的转录组数据集[38] - [40]。Meg1表达在4dap时被母系印迹,但在12dap时变成双等位基因[14]。转录组数据集是在7 DAP和10 DAP的胚乳样本中生成的Meg1的表情消失了。首先,我们比较了所有的编码序列梅格检测B73和Mo17自交系的单核苷酸多态性(SNPs)。我们在8个样本中鉴定出了snp梅格B73和Mo17的等位基因3.:图S4)和8个基因在Xin数据集中的父母表达比et al。[39]。不像Meg18个基因均无亲本特异性表达。相反,Mo17的等位基因Meg2,Meg7,Meg11的B73等位基因表现出较强的显性Meg3,Meg4,Meg13超过Mo17(图6一个)。Meg6而且Meg12没有表现出等位基因特定的表达模式。B73和Mo17等位基因均未鉴定出snpMeg1,Meg3,Meg9而且Meg10我们无法在数据集中找到他们的父母特定表达的信息。表达式数据Meg3、Meg4、Meg13均可从沃特斯购买et al。[38],与图中的结果一致6A.这些表明原点亲本的特定表达Meg1在8中不守恒梅格我们在B73XMo17表达数据集中检查的重复。
印记状态梅格基因和胚乳表达模式的非梅格基因梅格地区。(一)母系表达率梅格从B73XM17互惠杂交的胚乳中提取7 DAP(左图)和10 DAP(右图)的基因。水平和垂直的虚线标记了每个交叉中3:1的母体和父亲表达比例的边界。如果一个基因的母系等位基因的表达量是父系等位基因的3倍,则该基因应出现在右上角(红色方框)。这些比值由Xin根据胚乳转录组数据计算得出et al。[39]。15个DAP胚乳中未检测到Meg基因的表达。(B)描述基因在BETCs转录活性的热图在~9.4 mb的区域横跨梅格基因聚集。采用归一化基因表达水平(FPKM)生成图谱。梅格基因用绿色箭头标出。FPKM值的6个高表达梅格在9.4 Mb区间内,基因的数量(>3000)远远大于其他基因。在9个样本中,FPKM < 20的基因从热图中被省略。
的梅格基因区包括B73基因组数据库中的48个注释(AGPv2,工作基因集),其中13梅格基因。在其他35个注释中,13个是转座子,11个是假基因或缺乏编码序列,11个被预测为具有完整开放阅读框的蛋白质编码基因。为了确定这11个假定的蛋白质编码基因是否在胚乳中具有转录活性,我们使用BLAST程序搜索了我们的胚乳转录组数据。有3个基因(GRMZM2G553132、GRMZM2G144653、GRMZM2G150091)在胚乳中被鉴定为转录本,但转录量在5% ~ 20%之间Meg6成绩单(图2B,6B). GRMZM2G144653在三种细胞类型中均有表达,而GRMZM2G553132和GRMZM2G150091在BETCs中均有特异性表达。高水平的梅格BETCs转录本提示梅格该区域与BETCs中的转录“热点”相对应,即使该区域表现出熵点周围异染色质的特征。
结论
的梅格自玉米与高粱分化以来,玉米的基因家族急剧扩大。然而,这种扩张的功能后果尚不清楚。Meg蛋白是CRP超家族的成员,其他成员在真核细胞的细胞信号传导和防御中发挥着不同的作用[3.]。大部分的玉米梅格基因只在BETL中表达,很明显Meg1通过促进BETL的形成而参与控制营养物质的转运[20.]。高粱和玉米都有betl [41]、[42),但梅格基因只在玉米中扩增。这表明通过BETL控制种子发育和养分分配的细胞信号网络在玉米中可能是多样化的。另外,梅格基因家族的扩展可以改变玉米母体组织中负责隔离发育中的种子与感染的分子机制。印痕的丢失梅格基因的概念是符合功能多样性的梅格家庭在发展过程中不断扩大。所起的功能作用的进一步检查梅格家族基因可能会增强我们对串联基因复制事件如何促进植物物种特异性适应的理解。
在这项研究中,我们检查了最近复制的基因的进化,通过系统发育和群体遗传分析的结合来识别分子选择,并通过表征它们的表达、定位、印迹和蛋白质结构来识别重复基因之间的功能差异。我们观察到编码外显子和启动子序列的变化梅格玉米中的基因阵列,与串联复制基因阵列生产过程中引入的错误可能是基因表达和蛋白质功能差异的重要来源的模型一致。我们期望对基因复制过程的全面理解将阐明“不完美”基因复制在产生驱动进化和适应所必需的分子多样性方面的潜在作用。
方法
细胞类型特异性样品制备和测序文库构建
这是不可能区分每一个转录水平梅格由于它们的核苷酸序列高度相似,可以通过RNA印迹分析或定量RT-PCR进行分析。我们在所有的两个外显子中确定了核苷酸多态性梅格基因和分化的Illumina reads映射到不同的外显子梅格基因根据他们的序列多态性(附加文件3.:图S4)。因此,我们使用Illumina测序技术来识别和量化每个成员的转录本梅格家庭。分离出三种胚乳细胞类型,并制备每种细胞类型的总RNA样本,如[21]、[43]。简而言之,将8、12和16 DAPs的玉米B73籽粒冷冻在液氮中,并用Tissue-Tek OCT化合物安装在标本架上(Ted Pella, CA)。在−15℃下采集50 μm切片,冷冻切片在乙醇中脱水,用HistoGene LCM冷冻切片染色试剂盒(MDS Analytical Technologies, CA)染色。BETL、SEC和PE样品在解剖立体显微镜下使用外科手术刀从切片中切除,直到每个样品获得约2 μg总RNA。总RNA样本用Arcturus PicoPure RNA分离试剂盒(Life Technologies, CA)纯化,RNA-seq文库根据Zhong先前发表的方案构建et al。[43],略有改动。使用随机六聚体引物反转录生成第一链cDNA,随后进行第二链cDNA合成和适配器结扎。用Ampure XP (Beckman Coulter)分离约200-500 bp的cDNA片段,经15次PCR扩增。在Illumina HiSeq 2000平台上测序之前,该文库通过定量PCR和生物分析仪(Agilent, CA)进行定量。图中的亮场显微照片5按[44]。Illumina reads已保存在NCBI Sequence Read Archive(登录号SRA175303)。
基因表达的序列预处理、定位和定量
原始数据按条形码分组到单独的文件中。使用CUTADAPT删除适配器序列[45]、[46]然后是条形码修剪,质量修剪(−t 20,−l 50),工件去除和质量过滤(−q 20,−p 90)使用FASTX-TOOLKIT (http://hannonlab.cshl.edu/fastx_toolkit/).用bowti2去除线粒体、叶绿体和核糖体[47]使用默认设置。如果只剩下一端,则将处理后的读取放入成对端模式或单端模式。
读取数据使用GSNAP对齐[48]使用RefGen_v2工作基因集中提供的已知拼接结,参数为:−-nthreads 12,−-batch 5,−-max-mismatches 0.05,−-npaths 5,−-quiet-if-excess,−novelsplicing yes,−-split-output。只有文件concordant_uniq、concordant_mult、halfmapping_uniq、unpaired_uniq和unpaired_mult中的读取被合并用于下游分析。在此过程中,下游分析排除了易位、置乱和反转读数。皮卡德(http://picard.sourceforge.net/)用于去除每个样本组合对齐中的重复读取。
使用袖扣(CUFFLINKS)组装最终清洗的对准线[49-max-intron-length 8000, -min-intron-length 20, -GTF ZmB73_5a_WGS-chr1-10。−b ZmB73_5b-chr1-10.fasta. gff,−b ZmB73_5b-chr1-10.fasta. gff,通过FPKM(每千碱基外显子每百万片段的片段)测量表达水平[49]为每个被识别为在梅格基于CUFFLINKS结果的基因家族。对于印迹分析,数据集来自Xinet al。[39),水et al。[38],张et al。[40]从植物细胞网站和每个梅格从互交中寻找其表达率。
序列注释
玉米自交系B73的基因密度是根据玉米基因组组装的大小(版本AGPv2)和过滤基因的总数(版本5b)估算的(http://www.maizesequence.org/).通过检索玉米转座因子(TE)数据库(http://maizetedb.org/)使用BLASTN程序,e值截止为1E-20。
系统发育重建
的翻译氨基酸梅格基因在MUSCLE [50],然后进行人工校正,然后使用基于蛋白质的比对构建DNA比对。使用Phyml v3.0构建最大似然系统发生[51],采用赤池信息准则(AIC)选择进化模型[52]。最适合的进化模型是蛋白质序列的JTT + G + F和核苷酸序列的HKY + G。分支支持度使用sh样近似似然比检验(aLRTs)来推断[28]。
编码蛋白质的适应
我们使用分支特定模型来推断蛋白质编码适应,以推断具有过量正选择的分支,在PAML v4.5中实现[30.]、[53]。将模型M2a(阳性选择)应用于系统发育上的每个分支,并与模型M1a(接近中性)进行比较,以确定经历正向蛋白质编码适应的特定分支。采用2自由度卡方检验评估显著性[54]。我们使用Bonferroni校正对多个测试进行校正;我们只报告那些确定为自适应的结果p经多次试验校正后,< 0.05。利用分支位点分析,适应进一步定位到特定位置(Zhang等) [30.],推断后验概率为>0.95的位置为自适应。
二级结构预测
在网络序列分析服务器上预测并生成共识二级结构[55网络蛋白序列分析,NPS@http://npsa-pbil.ibcp.fr/).利用层次神经网络、双预测法、蛋白质二级结构类判别、Garnier、Gibrat、多元线性回归组合、PHD、Predator、NPS@服务器上的SOPM等多种算法预测蛋白质二级结构。
识别选择性扫描
利用玉米HapMap数据库中的单核苷酸多态性数据(http://www.panzea.org)(贾等;高度等) [35]、[36]。我们切除了周围30mb的基因组DNA区域梅格使用复合似然比检验(CLRT)评估自适应扫描的支持度(Nielsen等) [37]。CLRT在两种模型下计算了基因组中特定位置的局部位点频谱(SFS)的可能性:1)整个区域计算的背景SFS和2)在背景SFS中诱导特征扫样倾斜的单参数模型。对扫描模型的支持报告为扫描模型与背景SFS的对数似然比。我们扫描30 mb基因组区域,每100 bp进行自适应扫描采样。
在标准中性模型下使用100,000个联合模拟评估显著性,模拟条件是观察到的分离位点数量和每个区域的测序覆盖模式。对于每个模拟复制数据集,我们使用CLRT计算有利于自适应扫描的对数似然比,产生一个null分布,从中估计P -观测到的对数似然比值。这种方法已被证明对人口历史的变化(如人口瓶颈)是稳健的(Nielsen et al. [37])。
附加文件
参考文献
- 1.
Talbot MJ, Offler CE, McCurdy DW:转移细胞壁结构:对理解局部壁沉积的贡献。中国生物工程学报,2002,29(4):379 - 379。10.1007 / s007090200021。
- 2.
Offler CE, McCurdy DW, Patrick JW, Talbot MJ:转移细胞:有特殊用途的细胞。植物学报,2003,29(4):344 - 344。10.1146 / annurev.arplant.54.031902.134812。
- 3.
Marshall E, Costa LM, Gutierrez-Marcos J:富含半胱氨酸的多肽(CRPs)在植物繁殖和发育过程中介导细胞-细胞通讯的各个方面。实验学报,2011,62(5):1677-1686。10.1093 / jxb / err002。
- 4.
Silverstein KA, Moskal WA, Wu HC, Underwood BA, Graham MA, Town CD, VandenBosch KA:类似抗菌肽的富含半胱氨酸的小肽在植物中被低估了。植物学报,2007,26(2):366 - 366。10.1111 / j.1365 - 313 x.2007.03136.x。
- 5.
Wheeler MJ, Vatovec S, Franklin-Tong VE:罂粟花粉S决定因子:与已知植物受体和蛋白质配体伙伴的比较。实验学报,2010,61(7):2015-2025。10.1093 / jxb / erp383。
- 6.
杨晓明,王晓明,王晓明,王晓明,等。植物防御素抗真菌活性和钙通道阻断活性的研究进展。中国生物医学工程学报,2004,35(4):344 - 344。10.1104 / pp.104.040873。
- 7.
Tailor RH, Acland DP, Attenborough S, Cammue BP, Evans IJ, Osborn RW, Ray JA, Rees SB, Broekaert WF:从凤仙花(Impatiens balsamina)种子中提取的富含半胱氨酸的小抗菌肽是由单一前体蛋白衍生而来的。中国生物医学工程学报,1997,27(4):344 - 344。10.1074 / jbc.272.39.24480。
- 8.
Sugano SS, Shimada T, Imai Y, Okawa K, Tamai A, Mori M, Hara-Nishimura I:拟南芥气孔原正调控气孔密度自然科学学报,2010,38(4):344 - 344。10.1038 / nature08682。
- 9.
Okuda S, Tsutsui H, Shiina K, Sprunck S, Takeuchi H, Yui R, Kasahara RD, Hamamura Y, Mizukami A, Susaki D, Kawano N, Sakakibara T, Namiki S, Itoh K, Otsuka K, Matsuzaki M, Nozaki H, Kuroiwa T, Nakano A, Kanaoka MM, Dresselhaus T, Sasaki N, Higashiyama T:防御素样多肽诱导剂是由助细胞分泌的花粉管引诱剂。自然科学进展,2009,37(1):1 - 7。10.1038 / nature07882。
- 10.
Wheeler MJ, de Graaf BH, Hadjiosif N, Perry RM, Poulter NS, Osman K, Vatovec S, Harper A, Franklin FC, Franklin- tong VE:罂粟属花粉自交不亲和决定因素的鉴定。自然科学进展,2009,39(1):1 - 4。10.1038 / nature08027。
- 11.
Costa LM, Marshall E, Tesfaye M, Silverstein KA, Mori M, Umetsu Y, Otterbach SL, Papareddy R, Dickinson HG, Boutiller K, VandenBosch KA, Ohki S, Gutierrez-Marcos JF:开花植物中中央细胞来源的肽调控早期胚胎模式。科学通报,2014,38(4):344 - 344。10.1126 / science.1243005。
- 12.
蔡g, Faleri C, Del Casino C, Hueros G, Thompson RD, Cresti M:玉米胚乳中BETL-1, - 2和- 4的亚细胞定位。性植物繁殖,2002,15(2):85-98。10.1007 / s00497 - 002 - 0141 - 9。
- 13.
Serna A, Maitz M, O'Connell T, Santandrea G, Thevissen K, Tienens K, Hueros G, Faleri C, Cai G, Lottspeich F, Thompson RD:玉米胚乳分泌一种新的抗真菌蛋白到邻近的母质组织。植物学报,2001,25(6):687-698。10.1046 / j.1365 - 313 x.2001.01004.x。
- 14.
Gutierrez-Marcos JF, Costa LM, Biderre-Petit C, Khbaya B, O'Sullivan DM, Wormald M, Perez P, Dickinson HG:母系表达基因1是一种新的玉米胚乳转移细胞特异性基因,具有母系亲本表达模式。植物细胞。2004, 16(5): 1288-1301。10.1105 / tpc.019778。
- 15.
郭艳,郭艳,王晓明。玉米转移细胞特异转录因子ZmMRP-1表达域的构建与鉴定。植物细胞。2002, 14(3): 599-610。10.1105 / tpc.010365。
- 16.
Barrero C, Royo J, Grijota-Martinez C, Faye C, Paul W, Sanz S, Steinbiss HH, Hueros G:玉米转移细胞特异性转录激活子ZmMRP-1的启动子在溶质交换表面被诱导并响应运输需求。植物学报,2009,229(2):235-247。10.1007 / s00425 - 008 - 0823 - 0。
- 17.
罗宇J, Gómez E, Barrero C, Muñiz LM, Sanz Y, Hueros G: ZmMRP-1对玉米胚乳转移细胞特异性基因BETL1转录激活的影响植物学报,2009,30(4):344 - 344。10.1007 / s00425 - 009 - 0987 - 2。
- 18.
Sabelli PA, Larkins BA:草类胚乳的发育。植物学报,2009,49(1):344 - 344。10.1104 / pp.108.129437。
- 19.
胚乳印迹:儿童监护权之争?动物学报,2012,22 (3):R93-R95。10.1016 / j.cub.2011.12.043。
- 20.
刘志刚,王志刚,王志刚,王志刚,Gutiérrez-Marcos植物种子养分分配基因印迹的母本调控。中国生物医学杂志,2012,22:160-165。10.1016 / j.cub.2011.11.059。
- 21.
熊永强,李秋波,康波峰,Chourey PS:利用454转录组测序发现玉米基底胚乳转移细胞表达基因。植物分子生物学研究,2011,29(4):835-847。10.1007 / s11105 - 011 - 0291 - 8。
- 22.
傅华,朴玮,闫旭,郑智,沈波,杜纳HK:高重组基因bz位点位于玉米基因组中基因异常丰富的区域。中国科学:自然科学,2001,29(3):389 - 397。10.1073 / pnas.141221898。
- 23.
Schnable PS, Ware D, Fulton RS, Stein JC, Wei F, Pasternak S, Liang C, Zhang J, Fulton L, Graves TA, Minx P, Reily AD, Courtney L, Kruchowski SS, Tomlinson C, Strong C, Delehaunty K, Fronick C, Courtney B, Rock SM, Belter E, Du F, Kim K, Abbott RM, Cotton M, Levy A, Marchetto P, Ochoa K, Jackson SM, Gillam B,等:B73玉米基因组:复杂性、多样性和动态。科学通报,2009,26(5):528 - 528。10.1126 / science.1178534。
- 24.
刘珊,叶春涛,季涛,应凯,吴海红,唐宏宏,付勇,Nettleton D, Schnable PS: Mu转座子插入位点和减数分裂重组事件与玉米基因组开放染色质表观遗传标记共定位。公共科学学报,2009,5 (11):e1000733-10.1371/journal.pgen.1000733。
- 25.
高志刚,李志刚,李志刚,李志刚。植物基因重组:植物基因组进化中一个被忽视的因素。植物学报,2007,8(1):77-84。10.1038 / nrg1970。
- 26.
Swigonová Z,赖军,马军,Ramakrishna W, Llaca V, Bennetzen JL, Messing J:高粱和玉米基因组祖细胞的紧密分离。中国生物医学工程学报,2004,14(10):1616 -1923。10.1101 / gr.2332504。
- 27.
Gaut BS, Doebley JF:玉米片段异体四倍体起源的DNA序列证据。中国科学:自然科学,2007,26(3):389 - 397。10.1073 / pnas.94.13.6809。
- 28.
Anisimova M, Gil M, Dufayard JF, Dessimoz C, Gascuel O:分支支持方法的调查证明了快速基于似然逼近方案的准确性、威力和鲁棒性。中国生物医学工程学报,2011,60(5):685-699。10.1093 / sysbio / syr041。
- 29.
Castresana J:从多个比对中选择保守块用于系统发育分析。分子生物学杂志,2000,17(4):540-552。10.1093 / oxfordjournals.molbev.a026334。
- 30.
张军,杨震,尼尔森R:一种改进的分支位点似然方法在分子水平上检测正选择。分子生物学杂志,2005,22(12):2472-2479。10.1093 / molbev / msi237。
- 31.
陈凯,杜兰·D, Farach-Colton M: NOTUNG:一种用于基因重复定年和优化基因家族树的程序。中国生物医学工程学报,2000,7(3-4):429-447。10.1089 / 106652700750050871。
- 32.
Radhakrishnan Y, Hamil KG, Yenugu S, Young SL, French FS, Hall SH:灵长类β -防御素基因簇的鉴定、特征和进化。基因学报,2005,6(3):203-210。10.1038 / sj.gene.6364184。
- 33.
Graham MA, Silverstein KAT, Vandenbosch KA:类防御素基因:植物中一个不同超家族的基因组视角。作物科学,2008,48(增刊1):S3-S11。
- 34.
Silverstein KAT, Graham MA, Paape TD, VandenBosch KA:拟南芥300多个防御素样基因的基因组组织。植物学报,2005,38(3):344 - 344。10.1104 / pp.105.060079。
- 35.
Chia JM, Song C, Bradbury PJ, Costich D, de Leon N, Doebley J, Elshire RJ, Gaut B, Geller L, Glaubitz JC, Gore M, Guill KE, Holland J, Hufford MB, Lai J, Li M, Liu X, Lu Y, McCombie R, Nelson R, Poland J, Prasanna BM, Pyhajarvi T, Rong T, Sekhon RS, Sun Q, Tenaillon MI, Tian F, Wang J, Xu X,等:玉米happmap2识别通量基因组的存在变异。植物学报,2012,44(7):803-807。10.1038 / ng.2313。
- 36.
Hufford MB, Xu X, van Heerwaarden J, Pyhajarvi T, Chia JM, Cartwright RA, Elshire RJ, Glaubitz JC, Guill KE, Kaeppler SM, Lai J, Morrell PL, Shannon LM, Song C,施普林格NM, swson - wagner RA, Tiffin P, Wang J, Zhang G, Doebley J, McMullen MD, Ware D, Buckler ES, Yang S, Ross-Ibarra J:玉米驯化与改良的比较群体基因组学。植物学报,2012,44(7):808-811。10.1038 / ng.2309。
- 37.
Nielsen R, Williamson S, Kim Y, Hubisz MJ, Clark AG, Bustamante C:使用SNP数据的选择性扫描基因组扫描。中国生物工程学报,2005,15(11):1566-1575。10.1101 / gr.4252305。
- 38.
Waters AJ, Makarevitch I, Eichten SR, swson - wagner RA, Yeh C-T, Xu W, Schnable PS, Vaughn MW, Gehring M,施普林格NM:玉米胚乳基因表达和DNA甲基化的亲本效应。植物细胞。2011, 23(12): 4221-4233。10.1105 / tpc.111.092668。
- 39.
李鑫M,杨R, G,陈H,劳里J,马C, D,姚明Y,拉金斯英航,太阳Q, Yadegari R,王X, Z倪:印迹基因的动态表达与母亲般地associates控制养分分配在玉米胚乳发育。植物细胞。2013, 25(9): 3212-3227。10.1105 / tpc.113.115592。
- 40.
张明明,赵辉,谢松,陈娟,徐勇,王凯,赵辉,关辉,胡霞,焦勇,宋伟,赖俊:玉米胚乳发育过程中蛋白质编码和非编码rna的广泛聚集亲本印迹。《美国国家科学院学报》上。2011, 38(5): 528 - 528。10.1073 / pnas.1112186108。
- 41.
Kang BH, Xiong YQ, Williams DS, pozuita - romero D, Chourey PS:玉米胚乳转移细胞的壁内转化酶对壁内生长的组装和功能至关重要。中国生物医学工程学报,2009,29(3):366- 366。10.1104 / pp.109.142331。
- 42.
王红辉,王忠,王峰,顾玉杰,刘忠:高粱基底胚乳转移细胞的发育门氏菌及其与颖果生长的关系。中国生物医学工程学报,2011,32(2):379 - 379。10.1007 / s00709 - 011 - 0281 - 6。
- 43.
钟山,钟建刚,郑勇,陈玉茹,刘波,邵毅,向建正,费志,Giovannoni JJ:高通量illumina链特异性RNA测序文库制备。冷泉港协议。2011,ᅟ:ᅟ-doi:10.1101/pdb.prot5652(8)
- 44.
李克红,朴杰,李志勇,黄毅,姜波峰:拟南芥RH3 DEAD-box蛋白突变体叶绿体发育缺陷抑制了突变体脱落酸的正常水平。植物学报,2013,29(5):344 - 344。10.1111 / tpj.12055。
- 45.
Shimodaira H, Hasegawa M:对数可能性的多重比较及其在系统发育推断中的应用。中华生物医学杂志,1999,16(8):1114-1116。10.1093 / oxfordjournals.molbev.a026201。
- 46.
Martin M: Cutadapt从高通量测序读取中移除适配器序列。电子工程学报,2011,17(1):10-12。10.14806 / ej.17.1.200。
- 47.
朗米德B,萨尔茨堡SL:快速gap -read对齐领结2。光子学报,2012,9(4):357-359。10.1038 / nmeth.1923。
- 48.
Wu TD, Nacu S:短读中复杂变异和剪接的快速和耐snp检测。生物信息学,2010,26(7):873-881。10.1093 /生物信息学/ btq057。
- 49.
Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR, Pimentel H, Salzberg SL, Rinn JL, Pachter L: TopHat和Cufflinks RNA-seq实验的差异基因和转录本表达分析。Nat协议,2012,7(3):562-578。10.1038 / nprot.2012.016。
- 50.
Edgar RC: MUSCLE:多序列比对,高精度,高通量。中国生物医学工程学报,2004,32(5):1792-1797。10.1093 / nar / gkh340。
- 51.
Guindon S, Dufayard J-F, Lefort V, Anisimova M, Hordijk W, Gascuel O:估计最大似然系统发生的新算法和方法:评估PhyML 3.0的性能。系统生物学,2010,59(3):307-321。10.1093 / sysbio / syq010。
- 52.
Darriba D, Taboada GL, Doallo R, Posada D: ProtTest 3:蛋白质进化的最佳拟合模型的快速选择。生物信息学,2011,27(8):1164-1165。10.1093 /生物信息学/ btr088。
- 53.
杨忠:PAML 4:最大似然系统发育分析。分子生物学杂志,2007,24(8):1586-1591。10.1093 / molbev / msm088。
- 54.
王卫伟,杨震,高德曼,尼尔森R:检测蛋白质编码序列自适应进化和识别正向选择位点的统计方法的准确性和力量。中国生物医学工程学报,2004,18(2):344 - 344。10.1534 / genetics.104.031153。
- 55.
Combet C, Blanchet C, Geourjon C, Deleage G: NPS@:网络蛋白序列分析。生物化学,2000,25(3):147-150。10.1016 / s0968 - 0004(99) 01540 - 6。
致谢
这项工作得到了农业和食品研究计划竞争性赠款2010-0496的支持,该赠款来自美国农业部国家食品和农业研究所,植物生物学计划和国家科学基金会授予b.h h k ISO 1025976。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们没有竞争利益。
作者的贡献
YX和B-HK设计了这项研究。YX获得了大部分数据。YX, WM, EK, HH, BB, SB和B-HK分析梅格基因座,他们的基因组区域和表达数据。YX, KM, BK和B-HK进行进化分析。YX, BK和B-HK撰写了手稿。所有作者都阅读并批准了最终的手稿。
电子辅助材料
Meg区与高粱2号染色体、水稻7号染色体、玉米2号和7号染色体的同源区有共线关系。
附加文件3:的同源和同源区域梅格聚类由GEvo分析确定(Lyons和Freeling, 2008)。梅格基因模型用红色条表示,其他基因用绿色条表示。高粱2号染色体和玉米7号染色体在这一区域都含有大量的F-box基因拷贝。这两个区域的F-box基因用蓝线相连。所有其他锚定基因都与灰色线相连。所有的基因模型和区间都根据其物理大小按比例绘制。该图改编自GEvo分析结果。(docx981 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。
本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。
如欲查看本牌照的副本,请浏览https://creativecommons.org/licenses/by/4.0/.
创作共用公共领域奉献弃权书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
熊勇,梅伟,金,ED。et al。玉米的适应性扩张母系表达基因(梅格)家族涉及其成员的表达模式和蛋白质二级结构的变化。BMC植物生物学14日,204(2014)。https://doi.org/10.1186/s12870-014-0204-8
收到了:
接受:
发表:
关键字
- ■■■