摘要
背景
甜橙(素类)是世界上最重要的水果之一。由于甜橙是一种生长周期较长的木本植物,因此对甜橙的遗传研究滞后于其他物种。
结果
在本分析中,我们采用同源鉴定和结构域组合方法来预测甜橙的蛋白-蛋白相互作用(PPI)网络。采用k近邻(KNN)分类方法对网络进行验证和过滤。最终预测的PPI网络CitrusNet包含8195个蛋白,相互作用124491个。利用基因本体(GO)和Mapman注释对CitrusNet的质量进行评价,验证了该网络的可靠性。此外,我们还计算了互作基因的表达差异(EDI利用RNA-seq数据对香橙4个组织进行RNA-seq分析,并对香橙的dna序列进行分析EDI在不同子网络中的分布和变化。
结论
CitrusNet的基因表达具有显著的模块化特征。TOR蛋白靶蛋白(Target of rapamycin, TOR)是激素信号亚网络的中心节点。所有的证据都支持TOR可以整合各种激素信号并影响植物生长的观点。CitrusNet为研究甜橙的生物学功能提供了宝贵的资源。
背景
蛋白质-蛋白质相互作用(PPIs)几乎参与了细胞过程的所有方面。了解蛋白质之间的相互作用是系统生物学的一个重要目标,这些知识可以为蛋白质功能和分子机制提供关键的见解。各种实验技术,如亲和纯化质谱(AP-MS) [1],酵母双杂交(Y2H)系统[2和蛋白质阵列[3.] - [5]已被应用于许多模式物种的全基因组PPIs检测,包括智人[6],黑腹果蝇[7],酿酒酵母[8),而秀丽隐杆线虫[9].然而,技术问题限制了上述一些高通量筛选方法的有效性。例如,迄今为止确定的PPI仅代表相关物种中完整PPI网络的一小部分。此外,用于确定PPIs的技术是劳动密集型和昂贵的,进一步限制了这些方法的应用。此外,大多数实验方法具有条件或方法特定的特征;因此,即使在同一物种内,用各种方法获得的数据有时也显示出最小的重叠。
为了克服现有实验方法的不足,扩大PPIs的覆盖范围,各种生物信息学方法被开发出来,包括基于同源和域的方法、基因邻接方法和基因融合方法[10] - [16].各种类型的数据被用来预测PPIs,包括蛋白质序列和功能,蛋白质结构,基因表达,等.已经建立了许多公共数据库来存储实验和理论预测的PPIs,包括相互作用蛋白质数据库(DIP) [17],分子相互作用资料库(MINT) [18],蛋白质相互作用数据库(完整)[19]、互作基因/蛋白质检索工具(STRING) [20.],生物交互数据集通用储存库(BioGRID) [21]及人类蛋白质参考数据库(HPRD) [22].
目前,植物物种蛋白质相互作用组的综合测定落后于模型动物的类似工作。利用Y2H, Dreze和他的同事们生成了一个高度可靠的二进制PPI网络拟南芥包含2,700个蛋白质间约6,200个相互作用[23].迄今为止,PPIs的计算预测主要集中在几种模式植物上,如答:芥[24] - [26),栽培稻[27].对多年生木本植物蛋白质互作体的研究缺乏,部分原因是对这些物种的遗传研究有限。
柑橘类在130多个国家和地区有商业种植,柑橘类水果在全球水果产业中占据主导地位(http://faostat.fao.org).尽管存在许多种质和栽培品种,但综合形态学、解剖学和分子生物学的证据已经证实,所有现代栽培品种(如甜橙)都是两个原始物种pummel(甜橙)的衍生后代。c .茅)和普通话(c .试) [28]、[29].基因组证据,包括大量的遗传信息柑橘类,已经证明甜橙是柚子和柑橘的天然杂交[28]、[30.].鲜橙作为果汁食用,含有大量维生素C等对人体健康有益的化合物;该物种占世界水果产量的60%以上(http://faostat.fao.org).因此,由于其经济和遗传的重要性,甜橙是一种重要的木本模式植物。这个物种的基因组草案为研究它的遗传学、生物化学和进化提供了一个前所未有的机会。30.,也为研究蛋白质-蛋白质相互作用提供了一个很好的机会柑橘类.
在本分析中,我们在精心准备和选择可靠的数据资源的基础上,采用基于正交和基于域的相互作用方法对甜橙PPI网络进行预测,然后采用k近邻(K-nearest neighbors, KNN)方法对预测的PPI网络进行验证和过滤,得到一个更有信心的网络。甜橙中最终的PPI网络CitrusNet包含8195个蛋白和124491个相互作用。这是首次报道木本植物的全基因组PPI网络。仔细评估了该PPIs网络的质量。此外,利用来自4个组织的RNA-seq数据,我们分析了重要的功能模块,并深入了解了甜橙蛋白互作体的组织特异性特征。
结果
PPI网络的拓扑性质
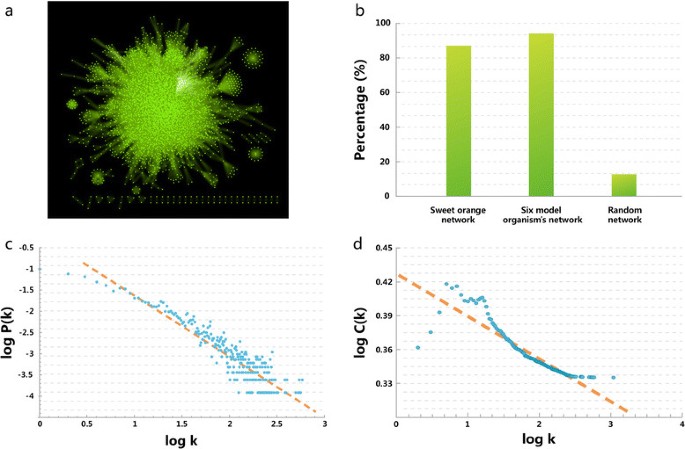
基于同源和域相结合的预测方法,我们获得了146056个PPIs,然后对所有预测的PPIs进行KNN评估和过滤。最终的CitrusNet包含8195个蛋白质和124,491个相互作用(图1a). KNN验证了85.2%的预测PPIs,与其他6种模式生物的PPIs验证率(93.3%)相似,远高于随机PPIs验证率(13.4%,图1b),说明CitrusNet的准确率很高。当我们使用Cytoscape可视化和分析CitrusNet的拓扑性质时[31,共8195个蛋白质中有7885个被连接到一个大的互联子网络中。拓扑性质表明CitrusNet是一个无标度网络,即。大多数节点的连接度较低,而少数集线器节点的连接度很高。CitrusNet中有602个蛋白有100多个连接。表格1列出了十大中枢节点,包括8种热休克相关蛋白,1种多泛素A蛋白和1种dna导向RNA聚合酶II亚基蛋白。这些数据表明热休克蛋白在柑橘互作体中起着重要的作用。通过回归方程拟合,柑橘网节点度服从幂律分布y= 4885.8 *x-1.407(右2= 0.851, P < 0.0001,图1c),x表示程度和y表示给定程度的节点数量。
具有高中间中心性的节点(BC,它计算通过一个节点的最短路径的分数)对网络也非常重要,因为即使在没有集线器的情况下,这些节点也会成为网络中的瓶颈[32].表格2列出了CitrusNet中BC最大的10个节点。其中5个节点也是10个轮毂节点中度最高的节点(Cs4g11150.3, Cs4g08220.1, Cs4g08220.3, Cs9g19220.1和Cs5g03150.1);其中四种是热休克相关蛋白,另一种是多泛素A蛋白。因此,所有这些蛋白在CitrusNet节点之间的连接和通信中发挥着关键作用。结合度和BC分析,我们推测热休克蛋白是甜橙PPI网络中最重要的蛋白。
CitrusNet的聚类系数为0.301,远高于同等度分布的随机网络的聚类系数(CC = 0.014)。这一特征表明CitrusNet是一个非常密集的网络,可以识别出多个包含蛋白复合物或信号通路的子网络。CitrusNet中CC与度的关系如图所示1d.从分布图中,我们观察到度小的节点分布密集,而度大的节点非常稀疏,进一步证实了CitrusNet的无标度特性。
柑桔网的质量评价
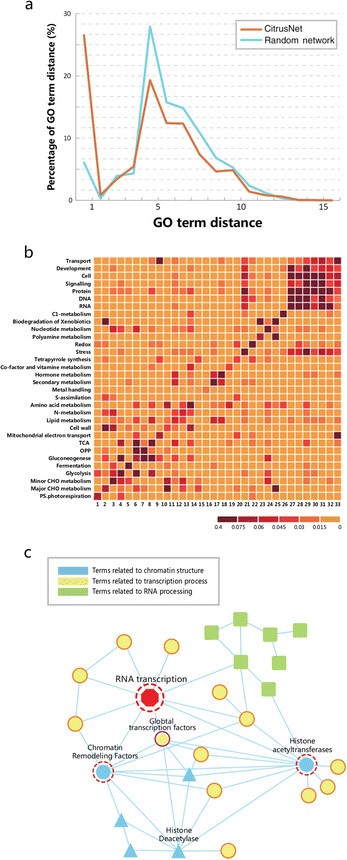
由于甜橙中没有实验性PPIs,我们采用基因本体(GO)注释分析对CitrusNet进行评价。因为相互作用的蛋白质往往具有相似或相关的功能,一个可靠的PPI网络应该比一个随机网络包含更多功能相关的蛋白质。我们进行GO分析,比较CitrusNet和随机网络中蛋白质节点之间的功能关系,然后从GO数据库中获得GO注释[33].甜橙中总共有27943个蛋白有GO注释,其中7327个(构成109409个相互作用)被纳入CitrusNet。为了进行这些比较,我们计算了CitrusNet和5个随机网络中蛋白质对GO项的最短距离。随机网络中蛋白质和蛋白质对的数量与CitrusNet相同,但值得注意的是,随机网络中蛋白质对没有相互作用。数字2a表明CitrusNet中GO项的最短距离远小于5个随机网络中的平均距离。
此外,我们分析了CitrusNet中的多个基因模块,以确定它们是否包含已知的甜橙生物通路。为此,我们使用Mapman注释,它表示不同术语之间没有冗余的生物功能[34].我们为CitrusNet构建了一个Mapman网络,并采用文献[]中描述的聚类算法[35]来提取相关模块。数字2b显示Mapman相关项相关度高,而没有功能关系的项相关度低。对RNA转录子网络的详细分析表明,PPIs提供了有用的生物学信息2c).在RNA转录子网络中,与RNA转录相关的术语与全局转录因子之间存在复杂的相互作用(图2c).此外,染色质重塑因子、组蛋白去乙酰化酶活性和组蛋白乙酰转移酶活性与RNA转录亚网络有许多联系,提示染色质重塑和组蛋白乙酰化/去乙酰化可能与转录过程密切相关。Mapman注释网络显示CitrusNet包含相当数量的生物模块,聚类算法成功地将之前分类为多体复合物的术语分组。
RNA-seq数据分析揭示了蛋白质相互作用网络的模块化特征
对基因表达与蛋白质相互作用的关系进行了全面分析[36]、[37].一般来说,编码相互作用蛋白的基因在mRNA表达水平上是相关的[38];因此,基因表达的相关性应反映预测的PPI网络的可靠性。基于甜橙4个组织(愈伤组织、叶片、花和果实)的RNA-seq数据,我们提出了一个相互作用蛋白对(EDI).具有相似表达水平的相互作用基因有EDI值接近于零。我们采用马尔可夫聚类(MCL)模块化方法[39]以从CitrusNet中提取相关模块。MCL是一种从构建的交互组中提取相关模块的聚类方法,在大多数情况下优于其他聚类方法[37].我们比较了EDI四个CitrusNet相关网络的分布,包括CitrusNet、一个随机网络、一个mcl -模块化网络和一个从酵母蛋白复合物推断出的网络[40].通过与酵母复合体序列比对,我们总共获得了159个甜橙蛋白复合体1).数字3.一个显示的是EDI这四种网络的分布。在蛋白质复合网络中,与基因的比例最高EDI在0左右,表明蛋白复合体具有最高比例的基因具有相似的表达水平。mcl -模块化网络表达相似基因的比例高于CitrusNet,而随机网络表达相似基因的比例最低EDI说明MCL算法提取相关模块的效率高,预测的CitrusNet是可靠的。目前,在甜橙中还没有实验测定的蛋白复合物,因此,本次分析中预测的159个蛋白复合物对相关研究非常有帮助。
的EDICitrusNet的分布和模块化特性。(一)EDICitrusNet、随机网络、mcl -模块化网络和蛋白复合物网络的分布。(b)EDI基于拓扑性质的柑橘网分布。红线表示低度节点,橙色线表示高度节点,黄色线表示高度节点,BC高,草绿色线表示高度节点,BC低。低度节点的百分比最高EDI值在零附近。(c)比较CitrusNet和随机网络之间的平均最短路径,基于去除不同的基因EDI变化值。(d)CitrusNet的子网络模块,由四个组织中相对高表达的基因组成。节点颜色表示不同的基因功能。橙色,合成;红色,DNA合成和染色质结构;蓝色、降解和泛素化;紫色,代谢物运输;草绿、细胞周期和细胞分裂;黄色,其他功能。
我们还分析了EDI基于其拓扑性质在柑橘网中的分布。首先,我们将网络中的所有基因分为两组(将度大于10的基因定义为高度基因,将其他基因定义为低度基因),然后将高度基因分为高BC和低BC两个亚组。数字3.B表示低度基因与EDI0左右的比例远高于高阶基因的比例,这是因为高阶基因有很多功能和表达水平不同的互作伙伴,而低阶基因有很少的互作伙伴,它们的功能和表达水平更可能相似。这一现象反映了基因相互作用的“经济”原则,即。在美国,基因倾向于与尽可能少的伴侣相互作用,以履行其生物功能。我们也观察到了相同的特征EDIBC拓扑参数。高度/低bc基因的百分比与EDI0左右高于高度/高BC基因,提示低BC基因可能具有相似的表达水平。
除了学习EDI在不同类型的子网络中,我们也分析了分布情况EDI四种组织中基因的变异。我们执行一个在网上分析模拟去除网络中特定节点的效果。我们定义了不同的阈值EDI变异取值范围为10到100,步长为1。基于这些阈值,我们移除具有的节点EDI变异值高于阈值,并在CitrusNet中随机删除相同数量的节点后,比较平均最短路径长度。两个节点之间的最短路径长度反映了整个网络的连通性。总共移除1658个节点EDI变异= 10,为117个节点EDI变异= 100。这两类交互体网络的平均最短路径长度分布如图所示3.的平均最短路径EDI变异-去除网络的长度均大于随机去除网络的长度,说明节点的高EDI变异值对于网络连接非常重要。数字3.C也表示节点与EDI变异10-20区间的值对网络的稳定性至关重要,删除这些节点会产生最大的最短路径长度。
最后,我们分析了CitrusNet基因表达的模块化特征。数字3.d说明了在至少三个组织中包含高表达基因的子网络(附加文件2).将功能相似的基因聚类成模块。例如,蛋白质降解和泛素相关的基因(蓝色),DNA合成和染色质结构相关的基因(红色),细胞周期和细胞分裂相关的基因(草绿色),蛋白质合成相关的基因(橙色),代谢物运输相关的基因(紫色),都聚集在功能群中。的EDI这些模块的基因值均小于不同功能的基因。因此,EDI值反映了相互作用的基因对之间的功能关系。综合分析结果表明,CitrusNet的基因表达模式具有模块化特征。
植物激素相关相互作用网络分析
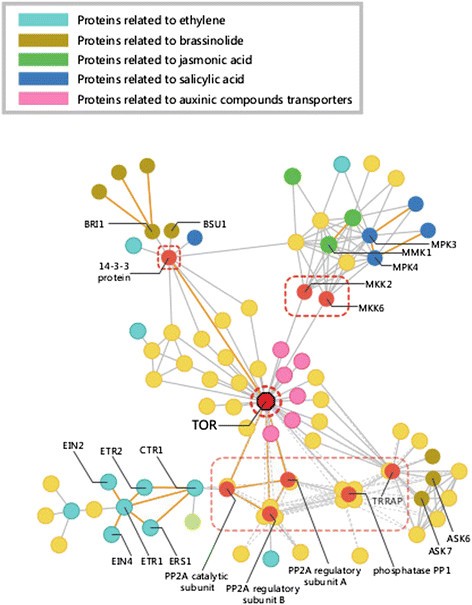
植物激素如乙烯、脱落酸、生长素、赤霉素、细胞分裂素和油菜素内酯等在植物生物学中发挥着重要作用。为了鉴定甜橙中重要的激素信号蛋白,我们构建了与激素信号通路相关的PPI子网络,信息来自拟南芥激素数据库(AHD2.0) [41].这些子网络通过过滤参与基本生物过程的蛋白质而简化。在激素信号亚网络中,乙烯、茉莉酸、水杨酸、生长素、油菜素内酯、细胞分裂素等相关蛋白形成了一个高度互联的网络,以雷帕霉素靶蛋白(TOR)为中心(图)4).在这个子网络中,TOR的BC最高,因此它是枢纽蛋白。TOR是一种保守蛋白,在酵母、人类和拟南芥[42]、[43].虽然TOR在植物中还没有得到广泛的研究,但有实验证据表明拟南芥研究表明,这种蛋白质是调控生长、抗逆性和mRNA翻译的枢纽[44].与激素信号和激素受体相关的蛋白质并不直接与TOR相互作用,而是通过其他连接蛋白与TOR相互作用。如图所示4,蛋白磷酸酶2A (protein phosphatase 2A, PP2A)调控亚基和催化亚基均与TOR有直接的相互作用,催化亚基还与raf样蛋白激酶CTR1相互作用,后者是乙烯反应通路的负调控因子拟南芥[45].此外,茉莉和水杨酸信号蛋白之间存在多种相互作用,这些相互作用大部分是由丝裂原活化蛋白激酶(MPK4, MMK1)介导的,其中许多还与丝裂原活化蛋白激酶激酶MKK2和MKK6相互作用,MKK6和MKK2直接与TOR相互作用。三种不同的连接途径将油菜素甾醇信号蛋白和TOR连接起来。蓬松相关蛋白iota/激酶eta具有相似的互作模式,均通过蛋白磷酸酶PP1和磷脂酰肌醇3-/4-激酶(TRRAP)与TOR互作。油菜素甾醇不敏感-1 (BRI1)及其下游蛋白BSU1通过14-3-3蛋白与TOR连接。6种生长素化合物转运体直接与TOR相互作用;因此,我们推测TOR调控生长素信号转导。
不同激素信号蛋白之间的联系非常复杂,因为几个相互作用的蛋白(PP2A、蛋白磷酸酶PP1、TRRAP、14-3-3蛋白和MAP激酶)之间有相当程度的连接。许多激素相关蛋白相互作用,表明各种激素信号通路在影响TOR之前交换信息(反之亦然)。虽然很多植物激素相关蛋白是相互连接的,但TOR无疑是激素相互对话的枢纽。TOR的中心作用是整合激素传递的环境信息,并利用这些信息调控植物的生长。
讨论
在本研究中,我们利用同源鉴定和区域组合方法预测了甜橙的全基因组PPI网络。由于在甜橙中没有黄金标准的PPI数据集,我们使用了一个高度精确的KNN算法来过滤预测的相互作用。合成的PPI网络CitrusNet包含8195个蛋白,相互作用124491个。然后我们使用GO和Mapman注释来评估预测的网络。CitrusNet中GO项的最短距离远小于随机网络,MapMan生物学网络显示CitrusNet中的基因模块反映了甜橙中已知的生物通路。我们还利用酵母蛋白复合物同源物预测了甜橙中的159个蛋白复合物,并利用它们来评估CitrusNet;结果表明,蛋白质复合物具有相对紧密的连接。总之,这些分析证实CitrusNet是高度可靠的。
基于CitrusNet中的连接,分析了PPI网络的模块化特点。我们提出了EDI指数分析基因表达与蛋白质互作的关系。在citrusnet衍生的子网络中,具有相似基因表达水平的蛋白节点的比例远远高于随机网络,尤其是在蛋白复合物和mcl模块网络中。蛋白质复合物是由两个或多个相关的多肽链组成的一组,预计这些多肽链具有紧密的网络连接,并由表达水平相似的基因编码。的分析EDI基于拓扑性质的分布揭示了基因与尽可能少的伴侣相互作用(“经济”原则)。此外,EDI0附近的值与度、BC等拓扑参数呈负相关。除了分析EDI分布,我们也进行了比较EDI四种组织间变异,发现基因与低EDI变异值对柑桔网的稳定性有重要影响。
蛋白质相互作用在许多不同的信号转导过程中发挥核心作用,包括蛋白质结构转化,蛋白质磷酸化和去磷酸化,以及激素信号处理。许多受激素信号调节的基因和生物过程已被确定[46,越来越多的研究集中在不同激素信号之间的相互影响[47] - [49].我们构建了一个与激素信号蛋白相关的PPI子网络,观察到TOR是激素互扰的中心枢纽。一般来说,TOR通过调节蛋白质翻译、RNA转录和蛋白质降解等基本过程来控制细胞生长[50]、[51].激素信号通过接力蛋白传递,最终影响TOR活性。虽然TOR在酵母,人类和拟南芥,其在激素信号通路中的功能尚未被研究。虽然我们确定了TOR和激素信号蛋白之间的可靠联系,但其潜在的调节模式尚未被阐明。CitrusNet中许多预测的植物激素相互作用都得到了其他物种的实验证据的支持[44]、[50]、[51].例如,PP2A可以下调TOR的活性[52],而PP2A与另一个相互作用蛋白CTR1之间的关系尚未被报道。考虑到乙烯对植物生长具有负向调控作用,我们推测CTR1可以下调PP2A催化亚基的活性。既往研究表明14-3-3蛋白在油菜素内酯信号通路中发挥重要作用[53];然而,14-3-3蛋白与BRI1相互作用的生物学意义尚不清楚。此外,14-3-3蛋白对TOR有正向影响[54];因此,14-3-3蛋白可能与BRI1有调控关系。TOR与其他植物激素相关蛋白的关系尚未见报道。我们对全基因组PPI网络的分析说明了TOR在激素信号通路中的核心作用。在未来,CitrusNet将为甜橙的功能基因组学和系统生物学研究提供新的思路。
结论
本研究利用同源识别和域组合方法对甜橙的全基因组PPI网络进行预测,然后采用高精度的KNN算法对预测的相互作用进行过滤。最终的PPI网络包含8195个蛋白质和124491个相互作用。我们使用GO和Mapman注释来评估预测的网络。我们进一步利用酵母蛋白复合物同源物预测了甜橙中的159个蛋白复合物,并利用它们来评估CitrusNet。我们最终构建了一个与激素信号蛋白相关的PPI子网络,发现TOR是激素串扰的中心枢纽。CitrusNet为甜橙的蛋白质-蛋白质相互作用提供了宝贵的资源。
方法
数据源
甜橙基因组完整序列、注释、转录数据来源于甜橙基因组注释项目[30.].RNA-seq数据来自四个不同的组织:花,叶,果实和愈伤组织。6个参考模型生物的高质量实验PPI数据,拟南芥,酿酒酵母,秀丽隐杆线虫,D. melanogaster, H. sapiens而且亩,是从公共数据库BIOGRID [21完好无损,19], [17), STRING [20.)、薄荷(18], HPRD [22和TAIR [55,为了确保数据的可靠性,我们进行了验证。利用BLAST将来自不同PPI数据库的冗余序列分配到唯一的识别号,参数如下:identity = 100%, coverage >99%。最终数据在附加文件中提供3..
预测PPIs的方法
直接同源识别
在全基因组统计趋势中,同源基因是不同物种中最相似的基因,通常被认为保持相同的功能[56].因此,如果两个同源蛋白在一个物种中相互作用,它们很容易在其他物种中相互作用。我们使用Inparanoid(独立版本4.1)[57].在这个分析中,答:芥比其他五种生物都要高。首先,确定甜橙与6个参考基因组之间的所有潜在同源基因,然后将不同生物体中的潜在同源基因分组在一起。偏执型得分归一化在0-1范围内,保留保守基因的最大阈值较高(4600)。如果一个蛋白在一个参考基因组中与多个同源蛋白相对应,得分最高的同源蛋白被保留。对于甜橙中的任意两个蛋白质,如果它们在六个参考基因组中的同源物至少有一个实验验证的相互作用,则预测这两个蛋白质会相互作用。为了对候选蛋白质相互作用进行排序,定义了一个归一化分数:
在方程(1),InScore一个而且InScoreB表示相互作用的蛋白质对的标准化Inparanoid评分(一个而且B)在一个参照有机体中。一个蛋白质对的最终得分是6个参考基因组得分的总和。根据评分分布规律,将阈值设置为0.15。
Domain-combination方法
结构域是蛋白质的基本功能单位,参与相关蛋白质之间的分子间相互作用。如果确信两个蛋白质的结构域会相互作用,那么就可以预测这些蛋白质本身会相互作用[11]、[58]、[59].因此,可以从它们的域-域相互作用(ddi)推断出PPIs。Han和他的同事认为,PPIs可能是一组域或多个域相互作用的结果;因此,我们采用他们的方法获得了基于ddi的精确PPI网络[11].我们提出了域组合和域组合对(dc-pair)的概念。为了克服传统领域预测方法的局限性,我们的模型框架将dc-对作为PPIs的基本单元。我们建立了以下概率模型来预测蛋白质对的相互作用概率。利用Hmmer工具对甜橙蛋白的结构域进行了鉴定[60],以搜寻Pfam资料库(包括PfamA及PfamB) [61].Hmmer的阈值设为1e-3,多结构域蛋白的结构域比对覆盖率设为>0.6,单结构域蛋白的结构域比对覆盖率设为>0.9。
假设有一对蛋白质一个而且B包含米而且n域,分别。然后,蛋白质一个而且B有2个米1和2n-1种可能的定义域组合米1)×(2n-1)得到dc对。每对dc-pair的评分定义如下:
使用方程(2)时,计算6种模式生物的dc-pair评分。考虑到一个dc-对可能会出现在许多不同的得分不同的蛋白质对中,每个dc-对选择得分最高的。在得到所有dc-对的得分后,我们计算甜橙中潜在的相互作用蛋白对的得分。每对蛋白质的最终得分(一个而且B)的定义如下。
在方程(3.),rated-dc-pairs表示蛋白质对的dc-pairs一个而且B在六个参考生物中all-dc-pairs表示所有dc-对蛋白质一个而且B.方程式的最终分数(3.)表示蛋白质对(一个而且B,由域组合法得到。为了获得可信的结果,设置了适当的阈值。
PPI数据的过滤和评估方法
由于没有甜橙的实验性PPI数据集,我们采用第三方交叉验证方法,基于同源识别和域组合对预测的PPI进行过滤和评估。k -最近邻(KNN)是一种验证预测PPIs的简单而准确的方法,需要正训练数据集和负训练数据集[62].在本分析中,阳性样本通过实验确定了6个参照生物中的PPIs,这些参照生物被分为5个相等的组。相应的阴性样本从甜橙中随机抽取蛋白质对,阳性样本中排除真实或相关的蛋白质对(Pearson相关性>0.3)。在阳性样本集和阴性样本集中,五组每组的蛋白质对数量相同。通过5个独立的训练数据集对预测结果进行检验,并保留至少2个数据集支持的预测交互。为了比较PPI网络与随机网络的差异,我们还对随机网络进行了此过程。
处理RNA-seq数据的方法
RNA-seq读取由Illumina和SOLiD平台生成,使用TopHat (v1.2.0)和BioScope (v1.3)与参考基因组进行对齐,并使用默认参数[63].每个RNA-seq文库的基因表达水平计算为每千碱基外显子读取量模型每百万映射读取量(RPKM) [64].在层次聚类分析中,我们使用聚类软件Cluster 3.0对基因进行完全连锁层次聚类,以非中心Pearson相关性为距离测度。假设基因一个与之交互米基因,我们定义了相互作用基因的表达差异(EDI)如下。
在方程(4),RPKM一个而且RPKM我表示基因的RNA-seq表达值一个以及它的互动伙伴我,D一个而且D我表明基因的程度一个和基因我.如果基因一个有EDI值接近于零,表示基因的表达水平一个它的互动伙伴也是类似的。
在获得EDI,我们计算EDI变异四个组织中每个基因的值;愈伤组织,叶,花,果。的EDI变异值定义为式(5).
EDI最高而且EDI最低代表最高和最低EDI在四个组织中分别有一个基因。如果一个基因没有在一个组织中表达,我们定义EDI最低为零。
附加文件
缩写
- PPI:
-
蛋白质相互作用
- 资讯:
-
再邻居
- 走:
-
基因本体论
- 艾迪:
-
互作基因表达差异
- TOR:
-
雷帕霉素靶
- AP-MS:
-
亲和纯化质谱法
- Y2H:
-
酵母2台混合动力
- 公元前:
-
中间性中心
- 答:
-
聚类系数
- ddi:
-
Domain-domain交互
参考文献
黄晓燕,刘志强,刘志强,等:拟南芥聚囊藻PCC6803脂肪酸生物合成酶的亲和纯化。中国生物医学工程学报,2006,26(5):357 - 361。
病因B, Davies B:分析酵母双杂交系统的蛋白质-蛋白质相互作用。植物学报,2002,30(3):393 - 397。
Angenendt P, Kreutzberger J, Glokler J, Hoheisel JD:通过未纯化PCR产物的无细胞原位表达生成高密度蛋白芯片。蛋白质组学。2006,5:1658-1666。
Popescu SC, Popescu GV, Bachan S, Zhang Z, Seay M, Gerstein M, Snyder M, Dinesh-Kumar SP:通过高密度拟南芥蛋白微阵列揭示钙调素相关蛋白与靶蛋白的差异结合。中国科学:地球科学,2007,29(3):349 - 356。
刘毅,刘志强,刘志强,刘志勇:自组装蛋白微阵列。科学通报。2004,34(5):393 - 393。
Rual JF, Venkatesan K, Hao T, Hirozane-Kishikawa T, Dricot A, Li N, Berriz GF, Gibbons FD, Dreze M, Ayivi-Guedehoussou N, Klitgord N, Simon C, Boxem M, Milstein S, Rosenberg J, Goldberg DS, Zhang LV, Wong SL, Franklin G, Li S, Albala JS, Lim J, Fraughton C, Llamosas E, Cevik S, Bex C, Lamesch P, Sikorski RS, Vandenhaute J, Zoghbi HY,等:构建蛋白质组尺度的人类蛋白质-蛋白质相互作用网络图谱。《自然》,2005,437:1173-1178。
Giot L, Bader JS, Brouwer C, Chaudhuri A, Kuang B, Li Y, Hao YL, Ooi CE, Godwin B, Vitols E, Vijayadamodar G, Pochart P, Machineni H, Welsh M, Kong Y, Zerhusen B, Malcolm R, Varrone Z, Collis A, Minto M, Burgess S, McDaniel L, Stimpson E, Spriggs F, Williams J, Neurath K, Ioime N, Agee M, Voss E, Furtak K,等:黑果果蝇的蛋白质相互作用图谱。科学。2003,302:1727-1736。
Uetz P、Giot L、Cagney G、Mansfield TA、Judson RS、Knight JR、Lockshon D、Narayan V、Srinivasan M、Pochart P、Qureshi-Emili A、Li Y、Godwin B、Conover D、vibfleisch T、Vijayadamodar G、Yang M、Johnston M、Fields S、Rothberg JM:酵母蛋白质-蛋白质相互作用的综合分析。自然。2000,403:623-627。
Walhout AJM, Sordella R, Lu X, Hartley JL, Temple GF, Brasch MA, Thierry-Mieg N, Vidal M:利用参与外阴发育的蛋白质来绘制秀丽隐杆线虫的蛋白质相互作用图谱。科学通报。2000,29(4):416 - 422。
Enright AJ, Iliopoulos I, Kyrpides NC, Ouzounis CA:基于基因融合事件的全基因组蛋白质相互作用图。自然。1999,402:86-90。
Han DS, Kim HS, Jang WH, Lee SD, Suh JK: PreSPI:基于结构域组合的蛋白质-蛋白质相互作用预测系统。中国生物医学工程学报,2004,24(3):323 - 329。
黄tw, Tien AC, Huang WS, Lee YC, Peng CL, Tseng HH, Kao CY, Huang CY: POINT:基于同源互作体的蛋白质-蛋白质相互作用预测数据库。生物信息学。2004,20:3273-3276。
李萨,陈昌昌,蔡昌昌,赖建明,王凤飞,高颖华,黄颖华:基于同源蛋白的蛋白质相互作用预测及其在物种间相互作用中的应用。生物信息学。2008,9(增刊12):S11-
Tsoka S, Ouzounis CA:蛋白质相互作用的预测:代谢酶经常参与基因融合。王志军。2000,26:141-142。
预测蛋白质相互作用的计算方法。中国生物医学工程学报,2002,12:368-373。
Wojcik J, Schächter V:使用相互作用域配置文件对的蛋白质-蛋白质相互作用图谱推断。生物信息学。2001,17:S296-S305。
李志强,段晓军,李志强,李志强,李志强。蛋白质相互作用的蛋白质数据库:一种研究蛋白质相互作用细胞网络的工具。中国生物医学工程学报,2002,30:303-305。
Licata L、Briganti L、Peluso D、Perfetto L、Iannuccelli M、Galeota E、Sacco F、Palma A、Nardozza AP、Santonico E、Castagnoli L、Cesareni G: MINT,分子相互作用数据库:2012年更新。中国生物医学工程学报,2012,30(3):393 - 393。
Kerrien S, Aranda B, Breuza L, Bridge A, brokes - carter F, Chen C, Duesbury M, Dumousseau M, Feuermann M, Hinz U, Jandrasits C, Jimenez RC, Khadake J, Mahadevan U, Masson P, Pedruzzi I, Pfeiffenberger E, Porras P, Raghunath A, Roechert B, Orchard S, Hermjakob H: 2012年完整分子相互作用数据库。中国生物医学工程学报,2012,30(3):393 - 393。
简森·李、库恩·M、斯塔克·M、Chaffron S、Creevey C、Muller J、Doerks T、Julien P、Roth A、Simonovic M、Bork P、von Mering C: STRING 8 - 630种生物中蛋白质及其功能相互作用的全局研究。中国生物医学工程学报,2009,30(3):412- 416。
王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰,王晓峰。中国生物医学工程学报,2011,29(3):393 - 393。
Goel R, Harsha HC, Pandey A, Prasad TS:人类蛋白质参考数据库和人类蛋白质百科作为磷蛋白组分析资源。生物学报,2012,31(3):393 - 393。
刘志强,刘志强,刘志强,刘志强,刘志强。拟南芥网络进化的证据及其在拟南芥中的应用。科学。2011,333:601-607。
Brandao MM, Dantas LL, Silva-Filho MC: AtPIN:拟南芥蛋白相互作用网络。生物信息学。2009,10:454-
张志刚,张志强,张志强,等:拟南芥与植物间相互作用体的关系。植物生理学报。2007,34(3):317-329。
李鹏,臧伟,李勇,徐峰,王杰,史涛:拟南芥层次功能蛋白互作网络总体接口及分析平台。中国生物医学工程学报,2011,30(3):393 - 393。
朱鹏,顾辉,焦艳,黄东,陈明:基于预测互作体网络的水稻蛋白质-蛋白质相互作用的计算识别。生物信息学与蛋白质组学。2011,9:128-137。
程勇,de Vicente MC,孟H,郭伟,陶楠,邓欣:一套用于柑橘及其相关属叶绿体DNA多样性分析的引物。植物学报,2005,25:661-672。
潘德萨A,施韦泽D,葛拉M:甜橙的细胞学杂合性和杂种起源[j]。等)。中国科学:地球科学,2000,30(3):361-367。
徐强,陈丽丽,阮欣,陈东,朱安,陈晨,Bertrand D,焦文斌,郝波黑,Lyon MP,陈俊杰,高松,邢峰,兰华,常景文,葛晓华,雷勇,胡强,苗洋,王磊,肖世祥,Biswas MK,曾文峰,郭峰,曹海斌,杨晓明,徐晓伟,程文杰,徐健,刘建华,等:甜橙(Citrus sinensis)基因组草图。王志军。2013,45:59-66。
Cytoscape:可视化和分析生物网络的软件。方法生物医学杂志,2011,696:291-303。
Joy MP, Brock A, Ingber DE, Huang S:酵母蛋白相互作用网络中的高间质蛋白。生物医学与生物技术。2005,2005:96-103。
Harris MA、Clark J、Ireland A、Lomax J、Ashburner M、Foulger R、Eilbeck K、Lewis S、Marshall B、Mungall C、Richter J、Rubin GM、Blake JA、Bult C、Dolan M、Drabkin H、Eppig JT、Hill DP、Ni L、Ringwald M、Balakrishnan R、Cherry JM、Christie KR、Costanzo MC、Dwight SS、Engel S、Fisk DG、Hirschman JE、Hong EL、Nash RS等:基因本体(GO)数据库和信息学资源。中国生物医学工程学报,2004,32:D258-D261。
Thimm O, Blasing O, Gibon Y, Nagel A, Meyer S, Kruger P, Selbig J, Muller LA, Rhee SY, Stitt M: MAPMAN:一个用户驱动的工具,将基因组数据集显示到代谢途径和其他生物过程的图表上。植物学报,2004,24(3):393 - 393。
Li Y, Agarwal P, Rajagopalan D:全球通路相声网络。生物信息学。2008,24:1442-1447。
Jansen R, Greenbaum D, Gerstein M:将全基因组表达数据与蛋白质-蛋白质相互作用联系起来。中国生物医学工程学报,2002,32(3):329 - 336。
王海燕,张晓燕,张志强:从蛋白质相互作用和基因表达数据中发现分子途径。生物信息学。2003,19(增刊1):i264-i271。
与蛋白质相互作用网络和基因表达相关的功能模块。中国生物医学工程学报,2003,29(3):393 - 393。
蛋白质-蛋白质相互作用网络聚类算法的评价。生物信息学。2006,7:488-
张志强,王志强,张志强,张志强。酵母蛋白复合体的研究进展。中国生物医学工程学报,2009,37:825-831。
蒋智,刘旭,彭智,万勇,季勇,何伟,万伟,罗杰,郭洪:AHD2.0:拟南芥植物系统研究激素数据库的更新版本。中国生物医学工程学报,2011,29(3):393 - 393。
Diaz-Troya S, Perez-Perez ME, Florencio FJ, Crespo JL: TOR在酵母、植物和哺乳动物自噬调节中的作用。自噬。2008,4:851-865。
Raught B, Gingras AC, Sonenberg N:雷帕霉素(TOR)蛋白的靶标。中国科学:地球科学,2001,32(3):349 - 356。
Deprost D, Yao L, Sormani R, Moreau M, Leterreux G, Nicolai M, Bedu M, Robaglia C, Meyer C:拟南芥TOR激酶与植株生长、产量、抗逆性和mRNA翻译有关。环境与经济研究,2007,8:864-870。
Kieber JJ, Rothenberg M, Roman G, Feldmann KA, Ecker JR: CTR1,拟南芥乙烯反应途径的负调控因子,编码蛋白质激酶raf家族的一个成员。细胞。1993,72:427-441。
张建平,刘志刚:植物激素信号转导的研究进展。自然。2009,459:1071-1078。
Gazzarrini S, McCourt P:植物激素信号传递中的交叉对话:拟南芥突变体告诉我们什么。机器人。2003,91:605-612。
Ludwig AA, Romeis T, Jones JD: cdpk介导的信号通路:特异性和串扰。中国生物医学工程学报,2004,24(3):391 - 396。
Spoel SH, Dong X:植物免疫反应中激素相互作用的意义。微生物学报。2008,3:348-351。
Dennis PB, Fumagalli S, Thomas G:雷帕霉素的靶点(TOR):平衡蛋白质合成和降解的反作用力。中国科学:地球科学,1999,9:49-54。
雷帕霉素靶标(TOR):营养和生长因子信号的整合者,细胞生长和细胞周期进程的协调者。肿瘤基因。2004,23:3151-3171。
Selvaraj A, Thomas G:磷酸酶2A阻断mTORC1营养信号。中国科学:地球科学,2010。
张慧玲,刘志强,刘志强,刘志强,刘志强。拟南芥亲和纯化14-3-3蛋白复合物的蛋白质组学分析。蛋白质组学。2009,9:2967-2985。
郑晓峰,曾昌,郑晓峰,郑晓峰,郑晓峰。14-3-3蛋白正调控rapamycin敏感信号转导。中国生物医学工程学报,1998,8:1259-1267。
李D、李D、Swarbreck D、Wilks C、Sasidharan R、Muller R、Dreher K、Alexander DL、Garcia-Hernandez M、Karthikeyan AS、Lee CH、Nelson WD、Ploetz L、Singh S、Wensel A、Huala E:拟南芥信息资源(TAIR):改进的基因注释和新工具。中国生物医学工程学报,2012,30(3):393 - 393。
Gabaldón T, Koonin EV:基因同源的功能和进化意义。王文杰。2013,14:360-366。
Ostlund G, Schmitt T, Forslund K, Kostler T, Messina DN, Roopra S, Frings O, Sonnhammer EL: InParanoid 7:用于真核同源分析的新算法和工具。中国生物医学工程学报,2010,29(3):353 - 356。
韩东,金红辉,徐建军,张伟:基于结构域组合的蛋白质相互作用预测的概率框架。基因工程学报,2003,14:250- 257。
Ta HX, Holm L:蛋白质相互作用预测的不同结构域方法的评价。中国生物医学工程学报,2009,35(3):357-362。
Finn RD, Clements J, Eddy SR: HMMER web服务器:交互式序列相似度搜索。中国生物医学工程学报,2011,39:W29-W37。
Finn RD, Tate J, Mistry J, Coggill PC, Sammut SJ, Hotz HR, Ceric G, Forslund K, Eddy SR, Sonnhammer EL, Bateman A: The Pfam蛋白家族数据库。中国生物医学工程学报,2008,29(3):353 - 356。
Guarracino M, Nebbia A:用k近邻分类算法预测蛋白质-蛋白质相互作用。2010.
Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR, Pimentel H, Salzberg SL, Rinn JL, Pachter L: TopHat和Cufflinks RNA-seq实验的差异基因和转录本表达分析。中国生物医学工程学报,2012,7:562-578。
Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B: RNA-Seq定位和定量哺乳动物转录组。物理学报,2008,5:621-628。
确认
本研究由国家自然科学基金项目(31271968、31271406)、高校新世纪优秀人才计划项目(NCET-13-0807)和中央高校基本科研业务费项目(2013PY004、2014PY018)资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明他们之间没有利益冲突。
作者的贡献
DYD和CJW收集数据,进行分析,撰写初稿。CLL和CYJ指导项目并撰写稿件。GJ和CDJ建立了网络服务器并帮助收集数据。LS帮助准备了这些数据。XQ和DXX提供了测序资料,并对稿件进行了修改。所有作者仔细阅读并通过了最终稿。
电子辅料
12870 _2014_213_moesm1_esm.xls
附加文件1:甜橙中159个蛋白质复合物的预测,使用酵母复合物的最佳序列比对。(XLS 974 KB)
12870 _2014_213_moesm2_esm.xls
附加文件2:基因在四个组织中至少三个高度表达的基因;愈伤组织,花,果,叶。(XLS 201 KB)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇根据知识共享署名许可条款发布的开放获取文章(http://creativecommons.org/licenses/by/2.0),允许不受限制地在任何媒体上使用、分发和复制,前提是正确地注明原创作品。
关于这篇文章
引用这篇文章
丁永德,张继伟。郭,J。et al。甜橙蛋白-蛋白相互作用网络的预测与功能分析。BMC植物杂志14日,213(2014)。https://doi.org/10.1186/s12870-014-0213-7
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-014-0213-7
关键字
- 蛋白质相互作用
- 直接同源
- 域
- 模块化
- 植物激素