摘要
背景
等位基因变异和连锁不平衡的全基因组模式的特征可以用来检测可靠的表型-基因型关联和分子选择的特征。然而,使用胡麻属indicum由于缺乏多态性数据,育种的种质资源受到限制。
结果
在这里,我们描述了来自12个国家的29个芝麻菌株的大规模并行重测序,每个测试样本的覆盖深度≥13倍。每个样本平均检测到127347个SNPs, 17961个小InDels和9266个结构变异。种群SNP率、种群多样性(π)和分离位点的Watterson估计量(θw)估计为8.6 × 1032.5 × 1033.0 × 103英国石油公司-1,分别。这些snp中,23.2%位于编码区内。多态性模式在基因家族中是非随机的,与生物或非生物环境相互作用的基因表现出高水平的多态性。连锁不平衡(LD)衰减距离估计为150kb,在种群中未观察到明显的结构。29个芝麻品系之间的系统发育关系符合芝麻起源于印度次大陆的假设。此外,我们还提出了腺苷酸异戊烯基转移酶(ITP)基因通过介导玉米素生物合成来决定芝麻叶腋花数的新作用。
结论
该研究首次报道了芝麻遗传变异的全基因组模式。这里描述的高LD距离和丰富的多态性增加了我们对影响芝麻全群体序列变异的力量的理解,这将是未来基因-表型和全基因组关联研究(GWAS)的宝贵资源。
背景
胡麻属indicum(芝麻)是一种古老的作物,基因组大小约为357 Mb,含有高浓度的油脂和蛋白质,具有药用价值。然而,这种植物容易遭受内涝,特别容易受到许多真菌和细菌疾病的影响,如茎腐和根腐病,镰刀菌素枯萎病,白粉病等。这些生物和非生物胁迫会导致总体产量下降,产量与生长条件密切相关。为了克服环境压力和提高产量,植物育种项目需要丰富的种质资源和遗传信息[1],等位基因变异和连锁不平衡的全基因组模式的表征确保了检测可靠的表型-基因型关联和分子选择的特征[2].印度、中国和韩国是芝麻种质收集、保存和研究的领先国家[3.].在中国,约6000株芝麻已存放在中国国家基因库(武汉,中期基因库;北京,长期资源库)。在韩国,位于韩国水原的农村振兴厅(RDA)的基因库中保存了> 7,698个变体[4],在印度,国家植物遗传资源局(印度新德里)已存档了> 1万个变体。然而,由于基因组信息的缺乏和合适的生物标记物的缺乏,在全基因组尺度上对芝麻种质资源遗传多样性的研究较少。1]、[5] - [7].
芝麻是该属中最常见的品种胡麻属,园内有超过20种开花植物。与芝麻不同的是,该属的大多数物种都没有被驯化,在多倍体水平上存在显著差异[1].由于这些野生物种大多原产于撒哈拉以南的非洲,芝麻最初被认为起源于非洲;然而,驯化的芝麻被证明起源于印度次大陆。8]、[9].由于缺乏多个芝麻品系的详细分子数据,对芝麻进化的进一步研究受到了阻碍。
参考基因组的完成为探索野生和驯化芝麻的遗传变异提供了必要的资源美国indicum(http://www.ocri-genomics.org/Sinbase/).我们分析了来自12个国家的29个重测序芝麻品系,覆盖≥13倍。根据这些数据,我们构建了首个芝麻单倍型图谱,为了解芝麻跨多品系的遗传多样性提供了依据。这些数据可用于全基因组关联研究的发展,进而促进与简单和复杂性状相关的基因的定位。
结果与讨论
重测序芝麻品系的表型多样性
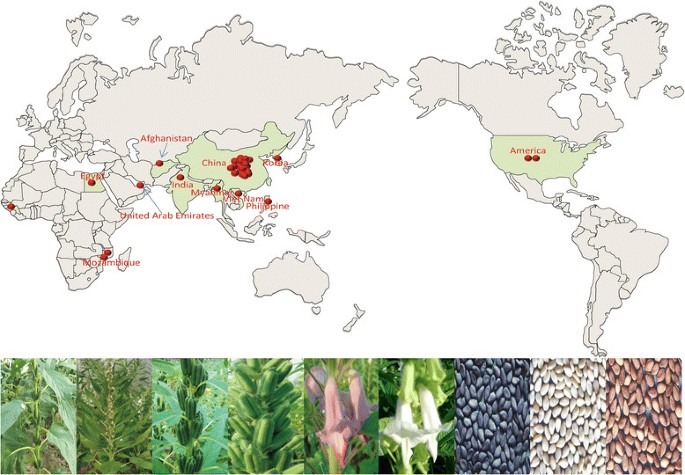
我们手工选择了29个芝麻品系进行基因组重测序,其中6个来自印度和非洲,16个来自中国,2个来自美国,1个来自阿富汗、阿拉伯联合酋长国、韩国、缅甸、菲律宾和越南1:数据S1)。这些品系表现出广泛的表型,包括确定和不确定的生长习性、株高和株高的高低、开花的早和晚、种皮颜色的不同、每叶腋开一朵花和三朵花、单株和分枝风格等。遥远的地理关系和广泛的表型变异使这些菌株成为探索栽培芝麻遗传多样性的理想模型(图1).
芝麻品种遗传多样性景观研究
为了识别大规模的多态性,更好地了解芝麻种质的遗传结构,对29个芝麻品系进行了重新测序,每个品系的覆盖深度≥13×,过滤后的数据超过120gb(附加文件2:图S1;额外的文件1:数据S1)。所有的序列都与“中子13号”的参考基因组进行比对,该基因组的有效长度为274 Mb (http://www.ocri-genomics.org/Sinbase/),使用BWA软件[10].不同品系间的映射率在88.8% ~ 95.2%之间,平均为91.4%。映射结果与GATK软件的映射结果一致1:数据S1)。
使用严格的流水线,我们使用SAMtools软件平均识别出每个菌株127,347个单核苷酸多态性(SNPs) [11],范围为40,925至392,5441;额外的文件2:图S2)。总体而言,SNP率在1.5 × 10之间4到14.3 × 104,其中G:A, A:G, C:T和T:C替换是最常见的(附加文件2:图S3)。通过组合所有菌株的SNP,我们鉴定出2,348,008个独特SNP,群体SNP率为8.6 × 103英国石油公司-1.接下来我们使用GATK软件调用并计算SNP种群,结果共生成2,003,821个种群SNP。GATK和SAMtools的符合率在80.0% ~ 89.9%之间,16个连锁组(假分子染色体)的符合率平均为85.4%1:数据S2)。Sanger测序结果显示,SNP呼叫准确率约为93.7%2:图S4;额外的文件1:数据S3)。这些结果表明,这里检测到的大多数snp是可靠的。在这些snp中,25.1%位于基因编码区,1.5%、7.5%和1.7%分别位于5 '非翻译区(UTR)、编码序列(CDS)和3'UTR2:图S5;额外的文件1:数据S4)。分别来自印度、缅甸和阿联酋的菌株20、24和26的SNP阳性率居前三;因此,这些地区可能蕴藏着更多样化的芝麻种质资源。
尽管芝麻传统上被认为是自花授粉的植物,但它也受到蝴蝶和蜜蜂等昆虫的交叉授粉。这与观察到的杂合度为12.37 ~ 49.30%,平均25.39%的杂合度率相一致2:图S6;额外的文件1:数据S5)。16个中国品种中有5个(品系1 ~ 5)杂合子率低于平均值(16.82 ~ 23.25%)和其他非中国品系。菌株16、22和26异常高的杂合子率表明这些菌株对昆虫更有吸引力;然而,更琐碎的解释,如测序和对齐错误,不能排除,特别是在重复区域。
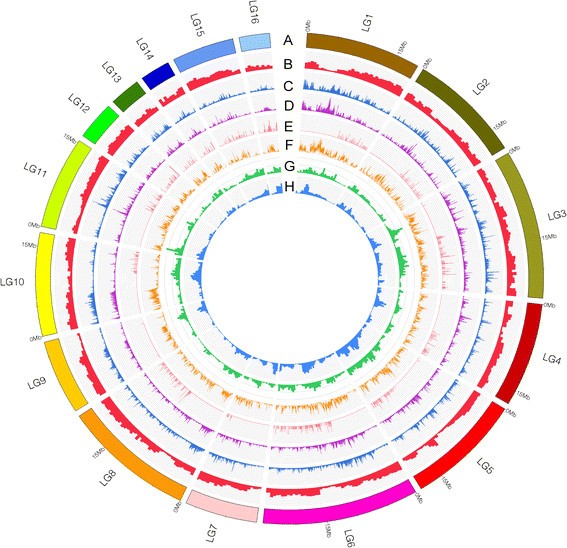
种群snp用于计算两个常用的种群遗传统计数据:种群多样性(π)和分离位点的Watterson估计(θw).平均π而且θw所有29个品系的值分别为2.5和3.0 / kb,低于水稻[12但比鹰嘴豆高(中投arietinum)[13)、西瓜(Citrullus lanatus)[14和大豆[15)(附加文件1:数据S6)。我们观察到许多连续滑动窗口伴随着假分子(LG1-LG16),这些假分子包含的snp比正常snp少,反过来也更低π而且θw值(图2),表明遗传多样性沿芝麻伪分子分布不均匀。我们检查了基因组中最高和最低的多态区域(位于顶部和底部5%的块)π值),并发现最高多态区域的基因数量小于最低区域(524 vs. 1308)(附加文件1:资料S7和S8),类似于其他物种,如鹰嘴豆[13].在最高多态区域的许多基因与环境适应性有关,包括应激反应途径1:数据S9)。这些基因可能为研究芝麻的生物和非生物胁迫提供有价值的资源。值得注意的是,尽管在最低多态性区域的基因数量更多,但只有5个基因在两个基因本体(GO)术语中富集,所有这些基因都与基本生物功能有关;即核糖体结合(附加文件1:数据S10)。
接下来,我们使用满足所有对端要求的映射读数,但在contig的一端包含对齐间隙,以检测每个菌株的短InDels (1 - 5 bp)。检测到的InDel的总数与InDel(附加文件)的长度成反比2:图S7)。在所有29个品系中鉴定出的数量从4,495 - 56,594(平均为17,961),总共有520,880个独特的InDels1:数据S11)。与SNP相似,InDels在基因组中的分布并不均匀,高密度区域与SNP高密度区域密切相关(图2).在这些indel中,插入和删除的数量相似(分别为48.8%和51.2%)。纯合子内嵌子的比例是杂合子内嵌子的1.5倍以上。其中71.7%位于基因间区,1.5%(8221)位于CDS区,5.0%位于utr区。
结构变异(SV)最初定义为插入、删除、DNA倒置和其他大于1kb大小的序列重排[16].测序现在已成为常规[17],结构变异的操作范围已扩大到包括小得多的事件[18]、[19].在本研究中,我们使用软件包Breakdancer v1.2检测了10 bp到1 Mb之间的SVs [20.设置为默认参数。与参考基因组相比,我们在所有29个菌株中发现了7220 - 12,458个SVs(平均= 9,266)1:数据S12)。对于这些sv,删除事件的数量几乎是插入事件的二比一(附加文件2:图S8)。除InDels外,其他sv(包括DNA倒置、染色体内易位和染色体间易位)的发生率相对较低,在739 ~ 2360之间(平均= 1140)。大多数sv的大小在100 - 1000 bp之间,较长的变异(>1 kb)较少,尤其是大于10 kb的变异(附加文件)2:图S9),与高粱的情况一致[21].
变异对基因的影响
基因内DNA序列的变化对植物的形态和进化起着至关重要的作用。芝麻基因组中有27148个注释基因(http://www.ocri-genomics.org/Sinbase/),与参考基因组相比,74.8%(20,311)包含一个或多个snp。此外,62.6%(16,997)、15.5%(4,218)和18.0%(4,892)的基因在CDS、5 ' utr和3 ' utr中分别含有SNPs。这些基因被分为43个分子功能基团,其中30%与蛋白质结合、水解酶活性和ATP结合有关;然而,所有预测水解酶活性的基因仅在其CDS区域内包含snp2:图S10)。进一步的分析发现,在其CDS区域有258个snp基因显著富集(P< 0.01)为生物过程细胞死亡和凋亡过程(附加文件1:数据向)。在编码区识别出的136,130个非同义snp和142,103个同义snp表示非同义与同义替换比为0.99(附加文件1: S4数据;额外的文件2:图S11),与高粱(1.0)相似[22,但高于拟南芥(0.83) (23低于大豆(1.38)[15]和大米(1.2)[24].具有非同义SNPs基因的氧化石墨烯术语富集与细胞死亡、凋亡和防御反应密切相关(附加文件1:数据S14),特别是编码抗病蛋白、udp -糖基转移酶或含有富含亮氨酸重复序列和NB-ARC结构域的蛋白(附加文件)2:图S12;额外的文件1:数据S15)。这些结果表明,参与生物胁迫反应的基因突变率较高,这与植物-病原体相互作用导致病原体相关分子模式识别受体多样化的理论一致[25]、[26].
位于关键结构位置的编码区snp可以导致蛋白质形态的显著变化,进而导致蛋白质整体功能的变化。在研究的29个芝麻菌株中,我们发现了1281个与过早停止密码子形成相关的snp和246个与停止密码子到不停止密码子突变相关的snp。在186个基因中观察到起始密码子到非起始密码子突变,以及额外的404个剪接位点突变2:图向)。这些大效应snp大多位于假分子(LG)的近端(图)2).这四种大效应snp的注释显示了不同的功能富集模式。例如,起始密码子到非起始密码子突变主要出现在涉及转运、凋亡和防御反应的基因中,而剪接位点突变则更常见于与细胞代谢、氧化还原、有机物质代谢和氮化合物代谢相关的基因中2:图S14系列)。在四种大效应snp中,过早停止密码子尤其有趣,因为这些突变通常与功能丧失有关。大多数突变是在与氧化石墨烯生物过程相关的基因中发现的,这些生物过程与逆境有关,包括细胞死亡、凋亡和防御反应1:数据S16)。
尽管大多数SNPs在CDS区域被检测到,但CDS区域仅占12651个InDel突变的14.3%,低于5 '和3 ' utr(分别为18.4%和19.6%)。5’和3’UTRs中包含InDels的基因数量随着InDel大小从1到5 bp的增加而减少,但在3 bp的InDels中,CDS InDels显著增加,这与在高粱中观察到的相似[21)(附加文件2:图S15)。这种3-bp InDels的富集是意料之中的,因为InDels如果不是3-bp的倍数,就会导致帧移位,通常是致命的。最后,我们在基因本体的基础上分析了InDels的分布,发现它们与导致过早停止密码子的SNPs相似,且富集具有统计学意义(P< 0.001)的基因参与细胞死亡,凋亡和防御反应(附加文件1:数据肌力表现)。
29个芝麻品系间的亲缘关系
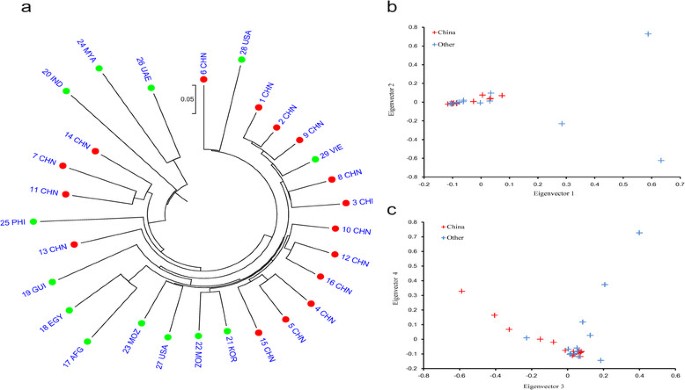
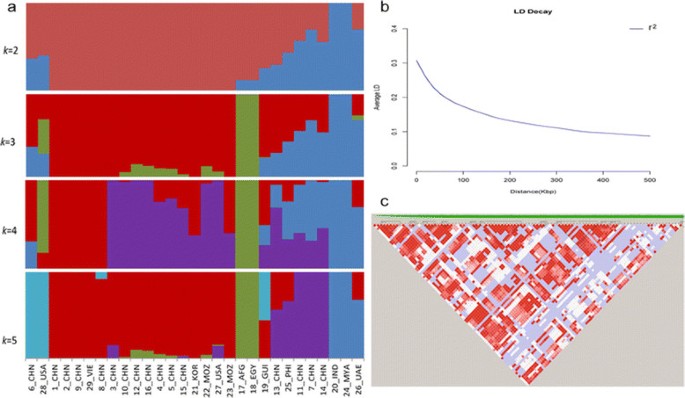
考虑到芝麻最初是在非洲还是在印度次大陆种植的时候[8]、[9因此,研究地理环境对芝麻遗传多样性的影响具有重要意义。采用邻居联结法构建了包含29个芝麻品系的系统发育树。该分析揭示了中国菌株之间的最高亲缘度,而来自其他国家的菌株则遍布全球(图3.a).基于主成分分析(PCA),来自不同地理位置的芝麻品系的这种相互交织的特性也很明显(图)3.b和c).使用贝叶斯聚类软件STRUCTURE观察模糊组[27], K由2 - 5逐渐变化(图4一个)。
由于本研究没有包括芝麻的任何亲缘种或野生种,因此不可能对芝麻的起源做出明确的结论。然而,观察到的29个芝麻品系之间的系统发育关系为芝麻的进化提供了一些线索。这三株来自印度、缅甸和阿拉伯联合酋长国的菌株相对于其他菌株表现出更高的遗传距离(图3.a).根据多样性理论的瓦维洛夫中心(Vavilov center of diversity theory),该理论认为,在植物最初被驯化的地方,可以观察到更丰富的遗传多样性[28,这些结果表明芝麻起源于印度次大陆。
芝麻的高度连锁不平衡
在设计关联研究时,LD模式对于确定映射分辨率是必要的[29]、[30.和解释联想峰值[31].为了估计芝麻的LD,我们计算了r2使用Haploview在snp对之间进行分析[32],并发现在大约150 kb的过程中,它从初始值0.30衰减到~0.15(图4b和c)。芝麻的LD衰减估计与自花大豆相当(约150kb) [15,但比在答:芥(~ 4 kb) (29],籼稻(约65 kb) [12)和谷子(约100 kb)。也显著高于高粱等异花授粉植物(1 kb) [33]和玉米(< 1kb) [34].芝麻的高LD不仅使它成为研究极端LD对基因组和群体结构影响的好植物[15],但也适用于多态标记相对较少的GWAS。
芝麻叶腋花数候选位点的聚类分离分析
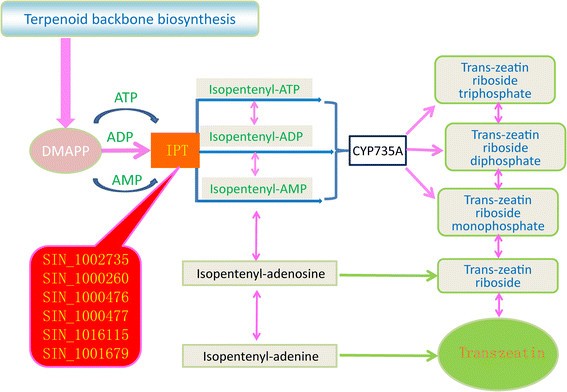
大块分离分析(BSA)是一种快速检测特定基因组区域标记的方法[35],并已成功应用于检测水稻各种性状的数量性状位点(QTL)或基因[36)、玉米(37和小麦[38].结合高通量测序技术,BSA已被用于鉴定木糖利用的新基因酿酒酵母.在此,我们利用BSA方法探索可能负责每叶腋花数的候选基因。这种表型是芝麻的一个重要农艺性状,因为它在预测产量中起着作用。29株芝麻菌株根据单花与三花(13株与16株)分为两组1:数据S1)。我们在两个基因库中鉴定出695个snp一致的基因。在这些基因中,有181个、21个和31个分别在cd中含有SNPs, 5'UTR和3'UTR1S18:数据美国)。GO术语注释主要与ATP结合、锌离子结合、核酸结合和热休克蛋白结合有关。特别感兴趣的是6个腺苷酸异戊烯基转移酶(ITP)同源物(SIN_1002735;SIN_1000260;SIN_1000476;SIN_1000477;SIN_1016115和SIN_1001679),它们在玉米素生物合成途径中显著富集(图5).玉米素是细胞分裂素植物激素家族的一员,已知其参与多种与植物生长发育相关的过程,包括促进侧芽生长和刺激细胞分裂以产生更浓密的植物[39]、[40].本研究结果表明,ITP基因可能通过介导玉米素的生物合成而影响芝麻叶腋花数。然而,还需要利用转基因模型或双亲杂交群体进行进一步的研究。
结论
下一代测序正迅速增进我们对作物遗传变异的了解[41].本研究首次对高油料作物芝麻进行了全面的重测序分析。这些数据来自12个国家的29个品系,提供了对芝麻种质基因组遗传变异的洞察,并促进了广泛的功能和进化研究,包括基因组进化、群体遗传学、标记辅助育种和基因鉴定。在芝麻基因组中发现高LD,表明标记辅助育种是芝麻改良的较好选择。这里提供的数据为芝麻起源于印度次大陆的假说提供了新的证据。在编码和非编码区域,我们发现了数十万个多态性,这为进化遗传学和功能研究提供了重要的资源。特别令人感兴趣的是含有非同义突变的基因,包括可能介导与环境相互作用的大效应snp。本研究还表明,ITP基因可能在决定芝麻叶腋花数方面起一定作用。然而,还需要进一步的研究来充分了解本研究中确定的遗传变异的功能相关性。
方法
选取29个栽培芝麻品系进行基因组重测序,其中16个来自中国,2个来自美国,1个来自阿富汗、阿拉伯联合酋长国、韩国、缅甸、菲律宾和越南。
图书馆建设与排序
用CTAB法从各品系的鲜叶和黄化叶中提取基因组DNA。根据制造商说明书(Illumina),使用5 μg基因组DNA构建每个菌株的成对端测序文库,嵌入大小约500 bp。测序使用Illumina Hiseq 2000平台。然后,原始测序读取经过一系列严格的过滤步骤,根据以下标准删除读取:
类型(1):对于短插入库和长插入库,分别具有≥10%和≥3%的未识别核苷酸。
类型(2):40%的基础阅读有>,质量分数< 7。
类型(3):>的读取10bp对齐适配器序列,允许≤2-bp的不匹配。
类型(4):与对应配对端重叠≥10bp的配对端读数。
Type(5):两个完全相同的对端reads的Read1和read2(认为是PCR复制的产物)。
在所有过滤步骤之后,在深度≥13倍处生成了> 120 Gb(附加文件1:数据S1)。
SNP打电话
利用BWA软件将Reads映射到“中植13号”芝麻基因组组装图上[10].使用的详细参数如下:
" bwa aln -m 200000 -o 1 -e 30 -i 15 -l 35-L -i -t 4 -n 0.04 -R 20 -f "
" bwa sampe -a 800 "
考虑到所有菌株都是一组,我们使用SAMtools函数“mpileup”[11使用映射质量≥20的reads检测原始种群snp。所用的参数如下:
" samtools mmpeup -uf -b -D | bcftools view -bvcgI -p 0.99 "
使用SAMtools程序“vcfutils”,使用上述过程提取的snp首先被过滤,得到30到581之间的测序深度。所用的参数如下:
“perl vcfutils.pl varFilter -d 30 -d 581”
根据以下标准进一步筛选原始SNP位点:拷贝数≤2,间隔至少5 bp,除了少数等位基因频率(MAF≥0.05),当SNP之间的距离< 5 bp时,SNP会被保留。多样性参数π而且θw使用10 KB的窗口和1 KB的滑动窗口进行测量[12]、[14].
为了检验SAMtools的SNP调用准确性,我们随机选取4个大小为4.5 ~ 8.1 kb的片段,用重叠引物进行扩增,得到的PCR产物进行Sanger测序。两种方法检测snp的符合率为92.3 ~ 95.2(平均为93.7%)2:图S4;额外的文件1:数据S3)。
此外,GATK工具包[42也被用来调用snp,如下所示:
我们首先使用BWA软件将干净的reads映射到芝麻基因组,参数如下:
" bwa aln -m 200000 -o 1 -e 30 -i 15 -l 35-L -i -t 4 -n 0.04 -R 20 -f "
" bwa sampe -a 800 "。
SAMtools用于分离、排序、rmdup和合并SAM对齐结果,picard-tools用于对bam结果进行排序并标记为副本。接下来,我们使用GATK程序从统一的基因型原始VCF中使用以下参数重新排列和过滤snp:
-T selectvariables -R -variant -concordance -o .jar
java -jar GenomeAnalysisTK.jar -T variantfiltering -R——filterExpression " QD < 20.0 || ReadPosRankSum < -8.0 || FS > 10.0 || QUAL < $MEANQUAL "——filterName LowQualFilter——missingValuesInExpressionsShouldEvaluatAsFailing——logging_level ERROR -o .jar
-T CombineVariants -R -V sample1. jar GenomeAnalysisTK.jarvcf - v sample2。vcf - gentypemergeoptions unique ify -o *。
利用GATK从16个连锁群中共获得2,003,821个群体snp。
短InDel检测
使用软件SOAPInDel的默认参数[43],根据满足对端要求并包含对齐间隙的映射读提取至多5 bp的主短插入或删除,所有间隙都由至少3个非冗余的对端读支持。然后过滤主要InDel集,包括读取质量值> 20和InDels < 5 bp。
结构变异检测
根据配对端测序的原理,配对端读取必须有一个对正向序列,另一个对反向序列。两个对准位置之间的距离应与插入件尺寸一致。因此,与基因组对齐的两个对端reads应该具有正常的方向和适当的跨度。通过聚类分析异常配对端对齐,并与之前使用Breakdancer软件定义的结构变化类型进行比较[20.使用默认参数运行。所得的SV数据集包括INS(插入)、DEL(删除)、ITX(染色体内易位)、INV(倒置)和CTX(染色体间易位),范围从10 bp到1 Mb。
连杆不平衡的计算
为了测量种群中的LD,我们计算了相关系数(r2)使用Haploview软件[32),如下:
- (1)
Ped和info文件作为输入文件生成。
- (2)
对于每一条染色体,例如LG1,参数设置为“java -jar haploview.jar -n -log LG1.log -pedfile LG1. genetype”。ped -info lg1 . gene - type.info -dprime -minGeno 0.6 -minMAF 0.01 -hwcutoff 0.001 -memory 2000 -maxdistance 500 "。
- (3)
然后用R脚本绘制曲线,绘制平均(R2)对一对明智的标记距离。
群体遗传学分析
多样性参数π而且θw使用10 KB的窗口和1 KB的滑动窗口进行测量[12]、[14].顶部和底部的5%块基于π值,将这些区块中的基因分别定义为高发散度基因和低发散度基因(附加文件1:数据S7和S8)。
单个snp被用来计算样本之间的距离。在带引导(1000)的p-distance模型下,使用TreeBest (http://sourceforge.net/projects/treesoft/files/treebest/)对29个芝麻品系进行筛选。系统发育树显示软件MEGA5 [44].使用软件EIGENSOFT [45].软件FRAPPE [46]被用来确定种群结构。
附加文件
缩写
- BSA:
-
的隔离分析
- cd:
-
编码序列
- CTX:
-
染色体间易位
- InDel:
-
插入和删除
- 走:
-
基因本体论
- GWAS:
-
全基因组关联研究
- INS:
-
插入
- 财年:
-
染色体内易位
- 发票:
-
反演
- 国际旅游业伙伴关系:
-
Isopentenyltransferase
- LD:
-
连锁不平衡
- 主成分分析:
-
主成分分析
- RDA:
-
农村发展管理
- SNP:
-
单核苷酸多态性
- SV:
-
结构变化
- UTR:
-
翻译区
参考文献
Carlsson AS, Pham TD, Bui TM, Werlemark G, Bui TC, Merker A:芝麻遗传多样性的研究(胡麻属indicumL.)在越南和柬埔寨通过RAPD标记估计。植物资源学报,2009,36(5):679-690。10.1007 / s10722 - 008 - 9393 - z。
Slavov GT、DiFazio SP、Martin J、Schackwitz W、Muchero W、罗杰斯- melnick E、Lipphardt MF、Pennacchio CP、Hellsten U、Pennacchio LA、Gunter LE、Ranjan P、Vining K、Pomraning KR、Wilhelm LJ、Pellegrini M、Mockler TC、Freitag M、Geraldes A、El-Kassaby YA、Mansfield SD、Cronk QC、Douglas CJ、Strauss SH、Rokhsar D、Tuskan GA:基因组重序列揭示了林树的多尺度地理结构和广泛的连锁不平衡杨树trichocarpa.植物生态学报,2012,32(3):713-725。10.1111 / j.1469-8137.2012.04258.x。
张颖,张旭,车铮,王磊,魏伟,李东:中国芝麻核心群体遗传多样性的表型和分子标记评价及小核心群体的提取。中国生物医学工程学报,2012,29(3):366 - 366。
Kim SY, Kang CW, Lee SW, Mathur PN, Hodgkin T, De Zhou M, Lee JR:用逐步聚类法筛选韩国芝麻核心种质。品种科学,2006,56(1):85-91。10.1270 / jsbbs.56.85。
Bhat KV, Babrekar PP, Lakhanpaul S:印度和外来芝麻的遗传多样性研究(胡麻属indicum利用随机扩增多态性DNA (RAPD)标记对L.)种质资源进行分析。植物生态学报,1999,29(1):433 - 433。10.1023 /: 1003724732323。
Cho YI, Park JH, Lee CW, Ra WH, Chung JW, Lee JR, Ma KH, Lee SY, Lee KS, Lee MC, Park YJ:芝麻遗传多样性与群体结构评价(胡麻属indicumL.)使用微卫星标记。基因与基因组学,2011,33(2):187-195。10.1007 / s13258 - 010 - 0130 - 6。
王亮,张勇,齐鑫,高原,张鑫:59个食用油作物多态cDNA-SSR标记的开发与鉴定胡麻属indicum(胡麻科)。中国生物医学工程学报,2012,29(5):394- 398。10.3732 / ajb.1200081。
Bedigian D, Harlan J:古代世界种植芝麻的证据。环境科学与技术,2004,25(2):1 - 6。10.1007 / BF02859136。
Bedigian D:芝麻的特性(胡麻属indicumL.)种质:评论。植物资源学报,2010,29(5):641-647。10.1007 / s10722 - 010 - 9552 - x。
Li H, Durbin R: Burrows-Wheeler变换快速准确的短读对齐。生物信息学,2009,25(14):1754-1760。10.1093 /生物信息学/ btp324。
李H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, Marth G, Abecasis G, Durbin R:序列对齐/地图格式和SAMtools。生物信息学,2009,25(16):2078-2079。10.1093 /生物信息学/ btp352。
徐鑫、刘鑫、葛森、Jensen JD、胡峰、李鑫、董勇、Gutenkunst RN、方磊、黄磊、李俊、何伟、张刚、郑鑫、张峰、李勇、于超、Kristiansen K、张鑫、王俊、Wright M、McCouch S、Nielsen R、王伟:对50份栽培稻和野生稻产量标记进行重测序,鉴定农艺重要基因。中国生物工程学报,2012,30(1):105-111。10.1038 / nbt.2050。
Varshney RK, Song C, Saxena RK, Azam S, Yu S, Sharpe AG, Cannon S, Baek J, Rosen BD, Tar an B:鹰嘴豆基因组序列草稿(中投arietinum)提供了品质提升的资源。中国生物技术,2013,31(3):240-246。10.1038 / nbt.2491。
郭硕、张杰、孙浩、Salse J、Lucas WJ、张浩、郑勇、毛亮、任勇、王铮、闵杰、郭欣、Murat F、Ham BK、张铮、高松、黄明、徐勇、钟森、Bombarely A、Mueller LA、赵红、何红、张勇、黄松、谭涛、庞娥、林凯、胡琪、匡辉:西瓜基因组草案(Citrullus lanatus),并对20个不同的资料进行了重新排序。植物学报,2013,45(1):51-58。10.1038 / ng.2470。
林敏敏,徐鑫,刘鑫,陈伟,杨刚,王福林,李明伟,何伟,秦宁,王斌,邵刚,un SS,张刚:31个野生大豆和栽培大豆基因组的重测序鉴定遗传多样性和选择模式。植物学报,2010,42(12):1053-1059。10.1038 / ng.715。
Feuk L, Carson AR, Scherer SW:人类基因组的结构变异。科学通报,2006,7(2):85-97。10.1038 / nrg1767。
Abecasis GR, Altshuler D, Auton A, Brooks LD, Durbin RM, Gibbs RA, Hurles ME, McVean GA:从人口规模测序得出的人类基因组变异图谱。自然学报,2010,46(6):769 - 769。10.1038 / nature09534。
Alkan C, Coe BP, Eichler EE:基因组结构变异的发现与基因分型。科学通报,2011,12(5):363-376。10.1038 / nrg2958。
李艳、郑辉、罗蓉、吴红、朱红、李锐、曹红、吴斌、黄松、邵红、马红、张芳、冯松、张伟、杜红、田刚、李俊、张欣、李松、Bolund L、Kristiansen K、de Smith AJ、Blakemore AI、Coin LJ、杨红、王娟:全基因组从头组装在单核苷酸分辨率上绘制的两个人类基因组的结构变异。中国生物技术,2011,29(8):723-730。10.1038 / nbt.1904。
Chen K, Wallis JW, McLellan MD, Larson DE, Kalicki JM, Pohl CS, McGrath SD, Wendl MC, Zhang Q, Locke DP, Shi X, Fulton RS, Ley TJ, Wilson RK, Ding L, Mardis ER: BreakDancer:一种用于基因组结构变异高分辨率映射的算法。物理学报,2009,6(9):677-681。10.1038 / nmeth.1363。
郑丽丽,郭秀贤,何斌,孙丽娟,彭莹,董珊珊,刘富峰,蒋珊,Ramachandran S,刘春昌,景洪昌:甜高粱和籽粒高粱全基因组遗传变异模式(高粱二色的).基因组生物学,2011,12 (11):R114-10.1186/gb-2011-12-11-r114。
Mace ES, Tai S, Gilding EK, Li Y, Prentis PJ, Bian L, Campbell BC, Hu W, Innes DJ, Han X, Cruickshank A, Dai C, Frere C, Zhang H, Hunt CH, Wang X, Shatte T, Wang M, Su Z, Li J, Lin X, Godwin ID, Jordan DR, Wang J:全基因组测序揭示了非洲本土谷物作物高粱未开发的遗传潜力。通讯学报,2013,4:2320-
Clark RM, Schweikert G, Toomajian C, Ossowski S, Zeller G, Shinn P, Warthmann N, Hu TT, Fu G, Hinds DA, Chen H, Frazer KA, Huson DH, Scholkopf B, Nordborg M, Ratsch G, Ecker JR, Weigel D:影响拟南芥遗传多样性的常见序列多态性。科学通报,2007,36(5):533 - 536。10.1126 / science.1138632。
McNally KL、Childs KL、Bohnert R、Davidson RM、Zhao K、Ulat VJ、Zeller G、Clark RM、Hoen DR、Bureau TE、Stokowski R、Ballinger DG、Frazer KA、Cox DR、Padhukasahasram B、Bustamante CD、Weigel D、Mackill DJ、Bruskiewich RM、Ratsch G、Buell CR、Leung H、Leach JE:全基因组SNP变异揭示了地方品种与现代水稻品种之间的关系。中国生物医学工程学报,2009,29(6):369 - 369。10.1073 / pnas.0900992106。
王晓燕,王晓燕,王晓燕,等。水稻Rpm1位点抗病多态性的动态分析拟南芥.自然,1999,400(6745):667-671。10.1038/23260。
McDowell JM, Dhandaydham M, Long TA, Aarts MG, Goff S, Holub EB, Dangl JL:基因内重组和多样性选择促进了霜霉病抗性的进化拟南芥.植物学报,1998,10(11):1861-1874。10.1105 / tpc.10.11.1861。
王晓燕,王晓燕,王晓燕。基于多位点基因型数据的群体结构推断。中国生物医学工程学报,2000,38(2):359 - 359。
TURRIL WB:栽培植物起源研究。自然杂志,1926,118:392-393。10.1038 / 118392 a0。
Kim S, Plagnol V, Hu TT, Toomajian C, Clark RM, Ossowski S, Ecker JR, Weigel D, Nordborg M:重组与连锁不平衡拟南芥.植物学报,2007,39(9):1151-1155。10.1038 / ng2115。
Mather KA, Caicedo AL, Polato NR, Olsen KM, McCouch S, Purugganan MD:水稻连锁不平衡程度(栽培稻l .)。中国生物医学工程学报,2007,29(4):378 - 378。10.1534 / genetics.107.079616。
赵魏黄X, X,唱T, Q,冯Q,赵Y,李C,朱C,陆T,张Z,李米,风扇D,郭Y,小王,小王L,邓L,李W,陆Y,翁Q,刘K,黄T,周T, Jing Y, Z,盾牌,钱Q,张QF,李J,汉族B: 14个农艺性状的全基因组关联研究大米一起。植物学报,2010,42(11):961-967。10.1038 / ng.695。
Barrett JC, Fry B, Maller J, Daly MJ:单倍视图:LD和单倍型图的分析和可视化。生物信息学,2005,21(2):263-265。10.1093 /生物信息学/ bth457。
Morris GP、Ramu P、Deshpande SP、Hash CT、Shah T、Upadhyaya HD、Riera-Lizarazu O、Brown PJ、Acharya CB、Mitchell SE、Harriman J、Glaubitz JC、Buckler ES、Kresovich S:高粱农业气候性状的群体基因组和全基因组关联研究。中国生物医学工程学报,2013,29(2):433 - 433。10.1073 / pnas.1215985110。
Gore MA, Chia JM, Elshire RJ, Sun Q, Ersoz ES, Hurwitz BL, Peiffer JA, McMullen MD, Grills GS, Ross-Ibarra J, Ware DH, Buckler ES:玉米第一代单倍型图谱。科学通报,2009,29(5):527 - 527。10.1126 / science.1177837。
Michelmore RW, Paran I, Kesseli RV:通过散装分离分析识别与抗病基因相关的标记:一种通过分离群体在特定基因组区域检测标记的快速方法。中国科学:地球科学,1998,21(3):369 - 369。10.1073 / pnas.88.21.9828。
Vikram P, Swamy BPM, Dixit S, Ahmed H, Cruz MTS, Singh AK, Ye G, Kumar A:批量分离分析:“水稻干旱产量一致性效应qtl的一种有效制图方法”。农学通报,2012,38(4):559 - 561。10.1016 / j.fcr.2012.05.012。
Quarrie SA, lazii - janiich V, kovavievvic D, Steed A, pekiic S:分子标记的体分离分析及其在玉米抗旱性改良中的应用。中国生物医学工程学报,1999,30(3):369 - 369。10.1093 / jxb / 50.337.1299。
Lanning SP, Fox P, Elser J, Martin JM, Blake NK, Talbert LE:小麦次生茎固性位点的微卫星标记。作物科学,2006,46(4):1701-1703。10.2135 / cropsci2005.10 - 0379。
Sasaki E, Ogura T, Takei K, Kojima M, Kitahata N, Sakakibara H, Asami T, Shimada Y:一种抑制拟南芥反式玉米素生物合成的细胞色素P450抑制剂Uniconazole。植物化学学报,2013,29(4):359 - 361。10.1016 / j.phytochem.2012.11.023。
kamada nobusada T, Sakakibara H:细胞分裂素生物合成的分子基础。植物生态学报,2009,30(4):444-449。10.1016 / j.phytochem.2009.02.007。
Flintoft L:作物遗传学:重测序播种种子。中国生物工程学报,2010,11(12):816-817。
McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M, DePristo MA:基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。中国生物医学工程学报,2010,20(9):1297-1303。10.1101 / gr.107524.110。
李松,李锐,李红,陆娟,李勇,Bolund L, Schierup MH,王娟:SOAPindel:短配对读的高效内链识别。基因组学报,2013,23(1):195-200。10.1101 / gr.132480.111。
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S: MEGA5:使用最大似然、进化距离和最大简约法的分子进化遗传学分析。分子生物学与病原学杂志,2011,28(10):2731-2739。10.1093 / molbev / msr121。
Patterson N, Price AL, Reich D:种群结构与特征分析。科学通报,2006,2 (12):e190-10.1371/journal.pgen.0020190。
唐宏,彭军,王萍,Risch NJ:单掺合料的估算:分析和研究设计的考虑因素。中华流行病学杂志,2005,28(4):289-301。10.1002 / gepi.20064。
确认
中国农业科学院油料作物研究所的研究得到国家作物种质资源基础设施项目(NB2012-2130135)、国家自然科学基金项目(no:31271766,没有。31101182也没有。国家作物种质资源保护(2012-014);中国农业科学研究系统(no. 31201242);汽车−15)。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明他们没有竞争利益。
作者的贡献
XRZ和LHW对研究的设计做出了贡献。LHW和XRZ撰写了手稿。XLH和LHW分别进行测序实验和数据分析。YXZ和DHL制备了材料并进行了实验。JYY执行数据库构建。所有作者阅读并批准了最终稿件。
电子辅料
12870 _2014_225_moesm1_esm.xlsx
额外的文件1:由补充资料S1至S18组成。数据S1。29个重测序芝麻品系的信息。数据S2。GATK和Samtools调用snp的比较。S3数据。用NGS对4个DNA片段进行Sanger测序,验证检测到的snp。S4数据。基因区snp的数量。数据S5。29株候选SNPs的总结。数据S6。芝麻和其他物种居群的多样性水平。S7数据。π值最高的区块中的基因列表(前5%)。S8的数据。π值最低的区块中的基因列表(前5%)。数据S9。在π最高的区域(前5%)基因富集氧化石墨烯。S10数据。在π最低的区域(前5%)基因富集氧化石墨烯。S11数据。29株菌株中检测到的InDels摘要。S12数据。29株sv检测结果总结。向的数据。富集氧化石墨烯术语指的是CDS中含有SNPs的基因。数据S14系列。富集氧化石墨烯术语指的是CDS中含有非同义SNPs的基因。S15数据。丰富了CDS中含有非同义snp的基因的知识产权术语。S16数据。富含氧化石墨烯的术语指的是含有大效应snp的基因。数据肌力。丰富的GO术语,用于CDS中含有移码indel的基因。S18数据美国。使用牛血清蛋白预测与各种表型相关的突变基因数量。(XLSX 408 KB)
12870 _2014_225_moesm2_esm.docx
额外的文件2:由补充图S1至S15组成。图S1。使用下一代测序技术获得的29个芝麻品系的干净数据。图S2。29个芝麻品系的总SNPs数和位于mRNA区域的SNPs数图S3。芝麻中不同SNP风格的频率比较。图S4。说明NGS测序和Sanger测序之间snp不一致的色度。图S5。29个芝麻品系UTR和CDS位点snp的统计。图S6。29个芝麻品系杂合子和纯合子snp的比例。图S7。29个芝麻品系1 ~ 5 bp的InDels数。图S8。29个芝麻品系中每一个sv的数量。图S9。29个芝麻品系sv的长度分布。图S10。5 ' -UTR和3 ' -UTR中snp基因的GO-SLIM分类。图S11。同义snp和非同义snp在CDS区域的比例。图S12。不同基因家族中含有非同义snp的基因比例。图向。含有大效snp的基因比例。图S14系列。含有大效应snp的GO-SLIM基因类别。图S15。CDS中不同InDels的基因数,5 ' -UTR和3 ' -UTR。(多克斯19 MB)
权利和权限
开放获取本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(https://creativecommons.org/licenses/by/2.0),它允许在任何媒体上不受限制地使用、分发和复制,只要原始作品的名称正确。创作共用公共领域奉献放弃书(https://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条提供的资料。
关于这篇文章
引用这篇文章
王磊,韩X,张燕。et al。深度重测序揭示等位基因变异胡麻属indicum.BMC植物杂志14日,225(2014)。https://doi.org/10.1186/s12870-014-0225-3
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-014-0225-3
关键字
- 胡麻属indicum
- 重新排序
- 变异
- 连锁不平衡