摘要
背景
许多研究表明大豆WRKY转录因子参与了植物对生物和非生物胁迫的响应。Phakopsora pachyrhizi是大豆最重要病害之一亚洲大豆锈病的病原。有证据表明wrkyys参与了一些大豆基因型对该真菌的抗性。的数量WRKY已在大豆基因组中标注的基因代表性不足。在本研究中,对大豆WRKY家族进行了全基因组注释,成员参与了对WRKY的响应p . pachyrhizi被确定。
结果
根据大豆基因组数据库的搜索结果,182WRKY标记-编码基因,鉴定出33个伪基因。基因参与了对p . pachyrhizi感染被确认使用微解剖病变的superSAGE、RNA-Seq及微阵列实验。真菌感染过程中有75个基因存在差异表达。8的表达式WRKY基因经RT-qPCR验证。这些基因在抗性基因型中的表达较早/或者比易感基因型更强p . pachyrhizi感染。对大豆体细胞胚进行过表达或沉默WRKY基因。胚胎过度表达WRKY虽然获得了基因,但无法转化成植物。当感染p . pachyrhizi沉默转基因株系叶片的病变数量高于野生型株系.
结论
本研究报道了大豆WRKY家族的全基因组注释。部分成员参与回应p . pachyrhizi显示感染。这些结果有助于阐明WRKYs的基因功能,并提示通过调控WRKYs来提高大豆植株的抗真菌能力。
背景
大豆(大豆玉米是世界上最重要的作物之一。目前影响大豆生产的主要病害之一是大豆锈病(ASR)Phakopsora pachyrhizi[1]。在有利于真菌繁殖的条件下,感染会导致产量损失10%至80% [2]-[4]。
在大豆种质上接种了三种侵染类型p . pachyrhizi(1)易感反应为“棕褐色”病变,尿路多,产孢多;(2)耐药反应,表现为红褐色病灶,尿路少,产孢量少至中度;(3)耐药反应,无明显病变或尿尿,具有免疫表型[5],[3.]。六个单一显性基因(Rpp1来Rpp6)调节大豆的抗性和/或免疫p . pachyrhizi已被确认[5]-[14]。这些基因的有效性在毒性ASR分离株中受到限制,而毒性ASR分离株能够克服它们各自赋予的耐药机制[1],[15]。因此,控制真菌传播最成功的方法是使用杀菌剂,但杀菌剂价格昂贵,对环境有负面影响,有利于病原体抗性的选择,在严重情况下是无效的[16]。在这种情况下,了解大豆防御真菌感染和生长的分子基础,识别敏感或抗性反应的基因,并描述它们的个体作用是工程持久和定量抗病性的关键步骤。因此,遗传转化是功能研究的有力工具。
许多研究表明大豆WRKY转录因子在对p . pachyrhizi感染(17]-[22]。WRKY基因可能调节防御基因的表达,调节直接下游靶基因或激活/抑制其他转录因子[23]。
WRKY转录因子是植物中最大的调控蛋白家族之一。之前的研究确定了72个WRKY-编码基因拟南芥[24],约100个米[25]-[28], 104杨树[29], 86英寸Brachypodium distachyon[30.], 80 in grape [31]以及两种不同棉花的116和102个基因[32]。原始真核生物全基因组分析[33揭示了WRKY蛋白的广泛存在。
这些蛋白质最显著的特征是WRKY结构域,这是一个高度保守的60个氨基酸区域,其特征是七肽WRKYGQK后面跟着一个C2H2-或C2HC锌手指图案。从核磁共振分析的结果推断的WRKY结构域在WRKY4,保守的WRKYGQK序列直接参与DNA结合[34],但也需要锌指图案[35]。大多数特征良好的WRKY蛋白与靶基因启动子区W-box元件(C/T)TGAC(C/T)结合[36]。结合位点的特异性部分取决于w盒核心附近的DNA序列,蛋白质复合物中WRKY因子的参与可能是决定启动子选择性的主要标准[37]。
64的识别WRKY在不同大豆组织中表达的基因和对非生物胁迫的反应,之前使用RT-PCR进行了评估[38]。然而,由于当时没有完整的大豆基因组序列,该基因家族的成员数量代表性不足。Yin等。[39]在大豆基因组中鉴定出133个WRKY成员。现在,几个用于大豆基因组分析的数据库已经公开了。PlantTFDB [40]大豆资料库[41]及SoyTFKB [42]是包含有价值信息的转录因子数据库,包括蛋白质序列、蛋白质结构域、预测的三级结构和与外部数据库的链接。然而,尽管这些数据库很有用,但它们都进行了系统的注释,导致大豆WRKY转录因子数量不同,一些基因模型也不正确。因此,到目前为止,还没有一个比较全面的大豆品种目录WRKY基因。此外,文献中对大豆WRKY成员的命名也不一致。Phytozome数据库(http://www.phytozome.org)从拟南芥而周等。[38]鉴定的64大豆WRKY基因(沉积于http://www.ncbi.nlm.nih.gov/),并给每个基因随机分配一个数字。此外,对单个基因的研究[43],[44]分配的数字与Zhou等人提出的不同。[38]。本研究报道了大豆WRKY家族的全基因组注释,并对一些参与大豆WRKY应答的基因进行了功能分析p . pachyrhizi感染。
结果
注释和在硅表征
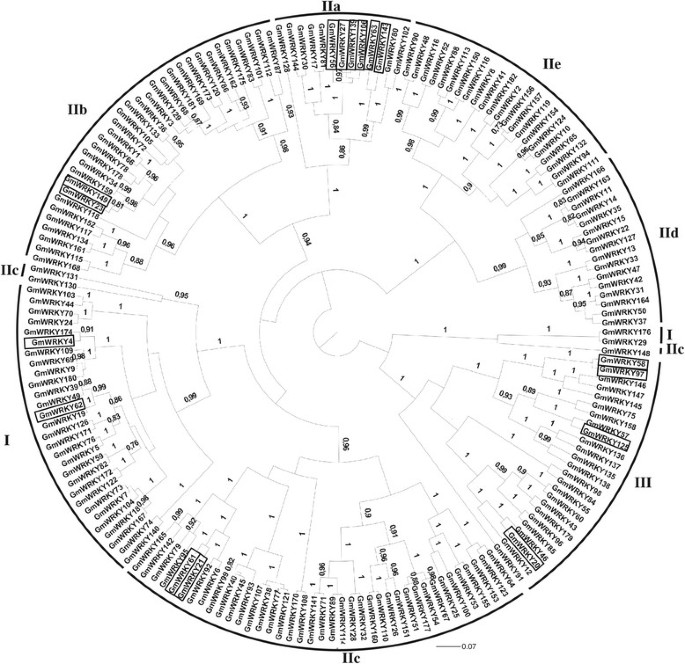
总共可能有182个WRKY-编码基因在目前的工作中被鉴定和注释(表1和附加文件1)。此外,共有33个推定的WRKY假基因被发现(附加文件2)。其中一些在我们的搜索中被识别出来,其他的在USM数据集中已经被描述过[45]。抄本为152个批注WRKY基因在SoyBase EST数据库(http://soybase.org/)和/或5个整体表达实验:大豆叶片接种(hai)后12、24和48小时的SuperSAGEp . pachyrhizi[46],接种后10 d微解剖病变的RNA-Seqp . pachyrhizi,两种不同的微阵列叶片12和120海的p . pachyrhizi(现有文献可查)和健康植物不同发育阶段RNA-Seq表达数据[47],可在SoyBase [48]。的GmWRKY基因分布在20条大豆染色体上,蛋白质序列长度从121到1356个氨基酸(表2)1和附加文件1)。平均得分为9.1分WRKY每个染色体上的基因,最多的基因(15个基因)位于染色体6上。
按照Eugelm等人的方法,将蛋白质分为三个主要的组和亚组。[24]。I、II和III组分别含有31、126和25个大豆WRKY基因,分别(表1和附加文件1)。分别有13、33、42、16和22个蛋白归属于IIa、IIb、IIc、IId和IIe亚群。
虽然WRKYGQK签名在大豆wrky中高度保守,但在c端结构域的签名中鉴定出15个氨基酸取代蛋白。这些变异蛋白分布在除IId亚群外的所有组中。WRKYGKK是最常见的变异,共有11个基因。其他非典型序列如WRKYGEK, WRKYEDK, WKKYGQK, CRKYGQK和WHQYGLK出现在单个蛋白中。九个WRKY蛋白在锌指序列中含有不完全和/或氨基酸取代(表1和附加文件1)。其中一些蛋白质含有锌指图案,在文献中尚未报道。检测到9个WRKY标记修饰基因和6个锌指修饰基因的表达,表明这些基因可能具有功能。此外,在IId基因成员的WRKY结构域上游发现了另一个高度保守的结构域,即锌簇。
用WRKY结构域序列进行的系统发育方法证实了分化通用汽车五组WRKY成员(I、IIa + IIb、IIc、IId + IIe和III)(图1和附加文件3.)。这些组对应的WRKY域分类(组和子组I, IIa, IIb, IIc, IId, IIe和III)已经在其他研究中得到了证明。IIa族基因与IIb族基因亲缘关系密切,IId族基因与IIe族基因亲缘关系密切。
基因表达数据
大豆差异表达研究综述WRKY被调节的基因p . pachyrhizi感染情况见表2和附加文件4.表达数据来自4个全局表达实验:接种后12、24和48小时叶片的SuperSAGE,接种后10天微解剖病变的RNA-Seq,以及目前文献中可获得的12和120海叶片的两种不同微阵列[17],[22]。75个基因在至少一个实验中表现出差异表达,16个基因在多个实验中表现出差异表达。来自I、II和III组的基因对这种应激条件有反应。
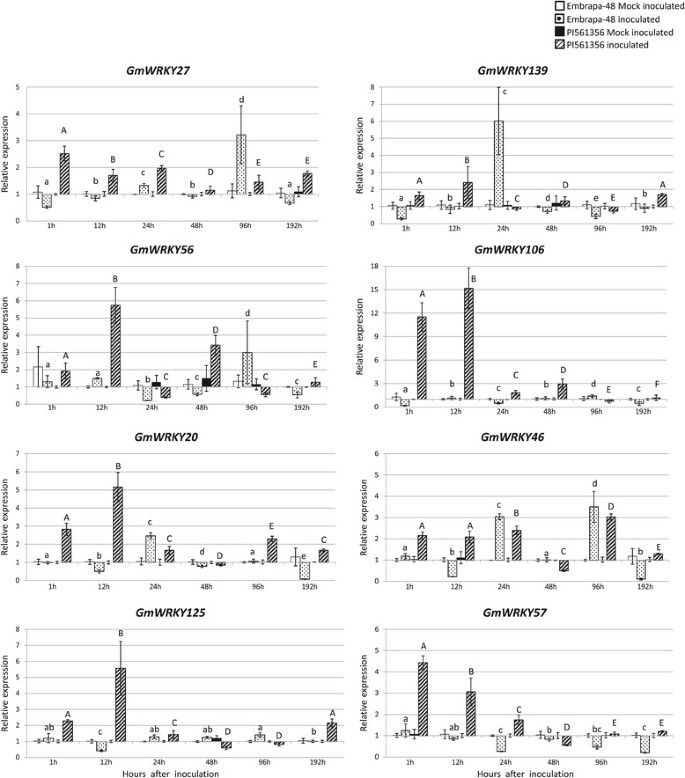
从每个分类组中随机选择一些对真菌反应表现出差异表达谱的基因进行更详细的分析。GmWRKY27(Glyma15g00570)和GmWRKY125(Glyma09g41050)在四次实验中的三次均有差异表达GmWRKY56(Glyma08g23380),GmWRKY106(Glyma07g02630)和GmWRKY20(Glyma08g02580)。GmWRKY139(Glyma13g44730),GmWRKY46(Glyma05g36970),GmWRKY57(Glyma18g44560)也被分析,因为它们与上述评估的至少一个基因密切相关。有趣的是,这些基因在真菌接种后10天的锈病感染皮损中均无表达(RNA-Seq)。
RT-qPCR检测这些基因的表达差异。在真菌侵染过程中,抗性基因型(PI561356)和易感基因型(Embrapa-48)的转录水平与模拟接种植物的转录水平进行了比较2)。
表达模式WRKY3周龄大豆植株感染p . pachyrizi。易感基因型(Embrapa-48)和耐药基因型(PI 561356)的基因反应p . pachyriziRT-qPCR检测感染(接种)情况。以模拟接种的植物作为对照。在3个生物重复和4个技术重复的基础上计算这些值(平均值±标准差)。3个因素(基因型、治疗和时间)的多因素分析具有高度显著性:GmWRKY57,GmWRKY27,GmWRKY125,GmWRKY20和GmWRKY46P = 0.0001;GmWRKY139P = 0.0265;GmWRKY56P = 0.0003。在相同品种和处理条件下,相同字母表示的平均值无显著差异(Tukey's多重比较检验,p < 0.05)。用小写字母区分接种的Embrapa-48植株之间的差异,用大写字母区分接种的PI561356植株之间的差异。F-Box蛋白和金属蛋白酶内参基因作为内对照,使每个样品中mRNA的含量归一化。转录水平WRKY利用模拟接种植株中存在的基因来计算接种植株中转录本的积累。
基因型、时间病程和病原体存在之间的相互作用极显著(p < 0.0001)。在接种植株中,这8个基因在PI561356(抗性)中比Embrapa 48(易感)中提早表达。在Embrapa 48中,24和24的表达峰较高/而在PI561356中,这些峰从1海到24海不等。此外,GmWRKY56,GmWRKY106,GmWRKY20和GmWRKY125在耐药基因型中表现出较强的反应。有趣的是,同源基因(GmWRKY27和GmWRKY139,GmWRKY125和GmWRKY57)与耐药基因型的表达峰值不重叠。GmWRKY27和GmWRKY57在一个海表现出较高的表达水平,然后表达下降,而GmWRKY139和GmWRKY12512海呈现更高的转录水平。
GmWRKY27在大豆植株中的过表达与沉默
GmWRKY27之所以选择该基因进行进一步的功能表征,是因为它是在不同实验中表现出差异表达的基因之一。此外,该基因还参与了不同的非生物胁迫[38]。确定功能的作用GmWRKY27作为对p . pachyrhizi对大豆体细胞胚进行转染,获得基因过表达和沉默。在过表达实验中,在耐湿霉素球状胚胎中检测到GFP的表达(附加文件)59个独立转基因株系的组织分化胚胎(7个来自Biobalistic, 2个来自/农杆菌属)。T-DNA在胚胎基因组中的存在通过PCR得到证实GmWRKY27在四个独立的转基因株系的胚胎中表达显著升高(附加文件)5C).然而,转基因胚胎过度表达的发展GmWRKY27没有成功。结果,这些胚胎无法发育成植物。
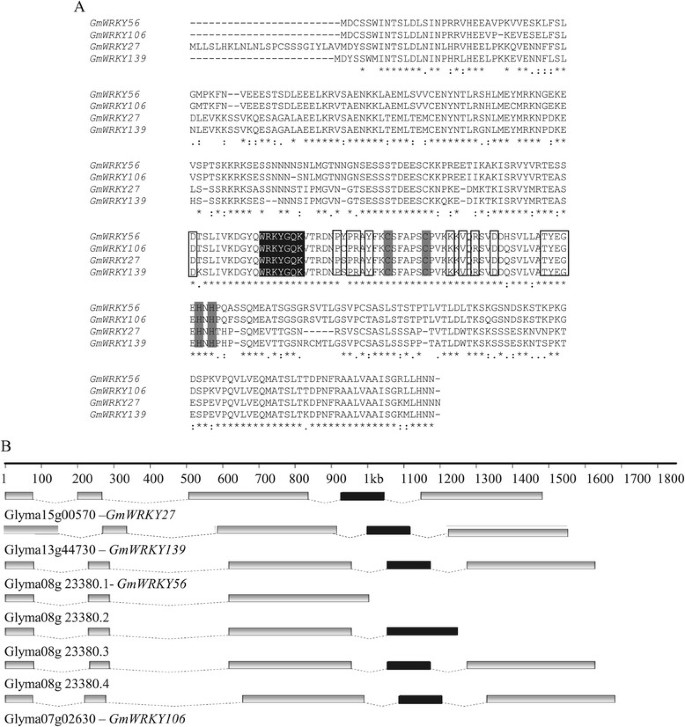
对于基因沉默,载体携带176 bp的反向重复序列从GmWRKY27构造。该片段与同源区域的相似性为83%GmWRKY13970%和67%的相似度GmWRKY56和GmWRKY106分别。这些数据证实了基因之间的密切关系,在系统发育分析中也观察到了这一点(图1)。这种高序列相似性表明沉默结构将针对这四个基因。
对4个同源基因进行更详细的结构分析,发现序列中的WRKYGQK签名、锌指基序等残基在4个对应蛋白中高度保守(图3.A).全蛋白序列同一性从66% ~ 94%不等(表3.)。4个大豆基因被推定为同源基因AtWRKY40, AtWRKY18和AtWRKY60拟南芥基因,如系统发育树所示(附加文件3.)。基因结构GmWRKY27,GmWRKY139, GmWRKY56和GmWRKY106相似,WRKY结构域存在于第4外显子中(图3.B)。有趣的是,GmWRKY56有四个可选的转录本,其中一个转录本缺乏WRKY域。
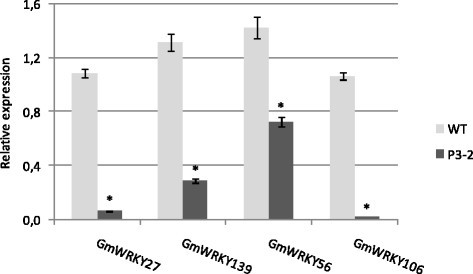
获得了两个独立的转基因株系(品种BRSMG 68 Vencedora)。分子分析显示,从第一行中消除了一个重复序列(176 bp片段)。因此,没有触发转录后沉默,这是通过RT-qPCR证实的(数据未显示)。在第二个转基因株系(P3-2)中,完整的盒体被成功整合(数据未显示)。正如预期的那样,RT-qPCR分析结果显示,四个同源基因的表达显著降低(图4)。转基因株系表型无明显改变。
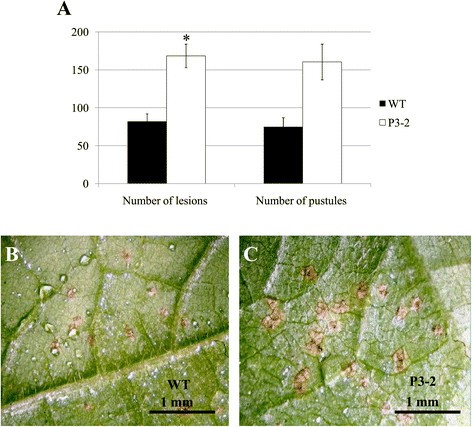
结果表明,沉默系对肿根P. pachyrhizi更敏感
进行了离体叶试验,以确认涉及GmWRKY27,GmWRKY139,GmWRKY56和GmWRKY106在大豆方面的反应p . pachyrhizi感染。如前所述,分离的叶片和完整的植物生物测定显示出高度相关性[49]。在本研究中,转基因和野生型样品的所有离体叶片在12天后都能观察到“棕褐色”病变p . pachyrhizi接种。然而,转基因株系叶片的病变数量明显更高(图5)。病变的外观和脓疱的形成或爆发没有明显的差异(数据未显示)。
讨论
大豆WRKY基因
全基因组测序[50促进了大豆基因家族的准确注释。在本研究中,我们对大豆中182个WRKY转录因子进行了注释。检测到152个基因的转录本,表明它们可以在蛋白质水平上表达;然而,剩余基因的成功转录可能需要特定的条件。
如前所述,文献中对大豆WRKY成员的命名不一致。为了统一术语,我们在前面描述的基础上提出了一个命名法WRKY-编码基因[38,并作了一些修改。序列比较的数据表明GmWRKY18和GmWRKY35是同一个基因。此外,GmWRKY3在大豆基因组中不存在;事实上,这个序列代表了通过反式分别在Glyma02g46690和Glyma14g01980的n端和c端序列之间进行-拼接。其余118个基因按照染色体的顺序进行编号1和附加文件1)。
更多的WRKY在大豆中发现的基因比在其他物种中发现的要多,比如水稻,拟南芥,棉花,葡萄和b . distachyon[24]-[28]。复制事件被大大过度保留,特别是在转录因子的情况下[51]。因此,功能冗余是植物物种的共同特征。然而,同源基因在功能上可能存在分歧,这为进化提供了新的来源[52]。
本研究中使用的系统发育方法允许对大豆进行分化WRKY先前报道的五组基因[26],[53],[54]。
在大豆中,第I组成员的结构域带有一个C2H2型锌手指图案。同样的特征在拟南芥而在水稻中,组I成员的WRKY结构域包括两种类型的锌指基序:C2H2和C2HC (25],[27]。
尽管WRKYGQK特征在大豆WRKY蛋白中高度保守,如图所示6,在21个基因中鉴定出变异。Zhou等。[38之前的研究表明GmWRKY6(Glyma08g15050)和GmWRKY21(Glyma04g39650)包含变体WRKYGKK而不是保守的WRKYGQK基序。在该地区也有轻微变化的报道拟南芥、大米、烟草、大麦、油菜籽及向日葵[25],[26],[55]-[58]。与含有4个WRKYGKK变异体的拟南芥相比,大豆中WRKYGQK修饰基序的基因数量更多。
一些不寻常的GmWRKY-编码基因(即含有修饰的WRKY标记和/或锌指基序)产生mRNA(表2和附加文件4)。如前所述,需要进一步分析以确定这些基因是否作为转录因子发挥作用,或是否通过RNAi诱导转录后调控[23]。变异蛋白可能已经取消或降低了与w盒结合的能力[35],[37]。有研究表明,没有典型WRKYGQK基序的WRKY蛋白可能具有不同的结合位点[37],[56]、目标基因及可能的不同作用[57]。
功能分析
尽管事实证明或预言了许多WRKY以前已经从不同物种中获得了基因,但其中只有一小部分具有功能特征。有关大豆基因(Glyma13g00380-)作用的信息GmWRKY13Glyma04g39650 -GmWRKY21Glyma10g01450 -GmWRKY54和Glyma18g44560 -GmWRKY57)基于从异源表达系统获得的数据[38],[43]。来自表达式分析的数据[17],[44]或使用瞬时基因沉默[59的角色WRKY基因对生物压力的反应。关于全局表达分析的研究已经证明了的重要性WRKY在转录重编程过程中编码基因p . pachyrhizi大豆植株的感染[17]-[22]。
来确定哪种大豆WRKY基因参与植物的防御p . pachyrhizi感染后,我们进行了一系列分析,以检查感染后的表达模式。我们最初比较了文献中可用的微阵列数据[17],[22]以及另外两个实验的结果:SuperSAGE和RNA-Seq。许多基因仅在一个文库中存在差异表达,少数基因在多个文库中存在差异表达。验证了8个基因转录水平的调节,表明了数据挖掘的可靠性。相似的表达模式对p . pachyrhizi在密切相关的基因中观察到感染(图1),例如GmWRKY61(Glyma06g15220)和GmWRKY21(Glyma04g39650),GmWRKY143(Glyma14g11920)和GmWRKY63(Glyma17g33920),GmWRKY106和GmWRKY56,GmWRKY58(Glyma04g40130)和GmWRKY97(Glyma06g14720)。这种相似的表达模式表明,这些基因在抗病方面可能具有相似的功能。的冗余函数GmWRKY基因可能有利于在各种应激条件下保护细胞或有机体,并引发多种途径,导致病原体感染后发生的各种生理反应[60]。
全球表达数据表明,防御途径的诱导时间和程度是大豆抗性诱导的决定因素p . pachyrhizi[17],[20.],[22],[60]。在我们的研究中,诱导表达GmWRKY20, GmWRKY27, GmWRKY46, GmWRKY57,GmWRKY56, GmWRKY106, GmWRKY125和GmWRKY139作为对p . pachyrhizi更早的时候/或者抗性基因型更强。在抗性基因型中,大多数基因的表达在12海达到峰值;因此,我们认为这些基因可能参与了非特异性防御反应。Van de Mortel等人。[17]和施耐德等人。[22报告说p . pachyrhizi感染诱导双相全局表达。基因表达最早在12海达到峰值,这与附着胞形成和表皮细胞渗透的早期侵染过程相对应。作者认为,这一峰值对应于一种类似于病原体触发的免疫的非特异性防御反应。基因表达的第二阶段开始于72海,一直持续到288海,与吸器的形成和效应蛋白的分泌相一致。作者认为,这种反应是一致的激活RPP2和RPP3介导的耐药性。已有研究表明,在不相容相互作用中,基因表达迅速并增加[17],[18],[22]。
密切相关的基因GmWRKY27,GmWRKY139,GmWRKY56和GmWRKY106是推定的正交体吗AtWRKY40, AtWRKY18和在WRKY60拟南芥基因。在这两个物种中,这些基因被归类为IIa组。这三个拟南芥wrky参与应激反应,包括对细菌的抗性两和真菌葡萄孢菌[61],[62]。在WRKY18是一种水杨酸诱导基因,正向调控SAR [63],[64]并调节PR基因的表达;在WRKY18过度表达会增加抵抗p .两[65]。在WRKY40和在WRKY60蛋白拮抗在WRKY18期间p .两感染。单、双或三重组合突变体基因功能的获得或丧失导致易感性增加b .灰质[61]。一些大米,大麦和芸苔属植物显著来自IIa组的WRKY成员也参与了对真菌和细菌病原体的反应,如表达研究所证明的那样。OsWRKY62和OsWRKY76是上调的稻瘟病菌感病叶片和表达下调的黄oryzae-接种叶片[66]。HvWRKY1和HvWRKY2在应对中发挥重要作用Blumeria茎感染(55),而BnWRKY18和BnWRKY40在应对中发挥作用菌核病sclerotiorum和链格孢属brassicae感染(57]。
大多数关于大豆基因功能的信息是基于从异源表达系统获得的数据。然而,由于许多蛋白质的活性往往依赖于仅在同源背景下发现的特定相互作用,本研究基于同源表达系统。采用RNA干扰方法沉默4个大豆同源基因(GmWRKY27,GmWRKY139,GmWRKY56和GmWRKY106)。四重沉默是一种优势,因为由于密切相关成员之间的功能冗余,单次敲除转录因子很少会导致表型改变[65]。本研究中使用的转基因RNAi系显著降低了四个靶基因的转录水平。当感染p . pachyrhizi,转基因株系对真菌的敏感性增加。综上所述,这些结果有力地表明,这四个基因中至少有一个可能与大豆抗性表型有关。
潘迪等人[59]采用病毒诱导基因沉默(VIGS)方法,分别沉默64个大豆wrkyy,以检测其参与Rpp2 -介导性抵抗p . pachyrhizi感染。其中三个基因(GmWRKY45,GmWRKY40和GmWRKY36)沉默时,会损害抗性表型。表型改变没有被证明GmWRKY56和GmWRKY106基因被单独沉默.然而,在目前的研究中,易感性增加p . pachyrhizi在四组沉默(GmWRKY27,GmWRKY139, GmWRKY56和GmWRKY106)行,表明这种表型是由GmWRKY27和/或GmWRKY139沉默。此外,本研究分析的4个基因也可能在致病防御反应中发挥协同作用。
之前的一项研究表明GmWRKY27在干旱和盐胁迫条件下大豆也强烈诱导[38]。总之,这些数据表明,该基因可能参与了一种发生在生物和非生物应激防御路线上游的非特异性反应,与特异性反应相反Rpp2 -Pandey等人鉴定的基因的反应[59对真菌感染的反应。
GmWRKY27用于过表达研究。通过4个独立的转化实验获得过表达该基因的组织分化胚胎。然而,这些植物并没有被回收。最可能的解释是GmWRKY27可能会影响植物的再生。在研究持续过表达对植物有有害影响的基因时,使用本构启动子是一个主要的限制[67]。陈与陈[65]报告了高水平的在WRKY18导致植物生长严重异常。即使在中等水平,个体或组合的过度表达AtWRKY18,AtWRKY40和AtWRKY60导致植物生长较小或发芽后不久死亡[61]。
这些WRKYs在植物生长过程中过量产生的有害影响表明,在激活植物防御反应时,该基因的表达可能需要适当的调控。然而,在健康植物中,这些基因的表达受到负调控,如Chen和Chen [65]为AtWRKY18.
在一定程度上,本研究中观察到的致命性问题可以部分克服使用组织特异性、发育调节或诱导启动子。虽然近年来组织特异性启动子的数量有所增加,但大豆叶片特异性启动子仍然缺乏。
结论
在本研究中,从大豆中鉴定了182个WRKY转录因子。有75个基因被鉴定出与大豆对p . pachyrhizi基于转录调节的感染.使用RNAi方法证实了四个基因参与对病原体感染的反应。需要进一步的研究来提供关于单个基因功能的线索。这些结果有助于阐明WRKYs的基因功能,并提示通过调控WRKYs来提高大豆植株的抗真菌能力。
方法
数据库搜索和序列注释
搜索大豆(通用汽车)WRKY我们使用了两种不同的方法:首先我们从Phytozome下载了大豆蛋白质组(http://www.phytozome.org)及SoyBase (http://soybase.org/)数据库,使用BLASTALL软件执行批处理BLAST [68]。中的WRKY域拟南芥[40],杨树[40]和大豆[40]-[42],[45]基因组在SMART网站上进行检查,并被用作执行tblastp (e-value cut off of 10)搜索的查询。在进行Batch BLAST搜索后,我们检查了大豆WRKY植物ttfdb基因(http://planttfdb.cbi.pku.edu.cn/)转录因子数据库及USM数据集[45]。
此外,我们使用编码序列(CDS)对Phytozome数据库(www.phytozome.org)及广场(http://bioinformatics.psb.ugent.be/plaza/)检索任何额外的WRKY基因。利用Phytozome数据库获取基因结构。自动WRKY-使用GENSCAN重新注释包含不正确基因模型(错误的开始/停止密码子或截断的蛋白质)的预测基因序列[69]和FEGENESH [70]预测因子,考虑从Gbrowse中获得的2,5或10kb DNA序列。序列与ClustalX v2.1进行比对[71],以及手动检查的域。没有保守的WRKYGQK域签名的序列被丢弃。利用MEME套件分析WRKYGQK和锌指结构域的保守程度(http://meme.sdsc.edu/meme/)。与Eugelm等人提出的方法一致,将注释的基因按组和亚组进行分类。[24]拟南芥。的命名法WRKY-编码基因在这项工作中被采用,根据染色体的顺序。四种大豆的结构WRKY-编码基因选择进行功能分析,并使用Fancy Gene v1.4分析其替代转录本[72]。
大豆WRKY关系
以便对大豆进行分类WRKY在两个数据集上进行了系统发育方法:第一个数据集只包含大豆WRKY序列,第二个数据集也包含拟南芥和杨树trichocarpaWRKY序列,从PlantTFDB数据库下载。采用MUSCLE软件进行多序列比对[73],由MEGA5(分子进化分析)软件实现[74]。利用BEAST1.7软件中实现的贝叶斯方法对WRKY结构域序列进行系统发育分析[75]。蛋白质进化的最佳拟合模型是使用抗议来确定的[76],选择JTT模型进行蛋白质基质置换。选择圣诞树作为贝叶斯分析的先验树,使用马尔科夫链蒙特卡罗(MCMC)算法进行了30.000.000代。在FigTree v1.3.1软件中对树进行可视化和编辑[77]。
基因表达数据挖掘
的通用汽车将WRKY cds搜索到RNA-Seq表达数据中[47],可在SoyBase [48]。的表达式配置文件WRKY被调节的基因p . pachyrhizi感染来自四个不同的来源。在前三个试验中,大豆植株对锈病感染的反应是通过接种p . pachyrhizi在野外收集的孢子进入在巴西PR Londrina Embrapa Soja的温室条件下保存的植物中。控件的表达式概要文件所使用的源WRKY基因被描述为:
a) SuperSAGE:文库采用大豆抗性基因型(PI561356)的叶片构建Rpp1抗性基因,感染了p . pachyrhizi与接种后12、24和48小时采集的未感染叶片(模拟接种/对照)相比(hai)。使用Plant RNeasy试剂盒(Qiagen)进行RNA提取,从每个样品中提取等量的RNA用于构建RNA池。文库(接种和模拟)在GenXPro GmbH(法兰克福,德国)使用先前描述的方法构建[78],随后使用Illumina基因组分析仪IIx进行测序。使用DiscoverySpace软件v.4.01分析SuperSAGE标签[79]来识别唯一的(unitags)和差异表达的标签(p≤0.05)。这些文库是作为GenoSoja项目(巴西大豆基因组联盟)的一部分构建的,结果可在LGE (Laboratório de Genômica e Expressão, UNICAMP)大豆基因组数据库[80]为财团成员提供服务。
b)病灶LCM(激光捕获显微解剖)RNA-Seq:叶段(1 cm2)包含p . pachyrhizi两种大豆抗性(PI561356)和易感BRS231 [81在感染后10天采集V2生长阶段的基因型。叶段立即在法默溶液中固定在冰上[82],按照Cai和Lashbrook的方法脱水后包埋在石蜡上[83]。使用旋转切片机生成12 μm厚度的系列切片,并转移到显微镜膜载玻片上。为每次生物复制/处理准备20个包含不同数量锈蚀病变的切片。使用PixCell II LCM系统(Arcturus)和CapSure Macro LCM (Arcturus)收集病变内的叶细胞。使用PicoPure RNA分离试剂盒(Arcturus)从每个生物复制不同数量的感染位点收集的细胞中提取总RNA。合成cDNA,在Illumina基因组分析仪GAAllx上进行108 bp的高效配对测序。低质量的RNA-Seq读取被丢弃。将reads(总共86,301,242)与大豆基因组进行比对,并使用TopHat [84]和SOAP2 [85对齐程序。使用FPKM(每千碱基外显子每百万个映射片段的片段数)值计算基因表达[86]。为了识别差异表达基因,两种基因型的FPKM值之间进行了成对比较t-在99%置信水平下进行测试。该库是作为Biotecsur联盟的一部分构建的,其结果是可用的[87]为财团成员提供服务。
c)微阵列[17:表达式oWRKY在大豆抗性基因型(PI970230)的叶片中发现了基因Rpp2基因,并在大豆易感基因型(Embrapa 48)中响应p . pachyrhizi将侵染与未侵染叶片(模拟接种)进行比较。在本研究中,考虑在12和120海获得的数据,因为在这些时间点表现出最高的基因表达。只检查了46个以前被描述为wrky的探针。利用SoyBase和Phytozome数据库分析探针的特异性。探针与e-values <0.05为考虑因素。
d)微阵列[22]:大豆品种Ankur (PI462312)的全局表达Rpp3分别用夏威夷94-1和台湾80-2菌株接种p . pachyrhizi,进行分析。使用Soybase数据库中可用的工具搜索Affy探测集。在本研究中,只选择了与大豆基因组中单个位点杂交的WRKY探针。考虑在12和120海获得的数据,因为在这两个时间点出现了最高的基因表达。p值<0.05的基因被认为是差异表达。
厚根P. pachyrhizi的基因表达分析
在温室条件下(28±1℃)盆栽系统中,光照强度为22.5 μEm,光照周期为16/8 h2年代1在Embrapa Soja, Londrina, PR,巴西。Embrapa-48基因型,形成“棕褐色”病变[17],作为易感标准,PI561356基因型携带Rpp1抗性基因[88],作为耐蚀性标准。从巴西田间分离的ASR在一个敏感品种上得到了保存。从叶片上收获孢子,在含有0.05%吐温-20的蒸馏水中稀释至最终浓度为3 × 105孢子/毫升。孢子悬浮液在植株V2发育阶段喷洒。用不含孢子的相同溶液进行模拟接种。随后,将水雾袋放置在每个罐子上一天。分别于1、12、24、48、96、192海分别采集三叶叶1片,液氮冷冻,-80℃保存。分析每个基因型/处理的3个生物学重复。
利用逆转录和实时荧光定量PCR (real-time RT-qPCR)分析表达模式
使用TRIzol试剂(Invitrogen)提取总RNA,并根据制造商的说明使用DNAse (Promega)进一步处理。使用M-MLV逆转录酶系统(Invitrogen)和24-polyVT引物提取2 μg无dna RNA,获得第一链cdna。RT-qPCR使用StepOne Applied Biosystems Real-Time cycler™进行。pcr循环条件如下:94℃下5 min, 94℃下10 s, 60℃下15 s, 72℃下15 s, 40次循环,60℃下2 min。在55-99°C的PCR运行结束时进行熔解曲线分析,每1 s逐步增加0.1°C的温度。每个25 μL反应由12.5 μL稀释DNA模板、1X PCR缓冲液(Invitrogen)、2.4 mM MgCl组成2, 0.024 mM dNTPs, 0.1 μM每个引物,2.5 μL SYBR Green(1:10万- molecular Probes Inc.)和0.03 U铂Taq DNA聚合酶(Invitrogen)。对cDNA(1:100)模板进行鉴定。所有PCR反应均采用技术四重复。缺乏模板的反应被用作阴性对照。
PCR反应使用基因特异性引物进行(表4)。设计用于扩增F-Box蛋白和金属蛋白酶序列的引物对用于标准化每个样品中存在的mRNA的数量。这些基因之前已被证实为本研究所用实验条件的良好内参基因[89]。对扩增产物用2——ΔΔCt方法(90]。结果采用方差分析与基因型、时间和病原体存在三因素处理进行统计学比较。采用加权最小二乘法对数据进行变换。均数比较采用Tukey多元比较检验。
沉默和过表达载体的构建
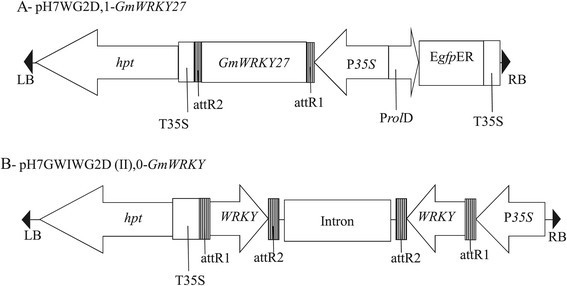
的开放阅读框GmWRKY27(Glyma15g00570),根据Phytozome v1.0,是从MGBR-46 Conquista大豆品种中使用高保真Taq DNA聚合酶(空斑形成单位-Fermentas)。使用Gateway®系统(Invitrogen)将PCR产物重组成过表达pH7WG2D,1个载体[91]。得到的pH7WG2D的T-DNA区,1-GmWRKY27向量包含GmWRKY27CaMV控制下的基因ORF35个年代启动子,即湿霉素-磷酸转移酶标记基因(成)和绿色荧光蛋白报告基因(绿色荧光蛋白)(图7A).使用pH7GWIWG2(II)构建RNAi沉默载体,0 [91]。得到的pH7GWIWG2(II)的T-DNA区,0-GmWRKY27含有反向重复片段(176 bp)GmWRKY27序列,由内含子从拟南芥基因组DNA序列,在CaMV的控制下35个年代启动子与湿霉素-磷酸转移酶标记基因(成)(图7B).这两种构造都是通过DNA测序确认的。
T-DNA区域的二进制向量用于通用汽车WRKY27过表达或通用汽车大豆中WRKY沉默。(一)过表达构造- pH7WG2D.1-GmWRKY27.的全长ORFGmWRKY27被克隆到载体中。(B)RNAi抑制结构- pH7GWIWG2(II).0-GmWRKY.将一个176 bp的WRKY片段反向重复序列克隆到该载体中。RB - T-DNA右缘,LB -左缘,成湿霉素磷酸转移酶基因,花椰菜花叶病毒(CaMV) 35S启动子,T35S - CaMV 35S终止子,EgfpER增强绿色荧光蛋白,ProlD -根位点D启动子,WRKY-大豆176 pb WRKY片段,attB1和attB2 - LR反应位点。
大豆转化与植株再生
大豆品种MGBR 46 (Conquista)、BRSMG 68 (Vencedora)和IAS5的豆荚中含有3-5毫米长的未成熟种子。他们都易受影响p . pachyrhizi.利用Droste等的方法,从未成熟的子叶诱导体细胞胚胎发生并增殖。[92]。使用颗粒流入枪(PIG)对增殖的胚胎发生组织进行颗粒轰击转化[93]遵循Droste等人的程序。[92]或使用无dna粒子轰击和农杆菌属变换(94]。在湿霉素b选择培养基中培养3个月后,目视选择耐湿霉素胚性大豆组织,进行单独培养,建立与假定的独立转化事件相对应的品系。
胚胎组织分化、转化为植物和驯化如前所述[92]。所有来自独立的抗湿霉素组织样本的植物都被认为是克隆植物。在同样的培养条件下,从未转化胚胎发生组织中提取的植物被回收,并用作分子表征和生物测定的对照。
转基因胚胎和植物的筛选
提取总DNA [95]来自抗湿霉素组织分化的胚胎和植物叶子。使用不同引物组合对假定的转基因胚胎/植物进行pcr筛选,以确定是否存在完整的T-DNA(表2)5)。PCR混合物由200 ng模板DNA、0.4 mM dNTPs、每个引物0.4 μM、2.5 mM MgCl组成2,1X Taq Buffer, 1u Taq DNA聚合酶(Invitrogen),蒸压蒸馏水,最终体积为25 μl。反应初始加热(94°C下5分钟),并在以下条件下进行30个循环:94°C下45秒,58°C下45秒,72°C下1分钟。在含溴化乙锭(0.01 mg/L)的1%琼脂糖凝胶上电泳后,在紫外线下观察PCR产物。
在蓝光下使用Olympus®荧光立体显微镜检测GFP表达,该显微镜配备了BP滤波器组,其中包含488 nm激发滤波器和505-530 nm发射滤波器。使用QCapture Pro™6软件(QImaging®)捕获图像。
RT-qPCR检测基因过表达或沉默。RNA提取,cDNA合成,qPCR分析。
真菌生物测定
采用离叶法评价p . pachyrhizi感染(49]。收集1株转基因株系和2株野生株系(2月龄)3片充分展开的叶片,用无菌蒸馏水冲洗,切成5 cm × 5 cm的薄片。接种用1 mL脲孢子悬液(105孢子/mL)滴到每个叶片上,随后将叶片背面朝上放置在覆盖湿滤纸的培养皿中。材料在20°C下12/12小时光/暗循环下孵育。接种后12天,记录病变和脓疱(尿道)数量。一个非参数的学生t的效果进行了比较p . pachyrhizi转基因和非转基因植物。p < 0.05为显著。
附加文件
参考文献
- 1.
杨建平,李志强,李志强:大豆种质资源抗病性评价Phakopsora pachyrhizi.植物健康研究2006年,doi: 10.1094 / php - 2006 - 0104 - 01 - rs。
- 2.
王志强,王志强,王志强:铁锈的影响(Phakopsora pachyrhizi)对昆士兰州东南部大豆产量和品质的影响。澳大利亚农业学报,1999,30:883-893。10.1071 / AR9790883。
- 3.
Bromfield KR:大豆锈病。专著第11号。美国植物病理学学会,圣保罗,明尼苏达州;1984.
- 4.
Patil VS, Wuike RV, Thakare CS, Chirame BB:尿道孢子活力Phakopsora pachyrhizi悉德。在不同的储存条件下。中国农业大学学报。1997,22:260-261。
- 5.
大豆锈病的抗性及其遗传模式。作物科学,1980,20:254-255。10.2135 / cropsci1980.0011183X002000020026x。
- 6.
郑玉文,陈建林:‘台农3号’大豆的选育。台湾农业学报,1968,17:30-35。
- 7.
Hidayat OO, Somaatmadja S:大豆抗锈病育种选育(Phakopsora pachyrhiziSydow)。大豆锈病通报,1977,1:9-22。
- 8.
Singh BB, Thapliyal PN:印度大豆锈病抗性育种。大豆锈病:问题与研究需要。编辑:Ford RE, Sinclair JB。伊利诺伊大学厄巴纳-香槟分校农学院;1977:62 - 65。
- 9.
陈晓明,陈晓明,陈晓明:抗锈病遗传研究(Phakopsora pachyrhizi)。德国农业学报,1980,31:951-956。10.1071 / AR9800951。
- 10.
Hartwig EE, Bromfield KR:大豆特定抗锈病基因之间的关系。作物科学,1983,23:237-239。10.2135 / cropsci1983.0011183X002300020012x。
- 11.
Hartwig EE:鉴定第四个主要基因赋予抗大豆锈病。作物科学,1986,26:1135-1136。10.2135 / cropsci1986.0011183X002600060010x。
- 12.
Monteros MJ, Missaoui AM, Phillips DV, Walker DR, Boerma HR:亚洲大豆锈病“Hyuuga”红褐病抗性基因的定位和确认。作物科学,2007,47:829-834。10.2135 / cropsci06.07.0462。
- 13.
Garcia A, Calvo ES, de Souza KRA, Harada A, Hiromoto DM, Vieira LG:大豆锈病的分子定位(Phakopsora pachyrhizi)抗性基因:新位点和等位基因的发现。应用理论与实践,2008,29(3):344 - 344。10.1007 / s00122 - 008 - 0798 - z。
- 14.
李S, Smith JR, Ray JD, Frederick RD:大豆抗锈病基因PI 567102B的鉴定。应用理论,2012,25(1):1 - 2。10.1007 / s00122 - 012 - 1821 - y。
- 15.
Bonde MR, Nester SE, Austin CN, Stone CL, Frederick RD, Hartman GL, Miles MR:毒力评价Phakopsora pachyrhizi和p . meibomiae隔离。植物科学学报,2006,29(3):344 - 344。10.1094 / pd - 90 - 0708。
- 16.
Sconyers LE, Kemerait RC, Brock J, Phillips DV, Jost PH, Sikora EJ, Gutierrez-Estrada A, Mueller JD, Marois JJ, Wright DL, Harmon CL: 2005年亚洲大豆锈病的发展:美国东南部的视角。在2006年APSnet特色。doi: 10.1094 / apsnetfeatures - 2006 - 0106。
- 17.
van de Mortel M, Recknor JC, Graham MA, Nettleton D, Dittman JD, Nelson RT, Godoy CV, Abdelnoor RV, Almeida AMR, Baum TJ, Whitham SA:对亚洲大豆锈病感染的明显双相mRNA变化。植物与微生物的相互作用,2007,20:887-899。10.1094 / mpmi - 20 - 8 - 0887。
- 18.
潘西DR,袁JS, Wright DL, Marois JJ, Mailhot D, Stewart CN:大豆对亚洲大豆锈病致病因子的基因表达分析(Phakopsora pachyrhiziSydow)生长初期。中国生物医学工程学报,2007,27(3):344 - 344。10.1007 / s10142 - 007 - 0045 - 8。
- 19.
Panthee DR, Marois JJ, Wright DL, Narvaez D, Yuan JS, Stewart CN:大豆对亚洲大豆锈病病原基因的差异表达(Phakopsora pachyrhiziSydow)是大豆生长阶段特有的。应用理论,2009,18(3):359- 366。10.1007 / s00122 - 008 - 0905 - 1。
- 20.
崔俊杰,alharouf NW, Schneider KT, Matthews BF, Frederick RD:大豆抗病性的表达模式Phakopsora pachyrhizi揭示过氧化物酶和脂氧合酶的重要性。中国生物医学工程学报。2008,8:341-359。10.1007 / s10142 - 008 - 0080 - 0。
- 21.
张晓峰,李晓峰,李晓峰,李晓峰,李晓峰,李晓峰,李晓峰:大豆对巨噬菌侵染的转录组分析。中国植物科学,2010,39(3):344 - 344。10.1016 / j.plantsci.2010.04.011。
- 22.
Schneider KT, van de Mortel M, Bancroft TJ, Braun E, Nettleton D, Nelson RT, Frederick RD, Baum TJ, Graham MA, Whitham SA:诱导双相基因表达变化Phakopsora pachyrhizi在大豆中与真菌渗透和吸器的形成有关。中国生物医学工程学报,2011,27(3):344 - 344。10.1104 / pp.111.181149。
- 23.
潘迪SP, Somssich IE: WRKY转录因子在植物免疫中的作用植物科学进展,2009,30(4):457 - 457。10.1104 / pp.109.138990。
- 24.
Eulgem T, Rushton PJ, Robatzek S, Somssich IE:植物转录因子WRKY超家族。植物科学进展,2000,5:199-206。10.1016 / s1360 - 1385(00) 01600 - 9。
- 25.
谢震,张志林,邹霞,黄娟,Ruas P, Thompson D,沈庆军:水稻WRKY基因超家族的注释与功能分析,揭示糊粉层细胞脱落酸信号的正、负调控因子。植物科学进展,2005,27(3):344 - 344。10.1104 / pp.104.054312。
- 26.
张艳,王玲:WRKY转录因子超家族的起源及其在植物中的扩增。中国生物医学工程学报,2005,5:1-12。10.1186 / 1471-2148-5-1。
- 27.
吴克林,郭志军,王海辉,李娟:水稻和拟南芥WRKY转录因子家族及其来源。DNA决议,2005,12:9-26。10.1093 / dnares / 12.1.9。
- 28.
Ross CA,刘勇,沈庆杰:水稻WRKY基因家族(栽培稻)。中国生物工程学报,2007,29(3):344 - 344。10.1111 / j.1744-7909.2007.00504.x。
- 29.
何海生,董强,邵永华,蒋海燕,朱文文,程斌,向勇:白杨WRKY基因家族的全基因组调查与鉴定。植物细胞代表2012年,doi: 10.1007 / s00299 - 012 - 1241 - 0。,
- 30.
温峰,朱辉,李鹏,姜敏,毛伟,王超,褚震:短柄藻wrky家族基因的全基因组进化特征及表达分析。DNA Res2014年,1-13。doi: 10.1093 / dnares / dst060。,
- 31.
张勇,冯俊:葡萄WRKY家族的鉴定与鉴定。生物医学研究国际。文章的ID。2014, 787680: 14-
- 32.
窦玲,张旭,庞超,宋敏,魏华,樊胜,余胜:棉花WRKY基因家族的全基因组分析。Mol基因基因组学doi: 10.1007 / s00438 - 014 - 0872 y。
- 33.
Ülker B, Somssich即:WRKY转录因子:从DNA结合到生物学功能。植物学报,2004,7:491-498。10.1016 / j.pbi.2004.07.012。
- 34.
Yamasaki K, Kigawa T, Inoue M, Tateno M, Yamasaki T, Yabuki T, Aoki M, Seki E, Matsuda T, Tomo Y, Hayami N, Terada T, Shirouzu M, Tanaka A, Seki M, Shinozaki K, Yokoyama S:拟南芥WRKY DNA结合域的溶液结构。植物学报,2005,17:944-956。10.1105 / tpc.104.026435。
- 35.
Maeo K, Hayashi S, Kojima-Suzuki H, Morikami A, Nakamura K: WRKY结构域保守残基在烟草WRKY家族蛋白dna结合中的作用。生物工程学报,2001,29(3):344 - 344。10.1271 / bbb.65.2428。
- 36.
Eulgem T, Somssich IE: WRKY转录因子在防御信号中的网络。植物学报,2007,10:366-371。10.1016 / j.pbi.2007.04.020。
- 37.
Ciolkowski I, Wanke D, Birkenbihl RP, Somssich IE: WRKY转录因子的dna结合选择性研究为WRKY结构域功能的研究提供了结构线索。中国生物医学工程学报,2008,29(3):344 - 344。10.1007 / s11103 - 008 - 9353 - 1。
- 38.
周强,田安,邹宏,谢震,雷刚,黄娟,王超,王辉,张娟,陈松:大豆wrky型转录因子基因,通用汽车WRKY13,通用汽车WRKY21,通用汽车WRKY54基因使转基因拟南芥植株对非生物胁迫具有不同的耐受性。中国生物工程学报,2008,6:486-503。10.1111 / j.1467-7652.2008.00336.x。
- 39.
黄松,高原,刘娟,彭霞,牛霞,费智,曹松,刘颖:番茄WRKY转录因子的全基因组分析。中国生物医学工程学报,2012,27(3):344 - 344。10.1007 / s00438 - 012 - 0696 - 6。
- 40.
张H,金JP,唐L,赵Y,顾我,高G,罗JC: PlantTFDB 2.0:综合植物转录因子数据库的更新和改进。中国生物医学工程学报,2011,29(4):344 - 344。10.1093 / nar / gkq1141。
- 41.
王忠,李宝明,程俊,王志勇,阮红,徐东,程杰:大豆转录因子知识库的构建。植物学报,2010,10:14-26。10.1186 / 1471-2229-10-14。
- 42.
大豆转录因子知识库。[], [http://www.igece.org/Soybean_TF/]
- 43.
张玲,王霞,毕勇,张超,范勇,雷伟:转录因子的分离与功能分析GmWRKY57b从大豆。科学通报,2008,53:3538-3545。10.1007 / s11434 - 008 - 0483 - 2。
- 44.
姜树生,朴e,杜ks:病原菌诱导的鉴定大豆转录因子GmWRKY1.植物病理学杂志,2009,25:381-388。10.5423 / PPJ.2009.25.4.381。
- 45.
伊格希:……http://systemsbiology.usm.edu/BrachyWRKY/WRKY/Soybean.html]
- 46.
LGE genômica e expressão。[], [http://www.lge.ibi.unicamp.br/soja/]
- 47.
Severin AJ, Woody JL, Bolon Y, Joseph B, Diers BW, Farmer AD, Muehlbauer GJ, Nelson RT, Grant D, Specht JE, Graham MA, Cannon SB, May GD, Vance CP, Shoemaker RC: RNA-Seq图谱大豆:大豆转录瘤指南。植物学报,2010,10:160-176。10.1186 / 1471-2229-10-160。
- 48.
Grant D, Nelson R, Cannon S, Shoemaker R: SoyBase, USDA-ARS大豆遗传学和基因组数据库。核酸研究,2010,38(数据库issue): D843-846。10.1093 / nar / gkp798。
- 49.
崔泽依马纳M,黄文杰,黄文杰,黄文杰,黄文杰,等。大豆抗锈病的分离叶方法研究。铁锈研讨会。2006年全国大豆锈病研讨会论文集,圣路易斯,2006。
- 50.
污物J,大炮某人,马Schlueter J, J, Mitros T,纳尔逊W,海顿耐DL,歌Q, JJ,泰伦程J,徐D, Hellsten U, GD,于Y,樱井T, Umezawa T, Bhattacharyya可,Sandhu D, Valliyodan B,林奎斯特E,皮托,格兰特D,蜀年代,古德斯坦D,巴里·K Futrell-Griggs M B,阿伯纳西Du J,田Z,朱L,吉尔N, Joshi T, Libault M, Sethuraman,张XC, Shinozaki K,阮HT,翅膀RA, Cregan P, Specht J, Grimwood J, Rokhsar D,斯泰西G,鞋匠RC,杰克逊山:古多倍体大豆基因组序列。自然科学进展,2010,38(4):344 - 344。10.1038 / nature08670。
- 51.
Freeling M:植物基因含量在不同类型复制后的偏倚:串联、全基因组、节段或转位。植物学报,2009,30(4):433-453。10.1146 / annurev.arplant.043008.092122。
- 52.
cartero - paulet L, Galstyan A, rogg - villanova I, Martinez-Garcia JF, Bilbao-Castro JR, Robertson DL:拟南芥、杨树、水稻、苔藓和藻类中bHLH转录因子家族的基因组分类和进化分析。中国生物医学工程学报,2010,29(3):344 - 344。10.1104 / pp.110.153593。
- 53.
陈晓霞,韩s,张华,陈晓霞,陈晓霞,陈晓霞,陈晓霞,陈晓霞:烟草转录因子对茄科植物转录调控的研究。中国生物医学工程学报,2008,27(3):344 - 344。10.1104 / pp.107.114041。
- 54.
Rushton PJ, Somssich IE, Ringler P, Shen QJ: WRKY转录因子。植物科学进展,2010,15:247-258。10.1016 / j.tplants.2010.02.006。
- 55.
Mangelsen E, Kilian J Berendzen千瓦,Kolukisaoglu呃,哈特K,简颂C,万科D:系统发育和大麦比较基因表达分析(大麦芽WRKY转录因子家族揭示了单子叶和双子叶之间保留的功能。中国生物医学工程学报,2008,9:194-211。10.1186 / 1471-2164-9-194。
- 56.
van Verk MC, Pappaioannou D, Neeleman L, Bol JF, Linthorst HJM:水杨酸和细菌激发子诱导PR-1a基因表达需要一种新的WRKY转录因子。中国农业科学,2008,29(3):344 - 344。10.1104 / pp.107.112789。
- 57.
杨波,姜艳,Rahman MH, Deyholos MK, Kav NNV:油菜WRKY转录因子的鉴定与表达分析(芸苔属植物显著L.)对真菌病原体和激素治疗的反应。中国生物医学工程学报,2009,9:68-87。10.1186 / 1471-2229-9-68。
- 58.
Giacomelli JI, Ribichich KF, Dezar CA, Chan RL:表达分析表明向日葵WRKY转录因子参与了胁迫响应,系统发育重建揭示了菊科植物中存在一个新的分支。植物科学学报,2010,38(3):339 - 341。10.1016 / j.plantsci.2010.02.008。
- 59.
Pandey AK, Yang C C, Zhang C C, Graham MA, Horstman HD, Lee Y, Zabotina OA, Hill JH, Pedley KF, Whitham SA: Rpp2介导亚洲大豆抗锈病途径的功能分析。植物与微生物相互作用,2011,24:194-206。10.1094 / mpmi - 08 - 10 - 0187。
- 60.
Goellner K, Loehrer M, Langenbach C, Conrath U, Koch E, Schaffrath U:Phakopsora pachyrhizi是亚洲大豆锈病的致病因子。中国植物病理学杂志,2010,11:169-177。10.1111 / j.1364-3703.2009.00589.x。
- 61.
徐旭,陈超,范斌,陈震:病原菌诱导的拟南芥WRKY18、WRKY40和WRKY60转录因子的物理和功能相互作用。植物科学学报,2006,18:1310-1326。10.1105 / tpc.105.037523。
- 62.
陈华昌,赖志,史俊,肖勇,陈志,徐旭:WRKY18、WRKY40和WRKY60转录因子在植物对脱落酸和非生物胁迫响应中的作用。中国生物医学工程学报,2010,10:281-296。10.1186 / 1471-2229-10-281。
- 63.
于东,陈晨,陈震:WRKY DNA结合蛋白在NPR1基因表达调控中的重要作用。植物学报,2001,13:1527-1539。10.1105 / tpc.13.7.1527。
- 64.
王东,董晓霞,王东:植物系统性获得性抗性转录网络调控节点的研究。科学通报,2006,30(4):457 - 457。10.1371 / journal.ppat.0020123。
- 65.
陈超,陈震:植物防御发育调控反应的增强作用在Wrky18,病原体诱导的拟南芥转录因子。植物科学学报,2002,29(3):366 - 366。10.1104 / pp.001057。
- 66.
Ryu H, Han M, Lee S, Cho JI, Sunggi HNR, Lee YH, Bhoo SH, Wang GL, Hahn TR, Jeon JS:水稻WRKY基因超家族在防御反应中的综合表达分析。植物学报,2006,25:836-847。10.1007 / s00299 - 006 - 0138 - 1。
- 67.
左娟,蔡宁:植物基因表达的化学诱导系统。中国生物医学工程学报,2000,29(4):344 - 344。10.1016 / s0958 - 1669(00) 00073 - 2。
- 68.
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。中华分子生物学杂志,1999,15(3):403-410。10.1016 / s0022 - 2836(05) 80360 - 2。
- 69.
麻省理工学院的GENSCAN网络服务器。[], [http://genes.mit.edu/GENSCAN.html]
- 70.
FEGENESH。[], [http://linux1.softberry.com/]
- 71.
Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG: ClustalX窗口界面:由质量分析工具辅助的多序列比对的灵活策略。中国生物医学工程学报,1997,27(4):457 - 457。10.1093 / nar / 25.24.4876。
- 72.
FancyGENE。[], [http://bio.ieo.eu/fancygene/]
- 73.
Edgar RC: MUSCLE:多序列比对,高精度,高通量。中国生物医学工程学报,2004,29(4):344 - 344。10.1093 / nar / gkh340。
- 74.
田村K,彼得森D,彼得森N, Stecher G, Nei M,库马尔S: MEGA5:利用最大似然,进化距离和最大简约方法的分子进化遗传学分析。分子生物学杂志,2011,28:2731-2739。10.1093 / molbev / msr121。
- 75.
Drummond AJ, Rambaut A: BEAST:抽样树的贝叶斯进化分析。中国生物医学工程学报,2007,29(4):344 - 344。
- 76.
Abascal F, Zardoya R, Posada D: ProtTest:蛋白质进化最佳拟合模型的选择。生物工程学报,2005,21:2104-2105。10.1093 /生物信息学/ bti263。
- 77.
分子进化,系统发育和流行病学-无花果树。[], [http://tree.bio.ed.ac.uk/software/figtree/]
- 78.
Matsumura H, Krueger DH, Kahl G, Terauchi R: SuperSAGE:全基因组定量转录谱分析的现代平台。中国生物医学工程学报。2008,9:368-374。10.2174 / 138920108785915157。
- 79.
Robertson N, ovesi - fordorei M, Zuyderduyn SD, Varhol RJ, Fjell C, Marra M, Jones C, Siddiqui A: DiscoverySpace:一个交互式数据分析应用程序。中国生物工程学报,2007,8:R6-10.1186/gb-2007-8-1 r6。
- 80.
LGE Genômica e Expressão。[], [http://www.lge.ibi.unicamp.br/soja/]
- 81.
Ribeiro AS, Moreira JUV, Pierozzi PHB, Rachid BF, Toledo JFF, Arias CAA, Soares RM, Godoy CV:大豆亚洲锈病的遗传控制。《植物学报》,2007,27(3):344 - 344。10.1007 / s10681 - 007 - 9404 - 8。
- 82.
Kerk NM, Ceserani T, Tausta SL, Sussex IM, Nelson TM, Ceserani T:植物组织细胞的激光捕获显微解剖。植物科学学报,2003,27(3):344 - 344。10.1104 / pp.102.018127。
- 83.
Cai S, Lashbrook CC:从带转移石蜡切片中激光捕获植物细胞的显微解剖促进了结构完整的RNA的恢复,用于全球基因分析。植物学报,2006,38(4):344 - 344。10.1111 / j.1365 - 313 x.2006.02886.x。
- 84.
Trapnell C, Pachter L, Salzberg SL: TopHat:发现RNA-Seq剪接连接。中国生物医学工程学报,2009,26(3):344 - 344。10.1093 /生物信息学/ btp120。
- 85.
李锐,于超,李勇,林文伟,姚明生,王娟:SOAP2:一种改进的短读对齐超快工具。生物信息学,2009,25:1966-1967。10.1093 /生物信息学/ btp336。
- 86.
Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B:利用RNA-Seq绘制和量化哺乳动物转录组。光子学报,2008,29(3):344 - 344。10.1038 / nmeth.1226。
- 87.
Biotecsur。[], [http://bioinfo.cnpso.embrapa.br/biotecsoja/sistema_autenticacao/login.php]
- 88.
Kim KS, Unfried JR, Hyten DL, Frederick RD, Hartman GL, Nelson RL, Song Q, Diers BW:大豆抗锈病基因PI 561356的分子定位及单倍型SNP分析Rpp1区域内种质资源多样。应用理论与实践,2012,29(4):457 - 457。10.1007 / s00122 - 012 - 1932 - 5。
- 89.
Libault M, Thibivilliers S, Bilgin DD, Radwan O, Benitez M, Clough SJ, Stacey G: 4个大豆内参基因的鉴定。植物基因组学报,2008,1:44-54。10.3835 / plantgenome2008.02.0091。
- 90.
Livak KJ, Schmittgen TD:利用实时定量PCR和2(-Delta Delta C(T))分析相关基因表达数据。方法,2001,25:402-408。10.1006 / meth.2001.1262。
- 91.
Karimi M, Inze D, Depicker A: GATEWAY向量农杆菌属介导植物转化。植物科学进展,2002,7:193-195。10.1016 / s1360 - 1385(02) 02251 - 3。
- 92.
大豆转基因可育植株(Glycine max (L.))梅里尔)从轰击胚胎发生组织中获得。《自然科学》,2002,27(3):366 - 366。10.1023 /: 1020370913140。
- 93.
Finer JJ, Vain P, Jones MW, McMullen MD:用于DNA传递到植物细胞的颗粒流入枪的开发。植物科学进展,2002,23(4):366 - 366。10.1007 / BF00233358。
- 94.
Wiebke-Strohm B, Droste A, Pasquali G, Osório MB, Bucker-Neto L, Passaglia LMP, Bencke M, Homrich MS, Margis-Pinheiro M, Bodanese-Zanettini MH:通过无dna粒子轰击结合转化体细胞胚获得的转基因可生育大豆植株农杆菌属系统。《植物科学》,2011,27(3):344 - 344。10.1007 / s10681 - 010 - 0249 - 1。
- 95.
Doyle JJ, Doyle JL:一种用于少量新鲜叶片组织的快速DNA分离程序。植物化学通报,1987,19:11-15。
致谢
我们感谢来自南联邦大学“Núcleo de Assessoria Estatística”的Elsa Mundstock博士和Gilberto P. Mesquita博士的统计支持;Cláudia Godoy博士提供真菌分离物,Emerson Del Ponte博士,Cláudia Godoy博士,Juliano dos Santos博士,Larissa Bittecourt和Silvia Richter提供技术援助。这项工作得到了国家生物科学委员会Científico e Tecnológico和Consórcio国家大豆Genoma da Soja (CNPq-GENOSOJA)和BIOTECSUR(欧盟/南方共同市场)的资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们没有竞争利益。
作者的贡献
构思设计实验:MB-M, BW-S, LBN, MM-P, MHB-Z, APC。进行实验:MB-M、BW-S、FCM-G、MCCGdeC、RS、MBO。载体构建:MSH。进行数据分析:MB-M, CC, BW-S, MCCGdeC, LBN, EM, GW, ACT-Z, MSH, RLMW。撰写论文:MB-M, BW-S, LBN。修改论文:MM-P, MHB-Z, RVA, ACT-Z。监督和协调研究:MHB-Z。所有作者都阅读并批准了最终的手稿。
电子辅助材料
注释的
附加文件1:大豆WRKY转录因子(4 ~ 20号染色体)。(docx62 kb)
代表三个物种WRKY蛋白之间关系的系统发育树。
附加文件3:使用贝叶斯(BA)方法重建树。共得到289个氨基酸序列拟南芥甘氨酸max和杨树trichocarpaWRKY域对应的位点有65个。后验概率值标记在分支上方,仅显示高于70%的值。表示I、IIa、IIb、IIc、IId、IIe和III组。*差异表达基因的反应p . pachyrhizi感染。(pdf 782 kb)
表达模式
附加文件4:WRKY编码基因在p . pachyrhizi感染(IIb ~ III组)。(docx30kb)
大豆转基因株系过表达的研究
附加文件5:GmWRKY27.野生型中GFP的表达分析(一)以及抗湿霉素胚胎发生组织(B).在蓝光下使用Olympus®荧光立体显微镜检测GFP的表达,并配备了包含488 nm激发滤波器和505-530 nm发射滤波器的BP滤波器组。(C)表达水平(RT-qPCR)GmWRKY27在野生型(WT)大豆植株和不同转基因大豆株系的组织分化胚胎中。Venc (BRSMG68 venencedora) P2-1、as -5 P1-1、Conq (MGBR-46 Conquista) P1-1系来自Biobalistic,而as -5 P3-1系来自Biobalistic/农杆菌属转换实验。F-Box蛋白和金属蛋白酶内参基因作为内对照,使每个样品中mRNA的含量归一化。利用wt中WRKY基因的转录水平来校准转基因胚胎中的转录量。* wt和转基因株系的平均值显著不同(学生的t-test, p < 0.05)。(pdf 140kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。
本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。
如欲查看本牌照的副本,请浏览https://creativecommons.org/licenses/by/4.0/.
创作共用公共领域奉献弃权书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
本克-马拉托,M.,卡布雷拉,C.,维伯克-斯特罗姆,B.。et al。大豆WRKY家族的全基因组注释和参与响应的基因的功能表征Phakopsora pachyrhizi感染。BMC植物生物学14日,236(2014)。https://doi.org/10.1186/s12870-014-0236-0
收到了:
接受:
发表:
关键字
- 大豆
- 遗传转化
- 真菌耐药性
- 转录因子
- 亚洲大豆锈病
- 功能分析