摘要
背景
提高抗病性是谷物育种计划的一个关键目标,因为疾病暴发继续威胁着全球粮食生产,特别是在非洲。在抗病基因家族中,核苷酸结合位点+富亮氨酸重复序列(NBS-LRR)家族是最常见和最古老的,也是已知植物中最大的基因家族之一。通过与水稻和玉米进行比较,并与真菌病原体抗性QTL进行比较,研究了高粱(非洲重要的主食)中nbs编码基因的序列多样性。
结果
在高粱中,nbs编码基因与非nbs编码基因相比具有更高的多样性,并且通过驯化和改良在提纯和平衡选择的基因组区域显著富集。祖先基因在物种分化之前,在有选择特征的区域比没有选择特征的区域更丰富。高粱nbs编码基因在真菌病原抗病QTL的基因组区域也显著富集;nbs编码基因的多样性受共定位生物抗逆性QTL类型的影响。
结论
在高粱中,nbs编码基因承受着强大的选择压力,通过净化和平衡选择的对比进化过程。这种截然不同的进化过程对祖先基因的影响大于对物种特定基因的影响。基因组中对多种病原体具有抗性的真菌抗病热点,除了为植物育种家提供快速跟踪开发具有更持久病原体抗性的高性能品种的选择目标外,还可以进一步了解谷物在与快速进化的病原体“军备竞赛”中使用的机制。
背景
禾草,包括主要谷物小麦、大麦、玉米、水稻和高粱,是最重要的农艺和经济品种,总共养活了世界上三分之二以上的人口[1].然而,这些作物的生产受到对全球人类粮食供应构成重大威胁的病原体的挑战。全球至少30%的粮食产量因病原体而损失[2]、[3.]疾病爆发的影响在发展中国家可能尤其严重[4].在谷物中,高粱是非洲和亚洲半干旱热带地区5亿多人口的主食,除了是牲畜饲料的重要来源外,它是最适应干旱和高温的谷物之一,在应对世界不断增长的人口的挑战方面将发挥越来越重要的作用。然而,其生产力经常受到叶片真菌疾病的影响。最有利可图和可持续的疾病最小化策略是种植具有遗传抗性的品种;因此,抗病选择几乎是所有植物育种计划的一个关键组成部分。
在所有抗病基因中,核苷酸结合位点+富亮氨酸重复基因(NBS-LRR)是最常见和最古老的,是植物中已知的最大的基因家族之一[5].这些基因参与检测和应对各种病原体,包括细菌、病毒、真菌、线虫、昆虫和卵菌[5].NBS-LRR基因编码n端可变结构域、中央核苷酸结合位点(NBS)结构域和c端富亮氨酸重复序列(LRR)结构域[6].此外,基于n端Toll/白细胞介素-1受体(TIR)结构域的分类将nns编码基因分为TIR和非TIR亚类,尽管先前的研究表明TIR亚类在谷类和一般单子叶植物中代表性不足[7]、[8].
nbs编码基因最显著的结构特征是LRR结构域数量可变,有些基因完全缺乏LRR编码结构域[8].这些结构域是高度可变的区域,被认为负责识别病原体编码的配体[9].相比之下,参与信号传递的NBS结构域包括几个高度保守和严格有序的基序[10].
先前的研究发现,植物基因组中nbs编码基因的数量变化很大,例如,拟南芥中大约有150个[11]到将近500吨的大米[12],高粱的营养含量介于211 [13]及348 [14NBS-LRR基因。据推测,如此快速的拷贝数进化是由一个物种内的基因丢失或扩张所驱动的,这种基因丢失或扩张是通过重复的复制、分化和最终的假基因形成或缺失来应对不同的病原体[15].这些基因被认为处于持续的等位基因选择压力下,从而使植物抵御病原体的攻击。初步研究表明,编码nbs的基因比非编码nbs的基因更常成为选择的目标[16],但这些编码nbs的基因显示了与不同类型选择作用一致的分子证据。一些进化相对缓慢,而另一些则表现出与快速进化相关的典型模式,包括多个和可变的拷贝数,非同义替换与同义替换的高比例,以及高水平的物种内多态性[17].
若干谷类作物全基因组序列的可用性[13]、[18]、[19]引发了一系列评估物种内全基因组序列多态性的新研究[20.] - [22].最近一项高粱重测序研究[23]为44种野生、杂草和栽培基因型的不同群体生成了高覆盖率(>20×)数据,涵盖地理起源、作物管理和亚群/种等维度。本研究利用这一资源提供了新的机会来探索高粱野生型和杂草型基因型中nbs编码基因的进化可塑性和由此产生的变异,与栽培基因型相比:1)先前确定的基因组区域在驯化和改良过程中处于选择状态;(2)高粱真菌病原抗病QTL;(3)与玉米、水稻共有的祖先基因家族。这些见解将为阐明nbs编码基因家族如何成为谷物基因组中最重要的病原体监测系统提供新的线索。它还将为育种家提供新的知识和工具,用于估计抗性种质的丰富度和针对特定的基因组区域,以便更有效地利用这些资源。
结果
高粱nbs编码基因的多态性分析
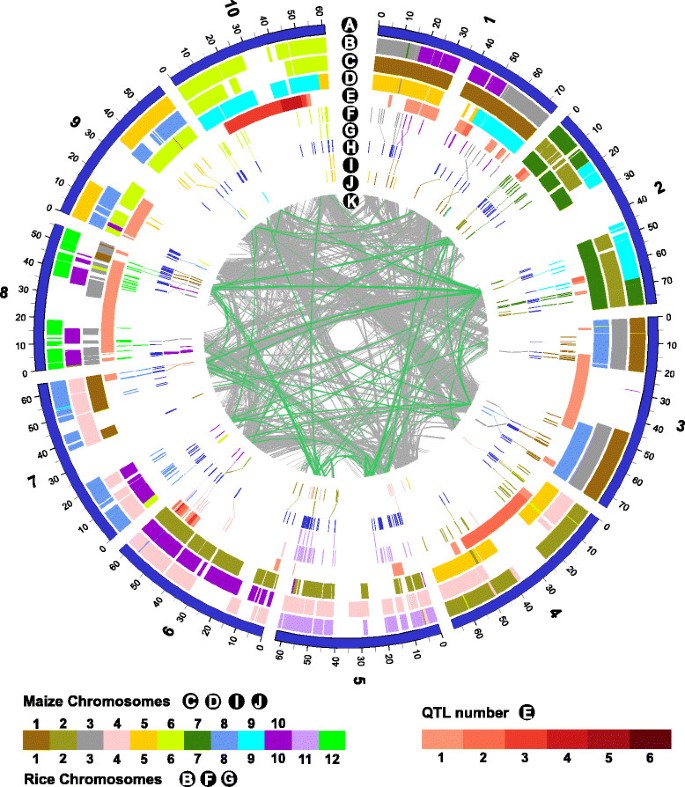
在约700 Mb的高粱基因组中,共鉴定出346个编码NBS的基因,这些基因具有高度保守的NBS区域,占高粱参考基因组中所有预测基因模型的1.2% [13](附加文件1:表S1),与最近对高粱的研究相当[14]鉴定出348个nbs编码基因。根据n端和LRR结构域的序列相似性,346个基因可分为14种不同的NBS类型(表2 - 4)1);其中228个为LRR域。nbs编码基因在整个基因组中分布不均匀(图1),超过60%位于3条染色体上(SBI-02, SBI-05和SBI-08)。此外,超过三分之二的nbs编码基因(68.7%)位于染色体上的簇(附加文件)2:表S2)。nbs编码基因在包含真菌病原抗病QTL (Χ)的基因组区域显著富集2p -值0.00272)。此外,nbs编码基因在被鉴定为纯化选择的基因组区域显著富集。这种模式是通过驯化(Χ2p -值0.000539)和改进(Χ2p -值0.0000046),其特征是野生、长地种和改良类群之间的分化增强,核苷酸多样性低,等位基因频谱呈负偏[23].在平衡选择条件下,nbs编码基因也在染色体区域富集(Χ2p -值为0.0323),与净化选择的多样性和分化特征形成对比。作为比较,高粱与拟南芥中鉴定的176个管家基因同源基因的分布[24]进行了分析,发现在被鉴定为净化或平衡选择的基因组区域中没有显著富集(Χ2p -值分别为0.956和0.202)或在含有真菌病原抗病QTL的基因组区域(Χ2p -值0.079)。
nbs编码基因的多态性模式也不均匀地分布在整个基因组中。总体而言,nbs编码基因具有显著较高的多样性(P< 3.18e-9通过配对t检验),与高粱筛选基因集中随机选择的346个非nbs编码基因进行比较。在高粱筛选的基因集中,nbs编码基因在核苷酸多样性测量值(θπ)经验分布的上5%尾部显著富集(n = 29,346)。这一模式观察到所有三组(Χ2p -值<0.0001)(图2);野生和杂草,地方种族和改良的自交系。基因型组之间的差异还观察到nbs编码基因的核苷酸多样性水平(附加文件)2:图S1)与野生和杂草基因型相比,栽培组的多样性始终较低,这是先前在基因区和非基因区全基因组范围内的观察结果[23].相对于所有基因型组,NBS基因多样性的显著减少,仅在高粱基因型的改良自交系组中观察到(θπ经验分布的低5%尾部富集;Χ2p -值0.046)(图2).
总的来说,只有不到20%的nbs编码基因具有与选择作用一致的分子变异模式,通过倾斜多样性(θπ和θw)、等位基因频谱(Tajima’s D)和组间分化(FST)值来测量。在这些nbs编码基因中,有超过一半(38个)的基因具有净化选择的特征,通过自然和人类介导的选择,驱动有益的等位基因固定和选择性去除有害的等位基因。11个nbs编码基因在栽培组和野生组中完全不变,另有10个基因仅在栽培组中固定(附加文件)1:表S1)。这些不变基因大部分(86%)出现在基因簇中。mlHKA检验用于验证编码nbs的驯化和改良候选者是否表现出与正选择一致的遗传变异模式[25].定向选择模型较好地解释了17个驯化和改良变异候选基因相对于38个中性位点的多态性与分化的模式(平均对数似然比检验统计量= 372;P所有比较均< 0.0001;额外的文件2:表S3)。相比之下,23个nbs编码基因的分子特征与平衡选择一致,其中多个等位基因在祖先和后代群体中保持在中间频率。mlHKA检验发现,平衡选择模型最好地解释了这23个编码nbs的驯化和改良候选者的遗传变异模式(平均对数似然比检验统计量= 493.2;P所有比较均< 0.0001;额外的文件2:表S3)。在23个具有平衡选择特征的nbs编码基因中,超过90%的基因具有LRR结构域,而在38个具有净化选择特征的nbs编码基因中,只有52%的基因具有LRR结构域2:图S2)。总的来说,具有LRR结构域的NBS基因比不具有LRR结构域的NBS编码基因更具多样性,无论是在培养组(具有LRR结构域的θπ = 0.00278 vs不具有LRR结构域的θπ = 0.002203)还是在野生组和杂草组(具有LRR结构域的θπ = 0.00384 vs不具有LRR结构域的θπ = 0.00364)。多样性也随着三组LRR域数量的增加而增加(附加文件)2:图S3)。
在纯化和平衡选择下,nbs编码基因在两个区域的多样性更大2),与具有选择特征的非nbs编码基因相比,提供了nbs编码基因在选择后增加多样性的证据。选择后的多样性恢复程度也随编码nbs基因类型的不同而不同,编码nbs基因的N类在改良自交系组中多样性恢复量最大,CNL类在长白种组中多样性恢复量最大,XN类在野生和杂草组中多样性恢复量最大(补充文件)2:图S4)。尽管在少数群体中,在纯化选择的区域中,也有编码nbs基因的多样性低于非编码nbs基因的例子。在所有情况下,在与真菌病原生物胁迫QTL共存的选择区域,nbs编码基因的多样性低于非nbs编码基因,特别是抗炭疽病和抗锈病(附加文件)2:图s4 -6)。
与纯化选择下的nbs编码基因相比,具有平衡选择特征的nbs编码基因具有更高数量的蛋白质变异(分别为7.6对4.3),这与平衡选择下维持过多多态性的预期一致。此外,Ka:Ks比值检验比较了非同义替换的数量(潜在的适应性氨基酸替换变化)与同义替换的数量(假设是中性进化),进一步证明了适应性替换在具有平衡选择特征的nbs编码基因中以更高频率积累,而与具有净化选择特征的nbs编码基因相比(表2)3.),这可能是由于与中性位点相比,有利的非同义突变更频繁地发生。相比之下,非nbs编码基因的Ka:Ks比值值在整个基因组中始终较低,无论是处于选择状态还是中性状态。
在所有346个nbs编码基因中,非功能性等位基因的发生,无论是通过框架转移还是大效应snp(过早终止密码子,开始密码子到非开始密码子,停止密码子到不间断,剪接位点),在高粱基因型中从2.17%到86.95%不等,与栽培组相比,野生和杂草基因型的频率更高(附加文件)2:表S4)。
与纯化选择或中性期望下进化的nbs编码基因相比,具有平衡选择特征的nbs编码基因也具有更高的非功能等位基因的总体比例(附加文件)2:图S7)。在大多数情况下(19/23个处于平衡选择的基因),不到50%的基因型受到框架转移或大效应snp的影响,多种功能蛋白变体仍然存在。在纯化选择下,nbs编码基因中很少出现非功能等位基因。高粱中4个编码nbs的基因(约1.2%)似乎是伪基因,即所有等位基因都因帧移位或大量缺失(>50%的基因)而丧失功能。其中一种基因(Sb05g007560;NXL分类)也具有平衡选择的特征,并且在高粱过滤基因集中的所有基因的核苷酸多样性测量(θπ)的经验分布的上1%尾部,横跨栽培和野生高粱基因型。该基因没有与先前鉴定的真菌病原体生物胁迫抗性QTL共定位;然而,在纯化选择和平衡选择条件下,与nbs编码基因共定位的真菌病原生物抗性QTL类型发生了分化。在纯化选择中,与nbs编码基因共定位的QTL中,近80%与抗锈病或炭疽病相关。与nbs编码基因共定位的QTL中,80%以上与麦角菌抗性相关,而与锈病和炭疽病基因共定位的QTL中仅有1个与麦角菌抗性相关。
为了研究选择性横扫的特征,分析了nbs编码基因侧边100 kb区域的基因多样性,发现与全基因组平均水平相比,基因多样性显著降低;侧翼基因富集在所有三组核苷酸多样性的低5%尾部(附加文件)2:图S8)。总共有69个nbs编码基因位于先前在高粱中筛选的候选基因的100 kb内[23],包括先前描述的驯化基因,如涉及分蘖角的LA1 [26]和与果皮颜色有关的Rd [27].
高粱同源基因、副同源基因和新型nbs编码基因的多态性
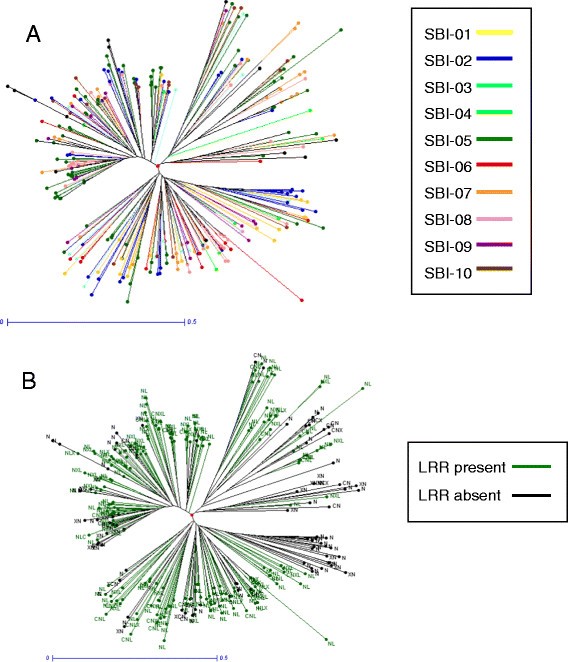
为高粱、玉米和水稻物种内部和之间的nbs编码基因构建了系统发育树(图3.和附加文件2:图S9-11),允许跨种(同源)基因家族的鉴定。根据支间核苷酸相似度<70%的支定义(支中的基因被鉴定为多基因家族),共鉴定出647支支;137个是多基因家族分支,共包含404个nbs编码基因;其中85个属于20个祖先基因家族,早于物种分化(附加文件)2:图S12)。玉米祖先基因所占比例最高(21.1%),其次是高粱(8.8%)和水稻(6.3%)。祖先基因家族主要出现在种间共线基因簇中(67.1%)。总的来说,在高粱中,带有选择(纯化或平衡)特征的nbs编码基因比中性的nbs编码基因更有可能是同源的,没有被选择(附加文件)2:图S13)。在高粱中,具有平衡选择特征的NBS编码基因与中性选择或纯化选择的NBS基因相比,祖先基因家族成员的比例最高。
高粱nbs编码基因的系统发育树。一个.按指示由染色体编码的颜色。B.根据有无LRR结构域进行颜色编码,如表所示,基因字母编码详见表1.
对高粱基因家族进行更详细的系统发育分析表明,在70%的核苷酸相似性和基因覆盖率下,高粱中大约三分之一的nbs编码基因属于同源的多基因家族(附加文件)2:图S14)。大多数(76%)的副同源基因位于同一基因簇内,尽管超过12%的基因家族具有跨多条染色体的副同源基因(附加文件)2:图S15)。高粱中55%的副同源nbs编码基因也出现在最近重复的基因组超基因区域,定义为4DTv < 0.497 [23].总体而言,与非副同源的单基因(0.0028;额外的文件2图S16),在高粱中,副亲缘nbs编码基因被选择的比例是单株nbs编码基因的两倍。此外,Ka:Ks比值测试在副同源nbs编码基因中发现了比单例基因中更多的非同义snp(附加文件)2:图S17)。
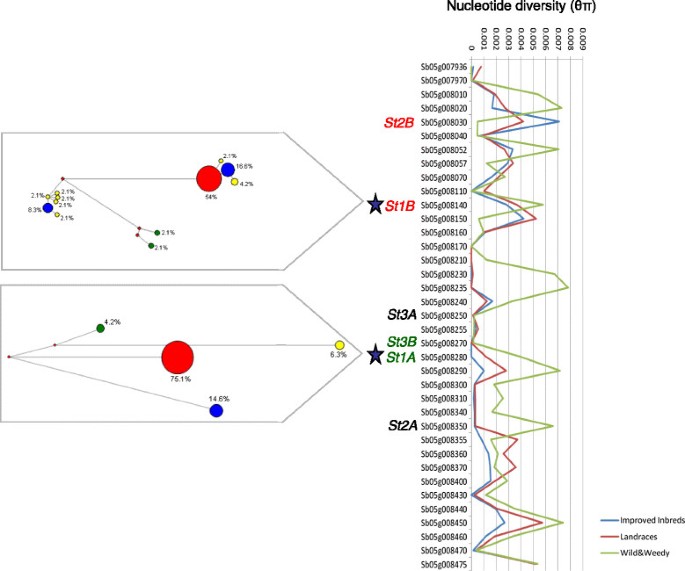
通过比较水稻中nbs编码基因的选择,确定了跨物种的同步选择横扫[21]和玉米[22]从以前的研究。总的来说,从75个野生、地方和改良玉米品系的重测序数据分析中鉴定出的1508个改良候选基因和1764个驯化候选基因[22],鉴定出6个玉米nbs编码基因处于纯化选择状态(5个为改良基因,1个为驯化基因)。这些玉米nbs编码基因与5个高粱nbs编码基因同源,包括抗炭疽病候选基因SbCg1SBI-05。在最近一项关于大米的类似研究中[21],分析了40个栽培祖细胞和10个野生祖细胞的重测序数据,在人工选择下鉴定出2506个候选基因,其中8个为nbs编码基因。水稻nbs编码基因全部位于11号染色体上的3个基因簇,与高粱SBI-05上的3个基因簇同源SbCg1候选基因反对Setosphaeria turcica玉米北叶枯病的致病因子[28].高粱基因对St1A而且St1B确定(28]属于在高粱、玉米和水稻中发现的20个祖先基因家族之一,其中包括玉米同源基因,发现在真菌挑战后转录物上调Setosphaeria turcica。而同源玉米基因未被鉴定为处于选择状态[22],在这个祖先基因家族中,水稻和高粱的亲缘关系先前已被确定为处于选择状态。在高粱中,有两种截然不同的选择特征St1A而且St1B基因对,与St1A(Sb05g008280)St1B(Sb05g008140)具有平衡选择签名(图4).第二个祖先基因家族也被鉴定为跨物种选择,包括玉米Rp1-D基因(GRMZM5G879178),高粱基因Rp1-dp3 (Sb03g036450)和rph1-3 (Sb08g002410),以及水稻Pi37 (Os01g57310)基因,用于抗稻瘟病。最近,这被确定为一个快速进化的基因家族,称为“Rp1/Pi37”[17],它有一种效应反应,可以对跨物种的多种病原体产生抗性。
SBI-05上nbs编码基因簇的核苷酸多样性。该基因簇与水稻(chr 11: Os11g11950, Os11g11960, Os11g11990)和玉米(chr 4: GRMZM2G005347, GRMZM2G005452)的基因簇同向,有6对候选基因对Setosphaeria turcica[28]并以颜色编码(红色文字:处于平衡选择,绿色文字:处于净化选择,黑色文字:未处于选择)。高粱nbs编码基因属于祖先基因家族,在水稻中也被鉴定为纯化选择[21用蓝色的星星突出显示。祖先基因对St1B和St1A详细描述了中值连接网络树。红色节点代表高频单倍型。蓝色节点代表中频单倍型。黄色节点代表低频单倍型。绿色节点表示美国propinquum。
先前在高粱中发现的新基因[23]富集了nbs编码基因(Χ2p -值0.0051)。共鉴定出10个新的nbs编码基因;所有都没有LRR域(其中8个被归为N, 2个被归为CN;额外的文件2:表S5)。四种新基因在任何栽培系中都不存在(附加文件)2:图S18);一个只出现在S.propinquum其中两种只属于野生和杂草类,两种都有一种美国propinquum野生和杂草组。在至少一个栽培组中出现的6个新基因中,有一个(novel_seq4_GLEAN_10000166)在所有基因型组中都高频率出现美国propinquum.所鉴定的新基因均属于高粱特有基因家族。
讨论
本研究以最普遍和最古老的抗病基因家族为重点,提供了新的数据,表明通过纯化和平衡选择的对比进化过程,nbs编码基因在高粱中处于强大的选择压力下,并且它们在与真菌病原体抗病相关的基因组区域富集。本研究还发现,谷物祖先nbs编码基因的多样性低于高粱谱系特有的nbs编码基因的多样性,具有选择特征(净化或平衡)的区域比未受选择特征的区域更丰富。这种对不同类型抗病基因变异模式的认识表明,nbs编码基因家族成员对农业具有真正的价值,可以为植物育种家提供新的工具,以更有效地开发对植物真菌病原体具有更持久抗性的增强型作物品种。
nbs编码基因通过不同的机制在高粱的驯化中发挥作用
驯化综合征传统上与分蘖和种子粉碎等性状有关,迄今为止只有有限的研究支持其与抗病性的关系(例如[29])。目前的研究发现,通过驯化和改良进行纯化和平衡选择的基因组区域富集了nbs编码基因。在选择中,三分之二的nbs编码基因与驯化有关,而不是与改良有关,这可能是受高粱野生近缘的多年生性质的影响,与大多数栽培类型的年度生命周期相反,在面对持续的抗病选择压力时,通过延长植物寿命。也有证据表明,编码nbs的基因在这些区域比非nbs基因更快地恢复和维持多样性。只有少数例外,特别是nbs编码基因与QTL共定位的抗炭疽病基因,这反映了不同病原体的不同选择压力。此外,作为澳大利亚高粱预育种计划初步产量试验的一部分,在昆士兰州对一组539种高级高粱基因型中选择的基因组区域进行了分析,确定了大多数(58.3%)选择的基因组区域与nbs编码基因位于同一位置(数据未显示)。
尽管高粱中约12%的nbs编码基因具有纯化选择的特征,且多样性一致降低,总体上nbs编码基因的核苷酸多样性是非nbs编码基因的三倍(分别为θπ = 0.0031和θπ =0.0018),另有约8%表现出特定的平衡选择特征。这种选择对nbs编码基因影响的异质性此前已被报道[5NBS结构域更常受到纯化选择的影响,而LRR结构域则更可变。这可能反映了LRR结构域在识别不断进化的病原体配体中的作用,以及NBS结构域在识别信号传递中的作用[30.].目前的研究结果与这一发现一致,并证明了超过90%的具有平衡选择特征的nbs编码基因具有LRR结构域,而在纯化选择下的nbs编码基因中,这一比例仅为50%。观察到的核苷酸多样性的增加伴随着每个基因LRR结构域数量的增加,强调了LRR蛋白的高度可变性质。
综合真菌病原病qtl图和nbs编码基因揭示了高粱基因组中与多种定量抗病性状相关的区域
基因组中与真菌病原体相关的生物胁迫抗性QTL区域被发现显著富集nbs编码基因。我们还发现,nbs编码基因的多样性可以根据共定位生物抗逆性QTL的特定类型而变化,例如,炭疽QTL下的nbs编码基因的多样性明显低于栽培高粱中nbs编码基因的平均多样性(分别为θπ = 0.0007和θπ = 0.0026)。
以前的研究在包括水稻在内的其他物种中研究nbs编码基因与抗病QTL的一致性[31]和大豆[32]还发现了基因组QTL部分中nbs编码基因的显著富集。大约36%的高粱基因组涉及对真菌病原体的定量疾病反应(QDR),比最近报道的水稻基因组要少,54% [31].此外,近一半的QDR基因组空间由同一性状的共定位QTL组成,这表明所识别的抗病QTL的鲁棒性。大多数共定位QTL(> ~ 90%)具有对多种疾病的抗性,在SBI-06的长臂上有一个突出的热点区域,包含4项研究中的6个性状的15个QTL [33] - [36].在玉米等其他物种中也发现了这种多重疾病抗性的热点[37],大米[31]、马铃薯[38].据认为,这可能是由于单基因效应,其中抗性基因和QTL是等位基因,或由基因集群的影响。在SBI-06的病害QTL热点区有7个nbs编码基因;2个单基因和1个由5个基因组成的簇,表明单个基因可以提供对多种病原体的抗性或涉及其他抗性机制。研究人员研究了防御相关转录子家族WRKY (n = 69)和MYB基因(n = 110)与高粱真菌病原体相关生物胁迫QTL的对应关系,但没有发现这些基因显著富集(Χ)2p -值分别为0.21和0.052)。此外,高粱基因的分布与拟南芥中鉴定的176个管家基因同源[24]与QDR基因组空间进行了比较,也没有发现显著相关。
有害突变和存在/缺失变异导致了草中nbs编码基因的快速变异
与先前的研究结果相似[13]、[39]、[40],水稻中nbs编码基因的数量(503个)几乎是玉米(137个)的4倍,是高粱(346个;表格1),在高粱、水稻或玉米中均未发现tir编码的NBS基因[39]、[41].通过检查相似性/覆盖阈值中多基因家族的比例,估计了物种之间nbs编码基因扩展的时间差异(附加文件)2:图S19)。根据最近的研究结果[15],与高粱和水稻相比,玉米中nbs编码基因的相似性比例要小得多,在60%到80%之间,这表明在玉米基因组中,最近的重复很可能主导了nbs编码基因,而在高粱和水稻中观察到更多的古老重复。高聚类程度也是三个基因组中nbs编码基因的一个显著特征,与高粱(68.7%)和玉米(51.4%)相比,水稻的nbs编码基因在聚类中比例更高(72.9%),这可能是由于其局部串联重复序列的数量更高。在跨基因组定义的基因簇中,玉米在物种间是100%的同位基因,相比之下,高粱(75.6%)和水稻的同位基因簇比例最低(58%)(附加文件)2:图S20)。与高粱(42.8%)和玉米(43.4%)相比,水稻的种特异性基因簇(72.4%)几乎是其两倍(附加文件)2:图S21)。有趣的是,在高粱中,具有平衡选择特征的nbs编码基因比具有净化选择特征的nbs编码基因或中性进化的nbs编码基因更不可能位于簇中(分别为61%,92%和70%),这可能表明nbs编码基因存在适应度成本。具有纯化选择特征的nbs编码基因的高比例位于集群中,这进一步支持了驱动快速进化和有益等位基因固定的谱系特异性重排。与最近的研究结果一致,与高粱和玉米相比,这种谱系特异性串联复制导致水稻中基因拷贝变异的数量更高[40].这种通过谱系特异性重排的高度动态聚类,可能是通过存在/缺失变异(如基因丢失事件、伪基因化和新基因)和拷贝数变异驱动草中nbs编码基因可塑性的关键机制。
此前也有研究指出,植物抗病基因的可塑性可由基因调控因子介导,包括microRNAs (miRNA) [40].mirna已被证明在真核生物的生长和发育中起着重要的调节作用[42].具体而言,它们已被证明可以调节植物中一些关键胁迫相关基因的表达(例如:[43])。在谷物中,高粱和水稻的nbs编码基因被miRNA靶向的比例(分别为37.5%和36.4%)高于玉米(28.94%)[40].与玉米相比,如此高比例的靶向抗病基因的miRNA可能与水稻和高粱中功能冗余的增加有关,这是通过更高比例的物种特异性nbs编码基因拷贝来实现的;水稻84.6%,高粱71.3%,玉米52.9%。据推测,miRNAs通过在大规模或局部基因组复制事件后抑制表达水平发挥剂量调节作用[40].
先前的研究观察了草种中nbs编码基因的数量,发现nbs位点的核苷酸进化迅速,与家养基因相比,nbs位点的基因丢失或基因数量变异的趋势更大[15]、[39].它被假定为[15自然选择可能是导致水稻、玉米、高粱和短茎类植物中nbs编码基因数量急剧变化的原因,在这些植物中,快速扩张和/或收缩是一个物种适应快速变化的病原体谱的基本重要策略。nbs编码基因与非功能等位基因的高频率可能表明存在与抗病基因相关的适应度成本。例如,在缺乏特定病原体的环境中没有有用功能的nbs编码基因更有可能丢失或通过功能突变的丢失成为假基因,以避免适应度成本。在大多数情况下,没有观察到零等位基因的固定,然而在一个案例中,Sb08g005620中由过早停止SNP引起的零等位基因被发现在整个培养基因型中固定(附加文件)2:图S22)。该基因也具有平衡选择的特征,然而,在培养系中固定零等位基因可能表明了净化选择的双重作用,以驱动新的有利突变频率的增加和平衡选择,以维持不同的等位基因。
以前曾有人猜测[15]多基因家族通过频繁的序列交换促进nbs编码基因的快速进化,从而产生可能编码改变特异性的新基因序列。我们发现多态性增加,特别是非同义多态性增加(多基因家族栽培高粱的Ka:Ks比值为0.74,而多基因家族单基因的Ka:Ks比值仅为0.57)支持这一假设。来自跨物种家族的nbs编码基因在高粱的选择区域更为丰富,这一发现支持了先前的结果(例如[17])的研究表明,编码nbs的基因不仅存在于物种内部,而且存在于物种之间,这些基因可以抵抗快速进化的病原体。
跨物种识别的nbs编码基因进化中的复杂进化动力学
尽管已经有许多研究描述了跨物种的nbs编码基因,但迄今为止,描述物种内nbs编码基因多态性的可比研究非常有限。现有的研究也报告了拟南芥中nbs编码基因的核苷酸多样性较高[16]和大米(例如:[12]、[21]、[44])与全基因组值进行比较。拟南芥的研究[16]特别关注27个nbs编码基因子集的进化动力学,通过对96个LRR域进行测序答:芥并观察到进化过程中可能状态的连续统,包括选择性横扫和介于许多阶段之间的平衡选择。尽管有几个位点可以被确定为最近选择性横扫的候选基因,但他们发现这种情况并不常见,总体上发现了有限的选择性横扫的证据,因此,不支持共同进化军备竞赛假说作为这组基因的一般进化模型。此外,只有微弱的证据被发现的签名平衡选择作用,以维持多个等位基因在中间频率在较长一段时间。然而,最近一项水稻全基因组重测序研究[21],研究了102个nbs编码基因的子集,发现了一些支持栽培水稻选择性扫描的证据,其中23个nbs编码基因显示出明显低于基因组平均水平的多样性。最近的一项研究也证实了水稻中725个nbs编码基因的动态特性[44],结果表明,存在/缺失多态性不仅在不同草种间普遍存在,而且在不同水稻种质间也普遍存在,这是由频繁的缺失和易位引起的。另一项关于稻米的研究[12]还发现了21个栽培水稻和14个野生水稻群体中nbs编码基因亚群的高频率存在/缺失变异,并假设这种变异可能是地理分化的结果。病原体流行率和毒力无疑受到地理分化的影响,因此nbs编码基因数量的这种扩张和/或收缩似乎是许多谷物品种适应快速变化的病原体谱的重要策略。片段复制和串联复制以及基因转换可能促成了跨物种的nbs编码基因的高度聚类(例如[40]),进一步导致共统侵蚀和基因丢失。目前的研究还确定了高粱中约5%的nbs编码基因的存在/缺失变异,鉴定出10 10个新的nbs编码基因,此外还观察到8个基因发生了基因丢失事件。这证明了最大的抗病基因家族的持续动态和高度可塑性。
结论
驱动谷物中nbs编码基因快速变异的一个关键机制是高度动态的聚类,通过存在/缺失变异(如基因丢失事件、伪基因化和新基因)和拷贝数变异进行谱系特异性重排。高粱nbs编码基因的可塑性受多种进化过程的影响;核苷酸序列汇总统计描述了进化过程中可能的连续状态,包括净化选择和平衡选择。这种截然不同的进化过程影响了所有谷物的祖先基因,而不是高粱的物种特异性基因。
当植物育种家寻求识别和部署强大的抗病能力时,这项研究使他们清楚地了解了这个丰富基因家族的起源和等位基因多样性,这对高粱的过去、现在和未来非常重要,高粱是5亿人的主食作物。更广泛地说,通过复制和重排,通过nbs编码基因家族的扩展,了解谷物在与快速进化的真菌病原体的“军备竞赛”中使用的机制,研究人员和育种人员可以更好地在这场持续的战斗中有效地操纵植物的防御。
方法
nbs编码基因鉴定
高粱基因组组合及预测基因模型(v1.4) [13]、玉米(v1.0) [19]和米(o .漂白亚麻纤维卷无性系种群。粳稻;第7版)[18]、[45]的资料,由JGI (http://www.phyotozome.net/x), Maizesequence.org (http://ftp.maizesequence.org/current/filtered-set/)及密歇根州立大学(ftp://ftp.plantbiology.msu.edu/pub/data/Eukaryotic_Projects/o_sativa/annotation_dbs/pseudomolecules/version_7.0/all.dir/分别)。为了鉴定3种草中nbs编码基因,使用了具有NB-ARC结构域氨基酸序列的BLASTp (Pfam: PF00931);阈值期望值在10-4上进行过滤。随后利用Pfam (Protein family)数据库确定相应的候选NBS蛋白是否编码TIR、NBS或LRR基序。随后,通过使用GenBank中现有的注释手动检查每个候选基因,以确认它们编码了预期的NBS蛋白。然后使用阈值为0.9的线圈专门检测CC(线圈)域。在每个物种基因组中,如果有两个或两个以上的nbs编码基因位于200 kb内,则定义为一个基因簇[46].
序列比对和系统发育分析
使用ClustalW对保守的NBS结构域(PFAM00931)的预测氨基酸序列进行多次比对[47]及Mega5.0 [48].使用前面描述的perl脚本计算核苷酸分化程度和基因覆盖范围,以识别每个物种内和所有三个物种中所有已识别的nbs编码基因之间的基因家族[4].利用TreeBest中NBS蛋白结构域(PFAM00931)的核苷酸序列,采用邻域连接法构建系统发育树。使用DARwin5.0软件显示该树[49].高粱、水稻和玉米全基因组的共线区[50],并突出显示已识别的nbs编码基因的位置,使用Circos软件[51].
数据分析
高粱中所有已鉴定的nbs编码基因的序列数据是从44个高粱基因型的全基因组重测序数据中提取的[23],代表三个类群(野生和杂草类群、长种类群和改良近交系类群)(补充文件3.:表S7)。如前所述,计算了以下总结统计数据:对已识别的nbs编码基因估计了组内的平均成对发散度(θπ), Watterson 's estimator (θw)和Tajima 's D,并使用BioPerl模块和内部perl脚本计算了三个组的周围10 kb基因组间隔。F圣基于相同的窗口计算,使用另一个BioPerl模块测量种群分化。涉及编码区域的汇总统计包括同义替换(Ks)和非同义替换(Ka)的数量,使用KaKs_Calculator1.2软件计算(MYN方法)[52],以及基于非同义替换的蛋白质变体的数量。
在提纯选择下的基因组区域先前使用群体遗传学总结统计(θπ, θw, Tajima’s D和F)确定圣)在以下三个种群的成对比较中:(i)野生和杂草与本地品种,以识别驯化事件;(ii)本地品种与改良自交系,以识别改良事件;(iii)野生和杂草与改良自交系,以识别驯化和改良事件。从先前描述的候选基因中鉴定出具有纯化选择特征的nbs编码基因[23],并在种群汇总统计的经验分布的顶部和底部5%的尾部鉴定了nbs编码基因。利用群体遗传学汇总统计,根据以下标准确定了平衡选择的特征;θπ和θw在经验分布的25%以上;田岛的D在经验分布的前5%和F圣值<90%的种群成对分布。选取nbs编码基因分别在Network [53].以纯化选择的17个非不变候选基因和38个中性基因作为输入,进行mlHKA检验以进行验证[25];除了23个平衡选择的候选基因外,还有38个中性基因。mlHKA程序在中性模型下运行,其中numselectedloci = 0,然后在选择模型下运行,其中numselectedloci > 0。显著性通过平均对数似然比检验统计量进行评估,其中模型之间对数似然差的两倍近似于卡方分布,df等于参数数量的差值。重复的基因对通过之前计算的四倍简并转换(4DTv)比率来识别[23].
在重测序的44种高粱基因型中鉴定了存在/缺失模式。基于NB-ARC结构域氨基酸序列(Pfam: PF00931)的BLASTp分析被用于之前鉴定的101个新基因[23];和以前一样,阈值期望值是在10-4上筛选的。此外,如前所述,所有基因型中所有已识别的nbs编码基因使用100 bp分辨率的读取深度来识别基因丢失事件[23].
综合疾病qtl图谱
从高粱共识图谱上投影的771个QTL中筛选出26个高粱真菌病原抗性QTL [54].另外4项研究发现40个真菌病原体抗病QTL和/或主要影响基因[35]、[36]、[55]、[56同样的策略也被投射到高粱共识地图上。来自11项研究的66个真菌病原抗病QTL的物理位置,代表9个性状(附加文件)2:表S6)使用已知遗传连锁距离的504个测序标记的框架图进行预测,如前所述[54].
支持数据的可用性
支持本文结果的数据集可从Dryad: doi:10.5061/ Dryad .d334b获得。
作者的贡献
ESM、SST、DJI、WSH进行数据分析;ESM、IDG、BCC、EKG、PJP、AC、DRJ撰写稿件;ESM和DRJ构想并设计了该项目;ESM, DRJ, AC和JW管理该项目。所有作者都阅读并批准了最终的手稿。
附加文件
缩写
- NBS-LRR:
-
核苷酸结合位点加上富亮氨酸重复序列
- QTL:
-
数量性状位点
- 行动:
-
人数/ interleukin-1受体
- 评估报告:
-
定量疾病反应
- microrna的:
-
小分子核糖核酸
参考文献
- 1.
Borlaug NE:养活100亿人口:未来的奇迹。中国生物医学工程学报,2002,38(3):344 - 344。10.1079 / IVP2001279。
- 2.
Christou P, Twyman RM:基因增强植物解决粮食不安全的潜力。环境科学学报,2004,17:23-42。10.1079 / NRR200373。
- 3.
Oerke EC:害虫造成的作物损失。中国农业科学,2006,44(3):344 - 344。10.1017 / S0021859605005708。
- 4.
杨松,冯志,张旭,姜坤,金霞,航勇,陈建强,田东:水稻抗病基因遗传变异的全基因组研究。植物化学学报,2006,29(3):344 - 344。10.1007 / s11103 - 006 - 9012 - 3。
- 5.
陈晓明,陈晓明,陈晓明,等。植物NBS-LRR蛋白的研究进展。中国生物工程学报,2006,7:212-10.1186/gb。
- 6.
岳建新,Meyers BC,陈建强,田东,杨松:植物核苷酸结合位点-亮氨酸水稻重复序列(NBS-LRR)基因起源与进化历史的追溯。环境科学学报,2012,29(3):344 - 344。10.1111 / j.1469-8137.2011.04006.x。
- 7.
周涛,王勇,陈建强,Araki H,景忠,姜凯,沈娟,田东:粳稻NBS基因的全基因组鉴定显示非tir NBS- lrr基因显著扩增。中国生物医学工程学报,2004,27(3):344 - 344。10.1007 / s00438 - 004 - 0990 - z。
- 8.
杨晓明,王晓明,王晓明,等:水稻抗性基因同源物的全基因组分析。应用理论与实践,2004,29(4):344 - 344。10.1007 / s00122 - 004 - 1758 - x。
- 9.
Hammond-Kosack KE, Jones JD:植物抗病基因。植物化学学报,1997,33(4):366 - 366。10.1146 / annurev.arplant.48.1.575。
- 10.
谭山,吴山:植物核苷酸结合位点抗病基因全基因组分析短柄藻基因组学2012, 418208:, [http://www.hindawi.com/journals/ijg/2012/418208/]
- 11.
王晓明,王晓明,王晓明,王晓明,王晓明。拟南芥nbs - lrr基因序列分析。植物学报,2003,29(3):344 - 344。10.1105 / tpc.009308。
- 12.
杨松,张旭,岳建新,田东,陈建强:最近的重复在两个木本物种中主导着nbs编码基因的扩增。中国生物医学工程学报,2008,29(3):344 - 344。10.1007 / s00438 - 008 - 0355 - 0。
- 13.
Paterson AH, Bowers JE, Bruggmann R, Dubchak I, Grimwood J, Gundlach H, Haberer G, Hellsten U, Mitros T, Poliakov A, Schmutz J, Spannagl M, Tang H, Wang X, Wicker T, Bharti AK, Chapman J, Feltus FA, Gowik U, Grigoriev IV, Lyons E, Maher CA, Martis M, Narechania A, Otillar RP, Penning BW, Salamov AA, Wang Y, Zhang L, Carpita NC,等高粱二色的基因组与草的多样化。自然科学进展,2009,37(4):344 - 344。10.1038 / nature07723。
- 14.
谭欣,王霞,王震,李娟,Paterson AH: NBS基因在基因组水平上的进化比较玉米而且高粱二色的[摘要]。植物动物基因组会议XX2012, [], [https://pag.confex.com/pag/xx/webprogram/Paper4080.html]
- 15.
李娟,丁娟,张伟,张勇,唐鹏,陈建强,田东,杨松:水稻NBS-LRR基因数量的独特进化模式。中国生物医学工程学报,2010,29(3):344 - 344。10.1007 / s00438 - 010 - 0527 - 6。
- 16.
杨晓燕,王晓燕,王晓燕,等。拟南芥R基因多态性的研究进展。植物科学学报,2006,30(3):344 - 344。10.1105 / tpc.106.042614。
- 17.
杨松,李娟,张鑫,张强,黄娟,陈建强,Hartl DL,田东:不同草种R基因的快速进化与水稻稻瘟病抗性。自然科学进展,2013,29(2):366 - 366。10.1073 / pnas.1318211110。
- 18.
于娟,胡松,王娟,黄国坤,李松,刘波,邓勇,戴丽,周勇,张鑫,曹敏,刘娟,孙娟,唐娟,陈勇,黄鑫,林伟,叶超,童伟,丛林,耿建,韩艳,李磊,李伟,胡刚,黄霞,李伟,李娟,刘震,李磊,等:水稻基因组序列草案(栽培稻l . ssp。籼稻).科学通报,2002,29(3):344 - 344。10.1126 / science.1068037。
- 19.
Schnable PS, Ware D, Fulton RS, Stein JC, Wei F, Pasternak S, Liang C, Zhang J, Fulton L, Graves TA, Minx P, Reily AD, Courtney L, Kruchowski SS, Tomlinson C, Strong C, Delehaunty K, Fronick C, Courtney B, Rock SM, Belter E, Du F, Kim K, Abbott RM, Cotton M, Levy A, Marchetto P, Ochoa K, Jackson SM, Gillam B,等:B73玉米基因组:复杂性、多样性和动态。科学通报,2009,29(4):344 - 344。10.1126 / science.1178534。
- 20.
赖军,李荣,徐旭,金伟,徐明,赵辉,向志,宋伟,应坤,张军,李丹,郭旭,叶凯,简明,王波,郑辉,梁辉,张鑫,王松,陈松,李娟,付勇,施普林格nm,杨辉,王俊,戴俊,Schnable PS,王俊:玉米优良自交系遗传变异的全基因组模式。中国科学,2010,30(4):344 - 344。10.1038 / ng.684。
- 21.
徐旭,刘霞,葛松,Jensen JD,胡峰,李霞,董艳,Gutenkunst RN,方琳,黄玲,李娟,何伟,张刚,郑霞,张峰,李艳,于超,Kristiansen K,张欣,王杰,Wright M, McCouch S, Nielsen R,王杰,王伟:50份栽培和野生稻种质重测序可获得农艺重要基因的标记。生物技术学报,2011,30:105-111。10.1038 / nbt.2050。
- 22.
Hufford MB, Xu X, van Heerwaarden J, Pyhäjärvi T, Chia JM, Cartwright RA, Elshire RJ, Glaubitz JC, Guill KE, Kaeppler SM, Lai J, Morrell PL, Shannon LM, Song C,施普林格NM, swson - wagner RA, Tiffin P, Wang J, Zhang G, Doebley J, McMullen MD, Ware D, Buckler ES, Yang S, Ross-Ibarra J:玉米驯化与改良的比较群体基因组学。中国科学,2012,34(4):344 - 344。10.1038 / ng.2309。
- 23.
Mace ES, Tai S, Gilding EK, Li Y, Prentis PJ, Bian L, Campbell BC, Hu W, Innes DJ, Han X, Cruickshank A, Dai C, Frère C, Zhang H, Hunt CH, Wang X, Shatte T, Wang M, Su Z, Li J, Lin X, Godwin ID, Jordan DR, Wang J:全基因组测序揭示了非洲本土谷物作物高粱未开发的遗传潜力。自然科学进展,2013,30 (4):344 - 344
- 24.
Scheideler M, Schlaich NL, Fellenberg K, Beissbarth T, Hauser NC, Vingron M, Slusarenko AJ, Hoheisel JD:监测从家政到病原体防御代谢的转变拟南芥使用cDNA阵列。中国生物医学工程学报,2002,27(3):344 - 344。10.1074 / jbc.M104863200。
- 25.
Wright SI, Charlesworth B:重新审视HKA检验:标准中性模型的最大似然比检验。遗传学报,2004,38(4):344 - 344。10.1534 / genetics.104.026500。
- 26.
李鹏,王勇,钱强,付忠,王明,曾东,李波,王霞,李娟:LAZY1通过调节生长素的极性转运控制水稻笋向地性。海洋学报,2007,17:402-410。
- 27.
Furukawa T, Maekawa M, Oki T, Suda I, Iida S, Shimada H, Takamure I, Kadowaki K: Rc和Rd基因参与水稻果皮原花青素合成。植物学报,2007,29(3):344 - 344。10.1111 / j.1365 - 313 x.2006.02958.x。
- 28.
Martin T, Biruma M, Fridborg I, Okori P, Dixelius C:一种高度保守的NB-LRR编码基因簇Setosphaeria turcica在高粱。中国生物医学工程学报,2011,11:151-10.1186/1471-2229-11-151。
- 29.
Córdova-Campos O, Adame-Álvarez RM, Acosta-Gallegos JA, Heil M:驯化对普通菜豆基础抗病性和诱导抗病性的影响(Phaseolous寻常的).中国植物学报,2012,34(3):366 - 366。10.1007 / s10658 - 012 - 9995 - 3。
- 30.
白j, Pennill LA, Ning J, Lee SW, Ramalingam J, Webb CA,赵b, Sun Q, Nelson JC, Leach JE, Hulbert SH:谷物中富含核苷酸结合位点亮氨酸重复基因的多样性。中国生物工程学报,2002,29(4):344 - 344。10.1101 / gr.454902。
- 31.
孙倩,孙强,李志刚,李志刚,李志刚:水稻基因组中与广谱定量抗病性相关区域的鉴定与鉴定。中国生物医学工程学报,2005,29(3):357 - 357。10.1534 / genetics.104.036327。
- 32.
姜玉杰,金克勤,沈森,尹美娟,孙珊,金美娟,范凯,李世华:大豆NBS-LRR基因的全基因组定位及其与抗病性的关系。中国生物医学工程学报,2012,29(3):349 - 349。10.1186 / 1471-2229-12-139。
- 33.
Klein RR, Rodriguez-Herrera R, Schlueter JA, Klein PE, Yu ZH, Rooney WL:影响高粱籽粒霉变发生率和其他农艺重要性状的基因组区域鉴定。应用理论,2001,32(3):397 - 397。10.1007 / s001220051647。
- 34.
Mohan SM, Madhusudhana R, Mathur K, Howarth CJ, Srinivas G, Satish K, Reddy RN, Seetharama N:高粱叶片抗病数量性状位点的共定位。中国植物学报,2009,29(3):344 - 344。10.1111 / j.1439-0523.2008.01610.x。
- 35.
Mohan SM, Madhusudhana R, Mathur K, Chakravarthi DVN, Rathore S, Reddy RN, Satish K, Srinivas G, Mani NS, Seetharama N:高粱叶片抗病相关数量性状位点的鉴定[高粱二色的(l)Moench]。《植物科学》,2010,27(3):344 - 344。10.1007 / s10681 - 010 - 0224 - x。
- 36.
王永华,王永华,王晓明,王晓明:高粱抗炭疽病遗传标记的关联分析。理论与实践,2013,26(1):1 - 7。10.1007 / s00122 - 013 - 2081 - 1。
- 37.
McMullen MD, Simcox KD:玉米抗病和抗虫基因的基因组组织。植物与微生物的相互作用,1995,8:811-815。10.1094 / mpmi - 8 - 0811。
- 38.
Gebhardt C, Valkonen JP:马铃薯基因组中控制抗病基因的组织。植物学报,2001,39:79-102。10.1146 / annurev.phyto.39.1.79。
- 39.
程燕,李霞,姜辉,马伟,苗伟,山田涛,张敏:玉米核苷酸结合位点抗病基因的系统分析与比较。中国生物医学工程学报,2012,29(3):344 - 344。10.1111 / j.1742-4658.2012.08621.x。
- 40.
张荣,张建平,王晓明,王晓明,王晓明,等。植物抗病基因的古进化可塑性研究。BMC基因组学杂志,2014,15:187-10.1186/1471-2164-15-187
- 41.
程旭,姜辉,赵艳,钱艳,朱松,程斌:水稻抗病基因核苷酸结合位点的基因组分析高粱二色的.中华生物医学杂志,2010,33:292-297。10.1590 / s1415 - 47572010005000036。
- 42.
Eckardht NA:植物防御中的microRNA级联。植物学报,2012,24:840-10.1105/tpc.112.240311。
- 43.
李芳,孙海燕,杨晓明,杨晓明,杨晓明,杨晓明,杨晓明,杨晓明,杨晓明。植物天然免疫受体的MicroRNA调控。自然科学进展,2012,29(4):489 - 497。10.1073 / pnas.1118282109。
- 44.
罗松,张勇,胡强,陈娟,李凯,陆超,刘红,王伟,匡宏:草科植物核苷酸结合位点和富亮氨酸重复序列动态编码基因。中国生物医学工程学报,2012,29(3):357 - 357。10.1104 / pp.111.192062。
- 45.
栽培稻l . ssp。粳稻).科学通报,2002,29(3):344 - 344。10.1126 / science.1068275。
- 46.
Holub EB:在拟南芥这种野花中,军备竞赛是古老的历史。中国科学,2001,(2):516-527。10.1038 / 35080508。
- 47.
拉金MA,布莱克希尔兹G,布朗NP,陈纳R,麦克格蒂根PA,麦克威廉H,瓦伦丁F,华莱士IM,威尔姆A,洛佩兹R,汤普森JD,吉布森TJ,希金斯DG: Clustal W和Clustal X版本20。生物信息学,2007,23:2947-2948。10.1093 /生物信息学/ btm404。
- 48.
田村K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S: MEGA5:利用最大似然,进化距离和最大简约方法进行分子进化遗传学分析。分子生物学杂志,2011,28:2731-2739。10.1093 / molbev / msr121。
- 49.
Perrier X, Jacquemoud-Collet JP: DARwin软件。2006 [], [http://darwin.cirad.fr/]
- 50.
杨晓明,王晓明,李晓明,等:牧草共链基因缺失的全基因组分析。中国生物医学工程学报,2012,29(4):366 - 366。对10.1093 / gbe / evs009。
- 51.
Kryzwinski MI, Schein JE, Birol I, Connors J, Gascoyne R, Horsman D, Jones SJ, Marra MA: Circos:比较基因组学的信息美学。中国生物工程学报,2009,29(4):344 - 344。10.1101 / gr.092759.109。
- 52.
张震,李娟,赵小强,王杰,王桂坤,余杰:KaKs计算器:通过模型选择和模型平均计算Ka和Ks。生物信息学杂志,2006,(4):357 - 357。10.1016 / s1672 - 0229(07) 60007 - 2。
- 53.
刘志刚,杨晓明,杨晓明,Rãhl A:基于中值连接网络的种内系统发育分析。中华生物医学杂志,1999,16:37-48。10.1093 / oxfordjournals.molbev.a026036。
- 54.
Mace ES, Jordan DR:将高粱全基因组序列信息与高粱QTL研究概要整合,揭示了QTL和基因丰富区域的不均匀分布,这对作物改良具有重要意义。应用理论与实践,2011,29(1):1 - 7。10.1007 / s00122 - 011 - 1575 - y。
- 55.
Mace ES, Jordan DR:高粱主要效应基因的定位(高粱二色的(l)Moench)。应用物理学报,2010,29(3):344 - 344。10.1007 / s00122 - 010 - 1392 - 8。
- 56.
王永华,王永华,王永华,夏尔梅:高粱叶锈病和籽粒霉变抗性的SNP标记。分子生物学学报,2013,32:451-462。10.1007 / s11032 - 013 - 9883 - 3。
确认
我们要感谢Colleen Hunt提供的统计支持和Sylvia Malory提供的初步序列分析。我们感谢昆士兰大学、农业、渔业和林业部、粮食研究与开发公司(GRDC)和北京基因组研究所的资金支持。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
电子辅助材料
12870 _2014_253_moesm1_esm.xlsx
附加文件1:表S1。:Detailing the location, type and selection status of 346 NBS-encoding genes identified in sorghum. (XLSX 35 KB)
12870 _2014_253_moesm2_esm.pdf
附加文件2:包含22个补充图和5个补充表,均在正文中引用。(pdf 810kb)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/4.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
梅斯,E,泰,S,英尼斯,D。et al。高粱抗NBS基因的可塑性是由多个进化过程驱动的。BMC植物生物学14日,253(2014)。https://doi.org/10.1186/s12870-014-0253-z
收到了:
接受:
发表:
关键字
- 谷物
- 抗病性
- 驯化
- 玉米
- NBS-LRR基因
- QTL
- 大米
- 选择
- 高粱