摘要
背景
新的SNP标记平台为研究来自不同地区的小麦品种之间的关系提供了机会,并评估了导致小麦适应特定生产环境的机制和过程。哈萨克斯坦的小麦育种历史悠久,本研究的目的是探讨哈萨克斯坦的关键品种与其他地区育种项目的种质之间的关系。
结果
在10个品种中共发现5898个多态标记,其中2730个在共识遗传图谱中。映射的SNP标记在A和B基因组中几乎平均分布,每个染色体上有279到484个标记。标记覆盖在D基因组中约低10倍。有863个SNP标记被鉴定为特定品种所特有的,这些标记的集群(包含三个以上紧密映射的独特SNP位点的区域)在每个品种的共识遗传图谱上显示出特定的模式。在3个品种(Tzelinnaya 3C、Kazakhstanskaya rannespelaya和Kazakhstanskaya 15)中发现了显著的品种内遗传多态性。基于品种间多态性的系统发育分析表明,非常古老的红精系841与其他9个哈萨克斯坦品种的遗传差异最大,与美国品种Sonora和中亚和南亚的基因型同属一个分支。现代品种Kazakhstanskaya 19也与美国品种撒切尔(Thatcher)属于一个单独的分支。其余8个品种共享一个亚枝,但分为4个类群。
结论
积累的哈萨克斯坦面包小麦基因型间SNP标记多态性数据可用于研究面包小麦的遗传多样性,并有可能应用于标记辅助选择和一套基因型特异性标记的制备。
背景
利用分子标记对品种和育种材料进行基因分型是现代植物育种的重要工具。分子标记作为标记辅助选择(MAS)的一个组成部分,可以改善和加速新品种的发育和释放过程[1] - [4].分子标记还有助于明确的品种识别和解决潜在的所有权冲突,通过阐明一个给定基因型的谱系/祖先。它们还用于调查种质收集内的遗传距离和相似性[5].
单核苷酸多态性(SNP)标记是一种最重要的分子标记类型,近年来栽培面包小麦的单核苷酸多态性技术取得了重大进展。小麦l . (6] - [9].SNP标记的最初开发是繁琐和低通量的,使用EST [10] - [13]和BAC测序信息[14].然而,全基因组分析[15]通过下一代测序(NGS)平台,极大地促进了植物育种SNP平台的开发和部署[2]、[16],特别是对小麦等作物[3.]、[17]、[18].异源多倍体物种基因分型的NGS技术及相关数据管理工具的开发[19]对面包小麦至关重要,面包小麦具有非常大的基因组和三组同源染色体:AA、BB和DD [20.] - [24].小麦基因分型的NGS-SNP技术有三种一般类型[9].单纯形技术通常与TaqMan分析一起用于多个样本中的单个SNP基因分型[25].多重基因分型方法适用于在有限数量的样本中研究更多的SNP标记(30-60),通常少于几百个[13]、[26].在我们的研究中,我们选择了第三种基于阵列的技术,它可以在基因组中检测数千个SNP标记。我们使用了Infinium 9k SNP阵列[3.]、[22]、[27]来调查来自哈萨克斯坦的一小批面包小麦的遗传多样性,以了解哈萨克斯坦小麦与来自其他地区的精品系之间的关系。类似的SNP基因分型策略也用于硬粒小麦[28] - [30.,二粒小麦[31]、合成小麦及其野生祖先[32]、[33].
SNP基因型在将标记与农艺性状关联的连锁和关联作图方法中都很有用。利用KASPar技术,建立了双单倍体群体的高密度SNP遗传图谱[24]和排序后的3B染色体[34].对172个欧洲冬面包小麦品系的遗传关系进行了研究,尽管在1395个高质量SNP标记中,只有518个被发现适合用于关联作图[35].
对NGS-SNP技术进行了进一步的修饰,并应用于硬粒小麦遗传多样性的研究。利用单链构象多态性(SSCP)和KASPar技术对小麦硬粒系的“感兴趣基因”进行了批量分离分析[36].序列相关扩增多态性(SRAP)标记和测序基因分型(GBS)技术已成功用于小麦种质、栽培品种和定位群体的基因分型[37] - [39].在二倍体物种山羊草属tauschii7 K - 185 K多态性和高置信度SNP标记已被报道,并用于研究不同品系的基因组进化、遗传多样性和地理起源[33]、[40] - [42].包括CerealsDB和SNPMeta在内的生物信息学数据库提供了有关NGS-SNP、GBS和KASPar技术的公开可访问数据[43]、[44].
本研究的目的是:(1)评估SNP平台的适用性,以哈萨克斯坦为样本,探讨相对孤立的小麦改良项目的起源和选择基础;(2)对哈萨克斯坦10个面包小麦品种(包括8个近期培育的品种和2个历史参考系谱品种)的多态性和独特SNP标记进行鉴定,并研究其在基因组和染色体中的分布;(3)对选择的哈萨克小麦品种进行品种内和品种间的遗传多样性分析,以探索哈萨克小麦关键品系的起源。这项初步研究可以作为进一步更广泛的种质研究的基础,并开发一套对哈萨克斯坦育种项目有用的SNP标记。
方法
植物材料
10个面包小麦品种的种子从哈萨克斯坦农业和作物生产研究所(Almalybak, Kazakh Research Institute of Agriculture and Crop Production)获得,并在表中列出了它们的谱系描述1.在种植该品种的哈萨克斯坦,农业生态条件可以被描述为整个植被期的强烈干旱和局部的盐胁迫。
选择已发表资料的小麦系系进行系统发育比较
从已发表数据的2,994份小麦资料中选择了一小组地理来源广泛的小麦[3.]与来自哈萨克斯坦的研究品种进行比较。从亚洲(4个)、澳大利亚(9个)、加拿大(8个)、中国(6个)和美国(10个)选择了基因多样性的样本(附加文件)1).除来自乌兹别克斯坦的两个地方品种外,其余均为春小麦品种。用该面板与哈萨克斯坦的10个品种进行系统发育比较。
DNA提取和9k Infinium SNP标记分析
植株在温室中生长,直到分蘖,每个品种选择5株均匀植株,除了品种TZE-3C,选择6株。使用苯酚-氯仿提取方法从每株植物的单个嫩叶中提取DNA [48].DNA浓度调整至50 ng μl-1用凝胶电泳和PCR检测DNA质量。DNA样本(共51个)提交给维多利亚州(澳大利亚)第一产业部(DPI),使用9k Infinium SNP阵列进行基因分型[3.]、[18]、[22]、[27],详情见附加文件2.
对所有SNP标记进行评估,并在至少一个品种的等位基因差异处确定多态性标记。将独特的SNP标记与多态标记区分开来,定义为对单一品种(Singleton)的诊断。此外,为每个品种定义和识别由三个或更多的独特标记组成的簇,这些标记映射到一致图谱上单个染色体的小区域(相邻snp之间小于或等于2.5 cM)。在确定SNP标记簇时,在遗传图谱上具有相同位置的唯一SNP标记被计算为单个标记。所有独特的SNP标记及其集群都进行了手工评估和检查。在特定品种内5-6株间具有多态性的标记被归类为“品种内”,而不用于“品种间”分析。
计算机地图成像和分子系统发育
计算机软件程序FlapJack [49]用于所有样本的图形基因分型和遗传图谱的可视化,并显示已知染色体位置的SNP标记的位置。从FlapJack和SNP标记表导出的相似矩阵[3.]转换为距离矩阵,然后应用该距离矩阵构建BioNJ树[50使用SplitsTree4程序[51]、[52].Nei’s基因多样性和多态性信息含量(PIC)计算使用PowerMarker,版本3.25 [53].Nei的基因多样性决定了样本中两个随机选择的等位基因不同的概率。PIC表示两个随机选择的样本显示多态等位基因的概率。MapChart 2.2版本用于遗传图谱的制备和可视化[54].
结果
多态SNP标记在小麦基因组和染色体组上的分布
9k Infinium SNP分析共鉴定8632个SNP标记,其中有效SNP标记5898个。支持本文结果的数据集包含在附加文件中3..Nei基因多样性较高,但在不同品种间比较相似,范围为0.5044 (paf -93) ~ 0.5309 (红霉素- 841), PIC值在0.3832 ~ 0.4224之间。
然而,只有2730个多态SNP标记(46.3%)可以被定位到小麦的染色体位置并用于进一步的研究2).这些标记在A和B基因组中几乎平均分布(分别为1341和1242个标记),而在D基因组中定位的SNP标记约为10倍。SNP标记在同源染色体上的分布变化不大,在279(组4)~ 484(组2)个SNP标记之间。
独特SNP标记映射到单个染色体的分布
唯一标记被定义为本研究中十个品种中的单个品种的诊断标记。共鉴定出885个独特的SNP标记,占标记总数的32.4%(表4)3.).
独特SNP标记在不同品种间的分布不均匀。超过一半(53.6%)是针对老参考系谱品种的红霉素- 841表明该基因型与其他小麦品种的遗传差异最大。其次和第三位的品种是kaz19和AKT-39,分别占有19.2%和9.8%的独特SNP标记。KAZ-R和PAV-93是遗传多态性最少的品种,分别与1.2%和0.8%的独特SNP标记相关(表2)3.).
独特SNP标记簇在染色体上的分布
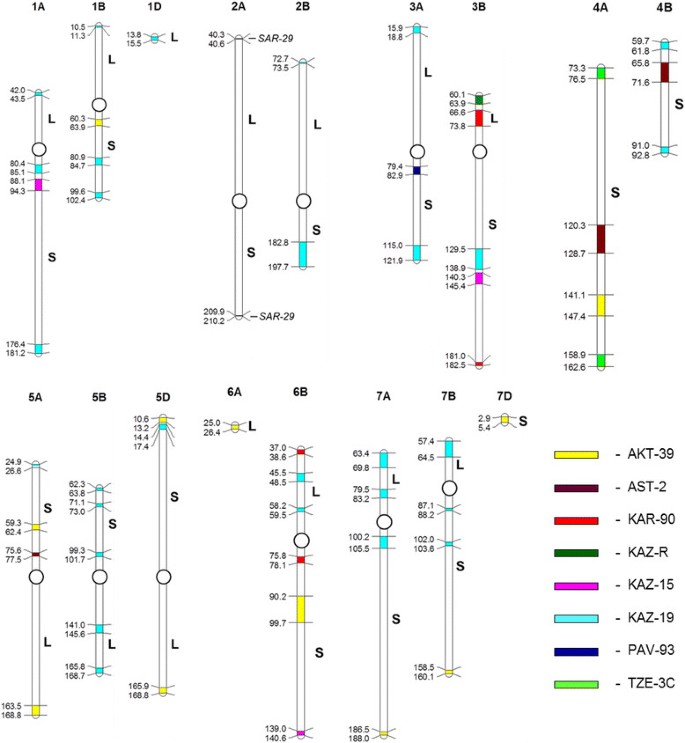
独特的SNP标记映射到个体染色体的分布对每个品种都是特定的。所有的染色体都被唯一的snp覆盖,但它们的分布频率不同。与单个独特的SNP标记相比,每个品种可以识别出3个或更多紧密映射标记的集群,它们的分布模式更具体,可以用于研究的种质特征(图1)1).独特的SNP标记簇可能代表来自外来祖先种质的小麦染色体片段。这种独特的SNP标记簇是诊断性的,因为它们只存在于一个品种中。表中几乎所有三种或三种以上SNP标记的组3.被映射到染色体的小区域,因此可以定义为集群,只有少数snp例外,分散在不同品种的染色体上。最多样化的品种红霉素- 841它也有35个独特SNP簇,几乎覆盖了所有的染色体。红霉素- 841没有显示在图中1因为在这个品种中发现了广泛的多样性。就独特的SNP而言,紧随其后的两个品种(kz -19和AKT-39)分别有29和11个独特的SNP标记簇,分布在基因组和染色体上(图1).重要的是,独特的SNP标记簇对每个研究的基因型分布非常明确。例如,在3AS染色体和3BL染色体上分别发现了PAV-93和kazo - r的单个SNP标记簇,而在TZE-3C中发现了两个簇,均位于4AS染色体上。每个簇内独特SNP标记的密度也各不相同。例如,在AST-2中,4个SNP标记以大约2 cM的间隔在4A染色体上形成了一个簇(120.3-128.7 cM),而在4B染色体上一个类似大小的区域(65.8-71.6 cM)包含了17个SNP标记。同样,在AKT-39中,沿着9条染色体的11个簇中,SNP标记的密度是可变的。有趣的是,这两簇10个独特的SNP标记SAR-29特别小,在2A染色体的最远端各有0.3 cM的片段(这不能在图1).在D基因组的部分染色体上未发现独特的SNP标记簇,而在6AL、1DL和7DS染色体上仅发现单个簇。本研究中最多样化的染色体是3B和6B,每个染色体都有4个品种的独特snp簇(图1).
在共识遗传图谱上按颜色编码的独特SNP标记簇(包含三个或更多紧密映射的SNP的区域)的分布。来自哈萨克斯坦的8个品种的彩色染色体片段编码如图片右侧的键所示。reference-pedigree的品种红霉素- 841没有显示,因为在这个基因型中存在广泛的多样性。集群发现SAR-29直接显示在染色体2A上。遗传距离以每条染色体左侧的cM表示;染色体的方向与一致性图中描述的一样,并显示长臂(L)和短臂(S)。着丝粒的大致位置用圆表示。该地图是使用MapChart软件绘制的[54],其中唯一SNP标记簇的初始数据来自维多利亚(澳大利亚)DPI生成的矩阵数据。
基因分型和品种内多态性
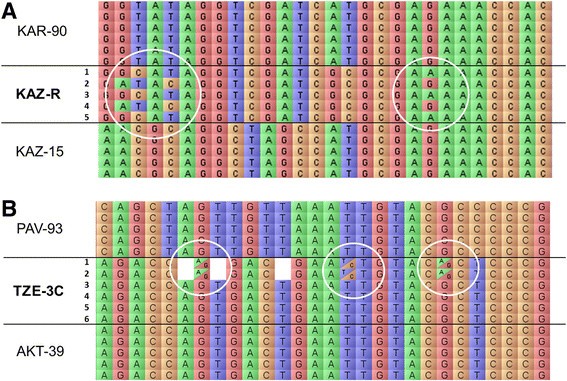
来自单株植物的5 ~ 6个独立DNA样本被用于通过SNP标记分析评估每个品种的品种内遗传多态性(图2).这十个品种被分为三个组。五个基因型(AKT-39、红霉素- 841KAZ-19,SAR-29和PAV-93)的品种内多态性不显著或极低,植株的遗传特征基本一致(图1)2A).第二组由两个品种(AST-2和KAR-90)代表,具有中等的品种内多态性,其中5个研究个体中有一个在不同染色体的少数遗传区域显示出不同的SNP标记等位基因(图2B).这种多态可能是品种间残留的杂合性,仍处于分离状态。kz - r、kz -15和TZE-3C属于第三组,表现出显著的品种内遗传多态性,表明它们是遗传差异个体的混合物(图1)2C). SNP标记分析表明,其中两个品种(kazo - r和kazo -15)的遗传多样性较小,而TZE-3C的遗传多样性较大,该品种内两组个体间的遗传变异与品种间多态性相当。我们认为第三组的高水平的品种内遗传多态性可能反映了培育这些品种所采用的方法(如群体而不是线性选择方法)。
图中给出了kazu - r品种内多态性的一个例子3.A.该品种5个个体的基因分型可以清楚地区分两组遗传多样性植物的组合:(i) 1、3、5号,(ii) 2、4号。3个品种中有时也有SNP杂合子标记,表现出显著的品种内多态性,其中以TZE-3C品种多见(图1)3.B).然而,它们的频率不超过映射多态snp的1%。
使用FlapJack软件可视化的面包小麦品种内多态性示例[[49]].列表示每个标记SNP位置上的核苷酸,每一行表示单个植物的基因分型。品种名称在图的左边给出,其中具有品种内多态性的品种以粗体表示。(一)KAZ-R 5个品种间3B染色体的品种内多态性用白色圆圈表示。(B)品种TZE-3C的6个植株的4A染色体上的杂合子SNP标记为白色圆圈内的对角线框。一些snp的无评分结果显示为空框。
品种间多态性与分子遗传系统发育
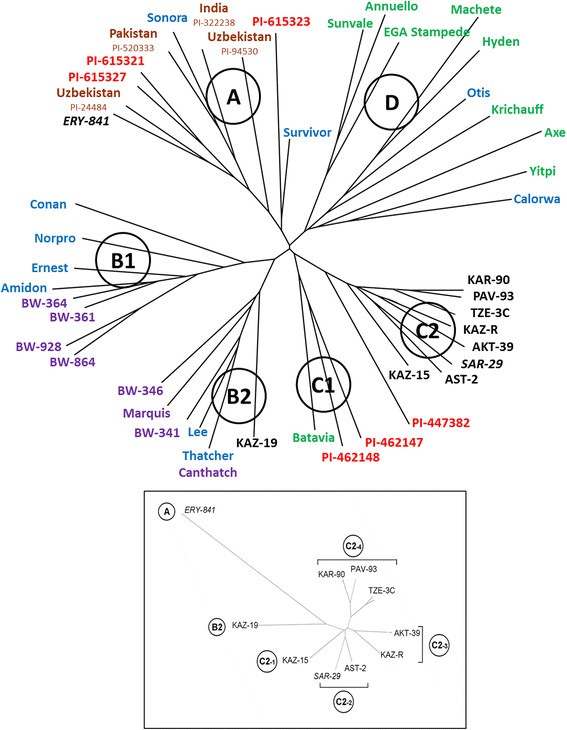
尽管所有研究的小麦品种具有共同的地理来源,但SNP标记分析表明它们具有广泛的品种间遗传多态性(图4).与预期的一样,遗传距离最远的基因型是参照系谱品种红霉素- 841与kz -19亲缘关系最为密切红霉素- 841,但在遗传上也不同于本研究中的所有其他品种(亚分支B2)。其余8个基因型被归类为一个C2子分支的4个簇。虽然kazu -15(群集C2-1)与kazu -19在同一个育种中心培育,但它们存在遗传差异。两个品种(AST-2和参考系谱品种)SAR-29)属于C2-2类,两个品种(AKT-39和KAZ-R)属于C2-3类。其余3个品种(tse - 3c、paf -93和KAR-90)属于C2-4聚类,其中paf -93和KAR-90亲缘关系密切(图2)4).
哈萨克斯坦10个面包小麦品种的分子遗传系统发育。国际小麦基因型面板和来自哈萨克斯坦的十个研究品种显示在图的顶部,系统发育演化支和亚演化支的圈号。参考小麦基因型的地理起源用颜色编码:红色(中国)、棕色(其他亚洲国家)、蓝色(美国)、紫色(加拿大)、绿色(澳大利亚)和黑色(哈萨克斯坦)。从哈萨克斯坦提取的十个品种的结果分别显示在图的底部有框的插入,其中的演化支、子演化支和簇用圆圈表示。八个在哈萨克斯坦培育的新品种用正常字体显示,而两个较老的参考系谱品种用斜体字显示。数据由本研究中基于5,898个多态SNP标记的BioNJ树显示,并从已发表的数据中提取[3.].
将10个研究品系的遗传多态性分析与一组从已发表的资料中提取的具有广泛地理来源的春面包小麦的代表性品种进行比较[3.)(图4).Reference-pedigree品种红霉素- 841是最明显的,与非常古老的美国品种Sonora共享A支,以及来自中国和中南亚的基因型。B支的两个部分都代表来自北美(美国和加拿大)的基因型,但也包括现代哈萨克品种kazz -19(亚支B2)。C支系的多样性相当大,所有来自哈萨克斯坦的剩余品种都属于一个单一的亚支系C2。C2亚分支簇间的遗传差异显著,但与其他分支的基因型相比要小得多。D支中只发现了来自澳大利亚和美国的栽培品种,表明与哈萨克斯坦的基因型的遗传关系极小(图1)4).
讨论
SNP标记是研究遗传多态性、分子系统发育和MAS的有力工具[6] - [9].利用8632个SNP标记对哈萨克斯坦10个面包小麦品种的品种内和品种间遗传多态性进行了分析。超过一半(68.3%)的标记是多态标记,这些标记被用于进一步的分子遗传学研究。多态SNP标记的比例与之前发表的面包小麦种质的小组和大组相似:20个美国小麦品种和13个不同基因型[12和478个春小麦和冬小麦品系[21),分别。使用相同的9k Infinium SNP标记阵列对172个优质面包小麦品系进行标记,Würschum等。[35]报告说,只有16.2%的SNP标记在整个收集中是多态的。然而,只有1395个被鉴定的SNP是“高质量”标记,因为相对大部分的SNP标记要么缺失值要么杂合度高[35].对550份六倍体和55份四倍体小麦的90 K SNP序列的最新结果显示,澳大利亚和欧洲材料的SNP标记总数中,分别有15.4%和25.9%具有多态性[18].这些结果与早先的一份报告相似[23],在8个英国面包小麦品种中,500,000 K snp中有19.5%被鉴定为多态性。利用NGS技术获得的有用多态标记的比例很大程度上取决于数据质量。据报道,多态SNP标记的比例可以随着序列覆盖的加深而增加。例如,当序列覆盖率从12倍增加到16倍时,硬粒小麦的总SNP标记从66%增加到83% [36].然而,与增强序列覆盖相关的成本增加可能不能证明多态snp相对适度的增加是合理的。
在本研究中,2730个(46.3%)多态性SNP标记可映射到染色体位置,高于类似研究报道的37.1%的比例[35]及22.5% [18并且在A、B和D基因组中映射的多态SNP标记的分布与之前关于这两种SNP标记的报道相似[12]、[18]、[27]、[32]、[35]、[38和SSR标记[55].多态性SNP标记在A和B基因组之间分布均匀,而在D基因组上定位的标记较少。六倍体小麦的D基因组是三个基因组中最年轻的一个,被认为是在大约8000年前引入六倍体小麦的,是与六倍体小麦自然杂交的结果山羊草属tauschii(DD) [56].相比之下,A和B基因组祖细胞之间的杂交估计发生在58- 82万年前,这比之前提出的要早得多[57].D基因组相对较近的基因渗入和狭窄的遗传多样性可能导致了我们和其他人观察到的低SNP率,因为遗传多样性可能依赖于小麦进化过程中的随机过程[32].
在不同的染色体中SNP标记的频率没有明显的变化趋势。表格4列出了六倍体和四倍体小麦基因组中SNP标记频率最高和最低的染色体。观察到变异的原因可能与所开发的SNP标记的不同,以及不同研究中使用的小麦基因型的广泛多样性有关。有趣的是,在面包小麦的研究中,有一个趋势是在染色体4A、4B、7B、3D和4D上的snp频率较低4).面包小麦染色体4A上的低频率分子标记可能与该染色体上发生的古老的非同源易位有关[32]、[58]、[59].
本研究的小麦系谱及分子系统发育
来自哈萨克斯坦的10个品种被特别挑选用于这项研究,因为它们在干旱环境中的生产潜力和多代稳定产量的报告[60].哈萨克斯坦的育种家最近开发和生产了8个受欢迎的品种,同时还选择了两个更老的参考系谱品种。
Erythrospermum 841 (红霉素- 841)是最古老的参考系谱品种之一,实际上可能代表一个地方品种。它最初的形态是1913年在阿什哈巴德地区(土库曼斯坦)发现的当地种质资源,1929年作为一个品种发布(表2)1).红霉素- 841具有耐旱的特点,在苏联时期被作为标准品种使用多年[61]、[62].红霉素- 841还具有潜在的耐盐性,尽管这还没有很好地记录。它被用作至少37个品种的亲本,最后一个登记品种的报告是在1939年,Lutescence 96 [63].红霉素- 841自1978年以来,由于许多具有更先进特征的新品种的可用性(P. Malchikov, Personal com.),没有直接用于产生育种系。本研究中使用的一些品种可能是由早期杂交的育种材料发展而来的红霉素- 841但谱系分析表明红霉素- 841在遗传上与所有其他研究的基因型都有距离(图4),这将意味着对红霉素- 841近年来的等位基因。与一小批国际小麦的比较分析表明红霉素- 841是“历史地理”分支a的一员,包含较古老的亚洲小麦基因型。中亚国家土库曼斯坦(原始种质的起源红霉素- 841)在地理上靠近乌兹别克斯坦、中国、印度和巴基斯坦,因此,它们的相似性并不令人意外。索诺拉是最古老的小麦品种之一,最早可追溯到1770年的墨西哥,然后才被转移到美国[47].目前还不清楚为什么索诺拉和红霉素- 841和其他亚洲基因型。我们推测在A支中有亚洲基因型红霉素- 841和索诺拉有共同的祖先现代美国品种幸存者(1991年发布)可能有助于进一步阐明这个系统发育故事,但目前的谱系记录并没有指向索诺拉[47].
第二个参照系谱品种为Saratovskaya 29 (SAR-29),它的历史比红霉素- 841(1957年发布)。这个品种在前苏联非常受欢迎,但它已被基于相同种质的更先进的品种所取代,如Saratovskaya 60(1995年发布[46])。SAR-29直到最近,它才被广泛用于新品种的杂交和选育。SAR-29被纳入一项利用SRAP标记进行的遗传多态性研究,证实了该种质在俄罗斯广泛用于生产面包小麦新品种[37].在我们的研究中,SAR-29具有独特SNP标记的特定模式(表3.)和SNP集群分布(图1),没有出现在任何其他研究的系。这可能表明,一些基因片段,特别是来自2A染色体两端末端的基因片段,从未渗透到哈萨克斯坦的现代小麦中。我们推测,这些相对较小的基因片段可能包含有害的等位基因,或代表来自其他来源的重要新等位基因的区域。
在我们的研究中,SAR-29聚在C2亚枝中间,与系统发育树中8个现代栽培品种的距离较近红霉素- 841(图4).C2亚枝非常独特,包含来自哈萨克斯坦的栽培品种和一个中国的附加品种(PI-447382)。相比之下,最接近的亚分支C1包含澳大利亚和中国的基因型,但没有来自哈萨克斯坦的基因型。这可能是共同的遗传来源。SAR-29与AST-2形成C2-2簇。AST-2是在与一个先祖品种Tzelinnaya yubileinaya进行三轮回交后产生的SAR-29在它的血统。SAR-29也是kaz19的直系亲本(表1),尽管我们的分析表明它与AST-2的基因关系不一样。事实上,kaz19和来自北美(美国和加拿大)的基因型一样,属于一个单独的B2亚分支。这一有趣的观察结果可能是由于kaz19和美国品种Thatcher(1934年发布)的系谱中存在一个共同的祖先Kanred(美国,1917年发布)[47].然而,kazu -15有相同的祖先,但属于另一个子分支C2。结果表明,在哈萨克斯坦育种计划中,对北美种质中发现的一些基因组区域有很强的选择,导致了观察到的kazk -19在系统发育中的位置。这可能是进一步分析和育种的一个富有成效的途径。
2个分支(B1和D)分别含有来自北美(美国和加拿大)、澳大利亚和美国的栽培品种,说明哈萨克斯坦的研究基因型分布在其他分支中。有趣的是,在我们的研究中,中国品种在A、C1和C2分支中分布相对广泛(图1)4).
以前对哈萨克斯坦小麦基因型的亲本分析要么仅仅基于系谱研究[64],或使用24个SSR标记进行分子遗传分析[60]和33个SSR标记[65,在这些研究中有8个小麦品种与这里包含的品种相匹配。不同的方法有一些相似之处。例如,在本研究中,有两个品种红霉素- 841和kz -19(演化支A和B2,图4)的基因差异最大,这些结果与家谱研究的结果相似[64]和我们之前发表的使用24个SSR标记的分析[60].相比之下,本研究中来自C2-4亚分支的3个品种(karl -90、paf -93和tse - 3c)也在分子遗传分析中组合为第三类,使用33个SSR标记[65],但在家谱研究中被分散为三组(B、C和F) [64].KAR-90、tse - 3c和PAV-93的谱系没有表明它们有共同的遗传历史,但它们确实来自哈萨克斯坦的近地理位置。因此,育种者有可能为他们的选择寻找类似的亲本材料。我们观察到的分子和系谱分析之间的不完全重叠与之前的一项研究相似,该研究使用了来自墨西哥(CIMMYT)、印度和中国的20个优秀小麦品种[66].作者报告了使用93个SSR标记进行的系谱研究和分子分析之间相对较低但显著的相关性。在两项研究中,基因距离较远的材料比基因关系较近的材料具有更大的一致性。66].然而,系谱记录并不总是准确的,特别是对于一些较老的品种,育种项目中使用的许多系具有相当高水平的杂合性或系的混合。利用SNP标记技术进行品种内多态性研究的结果证实了我们之前的发现[60].品种TZE-3C具有显著的遗传多态性,而kz - r和kz -15的品种内多态性较为适中([60]和目前的研究)。品种内多态性在商品型育种中具有重要的应用价值。
因此,随着时间的推移,很可能会有一些基因漂移,与多年前育种计划中使用的同一品系相比,今天进行的研究中包含的品系可能具有非常不同的基因组成。
独特的SNP标记及其簇分布
本研究的目的之一是为每个品种识别独特的SNP标记;共鉴定885只,占测绘总数的32.4%。这一比例略低,但与其他已发表的比例相当:172份面包小麦资料的58.5%(518个映射多态snp中的303个独特snp [35])和近等基因系的56.1%(82个多态snp中43个独特snp [36])。这可能是由于在不同的研究中使用的SNP标记和种质的不同。
独特的SNP标记及其聚类倾向于分布在每条染色体的双臂远端区域,而不是在中心点区域,反映了重组事件的分布。我们的结果与报道的225份的D基因组相似山羊草属tauschii使用7,185个SNP标记[42],与面包小麦编码序列的分布一致[67].
据我们所知,这是对一组种质中特定染色体区域的独特SNP簇分布的第一个描述(图1).在我们的研究中,两个品种kazo - r和PAV-93分别在3BL和3AS染色体上有一个单簇。在TZE-3C的4AS染色体和2A染色体的不同区域发现了两个簇SAR-29.AST-2有3个簇(染色体4AS、4BS和5AS),而kz -15和KAR-90各有4个簇。遗传多样性最大的品种是AKT-39、kz -19和kz -19红霉素- 841分别有11个、29个和35个独特的SNP标记簇。识别独特的SNP标记和了解独特簇的位置可以作为制备面包小麦品种特异性标记的第一步。在其他种群中识别类似的独特SNP簇区域可能有助于研究这些片段的祖先,并表明它们在适应特定环境中的重要性。
结论
由于数据的准确性和较高的通量,该SNP标记平台为面包小麦等重要农作物的分子遗传分析提供了强大的工具。SNP标记可用于细致和精确的遗传系统发育和基因型之间的差异鉴定。独特SNP标记及其簇(包含三个以上紧密映射SNP的区域)在染色体上的分布是特定基因型的另一个决定性特征。目前的研究结果报道了来自哈萨克斯坦10个面包小麦品种的SNP标记,支持9k Infinium SNP标记技术在哈萨克斯坦小麦基因型大面板研究和在该国育种项目中应用的潜力。
对目前和历史上在哈萨克斯坦种植的小麦品种的初步分析证实了哈萨克斯坦的种质资源与世界上其他小麦种植区的相对隔离。然而,该研究也揭示了一些可能的外来种质入侵,尤其是来自北美的。这些发现的含义是,通过对来自其他项目的精粹品系的系统渗透和评估,在哈萨克育种项目中可能有相当大的遗传增益机会。哈萨克斯坦的小麦生产面积非常大,但目前产量相对较低,因此有很大的机会提高小麦产量。哈萨克斯坦育种家似乎很少利用全球精英小麦种质资源,因此有机会获得显著的遗传收益。哈萨克斯坦的情况很可能反映在获得新种质资源受到限制的其他一些区域。
附加文件
缩写
- BAC:
-
细菌人工染色体
- DPI:
-
第一工业部
- 美国东部时间:
-
表达序列标签
- GBS:
-
Genotyping-by-sequencing
- 卡斯帕·:
-
竞争性等位基因特异性聚合酶链反应
- MAS:
-
标记辅助选择,NGS,下一代测序
- 图片:
-
多态信息含量
- SNP:
-
单核苷酸多态性
- SSCP:
-
单链确认多态性
- 苏维埃社会主义共和国:
-
简单序列重复
- SRAP:
-
序列相关扩增多态性
参考文献
- 1.
莫汉M, Nair S, Bhagwat A, Krishna TG, Yano M, Bhatia CR, Sasaki T:作物基因组定位、分子标记和标记辅助选择。动物学报,1997,3:87-103。10.1023 /: 1009651919792。
- 2.
Bevan MW, Uauy C:基因组学揭示了作物改良的新前景。中国生物医学工程学报,2013,34(5):366 - 366。
- 3.
Cavanagh CR、Chao S、Wang S、Huang BE、Stephen S、Kiani S、Forrest K、Saintenac C、Brown-Guedira GL、Akhunova A、See D、Bai G、Pumphrey M、Tomar L、Wong D、Kong S、Reynolds M、da Silva ML、Bockelman H、Talbert L、Anderson JA、Dreisigacker S、Baenziger S、Carter A、Korzun V、Morrell PL、Dubcovsky J、Morell MK、Sorrells ME、Hayden MJ:全基因组比较多样性揭示了小麦六倍体地方品种和栽培品种改良的多重选择靶点。中国生物医学工程学报,2013,29(5):557 - 557。10.1073 / pnas.1217133110。
- 4.
Randhawa HS、Asif M、Pozniak C、Clarke JM、Graf RJ、Fox SL、Humphreys DG、Knox RE、DePauw RM、Singh AK、Cuthbert RD、Hucl P、Spaner D:分子标记在加拿大小麦育种中的应用。中国植物学报,2013,32(5):558 - 571。
- 5.
Gupta PK, Varshney RK, Sharma PC, Ramesh BL:分子标记及其在小麦育种中的应用。植物育种,1999,18:369-390。10.1046 / j.1439-0523.1999.00401.x。
- 6.
Khlestkina EK, Salina EA:一个普通小麦实例的SNP标记:分析方法、开发方法和比较。中国科学(d辑),2006,42:585-594。10.1134 / S1022795406060019。
- 7.
单核苷酸多态性(SNP)在植物遗传学中的应用研究进展。生物技术学报,2006,5:435-459。
- 8.
廖鹏飞,李凯:从snp到功能多态性:生物技术应用的洞察。中国生物工程学报,2010,29(5):559 - 561。10.1016 / j.bej.2009.12.021。
- 9.
Paux E, Sourdille P, Mackay I, Feuillet C:基于序列标记的小麦育种进展及其应用。生物技术进展,2012,30:1071-1088。10.1016 / j.biotechadv.2011.09.015。
- 10.
Somers DJ, Kirkpatrick R, Moniwa M, Walsh A:从六倍体小麦ESTs中挖掘单核苷酸多态性。中国生物医学工程学报,2003,36(4):431-437。10.1139 / g03 - 027。
- 11.
Barker GLA, Edwards KJ:对世界主要谷物作物单核苷酸多态性多样性的全基因组分析。植物工程学报,2009,29(3):369 - 371。10.1111 / j.1467-7652.2009.00412.x。
- 12.
Chao S, Zhang W, Akhunov E, Sherman J, Ma Y, Luo MC, Dubcovsky J:美国小麦基因来源SNP标记多态性分析(小麦l .)品种。动物育种,2009,23:23-33。10.1007 / s11032 - 008 - 9210 - 6。
- 13.
Kozlova SA, Khlestkina EK, Salina EA:利用异源多倍体小麦SNP标记的特异性特征。中国科学(d辑),2009,45:81-84。10.1134 / S1022795409010116。
- 14.
Ravel C、Praud S、Murigneux A、Canaguier A、Sapet F、Samson D、Balfourier F、Dufour P、Chalhoub B、Brunel D、Beckert M、Charmet G:面包小麦选系中单核苷酸多态性的频率(小麦l .)。中国生物医学工程学报,2006,29(4):369 - 369。10.1139 / g06 - 067。
- 15.
Kilian A, Huttner E, Wenzl P, Jaccoud D, Carling J, Caig V, Evers M, Heller-Uszynska K, Cayla C, Patarapuwadol S, Xia L, Yang S, Thomson B:快速和廉价:基于SNP和dart的作物改良全基因组分析。国际会议论文集“双螺旋:从绿色革命到基因革命”,2003年5月27-31日。编辑:Tuberosa R, Phillips RL, Gale M. Avenue Media,博洛尼亚,意大利;2005:443 - 461。
- 16.
Mammadov J, Aggarwal R, Buyyarapu R, Kumpatla S: SNP标记及其对植物育种的影响。植物基因组学学报,2012,29(5):728398-10.1155/2012。
- 17.
Berkman PJ, Lai K, Lorenc MT, Edwards D:下一代测序在小麦作物改良中的应用。中国生物医学工程学报,2012,32(5):369 -371。10.3732 / ajb.1100309。
- 18.
Wang S, Wong D, Forrest K, Allen A, Chao S, Huang BE, Maccaferri M, Salvi S, Milner SG, Cattivelli L, Mastrangelo AM, Whan A, Stephen S, Barker G, Wieseke R, Plieske J, Lillemo M, Mather D, Appels R, Dolferus R, Brown-Guedira G, Korol A, Akhunova AR, Feuillet C, Salse J, Morgante M, Pozniak C, Luo MC, Dvorak J, Morell M:利用高密度9万单核苷酸多态性分析小麦多倍体基因组多样性植物生物技术J2014.doi: 10.1111 / pbi.12183。
- 19.
王晓燕,王晓燕,王晓燕。异源多倍体作物单核苷酸序列变异的鉴定、表征和解释。植物工程学报,2012,30(5):559 - 561。10.1111 / j.1467-7652.2011.00644.x。
- 20.
Duran C, Appleby N, Edwards D, Batley J:分子遗传标记:发现、应用、数据存储和可视化。生物学报,2009,4:16-27。10.2174 / 157489309787158198。
- 21.
Chao S, Dubcovsky J, Dvorak J, Luo MC, Baenziger SP, Matnyazov R, Clark DR, Talbert LE, Anderson JA, Dreisigacker S, Glover K, Chen J, Campbell K, Bruckner PL, Rudd JC, Haley S, Carver BF, Perry S, Sorrells ME, Akhunov ED:春冬小麦连锁失衡和SNP变异的群体和基因组特异性模式(小麦l .)。中国生物医学工程学报,2010,29(6):727-10.1186/1471-2164-11-727。
- 22.
Lai K, Duran C, Berkman PJ, Lorenc MT, Stiller J, Manoli S, Hayden MJ, Forrest KL, Fleury D, Baumann U, Zander M, Mason AS, Batley J, Edwards D:从小麦下一代序列数据中发现单核苷酸多态性。植物工程学报,2012,32(4):449 - 449。10.1111 / j.1467-7652.2012.00718.x。
- 23.
Winfield MO, Wilkinson PA, Allen AM, Barker GLA, Coghill JA, Burridge A, Hall A, Brenchley RC, D 'Amore R, Hall N, Bevan MW, Richmond T, Gerhardt DJ, Jeddeloh JA, Edwards KJ:小麦异allo六倍体外显子组的靶向重测序。植物工程学报,2012,32(3):366 - 366。10.1111 / j.1467-7652.2012.00713.x。
- 24.
Allen AM, Barker GLA, Wilkinson P, Burridge A, Winfield M, Coghill J, Uauy C, Griffiths S, Jack P, Berry S, Werner P, Melichar JPE, McDougall J, Gwilliam R, Robinson P, Edwards KJ:基于外显子的六倍体小麦共显性单核苷酸多态性标记的发现与发展(小麦l .)。植物工程学报,2013,29(4):379 - 379。10.1111 / pbi.12009。
- 25.
CerealsDB.uk.net-TaqMan:。http://www.cerealsdb.uk.net/cerealgenomics/CerealsDB/SNPs/Documents/FORM_TaqMan.php]
- 26.
Bérard A, Le Paslier MC, Dardevet M, Exbrayat-Vinson F, Bonnin I, Cenci A, Haudry A, Brunel D, Ravel C:小麦高通量单核苷酸多态性基因分型(小麦属植物spp)。植物工程学报,2009,29(3):374 -374。10.1111 / j.1467-7652.2009.00404.x。
- 27.
Edwards D, Wilcox S, Barrero RA, Fleury D, Cavanagh CR, Forrest KL, Hayden MJ, Moolhuijzen P, Keeble-Gagnère G, Bellgard MI, Lorenc MT, Shang CA, Baumann U, Taylor JM, Morell MK, Langridge P, Appels R, Fitzgerald A:面包很重要:澳大利亚小麦遗传多样性的国家倡议。植物工程学报,2012,30(4):366 - 366。10.1111 / j.1467-7652.2012.00717.x。
- 28.
Trebbi D, Maccaferri M, de Heer P, S·rensen A, Giuliani S, Salvi S, Sanguineti MC, Massi A, van der Vossen EAG, Tuberosa R:硬粒小麦高通量SNP的发现与基因分型(小麦属植物硬质Desf)。中国生物工程学报,2011,29(5):555-569。10.1007 / s00122 - 011 - 1607 - 7。
- 29.
van Poecke RMP, Maccaferri M, Tang J, Truong H, Janssen A, Orsouw N, Salvi S, Sanguineti MC, Tuberosa R, Vossen EAG:基于序列的小麦硬粒SNP基因分型。植物工程学报,2013,29(4):369 - 371。10.1111 / pbi.12072。
- 30.
任军,孙东,陈磊,尤丰芳,王杰,彭勇,Nevo E,孙东,罗春春,彭杰:利用单核苷酸多态性标记揭示世界范围内硬粒小麦种质资源的遗传多样性。中华分子生物学杂志,2013,31(4):371 - 371。10.3390 / ijms14047061。
- 31.
任军,陈亮,孙东,尤丰梅,王杰,彭勇,Nevo E, Beiles A,孙东,罗MC,彭杰:野生二粒小麦snp位点遗传多样性与生态因子的相关性。中国生物医学工程学报,2013,29(3):366 - 366。
- 32.
Akhunov ED、Akhunova AR、Anderson OD、Anderson JA、Blake N、Clegg MT、Coleman-Derr D、Conley EJ、Crossman CC、Deal KR、Dubcovsky J、Gill BS、Gu YQ、Hadam J、Heo H、Huo N、Lazo GR、Luo MC、Ma YQ、Matthews DE、McGuire PE、Morrell PL、Qualset CO、Renfro J、Tabanao D、Talbert LE、Tian C、Toleno DM、Warburton ML、You FM:核苷酸多样性图谱揭示了小麦基因组和染色体的多样性变化。中国生物医学工程学报,2010,26(6):366 - 366。
- 33.
Iehisa JCM, Shimizu A, Sato K, Nishijima R, Sakaguchi K, Matsuda R, Nasuda S, Takumi S:基于野生小麦前体转录本单核苷酸多态性的小麦D基因组标记开发山羊草属tauschii.中国生物医学工程学报,2014,29(4):371 - 371。10.1007 / s00122 - 013 - 2215 - 5。
- 34.
Shatalina M, Wicker T, Buchmann JP, Oberhaensli S, Šimková H, dolezel J, Keller B:基于全染色体3B序列信息的小麦品种Arina和Forno基因型特异性SNP图谱。植物工程学报,2013,11:23-32。10.1111 / pbi.12003。
- 35.
Würschum T、Langer S、Longin C、Korzun V、Akhunov E、Ebmeyer E、Schachschneider R、Schacht J、Kazman E、Reif J:利用SNP和SSR标记评价优秀冬小麦群体结构、遗传多样性和连锁不平衡。中国科学(d辑),2013,26(4):557 - 557。10.1007 / s00122 - 013 - 2065 - 1。
- 36.
Trick M, Adamski NM, Mugford SG, Jiang CC, feb M, Uauy C:结合新一代测序数据中的SNP发现和散装分离分析(BSA)对多倍体小麦基因进行精细定位。植物工程学报,2012,29(3):366 - 366。
- 37.
Filiz E: CIMMYT面包小麦遗传多样性分析(小麦L.)系的SRAP标记。植物育种学报,2012,3:956-963。
- 38.
Poland JA, Brown PJ, Sorrells ME, Jannink JL:用一种新的双酶基因测序分型方法建立大麦和小麦高密度遗传图谱。科学通报,2012,7:e32253-10.1371/journal.pone.0032253。
- 39.
王晓燕,王晓燕,王晓燕,王晓燕。标记确定偏倚对基因组选择准确性和遗传多样性估计的影响。科学通报,2013,8:e74612-10.1371/journal.pone.0074612。
- 40.
You FM, Huo N, Deal KR, Gu YQ, Luo MC, McGuire PE, Dvorak J, Anderson OD:基于注释的大而复杂基因组SNP发现山羊草属tauschii使用下一代基因组测序而没有参考基因组序列。中国生物医学工程学报,2011,29(5):563 - 566。
- 41.
罗MC、顾永强、尤fm、Deal KR、马勇、胡勇、霍宁、王勇、王杰、陈s、Jorgensen CM、张勇、McGuire PE、Pasternak S、Stein JC、Doreen W、Kramer M、McCombie WR、Kianian SF、Martis MM、Mayer KFX、Sehgal SK、Li W、Gill BS、Bevan MW、Šimková H、dolezel J、Weining S、Lazo GR、Anderson OD:一个4千兆酶物理图谱解开了复杂基因组的结构和进化山羊草属tauschii即小麦d基因组祖先。中国生物工程学报,2013,38(4):379 - 379。10.1073 / pnas.1219082110。
- 42.
王杰、罗mc、陈铮、尤美芳、魏勇、郑勇、德沃夏克:山羊草属tauschii单核苷酸多态性揭示了小麦d基因组遗传多样性的起源,并确定了六倍体小麦的地理起源。新植物学报,2013,32(4):394 - 394。10.1111 / nph.12164。
- 43.
Wilkinson PA, Winfried MO, Barker GLA, Allen AM, Burridge A, Coghill JA, Edwards K: CerealsDB 2.0:植物育种家和科学家的综合资源。生物信息学,2012,13:219-10.1186/1471-2105-13-219。
- 44.
Kono TJY, Seth K, Poland JA, Morrell PL: SNPMeta:没有参考基因组的SNP注释和SNP元数据收集。中国生物医学工程学报,2014,31(4):419-425。10.1111 / 1755 - 0998.12183。
- 45.
Goncharov NP, Goncharov PL:植物育种的方法基础。地质学术出版社,新西伯利亚;2009.
- 46.
Wheat-atlas:。http://wheatatlas.org/country/varieties]
- 47.
Wheat-pedigree:。http://www.wheatpedigree.net]
- 48.
夏普PJ,克瑞斯M,谢瑞PR,盖尔MD:位置β-淀粉酶序列。应用科学学报,1988,30(4):369 - 371。10.1007 / BF00303966。
- 49.
- 50.
Gascuel O: BIONJ:基于序列数据的简单模型的NJ算法的改进版本。中华分子生物学杂志,1997,14:685-695。10.1093 / oxfordjournals.molbev.a025808。
- 51.
胡森DH,布莱恩特D:系统发育网络在进化研究中的应用。中华分子生物学杂志,2006,23:254-267。10.1093 / molbev / msj030。
- 52.
SplitsTree4:。http://www.splitstree.org]
- 53.
PowerMarker, 3.25版本:.,[http://statgen.ncsu.edu/powermarker]
- 54.
- 55.
郝超,王磊,葛辉,董艳,张鑫:中国面包小麦遗传多样性与连锁失衡(小麦L.)。科学通报,2011,6:e17279-10.1371/journal.pone.0017279。
- 56.
Gill BS, Appels R, bota - oberholster AM, Buell CR, Bennetzen JL, Chalhoub B, Chumley F, Dvořák J, Iwanaga M, Keller B, Li W, McCombie WR, Ogihara Y, Quetier F, Sasaki T:小麦基因组测序研讨会报告:国际小麦基因组联盟研究。遗传学报,2004,38(4):369 - 369。10.1534 / genetics.104.034769。
- 57.
Marcussen T, Sandve SR, Heier L, Spannagl M, Pfeifer M, Jakobsen KS, Wulff BBH, Steuernagel B, Mayer KFX, Olsen OA:面包小麦祖先基因组中的古代杂交。科学通报,2014,38(5):559 - 559 /科学通报。
- 58.
刘长杰,Atkinson MD, Chinoy CN, Devos KM, Gale MD:小麦和黑麦4、5和7组染色体间的非同源易位。应用理论与实践,1998,30(3):354 - 354。10.1007 / BF00224276。
- 59.
Devos KM, Dubcovsky J, Dvořák J, Chinoy CN, Gale MD:小麦染色体4A、5A和7B的结构演化及其对重组的影响。《应用科学学报》,1995,29(6):676 - 682。10.1007 / BF00220890。
- 60.
Eliby S, Shavrukov Y, Ismagul A, Langridge P, Abugalieva AI, Kenenbaev SB:遗传作图方法在春小麦育种中的应用。生物技术理论实践,2012,2:46-59。doi: http://dx.doi.org/10.11134/btp.2.2012.5 [http://dx.doi.org/10.11134/btp.2.2012.5]
- 61.
《农作物鉴定方法说明》,1947年,国家农业出版社,《谷物》,莫斯科
- 62.
Dorofeev VF:抗冻抗旱材料的评价。作物遗传资源的今天和明天。编辑:Frankel OH, Hawkes JG。CUP档案馆,剑桥,伦敦,纽约,墨尔本;1975:211 - 222。
- 63.
- 64.
Martynov SP, Dobrovotvorskaya TV, Morvounov AI, Urazaliev RA, Absattarova AS: 1929-2004年哈萨克斯坦春面包小麦品种多样性的系谱分析。中国农学通报,2005,29(5):371 - 371。10.1556 / AAgr.53.2005.3.3。
- 65.
Abugalieva SI、Volkova LA、Ermekbaev KA、Turuspekov EK:利用微卫星dna标记对哈萨克斯坦春面包小麦商品品种进行基因分型。生物技术理论实践,2012,2:35-45。doi: http://dx.doi.org/10.11134/btp.2.2012.4 [http://dx.doi.org/10.11134/btp.2.2012.4]
- 66.
苏德斯、贝恩斯、南达GS:基于微卫星标记和谱系分析的小麦基因型间遗传关系。中国生物医学工程学报,2005,29(4):379 -379。
- 67.
Kumar A, Sharma M:小麦基因组系统发育和改良。作物学报,2011,5:1120-1126。
确认
我们感谢来自澳大利亚维多利亚州DPI的Mathew J. Hayden对SNP标记的分析,以及来自澳大利亚阿德莱德大学ACPFG的Anzu Okada和Julie Hayes对DNA提取的帮助和对手稿的批评意见。该研究得到了KazAgroInnovation(哈萨克斯坦阿斯塔纳)和阿德莱德大学(澳大利亚)澳大利亚植物功能基因组学中心(ACPFG)的资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明他们没有竞争利益。
作者的贡献
YS、SE、PL和AA设计了研究方案;YS进行了研究;RS进行计算机系统发育分析;YS、SE、AA、SK对结果进行分析;YS撰写了手稿,PL对结果的解释做出了贡献。所有作者阅读并批准了最终稿件。
电子辅料
权利和权限
本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(http://creativecommons.org/licenses/by/4.0),它允许在任何媒体上不受限制地使用、分发和复制,只要原始作品的名称正确。创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条提供的资料。
关于这篇文章
引用这篇文章
Y.沙夫鲁科夫,R.苏切奇,S.埃利比。et al。应用下一代测序技术研究哈萨克斯坦面包小麦的遗传多样性和识别独特的SNP标记。BMC植物杂志14,258(2014)。https://doi.org/10.1186/s12870-014-0258-7
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-014-0258-7
关键字
- 面包小麦
- 遗传多态性
- 独特标记的集群
- 基因种系发生
- 哈萨克斯坦
- 下一代测序(门店)
- 单核苷酸多态性(SNP)
- 独特的标记