- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
芝麻籽粒产量相关性状高密度遗传图谱构建及qtl分析gydF4y2Ba胡麻属indicumgydF4y2BaL.)基于RAD-Seq技术gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba14gydF4y2Ba文章编号:gydF4y2Ba274gydF4y2Ba(gydF4y2Ba2014gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
芝麻(gydF4y2Ba胡麻属indicumgydF4y2BaL., 2n = 26)是一种重要的油料作物,估计基因组大小为369 Mb。芝麻籽粒产量和品质的遗传基础,包括定量性状位点(qtl)的数量和位置仍然知之甚少,部分原因是缺乏可靠的标记和遗传图谱。本文报道了利用限制性位点相关DNA测序(RAD-seq)结合89个PCR标记构建迄今为止密度最高的芝麻遗传图谱,并利用重组自交系(RIL)群体鉴定了与籽粒产量相关的qtl。gydF4y2Ba
结果gydF4y2Ba
从RAD-seq中共鉴定到3769个单核苷酸多态性(SNP)标记,89个多态PCR标记,包括44个表达序列标记-简单序列重复序列(EST-SSRs), 10个基因组- ssrs和35个插入-缺失标记(InDels)。最终图谱包含1230个标记,分布在14个连锁群上,长度为844.46 cM,相邻标记间平均为0.69 cM。利用该图谱和RIL群体,利用多重区间映射(MIM)和混合线性复合区间映射(MCIM)分别在7个籽粒产量相关性状的7个LGs上检测到13个qtl,在10个LGs上检测到17个qtl。采用MIM + R法鉴定了3个主要qtlgydF4y2Ba2gydF4y2Ba> 10.0%或MCIM与hgydF4y2Ba一个gydF4y2Ba2gydF4y2Ba> 5.0%。两个共定位QTL组可以部分解释5个产量相关性状之间的相关性。gydF4y2Ba
结论gydF4y2Ba
RAD-seq开发了包括SNPs和InDels在内的3,800对新的DNA标记,并结合SSR标记构建了迄今为止密度最高的遗传图谱。利用该群体和遗传图谱鉴定了几个与粮食产量相关的qtl。本文首次报道了利用芝麻RIL群体的高密度遗传图谱对产量相关性状进行QTL定位。本研究结果为研究芝麻重要农艺性状和应用标记辅助选择技术进行遗传改良奠定了基础。gydF4y2Ba
背景gydF4y2Ba
芝麻(gydF4y2Ba胡麻属indicumgydF4y2BaL.)是一种重要而古老的油料作物[gydF4y2Ba1gydF4y2Ba].它是一个二倍体物种(2n = 26),估计基因组大小为369 Mb [gydF4y2Ba2gydF4y2Ba].与油菜籽、花生、大豆等油料作物相比,芝麻含油量最高[gydF4y2Ba3.gydF4y2Ba].它还富含蛋白质、维生素和特定的抗氧化剂,如芝麻素和芝麻素[gydF4y2Ba4gydF4y2Ba]、[gydF4y2Ba5gydF4y2Ba,是健康食品的最佳选择之一。随着芝麻市场需求的迅速增长,利用遗传方法稳定提高芝麻籽粒产量已成为重要目标之一。单株芝麻籽粒产量由单株荚果数、单株荚果粒数和粒重三部分组成。株高、蒴果(花)长和第一蒴果轴高与芝麻籽粒产量密切相关[gydF4y2Ba6gydF4y2Ba].由于与粮食产量相关的性状是定量遗传的,受多个对环境敏感的基因控制,因此需要对这些性状的遗传进行qtl定位分析[gydF4y2Ba7gydF4y2Ba].高密度遗传图谱是检测水稻qtl的一种非常有效和重要的方法[gydF4y2Ba8gydF4y2Ba] - [gydF4y2Ba11gydF4y2Ba和其他作物[gydF4y2Ba12gydF4y2Ba] - [gydF4y2Ba14gydF4y2Ba].遗憾的是,由于缺乏可靠的DNA标记和基于永久群体构建的遗传图谱,目前在芝麻中尚未发现与产量相关的qtl或基因。gydF4y2Ba
利用F基因构建了第一个芝麻遗传连锁图谱gydF4y2Ba2gydF4y2Ba由' COI1134 '(白色种皮)和' RXBS '(黑色种皮)杂交而来的居群[gydF4y2Ba15gydF4y2Ba].该图谱遗传长度为936.72 cM,平均标记距离为4.93 cM。它包含220个标记,包括8个表达序列标记简单序列重复序列(EST-SSRs), 25个扩增片段长度多态性(AFLPs)和187个随机选择微卫星多态性位点(RSAMPLs),分布在30个连锁组上,是芝麻单倍体基因组染色体数量的2倍多。后来,又有14个由RNA-seq发展而来的基因ssrs被整合到这张地图上[gydF4y2Ba16gydF4y2Ba].最近,这张地图通过使用放大F放置更多的标记得到了实质性的改进gydF4y2Ba2gydF4y2Ba人口(gydF4y2Ba17gydF4y2Ba].这使得LG的数量减少到14个,只比芝麻的单倍体染色体多1个LG。新图谱的遗传长度为1216 cM,标记密度为1.86 cM /标记区间。检测到4个控制种皮颜色的qtl,遗传率为59.33% ~ 69.89%gydF4y2Ba3.gydF4y2Ba人群。gydF4y2Ba
大规模并行的下一代测序(NGS)平台的出现不断降低了成本,为全基因组标记物的开发和通过测序进行基因分型(GBS)提供了前所未有的机会。几种NGS方法与限制性内切酶酶切相结合,降低了目标基因组的复杂性,使测序负载和成本显著下降[gydF4y2Ba18gydF4y2Ba],同时仍然能够发现数千个单核苷酸多态性(SNPs)或插入缺失(InDels)标记[gydF4y2Ba19gydF4y2Ba] - [gydF4y2Ba21gydF4y2Ba].限制性内切位点相关DNA测序(RAD-seq)是一种NGS方法,它只对特定限制性内切酶位点侧的DNA进行测序,以产生基因组的简化表示,该方法连接了一个包含简化表示文库(RRLs)中的多重标识符(mid)的连接器[gydF4y2Ba22gydF4y2Ba] - [gydF4y2Ba27gydF4y2Ba].通过这些方法,在茄子中构建了几个高密度遗传图谱[gydF4y2Ba28gydF4y2Ba],黑麦草[gydF4y2Ba13gydF4y2Ba],大麦[gydF4y2Ba14gydF4y2Ba)、葡萄(gydF4y2Ba27gydF4y2Ba甚至还有芝麻[gydF4y2Ba29gydF4y2Ba].近年来,以F为基础构建了芝麻高密度遗传图谱gydF4y2Ba2gydF4y2Ba采用特定长度扩增片段测序(SLAF-seq)技术,该技术是一种增强的RRL测序策略gydF4y2Ba新创gydF4y2Ba在大人群中发现SNP [gydF4y2Ba21gydF4y2Ba]、[gydF4y2Ba29gydF4y2Ba].该图谱由1233个SLAF标记组成,分布在15个连锁群上,全长1474.87 cM,平均标记间距为1.20 cM。总的来说,所有三个发表的芝麻遗传图谱都不是理想的定量性状图谱,因为它们都是基于一个临时群体(FgydF4y2Ba2gydF4y2Ba),使重复表现型变得不可行[gydF4y2Ba30.gydF4y2Ba].此外,由于这些地图缺乏共同的标记,所以没有可比性。gydF4y2Ba
本研究通过对“中植14号”和“妙前芝马”两个芝麻品种的RAD-seq分析,共鉴定出37069对SNP标记。这些标记与之前报道的1,195个EST-SSR或基因组- ssr和79个InDel标记结合[gydF4y2Ba31gydF4y2Ba],利用重组自交系(RIL)群体构建了芝麻高密度遗传图谱。我们进一步提出了基于这些新的基因组资源的谷物产量相关qtl的鉴定。gydF4y2Ba
结果gydF4y2Ba
RAD测序,snp和InDels发现gydF4y2Ba
共获得62.57 Gb高质量序列数据,包含312,829,823对端读。224个RILs的读数从598,119到3,483,606不等,平均为1,644,718。对于两个父母来说,3,030,776次读取来自于女性父母,3,881,579次读取来自于男性父母。之后,从男性和女性父母身上识别出的rad标签数量分别为23.1万个和20.7万个。雄性亲本个体标签的平均覆盖率为16.80倍,雌性亲本为14.64倍。两个亲本之间可比较的rad -tag数为47247。然而,在RIL人群的两个亲本中,仅鉴定出3769个SNP。这些snp多为过渡型snp, Y(T/C)型和R(G/A)型分别占30.43%和30.78%gydF4y2Ba1gydF4y2Ba).除snp外,还鉴定出97个≥2 bp的InDels,其中79个成功设计用于进一步PCR验证和群体基因型分析[gydF4y2Ba31gydF4y2Ba].gydF4y2Ba
结合已发表的芝麻ssr,在两个亲本的基因组DNA上共检测了1061个EST-SSRs、134个基因组-SSRs和79个InDels。89个PCR标记检测到多态性,包括44个EST-SSRs, 10个基因组- ssrs和35个InDels。EST-SSRs、基因组- ssrs、InDels和SNPs标记检测亲本间多态性的效率从EST-SSRs的5.0%到InDels的46.7%不等。这些多态SSR和InDel标记均检测到共显性位点。gydF4y2Ba
基因映射gydF4y2Ba
在对这些标记进行基因定位之前,我们排除了在RIL人群中缺失数据超过40%的656个SNP标记和1个InDel标记。另外,1786个SNPs、15个InDels、24个EST-SSRs和4个基因组- ssrs也被排除在外,因为它们的模式过于扭曲,次要等位基因频率分离比小于0.29。因此,使用最终的1327个SNPs, 19个InDels和26个SSRs,其中大部分以共显性方式遗传,用于遗传图谱的构建(TablegydF4y2Ba1gydF4y2Ba).gydF4y2Ba
结果,1230个标记,包括1190个SNPs, 22个SSRs和18个InDels被映射到14个不同的LGs上,覆盖芝麻基因组844.46 cM,相邻标记之间的平均距离仅为0.69 cM(图1)gydF4y2Ba1gydF4y2Ba额外的文件gydF4y2Ba2gydF4y2Ba).每个标尺的长度在6.08厘米到130.52厘米之间,每标尺的平均标记距离在0.23厘米到1.92厘米之间,每标尺的标记数量在26到227厘米之间gydF4y2Ba2gydF4y2Ba).除lgg2、LG8、LG9、LG10和LG14外,9个lgg上分布有16个大于10 cM的缝隙,最大的缝隙位于LG6,为22.54 cM。这些空白大部分位于接近连接组的末端(图gydF4y2Ba1gydF4y2Ba),这被认为反映了染色体远端区域的高水平重组[gydF4y2Ba39gydF4y2Ba]、[gydF4y2Ba40gydF4y2Ba].此外,SSR、InDel和SNP标记在不同lgg上的分布是随机的,每个lgg的SSR和InDel标记的分布都不超过10%。gydF4y2Ba
一千一百一十五个标记以预期的1:1比例在种群中分离。然而,包括4个SSRs, 2个InDels和109个SNPs在内的115个标记的分离明显偏离了这一比例(gydF4y2BaPgydF4y2Ba< 0.05)(表gydF4y2Ba2gydF4y2Ba).77个(61.1%)分离畸变标记的基因型频率向‘中植14号’倾斜,49个(38.9%)向‘妙前芝马’倾斜。除SBN1614、SBN3567和GSSR074外,大多数标记对地图距离的计算没有影响。与SNP和InDel标记相比,SSR标记的倾斜率最高,为17.4%。这些分离畸变标记分布在除LG14外的13个lgg上。最大的LG4有227个标记,分离畸变标记最多。LG12的偏析畸变标记频率为39.4%,远高于其他LG12。在LG2、LG4、LG6和LG12 4个lgg上检测到4个分离畸变区(SDR)gydF4y2Ba2gydF4y2Ba).这些sdr大多分布在其gs的末端附近,每个sdr有3 ~ 5个倾斜标记,占图中倾斜标记总数的14.3%。4个ssr的偏斜标记多为SNP型,其中一个EST-SSR标记(ZM1197)和一个InDel标记(SBI035)在ssr - lg4中偏斜。在SDR-LG2、SDR-LG6和SDR-LG12中,所有标记的基因型频率均向‘中植14’倾斜,而在SDR-LG4中向‘苗前芝马’倾斜。gydF4y2Ba
表型分析gydF4y2Ba
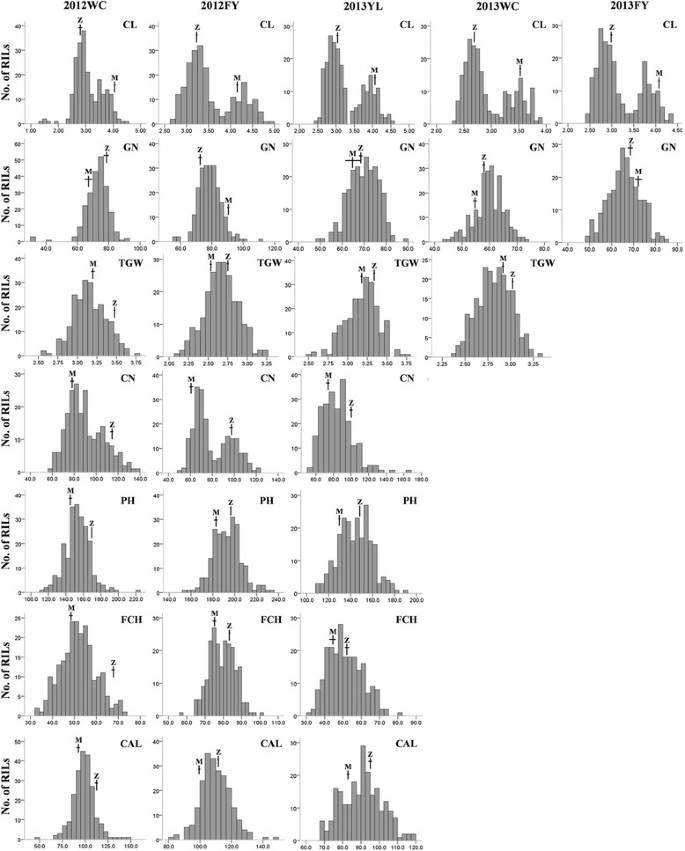
在所有试验中,7个与产量相关的性状在亲本系间表现出显著差异。与苗前芝马相比,父本中植14株高(PH)、第一蒴果高(FCH)、蒴果轴长(CAL)、单株蒴果数(CN)、蒴果长(CL)和千粒重(TGW)显著增加(图)gydF4y2Ba2gydF4y2Ba).2013财年和2013年wc的PH、FCH、CAL和TGW由于极端天气导致的野外表现不佳而被遗漏。有趣的是,中植14号的平均粒数(GN)在武昌(2012WC、2013WC)高于妙前芝马,而在阜阳(2012财年、2013财年)则低于妙前芝马。所有性状在RIL群体中均呈连续分布和海侵分离gydF4y2Ba2gydF4y2Ba),表明由多个基因控制。PH、FCH、CAL、GN和TGW的近正态曲线分布表明遗传控制为多基因模式;CL和CN呈双峰分布,提示主效基因参与。方差分析(ANOVA)表明,各试验性状的行间变异均显著gydF4y2BaPgydF4y2Ba= 0.001。7个性状的广义遗传力介于29.8% (FCH)至95.7% (CN)之间(表2)gydF4y2Ba3.gydF4y2Ba).各性状的遗传力与其相应的分布是一致的。gydF4y2Ba
试验范围内所有七个性状的相关系数在gydF4y2BaPgydF4y2Ba= 0.01(附加文件gydF4y2Ba3.gydF4y2Ba).不同环境(年份或地点)间的关联度均在0.80以上,相关性较弱,关联度在0.27 ~ 0.35之间。在可获得表型数据的三个环境(2012WC, 2012FY和2013YL)中,PH与FCH (gydF4y2BaPgydF4y2Ba≤0.01),PH和CAL (gydF4y2BaPgydF4y2Ba≤0.01),PH和TGW (gydF4y2BaPgydF4y2Ba≤0.05),FCH和TGW (gydF4y2BaPgydF4y2Ba≤0.05),甚至CL和GN (gydF4y2BaPgydF4y2Ba≤0.01),CN与TGW呈显著负相关(gydF4y2BaPgydF4y2Ba≤0.05)(表gydF4y2Ba4gydF4y2Ba).更有趣的是,GN与TGW在2012FY (gydF4y2BaPgydF4y2Ba≤0.01),但与2013YL (gydF4y2BaPgydF4y2Ba≤0.01)。gydF4y2Ba
QTL分析gydF4y2Ba
利用多区间映射(MIM)方法在7个连锁群上共发现13个产量相关qtl。单个性状检测到的qtl范围为1 ~ 3个gydF4y2Ba5gydF4y2Ba).6个qtl可在多个试验中检测到,包括gydF4y2BaQph-12gydF4y2Ba,gydF4y2BaQtgw-11gydF4y2Ba,gydF4y2BaQgn-1gydF4y2Ba,gydF4y2BaQgn-6gydF4y2Ba,gydF4y2BaQgn-12gydF4y2Ba而且gydF4y2BaQcl-12gydF4y2Ba,而其他的则可以通过两个软件进行重复。除中植14外,其余大部分均表现出正加性效应gydF4y2BaQgn-12gydF4y2Ba而且gydF4y2BaQcl-12。gydF4y2Ba检测到6个主效qtl,表型效应(RgydF4y2Ba2gydF4y2Ba)超过10%,包括1个QTL,gydF4y2BaQcl-12gydF4y2Ba,显示RgydF4y2Ba2gydF4y2Ba范围在52.2%到75.6%之间。gydF4y2Ba
在混合线性复合区间映射(MCIM)算法下,用QTLNetwork 2.0进行QTL映射,分析主要加性效应(gydF4y2Ba一个gydF4y2Ba),加性-加性上位效应(gydF4y2BaaagydF4y2Ba)和环境加性交互效应(gydF4y2BaaegydF4y2Ba在multi-trials)。在10个连锁群上共检测到17个qtlgydF4y2Ba3.gydF4y2Ba).他们都有显著的gydF4y2Ba一个gydF4y2Ba效果,gydF4y2BaQgn-6gydF4y2Ba也有重要的gydF4y2BaaegydF4y2Ba影响在gydF4y2BaPgydF4y2Ba在2013财政年度≤0.05。均表现出显著的加性效应gydF4y2BaPgydF4y2Ba≤0.001,解释了1.70 ~ 45.39%的表型变异,4个主要qtl均大于5.0%。首囊高度有2个qtl,gydF4y2BaQfch-4gydF4y2Ba而且gydF4y2BaQfch-12gydF4y2Ba,均检出显著gydF4y2BaaagydF4y2Ba效应解释了1.59%的表型变异(表gydF4y2Ba3.gydF4y2Ba).gydF4y2Ba
我们还比较了使用MIM和MCIM识别的7个不同产量相关性状的qtl。两种方法共检测到13个QTL, QTL区域相似gydF4y2BaQcl-3gydF4y2Ba,gydF4y2BaQcl-4gydF4y2Ba,gydF4y2BaQcl-7gydF4y2Ba而且gydF4y2BaQcl-8gydF4y2Ba只有MCIM检测到。用两种方法检测3个主效qtlgydF4y2Ba2gydF4y2Ba> 10.0%或hgydF4y2Ba一个gydF4y2Ba2gydF4y2Ba> 5.0%,包括gydF4y2BaQtgw-11gydF4y2Ba,gydF4y2BaQgn-6gydF4y2Ba而且gydF4y2BaQcl-12gydF4y2Ba.此外,gydF4y2BaQph-12gydF4y2Ba而且gydF4y2BaQfch-12gydF4y2Ba,由仲植14贡献gydF4y2BaQcl-12gydF4y2Ba由Miaoqianzhima贡献,均位于同一地点。三个主要gydF4y2BaQfch-11gydF4y2Ba而且gydF4y2BaQtgw-11gydF4y2Ba忠植14、gydF4y2BaQcn-11gydF4y2Ba由Miaoqianzhima贡献,位于LG11连锁群上。gydF4y2Ba
讨论gydF4y2Ba
芝麻高密度遗传图谱的构建gydF4y2Ba
在本研究中,只有44个(5.0%)EST-SSRs和10个(9.3%)基因组- ssrs在作图人群中具有多态性,因此可用于遗传图谱的构建。这一多态率远远低于之前芝麻的许多报告[gydF4y2Ba16gydF4y2Ba]、[gydF4y2Ba32gydF4y2Ba]、[gydF4y2Ba34gydF4y2Ba,表明父母之间的基因差异较小。然而,多亏了高通量的RAD-Seq技术,我们能够从约40k可比的rad -标签中发现超过3000个snp和几十个InDels。snp在全基因组中的阳性率为7.98%,高于Zhang等报道的5.12%。[gydF4y2Ba29gydF4y2Ba].观察到大多数snp属于Y(T/C)型(30.43%)和R(G/A)型(30.78%),与之前在芝麻中报道的情况一致[gydF4y2Ba29gydF4y2Ba和其他物种,甚至包括人类[gydF4y2Ba41gydF4y2Ba].gydF4y2Ba
此外,本研究的映射居群是芝麻中首次报道的,也是最大的永久性映射居群。与其他已发表的芝麻遗传图谱相比,本文构建的图谱具有最高的标记密度、相似的连锁群数量gydF4y2Ba胡麻属indicumgydF4y2BaL.染色体(2n = 26),畸变标记更少,间隙更小[gydF4y2Ba15gydF4y2Ba]、[gydF4y2Ba17gydF4y2Ba]、[gydF4y2Ba29gydF4y2Ba].此外,本研究排除了2442个(64.8%)SNP标记和44个(49.4%)多态性PCR标记过度缺失或扭曲的标记,而Zhang等报道的超过65.4%的标记因其出乎意料的分离模式被丢弃。[gydF4y2Ba29gydF4y2Ba].115个(9.35%)标记表现出明显的分离畸变(gydF4y2BaPgydF4y2Ba<0.05), 205例(16.63%)[gydF4y2Ba29gydF4y2Ba]及79 (10.91%)[gydF4y2Ba17gydF4y2Ba)在芝麻的另外两个基因图谱上。在我们地图的4个lg上检测到4个sdr,在SLAF地图的11个lg上检测到18个sdr [gydF4y2Ba29gydF4y2Ba].多数分布在LGs末端,可能参与配子、合子或其他选择[gydF4y2Ba42gydF4y2Ba]、[gydF4y2Ba43gydF4y2Ba].这里报告的地图尺寸为844.46厘米,比之前出版的1216厘米和1474厘米明显短。这可能是由于丢弃了少于20个标记的链接组,以及在我们的地图中分离失真标记和sdr较少。更重要的是,我们图谱上的几个PCR标记将为芝麻的图谱、基因或qtl的比较提供非常有用的信息。因此,本研究构建的高密度遗传图谱结合了芝麻两种老图谱的优点,将是QTL/基因定位、比较基因组学分析、基于图谱克隆等方面的理想图谱。但需要指出的是,该基因图谱主要基于两个芝麻品种之间的SNP,且SNP侧翼序列仅为85 bp,因此作为研究群体的通用工具存在一定的局限性。gydF4y2Ba
利用芝麻高密度遗传图谱鉴定籽粒产量相关qtlgydF4y2Ba
由于籽粒产量是一种复杂的数量性状,受多基因控制,且对环境敏感,因此,为了获得可靠的QTL定位,必须对产量相关性状进行重复表型分析。在这里,永久分离群体(RIL)的可用性使得随着时间和位置的重复显型成为可能。因为明显(gydF4y2BaPgydF4y2Ba= 0.01)各性状在不同环境间均存在相关性,为QTL定位提供了可靠的表型数据。然而,试验范围内的相关系数CAL低于0.351,CN低于0.509分别表示弱相关或中等相关。在MIM和MCIM均可检测到CAL和CN的3个qtl,但仅在一种环境中检测到。gydF4y2Ba
采用MIM和MCIM方法分别检测到7个LGs上的13个和10个LGs上的17个产量相关qtl。这些都是首次报道的芝麻籽粒产量相关qtl,它们都可以通过多个试验或两种算法检测到。7个产量相关性状的遗传控制主要由几个主qtl和几个小qtl组成。MIM + R法检测出3个主要qtlgydF4y2Ba2gydF4y2Ba> 10.0%或MCIM与hgydF4y2Ba一个gydF4y2Ba2gydF4y2Ba> 5.0%。利用MIM和MCIM共鉴定出7个产量相关性状的10个小qtl。另一方面,我们发现了一个QTL (gydF4y2BaQgn-6gydF4y2Ba显示重要gydF4y2BaaegydF4y2Ba有一对qtl对FCH具有显著性gydF4y2BaaagydF4y2Ba的效果。几个gydF4y2BaaegydF4y2Ba或gydF4y2BaaagydF4y2Ba与产量相关的qtl在小麦中的作用也有报道[gydF4y2Ba44gydF4y2Ba,大豆gydF4y2Ba45gydF4y2Ba]、油菜[gydF4y2Ba46gydF4y2Ba等等。这些法gydF4y2Ba一个gydF4y2Ba,gydF4y2BaaegydF4y2Ba或gydF4y2BaaagydF4y2Ba效应将是芝麻增产的重要共性和特殊性信息。gydF4y2Ba
此外,一些与产量相关的性状之间存在显著的相关性,这表明这些性状是由密切相关或多效性的遗传因素控制的。然后通过这些性状的几个qtl的共定位进行了验证。了的gydF4y2BaQph-12gydF4y2Ba而且gydF4y2BaQfch-12gydF4y2Ba,均来自中植14等位基因,均符合PH与FCH显著正相关关系。FCH与TGW呈正相关,而CN与TGW或CN与FCH呈负相关。相应地,gydF4y2BaQfch-11gydF4y2Ba而且gydF4y2BaQtgw-11gydF4y2Ba中植14等位基因具有正相加效应gydF4y2BaQcn-11gydF4y2Ba具有负相加效应的苗前芝马等位基因,均紧密分布在LG11上。然而,并不是所有的相关性都可以用QTL共定位来解释,如CL和GN, PH和CN。这些矛盾可能是由于未检测到的qtl的影响,也可能是多效性或连锁以外的原因。gydF4y2Ba
芝麻育种的未来展望与挑战gydF4y2Ba
提高产量是芝麻育种的重要目标之一;然而,这是一个耗时和繁琐的项目,因为在这个过程中涉及多个复杂和环境敏感的组件。本研究的产量相关qtl的鉴定为芝麻产量性状的标记辅助选择奠定了初步基础。尽管如此,对于一些LOD评分较低的小qtl,在将其用于育种之前需要进一步验证。另一方面,产量相关qtl的上位互作和共定位可能有利于或不利于期望位点的金字塔,这取决于它们的模式。积极的gydF4y2BaaagydF4y2Ba的影响gydF4y2BaQfch-4gydF4y2Ba而且gydF4y2BaQfch-12gydF4y2Ba提示这两个qtl的整合将有利于本研究FCH的改善。密切位于gydF4y2BaQtgw-11gydF4y2Ba而且gydF4y2BaQcn-11gydF4y2Ba对TGW和CN均有显著的加性效应,但有利等位基因由不同亲本携带。因此,对连锁或上位性qtl进行精确解剖,或筛选具有独立有利等位变异的种质,以促进育种仍有很多工作要做。gydF4y2Ba
本研究发现,具有正向加性效应的qtl大多来自具有多个高产性状的优良商品品种中植14号的等位基因。而在GN和CN中,有两个鉴定的qtl由苗千芝马贡献。说明利用妙前芝马等位基因引入这两个qtl将进一步提高中植14号的GN和CN。此外,我们还发现,使用QTLNetwork 2.0预测的“优势系”对GN值的基因型效应显著高于两个亲本[gydF4y2Ba47gydF4y2Ba)(数据未显示)。因此,该RIL群体在提高蒴果粒数方面具有很大的育种潜力。该基因分型群体与高密度遗传图谱相结合,也将为研究产量、籽粒含油量、蛋白质含量、耐胁迫性等重要农业性状提供有效的研究体系。gydF4y2Ba
结论gydF4y2Ba
本报告首次利用RIL群体对芝麻产量相关性状进行QTL定位,并构建了高密度遗传图谱。我们通过RAD标记测序开发了3769个SNPs标记,并结合SSR和InDel标记构建了迄今为止最多的14个LGs的高密度遗传图谱。利用该RIL群体和遗传图谱,在多个试验中或采用MIM和MCIM方法同时检测到多个与粮食产量相关的qtl,包括3个带R的主要效应qtlgydF4y2Ba2gydF4y2Ba> 10.0%或hgydF4y2Ba一个gydF4y2Ba2gydF4y2Ba> 5.0%。3个qtl显著gydF4y2BaaegydF4y2Ba或gydF4y2BaaagydF4y2Ba采用MCIM算法对效果进行了识别。共定位的qtl可以部分解释7个产量相关性状之间的相关关系。本研究获得的高密度遗传图谱和产量相关qtl,为芝麻重要农业性状研究、基于图谱的籽粒产量相关基因克隆和MAS遗传改良奠定了基础。gydF4y2Ba
方法gydF4y2Ba
植物材料和田间试验gydF4y2Ba
本研究使用的制图人群包括224个FgydF4y2Ba8:9gydF4y2Ba重组自交系由'妙千芝马'和'中植14 '杂交的单粒后代衍生而来,均为白色包衣种。父本“中植14号”是在中国广泛种植的商业品种,而母本“妙千芝马”是源于中国安徽省的地方增种。这两个品种在许多形态性状上有差异,包括株高、生长习性、蒴果形状、叶片形状和颜色以及对多种疾病的抵抗力。gydF4y2Ba
2012 - 2013年正常种植季(6 - 9月)在5个环境中进行了5次田间试验,其中武昌2次(2012WC、2013WC),阜阳2次(2012FY、2013FY),杨洛1次(2013YL)。武昌(30°52′n, 114°32′e)和杨洛(30°73′n, 114°62′e)相距约38.6 km,均位于长江中游夏芝麻区,阜阳(32°93′n, 115°81′e)位于黄淮盆地夏芝麻区。上述两个区占中国芝麻种植面积的50%以上。所有试验均采用随机完全区组设计,每个环境3个重复。每个地块有两个2.0米的行,间隔0.4米。在双幼叶期,植株被稀释,每行只保留13株均匀分布的植株进行进一步分析。gydF4y2Ba
特征评价gydF4y2Ba
在每个小区或基因型中,只使用6株均匀植株进行性状评价。为了避免边缘效应,不选择每行两端的植物。评价性状包括株高(PH, cm)、第一蒴果高(FCH, cm)、蒴果轴长(CAL, cm)、单株蒴果数(CN)、蒴果长(CL, mm)、每蒴果粒数(GN)和千粒重(TGW, g)。CAL为从最低的蒴果到最高的蒴果轴长。用6种植物的18个均匀胶囊的平均值测定CL和GN。TGW的一半被测量为三个500粒独立样品的平均重量。其他性状以6株植株的平均值进行测定。所有这些都是在收获阶段之前测量的。gydF4y2Ba
基因组DNA提取和PCRgydF4y2Ba
用DNA提取试剂盒(北京天根股份有限公司)从幼叶中提取基因组DNA。共使用1274个PCR标记(包括134个基因组- ssrs、1061个EST-SSRs和79个InDels)进行基因图谱构建(表2)gydF4y2Ba1gydF4y2Ba) [gydF4y2Ba31gydF4y2Ba].聚合酶链反应(PCR)对SSRs和InDels进行10 μl反应,包含10 ng DNA,每个引物2 pmol, 2 nmol dNTPs, 15 nmol MgClgydF4y2Ba2gydF4y2Ba0.2 U Taq DNA聚合酶(美国赛默飞世尔科学公司)和1 × PCR缓冲液随酶一起提供。PCR循环时间为94°C 3 min, 36个循环时间为94°C 20 s, 55°C ~ 60°C(根据引物不同)30 s, 72°C 40 s, 72°C 5 min。PCR产物用8%非变性聚丙烯酰胺凝胶(Acr:Bis =19:1或29:1)在180v恒定电压下分离2 ~ 3 h,银染色观察[gydF4y2Ba48gydF4y2Ba].gydF4y2Ba
RAD测序,InDel和SNP标记的开发gydF4y2Ba
采用限制性位点相关DNA (RAD)方法结合Illumina DNA测序,可快速有效地发现InDel和SNP标记。RAD库的构建,样本索引和池遵循Baird等。[gydF4y2Ba49gydF4y2Ba].的限制性内切酶gydF4y2Ba生态gydF4y2BaRI用于切割双亲和RIL群体的DNA [gydF4y2Ba50gydF4y2Ba].构建了22个多重测序文库,每个DNA样本分配一个唯一的核苷酸MID进行条形码编码。使用Illumina NGS平台HiSeq2000进行单端(101 bp)测序,总通量为22个通道。gydF4y2Ba
没有MID条形码的原始序列读取从3 '端开始裁剪到85个核苷酸,以确保超过90%的核苷酸的质量值高于Q30(等于0.1%的测序误差),超过99%的核苷酸的质量值高于Q20(等于1%的测序误差)。低质量的读取,包括修剪后<85 bp的读取或条形码不明确的读取,将被丢弃。对于InDels和snp调用,根据序列相似度将裁剪后的reads聚类到rad -tag中gydF4y2Ba栈gydF4y2Ba默认参数[gydF4y2Ba51gydF4y2Ba].排除了读取深度非常高(>500)的聚类rad标签[gydF4y2Ba51gydF4y2Ba].在两株亲本植株之间进行rad标记序列爆破。在比对结果中鉴定出InDels(≥2 bp)或snp,当每个等位基因至少观察3次时,视为真实多态性。InDel标记被开发用于PCR分析,因为与另一种协议的比对结果存在空白[gydF4y2Ba31gydF4y2Ba].在RIL植株中比较了含有snp的序列reads。只有在亲代和子代中始终被发现的snp才会被保留[gydF4y2Ba50gydF4y2Ba].将224个RILs的SNP或PCR标记的基因型用于遗传图谱的构建。gydF4y2Ba
键映射gydF4y2Ba
标记物分离比用卡方检验检验。表现不佳的标记在构建图谱前被剔除,在RIL人群中过度缺失,缺失数据超过40%,或过度扭曲,分离比超过次要等位基因频率小于0.29 [gydF4y2Ba13gydF4y2Ba].一个至少有三个相邻位点显示明显偏析畸变的区域(gydF4y2BaPgydF4y2Ba<0.05)定义为分离扭曲区(SDR) [gydF4y2Ba52gydF4y2Ba].使用JoinMap 4 (Kyazma, Wageningen, Netherlands)构建遗传连锁图谱。连锁组的确定采用最小LOD值为5.0,最大重组值为45%。在LOD阈值为3.0的情况下,采用回归映射算法确定各连锁组中标记的排序。含有少于20个标记的链接组被丢弃。每个位点相加后进行一个ripple,去除位点的拟合优度跳跃阈值为5.0,第三轮= Yes。利用Kosambi映射函数将重组频率转换为映射距离。每个连锁组的最终标记顺序通过软件程序RECORD [gydF4y2Ba53gydF4y2Ba].使用MapChart 2.2图形化可视化链接图[gydF4y2Ba54gydF4y2Ba].gydF4y2Ba
QTL分析gydF4y2Ba
利用SAS统计软件包对224个株系(基因型)在不同试验(环境)下3个重复(组)的平均表型数据进行频率分布、标准差、pearson相关系数和方差分析[gydF4y2Ba55gydF4y2Ba].广义遗传力(gydF4y2BaHgydF4y2Ba2gydF4y2Ba)用公式计算gydF4y2BaHgydF4y2Ba2gydF4y2Ba=gydF4y2BaσgydF4y2BaggydF4y2Ba2gydF4y2Ba/(gydF4y2BaσgydF4y2BaggydF4y2Ba2gydF4y2Ba+gydF4y2BaσgydF4y2BaegydF4y2Ba2gydF4y2Ba/gydF4y2BargydF4y2Ba),其中σgydF4y2BaggydF4y2Ba2gydF4y2Ba表示遗传方差σgydF4y2BaegydF4y2Ba2gydF4y2Ba残差方差,和gydF4y2BargydF4y2Ba是每个基因型的重复次数。gydF4y2Ba
利用Windows QTL Cartographer 2.5中实现的MIM方法检测7个性状的QTL [gydF4y2Ba56gydF4y2Ba]和QTLNetwork 2.0中的MCIM [gydF4y2Ba57gydF4y2Ba].在Windows QTL Cartographer 2.5中,首先对一个性状独立使用模型6进行复合区间映射(CIM)分析,在1 cM步长和10 cM窗口大小下进行正向和向后逐步回归。LOD显著性阈值(gydF4y2BaPgydF4y2Ba<0.05)通过进行1000次排列试验[gydF4y2Ba14gydF4y2Ba].随后使用MIM对qtl进行更精确的定位。将CIM中识别的QTL峰作为MIM的初始模型,并利用贝叶斯信息准则(bici - m0)逐步细化模型。QTL效应包括其表型变异的百分比(总gydF4y2BaRgydF4y2Ba2gydF4y2Ba)对MIM拟合的最终模型进行了估计,而gydF4y2BaRgydF4y2Ba2gydF4y2Ba使用CIM对单个QTL进行估计。以LOD值衰减值= 1的位置估计qtl的置信区间边界[gydF4y2Ba58gydF4y2Ba].gydF4y2Ba
采用10 cM检测窗口、1 cM行走速度和10 cM过滤窗口的基因组扫描参数,在3个重复的多个试验中,利用QTLNetwork 2.0识别一个性状的QTL互作和QTL-环境互作。采用二维(2D)基因组扫描来寻找多个相互作用的qtl。的全基因组阈值gydF4y2BaFgydF4y2Ba-统计量(α = 0.01)通过1000个随机排列估计QTL的存在。采用吉布斯样本量为20,000的蒙特卡洛马尔可夫链方法来估计QTL效应[gydF4y2Ba59gydF4y2Ba].每个QTL解释的个体表型方差之和作为每个性状的所有QTL解释的表型方差之和。gydF4y2Ba
支持数据的可用性gydF4y2Ba
RAD测序的原始序列数据已存入国家生物技术信息中心(NCBI)序列读取档案(SRA)数据库,登录号为SRA100255。gydF4y2Ba
附加文件gydF4y2Ba
缩写gydF4y2Ba
- 一个gydF4y2Ba:gydF4y2Ba
-
添加剂的影响gydF4y2Ba
- aagydF4y2Ba:gydF4y2Ba
-
Additive-additive上位性效应gydF4y2Ba
- aegydF4y2Ba:gydF4y2Ba
-
Additive-environmental交互作用gydF4y2Ba
- 方差分析:gydF4y2Ba
-
方差分析gydF4y2Ba
- 卡尔:gydF4y2Ba
-
囊轴长度gydF4y2Ba
- 肤色线:gydF4y2Ba
-
胶囊的长度gydF4y2Ba
- CN:gydF4y2Ba
-
每株蒴果数gydF4y2Ba
- 美国东部时间:gydF4y2Ba
-
表达序列标签gydF4y2Ba
- FCH:gydF4y2Ba
-
第一个胶囊的高度gydF4y2Ba
- GN:gydF4y2Ba
-
每粒粒数gydF4y2Ba
- InDel:gydF4y2Ba
-
Insertion-deletiongydF4y2Ba
- 格林:gydF4y2Ba
-
连杆组gydF4y2Ba
- MCIM:gydF4y2Ba
-
混合线性复合区间映射gydF4y2Ba
- MIM:gydF4y2Ba
-
多个区间映射gydF4y2Ba
- 门店:gydF4y2Ba
-
新一代测序gydF4y2Ba
- PH值:gydF4y2Ba
-
株高gydF4y2Ba
- QTL:gydF4y2Ba
-
数量性状位点gydF4y2Ba
- RAD-seq:gydF4y2Ba
-
限制性位点相关DNA测序gydF4y2Ba
- 瑞来斯:gydF4y2Ba
-
重组自交系gydF4y2Ba
- 特别提款权:gydF4y2Ba
-
隔离变形区域gydF4y2Ba
- SNP:gydF4y2Ba
-
单核苷酸多态性gydF4y2Ba
- 苏维埃社会主义共和国:gydF4y2Ba
-
简单序列重复gydF4y2Ba
- TGW:gydF4y2Ba
-
千粒重gydF4y2Ba
参考文献gydF4y2Ba
- 1.gydF4y2Ba
Bedigian D:芝麻的进化:驯化、多样性与展望。中国农业科学,2004,25(3):378 - 378。10.1023 /: 1025029903549。gydF4y2Ba
- 2.gydF4y2Ba
张辉,苗辉,王磊,曲磊,刘辉,王强,岳明:重要油料作物的基因组测序gydF4y2Ba胡麻属indicumgydF4y2BalgydF4y2Ba基因组医学杂志gydF4y2Ba2013年,14(1):401。gydF4y2Ba
- 3.gydF4y2Ba
Anilakumar KR, Pal A, Khanum F, Bawa AS:芝麻的营养、药用和工业用途(gydF4y2Ba胡麻属indicumgydF4y2Bal .) seeds-an概述。科学农业概论(ACS)。2010, 75(4): 159-168。gydF4y2Ba
- 4.gydF4y2Ba
芝麻的化学和生理功能。食品科学,1995,11(2):281-329。10.1080 / 87559129509541043。gydF4y2Ba
- 5.gydF4y2Ba
Moazzami AA, Kamal-Eldin A:芝麻是膳食木酚素的丰富来源。石油化工学报,2006,33(6):719-723。10.1007 / s11746 - 006 - 5029 - 7。gydF4y2Ba
- 6.gydF4y2Ba
Biabani AR, Pakniyat H:芝麻种子产量相关性状的评价(gydF4y2Ba胡麻属indicumgydF4y2BaL.)使用因子和通径分析。中国生物工程学报,2008,11(8):1157-1160。10.3923 / pjbs.2008.1157.1160。gydF4y2Ba
- 7.gydF4y2Ba
王晓燕,王晓燕,王晓燕。作物基因组学研究进展与应用。科学通报,2012,13(2):85-96。gydF4y2Ba
- 8.gydF4y2Ba
李勇,范超,邢勇,蒋勇,罗亮,孙亮,邵东,徐超,李旭,肖军,何勇,张强:GS5基因的自然变异在水稻粒级和产量调控中起着重要作用。植物学报,2011,43(12):1266-1269。10.1038 / ng.977。gydF4y2Ba
- 9.gydF4y2Ba
于宏,谢伟,王娟,邢勇,徐超,李旭,肖军,张强:基于群体测序的超高密度SNP图谱在QTL检测中的应用。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba2011年,6 (3):e17595。gydF4y2Ba
- 10.gydF4y2Ba
马拉蒂B、古莱里亚S、莫哈帕特拉T、帕sad R、Mariappan N、库伦加拉VK、Atwal SS、Prabhu KV、Singh NK、Singh AK:重组水稻新株型自交系产量及产量相关性状新基因组区域QTL分析(gydF4y2Ba栽培稻gydF4y2Bal .)。gydF4y2BaBMC植物杂志gydF4y2Ba2012年,12:137。gydF4y2Ba
- 11.gydF4y2Ba
中国生物医学工程学报,2013,29(5):369 - 369。10.1073 / pnas.1306579110。gydF4y2Ba
- 12.gydF4y2Ba
gydF4y2BaBMC麝猫gydF4y2Ba2013年,十四4。gydF4y2Ba
- 13.gydF4y2Ba
Pfender WF, Saha MC, Johnson EA, Slabaugh MB:利用限制性位点相关DNA标记快速定位黑麦草茎锈病抗性QTL。应用科学学报,2011,32(6):669 - 671。10.1007 / s00122 - 011 - 1546 - 3。gydF4y2Ba
- 14.gydF4y2Ba
Chutimanitsakun Y, Nipper RW, Cuesta-Marcos A, Cistue L, Corey A, Filichkina T, Johnson EA, Hayes PM:大麦限制性内切位点相关DNA (RAD)连锁图谱的构建及其QTL分析应用。gydF4y2BaBMC基因组学gydF4y2Ba2011年,12:4。gydF4y2Ba
- 15.gydF4y2Ba
魏伯林,张海燕,郑永忠,苗敏敏,张志忠,郭文忠:芝麻遗传连锁图谱的构建(gydF4y2Ba胡麻属indicumgydF4y2Bal .)。基因学报,2009,31(2):199-208。10.1007 / BF03191152。gydF4y2Ba
- 16.gydF4y2Ba
张海燕,魏伯伦,苗红梅,张道堂,王春春:利用RNA-seq建立芝麻遗传ssr标记及验证。gydF4y2BaBMC基因组学gydF4y2Ba2012年,13:316。gydF4y2Ba
- 17.gydF4y2Ba
张红,苗红,魏磊,李超,赵锐,王超:芝麻种皮颜色的遗传分析及QTL定位(gydF4y2Ba胡麻属indicumgydF4y2Bal .)。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba2013、8 (5):e63898。gydF4y2Ba
- 18.gydF4y2Ba
Davey JW, Hohenlohe PA, Etter PD, Boone JQ, Catchen JM, Blaxter ML:使用下一代测序发现全基因组遗传标记和基因分型。科学通报,2011,12(7):499-510。10.1038 / nrg3012。gydF4y2Ba
- 19.gydF4y2Ba
Hyten DL, Cannon SB, Song Q, Weeks N, Fickus EW, Shoemaker RC, Specht JE, Farmer AD, May GD, Cregan PB:通过对大豆全基因组序列中简化表达库的深度重测序来锚定和定位支架,发现高通量SNP。gydF4y2BaBMC基因组学gydF4y2Ba2010年,三十八分。gydF4y2Ba
- 20.gydF4y2Ba
陈松,黄铮,戴勇,秦松,高原,张磊,陈娟:7E染色体特异性分子标记的研究进展gydF4y2BaThinopyrum elongatumgydF4y2Ba基于SLAF-seq技术。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba2013年,8 (6):e65122。gydF4y2Ba
- 21.gydF4y2Ba
孙鑫,刘东,张鑫,李伟,刘宏,洪伟,蒋超,关宁,马超,曾浩,徐超,宋俊,黄磊,王超,石俊,王锐,郑鑫,陆超,王鑫,郑辉:SLAF-seq:一种利用高通量测序技术进行大规模SNP新发现和基因分型的有效方法。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba2013年,8 (3):e58700。gydF4y2Ba
- 22.gydF4y2Ba
王晓强,赵玲,Eaton DA,李德志,郭志华:基于RAD测序的温带竹科植物系统发育推断的SNP标记。动物生态学报,2013,13(5):938-945。10.1111 / 1755 - 0998.12136。gydF4y2Ba
- 23.gydF4y2Ba
Barchi L、Lanteri S、Portis E、Acquadro A、Vale G、Toppino L、Rotino GL:茄子SNP和SSR标记的RAD标记测序gydF4y2BaBMC基因组学gydF4y2Ba2011年,12:304。gydF4y2Ba
- 24.gydF4y2Ba
Miller MR, Dunham JP, Amores A, Cresko WA, Johnson EA:使用限制性内切位点相关DNA (RAD)标记快速且经济的多态性鉴定和基因分型。基因组学报,2007,17(2):240-248。10.1101 / gr.5681207。gydF4y2Ba
- 25.gydF4y2Ba
Hegarty M, Yadav R, Lee M, Armstead I, Sanderson R, Scollan N, Powell W, Skot L:利用RAD测序进行基因分型,可以绘制多年生黑麦草脂肪酸组成性状的图谱(gydF4y2Ba多年生黑麦草gydF4y2Bal .)。植物生态学报,2013,11(5):572-581。10.1111 / pbi.12045。gydF4y2Ba
- 26.gydF4y2Ba
Pegadaraju V, Nipper R, Hulke B, Qi L, Schultz Q:利用限制性内切位点相关DNA (RAD)方法对向日葵基因组进行从头测序以发现SNP。gydF4y2BaBMC基因组学gydF4y2Ba2013年,14(1):556。gydF4y2Ba
- 27.gydF4y2Ba
王宁,方丽,辛红,王磊,李松:利用下一代限制性位点相关DNA测序构建葡萄高密度遗传图谱。gydF4y2BaBMC植物杂志gydF4y2Ba2012年,12:148。gydF4y2Ba
- 28.gydF4y2Ba
Barchi L、Lanteri S、Portis E、Vale G、Volante A、Pulcini L、Ciriaci T、Acciarri N、Barbierato V、Toppino L、Rotino GL:基于RAD标记的茄子连锁图谱和决定花青素色素的qtl定位。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba2012年,7 (8):e43740。gydF4y2Ba
- 29.gydF4y2Ba
张颖,王磊,辛红,李东,马晨,丁旭,洪伟,张鑫:基于SLAF标记的芝麻高密度遗传图谱构建。gydF4y2BaBMC植物杂志gydF4y2Ba2013,13(1): 141。gydF4y2Ba
- 30.gydF4y2Ba
华建平,邢永忠,徐贵刚,孙晓玲,余顺生,张秋峰:对一株水稻杂种的遗传分析表明,杂合子并不总是有利于水稻的生产性能。中国生物医学工程学报,2017,38(4):359 - 359。gydF4y2Ba
- 31.gydF4y2Ba
杨吴K、M,刘H,道Y,梅J,赵Y:遗传分析和分子特征的中国芝麻(gydF4y2Ba胡麻属indicumgydF4y2Ba利用插入-删除(InDel)和简单序列重复(SSR)标记对L.)栽培品种进行筛选。gydF4y2BaBMC麝猫gydF4y2Ba2014年,15(1):35。gydF4y2Ba
- 32.gydF4y2Ba
郑俊伟,郑俊伟,郑洪红,郑俊伟,郑俊伟,马海红,朴永杰,赵EG:芝麻多态微卫星标记的研究进展(gydF4y2Ba胡麻属indicumgydF4y2Bal .)。中国生物医学工程学报,2005,5(4):736-738。10.1111 / j.1471-8286.2005.01048.x。gydF4y2Ba
- 33.gydF4y2Ba
Cho YI, Park JH, Lee CW, Ra WH, Chung JW, Lee JR, Ma KH, Lee SY, Lee KS, Lee MC, Park YJ:芝麻遗传多样性与群体结构评价(gydF4y2Ba胡麻属indicumgydF4y2BaL.)使用微卫星标记。基因学报,2011,33(2):187-195。10.1007 / s13258 - 010 - 0130 - 6。gydF4y2Ba
- 34.gydF4y2Ba
Spandana B, Reddy VP, Prasanna GJ, Anuradha G, Sivaramakrishnan S:芝麻植物微卫星标记(SSR)的建立与鉴定(英文)gydF4y2Ba胡麻属indicumgydF4y2Bal .)物种。中国生物医学工程学报,2012,38(6):369 - 369。10.1007 / s12010 - 012 - 9881 - 7。gydF4y2Ba
- 35.gydF4y2Ba
岳卫东,魏伯林,张道东,李超,苗敏敏,张海燕:芝麻种质资源遗传多样性及群体结构分析(gydF4y2Ba胡麻属indicumgydF4y2BaL.)的SSR标记。作物学报(中文)。2012, 38(12): 2286-2296。10.3724 / SP.J.1006.2012.02286。gydF4y2Ba
- 36.gydF4y2Ba
魏伟,齐鑫,王磊,张颖,华伟,李东,吕红,张鑫:利用Illumina配对端测序和EST-SSR标记分析芝麻(Sesamum indicum L.)全局转录组。gydF4y2BaBMC基因组学gydF4y2Ba2011年,12:451。gydF4y2Ba
- 37.gydF4y2Ba
王亮,张勇,齐鑫,高原,张鑫:59个食用油作物多态cDNA-SSR标记的开发与鉴定gydF4y2Ba胡麻属indicumgydF4y2Ba(胡麻科)。中国生物医学工程学报,2012,29(5):394- 398。10.3732 / ajb.1200081。gydF4y2Ba
- 38.gydF4y2Ba
Yepuri V、Surapaneni M、Kola V、Vemireddy LR、Jyothi B、Dineshkumar V、Anuradha G、Siddiq EA:芝麻遗传多样性评价(gydF4y2Ba胡麻属indicumgydF4y2BaL.)基因型,使用est衍生的SSR标记。作物科学与生物技术学报,2013,16(2):93-103。10.1007 / s12892 - 012 - 0116 - 9。gydF4y2Ba
- 39.gydF4y2Ba
薛松,张铮,林芳,孔智,曹勇,李超,易华,梅梅,朱辉,吴娟,徐辉,赵东,田东,张春,马铮:表达序列标记富集的小麦基因组高密度品种间图谱。应用科学学报,2008,(2):181-189。10.1007 / s00122 - 008 - 0764 - 9。gydF4y2Ba
- 40.gydF4y2Ba
gydF4y2Ba豇豆属。unguiculatagydF4y2Bassp。gydF4y2BasesquipedialisgydF4y2Ba)并与更广泛的物种进行比较。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba2011年,6 (1):e15952。gydF4y2Ba
- 41.gydF4y2Ba
Brookes AJ: snp的本质。中国生物医学工程学报,1999,34(2):376 - 376。10.1016 / s0378 - 1119 (99) 00219 - x。gydF4y2Ba
- 42.gydF4y2Ba
吕洪华,李文华,李文华,等。玉米分离畸变的相关染色体区域。应用理论与实践,2002,10(4):622-628。10.1007 / s00122 - 002 - 0970 - 9。gydF4y2Ba
- 43.gydF4y2Ba
法瑞斯JD, Laddomada B, Gill BS: tauschii植物分离畸变位点的分子定位。中国生物医学工程学报,1998,32(1):319-327。gydF4y2Ba
- 44.gydF4y2Ba
贾辉,万辉,杨松,张铮,孔铮,薛松,张磊,马铮:中国小麦育种关键亲本重组自交系群体产量相关性状的遗传解剖。中国生物医学工程学报,2013,29(4):369 - 369。10.1007 / s00122 - 013 - 2123 - 8。gydF4y2Ba
- 45.gydF4y2Ba
Palomeque L, Li- jun L, Li W, Hedges B, Cober ER, Rajcan I:超级环境下的QTL: I.在一个高产适应大豆与高产外来大豆杂交群体中检测到的普遍和特异性种子产量QTL。应用科学学报,2009,29(3):417-427。10.1007 / s00122 - 009 - 1049 - 7。gydF4y2Ba
- 46.gydF4y2Ba
巴苏南达·P, Radoev M, Ecke W, Friedt W, Becker HC, Snowdon RJ:油菜苗期和产量性状杂种优势数量性状位点的比较定位(gydF4y2Ba芸苔属植物显著gydF4y2Bal .)。中国生物医学工程学报,2010,29(2):381 - 381。10.1007 / s00122 - 009 - 1133 - z。gydF4y2Ba
- 47.gydF4y2Ba
杨军,朱军,Williams RW:实验群体复杂性状的遗传结构图谱。生物信息学,2007,23(12):1527-1536。10.1093 /生物信息学/ btm143。gydF4y2Ba
- 48.gydF4y2Ba
梁红伟,王长忠,李铮,罗晓珍,邹格光:聚丙烯酰胺凝胶电泳银染技术的改进。易川。2008,30(10):1379-1382。10.3724 / SP.J.1005.2008.01379。gydF4y2Ba
- 49.gydF4y2Ba
Baird NA, Etter PD, Atwood TS, Currey MC, Shiver AL, Lewis ZA, Selker EU, Cresko WA, Johnson EA:使用测序RAD标记快速发现SNP和基因制图。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba2008年,3 (10):e3376。gydF4y2Ba
- 50.gydF4y2Ba
道徐许P, S,吴X, Y, B王,王,秦D,陆Z,李G:人口基因组分析信号低RAD-Seq数据:一个案例研究在non-model葫芦葫芦。植物生态学报,2014,32(3):441 - 441。10.1111 / tpj.12370。gydF4y2Ba
- 51.gydF4y2Ba
Catchen JM, Amores A, Hohenlohe P, Cresko W, Postlethwait JH:栈:短读序列中新位点的构建和基因分型。G3(贝塞斯达)。2011, 1(3): 171-182。10.1534 / g3.111.000240。gydF4y2Ba
- 52.gydF4y2Ba
Paillard S, Schnurbusch T, Winzeler M, Messmer M, Sourdille P, Abderhalden O, Keller B, Schachermayr G:冬小麦综合遗传连锁图谱(gydF4y2Ba小麦gydF4y2Bal .)。应用理论与实践,2003,27(6):669 - 672。10.1007 / s00122 - 003 - 1361 - 6。gydF4y2Ba
- 53.gydF4y2Ba
Van Os H, Stam P, Visser RG, Van Eck HJ: RECORD:一种新的遗传连锁图谱上的位点排序方法。中国科学(d辑),2005,30(1):359 - 361。10.1007 / s00122 - 005 - 0097 - x。gydF4y2Ba
- 54.gydF4y2Ba
Voorrips RE: MapChart:用于链接图和qtl的图形表示软件。中华医学杂志,2002,93(1):77-78。10.1093 / jhered / 93.1.77。gydF4y2Ba
- 55.gydF4y2Ba
schlotzhaer SD, Littell RC: SAS基本统计分析系统。SAS研究所,卡里,北卡罗来纳州;1997.gydF4y2Ba
- 56.gydF4y2Ba
王s, Basten J,曾铮:Windows QTL制图师,北卡罗莱纳州立大学统计系,罗利。数控;2010:2.5。gydF4y2Bahttp://statgen.ncsu.edu/qtlcart/WQTLCart.htmgydF4y2Ba
- 57.gydF4y2Ba
杨娟,朱娟:基于QTL效应的多环境优势基因型预测方法。中国科学(d辑),2005,38(6):669 - 669。10.1007 / s00122 - 005 - 1963 - 2。gydF4y2Ba
- 58.gydF4y2Ba
Lander ES, Botstein D:利用RFLP连锁图谱绘制数量性状背后的孟德尔因子。中国生物医学工程学报,2004,25(1):381 - 381。gydF4y2Ba
- 59.gydF4y2Ba
蒋超,曾志斌:两个自交系的优势标记和缺失标记的数量性状位点的定位。中国生物医学工程学报,2017,38(1):449 - 452。10.1023 /: 1018394410659。gydF4y2Ba
致谢gydF4y2Ba
本研究得到国家自然科学基金项目(No. 31201243)、国家重点基础研究计划项目(2011CB109304)、农业部油料作物生物学与遗传改良重点实验室开放项目(201210)、中国农业科学研究体系项目(car -15)和中国农业科学院油料作物研究所所长基金项目(1610172011007)资助。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
相应的作者gydF4y2Ba
额外的信息gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明他们没有竞争利益。gydF4y2Ba
作者的贡献gydF4y2Ba
KW和YZZ设计了本研究;HYL和YZZ构建了重组自交系群体;MMY进行PCR标记分析;YT行RAD测序;HHM、WXW和YZ对产量相关性状进行评价;KW进行了连锁映射、数据分析和qtl检测,并撰写了稿件。所有作者阅读并批准了最终稿件。gydF4y2Ba
作者提交的图片原始文件gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。gydF4y2Ba
本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。gydF4y2Ba
欲查看此许可证的副本,请访问gydF4y2Bahttps://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
创作共用公共领域奉献放弃书(gydF4y2Bahttps://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文提供的数据,除非在数据的信用额度中另有说明。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
吴凯,刘海华,杨明。gydF4y2Baet al。gydF4y2Ba芝麻籽粒产量相关性状高密度遗传图谱构建及qtl分析gydF4y2Ba胡麻属indicumgydF4y2BaL.)基于RAD-Seq技术。gydF4y2BaBMC植物杂志gydF4y2Ba14日,gydF4y2Ba274(2014)。https://doi.org/10.1186/s12870-014-0274-7gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-014-0274-7gydF4y2Ba
关键字gydF4y2Ba
- 遗传图谱gydF4y2Ba
- 法gydF4y2Ba
- RAD-seqgydF4y2Ba
- 瑞来斯gydF4y2Ba
- 芝麻gydF4y2Ba
- 粮食yield-related特征gydF4y2Ba