摘要
背景
在植物中,非特异性脂质转移蛋白(non-specific脂质转移蛋白,nsLTPs)是一种小而基本的蛋白质,已被报道参与许多生物过程,如磷脂转移、生殖发育、病原体防御和非生物应激反应。迄今为止,只有一小部分植物nsLTPs被功能鉴定出来,在玉米中鉴定出来的就更少了[玉米Zm评选)。
结果
在本研究中,我们对玉米nsLTP基因家族进行了全基因组分析,共鉴定出63个nsLTP基因,可分为1、2、C、D和G 5种类型,在同一类型中观察到相似的内含子/外显子结构模式,有力地支持了它们之间密切的进化关系。基因复制分析表明,串联复制和片段复制都有助于该基因家族的多样化。此外,通过同源建模研究了具有代表性的nsLTPs的三维结构,以了解其分子功能。通过对玉米nsLTPs (ZmLTPs)进行基因本体分析,获得玉米nsLTPs生物学功能的线索。上游调控元件的分析显示了转录调控基序的共享性和独特性ZmLTPs,进一步表明ZmLTPs可能在多种生物过程中发挥作用。的动态表达模式ZmLTPs不同发育阶段的家系研究表明,其中几个基因表现出组织特异性表达,表明它们在玉米生命周期中发挥着重要作用。此外,我们还研究了玉米nsLTPs在生物和非生物胁迫响应中的作用。我们的分析证明了一些ZmLTPs感染后表现为延迟表达模式黑粉菌属maydis在干旱、盐胁迫和冷胁迫下均有差异表达,这对进一步提高玉米抗逆性和耐受性的研究有很大的帮助。
结论
我们的研究结果为玉米nsLTPs的系统发育关系和特征功能提供了新的见解,并将有助于揭示玉米发育和胁迫响应的全球调控网络,从而有助于提高玉米品质性状的分子育种。
背景
顾名思义,植物脂质转移蛋白(LTPs)因其在膜间转移磷脂和脂肪酸的功能而得名在体外[1].由于它们与不同脂类的非特异性结合,也被称为非特异性LTPs (nsLTPs)。植物nsLTPs是一种小而基本的蛋白质,大小通常在6.5 ~ 10.5 kDa左右,具有8个半胱氨酸基序(8CM)的主链,一般形式为C-Xn-C-Xn-CC-CXC-Xn-C-Xn-C - xn - c - xn - c [2].几乎所有的nsLTPs在其新生多肽中都带有一个n端信号肽。因此,这些蛋白质可能被分泌到细胞外部以发挥作用。许多nsLTPs还具有翻译后添加糖基磷脂酰肌醇(GPI)锚的序列,该锚将蛋白质连接到质膜的外侧[3.].植物nsLTPs的保守特征包括由8个Cys残基形成的4个二硫键。此外,植物nLTPs的晶体结构由4或5个α螺旋(α-螺旋)组成,中心有一个疏水腔,脂质结合在此发生[4].
根据成熟蛋白的分子量(Mw),植物nsLTPs可分为两种主要类型,nsLTP1 (9 kDa)和nsLTP2 (7 kDa) [5].根据序列相似性,Boutrot等从52种水稻中筛选出49种nsLTPs49株拟南芥中的45株nsLTPs分为九类(I、II、III、IV、V、VI、VII、VIII及Y) [2].最近,nsLTPs根据序列相似性、内含子位置和半胱氨酸残基间距,以及编码蛋白中潜在的糖磷脂酰肌醇修饰位点,将其分为四种主要类型和几种次要类型[6].在体外研究表明,植物nsLTPs能够促进磷脂、糖脂、类固醇、酰基辅酶as和脂肪酸等多种两亲性分子的膜间交换和转移。植物nsLTPs的几种结构已经通过x射线和核磁共振光谱技术得到了解析[4]、[7]、[8].nsLTP1的典型折叠特征是由4 - 5个α-螺旋组成,由4个二硫键桥连接,部分被长c端段包裹。整体结构划定了一个大的中央疏水腔,其中烷基部分的脂类被插入。nsLTP1和nsLTP2的主干褶皱结构相似,但其中心疏水腔发生了剧烈变化。与这两种最早的nsLTP类型相比,拟南芥DIR1的三维模型(AtDIR1)遵循了一般的nsLTP折叠,由四个二硫键围绕中心隧道状腔稳定的五个α-螺旋。据报道,nsLTPs的大多数假定功能与它们在疏水腔中结合脂质的能力有关[9] - [11].
在随后的几年里,一些nsLTPs成员在植物物种中被功能鉴定,包括角质层蜡和角质层合成的参与[12].最近的一项研究表明,LTPg1,一个来自拟南芥的G型nsLTP,直接或间接地促进角质层脂质积累[13].此外,nsLTPs在不同的器官和细胞中表达,包括愈伤组织、发芽和成熟的种子、叶、根、茎、子房、花药和花粉[6]、[14].在拟南芥和水稻中,nsLTP转录本在花药中的定位已经得到了很好的报道,大量的III型nsLTPs(也称为C型nsLTPs)在花药绒毡层中特异性表达,在花粉壁外壁最大合成的发育阶段达到峰值[15].脂质转移蛋白OsC6广泛分布在绒毡层等花药组织中,是水稻减数分裂后花药发育所必需的[16].在植物防御方面,nsLTPs也被认为是与致病相关的蛋白质,并构成PR-14家族[17].其中一些被证明与卵菌诱导素具有结构相似性,并作为膜上特定受体的诱导素的竞争对手[18]、[19].此外,nsLTPs在病原体防御过程中参与长距离信号传递[9].纯化的nsLTPs的研究证实了它们在非生物胁迫耐受中的作用[20.].转录水平nsLTPs在许多情况下,对干旱、盐和冷胁迫的反应增强[21].膜的稳定、角质层沉积和/或细胞壁组织的变化被认为是它们对这些应激因素的反应的假定角色。干旱诱导烟草表皮蜡沉积过程中nsLTP基因上调[22].辣椒CALTP1基因在拟南芥各营养生长阶段均能提高其对NaCl和干旱胁迫的耐受性[23].拟南芥LTP3作为MYB96的靶标参与植物对冰冻和干旱胁迫的耐受性[24].
在之前的研究中,只有一小部分nsLTPs已经从玉米中得到了很好的特征。这些包括,MZm3-3,在雄性配子体发生过程中特异性表达于绒毡层[25];Zm评选ltp (ZmLTP1.2;GRMZM2G010868)与钙调素(CaM)结合2 +-独立方式[26];ZmLTP3(ZmLTP1.1;GRMZM2G126397),由甘露醇、盐和SA处理诱导[27];BETL-9(ZmLTPd6;GRMZM2G087413),在发育中的胚乳外表面转录[28].随着自交系B73基因组测序的完成,是时候对玉米nsLTP基因家族进行更全面的功能注释和分析了。迄今为止,玉米nsLTP基因家族的全基因组综述尚未报道。因此,作为阐明ZmLTPs功能的第一步,对该基因家族进行全基因组研究是必要的。在这项研究中,我们在玉米基因组中鉴定了63个基因,编码77个假定的nsLTPs,可分为5种类型。详细分析包括主序列、系统发育关系、基因结构、染色体定位、基因重复和分化、三维结构、基因本体、启动子和表达谱。基于基因芯片、rna测序和实时定量PCR (qRT-PCR)技术,我们分析了ZmLTP基因在不同组织和发育阶段的表达模式,进一步了解了植物对生物和非生物胁迫的响应。我们的分析提供了对ZmLTP基因家族的新见解,为进一步研究植物中nsLTP基因家族的功能提供了支持,特别是在响应生物和非生物胁迫中可能具有重要功能的蛋白质。
结果与讨论
玉米和高粱中nsLTP成员的鉴定
为了鉴定玉米中完整和非冗余的nsLTPs,对玉米蛋白质组进行了精确的扫描。最初,除去8CM结构域不完整的序列后,在玉米基因组中鉴定出130个潜在的ZmLTPs。然后,通过分析半胱氨酸残基模式手动评估每个推导出的蛋白质序列。随后,去除40个富脯氨酸或杂交富脯氨酸蛋白,这些蛋白在信号肽和8CM之间的序列中具有高比例的脯氨酸、组氨酸和甘氨酸残基(附加文件)1:表S1)。然后,2个α-淀粉酶/胰蛋白酶抑制剂,3个脯氨酸存储蛋白和3个2S白蛋白存储蛋白也被丢弃。另外,缺失GPI-anchor信号肽的5种转录本形式(GRMZM2G174680_P02、GRMZM2G083725_P02、GRMZM2G006047_P01、GRMZM2G005991_P02、GRMZM2G116167_P02)未被考虑。结果,77个蛋白被确认为玉米nsLTP,它们由63个nsLTP基因编码(附加文件)2:表S2)。此外,同样的方法被用于鉴定高粱中的nsLTP基因,结果鉴定出58个nsLTP基因(附加文件3.:表S3和附加文件4:表S4)。
玉米nsLTPs的序列分析与分类
此前,Edstam等人提出植物nsLTPs根据序列相似性、内含子位置和半胱氨酸残基间距可分为四种主要类型和几种次要类型[6].在主要类型之一的G型中,转录本除了编码n端信号序列外还编码c端信号序列,导致翻译后修饰,其中gpi -锚被添加到蛋白质上。所识别的G型ZmLTPs的分类也是基于GPI修饰位点的存在。在第二轮分类中,根据多个序列比对计算出的单位矩阵对剩余序列进行分组。与Edstam等人提出的分类方法进行比较,我们发现77个ZmLTPs中有73个可以分为5种类型(1、2、C、D和G)。在四种开花植物(玉米、高粱、水稻和拟南芥)中,检测到nsLTPs在不同类型和物种之间的数量差异(附加文件)5:表S5)。在G型中,玉米、高粱、水稻和拟南芥中分别有26、24、27和29个nsLTPs。而拟南芥中只有2个nsLTP基因分布在E型。值得注意的是,在1型和2型中发现了相似数量的nsLTP基因。因此,可以想象,这种差异可能是由于基因的复制和丢失。植物nsLTPs的主要特征是在高度保守的区域存在8个半胱氨酸残基(C-Xn-C-Xn-CC-Xn-CXC-Xn-C-Xn-C - xn - c)。为了为每个获得的nsLTP类型建立一个特定的8CM共识,我们使用来自77个zmltp(附加文件)的8CM进行了多个序列比对6:图S1)。ZmLTPs的8cm的氨基酸序列比对揭示了半胱氨酸间氨基酸残基的可变数量(表1).
在本研究中,77个zmltp的所有特征都总结在附加文件中2:表S2。96%的nsLTP前体最初是由16-46个氨基酸的信号肽合成的。分析了77个ZmLTP前蛋白序列的亚细胞靶向性。正如预期的那样,除了ZmLTPg21被预测为细胞质蛋白外,大多数蛋白被预测为分泌蛋白(附加文件)2:表S2)。在蛋白前水平,ZmLTPd1和ZmLTPd4推导出的蛋白是相同的,ZmLTPd2和ZmLTPd10推导出的蛋白也是相同的。信号肽裂解后,ZmLTPg20.1和ZmLTPg20.2成熟蛋白完全相同。因此,在潜在的翻译后修饰之前,63个ZmLTP基因编码了74种不同的成熟蛋白。29个zmltp被发现有一个或多个磷酸化位点,大多数磷酸化位点位于c端丝氨酸残基上(补充文件)7:表S6)。为了清楚地了解ZmLTPs的序列特征,我们进一步分析了所有可用的ZmLTPs的pI(等电点)值、Mw值和CXC基序。如附加文件所示2:表S2中,玉米nsLTPs分子量较小,除g型外,其分子量一般在6854 Da至11107 Da之间。1型和D型nsLTPs多为9 kDa蛋白,2型和C型nsLTPs多为7 kDa蛋白。G型nsLTPs的Mw值远高于其他类型nsLTPs,这是由于在推导的成熟蛋白的c端存在多余的氨基酸残基。从pI值判断,1、2、C、D型nsLTPs多为碱性蛋白。大多数G型nsltp呈弱碱性或弱酸性(附加文件)2:表S2)。平均分子质量为8963 Da,理论pI为9.27 pI。对于CXC基序,1型nsLTPs的X位残基大多为亲水性残基,而2型、C型、D型和G型nsLTPs的X位残基通常为疏水性残基(附加文件)6:图S1)。这些保守的疏水或亲水残基可能在ZmLTPs的生物学功能中发挥重要作用[29].
玉米、高粱、水稻和拟南芥nsLTPs的系统发育分析
为了分析玉米、高粱、水稻和拟南芥中nsLTPs的系统发育关系,对这4个物种的274个nsLTPs进行了分析8:图S2)。我们对玉米、高粱、水稻和拟南芥的8CM结构域序列进行了多序列比对,并采用邻居连接法生成了系统发育树。在此之前,Edstam等人将植物nsLTPs分为十种类型[6].在前期数据集与我们数据集比较的基础上,本文划分的6个类群与nsltp的1、2、C、D、E、G类型一致。如附加文件所示8:图S2中,Type 1和Type 2的成员形成了特定的演化支,这表明这些主要nsLTP类型的基因具有共同的祖先。小C型和E型的序列也形成了独立的演化支。由于在1型和C型的内部分支中观察到相对较高的自举值,这清楚地表明从一个共同祖先衍生出统计上可靠的可能同源蛋白对,它们具有相似的起源。值得注意的是,E型nsLTPs似乎是单子叶植物,这可能意味着单子叶植物在单子叶植物和双子叶植物进化分化过程中丢弃了这些基因。
玉米nsLTP基因家族的内含子-外显子结构
内含子-外显子排列作为一种进化遗迹,携带着基因家族进化的印记。对ZmLTP基因结构的研究显示,在外显子序列中内含子区域的分布多样性较低,预计31个ZmLTP基因(36种可选剪接形式)被位于8CM中编码第8个半胱氨酸的密码子下游4至67 bp的1-2个内含子所中断9:图S3)。同一基因的不同剪接形式通常具有相似的内含子-外显子结构,这表明来自单个转录本的不同蛋白质可能具有相似的功能。此外,有趣的是在每组中都发现了相似的外显子/内含子模式。例如,Type 1,2和C中的nsLTP基因缺少一个内含子,而Type G中的所有成员都含有1-3个内含子。除了ZmLTPd6,ZmLTPd13,ZmLTPd14和ZmLTPd15, D型基因编码区未检测到内含子。我们与基因结构的比较分析AtLTPs而且OsLTPs表明该基因的外显子/内含子结构ZmLTPs与拟南芥和水稻的基因相似(附加文件10:图S4)。
ZmLTP基因的染色体定位和基因复制
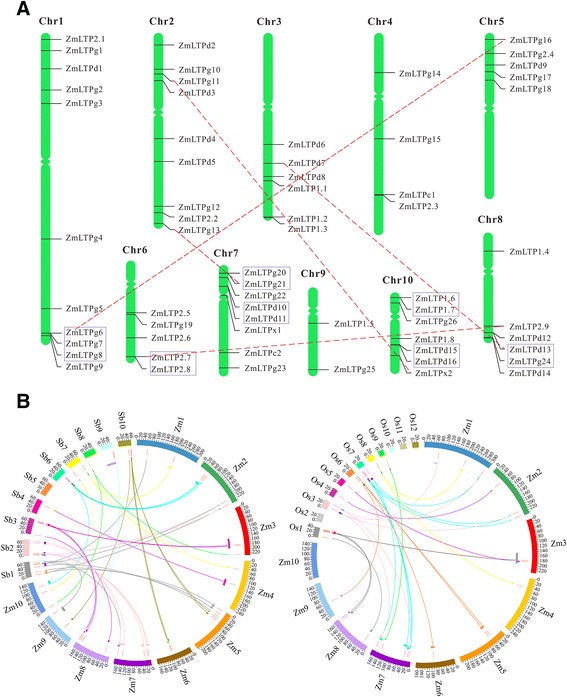
在网上基因位点图谱显示,63ZmLTPs不均匀地分配到玉米的10条染色体上(图1A). 1号染色体含有最大数目的ZmLTPs(11),最小数目(2)出现在9号染色体上。然而,有几条染色体缺乏ZmLTPs在某些区域,如5号染色体的长臂和3号、6号和9号染色体的短臂。每一个的确切位置(单位:bp)nsLTP玉米染色体上的染色体数目见附档2:表S2。
基因复制通常被认为是进化创新的主要驱动力,导致基因组的复杂性。玉米起源于一种古老的异体四倍体,在基因进化过程中经历了多轮全基因组复制事件[30.]、[31].节段复制和串联复制是导致基因家族扩张的主要原因。在本研究中,共有7个串联重复序列(ZmLTPg6 / ZmLTPg7 / ZmLTPg8;ZmLTP2.7 / ZmLTP2.8;ZmLTPg20 / ZmLTPg21;ZmLTPd10 / ZmLTPd11;ZmLTPd12 / ZmLTPd13;ZmLTP1.6 / ZmLTP1.7而且ZmLTPd15 / ZmLTPd16)在玉米基因组中被发现。在1号染色体上发现了一个由3个G型基因组成的重要集群。此外,在第6、7、8和10号染色体上发现了6个直接重复串联。同一簇中的基因彼此密切相关。例如,ZmLTPd15和ZmLTPd16蛋白质序列有95%的相似性,这两个基因在物理上位于同一染色体区域中彼此相邻。此外,五对姐妹(ZmLTPg6/ZmLTPg16,ZmLTPg11/ZmLTPx2,ZmLTPg13/ZmLTPg21,ZmLTPd7/ZmLTPd13而且ZmLTP2.7/ZmLTP2.9)似乎是由片段复制事件产生的,因为它们位于相同的复制基因块上(图1A和附加文件11:表S7)。此外,我们还分析了nsLTP基因在玉米、高粱和水稻中的进化(图1B和附加文件11:表S7)。63个玉米nsLTP基因中22个在水稻中有共线基因,35个在高粱中有共线基因。根据分析,两个成对的nsLTP基因(ZmLTPg21/OsLTPg17和ZmLTPg13/OsLTPg17)均位于玉米和水稻基因组的同位区。七对nsLTP基因(ZmLTPg16/SbLTPx1,ZmLTPg11/SbLTPg18,ZmLTPx2/SbLTPg18,ZmLTP2.9/SbLTP2.5,ZmLTPg21/SbLTPg10,ZmLTPg13/SbLTPg10而且ZmLTPg13/SbLTPg4)位于玉米和高粱基因组的同位位点,表明这些基因可能来自共同的祖先。有趣的是,其中一对姐妹(ZmLTPg6/ZmLTPg16)有一个独特的,共音的nsLTPs在高粱中,这表明它们可能是在玉米和高粱分化后通过节段复制产生的。
玉米nsLTP基因的分化率
非同义替代率(Ka)与同义替代率(Ks)的比值(Ka/Ks)被广泛用作选择压力的指标[32].如表所示2, 5对重复基因的Ka/Ks比值均小于1,表明它们似乎处于纯化选择状态;但9对重复基因的Ka/Ks比值均大于1,说明它们经历了正向选择。正向选择被认为是基因复制后蛋白质中出现新基序/功能的主要动力之一,而重复基因的分化是由正向选择驱动的[33].基于替代率分析,我们发现64.29%的重复基因对具有较强的正选择压力,这表明正选择有助于进一步的基因多样化ZmLTP家庭。此外,还分析了重复基因对之间的发散次数(表2).5个分段重复的重复事件大约发生在943 ~ 3406万年(Mya)之间,而9个串联重复的重复事件大约发生在9.13 ~ 50.46 Mya之间。化石数据和系统发育研究估计,不同的草科从50至70亿年的共同祖先中分离出来[34].假设玉米和水稻的散度为50 Mya[,采用Sanderson的非参数速率平滑方法估计树节点上的散度时间。35].一个串联复制基因对的重复事件(ZmLTPg20/ZmLTPg21)发生在约50.46 Mya,在草的起源之后,在水稻和玉米的分化之前(近7000万至5000万年)。玉米和高粱的祖先大约在12 Mya发生了分化,随后大约5 Mya在玉米中发生了全基因组三倍繁殖(WGT)事件[34].10对基因的重复事件(ZmLTPg11/ZmLTPx2,ZmLTPg13/ZmLTPg21,ZmLTPd10/ZmLTPd11,ZmLTP2.7/ZmLTP2.9,ZmLTPd7/ZmLTPd13,ZmLTP1.6/ZmLTP1.7,ZmLTP2.7/ZmLTP2.8,ZmLTPd12/ZmLTPd13,ZmLTPg6/ZmLTPg7而且ZmLTPg6/ZmLTPg8)发生在3991 ~ 1812万年之间,发生在水稻和玉米分化之后,但在玉米和高粱分离之前。其他两个基因对的复制事件(ZmLTPg6/ZmLTPg16而且ZmLTPd15/ZmLTPd16)发生在大约过去900万年前,在玉米和高粱被分离之后,在玉米发生全基因组复制(WGD)事件之前。因此,玉米nsLTP基因的进化起源可能经历三个进化阶段。
主要nsLTPs的三维结构特征
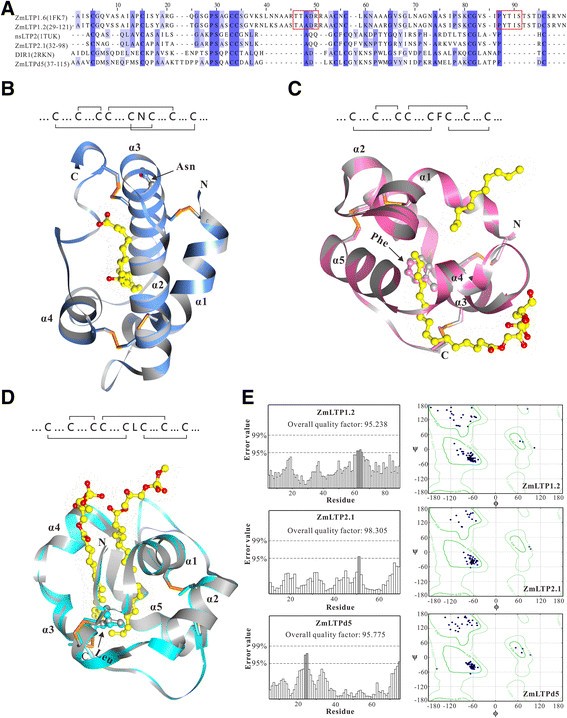
为了更好地理解疏水配体与ZmLTPs之间的非特异性结合,选择ZmLTP1.2、ZmLTP2.1和ZmLTPd5作为Type 1、2和D的代表序列进行结构建模(附加文件)2:表S2)。玉米ZmLTP1.6 (PDB ID: 1FK7)晶体结构研究[7],小麦nsLTP2 (PDB ID: 1TUK) [8], AtDIR1 (PDB ID: 2RKN) [4]被选为结构建模模板,基于使用NCBI的基本局部对齐搜索工具(BLAST)对PDB进行搜索(http://blast.ncbi.nlm.nih.gov/),以目标1型、2型和D型蛋白序列为诱饵。
基于ZmLTP1.6、nsLTP2和AtDIR1结构,ZmLTP1.2的29-121氨基酸、ZmLTP2.1的32-98氨基酸和ZmLTPd5的37-115氨基酸的序列一致性分别为88.17%、62.69%和51.95%。我们的结构分析表明,ZmLTP1.2和ZmLTP1.6具有典型的植物nsLTPs特征,包括两个保守的五肽T-T/A-A-D-R(位置46-50)和P-Y-T-I-S(位置87-91)。据报道,这两种共识五肽(T/S-X-X-D-R/K和P-Y-X-I-S)在催化或结合中很重要[5].ZmLTPd5和AtDIR1含有高脯氨酸含量(位置25-32),这在nsLTP2和ZmLTP2.1中没有突出显示。值得注意的是,脯氨酸丰富区域参与蛋白质-蛋白质相互作用[36].如前所述,X是1型nsLTPs CXC基序中的亲水残基。然而,在2型和D型nsLTPs的X位置发现了疏水残基。如图所示2A,在CXC基序中,ZmLTP1.2和ZmLTP1.6中两个半胱氨酸之间的天门酰胺被ZmLTP2.1和nsLTP2中的疏水氨基酸苯丙氨酸取代,而在ZmLTPd5和AtDIR1中,X位被疏水残基亮氨酸取代。ZmLTP2.1、nsLTP2、ZmLTPd5和AtDIR1 CXC基序中的疏水残基埋藏在分子内部,而ZmLTP1.2和ZmLTP1.6的亲水残基位于分子表面(图2在模板蛋白ZmLTP1.6中,Cys残基1-6、2-3、4-7和5-8配对,而在模板蛋白AtDIR1中,Cys残基1-5、2-3、4-7和6-8配对,与2型nsLTPs相似。这些观察结果表明CXC基序的中心残基可能调控半胱氨酸配对并影响蛋白质的整体折叠。通过Ramachandran图和ERRAT预测,从立体化学质量方面验证了晶体结构同源模型(图2E)。
玉米nsLTPs的结构特征。(一)玉米ZmLTP1.6 (1FK7;氨基酸(aa) 1-93),小麦nsLTP2 (1TUK;aa 1-67), AtDIR1 (2RKN;aa 1-77), ZmLTP1.2 (aa 29-121), ZmLTP2.1 (aa 32-98)和ZmLTPd5 (aa 37-115)。相同的残留物由蓝色背景突出显示。共识残基Thr-X-X-Asp-Arg和pro - tir - x - ile - ser标记在方框中。(B)ZmLTP1.2(蓝色)和ZmLTP1.6(灰色)与蓖麻油酸络合的半胱氨酸配对模式和骨架痕迹的叠加示意图(以球棒状表示;黄色是碳,红色是氧)。四个二硫键用橙色表示。(C)ZmLTP2.1(粉红色)和nsLTP2(灰色)配合LPG (L-α-棕榈酰甘油)脂质配体的半胱氨酸配对模式和主链痕迹的叠加示意图(显示为球棒状;黄色是碳,红色是氧)。四个二硫键用橙色表示。(D)半胱氨酸配对模式和ZmLTPd5主链迹(青色)和AtDIR1晶体结构(灰色)与溶血磷脂酰胆碱分子配合物的叠加示意图(显示为球和棒;黄色是碳,红色是氧)。四个二硫键用橙色表示。(E)描述ZmLTP1.2、ZmLTP2.1和ZmLTPd5的总体质量因子和Ramachandran图的ERRAT结果。
从叠加结构中,我们发现ZmLTP1.2和ZmLTP1.6有一个共同的由四个二硫键稳定的结构褶皱,四个突出的α-螺旋被包裹在由一系列旋转形成的柔性c端臂上,这在疏水腔上形成了一个帽状结构(图2B). ZmLTP2.1的三维结构表明,相邻的两个疏水腔似乎能够扩大疏水腔以容纳烷基配体(图2C). ZmLTPd5的结构在顺序和方向上与AtDIR一致。它们的α-螺旋拓扑结构很保守,四个半胱氨酸桥很好地叠加,在三维结构上表现出很高的相似性(图2D).有趣的是,拟南芥突变体存在缺陷(dir1-1)在制作或传送重要的流动信号以推广远距离信号时受到损害[9].其他D型nsLTPs是否具有与全身抗性信号传导相关的功能仍有待进一步研究。
玉米nsLTPs的基因本体分析
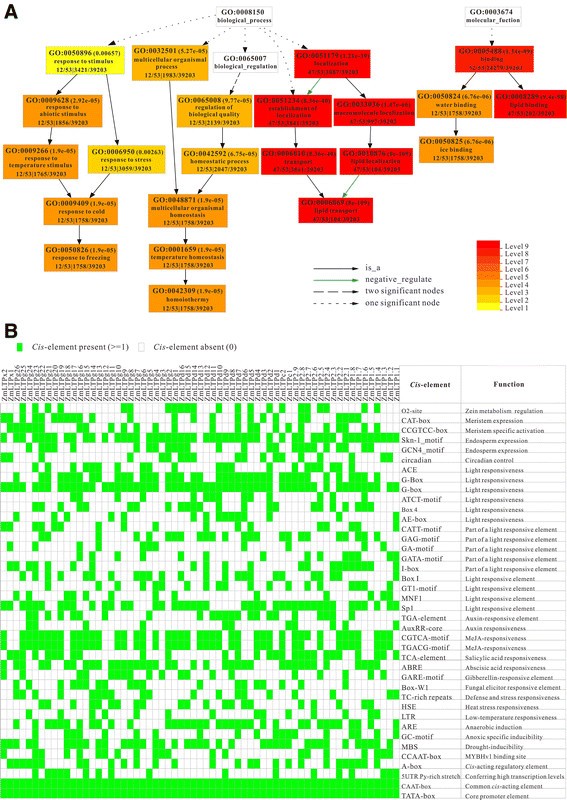
为了了解玉米nsLTPs的独特之处,对其功能意义进行了基因本体(GO)富集分析。在77个ZmLTP蛋白中,有24个蛋白无法进行注释。此外,53个zmltp在22个重要的GO术语中被定义(附加文件12:表S8)。分析显示,53个ZmLTPs被分为两大类(生物过程和分子功能),其中分别包含18个和4个显著的GO术语(图3.A).对于丰富的生物过程,常见的类别是“刺激反应”,其次是“非生物刺激反应”和“应激反应”,“多细胞有机体过程”,“生物调节”和“定位建立”。值得注意的是,大约有47个ZmLTPs参与了“脂质转运”(GO: 0006869),这与nsLTP在转运疏水分子中的分子作用是一致的在体外表明ZmLTPs在将膜成分运送到伸长细胞生长部位的过程中起着重要作用。富集的GO术语还包括父术语“对非生物刺激的反应”和子术语“对冷的反应”,其中22.64%(12 / 53)的ZmLTPs参与了“应激刺激的反应”(特别是在冷的情况下)。这突出了nsLTP蛋白在玉米抗逆性行为中的假定关联。在分子功能方面,约88.68%(47 / 53)的ZmLTPs参与了“脂质结合”(GO: 0008289)。一个有趣的观察结果是,“水结合”和“冰结合”涉及到一些显著丰富的术语。综上所述,GO分析表明ZmLTPs可能参与了多种生物过程。
关键独联体- ZmLTP基因启动子区域内的元件
In-silico对63个玉米nsLTP基因上游1 kb区域(从翻译起始位点)的分析揭示了各种调控元件的存在,这些调控元件与发育、非生物或生物胁迫信号转导以及激素信号转导有关(附加文件)13:表S9)。为避免分析中的偏差,只有独联体-在至少10个不同的基因中发现的元素被考虑。如图所示3.B,除了TATA-box和CAAT-box, G-box是最常见的类型独联体-元素,而涉及光响应性的调节元素似乎富集ZmLTPs.近80%ZmLTPs含有Skn-1_motifs,可能解释胚乳表达。逆境应答独联体本研究鉴定的调控元件包括真菌激发子响应元件(Box-W1)、防御和应激响应元件(TC-rich repeats)、热应激响应元件(HSE)、低温响应元件(LTR)、厌氧响应元件(ARE和GC-motif)、涉及干旱诱导和植物激素响应元件的MYB结合位点(MBS),如生长素响应元件(tga元件和AuxRR-core)、茉莉酸甲酯响应元件(CGTCA-motif和TGACG-motif)、水杨酸响应元件(TCA-element)、脱落酸响应元件(ABRE)和赤霉素酸响应元件(GARE)。除了少数例外,ZmLTPs含有昼夜因素,这可能是其独特的昼夜表达模式的原因。其他调控元件如O2-site(参与玉米醇溶蛋白代谢调节)、DRE(参与脱水、低温和盐的响应)、CAT-box(与分生组织表达相关)和CCGTCC-box(与分生组织特异性激活相关)也被提出。尤其是39号的发起人ZmLTPs包含MBS元素,范围从1到5份(附加文件13:表S9),表明MYB转录因子在调控中的重要作用ZmLTPs.最近的一些报道表明,nsLTP可以作为MYB的靶点,调节植物对冰冻和干旱胁迫的耐受性[24]、[37],表明ZmLTP成员参与了一些非生物胁迫信号的传递。
nsLTPs在玉米发育中的不同作用
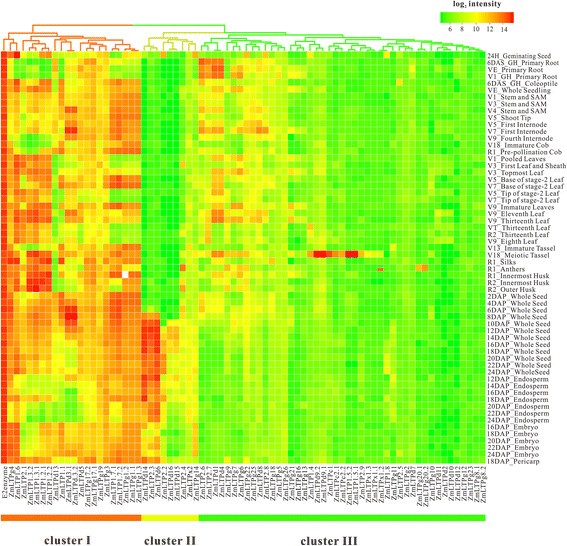
对于多基因家族,基因表达模式的分析往往为确定基因功能提供有用的线索。为了研究nsLTP基因在玉米生命周期中的时空表达模式,通过层次聚类,可视化了ZmLTP基因在11个器官不同发育阶段的整体转录谱。如图所示4在美国,热图可以明显地分为三组。簇I包含21个成员(不包括作为参考的E2酶)。第二组有9个成员,第三组有45个成员。簇I中的基因明显具有较高的表达水平,每个基因的平均log-signal值在9.0 ~ 14.9之间(附加文件)14:表S10)。聚类III包含表达水平相对较低的基因,平均日志信号值范围在5.2 - 9.2之间。在聚类II中,基因表达水平中等,平均日志信号值在6.1 ~ 8.5之间。为了阐明特定器官或阶段推定的差异表达基因,变异系数(CV值;CV = s / x的意思是,其中S为标准差,X为的意思是描述了一个基因在所有组织中的平均表达水平),计算了三个簇中的每个基因(附加文件14:表S10)。在我们的表达分析中,玉米中一种编码泛素偶联酶(E2)的家政基因被用作内参[38].结果表明,各基因间变异较大,CV值在1.01% ~ 52.97%之间。簇I的表达变异性最小,为1.01% ~ 26.80%,表明其相对于其他ZmLTP基因的表达模式最为稳定。ZmLTP1.1是I簇中最易变的基因,在水稻中有同源基因,光周期敏感侏儒1(Psd1,Os01g60740),先前被证明编码一种脂质转移蛋白,该蛋白可能参与调节植物细胞分裂和伸长[39].一个基因(ZmLTP1.2),可与钙调素结合2 +-独立方式[26],除根系、未成熟穗轴和胚乳外,在玉米几乎所有器官和/或发育阶段均表现出高表达。共发现12个nsLTP蛋白编码基因在花药中高表达。7ZmLTPs在叶片不同发育阶段均有高表达,而5ZmLTPs特别是积累在2期叶V5_Base、2期叶V7_Base、v9_未成熟叶、v9_11叶和v9_13叶。聚类II表达变异性最高,为11.95% ~ 52.97%。聚类II中CV值大于15%的基因有8个,其中3个基因(ZmLTPd14,ZmLTP2.3而且ZmLTPd6) CV值大于45%,在种子发育后期(10 ~ 24dap)和胚乳均有转录本积累。据报道BETL9(ZmLTPd6)只在胚乳转移细胞中表达,胚乳转移细胞同为发育中的种子的入口,以吸收来自母体组织的营养物质[28].越来越多的证据表明,这种蛋白质可能参与了发育中的种子对母体植物传播病原体的防御[40]、[41].聚类III的基因波动明显,CV值为4.82% ~ 31.83%。在这种情况下,簇III可以进一步分为两个子簇,第一个子簇中的基因比第二个子簇中的基因具有更高的信号强度值。在第一个子簇中,是4ZmLTPs(ZmLTP2.6,ZmLTP2.7,ZmLTPd1而且ZmLTPd4)在玉米根系发育过程中表现出较高的表达水平,这表明ZmLTPs可能参与玉米根系发育。器官特异性表达动态揭示了ZmLTP基因在玉米整个生命周期中不同的表达模式。例如,两个ZmLTP基因,ZmLTPg20而且ZmLTP2.5,分别在花药和胚芽鞘中特异性表达。有6个基因(ZmLTP1.4,ZmLTP1.5,ZmLTP2.9,ZmLTPc1,ZmLTPc2而且ZmLTPd9), CV值在15.54% ~ 23.92%之间,显著提高了减数分裂流苏的表达水平。其中,ZmLTPc2高度同源于MZm3-3,在玉米雄配子体发生过程中特异性表达于绒毡层[25].此外,四个nsLTP基因(Os09g35700,Os01g49650,Os01g12020而且Os08g43290)均在水稻花药中表达,推测其与渐进性发育阶段孢子体花药的外氨酸合成有关[42].有趣的是,这四种大米nsLTPs的同源物是ZmLTPc2,ZmLTP2.9,ZmLTP1.4而且ZmLTPc1,分别。包括花粉外壁在内的花药中脂质成分的合成对植物雄性生殖发育至关重要。因此,这一发现进一步表明nsLTPs在男性生殖发育中具有重要作用。
病原体反应ZmLTPs真菌感染
植物nsLTPs能够抑制细菌和真菌病原体,因此被认为在植物防御中发挥重要作用[18].为了发现ZmLTP基因参与玉米病原菌反应,分别在处理12、24 h、2、4、4.5、8 d后采集微阵列数据黑粉菌属maydis(美国maydis)感染情况进行分析。美国maydis是一种普遍存在的玉米病原菌,依赖于活组织进行增殖发育。在玉米18k基因芯片上发现的41个探针组可被分配到36个不同的ZmLTP基因(附加文件)15:表S11)。log2比值值(处理/对照)由热图表示,显示了每个表达的折叠变化ZmLTP.如附加文件所示16:图S5,大部分ZmLTPs感染后表现为延迟表达模式美国Maydis.23ZmLTPs在感染后第4和第8天,其表达量显著增加,且主要聚集在感染后第4和第8天ZmLTPg1,ZmLTP1.1而且ZmLTP1.7这表明这些基因可能参与了病原体的反应。有趣的是,一些ZmLTP基因的表达水平随着时间的推移而上调,特别是ZmLTP2.8,对这种真菌感染表现出类似的反应模式。此外,表达水平ZmLTPd2而且ZmLTP2.1感染后迅速增加,说明它们对美国maydis感染。拟南芥中的一个nsLTP (AtDIR1)被认为参与了病原体防御过程中的长距离信号传递[9].因此,寻找一份成绩单的积累是合理的ZmLTPd5,是来自玉米的AtDIR1同源物美国maydis.除了基因上调后美国maydis感染时,也有一些ZmLTP基因的表达水平随着时间的推移而下降,如ZmLTPg23而且ZmLTPd6.这些基因可能在其他生物过程中发挥作用,或对其他种类的病原体攻击做出反应。综上所述,这些结果表明,该家族中的许多基因可能参与了病原体反应。
玉米nsLTP基因对非生物胁迫的转录应答
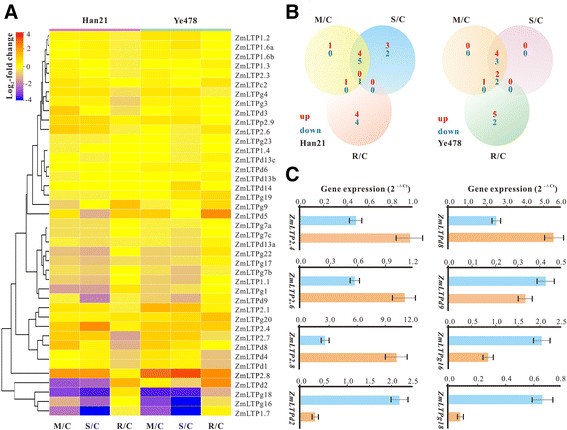
除了对病原体感染的诱导外,nsLTP基因还对干旱、寒冷和盐等非生物胁迫有反应[21]、[43].调查…的可能作用ZmLTPs在干旱胁迫下,利用抗旱系Han21和抗旱敏感系Ye478两个自交系在不同干旱处理下的微阵列数据[44].经过全芯片数据处理,在微阵列中提取了57.14%(63个中的36个)的ZmLTP基因,约41个探针(有些基因有两个或多个探针集)用于分析(附加文件)15:表S11和图5A).使用折叠改变>2和P值<0.05时,在至少一种条件下表达谱变化的ZmLTP基因被认为是差异表达基因(图5B).结果显示14和13ZmLTPs干旱和/或复水处理分别对汉21和叶478玉米自交系的光合速率有差异调节。有趣的是,一些ZmLTPsHan21在干旱胁迫下表达上调,表明它们可能在玉米干旱胁迫反应中起作用。例如,转录水平ZmLTP2.4,ZmLTP2.7而且ZmLTP2.8,在中度和重度干旱胁迫下均显著上调5A)。此外,ZmLTP2.6而且ZmLTPd8与未处理的样品相比,在中度干旱条件下显示高丰度的转录水平。为了验证这些预测基因在干旱胁迫下的表达模式,我们选择了8个差异表达基因进行qRT-PCR分析,所用引物在附加文件中列出17:表S12。如图所示5C,ZmLTP2.4,ZmLTP2.6,ZmLTP 2.8而且ZmLTPd8在缺水时明显上调,而ZmLTPd2,ZmLTPd9,ZmLTPg16而且ZmLTPg18在干旱胁迫下下调。综上所述,基于qrt - pcr的这些基因表达谱证实了微阵列分析的结果。
差异基因表达ZmLTPs在干旱胁迫下。(一)36例表达谱ZmLTPs汉21和叶478在中度干旱胁迫(M/C)、重度干旱胁迫(S/C)和复水(R/C)下处理。基于log2的折叠变化被用于创建热图。(B)汉21和叶478在中度干旱胁迫(M/C)、重度干旱胁迫(S/C)和复水胁迫(R/C)下ZmLTP基因差异表达数的维恩图。如果一个基因在任何给定条件下上调或下调至少两倍且P值<0.05,则认为该基因存在差异表达。(C)选择性ZmLTP基因在干旱处理中的定量RT-PCR分析。以玉米UBQ1基因为内源对照,进行数据归一化。所表示的值是两个生物重复的平均值,每个重复有三个技术重复。误差条表示由两次生物重复确定的标准偏差。
除了DNA微阵列分析外,还进行了RNA-seq分析以研究其功能ZmLTPs玉米生殖组织(子房)和营养组织(叶片)在干旱和充足水条件下的干旱敏感性差异[45].的近66.67% (42)ZmLTPs通过RNA-seq数据可靠地检测到(附加文件18:表S13)。在42个表达基因中,3个基因(ZmLTP2.8,ZmLTPg14而且ZmLTP2.2)在干旱胁迫下的子房组织中表达明显上调,17个基因表达下调,10个基因(ZmLTPd3,ZmLTP2.8,ZmLTPd4,ZmLTPd1,ZmLTP2.6,ZmLTPg2,ZmLTP1.1,ZmLTPg3,ZmLTPg1而且ZmLTPd5)显著上调,而2 (ZmLTP2.9而且ZmLTPg24)在干旱胁迫下叶片分生组织中表达下调,表明这些ZmLTP基因在干旱胁迫下可能表现出组织特异性表达。
植物中许多nsLTPs已被报道与干旱胁迫密切相关。植物的叶片表面有一层角质层,可以抵御环境逆境,如干旱胁迫。在拟南芥中,AtLTPg4与玉米中的ZmLTPg14具有密切的系统发育关系,在蜡和/或角质单体运输中发挥作用[46].在我们的研究中,表达水平ZmLTPg14在干旱胁迫下的子房组织中增加,这意味着它可能在抗旱性中起作用。ZmLTP1.2在干旱胁迫下,钙调素在子房组织中表达显著下调,而在叶片分生组织中表达上调。不出所料,应激反应基因,ZmLTP1.1干旱胁迫下,其在叶片分生组织中的表达量增加,而在子房组织中表达量下降。无论如何,基于微阵列和RNA-seq方法的表达谱分析证明,ZmLTPs可能在玉米抗旱性中发挥重要作用。然而,还需要进一步的工作来了解ZmLTPs与玉米干旱胁迫反应的机制联系。
除了了解干旱胁迫对玉米nsLTP基因转录水平的影响外,了解这些基因在盐胁迫下的表达水平也很重要。根是第一个直接受到盐胁迫的器官。为了更好地了解ZmLTP基因在盐胁迫响应中的作用,我们进行了RNA-seq分析,以探索ZmLTP基因在盐胁迫响应中的表达谱ZmLTPs在盐胁迫下,玉米三种根类型[主根(PR)、种子根(SR)和冠根(CR)]均有显著差异。近60.32% (38 / 63)ZmLTPs通过RNA-seq检测。如附加文件所示19:图S6, 3个ZmLTP基因(ZmLTP2.4, ZmLTPd3而且ZmLTPg17)和4个基因(ZmLTP2.8,ZmLTPd3,ZmLTPd11而且ZmLTPg17)在盐胁迫下,PR和SR分别上调至少2倍。盐胁迫下,11个ZmLTP基因(ZmLTP1.1,ZmLTP1.5,ZmLTP2.4,ZmLTP2.8,ZmLTPd3,ZmLTPd9,ZmLTPd11,ZmLTPg2,ZmLTPg4,ZmLTPg5而且ZmLTPg10)显著增加了其在CR中的表达水平ZmLTPs在盐胁迫下,CR的变化大于PR和SR。一个ZmLTP基因,ZmLTPd3在PR、SR和CR中均表现为盐胁迫的高度诱导,表明其可能在玉米盐胁迫响应中发挥重要作用。此外,ZmLTP1.1在CR中明确表达,这与之前的报告一致,ZmLTP1.1可能参与耐盐[27].
众所周知,植物经常受到各种各样的环境胁迫。其中,低温是影响植物生长发育和作物产量的关键因素。可溶性糖和脯氨酸的积累对植物抵御低温胁迫很重要[47].在以往的研究中,水稻冷诱导nsLTP基因在转基因中的表达蝴蝶兰属amabilis提高了对冷应激的适应性反应。对转基因植物的检查表明,几种相容溶质如总可溶性糖和脯氨酸的积累增加了[48].为了进一步解析ZmLTPs在抗寒性中的生物学功能,我们研究了低温敏感玉米系ETH-DL3和耐冷玉米系ETH-DH7在低温胁迫条件下ZmLTPs可能发挥的作用[49].采用全芯片数据处理后,54%(63人中的34人)ZmLTPs在玉米58 k NSF阵列上共提取了36个探针集(部分基因有两个探针集)进行分析(附加文件20.:表S14)。三个ZmLTPs(ZmLTPd12,ZmLTPg9而且ZmLTP1.5)被定义为响应冷应激的差异表达基因(附加文件21:图S7)。令人惊讶的是,所有三个差异表达的基因在严寒胁迫下都上调了。其中,ZmLTPd12而且ZmLTPg9在重度冷胁迫条件下,ETH-DH7表达差异显著ZmLTP1.5而且ZmLTPd12在重度冷胁迫条件下,ETH-DL3表达上调。
本研究揭示了干旱、盐胁迫和冷胁迫对玉米nsLTP基因家族几个成员转录丰度的影响,表明它们在非生物胁迫中具有重要作用。而且,促进者居多ZmLTPs被发现包含一个或多个重要的独联体- ABRE、MBS、TC-rich repeat和C-repeat/DRE等监管元素(附加文件13:表S9),这可能与调控的表达有关ZmLTPs在这样的压力下。因此,抗非生物胁迫的表达谱分析为nsLTP基因的特定和/或重叠表达模式提供了新的见解。这些分析将有助于揭示信号转导、应激反应和发育过程的分子机制。
结论
我们在玉米基因组中鉴定了63个nsLTP基因,该家族成员可分为1、2、C、D和G 5种类型。对ZmLTPs的全面研究揭示了该基因家族的一些重要特征,如基因结构、重复事件和三维结构。此外,还进行了基因本体分析,以获得有关ZmLTPs生物学功能的线索。对上游调控元件的分析进一步表明,ZmLTPs可能参与了多种生物学过程,并为研究ZmLTPs共享和不同的转录调控机制提供了新的视角ZmLTPs.的表达式配置文件ZmLTPs在不同的发育阶段表明,该家族的一些成员表现出组织特异性表达,表明它们在玉米生命周期中发挥不同的发育和生理功能。此外,我们着重讨论了玉米的作用nsLTPs在压力反应中。结果表明,一些ZmLTPs感染后表现为延迟表达模式黑粉菌属maydis在干旱、盐胁迫和冷胁迫下均有差异表达,可用于进一步提高玉米抗逆性的研究。综上所述,本研究结果对玉米优质性状分子育种具有重要意义。
方法
玉米和高粱基因组中nsLTPs的提取和鉴定
我们的搜索策略使用隐马尔可夫模型(HMM)和BLAST搜索的连续迭代来识别玉米中小的、分泌的nsLTPs。从玉米序列数据库(release version 5b;http://www.maizesequence.org/index.html).从用Pfam结构域PF00234(植物脂质转移/种子储存/胰蛋白酶- α淀粉酶抑制剂)注释的蛋白质序列开始,我们使用HMMER 3.0包中的hmmsearch扫描玉米蛋白质组序列[50使用默认参数。为了增加在玉米中检测假定的nsLTP的概率,以拟南芥和水稻中所有已知的nsLTP氨基酸序列为诱饵,进一步对玉米蛋白质组序列进行BLASTP搜索。结果被合并,冗余序列被删除。然后,人工检查候选nsLTPs的蛋白质序列,以包含8CM,并去除缺乏必需半胱氨酸残基的蛋白质。此外,编码假定的富含脯氨酸的蛋白质被排除在进一步的分析之外。剩余的候选蛋白被提交到InterProScan (http://www.ebi.ac.uk/InterProScan)来验证nsLTP域的存在。2s蛋白蛋白序列(At2S1 ~ At2S4) [51],以及alpha淀粉酶抑制剂(RATI) [52]与其他候选nsLTP蛋白进行blast搜索,以排除可能的谷物储存蛋白。此外,高粱基因组序列从联合基因组研究所植物基因组数据库(发布版本1.4;http://www.Phytozome.net),同样的方法用于鉴定高粱中的nsLTPs。
主序列分析
使用SignalP v4.0程序分析所有鉴定出的nsLTPs是否存在潜在的信号肽切割位点[53].通过LocTREE 3预测nsLTP的亚细胞定位(https://rostlab.org/services/loctree3/).去除信号肽后,用ExPasy的pI/Mw工具计算成熟蛋白的分子量和等电点(http://web.expasy.org/compute_pi/).pI计算时,8CM结构域的8个Cys残基被排除在外,因为二硫键中的Cys残基不带电荷。三大预测工具,PredGPI [54],大pi植物预测器[55]及GPI-SOM [56]被用来确定gpi -锚点的存在。通过P3DB预测nsLTPs的磷酸化位点(http://www.p3db.org/).使用BioEdit v7.0.9.0计算成熟nsLTP序列的序列单位矩阵(http://www.mbio.ncsu.edu/bioedit/bioedit.html),使我们能够确定基因亚家族分配。命名是基于Edstam等人提出的指导方针。[6].
序列比对和系统发育重建
拟南芥nsLTP氨基酸序列(答:芥)和米(o .漂白亚麻纤维卷)已从TAIR (http://www.Arabidopsis.org/)及TIGR (http://rice.plantbiology.msu.edu/)数据库。利用MEGA 5软件中实现的ClustalW对成熟蛋白的8CM部分进行多次比对。在ProtTest v2.4中,手动细化的对齐被用作输入[57],设置为所有候选模型、BIONJ树和缓慢优化策略。采用在ProtTest中实现的Akaike信息准则选择最佳模型(WAG + I + G)。然后,将玉米、水稻和拟南芥的8CM氨基酸序列共274条序列加载到PhyML v3.0软件中[58].每个节点的置信度由使用100次重采样重复数据的bootstrap程序估计[59].
基因结构及保守内含子剪接位点分析
为了探索玉米nsLTP基因的不同外显子-内含子组织,我们使用GSDS软件(http://gsds.cbi.pku.edu.cn).为了更好的可视化和比较,5' UTR序列事先被删除。8CM的位置用PAL2NAL (http://www.bork.embl.de/),将8CM序列与相应基因序列的多序列比对转换为密码子比对,然后用红色标记[60].
基因组分布和基因重复分析
根据物理位置(bp)从短臂端粒到长臂端粒的递增顺序,将这些基因分别绘制在10条玉米染色体上。由于一个基因家族可以通过串联和片段复制事件来扩展,我们打算确定ZmLTP基因家族扩展的机制。插入基因不超过10个的ZmLTP基因被认为是串联复制[61].根据TIGR数据库的搜索结果,将水稻、高粱和拟南芥中假定的othologs分配到相应的玉米nsLTP蛋白(http://rice.plantbiology.msu.edu/annotation_pseudo_apk.shtml).从植物基因组复制数据库(PGDD,http://chibba.agtec.uga.edu/duplication).这些表意文字是用Circos软件[62].
同义和非同义替换率的估计
采用DnaSP v5.0计算重复基因的同义和非同义替代率[63].通过分析非同义核苷酸置换率与同义核苷酸置换率的比值来检测选择模式。Ka/Ks比值>1、<1和=1分别代表阳性选择、净化选择和中性选择。用方程T = Ks/2λ × 10计算了重复事件发生的日期6Mya, (λ =6.5 × 109) [64]、[65].
玉米nsLTPs的同源性建模
用BLASTP软件在蛋白数据库(Protein Data Bank, PDB)中以默认参数对所有玉米nsLTP蛋白进行搜索,以确定最佳模板。数据采用SWISS-MODEL [66]通过同源建模来预测蛋白质结构。为了可视化和编辑PDB模型,使用交互式分子图形程序Chimera v1.8 [67].使用Chimera计算用于评估模型的Ramachandran图,并使用UCLA-DOE结构分析与验证服务器(http://nihserver.mbi.ucla.edu/ERRAT/).利用MEGA 5软件中实现的ClustalW对各nsltp进行序列比对,并通过Jalview (http://www.jalview.org/).
基因本体分析
GO富集分析使用基于网络的工具AgriGO (http://bioinfo.cau.edu.cn/agriGO/index.php).采用奇异富集分析(SEA)方法鉴定富集的GO项,选取显著富集的功能簇,调整后的q值小于0.05。该程序提供了定义GO分类的三类输出,即生物过程、细胞成分和分子功能。
玉米nsLTP基因启动子分析
调查独联体在玉米nsLTP基因启动子序列中,每个启动子序列上游(~1000 bp)被鉴定ZmLTPs使用Perl脚本从B73玉米基因组序列中检索。PlantCARE网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)被用来识别独联体-启动子中的元素[68].为避免分析上的偏差,只能独联体-在至少10个不同基因中发现的元素被进一步考虑。
基于微阵列的基因表达分析
为了深入了解整个家族的表达谱,进行了基于微阵列的发育和应激反应数据分析。为了实现ZmLTP基因在不同器官和发育阶段的表达谱分析,从公开数据库PLEXdb(登录号ZM37)获取玉米自交系B73全基因组基因表达图谱的转录组数据。从GEO数据库(登录号分别为GSE16567和GSE10023)中下载干旱胁迫和病原侵染的基因表达数据。冷应力微阵列数据从ArrayExpress数据库下载,登录号为E-MTAB-1315。为了鉴定ZmLTP基因的探针集,在Affymetrix netafffx网站(http://www.affymetrix.com/estore/)和PLEXdb。使用稳健多芯片平均(RMA)算法对数据进行归一化[69].数据处理采用Limma包,用gplots包生成表示log2转换后探头强度的热图[70].应用层次聚类算法确定表达式配置文件中的相似模式。使用折叠改变>2和P值<0.05时,ZmLTP基因在干旱胁迫下被认为是差异表达基因,而在冷胁迫下则至少是差异表达基因的2.8倍。
基于RNA-seq的差异基因表达谱
此前,通过RNA-seq分析的玉米干旱转录组研究了干旱处理和充分浇水的受精生殖组织(子房)和营养组织(基叶分生组织)中干旱响应基因的表达模式[45].子房和基叶分生组织各有四个文库,分别对应于水分充足和干旱胁迫的组织,各有一个生物复制。每个基因的转录丰度由每千碱基片段外显子每百万片段映射(FPKM)估计。为了获得盐胁迫下三种根类型(PR、SR和CR) ZmLTP基因的表达模式,从登录号为E-GEOD-53995的ArrayExpress数据库中下载了RNA-seq数据。每个基因的转录丰度由reads per kilobase per million mapped reads (RPKM)估算。
实时PCR分析
玉米种子在30°C下孵育发芽,播种于含0.1 m砂壤土的塑料盆中3.每一个。将每株盆栽分为3组,每组2个重复。在受控光周期14 h、昼夜温度28 × 2℃、空气湿度55%的条件下进行植株生长。从播种之日起34天(此时所有植株都有3-4片发育完全的叶片),保留一套花盆作为对照(充分浇水),另外两套花盆用于干旱,闭水6天。在此期间结束时,干旱胁迫植物的土壤体积含水量(从土壤顶部5°Cm深度测量)约为12.5%,而对照植物约为30%。收获幼苗的叶子,立即在液氮中冷冻,并在-80°C保存。然后,用Trizol试剂从干旱胁迫后的叶片中提取总RNA,然后用DNase I处理去除基因组DNA污染。利用Takara RNA PCR试剂盒反转录总RNA约2 μg,得到cDNA。QRT-PCR在iCycler iQ5多色实时PCR检测系统(Bio-Rad)上进行,使用Power SYBR Green PCR Master Mix (Applied Biosystems)。每个样品使用两个生物重复,每个生物重复使用三个技术重复。 The thermal cycling conditions were as follows: 50°C for 2 min, 95°C for 10 min, 40°Cycles of 95°C for 15's and 60°C for 1 min. Quantification of gene expression was performed by 2——ΔCt方法。以玉米biquilin-1 (UBQ1)基因作为内控进行归一化。
支持数据的可用性
所有支持性数据都包含在附加文件中。
作者的贡献
KW概念化,设计项目。XZ和KW进行了计算分析、微阵列和RNA-seq表达分析。KW和XZ进行实验。XZ和KW撰写了手稿。两位作者都阅读并批准了最终的手稿。
附加文件
参考文献
- 1.
植物中的脂质转移蛋白。植物化学学报,2004,27(4):344 - 344。10.1146 / annurev.arplant.47.1.627。
- 2.
Boutrot F, Chantret N, Gautier MF:水稻和拟南芥非特异性脂质转移蛋白(nsLtp)基因家族的全基因组分析和小麦nsLtp基因的EST数据挖掘鉴定。中国生物医学工程学报,2008,29(4):344 - 344。
- 3.
Debono A, Yeats TH, Rose JK, Bird D, Jetter R, Kunst L, Samuels L:拟南芥LTPG是一种糖基磷脂酰肌醇锚定的脂质转移蛋白,用于将脂质输出到植物表面。植物学报,2009,21:1230-1238。10.1105 / tpc.108.064451。
- 4.
Lascombe MB, Bakan B, Buhot N, Marion D, Blein JP, Larue V, Lamb C, Prange T:“诱导抗性缺陷”蛋白的结构拟南芥DIR1揭示了一种新型脂质转移蛋白。蛋白质学报,2008,17:1522-1530。10.1110 / ps.035972.108。
- 5.
杜利兹,李丽娟,李丽娟,马丽娟:谷物籽粒中脂质结合蛋白的结构、生物学和技术功能。粮食科学,2000,32:1-20。10.1006 / jcrs.2000.0315。
- 6.
李志强,李志强,李志强,等。非特异性脂质转移蛋白的研究进展。植物学报,2011,4:947-964。10.1093 / mp / ssr019。
- 7.
韩gw,李建勇,宋hk, Chang C, Min K, Moon J, Shin DH, Kopka ML, Sawaya MR, Yuan HS, Kim TD, Choe J, Lim D, Moon HJ, Suh SW:高分辨率x射线晶体学揭示玉米脂质转移蛋白复合物非特异性脂质结合的结构基础。中华生物医学杂志,2001,30(3):344 - 344。10.1006 / jmbi.2001.4559。
- 8.
Hoh F, Pons JL, Gautier MF, De Lamotte F, Dumas C:小麦配体2型非特异性脂质转移蛋白的结构及脂质结合的分子基础。生物晶体学报,2005,61:397-406。10.1107 / S0907444905000417。
- 9.
Maldonado AM, Doerner P, Dixon RA, Lamb CJ, Cameron RK:拟南芥系统性抗性信号的脂质转移蛋白。自然科学,2002,29(3):349 - 349。10.1038 / nature00962。
- 10.
杨晓明,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚,李志刚:烟草LTP1生物活性的研究进展。中国生物医学工程学报,2004,29(3):349 - 349。10.1091 / mbc.e04 - 07 - 0575。
- 11.
倪志刚,刘志刚,王志刚,王志刚。脂质转移蛋白对烟草细胞壁延伸的促进作用。中国生物医学工程学报,2005,17:2009-2019。10.1105 / tpc.105.032094。
- 12.
王晓燕,王晓燕,王晓燕,王晓燕,王晓燕。胡萝卜EP2脂质转移蛋白基因的细胞特异性表达。植物科学学报,2004,27(3):374 - 374。10.1105 / tpc.3.9.907。
- 13.
李珊珊,郭玉生,裴海杰,朴俊华,赵海杰,赵海杰,李世德,朴ok,黄毅,Suh MC:糖基磷脂酰肌醇-脂质转移蛋白基因的破坏改变了表皮脂质组成,增加了质体蛋白,并增强了真菌病原体十字花型油菜链孢菌(Alternaria brassicicola)的易感性。植物科学进展,2009,30(4):344 - 344。10.1104 / pp.109.137745。
- 14.
Edstam MM, Blomqvist K, ekklof A, Wennergren U, Edqvist J:共表达模式表明gpi -定位的非特异性脂质转移蛋白参与角质层蜡、子皮素和孢粉素的积累。中国生物医学工程学报,2013,29(3):344 - 344。10.1007 / s11103 - 013 - 0113 - 5。
- 15.
黄冬梅,陈立林,黄亚华:拟南芥绒毡层中丰富的III型脂质转移蛋白分泌到房室,成为花粉外壁的组成部分。中国生物医学工程学报,2013,29(3):344 - 344。10.1104 / pp.113.225706。
- 16.
张东,梁伟,尹超,宗杰,顾峰,张东:OsC6编码脂质转移蛋白,是水稻减数分裂后花药发育所必需的。中国生物医学工程学报,2010,29(4):344 - 344。10.1104 / pp.110.158865。
- 17.
Van Loon LC, Van Strien EA:致病相关蛋白家族及其活性,以及PR-1型蛋白的比较分析。植物病理学杂志,1999,29(3):344 - 344。10.1006 / pmpp.1999.0213。
- 18.
Buhot N, Douliez JP, Jacquemard A, Marion D, Tran V, Maume BF, Milat ML, Ponchet M, Mikes V, Kader JC, Blein JP:一种脂质转移蛋白与控制植物防御反应的受体结合。农学通报,2001,39(2):344 - 344。10.1016 / s0014 - 5793(01) 03116 - 7。
- 19.
blin JP, Coutos-Thevenot P, Marion D, Ponchet M:从诱导素到脂质转移蛋白:植物防御机制中细胞信号通路的新认识。植物科学进展,2002,7:293-296。10.1016 / s1360 - 1385(02) 02284 - 7。
- 20.
Hincha DK, Neukamm B, Sror HA, Sieg F, Weckwarth W, Ruckels M, Lullien-Pellerin V, Schroder W, Schmitt JM:卷心菜低温保护蛋白是非特异性植物脂质转移蛋白基因家族成员。中国农业科学,2001,29(4):457 - 457。10.1104 / pp.125.2.835。
- 21.
Jung HW, Kim W, Hwang BK:编码辣椒脂质转移蛋白的三个病原体诱导基因在病原体、非生物和环境胁迫下被不同的激活。植物细胞环境学报,2003,26:915-928。10.1046 / j.1365-3040.2003.01024.x。
- 22.
Cameron KD, Teece MA, Smart LB:烟草叶片表皮蜡积累和脂质转移蛋白表达增加对周期性干燥事件的响应。植物科学学报,2006,30(4):366 - 366。10.1104 / pp.105.069724。
- 23.
黄柏凯,金玉杰,黄柏凯,陈晓明,辛杰:烟草脂质转移蛋白(LTP)基因的过表达及其在烟草长距离系统信号转导中的作用。植物细胞学报,2009,28:419-427。10.1007 / s00299 - 008 - 0653 - 3。
- 24.
郭林,杨华,张旭,杨松:脂质转移蛋白3作为MYB96的靶点介导拟南芥的冻结和干旱胁迫。应用物理学报,2013,32(3):344 - 344。10.1093 / jxb / ert040。
- 25.
Lauga B, charbonnell - campa L, Combes D:特征MZm3-3,一个玉米tapetum-specific成绩单。中国植物科学,2000,29(3):344 - 344。10.1016 / s0168 - 9452(00) 00267 - 3。
- 26.
李C,谢W,白W,李Z,赵Y,刘H:钙调蛋白结合玉米脂质转运蛋白和调节其脂质结合能力。中国生物医学工程学报,2008,27(3):344 - 344。10.1111 / j.1742-4658.2008.06660.x。
- 27.
邹华文,田晓辉,马国华,李宗祥:聚硅酸酯的分离与功能分析ZmLTP3,与拟南芥同源LTP3.中华分子生物学杂志,2013,14:5025-5035。10.3390 / ijms14035025。
- 28.
Royo J, Gomez E, Sellam O, Gerentes D, Paul W, Hueros G:两种玉米END-1直接同源,BETL9而且BETL9like,在发育中的胚乳外表面以不重叠的空间模式转录。植物科学,2014,5:180-10.3389/fpls.2014.00180。
- 29.
李晓明,李晓明,李晓明,李晓明,等。大豆疏水蛋白的晶体学研究。中华分子生物学杂志,1998,21(3):349 - 349。10.1016 / 0022 - 2836(89) 90303 - 3。
- 30.
Wei F, Coe E, Nelson W, Bharti AK, Engler F, Butler E, Kim H, Goicoechea JL, Chen M, Lee S, Fuks G, Sanchez-Villeda H, Schroeder S, Fang Z, McMullen M, Davis G, Bowers JE, Paterson AH, Schaeffer M, Gardiner J, Cone K, Messing J, Soderlund C, Wing RA:玉米基因组的物理和遗传结构反映了其复杂的进化史。公共科学学报,2007,3:e123-10.1371/journal.pgen.0030123。
- 31.
Schnable PS, Ware D, Fulton RS, Stein JC, Wei F, Pasternak S, Liang C, Zhang J, Fulton L, Graves TA, Minx P, Reily AD, Courtney L, Kruchowski SS, Tomlinson C, Strong C, Delehaunty K, Fronick C, Courtney B, Rock SM, Belter E, Du F, Kim K, Abbott RM, Cotton M, Levy A, Marchetto P, Ochoa K, Jackson SM, Gillam B: B73玉米基因组:复杂性、多样性和动态。科学通报,2009,29(4):344 - 344。10.1126 / science.1178534。
- 32.
魏坤,陈勇,谢东:1996-2012年中国H5N1流感病毒基因组尺度演化与系统动力学。中国畜牧兽医杂志,2013,27(3):344 - 344。10.1016 / j.vetmic.2013.09.030。
- 33.
杨旭,葛图坎,程明志:杨树、拟南芥和水稻Dof基因家族的差异表明基因复制后存在多种进化模式。植物科学学报,2006,29(3):344 - 344。10.1104 / pp.106.083642。
- 34.
Salse J, Bolot S, Throude M, Jouffe V, Piegu B, Quraishi UM, Calcagno T, Cooke R, Delseny M, Feuillet C:水稻和小麦共享重复的鉴定和表征为草基因组进化提供了新的视角。植物学报,2008,20:11-24。10.1105 / tpc.107.056309。
- 35.
王志刚,王志刚,王志刚,王志刚,王志刚。高粱和玉米基因组祖细胞的分离。基因组学报,2004,14:1616 -1923。10.1101 / gr.2332504。
- 36.
Williamson MP:蛋白质中富含脯氨酸区域的结构和功能。生物化学学报,2004,27(2):379 - 379。
- 37.
徐普杰,李树生,徐春梅,朴俊杰,高玉生,朴春梅:干旱条件下MYB96转录因子对拟南芥角质层蜡生物合成的调控作用。植物学报,2011,23:1138-1152。10.1105 / tpc.111.083485。
- 38.
魏克峰,王艳梅,谢晓东:玉米发育过程中蛋白激酶基因超家族的鉴定及表达谱分析。动物遗传学报,2014,33:155-172。10.1007 / s11032 - 013 - 9941 - x。
- 39.
李锐,夏娟,徐勇,赵旭,刘永刚,陈勇:a .植物的遗传定位与鉴定光周期敏感侏儒1水稻基因座(栽培稻l .)。应用理论与实践,2014,27(1):1- 4。10.1007 / s00122 - 013 - 2213 - 7。
- 40.
Serna A, Maitz M, O°Connell T, Santandrea G, Thevissen K, Tienens K, Hueros G, Faleri C, Cai G, Lottspeich F, Thompson RD:玉米胚乳分泌一种新的抗真菌蛋白到邻近母质组织。植物学报,2001,25:687-698。10.1046 / j.1365 - 313 x.2001.01004.x。
- 41.
Gomez E, Royo J, Muniz LM, Sellam O, Paul W, Gerentes D, Barrero C, Lopez M, Perez P, Hueros G:玉米转录因子myb相关蛋白-1是转移细胞分化的关键调节因子。中国生物医学工程学报,2009,29(4):344 - 344。10.1105 / tpc.108.065409。
- 42.
黄冬梅,魏富军,吴春成,邢毅,黄亚华:水稻花药转录组分析揭示了整体绒毡层分泌功能和脂质外膜形成的潜在蛋白。植物科学学报,2009,29(3):344 - 344。10.1104 / pp.108.131128。
- 43.
张春春,李海杰,张世杰,徐耀文:基因表达及启动子分析TaLTP1小麦干旱和盐胁迫诱导的基因(小麦l .)。植物科学学报,2004,27(3):344 - 344。10.1016 / j.plantsci.2004.05.019。
- 44.
郑洁,付洁,勾明,怀娟,刘勇,简明,黄强,郭霞,董志,王宏,王刚:干旱胁迫下两个玉米自交系的全基因组转录组分析。中国生物医学工程学报,2010,27(3):344 - 344。10.1007 / s11103 - 009 - 9579 - 6。
- 45.
Kakumanu A, Ambavaram MM, Klumas C, Krishnan A, Batlang U, Myers E, Grene R, Pereira A:干旱对玉米生殖组织和叶片分生组织基因表达的影响植物科学进展,2012,29(4):344 - 344。10.1104 / pp.112.200444。
- 46.
金海红,李雪峰,黄毅,黄秀珍:糖基磷脂酰肌醇锚定脂质转移蛋白2 (LTPG2)的研究及其在角质层蜡输出或堆积中的重叠作用拟南芥.中国生物医学工程学报,2012,29(3):344 - 344。10.1093 /卡式肺囊虫肺炎/ pcs083。
- 47.
Wanner LA, Junttila O:拟南芥低温诱导的抗冻性。植物营养学报,2004,27(3):366 - 366。10.1104 / pp.120.2.391。
- 48.
秦霞,刘勇,毛松,李涛,吴红,褚超,王勇:脂质转移蛋白编码基因在植物中的遗传转化蝴蝶兰属amabilis增强抗寒能力。《植物科学》,2011,37(3):344 - 344。10.1007 / s10681 - 010 - 0246 - 4。
- 49.
Sobkowiak A, Jonczyk M, Jarochowska E, Biecek P, Trzcinska-Danielewicz J, Leipner J, Fronk J, Sowinski P:玉米自交系低温响应的全基因组转录组分析揭示了决定差异性冷敏感性的候选基因。中国生物医学工程学报,2014,34(4):317-331。10.1007 / s11103 - 014 - 0187 - 8。
- 50.
Finn RD, Clements J, Eddy SR: hmm web服务器:交互式序列相似性搜索。生物工程学报,2011,39:W29-W37。10.1093 / nar / gkr367。
- 51.
Guerche P, Tire C, De Sa FG, De Clercq A, Van Montagu M, Krebbers E:拟南芥2S白蛋白基因的差异表达及增加基因家族大小的影响。植物科学学报,2000,27(2):366 - 366。10.1105 / tpc.2.5.469。
- 52.
strobel S, Maskos K, Wiegand G, Huber R, Gomis-Ruth FX, Glockshuber R:一种抑制α -淀粉酶的新策略:黄粉虫α -淀粉酶与Ragi双功能抑制剂在2.5 A分辨率下的复合物。结构学报,1998,6:911-921。10.1016 / s0969 - 2126(98) 00092 - 6。
- 53.
陈晓明,陈晓明,陈晓明,等。跨膜区信号肽识别技术研究进展。岩石力学与工程学报,2011,29(3):344 - 344。10.1038 / nmeth.1701。
- 54.
Pierleoni A, Martelli PL, Casadio R: PredGPI:一个gpi锚预测器。中国生物医学工程学报,2008,29(4):392-10.1186。
- 55.
艾森哈伯B, Wildpaner M, Schultz CJ, Borner GH, Dupree P, Eisenhaber F:植物蛋白的糖基磷脂酰肌醇脂质锚定。拟南芥和水稻全序列和全基因组研究的敏感预测。植物科学学报,2003,23(3):366 - 366。10.1104 / pp.103.023580。
- 56.
范凯泽,马泽P:基于Kohonen自组织映射的GPI锚附着信号识别。生物信息学,2005,21:1846-1852。10.1093 /生物信息学/ bti299。
- 57.
Abascal F, Zardoya R, Posada D: ProtTest:蛋白质进化最佳拟合模型的选择。生物信息学,2005,21:2104-2105。10.1093 /生物信息学/ bti263。
- 58.
Guindon S, Gascuel O:通过最大似然估计大系统发育的简单,快速和准确的算法。中国生物医学工程学报,2003,29(3):344 - 344。10.1080 / 10635150390235520。
- 59.
魏凯,陈娟,王艳,陈艳,陈松,林艳,潘松,钟霞,谢东:玉米bzip编码基因的全基因组分析。DNA, 2012, 19: 463-476。10.1093 / dnares / dss026。
- 60.
魏凯,陈娟,陈勇,吴玲,谢东:ZmWRKY亚群多策略分析及ZmWRKY基因在病原应答中的功能探索。中国生物工程学报,2012,30(4):349 - 349。10.1039 / c2mb05483c。
- 61.
魏凯,潘s:玉米蛋白磷酸酶基因家族的鉴定与分子特性。BMC基因组学杂志,2014,15:773-10.1186/1471-2164-15-773。doi: 10.1186 / 1471-2164-15-773
- 62.
Krzywinski M, Schein J, Birol I, Connors J, Gascoyne R, Horsman D, Jones SJ, Marra MA: Circos:比较基因组学的信息美学。中国生物工程学报,2009,29(4):344 - 344。10.1101 / gr.092759.109。
- 63.
Librado P, Rozas J: DnaSP v5: DNA多态性数据综合分析软件。生物信息学,2009,25:1451-1452。10.1093 /生物信息学/ btp187。
- 64.
林奇M, Conery JS:重复基因的进化人口统计学。中华基因工程学报,2003,3:35-44。10.1023 /: 1022696612931。
- 65.
杨智,顾松,王旭,李伟,唐智,徐超:植物cpplike基因家族的分子进化:来自拟南芥和水稻比较基因组学的研究。中华药理学杂志,2008,29(4):366 - 366。10.1007 / s00239 - 008 - 9143 - z。
- 66.
Arnold K, Bordoli L, Kopp J, Schwede T: SWISS-MODEL工作空间:基于网络的蛋白质结构同源建模环境。生物信息学,2006,22:195-201。10.1093 /生物信息学/ bti770。
- 67.
Pettersen EF, Goddard TD, Huang CC, Couch GS, Greenblatt DM,孟EC, Ferrin TE: UCSF chimera -一个用于探索性研究和分析的可视化系统。计算化学,2004,25:1605-1612。10.1002 / jcc.20084。
- 68.
Lescot M, Dehais P, Thijs G, Marchal K, Moreau Y, Van de Peer Y, Rouze P, Rombauts S: PlantCARE,植物顺式作用调控元件数据库和启动子序列的计算机分析工具门户。中国生物医学工程学报,2002,30:325-327。10.1093 / nar / 30.1.325。
- 69.
魏克峰,陈娟,陈云峰,吴丽娟,谢晓东:玉米WRKY全转录因子家族的分子系统发育及表达分析。DNA, 2012, 19: 153-164。10.1093 / dnares / dsr048。
- 70.
魏坤,陈艳,林艳,潘艳:中国甲型H5N1流感病毒NS1基因的遗传动态分析。公共科学学报,2014,9:e101384-10.1371/journal. psci .0101384。
致谢
我们非常感谢将微阵列和RNA-seq数据提交到公共表达数据库的提供商,这些数据库可以自由应用。我们衷心感谢清华大学王艳梅博士对我们稿件的修改。我们也感谢陈彦辉、林依娜、潘思和刘晓尧在我们实验室进行的有益讨论。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
电子辅助材料
12870 _2014_281_moesm1_esm.pdf
附加文件1:表S1。:Maize genes encoding proteins with a Pfam domain PF00234 which belong to hybrid proline-rich proteins, alpha-amylase/trypsin inhibitors, prolamin storage proteins and 2S albumin storage proteins. (PDF 47 KB)
12870 _2014_281_moesm3_esm.pdf
附加文件3:表S3。:Designations used for nsLTP sequences in this report, and their corresponding database accession numbers. (PDF 55 KB)
12870 _2014_281_moesm4_esm.pdf
附加文件4:表S4。:Sorghum genes encoding proteins with a Pfam domain PF00234 which belong to hybrid proline-rich proteins, alpha-amylase/trypsin inhibitors, prolamin storage proteins and 2S albumin storage proteins. (PDF 44 KB)
12870 _2014_281_moesm6_esm.pdf
附加文件6:图S1。:Multiple sequence alignment of ZmLTPs. Amino acid sequences of the 8CMs were aligned using ClustalW implemented in MEGA5 software suite program and manually refined. The conserved cysteine residues are blue boxed. (PDF 1 MB)
12870 _2014_281_moesm8_esm.pdf
附加文件8:图S2。:Unrooted phylogenetic tree between maize, sorghum, rice and Arabidopsis nsLTP gene families. Different species are denoted with different colors (maize in black, sorghum in green, rice in blue and Arabidopsis in red). (PDF 57 KB)
12870 _2014_281_moesm9_esm.pdf
附加文件9:图S3。:的map of intron-exon arrangement of maize nsLTP genes. Introns and exons are drawn to scale with the full encoding regions of their respective. Exons are depicted as green boxes, introns as connecting thin lines and 8CM as red boxes. Non-translated regions, when supported by full-length cDNA sequences, are shown in blue boxes. (PDF 41 KB)
12870 _2014_281_moesm10_esm.pdf
附加文件10:图S4。:的map of intron-exon arrangement of Arabidopsis and rice nsLTP genes. Introns and exons are drawn to scale with the full encoding regions of their respective. Exons are depicted as green boxes, introns as connecting thin lines and 8CM as red boxes. Non-translated regions, when supported by full-length cDNA sequences, are shown in blue boxes. (PDF 69 KB)
12870 _2014_281_moesm12_esm.pdf
附加文件12:表S8。:Genes list corresponding to the GO enrichment analysis by the AgriGO program. (PDF 57 KB)
12870 _2014_281_moesm13_esm.pdf
附加文件13:表S9。:分析独联体在ZmLTP基因翻译起始密码子上游1kb序列中的-元件。(pdf 106kb)
12870 _2014_281_moesm14_esm.pdf
附加文件14:表S10。:Average log2 signal intensity values and CV values of 75 detected ZmLTP transcripts in sixty distinct tissues representing eleven major organ systems of inbred line B73 ordered by three clusters. (PDF 144 KB)
12870 _2014_281_moesm16_esm.pdf
附加文件16:图S5。:Normalized signal intensities of 41 probe sets representing 36 ZmLTP genes displayed for the microarray experiment, which was treated with黑粉菌属maydis.Log2比值值(由绿-红梯度表示)表示真菌感染中基因表达的折叠变化。(pdf 24kb)
12870 _2014_281_moesm18_esm.pdf
附加文件18:表S13。:List of FPKM values of 42 ZmLTP genes in maize reproductive (ovary) and vegetative tissue (leaf) under both drought and well-watered conditions. (PDF 63 KB)
12870 _2014_281_moesm19_esm.pdf
附加文件19:图S6。:Expression profiles of 38 ZmLTP genes in three root types [primary root (PR), seminal roots (SR) and crown roots (CR)] under salt stress. (PDF 20 KB)
12870 _2014_281_moesm21_esm.pdf
附加文件21:图S7。:Microarray-based expression profiles of 36 probe sets representing 34 ZmLTP genes under cold stress condition in two inbred maize lines, chilling-sensitive ETH-DL3 and chilling-tolerant ETH-DH7. (PDF 49 KB)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/4.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
魏凯,钟霞。玉米非特异性脂质转移蛋白。BMC植物生物学14日,281(2014)。https://doi.org/10.1186/s12870-014-0281-8
收到了:
接受:
发表:
关键字
- 基因本体论
- 盐胁迫
- 干旱胁迫
- 冷应激
- 的根