摘要
背景
亲环蛋白(的CYP)属于免疫亲家族,并具有肽脯氨酰CIS-TRANS.异构酶(PPI酶)活性。PPI酶催化CIS-和反式肽的肽 - 脯氨酰酰胺肽的旋转式互连,蛋白质折叠的速率限制步骤。研究表明了许多Ppiases在植物生物学中的重要性,但没有基因组分析CYP.对一种豆科植物进行了基因家族研究。
结果
在这里,我们进行了一个综合的数据库调查,并确定了62次CYP.基因,位于大豆基因组的18个不同染色体上(GmCYP1到目前为止GmCYP62),其中10个是多结构域蛋白,52个是单结构域蛋白。大多数预测的GmCYPs成对聚集在一起,反映了古老的基因组复制事件。基因结构分析显示,蛋白编码区以及5'和3'非翻译区都存在内含子,它们的大小、丰度和分布在基因家族中存在差异。表达式的分析GmCYP大豆组织中的基因显示它们的差异组织特异性表达模式。
结论
总体而言,我们已经确定62CYP.大豆基因组中的基因,最大CYP.已知的基因家族。这是第一次全基因组研究CYP.基因家族的豆类物种。扩张GmCYP基因大豆,而染色体上的分布格局强烈建议全基因组段和串联重复。
背景
亲环蛋白(的CYP)是生物体从古细菌中发现的蛋白质普遍存在和细菌对植物和动物[1],[2].由于最初被鉴定为免疫抑制药物环孢素A (CsA)的受体,CYPs属于具有肽基脯氨酸的免疫亲和蛋白家族顺/异构酶活性。多的CYP已在多种原核生物基因组研究中发现,但只有少数人在详细研究。的大肠杆菌基因组编码两个cyp,一个胞质形式(ecypp -18)和它的周质对等物(ecypp -20) [3.].在酵母酿酒酵母酿酒酵母有至少8周不同的CYP,CPR1到目前为止CPR8.[4].这些蛋白质对生长不是必需的,但对热应激后的生存至关重要[5].人类基因组编码16种独特的CYP,分为7大类,即人类CYP A (hcypa)、hcypb、hcypc、hcypd、hcype、hcyp40和hcypnk [6].hcypa与CsA结合,与钙调磷酸酶形成三元复合物。csa - hcypa与钙调磷酸酶结合抑制钙调磷酸酶的活性,从而导致一系列活动导致t细胞失活[7].
与人类CYP相比,很少有关于植物CYP的含量。第一植物CYPS.同时从番茄(Lycopersicon esculentum),玉米(玉米),和油菜(芸苔栗鸟)[8].近年来,随着全基因组测序技术的发展,植物cyp的鉴定和表征取得了很大进展。然而,与其他生物相比,植物的总数量CYPS.在数据库中仍然很小,这表明许多植物CYPS.仍然有待鉴定[9].到目前为止,拟南芥和米饭(栽培稻)是据报道cyp数量最多的两种植物,为35个AtCYPs[10.],[11.]和28OsCYPs[10.],[12.]中。其中标识AtCYPs,只有15个在分子水平上有特征[11.],[13.] - [21.].它们编码的蛋白质存在于细胞质中[17.],[19.],[20.,内质网(ER) [18.],[21.],叶绿体[15.],[16.]和核[13.].的表达的增加ROC1,一个AtCYP,响应于光与植物光敏素和隐花色素[相关联19.],[22.].ROC1突变体显示早期开花表型[22.],虽然功能突变突变Roc1.减少茎伸长和增加枝条分支[23.].相反,功能缺失突变AtCYP40幼叶数减少,花序形态和开花时间无明显变化拟南芥有缺陷的植物AtCYP20-3被发现是过敏的由高亮度,高盐水平,和渗透压休克[创建氧化应激条件24.].除了在各种发育过程中发挥作用的AtCYPs, AtCYP59,一个具有RNA识别基元(RRM)的多域CYP,通过结合RNA聚合酶II的c端结构域调控转录和mrna前加工[13.].集体,这些结果表明了角色拟南芥的CYP在不同细胞途径,从而需要进一步的工作来探索与每个的CYP的相关联的功能。
与这一点相比拟南芥的CYP,很少工作已经对水稻的CYP完成。大多数对后者的研究表明:在不同类型的应力自己的角色。OsCYP2据报道,在不同的非生物压力反应中具有作用[25.].表达OsCYP2在盐胁迫下表达上调,在水稻中过表达增强了对盐胁迫的耐受性。同样,过度的拟南芥以及类囊体定位的烟草OsCYP20-2增加的耐受性对渗透胁迫,和极高的光条件[26.].其他几个的表达水平OsCYPs增加了非生物胁迫,如干燥和盐胁迫[10.],[12.,表明OsCYPs在应力条件下起着关键作用。
大豆(甘氨酸最大[L.] MERR)是属于豆科植物Papilionoideae富含蛋白质、油脂和异黄酮等植物天然产物。大豆基因组包含56,044个蛋白质编码位点,分布在20条不同的染色体上。大豆大约在5900万年前和1300万年前经历了两次全基因组复制事件,结果是75%的基因有多个副本[27.].直到现在,人们对大豆CYPs所知不多,除了那一小部分CYP.基因序列已被存放在公共数据库中。我们在座的大豆全基因组鉴定CYP.S,它们的系统发育分析,染色体分布,结构和表现的分析.结果表明,大豆中含有62个CYP,是目前已知的最大的CYP家族。此外,该研究还描述了基因组范围内的节段复制和串联复制GmCYP基因家族。
结果与讨论
大豆基因组包含62个假定的GmCYPs
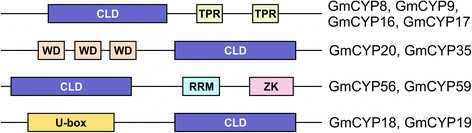
要识别的所有成员CYP.大豆的基因家族,一个爆破的大豆基因组数据库G. Max.Wm82.a2.v1(http://phytozome.jgi.doe.gov/pz/portal.html#!search?show=BLAST&method=Org_Gmax)用先前识别大豆的核苷酸序列进行CYP.的cDNA(GenBank登录号:AF456323)作为查询。该搜索确定了11个独特的CYP.基因。每个11CYP.基因被单独用作BLAST检索大豆基因组数据库的一个查询序列。重复这个过程,直到没有新CYP.基因被发现。大豆共62种的CYP,位于18组不同的染色体,鉴定并命名GmCYP1到目前为止GmCYP62(表1).在62GmCYPs,52编码具有单环蛋白样结构域(CLD)一种蛋白质,其负责顺/肽基脯氨酰肽键的异构化。其余的10个GmCYPs包含的CLD和额外结构域。如图所示1,GmCYP8,GmCYP9,GmCYP16和GmCYP17各自在C-末端包含在两个三十四肽重复序列(的TPR)。所述TPR基序是在本质简并和由一个34的氨基通常布置成串联排列[酸重复单元28.].这种含有蛋白质的TPR基序介导蛋白质-蛋白质相互作用,并经常帮助多蛋白质复合物的组装。AtCYP40 (AGI: At2g15790)拟南芥GmCYP8,GmCYP9,GmCYP16,和GmCYP17的直向同源物,含有3点的TPR并且涉及微小RNA介导的基因调控[29.].丧失功能的突变AtCYP40展示了一种早熟的相变,减少了少年叶,但没有开花时间的改变[20.].此外,AtCYP40与细胞质Hsp90蛋白的相互作用需要AtCYP40 TPR结构域的保守氨基酸。这种相互作用对AtCYP40的功能至关重要在Planta.[29.建议在MicroRNA介导的基因调节中对TPR结构域的关键作用。在这里,我们为GMCYP8,GMCYP9,GMCYP16和/或GMCYP17中的TPR域推测了可能类似的功能。
GmCYP20和GmCYP35含有N-末端(3色氨酸天冬氨酸(WD)重复图1).WD重复含蛋白参与了多种细胞功能,提供结合位点的两个或更多的蛋白质,或促进短暂的相互作用与其它蛋白质[30.],[31.].的拟南芥CYP, AtCYP71 (AGI:At3g44600),包含2个WD重复[11.]和功能在染色质重塑[14.].AtCYP71与GmCYP20(87%)和GmCYP35(83%)序列高度一致,提示这两个GmCYPs可能在大豆中发挥相似的作用。
序列分析确定了两个大豆的CYP,GmCYP56和GmCYP59,具有RNA识别基序(RRM)和锌指关节(ZK)在C-末端与CLD在N末端端(图沿1).RRM是90个氨基酸的小RNA结合主题,并在各种各样的生物中保守[32.].AtCYP59(AGI:At1g53720)中,拟南芥GmCYP56(80%)和GmCYP59(66%)的直系同源物(附加文件1)[11.],包含一个RRM基序,并且是转录调节[13.与未加工的mRNA的保守序列相互作用,导致PPIase活性的抑制在体外[33.].基于AtCYP59在转录调控中的功能关联,我们推测多结构域大豆CYPs GmCYP56和GmCYP59可能在大豆的转录调控中发挥作用通过他们RRMS。
最后,GmCYP18和GmCYP19在蛋白的n端含有一个U-box。U-box结构域在一些泛素连接酶中高度保守,并有望成为泛素化机制的一部分。哺乳动物CYC4和拟南芥ATCYP65是包含CYP的U形盒,其中CYP域预测有伴侣函数[11.],[34.].
在62GmCYPs,可以确认13含有叶绿体转运肽,13包含信号肽,5含有线粒体靶向肽,10含有核定位信号,并且剩余的21是胞质(表1).不像拟南芥和大米的CYP [10.],预计没有大豆Cyps局部地定位于ER或GOLGI或血浆膜。仅预测线粒体内膜或血浆膜中的定位预测一个分泌gmcyp39。搜索ER保留信号未在任何GMCYP中定位KDEL或HDEL。我们也搜索了CYP.基因包含表示73178个TC序列1354268分的EST DFCI大豆基因指数(http://compbio.dfci.harvard.edu/tgi/).对这个数据库的筛选证实了62人中有15人GmCYP我们鉴定的基因由99-100%的身份代表,100%覆盖率暗示,这意味着至少25%的覆盖率GmCYPs在正常生长和发育过程中,或在应激反应中,在各种大豆组织中转录。此外,33GmCYPs在大豆EST数据库中使用TC序列显示大于95%的序列同一性,但查询覆盖范围小于100%。较低的序列同一性可能是由于两种数据库之间的品种特异性序列差异,全基因组序列仅来自品种威廉姆斯82 [27.],和包括从几个不同的大豆品种的cDNA文库EST数据的DFCI大豆基因索引,转录的数目GmCYPs大豆可以预计将超过15个全部大豆的列表CYP.基因家族成员及其详细信息详见附加文件1.
染色体的分布和大豆的系统发育分析CYP.基因
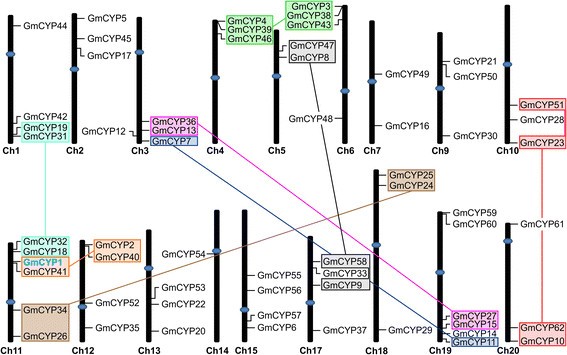
来确定基因组的组织和分布GmCYPs在大豆的不同染色体上,构建了一种染色体图。结果表明62GmCYPs位于18个不同的染色体上。如图所示2,每个染色体的基因密度不均匀。11号染色体和19包含最,并显示相对密集发生CYP.基因(6个),而只有一个CYP.(GmCYP54)存在于14染色体上。没有CYPS.在染色体上发现了8或16次。大多数GmCYPs分别定位对染色体末端,只有GmCYP52,GmCYP49和GmCYP54在着丝粒附近发现(图2),表明染色体重排间的可能性,基因组复制后,不同大豆染色体之间。
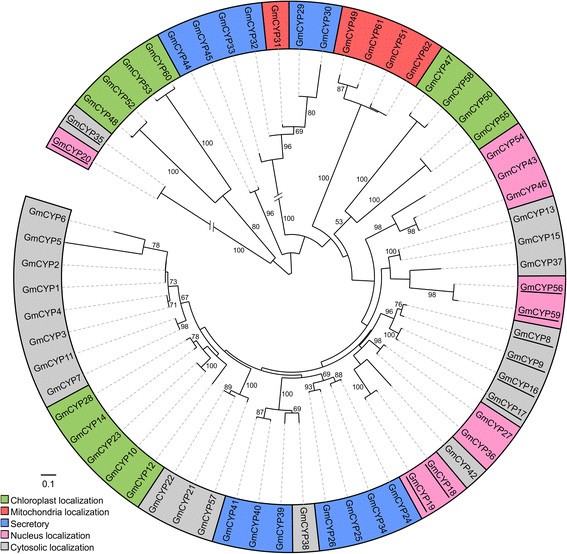
为了探究大豆CYPs之间的进化关系,利用预测的氨基酸序列进行系统发育分析(图)3.).正如在大豆中观察到的许多其他基因,大多数预测的GmCYPs成对聚集在一起,反映了古老的基因组复制事件[27.],[35.].这些事件导致其经历洗牌和重新排列每个基因的两个副本,创建新的多元化的潜力。有重复基因的四种可能的命运[36.].首先,可以在进化过程中删除基因的一种拷贝,导致功能冗余丧失。其次,可以保留基因的两种拷贝并分享祖先功能,但逐渐发展部分不同的功能(副官能化)。第三,在进化过程中,基因的一份副本可以在进化过程中获得新功能(新官能化)。最后,可以存在子和新功能化之间的中间阶段。发生这些结果中的哪一个取决于特定基因在植物生长和发育中的作用。只保留了与正常植物生长和发育的关键功能相关的那些基因,而其他基因则可能会丢失。大量的CYP.S出现在大豆基因组中从而可能反映重复的组合中的重要作用GmCYPs在大豆正常生长发育过程中,以及对环境刺激的反应中。
62个GmCYPs的,54被聚集在系统发生树对(27对)。其余8个GmCYPs分叉的另一对GmCYPs的端子分支。此分析进一步揭示了在多域GmCYPs聚集在一起。
我们还试图将动态血液发育树中的Gmcyps聚类与预测的亚细胞定位相关联。有趣的是,预测的Gmcyps预计将被靶向与单独的疏水板一起分组的相同亚细胞室。例如,用叶绿体过渡肽(GMCOP10,GMCOP23,GMCOP14,GMCOP14,GMCOP28和GMCOP12)的GMCYP在树上形成了不同的疏水性。另外4个叶绿体 - 定位的Gmcyps(Gmcyp48,Gmcyp52,Gmcyp53和Gmcyp60)也形成了一个不同的疏水性,但在树上的不同位置。类似地,具有核定位信号的GMCYP(GMCYP27,GMCYP36和GMCYP54,GMCYP43,GMCYP46)也在树上形成了单独的片状(图3.).预测定位于线粒体或分泌的GmCYPs也观察到类似的基因聚类模式。
通过比较GmCYPs在染色体图谱上(图2),并在系统发生树(图3.),观察到一个有趣的分组模式。如果是GmCYPs在染色体上局部地定位,他们的副病也被发现在不同的染色体上。例如,gmcyp4,gmcyp39,和GmCYP46聚集在第4染色体的次端粒区域,与GmCYP3,GmCYP38,和GmCYP43分别,其在6号染色体的亚端粒区聚集在一起(图2).同样的,gmcyp36,gmcyp13,和GmCYP7(染色体3)配对GmCYP27 GmCYP15,和gmcyp11,分别从19号染色体,并gmcyp18和GmCYP32(染色体11)配对gmcyp19和GmCYP31分别(染色体1),而GmCYP1和GmCYP41(染色体11)配对GmCYP2和GmCYP40(12号染色体)。此外,GmCYP34和GmCYP26,从11号染色体,配对了GmCYP24和GmCYP25分别来自染色体18.这些发现提供了含有其染色体区域的细分重复的强有力证据GmCYPs如已被证实在进化生成其它的基因家族的成员[中发挥重要作用37.],[38.].
基因的结构GmCYPs
外显子-内含子结构分析GmCYP基因显示出几种变化(图4).六个GmCYP基因(GmCYP1——GmCYP4,GmCYP6和GmCYP7在其开放阅读框(ORF))不含有内含子。内含子的数量从1中的其他的ORF的变化,以13GmCYP基因。的GmCYP5,GmCYP47,GmCYP50,GmCYP55和GmCYP58ORF中含有一个内含子,而内含子数量最多的是ORF中GmCYP56.内含子的大小也不同之间变化相当大GmCYP基因家族成员的大小从39 bp (GmCYP5)至9359碱基对(GmCYP56)。其他一些基因,比如GmCYP22, GmCYP30, GmCYP34, GmCYP39-GmCYP42, GmCYP45, GmCYP49, GmCYP51- GmCYP53, GmCYP55- GmCYP57和GmCYP59在其ORF中含有大于4.0 kB的内含子。提出,基因组大小可以与内含子大小相关,并且在基因内发生基因组大小演化的一些元素[39.].但是,在Gossypium sps。内含子和基因组大小进化不耦合[40].在低重组区域,较长的内含子具有选择性优势,因为它们可以改善重组,并可能抵消对缺失的突变偏见[41.].对不同王国(动物,植物和真菌)之间的内含子位置的大规模比较分析确定了大量可能是祖先的职位[42.].内含子大小和位置的分析GmCYP家庭没有显示任何特定模式。然而,在大多数情况下,外显子内数在系统发育树中聚集在一起的基因中相似(图3.),例如,GmCYP25和GmCYP26或者GmCYP47和GmCYP58或者GmCYP20和GmCYP35.5' 和3' 非翻译区(UTR),该边界蛋白质编码序列是真核基因的重要的结构和调控元件[43.]并且也含有大量的内含子[44.].出62GmCYPs,12中所含的5'UTR区的单一内含子,而其余GmCYPs包括内含子5'UTR的。两个的3'UTRGmCYPs,GmCYP16和GmCYP50,由单一的内含子而被中断gmcyp17和GmCYP44分别含有2个和5个内含子。每个外显子和内含子的数量GmCYP基因显示在附加文件中2.
外显子 - 内含子结构的示意图,和的剪接变体GmCYPs.Exon-intron结构GmCYPs从Phyotozome数据库进行了汇编(http://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Gmax).GmCYP具有预测的替代转录物如下所示的相应基因。绿色盒子,黑匣子和线条分别表示外显子,UTR和内含子。左右转录方向显示“+”股线,而左侧显示“ - ”股线,相对于基因组序列的注释。基因结构图像被绘制到尺度之外GmCYP50,GmCYP56,GmCYP59,其中图分别减少到0.5倍,0.35倍和0.5倍。
表达式的分析GmCYP基因
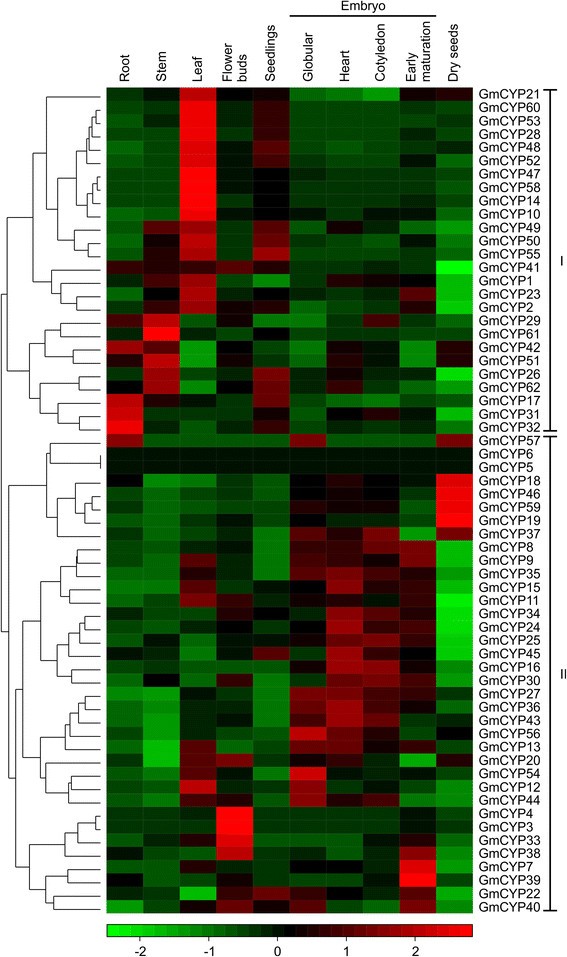
确定的表达模式GmCYP基因,我们使用公开可用的全基因组转录谱描述大豆组织的数据作为一种资源(http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE29163).该数据集包含来自种子发育的几个阶段的大豆种子(球形,心脏,子叶,早期成熟,干)和生殖(花芽)和植物(叶,根,茎,幼苗)组织的rnaseq。如图所示5,大部分GmCYP基因显示出不同的组织特异性表达模式。在62中GmCYP基因,26营养组织中表达,而在34花芽和种子发育的不同阶段中表达。二GmCYPs,GmCYP5和GmCYP6,不含任何列入研究大豆组织的阅读顺序。此外,有在DFCI基因索引数据库没有EST或TC序列与完美匹配GmCYP5和GmCYP6(附加文件1).这些证据表明GmCYP5和GmCYP6是假基因,或在特定条件下或在特定发育阶段表达。基因表达数据显示,大多数GmCYPs(41%)具有最高转录累加级叶组织中均有表达。此外,有趣的是,预测叶绿体本地化GmCYPs叶组织中表达,这表明在光合作用其可能发挥的作用。几种表达GmCYP在发育过程中种子组织的基因表明这些基因在种子发育中的重要作用。
大豆的表达分析CYP.基因。大豆不同组织和发育阶段的转录组数据来自美国国家生物技术信息中心(http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE29163)来生成热图。热图下方的色标表示表达值,绿色表示低转录本丰度,红色表示高转录本丰度。聚类的GmCYPsin.(i)植物组织或(ii)显示了生殖组织或种子组织。
结论
总之,我们已经完成大豆的综合序列分析CYP.基因(GmCYPs),并提供有关的详细资料。具体来说,我们的结果表明,大豆基因组包含62CYP.基因,最大的CYP.基因家族中的任何生物的最新标识。预测图案,亚细胞定位和与来自其他生物的其他所识别的CYP其序列同源性的存在提供了深入了解它们的推定的功能。本研究的结果表明的膨胀过程中全基因组节段和串联重复GmCYP基因家族。
方法
数据库搜索CYP.基因大豆
要确定所有的CYPS.存在于大豆基因组中,核苷酸序列GmCYP1(Genbank:AF456323)用于爆炸[45.对新大豆基因组数据库]查询(Wm82.a2.v1)(http://phytozome.jgi.doe.gov/pz/portal.html.)[46.].新发现的序列随后用作查询找到其他类似的少GmCYPs。所有的染色体位置GmCYPs从大豆基因组数据库中获得染色体图谱。使用ProtParam软件计算每个GmCYP的分子量[47.](http://web.expasy.org/protparam/).TargetP1 [48.](http://www.cbs.dtu.dk/services/TargetP/)及WoLF-PSORT [49.](http://wolfpsort.org/)被用来确定预测的蛋白质序列的推定的亚细胞定位,并从大豆基因组数据库获得域信息[27.].识别转录GmCYPS的编码序列GmCYP被用作一个查询来针对BLAST大豆基因指数(http://compbio.dfci.harvard.edu/tgi/).大豆基因指数数据库中的暂定折曲(Tc)序列与相应的相应对齐GmCYP序列识别的百分比同一性和覆盖范围。同样,以找到同源基因GmCYP拟南芥,将GMCYP的氨基酸序列用作爆炸的疑问拟南芥蛋白质数据库(http://www.arabidopsis.org/)[50.].
多序列比对和系统发育分析
为了研究GmCYP蛋白之间的系统发育关系及其分子进化,建立了系统发育树。通过Clustal X对所有GmCYP蛋白推导的氨基酸序列进行多序列比对,并将比对结果导入MEGA5.1中,形成系统发育树[51.].用1000mL引导重复使用邻接法。树被导出到互动生命之树(http://itol.embl.de),用于注释和处理[52.].
大豆中的表达分析CYP.基因
确定表达式模式CYP.基因大豆组织中,公开可用的转录组数据(http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE29163)作为一个主要来源。来自十个不同的大豆组织的转录的Illumina测序是从NCBI数据库下载(http://www.ncbi.nlm.nih.gov/)与登录号SRX062325-SRX062334。数据集的归一化之后,每个基因的值,是通过减去每个基因的平均归一化的值为中心,并通过由该基因的下列Eisen等人的标准偏差除以中心值进行缩放。[53.].热线图GmCYP利用热图在R中生成。从gplot CRAN库中获得的函数(http://CRAN.R-project.org/package=gplots).
可用性支持数据
系统存储库中的系统发育数据(用于生成它们的树和数据)已存放在TreeBase存储库中,并在URL下提供http://purl.org/phylo/treebase/phylows/study/tb2:s16455.
附加文件
参考文献
- 1。
维持其生物学功能的蛋白质的序列和氨基酸组成的变化:对蛋白质的亲环蛋白家族的分析。中国生物化学学报,1998,31(2):441 - 446。10.1006 / abbi.1999.1434。
- 2。
Maruyama T, Suzuki R, Furutani M: Archaeal peptidyl prolyl顺反异构酶(PPIases)更新2004。生物工程学报,2004,29(4):457 - 461。10.2741/1361。
- 3.
Hayano T,Takahashi N,Kato S,Maki N,Suzuki M:两种不同形式的肽基脯氨酰-CIS-反式异构酶在大肠杆菌细胞的周质和细胞质隔室中分别表达。生物化学。1991,30(12):3041-3048。10.1021 / bi00226a009。
- 4.
研究了酵母中脯氨酸异构酶的研究进展。生物工程学报,2004,29(4):457 - 461。10.2741/1405。
- 5。
赛克斯K,见Gething MJ,SAMBROOK Y:脯氨酸热休克期间异构酶功能。美国国家科学院学报。1993年,90(12):5853-5857。10.1073 / pnas.90.12.5853。
- 6。
肽基丙醇顺/反式异构酶(免疫蛋白):生物多样性-靶点-功能。中华医学杂志。2003,3(12):1315-1347。10.2174 / 1568026033451862。
- 7。
刘俊,农民JD,泳道WS,弗里德曼Ĵ,威斯曼I,施雷伯SL:钙调磷酸酶是亲环环孢菌素A和FKBP-FK506复合物的一个共同的目标。细胞。1991年,66(4):807-815。10.1016 / 0092-8674(91)90124-H。
- 8。
高等植物胞质嗜环素/肽丙基顺反异构酶的结构和表达及番茄活性嗜环素在大肠杆菌中的生产。美国国家科学院学报。[j] .地理科学进展,2019,38(4):593 - 598。10.1073 / pnas.87.24.9519。
- 9。
Opiyo SO, Moriyama EN:挖掘拟南芥和水稻基因组中的亲环蛋白家族。中国生物医学工程学报,2009,27(3):337 - 341。10.1504 / IJBRA.2009.026421。
- 10。
水稻和拟南芥嗜环素基因家族的全基因组分析及其与酵母的比较。植物信号学报。2012,7(12):1653-1666。10.4161 / psb.22306。
- 11.
拟南芥嗜环素基因家族。植物营养与肥料学报。2004,29(4):429 - 434。10.1104 / pp.103.022160。
- 12.
安JC,金DW,你YN,锡MS,公园JM,黄H,金BG,栾S,公园HS,卓HS:稻的分类(水稻粳稻日本晴)免疫亲和素(FKBPs,的CYP)和表达模式水分胁迫下。BMC植物BIOL。2010年,10:253-10.1186 / 1471-2229-10-253。
- 13。
Gullerova男,巴塔A,Lorkovic ZJ:AtCyp59是来自拟南芥多畴亲环与SR蛋白质和RNA的C末端结构域相互作用聚合酶II。RNA。2006年,12(4):631-643。10.1261 / rna.2226106。
- 14。
李H,栾S:在多个重塑染色质与处理CAF-1和LHP1的亲环AtCYP71相互作用和功能。分子植物。2011年,4(4):748-758。10.1093 / MP / ssr036。
- 15.
Schubert M,Petersson Ua,Haas BJ,Funk C,Schroder WP,Kieselbach T:拟南芥叶绿体腔的蛋白质组地图。J Biol Chem。2002,277(10):8354-8365。10.1074 / JBC.M108575200。
- 16。
Lippuner V,周杰伦IT,斯科特SV,艾丁格WF,Theg SM,加塞CS:克隆与叶绿体和拟南芥环素的胞质形式的表征。J Biol Chem。1994年,269(11):7863-7868。
- 17。
海曼GT,Miernyk JA:核苷酸和推导的氨基酸来自拟南芥一个肽基 - 脯氨酰顺反异构酶的氨基酸序列。Biochim Biophys Acta。1994,1219(2):536-538。10.1016 / 0167-4781(94)90083-3。
- 18。
Grebe M, Gadea J, Steinmann T, Kientz M, Rahfeld JU, Salchert K, Koncz C, Jurgens G:拟南芥GNOM蛋白的保守结构域介导了亚基相互作用和亲环蛋白5的结合。植物学报。2000,12(3):343-356。10.1105 / tpc.12.3.343。
- 19。
Chou It,Gasser Cs:拟南芥拟南芥基因系列的表征和已知环疗蛋白的系统发育分析。植物mol biol。1997,35(6):873-892。10.1023 /答:1005930024796。
- 20.
杨志强,杨志强,杨志强:嗜环素40对拟南芥营养阶段变化的调控。中国科学:地球科学。2001,29(5):591 - 598。10.1126 / science.1057144。
- 21。
杰克逊K,Soll D:新拟南芥细胞苷中的突变破坏了与蛋白质磷酸酶2a的相互作用。Mol Gen Genet。1999,262(4-5):830-838。10.1007 / S004380051147。
- 22。
Trupkin SA, Mora-Garcia S, Casal JJ:嗜环蛋白ROC1将光敏色素和隐色素与油菜素内酯敏感性联系起来。植物学报,2012,71(5):712-723。10.1111 / j.1365 - 313 x.2012.05013.x。
- 23。
马晓霞,宋磊,杨永强,刘东东:拟南芥ROC1基因功能获得突变对植物结构的影响。植物营养学报。2013,19(3):751-762。10.1111 / nph.12056。
- 24。
格斯-索利斯JR,他Z,利马A,亭Ĵ,布坎南BB,栾S:亲环链接氧化还原和光信号成半胱氨酸的生物合成和在叶绿体应激反应。美国国家科学院学报。2008年,105(42):16386-16391。10.1073 / pnas.0808204105。
- 25。
阮SL,马协,王SH,傅YP鑫Y,刘WZ,王楼,堂JX,王SZ,陈灏珠:OsCYP2的蛋白质组鉴定,水稻亲环素,在赋予水稻耐盐性(水稻)苗时表达。BMC植物BIOL。2011年,11:34-10.1186 / 1471-2229-11-34。
- 26。
Kim SK, You YN, Park JC, jong Y, Kim BG, Ahn JC, Cho HS:水稻类囊体腔内亲环素OsCYP20-2对烟草和拟南芥的环境胁迫耐受性增强。植物学报,2012,31(2):417-426。10.1007 / s00299 - 011 - 1176 - x。
- 27。
污物J,大炮某人,马Schlueter J, J, Mitros T,纳尔逊W,海顿耐DL,歌Q, JJ,泰伦程J,徐D, Hellsten U, GD,于Y,樱井T, Umezawa T, Bhattacharyya可,Sandhu D, Valliyodan B,林奎斯特E,皮托,格兰特D,蜀年代,古德斯坦D,巴里·K Futrell-Griggs M B,阿伯纳西Du J,田Z,朱L,等:古多倍体大豆的基因组序列。自然科学学报。2010,463(7278):178-183。10.1038 / nature08670。
- 28。
布拉奇GL,Lassle玛:十四肽重复:结构基序介导的蛋白质 - 蛋白质相互作用。生物。1999年,21(11):932-939。10.1002 /(SICI)1521年至1878年(199911)21:11 <932 :: AID-BIES5> 3.0.CO; 2-N。
- 29。
Earley KW, Poethig RS:拟南芥中斜视蛋白与Hsp90蛋白的结合是斜视蛋白功能的必需条件。中国生物医学工程学报。2011,31(4):591 - 598。10.1074 / jbc.M111.290130。
- 30.
Van Nocker S,Ludwig P:拟南芥的WD重复蛋白超家族:结构和功能的保护和分歧。BMC基因组学。2003,4(1):50-10.1186 / 1471-2164-4-50。
- 31.
Stirnimann CU,Petsalaki E,罗素RB,穆勒CW:WD40蛋白质推进蜂窝网络。趋势Biochem SCI。2010年,35(10):565-574。10.1016 / j.tibs.2010.04.003。
- 32.
Bandziulis RJ,Swanson的MS,Dreyfuss的G:RNA结合蛋白作为发育调节。基因开发。1989年,3(4):431-437。10.1101 / gad.3.4.431。
- 33。
Bannikova O, Zywicki M, Marquez Y, Skrahina T, Kalyna M, Barta A:核多域嗜环蛋白atCyp59 RNA靶点的鉴定及其对PPIase活性的影响。中国生物医学工程学报,2013,41(3):1783-1796。10.1093 / nar / gks1252。
- 34。
CYR DM,霍菲尔德Ĵ,帕特森C:蛋白质量控制:包含U盒-E3泛素连接酶加入折。趋势Biochem SCI。2002年,27(7):368-375。10.1016 / S0968-0004(02)02125-4。
- 35。
李X,Dhaubhadel S:大豆14-3-3基因家族:鉴定和分子表征。Planta。2011,233(3):569-582。10.1007 / s00425-010-1315-6。
- 36。
Charon C, Bruggeman Q, Thareau V, Henry Y:绿色谱系中的基因复制:TEL基因的案例。实验机器人。2012,63(14):5061-5077。10.1093 / jxb / ers181。
- 37。
佳能S,米特拉A,鲍姆加滕A,年轻的N,五月G:在拟南芥大基因家族的进化段和串联基因重复的角色。BMC植物BIOL。2004年,4(1):10-10.1186 / 1471-2229-4-10。
- 38。
穆尔RC,Purugganan介绍MD:植物重复基因的进化动力学。CurrOp植物BIOL。2005年,8(2):122-128。10.1016 / j.pbi.2004.12.001。
- 39。
河豚(河豚)与人类的同源性保护和基因组压缩。酵母。2000,1(1):22-36。10.1002 / (SICI) 1097 - 0061 (200004): 1 < 22:: AID-YEA5 > 3.0.CO; 2 s。
- 40。
温德尔JF,Cronn RC,阿尔瓦雷斯我,刘B,小RL,Senchina DS:内含大小和在植物基因组大小。mol Biol Evol。2002年,19(12):2346至2352年。10.1093 / oxfordjournals.molbev.a004062。
- 41。
遗传重组:内含子大小与自然选择。自然科学学报。1999,31(4):591 - 598。10.1038/43827。
- 42.
Fedorov A,美国AF, Gilbert W:动物、植物和真菌基因内含子位置的大规模比较。美国国家科学院学报。[j] .地理科学进展,2019,38(4):457 - 461。10.1073 / pnas.242624899。
- 43.
Wilkie GS,Dickson Ks,Gray NK:通过5'-和3'-UTR结合因子调节mRNA翻译。趋势Biochem SCI。2003,28(4):182-188。10.1016 / s0968-0004(03)00051-3。
- 44.
Pesole G, Mignone F, Gissi C, Grillo G, Licciulli F, Liuni S:真核生物mRNA非翻译区的结构和功能特征。基因工程学报。2001,27(2):73-81。10.1016 / s0378 - 1119(01) 00674 - 6。
- 45。
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。中华生物医学杂志,1996,15(4):427 - 434。10.1016 / s0022 - 2836(05) 80360 - 2。
- 46。
Goodstein DM, Shu S, Howson R, Neupane R, Hayes RD, Fazo J, Mitros T, Dirks W, Hellsten U, Putnam N, Rokhsar DS: Phytozome:绿色植物基因组学的比较平台。核酸学报2012,40(1):1 - 4。10.1093 / nar / gkr944。
- 47。
Gasteiger E, Hoogland C, Gattiker A, Duvaud S, Wilkins M, Appel R, Bairoch A: ExPASy服务器上的蛋白质鉴定和分析工具。蛋白质组学协议手册。编辑:Walker JM。Humana出版社,新泽西州托托瓦;2005:571 - 607。10.1385 / 1 - 59259 - 890 - 0:571。
- 48。
Emanuelsson O, Brunak S, von Heijne G, Nielsen H:使用TargetP, SignalP和相关工具定位细胞中的蛋白。Nat协议。2007,2(4):953-971。10.1038 / nprot.2007.131。
- 49。
Horton P,Park Kj,Obayashi T,Fujita N,Harada H,Adams-Collier CJ,Nakai K:WOLF Psort:蛋白质定位预测指标。核酸RES。2007,35(Web服务器问题):W585-W587。10.1093 / nar / gkm259。
- 50.
Swarbreck D, Wilks C, Lamesch P, Berardini TZ, garia - hernandez M, Foerster H, Li D, Meyer T, Muller R, Ploetz L, Radenbaugh A, Singh S, Swing V, Tissier C, Zhang P, Huala E:拟南芥信息资源(TAIR):基因结构和功能注释。核酸学报2008,36(增刊1):D1009-D1014。
- 51。
田ura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S: MEGA5:利用最大似然、进化距离和最大简约法的分子进化遗传学分析。中国生物医学工程学报,2011,28(10):2731-2739。10.1093 / molbev / msr121。
- 52。
LeetUnic I,Bork P:生活中的互动树v2:在线注释和系统发育树的展示变得简单。核酸RES。2011,39(Web服务器问题):W475-W478。10.1093 / nar / gkr201。
- 53。
艾森MB,斯派曼PT,布朗PO,博茨泰因d:群集分析和全基因组表达模式的显示。美国国家科学院学报。1998年,95(25):14863-14868。10.1073 / pnas.95.25.14863。
致谢
作者感谢凯瑟琳·多宾森博士对手稿的批判性审查和有帮助的评论,凌辰的技术援助,以及亚历克斯·莫尔纳的平面设计。这项研究由加拿大自然科学和工程研究委员会的发现基金和农业和农业食品加拿大的作物基因组计划资助SD。
作者信息
从属关系
相应的作者
附加信息
相互竞争的利益
作者宣称,他们没有竞争的利益。
作者的贡献
人力资源管理设计并进行了实验,分析数据并撰写手稿。PC进行RNA测序数据的表达分析,和SD构思和设计的实验中,分析数据并写在原稿的最后草案。所有作者均阅读并批准最终手稿。
电子补充材料
大豆
附加文件1:cyclophilin.基因家族。(DOCX 36 KB)
外显子/内含子和剪接的变种数在
附加文件2:GmCYP基因。(DOCX 20 KB)
作者的原始提交的图像文件
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。
本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。
如欲查阅本许可证副本,请浏览https://creativecommons.org/licenses/by/4.0/.
创作共用及公共领域专用豁免书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
Mainali,H.R.,Chapman,P.&Dhaubhadel,S.基因组广泛分析还有大豆基因家族(甘氨酸最大).BMC植物BIOL.14,282(2014)。https://doi.org/10.1186/s12870-014-0282-7
收到了:
接受:
发表:
关键字
- 大豆基因组
- 大豆组织
- 进化基因组大小
- 基因索引数据库
- 大豆基因组数据库