摘要
背景
在昆虫与植物的共同进化过程中,花对称的建立是被子植物发育的重要一步,因为它促进了与昆虫传粉者的相互作用,从而可能促进被子植物的多样化。在金鱼草majus,TCP转录因子CYCLOIDEA是驱动花对称形成的分子关键调节因子。虽然大多数芸苔科形成一个多对称花冠,六个属发展单对称花与两个花瓣对大小不等。在单对称十字架中Iberis amara,形成不同花瓣对的同时,表达了较强的赛克同族体IaTCP1在小的,正面花瓣上。
结果
在本研究中,RNA-Seq用于重建非模式种的花瓣转录组Iberis amara.大约9 Gb的序列数据被生成,处理和重新组装成18,139个可能IberisUnigenes中有15983个同源性较高拟南芥蛋白质。转录组为调控花瓣发育后期的分子机制提供了详细的见解。此外,它还被用作支架,检测小的,正面的和大的,背面的花瓣之间差异表达的基因,以了解驱动不平等花瓣生长的分子机制。在花瓣正面表达的基因远多于背面表达的基因,这意味着IaTCP1激活的基因比抑制的要多。在近轴瓣上调的基因中,显著上调的基因比例与细胞壁修饰和细胞-细胞信号转导过程有关。此外,利用微阵列检测和比较转基因TCP靶基因的定量差异拟南芥植物异位表达不同TCP转录因子。
结论
与细胞壁修饰和信号传递有关的基因的增加表明,不平等的花瓣生长是通过在小的、正面的花瓣中细胞增殖阶段的较早停止来实现的,随后是细胞扩张的开始。这个过程,就形成了单对称的花冠Iberis amara,很可能是由增强的活动IaTCP1在花瓣正面。
背景
在植物与昆虫的共同进化过程中,花对称的发展是重要的一步,它促进被子植物的物种形成,以适应昆虫传粉者的视觉感官[1].因此,单对称性很可能是形态上的关键创新,并在不同被子植物谱系中独立进化[2]、[3.].
近20年前的开创性研究发现TCP转录因子CYCLOIDEA(本体)作为单对称性发育的关键调控分子金鱼草属植物[4].赛克以及它的para - logueDICHOTOMA表现在显影的正面金鱼草属植物花,它们引导获得第二和第三轮器官的正面特征[4]、[5].赛克属于CYC2TCP转录因子家族分支[6]在迄今所分析的所有核心种中,单对称性发育受CYC2演化支基因(例如:[7] - [11])。
大多数十字花科植物(芸苔科)都有多对称花冠,只有六个属的花有两对大小不同的花瓣[12].在Iberis amara,不平等的花瓣对形成与较强的表达相关CYC2进化枝基因IaTCP1在较小的,正面花瓣。正面和背面表皮细胞大小的比较表明,花瓣大小的差异是由于细胞增殖速度的差异[10].在一个peloric花的变种,只形成大的,背面的花瓣IaTCP1表达明显减少。转基因拟南芥十字花科植物CYC2转录因子IaTCP1从Iberis amara而且TCP1从拟南芥,两者都能开出相似的花,但花瓣更小。对于植物过度表达IaTCP1这是由于细胞数量的减少[10].相反,异位表达赛克从金鱼草属植物结果,转基因植物形成花瓣更大的花,这是细胞大小增加的结果[13].这说明两种十字花科蛋白质的功能主要是保守的,而来自较远的物种的CYC的功能则是保守的金鱼草属植物可能有分歧[10].Iberis花瓣同时以小凸起的形式开始发育,在雄蕊分化开始前后可以发现大小不等的发育。从这一点开始,正面花瓣和背面花瓣在整个花的发育过程中发育不同。然而,花瓣大小的主要差异出现在花的发育后期,在开花后(A1阶段)大小差异为1.6倍,在完全成熟的花(A2阶段)中增加到3.7倍[10].
这就提出了一个关于实现不同花瓣生长的分子网络的问题。研究人员对一般花器官大小决定的遗传基础进行了全面分析,这是通过几种独立的途径调节的(综述于[14]、[15])。最初的花瓣生长是通过细胞增殖来实现的,后来只有在某些特定区域才能维持生长[14].通过细胞分裂的生长停止,花瓣通过细胞伸长获得最终大小,这一转变似乎发生在花发育的后期,即小孢子成熟后[16] - [19].细胞伸长的转变伴随着细胞壁合成和细胞壁代谢基因表达的增加[20.].
因此,最终器官大小的重要决定因素是控制细胞增殖停止时间和细胞扩张开始的因素。在大多数情况下,植物生长的控制取决于不同激素之间的相互作用,这些激素可以影响细胞分裂(如细胞分裂素)、伸长(如油菜素内酯、赤霉素),或者像生长素一样,影响这两种过程[21].生长调节剂,比如转录因子AINTEGUMENTA(蚂蚁)而且锯齿状的,细胞色素P540单加氧酶KLUH或者生长素诱导基因阿哥斯(通过蚂蚁)对花瓣细胞增殖有积极影响,主要是通过影响其持续时间[22] - [25].另一方面,在花瓣发育过程中,细胞分裂期的限制是通过E3泛素连接酶降解蛋白质来实现的大哥哥通过预测的泛素受体DA1[26]、[27].MED25是中介复合物的组成部分,具有限制细胞增殖和细胞扩张的双重功能[28].同样,也AUXIN-RESPONSE FACTOR8是花瓣发育早期细胞分裂的负调节因子,并与转录因子一起抑制晚期细胞扩张大的花瓣[29]、[30.].相比之下,阿哥斯像促进细胞扩张[31].
采用RNA-Seq, a新创重新组装的花瓣转录组被生成用于全局分析Iberis amara花瓣发育和鉴定两对花瓣中差异表达的基因。这使得我们可以详细地了解在非模式生物中实现不平等花瓣生长的分子网络Iberis amara.在正面花瓣中,参与细胞壁组织和修饰过程的基因显著增强,这意味着与大的背面花瓣相比,更小的正面花瓣的形成是更早开始细胞扩张的结果。此外,对转基因进行了芯片分析拟南芥植物异位表达不同CYC2转录因子检测是为了检测靶基因调控的差异。
结果
过程中细胞分裂的时空模式分析Iberis amara花瓣发展
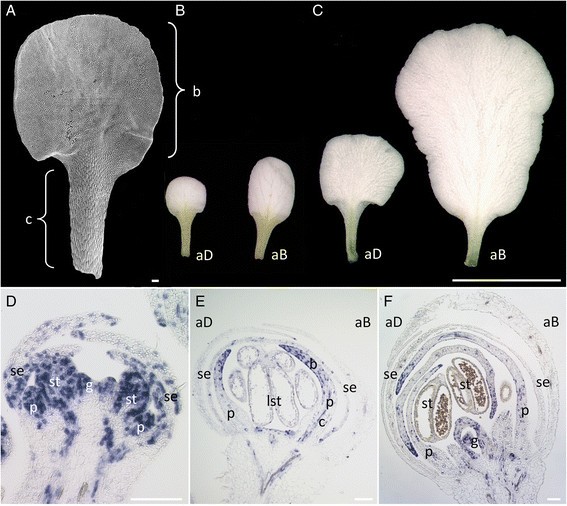
正面和背面的花瓣都形成一个上部叶片,表皮细胞圆锥形,下部有一个爪,表皮细胞长(图1A).伸长的爪状细胞呈绿色,表明叶绿体的存在(图1B和C)。花朵开放后,从A1阶段开始不平等的生长(图1B)直至开花成熟A2期(图1C),并建立了两对花瓣之间的主要大小差异,可能是通过差异细胞分裂,如[10].
Iberis amara花瓣的发展。(一)A1花瓣近轴段扫描电镜图像。正面和背面A1阶段(B)A2阶段(C)花瓣。(D-F):原位杂交与IaH4纵截面通过Iberis amara开花前的花蕾。(D)在年轻的花朵中,IaH4在整个新生的花瓣芽中表达。(E)后来,在区分花瓣时,IaH4定位于上部,远端花瓣部分,形成叶片,并在近端部分减少,发展成爪。(F)IaH4在较老的花中,转录本在开花前在花瓣尖端积累。散布在整个花瓣长度的一些细胞仍然表达IaH4.广告,向轴的;aB,远轴的;b,叶片;c,爪;p,花瓣;se,萼片;顶生,侧生雄蕊;圣,雄蕊;g,雌蕊。 Scale bars:, D-F, 100 μm;B而且C, 5毫米。
通过导联法分析了花瓣发育过程中细胞分裂的时空格局原位杂交研究的年轻人Iberis花前的花蕾带有细胞周期标记基因Histone4从Iberis amara(IaH4).最初,IaH4在花瓣原基中一致表达(图1D).在较老的花中,花瓣分化开始后,IaH4近端表达下降,远端花瓣部分表达增加(图1E).这表明细胞分裂首先在近端区域停止,细胞扩张成细长的爪状细胞,而在叶片中细胞分裂仍在继续。此外,有更多的细胞表达IaH4与花瓣正面叶片相比,花瓣背面叶片中有更多的花粉1E和F),如较早阶段所示[10].在开花前后,当花瓣完全覆盖繁殖结构时,IaH4表达主要集中在远端花瓣尖端和少数IaH4在近爪区可见表达细胞(图1F)。
这些数据表明Iberis花瓣沿近端-远端梯度成熟,细胞分裂下降,从基部移动到花瓣尖端。此外,在叶片尖端的花周围,细胞分裂仍在持续,促使成熟的正面花瓣和背面花瓣最终形成3.7倍大小的差异。
重建Iberis amara花瓣转录组
之前检测的基因,其中有差异表达Iberis amara通过RNA-Seq,重建了参考花瓣转录组。分别从A1期花的正面和背面花瓣上分别取2个重复IaTCP1在此阶段,正面花瓣的表达量最高,正面和背面花瓣的表达量差异最大[10].为了避免这种几乎不自花授粉的非模式生物缺乏等基因系造成的遗传差异,所有的花瓣材料都是从一株植物上连续采集的。
Illumina对所有四个文库的测序总共产生了184,515,960个高质量的过滤成对末端reads。50个核苷酸(nt)的长度,相当于大约9gb的序列数据。其中,156,818,896个读取(约85%)用于重新组装(表1A),这与其他RNA-Seq研究相当[32]、[33].获得的52,081个contigs(表1B)不能再通过串联化扩展,因此,代表用于进一步分析的花瓣转录本。平均contig大小为677 nt,范围从191到12,804 nt。
大约59%的contigs小于500个核苷酸,这表明可能有更大一部分转录本没有被相应的contigs完全覆盖。在所有的contigs中,大约22%的长度在1000个核苷酸以上,14%的长度在1000 - 2000个核苷酸之间,其次是大约5%的长度在2000 - 5000个核苷酸之间,0.2%的长度在5000 - 10000个核苷酸之间1C).通过建立的花瓣转录组重组,下一步可以完成花瓣发育的整体分析。
注释的Iberis与花瓣发育有关的基因
所有52081个contigs都是基于BLASTX搜索和拟南芥蛋白质数据库,以允许功能分类Iberis并与花瓣转录本进行进一步比较拟南芥表达数据。45001年IberisContigs匹配到一个对应的拟南芥基因组计划(AGI)代码。59.6%的匹配contigs检索到的AGI代码也被发现与其他contigs匹配。这很可能是因为有很大比例的Iberis转录本没有被它们各自的contigs完全覆盖,因此几个contigs被分配给相同的AGI代码。同样,检测到大量编码转录因子的基因MarchantiaRNA-Seq被解释为由“零碎”数据集引起的,可能超过了实际存在的转录因子的数量[34].因此,在这里,具有相同AGI代码的contigs被视为同一基因的部分转录本,并被减去,得到18,139个假定的unigenes。接下来,应用e值截断e−4结果显示15,983个(88%)可能的unigenes具有假定的保守功能拟南芥.没有命中的contig可能是组装工件或者是唯一的Iberis.
根据他们各自的AGI代码,15983Iberisunigenes通过MapMan提供的分类superviewer工具进行功能分类[35]、[36].在种间转录组比较中,来自13492个基因表达的微阵列数据拟南芥第15阶段的花瓣被拿走了。第15阶段大约在花开花后一天开始[37],因此,在发展上最接近于IberisA1期,定义为花期后的阶段[10].
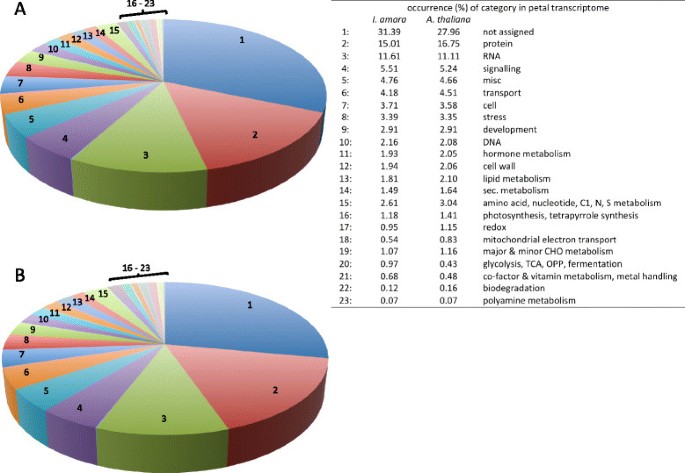
两种转录组均显示出属于各自类别的基因的总体相似分布,这表明全球十字花科植物的花瓣发育程序是相似的(图2有趣的是,最大比例的基因被归类为不分配.这个班级占28%拟南芥花瓣转录组(图2B)和单对称花冠中31%的转录本Iberis(图2A).此外,两者的花瓣发育较晚,Iberis而且拟南芥,主要是由分配到类别的基因决定的吗蛋白质,RNA,信号,杂项(包括参与不同过程的各种酶)、运输而且细胞,占所有类别的75%。
描述的Iberis花瓣发展
为了分析花瓣正面和背面的基因表达水平,将四个样本的干净读数分别映射到新创重组花瓣转录组。只读取唯一映射到引用的单个位点的值Iberis转录组被考虑在内,占所有reads的63-64%2).每个样本库的基因表达水平显示为RPKM(每千碱基每百万映射reads)值。在对乘法误差应用临界值0.01的p值后,1600个基因仍然具有可靠的表达值。所有基因及其表达值的完整列表在附加文件中给出1.
在这1600个转录本中,从两种花瓣类型中最强表达的前5%的基因进行了功能分类,以了解哪种生物过程对晚期花瓣发育最重要Iberis(表3.,附加文件2).类别misc包含最多数量的高丰度转录本。此外,花瓣的发育似乎依赖于基因的强烈表达,这些基因也在适应生物/非生物胁迫和激素代谢中发挥作用。有趣的是,上述类别的成绩单不分配在前5%的最强表达基因中,无论在正面还是背面Iberis花瓣。这进一步证实了一种观察,即花瓣的晚期发育似乎需要大量迄今尚未鉴定的基因的活动。
在花瓣正面和背面差异表达的转录本鉴定
对于所有1600个基因,分别比较了花瓣正面和背面的表达值(表1)4A), 1141个基因在近轴花瓣中表达较强,459个转录本在背向花瓣中RPKM值较高。大多数基因(1266个)的表达倍数变化小于2倍,其中828个基因在近轴花瓣中表达较高,438个基因在背向花瓣中表达较高。334个基因的表达在两种花瓣类型之间发生了2倍或更多的变化,其中313个基因在近轴花瓣中转录量更高,只有21个基因在背向花瓣中表达量更高。这些基因中的大多数表达差异可达5倍(花瓣正面240倍,花瓣背面20倍)。只有一小部分的折叠变化等于或大于5(正面花瓣64和9,背面花瓣1个基因),包括alsoIaTCP1,在近轴花瓣中上调约27倍,证实了之前的观察结果[10](附加文件3.).
总的来说,这些数据表明,与控制大的背向花瓣形成相比,控制小的正面花瓣发育的基因数量明显更高。
正面花瓣转录组中基因类别的测定
为了了解参与两种花瓣类型形成的发育过程,我们将折叠变化最小的差异表达基因分为功能MapMan分类。在花瓣背面上调的21个转录本中,没有发现包含超过3个成员的类别,这是应用的阈值标准之一。在近轴花瓣上调的313个转录本中,约有25%(81个)未被分配到任何类别,编码功能未知的蛋白质(表2)4B和附加文件3.).大量的基因(26)被归为一类,编码各种酶misc其中涉及25个转录本蛋白质代谢,如蛋白质修饰,靶向和降解。类别运输包含24个基因,编码转运无机化合物、初级代谢物、离子、金属或生长素的蛋白质。类别细胞壁,信号而且光合作用在应用与正面特异性转录本相关的AGI编码的基因本体(GO)术语时,也被检测到显著富集,并在下面进行描述。在类别中核糖核酸(23个成员),20个编码各种转录因子,包括IaTCP1.此外,16个正面上调基因在生物和非生物胁迫的适应中发挥作用。
为了更详细地了解产生更小的正面花瓣的发育机制和相关基因,使用FatiGO揭示了313个正面特异性转录本中显著丰富的GO项。38].
个体基因被反复分配到相关的GO项上。GO术语与细胞壁相关的过程相关联,例如细胞壁组织而且修改在花瓣正面显著富集(表4C).发挥细胞壁相关功能的15个基因编码果胶裂解酶、果胶甲酯酶/果胶降解抑制剂[39以及其他在细胞壁重塑和松动中起作用的酶。甚至更多的基因被分配到MapMan类别细胞壁(21)(表4B)除细胞壁降解/修饰酶外,还包括COBRA 10和阿拉伯半乳聚糖蛋白。这两种蛋白质都构成了细胞壁和细胞质之间的假定链接[40].
GO项信息的信号含有5个转录本,编码在植物细胞生长中起作用的小信号肽(表4C).这些肽最初是在烟叶提取物中发现的,因为它们能够诱导细胞悬浮培养中的pH值迅速升高,因此被称为RALFs(快速碱化因子)[41]、[42].
GO项花形态发生包括三个参与花瓣发育的转录因子,即花瓣损失,叶柄上的叶片1而且2.的类别内吞作用,膜出芽而且网格蛋白涂层组件包括编码ENTH/ANTH/VHS超家族蛋白的基因,这些蛋白是参与膜运输的核内体系统的关键调节因子[43].
出乎意料的是,包括GO项在内的17个基因被发现与光合作用有关光合作用,光收集,光反应而且碳固定。为了分析光合作用相关过程是否有助于花的单对称性形成Iberis这一发现通过详细的qPCR分析得到了验证。因此,磷酸激酶同源物从Iberis作为该类别的代表进行分析光合作用并证实RNA-Seq数据显示在近轴花瓣中有更强的表达(附加文件)4A).接下来,分别采集两种花瓣类型的花瓣叶片和花瓣爪材料进行qPCR,检测两种花瓣类型中是否都存在绿色的花瓣爪细胞(图1B),有助于光合作用相关基因的检测。磷酸激酶转录本在正面和背面花瓣叶片或花瓣爪之间没有差异表达(附加文件)4B),说明光合作用基因对正面花瓣较小的形成没有贡献。在两种类型的花瓣中,绿色的爪状区域,磷酸激酶表达更强(附加文件)4C),在尺寸和形状上相似,但叶片尺寸差异显著。因此,RNA-Seq检测到的光合作用相关基因的表达差异可能是由于收获整片花瓣稀释了大背面花瓣比小正面花瓣更强的爪表达基因。因此,光合作用相关基因被排除在进一步的分析之外。
正面上调基因的表达分析IberisqPCR检测花瓣
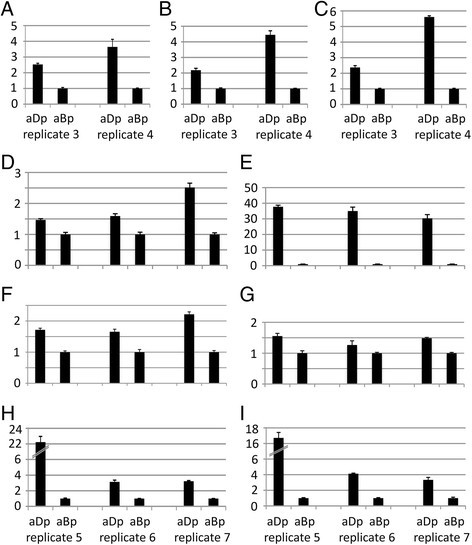
接下来,来自其他显著富集的GO术语的转录本(表4C)用qPCR进一步分析。从类别来看细胞壁组织选取了5个在细胞大小控制中具有可能功能的代表。所有细胞壁相关基因均在两种花瓣类型的A1阶段的叶片材料上进行检测,以排除长爪细胞介导的影响(图1A).全部经过测试Iberis的基因,VANGUARD1同源物2,一种聚半乳糖醛酸酶,一种糖基水解酶,一种果胶裂解酶家族成员和一种转化酶/果胶甲基酯酶抑制剂,在正面花瓣叶片中的表达比背面花瓣叶片强1.5至3.6倍(图2)3.A-E),表明细胞壁相关过程在正面花瓣形成过程中的参与增强。从类别来看信息的信号(表4C) 5个转录本中的2个被选作进一步的qPCR分析。这两个Iberis拉尔夫基因在近轴花瓣中表达较强(图4A和B),表示该组的一个可能的函数信息的信号形成小花瓣的基因。三螺旋转录因子花瓣损失(PTL)属于显著富集类花的形态(表4C),在近轴花瓣中表达增强(图4D).此外,强烈的差异花瓣的表达TCP转录因子IaTCP1[10]的qPCR验证结果显示,在所有测试基因中,两种花瓣类型的表达差异最大(图4E)。
从这些数据中,可以得出一个大致的情况,即正面花瓣的特征是含有丰富的转录本,这些转录本涉及细胞壁组织和信号传递的基因。这意味着,相对于较大的花瓣,较小的花瓣是细胞分裂较早停止的结果。与此同时,参与细胞伸长和分化的基因在小的、正面的花瓣中比大的、背面的花瓣出现得更早,后者在细胞增殖期停留的时间更长。进一步从近轴花瓣中上调的转录本中选择基因(附加文件)3.)基于它们的注释和与这些过程的假定关系。
参与细胞扩张过程的第一个有趣的候选人是类眼镜蛇10 (COBL10)的成员结实的矮基因家族。眼镜蛇,是第一个被分析的成员,在根伸长区强烈表达,被认为是定向细胞扩张的关键调节因子[44].同样的,COBL10参与控制定向和可能的雌性引导花粉管生长[45].在两种生物样本中Iberis COBL10与背面花瓣相比,正面花瓣的表达上调(图4C),如RNA-Seq所示。另一基因在正面上调Iberis花瓣是p糖蛋白(PGP(图4F),属于植物ABC转运蛋白超家族的范畴运输(表4B)。PGPS已被证明在植物生长发育中发挥作用,因为它们介导生长素的细胞和长距离运输[46].细胞分裂素氧化酶3(CKX 3)催化植物激素细胞分裂素的分解,从而对细胞分裂产生负面影响[47].因此,CKX 3其RNA-Seq表达fold change为1.9,略低于2倍阈值,纳入进一步qPCR分析。更强的表达伊比利亚CKX 3在花瓣正面同源(图4G)可能与降低细胞分裂速率有关,在细胞伸长之前。也Rcd1-like,一种参与细胞分化的蛋白质核糖核酸(表4B)在近轴花瓣中表达较高(图4H).哺乳动物Rcd1基因是一种转录辅助因子,在细胞向分化的转变中发挥作用[48].在正面花瓣中较强的基因表达,显示出最高的同源性拟南芥基因At5G42680编码一种功能未知的蛋白质(图4I),证实了这种高度丰富的转录本在近轴花瓣中上调的成员的差异表达。
综上所述,qPCR分析强化了以下观察结果:在较小的近轴花瓣中,差异花瓣大小的发育是通过较早开始的细胞伸长和分化来完成的,这表明与细胞壁重塑、细胞分裂抑制、细胞分化和信号传导有关的基因上调。
拟南芥异源系统CYC2靶基因异位表达的种间比较
转基因拟南芥植物过度表达两种十字花科CYC2转录因子IaTCP1而且TCP1产生花瓣较小的花,而异位表达的赛克从金鱼草属植物形成更大的花瓣[10]、[13].比较CYC、IaTCP1和TCP1蛋白在中药下游调控网络中的作用拟南芥,进行微阵列分析。用于RNA提取的材料取自过度表达这些基因的转基因T2植物的年轻花序,包括开花前的花芽阶段。
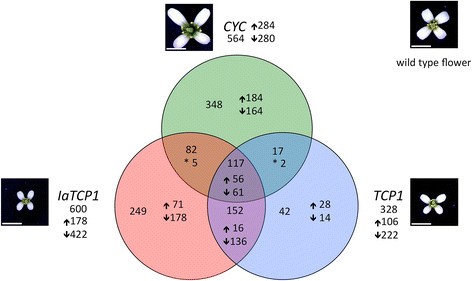
总共有600个基因被确定在异位后被差异调节IaTCP1表达,大多数(422)受到负面影响(图5).略少的基因(564)对过度表达的赛克(上调284个,下调280个)。最低数量的基因(328)是由异位处理的TCP1活跃度(106点上调,222点下调)。
的CYC2转录因子赛克而且IaTCP1,控制单对称发育,在异种中处理大量的靶基因拟南芥系统比TCP1.这种趋势仅在单个CYC2蛋白只处理的靶基因数量上更为明显,即CYC处理348个,IaTCP1处理249个,TCP1仅处理42个。的能力赛克而且IaTCP1处理大量下游靶标的基因可能反映了它们在控制单对称形成中的功能,即使在形成多对称花冠的异源种中表达,如拟南芥.117个靶基因通常由所有这三种转录因子处理,因此可能与对称调节中的特定功能无关。两个十字花科转录因子之间重叠调控的基因数量最多(152),而CYC和TCP1相互调控的靶标数量最少(19)。
深入了解下游的分子网络IaTCP1,检测到明显累积的GO项。422个基因被IaTCP1,大多数富集的GO项与细胞壁相关的过程相关(细胞壁组织,细胞壁修饰,植物型细胞壁组织,单维细胞生长而且植物型细胞壁松动).其他丰富的GO术语与次生代谢过程,H2O2分解代谢的过程而且对红灯的反应(表5).额外的文件5总结了单个基因,其中有11个细胞壁基因通过异位表达相互处理IaTCP1在拟南芥花序和由内生IaTCP1在近轴的Iberis花瓣。
RNA-Seq和微阵列分析都检测到参与细胞壁过程的基因的差异调节。然而,在年轻的转基因拟南芥花序所有细胞壁相关转录本在异位后下调IaTCP1活动,而它们在老年人中上调Iberis花后的花瓣,与高内源性相关,大概也对其作出反应IaTCP1表达式。这表明了的时空脉络CYC2活性对于其对细胞生长和分化的作用至关重要。
讨论
单轴对称的Iberis是通过花发育后期的分化细胞增殖实现的吗
Iberis花瓣同时开始生长,不平等的生长开始于雄蕊分化的开始,从那里开始,并在开花后获得最大的大小差异,可能是由不同的细胞增殖驱动[10].这需要细胞分裂在花发育的后期阶段继续进行,这可以通过监测细胞分裂模式来证明Histone4表达的Iberis在不同的花瓣阶段。在较老的花瓣中,两种花瓣类型的细胞分裂仍然发生在接近开花的地方,细胞分裂主要局限在花瓣尖端。类似于Iberis,细胞分裂拟南芥花瓣在花的发育过程中不断增加,直到第11阶段,在第12阶段,即开花前仍然可以看到[23].
花后,正面Iberis花瓣增加2.8倍,而背面的花瓣增加6.7倍,达到最终的大小。相比之下,两种花瓣类型的细胞从开花到成熟只会增加一倍[10].这些数据表明,至少在背向花瓣中,细胞分裂在开花之后继续进行。
植物花瓣发育的转录组分析Iberis amara
RNA-Seq被越来越多地用作一种方便的工具,用于对无法获得基因组序列的物种进行比较转录组分析[49] - [51].应用RNA-Seq方法,我们研究了Iberis转录组在花瓣发育后期A1阶段,即花开花后。
比较拟南芥而且Iberis花瓣转录本在功能分类上的分布表明,两种十字花科植物花瓣发育后期的分子过程是相似的。有趣的是,对于最大的群体Iberis花瓣转录组占转录本的31%,尚未确定其功能。在28%的这些基因中观察到类似的情况拟南芥.有趣的是,比较分析了不同的花瓣阶段金鱼草属植物研究发现,在花瓣中表达的所有转录本中,未分类蛋白质组的频率占50%以上[16].这表明,很大一部分基因在花瓣形态发生过程中发挥着有趣的,但尚未被表征的功能。
中最丰富的转录本分析Iberis花瓣A1期显示出多种酶,归为该类misc,在两种花瓣类型中均有高表达,其中包括gdsl -脂肪酶和脂质转移蛋白。这两种酶类型都在脂质代谢中发挥作用,并被描述为花瓣发育过程中花瓣和雄蕊指定b类MADS转录因子的推测靶基因金鱼草属植物而且拟南芥[16]、[52].细胞色素P450基因CYP76C3,这是表达率最高的语言之一Iberis花瓣基因,也强烈表达在各种拟南芥花器官,在那里它参与芳樟醇代谢。芳樟醇是一种已被充分研究的传粉媒介引诱剂,在自交配中通过CYP76C3进一步转化为其氧化衍生物拟南芥花(53],它们可能在植物防御病原体和食草昆虫方面发挥作用[54].两个脂肪氧合酶,LOX1而且LOX2,在激素代谢中起作用,在Iberis花瓣。LOX2蛋白催化茉莉酸(JA)生物合成的第一个专用步骤,并被叶片中的转录因子TCP4激活[55].TCP4也表示为bothIberis花瓣类型(附加文件1),由于LOX2定位于叶绿体[56),LOX2JA的依赖诱导可能发生在绿色花瓣的爪子中,在那里JA可能对细胞增殖产生负面影响[57].
的单对称花冠形成Iberis是通过从细胞分裂到细胞延伸的不同时间切换来实现的吗
近轴小花瓣中上调的基因几乎是大花瓣的15倍,似乎需要更多的基因来限制近轴花瓣的大小Iberis花冠的发展。事实是,单对称键调节器IaTCP1在近轴花瓣中高度表达,是表达差异最大的基因之一,这意味着IaTCP1激活的靶基因数量比其抑制转录效应要多。
微分Iberis花瓣的发育可能是由不平等的细胞分裂过程驱动的。一直以来,对细胞分裂产生负面影响的基因都存在于近轴花瓣中上调的转录本中。在拟南芥细胞分裂素氧化酶(CKX催化植物激素细胞分裂素的代谢分解和植物缺陷ckx由于细胞数量的增加,基因形成了花瓣变大的花[47].三螺旋转录因子PTL表现在发展中地区拟南芥花,生长被抑制的地方。PTL萼片间区的表达被认为可以控制细胞增殖,从而使花瓣原基得以生长。损失PTL功能导致该区域的细胞增殖增加[58].花瓣是对局部生长素积累的反应,Lampugnani和同事[59]推测这可能是通过支持的作用引起的PTL通过PGPs进行非极性生长素外排。PGPs属于abc转运蛋白超家族,在生长素转运中起介导作用[60].PGP13,它也在正面转录富集Iberis花瓣,属于同一支生长素出口商PGP1而且PGP19[46],这种双突变植物会出现严重的生长缺陷[60].
除了转录本对细胞增殖有负面影响外,大量与细胞壁修饰过程相关的基因在正面花瓣中表达上调。在通过细胞分裂实现器官生长的初始阶段之后,通过细胞伸长实现器官的最终大小[14]、[15].陆生植物的初生细胞壁由木质葡聚糖(XG)链交联的强纤维素微纤维框架组成,并嵌入果胶多糖和结构蛋白的稳定基质中。植物细胞壁通过承受拉伸应力来提供稳定性,细胞的生长是由于细胞壁的可伸长性因适应这种应力而发生变化[61]、[62].在近轴细胞壁修饰蛋白组中Iberis花瓣是几种果胶酶,在果胶的去甲基酯化或降解中起作用的酶[39]并通过精确调节细胞壁中果胶酸的比例来促进细胞生长[62].一个木葡聚糖内转糖酶(XET),它会裂解并重新连接木葡聚糖链在体外在正面花瓣中表达上调。XET活性与正在进行细胞扩张的区域相关,这表明XETs在细胞扩张中发挥作用,例如通过新形成的细胞壁的生物发生(综述于[63])。此外,还有几个伊比利亚糖基水解酶在近轴花瓣中上调,形成了一组不同的酶,水解糖苷键[64].一个拟南芥的同系物“大酒店”属于endo-1,4-ß-葡聚糖酶亚家族KORRIGAN,对细胞伸长和纤维素合成至关重要拟南芥下胚轴(65].近轴花瓣中编码一种类似bra蛋白、两种富含脯氨酸的延伸样受体激酶(PERKs)、一种阿拉伯半乳聚糖蛋白和一种双胍的转录本上调,可能是细胞质和细胞壁之间的潜在连接。因此,这些蛋白质是连接细胞骨架网络、细胞质和细胞壁的分子连续体的一部分,并有助于控制细胞的扩张拟南芥(见[40])。
拟南芥一些花细胞壁相关基因的同源物,如聚半乳糖醛酸酶(At1G02790)、XET (At1G65310)和果胶裂解酶样成员(At5G07430)也主要表达在根尖的细胞伸长区(用图1所示)拟南芥eFP浏览器bar.utoronto.ca),并且在前面的分生区只有弱的,在那里生长是通过细胞分裂实现的。这支持了它们在花和根环境中细胞壁修饰的一般功能,通过介导细胞伸长过程促进组织生长。
最终器官大小的确定需要细胞分裂和细胞伸长过程的精确协调,并且可能依赖于紧密的细胞通信。在所有正面花瓣特异性基因中,一组参与细胞-细胞信号传递的转录本显著富集。这些ralf是小肽,在许多发育过程中都很重要,被认为是一种新型的植物激素[41].在草叶中,RALFs主要在分生区以外的年轻伸长细胞中表达,而在成熟细胞中几乎不表达[66].由于RALFs被认为对细胞生长有负面影响,它们的表达强度和位置可能在器官发育过程中被精确调整,以实现对细胞扩张的精确引导,而不会完全阻断细胞扩张[66]、[67].
综上所述,在小型、近轴富集的313个转录本中,涉及细胞壁修饰和细胞-细胞信号转导的转录本数量显著增加,以及调节细胞分裂和生长素运输限制的基因被检测到Iberis花瓣。因此,不均匀的花瓣生长可能是通过在近轴花瓣中较早地停止细胞分裂,随后较早地开始细胞扩张来实现的,而在背向花瓣中细胞继续分裂。在Iberis时,细胞分裂终止于顶瓣梯度,顶端的细胞分裂持续时间最长,与其他植物物种相似[23]、[68].因此,细胞分裂的不平等停止可能是通过近轴花瓣的细胞分裂停止比背面花瓣更快地进行而实现的。
iatcp的异位表达引起拟南芥细胞壁相关基因的表达变化
微阵列分析的目的是比较分子网络在花序的地址IaTCP1而且TCP1造成较小的花瓣形成,并由赛克,从而产生更大的花瓣。
相当数量的基因(分别为600、564和328中的117个)由所有三个CYC2蛋白一起处理。这些转录本可能构成一组共同的基础TCP / CYC2转录因子靶向基因拟南芥.有趣的是,在异源中拟南芥系统中,更多的基因被专门处理IaTCP1从Iberis(249)和赛克从金鱼草属植物(348),发育单对称花,比TCP1从拟南芥(42),形成多对称花冠。这些数量上的差异表明IaTCP1而且赛克可能调控更多的靶基因,这意味着与多对称花冠发育相比,实现单对称花冠发育的下游调控网络更大。因此,除了顺式调控的变化之外,还导致了异质性CYC2芸苔科基因表达变化促进花冠单对称性进化[10]、[12],也是反式作用的发散CYC2蛋白质很可能促成了这种形态上的创新。
其中重叠最大的靶基因数为IaTCP1而且TCP1.这152个基因包括参与细胞壁相关和信号过程的几个转录本(附加文件)6而且7)和这些专属的十字花科目标的活动可能是必不可少的花瓣大小限制拟南芥,因为只有异位表达十字花科CYC2蛋白的转基因植物才能开出花瓣更小的花[10].在目标基因群体中,响应异源基因下调IaTCP1表达拟南芥,同样高比例参与细胞壁修饰过程。这表明IaTCP1处理这些基因拟南芥并且在近轴RNA-Seq检测到该基因类的高丰度Iberis花瓣的效果是坚强的IaTCP1表达式。而细胞壁基因在老的正面影响Iberis花瓣,IaTCP1表达强烈增强,IaTCP1活动对幼鼠细胞壁基因有负面影响拟南芥花序overexpressingIaTCP1.已知TCP转录因子对器官生长具有激活或抑制作用,这取决于其组织特异性表达[7]、[13]、[69].TCP蛋白作为二聚体[70],它们活动的结果可能因在不同器官或发育阶段对可用相互作用伙伴的依赖而不同。KNOX/BEL转录因子BELLRINGER也有类似的发育依赖性调控作用[71] (BLR),与其他BEL和KNOX蛋白相互作用[72]、[73].它的目标之一,果胶甲基酯酶PME5在芽分生组织中被BLR抑制,以允许原基的协调形成,但在节间被BLR激活,促进细胞伸长[71].
结论
A1前后期RNA-Seq表达分析Iberis在花瓣中,正面花瓣的细胞壁相关转录本数量显著增加。这说明花瓣生长差异大,具有单对称的特征Iberis花冠,是通过细胞增殖阶段的早期停止,随后在较小的正面花瓣中开始细胞扩张和分化,而背面花瓣的细胞可能仍然通过细胞分裂继续生长。作为异位表达的IaTCP1在拟南芥与细胞壁相关的基因,增强了这类基因在正面的发生Iberis花瓣也很有可能是直接的强烈后果IaTCP1表达式。此外,十字花科单对称进化可能依赖于顺式调节变化的组合,导致了晚期的近轴IaTCP1CYC2蛋白本身的差异介导的跨调控变化,改变了导致大花瓣大小差异的潜在靶基因集Iberis amara.
方法
植物材料和生长条件
Iberis amara种子是从Saatgut Kiepenkerl(德国everwinkel)购买的,并在温室中种植了几代。植物是在Osnabrück大学的标准温室条件下种植的。转基因拟南芥(col)植物在生长室中在20°C光照13小时/黑暗11小时的循环条件下生长。
Illumina公司测序,新创组装和RNA-Seq分析
对于RNA-Seq,两个A1期近轴技术重复(样本aD_1和aD_2,表2)和aB_1、aB_2样品,表2)的花瓣从一株植物中采集,以避免遗传变异,这会阻碍后续的转录组新创组装。植物材料在液氮中快速冷冻,用RNeasy Plant Mini试剂盒(Qiagen, Valencia, CA)分离总RNA,包括柱上DNA消化步骤。
文库准备,Illumina测序,新创组装,读映射和显著性统计,如下所述,由BaseClear BV,荷兰(www.baseclear.com).利用Illumina TruSeq RNA- seq文库制备试剂盒从总RNA中纯化、片段化mRNA,转化为双链cDNA。在TruSeq Illumina测序仪上循环50次后获得配对末端读数。序列读取使用Illumina Casava流水线版本1.8.2生成,在通过质量评估后,过滤和修剪读取,排除重复,使用CLC Genomics Workbench版本5.1.0组装。每个样本的干净reads(包括重复)分别映射到一致转录组,并使用CLC Genomics Workbench计算每个样本的contigs的表达值。一个读由完整的成对端读对和单个读组成,当唯一映射时,它们都被算作一次命中。破碎的配对,其中一个伴侣缺失,从量化中被省略。每个contig的表达式通过每个花瓣类型的RPKM值显示,RPKM值是来自两个技术重复的平均表达式。每个contig的显著性表达进行Baggerley检验[74],然后对乘法误差进行Bonferroni修正。
数据分析和全局基因表达比较
所有contig序列对an进行爆破拟南芥蛋白质数据库(TAIR10_pep_20110103_representative_gene_model)多个检索相同的contigs拟南芥agi编码被视为同一基因的部分转录本。因此,来自具有相同AGI代码的contig的各自表达式值被合计。差异表达基因定义为具有统计学意义的p值低于0.01,表达倍数变化等于或大于2。
的功能注释Iberis序列是基于每个contig对其各自的赋值拟南芥通过BLAST-search进行正交。对于所有下游分析,只有e值等于或低于e时支持contig/AGI代码匹配4被认为是。使用bar.utoronto.ca的分类superviewer工具进行功能分类[36],基于MapMan标注(Ath_AGI_LOCUS_TAIR10_Aug2012.txt文件)。只有p值低于0.05且成员超过3个的类别才会被列出。注意,在图中2组合了几个MapMan类别。
采用FatiGO对GO术语富集进行统计学分析(p值<0.05)[38]对抗15983种unigenesIberis转录组(表4C)反对拟南芥从TAIR下载的花序转录组(表5).
的拟南芥第15阶段花瓣和花序转录组数据来自微阵列分析(TAIR-ME00319;AtGenExpress:的表达图谱拟南芥开发)和从TAIR下载(注册编号:HybData: 1007126238-40和HybData: 1007126271-73)。只有在所有三个重复中都出现的元素被认为是表达的,出现一次以上的AGI代码被删除。
定量实时PCR验证
使用RNeasy Plant Mini试剂盒(Qiagen, Valencia, CA)分离总RNA,包括柱上DNA消化步骤。定量逆转录酶PCR方法,详见[12].引物通过Primer3程序在线设计[75]和引物序列在附加文件中给出8.
通过凝胶分析和每次反应后产生的熔体曲线检查来评估每个PCR的特异性。每个样本的平均归一化表达式(MNE)(计算方法为[12])基于每个反应的三次技术重复,除了contigs 5537, 12149, 16003, 6640和7844(附加文件1而且3.),其中一个样本只使用两次技术重复。错误条是来自技术复制的标准错误。的伊比利亚基因Ran3 GTPase[GenBank: EU145777]作为管家基因进行归一化。
对A1阶段的花瓣正面和背面进行实时PCRIberis3个生物重复(图中重复5-7)4D-I和附加文件4A).表达拟南芥直接同源的Iberis对待测基因进行分析拟南芥eFP浏览器在bar.utoronto.ca [76].在花粉中强烈表达同源物的情况下,使用来自A1期花的无花粉的正面和背面花瓣的两个生物重复(图中重复3,4)4a - c)。表达差异拟南芥直接同源的Iberis基因分配到功能go类连接细胞壁而且光合作用在A1期花瓣正面和背面(无花粉)的两次生物重复上进行了验证Iberis花,被切割成上部花瓣叶片和下部花瓣爪(图中重复1、2)3.和附加文件4B和C)。
为了验证RNA-Seq方法检测到的基因表达差异,对至少两个生物重复的A1期花瓣对进行qPCR。对给定基因进行配对表达比较,将背面花瓣(或花瓣爪,在叶片/爪比较中)的表达值设为1。近轴花瓣在至少两次生物重复中表达增加1.5倍或以上被认为是差异表达,因此进一步证实了RNA-Seq数据。
微阵列分析
从每个表达的转基因T2系中制备两个总RNA技术样本IaTCP1,TCP1而且赛克,分别控制下的CaMV35S启动子(10]或空向量pBAR35S [GenBank: AJ251014]作为参考。从主花序中显示出清晰花表型的植物中提取总RNA,收获只携带年轻的闭合花蕾的次生花序。RNA分离使用RNA分离试剂盒NucleoSpin RNA XS (Macherey and Nagel)进行。探针制备,与ATH1杂交拟南芥基因组阵列(Affymetrix, Santa Clara, California)和统计数据分析在Münster大学(德国)的综合功能基因组服务单位进行。只有表达倍数变化最小为2且p值≤0.05的基因才被认为是差异表达。将微阵列表达数据与含有空载体的转基因植物进行比较。通过逆转录酶PCR验证所选基因的数据,如[10](附加文件9),使用与微阵列分析相同的植物材料。转录累积值来自于3次PCR重复,退火温度为58℃,归一化到的表达强度Ran3 GTPase(At5g55190)。引物序列在附加文件中给出8,微阵列数据在附加文件中给出6,10,7.
原位杂交
为原位杂交实验IaH4[GenBank: EU145778]反义探针杂交到我阿玛拉。花蕾在开花前,按[10].
图像取得
用于扫描电子显微镜,将花瓣冷冻在液氮中,溅射在低温制备系统(K1250X, Emitech)中涂有钯,并用数字扫描电子显微镜(Zeiss Auriga)进行检查。A1和A2阶段的花瓣图片使用双筒相机(徕卡M165 FC, Wetzlar)拍摄。
支持数据的可用性
支持本文结果的序列数据集可从NCBI的短读档案(SRA)数据库中获得,编号为SRP048782。组装的contigs在附加文件中以FASTA格式提供11.微阵列数据已保存在NCBI的基因表达综合(GEO)数据库中,并可在GSE62213下访问。
附加文件
参考文献
- 1.
Dilcher D:朝着一个新的合成:被子植物化石记录的主要进化趋势。中国科学:自然科学,2000,29(3):344 - 344。10.1073 / pnas.97.13.7030。
- 2.
Sargent RD:花的对称性影响被子植物的物种形成率。中国生物科学,2004,27(3):357 - 357。10.1098 / rspb.2003.2644。
- 3.
Stebbins G:开花植物:物种水平以上的进化。1974年,哈佛大学出版社,剑桥(马萨诸塞州)
- 4.
罗东,张志刚,李志刚,张志刚。中国植物不对称的研究进展金鱼草属植物.自然科学,1996,37(3):344 - 344。10.1038 / 383794 a0。
- 5.
罗东,刘志刚,刘志刚,刘志刚,刘志刚,黄志刚:植物器官不对称的控制金鱼草属植物.细胞学报,1999,39(3):367-376。10.1016 / s0092 - 8674(00) 81523 - 8。
- 6.
Howarth DG, Donoghue MJ:“ECE”(CYC/TB1)演化支的系统发育分析揭示了早于核心euudicots的重复。自然科学进展,2006,29(4):344 - 344。10.1073 / pnas.0602827103。
- 7.
Broholm SK, Tähtiharju S, Laitinen RAE, Albert VA, Teeri TH, Elomaa P: TCP结构域转录因子控制花型沿径向轴的规格非洲菊(菊科)花序。自然科学进展,2008,26(2):339 - 344。10.1073 / pnas.0801359105。
- 8.
金敏,崔明明,陈志伟,陈志伟,陈志伟,陈志伟,陈志伟,陈志伟,陈志伟。植物间形态和生态特性的调控基因研究进展。科学通报,2008,37(3):344 - 344。10.1126 / science.1164371。
- 9.
王忠,罗勇,李霞,王玲,徐松,杨娟,翁玲,Sato S, Tabata S, Ambrose M, Rameau C,冯欣,胡霞,罗东:豌豆花对称形态的遗传控制(Pisum一l .)。自然科学进展,2008,29(4):344 - 344。10.1073 / pnas.0803291105。
- 10.
芸苔科花冠单对称性的控制Iberis amara.中国科学:自然科学,2007,29(2):344 - 344。10.1073 / pnas.0705338104。
- 11.
冯欣,赵忠,田志,徐松,罗勇,蔡忠,王勇,杨娟,王震,翁磊,陈娟,郑林,郭欣,罗杰,佐藤生,Tabata S,马伟,曹欣,胡欣,孙超,罗东:花形与花形的控制Lotus粳稻.自然科学进展,2006,29(4):457 - 457。10.1073 / pnas.0600681103。
- 12.
Busch A, Horn S, Mühlhausen A, Mummenhoff K, Zachgo S:花冠单对称性:芸苔科一个形态学新物种的进化。分子生物学杂志,2012,29(4):1241-1254。10.1093 / molbev / msr297。
- 13.
Costa MM, Fox S, Hanna AI, Baxter C, Coen C:控制花不对称的调控相互作用的进化。《科学进展》,2005,32(3):344 - 344。10.1242 / dev.02085。
- 14.
Krizek BA, Anderson JT:花的大小控制。应用物理学报,2013,32(4):344 - 344。10.1093 / jxb / ert025。
- 15.
李文杰,李志刚,李志刚,李志刚:花大小和比例的遗传调控。中国生物医学工程学报,2005,29(4):513-525。10.1387 / ijdb.051998jw。
- 16.
Bey M, Stüber K, Fellenberg K, Schwarz-Sommer Z, Sommer H, Saedler H, Zachgo S:抗rrhinum花瓣发育特征及B类MADS box基因靶基因鉴定DEFICIENS.植物学报,2004,16:3197-3215。10.1105 / tpc.104.026724。
- 17.
文森特·CA,科恩·ES:植物花发育的时间和形态框架金鱼草majus.机械工程学报,2004,29(3):344 - 344。10.1139 /当- 042。
- 18.
Izhaki A, Borochov A, Zamski E, Weiss D:赤霉素调控矮牵牛花药小孢子发生后过程。中国生物医学工程学报,2002,29(3):344 - 344。10.1034 / j.1399-3054.2002.1150314.x。
- 19.
Martin C, Gerats T:花瓣发育过程中色素生物合成基因的控制。中国生物医学工程学报,2003,23(4):357 - 357。10.1105 / tpc.5.10.1253。
- 20.
科斯格罗夫DJ:植物细胞壁的扩张生长。植物化学学报,2000,38(1/2):109-124。10.1016 / s0981 - 9428(00) 00164 - 9。
- 21.
杨晓明,杨晓明,杨晓明,杨晓明。植物发育过程中细胞分裂与分化的关系。中国生物医学工程学报,2005,29(4):457 - 457。10.1387 / ijdb.052001er。
- 22.
杨晓明,杨晓明,杨晓明,杨晓明,杨晓明,杨晓明。植物器官大小的调控作用KLUH / CYP78A5依赖细胞间信号。生物工程学报,2007,29(3):344 - 344。10.1016 / j.devcel.2007.10.001。
- 23.
Dinneny JR, Yadegari R, Fischer RL, Yanofsky MF, Weigel D:角色锯齿状的侧面器官的形成。科学进展,2004,31(3):344 - 344。10.1242 / dev.00949。
- 24.
胡艳,谢强,蔡新华:拟南芥生长素诱导基因阿哥斯控制侧器官大小。植物学报,2003,15:531 - 531。10.1105 / tpc.013557。
- 25.
Mizukami Y, Fischer RL:植物器官大小控制:AINTEGUMENTA在器官发生过程中调节生长和细胞数量。中国科学:自然科学,2000,30(4):344 - 344。10.1073 / pnas.97.2.942。
- 26.
李勇,郑林,Corke F, Smith C, Bevan MW:种子终粒和器官大小的控制DA1基因家族拟南芥.基因开发,2008,22:1331-1336。10.1101 / gad.463608。
- 27.
Disch S, Anastasiou E, Sharma VK, Laux T, Fletcher JC, Lenhard M: E3泛素连接酶BIG BROTHER控制拟南芥器官大小以剂量依赖的方式。中国生物医学工程学报,2006,16:272-279。10.1016 / j.cub.2005.12.026。
- 28.
徐锐,李勇:介导复合亚基25对终器官大小的控制拟南芥.发展,2011,138:4545-4554。10.1242 / dev.071423。
- 29.
Varaud E, Brioiudes F, Sz°Csi J, Leroux J, Brown S, Perrot-Rechenmann C, Bendahmanea M:生长素反应因子8调控拟南芥通过与bHLH转录因子BIGPETALp相互作用来促进花瓣生长。植物学报,2011,23:973-983。10.1105 / tpc.110.081653。
- 30.
Sz°Csi J, Joly C, Bordji K, Varaud E, Cock M, Dumas C, Bendahmane M:BIGPETALp,一个bHLH转录因子参与控制拟南芥花瓣大小。中国生物工程学报,2006,26(3):349 - 349。10.1038 / sj.emboj.7601270。
- 31.
胡勇,保敏,蔡恩华:拟南芥ARGOS-LIKE基因调节器官生长过程中的细胞扩张。植物学报,2006,47:1-9。10.1111 / j.1365 - 313 x.2006.02750.x。
- 32.
管生,陆勇:通过rna测序分析器官特异性转录组。工厂方法2013年,42。
- 33.
Bräutigam A, Kajala K, Wullenweber J, Sommer M, Gagneul D, Weber KL, Carr KM, Gowik U, Mass J, Lercher MJ, Westhoff P, Hibberd JM, Weber APM:从密切相关的C3和C4物种的比较转录组中获得C4光合作用的mRNA蓝本。中国生物医学工程学报,2011,29(3):344 - 344。10.1104 / pp.110.159442。
- 34.
沙尔玛N, Bhalla PL, Singh MB:苔类转录因子家族的转录组分析与表达分析,Marchantia polymorpha.BMC基因组学2013年,14:915。
- 35.
Thimm O, Bläsing O, Gibon Y, Nagel A, Meyer S, Krüger P, Selbig J, Müller LA, Rhee SY, Stitt M: MAPMAN:一种用户驱动的工具,可以将基因组学数据集显示在代谢途径和其他生物过程的图表上。植物学报,2004,37:914-939。10.1111 / j.1365 - 313 x.2004.02016.x。
- 36.
教授,朱涛:基于浏览器的功能分类SuperViewer拟南芥基因组学。Curr compput Mol生物学2003年,271 - 272。
- 37.
史密思D,鲍曼J,梅耶罗维茨E:植物早期花发育拟南芥.植物科学学报,1999,25(2):366 - 366。10.1105 / tpc.2.8.755。
- 38.
Al-Shahrour F, Dfaz-Uriarte R, Dopazo J: FatiGO:一个发现基因本体术语与基因组显著关联的网络工具。生物信息学,2004,20:578- 588。10.1093 /生物信息学/ btg455。
- 39.
曹静:果胶裂解酶拟南芥:进化、选择和表达谱。公共科学学报,2012,7 (10):e46944-10.1371/journal.pone.0046944。
- 40.
Kanneganti V, Gupta AK:植物壁相关激酶概述。生物化学学报,2008,14(1&2):109-118。10.1007 / s12298 - 008 - 0010 - 6。
- 41.
曹军,史峰:中国历史的演变拉尔夫植物基因家族:基因复制与选择模式。生物信息学杂志,2012,8:271-292。10.4137 / EBO.S9652。
- 42.
Pearce G, Moura DS, Stratmann J, Ryan CA: RALF是一种在植物中普遍存在的抑制根系生长发育的多肽。中国科学院学报,2001,29(3):344 - 344。10.1073 / pnas.201416998。
- 43.
De Craene J-O, Ripp R, Lecompte O, Thompson JD: ENTH/ANTH/VHS蛋白超家族的进化分析揭示了膜运输和代谢之间的共同进化。BMC基因组学2012年,13:297。
- 44.
Schindelmann G, Morikami A, Jung J, Baskin TI, Carpita NC, Derbyshire P, McCann MC, Benfey PN: COBRA编码了一种假定的gpi固定蛋白,该蛋白在细胞定向扩张中具有极性定位拟南芥.基因工程学报,2001,29(4):344 - 344。10.1101 / gad.879101。
- 45.
李松,葛凤荣,徐敏,赵晓燕,黄国强,周丽珍,王金光,Kombrink A, McCormick S,张xs,张艳:拟南芥眼镜蛇样10蛋白是一种gbi锚定蛋白,介导花粉管定向生长。植物学报,2013,29(4):344 - 344。10.1111 / tpj.12139。
- 46.
黄志刚,李志刚,李志刚。p-糖蛋白在植物生长发育中的作用。清华大学学报(自然科学版),2006,30(4):344 - 344。10.1016 / j.febslet.2005.11.054。
- 47.
Bartina I, Otto E, Strnad M, Werner T, Schmülling T:细胞分裂素调节植物生殖分生组织的活性、花器官大小、胚珠形成和种子产量拟南芥.植物学报,2011,23:69-80。10.1105 / tpc.110.079079。
- 48.
Hiroi N, Ito T, Yamamoto H, Ochiya T, Jinno S, Okayama H: Rcd1是介导维甲酸诱导细胞分化的一种新的转录辅因子。EMBO。2002, 21(19): 5235-5244。10.1093 / emboj / cdf521。
- 49.
李霞,罗娟,闫涛,向亮,冯洁,秦东,孙超,谢敏:基于深度测序的植物生长特征分析兰花ensifolium植物转录组。公共科学学报,2013,8 (12):e85480-10.1371/journal.pone.0085480。
- 50.
刘智,马琳,南智,王勇:比较转录谱分析为研究植物双形花的进化和发育提供了新的思路野豌豆属漂白亚麻纤维卷(Papilionoideae)。公共科学学报,2013,8 (2):e57338-10.1371/journal.pone.0057338。
- 51.
Soeraert SSA, Neste CMF, Vandewoestyne ML, Head SRm Goossens A, van Nieuwerburgh CW, Degorce DLD:植物腺状毛状体和丝状毛状体的差异转录组分析青蒿.BMC植物生物学2013年,13:220。
- 52.
Zik M,爱尔兰VF:调控的靶基因的全局识别APETALA3而且PISTILLATA花同源异型基因作用。中国生物医学工程学报,2003,29(3):344 - 344。10.1105 / tpc.006353。
- 53.
Ginglinger J-F, Boachon B, Höfer R, Paetz C, Köllner TG, Miesch L, Lugan R, Baltenweck R, Mutterer J, Ullmann P, Beran F, Claudel P, Verstappen F, Fischer MJC, Karst F, Bouwmeester H, Miesch M, Schneider B, Gershenzon J, Ehlting J, Werck-Reichhart D:基因共表达分析揭示了单萜醇芳醇的复杂代谢拟南芥花。中国生物医学工程学报,2013,29(4):357 - 357。10.1105 / tpc.113.117382。
- 54.
Herken EN, Celik A, Aslan M, Aydinlik N:麻粉菊精油的抗菌和抗氧化活性。& Hausskn。木香。来自东安纳托利亚。中国医药食品杂志,2012,15(9):835-839。10.1089 / jmf.2011.0315。
- 55.
Schommer C, Palatnik JF, Aggarwal P, Chételat A, Cubas P, Farmer EE, Nath U, Weigel D: miR319靶点对茉莉酸盐生物合成和衰老的控制。公共科学图书馆,2008,6 (9):e230-10.1371/journal.pbio.0060230。
- 56.
Bell E, Creelmann RA, Mullet JE:创伤诱导茉莉酸积累需要叶绿体脂氧合酶拟南芥.自然科学进展,2004,27(3):377 - 377。10.1073 / pnas.92.19.8675。
- 57.
Pauwels L, Morreel K, De Witte E, Lammertyn F, van Montagu M, Boerjan W, Inzé D, Goossens A:茉莉酸甲酯介导的代谢产物转录重编程与培养细胞周期进程拟南芥细胞。中国科学:自然科学,2008,29(2):366 - 366。10.1073 / pnas.0711203105。
- 58.
Lampugnani ER, Kilinc A, Smyth DR:花瓣损失在发育中的萼片之间是否存在抑制生长的边界基因拟南芥.植物学报,2012,29(3):344 - 344。10.1111 / j.1365 - 313 x.2012.05023.x。
- 59.
李志刚,李志刚,李志刚。植物生长素控制植物花瓣形成拟南芥.发展,2013,40(1):1 - 4。10.1242 / dev.084582。
- 60.
Noh B, Murphy AS, Spalding EP, Cosgrove DJ:多药耐药性类基因拟南芥生长素运输和生长素介导的发育所必需的。植物细胞壁学报,2005,6:850- 850。
- 61.
科斯格罗夫DJ:植物细胞壁的生长。自然科学进展,2005,30(6):366 - 366。10.1038 / nrm1746。
- 62.
Peaucell A, Braybrook S, Höfte H:植物细胞壁力学和生长控制:果胶的作用再探讨。2012,植物科学前沿
- 63.
陈建平,陈建平,陈建平。木葡聚糖内转糖基酶的基因多样性和酶的潜在壁修饰功能。植物科学进展,1999,4(9):361-366。10.1016 / s1360 - 1385(99) 01468 - 5。
- 64.
Urbanowicz BR, Bennett AB, del Campillo E, Catalá C, Hayashi T, Henrissat B, Höfte H, McQueen-Mason SJ, Patterson SE, Shoseyov O, Teeri TT, Rose JKC:糖基水解酶家族的植物endo1 -4-ß-葡聚糖酶(纤维素酶)的结构组织和标准化命名法9。中国农业科学,2007,29(4):344 - 344。10.1104 / pp.107.102574。
- 65.
Sato S, Kato T, Kakegawa K, Ishii T, Liu Y- g, Awano T, Takabe K, Nishiyama Y, Kuga S, Sato S, Nakamura Y, Tabata S, Shibata D:膜结合内-1,4-葡聚糖酶KORRIGAN在细胞延伸和纤维素合成中的作用拟南芥.植物细胞生理学杂志,2001,42(3):251-263。10.1093 /卡式肺囊虫肺炎/ pce045。
- 66.
Mingossi FB, Matos JL, Rizzato AP, Medeiros AH, Falco MC, Silva-Filho MC, MourA DS:草甘蔗肽信号SacRALF1 (糖Spp .)可能参与组织扩张的调节。中国生物医学工程学报,2010,29(3):344 - 344。10.1007 / s11103 - 010 - 9613 - 8。
- 67.
Covey PA, Subbaiah CC, Parsons RL, Pearce G, Lay FT, Anderson MA, Ryan CA, Bedinger PA:番茄花粉特异性RALF调节花花管伸长。中国生物医学工程学报,2010,29(3):344 - 344。10.1104 / pp.110.155457。
- 68.
Reale L, Porcessu A, Lanfaloni L, Moretti C, Zenoni S, Pezzotti M, Romano B, Ferranti F:植物花瓣发育过程中细胞分裂和扩张的模式佩妮矮牵牛.植物生殖学报,2002,15:123-132。10.1007 / s00497 - 002 - 0150 - 8。
- 69.
克劳福德BCW, Nath U, Carpenter R, Coen ES:CINCINNATA控制莲叶和叶的细胞分化和生长。植物科学学报,2004,27(3):344 - 344。10.1104 / pp.103.036368。
- 70.
Kosugi S, Ohashi Y: TCP蛋白家族的DNA结合和二聚化特异性和潜在靶点。植物学报,2002,30(3):337-348。10.1046 / j.1365 - 313 x.2002.01294.x。
- 71.
Peaucelle A, Louvet R, Johansen JN, Salsac F, Morin H, Fournet F, Belcram K, Gillet F, Höfte H, Laufs P, Mouille G, Pelloux J:转录因子BELLRINGER通过调节果胶甲基酯酶的表达来调节叶向性拟南芥.发展,2011,138:4733-4741。10.1242 / dev.072496。
- 72.
Rutjens B, Bao D, van Eck-Stouten E, Brand M, Smeekens S, Proveniers M:拟南芥芽尖分生组织功能需要三种bel1样同源结构域蛋白的联合活性。植物学报,2009,38(4):344 - 344。10.1111 / j.1365 - 313 x.2009.03809.x。
- 73.
Smith HMS, Hake S:两个同源盒基因的相互作用BREVIPEDICELLUS而且小精明的节点间模式拟南芥花序。植物科学学报,2003,22(3):344 - 344。10.1105 / tpc.012856。
- 74.
邓丽娟,张志刚,张志刚:SAGE基因的差异表达及其对基因库间变异的影响。生物信息学,2003,19(12):1477-1483。10.1093 /生物信息学/ btg173。
- 75.
Rozen S, Skaletsky HJ:面向普通用户和生物学家程序员的WWW上的Primer3。生物信息学方法与协议:分子生物学方法。编辑:Krawetz S, Misener S. 2000, Humana出版社,托托瓦(新泽西州),365-386。
- 76.
Winter D, Vinegar B, Nahal H, Ammar R, Wilson GV, Provart NJ:用于探索和分析大规模生物数据集的“电子荧光象形文字”浏览器。公共科学学报,2007,2 (8):e718-10.1371/journal.pone.0000718。
确认
我们非常感谢Claudia Gieshoidt在收获植物材料、qPCR和植物栽培方面的帮助。我们也非常感谢库尔特博士Stüber在科隆育种研究,他对BLAST分析的友好帮助IberisContigs对拟南芥蛋白质数据库。这项研究得到了德国Forschungsgemeinschaft的支持
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
AB和SZ设计了项目和实验装置。AB进行RNA-Seq数据分析,qPCR和原位杂化。SH进行微阵列分析和RT PCR。AB和SZ对数据进行解读并撰写稿件。所有作者都阅读并批准了手稿。
电子辅助材料
所有1600人的名单
附加文件1:Iberis中有显著表达的基因IberisA1期花瓣正面和背面。名单包括所有Iberis amara在花瓣中有显著表达的转录本,即在“Bonferroni检验”一栏中,乘法误差的p值低于0.01。所有Iberis基因,由各自的contig数表示,被分配到AGI代码,基于对基因的blast搜索拟南芥蛋白质数据库。从搜索中获得的e值列在“BLAST命中”列中。只有e值截断值为e的基因4列出。每一个Iberis-gene由其各自AGI代码的注释和MapMan分类描述。每个基因在正轴(aD)和反轴(aB)花瓣中的表达用RPKM值(每千碱基每百万的映射reads)描述,并总结为折叠变化。(xl2020kb)
背轴和正面5%高表达基因列表
附加文件2:Iberis amaraA1期花瓣。作为附加文件的子集1,此表列出所有Iberis amara花瓣转录本,是背轴和正面A1期花瓣中表达量最多的5%基因之一。每一个Iberis抄本由其对应的AGI代码、Annotation和MapMan bin表示。此外,还给出了相应的RPKM值和折叠变化值。(xlsx49 kb)
A1期前后基因表达差异
附加文件3:Iberis花瓣。作为附加文件的子集1,该列表包含所有334个转录本,由各自的contig数表示,两种花瓣类型之间的表达折叠变化最小为2。为每一个Iberis给出了一个AGI代码,并给出了相应的注释和MapMan特征。用RPKM和折叠变化值描述花瓣正面(aD)和背面(aB)的基因表达。“Bonferroni测试”和“BLAST命中”列出了乘法错误的p值和由爆炸搜索产生的e值IberisContigs对拟南芥蛋白质数据库。(xlsx78 kb)
12870 _2014_285_moesm4_esm.tiff
附加文件4:光合作用相关基因不促成单对称性的形成。选择磷酸激酶作为光合作用相关过程的代表,以验证RNA-Seq数据。(A)从三个独立的生物样本中比较了正面花瓣和背面A1期花瓣的表达(重复5-7)。(B, C)将取自两个生物重复(重复1和2)的正面和背面花瓣解剖为上部花瓣叶片和下部花瓣爪。(B)比较了正面花瓣和背面花瓣叶片和爪部磷酸激酶的表达。(C)比较两种花瓣类型的花瓣叶片和花瓣爪之间磷酸化激酶转录本的丰度。误差条是来自三个技术重复的标准误差。aBp,花瓣背面;aBpb,花瓣叶片背面;aBpc,背面花瓣爪; aDp, adaxial petal; aDpb, adaxial petal blade; aDpc, adaxial petal claw. (TIFF 184 KB)
的常见假定靶基因
附加文件5:IaTCP1在Iberis amara花瓣和异位表达的IaTCP1, TCP1而且赛克在拟南芥花序。列出了在A1期花瓣的正面和背面差异表达(≥2倍)的基因Iberis amara在植物中表达这两种IaTCP1, TCP1或赛克在控制之下35个年代启动子的拟南芥与携带空载体的植物相比。-或+表示激活或抑制。粗体字体突出了与细胞壁过程相关的基因。(pdf 66kb)
对异位表达有差异调节的600个基因列表
附加文件6:IaTCP1在转基因拟南芥.差异表达基因来自转基因芯片分析拟南芥花序携带年轻的花蕾,异位表达IaTCP1在控制之下35 s -启动子。与空载体的转基因植物相比,差异表达被推导出来。所有600个基因都表现出最小的表达折叠变化为2,p值等于或小于0.05。对于每个探测集标识符,给出了各自的AGI代码、Annotation、MapMan bin和折叠变化值(红色)。此外,利用转基因植株过表达的花序材料进行杂交分析,得到相应的折叠变化值(和p值)TCP1而且赛克列出。(xlsx199 kb)
对异位表达有差异调控的328个基因列表
附加文件7:TCP1在转基因拟南芥.差异表达基因来自转基因芯片分析拟南芥花序携带年轻的花蕾,异位表达TCP1在控制之下35 s -启动子。与空载体的转基因植物相比,差异表达被推导出来。所有328个基因都表现出最小的表达折叠变化为2,p值等于或小于0.05。对于每个探测集标识符,给出了各自的AGI代码、Annotation、MapMan bin和折叠变化值(蓝色)。此外,利用转基因植株过表达的花序材料进行杂交分析,得到相应的折叠变化值(和p值)IaTCP1而且赛克列出。(xlsx76 kb)
引物列表。
附加文件8:用于确认RNA-Seq数据的引物列表Iberis amara并在微阵列中表达数据拟南芥.(pdf 75kb)
微阵列表达数据确认。
附加文件9:由于异位活性而差异表达的28个基因的微阵列表达数据IaTCP1,TCP1或赛克在拟南芥花序。利用半定量RT - PCR技术对基因表达数据进行了验证IaTCP1或赛克.在所有三个重复中,最小表达倍数变化≥2表示为+。(pdf 52 kb)
564个基因的表差异调节异位表达
附加文件10:赛克在转基因拟南芥.差异表达基因来自转基因芯片分析拟南芥花序携带年轻的花蕾,异位表达赛克在控制之下35 s -启动子。与空载体的转基因植物相比,差异表达被推导出来。所有564个基因的最小表达折叠变化为2,p值等于或小于0.05。对于每个探测集标识符,给出了各自的AGI代码、Annotation、MapMan bin和折叠变化值(绿色)。此外,利用转基因植株过表达的花序材料进行杂交分析,得到相应的折叠变化值(和p值)IaTCP1而且TCP1列出。(xlsx106 kb)
的
附加文件11:Iberis amaraA1期花瓣转录组。此列表包含所有新创组装52081Iberis amaraA1期花瓣序列和相应的contig数。(zip 11mb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。
本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。
如欲查看本牌照的副本,请浏览https://creativecommons.org/licenses/by/4.0/.
创作共用公共领域奉献弃权书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
Busch, A., Horn, S. & Zachgo, S.差异转录组分析揭示了十字花科植物单对称花冠发育的洞察Iberis amara.BMC植物生物学14,285(2014)。https://doi.org/10.1186/s12870-014-0285-4
收到了:
接受:
发表:
关键字
- 十字花科
- 单轴对称
- 赛克
- TCP1
- RNA-Seq
- 微阵列