摘要
背景
几十年来,利用野生番茄种质资源进行的密集育种已经产生了驯化种质资源的基因组(茄属植物lycopersicum)与它们的野生亲戚的渗透交织在一起。比较分析有遗传变异的栽培番茄和野生番茄的基因组可以帮助确定理想的基因,比如那些赋予抗病能力的基因。识别渗入位置、边界和内容的能力可以揭示祖先的起源,并有助于在作物育种中利用野生变异。
结果
本文报道了两个番茄自交系Gh13和BTI-87的全基因组序列,这两个自交系都携带有豆状病毒抗性位点Ty-3从野生番茄品种中渗入的。Gh13和BTI-87的6号染色体均有不同大小的渐渗,均对应于Ty-3在该地区,被鉴定为来自一个来源接近野生的物种美国chilense.其他渐渗现象在自交系的基因组中被发现,并在两个系的育种系谱中显示出重大差异。有趣的是,来自番茄近亲的额外大量的基因渗入美国pimpinellifolium在两行都被识别。一些多态区域被认为是参考Heinz 1706基因组的渐进,表明参考番茄基因组中的野生基因组序列。
结论
在这项工作中开发的方法可以用来描绘基因组渐渗入,并随后有助于分子标记的发展,以帮助表型选择,精细定位和发现重要表型的候选基因,并鉴定番茄改良的新变异。这些通用方法可以很容易地应用于其他作物。
背景
现代植物育种的一个重点是引进理想性状的新变异;生物和非生物胁迫是提高产量和提供可靠粮食生产的最关键因素。番茄(茄属植物lycopersicum)是一种重要的粮食作物,也是研究肉质果实成熟、果实发育等过程的模式物种[1],以及抗病的分子基础[2]、[3.]。
番茄原产于南美洲的安第斯山脉、沙漠和沿海平原[4]。在将番茄从其原始野生物种驯化的过程中,番茄基因组经历了一个遗传瓶颈,将其遗传多样性降低到不到其最近的野生亲缘物种多样性的5% [5]、[6]。此外,人类对与产量和果实质量相关的性状,如大小、重量、颜色、糖含量和保质期的选择,忽视了抗病性性状。因此,番茄传家宝品种易受多种病原体的影响,包括细菌、病毒、真菌、线虫和害虫,抗性等位基因仅存在于野生番茄亲缘种[7]。由于这些物种可以与栽培品种异交,自1917年以来,育种者已将野生基因组渗入栽培品种[8]、[9],这种做法一直延续至今[7]。大多数抗病基因都是从野生物种如茄属植物chilense[10] - [12),美国peruvianum[13] - [15),年代.habrochaites[16),美国pennellii[17),而美国pimpinellifolium[7]、[18]。
海湾病毒是影响热带和亚热带地区番茄的主要疾病。症状各不相同,但都涉及一定程度的叶片变形和生长和产量下降[19] - [21]。控制由生豆病毒引起的番茄病害的管理策略传统上侧重于昆虫病媒[22]。从三个来源的番茄中,至少有四个基因座已渗入到番茄中美国chilense而且美国habrochaites[11]、[16]、[21]、[23]。
2012年初发布的参考番茄基因组序列(品种Heinz 1706)使许多新的遗传和基因组方法成为可能[24],例如从重新排序的育种系中映射读取。使用作图方法,包含有限数量snp的基因组区域可以有效地与参考序列对齐,并使用配对端测序,可以检测插入和删除。然而,大的插入和高度发散的区域不容易用这种映射方法来描述。需要更多高质量的参考基因组从头组装,特别是野生种质资源,以分析利用现有资源无法绘制的重新测序的基因组区域[25]。
由于几乎所有的番茄抗病基因都来自野生亲缘,进一步了解这些基因组将有助于多种抗病基因渗透到优良品种中。此外,虽然所有的番茄品种都有大量的同位基因组,并且可以异交,但参考基因组的基因组含量甚至与其他商业番茄品种也不完全相同。例如,水果形状基因太阳已在某些品种中复制,但在亨氏1706 (H1706)中不存在其功能性副本[26]。另一个例子是细菌抗性基因美国专利商标局这是从野生番茄物种中渗入的,美国pimpinellifolium,在20世纪30年代和后来的位置克隆[2]、[27]。在H1706中也缺少该基因的功能版本。
将野生物种基因组区域渗入驯化物种是增加番茄和其他作物物种多样性的一种广泛使用的做法[28]。经过几代的回交和选择,携带有利性状的更大的渐渗,以及隐性渐渗,出现在整个基因组中。虽然番茄有很好的基因图谱[29],许多可用的地图不是很密集,不能精确地定义渗入点。选择过程可能伴随着连锁-拖累,产生具有紧密联系的有害等位基因的基因组,这需要许多轮回交和精细映射来消除[30.]。因此,定义野生物种渐进的边界和内容的能力可以显著减少选择有利等位基因所需的代数,同时最大限度地减少负面变异。基因渗入的鉴定可以帮助鉴定对抗病等有益性状负责的候选基因[31]。
其他作物,如玉米、水稻、大麦[32], bean [33],和瓜[34],表现出与番茄相似的野生渗入模式。这些基因组和番茄的基因组[35],最近已经使用高密度SNP芯片进行了研究。然而,尽管这些技术在检测种群特征和揭示种群结构方面非常出色[36],它们在界定渗透边界及其内容方面的信息量较少。另一方面,全基因组测序方法通过与参与育种过程的野生物种的基因组进行比较,提供了有关基因含量和渐渗区域起源的更详细信息[37]。最近发表了与番茄基因组重测序相关的其他工作,并证明了在驯化番茄系中SNP是如何调用的,可以揭示由于野生渐入导致的驯化种质之间的实质性差异[38]。对番茄种质的重新测序也被用于全基因组关联研究(GWAS),以将SNPs与农艺上的重要性状联系起来[39]。
在这项研究中,选择了两个抗begomovirus的自交系,Gh13 [40]和BTI-87 (D.P. Maxwell,未发表的数据),这些数据被认为来自不同的资料。Gh13是在危地马拉开发的[41经过多个季节的测试,始终表现出对高begomavirus pressure的非常好的抵抗力。到目前为止,Gh13的抗性大概来自于美国habrochaites[42]。BTI-87也是在危地马拉开发出来的,它保持了来自具有begomovirus抗性的近交系Gc171的高水平抗性美国chilense加入LA1932 [43]。两个自交系都携带aTy-3抗性等位基因,以及其他几个抗性基因来自几个野生接入源。
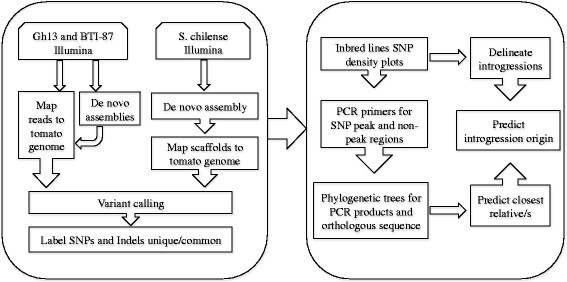
我们使用全基因组测序(WGS)检测了来自野生物种的两个抗begomovirus自交系的渐渗入。确定了渗进的边界,并确定了几个渗进的来源(图1).这些发现为从育种项目中获得的番茄自交系的基因组结构提供了深入的了解,并证明了育种如何从WGS中获得极大的好处,这可以减少耗时的表型筛选。
结果
测序与装配
Gh13和BTI-87基因组的配对端文库分别在Illumina HiSeq通道中测序。将Gh13基因组映射到参考番茄H1706基因组,在去除低质量reads和重复序列后,H1706基因组的覆盖率为14.7倍,参考基因组的覆盖率为97.6%。Gh13基因组的缺口估计跨越9.2 Mb, SNPs总数为288,640个(表2)1).将BTI-87基因组定位到参考番茄基因组的覆盖率为32.3×,代表了H1706基因组的96.5%,装配中存在79.9 Mb的缺口,有702560个SNPs(表2)1),与参考番茄基因组相比,有77,652个与Gh13共享snp。
Gh13和BTI-87的覆盖深度差异(分别为14.7×和32.3×)主要归因于基因组DNA的质量。与Gh13相比,BTI-87的DNA文库的质量更高,因为它包含更少的精确重复reads。覆盖范围的差异并不影响将reads映射到参考基因组的能力,以及使用相同的标准以高可信度调用snp的能力。这些基因组产生了相似的基因组覆盖率水平(97.6%和96.5%),但Gh13的覆盖率略高,因为它比BTI-87具有更少的snp和缺口,这主要是由于野生物种的渗透区域更少。
Gh13和BTI-87基因组序列均可在Sol基因组网络(SGN;http://solgenomics.net).两个基因组中snp的位置都可以在基因组浏览器轨道中找到,并可用于设计新的标记。
单核苷酸多态性分布
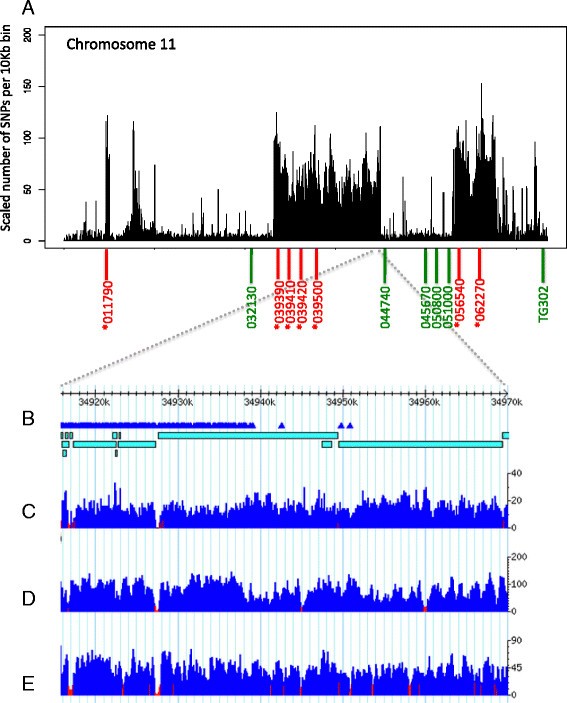
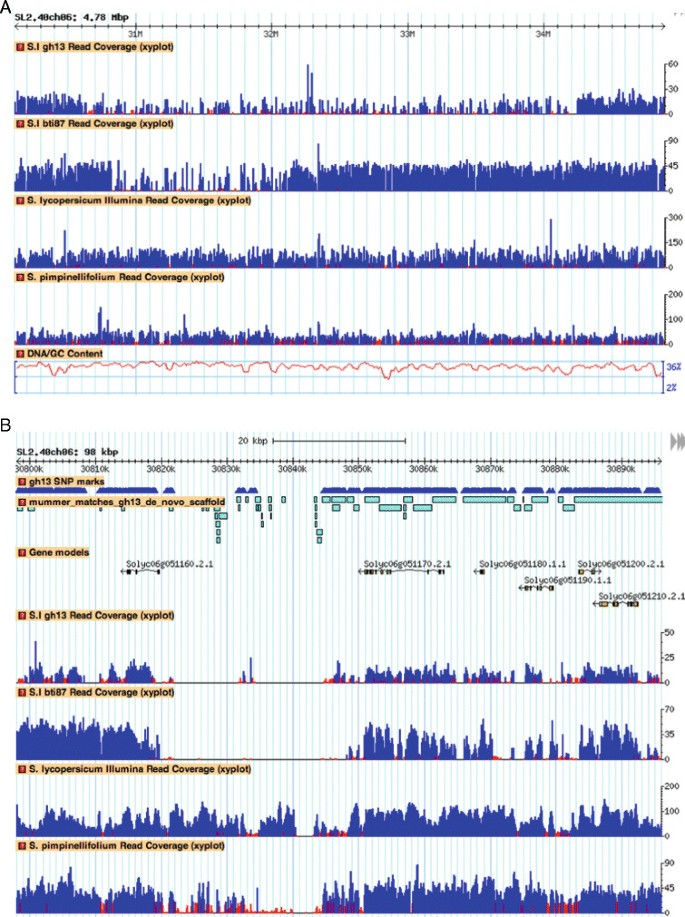
在Gh13的6号染色体上有较大的SNP密度峰区,跨越了Ty-3地区(21] (30.6-34.22 Mb;数字2一个;额外的文件1:图S1),表明该SNP分析方法可以有效地识别渐渗的基因组区域。此外,我们在BTI-87系中发现了一个具有Ty-3a轨迹从美国chilenseLA1932。BTI-87在6号染色体上也有类似的SNP密度峰值,在染色体周围的区域较小,为1.33 MbTy-3基因座区域(30.81-32.14 Mb。附加文件2:图S2)。
我们还在整个Gh13基因组中发现了许多其他不同的SNP密度峰值区域,其中最显著的是在11号染色体上,有两个较大的峰值区域,跨度为11.76 Mb (23.18-34.94 Mb)和4.49 Mb (43.18-47.67 Mb)(图18)3.一个;额外的文件3.:表S1)。在第4号染色体(2.17 Mb和2.11 Mb)、第7号染色体(1.29 Mb)和第10号染色体(1.79 Mb)上发现了其他显著的SNP峰值区域。在所有染色体上都发现了其他候选SNP峰值区域,长度在50 Kb到11.76 Mb之间2).我们将SNP峰值定义为在5个或更多连续的10 Kb窗口中有10个或更多SNP的区域,允许高达40 Kb的间隙,包括由于读取数量不足或无法映射到参考基因组中的区域而可能具有低覆盖率的区域,同时不允许最大间隙大小超过最小SNP峰值大小50 Kb。我们的目标是通过定义最小窗口大小为50 Kb来测试是否有可能揭示相对较小的渐渗。采用H1706基因组分析中使用的150 Kb标准[24],将只得到Gh13中32个snp峰值区域,而忽略了许多snp数量显著较高的区域。为了测试为定义snp -峰值区域而选择每10 Kb窗口的最小SNPs数量的截止点,我们计算了Gh13整个基因组中每10 Kb窗口的平均SNPs数量,并将其与非峰值区域的平均SNPs数量进行比较,使用每10 Kb的最小数量3,5,10,15和20个SNPs来调用峰值区域。我们的统计分析表明,当使用最小数量的3和5个SNPs时,整个基因组的平均SNPs数量与非峰值区域没有显著差异(p?
使用这些标准识别的snp峰值区域总数为144个,跨度49.42 Mb,共有171,711个snp,其中94个区域为100kb或更大(表2)2;额外的文件3.:表S1)。使用相同的标准来调用BTI-87中的SNP峰,我们还在其基因组中检测到146个区域,跨度为150.16 Mb,总共有641,454个SNP(表2)2;额外的文件4:表S2)。苏格兰国民党的高峰侧翼Ty-36号染色体上的位点区域为1.33 Mb。两个基因组中snp分布的显著差异是在BTI-87的4、6和9号染色体上检测到大量的渐进(4号染色体上11个区域共48.89 Mb, 6号染色体上47个区域共18.51 Mb, 9号染色体上10个区域共53.39 Mb)。
检测假定的渗透
为了鉴定潜在的基因渐渗,我们鉴定了Gh13和参考基因组之间的snp,并发现了与参考基因组显著不同的区域(番茄SL2.40基因组构建,http://solgenomics.net/organism/Solanum_lycopersicum/genome).这些区域可能表明在被分析基因组或参考基因组中存在渐渗。通过绘制Gh13和BTI-87基因组中10 Kb窗口内snp的数量,确定了基因组中可能来自野生物种的潜在渗入的一些区域(附加文件)1:图S1,附加文件2:图S2)。
为了验证高SNP密度区域对应野生物种渐进的假设,将每个自交系Gh13和BTI-87之间的SNP与参考番茄基因组中的SNP进行比较美国pimpinellifoliumLA1589 [24]和传家宝品种黄梨(Yellow Pear, YP)。美国pimpinellifolium是驯化番茄的近亲,美国lycopersicum[4],而参考番茄基因组H1706有一个美国pimpinellifolium背景中的父元素[24]、[44]。因此,我们期望从美国pimpinellifolium在参考番茄基因组中,也可能来自其他野生物种。YP没有显示任何野生物种渗入的痕迹[37]。因此,任何在YP和H1706之间显示高密度snp的区域都可能表明H1706中不起源于此的区域美国lycopersicum,并且很可能是在这个品系的繁殖过程中渗入的[24]、[44]。Gh13和BTI-87的SNP密度图显示了每个基因组与参考番茄基因组之间存在重大差异的区域,但仅从这一信息无法确定SNP峰值是否代表自交系或H1706基因组的渐进。通过测定Gh13和美国pimpinellifolium,就有可能预测Gh13中哪些元素的渗入最有可能来自美国pimpinellifolium.在Gh13和YP (Gh13 X YP)之间共享但在H1706 (H1706 X Gh13和H1706 X YP)中不同的SNP峰区域最有可能代表H1706基因组中的野生基因渗入。
Gh13中SNP的峰值区域与YP中的峰值不对应美国pimpinellifolium基因组,可以指定为Gh13的渐进源于不同的野生物种(附加文件3.:表S1)。H1706不是无渐渗的,包含来自的渐渗美国pimpinellifolium[24]、[44可能还有其他野生物种。我们在Gh13个snp峰区域检测到与YP共享snp(在144个检测到的候选渐渗区域中有60个)。因为YP没有野生基因渗入,被认为是100%?美国lycopersicum基因组(37我们可以推断近交系Gh13的这些区域对应于无渐渗的区域美国lycopersicum基因组(附加文件)3.:表S1;表格2).例如,在Gh13的10号染色体上,15个SNP峰值区域中有5.18 Mb与YP共享,而与YP不共享美国pimpinellifolium,表明所有这些区域都是H1706的未知野生物种的渗透,而这些物种在其谱系中没有记录[44]。血统起源也不总是可靠的,正如我们已经证明了Ty-3基因在Gh13系,据报道有美国habrochaites作为阻力的来源,但Ty-3基因座是从美国chilense这一点并没有记录在该品系的谱系中。
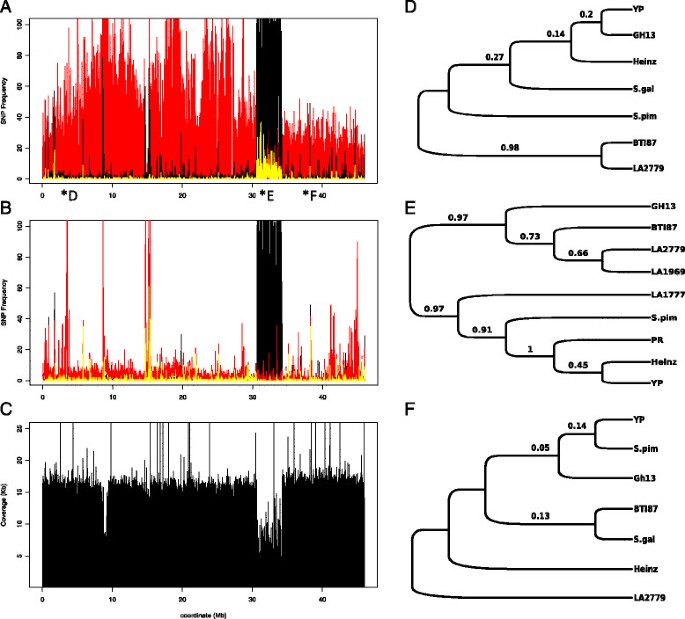
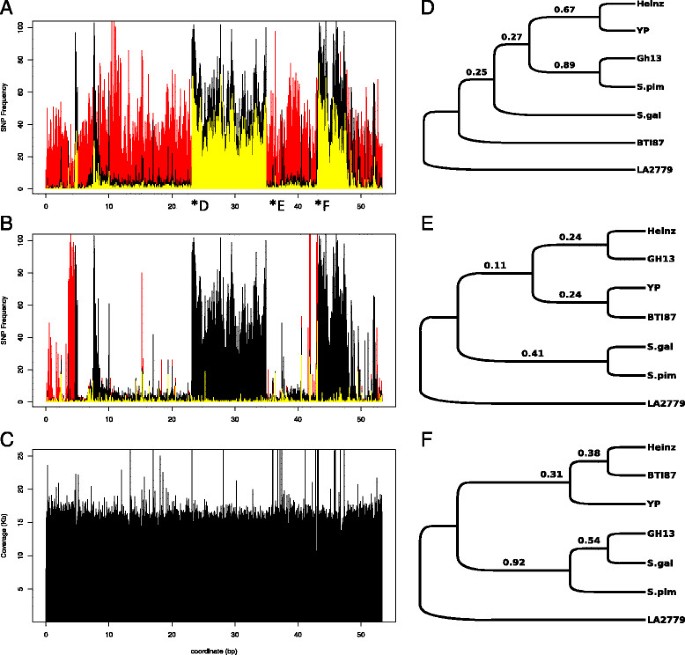
在Gh13的6号染色体上检测到SNP峰值(图2A)和BTI-87(附加文件2:图S2)与的SNPs也没有明显重叠美国pimpinellifolium或与YP的杂交,表明这些是其他野生物种的渗透美国pimpinellifolium(图4一个;额外的文件3.:表S1)。Gh13系的11号染色体显示了三个不同的区域,我们认为这些区域是遗传的美国pimpinellifolium,因为大多数SNPs在两者之间共享(图5A).相反,BTI-87的11号染色体的SNP渐渗与Gh13的不同(附加文件2:图S2;额外的文件3.:表S1,附加文件4:表S2)。
在Gh13的第4染色体上,我们检测到一个较大的2.17 Mb的渐渗(从53.35 Mb到55.52 Mb),这是最接近美国pimpinellifolium.然而,这种渗透包括一些大小在10到200 Kb之间的片段,YP有大量匹配的snp (10 Kb中超过10个snp)。第4号染色体的第二大SNP峰与美国pimpinellifolium从57.53 Mb到57.91 Mb,紧接着是1.73 Mb区域(57.91 Mb到59.64 Mb),这很可能对应于H1706中由于Gh13和YP之间共享的高SNP密度而导致的基因渐渗(补充文件)3.:表S1)。在YP中一些高SNP密度的区域,尚不清楚Gh13基因渐渗的起源(附加文件)3.:表S1)。需要对每个区域进行进一步的系统发育分析,以澄清其起源。
PCR测序和基因树
为了研究Gh13染色体6和11上每个检测到的SNP峰值区域的起源,设计了PCR引物,用于扩增所选SNP峰值区域内外的片段(图2一个,3.A).对PCR序列进行比对,分析snp(表3.)和indels,用于建立系统发育基因树,包括H1706、传家宝系YP和Purple Russian (PR)、自交系Gh13和BTI-87以及野生种的序列美国pimpinellifolium,美国galapagense,美国chilense,美国habrochaites.
在第6号染色体上,SNP峰值之外的三个选定区域(标记物REX, T0774, TG472;数字2A)显示,正如预期的那样,Gh13序列与来自两者的序列相同美国lycopersicum基因组,H1706和YP,与野生物种非常不同美国chilense而且美国galapagense.Gh13的非峰序列也几乎相同美国pimpinellifolium序列(REX片段有1个SNP,其余两个标记完全相同)(图4A, D, E)。在SNP峰值区域检测的三个标记TG590, T0834, P6_051570(图2A),表明Gh13的序列不同于美国lycopersicumTG590和T0834的基因序列为H1706、YP和Purple Russian美国pimpinellifolium而且美国galapagense.在6号染色体SNP峰区检测的其他野生物种有两个报道的Gh13谱系亲本系美国habrochaites(登记号LA1777及LA0386) [42]和另外两个茄属植物chilenseLA2779和LA1969)被认为是该基因等位基因的来源Ty-3轨迹(21]。系统发育分析表明,Gh13序列总是最接近这两个标记美国chilense登记入册(图4E)而不是预期的野生物种。habrochaites.
类似的方法应用于11号染色体,我们在Gh13基因组中检测到三个候选渐渗区域(图13)3.A). Gh13的SNP图,美国pimpinellifoliumH1706基因组显示Gh13渐渗区大多与美国pimpinellifolium单核苷酸多态性(图5A).正如预期的那样,在3个SNP峰值区域检测的7个标记表明Gh13序列具有最高的同源性美国pimpinellifolium(数据-5D和F)。在非snp峰侧区检测的6个标记均显示Gh13序列与美国lycopersicum基因组H1706和YP(表3.,图5E).将11号染色体上所有13个标记的序列与另外两个野生番茄品种的序列进行比较。美国chilense所有标记的序列大多不同于所有其他基因组序列美国galapagense序列介于美国lycopersicum而且美国pimpinellifolium(数据5D, E,和F;表格3.).
SNP芯片基因分型
包含7,720个SNP标记的SolCAP SNP芯片阵列[45]用于Gh13和HUJ-VF基因分型,HUJ-VF是一种对begomovirus敏感的近交系。我们将在100 Kb内具有3个或更多多态性SNPs的区域定义为候选渐渗,并发现共49个区域,跨度96.76 Mb,有968个多态性SNPs(附加文件)5:表S3),与WGS预测的跨度49.42 Mb的171711个snp相比。在SolCAP芯片检测到的49个渐渗区中,25个至少与Gh13渐渗区有部分重叠,如预期的那样,包括与预测的含有Gh13的6号染色体渐渗区完全重叠Ty-3轨迹。未被WGS检测到的SolCAP渐渗入可能是由于与基因组含量不同的两个不同易感系(H1706和HUJ-VF)比较所致。
讨论
在这项研究中,通过全基因组序列分析(重测序)、SNP调用、PCR测序和系统发育,检测到基因渐渗,并推断其起源。2个番茄自交系(Gh13和BTI-87)在海棠病毒抗性位点上有等位基因Ty-3被用来证明一个已知的渗透为Ty-3可以检测到6号染色体上的基因座,并确定边界(图6该重测序策略提供了参考基因组与重测序系Gh13和BTI-87之间丰富的多态性数据(snp)。为了评估SNP区域,染色体被划分为10 Kb的连续窗口。沿着参考序列绘制每个窗口的SNP频率,揭示了SNP密度较高的区域。这些区域暂时被标记为渐渗区。然而,有许多较小的区域,从40 Kb到几百Kb不等,显示出较高的SNP密度。这些区域可能代表更小的“隐秘”渐渗,或者可能是由于其他因素(如转座子序列)导致的高度分化区域。在不同野生物种的整个Gh13基因组中,总共检测到144个迄今未知的假定基因渗入,大小从50 Kb到超过11 Mb不等,在BTI-87基因组中检测到146个预测基因渗入(范围从50 Kb到42.87 Mb)。
Gh13和BTI-87中Ty-3渐渗的基因组区域。一)6号染色体渐渗的基因组覆盖图(Gh13和BTI-87)。B)放大图中的80kb区域5A,横跨Ty-3区域。
我们在两个自交系中都检测到包含Ty-3位点的6号染色体渐渗入。由于这些抗始形病毒株系的育种谱系大多未知,但它们都来自许多野生番茄品种,我们通过基于PCR片段测序构建系统进化树确定了渐进的起源。我们的结果表明,BTI-87和Gh13的渐渗区域紧密地聚集在一起美国chilense,确定这种野生物种为源头Ty-3轨迹。其他显著的基因渗入在染色体4和11上被检测到,这是它们最有可能的起源美国pimpinellifolium.Gh13和YP之间的SNP峰区高度相似,表明H1706中有来自未知来源或不同来源的渐渗区域美国pimpinellifolium加入。与Gh13相比,BTI-87的SNPs数量增加了一倍以上1;额外的文件2:图S2)归因于染色体4、6和9的大量渐渗。这些结果表明,番茄育种导致了许多来自各种野生物种的隐性渗入。目前的基因组测序技术,加上现有的基因组资源,可以快速发现这种候选渐进,可以进一步帮助育种计划,并促进发现新的遗传变异和基因功能的研究。
渐渗检测的一个重要特性是能够准确地确定其边界。的开始和结束核苷酸的检测能力美国chilense通过提取Gh13基因6号染色体的独特snp,检测Gh13基因的渐渗美国chilense在Gh13基因组中,只选择在其他被测基因组中不存在的唯一snp,覆盖范围大于10×,等位基因频率大于90%。这项分析得出了4931个独特的结果美国chilenseSNP在Gh13基因组中的位置,预测的6号染色体30.6- 34.22 mb区域有148个SNP美国chilense渐渗现象。该区域内的第一个SNP位点位于核苷酸30,620,481,最后一个SNP位点位于核苷酸34,051,365。该分析应与完全测序的参考基因组重复美国chilense和其他野生亲本系,以描绘整个基因组的精确渗入。SolCAP SNP芯片也给出了类似的结果Ty-3基因渐渗(30,623,784到33,972,992个核苷酸);然而,与WGS检测到的超过35000个snp相比,只有29个snp是多态的,因此提供了与渐渗含量相关的更广泛的数据。
的Ty-1而且Ty-3基因座最近被定位到6号染色体的同一区域[21],这在Gh13和BTI-87的6号染色体的渐渗范围内。映射Ty-1而且Ty-3基因座是耗时的,需要在多代选择中进行大量的定位种群[21]。通过重新测序和SNP分析,有可能促进目标基因的精细定位并最终克隆,因为来自野生物种的假定的基因渗入可以很容易地检测到,并可能缩小要筛选的基因组区域。
结论
我们利用H1706参考基因组和其他基因组序列美国pimpinellifolium,美国chilense和YP,以检测两个抗begomovirus自交系的遗传变异,并确定其中一些遗传变异的来源。所发现的基因渗入在大小、位置和内容上差异很大,我们对传家宝系YP的分析表明,许多基因渗入存在于已知的H1706基因组中美国pimpinellifolium在血统上。这些发现强调了对番茄野生种额外基因组序列的需求,这些基因组序列可用于鉴定番茄渐渗入的起源,并研究H1706基因组中可能不存在的基因组序列[46]。此外,本文概述的方法可用于开发特定区域的SNP标记,并确定渐渗的边界。在本报告中,我们的方法代表了一个概念的证明,可以很容易地应用于其他具有可用参考基因组的物种。
方法
植物材料
茄属植物lycopersicum近交系Gh13源自抗tylcv种质FAVI 9 [42]通过在危地马拉Sanarate的田间多代选择单株抗begomovirus植物[41]、[46]。AgBiotech, Inc.通过SNP分析检测Gh13中的抗病基因,结果为:begomovirus抗性位点为纯合子Ty-3在染色体6上;纯合子的已经在9号染色体上;杂合的来I2在11号染色体上,易患心肌梗死,Sw5,Ty2,Ph3,Tm2a,美国专利商标局.PCR片段测序分子扫描显示,Gh13在6号染色体20 ~ 32 cM处有一个渐进位点(C. Martin and D.P. Maxwell, personal communication),这与Gh13基因的位置相对应Ty-3轨迹(47]、[48]。Gh13被用于几个研究项目,以确定该药物的有效性Ty-3赋予生豆病毒抗性的基因座[40]、[49]。
专有的抗begomovirus美国lycopersicumBTI-87是由商业种子公司Semillas Tropicales, S.A.获得的。BTI-87对begomovirus抗性的来源是Gc171自交系,该自交系已知具有Gc171Ty-3a而且Ty-4抗性位点分别在6号染色体和3号染色体上[47]、[50]。这些抗性基因座是从美国chilenseLA1932 [43]。AgBiotech, Inc.通过SNP分析检测BTI-87中的抗病基因,结果为:begomovirus抗性位点为纯合子Ty-3或Ty-3a在染色体6上;杂合的来心肌梗死在染色体6上;基因为纯合子Tm2a在9号染色体上;易受感染I2而且Sw5.
获得的种子美国habrochaitesLA0386和LA1777,美国chilenseLA1932, LA1969, LA2779,还有美国galapagenseLA0436是从加州大学戴维斯分校的番茄遗传资源中心(http://tgrc.ucdavis.edu).
的种子美国lycopersicumH1706 (LA4345)和YP由博伊斯汤普森植物研究所(BTI)的Gregory Martin提供。美国lycopersicum紫色俄罗斯种子可从威斯康星大学麦迪逊分校道格拉斯·麦克斯韦尔的实验室获得。AgBiotech, Inc.的抗性位点SNP检测结果表明美国lycopersicumH1706、YP和紫俄系均有易感位点Ty-3,心肌梗死,I2,Sw5,Tm2a.
DNA提取
Gh13幼苗在威斯康星大学麦迪逊分校种植。采用CTAB法提取DNA [51],可获得约500 ng/ul的基因组DNA,用于全基因组测序。
在博伊斯汤普森植物研究所(Boyce Thompson Institute for Plant Research)的标准条件(22°C, 14 h light)温室中,种植了大约20株番茄株系BTI-87的幼苗。收集4周龄幼苗的幼叶,如前所述,使用CsCl梯度提取DNA [52]。紫俄国、LA0386、LA1777、LA1932、LA1969、LA2779和H1706 (LA4345)在与BTI-87相同的条件下生长,收集幼叶组织,用CTAB法提取DNA。
基因组测序
Gh13、BTI-87和美国chilenseLA1932在位于纽约的威尔-康奈尔基因中心的Illumina HiSeq 2000机器上生成并测序。每个PE库的插入大小为300 bp。参考基因组美国lycopersicum使用的H1706来自国际番茄基因组计划,版本SL2.40 (http://solgenomics.net/organism/Solanum_lycopersicum/genome).扎克·利普曼博士,在冷泉港实验室,对美国pimpinellifolium加入,LA1589, [24]。美国galapagense接入LA0436和美国lycopersicum传家宝系YP序列是从BTI之前的研究中获得的[37]。
基因组组装
使用FastQC检查Illumina读数的质量,并在清洗后重新检查。使用fastq-mcf (http://www.bioinformatics.babraham.ac.uk/projects/fastqc/).读取的数据被映射到美国lycopersicumH1706参考程序集版本2.40使用BWA [53使用默认参数。使用Picard软件进行变异分析时,删除重复的reads以及映射质量小于30的reads (http://picard.sourceforge.net)及Samtools (http://samtools.sourceforge.net/) [54),分别。使用Samtools mpileup检测SNPs和indes (http://samtools.sourceforge.net/mpileup.shtml).
全基因组新创Gh13和BTI-87的程序集是使用SOAPdenovo版本1.05创建的(http://soap.genomics.org.cn/) [55]。组件的生产使用克麦25至63之间的范围。SOAPdenovo包提供的脚本用于脚手架的错误纠正和间隙填充。从头reads被映射到参考H1706基因组,以增加bwa对齐序列映射不良区域的覆盖率。
来确定准确的美国chilenseGh13的渐进断点,加入LA1932的变体使用VarScan2调用[56]和Gh13基因组中唯一的LA1932 snp使用自定义Perl脚本提取(https://github.com/nmenda/GenomeTools).
SNP的情节
的单核苷酸多态性美国pimpinellifolium参考H1706调用的,Gh13和BTI87相互比较,并标记为“唯一的”或“共同的”。然后,使用自定义Perl脚本(https://github.com/nmenda/GenomeTools).每个比较的SNP密度沿每个美国lycopersicum使用R统计量的' Heinz '染色体(http://www.R-project.org).
渐渗现象检测
渐进被定义为每10 Kb窗口至少有10个SNPs的snp -峰值,最小大小为50 Kb,最长为40 Kb的连续间隙。选择最小尺寸来捕获小的渐渗,并引入间隙来抵消渐渗区域基因组覆盖的显著下降,这是由于难以将这些区域映射到参考H1706基因组。每个窗口的最小SNPs数量是根据假设选择的,即没有渗透意味着整个基因组中每10 Kb窗口的平均SNPs数量将与非峰值区域的这个数量相似。如果基因渗入可以被定义为在峰值区域snp数量显著增加,而在非峰值区域snp数量显著减少,那么整个基因组中每个窗口的平均snp数量应该高于非峰值区域的snp数量。我们使用每10 Kb最小数量的3、5、10、15或20个SNPs来测试渐进,提取每种条件下的snp峰值和非峰值区域,并将非峰值区域的10 Kb窗口中SNPs的平均数量与Gh13整个基因组中的SNPs数量进行比较,并使用Student 's t检验对每对进行比较[57]、[58]。
PCR和Sanger测序
在已有标记和基因区基础上,对感兴趣的区域开发PCR引物。PCR产物由美国chilense,美国habrochaites,美国lycopersicum(Gh13行和紫色俄罗斯行)。PCR在55℃下进行,扩增循环32次,扩增步骤60秒。所有设计的底漆均列于表中3..PCR产物用Qiagen QIAquick PCR纯化试剂盒清洗,并送往康奈尔大学生命科学核心实验室中心(Ithaca, NY)或威斯康星大学麦迪逊生物技术中心进行Sanger测序。序列从美国lycopersicumH1706和YP,近亲繁殖的BTI-87,美国pimpinellifolium,美国galapagense通过引物对的最佳BLAST匹配从其基因组组合中提取。
系统发育树
从草图基因组集合中获得感兴趣区域的假定同源序列美国lycopersicumH1706序列选择顶部BLAST命中,然后反向BLAST返回美国lycopersicumH1706。Gh13, BTI-87,美国lycopersicumH1706, YP和紫色俄罗斯,美国pimpinellifolium,美国galapagense,美国chilense,美国habrochaites可用时,使用ClustalW [59]使用默认设置。检查了校准以确保准确性。Mega5使用500个自举重复和Tamura-Nei替代模型构建最大似然树[60]。无花果树(http://tree.bio.ed.ac.uk/software/figtree/)绘制基因树形图。所有的树都被提交到TreeBasehttp://purl.org/phylo/treebase/phylows/study/TB2:S16453.
SNP阵列基因分型
株系Gh13和一个易受begomovirus感染的自交系HUJ-VF缺乏Ty-3根据Infinium试验(Illumina Inc., San Diego, CA, USA),使用含有7720个SNPs的番茄阵列进行基因分型。HUJ-VF是一种加工型番茄,由耶路撒冷希伯来大学的Favi Vidavsky博士提供。对于每一次加入,使用威斯康星大学麦迪逊分校的Qiagen DNeasy试剂盒(Qiagen, USA)从新鲜、年轻的叶片组织中分离出基因组DNA。双链DNA浓度用PicoGreen法(Life Technologies Corp., Grand Island, NY, USA)定量,用10 mM Tris-HCl pH 8.0, 1 mM EDTA归一化至50 ng/ul。按照制造商Infinium测定方案,每次加入250 ng DNA进行基因分型。对于SNP调用,产生的强度数据被加载到GenomeStudio版本1.7.4 (Illumina Inc., San Diego, CA, USA)。为了确定SNP基因型,首先使用自动聚类算法生成初始SNP呼叫。使用SolCAP聚类文件确定每个SNP的聚类[45]。
支持数据的可用性
Gh13系和BTI-87系的基因组可在Sol基因组网络网站(http://solgenomics.net/organism/Solanum_lycopersicum/inbred_genomes).设计的PCR产物和引物的序列以及本工作的序列可从NCBI GenBank核苷酸数据库获得,登录号为KF887310-KF887341。
自定义perl脚本可从GitHubhttps://github.com/nmenda/GenomeTools.
作者的贡献
NM对Gh13和BTI-87进行基因组组装,编写生物信息学分析脚本,进行PCR测序,并起草手稿。SR进行系统发育分析,PCR测序,编写生物信息学分析脚本。乙脑为生物信息学分析工具做出了贡献。AB为基因组的生物信息学分析编写了脚本。DD种植植物并提取DNA,并为PCR测序做出了贡献。GM有助于PCR测序和基因渐渗分析。LM对自交系进行了发育、表型和基因分型。对SH贡献的分析Ty-3渐渗现象。MH提取了基因组DNA,并对基因渗入的分析做出了贡献。DM对自交系进行PCR测序、开发、基因分型和表型分析,并对自交系基因组、渐进和系统进化树进行分析。LAM为基因组的生物信息学分析和基因渐渗分析做出了贡献。所有作者都阅读并批准了最终的手稿。
附加文件
缩写
- SNP:
-
单核苷酸多态性
- WGS:
-
全基因组测序
- YP:
-
黄色的梨
- H1706:
-
亨氏食品公司1706
- 公关:
-
紫色的俄罗斯
参考文献
- 1.
乔凡诺尼·JJ:果实成熟突变体为成熟控制提供了新的见解。植物学报,2007,10(3):283-289。10.1016 / j.pbi.2007.04.008。
- 2.
裴德利KF,马丁GB: pto介导的番茄细菌性斑点病抗性的分子基础。植物科学进展,2003,26(4):344 - 344。10.1146 / annurev.phyto.41.121602.143032。
- 3.
张建华,张建华,张建华,张建华,张建华,张建华。番茄细菌性斑疹病基因间特异性的分子生物学基础。科学通报,1996,29(4):344 - 344。10.1126 / science.274.5295.2063。
- 4.
Blanca J, Canizares J, Cordero L, Pascual L, Jose Diez M, Nuez F:通过SNP基因分型和形态学揭示的变异可以深入了解番茄的起源。公共科学学报,2012,7 (10):e48198-10.1371/journal.pone.0048198。
- 5.
王晓明,王晓明,王晓明,王晓明,王晓明:番茄种群结构和遗传分化与育种历史和选择关系。遗传学报,2010,106(6):927-935。10.1038 / hdy.2010.139。
- 6.
Tanksley SD, McCouch SR:种子银行和分子地图:释放来自野外的遗传潜力。科学通报,1997,27(4):344 - 344。10.1126 / science.277.5329.1063。
- 7.
傻瓜MR:番茄的基因组定位和分子育种。植物基因组学2007年,doi: 10.1155 / 2007/64358。
- 8.
Alexander LJ:番茄的叶霉抗性。俄亥俄州农业Exp Sta Bul1934年,539年。
- 9.
艾伦·EW:美国农业部:各州关系服务处实验站实验站记录。1919
- 10.
Grandillo S, Chetelat R, Knapp S, Spooner D, Peralta I, Cammareri M, Perez O, Termolino P, Tripodi P, Chiusano ML, Ercolano MR, Frusciante L, Monti L, Pignone D:茄属。野生作物近缘:基因组和育种资源。编辑:Chittaranjan K. 2011,施普林格,海德堡/多德雷赫特/伦敦/纽约,129-215。10.1007 / 978 - 3 - 642 - 20450 - 0 - _9。
- 11.
季勇,Scott JW, Hanson P, Graham E, Maxwell DP:番茄侵染begomovirus的抗性来源、遗传和遗传位点定位。番茄黄曲叶病毒病。编辑:Czosnek H. 2007,施普林格,荷兰,343-362。10.1007 / 978 - 1 - 4020 - 4769 - 5 - _20。
- 12.
Zamir D, Ekstein-Michelson I, Zakay Y, Navot N, Zeidan M, Sarfatti M, Eshed Y, Harel E, Pleban T, van-Oss H, Kedar N, Rabinowitch HD, Czosnek H:番茄黄卷叶病毒耐受基因TY-1的定位和基因导入。应用理论,1994,88(2):141-146。10.1007 / BF00225889。
- 13.
Barham WS, Winstead NN:番茄对根结线虫抗性的遗传。中国园艺学报,2004,27(3):377 -377。
- 14.
Lanfermeijer FC, Warmink J, Hille J:番茄Tm-2断裂基因和Tm-22耐受性基因的产物在4个氨基酸上存在差异。应用物理学报,2005,26(4):344 - 344。10.1093 / jxb / eri288。
- 15.
Seah S, Yaghoobi J, Rossi M, Gleason C, Williamson V:与抗性番茄相比,易感番茄的抗线虫基因Mi-1与一个倒置的染色体片段有关。应用理论与实践,2004,38(4):357 - 357。10.1007 / s00122 - 004 - 1594 - z。
- 16.
Hanson P, Green S, Kuo G: Ty-2, 11号染色体上调节番茄双病毒抗性的基因。番茄热内合作,2006,56:17-18。
- 17.
Parniske M, Wulff BB, Bonnema G, Thomas CM, Jones DA, Jones JD:在番茄1号染色体短臂的多个位点上存在Cf-9抗病基因(Hcr9s)的同源物。分子植物-微生物相互作用。1999,12(2):93-102。10.1094 / MPMI.1999.12.2.93。
- 18.
杨春春,李春春,李春春,李春春,等。番茄晚疫病抗性Ph-3基因的分子定位。中国园艺学报,2002,27(3):389 - 397。
- 19.
Anbinder I, Reuveni M, Azari R, Paran I, Nahon S, Shlomo H, Chen L, Lapidot M, Levin I:番茄品种TY172抗番茄卷叶病毒的分子解剖。理论应用,2009,19(3):519-530。10.1007 / s00122 - 009 - 1060 - z。
- 20.
Moriones E, Navas-Castillo J:番茄黄卷叶病毒,一种新出现的病毒复合物,在世界范围内引起流行。病毒决议2000,71(1):123-134。10.1016 / s0168 - 1702(00) 00193 - 3。
- 21.
Verlaan MG, Hutton SF, Ibrahem RM, Kormelink R, Visser RG, Scott JW, Edwards JD, Bai Y:番茄黄曲叶病毒抗性基因Ty-1和Ty-3是等位基因,编码dfdgd类RNA依赖性RNA聚合酶。公共科学学报,2013,9 (3):e1003399-10.1371/journal.pgen.1003399。
- 22.
Polston JE, Lapidot M:番茄黄曲叶病毒的管理:美国和以色列的观点。在番茄黄曲叶病毒病。Czosnek H.施普林格编辑;2007:251 - 262
- 23.
Leinonen R, Sugawara H, Shumway M:序列读取存档。核酸研究,2011,39(增编1):D19-D21。10.1093 / nar / gkq1019。
- 24.
番茄基因组序列为肉质水果的进化提供了见解。自然科学进展,2012,34(4):344 - 344。10.1038 / nature11119。
- 25.
黄霞,陆涛,韩波:水稻基因组重测序:水稻基因组学的新时代。植物学报,2013,29(4):225-232。10.1016 / j.tig.2012.12.001。
- 26.
肖华,姜宁,陈晓明,陈晓明,陈晓明,陈晓明。番茄果实形态变异与逆转录转座子基因多态性的关系。科学通报,2008,29(4):344 - 344。10.1126 / science.1153040。
- 27.
吴涛,吴涛,王志刚,王志刚,王志刚,王志刚。番茄抗病蛋白激酶基因的克隆。科学通报,1998,26(3):344 - 344。10.1126 / science.7902614。
- 28.
Hajjar R, Hodgkin T:利用野生亲缘在作物改良:过去20年的发展调查。生态学报,2007,34(1):1-13。10.1007 / s10681 - 007 - 9363 - 0。
- 29.
Tanksley SD, Ganal MW, Prince JP, de Vicente MC, Bonierbale MW, Broun P, Fulton TM, Giovannoni JJ, Grandillo S, Martin GB:番茄和马铃薯基因组的高密度分子连锁图谱。中国生物医学工程学报,1997,32(4):344 - 344。
- 30.
Labate JA, Robertson LD:番茄(Solanum lycopersicum L.)基于野生番茄品种等位基因的隐性渐渗的证据。中国生物医学工程学报,2012,12(1):133-10.1186/1471-2229-12-133。
- 31.
Viquez-Zamora M, Vosman B, van de Geest H, Bovy A, Visser RG, Finkers R, van Heusden AW:基因组学时代的番茄育种:来自SNP阵列的见解。中国生物医学工程学报,2013,34(4):354-10.1186/1471-2164-14-354。
- 32.
Henry RJ:用于理解和加速作物驯化的下一代测序。中国基因组学杂志,2012,11(1):51-56。10.1093 / bfgp / elr032。
- 33.
Blair MW, Cortés AJ, Penmetsa RV, Farmer A, Carrasquilla-Garcia N, Cook DR:一种用于普通菜豆亲本多态性筛选和多样性分析的高通量SNP标记系统。应用物理学报,2013,26(2):535-548。10.1007 / s00122 - 012 - 1999 - z。
- 34.
Esteras C, Formisano G, Roig C, Díaz A, Blanca J, Garcia-Mas J, Gómez-Guillamón ML, López-Sesé AI, Lázaro A, Monforte AJ:瓜类SNP基因分型:遗传变异、群体结构与连锁不平衡。应用理论与实践,2013,26(1):1 - 5。10.1007 / s00122 - 013 - 2053 - 5。
- 35.
Robbins MD, Sim S, Yang W, Van Deynze A, Van der Knaap E, Joobeur T, Francis DM:利用全基因组snp集检测栽培番茄多态性的映射和连锁不平衡分析。实验学报,2011,62(6):1831-1845。10.1093 / jxb / erq367。
- 36.
Sim SC, Van Deynze A, Stoffel K, Douches DS, Zarka D, Ganal MW, Chetelat RT, Hutton SF, Scott JW, Gardner RG, Panthee DR, Mutschler M, Myers JR, Francis DM:番茄(Solanum lycopersicum L.)的高密度SNP基因分型揭示了育种引起的遗传变异模式。公共科学学报,2012,7 (9):e45520-10.1371/journal.pone.0045520。
- 37.
史卓乐,孟克沃尔德,孟达,马廷吉,穆勒,李志刚:栽培番茄与近缘野生番茄的比较基因组学和系统发育不一致性。中国印刊杂志,2014,2:e377v1-
- 38.
Causse M, Desplat N, Pascual L, Le Paslier MC, Sauvage C, Bauchet G, Berard A, Bounon R, Tchoumakov M, Brunel D, Bouchet JP:番茄全基因组重测序揭示了与渗透和育种事件相关的变异。BMC基因组学杂志,2013,14(1):791-10.1186/1471-2164-14-791。
- 39.
Shirasawa K, Fukuoka H, Matsunaga H, Kobayashi Y, Kobayashi I, Hirakawa H, Isobe S, Tabata S:利用种植番茄基因组重测序开发的单核苷酸多态性标记进行全基因组关联研究。中国生物医学工程学报,2013,20(6):593-603。10.1093 / dnares / dst033。
- 40.
Mejía L, Garcia BE, Fulladolsa AC, Sánchez-Pérz A, Havey MJ, Teni R, Maxwell DP: Ty-3基因在危地马拉番茄重组自交系中抗性的研究。中国农业科学,2009,29(4):344 - 344。
- 41.
Mejía L, Teni R, Vidavski F, Czosnek H, Lapidot M, Nakhla M, Maxwell D:危地马拉番茄种质资源评价及抗豆状病毒育种选育。植物学报,2004,695:251-256。
- 42.
Vidavsky F, Czosnek H:抗和耐受番茄黄曲叶病毒的番茄育种系。植物病理学杂志,1998,29(3):344 - 344。10.1094 / PHYTO.1998.88.9.910。
- 43.
Scott JW, Schuster DJ: Gc9, Gc171和Gc173抗果腺病毒自交系。中国科学(d辑),2007,30(4):344 - 344。
- 44.
Ozminkowski R:亨氏1706品种谱系。《番茄报告》,2004,54:26-
- 45.
Sim S, Durstewitz G, Plieske J, Wieseke R, Ganal MW, Van Deynze A, Hamilton JP, Buell CR, Causse M, Wijeratne S, Francis DM:番茄大型SNP基因分型阵列的开发和高密度遗传图谱的生成。公共科学学报,2012,7 (7):e40563-10.1371/journal.pone.0040563。
- 46.
Finkers R, van Heusden S: 150+番茄基因组(重)序列项目;经验教训和潜在应用。2013年,番茄育种者圆桌会议,泰国清迈
- 47.
Ji Y, Salus M, Van Betteray B, Smeets J, Jensen K, Martin C, Mejia L, Scott J, Havey M, Maxwell D:番茄6号染色体25 cM处辣椒茄Ty-3和Ty-3a位点的共优势SCAR标记《番茄》,2008,57:25-29。
- 48.
季勇,Schuster DJ, Scott JW:番茄6号染色体上一个邻近番茄黄曲叶病毒抗性位点Ty-1的豆状病毒抗性位点Ty-3。Mol品种,2007,20(3):271-284。10.1007 / s11032 - 007 - 9089 - 7。
- 49.
Garcia BE, Mejia L, Melgar S, Teni R, Sanchez-Perez A, Barillas AC, Montes L, Keuler NS, Salus MS, Havey MJ, Maxwell DP: Ty-3遗传在危地马拉番茄F3科对二部begomovirus抗性的有效性。番茄Genet Coop . 2008, 58: 22-28。
- 50.
Ji Y, Scott JW, Maxwell DP, Schuster DJ:第3染色体上的番茄黄曲叶病毒抗性基因Ty-4。番茄Genet Coop . 2008, 58: 29-31。
- 51.
Doyle JJ:一种对少量新鲜叶片组织进行快速DNA分离的方法。植物化学通报,1987,19:11-15。
- 52.
Bombarely A, Rosli HG, Vrebalov J, Moffett P, Mueller LA, Martin GB: benthamiana的基因组序列草案,以加强分子植物-微生物生物学研究。植物微生物相互作用,2012,25(12):1523-1530。10.1094 / mpmi - 06 - 12 - 0148 - ta。
- 53.
李华,杜斌R:基于Burrows-Wheeler变换的快速准确短读对齐。生物信息学,2009,25(14):1754-1760。10.1093 /生物信息学/ btp324。
- 54.
Li H, Handsaker B, Wysoker A, Fennell T, ruj, Homer N, Marth G, Abecasis G, Durbin R:序列比对/映射格式与SAMtools。生物信息学,2009,25(16):2078-2079。10.1093 /生物信息学/ btp352。
- 55.
李锐,李勇,王娟:SOAP:短寡核苷酸比对程序。生物信息学,2008,24(5):713-714。10.1093 /生物信息学/ btn025。
- 56.
Koboldt DC, Zhang Q, Larson DE, Shen D, McLellan MD, Lin L, Miller CA, Mardis ER, Ding L, Wilson RK: VarScan 2:外显子组测序发现癌症体细胞突变和拷贝数改变。中国生物工程学报,2012,22(3):568-576。10.1101 / gr.129684.111。
- 57.
JMP®,版本11.2。1989-2007年,北卡罗来纳州卡里的SAS研究所公司。
- 58.
RStudio团队:RStudio: R.波士顿的集成开发:RStudio, Inc.;2012 [], [http://www.RStudio.com/ide]
- 59.
拉金MA, Blackshields G,布朗NP, Chenna R, McGettigan PA, McWilliam H, Valentin F, Wallace IM, Wilm A, Lopez R, Thompson JD, Gibson TJ, Higgins DG: Clustal W和Clustal X 2.0版本。生物信息学,2007,23(21):2947-2948。10.1093 /生物信息学/ btm404。
- 60.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S: MEGA5:利用最大似然,进化距离和最大简约方法进行分子进化遗传学分析。分子生物学杂志,2011,28(10):2731-2739。10.1093 / molbev / msr121。
致谢
我们感谢Mark Massoudi博士,AgBiotech Inc. (San Juan Bautista, California)为抗性位点提供的KASP SNP标记分析;Allen Van Deynze博士,加州大学戴维斯分校和SolCAP项目(美国农业部NIFA AFRI植物育种、遗传学和基因组资助2009-85606-05673),用于SNP阵列基因分型;野生番茄种子为TGRC。
我们感谢Martha Maxwell和Monica Franciscus对手稿的校对,以及Sarah Refi-Hind对手稿的批判性阅读。这项工作得到了BTI启动基金穆勒实验室(NM, SRS, JDE, AB)和国家科学基金会拨款IOS-1025642 (GBM)的支持。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
电子辅助材料
12870 _2014_287_moesm1_esm.pdf
附加文件1:图S1。:Gh13 SNP density and coverage plots. X axes are positions in bp, Y axes are number of SNPs, and negative Y axes are genome coverage. Introgression regions are highlighted in red. (PDF 2 MB)
12870 _2014_287_moesm2_esm.pdf
附加文件2:图S2。:BTI-87 SNP density and coverage plots. X axes are positions in bp, Y axes are number of SNPs, and negative Y axes are genome coverage. Introgression regions are highlighted in red. (PDF 2 MB)
12870 _2014_287_moesm3_esm.xls
附加文件3:表S1。:在trogressions in Gh13 by 10 Kb windows. Overlapping SNPs of YP,美国pimpinellifolium英国国际贸易局,- 87,美国chilenseLA1932,以及它们与Gh13重叠的snp。(xl50kb)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/4.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
门达,N.,斯特里克勒,s.r.,爱德华兹,J.D.et al。分析番茄自交系的野生物种渗入揭示了祖先起源。BMC植物生物学14,287(2014)。https://doi.org/10.1186/s12870-014-0287-2
收到了:
接受:
发表:
关键字
- 茄属植物lycopersicum
- 茄属植物pimpinellifolium
- 茄属植物chilense
- 基因渐渗现象
- 基因组测序
- 抗病性
- 单核苷酸多态性
- 野生物种
- 驯化
- 系统发生学