- 研究文章
- 开放访问
- 发布:
精确的叶绿体基因组莲属椰子(Nelumbonaceae)用Sanger,Illumina Miseq和PacBio RS II测序平台评估:深入了解基础申明岛的塑植演进
BMC植物生物学体积14.文章编号:289(2014年)
摘要
背景
叶绿体基因组对植物发育和进化具有重要意义。莲属椰子是白垩纪晚期遗留下来的植物吗.最近,一个新的测序平台PacBio RS II,被称为“SMRT (Single Molecule, Real-Time) sequencing”,已经开发出来。利用SMRT测序技术研究了黄花苜蓿叶绿体基因组n .椰子将有助于阐明基础实二体质体的进化。
结果
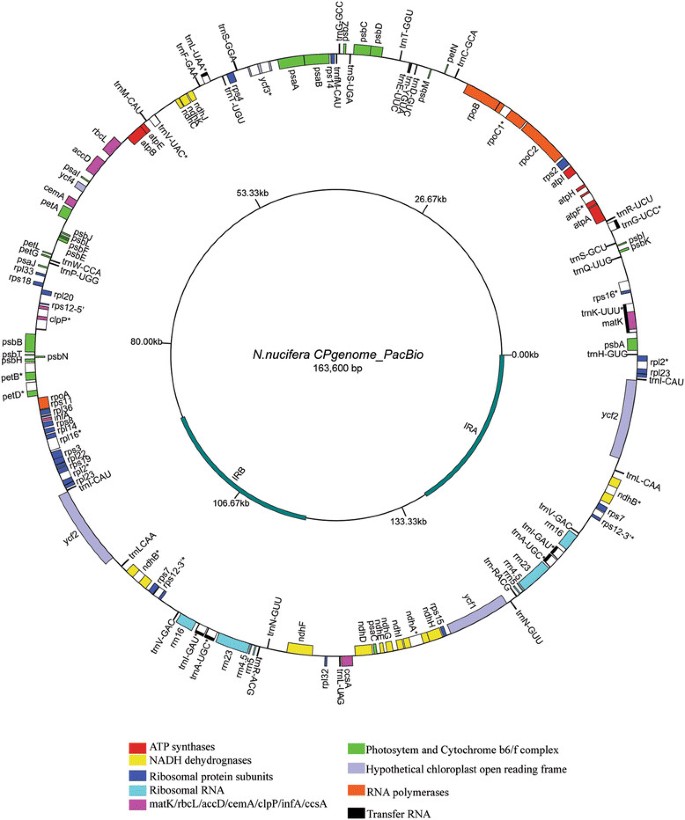
尺寸新创组装完全叶绿体基因组n .椰子是163,307bp,163,747bp和163,600 bp,分别由Sanger,Illumina Miseq和Pacbio RS II测序为7×,712×和105×的平均覆盖深度。精确的叶绿体基因组n .椰子由Illumina MiSeq reads从PacBio RS II数据校对中获得,该结构包含一个大的单拷贝区(91,846 bp)和一个小的单拷贝区(19,626 bp),两个反向重复区(26,064 bp)分开。基因组包含113个不同的基因,包括4个不同的rrna, 30个不同的trna和79个不同的肽编码基因。对56个目133个类群进行系统发育分析莲属一个人的妹妹是一个177万年的人Platanus.,属于基生双子科。根据MCMCTree估算,基底双二体在早侏罗世开始出现,分化时间为1.97亿年。基底双二二体的IR扩张/收缩似乎是独立发生的。
结论
由于富裕地区的覆盖范围长期读取和缺乏偏见,PACBIO RS II对高度准确的“完成”基因组表示了很大的承诺,特别是新创组装基因组。n .椰子是基础申逊的一个成员,然而,IR结构变化的进化分析n .椰子和其他基础实二二体表明,IR的扩张/收缩在这些基础实二二体中独立发生,或由独立的插入和缺失引起。精确的叶绿体基因组n .椰子这将为叶绿体基因组的结构变异提供新的信息,并在初级序列和结构水平上为基础双二体的进化提供新的见解。
背景
叶绿体基因组(CP Genome)编码一组用于光合作用的蛋白质和其他对植物开发至关重要的房屋保持功能[1].Cp基因组常被用于植物进化的研究。此外,cp基因组主要是单亲本遗传的[2],具有高度保守的基因含量和四重形组织,包括一个大的单拷贝(LSC),小单拷贝(SSC)和两个倒拷贝(IRS)。因此,CP基因组广泛用于追踪物种历史[3.] - [6].在过去的几年中,来自高等植物的完整叶绿体基因组的数量急剧增加[7] - [12.].到目前为止,随着新一代测序仪的出现,NCBI数据库中已经保存了437个完整的植物叶绿体基因组。这些数据库资源为更好地了解陆生植物cp基因组进化提供了信息。“活化石”莲属亚丹属是被子植物的一个小属,具有悠久的进化历史。它们是多年生水生植物,在阿尔比亚中部繁盛[13.],[14.].现在只有两种幸存的物种,莲属椰子Gaertn。和Nelumbo Lutea.威廉德分别。前者主要分布在亚洲和北澳大利亚,后者主要发现在北部和南美洲[15.].莲属是经济上重要的水生作物,具有观赏、食用和药用特性。1795年,林奈将莲属在睡莲属莲属林恩。在这之后的200年里,莲属一直被认为是睡莲属(睡莲)的一员,后来被认定为睡莲属的一个单独的科[16.].
在过去的二十年中,DNA序列被用来重新评估莲属.传统观点受到非分子研究的挑战[17.],[18.),rbcl.序列数据(19.].迄今为止,已经使用了五种不同的编码基因和几种非编码序列来重建关系莲属[19.] - [25.].除了核基因组[26.的完整cp基因组n .椰子应该执行来阐明基因组进化n .椰子.准确的cp基因组图谱对研究植物的系统发生、进化和资源保护具有重要意义n .椰子。
获得准确的CP基因组是了解其对高等植物的生物学功能和演化的先决条件。在开始时,大多数植物CP基因组新创由传统的桑格测序组装而成[27.] - [31.].该方法速度慢、成本高、费力且吞吐量低。最近,以高通量和低成本著称的Illumina等下一代测序仪已经被用于基于相关参考基因组组装基因组。由于其读取长度较短,无法解析长重复或低gc区域的基因组组装,导致缺口[32.].单分子实时测序技术(Single Molecule, Real-Time sequencing technology, SMRT)是由太平洋生物科学公司(PacBio)开发的第三代测序技术。流程如下:首先,DNA嵌入的DNA聚合酶附着在50纳米宽的孔的底部,称为零模波导(zero-mode waveguide, ZMWs),第二,聚合酶在ZMWs中使用γ-磷酸盐荧光标记的核苷酸来合成DNA,第三,ZMWs的宽度不允许光通过波导,但能量可以穿透很短的距离,并激发在井底聚合酶附近不断生长的DNA分子中的荧光团。与Sanger和Illumina平台相比,PacBio可以产生大约3000 bp的平均读取长度,目前的PacBio RS II平台的一些读取长度可达30 kb。对合并事件或未检测到的间隔事件引起的准确率和插入/删除(indel)事件存在一些担忧,但这些可以通过增加多个SMRT细胞的吞吐量来改善[33.],[34.].装配方法的优化[32.],[35.和准确率的提高使得该平台在基因组序列整理方面有很大的前景。目前,PacBio平台被广泛应用于新创针对各种生物的测序,包括人类[36.],[37.),微生物(38.],[39.]和植物[40].
在本研究中,我们达到了三个目标:n .椰子(代表莲属)作为评估和比较cp基因组的材料,包括Sanger、Illumina MiSeq和PacBio RS II三种测序平台的准确率和序列大小。第二,我们新创组装,注释和分析CP基因组n .椰子使用PacBio RS II数据。第三,评价我国的制度地位和分歧莲属的cp基因组n luteaSanger平台测序,平均覆盖深度为6×。我们构建了一个大的系统发育树,包括来自56个目的133个物种(附加文件)1).最后,我们还估计了基础的双二二体分化时间,并比较了cp基因组结构,以说明这些早期分化的双二体之间的IR扩张/收缩。
结果与讨论
新创Illumina MiSeq和PacBio RS II平台组装
使用HGAP方法[32., PacBio RS II的数据是新创组装到一个163,600 bp contig,105×覆盖度景深(图1)使用Celera汇编器7.0。illumina miseq数据是新创组装到一个163,747 bp contig(附加文件2:使用Celera汇编器7.0,图S1)具有712倍的覆盖深度。163,307 bp contig(附加文件3.:Sanger的图S2)用SealIncher软件组装。使用PCR填充了来自Illumina Miseq平台和Sanger技术的NNN的序列间隙。从illumina miseq和Pacbio RS II的数据统计数据和组装在表格中总结在表格中1.利用Sanger的数据,我们发现三个序列的同源性非常相似,但来自Sanger、Illumina MiSeq和PacBio RS II测序平台的三个contigs的长度不同。通过ClustalW比对和PCR验证,我们发现了一个282 bp的缺失ndha.内含子使用Sanger平台和使用Illumina MiSeq平台的反相重复中的152bp插入(附加文件2:图S1和附加文件3.:图S2)。这些错误可能是由于Sanger测序reads的低通量和下一代测序方法中读取长度较短造成的[41.].序列技术引起的错误插入和缺失通常导致基因组特征的不正确分析。此外,低通量技术和短读取长度不理想地达到具有高度重复序列的某些区域。然而,在一些小型基因组中,例如微生物的基因组,这种重复的序列似乎为细菌菌株之间的区别提供了关键的见解[42.].
目前,塑性测序的进步主要由下一代测序技术促进。已经进行了相关的报告比较塑性测序的下一代测序平台,例如GS20系统(454寿命公司)[43.] Illumina Ga II平台[44.],改进的传统桑格法[45.].此外,在去年进行了第三代技术(PACBIO)的下一代技术(Illumina)的第一次比较[40].但是,尚未确定排序时代,桑雷,illumina和PacBio的三个代表的优缺点的全面比较。特别是对于精度挑战的PACBIO平台,新开发的组装方法和升级化学(从C1到C2)将提高准确度和吞吐量[32.].我们应用了三个独立的测序平台来评估CP基因组n .椰子。PCR扩增证实的结果表明新创PacBio RS II平台的组装基因组最完整,覆盖率达到100%。在足够的深度(105×)下,PacBio RS II的SMRT测序提供了高度精确的cp基因组n .椰子,因为同一错误不太可能被随机观察多次[34.].含有大片段的深度测序覆盖和额外的illumina文库对于获得Sanger和Illumina Miseq的准确结构是必不可少的。尽管三者之间存在较小差异新创在植物发育过程中,精确的cp基因组结构可能比cp基因组序列具有更重要的作用。同时,不完整的cp基因组信息n .椰子分别由Sanger和Illumina的缺失和插入引起,不能反映cp基因组的真实结构体内.此外,PacBio的运行时间非常短,只有2小时[34.,可以为研究人员节省大量时间。因此,PacBio RS II平台具有长读和无at富区覆盖偏倚的特点,有望实现高度精确的“完成”基因组。
的一般特征和密码子用法n .椰子cp基因
最后叶绿体圆形图n .椰子从illumina miseq数据纠正的PacBio数据是163,600 BP。在结构和编码能力方面,CP基因组n .椰子类似于那些双二子体,由系谱特异性插入和删除引起的微小长度变化。该基因组呈典型的四部分结构,其中一个大的单拷贝区(LSC, 91,846 bp)和一个小的单拷贝区(SSC, 19,626 bp)被两个反向重复序列(IR, 26,064 bp)分开(图)1).CP基因组n .椰子含有最完整的113种不同基因,包括四种不同的RRNA(16s,23s,4.5s和5s),30个不同的TrNA和79个不同的肽编码基因(包括四个ycfs.).四个rrnas,七个trnas和六种肽编码基因(包括RPS12.)在IR区域复制,共产生130个基因(表2).
开始密码子使用n .椰子与其他8种基础双二酮组比较(表3.).在这些基础的双二核苷酸中,ACG、GTG或ATA被用作ATG的起始密码子。在起始密码子的变化中,rpl2和RPS19在所有被调查的基础上发现的,但是NDHB.和YCF2.只在毛茛属植物.在79个不同的叶绿体蛋白质编码基因中n .椰子只有三个基因(psbL, rpl2和RPS19)使用ATG的替代密码子作为起始密码子:ACG表示psbL和rpl2, GTG表示RPS19.一个ACG到AUG的编辑站点n,psbL和rpl2转录物存在于大多数血管植物体积中[46.],[47.,但我们只检测到两个RNA编辑位点(psbL和rpl2)在起始密码子区域中。丢失这样的编辑网站n转录本可能是由Nelumbonaceae在过去的1.6亿年中非常缓慢的进化速度或C到T的反向突变造成的n开始密码子。这种替代起始密码子,acgn可能会大大削弱NDH复合物在叶片中的积累[48.].此外,我们检查了79个不同叶绿体蛋白编码基因的密码子使用模式n .椰子.共22,902个密码子组成了79个不同的叶绿体蛋白编码基因n .椰子.密码子的总体用法n .椰子通常类似于其他基因组报告的,例如人参[22.),莲花[49.].相关同义密码子用法(RSCU)分析表明n .椰子CP基因组与A或U的第三位核苷酸比以G或C结尾的那些更频繁地使用(表4),如在土地植物的大多数CP基因组中所观察到的[30.].例如,对于编码的四个密码子,古胡和浦族的RSCU为1.43和1.5,但GUC和GUG的RSCU分别仅为0.49和0.58。
在缓解心肌的演变期间,最小测序的CP基因组的尺寸范围为约120kb至160kb。然而,寄生植物具有独特的生活方式存在一些例外,其中CP基因组的尺寸超出了120 kb至160kb的范围,例如Conopholis美国,具有45 kB土地植物的最小塑料[50.].此外,cp基因组中的基因数量在不同的谱系中也存在差异,如缺失NDH.基因。事件NDH.大多数非光合作用植物发生基因损失,如Cuscuta Reflexa.[51.和寄生植物,如Epifagus virginiana[52.],在一些非寄生的光合植物中,如蝴蝶氏植物阿芙罗狄蒂[53.]和geraniaceae [54.].其机理NDH.可以解释基因损失,以便将基因转移到核或者它们不参与特定谱系的关键生命发展[55.].除了NDH.基因,不同谱系中还有其他独立的基因损失,包括infA,rpl,石头剪刀,宠物,公安局等等(附加文件1).例如,rpl21.通过线粒体基因组的基因补偿祖先植物植物和高血管植物的祖先蛹中的CP基因组的基因丧失。独立丧失infA在被子植物(包括几乎所有蔷薇科)中,是叶绿体向细胞核转移事件的结果[56.].CP基因组n .椰子保留了一套完整的基因数据,表明这些基因可能对其发育至关重要。
在土地植物中广泛发生的CP基因组的替代起始密码子,例如Pteridophytes [30.].这种密码子的编辑模式似乎是在陆生植物进化过程中独立发生的,与植物界的系统发育树无关。密码子的总体用法n .椰子Cp基因组与其他已报道的Cp基因组相似[30.],[57.和线粒体基因组[58.].这些密码子的使用模式可能是由高比例的A/T的组成偏倚驱动的。
基生双二萜的系统发育和分子年代分析
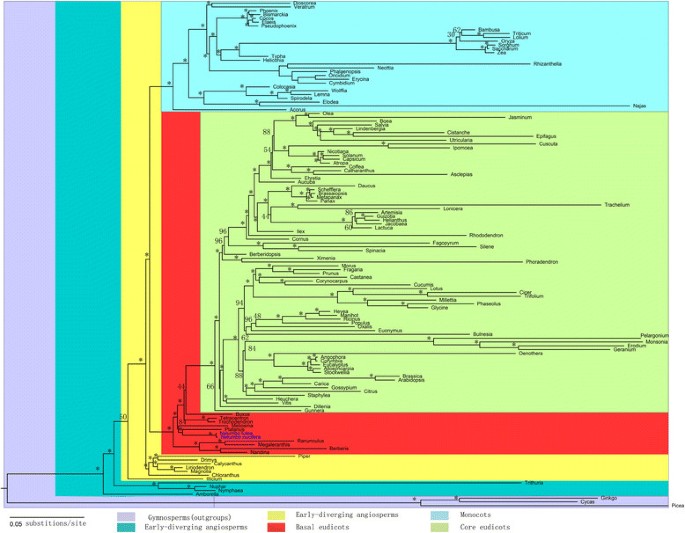
使用三个数据矩阵,使用来自56个种子植物的79个蛋白质编码基因进行最大似然(M1)系统发育分析。在使用ModelTest 3.7中搜索56型号之后,选择具有站点和不变站点(GTR + G + G + I)的速率变化的一般时间可逆(GTR)模型作为三个数据矩阵的最佳拟合。从三个数据矩阵推断的系统发育树显示出相同的拓扑。另外,由此产生的拓扑,与Angiosperm Phylogy基团(APG)的结果一致[59.],建议系统发育树是可靠的。作为姐妹Meliosma.,莲属和Platanus.用100%的引导值形成一个进化枝。这个结果证实了n .椰子是一个茎逃避,由特异性花粉颗粒的形态学证据支持[21.].由于收敛演变[60.在相同的水生环境中,类似的形态导致了误识n .椰子作为…的亲戚睡莲属阿尔巴.对56个目133个类群进行系统发育分析莲属她的姐妹进化到了哪里Platanus.这是一种高大的陆生树(图2).
申明队包括绝大多数现存的血症,估计有200,000种。片状可以分为基础销售和核心申办透视[61.],[62.].迄今为止,塑性基因组已完全测序八个基础销售,包括进行了[63.],Megaleranthis[64.],南娜[43.],Platanus.[43.],毛茛属植物[30.],Trochodendron[65.],四分离人体[65.),莲属(本研究)。添加了未取样的基础双子体cp基因组n .椰子将更好地了解基础才审美的演变。
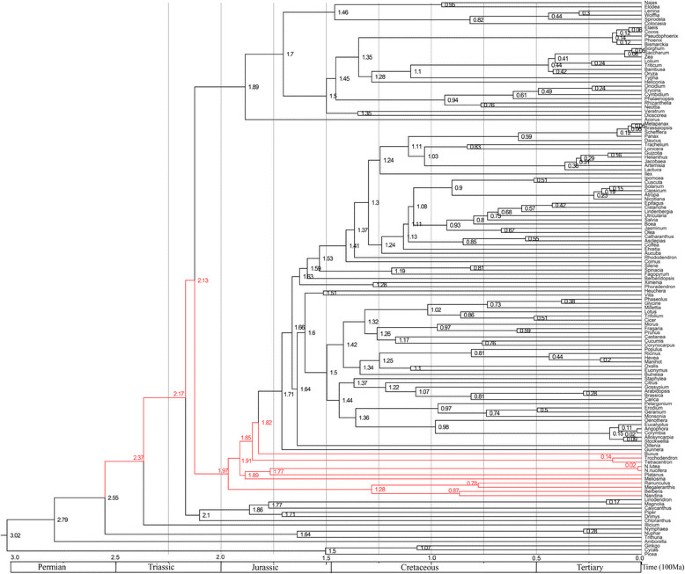
在我们研究中获得的系统发育树中,分析的基础销售,包括兔子(南娜,小檗属植物,Megaleranthis,毛茛属植物)、沙柏科、变形目(Platanus.,莲属),Trochodendres(Trochodendron,四分离人体)和Buxales (进行了)形成独立的演化枝(图2).为了估算这些曲折的分歧时间,PAML4.7的McMctree被使用与近似似然计算方法[66.].该分析将毛茛科、Sabiaceae、Proteales、Buxales和Trochodendrales的年代分别确定为197、189、189、185和1.82亿年前(Myr)。莲属有177岁的MYR,并且在唯一的两个现存物种之间分裂,n lutea和n .椰子,据估计大约发生在2myr之前(图3.).
近年来,随着NCBI发布的叶绿体数据,研究人员将这些cp基因组用于植物进化[67.],[68.].在我们的研究中,我们从NCBI数据库中仔细选择了不同的分类群,其中CP基因组可能出版。此外,长分支吸引力将误导错误的系统发育树。避免长分支吸引力[69.,选择均匀分布于种内的类群。同一顺序控制类群数量(不超过8个)。密码子位点取代率的饱和,特别是第三个位点,影响系统发育树的拓扑结构[28.].在我们的测试中,包含每个密码子所有三个位点的矩阵的系统发育拓扑与其他两个矩阵(1英石和2nd网站,和3rd.每个密码子的位点)验证了在我们分析的类群中没有饱和取代率。
在这里,采用了133个统一覆盖56个命令的分类率来进行系统发育分析和基础申销顾问的分歧时间。来自三种矩阵的系统发育分析(所有三个地点,1英石和2nd网站和3rd.79个叶绿体蛋白编码基因的密码子位点为叶绿体蛋白的系统发育提供了依据n .椰子作为一个基础奥尽管,姐姐Platanus..对散度时间的估计表明莲属和Platanus.大约在177myr之前开始分化[66.].此外,基底双二子体的分化(包括莲属) 从纽约巴妥(“早期分化的”被子植物),大约是255 Myr以前(图3.).形态相似之处莲属和纽约巴妥由收敛演变引起的通常与三个分类群中的分子序列的相似性相互矛盾Nelumbo,Nymphaea和Platanus..因此,这些物种的表型由其分子序列和生物环境的组合确定。
基生双二体内的结构演化
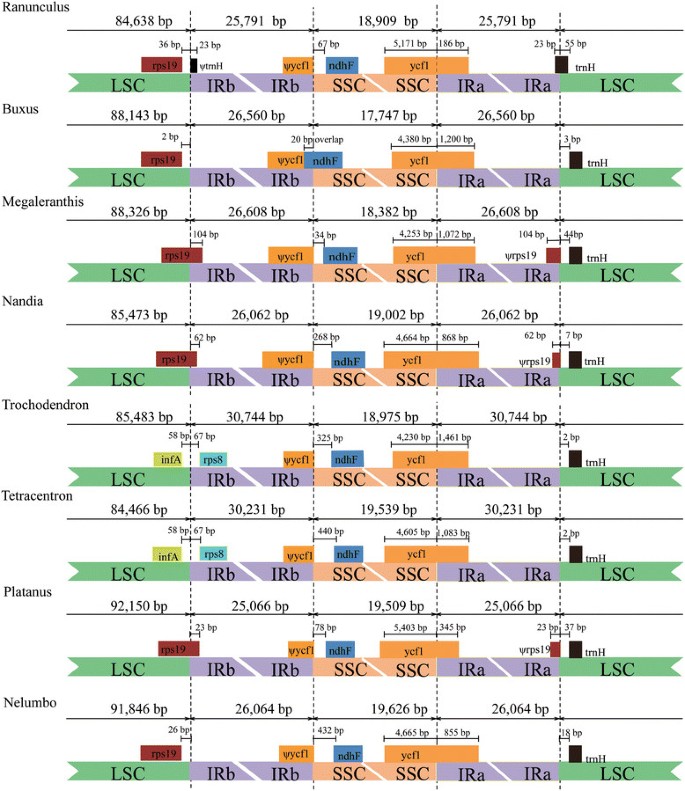
在Anuiaperms中,SSC和LSC结合的频繁收缩和扩展与IRS有助于CP基因组的尺寸变化。因此,这些连接点的收缩和扩展已被认为是用于说明分类群之间关系的进化标记[70].我们感兴趣的是n .椰子以及其他基础的双二基酯。的结构n .椰子Cp基因组与7个基础的双二体(Trochodendron,四分离人体,Platanus.,毛茛属植物,进行了,Megaleranthis,Nandia).与其他6种不同的是,LSC/IRb边界的扩张幅度最大Trochodendron和四分离人体,最大30 kb。LSC/IRb的连接Trochodendron和四分离人体扩展到两者之间的区域infA和RPS8..然而,其他6种物种的结似乎只保守了轻微的扩张(图4).IRB的Platanus.,Megaleranthis和Nandia扩展到3 '部分rpl19.分别由23 bp,104 bp和62 bp。LSC / IRB边界毛茛属植物,进行了和莲属位于基因间空间区域下游RPS19(图4).这些数据表明,即使在相同的序列中,也存在着不同的边界,如莲属(审查)和Platanus.(Proteales)。我们推测IR/LSC边界的位置可能与它们的系统发生位置无关。
n .椰子是陆生植物的一员[71.,在白垩纪繁盛。当第四纪冰期发生时,n .椰子因环境压力而被困在水域[72.].以往的报道指出,与非单子叶被子植物相比,单子叶被子植物中IR的扩张更为渐进,并提出了两种假设来解释单子叶被子植物中IR的扩张[70].17种测量的血管植物的IR边界在这些CP基因组之间变化,即使在相同家庭的密切相关属之间[22.].我们想知道这支基生双二体是否保持着保守的IR边界。在本研究中,这些基本双二体的IR边界的扩张和收缩也有所不同,这与谱系的系统发育无关。例如,IR边界莲属(Proteales)更接近于进行了(Buxales)比其密切相关的分类群岛,Platanus.(Proteales).尽管每个IR莲属比那个高于1 kbPlatanus.(图4),前者不包含的部分RPS19后者也是如此。我们发现,IRs的变化是由IR扩展到LSC或由独立的DNA片段插入IR区域。独立插入是如何发生的,在未来的研究中还有待阐明。
结论
我们首先应用三个测序平台来评估cp基因组n .椰子。利用PacBio RS II数据、Illumina MiSeq数据和Sanger数据,我们进行了数据分析新创组装,注释和分析CP基因组n .椰子.精确的CP基因组n .椰子是一个典型的四部结构的圆形分子,长度为163,600 bp,包含一个LSC区(91,846 bp)和一个SSC区(19,626 bp),由IR区(26,064 bp)分开,共130个基因。133个类群的79个组合叶绿体蛋白编码基因的ML树证实了这一点n .椰子是基友的姐妹吗Platanus..估计具有近似似然计算的McMctree中的分歧时间显示,基础拒于在197年的MYR分歧,莲属是177年最高产量研究。之间的分裂n lutea和n .椰子估计已经发生了大约2 myr。结构比较表明,基础才拒绝的IR边界发生在各种边界位置,并且发生了独立插入IR莲属.本研究表明,PacBio平台将对新创组装的基因组和cp基因组n .椰子提供了新的洞察力演变的新洞察力。我们认为,随着新的PACBIO测序平台的外观,将获得更准确的CP基因组以了解序列和结构水平的缓解植物的演变。
方法
材料
材料(莲属椰子试验用蔬菜由武汉市蔬菜科学研究所、武汉市水产蔬菜国家野外观测研究站(30°12′n, 111°20′e)维护。
叶绿体基因组DNA提取
如下取得高质量DNA:展开的嫩叶n .椰子收获并在4°C的黑暗中储存,以消除组织中的淀粉。采用不连续蔗糖梯度离心法,DNase I酶切分离叶绿体[73.].除非另有规定,否则必须在4°C下进行所有步骤。通过向200μg/ ml的终浓度增加一级裂解缓冲液和一流的蛋白酶K来轻轻裂解叶绿体溶液。然后在37℃下在30分钟的水浴中每15分钟轻轻倒置并混合一次,然后在37℃下每15分钟混合一次。添加冷NH后4三饱和酚/氯仿/异戊醇(25:24:1)和氯仿/异戊醇(24:1)分别提取核酸至最终浓度为0.8 M。
将叶绿体基因组DNA(CpdNA)在-20℃下以两体积的100%乙醇沉淀过夜,并以18,000×g离心20分钟。将粗CPDNA用70%乙醇洗涤两次,并在37℃下用RNase消化重新悬浮30分钟。最后,OD260 / 280和OD260 / 230的值分别为1.9和1.97。
图书馆建设,排序和新创组装
- (1)
Sanger图书馆建设
利用cpDNA远程PCR技术扩增叶绿体全长序列。PCR引物是根据已知的真双子叶植物叶绿体基因组序列进行比对而开发的(附加文件)4).我们覆盖了两种叶绿体基因组(n lutea和n .椰子)具有PCR产物,其显示在8至15 kB之间的长度。使用电泳在低熔点琼脂糖凝胶上洗脱PCR产物,然后用柱纯化(轴烯)。将纯化的产物剪切到约1.5kb的随机片段中,并克隆到PMD18-T载体(Takara)中。将重组质粒转化到TOP10细胞中并使用ABI 3730 DNA测序仪进行测序。
序列掩蔽和组装使用Sequencher软件(Gene Codes Corp., Ann Arbor, MI)。每个核苷酸大约有7倍的覆盖率n .椰子6 × inn lutea.用PCR方法填充空白和低覆盖区域,确保每个核苷酸至少有3个reads被覆盖。
- (2)
Illumina MiSeq库建设
使用约5μg的CPDNA构建含有400bp插入尺寸的文库。根据制造商的协议,使用Truseq DNA LT样品制备试剂盒V2(Illumina)构建基因组DNA测序文库。使用Covaris S220将基因组DNA剪在短片段中。DNA片段在末端修复后在3'末端腺苷酸。将比衔接子连接到DNA片段的两端,并且条形码序列包含在其中一种适配器中。通过凝胶切割选择靶向DNA片段,并使用通用引物(Illumina)通过10-循环PCR扩增。在纯化,定量和验证后,在制造商的标准工作流程后,在Illumina Miseq测序系统上测序了验证的DNA文库。
对原始数据进行过滤,得到的干净数据用FLASH进行连接。连接读数以bwa-0.7.3a对齐cpBase。然后选择对齐的读取新创使用Celera Assembler 7.0进行汇编。最后用SSPACE和lastZ组装成一个支架。利用PCR扩增技术填补两个空白。
- (3)
PacBio图书馆建设
需要20μg纯和高分子量DNA的样品来制备大小 - 选择约20kb的Smrtbell模板。使用纳米玻璃分光光度计和QUBit荧光计测量CpDNA浓度,并在场转换凝胶上运行大约200ng的CPDNA。随后的步骤基于PACBIO示例Net-Sharid协议,可提供http://pacificbiosciences.com/
Pacbio RS II的原始读数用SMRT分析2.1,共产生9,165个高质量的读数,高达42,623,117 BP。读数的平均长度为4,651 bp。使用Celera汇编器7.0组装这些读数。读取与BWA-0.7.3a的组装成簇对齐。最终,使用Lastz工具组装超过10x覆盖深度的4个具有超过10倍的覆盖深度的Contig。最后,为了产生CP基因组序列的最佳共识序列,使用QUIVIVE(新的多欧共识算法)来校正错误区域[32.].
基因组注释
整个叶绿体基因组序列n .椰子来自三个平台的注释使用DOGMA (Dual Organellar GenoMe Annotator) [74.].基因n lutea来自Sanger平台也被注释。对于一些具有很短的外显子的基因,例如PETB.和佩德,手工标注。我们使用tRNAscan-SE来确证tRNA边界[75.].使用GenomeVx绘制实体地图[76.].
密码子的使用
相对同义词密码子用法[77.] CP基因组n .椰子根据79个蛋白质编码基因的编码序列计算。IR区的基因仅计算一次。
系统发育分析
79个不同的肽编码基因被用于系统发育分析。除已测序的2个叶绿体基因组外,还选择了56个目的131个类群构建ML树。79个蛋白编码基因序列全部翻译为氨基酸序列,并在ClustalW中比对[77.]和手动调整。将这些基因的核苷酸序列限制在氨基酸序列上进行比对。那些发展速度更快、难以对齐的区域被排除在分析之外。连接67,862个核苷酸的FASTA文件(附加文件5)被产生,在附加文件中看到翻译的蛋白质编码序列6.我们提取了密码子的第一和第二位点,以及密码子的第三位点,形成另外两个新的矩阵。最后,利用三种基质进行系统发育分析。它们是使用我们的Perl脚本在DOS平台上建立的。
在推断系统发育树之前,对包含133个连接序列的核苷酸文件进行了测试。首先,在DAMBE中测试了取代饱和度[78.]横跨三个矩阵。结果,在P = 0.0000时,ISS
发散时间估计
对来自133个物种的79个基因序列和系统发育树进行了分子年代学分析。发散时间估计采用在PAML4.7的MCMCTree中实现的贝叶斯方法[81.].通过前一次纳入八个化石校准(附加文件7).采用近似似然计算方法,采用GTR + Γ5替代模型计算BASEML梯度g和Hessian H [82.].独立率(IR)模型[83.]和GTR + Γ5核苷酸替换模型在mcmctree中建立。ctl控制文件。为了确定是否达到收敛,两个独立的MCMC分析106丢弃10的刻录后的步骤5步骤,被模拟[84.],[85.].
提供支持数据的可用性
精确的叶绿体基因组n .椰子由PacBio RS II测序和Illumina MiSeq校正已提交到GenBank (accession KM655836)。的叶绿体基因组n .椰子和n lutea由Sanger测序的序列可在国家生物技术信息中心获取(登录NC_015610和NC_015605)。支持本文结果的其他数据集包括在本文及其附加文件中。
作者的贡献
YD和ZHW构思并设计了实验。ZHW、STG、ZWQ、LP、SZW和DQL进行了实验。WDK提供植物材料。本文由ZHW和YD撰写。所有作者阅读并批准了最终的手稿。
附加文件
参考
- 1。
刘静,齐志成,赵玉萍,付春霞,向清云:菝葜cpDNA全基因组序列与百合属系统发育定位——基因分区和分类单元取样的影响。中国生物医学工程学报。2012,32(3):457 - 462。10.1016 / j.ympev.2012.05.010。
- 2。
Sodmergen ZQ:为什么在Angiosperms中恢复胎儿塑料葡萄球菌葡萄酒?j植物res。2010,123(2):201-206。10.1007 / s10265-009-0291-z。
- 3。
Jansen RK, Cai Z, Raubeson洛杉矶,丹尼尔H, Depamphilis CW, Leebens-Mack J,穆勒KF, Guisinger-Bellian M, Haberle RC,汉森AK, Chumley TW,李某人,Peery R,麦克尼尔JR屈尔合资,Boore杰:分析从64年的81个基因在被子植物质体基因组解析关系和公司标识的进化模式。中国科学(d辑:地球科学),2007,33(4):531 - 534。10.1073 / pnas.0709121104。
- 4。
利用质体基因组规模数据分析基部被子植物之间的神秘关系。中国科学(d辑:地球科学),2007,33(4):593 - 598。10.1073 / pnas.0708072104。
- 5。
Moore MJ, Soltis PS, Bell CD, Burleigh JG, Soltis DE: 83个质体基因的系统发育分析进一步解决了eudicots的早期分化。中国科学(d辑:地球科学),2010,27(10):1423 - 1428。10.1073 / pnas.0907801107。
- 6。
Olmstead R,Palmer JD:Chorloplast DNA Systematics:方法和数据分析综述。我是J机器人。1994,81(9):1205-1224。10.2307 / 2445483。
- 7。
Li R,MA PF,Wen J,YI TS:完成五个araliaceae叶绿体基因组和系统发育的序列。Plos一个。2013,8(10):E78568-10.1371 / journal.pone.0078568。
- 8。
林CP,吴Cs,黄yy,Chaw Sm:Ginkgo Biloba的完整叶绿体基因组揭示了倒置重复收缩的机制。基因组Biol Evol。2012,4(3):374-381。10.1093 / GBE / EVS021。
- 9.
香蕉(Musa acuminata, Zingiberales)叶绿体基因组的完整:质体单子叶进化的深入研究。公共科学学报。2013,8 (6):e67350-10.1371/journal. pon.0067350。
- 10.
STRUB SC,Cronn RC,Edwards C,Fishbein M,Candon A:从线粒体的DNA水平转移到体液基因组及其随后的乳草(Apocynaceae)的演变。基因组Biol Evol。2013,5(10):1872-1885。10.1093 / GBE / EVT140。
- 11.
杨建斌,杨树新,李海涛,杨军,李德志:山茶属植物叶绿体基因组比较。公共科学图书馆。2013,8 (8):e73053-10.1371/journal.pone.0073053。
- 12.
易X,Gao L,王B,苏yj,王T:Cephalotaxus Oliveri(Cephalotaxaceae)的完整叶绿体基因组序列:脑毛细血管DNA的进化比较和洞察术中裸子植物中倒置重复拷贝的丧失。基因组Biol Evol。2013,5(4):688-698。10.1093 / GBE / EVT042。
- 13。
Crane Pr,Herendeen PS:含有Agiosperm花卉和北美洲的花群的白垩纪植物群。Rev Palaeobot Palyno。1996,90:321-340。10.1016 / 0034-6667(95)00090-9。
- 14。
Upchurch GR, Crane PR, Drinnan AN:弗吉尼亚州下白垩世波托马克群Quantico地区(Upper Albian)的巨型植物群。Mem Virginia Mus Nat Hist. 1994, 4: 1-57。
- 15.
中国莲属植物的分类与分布。(莲科)。《中国生物医学杂志》1994年第4期,第421-450页。
- 16。
一种有花植物的综合分类系统。哥伦比亚大学出版社,纽约;1981.
- 17。
若虫目的系统发生系统学。Bot Mag Tokyo. 1987, 100: 17-35。10.1007 / BF02488417。
- 18。
角叶科的起源和亲缘关系。分类学报。1988,37:326-345。10.2307 / 1222142。
- 19.
追逐MW, Soltis DE, Olmstead RG,摩根D, Les DH Mishler BD,杜瓦尔先生,价格RA,山汞、秋黄,Kron KA, Rettig JH,孔蒂E,帕尔默JD Manhart JR Sysma KJ, Michaels HJ,克雷斯WJ,卡罗尔公斤,克拉克WD Hedren M, Gaut BS,詹森RK, Kim KJ Wimpee CF,史密斯摩根富林明,Furnier GR,施特劳斯SH,湘QY,冷藏室通用,等:种子植物的系统发育:质体基因rbcL的核苷酸序列分析。安密苏里Bot Gard, 1993, 80: 528-580。10.2307 / 2399846。
- 20。
Hilu K,Borsch T,Muller K,Soltis de,PEA S:基于MATK序列信息的Angiosperm Phylogy推断。我是J机器人。2003,90:1758-1776。10.3732 / AJB.90.12.1758。
- 21.
基于三组分子数据:atpB, rbcL和18 S核糖体DNA序列的基础双二体的系统发育。安密苏里Bot Gard. 1999, 86: 1-32。10.2307 / 2666215。
- 22.
高丽君,李洪林:高丽参叶绿体全基因组序列及17种维管植物序列进化的比较分析。中国生物医学工程学报,2017,32(4):457 - 461。10.1093 / dnares / 11.4.247。
- 23.
Savolainen V, Chase MW, Hoot SB, Morton CM, Soltis DE, Bayer C, Fay MF, DE Bruijn AY, Sullivan S, Qiu YL:基于质体atpB和rbcL基因序列的开花植物系统发育研究。系统工程学报。2000,29(4):457 - 462。10.1093 / sysbio / 49.2.306。
- 24.
Soltis DE, Soltis PS, Chase MW, Mort ME, Albach DC, Zanis M, Savolainen V, Hahn WH, Hoot SB, Fay MF:从18S rDNA、rbcL和atpB序列推断被子植物系统发育。acta photonica sinica, 2012, 35(6): 741 - 748。10.1111 / j.1095-8339.2000.tb01588.x。
- 25.
Worberg A,Quandt D,Barniske A-M,LöhneC,Hilu KW,鲍尔斯T:基础奥德斯的系统发育:来自非编码和快速不断发展的DNA的见解。org潜水员evol。2007,7(1):55-77。10.1016 / J.ODE.2006.08.001。
- 26.
王Y,风扇GY,刘YM,太阳调频,CC,刘X,彭J,陈WB,黄XF,程科幻,刘YP,梁XM,朱HL、扁C,钟L, Lv T,董HX,刘WQ,钟X,陈J,关丽珍ZW,王,棕褐色,林CF,μF,徐X, Y叮,郭哦,王J,柯WD:神圣的lotus基因组提供了洞察开花植物的进化。植物学报,2013,76(4):557-567。10.1111 / tpj.12313。
- 27.
Goremykin VV, Hirsch-Ernst KI, Wolfl S, Hellwig FH: Amborella trichopoda叶绿体基因组序列分析表明,Amborella不是基部被子植物。中国生物医学工程学报,2003,20(9):1499-1505。10.1093 / molbev / msg159。
- 28.
白花睡莲(Nymphaea alba)叶绿体基因组:全基因组分析和最基础被子植物的鉴定问题。中国生物化学学报。2004,21(7):1445-1454。10.1093 / molbev / msh147。
- 29.
acta opsica sinica (acta opsica sinica), 2012, 34 (3): 442 - 442烟草叶绿体DNA。核酸学报1997,25:3681-3686。10.1093 / nar / 25.18.3681。
- 30.
Raubeson La,Peery R,Chumley Tw,Dziubek C,FourCade HM,Boore JL,Jansen RK:对比叶绿体基因组学:分析包括来自Angiosperms Nuphar Advena和毛茛属植物毛茛属植物的新序列。BMC基因组学。2007,8:174-10.1186 / 1471-2164-8-174。
- 31.
佐藤,中村,金子安,夏水泉拟南芥.基因工程学报,19(6):591 - 598。10.1093 / dnares / 6.5.283。
- 32.
Chin CS, Alexander DH, Marks P, Klammer AA, Drake J, Heiner C, Clum A, Copeland A, Huddleston J, Eichler EE, Turner SW, Korlach J:非杂交的,完成的微生物基因组组装来自长读SMRT测序数据。中国生物医学工程学报。2013,10(6):563-569。10.1038 / nmeth.2474。
- 33.
Quail MA, Smith M, Coupland P, Otto TD, Harris SR, Connor TR, Bertoni A, Swerdlow HP, Gu Y:三个下一代测序平台的故事:Ion Torrent的比较。太平洋生物科学公司和Illumina MiSeq测序仪。中国生物医学工程学报。2012,13:341-10.1186/1471-2164-13-341。
- 34.
Roberts RJ,Carneiro Mo,Schatz MC:SMRT测序的优势。基因组Biol。2013,14(6):405-10.1186 / GB-2013-14-6-405。
- 35.
karenn S, Schatz MC, Walenz BP, Martin J, Howard JT, Ganapathy G, Wang Z, Rasko DA, McCombie WR, Jarvis ED, Phillippy AM:单分子测序reads的混合错误校正和从头组装。生物工程学报,2012,30(7):693-700。10.1038 / nbt.2280。
- 36。
Loomis EID,EID JS,PELUSO P,YIN J,HICKEY L,RANK D,MCCALMON S,Hagerman RJ,Tassone F,Hagerman PJ:测序未曲折:扩展脆弱X基因的CGG重复等位基因。Genome Res。2013,23(1):121-128。10.1101 / gr.141705.112。
- 37。
Pugh TJ,Weeraratne SD,Archer TC,Pomeranz Krummel Da,Auclair D,Bochicchio J,Carneiro Mo,Carter SL,Cibulskis K,Erlich RL,Greulich H,Lawrence Ms,Lennon NJ,McKenna A,Meldrim J,Meldrim J,Ramos Ah,RossMG,Russ C,Shefler E,Sogachenko A,Sogoloff B,Sogjanov P,Tamayo P,Mesirov JP,Amani V,Teider N,Sengupta S,Francois JP,Northcott Pa,Taylor MD等:Medulloblastoma Exome测序源亚型 -特定的体细胞突变。自然。2012,488(7409):106-110。10.1038 / Nature11329。
- 38。
基于太平洋生物科学测序平台的微生物系统发育分析。微生物学报。2013,1(1):10-10.1186/2049-2618-1-10。
- 39。
Koren S, Harhay GP, Smith TP, Bono JL, Harhay DM, McVey SD, Radune D, Bergman NH, Phillippy AM:用单分子测序降低微生物基因组组装的复杂性。中国生物医学工程学报。2013,34 (9):1141 - 1146 /gb-2013, 14-9。
- 40。
Ferrarini M,Moretto M,Ward Ja,Surbanovski N,Stevanovic V,Giongo L,Viola R,Cavalieri D,Velasco R,Cestaro A,Sargent DJ:对PacBio Rs平台进行测序和叶绿体基因组的诺夫组装的评估.BMC基因组学。2013,14:670-10.1186 / 1471-2164-14-670。
- 41。
Metzker ML:测序技术 - 下一代。NAT Rev Genet。2010,11(1):31-46。10.1038 / NRG2626。
- 42。
Ricker N,Qian H,Fulthorpe Rr:用于了解原核适应和演化的组合草案的局限性。基因组学。2012,100(3):167-175。10.1016 / J.YGENO.2012.06.009。
- 43。
Moore MJ,Dhingra A,Soltis PS,Shaw R,Farmerie Wg,Folta Km,Soltis de:高级准确的Angiosperm Plastid Genomes的焦点测序。BMC植物BIOL。2006,6:17-10.1186 / 1471-2229-6-17。
- 44。
Straub SC, Fishbein M, Livshultz T, Foster Z, Parks M, Weitemier K, Cronn RC, Liston A:建立模型:利用低覆盖率基因组测序开发普通乳草(Asclepias syriaca)基因组资源。基因组学杂志。2011,12:211-10.1186/1471-2164-12-211。
- 45.
董伟,徐超,程涛,林凯,周胜:被子植物质体基因组测序:一套完整的通用引物和Saxifragales系统发育的实例研究。中国生物医学工程学报。2013,25(5):989-997。对10.1093 / gbe / evt063。
- 46.
Hirose T, Sugiura M: RNA编辑和RNA切割对烟草叶绿体ndhD mRNA的翻译是必需的:一种可能的调控机制,由功能不相关的基因组成的叶绿体操纵子的表达。中国海洋大学学报(自然科学版),1997,16(22):6804-6811。10.1093 / emboj / 16.22.6804。
- 47.
Tsudzuki T, Wakasugi T, Sugiura M:高等植物叶绿体RNA编辑位点的比较分析。中国科学:地球科学,2001,27(4):531 - 534。10.1007 / s002390010222。
- 48.
在盘基网柄细胞群体中,一种远距离自组织的自调节回路。自然科学进展。2005,33(7):423 - 426。10.1038 / nature03228。
- 49.
Kato, Kaneko, Sato S, Nakamura Y, Tabata S:豆科植物Lotus japonicus叶绿体基因组的完整结构。DNA资源学报2000,7:323-330。10.1093 / dnares / 7.6.323。
- 50。
张颖,张玉华,张玉华,张玉华,张玉华,张玉华,张玉华:帚状油菜科寄生性植物功能基因组和物理基因组的减少机制。植物学报,2013,25(10):3711-3725。10.1105 / tpc.113.113373。
- 51。
Funk HT,Berg S,Krupinska K,Maier UG,Krause K:两个寄生开花植物物种,Cuscuta Reflexa和Cuscuta Gronovii的完全DNA序列。BMC植物BIOL。2007,7:45-10.1186 / 1471-2229-7-45。
- 52。
寄生开花植物叶绿体基因组中光合作用和氯呼吸基因的丧失。自然。1999,348(6299):337-339。10.1038 / 348337 a0。
- 53。
张传聪,林慧琴,林叶,周天宇,陈洪辉,陈文辉,程春华,林春英,刘思明,张传聪,周思明:蝴蝶兰属植物叶绿体基因组与禾草进化速率的比较分析及其系统发育意义。生物化学学报,2006,23(2):279-291。10.1093 / molbev / msj029。
- 54.
翁曼丽,Blazier JC, Govindu M, Jansen RK:天竺葵科植物原始质体基因组的重建揭示了基因组重排、重复和核苷酸替换率之间的相关性。中国生物化学学报,2014,31(3):645-659。10.1093 / molbev / mst257。
- 55.
Wakasugi T, Tsudzuki J, Ito S, Nakashima K, Tsudzuki T, Sugiura M:通过测序黑松thunbergii的整个叶绿体基因组,确定了所有ndh基因的丢失。中国科学(d辑:地球科学),1994,21(4):794- 798。10.1073 / pnas.91.21.9794。
- 56.
Millen Rs,Olmstead RG,Adams Kl,Palmer JD,Lao Nt,Lao NT,Kavanagh Ta,Hibberd JM,Gray JC,Morden CW,Calie PJ,Jermiin LS,Wolfe Kh:Anviosperm Evolution期间叶绿体DNA的许多平行损失用多个独立的转移到核。植物细胞。2001,13(3):645-658。10.1105 / TPC.13.3.645。
- 57.
Delannoy E,Fujii S,Des Francs-Small CC,Brundrett M,小我:地下兰花的猖獗基因损失Rhizanthella Gardneri.突出塑性基因组的进化约束。mol Biol Evol。2011,28(7):2077-2086。10.1093 / molbev / msr028。
- 58.
Barth D,Berendonk Tu:Ciiliata副扫描素的线粒体基因组序列揭示了核苷酸组合物和阴性序列中的核苷酸组合物的转变。BMC基因组学。2011,12:272-10.1186 / 1471-2164-12-272。
- 59.
开花植物目和科被子植物系统发育类群的更新:APG III。引用本文:王志强,王志强。10.1111 / j.1095-8339.2009.00996.x。
- 60。
Kreunen SS,奥斯本JM:Nelumbo(Nelumbonaceae)的花粉和花药发育。我是J机器人。1999,86(12):1662-1676。10.2307 / 2656664。
- 61。
Drinnan An,Crane Pr,Hoot SB:非马酚二旋转龙(Eudicots)早期多样化的花卉演进模式。植物系统中的Evol。1994年,8:93-122。
- 62。
从多个基因推断被子植物系统发育是比较生物学的一个工具。自然科学进展。1999,402:402-404。10.1038/46528。
- 63。
Hansen Dr,Dastidar Sg,Cai Z,Penaflor C,Kuehl JV,Boore JL,Jansen RK:完全叶绿体基因组序列的四种早期分歧性植物植物的系统发育和进化意义:Buxus(Bulaceae),氯鱼(氯鱼植物),Dioscorea(Dioscoreacee)和illicium(schisandraceae)。mol phylocyet evol。2007,45(2):547-563。10.1016 / J.YMPEV.2007.06.004。
- 64。
Kim YK, Park CW, Kim KJ:韩国特有属大鼠叶绿体DNA的完整序列及其进化意义。中国生物医学工程学报,2009,27(3):365-381。10.1007 / s10059 - 009 - 0047 - 6。
- 65。
Sun YX,Moore MJ,Meng AP,Soltis PS,Soltis de,Soltis de,Li JQ,王HC:TrochodendRaceae的完全塑性基因组测序揭示了倒置重复的显着扩张,并表明两种外延物种之间的古常差异。Plos一个。2013,8(4):E60429-10.1371 / journal.pone.0060429。
- 66。
中B,Yonezawa T,Zhong Y,Hasegawa M:祖传草中叶绿体基因组的emisodic演变与调整。Plos一个。2009,4(4):E5297-10.1371 / journal.pone.0005297。
- 67。
Drew BT, Ruhfel BR, Smith SA, Moore MJ, Briggs BG, Gitzendanner MA, Soltis PS, Soltis DE:对被子植物根的另一个观察揭示了一个熟悉的故事。中国生物医学工程学报。2014,63(3):368-382。10.1093 / sysbio / syt108。
- 68。
从藻类到被子植物——从360个质体基因组推断绿色植物(Viridiplantae)的系统发育。BMC Evol Biol. 2014, 14: 23-10.1186/1471-2148-14-23。
- 69.
Bergsten J:对长分支吸引力的评论。克莱斯。2005,21(2):163-193。10.1111 / J.1096-0031.2005.00059.x。
- 70.
王荣军,程春林,常春春,吴春林,苏明明,周明明:单子叶植物叶绿体基因组中反向重复-大单拷贝连接的动态和进化。BMC Evol Biol. 2008, 8: 36-10.1186/1471-2148-8-36。
- 71.
Jia RZ, Ming R, Zhu YJ: linumbo nucifera Gaertn. Sacred Lotus (Nelumbo nucifera Gaertn.)抗病(R)基因的全基因组分析揭示了它们在陆地植物早期进化中的转变作用。热带植物学报。2013,6(2):98-116。10.1007 / s12042 - 013 - 9122 - 4。
- 72.
李家宽,周恩熙,李德兴,黄树清:亚洲莲花的北方复栖地。植物学报,2010,58(6):463-472。10.1071 / BT10002。
- 73。
Jansen RK,Raubeson La,Boore JL,De Pamphilis CW,Chumley TW,Haberle RC,Wyman SK,Alverson AJ,Peery R,Herman Sj,Fourcade HM,Kuehl JV,McNeal Jr,Leebens-Mack J,Cui Ly:方法获得和分析整个叶绿体基因组序列。方法苄醇。2005,395:348-384。10.1016 / S0076-6879(05)95020-9。
- 74。
基于DOGMA的细胞器基因组自动注释。生物信息学。2004,20(17):3252-3255。10.1093 /生物信息学/ bth352。
- 75。
Schattner P, Brooks AN, Lowe TM: tRNAscan-SE, snoscan和snoGPS网络服务器用于检测tRNAs和snoRNAs。核酸学报2005,33 (Web服务器版):W686-W689。10.1093 / nar / gki366。
- 76。
Conant GC,Wolfe KH:Genomevx:简单的基于Web的可编辑圆形染色体地图的创建。生物信息学。2008,24(6):861-862。10.1093 / Bioinformatics / BTM598。
- 77。
Tamura K,Peterson D,Peterson N,Stecher G,Nei M,Kumar S:Mega5:使用最大可能性,进化距离和最大判定方法的分子进化遗传学分析。mol Biol Evol。2011,28(10):2731-2739。10.1093 / Molbev / MSR121。
- 78.
夏旭,谢志:分子生物学与进化数据分析软件包。王志强,王志强,王志强,等。10.1093 / jhered / 92.4.371。
- 79.
Posada D,Crandall Ka:Modeltest:测试DNA替换模型。生物信息学。1998,14(9):817-818。10.1093 / Bioinformatics / 14.9.817。
- 80.
Stamatakis A,Hoover P,Rougemont J:RAXML Web服务器的快速引导算法。系统中的生物学。2008,57(5):758-771。10.1080 / 10635150802429642。
- 81.
杨Z:PAML 4:最大可能性的系统发育分析。mol Biol Evol。2007,24(8):1586-1591。10.1093 / molbev / msm088。
- 82.
杨志强,杨志强:离散时间贝叶斯估计系统演化的近似似然计算。中国生物化学学报,2011,28(7):2161-2172。10.1093 / molbev / msr045。
- 83.
杨志强:分子时钟对物种形成时间的影响。中国科学:地球科学,2019,39(3):457 - 461。10.1080 / 10635150701420643。
- 84。
杨Z:计算分子演变。牛津大学出版社牛津;2006年。
- 85。
Yang Z, Rannala B:基于软边界多化石标定的分子钟下物种分化时间的贝叶斯估计。中国生物化学学报,2006,23(1):212-226。10.1093 / molbev / msj024。
致谢
我们感谢伦敦大学学院的Mario dos Reis帮助使用PAML4.7包的MCMCTree。本研究由国家自然科学基金项目(31271310)资助。
作者信息
隶属关系
相应的作者
附加信息
相互竞争的利益
提交人声明他们没有竞争利益。
电子辅料
12870_2014_289_moesm1_esm.pdf.
附加档案1:133征集来自56个命令,包括APG III序数分类。(PDF 236 KB)
12870 _2014_289_moesm5_esm.pdf
附加文件5:DNA序列从系统发育分析中使用的133种的基因对齐。(PDF 9 MB)
12870_2014_289_moesm6_esm.pdf.
附加文件6:用于系统发育分析的133个物种的蛋白质的氨基酸序列比对。(PDF 5 MB)
作者的原始提交的图像文件
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。
除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。
要查看本许可证的副本,请访问https://creativecommons.org/licenses/by/4.0/.
创作共用及公共领域专用豁免书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
吴志强,桂顺,权志强。等等。精确的叶绿体基因组莲属椰子(Nelumbonaceae)用Sanger,Illumina Miseq和PACBIO RS II测序平台评估:深入了解基础才津代富的塑植演变。BMC植物BIOL.14,289(2014)。https://doi.org/10.1186/s12870-014-0289-0
收到了:
接受:
发表:
关键词
- n .椰子
- 叶绿体基因组测序
- 基底eudicots
- 系统的位置
- 散度的时间
- PacBio RS二世