摘要
背景
增加水稻产量潜力是水稻育种计划的一个主要目标,因为需要满足人口增长的需求,特别是在亚洲。利用基因组信息和高产品种进行遗传分析有助于理解水稻产量潜力的遗传机制。染色体片段替代系(CSSLs)是检测和精确定位大大小小的数量性状位点(qtl)的有力工具。此外,在两种亲本品种背景下形成的互反CSSLs可能适用于评估基因活性,作为一个单因素或上位相互作用。
结果
我们开发了从Takanari(最具生产力之一)的杂交中衍生出来的互惠cssl籼稻品种)和一个领先粳稻Koshihikari品种;这两个品种都是在日本培育的。41个CSSLs覆盖了Koshihikari背景下的大部分Takanari基因组,39个CSSLs覆盖了Takanari背景下的Koshihikari基因组。利用互惠CSSLs进行了稻田冠层条件下的产量试验。遗传分析发现,在小光和高光背景下,产量及其组成的qtl分别为48和47个。许多qtl表现出一种权衡,其中水槽大小性状(每穗小穗数或每平方米小穗数)增加的等位基因与成熟率或千粒重降低相关。这些结果表明,在这两种背景下,增加水槽大小并不足以提高水稻产量。此外,大多数qtl在两种遗传背景中的一种中被检测到,提示这些位点可能与其他基因处于上位控制状态。
结论
我们证明了互交CSSLs是理解高产水稻品种Takanari潜在产量潜力的遗传机制的有用工具。我们的研究结果表明,库大小的qtl与源强度或易位能力的qtl结合,以及对互作的重视是提高水稻产量的必要条件。本研究结果为今后选育具有高产潜力的水稻品种提供了基础。
背景
提高作物生产力是一项全球性挑战,是与全球人口增长保持同步的必要条件[1].世界上一半以上的人口在亚洲,那里种植水稻并将其作为主食消费[2].据预测,到2050年,亚洲的人口增长将要求水稻产量增加60 - 70%,但农业产量相应增加的空间不足[3.].为满足预期需求,有必要通过提高单位土地面积水稻潜在产量来增加水稻产量。
在热带地区,目前高产自交稻品种的产量潜力为10吨·公顷−1在良好灌溉条件下的无壳水稻[4].国际水稻研究所(IRRI)在20世纪60年代末发布的第一个现代高产品种IR8首次实现了这种产量潜力。在过去的半个世纪里,IR8和随后的高产品种的释放使水稻产量增加了一倍多。这种成功的增产被称为水稻的“绿色革命”[5].然而,最近热带环境的产量趋势表明,自IR8发布以来,产量潜力停滞不前。6].
在气候温和的日本,高产水稻已开发利用籼稻而且粳稻1980年代以来的品种[7].最新的产量试验使用了最近开发的高产品种,产量接近10 t·ha−1作为糙米(>12吨·公顷−1如未剥壳的水稻产量)在日本东部[8和>10 t·ha−1如日本西部的糙米[9].在个别试验中,糙米产量为11.7 t·ha−1在日本西部有报告[9].据我们所知,这是日本迄今为止记录的最高产量,是使用高产品种Takanari获得的籼稻品种。Takanari是由IR8等高产品种遗传而来的半矮型品种[10].生态生理学研究认为,由于高穗小穗数、强源特性(如高光合作用速率)和高碳水化合物易位能力,高根草具有较大的库尺寸[11] - [14].因此,了解高竹高产潜力的遗传机制对进一步提高高竹高产潜力具有重要意义。
在过去20年里,利用水稻全基因组序列的分子遗传学技术的进步促进了遗传分析,包括控制复杂性状的数量性状位点(qtl)的定位和克隆[15]、[16].染色体片段替代系(CSSLs)是一种具有遗传背景的特定供体染色体片段的遗传分析工具。CSSLs适用于检测具有大小效应的qtl,这些qtl通常被其他在初级群体中具有较大效应的qtl所掩盖,如F2群体和重组自交系[17]、[18].由于产量是一种高度复杂的性状,由大量的qtl控制,但效果很小,因此CSSLs有助于理解这一性状背后的遗传机制。迄今为止,已经在水稻中开发了几个用于不同杂交组合的CSSLs [17]、[19] - [23],包括相互的cssl [20.]、[21].互惠CSSLs的优势在于能够评估两种遗传背景下qtl等位基因效应的差异。然而,据我们所知,还没有利用互惠CSSLs进行水稻产量潜力的遗传分析。因此,开发利用Takanari进行产量试验的互惠CSSLs是一种很有前途的方法。
在本研究中,我们从Takanari和领先的Koshihikari的杂交中开发了互惠cssl粳稻通过重复回交、自花授粉和标记辅助选择(MAS)的方法获得的新品种。Koshihikari背景中的CSSLs由覆盖整个高竹基因组的41行组成,这些材料是检测高竹高产潜力的潜在qtl的有前途的材料。高竹本底的CSSLs由覆盖高竹全基因组的39行组成,这些CSSLs可用于检测高竹提高产量潜力的qtl。利用互反CSSLs的产量试验显示,在两种遗传背景下,许多与产量及其组成成分相关的qtl。本研究结果为今后培育具有高产潜力的水稻品种提供了基础。
方法
开发cssl
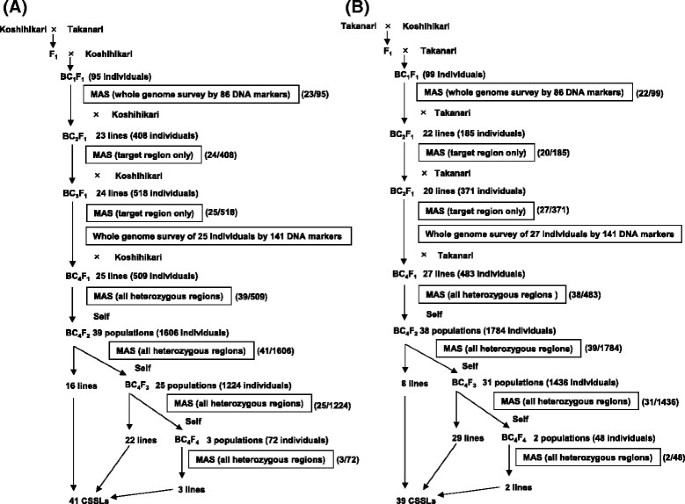
两个水稻品种,Takanari和Koshihikari,在日本开发(图1),使用图中总结的程序来开发互惠CSSLs2.我们对目标染色体段进行了重复的反向回交和前景(而不是背景)选择,直到BC3.F1的一代。从公元前4F1对所有杂合子区域进行调查,并结合前景和背景选择选择CSSLs。聚合酶链反应DNA标记物(n=141),包括先前开发的基因标记GN1a,sd1,APO1[10]、[16]、[19]、[24] - [26],用于MAS。
为了在Koshihikari遗传背景下开发cssli1从高光和高光杂交而来的植物被回杂交到高光,产生了公元前95年1F1植物。然后,我们用MAS选择BC 23年1F1基于分布在基因组中的86个DNA标记的基因型,植物携带一个或两个目标染色体段。这些公元前231F1植物再次回交到越光卡里生产BC2F1种子。我们后来发展到公元前408年2F1从公元前23年开始1F1植物,并选择公元前24年2F1携带一到两个杂合子靶段的植株,经MAS回交至越光,产生BC3.F1种子。同样的,公元前518年的25年3.F1从公元前24年开始2F1经MAS筛选的植株回交至越光,产生BC4F1种子。我们调查了公元前25年的基因型3.F1利用141个全基因组DNA标记对植物进行后续的靶标和背景选择。然后是公元前509年的39年4F1从公元前25年开始3.F1用MAS对杂合子的所有区域(包括目标段)进行筛选。为获得目标段高竹的CSSLs纯合子候选植物,公元前39年4F1植物是自花授粉的结果是公元前1606年4F2MAS对公元前41年的个体进行了调查4F2植物。非目标背景的杂合子片段保留在公元前25年4F2因此,为了减少背景中杂合子区域的比例,进行了额外的自花授粉和MAS。最终选取41株植物作为CSSLs(图2一个)。
Takanari遗传背景中的CSSLs是使用与Koshihikari背景相同的方法开发的(图2B).最后选择39株植物作为CSSLs。互惠CSSLs的种子可从水稻基因组资源中心(http://www.rgrc.dna.affrc.go.jp/index.html).
产量试验
2011年和2012年,在日本茨城县Tsukubamirai(36°02′n, 140°04′e)的NARO作物科学研究所的试验田进行了产量试验。土壤为Gleyic Fluvisols。在灌水条件下,将小光背景下的41个、高焰背景下的39个cssl与亲本栽培品种(小光和高焰)进行相互栽培。准备了两块水田,在每块水田中种植了每一个对等的csl。2011年4月26日和2012年4月25日在苗圃箱中播种,分别于2011年5月19日和2012年5月17日移栽(每山1株)。种植密度为22.2山m−2丘陵之间15厘米,行间30厘米。试验区(5.7 m2每个)被安排在一个随机的完整块设计中,有三次重复。基肥的施肥量为6克氮−2作为控释肥(2 g LP40, 2 g lp100, 2 g LP140), 5.2 g P m−27.5 g K m−2.在20-30°C条件下,LP40和LP140分别在施用后40天和140天内均匀释放其总氮含量的80%。LPs100在20-30°C施用后,以sigmoid速率释放其总氮含量的80%,最多可达100天。
离抽穗天数定义为每个clsl和亲本品种的5个植株从播种到第一个穗抽穗的天数。成熟期为9月中下旬,株长1.8米2(40座山)从每个地块收割,以确定产量及其组成部分。计数穗数,脱粒获得脱壳籽粒,称量后平均分成亚样品A和b,从亚样品A中选取约40 g的脱壳籽粒(亚样品C),使用电子种子计数器(KC-10S,藤原科学有限公司,日本东京)进行计数。单位面积的小穗数(m2)的计算方法为子样品C中的粒数除以子样品C的重量,再乘以单位面积脱壳的粒重。每穗小穗数用单位面积小穗数除以单位面积穗数计算。随后用稻壳机(25M, Ohya Tanzo G.K. Company,爱知,日本)将次样品B的稻壳取出,并对去壳的谷粒称重,以确定糙米产量。然后使用筛分机(TWS,佐竹株式会社,东京,日本)筛分1.6 mm,计算1000粒重。成熟率的计算方法是筛出的脱壳粒数除以单位面积上的小穗数。糙米产量和千粒重调整为含水量15%。测定成熟期各clssl和亲本5株植株的秆长。
统计和遗传分析
使用SPSS 22.0通用线性模型进行统计分析(IBM, Chicago, IL)。csl被认为是固定效应,年份和复制被认为是随机效应。采用方差分析(ANOVA)方法,考察了CSSL对产量及其组成因素的影响。基于方差分析结果,显著的csssl效应(P<0.05),采用Dunnett法测定产量及其组成成分。Dunnett’s检验中,Koshihikari在Koshihikari遗传背景中作为对照,Takanari在Takanari遗传背景中作为对照。为了勾画候选QTL区域,根据我们之前的研究,我们通过比较CSSLs之间的重叠片段来进行替代映射[17].
结果
互易型CSSLs的基因型
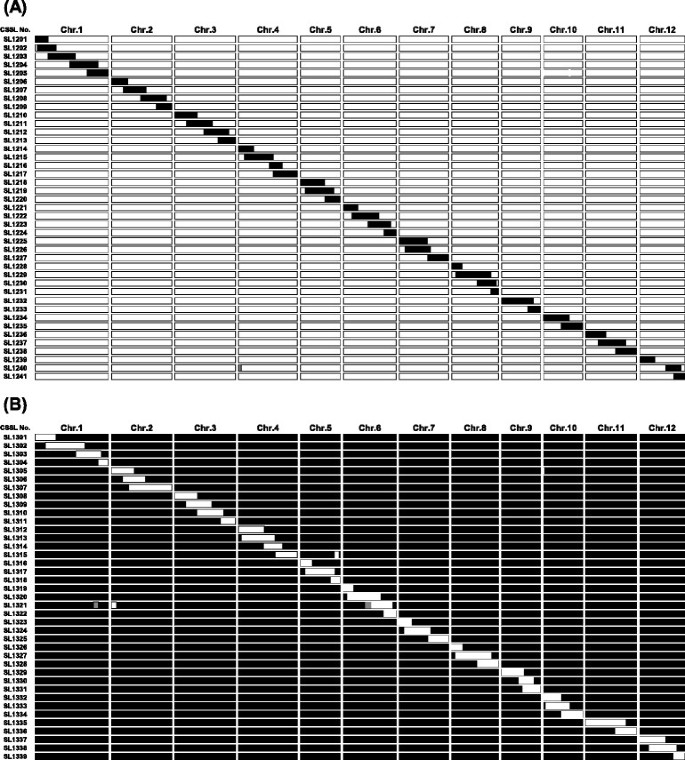
利用分布在12条水稻染色体上的141个DNA标记,测定了41个Koshihikari背景的CSSLs和39个Takanari背景的CSSLs的基因型图3.额外的文件1:图S1)。每个染色体都被2 - 5条携带重叠片段的细胞系覆盖,除了在越光背景的第12号染色体上的DNA标记RM2935和RM7344之间的一小部分区域,当我们为高雁选择携带片段纯合的细胞系时,由于不育而没有覆盖。大多数CSSLs只携带一个染色体片段。然而,在SL1240和SL1315的遗传背景中有一小部分被取代。SL1321还携带Koshihikari的两个杂合子段和一个纯合子段。每个csl的替换段大小在Koshihikari背景下从6.9 Mb到26.2 Mb,在Takanari背景下从7.4 Mb到27.1 Mb。
气候条件和对等CSSLs中天数到航向的变化
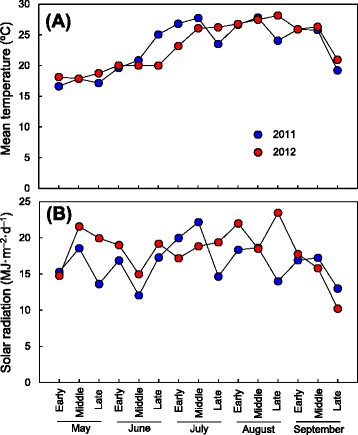
实验期间的平均气温与2011年和2012年相似,并随着季节的推移逐渐升高,直到9月中旬(图1)4A). 2012年5月下旬和8月下旬的太阳辐射较2011年相对较高(图1)4B). Koshihikari在播种后大约99天抽穗,Takanari在播种后102-103天抽穗2:图S2)。在越shihikari背景中,SL1222和SL1208分别比越shihikari早11天和晚11天左右到达。SL1222中较早的航向可能源自于即,因为SL1222中的取代段包含即[28].稍后将讨论SL1208中的后期标题。在剩下的39个cssl中,从95天到105天不等(在Koshihikari的6天内)。在Takanari背景下,SL1320和SL1323在实验条件下没有头部。在SL1320中没有标题可能是由即,因为SL1320中的取代段包含即[28].SL1323不抽穗可能是由一个新的QTL引起的,因为在第7号染色体短臂的取代区没有发现QTL。SL1335和SL1336分别比Takanari晚17天和29天。稍后讨论SL1335和SL1336的后期掘进。其余35个cssl在播种后97 ~ 108 d抽穗期,也在Takanari的6 d内。因此,我们认为大多数cssl和亲本品种生长在相似的气候条件下。
倒数CSSLs中的产量及其组成部分
Takanari的单位面积小穗产量比Koshihikari高出约40%,这是由于Takanari的穗数比Koshihikari少30%,但每穗小穗数却是前者的两倍5).尽管高粒重比高粒重低7%,但高粒重和小光果成熟率相同。最后,Takanari地区的糙米产量比Koshihikari地区高27%。
以往的研究鉴定和克隆了水槽大小性状(每穗小穗数)的qtl,包括GN1a[29),APO1[30.和半侏儒基因,sd1[31,作为一个单一基因。因为我们在这些基因中发现了Takanari和Koshihikari之间的序列差异3.图S3),我们首先关注基因的影响。SL1201和SL1202,携带的Takanari等位基因GN1a在光光背景下,每穗产量分别比光光多33%和38%,每平方米小穗产量分别比光光多22%和23%5A).但是,这两种CSSLs降低了成熟率和千粒重,最终糙米产量与越光无差异。同时,SL1301,携带的是GN1a在高花蓟背景中,每穗产生的小穗数比高花蓟少26%,每平方米产生的小穗数比高花蓟少20%(图5B). SL1301的成熟率和千粒重与高岭土相同,SL1301的最终糙米产量比高岭土低22%。类似的相互作用APO1观察每穗小穗数;的Takanari等位基因SL1223和SL1224APO1而SL1321和SL1322(含小穗光等位基因SL1321和SL1322)每穗的小穗数分别多出19%和13%APO1)每穗的小穗数分别减少17%和12%。然而,影响APO1没有导致最终糙米产量的变化。证实了茎秆长度对植株生长的相互作用sd1基因;SL1205(携带Takanari等位基因sd1而SL1303和SL1304(携带的是日本的Koshihikari等位基因)的秆比日本的短sd1与高根属植物相比,高根属植物的秆较长2:图S2)。然而,没有效果sd1在互作背景下观察了糙米产量及其构成因素。
除了cssl的携带GN1a,APO1,sd1,在Koshihikari遗传背景下,Koshihikari与部分cssli之间的糙米产量及其组成成分存在显著差异(图5A),以及Takanari和Takanari遗传背景中的一些CSSLs之间的关系(图5B).尽管一些CSSLs的产量成分为正,但它们在两种背景下的产量都没有显著高于重复亲本品种。例如,SL1310(高粒系)的每平方米穗数和小穗数比高粒系多19%,但成熟率和千粒重较低。因此,该区域的最终糙米产量与高雁区相近。
产量及其组成成分QTL定位
我们检测到的产量及其组分的qtl分别为48个和47个(图6).
在Koshihikari背景下,鉴定出3个穗数qtl,其中1个增加,2个减少6A).检测到12个每穗小穗数的qtl,其中7个对小穗数有积极影响,5个对小穗数有消极影响。考虑到qtl的作用和染色体区域,认为1号染色体短臂和6号染色体长臂上的位点为GN1a而且APO1,分别。每平方米小穗数共发现6个qtl;其中一半增加了小穗的数量,另一半减少了小穗的数量。鉴定的成熟率的5个qtl均对含有高竹属等位基因的植株有负面影响。千粒重检测到16个qtl;其中,有7个增加,9个减少了这种产量成分的值。6个与糙米产量相关的qtl被鉴定出来,它们都对携带Takanari等位基因的植株有负面影响(图6一个)。
在Takanari背景中,发现了4个穗数qtl,其中3个增加了穗数,1个减少了穗数6B).检测到9个每穗小粒数的qtl,且均对小穗小粒数有负面影响。考虑到qtl的作用和染色体区域,认为1号染色体短臂和6号染色体长臂上的位点为GN1a而且APO1,分别。每平方米小穗数有6个qtl;小穗数2个增加,4个减少。5个成熟率的qtl在带有小光光等位基因的植株中表现为负效应。检测到19个千粒重qtl;10个品种的千粒重增加,9个品种的千粒重减少。确定了4个影响糙米产量的qtl;所有这些都对带有Koshihikari等位基因的植株的产量有负面影响。
讨论
水稻产量是一个高度复杂的性状,由大量的qtl控制,个体效应小。CSSLs适用于检测具有大小效应的qtl,而互惠CSSLs具有评估两种遗传背景下qtl等位基因效应差异的优势。如果一个被检测的QTL在相互遗传背景下表现出相同的基因活性,则该位点应不与其他背景因子发生遗传互作或上位。在互反背景下表现出不同基因活性的qtl可能与其他背景因子参与遗传互作或上位作用[21].通过利用高光和高光杂交后代的互交CSSLs,我们检测了许多与糙米产量及其组成因素相关的qtl。在这些位点中,我们确认了高根的等位基因GN1a而且APO1增加了相互背景下每穗的小穗数(图5而且6).3号染色体上的单位面积小穗数QTL为RM3513, 8号染色体上的千粒重QTL为RM3634, 12号染色体上的千粒重QTL为RM1300,在两种遗传背景下均表现出相同的基因活性。这些结果表明,这5个qtl应该是一个单一的因素,除非与性状相关的多个基因位于取代段。因此,来自这些qtl的有利等位基因可以用于改善目标性状的互惠遗传背景。此外,最近的一项研究还克隆了8号染色体长臂上的一个QTLGW8控制粒度[32].因为千粒重QTL的位置接近GW8,本研究中检测到的QTL有可能为GW8.这需要进一步的研究来证实。
另一方面,其余qtl仅在两个遗传背景中的一个基因中检测到,提示这些位点可能与其他基因在背景中存在上位控制。在Koshihikari背景的第2染色体上RM3515-1位点和Takanari背景的第11染色体上RM1355位点的QTL集群观察到可能的上位相互作用(图6).这些QTL簇未在相反背景的相同基因组区域检测到。携带2号染色体上QTL簇的SL1208和携带11号染色体上QTL簇的SL1335和SL1336表现出杂种劣势(抽穗延迟、植株矮小、小穗少、成熟率低、产量低)(图)5额外的文件2:图S2)。之前的一项研究表明,杂交基因的破裂是由两个隐性基因的相互作用引起的,hbd2而且hbd3,以及Koshihikari所携带的hbd3和一个籼稻品种Habataki,它是Takanari的姐妹系,携带hbd2[33].的hbd2而且hbd3基因分别位于2号染色体和11号染色体QTL簇附近。假设Takanari也携带hbd2,在SL1208、SL1335和SL1336中观察到的两个QTL簇和杂交弱点可以很好地解释,因为SL1208应该携带hbd2而且hbd3在Koshihikari背景中,SL1335和SL1336也携带hbd2而且hbd3在Takanari的背景中。因此,两个QTL簇应该是相互作用的结果hbd2而且hbd3.虽然我们无法阐明仅在一种互作背景中检测到的其他qtl的基因相互作用,但仅在一种遗传背景中检测到许多qtl表明,高竹和越光之间的产量及其组成部分的很大一部分变异可能是由基因相互作用控制的。这是首次提出在互惠CSSLs的基础上对产量及其组成部分进行上位控制的可能性。需要进一步的研究来确定与检测到的qtl相互作用的背景因素。
qtl的图谱也揭示了产量组分之间的权衡关系。一个显著的例子是,与库大小增加相关的QTL等位基因与成熟率或千粒重降低相关。在Koshihikari背景的7个染色体区域观察到这种权衡(包括GN1a而且APO1)和Takanari背景中的6个(图6).这种权衡可能是由于缺乏来源强度或碳水化合物易位造成的,也可能是由于碳汇大小、来源强度和易位能力之间的不平衡造成的,最终产量没有增加。含有有利等位基因的NILs也缺乏显著的粮食增产GN1a而且APO1在其他粳稻遗传背景(34].然而,应该注意的是,亲本品种,Takanari,获得了高的成熟率,尽管从有利的等位基因的大汇大小GN1a而且APO1.高熟率被认为是由于高来源(高光合速率)和高碳水化合物易位能力[11] - [14].这些结果表明,增加源强度和易位能力的qtl应该存在于高竹属等位基因中GN1a或APO1在Koshihikari背景下获得更高的产量是必要的。最近,在高竹属植物中发现了一个与叶片高光合作用相关的QTL,并克隆为单基因(全球定位系统(GPS))的基因组合中,Takanari和Koshihikari的基因组合相同[35].我们目前正在开发金字塔线搬运GN1a而且全球定位系统(GPS)以测试在越光本底的产量增加。
虽然Takanari的等位基因GN1a在Koshihikari背景下,Koshihikari等位基因GN1a减少了库大小性状(每穗和每平方米的小穗数),从而降低了Takanari背景的糙米产量(图5B和图6B).这些结果表明GN1a是实现Takanari高产潜力所必需的。这些结果和高山草的强源也暗示了高山草的产量潜力可以通过扩大其汇的大小来提高。我们在高田背景中发现了一个增加3号染色体长臂上每平方米穗数和小穗数的QTL6B).携带该QTL的SL1310比Takanari每平方米多生产20%的小穗。这是一个很有前景的增加高竹菌库大小的QTL。然而,由于成熟率和千粒重降低,SL1310并没有获得比Takanari更高的糙米产量5B).这些结果表明,即使在Takanari背景下,也有必要将3号染色体上的QTL与增强源强度和易位能力的位点相结合,以提高产量潜力。尽管高蕉属植物的叶片光合作用低于高蕉属植物,但我们之前的研究发现,在高蕉属植物背景中,高蕉属植物的Koshihikari等位基因具有提高叶片光合作用的QTL [35].目前,我们正在将3号染色体上的QTL与高竹本底的高光合作用QTL结合,以测试高竹的增产潜力,并尝试克隆这两个QTL。
在高光和高光背景中分别检测到6个和4个产量相关qtl,但未检测到两种背景共有的产量相关qtl,且均为负效应qtl。如前所述,常见qtl的检测失败可能是由这些qtl的上位控制引起的。负面影响可能是由于一个QTL的替代引起的库大小、源强度和易位能力的不平衡。然而,本文给出的结果是基于在单一试验点进行的单一施肥处理的试验。由于产量经常受到环境条件的影响,需要在多种环境条件下进行进一步的试验,以确认本研究中检测到的qtl的效果。
结论
我们成功地从水稻品种Takanari和Koshihikari的杂交中获得了互惠CSSLs。通过互惠CSSLs的遗传分析证实了它们的有效性,并表明一些产量及其组分的qtl代表单一因素,而另一些则可能受上位相互作用的控制。替代作图还表明,在这两种遗传背景下,需要将库大小qtl与源强度或易位容量qtl结合,以提高水稻产量。研究结果为今后选育具有高产潜力的水稻品种提供了基础。
作者的贡献
TT和TY设计了这项研究并撰写了手稿。TT、TI、KK、YN、NO和TY形成了相互的CSSLs。TT、YS-A、SY、HN进行田间试验。MK和MY参与了研究的设计和协调。所有作者阅读并批准了最终稿件。
附加文件
参考文献
McCouch S、Baute GJ、Bradeen J、Bramel P、Bretting PK、Buckler E、Burke JM、Charest D、Cloutier S、Cole G、Dempewolf H、Dingkuhn M、Feuillet C、Gepts P、Grattapaglia D、Guarino L、Jackson S、Knapp S、Langridge P、Lawton-Rauh A、Lijua Q、Lusty C、Michael T、Myles S、Naito K、Nelson RL、Pontarollo R、Richards CM、Rieseberg L、ros - ibarra J等:农业:供养未来。自然学报,2013,499:23-24。10.1038 / 499023。
Khush GS:到2030年,养活50亿大米消费者需要多少钱?植物生物学杂志,2004,25(3):366 - 366。10.1007 / s11103 - 005 - 2159 - 5。
测试者M, Langridge P:在不断变化的世界中提高作物产量的育种技术。科学通报,2010,36(5):527 - 527。10.1126 / science.1183700。
Peng S, Cassman KG, Virmani SS, Sheehy J, Khush GS: IR8释放以来热带水稻产量潜力趋势及提高水稻产量潜力的挑战。作物科学,1999,39:1552-1559。10.2135 / cropsci1999.3961552x。
Evenson RE, Gollin D:评估1960 - 2000年绿色革命的影响。科学通报,2003,30(3):558 - 562。10.1126 / science.1078710。
彭硕,Khush GS, Virk P,唐强,邹勇:提高水稻产量潜力的理想型育种研究进展。作物学报2008,108:32-38。10.1016 / j.fcr.2008.04.001。
稻田K:水稻品种的历史变化。4.标题日本水稻品种的分类与分布。《水稻植物科学》(卷三)编辑:Matsuo T, Futsuhara Y, Kikuchi F, Yamaguchi H.东京粮食和农业政策研究中心;1997:854 - 875。
Yoshinaga S, Takai T, arai sanoh Y, Ishimaru T, Kondo M:新开发高产水稻品种的汇产和灌浆能力差异(栽培稻L.)的品种。农学通报,2013,38(3):447 - 447。10.1016 / j.fcr.2013.06.004。
永田K,佐佐木R,大田拉Y,吉永S:日本温暖地区高产水稻的生长、产量和干物质生产。作物学报,2009,78(增刊1):240-241。
Takai T, Arai-Sanoh Y, Iwasawa N, Hayashi T, Yoshinaga S, Kondo M:比较作图表明在水稻高产育种计划中重复选择相同的叶片光合速率数量性状位点。作物科学,2012,52:2649-2658。10.2135 / cropsci2012.03.0179。
陈CP,酒井H, Tokida T,臼井Y,中村H,长谷川T:富人总是变得更富吗?研究了高产水稻品种高凤梨叶片对自由空气CO的生理响应2浓缩。植物生理学报,2014,29(4):391 -391。10.1093 /卡式肺囊虫肺炎/ pcu009。
大美A,滨崎A,中川H,吉田H,白岩和T,堀文T:水稻叶片光合速率基因型和个体发育变异的解释模型(栽培稻)基于叶片含氮量和气孔导度。生物技术学报,2007,29(5):561 - 561。10.1093 / aob / mcl253。
高井T、松浦S、西尾T、大穗A、白岩T、堀江T:水稻生育后期产量潜力与作物生长速度密切相关。田间作物学报,2006,96:328-335。10.1016 / j.fcr.2005.08.001。
Taylaran RD, Adachi S, Ookawa T, Usuda H, Hirasawa T:水力导和氮积累在日本产量最高的水稻品种的叶片光合作用速率较高中发挥作用。中国生物医学工程学报,2011,32(4):366 - 366。10.1093 / jxb / err126。
Yano M:自然变异的遗传和分子解剖。植物学报,2001,4:130-135。10.1016 / s1369 - 5266(00) 00148 - 5。
基于地图的水稻基因组序列。自然学报,2005,436:793-800。10.1038 / nature03895。
Ebitani T, Takeuchi Y, Nonoue Y, Yamamoto T, Takeuchi K, Yano M:含有重叠染色体段的染色体段代换系的构建与评价籼稻水稻品种“Kasalath”的遗传背景粳稻精英品种“Koshihikari”。品种科学2005,55:65-73。10.1270 / jsbbs.55.65。
Keurentjes JJ, Bentsink L, Alonso-Blanco C, Hanhart CJ, Blankestijn-De Vries H, Effgen S, Vreugdenhil D, Koornneef M:植物近等基因系群体的发育拟南芥并与重组自交系群体的定位能力比较。遗传学报,2007,29(5):591 - 598。10.1534 / genetics.106.066423。
Ando T, Yamamoto T, Shimizu T, Ma XF, Shomura A, Takeuchi Y, Lin SY, Yano M:利用染色体片段替代系对水稻穗型数量性状进行遗传解剖和金字塔化。应用科学学报,2008,35(6):881-890。10.1007 / s00122 - 008 - 0722 - 6。
Hori K, Sugimoto K, Nonoue Y, Ono N, Matsubara K, Yamanouchi U, Abe A, Takeuchi Y, Yano M:利用回交群体检测收获前发芽抗性的数量性状位点粳稻水稻品种。中国生物工程学报,2010,29(5):557 - 557。10.1007 / s00122 - 010 - 1275 - z。
Kubo T, Aida Y, Nakamura K, Tsunematsu H, Doi K, Yoshimura A:水稻粳稻和籼稻杂交的互交染色体片段取代序列(栽培稻l .)。中国畜牧兽医学报,2002,32(4):319-325。10.1270 / jsbbs.52.319。
Takai T, Nonoue Y, Yamamoto SI, Yamanouchi U, Matsubara K,梁志伟,林HX, Ono N, Uga Y, Yano M:间回交衍生染色体片段取代系的发育籼稻供体水稻品种诺娜波克拉和粳稻受体品种“Koshihikari”。品种科学,2007,57:257-261。10.1270 / jsbbs.57.257。
徐娟,赵强,杜鹏,徐超,王斌,冯强,刘强,唐珊,顾敏,韩斌,梁刚:基于群体全基因组重测序的水稻高通量基因型染色体片段代换系(栽培稻l .)。中国生物医学工程学报,2010,26(6):656-10.1186/1471-2164-11-656。
McCouch SR, Teytelman L, Xu Y, Lobos KB, Clare K, Walton M, Fu B, Maghirang R, Li Z, Xing Y, Zhang Q, Kono I, Yano M, Fjellstrom R, DeClerck G, Schneider D, Cartinhour S, Ware D, Stein L:水稻2240个SSR新标记的开发与定位(栽培稻l .)。中国生物医学工程学报,2002,29(4):447 - 447。10.1093 / dnares / 9.6.199。
Monna L,北泽N,吉野R,铃木J,增田H,前原Y,谭吉M,佐藤M, Nasu S, Minobe Y:水稻半矮化基因的位置克隆,sd-1:水稻“绿色革命基因”编码一种与赤霉素合成有关的突变酶。DNA学报,2002,9:11-17。10.1093 / dnares / 9.1.11。
Terao T, Nagata K, Morino K, Hirose T:控制初级轴分枝数量的基因也控制维管束的形成,因此负责提高水稻的收获指数和产量。中国生物医学工程学报,2010,29(5):557 - 557。10.1007 / s00122 - 009 - 1218 - 8。
Kawahara Y, de la Bastide M, Hamilton JP, Kanamori H, McCombie WR, Ouyang S, Schwartz DC, Tanaka T, Wu J, Zhou S, Childs KL, Davidson RM, Lin H, Quesada-Ocampo L, vaillancourb, Sakai H, Lee SS, Kim J, Numa H, Itoh T, Buell CR, Matsumoto T:改进栽培稻使用下一代序列和光学图谱数据的日本基准基因组。水稻学报,2013,6:4-10.1186/1939-8433-6-4。
矢野M、片濑Y、足kari M、山内U、莫娜L、Fuse T、Baba T、山本K、梅原Y、长村Y、佐佐木T:即是水稻光周期敏感性的主要数量性状位点,与拟南芥花期基因密切相关君士坦斯.植物学报,2000,12:2473-2483。10.1105 / tpc.12.12.2473。
石野M、坂原H、林S、山本T、高志T、西村A、安吉尔斯尔、钱Q、北野H、松冈M:细胞分裂素氧化酶调节水稻产量。科学通报,2005,29(4):447 - 447。10.1126 / science.1113373。
Ookawa T、Hobo T、矢野M、村田K、安藤T、三浦H、浅野K、奥佳Y、池田M、西谷R、Ebitani T、尾崎H、Angeles ER、平泽T、松冈M:利用多效性QTL基因改良水稻抗倒伏和产量的新途径。通信学报,2010,1:132-10.1038/ncomms1132。
Sasaki A、ashhikari M、Ueguchi-Tanaka M、Itoh H、Nishimura A、Swapan D、Ishiyama K、Saito T、Kobayashi M、Khush GS、Kitano H、Matsuoka M:水稻赤霉素合成基因突变体。自然学报,2002,416:701-702。10.1038 / 416701。
王胜,吴凯,袁强,刘铮,林旭,曾锐,朱辉,董刚,钱强,张刚,付鑫:用OsSPL16在大米。中国生物工程学报,2012,38(4):359 - 359。10.1038 / ng.2327。
山本E,高志T,森中Y,林S,吴娟,松本T,北野H,松冈M, Ashikari M:水稻获得有害功能引起自身免疫反应和batson - dobzhansky - muller不亲和。中国生物医学工程学报,2010,29(5):559 - 561。10.1007 / s00438 - 010 - 0514 - y。
大隅A, Takai T, Ida M, Yamamoto T, arai sanoh Y,矢野M, Ando T, Kondo M:增加小穗数的水稻近等基因系产量性状评价。作物学报,2011,32(3):368 - 375。10.1016 / j.fcr.2010.08.013。
高井T,足立S,田口盐原F,三野新井Y,岩泽n,吉永S,广濑S,谷口Y,山内内U,吴J,松本T,杉本K,近藤K,伊卡T,安藤T,河野I,伊藤S,正村A, Ookawa T,平泽T,矢野M,近藤M,山本T:天然变体NAL1在高产水稻育种计划中选择的多向性增加了光合作用速率。科学通报,2013,3:2149-10.1038/srep02149。
致谢
这项工作得到了日本农林水产省的资助(基因组学促进农业创新,QTL1002和NVR-0001和基于基因组学的农业改进技术,RBS2005)。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明他们没有竞争利益。
电子辅料
12870 _2014_295_moesm1_esm.xlsx
附加文件1:图S1。:Genotype data for 41 chromosome segment substitution lines (CSSLs) in the Koshihikari genetic background and 39 CSSLs in the Takanari genetic background. Columns show CSSLs and rows show DNA markers. A, B, and H indicate homozygous for Koshihikari, homozygous for Takanari, and heterozygous, respectively. Approximate positions (Mb) of each marker are based on the physical map distance in Os-Nipponbare-Reference-IRGSP-1.0 [27].正向引物和反向引物序列分别为5 ' -CTCCATCCCAAAATAAGTTC-3 '和5 ' -GCTGGCCTGTCATCC-3 'GN1a5 ' -AGCTGGACATGCCCGTGGTC-3 '和5 ' -TTGAGCTGCTGTCCGCGAAG-3 '用于sd1和5 ' - ccggtttttttgtctag -3 '和5 ' -ATGAACACTGTCCAACAAATTGTTT-3 '用于APO1,分别。(XLSX 59 KB)
12870 _2014_295_moesm2_esm.pdf
附加文件2:图S2。:Days-to-heading and culm length of chromosome segment substitution lines (CSSLs) in the Koshihikari (A) and Takanari (B) backgrounds. Bars indicate mean values over two years. Dashed red lines denote trait values in Koshihikari (A) and Takanari (B). ***P< 0.001, * *P< 0.01, *P与Koshihikari (A)和Takanari (B)相比<0.05,由Dunnett检验确定。N / A,不可用。(PDF 38 KB)
12870 _2014_295_moesm3_esm.pdf
附加文件3:图S3。的序列多态性GN1a,APO1,sd1小光和高奈之间的关系。浅蓝色条代表外显子;白色条表示5 '和3 '未翻译区域。(PDF 33 KB)
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。
本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。
欲查看此许可证的副本,请访问https://creativecommons.org/licenses/by/4.0/.
创作共用公共领域奉献放弃书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
Takai, T, Ikka, T,近藤,K。et al。基于互交染色体片段替代系的水稻高产品种高根的遗传机制。BMC植物杂志14,295(2014)。https://doi.org/10.1186/s12870-014-0295-2
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-014-0295-2
关键字
- 染色体片段替代系(CSSLs)
- 数量性状位点(QTL)
- 大米
- 产量潜力