摘要
背景
植物已经进化了疾病抗性(R.用由损伤/白细胞介素-1受体(TIR)或卷轴 - 线圈(CC)结构域表示的N-末端编码用于核苷酸结合位点(Nb)和富含亮氨酸的重复(LRR)蛋白的基因。在此,在19次CC-NB-LRR和TIR-NB-LRR编码基因的存在和多样化的基因组研究,以及19中更短的域组合拟南芥蒂利亚纳加入和拟南芥,风疹芥,甘蓝和Eutrema salsugineum呈现。
结果
在528中R.分析的基因,19个CC-NB-LRR和17次TIR-NB-LRR基因在19中被保守答:芥基因型,而只有两个CC-NB-LRR,包括Zar1,5个物种比较,保留了3个tir - nb - lrr。这耐瘦性磷酸耐药3(RLM1.)位点具有对芸苔属病原菌的抗性L. MAMULANS.黑腿病的致病因素,尤其在我国经历了保护和多样化事件b·拉伯.相反,RLM3.免疫反应中重要的位点葡萄孢菌和链格孢属SPP。最近在演变中拟南芥属。
结论
我们的全基因组分析R.23个十字花科植物基因组序列差异较大。这些数据进一步深入了解了影响这一重要基因家族的进化过程。
背景
作为无梗死生物,植物适应其变化的周围环境,其存活主要是基于及时进化的免疫反应。在植物细胞表面发生第一防御线,识别保守的微生物组,例如脂多糖和肽聚糖,通常曾因于病原体或微生物相关分子图案(PAMPS / MAMPS)。通过同源模式识别受体(PRRS)识别MAMPS,并引发直接免疫应答,导致基底效果触发的免疫(PTI)[1],[2].已知的PRRS落入两种受体类中的一种:跨膜受体激酶和跨膜受体样蛋白,后者缺乏任何表观内部信号域的蛋白[3.].值得注意的是,PRRS是蛋白质磷酸酶和其他调节蛋白的紧密控制下血浆膜的多素蛋白复合物的组分[4.].在许多情况下,特异的病原体能够克服基础的PTI,要么绕过PTI的检测,要么通过延迟、抑制或重编程宿主细胞内的效应分子来干扰PTI。作为一种对抗机制,部署的细胞内抗性(R)蛋白直接或间接检测这些效应剂的存在,从而导致效应剂触发免疫(ETI)。rpm1 - interaction PROTEIN 4 (RIN4)在前一种情况下是一个被充分研究的关键角色[5.],[6.,而直接的互动可以用R.水稻的基因和效应magnaporthe oryzae.Pathosystem [7.],[8.].
植物抗性蛋白是模块化的,也就是说,它们由保守成分的组合组成,有些保守成分具有与动物共享的特征。9.] - [11.].大多数R蛋白通常由核苷酸结合位点(NB)和c端可变长度的富亮氨酸重复结构域(LRR)组成。这些NB-LRR蛋白根据其n端序列分为两类,其中一种是螺旋螺旋(CC)序列,另一种是具有序列相似性的结构域果蝇黑胶基TOLL和人白细胞介素-1受体,简称TIR。这些保守的序列块在整个进化过程中一直保持着,并且仍然可以在真细菌、古细菌、后生动物和苔藓植物等多种生物中被识别出来[12.].尽管高度保守,R蛋白提供了对广泛的植物病原体的抗性,包括病毒、细菌、真菌、卵菌和线虫[13.] - [15.].
Nb编码抗性基因已在许多单子叶和二萝萝卜物种中注释拟南芥蒂利亚纳[16.].目前丰富的植物基因组测序已经揭示R.基因是最大的植物基因家族之一。在参考基因组中答:芥,149 r-proteins含有LRR基序,其中83由TIR-NB-LRR和51组成,具有CC-NB-LRR结构域[17.],[18.].一些较短的蛋白也存在,由19个TIR-NB编码基因和30个TIR-X结构域代表的一个或两个结构域。总的来说,答:芥大约有200个蛋白质含有1到3个R.基因相关蛋白结构域组合。
在这项研究中,我们利用加速基因组信息答:芥并进行全基因组分析R.基因19答:芥基因组。我们进一步扩展了分析,包括相关的基因组拟南芥,风疹芥,甘蓝和Eutrema salsugineum物种。此外,我们选择了两个潜在的抵抗力芸苔属植物以追踪真菌病原体的进化模式。我们发现R.基因在里面形成了一个核心答:芥,而少数五R.从五种不同物种的基因组中检索基因。这五个基因中的一个,HOPZ-ACTIVATED电阻1(ZAR1.)已知具有新的信号传导要求的基因也存在于ROSID思工内的其他植物家族中。这耐瘦性磷酸耐药3(RLM1.)基因座部分被保守答:lyrata和c .风疹大大多样化b·拉伯和e . salsugineum而RLM3.洛卡最近在进行中拟南芥属。这项工作提供了方面R.比较基因组分析中参考基因型的基因多样性及选择。
结果
19 A.拟南芥基因组中存在核心29 r基因的核心组
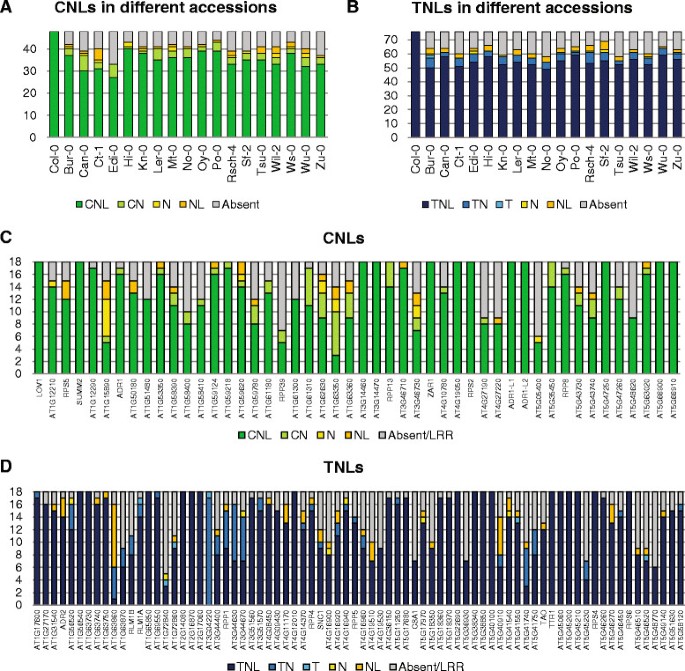
深入了解级别R.基因保护A. Thaliana,我们分析了Col-0和18额外附加的参考基因组(Bur-0,CAN-0,CT-1,EDI-0,HI-0,KN-0,L呃没有0,0,Mt-0 Oy-0、Po-0 Rsch-4, Sf-2, Tsu-0, Wil-2, Ws-0, Wu-0 Zu-0) [19.].选择这18个基因组的主要原因是它们的序列质量、高覆盖率、RNA测序数据新创组装。预测148个nb - lrr编码基因的Pfam同源性和线圈服务器搜索[18.]导致124的减少清单R.Col-0中的基因进一步分析,包含48个CC-NB-LRR(CNL)和76次TIR-NB-LRRS(TNL)(附加文件1:表S1)。在97 (edi0)至109 (Hi-0和Po-0)之间R.在新测序的18的基因组内发现基因答:芥进入(图1a,b)。没有额外的R.在18个基因组的trace sequence archive中发现了除Col-0外的其他基因。
NB-LRR编码域结构的多样性R.基因18答:芥与Col-0。在(一种)编码全长或碎片的CC-NB-LRR(CNL)基因的基因数量,以及(b)编码全长或碎片的TIR-NB-LRR(TNL)基因的基因数。124核的分布答:芥Col-0.R.基因18答:芥加入,在(C)CNL基因和(D)TNL基因。对于基因名称,请参阅支持信息附加文件2S2:表。只编码LRR的基因与缺失的基因一起分组。
在Col-0中的48个CNL编码基因的比较中,在所选的载体中回收27(EDI-0)至40(HI-0)(图1一种)。剩余基因的蛋白质产物在Col-0中的CNL蛋白质中缺少一个或多个结构域(CN,Nl,N或L),或者在至少一个加入中完全不存在(图1C)。没有包含的已知防御相关基因的代表RPS5.在edi0, No-0和Sf-2,和ADR1.在zu-0。对于基因缩写,请参阅附加文件2S2:表。在TNL组中,完全TNL基因的数量在49(NO-0)和59(PO-0和WU-0)之间变化(图1B,D)。缺失基因的实例是RPP5.在Ct-1, Mt-0, Oy-0和Wu-0SNC1.在CAN-0,EDI-0,NO-0,RSCH-4,TSU-0和WU-0中。
总之,分布相当广泛R.在19例中发现了基因序列答:芥登记入册。在124个编码中R.col0基因中有41个基因与其他18个基因同源。然而,这些基因中有12个至少在一次添加中缺少一个或两个结构域。例如,RPP13在NO-0,RSCH-4,WIL-2和ZU-0中丢失了其LRR域。在剩余的12个CNL和17 TNL编码基因的核心组中,所有这些都随机分布在基因组上(附加文件3.:图S1),九个基因(ADR1-L1那ADR1-L2那LOV1, RPS2那rps4.那rps6.那萨姆2.那TTR1和ZAR1.),已知涉及各种植物防御反应。
5个NB-LRR基因在5个成员中是保守的十字花科家庭
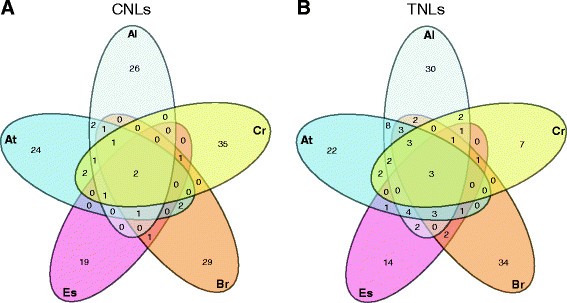
扩展分析R.基因在答:芥,我们监控了可能的保护R.基因跨谱系十字花科代表答:lyrata那c .风疹那b·拉伯和大肠salsugineum.PFAM同源性和线圈服务器搜索用CNL或TNL架构识别404蛋白(附加文件1:表S1)。预测的CNL和TNL编码基因数量差异较大:大肠salsugineum(67),c .风疹(75),答:芥COL-0(124),答:lyrata(127),和b·拉伯(135),不反映各种物种中预测基因模型的基因组尺寸或数量的数字。
通过CNL和TNL序列的NB域系统发育分析,鉴定了5个物种的同源序列。在由此产生的系统发育树中,形成了至少两种植物的57个分支(附加文件)4.:图S2和附加文件5.:表S3)。在该57片中,还发现来自单一物种的多拷贝基因在该特定物种中被鉴定为逐渐寄生序列。没有解决57个枝条外的序列的位置。在直晶序列中,观察到TNL组的偏差,76分中有52个答:芥在一种或多种物种中具有正直的TNL序列,而48个CNL中只有17个具有正直。不包括旁级基因,之间鉴定了最多的正交序列答:芥和答:lyrata(数字2),与早期调查结果同时[20.],[21.].从答:芥29个基因的核心组,7个CNL和9个TNL基因也发现了两种或更多种物种中,包括ADR1-L1那ADR1-L2那RPS2.那rps6.那TTR1和ZAR1..
R基因同源性之间答:芥那答:lyrata那c .风疹那b·拉伯和大肠salsugineum.在(一种)CNL的直系和在(b)直际TNL序列答:芥col-0(在),答:lyrata(AL.),c .风疹(Cr),b·拉伯(布尔) 和大肠salsugineum(西文).来自系统发育分析的数据(附加文件4.:图S2)。
鉴定了总共有两个CNL片状和三种TNL片状来自所有五种物种的序列。只有其中一个的片状(第5条;附加文件4.(图S2)包含一个与防御反应有关的基因,称为ZAR1.并且需要被认可假单胞菌含油T3se Hopz1A效应器[22.].ZAR1.在Rosid Clade内有几种物种的同源物,以及葡萄和Solanum.物种,以及我们的数据集ZAR1.保守良好,有一个卡/Ks比例为0.4支撑纯化选择。在同一地区发现另外两种基因,AT5G66900和AT5G66910(第12条;附加文件4.:图S2),表明它们之间是平行的,可能有冗余的函数。在这个进化枝,b·拉伯和大肠salsugineum分别有3个和2个基因,而来自答:lyrata和c .风疹.CDS序列的系统发育分析表明,在五种物种之间只保守AT5G66900基因(附加档案6.:图S3)。这RPS2.基因较早在几种芸苔属植物中发现,包括b·蒙大拿那b·拉伯和B. Oleracea.[23.],[24.]那它最有可能成为同源物(945467,身份为94%)答:lyrata[20.].在我们的数据集中答:芥RPS2还发现了基因大肠salsugineum但不是在c .风疹.然而,通过对BLASTN同源性的搜索,揭示了两者之间的相似性RPS2.以及一个在反义链上注释的区域,作为一个没有任何结构域的基因c .风疹(carubv10005994m)。88.7的高相似性和同一性提出了五种物种之间存在的第三种CNL基因。
综上所述,同源性与两个CNL基因(At3g50950和At5g66900),并可能添加RPS2.3个TNL基因(At4g19510、At5g45230、At5g17680)均存在于5个物种中。在19个基因组中答:芥在这个特定的基因组比较中,只有CNL基因是保守的。5个保守基因中的4个没有已知的功能,包括它们的同源基因。
RLMlocus基因的保存与多样化
L. MAMULANS.是一种血渗真菌病原体和广泛的Blackleg病的因果因子芸苔属植物作物[25.].这RLM1.轨迹in.答:芥Col-0早期被鉴定为在免疫反应中发挥重要作用[26.]并包含七个基因,其中TNL架构跨越AT1G63710和AT1G64360(附加文件7.:图S4)。两个基因,RLM1A和RLM1B被发现负责RLM1.活动,RLM1A作为免疫反应的主要参与者[26.].剩下的5个还没有函数RLM1C-RLM1G基因。在几种情况下,已经证明了不同载体中的抗性基因座的多样化[21.],[27.],[28.]并扩大我们的知识RLM1.,我们研究了存在和多样化RLM1.在我们的基因组数据集中。
在这里,我们发现了RLM1A出席全部18场答:芥编码所有三个域的14个accessions (Can-0, Ct-1,艾迪-0,Hi-0, L呃-0,MT-0,No-0,PO-0,SF-2,TSU-0,WIL-2,WS-0,WU-0和ZU-0(附加文件8.:表S4)。这与它们的抗性表型是一致的[29.].一般来说RLM1A17份供试材料的基因变异位点较少RLM1A在Col-0(P-距离0.2 ~ 0.9%)。Ws-0是非典型的,与之相比有230个可变位点RLM1A在col0中导致aP-距离为13.8%(图3.A和附加文件9.:表S5)。没有RLM1A同源物在答:lyrata那b·拉伯和大肠salsugineum基因组。一个RLM1A候选人被发现未注释c .风疹LRR区域的基因组序列和RNA表达数据[30.表明该基因表达,并可能在防御反应中发挥潜在作用。为了支持我们的发现,PCR扩增和测序RLM1A地区答:lyrata那b·拉伯和c .风疹确认只有c .风疹坚持了rlm1a。b·拉伯物种不知道抵抗抵抗力L. MAMULANS.[31]除了杂草亲戚b·拉伯ssp。结果表明[32],[33].为了弄清RLM1A我们使用RLM1A用于扩增该区域的特定引物显著简历。超过400种野生抗性性状b·拉伯相对,基因祖细胞,以及比较,已知的敏感b·拉伯基因型。在这里,只b·拉伯ssp。结果表明含有高度相似的基因组序列RLM1A基因答:芥(身份81%)。
TNL基因内部RLM1.Locus,TN基因19答:芥加入和答案RLM3.轨迹。在(一种)P-不同的距离TNL.编码蛋白质RLM1.轨迹在19答:芥登记入册。有关个体基因值的详细信息,请参阅支持信息附加文件9.:表S5。TIR-NB编码的域架构多样性R.基因18答:芥与Col-0相比的进一步(b)全长或碎片的TIR-NB(TN)基因,以及(C)在18中分布11COL-0 TN蛋白答:芥登记入册。只编码LRR的基因与缺失的基因一起分组。(D)同时在这方面RLM3.座位之间答:芥Col-0,答:芥Kn-0,答:lyrata(AL.),c .风疹(Cr),b·拉伯(布尔) 和大肠salsugineum(西文).*早期停止密码子;**RLM3.Rsch-4、su-0、will -2、Ws-0和Wu-0与Kn-0相同。
这RLM1B基因在免疫应答中具有次要作用,并侧翼RLM1C和RLM1D.与Col-0相比,这三种TNL基因编码缺乏一个或多个域中的蛋白质,特别是RLM1D(附加文件8.:表S4)。一个可能的候选人直观RLM1C在c .风疹但是使用诠释答:芥作为比较,潜在基因有多个终止密码子。相似性被发现RLM1B来RLM1C基因组中的基因答:lyrata那b·拉伯和大肠salsugineum(附加文件7.:图S4)。由于物种间缺乏同源性,该染色体区域似乎处于积极选择状态,显示出减少RLM1B来rlmd.基因内答:lyrata和大肠salsugineum.在b·拉伯相反,用五个TNL和一个TN基因观察到膨胀RLM1B-RLM1D区域,显示出与RLM1B和RLM1C基因的答:芥Col-0。
最保守的序列答:芥加入是RLM1E,F和G只有少量修饰的基因(P-距离0.5-0.8%)(附加文件9.:表S5)。进一步的保护观察RLM1F和RLM1G在答:lyrata,后者含有两个orthologs的RLM1F和RLM1G基因卡/Ks与...相比,1.3和0.8的比率答:芥Col-0。此外,相似性被发现RLM1G到基因组区域c .风疹(卡/Ks比率为0.7)和转录数据先前透露了这一点RLM1G表达了c .风疹[30.].在B. Rapa,发现五个编码基因被发现直观RLM1F和RLM1G(疏浚21,附加文件4.:图S2),但在RLM1.轨迹。其他三种编码基因位于其他地方,没有同步RLM1.轨迹。未发现同源性RLM1E来RLM1G基因在大肠salsugineum.
总的来说,在答:芥登记入册的RLM1.基因座在RLM1E来RLM1G地区和似乎经历了多样化RLM1A来RLM1D序列拉伸。例外是wu-0,其中RLM1.轨迹非常相似于RLM1.Col-0的轨迹,只有平均值P.-距离0.2%(附加文件9.:表S5)。在其他四个物种中,有几个RLM1.与...相比,基因经历了多样化答:芥对彼此也一样。保守的人是个例外RLM1G同时答:lyrata和c .风疹和RLM1F在答:lyrata尽管RLM1A也被发现c .风疹.
rlmlocus是A. Thaliana和A. Lyrata的独特之处
这RLM3.基因在免疫反应中起着重要作用,不仅仅是对L. MAMULANS.但也葡萄孢菌和链格孢属物种 [34].该基因编码TIR和NB结构域,但缺乏LRR结构域。相反,c端包含三个DZC(抗病、锌指、染色体凝聚)或BRX结构域(Brevis Radix.)最初描述在根本发展中具有作用[35].除了RLM3,18基因答:芥COL-0包含没有LRR域的TN基因[18.].然而,RLM3.是唯一的TN基因答:芥包含BRX域的参考基因组。获得更多关于编码基因的更多洞察力答:芥使用COL-0,PFAM同源性和线圈服务器搜索。这被设计为排除具有截短的TIR或NB结构域的基因,导致11N基因(附加文件1:表S1)。另外18个基因中进一步研究了TN编码基因的存在答:芥基因组。
总体而言,我们在六(WIL-2)和11(HI-0,PO-0和ZU-0和ZU-0)之间发现,编码整个TIR和NB域(图3.在Col-0的11个TN基因中,有7个存在于所有18个供体中,其中3个编码完整的TN。其余4个基因在至少一个供体中编码修饰(T或N)(图)3.C). At1g72850在大多数植物(Can-0, Edi-0, Mt-0, No-0, Oy-0, will -2和Ws-0)中不存在,bur0, Ct-1和Sf-2中仅编码TIR域。当我们扩展Pfam同源性搜索时,我们发现了7个TNs答:lyrata,一个在c .风疹, 16岁b·拉伯无TN编码基因大肠salsugineum.在系统发育树中,鉴定了五个具有同源蛋白的分支(附加文件)4.:图S2)。没有一个进化支含有所有四种物种的蛋白质。
一个完整的RLM3.19例中有13例存在序列答:芥鉴定了包括COL-0和缺少一个或多个域的成绩单。高卡/Ks2.3的比率表明RLM3.在13种附加方面处于正面选择。检查跨越致染色体区域RLM3.轨迹透露,在六种附加(KN-0,RSCH-4,TSU-0,WIL-2,WS-0和Wu-0)中,Col-0中大约8,200bp完全不存在,而侧翼基因;AT4G16980和AT4G17000存在(图3.d)。AT4G17000基因具有经历的突变和小缺失,导致早期的止损密码子。在Col-0基因组序列中未发现在AT4G16980和AT4G17000之间的大约400bp在这六种载体之间显示较小的多态性,表明删除缺失RLM3.由单一事件产生。
一种RLM3-类似的基因答:lyrata(进化枝。3;额外的文件4.:图S2)建议存在RLM3.在分裂之前答:芥~ 13米娅(36].相比之下,没有RLM3.同源物在c .风疹那b·拉伯和大肠salsugineum基因组序列。来进一步追溯其可能的来源RLM3.,BRX结构域用于系统发育分析,但不能发现王国植物(额外文件)内的序列10.:图S5)。我们得出这样的结论:RLM3.最有可能完全在属于属内进化拟南芥.
讨论
在本报告中,我们描述了对大型的基因组调查R.19岁的基因家庭答:芥植物资源及4个近缘种十字花科家庭。比较的答:芥加入揭示了基因数的巨大变化和LRR结构域的偏置损失。有趣的是,Col-0基因组最多R.数据集中的基因密集添加。我们检查了附加基因的重测序和基因注释过程中的偏差答:芥基因型,但无法确定任何明显的丧失解释R.基因。这与最近的基因组研究一致新创180年大会答:芥新序列1.3-3.3 Mb,每个基因型增加200-300个基因[37].然而,这些差异主要是由于45S rDNA的拷贝,而不是新的R.报道了Col-0中缺席的基因。
Col-0是Col-1的直系后代,根据其肥力和旺盛的植物生长从Landsberg种群中选择[16.].同样的种群被用于辐射实验,导致了Landsbergerecta.登记入册(左呃).现在已经很清楚,原来的兰德斯堡种群包含了略微不同的基因型的混合物,这解释了观察到的差异R.col0和L呃-0。遗传变异答:芥在我们的数据集中观察到的登录历史悠久的历史悠久R.基因定位和克隆。抗性基因的特性P. inringae.(rpm.那rps.) 和...一起rpp.基因到卵母细胞Hyaloperonospora Arabidopsidis.一直走在前沿,也促进了对病原体效应剂相互作用的理解。这RPP1.WS-0和ND-1加入的轨迹识别不同H. Arabidopsidis.分离株,一种导致发现无流动基因的观察ATR1和六个不同的等位基因[38].序列对齐ATR1同期基因Phytophthora sojae.和p . 5反过来揭示了RXLR易位核心图案,将另一个尺寸添加到宿主病原体对和效应生物学的遗传构成。
在18个国家中答:芥大量的R.与COL-0相比,基因缺少一个或多个结构域,随着LRR结构域的损失是最常见的改变。将LRR序列与基因转化,结构域交换和缺失事件的调节有关植物与病原体共同发展的策略。LRR结构域已在各种细菌,蛋白质和真菌物种中鉴定,共同代表成千上万的基因[12.].使用Nb结构域的LRR结构域的融合是比具有受体样激酶的LRR融合更新的原点,只能在土地植物谱系中看到。建议LRR域已演变几次,导致八个特定类,其在LRR域的可变段内的序列长度和相似性不同[39],[40].称为植物特异性LRRS的LRR类别之一已被证明在几个R蛋白中的不同选择下[41] - [44].这种类型的序列多样化很可能反映了与病原体效应体(已知与LRR基序直接或间接相互作用的蛋白质)的共同进化[7.],[45] - [47].还证明了存在或不存在特定LRR域的重要性。在缺少的情况下P. inringae.效应器AVRPHB,LRR域RPS5.抑制CC和NB域的活性[48].因此,LRR抑制因子活性的丧失导致植物细胞因结构性死亡RPS5.活动。因此,没有一个RPS5.在我们的数据集中,同源物缺少LRR域。RPS2.那rps4.和rps6.序列之间高度保守,LRR域的多态性程度较低(Ka / Ks比率在0.64和0.76之间)。的情况下rps4.LRR结构域对蛋白质稳定性很重要,但它缺乏抑制活性,比如RPS5.[49].
在许多人答:芥我们找到了数据集中的数据R.编码二部分蛋白的基因,与Col-0相比,通常以LRR结构域的缺失为代表。这类tn编码基因被推测为与TNL蛋白或下游信号元件相互作用的适配器蛋白[17.].例如,PBS1,一个重要的球员RPS5.与TN蛋白相互作用[50].CN和TN基因是否在识别病原体效应器的蛋白质复合物中普遍起作用还有待证实。植物R.编码二分蛋白的基因也被推测成是植物中进化储层的一部分,从而通过重复,易位和融合形成新基因[12.],[51],[52].RLM3中TN和BRX域之间的融合对于答:芥和A. Lyrata,可以与其他含Brx结构域的蛋白质二聚化,因为已经显示了单个蛋白质的Brx结构域之间的同源和异二聚化能力[53].此外,含有两个BRX结构域的转录因子BRX被证明控制油菜素内酯合成中一个重要基因的表达[54从而调节植物的根和茎的生长。
在我们的数据集中,我们观察到独特的CNL和TNL的数量有很大的变化R.基因,范围从33英寸大肠salsugineum到63 in.b·拉伯.不同种内的拷贝数差异R.提出基因家族通过通过重复事件和随后的分歧来通过基因丧失的基因丧失驱动和膨胀[12.].我们数据集中的五种物种代表两个谱系;谱系i(拟南芥和危害)及血统II (芸苔属植物和Eutrema.),在约43米亚分叉[36],[55].由于五种物种之间的密切关系,较高的保守数量R.预期基因,但没有谱系特异性R.发现了基因序列。比较基因组分析答:芥和b·拉伯已经在几种Nb-lrr基因之间建立了正面疗法[24.].但是,在我们的研究中,我们发现了11套,包括orthologsADR1-L1那ADR1-L2那RPP1.那RPP13和ZAR1..在528人中R.分析的基因仅在五种物种中仅保守了两种CNL和三个TNL。其中之一,ZAR1.,也存在于许多其他物种的双二体科中,主要在Rosid分支中[22.].罗西人从Caryophyllales分歧,分发口交超过110 mya [56的起源ZAR1.基因。最近的研究表明,ZAR1与伪激酶ZED1相互作用介导免疫P. inringae.[57].这一假激酶家族在开花植物中也很常见,我们可以推测,假激酶和ZAR1在植物的基础防御反应中发挥着普遍的作用,而在由假激酶触发的ETI反应中没有见过P. inringae.在A. Thaliana。
结论
在这里,我们揭示了一个大的变化R.基因曲目在答:芥加入,突出显示快速不断发展的性质R.基因家族,但在使用单一基因型进行基因组比较的潜在偏差。基因组测序技术的最新进展使得对作物改良相关基因型的重新测序成为可能,而且成本合理,快速生成与相关性状共分离的分子标记。因此,可以预见到芸苔属品种丰富的遗传资源提供的丰富的基因信息,以及丰富感兴趣的基因的方法。利用这些策略,马铃薯基因组中NB-LRR基因的数量从438个增加到755个[58[以自第一个开花植物排序以来,在相对较短的时间内通过下一代测序来展示新的途径和突破。
方法
数据采样
编码(CDS)和蛋白质序列答:芥col0参考基因组,18答:芥加入,A.风疹;C.风疹和大肠salsugineum(之前Thellungiella halophila)基因组从在线数据库下载[19.],[59] - [66].根据Pfam软件具有显著匹配的蛋白质[67选择TIR域(PF01582),NB-arc(NB)域(PF00931)和LRR域(LRR1-5,7-8),(PF00560,PF07723,PF07725,PF12799,PF13306,PF13504,PF13855)是.分析缺乏TIR结构域的所有蛋白质使用默认设置和置信阈值> 0.9 [68].为了答:芥选取Col-0和4个物种的参考基因组,分别编码NB和LRR结合TIR结构域(TNL)和NB和LRR结合CC结构域(CNL)的基因。在不同亚型的情况下,每个基因最长的转录本都包含在数据集中。所有的蛋白序列进行Pfam同源性和线圈服务器搜索,以识别CNL或TNL答:芥登记入册。
这耐瘦性磷酸耐药3(RLM1.) 和耐磷酸盐的耐药3(RLM3.)选择基因座进行详细分析。跨越两个基因的基因组和CDS序列上游(AT1G63710)和下游(AT1G64090)RLM1.基因座[26.[TAIR10数据库]检索[16.].利用Col-0中At1g63710至At1g64090的CDS序列,鉴定出相应的染色体区域答:lyrata那c .风疹那b·拉伯, 和大肠salsugineum通过BLAST搜索植物血统数据库[60],[69].同样,在AT4G16980-AT4G17000区域周围RLM3.基因座(AT4G16990)[34]被选中并确认答:lyrata那c .风疹那b·拉伯, 和大肠salsugineum.PFAM软件用于选择编码TIR和NB域(TN)组合的基因,在18中的后续直播答:芥登记入册被确定。对于NB-LRR基因的存在/缺失(P/A)多态性的定义[70使用了)。每址非同义及同义替换的平均比率(卡/Ks)使用与在Mega 5.2中实现的Nei-Gojobori距离方法的差异确定的每个基因进行测定[71].
多序列比对和系统发育分析
鉴定的CNL和TN1蛋白中的Nb结构域A.风疹;C.风疹和大肠salsugineum基因组与Clustalw对齐[72]使用默认设置和与翻译工具翻译成核苷酸的对齐[73].使用gblock 0.91b从数据集中删除对齐不良的站点[74[以下设置:-b1 = 282,-b2 = 283,-b4 = 5,-b5 = h,-b6 = y。相同的蛋白质减少到一个代表性。使用Paup *4.0β10构建邻居连接树[75]通过佐埃西7.0.4 [76使用GTR+G+I模型,其中0.1个不变站点和1000个引导复制。bootstrap置信度≥70的蛋白被选为同源蛋白。为了进一步分析结果树的部分内容,在MEGA 5.2中使用GTR+G+I模型和1000次bootstrap率重复进行了最大似然(ML)分析[71].利用BRX结构域序列(PF08381)的隐马尔可夫模型(HMM)在BLASTP同源性搜索中鉴定了具有BREVIS RADIX (BRX)结构域的蛋白。将BRX结构域序列与translatorX进行比对并翻译成核苷酸,在MEGA 5.2中使用GTR+G+I速率和1000个bootstrap重复构建ML树。
RLM1和RLM3基因座的分析
在中间地区之间答:芥Col-0,答:lyrata那c .风疹那b·拉伯, 和大肠salsugineum使用带有默认设置的synnorth v1.0工具进行识别[77[,通过将所选区域中的所有基因与所有物种之间的比较。蛋白质对E.-value临界值<1e-9被认为是同源的。所有非tnl蛋白RLM1.使用OrthoMCL 2.0版本服务器将不同物种的区域划分到同源组[78]后跟PFAM同源性搜索识别域架构。TNL蛋白和未在内部的未泛曲线区域RLM1.利用ClustalW对不同物种的基因座进行比对,人工检测并分类为高度相似(≥60% aa)或同源(≥80 aa)。进化P.- 在TNL基因之间的两个序列不同的氨基酸位点的比例除以转化为百分比的百分比)RLM1.十八区答:芥加入[19.与Col-0相比计算了[79].为了RLM3.轨迹,地区在AT4G16980-AT4G17000之间答:芥Col-0,A.风疹;C.风疹和大肠salsugineum使用Clustalw与默认设置对齐并手动检查。
PCR扩增RLM1A在不同物种中,通过溶解碎叶片提取DNAA. Lyrata,(i2_aut1 [80])c .风疹(CR1gr1,Samos,希腊),b·拉伯ssp。学报简历。“Granaat”,显著超越400和b·拉伯ssp。结果表明在提取缓冲液(50 mM Tris, pH 7.9;0.06 mM EDTA, pH 8;用苯酚/氯仿/异戊醇(25:24:1)、氯仿/异戊醇(24:1)纯化DNA,然后用3 M NaOAc (pH 5.2)和100%乙醇沉淀。这RLM1A含有部分侧翼基因的区域(AT1G64065和AT1G64080答:芥), PCR扩增c .风疹(CR),答:lyrata(半岛)和b·拉伯ssp。学报(BR)使用物种特定引物,CR_FW:GTTGTGGTTTGAGATCGGGTTC,CR_RV:TGTTGCACGAAAAGAGAGAAA,AL_FW:GaacctCCAGGAAATGTCT,AL_RV:CCATTGTCACTTCCGTTTC,BR_FW:CACTTCCCCCATTAACTCCT和BR_RV:TaaaAGCGGAGGGAGATC。在超过400和b·拉伯ssp。结果表明RLM1A使用RLM1A_FW3(Catcccattggtcttgatga)和RLMA_RV3:TGCTTTTCACAAGATCAGACA放大。使用Genejet PCR纯化试剂盒(Thermo Scientific)纯化PCR产物,然后进行测序(Macrogen Inc. Amsterdam,荷兰)。
提供支持数据的可用性
支持本文结果的数据包含在文章中。
附加文件
参考
- 1。
寄主-微生物的相互作用:塑造植物免疫应答的进化。细胞。2006,124:803-814。10.1016 / j.cell.2006.02.008。
- 2。
Jones JDG,Dangl JL:植物免疫系统。自然。2006,444:323-329。10.1038 / Nature05286。
- 3.
植物先天免疫中的模式识别受体。免疫学报。2008,20:10-16。10.1016 / j.coi.2007.11.003。
- 4.
Macho AP,Zipfel C:植物PRR和先天免疫信号的激活。Mol细胞。2014,54:263-272。10.1016 / j.molcel.2014.03.028。
- 5.
r4与r4相互作用假单胞菌含油III型效应分子,并且是RPM1介导的抗性所必需的拟南芥.细胞。2002,108:743-754。10.1016 / s0092 - 8674 (02) 00661 - x。
- 6.
Kim MG, da Cunha L, McFall AJ, Belkhadir Y, DebRoy S, Dangl JL, Mackey D:两个假单胞菌含油III型效应器抑制RIN4监管的基础防御拟南芥.细胞。2005,21:749-759。10.1016 / J.Cell.2005.03.025。
- 7.
贾Y,麦田斯萨,布莱恩GT,Hershey HP,Valent B:抗性基因的直接相互作用和无流动基因产品赋予稻瘟病耐药性。Embo J. 2000,19:4004-4014。10.1093 / EMBOJ / 19.15.4004。
- 8.
Kanzaki H,Yoshida K,Saitoh H,Fuijsaki K,Hirabuchi A,Alaux L,Fournier E,Tharrau D,Terauchi R:Arms Race Co-SevolutionMagnaporthe oryzae AVR-Pik和大米实物支付债券由物理相互作用驱动的基因。工厂J. 2012,72:894-907。
- 9.
Deyoung Bj,Innes Rw:植物NBS-LRR蛋白在病原体传感和主机防御中。NAT IMMUNOL。2006,7:1243-1249。10.1038 / Ni1410。
- 10。
Dodds Pn,Rthjen JP:植物免疫:迈向植物病原体相互作用的综合观点。NAT Rev Genet。2010,11:539-548。10.1038 / NRG2812。
- 11.
Maekawa T,Kufer Ta,Schulze-Lefert P:植物和动物免疫系统中的NLR功能:到目前为止,但又如此接近。NAT IMMUNOL。2011,12:818-826。10.1038 / NI.2083。
- 12.
岳建新,Meyers BC,陈建强,田东,杨胜:植物核苷酸结合位点-富亮氨酸重复序列的起源和进化历史(nbs-lrr.)基因。植物营养学报,2012,33(6):759 - 763。10.1111 / j.1469-8137.2011.04006.x。
- 13。
作者:张志刚,吴志刚,吴志刚。植物抗病分子育种的新进展。生物技术。2003,14:177-193。10.1016 / s0958 - 1669(03) 00035 - 1。
- 14.
Fuller VL, Lilley CJ, Urwin PE:线虫抗性。新植物学报,2008,30(4):514 - 514。10.1111 / j.1469-8137.2008.02508.x。
- 15.
Vleeshouwers VGAA, Raffaele S, Vossen JH, Champouret N, Oliva R, Segretin ME, Rietman H, Cano LM, Lokossou A, Kessel G, Pel MA, Kamoun S:了解和利用效应体的年龄抗晚疫病。植物病理学杂志。2011,49:507-531。10.1146 / annurev -发朵- 072910 - 095326。
- 16.
Tair10 [],[ftp://ftp.arabidopsis.org/home/tair/]
- 17.
TIR-X和TIR-NBS蛋白:编码在TIR-NBS- lrr蛋白中的两个与抗病相关的新家族拟南芥和其他植物基因组。植物学报,2002,32:77-92。10.1046 / j.1365 - 313 x.2002.01404.x。
- 18.
拟南芥nbs - lrr编码基因的全基因组分析acta botanica sinica(云南植物学报),2003,27(5):489 - 494。10.1105 / tpc.009308。
- 19.
Gan X,Stegle O,Behr J,Steffen JG,Drewe P,Hildebrand KL,Lyngsoe R,Schultheiss SJ,奥斯本ej,Sreedharan vt,Kahles A,Bohnert R,Jean G,Derwent P,Kersey P,Belfield EJ,Harberd NP,Kemen E,Toomajian C,Kover PX,Clark RM,Räqscg,Mott R:多个参考基因组和转录om拟南芥蒂利亚纳.自然。2011,477:419-423。10.1038 / Nature10414。
- 20.
陈强,韩振江,田东,杨胜:强正向选择驱动企业快速多元化R.基因在拟南芥不相关的亲戚(联系)。中国科学:地球科学,2010,30(4):457 - 461。10.1007 / s00239 - 009 - 9316 - 4。
- 21.
GUO YL,FITZ J,Schneeberger K,Ossowski S,Cao S,Weigel D:核苷酸结合位点的基因组比较富含少量的重复编码基因拟南芥.植物理性。2011,157:757-769。10.1104 / pp.111.181990。
- 22.
刘志强,刘志强,刘志强,等假单胞菌含油HopZ1a III型效应体通过拟南芥ZAR1抵抗蛋白质。公共科学图书馆。2010,6:e1000894-10.1371/journal.pgen.1000894。
- 23.
罗博列夫斯基,陈志强,陈志强,等:变异与系统发育的关系Arabidopsis Thaliana rps2.在各种各样的部落Brassiceae的同源物。mol phylocyet evol。2000,16:440-448。10.1006 / mpev.2000.0781。
- 24.
于军,Therim S .,张飞,童超,黄杰,程欣,董超,周勇,秦瑞,华伟,刘胜:两种nsbs编码基因的全基因组比较分析芸苔属植物物种和拟南芥蒂利亚纳.BMC基因组学。2014,15:3-10.1186 / 1471-2164-15-3。
- 25。
FITT BDL,Brun H,Barbetti MJ,Rimmer SR:世界各地的Phoma Phoma Step Canker(瘦性的黄瓜和l . biglobosa)以油菜籽(芸苔栗鸟).acta botanica sinica(云南植物学报),2006,34(4):427 - 434。10.1007 / s10658 - 005 - 2233 - 5。
- 26。
stal J, Kaliff M, Bohman S, Dixelius C:越界分离揭示了两个拟南芥TIR-NB-LRR抗性基因的有效性瘦性的黄瓜,Blackleg病的因果剂。工厂J. 2006,46:218-230。10.1111 / J.1365-313x.2006.02688.x。
- 27。
Hall Sa,Allen RL,Baumber Re,Baxter La,Fisher K,Bittner-Eddy PD,玫瑰乐,Holub EB,Beynon JL:植物和病原体的遗传变异的维护涉及复杂的基因基因相互作用网络。莫尔植物途径。2009,10:449-457。10.1111 / J.1364-3703.2009.00544.x。
- 28。
高志强,王志强,王志强,等:卵菌效应体和同源基因的计算预测和分子表征拟南芥耐药基因。公共科学图书馆。2012,8:e1002502-10.1371/journal.pgen.1002502。
- 29。
Bohman S,Staal J,Thomma BPHJ,Wang M,Dixelius C:表征拟南芥 - 瘦性的微米蛋白病理系统:抗性部分需要Camalexin生物合成,并且与水杨酸,乙烯和茉莉酸信号传导无关。工厂J. 2004,37:9-20。10.1046 / J.1365-313x.2003.01927.x。
- 30.
在交配系统进化和种群瓶颈之后,Capsella属的抗病位点保持了平衡选择的特征。BMC Evol Biol. 2012, 12: 152-10.1186/1471-2148-12-152。
- 31。
Warwick Si:农业的芸苔。粉的遗传学和基因组学。卷9。编辑:Schmidt R,Bancroft I. Springer Verlag,纽约;2011年:33-66。10.1007 / 978-1-4419-7118-0_2。
- 32。
Mithen RF,Lewis BG,Heaney RK,Fenwick Gr:叶片的抵抗芸苔属植物物种瘦性的黄瓜.acta photonica sinica, 2010, 39(5): 525-531。10.1016 / s0007 - 1536(87) 80036 - 0。
- 33。
研究了基因组替换对水稻抗性的影响芸苔栗鸟感染了瘦性的黄瓜.植物品种。1994年,112:265-278。10.1111 / J.1439-0523.1994.TB00684.x。
- 34。
Staal J,Kaliff M,Dewaele E,Persson M,Dixelius C:RLM3.,编码基因的TIR域编码基因涉及拟南芥的广泛免疫力,对病症进行真菌病原体。工厂J. 2008,55:188-200。10.1111 / J.1365-313x.2008.03503.x。
- 35。
Mouchel CF,Briggs GC,Hardtke CS:自然遗传变异拟南芥识别短基数,一种新的调节细胞增殖和伸长的根。中国生物医学工程学报,2004,18(1):17 - 21。10.1101 / gad.1187704。
- 36。
贝尔斯坦MA,Nagalingum NS,Clended Md,Manchester SR,Mathews S:日期分子系统表明中生起源拟南芥蒂利亚纳.Proc Natl Acad Sci U S A. 2010,107:18724-18728。10.1073 / pnas.0909766107。
- 37。
Long Q,Rabanal Fa,Meng D,Huber CD,Farlow A,Platzer A,Zhang Q,VilhjálmssonBJ,Korte A,Nizhynska V,Voronin V,Korte P,Sedman L,Mandáovát,Lysak Ma,Serenü,Hellmann i,北堡M:大规模的基因组变异和强烈的选择拟南芥蒂利亚纳来自瑞典的线条。NAT Genet。2013,45:884-891。10.1038 / ng.2678。
- 38.
Rehmany AP, Gordon A, Rose LE, Allen RL, Armstrong MR, Whisson SC, Kamoun S, Tyler BM, Birch PRJ, Beynon JL:高度分化霜霉病致病基因的差异识别RPP1.来自两个拟南芥系的抗性基因。acta botanica sinica(云南植物研究),2017,34(5):531 - 534。10.1105 / tpc.105.031807。
- 39.
作者简介:陈志刚,男,硕士研究生,主要研究方向为植物与细菌。PLoS ONE。2008年,3:e1694 - 10.1371 / journal.pone.0001694。
- 40.
3 .宫崎下,黑木,黄立新,马松岛,等:植物与细菌间富亮氨酸重复序列的水平基因转移。自然。2013,5:580-598。
- 41.
McDowell JM, Dhandaydham M, Long TA, Aarts MGM, Goff S, Holub EB, Dangl JL:基因内重组和多样化选择有助于霜霉病抗性的进化RPP8.拟南芥的基因座。植物细胞。1998,10:1861-1874。10.1105 / TPC.10.11.1861。
- 42.
Meyers BC, Shen KA, Rohani P, Gaut BS, Michelmore RW:生菜主要抗性位点的类受体基因受到分化选择的影响。植物学报。1998,11:1833-1846。10.1105 / tpc.10.11.1833。
- 43.
Ellis J,Dodds P,Pryor T:植物疾病抗性基因的结构,功能和演化。CurrOp植物BIOL。2000,3:278-284。10.1016 / s1369-5266(00)00080-7。
- 44.
NG A,Xavier RJ:富含亮氨酸的重复(LRR)蛋白:图案识别的集成商和免疫中的信号传导。自噬。2011,7:1082-1084。10.4161 / auto.7.9.16464。
- 45.
Deslandes L,Olivier J,Theulièresf,Hirsch J,Feng DX,Bittner-Eddy P,Beynon J,Marco Y:抵抗Ralstonia solanacearum.在拟南芥蒂利亚纳是由隐性的RRS1-R.基因,一种新型抗性基因家族的成员。Proc Natl Acad Sci U S A. 2002,99:2404-2409。10.1073 / pnas.032485099。
- 46.
AXTELL MJ,Staskawicz BJ:启动RPS2.- 精确的疾病抗性拟南芥与avrrpt2定向消除RIN4耦合。细胞。2003,112:369-377。10.1016 / s0092 - 8674(03) 00036 - 9。
- 47.
Mackey D, Belkhadir Y, Alonso JM, Ecker JR, Dangl JL:拟南芥RIN4是III型毒力效应器AVRRPT2的靶标,并调节RPS2介导的抗性。细胞。2003,112:379-389。10.1016 / S0092-8674(03)00040-0。
- 48.
QI D,Deyoung BJ,INNES RW:RPS5抗病蛋白的卷曲线圈和富含亮氨酸重复域的结构函数分析。植物理性。2012,158:1819-1832。10.1104 / pp.112.194035。
- 49。
张Y,Dorey S,Swiderski M,Jones JDG:表达rps4.在烟草中诱导一个独立于AVRRPS4的HR,需要EDS1,SGT和HSP90。工厂J. 2004,40:213-224。10.1111 / J.1365-313x.2004.02201.x。
- 50。
Nandety Rs,Caplan JL,Cavanaugh K,Perroud B,Wrooblewski T,Michelmore RW,Meyers BC:Tir-Nbs和Tir-X蛋白在植物基底防御反应中的作用。植物理性。2013,162:1459-1472。10.1104 / pp.113.219162。
- 51。
作者简介:陈志刚,男,博士,主要从事植物NLR功能的进化与保护研究。免疫学报。2013,4:297-10.3389/fimmu.2013.00297。
- 52。
许志强,王志强:基因组多样性与分子重组的研究进展R.-植物的基因进化。植物营养学报,2013,19(1):1-9。10.1007 / s12298 - 012 - 0138 - 2。
- 53。
Briggs GC,Mouchel CF,Hardtke CS:特定于植物的表征短基数尽管高序列保护,基因家族揭示了有限的遗传冗余。植物理性。2006,140:1306-1316。10.1104 / pp.105.075382。
- 54。
Mouchel CF,Osmont Ks,Hardtke CS:BRX在根生长中调节油菜素内酯水平和生长素信号之间的反馈。自然。2006,443:458-461。10.1038 / nature05130。
- 55。
Couvreur TLP,Franzke A,Al-Shehbaz Ia,Bakker Ft,Koch Ma,Mummenhoff K:Mustard家族中的分子系统多样化,时间多样化和进化原则(十字花科).生物科学进展。2010,27:55-71。10.1093 / molbev / msp202。
- 56。
Magallón S, Hilu KW, Quandt D:陆生植物进化时间表:在年龄和替代率的松弛时钟估计中,基因效应是次要的化石限制。中国生物医学工程学报,2013,31(6):566 -573。10.3732 / ajb.1200416。
- 57。
Lewis JD,Lee Ahy,Hassan Ja,Wan J,Hurley B,Jingree Jr,王PW,Lo T,Youn Jy,Guttman DS,Desveaux D:拟南芥ZED1假激酶在zar1介导的免疫中是必需的假单胞菌含油III型效应体HopZ1a。中国科学(d辑:地球科学),2013,32(4):591 - 598。10.1073 / pnas.1315520110。
- 58。
上衣F, Witek K, Verweij W,Śliwka J, Pritchard L -埃瑟林顿GJ, Maclean D,旋塞PJ, (RM,布赖恩•GJ Cardle L,嗯我,琼斯JD:耐药基因富集测序(RenSeq)使reannotation NB-LRR基因家族的从植物基因组测序和隔离种群的抗性位点的快速映射。植物学报,2013,76:530-544。10.1111 / tpj.12307。
- 59。
19的基因组拟南芥蒂利亚纳[],[http://mus.well.ox.ac.uk/19genomes/]
- 60.
Phytozome [],[http://www.phytozome.net/]
- 61.
如他胡TT, Pattyn P,曹J,程摩根富林明,克拉克RM, Fahlgren N,福塞特是的,Grimwood J, Gundlach H,哈贝雷尔G,霍利斯特JD,索夫斯基年代,Ottilar RP, Salamov AA, Schneeberger K, Spannagl M,王X,杨L,纳斯鲁拉,Bergelson J,卡灵顿JC, Gaut BS,污物J,梅耶尔KFX, Van de同行Y,眼镜IV, Nordborg M,魏盖尔D,郭黄:拟南芥lyrata基因组序列和快速基因组大小变化的基础。NAT Genet。2011,43:476-483。10.1038 / ng.807。
- 62.
JGI基因组门户[],[http://genome.jgi-psf.org/araly1/araly1.dowload.ftp.html.]
- 63.
Slotte T,Hazzouri Km,Ågrenja,koenig d,maumus f,gu yl,steige k,普拉斯·艾,eScobar js,纽曼lk,王W,Mandáovát,velloe,史密斯lm,henz sr,Steffen j,takuno s,Brandvain Y,Coop G,Andolfatto P,HU TT,Blanchette M,Clark RM,Quesneville H,Nordborg M,Gaut BS,Lysak Ma,Jenkins J,Grimwood J,Chapman J等:该危害风疹基因组和快速交配系统演化的基因组后果。NAT Genet。2013,45:831-837。10.1038 / ng.2669。
- 64.
王X,王H,王J,Sun R,Wu J,Liu S,Bai Y,Mun JH,Bancroft I,Cheng F,Huang S,Li X,Hua W,Wang J,Wang X,Freeling M,Pires Jc那Paterson AH, Chalhoub B, Wang B, Hayward A, Sharpe AG, Park BS, Weisshaar B, Liu B, Li B, Liu B, Tong C, Song C, Duran C, et al: The genome of the mesopolyploid crop speciesBrassica Rapa..NAT Genet。2011,43:1035-1040。10.1038 / ng.919。
- 65.
Yang R, Jarvis DE, Chen H, Beilstein MA, Grimwood J, Jenkins J, Shu S, Prochnik S, Xin M, MA C, Schmutz J, Wing RA, Mitchell-Olds T, Schumaker KS, Wang X:盐生植物的参考基因组Eutrema salsugineum.前植物SCI。2013,4:46-
- 66.
Sanseverino W,Hermoso A,D'Alessandro R,Vlasova A,Andolfo G,Frusciante L,Rowy E,Roma G,Ercolano Mr:PRGDB 2.0:朝向植物中R-基因分析的基于社区的数据库模型。核酸RES。2013,41:D1167-D1171。10.1093 / nar / gks1183。
- 67.
Punta M, Coggill PC, Eberhardt RY, Mistry J, Tate J, Boursnell C, Pang N, Forslund K, Ceric G, Clements J, Heger A, Holm L, Sonnhammer ELL, Eddy SR, Bateman A, Finn RD: Pfam蛋白家族数据库。核酸学报2012,40(4):593 - 598。10.1093 / nar / gkr1065。
- 68.
Lupas a,van dyke m,股票J:预测来自蛋白质序列的盘绕线圈。科学。1991,252:1162-1164。10.1126 / Science.252.5009.1162。
- 69.
Goodstein DM, Shu S, Howson R, Neupane R, Hayes RD, Fazo J, Mitros T, Dirks W, Hellsten U, Putnam N, Rokhsar DS: Phytozome:绿色植物基因组学的比较平台。中国生物化学学报,2011,30(4):591 - 598。10.1093 / nar / gkr944。
- 70。
谭胜,钟勇,侯华,杨胜,田东:存在/缺失基因的变异拟南芥人口。BMC EVOL BIOL。2012,12:86-10.1186 / 1471-2148-12-86。
- 71。
田ura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S: MEGA5:利用最大似然、进化距离和最大简约法的分子进化遗传学分析。中国生物科学(英文版),2011,28(6):731- 739。10.1093 / molbev / msr121。
- 72。
Clustalw [],[http://www.genome.jp/tools/clustalw/]
- 73。
Abascal F,Zardoya R,Telford MJ:Translatorx:氨基酸翻译引导的核苷酸序列的多重对准。核酸RES。2010,38:W7-W13。10.1093 / nar / gkq291。
- 74。
Castresana J:从多次对准中选择保守块用于其在系统发育分析中的使用。mol Biol Evol。2000,17:540-552。10.1093 / oxfordjournals.molbev.a026334。
- 75。
Swofford DL:Paup * v4.0b8:使用副锑*(和其他方法)。Sinauer Associates Inc,桑德兰;2002年。
- 76。
BioMatters,US [],[http://www.biomatters.com/#/]
- 77。
程飞,吴军,方磊,王雪梅:植物间共序基因分析Brassica Rapa.和别的十字花科物种。前植物SCI。2012,3:198-10.3389 / FPLS.2012.00198。
- 78。
Fischer S,Brunk BP,Chen F,Gao X,Harb OS,Iodice JB,Shanmugam D,Roos DS,Stoeckert CJ:使用Orthomcl将蛋白质分配给Orthomcl-DB组或将蛋白质群体分配到新的正脑中。Curr Protoc生物信息学。2011,35:6.12.1-6.12.19。
- 79。
王志强:分子进化与系统发育。纽约牛津大学出版社;2000.
- 80。
JørgensenMH,Ehrich D,Schmickl R,Kock Ma,Brysting Ak:中欧中的间隙和表圈基因流动拟南芥(十字花科)。中国生物医学工程学报。2011,11:346-10.1186/1471-2148-11-346。
- 81。
MUN JH,YU HJ,PARK S,PARK BS:全基因组鉴定NBS编码抗性基因Brassica Rapa..Mol Genet基因组学。2009,282:617-631。10.1007 / S00438-009-0492-0。
- 82。
植物抗性基因Wiki [],[http://prgdb.crg.eu/wiki/Main_Page]
- 83.
Uniprot [], [http://www.uniprot.org/]
- 84.
格兰特JJ,Chini A,Basu D,Loake GJ:有针对性的激活标记拟南芥NBS-LRR基因,ADR1.,对毒性病原体传达抗性。Mol植物微生物相互作用。2003,8:669-680。10.1094 / mpmi.2003.16.8.669。
- 85.
Bonardi V,Tang S,Stallmann A,Roberts M,Cherkis K,Dangl JL:扩展植物细胞内免疫受体家族的功能,超出了病原体效应的特定识别。Proc Natl Acad Sci U S A. 2011,108:16463-16468。10.1073 / PNA.1113726108。
- 86.
Borhan MH,Gunn N,Cooper A,Gulden S,TörM,Rimmer SR,Holub EB:WRR4编码促进型抗谱白色铁锈抗性的TIR-NB-LRR蛋白拟南芥蒂利亚纳四场生理比赛角膜白斑假丝酵母.植物营养与肥料学报,2008,21(4):457 - 461。10.1094 / mpmi - 21 - 6 - 0757。
- 87.
Faigón-Soverna A,Harmon FG,Storani L,Karayekov E,Staneloni RJ,Gassmann W,MásP,Casal JJ,Kay SA,Yanovsky MJ:构成阴影避免突变体意味着TIR-NBS-LRR蛋白拟南芥photomorphogenic发展。acta botanica sinica(云南植物研究),2006,28(4):429 - 434。10.1105 / tpc.105.038810。
- 88.
Lorang JM, Sweat TA, Wolpert TJ:一种“抗性”基因赋予的植物病害易感性。中国科学(d辑:地球科学),2007,31(4):591 - 598。10.1073 / pnas.0702572104。
- 89.
Botella MA, Parker JE, Frost LN, Bittner-Eddy PD, Beynon JL, Daniels MJ, Holub EB, Jones JDG:拟南芥的三个基因RPP1.复杂的抵抗基因座识别截然不同霜霉属parasiticaairulence决定簇。植物细胞。1998,10:1847-1860。10.1105 / TPC.10.11.1847。
- 90.
van der Biezen EA, Freddie CT, Kahn K, Parker JE, Jones JD:拟南芥RPP4.是RPP5.通过多信号传导组件,多硫代肾上腺系列的TIR-NB-LRR基因和柔软的霉菌抗性。工厂J. 2002,29:439-451。10.1046 / J.0960-7412.2001.01229.x。
- 91.
Parker JE, Szabò V, Staskawicz BJ, Lister C, Dean C, Daniels MJ, Jones JDG:表型特征和分子定位拟南芥蒂利亚纳轨迹RPP5.,确定疾病抵抗力霜霉属parasitica.工厂J.1993,4:821-831。10.1046 / J.1365-313x.1993.04050821.x。
- 92.
Bittner-Eddy P,Can C,Gunn N,Pinel M,TörM,Crute I,Holub EB,Beynon J:遗传和物理映射RPP13在拟南芥中的遗迹,负责特定的几个霜霉属parasitica(霜霉病)隔离。Mol植物微生物相互作用。1999,12:792-802。10.1094 / mpmi.1999.12.9.792。
- 93.
Mindrinos M,Katagiri F,Yu GL,Ausubel FM:A. Thilala抗病基因RPS2编码含有核苷酸结合位点的蛋白质和富含亮氨酸的重复。细胞。1994年,23:1089-1099。10.1016 / 0092-8674(94)90282-8。
- 94。
Gassmann W,Hinsch Me,Staskawicz Bj:拟南芥rps4.细菌耐药基因是TIR-NBS-LRR抗病基因家族的一个成员。植物学报1999,20:265-277。10.1046 / j.1365 - 313 - x.1999.t01 - 1 - 00600. x。
- 95。
Warren RF,Henk A,Mowery P,Holub E,Innes RW:富含拟拟氨酸抗性抗病基因的富含亮氨酸的重复域内的突变RPS5.部分抑制多种细菌和霜霉病抗性基因。acta botanica sinica(云南植物学报). 1998,10:1439-1452。10.1105 / tpc.10.9.1439。
- 96。
Kim SH, Kwon SI, Saha D, ananwu NC, Gassmann W:抵抗假单胞菌含油效应体HopA1受TIR-NBS-LRR蛋白RPS6调控,在突变时增强SRFR1..植物营养与肥料学报。2009,29(4):523 - 532。10.1104 / pp.109.139238。
- 97。
Stokes TL,Kunkel BN,Richards EJ:表观遗传变异拟南芥抗病性。基因开发。2002,16:171-182。10.1101 / gad.952102。
- 98。
张Z,吴y,高m,张俊,kong q,刘y,ba h,zhou j,zhhangy:破坏PAMP诱导的地图激酶级联的破坏假单胞菌含油效应器激活由Nb-LRR蛋白质序列2介导的植物免疫。细胞宿主微生物。2012,11:253-263。10.1016 / J.Chom.2012.01.015。
- 99。
Eitas TK,Nimchuk ZL,Dangl JL:拟南芥Tao1是一种促达NB-LRR蛋白,有助于抗病抗性假单胞菌含油效应AvrB。中国科学(d辑:地球科学),2008,32(5):591 - 598。10.1073 / pnas.0802157105。
- 100。
Nam M,Koh S,Kim Su,Domier Ll,Jeon Jh,Kim Hg,Lee Sh,Bent AF,Moon JS:拟南芥TTR1导致LRR依赖性致死的致命性质坏死,而不是系统性获得的抵抗力,而不是系统性的抗性。mol细胞。2011,32:421-429。10.1007 / s10059-011-0101-z。
致谢
作者要感谢Joel Sohlberg在系统发育分析方面的指导。这项工作得到了以下基金会的支持:nilson - ehle, Helge Ax:son Johnson;以及研究委员会VR和Formas,以及瑞典农业科学大学。
作者信息
从属关系
通讯作者
额外的信息
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
作者的贡献
HMP, NG和CD构思和设计了这项研究;HMP、NG、JF汇编分析数据;HMP和CD写了手稿。所有作者阅读并批准了最终的手稿。
电子辅料
12870 _2014_298_moesm3_esm.pdf
附加文件3:图S1。:Chromosomal distribution of conserved and selected NB-LRR genes in 19答:芥登记入册。在每种染色体的右侧,将29个保守的CNL和TNL基因与矫正器一起描绘答:lyrata那c .风疹那b·拉伯, 和大肠salsugineum蓝色的。红色的基因与这四种基因有同源性十字花科物种,但在一些答:芥登记入册。染色体左侧的基因归因于防御响应,但在19种附加方之间没有找到保守。R.基因信息在附加文件中编译2S2:表。(PDF 806 KB)
12870 _2014_298_moesm4_esm.pdf
附加文件4:图S2。:基于R蛋白的Nb结构域的系统发育分析答:芥那答:lyrata那c .风疹那b·拉伯, 和大肠salsugineum.使用GTR模型和1,000个Bootstrap复制构建邻居加入树。在单独的植物中以≥70的自举蛋白质在单独的蛋白质中鉴定出直晶蛋白,并突出显示并编号。标记如下:CNL蛋白(绿色),TNL蛋白(蓝色),TN蛋白(浅蓝色)和带自举<70(灰色)的枝条。每个基因的标识符在附加文件中描述1S1:表。(PDF 3 MB)
12870_2014_298_moesm7_esm.pdf.
附加文件7:图S4 .:同时在RLM1.五种物种之间的基因座。在(a)之间答:lyrata(AL.),答:芥(Col-0) (AL.) 和c .风疹(Cr)和(B)之间b·拉伯(Cr),答:芥(col-0)和大肠salsugineum(西文).七个RLM1.基因;RLM1A(At1g64070),RLM1B(B, At1g63880)RLM1C(C, At1g63870)RLM1D,(d,at1g63860),RLM1E(e,at1g63750),RLM1F(F, At1g63740)RLM1G(G, At1g63730)和其他四种TNL编码基因呈橙色(未注释为浅橙色)。非tnl基因用黑色(同同)或白色(不同同)表示。用虚线表示两个TNL蛋白具有60或更高的同源性。bp长度的减少用双前斜杠表示。(PDF 838 KB)
12870 _2014_298_moesm8_esm.docx
附加文件8:表S4 .:基因成员的存在和不存在的分布RLM1.轨迹在19答:芥登记入册。(docx 17 kb)
12870_2014_298_moesm9_esm.docx.
附加文件9:表S5 .:P-不同的距离TNL.编码基因RLM1.轨迹在19答:芥登记入册。(DOCX 22 KB)
12870 _2014_298_moesm10_esm.pdf
附加文件10:图S5。:Maximum likelihood analysis of the BRX domain. The GTR model was used and bootstrapping was with 1,000 replicates. Labeling is as follows: dicots (green), monocots (dark blue), green algae (orange), moss (pink) and lycophyta (light blue). The clades consisting of BRX domains of RLM3 is highlighted. (PDF 11 MB)
作者为图像提交的原始文件
权利和权限
本文在BioMed Central Ltd.的许可下发布了这是一个开放的访问文章,根据Creative Commons归因许可(http://creativecommons.org/licenses/by/4.0.)如果原始工作正确记入,则允许在任何媒体中进行无限制使用,分发和再现。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
Peele,H.M.,Guan,N.,Fogelqvist,J.et al。在五种植物中抗性基因的丢失和保留十字花科家庭。BMC植物杂志14,298(2014)。https://doi.org/10.1186/s12870-014-0298-z
已收到:
公认:
发表:
关键字
- 拟南芥蒂利亚纳
- 十字花科
- CC / TIR-NB-LRR域
- 基因组
- 瘦性的黄瓜
- 抗性基因