抽象的
背景
小种子Desi-型豆类作物核基因组序列的可用性中投arietinum为研究鹰嘴豆独特的基因组特征和评价其生物学意义提供了机会。越来越多的豆科植物基因组序列也为开发可靠的、信息驱动的生物信息学应用程序提出了挑战,这些应用程序适用于比较探索这类重要的作物植物。
结果
鹰嘴豆基因组网络资源(CGWR)是一套基于网络的应用程序,专门用于鹰嘴豆基因组可视化和比较分析,基于下一代测序和组装中投arietinumDesi-型基因型ICC4958。鉴于各种豆类基因组项目中的突变映射和克隆中的各种豆科基因组项目的重要性和潜在角色,设计和配置CGWR,用于映射,扫描和浏览显着的鹰嘴豆基因组特征。它还使ICC4958 DNA序列分析的比较信息学与其他野生和培养的鹰嘴豆,各种其他豆类物种以及几种非豆科模型植物的栽培类型分析,以使研究能够调查形状豆类基因组的进化过程。
结论
CGWR是一个在线数据库,提供鹰嘴豆基因组的全面视觉和功能基因组分析,以及定制的地图和基因聚类选项。它也是基于唯一的基于植物的网站资源,支持和分析了基因组中的核小体定位模式。CGWR的有用性已经通过使用此服务器的生物学意义发现。CGWR与所有可用的操作系统和浏览器兼容,可在开源许可证下免费提供http://www.nipgr.res.in/cgwr/home.php..
背景
经济上重要脉冲作物的基因组序列草案中投arietinuml .(鹰嘴豆;Desi基因型)最近通过全基因组深度测序完成[1].鉴于豆科植物在世界范围内的重要性、遗传种群的耐旱特性,以及为了促进鹰嘴豆改良品种的遗传改良和育种,我们小组对小种子鹰嘴豆ICC4958基因型开展了这项倡议。ICC4958和大种子卡布利型鹰嘴豆基因组序列的可用性[2]已经丰富了现有的可访问豆类序列数据,其中包括其他三种豆类粮食作物大豆(大豆)[3.], 木豆 (Cajanus毛竹)[4]和常见的豆子(菜豆)[5],以及两种非食用豆科植物,即桶医(Medicago truncatula)[6]和鸟脚三叶子(莲花japonicus.)[7].这种丰富的数据能够实现各种比较分析,并为豆科研究室提供了开发新型生物解释工具的机会,为启动豆科基因组学的新研究铺平了途径。
尽管最近鹰嘴豆基因组研究激增,尽管可以获得两个不同鹰嘴豆基因组的草图序列,但公共领域可用的软件对这些基因组的比较探索存在局限性,导致缺乏一个全面的基因组分析界面加工物种,或关于鹰嘴豆的多基因组数据处理。为了克服这种限制,我们开发了一个具有富有功能丰富功能的交互式网络服务器,用于详细分析和可视化鸡肝核基因组,还支持其他鹰嘴豆基因型(ICCV2,JG62和PI489777,Kabuli-的详细比较基因组学型鹰嘴豆型),豆类(Pigeonpea,大豆,常见豆,莲花和Medicago)及非豆类(拟南芥和葡萄)。此Web服务器已被命名为“Chickpea Genomic Web资源”或CGWR,并且在没有任何登录要求的情况下可以自由获取http://nipgr.res.in/CGWR/home.php.CGWR包括基于泛型基因组浏览器(Gbrowse)的浏览器[8以及用于查询、分析和下载可用数据的多种工具或界面。GBrowse是一个在Perl中实现的web服务器应用程序,非常适合作为一个独立的基因组浏览器,目前正在全球100多个生物基因组中使用[8].
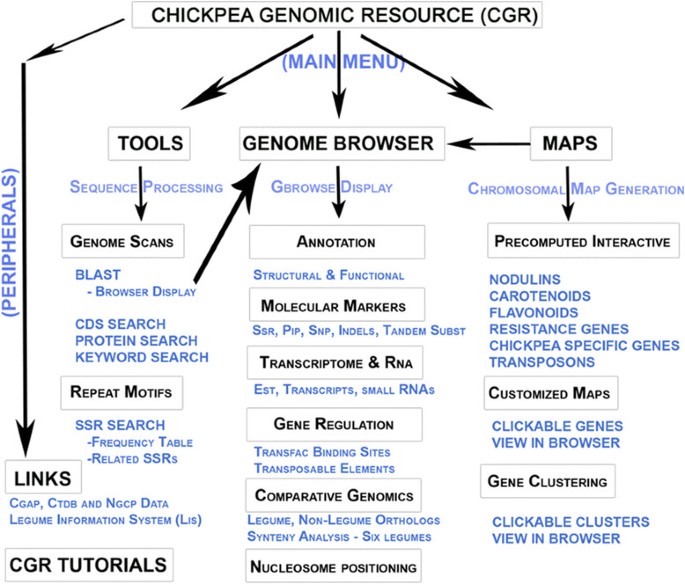
本报告提供了关于cggwr图形用户界面(GUI)基本元素的入门知识。对于每个菜单,我们提供了一个可以使用CGWR执行的例行任务的管道,并提供了具体的示例,提供了可以通过该资源解决的生物学兴趣问题的见解,最后我们讨论了我们的下一个CGWR发布计划。数字1提供CGWR及其组件的总体摘要。我们认为CGWR将鼓励研究人员进行基于豆类的信息学分析,因为它旨在易于使用和交互式图形显示多种基因组信息。
实现
CGWR分为三个主要部分。第一个是“工具”部分,可以快速扫描SSR重复,提取所需的CDS或基因,以及成对比对,这有助于物种之间的同源区域鉴定。这个菜单支持基于文本和序列的搜索,提供快速和精确的访问鹰嘴豆的任何想要的基因或蛋白质。第二部分是“地图”,出于需要有趣的单一视图快照的鹰嘴豆基因组地图——所需的基因的染色体定位(s)模型或基因家族的这个工具可以显示一个可点击基于基因交互式图像,可以下载。CGWR中的“浏览器”是一个用于基因组探索和解释的快速加载在线工具,可以在任何规模上可靠地显示鹰嘴豆基因组中给定的感兴趣区域,并可以在一个窗口中通过几十个注释“轨迹”来浏览、过滤和分析数据。除了这三个主要的部分,CGWR还为每个部分提供了详细的教程,并提供了到外部豆科研究工具和网站的重要链接,以及下载所有可用数据集的设施,从而成为一个豆科知识库。
数据源
如Jain等人所述,在NGCP项目下产生了关于鹰嘴豆核基因组序列,组装,注释,基因模型和参考序列本身的所有数据,如Jain等人所述。[1].用于比较分析的基因组数据取自Phytozome v9.0 (http://www.phytozome.net/).从NCBI和CTDB数据库获得转录组数据[9]、[10.].基因本体论从拟南芥去数据库[11.].另外六种豆科植物的核基因组从它们各自的数据库下载。中投arietinum卡布里型基因型http://www.icrisat.org/gt -bt/icgc/genomemanUscript.htm.),Cajanus毛竹(http://gigadb.org/dataset/100028),甘氨酸最大(ftp://ftp.jgi-psf.org/pub/compgen/phytozome/v9.0/gmax/assembly/),莲花japonicus.(ftp://ftp.kazusa.or.jp/pub/lotus/lotus_r2.5/pseudomolecule),Medicago truncatula(ftp://ftp.jgi-psf.org/pub/compgen/phytozome/v9.0/Mtruncatula/assembly/), 和菜豆(ftp://ftp.jgi-psf.org/pub/compgen/phytozome/v9.0/Pvulgaris/装配/)。
比较基因组学和变异
对于工具界面,创建命令行爆炸数据库C.Arietinum.ICC4958草案基因组序列,其多肽和CDS序列。为了进行同源性和种间多态性的鉴定,我们建立了上述8种植物和4个鹰嘴豆品种的BLAST数据库。BLAST版本2.2.27+ [12.用于这个目的。对于每次新的运行,输出都会使用PHP脚本转换为HTML和表格格式。后端perl脚本用于BLAST输出与基因组浏览器的集成,直接实现感兴趣序列的基因组区域的可视化。SSR搜索工具可以通过后端perl脚本,概述鹰嘴豆基因组上任何想要的SSR基序的迭代次数。为了识别同序区域,BLASTn分值(<10−10)应用于鹰嘴豆基因组和上述六种豆科植物基因组。用于计算同期区域链,Dagchainer软件[13.使用其中通过用C ++和Perl编写的Inhouse处理脚本编写了哪些输入文件。已删除包含九列(Chra,Inceptiona,Starta,Enda,ChrB,AcessBB,StartB,ENDB和E-Value)的输入文件中的重复匹配以减少数据噪声,并完成50 KB窗口长度的过滤。DAG(定向非环状图)和动态编程用于比较上述每对基因组序列。
映射和聚类
使用通过PHP编程生成的基因位置表生成基因映射和聚类数据。通过该工具生成的地图是交互式的,并且使用基于特定的基因ID的基因组位置,并按照八个LG的发生顺序排列它们。用八个垂直条创建图像,每个杠杆表示一个假组合(或连接组),并且基因模型在这些杆上标记为水平灰度线。地图中的每个水平标记都使用shell和perl脚本可以点击,以便用户可以推断各个或组映射基因ID的进一步细节。对于群集,每当发现两个或多个输入ID在相对于彼此的预计距离截止(0.3 MBP)内时,程序将它们分配给群集并返回用户的Web链接进一步分析此集群。映射到八个组合的假致散发器中的任何一个的每个基因模型或簇可以通过在CGWR后端的工具集成的链路上直接在Chickpea Genome浏览器上直接可视化,如上面的部分所述。In case, one or more input gene IDs do not map to any of the eight pseudomolecules or LGs, they are assigned to an ‘unassembled scaffold’ or a virtual LG termed as ‘UN’ which can be seen as the last (ninth) vertical bar on the map image. The program does not carry out clustering analysis of gene models mapped to this virtual pseudomolecule since the gene models are unassembled and their spatial locations are unknown.
监管元素识别
使用Perl脚本进行GFF文件过滤,数据归一化去除重叠的基因片段,并为每个注释的基因模型提取300 bp上游序列。从8个假分子和18,826个支架中共获得20,057条序列中投arietinum核基因组和这些被提交给转录因子结合位点(TFB)或独联体-element预测管道。关于监管区域的数据或独联体通过计算预测方法获得了注释鹰嘴豆基因模型上游调控区域的-元素。为此,使用PLACE确定了潜在的TFBSs [14.]及jasper [15.]数据库,两个程序使用不同的方法,即基于文学和位置的基于特定评分矩阵(PSSM)的方法。Jaspar包含用于多细胞真核生物的注释,基于矩阵的TFBS谱,来自芯片SEQ和芯片芯片的全基因组结合实验。简而言之,PSSM的元素对应于反映观察候选TFB的特定位置的可能性。用于Jaspar Core Plantae的参数包括八种植物种类,包括21种不同的转录因子,每个转录因子由非冗余轮廓表示,初始相对概况得分阈值为85%。使用75%阈值的刻度值7以7匹配预测的TFBS数据文件中获得的近似最低分数匹配所得到的数据。以这种方式获得的所有102,597次命中始终用GFF3,PHP和Perl纳入CGWR浏览器。基本上是基于文献的数据库,包含在植物中发现的愈合和非冗余核苷酸序列基序独联体- 从先前公布的报告,文章和评论中提取的调节DNA元素,以及关于各种植物基因的调节区域的评论[14.].然后使用机械化的Perl模块来通过进入20,057个鸡族上游调节区序列中的每一个进入通过信号分组的'放置网信号扫描'来获得位置预测。
核小体定位地图
使用基于fortran的R软件包NuPop对鹰嘴豆基因组的核小体起始位点和占据率进行了预测。该方法采用带有个体函数的持续时间隐马尔可夫模型计算核小体位置的维特比预测;给定DNA片段的占据状态和结合亲和力评分[16.]、[17.].拟南芥被发现是鹰嘴豆大多数相似的基础组合物的物种,因此使用的鸡眼的核小体预测拟南芥预培训的接头DNA长度分布模型。所使用的参数中,核体和接头DNA状态的第4阶Markov链模型。发现该模型在预测中稍微有效,尽管它需要额外的计算时间(未显示数据)。这些预测的输出被转换为标签分隔文件,此后要绘制可视化。对于以可能的核细胞核态认为的基因组学区,标准如下分隔:最小核小体起始现场评分(> = 0.45),然后是至少146个碱基对,具有高分性的核小体占用(平均> =0.8)。不满足该标准的地区被视为接头DNA状态。以这种方式,为了方便可视化,将原始的NUPOP分数转换为地块以使用内部壳牌脚本。轨道被呈现为曲线,其中具有接头DNA状态的区域出现在负y轴上,而具有高核小体态可能的区域出现在正y轴上。含有连续系列N的组件的区域显示为零分数,以避免与预测区域混淆。
存储、提取和GUI
所有分析都按照上述方法进行,并将结果转换为GFF格式进行存储、显示和提取。后端MySQL(版本5.5.29)用于存储支持序列搜索和基于基因映射的所有类别的数据。GBrowse用于构建和开发基因组浏览器,通过通用生物模型数据库(GMOD)项目,一个用于创建和管理基因组规模的生物数据的开源软件工具集合[18.].对于鹰嘴豆,gbrowser -2.27与Apache、标准perl库、libgd2和MySQL一起在Red Hat Linux平台上使用,并配置为既显示基因的剪切结构等定性数据,也显示微阵列表达水平等定量数据。为了提高资源的响应性,Apache配置文件被修改为用FastCGI协议代替常规的CGI实现,并安装了Perl FCGI模块。为了提高效率,特性和序列存储在关系数据库中,其模块和依赖关系是GBrowse中数据访问的基础。数据创建管道使用两种格式的输入数据进行浏览器操作,即GFF和FASTA,这两种格式都可以通过bioperl模块相互转换。后端数据加载管道使用MySQL和一个以tab分隔的文件,其中包含各种基因组特征的GFF格式,以及bioperl工具,用于加载Bio::DB::GFF数据库。通过FCGI、Javascript、PHP和HTML脚本实现了CGWR的整体配置和定制。前端页面是使用HTML脚本生成的。编写了不同的内部PHP和perl程序来创建输出。使用CPAN模块GD、ChromosomeMap-0.10和ImageMagick-6.8.5-6生成映射图像(http://www.cpan.org/).为了使cggwr的映射模块更加健壮,添加了shell脚本,允许多个用户同时访问和可视化映射结果。
结果
这项工作主要集中在核基因组组合草稿中的比较基因组分析中投arietinumGenotype ICC4958最近由我们的团体发布[1].在顶层,当前程序集被组织为Ca_LG_1到Ca_LG_8;表示与8个鹰嘴豆连锁基团匹配的WGS contigs,而Ca_LG_0表示无法与8个伪分子中的任何一个匹配的scaffold。鹰嘴豆基因组资源可以通过网页访问http://nipgr.res.in/CGWR/home.php.数字1提供CGWR及其组件的流程图摘要。在以下部分中,我们描述了单独的菜单项,然后在浏览器中的可用工具和基因组曲目的简要说明(用文本标签和斯特兰标记的彩色,共线块表示)以及它们的突出特征。
CGWR工具
CGWR的工具菜单包括简单的用户友好的GUI,可以快速扫描和提取基因组的所需区域,以及与用户指定序列的成对比对,以识别副病剂和矫正器。用户可以从此下拉菜单中使用各种选项,包括SSR搜索,Blast,CD,蛋白质和关键字搜索,如图所示2.
SSR搜索
此页面使微型卫星分析能够进行鹰嘴豆基因组中检测到的64418简单序列重复(SSR)在-硅鉴别。它包含一个表单,用户可以提供兴趣的SSR的主题,例如ACA。该工具在拼接输入字符串中找到鸡群基因组中的所有SSR,并拆分数据以易于解释,导致具有在SSR的各个迭代处发生频率的表。例如,ACA重复在鹰嘴豆核基因组中总共231个位置发生,其中ACA(即Acaacaacaaca)的五个迭代发生在112个职位,而十个迭代(ACA10.)仅发生在三个位置等。在结果页面的底部,此工具还返回所有“相关”SSR序列的评估,这是比输入SSR序列长的一个或两个核苷酸。对于ACA示例,“相关的”SSR“将包括ACAX,XACA和XACAX的所有实例,用于扫描和报告的数据以及重复数量和频率。(这里x是指四个核苷酸A / C / T / g中的任何一种。
爆破
基本局部比对搜索工具是检测序列相似性的常用比对程序。用户可以根据查询的性质选择特定的BLAST程序和数据库。查询的性质可以是DNA、蛋白质、翻译RNA或翻译DNA。该工具的数据库选项包括完整的CDS和ICC4958的蛋白质以及核基因组序列。输入序列可以输入文本,FASTA格式,或多个序列可以上传为文件,如果需要。该工具以HTML格式返回对齐结果,可以下载输出摘要。除了基于文本的摘要,这个页面还通过一个“浏览器”链接直接将工具菜单连接到cgwar中的鹰嘴豆浏览器,如图所示2,以便对感兴趣的基因组区域进行详细的研究。在每次BLAST搜索时,浏览器中会添加一个标有“BLAST”的额外“对齐轨迹”,用户可以看到与查询对齐的基因组的确切区域,无需任何人工干预。
CDS搜索
除了BLAST, CGWR还可以直接编码DNA序列(CDS)搜索已知基因模型,也可以通过BLAST搜索确定其ID,如上所述。基因的编码区是由外显子组成的部分,负责编码蛋白质。对于一个有机体来说,它代表由基因编码区组成的基因组的总和。鹰嘴豆的所有cds的计算预测[1]可以被此工具搜索,用户可以粘贴一个或多个感兴趣的id并获得相应的cds。结果直接连接到鹰嘴豆浏览器,以使详细的调查感兴趣的基因组区域,如上所述。
蛋白质搜索
类似于CDS搜索,计算鹰嘴豆基因组的翻译区域的计算上预测的补充(根据ref [1])可以通过ID号扫描。此菜单支持基于文本的搜索,提供快速和精确地访问任何所需的鸡豆蛋白。结果可以下载并直接在基因组浏览器中可视化,其中示例在表单中提供。
关键字搜索
如果用户没有任何先前的信息,例如感兴趣的序列或CID,则关键字工具允许搜索包含在其注释中的给定输入文本的所有潜在的鸡拨ID。例如,该工具返回六个潜在的匹配术语“reductoisomerase”,并且可以与每个匹配的ID的信息一起下载这六个列表,包括基因描述,轨迹,PFAM ID和GO SLIM术语。此外,CGWR算法自动生成搜索查询的交互式映射,使得用户可以直接可视化从他们搜索获得的ID列表的出现的空间模式。
CGWR地图
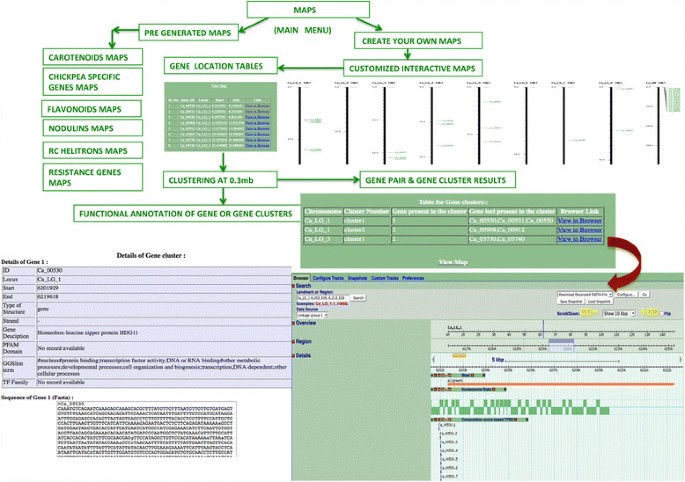
“地图”菜单提供基因家族的交互式染色体地图,即所需基因模型在各自伪分子上的位置。该工具可以生成基因组、基于序列的地图,并显示碱基对坐标的伪分子。它也可以用来点击任何想要的基因或地图上的群集,以评估和可视化鹰嘴豆基因组上的映射基因模型的群集。用户可以获得鹰嘴豆基因组有趣的单视图快照,其中所要求的基因的空间位置(s)可以通过八个伪分子同时显示。这个菜单提供了两种程序,一种用于显示已选鹰嘴豆基因家族的现有图谱,另一种用于用户定制构建所需基因模型集的图谱。
鹰嘴豆基因家庭地图
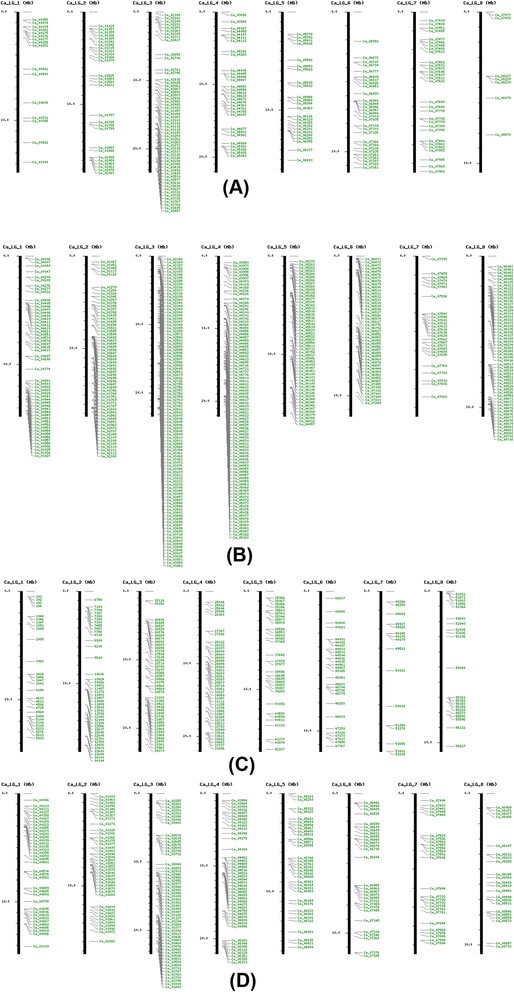
在鉴定的640种独特的基因模型中,与鸡眼的黄酮类化合物的代谢相关,可以在图中描绘了可以映射到八个不同的假组件的那些3.a,并且发现群体簇中的最高数量的黄酮类基因模型3.对于预测与类胡萝卜素代谢相关的基因模型,未观察到群体的趋势未观察到(在地图菜单下的CGWR网站上提供的数据)。通过单击地图图像上的相应栏可以详细分析每个基因或簇。例如,LG-3上的前三种类黄酮基因落入一个可以点击的簇中以查看群体每个成员的完整细节,包括基因名称,功能注释,基因本体,TF结合位点和完整序列。可以通过单击将每个基因或群集连接到CGWR基因组浏览器的链接来指出更多信息。我们对整个植物王国的分析显示9990种豆类特异性基因模型和2751个鹰嘴豆特异性基因模型在鹰嘴豆基因组和图中的图B中3.显示了鹰嘴豆特定基因的映射子集。此外,通过鹰嘴豆单基因集筛选确定的假定抗性相关基因模型(r基因)也被定位,这些基因似乎存在于鹰嘴豆基因组中,尽管聚类可能发生在特定的保守类中,这些r基因在分析中被分配(图)3.d)。鉴定了几乎三分之一的鹰嘴豆基因组重复是各种转换元素,其中大部分代表逆转录和约5%构成的DNA转座子。后期群体,图3.C显示映射的RC Helitrons,即被认为是通过滚动圈机制复制的转座子,并且可以看出它们在鹰嘴豆基因组上遍历并聚集在几个区域中。值得注意的是,几种类型的线和其他基因家族也似乎在鹰嘴豆基因组上聚集,并且可能有趣的是,了解群体是否发生在其他豆类基因组中。如下部分中所述,可以使用浏览器和工具菜单的组合在CGWR内查询这种可能性。
自定义地图
数字4显示了基于流程图的cgwar地图工具的轮廓。“创建你自己的地图”选项允许用户粘贴他们想要的一组基因模型的id,以可视化类似于图中描述的空间位置地图3..在提交表单上,用户可以在输入框中输入基因ID,然后按键盘上的回车键。表单本身提供了一组基因id示例。用户还可以通过关键词搜索确定感兴趣基因的cid。如图所示4,地图工具返回一个表列出了作为输入提供的指定基因ID的基因座,启动和结束位置。此表的顶部是查看导致映射图像的地图的链接。如果感兴趣的基因位于一个未组装的脚手架上,而不是八个伪模型中的一个,则程序将其分配给独立的未组装单元或虚拟LG称为“UN”。在CGWR预先生成的地图中提供这种情况的示例。这些自定义生成的映射是交互式的,允许用户单击地图上的任何感兴趣区域,以可视化与上一节中所述的各个区域的详细信息。此外,用户可以从本算法映射的任何输入基因模型直接查找链接到Chickpea浏览器,因为单击这些链接将用户重定向到鹰嘴豆基因组的相应区域,如图所示4.这些地图也可下载为高分辨率图像,供出版之用。因此,CGWR通过连接后台的工具、地图和浏览器,实现了各种功能之间的直接连接。
基因聚类
如图所示4当发现两个或更多个映射的基因模型在相互参考的预计距离切断内发生时,它们被认为是物理基因组簇的一部分。在这种情况下,地图的输出将提供另一个“群集”链接。在此功能的帮助下,用户可以直接可视化在基因模型的输入组中识别的每个集群的群集数和组合。地图是交互式的,并且可以手动单击地图上的每个群集,以便在鹰嘴豆基因组浏览器上识别到其成员的洞察中,而用户也可以查看包含此类基因簇的整个基因组区域,以进一步分析如图所示4,如存在共同的上游调控元件,或识别附近的基因模型及其功能。
鹰嘴豆基因组浏览器
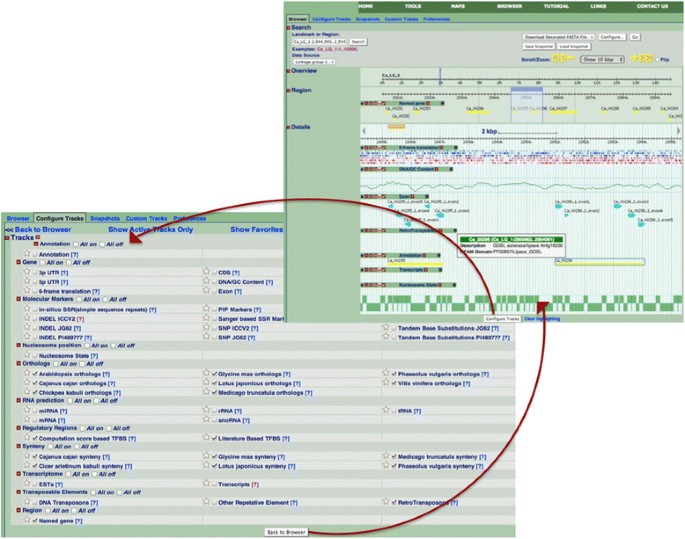
基因组浏览器是CGWR的主要功能之一。目前,2013年5月装配可用;在不久的将来,当它被释放的时候,下一次的冻结将会被实现。数字5shows the default browser display i.e. the first 10 kbp data on the first Chickpea pseudomolecule LG1, although users can select any of the eight LGs from the pull-down list in the Data Source, and positional information can also be typed into the landmark or position box on the top left corner, e.g., Ca_LG_1 for the whole of chromosome 1 and Ca_LG_2:1..10,000 for the region from position 1 to 10,000 on chromosome 2. The region expanded in the browser will be highlighted in pale blue in the Overview section as shown in Figure5.对于未组装的脚手架,用户可以从搜索部分中的下拉数据源列表中选择CA_LG_0,并键入所需脚手架的名称和位置。缩放和滚动控制有助于缩小或拓宽显示的染色体范围,以专注于所感兴趣的确切区域。默认浏览器显示可以根据“配置曲目”按钮在浏览器底部提供的轨道控件,其中大约五十个不同的轨道可供选择,如图所示5.
第一个LG区域中鹰嘴豆基因组浏览器的典型显示。在上面板的左上方可以看到四个主要区域,即搜索、概述、区域和详细信息。最上面的“搜索”部分确定了在浏览器中显示的确切基因组范围(参见左上角的“地标”文本框)。在接下来的两个部分,即“概述”和“区域”中以天蓝色高亮显示的区域,将在余下的浏览器视图中展开。因此,当前示例(' Details '部分)表示CA lg1的3mbp区域内的10kbp延伸。3mbp区域约有11个基因模型(见“区域”部分的黄色条带),其中只有两个位于扩展的Details部分(见注释轨道中的黄色条带)。通过单击扩展部分中的注释带,可以看到基因模型的注释,弹出框的形式如下所示。在这张图中,七个基因组轨迹被开启,包括反转录转座子、核小体状态和转录组。如左下角面板所示,用户可以从当前CGWR构建的48个选项中选择其他曲目。
为了避免由于这种大量曲目而过载,可以以这样的方式协调Gbrowse控件,可以关闭一些浏览器轨道的方式,并且其他可以折叠成浓缩的单行显示器。因此,可以根据用户偏好使用基于轨道的切换,用于开/关和隐藏/显示模式,除了下载,共享,密度和喜爱的模式之外,可以根据用户偏好隐藏或过滤。每个轨道上还有一个配置模式,允许用户在该轨道上编辑显示特征。悬停在对应于每个轨道显示器的彩色条上释放描述各个轨道的信息气泡及其数据源,无论适用。单击轨道内的各个彩色条或功能打开详细信息页面,其中包含轨道的各个属性的摘要,其中具有附加特征特定信息,例如对对齐或与外部信息的链接,具体取决于轨道的性质。在以下部分中,我们提供了一个轨道和典型交叉轨道分析的示例列表,即CGWR浏览器可以用于。
基因结构预测
目前浏览器有7个独立的基因和基因预测轨迹,描述了基因结构的各个方面,包括选择5 '和3 ' utr、编码区(CDS)、基因组DNA的外显子和内含子的轨迹。预测蛋白序列的mRNA序列,以及基因组DNA的GC含量和六帧翻译也可用。
功能注释
对于蛋白质或RNA编码基因,功能注释在主浏览器窗口的“区域”和“详细信息”部分提供。区域部分中最上面的“命名基因”轨道允许在所有后续(下)轨道中扩展的用户选择的高亮区域之外的基因模型的可视化。对于用户选择的高亮区域内的基因注释的可视化,可以使用“注释”轨迹。这些基因模型在黄色的条中,鼠标悬停将打开一个带有功能注释和PFAM域信息的气泡,在任何可用的地方。点击每个基因会返回一个页面,里面有详细的基因位点信息、基因描述、蛋白质家族分类和基因本体信息,以及各自基因的FASTA格式的核苷酸序列。
分子标记
在鹰嘴豆基因组水平上,CGWR共有12个单独的分子标记评估路径。这包括两种简单的序列重复,即:在-硅SSRS和基于SSR,PIP标记的SSR,SSR,以及串联基替代和参考其他三种鹰嘴豆品种。在这些轨道中描绘了总共1,644,016个标记。在基因组上识别的所有SSR可以通过SSR轨道可视化,可实现各种数据分析。在SSR上悬停将指定重复的数字和类型,以及基因组中存在的特定类型的SSR数量。例如,给定的SSR可以是第五次的四聚体SSR或千分之二聚体SSR等。单击SSR将返回一个页面详细说明SSR的轨迹信息,类型,长度,数量和迭代以及精确的SSR主题。该轨道还具有从特征的侧翼区域获得DNA的设施,包括100个上下底座,以实现底漆设计努力。另外,CGWR浏览器支持进一步的交互式SSR分析,其中用户可以找到任何所需SSR的数量和类型。此页面包含一个表单,其中SSR的SSR的长度和图案可以键入,并且返回一个表提供有关各种类型是否存在的SSR的信息,如果是,则会呈现出相关染色体中的SSR数量描绘了。The SSR search options in the pull-down ‘Tools’ menu on the home bar at the top of the browser further enables a scan of all kinds of ‘related’ SSRs that differ by a length of one or two form the input SSR, as described earlier. Nucleotide diversity has been measured at the genomic scale by comparing ICC4958 with three other cultivated and wild chickpea genotypes, namely, desi-type JG62/ICC4951, kabuli-type ICCV2/IC12968, and wild-type P1489777. Variations have been analyzed between these four varieties revealing 32,919 InDels, and 1,504,646 SNPs and 41,824 tandem base substitutions all of which can be browsed in the CGWR through nine individual tracks representing each of these three categories compared pairwise between IC4958 and one of the above-mentioned genotypes. Each track, when clicked, provides details of gene structural variation via alignments between IC4958 and the respective variety being compared, along with additional flanking alignments from upstream and downstream regions, in order to assist in marker based studies and for acquiring the DNA for additional features and reverse complementation. The potential intron length polymorphism marker track (PIP markers) shows the markers that have been predicted using the PIP database.
比较基因组学
如前所述,CGWR中最重要和最大量的数据是鹰嘴豆与其他豆科和非豆科植物基因组的比较评估。在所有的CGWR包含24个比较基因组学的个体轨迹,其中9个轨迹代表了4个鹰嘴豆基因型之间的核苷酸变异,已在上述的分子标记部分进行了描述。此外,还有9个保护轨迹用于描述ICC4958和其他6种豆类作物(包括卡布里基因型鹰嘴豆“CDC Frontier”)之间的同源基因模型,甘氨酸最大,Medicago truncatula,菜豆,Cajanus毛竹,莲花japonicus.,除了拟南芥和vitis Vinifera(non-legumes)。这些同源轨迹显示了进化保护的措施,并突出了基因组中可能在这对配对之间具有重要功能的区域。点击轨迹可以看到同源基因和鹰嘴豆基因的位点位置、基因id和FASTA格式序列的详细信息。在这个页面的底部是一个链接到对齐之间的直系亲属。爆炸(12.]工具菜单前面描述的搜索引擎也可用于满足比较和对齐的特定需求。除了这18个轨道之外,CGWR还具有六条基因型ICC4958的Synteny评估曲目,其中六种豆类基因组中的每一个。我们建议一个大的窗口尺寸,以实现给定区域的同步方向的可视化,以及探索给定区域中的两个基因组之间的多个同步匹配。单击同步区域弹出一个气泡,为每个匹配的轨迹提供启动和结束站点。在整个24个对比基因组轨道上的每个植物物种已经保持着彩色代码。例如,蓝色代表菜豆而红色代表Cajanus毛竹,等等。
转录组
浏览器包含了详细的转录组分析的轨迹,有超过27000个ESTs和2.74亿个过滤reads,代表来自独立组织/器官样本的鹰嘴豆转录本。对每一个转录本,跟踪提供基因位点信息、转录本描述、来自分子功能、生物过程和细胞定位的基因本体论数据。除了能让用户看到所研究的六个组织样本中的每个组织的表达。
监管区域
转录因子(TFs)与基因组上短而简并的寡核苷酸特异性结合是转录调控和基因表达的关键。CGWR浏览器包含了预测TF绑定位点(TFBS)的跟踪,根据PSSM基于计算分数(JASPAR跟踪)[15.[以及基于文献的数据对应(地方曲目)[14.].对于每个轨道,浏览器提供有关股线,基因座,TFBS序列基序,家庭的分类以及植物物种的信息,具有类似的结合位点的证据。基于家族的分类允许一个解码转录因子可能绑定到感兴趣区域,CGWR进一步提供标识符和每个家庭的细节。例如,已知网站'caactc'与转录因子careosrep1结合,并且来自在水稻中半胱氨酸蛋白酶(Rep-1)基因的启动子区中发现的关心(caactc调节元件)。监管区域分析通常可以导致同一站点的多种TFBS预测,因此在CGWR中为此目的的两个独立轨道提供了来自多种来源的交叉引用和证据的额外优势。我们建议在5到10 KB范围内的低窗口大小,以便以单独的方式可视化多个预测。
可转换元素
超过40%的组装草稿基因组代表了分散重复,包括各种转座元件,这些可以单独显示。这些轨迹可用于DNA转座子、反转座子(ltr)和其他重复元件。跟踪DNA转座子可以用来可视化超过80种不同的DNA转座子以及RC螺旋子。反转录转座子的轨迹可以可视化和分析不同家族的LINEs, sine和ltr。不能分为这两个轨道的元素被分配到转座轨道中的第三个轨道,即其他重复轨道,由未知数、简单重复、卫星等组成。每个转座子都有一个基于其基因组位置的唯一ID。在多个副本中出现的具有相同分数的转座元素id被分配了子id,如N.1, N.2, N.3等等,这取决于出现的次数。点击一个元素将返回一个页面,详细说明该特定转座子或逆转录转座子的基因座信息、类型和基于家族的分类,以及它的整个序列。
非编码RNA预测
鹰嘴豆基因组的非编码rna基因模型已被预测,共鉴定出约121个不同的Rfam家族,包括miRNA、snoRNA、rRNA、tRNA等。这些已经被绘制到基因组上,可以通过五个单独的轨迹来可视化。点击这些轨道返回一个包含各自RNA位点特征的页面,如反密码子和氨基酸规范(tRNA),独特的家族分类(mirna和snorna),链信息;完整序列以及二维结构表示法。
核小体定位
使用的鸡眼的核细胞核态和接头DNA状态的预测已经使用拟南芥作为索引物种[16.]、[17.[如方法中所述,结果已被标准化并映射到鹰嘴豆基因组。该轨道提供了一种典可,其中包含有关核小体和接头DNA状态的信息。它叠加占用和结合亲和力评分,用于最佳核小体定位的维特比预测和基因组位置的后概率是核心的开始。为了便于解释,在正y轴上描绘了具有较高核心态趋势的基因组的区域,而具有较高趋势的接头DNA状态的区域显示在负y轴上。核小体占用似然和基因结构之间的初步计算对应揭示了基因组的编码区域比调节区域显着富集核细胞核,而启动子显着耗尽核心。我们还发现内含子与任何其他基因组区域的核细胞态密度高得多(数据未显示)。目前正在在我们的实验室进一步调查鹰嘴豆核心定位预测的这些和其他有趣的方面。
组件集成和其他服务
为了促进其各种组件和能力之间的数据之间的无缝交换,CGWR后端可以实现动态连接和其三个主要部分,工具,映射和基因组浏览器之间的结果的频繁耦合。例如,用户可以进行BLAST运行,用于识别其感兴趣的序列的直字,以及输出页面上的链接,以便直接访问包含初探的兴趣的基因组区域。潜在的潜在地区的列表也可以通过键入感兴趣的关键字来获得。由此识别的ID可以通过地图菜单映射到高分辨率交互图像的组装基因组,其中再次,提供与浏览器的直接连接。例如,在Blast搜索中获得的鹰嘴豆喉咙的基因ID可以粘贴到地图菜单中以可视化这些域位于鹰嘴豆基因组上的位置,以及它们是否显示出任何朝向空间聚类的趋势。每当发现感兴趣的基因在同一系列的其他基因模型附近,它被分配给一个可以在交互式地图上可视化的群集以及CGWR浏览器,以便对聚类的其他方面进行详细分析地区。除了这些后端的规定外,CGWR的链接菜单可以访问各种数据集,并链接到鹰嘴豆和基于豆科的基因组学研究的重要外部信息。此菜单还可以下载此工作中使用的各种数据集。
典型分析的例子
用户可能有一个感兴趣的基因,他们想找到豆科和非豆科同源物。可以在工具菜单中对鹰嘴豆基因组序列使用BLAST,并在BLAST输出页面中找到浏览器的链接。这将导致鹰嘴豆的同源基因在浏览器显示,从这里,通过使用15个预先计算的比较基因组追踪之一,可以找到与六种豆科植物和两种非豆科植物的相似性。另外,9个预先计算的轨迹可以对鹰嘴豆及其野生和desi型品种之间的结构变异进行详细的核苷酸水平评估。点击这些轨道可以查看比较排列以及关于两个基因组的排列坐标的信息。如果在鹰嘴豆中发现了感兴趣的基因的多个同源物,这些都可以使用Maps菜单进行交互和方便地检测各自基因家族中的簇。如果有的话,可以通过地图和基因组浏览器之间的整合链接,进一步分析该区域及其周围的其他感兴趣的特征。例如,用户可以切换聚类的同源轨迹,以确定相关的基因模型集是否也聚类在其他九个植物基因组中的任何一个。在聚类基因模型的上游调控区域中,如果有共享的转录因子结合位点,也可以通过浏览器中的指定轨迹显示出来。通过对EST、mRNA和转录本的跟踪,可以看到这些基因模型在鹰嘴豆中的组织特异性或器官特异性表达的程度,如果有的话。 Additionally, turning on some of the TFBS prediction tracks would suggest whether there is evidence for any specific TF binding to the promoters of the identified homologs. Patterns of nucleosome arrays can also be visualized for assessment of DNA accessibility in these regions of the genome, and compared for overlap with other features such as coding and non-coding areas.
讨论
鹰嘴豆基因组网络资源的建设和发展是为了使豆科群落能够开展基于连贯的信息学的探索最近测序的鸡肝核基因组,包括底层DNA序列结构和功能方面的映射和可视化。CGWR对鹰嘴豆基因组提供了广泛的分析,以及对鹰嘴豆的11种其他物种或基因型进行比较交叉物种研究,是唯一的植物资源提供定制和互动染色体定位图对于所需的基因模型和基因家族。此外,CGWR内的浏览器是唯一的植物基因组浏览器,其提供核心定位模式的显示和分析,揭示沿编码区的规则间隔的核瘤阵列的情况,以及上游调节区域的核心的趋势.因此,CGWR结果可以为未来的研究努力提供了对核小体图的比较分析以及其基因组排列可以控制基因调控的比较分析的机会。CGWR的另一个独特功能是穷举的信息独联体-鹰嘴豆核基因组上游区域的调控元件数据,基于不同的预测方法。大多数现有的植物基因组浏览器提供传统的基因模型、转录本、复制、共线区域和重复元素的数据。在许多情况下,甚至连这些信息都无法获得。例如,鹰嘴豆基因组浏览器目前由豆科信息系统(LIS)托管http://cicar.comparative-legumes.org/gb2/gbrowse/ca1.0/[19.].它提供了对CDC-Frontier (kabuli型)鹰嘴豆基因型的基因组(1.0版)和转录组组装(v2版)的访问,但不支持基因组内或基因组间分析。另一个专门用于冷藏食品豆类基因组的数据库在http://coolseasonfoodlegume.org/[20.].CSFL在基于豆类的QTL方面是高度信息的,包括豌豆,扁豆,鹰嘴豆和鹰嘴豆和Medicago但是,虽然它没有Desi-型鹰嘴豆的数据,但其Kabuli型基因组浏览器的轨道少于十条曲目,没有任何其他凉爽的季节食物豆类的比较基因组学。然而,LIS和CFSL的这种比较技术讨论绝不会破坏任何一个资源,因为它们具有大量优势,包括最先进的可视化技术,高度高级QTL搜索选项以及豆类的常规全局更新相关新闻和研讨会,为社区提供了大量利益。总之,我们认为CGWR填补了不同的利基,而不是通过与其他平台竞争来创造冗余,因此豆类社区将受到欢迎。在CGWR中实施的创新方法将为不同类型的生物学相关数据进行深入显示和解释技术,从而鼓励多个未来的研究举措。CGWR中也可提供完整的文档和在线教程,以了解资源及其功能。
结论
CGWR是用于动态分析的多刻录网资源,并专门用于鹰嘴豆核基因组的最新组合组装的专门视野。如果没有任何登录要求,则可以自由使用http://www.nipgr.res.in/cgwr/home.php..每一个新的鹰嘴豆组装草案都将集成到CGWR中,并在发布时提供。在未来的版本中,我们希望修改和扩展浏览器,以添加豆科植物研究社区感兴趣的新功能,如纳入生化途径,以不同颜色同时可视化多个基因家族地图,以及与更多植物物种的比较基因组学轨迹。此外,我们希望将细胞器基因组整合到CGWR中,以实现更完整的全局分析。我们欢迎并鼓励我们的用户为新的和有趣的轨道提出建议。
可用性和要求
项目名称:鹰嘴豆基因组网络资源
项目主页:http://www.nipgr.res.in/cgwr/home.php..
操作系统 (年代):平台无关的
编程语言:Perl, PHP, FORTRAN, CGI
其他要求:零
许可证:开源许可GNU GPL v2
非的任何限制-学者:免费提供给社区
缩写
- 爆炸:
-
基本局部对齐序列工具
- ca_lg:
-
中投arietinum连锁群
- CD:
-
编码DNA序列
- CGI:
-
公共网关接口
- CGWR:
-
鹰嘴豆基因组网资源
- 重叠群:
-
由重叠的克隆DNA片段组装而成的DNA连续序列
- CPAN:
-
综合Perl归档网络
- CTDB:
-
Chickpea转录组数据库
- 脱氧核糖核酸:
-
脱氧核糖核酸
- 美国东部时间:
-
表达序列标签
- GFF:
-
通用功能格式
- GFF3:
-
通用功能格式版本3
- GMOD:
-
通用模型生物体数据库
- GUI:
-
图形用户界面
- gbrowse:
-
通用的基因组浏览器
- HTML:
-
超文本标记语言
- 线条:
-
长点缀元素
- LTRS:
-
长终端重复
- mirnas:
-
microRNA.
- mysql:
-
结构化查询语言
- NCBI:
-
国家生物技术信息中心
- NGCP:
-
下一代挑战计划
- 包含了:
-
拥有大量蛋白质家族的数据库
- PHP:
-
超文本预处理器
- PSL:
-
表示序列对齐的文本文件格式
- PSSM:
-
特定位置的评分矩阵
- RC Helitrons:
-
滚动圈子赫拉克里州
- RNA:
-
核糖核酸
- rrnas:
-
核糖体RNA.
- 锡:
-
短途元素
- Snornas:
-
小核仁RNA.
- SNP:
-
单核苷酸多态性
- SSR:
-
简单的序列重复
- STS:
-
序列标记站点标记表示基因组中的单个,唯一,序列定义的点
- SuperContig:
-
supercontig由一个或多个已知以特定顺序和方向发生的序列contig组成
- TFBS:
-
转录因子结合位点
- 图示:
-
转移RNA.
- WGS:
-
全基因组霰弹枪测序方法
参考文献
- 1。
Jain M,Misra G,Patel Rk,Priya P,Jhanwar S,Khan AW,Shah N,Singh VK,Garg R,Jeena G,Sharma P,康德C,Yadav M,Yadav G,Bhatia S,Tyagi AK,Chattopadhyay D.:脉冲作物鹰嘴豆草稿序列(中投arietinuml .)。植物学报,2013,74:715-729。10.1111 / tpj.12173。
- 2。
Varshney RK,歌C, Saxena RK, Azam年代,Yu年代,夏普AG)、大炮年代,门敏J,罗森BD,焦油国安B,文澜T,张X,拉姆齐LD,岩田聪,王Y,纳尔逊W,农民广告,白肢野牛点,C,索德伦德Penmetsa房车,徐C, Bharti AK, W,冬天P,赵年代,羽根JK, Carrasquilla-Garcia N, Condie是的,Upadhyaya高清,罗MC, et al:鹰嘴豆基因组序列(草案中投arietinum)为特质改进提供资源。NAT BIOTECHNOL。2013,31:240-246。10.1038 / NBT.2491。
- 3.
污物J,大炮某人,马Schlueter J, J, Mitros T,纳尔逊W,海顿耐DL,歌Q, JJ,泰伦程J,徐D, Hellsten U, GD,于Y,樱井T, Umezawa T, Bhattacharyya可,Sandhu D, Valliyodan B,林奎斯特E,皮托,格兰特D,蜀年代,古德斯坦D,巴里·K Futrell-Griggs M B,阿伯纳西Du J,田Z,朱L,等:古多倍体大豆的基因组序列。《自然》,2010,463:178-183。10.1038 / nature08670。
- 4.
李Varshney RK,陈W, Y, Bharti AK Saxena RK, Schlueter是的,多诺霍太,阿扎姆年代,风扇G,惠利,农民广告,谢里丹J,岩田聪,Tuteja R, Penmetsa房车,吴W, Upadhyaya高清,杨SP,沙T, Saxena KB, Michael T McCombie WR,杨B, G,杨H,王J,斯皮兰C,库克博士,可能GD,徐X, et al:木豆基因组序列(草案Cajanus毛竹),孤儿豆类群体的资源贫困农民。NAT BIOTECHNOL。2011,30:83-89。10.1038 / NBT.2022。
- 5。
菜豆v0.9, DOE-JGI and usa - nifa [http://www.phytozome.net/commonbean]
- 6.
吉尔茨奥尔德罗伊德年轻ND Debelle F,通用电气,R,大炮某人,Udvardi可,Benedito VA,梅耶尔KF, Gouzy J, Schoof H, Van de同行Y, Proost年代,库克博士,迈耶斯公元前Spannagl M,张F, de Mita年代,克里斯V, Gundlach H,周年代,玛吉J, Bharti AK,默里JD, Naoumkina妈,罗森B,西尔弗斯坦KA,唐H, Rombauts年代,赵PX,周P等:Medicago基因组提供了深入了解根瘤菌共生的进化。《自然》,2011,48(4):521 -524。10.1038 / 480162。
- 7。
佐藤年代,中村Y, T,金子Asamizu E,加藤T,地震区M, Sasamoto年代,渡边,小野,川岛K, Fujishiro T, Katoh M, Kohara M, Kishida Y,南城C, Nakayama年代,Nakazaki N,清水Y, Shinpo年代,高桥C,和田T,山田M, Ohmido N, Hayashi M,福井K,巴巴T, Nakamichi T, Mori H, Tabata S:豆类、基因组结构的莲花japonicus..DNA Res. 2008, 15: 227-239。10.1093 / dnares / dsn008。
- 8。
Donlin MJ:使用通用基因组浏览器(Gbrowse)。Curr Protoc生物信息学。2009,28:9.9.1-9.9。25。
- 9。
Jhanwar S,Priya P,Garg R,Parida Sk,Tyagi Ak,Jain M:野生鹰嘴豆的转录组测序作为标记开发的丰富资源。植物Biotechnol J. 2012,10:690-702。10.1111 / J.1467-7652.2012.00712.x。
- 10。
Garg R, Patel RK, Jhanwar S, Priya P, Bhattacharjee A, Yadav G, Bhatia S, Chattopadhyay D, Tyagi AK, Jain M:鹰嘴豆的基因发现和组织特异性转录组分析与大规模平行测序和网络资源开发。植物营养与肥料学报。2011,32(4):457 - 461。10.1104 / pp.111.178616。
- 11.
Swarbreck D,Wilk C,Lamesch P,Berardini Tz,Garcia-Hernandez M,Foerster H,Li D,Meyer T,Muller R,Ploetz L,Radenbaugh A,Singh S,Swing V,Tissier C,Zhang P,Huala E:拟南芥信息资源(TAIR):基因结构和功能注释。核酸RES。2008,36:D1009-D1014。10.1093 / nar / gkm965。
- 12.
Altschul SF, Madden TL, Schäffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ: gap BLAST和psp -BLAST:新一代蛋白质数据库搜索程序。核酸学报1997,25:3389-3402。10.1093 / nar / 25.17.3389。
- 13。
Haas BJ, Delcher AL, Wortman JR, Salzberg SL: DAGchainer:挖掘片段基因组复制和共同的工具。生物信息学。2004,20(18):3643-6。10.1093 /生物信息学/ bth397。
- 14。
Higo K, uawa Y, Iwamoto M, Korenaga T:植物顺式调控DNA元件(PLACE)数据库:1999。核酸学报1999,27:297-300。10.1093 / nar / 27.1.297。
- 15.
Portales-Casamar E, Thongjuea S, Kwon AT, Arenillas D, Zhao X, Valen E, Yusuf D, Lenhard B, Wasserman WW, Sandelin A: JASPAR 2010:极大扩展了转录因子结合谱的开放获取数据库。核酸学报2010,38(4):497 - 503。10.1093 / nar / gkp950。
- 16.
xie L, Fondufe-Mittendorf Y, Xia L, Flatow J, Widom J, Wang JP:利用持续时间隐马尔可夫模型预测核小体的位置。生物信息学。2010,11:346-10.1186/1471-2105-11-346。
- 17.
王立,王立,KO TP,李烨,雷耶,何居,黄WP,LO CF,Wang AH:白斑综合征病毒蛋白ICP11:破坏核心组装的组蛋白结合DNA模仿。Proc Natl Acad SCI U S A. 2008,105:20758-20763。10.1073 / pnas.0811233106。
- 18。
Papanicolaou A,Heckel DG:GMOD Drupal BioInformatic服务器框架。生物信息学。2010年,26:3119-3124。10.1093 / Bioinformatics / BTQ599。
- 19。
Gonzales MD,Archuleta E,Farmer A,Gajendran K,Grant D,Shoemaker R,Beavis WD,Whaugh Me:豆科信息系统(LIS):比较豆科生物学的综合信息资源。核酸RES。2005,33:D660-665。10.1093 / nar / gki128。
- 20.
Main, D, Cheng, C-H, flin, S.P, Jung, S, Zheng, P, Coyne, C.J, McGee, R, Mockaitis, K:清凉季节食用豆类数据库:基础、转化和应用研究综合资源。国际植物和动物基因组会议的诉讼程序:2013年1月12日至16日,美国圣地亚哥,美国
致谢
该项目是在印度政府生物技术部(DBT)资助的鹰嘴豆基因组学下一代挑战计划(NGCP)下开发的。BT / PR12919 AGR / 02/676/2009)。GM和PP在工作期间分别获得了印度政府DBT和印度政府CSIR的高级研究奖学金。GY是印度科学和技术部门科学和教育研究委员会(塞尔维亚)颁发的女性卓越奖的获得者。感谢印度政府生物技术发展局(DBT) NIPGR子分布式信息中心(sub dic)在生物技术信息系统网络(BTISNET)赠款下提供的设施。BT / BI / 04/069/2006)。我们也感谢CGWR用户委员会成员的有益投入和思想贡献。
作者信息
从属关系
相应的作者
附加信息
相互竞争的利益
提交人声明他们没有竞争利益。
作者的贡献
GM、PP、NB开发CGWR模块,浏览器跟踪核小体定位研究。PP构造了映射和聚类模块。NB进行了TFBS预测。MJ, SB和DC贡献了在这项工作中使用的各种鹰嘴豆特定数据集。GY和AKT构思并设计了软件。所有作者协调起草,阅读和批准最终稿件。
作者为图像提交的原始文件
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。
本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。
如欲查阅本许可证副本,请浏览https://creativecommons.org/licenses/by/4.0/.
创作共用及公共领域专用豁免书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
Misra,G.,Priya,P.,Bandhiwal,N。等等。鹰嘴豆基因组网资源:Desi-Type的可视化和分析中投arietinum核心基因组对比较探索豆类。BMC植物BIOL.14,315(2014)。https://doi.org/10.1186/s12870-014-0315-2
收到了:
接受:
发表:
关键词
- Cicer Arietinum ICC4958.
- 聚类
- 比较基因组学
- 基因组浏览器
- 映射