图3.
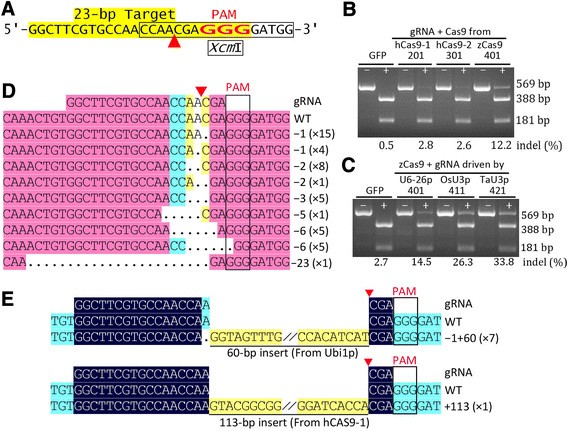
验证玉米密码子优化的CAS9和三种POL-III启动子在玉米原生质体中驾驶GRNA表达。(一种)来自ZM的目标网站的序列HKT1.轨迹。PAM,推定的乳沟网站(红色箭头),以及XCM.指示I网站(盒装)。(公元前)突变分析XCM.我消化了PCR片段。GFP,201,301,401(b):分别从用PUC-GFP(对照),PBUN201-ZT1,PBUN301-ZT1和PBUN401-ZT1分别转染的玉米原生质体的基因组DNA的PCR片段。三个CRISPR / CAS9载体具有相同的GRNA但不同Cas9.:HCAS9-1/2,两种类型的人密码子优化Cas9.;ZCAS9.那Zea Mays.密码子优化Cas9.。GFP,401,411,421(C):PCR片段来自PUC-GFP,PBUN401-ZT1,PBUN411-ZT1和PBUN421-ZT1转染;三个CRISPR / CAS9矢量具有相同的Z.Cas9.和GRNA,但GRNA由三种不同的POL-III启动子驱动。- 并且+表明PCR片段是否被消化XCM.I.基于残余未消化的PCR片段(+泳道:569bp)的百分比百分比计算突变效率(%indel)对总PCR产物( - 通道);WT Indel值应被视为背景级别。(d,e)抗克隆PCR片段抗性突变等位基因序列的对准XCM.我消化了。突变的等位基因包括缺失(d)和插入(e)。点,删除的基础。突出显示表示对齐片段的同源性,并且仅示出了对准的感兴趣区域。表示indel的类型和相同类型的indel的数量。
