- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
新测序生物的功能注释管道的评估和集成:马铃薯基因组作为测试用例gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba14gydF4y2Ba,文章号:gydF4y2Ba329gydF4y2Ba(gydF4y2Ba2014gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
对于大多数生物来说,即使它们的基因组序列是可用的,关于单个基因或蛋白质的功能信息也很少。基于序列、“组学”和文献数据,已经开发了几种用于功能分析的注释管道。然而,研究人员几乎没有得到关于他们表现如何的指导。在这里,我们使用最近测序的马铃薯基因组作为案例研究。之所以选择马铃薯基因组,是因为它的基因组是新测序的,即使单个马铃薯基因的信息相对充足,而且可以获得多个基因表达谱,但它也是一种非模式植物。gydF4y2Ba
结果gydF4y2Ba
我们表明,与基于实验验证的马铃薯基因的“金标准”相比,马铃薯的自动基因注释的准确性较低。此外,我们评估了六个最先进的注释管道,并表明它们的预测显著不同(管道之间的Jaccard相似系数平均为0.27)。为了克服这种差异,我们引入了一种简单的基于GO结构的算法,以协调不同管道的预测。我们表明,集成注释涵盖了更多的基因,将高共表达的GO过程数量增加了50%以上,并且与金标准的一致性更高。gydF4y2Ba
结论gydF4y2Ba
我们发现不同的注释管道产生不同的结果,并展示了如何将它们集成到一个统一的注释中,该注释的质量比每个单独的管道都要高。我们提供了PGSC和ITAG马铃薯基因模型的改进功能注释,以及可以应用于其他管道和改进其他生物注释的工具。这将极大地有助于未来对马铃薯和其他具有新测序基因组的生物的“组学”数据集的功能分析。本文提供了新的马铃薯注释。gydF4y2Ba
背景gydF4y2Ba
土豆(gydF4y2Ba茄属植物tuberosum)gydF4y2Ba是人类消费的第三大粮食作物[gydF4y2Ba1gydF4y2Ba].因此,它对我们的粮食安全非常重要,我们需要了解它的基因组。马铃薯研究面临的主要挑战是它对干旱胁迫的敏感性和对某些疾病(如卵菌)缺乏抵抗力gydF4y2BaPhytopthora 5种,gydF4y2Ba这导致了19世纪40年代的爱尔兰饥荒。农民需要使用大量的杀菌剂来保护他们的马铃薯作物,从而增加了种植成本并威胁到环境。例如,全球成本保护和产量损失所致gydF4y2Bap . 5gydF4y2Ba估计每年为4.8亿欧元[gydF4y2Ba2gydF4y2Ba].gydF4y2Ba
最近,马铃薯基因组(gydF4y2Ba茄属植物tuberosumgydF4y2Ba马铃薯基因组测序联盟(PGSC)对Phureja类群进行了测序。PGSC基因组分析报告了39031个代表性转录本的基因模型,56218个包括剪接变异[gydF4y2Ba3.gydF4y2Ba].在后来的努力中,国际番茄注释小组(ITAG)通过联合分析番茄和土豆基因组,产生了新的基因模型[gydF4y2Ba4gydF4y2Ba].这些新的基因模型分别覆盖了34,727个和35,004个预测的番茄和土豆基因组的蛋白质编码基因。不幸的是,在新测序的基因组中,很少有实验验证的基因(例如,通过荧光标记蛋白或基因敲除),其中与已建立的模式生物不同,很少有基因具有已验证的功能,如马铃薯的情况。全面和准确的功能注释这些最近测序的基因组基因是有效利用这些基因组数据的先决条件。gydF4y2Ba
功能注释的一个关键工具是基因本体(GO),它提供了一组结构化的已定义术语来表示基因属性[gydF4y2Ba5gydF4y2Ba].基因本体的结构由三个主要领域组成:gydF4y2Ba蜂窝组件gydF4y2Ba(CC),细胞或其细胞外环境的部分;gydF4y2Ba分子功能gydF4y2Ba(MF),指基因产物在分子水平上的基本活性;而且gydF4y2Ba生物过程gydF4y2Ba(BP),描述了一组功能相关的分子事件。因此,完整的GO结构提供了一个统一的生物术语词汇表,也可以用来评估不同术语的生物相似性[gydF4y2Ba6gydF4y2Ba].注释一个基因意味着将其置于三个基因本体域中的一部分或全部。gydF4y2Ba
植物科学的最新进展以高通量测序数据的可用性和质量的迅速提高为标志。这些数据最基本的用途是基因功能预测,其中GO起着关键作用。有几个计算套件,如EXPANDER [gydF4y2Ba7gydF4y2Ba], MapMan [gydF4y2Ba8gydF4y2Ba],墨卡托[gydF4y2Ba9gydF4y2Ba]和AmiGO [gydF4y2Ba10gydF4y2Ba]使生物学家能够在几种植物模型系统中进行氧化石墨烯富集分析。这通常是通过首先识别一组在给定表达数据集中表现相似的基因,寻找该组中高度丰富的本体术语,并将高度丰富的功能与属于同一组的未注释的基因联系起来。这个过程有时被称为“联想负罪感”。自动化基因功能注释也与充分研究的植物模式生物相关,例如gydF4y2Ba拟南芥,gydF4y2Ba西红柿,gydF4y2BaBrachypodiumgydF4y2Ba还有水稻,其中约40%的基因仍然没有任何已知的功能[gydF4y2Ba11gydF4y2Ba].gydF4y2Ba
为了将功能注释分配到已测序的植物转录本,研究人员可以使用几种基于序列的注释管道。有关自动功能注释背后的方法和原则的全面总结,请参阅[gydF4y2Ba12gydF4y2Ba].一些最近的努力已经作出了描述植物基因组的注释质量。例如,Jaramillo-Garzón等。[gydF4y2Ba13gydF4y2Ba]采用序列特征,MF和CC项的可预测性较高,BP项的可预测性较低。然而,分析仅限于GO术语的一个小子集(GO- slim)。拉姆萨克等人[gydF4y2Ba8gydF4y2Ba]介绍了GOMapMan,一种用于植物基因注释可视化和分析的工具。在马铃薯中,为了增加与GO项相关的马铃薯基因数量,对26个已测序植物基因组的同源基因家族信息进行了分析[gydF4y2Ba14gydF4y2Ba].尽管如此,仍然迫切需要一种强大的、自动化的方法来评估和比较全基因组注释管道。gydF4y2Ba
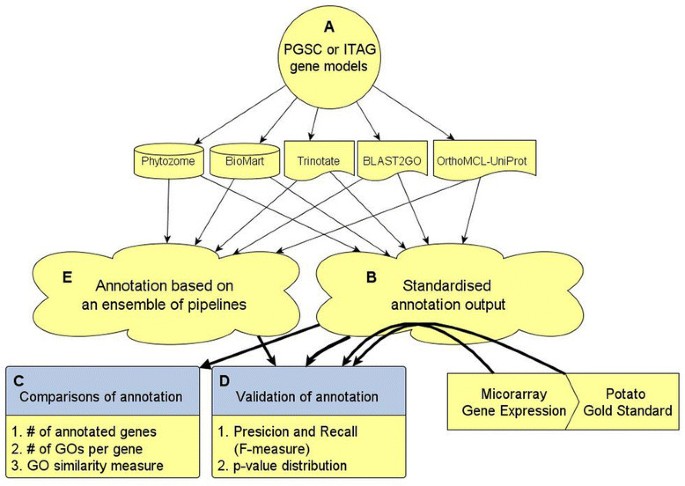
新测序生物的典型全基因组功能注释始于使用单一的“默认”管道。在这里,我们分析了两组马铃薯基因模型,来自ITAG和PGSC。我们比较了六种注释管道:Trinotate HMM, Trinotate BLAST [gydF4y2Ba15gydF4y2Ba], OrthoMCL-UniProt [gydF4y2Ba16gydF4y2Ba], blast2go [gydF4y2Ba17gydF4y2Ba], Phytozome [gydF4y2Ba18gydF4y2Ba]和BioMart提供的InterPro2GO [gydF4y2Ba19gydF4y2Ba)(图gydF4y2Ba1gydF4y2Ba).选择这些管道是因为它们试图提供整个基因组的全面注释。其中一些管道仅基于序列相似性(BLAST),另一些依赖于特定的结构域,还有一些基于同源基因家族组的聚类。正如我们将要展示的,这项工作的一个明确结论是,基因组的功能注释应该依赖于多个注释管道。gydF4y2Ba
管道比较,准确性验证和集成过程的概述。(一)gydF4y2BaPGSC和ITAG基因模型被用作评估6个管道的输入。gydF4y2Ba(B)gydF4y2Ba每个管道的注释被转化为基因ID - GO术语关联。gydF4y2Ba(C)gydF4y2Ba通过注释的基因模型数量、每个基因模型关联的GO术语数量以及GO相似性来比较注释。gydF4y2Ba(D)gydF4y2Ba每个管道注释的质量和全面性是通过将其预测与实验验证的注释(金标准)进行比较来计算的。此外,基因共表达数据被用于测试预测共享相同GO过程的基因是否显著共表达。gydF4y2Ba(E)gydF4y2Ba使用集成所有管道的结果创建一个集成注释,并使用d中相同的标准进行验证。将集成注释的结果与单个管道的结果进行比较。gydF4y2Ba
通过检查这些管道生成的GO项,我们证明它们预测了非常不同的注释(例如,平均而言,由两个管道注释的基因中只有不到30%被分配了相同的功能)。为了评估管道的性能,我们首先创建了一组具有已知功能特征的马铃薯基因(以下称为“金标准”),包括来自特征良好的生物合成类胡萝卜素途径的基因。我们表明,与黄金标准相比,管道可能具有相当低的准确性。由于金标准的大小相当适中(116个PGSC基因id),我们使用了基于基因表达数据的额外验证方案。在参与同一生物过程的基因的表达模式应该比偶然性预期的更相似的前提下,我们根据每个管道的过程内基因共表达水平来评估其预测。我们表明,虽然所有管道提供的进程内共表达都比预期的要高得多,但方法之间存在很大差异。我们介绍了一种简单的方法,将不同管道的结果组合到一个集成注释中。与单一管道相比,该方法提高了基因覆盖率、预测精度以及预测GO过程的整体共表达。除了改进马铃薯基因的注释,我们的分析还提供了通用工具,可用于改进其他新测序植物的注释。gydF4y2Ba
结果与讨论gydF4y2Ba
最先进的注释工具的概要gydF4y2Ba
在这项研究中,我们测试了马铃薯基因组的自动注释管道。我们使用了六种最先进的工具来预测氧化石墨烯基因功能:(1)Trinotate HMM, (2) Trinotate BLAST [gydF4y2Ba15gydF4y2Ba], (3) OrthoMCL-UniProt [gydF4y2Ba16gydF4y2Ba], (4) blast2go [gydF4y2Ba17gydF4y2Ba], (5) Phytozome [gydF4y2Ba18gydF4y2Ba],以及(6)InterPro2GO [gydF4y2Ba19gydF4y2Ba].参见方法和附加文件gydF4y2Ba1gydF4y2Ba方法S1-4。我们注意到,每个程序都有自己的一组参数,为特定的数据集拟合最佳参数组合需要大量的工作。在这一领域的常见做法是使用带有默认参数值的已发布工具(参见示例[gydF4y2Ba20.gydF4y2Ba],[gydF4y2Ba21gydF4y2Ba].如有必要,我们使用Pfam2GO等自动映射文件将其预测功能映射到GO术语,并将基因和转录本映射到蛋白质标识符。因此,在我们的分析中,基因对应于管道输出中出现的转录本或蛋白质。接下来,将每个管道的输出总结为一组预测的基因- go项对。对于每个基因,我们只保留最“具体”的GO术语。也就是说,如果一个基因与两个GO术语a和B相关联,但B是a的泛化(即在GO层次结构中a的祖先),我们将B排除在外。我们称之为这一步gydF4y2Ba祖先删除gydF4y2Ba.值得注意的是,在过滤之后,许多基因仍然与一个以上的GO术语相关联,因为一个基因可以有几个相关的注释,其中没有一个是另一个基因的祖先。有关所有管道的输出,请参阅附加文件gydF4y2Ba2gydF4y2Ba:表S1,附加文件gydF4y2Ba3.gydF4y2Ba:表S2,附加文件gydF4y2Ba4gydF4y2Ba:表S3,附加文件gydF4y2Ba5gydF4y2Ba:表S4,附加文件gydF4y2Ba6gydF4y2Ba:表S5和附加文件gydF4y2Ba7gydF4y2Ba表S6为PGSC和附加文件gydF4y2Ba8gydF4y2Ba:表S7,附加文件gydF4y2Ba9gydF4y2Ba:表S8,附加文件gydF4y2Ba10gydF4y2Ba:表S9,附加文件gydF4y2Ba11gydF4y2Ba:表S10,附加文件gydF4y2Ba12gydF4y2Ba:表S11和附加文件gydF4y2Ba13gydF4y2Ba表S12为ITAG。尽管基因本体论有其局限性,因为它偏向于已知的东西,但它仍然是基于序列身份、域和结构以及文献研究的功能注释推断功能的通用关键工具。gydF4y2Ba
管道之间的差异gydF4y2Ba
每个管道的输出可以表示为三元组(P, G, GO),其中P是所有预测的基因-GO项对的集合(去除祖先后),G是P覆盖的基因的集合,GO是P覆盖的GO项的集合。我们测量了从研究中使用的六个管道获得的三元组之间的成对相似性。使用三种不同的方法来比较两条管道的输出A = (PgydF4y2Ba一个gydF4y2BaGgydF4y2Ba一个gydF4y2Ba,去gydF4y2Ba一个gydF4y2Ba)和B = (PgydF4y2BaBgydF4y2BaGgydF4y2BaBgydF4y2Ba,去gydF4y2BaBgydF4y2Ba).首先,我们测量了管道P的预测之间的重叠gydF4y2Ba一个gydF4y2Ba和PgydF4y2BaBgydF4y2Ba.这是通过计算P的交点大小之间的比率来完成的gydF4y2Ba一个gydF4y2Ba和PgydF4y2BaBgydF4y2Ba和P的并集的大小gydF4y2Ba一个gydF4y2Ba和PgydF4y2BaBgydF4y2Ba.这一措施被称为gydF4y2BaJaccardgydF4y2Ba分数(gydF4y2Ba22gydF4y2Ba],[gydF4y2Ba23gydF4y2Ba].其次,我们测量了覆盖基因集G之间的相似性gydF4y2Ba一个gydF4y2Ba和GgydF4y2BaBgydF4y2Ba计算他们的杰卡德分数。这两个分数是互补的:第一个衡量的是A和B之间的总体相似性,而第二个衡量的是A和B覆盖相同基因的趋势。然而,这些分数忽略了GO结构,因此它们忽略了不同GO术语之间的功能相似性。因此,我们也使用了基于GO术语语义相似度的相似度评分[gydF4y2Ba24gydF4y2Ba].给定一个特定的GO类型GT (BP或MF),对于每个基因,我们测量其在a中的GO术语和在B中的GO术语之间的语义相似性,然后将所有基因的平均值作为a和B在GT中的相似性(详见方法)。由于这个分数使用了GO层次结构,我们称之为gydF4y2Ba小gydF4y2Ba.gydF4y2Ba
图中显示了预测的无结构相似性的一个例子gydF4y2Ba2gydF4y2BaA.该图显示了管道PGSC MF预测之间的成对Jaccard评分。总体上相似度很低,平均为0.27。然而,可以观察到局部模式。例如,InterPro2GO、Trinotate HMM和Phytozome更相似(平均0.46)。数字gydF4y2Ba2gydF4y2BaB为不同管道注释的PGSC基因之间的Jaccard相似性。平均相似度较高,为0.54,仍属较低水平。这表明不同的管道倾向于覆盖不同的基因,即使覆盖相同的基因,它们也经常将不同的注释与它们联系起来。即使重新计算仅限制每对管道共享基因的无结构相似性(考虑MF和BP预测),平均得分也只有0.27。gydF4y2Ba
PGSC基因基于结构的MF和BP相似性如图所示gydF4y2Ba2gydF4y2BaC和gydF4y2Ba2gydF4y2BaD.关于ITAG数据的类似矩阵在附加文件中显示gydF4y2Ba1gydF4y2Ba:图S1。同样,管道往往非常不同,BP的平均相似性为0.29,MF的平均相似性为0.42。分数高于无结构方法,因为当预测不同但生物学相似时,基于结构的方法会给出更高的分数。同样,就像图中的无结构分数一样gydF4y2Ba2gydF4y2BaA、InterPro2GO、Trinotate HMM和Phytozome在BP和MF中都形成了一个簇。综上所述,管道之间的差异表明管道所覆盖的基因集不同,相同基因在不同管道中的注释可能非常不同。gydF4y2Ba
管道组合gydF4y2Ba
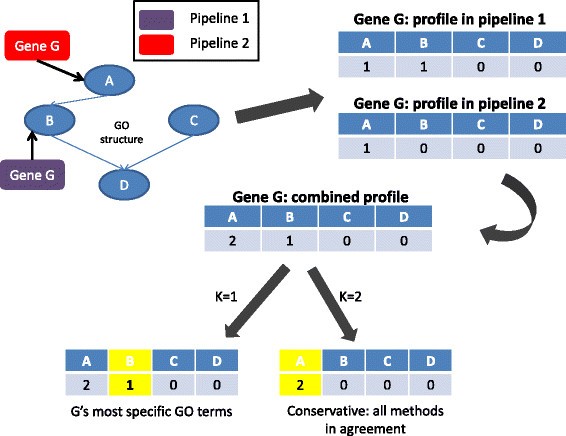
不同管道的基因注释的显著差异要求整合不同的预测,以提供统一的马铃薯基因注释。受之前研究的启发,我们开发了一种简单的集成算法[gydF4y2Ba25gydF4y2Ba].我们的算法将所有管道的预测作为输入,并将每个基因的预测合并到一个分数向量中,表示为基因的得分gydF4y2Ba结合剖面gydF4y2Ba(图gydF4y2Ba3.gydF4y2Ba).简单地说,我们首先计算gydF4y2Bapipeline-specificgydF4y2Ba基因资料。对于预测配对(G, t)的特定管道,其中G是一个基因,t是一个GO项,如果G与t或至少一个后代相关,则该轮廓的第t个位置为1,否则为0(图中右上方)gydF4y2Ba3.gydF4y2Ba).每个基因G的组合剖面是其管道特定剖面的和(图gydF4y2Ba3.gydF4y2Ba右)。在一个基因的组合剖面的值显示有多少管道同意每个基因- go术语关联。给定一个阈值k,对于每个基因,我们报告所有合并得分≥k的GO项。这个过程为每个基因生成一个GO术语列表。我们称之为变种gydF4y2BaEnsemble-k。gydF4y2Ba最后,我们应用上面描述的祖先删除过滤器。因此,k的每个值都产生了集成算法的不同变体。数字gydF4y2Ba3.gydF4y2Ba展示了Ensemble-1和2的一个玩具例子。为了清晰起见,在下一节中我们将使用该名称gydF4y2Ba注释的方法gydF4y2Ba对于管道和集成算法的变体。我们还测试了一种更复杂的监督集成方法,它根据管道的平均F-measure对黄金标准进行排名(见下文),但这并没有改善结果(见附加文件)gydF4y2Ba1gydF4y2Ba:方法S6)。gydF4y2Ba
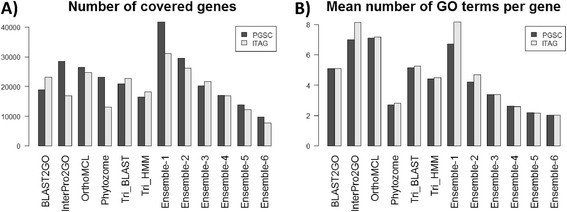
我们从基因覆盖和平均两方面比较了注释方法gydF4y2BangydF4y2BaGO项的个数gydF4y2BapgydF4y2Ba呃gydF4y2BaggydF4y2Baene,我们记作NGPG。理想情况下,基因覆盖率应尽可能高,而NGPG应较低[gydF4y2Ba26gydF4y2Ba].结果如图所示gydF4y2Ba4gydF4y2Ba一个和gydF4y2Ba4gydF4y2BaB.可以观察到不同管道之间以及ITAG和PGSC基因模型之间的显著差异。例如,基于PGSC数据,InterPro2GO和OrthoMCL-UniProt的基因覆盖率最高(分别为29,445和26,371),NGPG评分最高(分别为7和7.1)。然而,根据ITAG数据,OrthoMCL-UniProt的结果与PGSC相似,而InterPro2GO的基因数量下降到20,000以下,NGPG评分上升到8.1(图gydF4y2Ba4gydF4y2BaB)。gydF4y2Ba
数字gydF4y2Ba4gydF4y2Ba一个和gydF4y2Ba4gydF4y2BaB还显示了集成算法的基因覆盖率和NGPG。正如预期的那样,与使用ITAG和PGSC基因模型的单一管道相比,使用Ensemble-1或2增加了基因覆盖率。例如,基于PGSC,覆盖的基因模型(包括剪接变体)的数量为41,668 (k = 1)和29,495 (k = 2)。k值越大,基因覆盖率就会急剧下降,即使是单个管道也会覆盖更多的基因。使用Ensemble-1, NGPG得分与单一管道获得的最高得分相似,PGSC数据得分为6.70,ITAG数据得分为8.15。Ensemble-2导致NGPG急剧下降:PGSC为4.39,ITAG为4.68。gydF4y2Ba
总之,我们的结果表明,集成算法在不增加NGPG评分的情况下显著增加了基因覆盖率。集成-1在ITAG和PGSC数据上增加了超过5000个基因的基因覆盖,同时保持NGPG评分与最高的单个管道相似。与单个管道相比,ensemsil -2仅适度增加了基因覆盖率,但与所有管道相比(Phytozome除外,但后者的基因覆盖率较低),NGPG评分急剧下降,因此提供了更集中的注释。在下一节中,我们将演示上述改进并不是以牺牲精度为代价实现的。gydF4y2Ba
使用马铃薯金标准进行验证gydF4y2Ba
为了评估不同注释方法的预测,我们分别根据PGSC和ITAG数据编写了838和724个基因- go术语对的金标准,使用专家手动注释(见方法和附加文件)gydF4y2Ba14gydF4y2Ba:表S13,附加文件gydF4y2Ba15gydF4y2Ba:表S14和附加文件gydF4y2Ba16gydF4y2Ba:表S15)。金标准中包含的基因数量(43个文献参考,映射到116个PGSC基因id,参见附加文件gydF4y2Ba14gydF4y2Ba:表S13),它很小,但在像土豆这样的生物体中,它仍然含有大多数实验证据表明的基因。我们通过计算其基于go的精度和召回率来评估注释方法。使用GO结构来计算金标准验证的分数之前已经由[gydF4y2Ba27gydF4y2Ba].基于GO的基因回忆量衡量的是,根据黄金标准,基因的条款被预测的GO条款覆盖的程度。基于GO的基因精度衡量的是其预测的GO条款与金标条款的匹配程度。对于每个管道,我们计算平均精密度和平均召回率(在基因上),并报告f测度,这是精密度和召回率的调和平均值[gydF4y2Ba28gydF4y2Ba].有关这些计算的完整描述,请参阅方法。gydF4y2Ba
基于PGSC和ITAG数据的验证结果如图所示gydF4y2Ba5gydF4y2Ba和附加文件gydF4y2Ba1gydF4y2Ba:图S2。数字gydF4y2Ba5gydF4y2BaA表示BP GO项的f测度。Ensemble-1和2的f值分别为0.8和0.77,而表现最好的管道是InterPro2GO,仅为0.61。数字gydF4y2Ba5gydF4y2BaB表示MF金本位的f度量。集成1和2的f值分别为0.84和0.83,而性能最好的管道是InterPro2GO, f值仅为0.71。因此,结果与BP验证一致:Ensemble-1和ensemble - 2的性能最好,且在单管道条件下有所改善。综上所述,我们的结果表明,与单一管道相比,Ensemble-1和2提供了显著的改进。gydF4y2Ba
使用基因表达数据进行验证gydF4y2Ba
任何金标准的一个明显缺点是,它仅限于经过实验验证的基因,并受制于专家的意见。因此,我们增加了一个基于基因共表达分析的额外验证,其中我们测量了管道预测高共表达基因的相同go项的能力。我们的共表达分析基于来自20多个微阵列研究的326个表达谱中的12956个基因的基因表达。我们使用Pearson相关系数来衡量基因之间的共表达。gydF4y2Ba
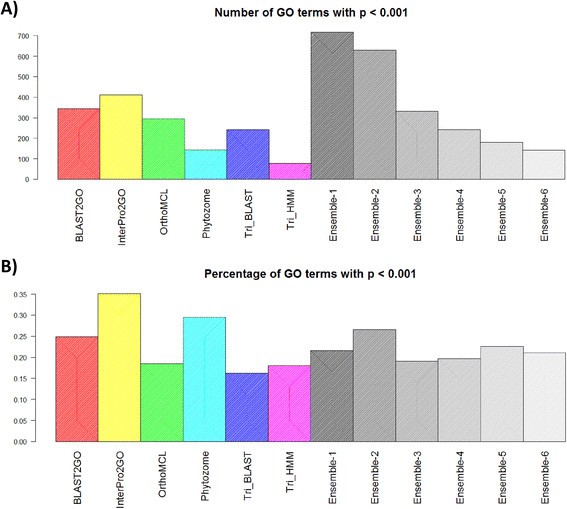
我们使用基因对共表达评分来验证预测的GO BP项。为了减少噪声,我们忽略了包含>500个基因或少于5个基因的项。给定一组根据特定的注释方法预测与同一GO术语相关的基因,我们测试其基因之间的共表达水平是否偶然高于预期(详见方法)。因此,对于特定注释方法中的每个项,我们计算了一个p值。为了在比较方法时总结这些值,我们计算了两个分数:(1)p <0.001的GO术语的数量,(2)p <0.001的GO术语的百分比(在所有至少有三个基因的预测术语中)。前者是衡量重要GO术语覆盖率的指标,而后者是衡量预测GO BP术语质量的指标。与黄金标准类似,该分析的目的只是比较管道。未来的工作可以使用类似的方法从不同的管道中选择高度共表达的GO术语进行后续分析。gydF4y2Ba
基于PGSC数据的基因共表达验证结果如图所示gydF4y2Ba6gydF4y2Ba.参见附加文件gydF4y2Ba1gydF4y2BaITAG结果见图S3。重要GO项数量最多的两个管道是InterPro2GO (n = 411)和BLAST2GO (n = 345)。重要氧化石墨烯术语百分比排名前两位的管道是InterPro2GO(35%)和Phytozome(30%)。集成算法显著提高了重要GO术语的数量:ensemble -1达到了718个,ensemble -2达到了650个。然而,集成方法在重要GO项的百分比方面并没有比单一管道更好:集成-1和2分别达到22%和27%。但是,Ensemble-2的评分优于除InterPro2GO和Phytozome外的所有管道。因此,集成方法在重要GO项的数量上提供了至少1.5倍的改进,代价是与最佳管道相比,重要GO项的百分比下降了8%。请注意,共表达和GO分析是互补的,因为金标准基因没有表现出异常高的共表达(见附加文件)gydF4y2Ba1gydF4y2Ba:方法S7)。gydF4y2Ba
使用基于排名的比较来合并不同的优点gydF4y2Ba
我们的分析表明,根据大多数标准,集成方法是有益的。但是,由于我们使用了多种方法来对方法进行评分,因此很难决定哪个k值是最好的,哪个管道更好。为了提供一个清晰统一的观点,我们使用了不同分数的非参数基于排名的合并[gydF4y2Ba29gydF4y2Ba].在前面的章节中,对于每种注释方法,我们在金标准分析中计算了两个F-measure分数,在基因共表达分析中计算了两个分数。此外,我们还比较了基因覆盖和NGPG的注释方法。注意,按NGPG分数排序时,分数越低越好。相比之下,当按照基因覆盖率对方法进行排名时,得分越高越好。为了巩固这些不同的分数,我们使用了六种排名:通过基因覆盖和NGPG得分,通过金标准验证的两个f测量,以及通过基因共表达验证的两个分数。我们在必要时将分数颠倒,以便每个方法的排名1是最好的,平均排名,并根据平均排名对方法进行排名。我们称之为分数gydF4y2Barank-merge。gydF4y2Ba
数字gydF4y2Ba7gydF4y2Ba显示PGSC (A)和ITAG (B)数据的rank-merge结果。前三种方法都是黑色的。在这两种情况下,最高的方法是Ensemble-2, PGSC的平均排名为1.66,ITAG的平均排名为1.16。在评估的不同管道中,Phytozome在PGSC数据中得分最高,平均排名3.66,BLAST2GO在ITAG数据中得分最高,平均排名3.50。请注意,Ensemble-1、2和3在两个测试中排名始终较高。参见附加文件gydF4y2Ba17gydF4y2BaPGSC和附加文件:表S16gydF4y2Ba18gydF4y2Ba:表S17为ITAG。因此,我们得出结论,集成方法,特别是当k = 2时,是有益的,可以帮助集成不同的基因功能预测管道。参见附加文件gydF4y2Ba1gydF4y2Ba:方法S5,详细重现结果并将管道应用到新的基因组。gydF4y2Ba
注意,使用k = 1相当于将来自所有管道(及其祖先)的所有注释分配给每个基因,然后执行祖先删除。虽然这种方法是最直观的集成,但我们在这里表明,改变k参数可以改善基因组的注释。gydF4y2Ba
对于我们的方法,一个看似自然的测试案例是评估它在预测拟南芥基因功能方面的价值。然而,目前尚不清楚如何以严格和公正的方式做到这一点。用于新测序植物基因功能注释的工具在很大程度上依赖于模式物种(如拟南芥)基因的序列相似性。为了测试这些工具在预测拟南芥基因功能方面的作用,必须排除所有直接或间接来源于拟南芥的注释。这样做需要跟踪间接注释源,这些源通常没有记录在管道中。相反,我们在评估中使用了新测序的马铃薯基因组以及实验验证的基因功能和丰富的基因表达数据。gydF4y2Ba
结论gydF4y2Ba
对于新近测序的非模式生物,基因的自动功能注释也主要依赖于基于序列的预测,往往存在基因覆盖率低、特异性差的问题。我们通过分析六个最先进的注释管道,证实了土豆基因组的情况。gydF4y2Ba
我们观察到,尽管所有管道都是基于序列分析,但不同管道对基因功能注释的预测存在显著差异。我们表明,结合来自几个管道的预测增加了基因本体预测的覆盖率和准确性。这里使用的简单集成方法可以很容易地应用于其他测序基因组,并通过利用不同的GO预测工具来改进功能注释。然而,当目标是选择最好的管道或整合不同的预测时,比较管道之间的一致性是不够的。管道也应该基于其预测的精度进行评估。最直观的方法是将管道与一组已知注释进行比较。然而,在新测序的生物中,如马铃薯,已知的注释在主要的公共数据库中很少。为了克服这一问题,我们编制了一个实验验证基因-氧化石墨烯关联的金标准。尽管这个黄金标准相对较小,但我们发现它对于比较管道是有用的。此外,为了克服金标准中基因数量有限的问题,我们使用了基于基因共表达测试管道预测与同一go术语相关的基因共表达的能力的第二种验证方法。gydF4y2Ba
最后,我们引入了不同管道的集成注释,它在金标准验证和共表达式验证中都优于单个管道。我们的整合方法依赖于选择一个参数k,它对应于我们过滤基因- go关联的严格程度。也就是说,当将一个基因与一个GO项关联时,必须至少有k条管道同意这种关联。因此,我们隐含地假设我们使用的每个管道都有有意义的预测。此外,在集成过程中,所有管道的重量相同。未来的分析可以寻求赋予更好的管道更多权重的方法,或者增加一个初始步骤,过滤掉预测质量异常低的管道。马铃薯基因组的新功能注释以及JHI茄粒微阵列探针的新功能注释可通过本文获得(附加文件)gydF4y2Ba17gydF4y2Ba:表S16,附加文件gydF4y2Ba18gydF4y2Ba:表S17和附加文件gydF4y2Ba19gydF4y2Ba:表S18)。我们还提供了开源R代码工具,用于实现附加管道和其他测序生物的方法。gydF4y2Ba
方法gydF4y2Ba
执行函数注释管道gydF4y2Ba
我们定义了gydF4y2Ba管道gydF4y2Ba作为一个自动化的过程,预测基因和功能之间的联系。管道的输入可以是DNA序列、蛋白质序列或蛋白质结构域。管道的输出是一组形式为(基因ID, GO术语ID)的对。我们分别运行ITAG (potato.Sotub.proteins.itag.v1.fasta)和PGSC (pgsc_dm_v3 . 3.4_pep_rep .fasta)基因模型的所有管道,使用默认设置如下:gydF4y2Ba
OrthoMCL-UniProt管道gydF4y2Ba
我们运行OrthoMCL [gydF4y2Ba16gydF4y2Ba管道分为两步:gydF4y2Ba
- 1.gydF4y2Ba
构建同源物簇:我们从Phytozome (v9.1)检索了16个植物蛋白质组,涵盖了整个植物系统发育。与马铃薯PGSC和ITAG基因模型预测的蛋白质组一起,我们使用blastp [gydF4y2Ba30.gydF4y2Ba];(参数:−e-value: 1e-05 -outfmt 6)。然后使用OrthoMCL v2构建同源蛋白簇。gydF4y2Ba
- 2.gydF4y2Ba
GO术语注释:为了用GO术语注释18个完整植物蛋白质组的每个蛋白质序列,我们对整个UniProt数据库(版本2013_08)进行了一次blast搜索[gydF4y2Ba31gydF4y2Ba], e值分界点为1e-10。对于每个蛋白质序列,我们都保留了一个十佳命中(即具有最低e值的命中)的排名列表。我们在UniProt中关联了列表中有GO注释的第一个命中。然后,OrthoMCL群集继承与其蛋白质相关的所有GO项,每个PGSC(和ITAG)蛋白质继承其群集的GO项。gydF4y2Ba
有关完整的协议细节,请参阅附加文件gydF4y2Ba1gydF4y2Ba:方法S2。gydF4y2Ba
BLAST2GO管道gydF4y2Ba
使用BLAST2GO接口[gydF4y2Ba17gydF4y2Ba],对NCBI NR数据库(blastp参数:−e-value: 1e-05 -max_target_seqs 20 -outfmt 5)爆破PGSC和ITAG蛋白序列。然后将blastp输出文件加载到Blast2GO (v2.6.6,默认参数)中,并根据其输出为PGSC和ITAG序列分配GO项。gydF4y2Ba
三酸盐管道gydF4y2Ba
在Trinotate套房里[gydF4y2Ba15gydF4y2Ba]我们使用NCBI-BLAST (SwissProt)的默认设置,HMMER [gydF4y2Ba32gydF4y2Ba],及Pfam [gydF4y2Ba33gydF4y2Ba].有关完整的协议细节,请参阅附加文件gydF4y2Ba1gydF4y2Ba:方法S3。gydF4y2Ba
phytozome管道gydF4y2Ba
我们从Phytozome v9.1下载了土豆注释[gydF4y2Bahttp://www.phytozome.net/potato.phpgydF4y2Ba;[gydF4y2Ba18gydF4y2Ba) (gydF4y2Bahttp://www.phytozome.net/potato.phpgydF4y2Ba).基因注释是gydF4y2Ba茄属植物tuberosumgydF4y2BaPhureja DM1-3 516R44 (CIP801092)gydF4y2Ba基因组注释v3.4映射到gydF4y2BapseudomoleculegydF4y2Ba序列gydF4y2Ba(PGSC_DM_v3_2.1.10_pseudomolecules.fa)。gydF4y2Ba
来自BioMart的InterPro2GO数据gydF4y2Ba
我们从(下载土豆数据gydF4y2Bahttp://central.biomart.org/gydF4y2Ba).BioMart中的GO术语来源于半自动化的InterPro2GO [gydF4y2Ba19gydF4y2Ba].gydF4y2Ba
格式化管道gydF4y2Ba
为了比较管道,我们将它们的预测注释映射到一组通用的基因本体(GO)术语。如果原始管道输出不在GO术语id中,则使用GO术语的基因本体联盟映射文件将其映射到GO id。我们将此方法应用于Trinotate, InterPro2GO, BLAST2GO, Phytozome管道,并通过OrthoMCL聚类方法定位18个已测序植物物种的同源和副同源基因家族。gydF4y2Ba
制定马铃薯“黄金标准”gydF4y2Ba
根据《PlantCyc》中报道的马铃薯湿实验室功能基因研究的文献证据,构建了一套马铃薯基因的“金标准”[gydF4y2Bahttp://pmn.plantcyc.org/PLANT/organism-summarygydF4y2Ba]及其他有关马铃薯的研究[gydF4y2Ba34gydF4y2Ba] - [gydF4y2Ba37gydF4y2Ba].总共创建了43个马铃薯基因/蛋白质的列表(附加文件)gydF4y2Ba14gydF4y2Ba:表S13)。这些蛋白质名称被搜索到PGSC发布的相应标识符[gydF4y2Ba3.gydF4y2Ba],得到116个独特的PGSC基因标识符。gydF4y2Ba
上述基因列表与所有6个测试管道中的1658个GO术语匹配。然后,在文献搜索的帮助下,以无偏倚的方式手动对每个基因- go术语关联进行评分,其中为go关联分配分数的专家不知道注释来自哪个管道。集合中的每个GO项都被分为“1”(低证据)、“2”(中性或未知)和“3”(高证据)。在最后的分析中,只有3分的关联分数被用于金标准,产生了838个注释(附加文件gydF4y2Ba15gydF4y2Ba:表S14)。为了对两种基因模型进行分析,PGSC基因使用BLAST(同源性>95%,长度>100个氨基酸)定位到ITAG基因。这产生了具有724个注释的ITAG金标准(附加文件gydF4y2Ba16gydF4y2Ba:表S15)。gydF4y2Ba
比较管道和金标准评估gydF4y2Ba
数学符号gydF4y2Ba
在“结果”一节中,我们概述了与金标准比较管道和评估管道的计算。在这里,我们提供了这些计算的完整描述。为此,我们从更详细的定义开始。gydF4y2Ba
设G为被测生物体中所有基因的集合,设T为所有GO项的集合。管道的输出gydF4y2BaPgydF4y2Ba是一对吗gydF4y2BaP = {PgydF4y2Ba1gydF4y2Ba,…,pgydF4y2BakgydF4y2Ba}gydF4y2Ba其中每个gydF4y2Ba注释对gydF4y2BapgydF4y2Ba我gydF4y2Ba= (ggydF4y2Ba我gydF4y2BatgydF4y2Ba我gydF4y2Ba)是基因g之间的关联gydF4y2Ba我gydF4y2Ba(G)和GO项tgydF4y2Ba我gydF4y2Ba(去)。设BP(P)为取P中所有t为生物过程的对所得到的P的子集。类似地,定义MF(P)为分子功能,CC(P)为细胞成分。下面我们定义管道的功能。注意,根据定义,BP(P)、MF(P)和CC(P)都是一组对。因此,在下面的定义中,P要么是管道的原始输出,要么是应用BP、MF或CC的结果。gydF4y2Ba
我们定义gydF4y2Ba基因(P)gydF4y2Ba为P和覆盖的基因集gydF4y2Ba术语(P)gydF4y2Ba所涵盖的GO项的集合gydF4y2BaPgydF4y2Ba.我们定义gydF4y2Ba基因(P t)gydF4y2Ba作为与GO术语相关的一组基因gydF4y2BatgydF4y2Ba根据gydF4y2BaPgydF4y2Ba,gydF4y2Ba术语(P, g)gydF4y2Ba作为与基因相关的GO项的集合gydF4y2BaggydF4y2Ba根据gydF4y2BaPgydF4y2Ba.最后,我们表示gydF4y2Ba扫描电镜(tgydF4y2Ba我gydF4y2BatgydF4y2BajgydF4y2Ba)gydF4y2Ba为两个GO术语之间的语义相似度gydF4y2BatgydF4y2Ba我gydF4y2Ba而且gydF4y2BatgydF4y2BajgydF4y2Ba.这里的语义相似性是一种度量,用于量化GO图中两个术语的接近程度。有几种方法可以计算GO术语之间的语义相似性。在本研究中,我们使用了Wang的方法[gydF4y2Ba6gydF4y2Ba],[gydF4y2Ba24gydF4y2Ba].gydF4y2Ba
两条管道间的杰卡德系数gydF4y2Ba
杰卡德系数是衡量两个集合之间相似性的通用指标。它被定义为集合的交集的大小与集合的并集的大小之间的比率。例如,给定两条管道gydF4y2BaPgydF4y2Ba1gydF4y2Ba而且gydF4y2BaPgydF4y2Ba2gydF4y2Ba,表示gydF4y2Ba相交(PgydF4y2Ba1gydF4y2BaPgydF4y2Ba2gydF4y2Ba)gydF4y2Ba作为注释对的集合,它们都在gydF4y2BaPgydF4y2Ba1gydF4y2Ba而在gydF4y2BaPgydF4y2Ba2gydF4y2Ba,让gydF4y2Ba联盟(PgydF4y2Ba1gydF4y2BaPgydF4y2Ba2gydF4y2Ba)gydF4y2Ba中的注释对的集合gydF4y2BaPgydF4y2Ba1gydF4y2Ba或在gydF4y2BaPgydF4y2Ba2gydF4y2Ba.杰卡德系数gydF4y2BaJgydF4y2Ba管道gydF4y2Ba(PgydF4y2Ba1gydF4y2BaPgydF4y2Ba2gydF4y2Ba)gydF4y2Ba注释对的数目之间的比率是gydF4y2Ba相交(PgydF4y2Ba1gydF4y2BaPgydF4y2Ba2gydF4y2Ba)gydF4y2Ba以及注释对的数目gydF4y2Ba联盟(PgydF4y2Ba1gydF4y2BaPgydF4y2Ba2gydF4y2Ba)gydF4y2Ba.此外,我们还计算了杰卡德系数gydF4y2BaJgydF4y2Ba基因gydF4y2Ba(PgydF4y2Ba1gydF4y2BaPgydF4y2Ba2gydF4y2Ba)gydF4y2Ba在基因集之间gydF4y2Ba基因(PgydF4y2Ba1gydF4y2Ba)gydF4y2Ba而且gydF4y2Ba基因(PgydF4y2Ba2gydF4y2Ba)gydF4y2Ba测量两条管道注释相同基因的倾向。gydF4y2Ba
两个管道之间基于结构的相似性gydF4y2Ba
上面的Jaccard度量不考虑GO术语之间的功能相似性。因此,我们使用语义相似性作为定义两个管道之间基于结构的相似性的方法gydF4y2BaPgydF4y2Ba1gydF4y2Ba而且gydF4y2BaPgydF4y2Ba2gydF4y2Ba.我们从定义单个基因注释集之间的相似性开始。给定一个基因g,我们的目标是测量两者之间的语义相似度gydF4y2Ba条款(PgydF4y2Ba1gydF4y2Ba, g)gydF4y2Ba而且gydF4y2Ba条款(PgydF4y2Ba2gydF4y2Ba, g)gydF4y2Ba.作为第一步,我们定义单个GO术语之间的相似性gydF4y2BatgydF4y2Ba和一组GO项gydF4y2BaT 'gydF4y2Ba为:gydF4y2Ba
只有当T '包含gydF4y2BatgydF4y2Ba或类似的GO术语。接下来,我们用这个分数来计算之间的相似度gydF4y2Ba条款(PgydF4y2Ba1gydF4y2Ba, g)gydF4y2Ba而且gydF4y2Ba条款(PgydF4y2Ba2gydF4y2Ba, g)gydF4y2Ba使用run -max-average [gydF4y2Ba6gydF4y2Ba]:gydF4y2Ba
这个分数只有在gydF4y2Ba条款(PgydF4y2Ba1gydF4y2Ba, g)gydF4y2Ba涵盖的生物功能gydF4y2Ba条款(PgydF4y2Ba2gydF4y2Ba, g)gydF4y2Ba反之亦然。最后,整体相似性之间gydF4y2BaPgydF4y2Ba1gydF4y2Ba而且gydF4y2BaPgydF4y2Ba2gydF4y2Ba是平均基因相似性:gydF4y2Ba
基于go的精度和召回gydF4y2Ba
上述计算衡量的是管道之间的相似性。这里定义了一种测量管道精度和召回率的方法gydF4y2BaPgydF4y2Ba与金本位制相比gydF4y2BaGSgydF4y2Ba.类似于gydF4y2BaP, GSgydF4y2Ba是一组注释对吗gydF4y2Ba{gsgydF4y2Ba1gydF4y2Ba,…,ggydF4y2BakgydF4y2Ba}gydF4y2Ba每对gydF4y2BagsgydF4y2Ba我gydF4y2Ba= (ggydF4y2Ba我gydF4y2BatgydF4y2Ba我gydF4y2Ba)gydF4y2Ba基因之间有关联吗gydF4y2BaggydF4y2Ba我gydF4y2Ba(在gydF4y2BaGgydF4y2Ba)和GO术语gydF4y2BatgydF4y2Ba我gydF4y2Ba(在gydF4y2BaTgydF4y2Ba).我们首先定义单个基因的精度gydF4y2BaggydF4y2Ba.的GO-basedgydF4y2Ba精度gydF4y2Ba管道gydF4y2BaPgydF4y2Ba基因gydF4y2BaggydF4y2Ba衡量的程度gydF4y2Ba术语(P, g)gydF4y2Ba是由gydF4y2Ba术语(g, g)gydF4y2Ba:gydF4y2Ba
的gydF4y2Ba精度gydF4y2Ba的gydF4y2BaPgydF4y2Ba定义为基因的平均精度gydF4y2Ba基因(G)gydF4y2Ba:gydF4y2Ba
的gydF4y2BaGO-based回忆gydF4y2Ba管道gydF4y2BaPgydF4y2Ba基因gydF4y2BaggydF4y2Ba衡量的程度gydF4y2Ba术语(P, g)gydF4y2Ba涵盖了gydF4y2Ba术语(g, g)gydF4y2Ba:gydF4y2Ba
的gydF4y2BaP的召回gydF4y2Ba定义为基因(G)中基因的平均召回率:gydF4y2Ba
微阵列数据预处理与归一化gydF4y2Ba
我们整合了来自20多项研究的马铃薯基因表达数据,这些研究基于安捷伦JHI茄类60 k v1微阵列(ArrayExpress ID: e - mtaba -1655),该微阵列由詹姆斯·赫顿研究所使用安捷伦推荐的标准方法处理[gydF4y2Ba38gydF4y2Ba].这些研究包括326种来自以下处理的条件:中度热应激[gydF4y2Ba38gydF4y2Ba]、短期和长期生长机制[gydF4y2Ba39gydF4y2Ba],瘀伤,磷生长机制,酸性,gydF4y2BaPhytopthora 5gydF4y2Ba感染(gydF4y2Ba40gydF4y2Ba]、亚磷酸盐[gydF4y2Ba41gydF4y2Ba, baba [gydF4y2Ba14gydF4y2Ba], ABA,油菜素类固醇,SA处理。包括品种差异和块茎、茎、叶组织。gydF4y2Ba
我们使用Limma包应用分位数归一化[gydF4y2Ba42gydF4y2Ba]并使用' normexp '方法从每个点的前景强度中减去背景强度[gydF4y2Ba43gydF4y2Ba].我们的归一化表达式矩阵包含52,998个探针。为了减少统计噪声并专注于高变异基因,我们删除了样本中始终低表达值的探针和低变异的探针。探头移除阈值已按[gydF4y2Ba44gydF4y2Ba],参见附加文件gydF4y2Ba1gydF4y2Ba:方法S4查询详情。数据中还有14000个探针。这些探针被定位到12956个基因,与Tzfadia等人分析的基因数量大致相同。[gydF4y2Ba44gydF4y2Ba].gydF4y2Ba
评估预测GO过程的共表达gydF4y2Ba
给定一个与特定GO项相关的基因集U,以及一个以基因为行的基因表达矩阵X,我们首先利用它们在X中的表达谱计算U中所有基因对之间的Pearson相关性。为了评估U中的相关性是否会偶然地高于预期,我们对X中的随机基因对取样,并计算它们的相关性以得到随机相关性得分的分布。我们使用Kolmogorov-Smirnov检验来比较U的真实相关得分和随机相关得分。为了提高稳健性,我们对每个基因集U重复该过程50次,并在所有重复中使用平均p值。gydF4y2Ba
附加文件gydF4y2Ba
缩写gydF4y2Ba
- 走:gydF4y2Ba
-
基因本体论gydF4y2Ba
- PGSC:gydF4y2Ba
-
马铃薯基因组测序联盟gydF4y2Ba
- ITAG:gydF4y2Ba
-
国际番茄注释组gydF4y2Ba
- 答:gydF4y2Ba
-
蜂窝组件gydF4y2Ba
- MF:gydF4y2Ba
-
分子功能gydF4y2Ba
- 英国石油公司:gydF4y2Ba
-
生物过程gydF4y2Ba
- NGPG:gydF4y2Ba
-
每个基因的GO项数gydF4y2Ba
参考文献gydF4y2Ba
- 1.gydF4y2Ba
Birch PR, Bryan G, Fenton B, Gilroy EM, Hein I, Jones JT, Prashar A, Taylor MA, Torrance L, Toth IK:养活世界的作物8:马铃薯:全球产量增长的趋势是可持续的吗?粮食安全,2012,4(4):477-508。10.1007 / s12571 - 012 - 0220 - 1。gydF4y2Ba
- 2.gydF4y2Ba
Haverkort A, Boonekamp P, Hutten R, Jacobsen E, Lotz L, Kessel G, Visser R, Van der Vossen E:马铃薯晚疫病的社会成本和顺基因修饰持久抗性的前景。马铃薯学报,2008,51(1):47-57。10.1007 / s11540 - 008 - 9089 - y。gydF4y2Ba
- 3.gydF4y2Ba
马铃薯基因组测序联盟:块茎作物马铃薯的基因组序列和分析。自然科学,2011,475(755):189-195。10.1038 / nature10158。gydF4y2Ba
- 4.gydF4y2Ba
Zouine M, Latché A, Rousseau C, Regad F, Pech J-C, Philippot M, Bouzayen M, Delalande C, Frasse P, Schiex T:番茄基因组序列为肉质水果进化提供了见解。自然科学学报,2012,34(4):344 - 344。10.1038 / nature11119。gydF4y2Ba
- 5.gydF4y2Ba
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT, Harris MA, Hill DP, Issel-Tarver L, Kasarskis A, Lewis S, Matese JC, Richardson JE, Ringwald M, Rubin GM, Sherlock G:基因本体论:生物学统一的工具。基因本体联盟。植物学报,2000,25(1):25-29。10.1038/75556。gydF4y2Ba
- 6.gydF4y2Ba
王继泽,杜震,Payattakool R,余PS,陈cf:一种新的GO术语语义相似度度量方法。生物信息学,2007,23(10):1274-1281。10.1093 /生物信息学/ btm087。gydF4y2Ba
- 7.gydF4y2Ba
Ulitsky I, Maron-Katz A, Shavit S, Sagir D, Linhart C, Elkon R, Tanay A, Sharan R, Shiloh Y, Shamir R:扩展器:从表达微阵列到网络和函数。光子学报,2010,35(2):339 - 344。10.1038 / nprot.2009.230。gydF4y2Ba
- 8.gydF4y2Ba
Ramsak Z, Baebler S, Rotter A, Korbar M, Mozetic I, Usadel B, Gruden K: GoMapMan: MapMan本体内植物基因注释的集成、整合和可视化。中国生物医学工程,2013,42:D1167-D1175。10.1093 / nar / gkt1056。gydF4y2Ba
- 9.gydF4y2Ba
Lohse M, Nagel A, Herter T, May P, Schroda M, Zrenner R, Tohge T, Fernie AR, Stitt M, Usadel B: Mercator:用于植物序列数据基因组规模功能注释的快速简单的web服务器。植物细胞环境,2014,37(5):1250-1258。10.1111 / pce.12231。gydF4y2Ba
- 10.gydF4y2Ba
Carbon S, Ireland A, Mungall CJ, Shu S, Marshall B, Lewis S: AmiGO:在线获取本体和注释数据。生物信息学,2009,25(2):288-289。10.1093 /生物信息学/ btn615。gydF4y2Ba
- 11.gydF4y2Ba
Lamesch P, Berardini TZ, Li D, Swarbreck D, Wilks C, Sasidharan R, Muller R, Dreher K, Alexander DL, Garcia-Hernandez M, Karthikeyan AS, Lee CH, Nelson WD, Ploetz L, Singh S, Wensel A, Huala E:拟南芥信息资源(TAIR):改进基因注释和新工具。核酸决议2012,40(数据库issue): D1202-D1210。10.1093 / nar / gkr1090。gydF4y2Ba
- 12.gydF4y2Ba
提示菌VJ, Ouzounis CA, Iliopoulos I:从基因融合事件验证功能关联的计算推断的实验证据:关键调查。生物信息学报,2012,15(3):443-454。10.1093 /龙头/ bbs072。gydF4y2Ba
- 13.gydF4y2Ba
Jaramillo-Garzón JA, Gallardo-Chacón JJ, Castellanos-Domínguez CG, Perera-Lluna A:从胚胎植物蛋白质的初级结构信息预测基因本体薄术语。生物医学工程学报,2013,14(1):68-10.1186/1471-2105-14-68。gydF4y2Ba
- 14.gydF4y2Ba
Bengtsson T, Weighill D, Proux-Wera E, Levander F, Resjo S, Burra DD, Moushib LI, Hedley PE, Liljeroth E, Jacobson D, Alexandersson E, Andreasson E:利用新的功能注释方法研究baba诱导的马铃薯抗性反应的蛋白质组学和转录组学。BMC基因组学杂志,2014,15(1):315-10.1186/1471-2164-15-315。gydF4y2Ba
- 15.gydF4y2Ba
Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I, Adiconis X, Fan L, Raychowdhury R, Zeng Q:基于RNA-Seq数据的无参考基因组转录组组装。生物技术学报,2011,29(7):644-652。10.1038 / nbt.1883。gydF4y2Ba
- 16.gydF4y2Ba
李丽玲,李志强,李志强,李志强,等。真核生物基因组正正交基团的鉴定。基因组学报,2003,13(9):2178-2189。10.1101 / gr.1224503。gydF4y2Ba
- 17.gydF4y2Ba
Conesa A, Götz S, García-Gómez JM, Terol J, Talón M, Robles M: Blast2GO:功能基因组学研究中注释、可视化和分析的通用工具。生物信息学,2005,21(18):3674-3676。10.1093 /生物信息学/ bti610。gydF4y2Ba
- 18.gydF4y2Ba
Goodstein DM, Shu S, Howson R, Neupane R, Hayes RD, Fazo J, Mitros T, Dirks W, Hellsten U, Putnam N: Phytozome:绿色植物基因组学比较平台。中国生物医学工程学报,2012,40 (D1): D1178-D1186。10.1093 / nar / gkr944。gydF4y2Ba
- 19.gydF4y2Ba
Kasprzyk A: BioMart:推动生物数据管理的范式变革。数据库(牛津大学)。2011, 2011: bar049-10.1093/database/bar049。gydF4y2Ba
- 20.gydF4y2Ba
赵凯,Bartley LE:拟南芥、杨树、水稻、玉米和柳枝稷R2R3 MYB次级细胞壁调控因子的比较基因组分析。植物学报,2014,14(1):135-10.1186/1471-2229-14-135。gydF4y2Ba
- 21.gydF4y2Ba
Kim HA, Lim CJ, Kim S, chojk, Jo S- h, Baek N, Kwon S- y:甘蓝转录组分析的高通量测序和De Novo Assembly。公共科学学报,2014,9 (3):e92087-10.1371/journal.pone.0092087。gydF4y2Ba
- 22.gydF4y2Ba
Jaccard P:gydF4y2Ba阿尔卑斯山脉和侏罗地区花卉分布比较练习曲。gydF4y2BaCorbaz;1901.gydF4y2Ba
- 23.gydF4y2Ba
P:高山地区植物群的分布。1.新植物学报,1912,11(2):37-50。10.1111 / j.1469-8137.1912.tb05611.x。gydF4y2Ba
- 24.gydF4y2Ba
于刚,李峰,秦勇,薄霞,吴勇,王松:GOSemSim:一种用于度量GO术语和基因产物语义相似性的R包。生物信息学,2010,26(7):976-978。10.1093 /生物信息学/ btq064。gydF4y2Ba
- 25.gydF4y2Ba
Khatri P, Done B, Rao A, Done A, Draghici S:人类基因组注释的语义分析。生物信息学,2005,21(16):3416-3421。10.1093 /生物信息学/ bti538。gydF4y2Ba
- 26.gydF4y2Ba
Klie S, Nikoloski Z: mapman和基因本体在植物科学基因功能自动化预测中的选择。植物学报,2012,3:115-10.3389/fgene.2012.00115。gydF4y2Ba
- 27.gydF4y2Ba
Defoin-Platel M, Hindle M, Lysenko A, Powers S, Habash D, Rawlings C, Saqi M: AIGO:面向GO函数注释分析与互比较的统一框架。生物医学工程学报,2011,12(1):431-10.1186/1471-2105-12-431。gydF4y2Ba
- 28.gydF4y2Ba
权力D:评估:从精度,召回和f-测量到roc。、知情性、标记性和相关性。机械工程学报。2011,2(1):37-63。gydF4y2Ba
- 29.gydF4y2Ba
Datta S, Pihur V:应用于高维数据的自适应最优集成分类器。中国生物医学工程学报,2010,27(4):427-10.1186。gydF4y2Ba
- 30.gydF4y2Ba
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。中华分子生物学杂志,1999,15(3):403-410。10.1016 / s0022 - 2836(05) 80360 - 2。gydF4y2Ba
- 31.gydF4y2Ba
Magrane M, Consortium U: UniProt知识库:集成蛋白质数据的枢纽。数据库(牛津大学)。2011, 2011: bar009-10.1093/database/bar009。gydF4y2Ba
- 32.gydF4y2Ba
Finn RD, Clements J, Eddy SR: hmm web服务器:交互式序列相似性搜索。核酸研究,2011,39(增刊2):W29-W37。10.1093 / nar / gkr367。gydF4y2Ba
- 33.gydF4y2Ba
彭塔M, Coggill PC, Eberhardt RY, Mistry J, Tate J, Boursnell C, Pang N, Forslund K, Ceric G, Clements J: Pfam蛋白家族数据库。中国生物医学工程学报,2012,40 (D1): D290-D301。10.1093 / nar / gkr1065。gydF4y2Ba
- 34.gydF4y2Ba
Pasare SA, Ducreux LJ, Morris WL, Campbell R, Sharma SK, Roumeliotis E, Kohlen W, van der Krol S, Bramley PM, Roberts AG, Fraser PD, Taylor MA:马铃薯CCD8基因在匍杆和块茎发育中的作用。植物学报,2013,198(4):1108-1120。10.1111 / nph.12217。gydF4y2Ba
- 35.gydF4y2Ba
沙尔玛,陈晓明,陈晓明,陈晓明:马铃薯体细胞胚胎发育过程中SERK基因的克隆及分子特性研究。植物学报,2008,228(2):319-330。10.1007 / s00425 - 008 - 0739 - 8。gydF4y2Ba
- 36.gydF4y2Ba
王晓明,王晓明,王晓明,王晓明,王晓明。马铃薯开花和贮藏器官形成的基因调控研究进展。中国农业科学,2011,29(4):344 - 344。10.1038 / nature10431。gydF4y2Ba
- 37.gydF4y2Ba
Kloosterman B, Abelenda JA, Gomez Mdel M, Oortwijn M, de Boer JM, Kowitwanich K, Horvath BM, van Eck HJ, Smaczniak C, Prat S, Visser RG, Bachem CW:自然发生的等位基因多样性允许在北纬地区种植马铃薯。自然科学进展,2013,39(4):344 - 344。10.1038 / nature11912。gydF4y2Ba
- 38.gydF4y2Ba
Hancock RD, Morris WL, Ducreux LJ, Morris JA, Usman M, Verrall SR, Fuller J, Simpson CG, Zhang R, Hedley PE, Taylor MA:马铃薯(Solanum tuberosum L.)对中等高温的生理、生化和分子响应。植物细胞环境,2014,37(2):439-450。10.1111 / pce.12168。gydF4y2Ba
- 39.gydF4y2Ba
Morris WL, Hancock RD, Ducreux LJM, Morris JA, Usman M, Verrall SR, Sharma SK, Bryan G, Mcnicol JW, Hedley PE:具有不同结节表型的马铃薯基因型叶片转录组和代谢组的日长依赖重组。植物细胞环境,2014,37(6):1351-1363。10.1111 / pce.12238。gydF4y2Ba
- 40.gydF4y2Ba
Ali A, Alexandersson E, Sandin M, Resjö S, Lenman M, Hedley P, Levander F, Andreasson E:马铃薯对疫霉菌的定量蛋白质组学和转录组学响应。BMC基因组学杂志,2014,15(1):497-10.1186/1471-2164-15-497。gydF4y2Ba
- 41.gydF4y2Ba
Burra DD, Berkowitz O, Hedley PE, Morris J, Resjö S, Levander F, Liljeroth E, Andreasson E, Alexandersson E:磷酸盐诱导的茄类转录组和分泌组变化导致对疫霉菌的抗性。生物工程学报,2014,14 (1):254-10.1186/s12870-014-0254-y。gydF4y2Ba
- 42.gydF4y2Ba
Smyth GK: Limma:微阵列数据的线性模型。使用R和Bioconductor的生物信息学和计算生物学解决方案。编辑:Gentleman R, Carey V, Dudoit S, R Irizarry WH。2005,施普林格,纽约,397-420。10.1007 / 0 - 387 - 29362 - 0 - _23。gydF4y2Ba
- 43.gydF4y2Ba
Ritchie ME, Silver J, Oshlack A, Holmes M, Diyagama D, Holloway A, Smyth GK:双色微阵列背景校正方法的比较。生物信息学,2007,23(20):2700-2707。10.1093 /生物信息学/ btm412。gydF4y2Ba
- 44.gydF4y2Ba
Tzfadia O, Amar D, Bradbury LM, Wurtzel ET, Shamir R: MORPH算法:拟南芥和番茄途径中候选基因的归属排序。植物细胞。2012, 24(11): 4389-4406。10.1105 / tpc.112.104513。gydF4y2Ba
确认gydF4y2Ba
作者要感谢SURF-sara (gydF4y2Bahttps://www.surfsara.nl/gydF4y2Ba)举办“黑客马拉松”活动及提供高性能计算服务。这项工作是Allbio计划的一部分,并得到了EU FP7号拨款的部分支持;289452;KBBE.2011.3.6-02)。David Amar的部分研究经费来自Azrieli基金会和特拉维夫大学Edmond J. Safra生物信息学中心。Erik Alexandersson和Itziar Frades得到了Crafoord基金(20120533)和瑞典战略研究基金会(RB608-0006)的支持,Estelle Proux-Wera由PlantLink资助。Agnieszka Danek获得POIG.02.03.01-24-099/13赠款:“GeCONiI -上西里西亚计算科学与工程中心”的支持。我们感谢MapMan团队的帮助。我们也要感谢Ashfaq Ali, Kate Dreher和Paul Kersey的有益讨论和输入,以及Efrat Weithorn的手稿编辑帮助。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
相应的作者gydF4y2Ba
额外的信息gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称他们之间没有利益冲突。gydF4y2Ba
作者的贡献gydF4y2Ba
OT和E Alexandersson设计了研究,DA, IF, AD, TG, SKS, EPW, OT和E Alexandersson进行了研究。DA、IF和AD贡献了新的分析计算工具。PH、E Andreasson和SKS提供基因表达数据集。DA, IF, AD, TG, SKS, EPW OT和E Alexandersson分析了数据。DA, IF, RS, OT和E Alexandersson写了这篇论文。所有作者都阅读并批准了最终的手稿。gydF4y2Ba
电子辅助材料gydF4y2Ba
图S2。gydF4y2Ba
附加文件1:图S1。:ITAG pipeline similarity, ITAG gold standard validation,图S3。gydF4y2BaITAG基因表达验证gydF4y2Ba方法S1-5gydF4y2Ba.(pdf 840kb)gydF4y2Ba
12870 _2014_329_moesm19_esm.xlsxgydF4y2Ba
附加文件19:基于集成k = 2的JHI茄属60 k v1微阵列(ArrayExpress ID: E-MTAB-1655) GO注释文件。(xlsx3mb)gydF4y2Ba
作者提交的图片原始文件gydF4y2Ba
下面是作者提交的原始图片文件的链接。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。gydF4y2Ba
本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。gydF4y2Ba
如欲查看本牌照的副本,请浏览gydF4y2Bahttps://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
创作共用公共领域奉献弃权书(gydF4y2Bahttps://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本条所提供的资料,除非在资料的信用额度中另有说明。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
阿玛,D,弗雷德斯,我,达内克,A。gydF4y2Baet al。gydF4y2Ba新测序生物的功能注释管道的评估和集成:马铃薯基因组作为测试用例。gydF4y2BaBMC植物生物学gydF4y2Ba14日,gydF4y2Ba329(2014)。https://doi.org/10.1186/s12870-014-0329-9gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-014-0329-9gydF4y2Ba
关键字gydF4y2Ba
- 功能注释gydF4y2Ba
- 基因本体论gydF4y2Ba
- 基因co-expressiongydF4y2Ba
- 土豆gydF4y2Ba
- 基因组学gydF4y2Ba