摘要
背景
在过去几年中,参考几个经济和科学重要的谷物和模型植物的基因组序列变得可用。尽管这些作物的农业意义只有少量的工具,但允许用户以交互式方式检查和可视化感兴趣基因的基因组位置。
描述
我们提出chromoWIZ,这是一种网络工具,可以可视化相关基因的基因组位置,并在不同植物基因组之间比较这些数据。可以使用基因标识符、功能注释或序列同源性查询四种草(小麦,普通大麦,短穗小麦,栽培稻).利用热图可以直观地看到锚定基因在染色体上的分布。可以上传自定义基因表达测量、差异表达信息和基因到组的映射,并可用于进一步过滤。
结论
该工具主要是为育种家和植物研究人员设计的,他们对候选基因的位置和分布以及不同草物种之间的共线关系感兴趣。chromoWIZ是免费提供的,并在网上访问http://mips.helmholtz-muenchen.de/plant/chromowiz/index.jsp.jsp..
背景
自从基因组测序公布以来拟南芥蒂利亚纳2000年(1],目前已有50多个植物参考序列[2].而被子植物的平均基因组大小约为6gb [3.,测序工作主要集中在较小尺寸的基因组(< 1 Gb),作为大型和仍未测序物种的模型,或更容易获得的作物植物基因组,如水稻(栽培稻).Poo eae亚家族的谷物种类,包括面包小麦(小麦), 大麦 (Hordeum Vulgare.),而水稻是最重要的作物之一,在全基因组水平上具有高度的同源性保护[4]、[5].在作物中,六倍磅面包小麦(t . aestivum,2n = 6x = 42,AABBDD)包含最大和最复杂的基因组,大小为约17 GB [6].尽管其高经济相关性 - 世界人口消耗的热量源于面包小麦的20% - 其基因组目前没有完全组装。已经花了几年的时间来提供甚至一个染色体的参考序列(3b,[7]),其本身超过米的基因组大小几乎是3倍。最近,霰弹枪测序和流式细胞仪为包含〜124k基因模型的完整面包小麦基因组的基因注释提供了基础[6].此外,对于选定的染色体或染色体臂,已经建立了物理图谱并进行了基因锚定(例如1A [8]、[9],1bs [10.), 3 b (7]、[11.), 6 (12.])。对于大麦,已经发布了一个固定的物理地图,覆盖3.9 Gb累积地图长度[13.]、[14.],包括26k高置信基因,包括来自三种品种的霰弹枪组件。大多数霰弹枪葡萄酒已经被人口遗传学锚定。这种方法,称为popseq [15.[还用于改善物理图的锚定[13.].像面包,小麦和大麦,Brachypodium(Brachypodium distachyon)也属于禾本科科的禾本科亚科。它的基因组相对较小(~300 Mb),已被广泛用作研究其他禾草物种结构和进化的模式生物[16.].水稻是禾本科的另一重要成员,是世界上最重要的主食之一。为了成功地整合所有不同的资源,例如基因信息和基因表达测量,需要对这些作物的异构数据集进行合并。因此,需要将锚定数据集互连的工具和标准(见[17.])。组合异构数据集的方法之一是“GenomeZipper”[4].它通过利用高度保守的同位染色体,建立了没有染色体组装的植物基因的虚拟顺序。大尺寸同线区域与遗传标记集一起可以锚定大尺寸谷物的大多数基因,如大麦[14.)、黑麦(Secale cereale)[18.),山羊草属tauschii[19.].由于从共同祖先分离后,大多数基因的位置是保守的,这种方法提供了基因位置和顺序的稳健逼近[20.].
存在少量工具,使用户能够检查靶基因组中查询基因的基因组位置。对于大麦,可以使用ipk viroblast映射查询序列(http://webblast.ipk-gatersleben.de/)或barleymap(http://floresta.eead.csic.es/barleymap/).然而,据我们所知,目前还没有网络工具可以覆盖多个基因组,并允许计算和可视化沿着染色体的基因密度。当需要定位几十个基因时,例如分析一个数量性状位点(QTL),这尤其重要。以转录组为导向的研究可能会揭示一套候选基因和相应的基因组位置,以支持去除假阳性候选基因和确定QTL的遗传或物理位置。列出的工具都没有提供基于功能注释或表达式数据集成的查询。作为GenomeZipper的一部分,我们之前已经实现了一个模块'chromoWIZ”引入了易于检测又一个未组装的基因组的同时性区域和几个测序和组装基因组Brachypodium[16.)、大米(21.]和高粱(高粱二色的)[22.].在这里,我们描述的是基于网络的版本chromoWIZ以及新功能。最初,chromoWIZ作为GenomeZipper包装的一部分,它被限制在本地使用,并允许对一个染色体或染色体臂的基因或猎枪组合体进行映射,以对抗参考基因组Brachypodium,大米和高粱。为了找到感兴趣基因的基因组位置,在最新的网络版中,可以使用功能注释和序列同源性来找到相应基因组中的相应区域。对成组或成簇的基因chromoWIZ现在用群体的方式来形象化物理位置。在最新版本中,chromoWIZ集成了的锚定结果国际大麦基因组序列联盟(IBSC [14.)和国际小麦基因组测序联盟(IWGSC) [6])并允许将序列与基因组进行比较Brachypodium和大米。该工具主要是为育种家和植物研究人员设计的,他们对候选基因的位置和分布以及不同草物种之间的共线关系感兴趣。为了说明的特征chromoWIZ并解释基本工作流,我们呈现不同的用例。可以访问应用程序网站:http://mips.helmholtz-muenchen.de/plant/chromowiz/index.jsp.jsp.没有任何限制。
建设和内容
chromoWIZ在PGSB网站的网络服务器上运行[23.].该工具的后端是用编程语言Python实现的。前端使用原生HTML和Javascript进行数据可视化和导航。测绘信息和基因信息都是从官方发布的Brachypodium,米饭,大麦和面包小麦基因组[6]、[14.]、[16.]、[21.].为了Brachypodium蛋白及其编码序列,以及功能注释信息均来源于PGSB数据库[23.使用基因模型的1.2版本。对于大麦,我们整合了提供基因锚定物理图的数据集[14.],被托管在ftp://ftpmips.helmholtz-muenchen.de/plants/barley/public_data.对于面包小麦,2.2版本的基因模型(ftp://ftpmips.helmholtz-muenchen.de/plants/wheat/IWGSC)都包括在内。该MSU7注释已集成水稻[21.].有关当前使用的数据集和相应更新的详细信息,请参见chromoWIZ网站。
公用事业
Chromowiz的应用
chromoWIZ允许在功能基因注释、序列同源性或基因列表的基础上可视地定位沿着染色体的锚定基因。到目前为止,这个网络工具包括面包小麦(t . aestivum), 大麦 (h . vulgare)和近亲,但小得多Brachypodium(B. Distachyon.)及米(o .漂白亚麻纤维卷)的基因组。锚定基因沿着染色体聚集在不重叠的基因组或遗传间隔,称为箱。在Brachypodium和水稻,每箱代表非重叠的染色体序列的一个兆碱基(MB)。对于大麦为10 Mb以及用于面包小麦5厘摩(CM)的间隔被使用。作为热映射到使沿着整个染色体的直观视图箱被可视化。在大麦的基因组的位置相对向其中串成虚拟染色体锚定的物理BAC重叠群被突出显示。内所有基因chromoWIZ连接到外部数据库,提供关于基因模型的额外信息(例如,面包小麦EnsemblePlantshttp://plants.ensembl.org/Triticum_aestivum/Info/Index).箱内标记基因的序列可以以快速格式下载。为了获得感兴趣基因的基因组位置,称为“标记基因”,chromoWIZ提供几种搜索方法(表1):通过序列同源性,可以使用核苷酸或蛋白质BLAST搜索将一组查询序列映射到注释的基因模型上,这需要一个预定义的e值和序列身份。或者,如果已知,可以直接提供物种特定的基因标识符,而不是序列。查询基因家族(例如共享特定基因本体论(GO)术语或PFAM域的基因[24.]、[25.,其中包含了一种基于注释的方法。查询基因的分布通过热图可视化,热图描述了查询匹配基因相对于染色体总数的相对分布。此外,由于锚定基因的数量在不同的容器之间是不同的,因此显示了总体的基因分布。为了了解某些染色体(−臂)是否对标记的基因富集,提供了富集分析。标记基因的过度代表性的意义是通过单侧Fisher精确检验和P值的Bonferroni调整来评估的。此外,可以包括标记的基因组,例如,基因被聚类或共同表达,或基于序列相似性被分组在一起,以允许群组可视化和分析。数据管理器是chromoWIZ这样就可以上传各种特定于用户的数据集,并在集成到chromoWIZ搜索界面。这些数据随后仅对各自的用户可见,并在被自动从服务器删除之前24小时可用。基因表达是判断候选基因相关性的重要因素。在chromoWIZ,通过使用数据管理器,用户可以选择上传他们感兴趣的基因的表达式值。类似于表达式数据,可以提供关于微分表达式的信息。有了表达数据在手,功能信息可以与基因组位置相结合。
下面的用例说明了的不同方面chromoWIZ.第一个用例描述了如何使用基因标识符、基于序列的搜索或功能注释将候选基因映射到参考基因组序列。第二个用例说明了如何根据基因的表达或包含关于差异表达的信息来过滤基因列表。在第三个用例中,我们将展示如何操作chromoWIZ允许突出面包小麦之间的同时性区域Brachypodium或大麦。在第四个用例中,我们使用已公布的表达数据来说明基因到组的信息如何有助于完善一个抗性QTL的基因组位置。这是通过将数据从古老的参考序列转移到最近的参考序列来实现的。第五个用例最后给出了一个示例chromoWIZ可用于比较基因组分析。
用例1:使用标识符、序列相似性或注释查找基因
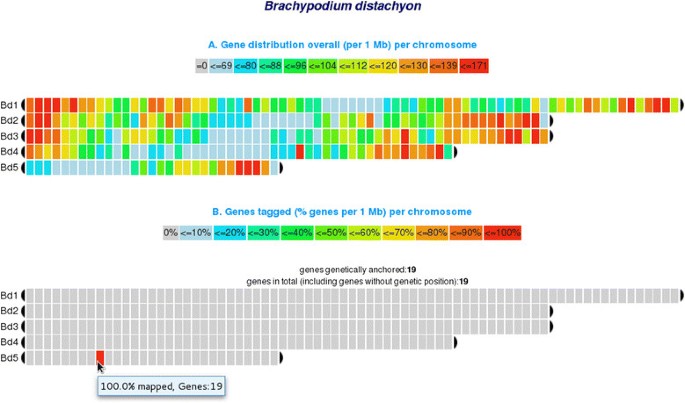
其中一个很基本功能的chromoWIZ正在寻找和它们的标识符可视化的基因。给定一组的物种特异性基因标识它们的基因组位置可以高亮显示。在情况下,没有标识符可用,另一种方法是提供一种用于在相应的基因序列信息。为了说明这个功能,我们使用下面的例子:从19个基因标识的列表Brachypodium,从一个特定的基因组区域中预先选择,提供给搜索界面(基因标识符在附加文件中给出1).chromoWIZ提供了两个输出:首先是一个热图,它描述了每个箱子中沿着染色体的所有锚定基因的数量(图1A),其次,只显示满足查询条件的锚定基因(标记基因)的热图(图)1B).对于给定的例子,5号染色体上对应的bin (bin9, 9-10 Mb)被突出显示。为了说明基于序列的搜索,我们首先从这个箱子中提取基因序列,通过使用FASTA输出功能chromoWIZ.然后将这组序列提供给搜索界面,以执行基于同源性的搜索。通过仅可视化匹配以下的e值10E-5,序列身份100%,并要求最佳双向匹配(旗帜' BBH '必须设置),我们再次检索包含基因的bin。
除了基因标识符和基于同源性的搜索,chromoWIZ还提供了基因注释功能的搜索。用户可能对某个特定的基因家族感兴趣,并想分析该家族的成员与其他基因组相比是否增加了或减少了拷贝数。分析复制数量差异的一种方法是根据蛋白质家族比较基因数量(PFAM [25.)或基因本体论(GO [24.])条款和chromoWIZ包括来自这些源的注释信息。在本例中,我们旨在可视化所有在基因本体论(GO)术语GO:0043565(序列特异性DNA结合)下注释的基因,例如包括转录因子。在Brachypodium在面包中,我们找到了349个匹配基因,在面包中,我们找到了421个(732个包括非锚定基因)基因,在大麦中我们找到了225个(340个)基因。
用例2:过滤差异表达基因和表达约束的使用
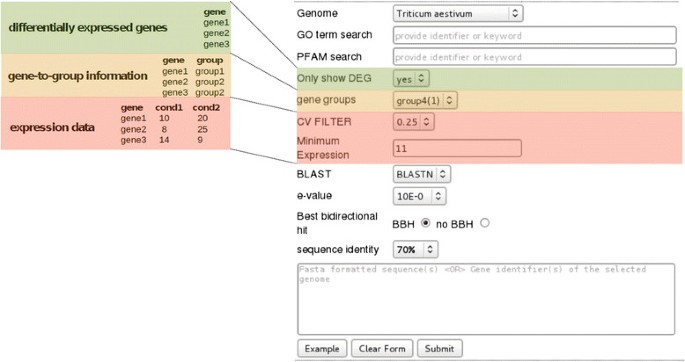
RNA-seq数据通常用于分析全基因组水平上的基因表达。它可以通过Cufflinks等分析管道有效地进行处理[26.]或HTSeq [27.].根据他们的表达模式发现的候选基因后,它往往是感兴趣的探索各自的基因组位置。chromoWIZ提供将表达式数据与位置信息结合的特性:(我)可以提供基因到群体的信息。(ii)可以包括差异表达基因列表,并且(3)可以整合所有基因的表达数据。数字2显示数据管理器输入和入口点扩展查询功能,一旦被列入数据集,其可用。基因对组信息,其中第一列包含基因标识符和所述第二列定义的组的输入文件中提供。差异表达的基因(DEGS)经由包含该基因的标识符的输入文件中提供。也可以在一个文件中,其中,列表示所关注的条件来提供表达信息。有关文件格式的细节上给定的chromoWIZ帮助页面。当包含差异表达基因信息时,用户可以指定是否只查询差异表达基因。如果包含表达信息,基因可以通过两种标准进行过滤:一种是“最小表达”标准,即至少在一种条件下表达必须超过给定的阈值。另一种方法是,在不同条件下寻找表达变异的基因,用户可以设置简历'(变异系数,由样本标准差除以样本均值得到)过滤器,只保留所需的最小基因简历价值。
为了说明这一点,我们提取了692份大麦转录本,这些转录本在两种西藏野生大麦基因型对低钾处理的反应中有差异表达[28.].[的表S3所示的转录本序列28.](http://www.plosone.org/article/fetchsinglerepresentation.action \ insuri=info:doi/10.1371/journal.pone.0100567.s009.使用BLASTN(序列同一性大于95%,E-10,BBH标准)的血管锚定的大麦基因模型映射.450个匹配基因被编译成差异表达基因列表(附加文件)2),并使用数据管理器上传。在大麦中寻找锚定差异表达基因时,我们获得了分散在不同染色体上的286个位点。
使用案例3:在草地中共享的发音相同步区域
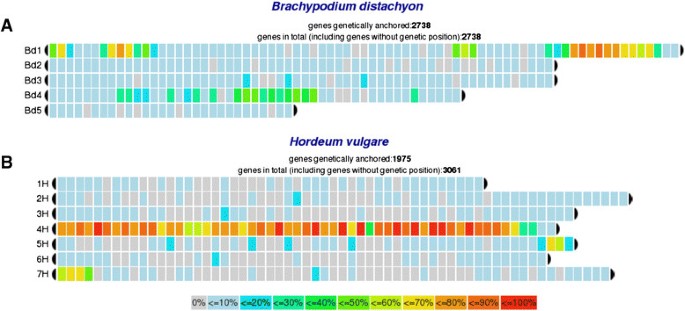
chromoWIZ已被反复用于定义和精炼相关参考基因组之间的同位区域[29.]、[30.].为了说明,我们用面包小麦4A染色体的基因模型[6进行序列同源性搜索Brachypodium和大麦基因。在4A染色体上共注释了4830个基因,提取了相应的序列,利用BLASTN对两个基因组进行比对(序列一致性至少为70%,e值为10E-5,最佳双向命中)。我们找到了匹配的1号和4号染色体Brachypodium以及一个约3mb基因组区域的重排,该区域从染色体1的短臂转移到长臂(图)3.此外,在4号染色体中,着丝粒和着丝粒周围附近区域被标记。将面包小麦染色体4A与大麦进行比较,除4H染色体上同源性较大外,在5H和7H染色体上还发现了同位区,基因组区分别为40mb(图)3.b)。这些发现与面包小麦4A染色体的记载的染色体重排是一致的[31].
用例4:为标记基因提供群集信息
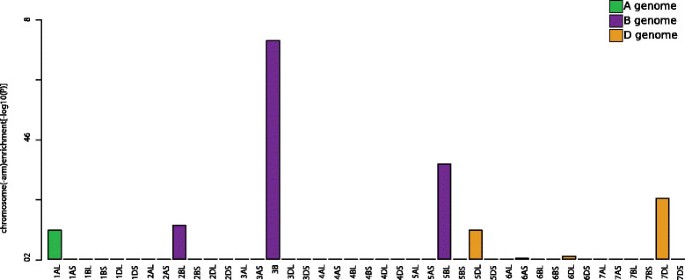
将全基因组表达数据聚类成有意义的子集已成为许多转录组导向研究的标准程序。有几种方法可以实现这样的数据分区,例如通过层次聚类、k-means聚类或基于网络的方法。chromoWIZ提供支持群体的分析,因为它允许上传基因到群体的信息。这个用例的示例数据来源于一个共表达网络研究,评估真菌病原体对不同面包小麦品种的影响[32].本研究中的五种面包小麦线的特征在于存在或不存在特定的定量性状基因座(QTL),其赋予不同的抗性水平。该数据已被用于推断使用加权相关网络分析方法(WGCNA,[33])。WGCNA可以通过推断一个基于相关性的加权基因网络来发现高度连接的基因簇,也就是所谓的网络模块。在将面包小麦转录组数据映射到454.基于整个基因组组件的测序[34然后用袖扣量化这个表达[26.],我们观察到8个不同的表达模式,共包含3273个基因。其中一个模块特别有趣,因为相关的基因表达描述了对真菌病原体的明显反应。相应的核苷酸序列在附加文件中给出3..使用chromoWIZ这些转录本与面包小麦基因组调查序列进行了比对[6通过要求最佳双向匹配和序列同一性至少为95%。发现染色体(arms)3b,5bl和7dl的显着富集(图4).这是为了支持实验设置作为主要的一个镰刀菌素头部枯萎电阻QTLS(Fhb1),并位于3B染色体的短臂上[35].
用例5:用chromoWIZ比较基因组学分析udp -酰基转移酶
chromoWIZ可以用来检测同源基因及其在四个谷物和模型基因组中的位置,使用实现的BLAST搜索。为了说明这一点,我们搜索Brachypodium水稻、大麦和面包小麦的udp -糖基转移酶(UGT)同源基因。的BrachypodiumUGT基因家族包含五个成员,其中几个编码的能力,以灭活霉菌毒素脱氧雪腐镰刀菌醇(附加文件4)[36].脱氧雪腐镰刀菌醇是一种有效的蛋白质生物合成抑制剂镰刀菌素graminearum,也是小麦和大麦的病原体[37].这种转移酶的存在/活性可以确认高电阻。然而,它们的识别遗体也是具有挑战性的,由于UGT超家族的庞大规模,该方法包括在178个成员Brachypodium可能是几百个面包小麦[36].chromoWIZ把这六个基因定位到五号染色体的第三和第四箱Brachypodium.为了找到推测的同源基因,我们提取了序列,并将它们映射到水稻、面包小麦和大麦上。在大麦中,以70%的同源性和10E-5的e值作为搜索标准,找到了与2H(3)和5H(1)染色体的匹配。此外,还发现了一种与基因上尚未固定的基因相匹配的基因。在水稻中,第4(8)号染色体和第9(1)号染色体上发现了匹配,证实了之前的发现[36].在面包小麦中,匹配2A(1)、2B(1)、2D(1)和5A(1)表明连锁群2上可能存在同源基因簇,但大多数基因(13)尚未获得任何遗传位置。在3B染色体中未观察到含有Fhb1轨迹(35],它以前被证明能控制较高的灭活毒素的能力[38].
讨论
chromoWIZ允许搜索候选基因和可视化他们的选择沿草基因组染色体密度和本地化。基因可以通过使用若干选项,例如被搜索通过基因标识,由功能注释,通过序列同源性搜索或通过基因对组映射。该工具以灵活的方式实施,以确保新的基因组或现有基因组的更新,可以很容易地进行。提供出口特征和如果提供基因表达数据或聚类信息扩展功能被激活。
chromoWIZ能够整合基于表达的信息来筛选候选基因
虽然有几种工具提供了关于植物基因组中的同时性关系的信息,映射和可视化能力[39]、[40[],缺乏对遗传和物理锚定的基因进行查询和交互式检查的工具。其中一个主要优势chromoWIZ超过其他工具,如Barleymap(http://floresta.eead.csic.es/barleymap/)或IPK病毒原体(http://webblast.ipk-gatersleben.de/barley/)可以将表达数据包括以通过若干标准滤波,从而选择最相关的基因。另外,可以包括诸如共表达基因,所选基因家族和/或差异表达基因的聚类信息和基因对组映射,并独立地分析。可以通过使用数据管理器作为内在部分来导入不同的数据集chromoWIZWeb应用程序。上传数据后,“附加筛选”和“入口”页面上会显示“搜索选项”(表1和图2).
chromoWIZ能够将以前的结果转移到当前的参考序列
chromoWIZ允许将基因锚定信息与注释的基因信息连接起来,并提供对候选基因及其定位及其邻近基因的访问。随着积极进行的项目和面包、小麦和大麦参考序列的相应更新,需要将数据映射到一个共同的参考序列,以便将以前的结果与当前的结果进行比较。我们通过使用一个特定的基因共表达模块演示了这种方法,该模块包含面包小麦基因对真菌病原体的主要反应[32].如用例4所示chromoWIZ容许转移先前的分析[32]通过绘制较早的面包小麦基因组草图而获得更新的资源[34]到更近的染色体臂分类散弹枪contigs [6].
Chromowiz可以在未完成的基因组内检测更大的同期块
对于(新的)草基因组,chromoWIZ可以用来检测和分析同序区域Brachypodium大米、大麦和面包用小麦。在案例3中,面包小麦染色体4A的注释基因模型被用来检测同线区域,与大麦和Brachypodium(图3.).这条染色体特别有趣,因为在大多数情况下大麦和小麦染色体共线[4].在大麦染色体5H和7H上也出现了这一特定的同线区域[31].此外,当ARM排序的染色体数据集可用于新测序但尚未组装的基因组时,chromoWIZ可以帮助将基因分配到大麦、大米、面包、小麦和Brachypodium.因此,它提供了这些植物的基因组结构的第一眼,特别是揭示重排和渐渗和分析更复杂的嵌套共线结构。
结论
chromoWIZ提供了一个有价值的和用户友好的界面来访问锚定基因的重要农作物和模型的基因组。通过使用不同的查询选项,可以灵活地缩小的利息和/或候选基因区域。随着未来更新,计划包括更多种类和之前延长了功能范围,允许在不断发展的庞大而复杂的作物植物基因组中的互动和整合搜索。
可用性和要求
chromoWIZ是免费的,没有任何限制,在http://mips.helmholtz-muenchen.de/plant/chromowiz/index.jsp.jsp..
许可:不是必需的。
对非学者使用的任何限制:没有。
可获得的支持数据
支持本文结果的数据集包含在文章(及其附加文件)中。
附加文件
缩写
- BBH:
-
最好的双向热门
- 走:
-
基因本体论
- IBSC:
-
国际大麦基因组测序联盟
- IWGSC:
-
国际小麦基因组测序联盟
- POPSEQ:
-
通过填充测序锚定和排序NGS集合
- QTL:
-
数量性状位点
- UGT:,
-
UDP依赖性糖基转移酶
- WGCNA:
-
加权相关网络分析
参考文献
- 1。
开花植物拟南芥基因组序列分析。自然。2000,408(6814):796-815。10.1038 / 35048692。
- 2。
Michael TP,Jackson S:前50种植物基因组。植物基因组2013,6(2)。。,[https://www.crops.org/publications/tpg/articles/6/2/plantgenome2013.03.0001in]
- 3.
Morrell Pl,Buckler Es,Ross-Ibarra J:作物基因组学:进步和应用。NAT Rev Genet。2011,13(2):85-96。
- 4.
Mayer KF,Marter M,Hedley Pe,Simkova H,刘H,莫里斯JA,Steuernagel B,Toudien S,Roessner S,Gundlach H,Kubalakova M,Suchancova P,Murat F,Felder M,Nussbaumer T,Graner A,Salse J,endo t,sakai h,tanaka t,Itoh T,Sato K,Platzer M,Matsumoto T,Scholz U,Dolezel J,Waugh R,Stein N:通过染色体和比较基因组学解锁大麦基因组。植物细胞。2011,23(4):1249-1263。10.1105 / TPC.110.082537。
- 5.
Bolot S, Abrouk M, Masood-Quraishi U, Stein N, Messing J, Feuillet C, Salse J:谷物基因组的“内圈”。植物学报,2009,12(2):119-125。10.1016 / j.pbi.2008.10.011。
- 6.
一种基于染色体的六倍体面包小麦(Triticum aestivum)基因组的序列。科学。2014,345(6194):1251788-10.1126 / Science.1251788。
- 7.
年代,Choulet F,阿尔贝蒂,赛尔Glover N, V, "达J, Pingault L, Sourdille P, Couloux, Paux E, Leroy P, Mangenot年代,Guilhot N, Le Gouis J Balfourier F,塞德cedreic Alaux M, Jamilloux V,也不会J,杜兰C, Bellec, Gaspin C,回历2月J, Dolezel J,罗杰斯J, Vandepoele K, Aury JM,梅耶尔K, berg H, Quesneville H, Wincker P,等:面包小麦3B染色体的结构和功能划分。中国科学(d辑):地球科学(d辑)。
- 8.
布林J,柳条T, Shatalina M,弗仑克尔Z,贝尔坦公司我,菲利普·R, Spielmeyer W, Simkova H,回历2月J, Cattonaro F, Scalabrin年代,马尼F,魏特琳年代,berg H, C国际小麦基因组测序,Paux E, Fahima T, Dolezel J,各地,Feuillet C,凯勒B:小麦染色体的短臂上的物理图谱1。公共科学图书馆。2013,8 (11):e80272-10.1371/journal.pone.0080272。
- 9.
卢卡斯SJ Akpinar英航,坎塔尔M,温斯坦Z, Aydinoglu F,回历2月J, Simkova H,弗仑克尔Z,各地,马尼F, F Cattonaro,魏特琳年代,Bellec, berg H, Dolezel J, Budak H:物理映射结合syntenic分析描述的基因空间1小麦染色体的长臂。公共科学图书馆。2013,8 (4):e59542-10.1371/journal. pon.0059542。
- 10。
Raats D,Frenkel Z,Krugman T,Dodek I,Sela H,Simkova H,Magni F,Cattonaro F,Vautrin S,Berges H,柳条T,Keller B,Leroy P,Philippe R,Paux E,Dolezel J,Feuillet C,Korol A,Fahima T:小麦染色体1BS的物理地图为其基因空间组织和进化提供了见解。基因组Biol。2013,14(12):R138-10.1186 / GB-2013-14-12-R138。
- 11.
Paux E, Sourdille P,泥火山J, Saintenac C, Choulet F, Leroy P,各地,旧事的M, Kianian年代,Spielmeyer W, Lagudah E,萨默斯D, Kilian,塞德cedreic Alaux M,魏特琳年代,berg H, Eversole K, appel R,回历2月J, Simkova H, Dolezel J,伯纳德·M Feuillet C:物理地图1-gigabase面包小麦染色体3 b。中国科学(d辑:地球科学),2008,32(6):591 - 598。10.1126 / science.1161847。
- 12.
Poursarebani N, Nussbaumer T, Simkova H, Safar J, Witsenboer H, van Oeveren J, Dolezel J, Mayer KF, Stein N, Schnurbusch T:全基因组分析和散列测序提供了一个锚定的,基因修饰的,面包小麦染色体6A的物理图谱组装。植物学报,2014,79(2):334-347。10.1111 / tpj.12550。
- 13.
Ariyadasa R,Mascher M,Nussbaumer T,Schulte D,Frenkel Z,Poursarebani N,周R,Steuernagel B,Gundlach H,Tudien S,Felder M,Platzer M,Himmelbach A,Schmutzer T,Hedley Pe,Muehlbauer GJ,Scholz U,Korol A,Mayer Kf,Waugh R,Langridge P,Graner A,Stein N:大麦的序列就绪物理图,遗传地锚定了200万单核苷酸多态性。植物理性。2014,164(1):412-423。10.1104 / pp.113.228213。
- 14.
Mayer KF, Waugh R, Brown JW, Schulman A, Langridge P, Platzer M, Fincher GB, Muehlbauer GJ, Sato K, Close TJ, Wise RP, Stein N:大麦基因组物理、遗传和功能序列组装。自然科学进展。2012,421(4):421 - 427。
- 15.
Mascher M,Muehlbauer GJ,Rokhsar DS,Chapman J,Schmutz J,Barry K,Munoz-Amatriain M,Close TJ,Wise Rp,Schulman啊,Himmelbach A,Mayer Kf,Scholz U,Poland Ja,Stein N,Waugh R:堆积和排序NGS Contig组件(POPSEQ)。工厂J. 2013,76(4):718-727。10.1111 / TPJ.12319。
- 16.
模型禾草的基因组测序与分析。自然。2010,463(7282):763-768。10.1038 / nature08747。
- 17.
Spannagl M, Martis MM, Pfeifer M, Nussbaumer T, Mayer K:分析复杂小麦科基因组的概念和策略。植物学报。2013,9(1):35-10.1186/1746-4811-9-35。
- 18.
Martis MM, Zhou R, Haseneyer G, Schmutzer T, Vrana J, Kubalakova M, Konig S, Kugler KG, Scholz U, Hackauf B, Korzun V, Schon CC, Dolezel J, Bauer E, Mayer KF, Stein N:黑麦基因组的网状进化。植物学报,2013,25(10):3685-3698。10.1105 / tpc.113.114553。
- 19.
李贾J,赵年代,香港的X, Y,赵G,他W, appel R, Pfeifer M,道Y,张X, Jing R,张C,马Y,高,高C, Spannagl M,梅耶尔KF,李D,锅年代,郑F,胡问,夏X,李J,梁问陈J,柳条T,郭台铭C,旷H, G,罗Y, et al:山羊草属tauschii草案基因组序列揭示了基因小麦改编的曲目。自然资源学报。2013,496(4):91-95。10.1038 / nature12028。
- 20。
Poursarebani N, Ariyadasa R, Zhou R, Schulte D, Steuernagel B, Martis MM, Graner A, Schweizer P, Scholz U, Mayer K, Stein N:大麦基因组物理图的保守共体锚定。基因组学杂志。2013,13(3):339-350。10.1007 / s10142 - 013 - 0327 - 2。
- 21。
基于地图的水稻基因组序列。自然。2005,436(7052):793-800。10.1038 / Nature03895。
- 22。
帕特森啊,鲍尔斯我,Bruggmann R, Dubchak我Grimwood J, Gundlach H,哈贝雷尔G, Hellsten U, Mitros T, Poliakov,污物J, Spannagl M,唐H,王X,柳条T, Bharti AK,查普曼J, Feltus FA, Gowik U,眼镜IV,里昂E,马赫CA, Martis M, Narechania, Otillar RP,彭宁BW, Salamov AA,王Y,张L, Carpita数控,等:高粱双色基因组与禾本科植物的多样性。自然。2009,457(729):551-556。10.1038 / nature07723。
- 23。
Nussbaumer T, Martis MM, Roessner SK, Pfeifer M, Bader KC, Sharma S, Gundlach H, Spannagl M: MIPS PlantsDB:比较植物基因组研究数据库框架。核酸学报2013,41(数据库版):D1144-D1151。10.1093 / nar / gks1153。
- 24。
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT, Harris MA, Hill DP, Issel-Tarver L, Kasarskis A, Lewis S, Matese JC, Richardson JE, Ringwald M, Rubin GM, Sherlock G:基因本论:生物学统一的工具。基因工程学报,2000,25(1):25-29。
- 25.
Finn RD, Bateman A, Clements J, Coggill P, Eberhardt RY, Eddy SR, Heger A, Hetherington K, Holm L, Mistry J, Sonnhammer EL, Tate J, Punta M: Pfam:蛋白家族数据库。核酸学报2014,42(数据库版):D222-D230。10.1093 / nar / gkt1223。
- 26.
Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR, Pimentel H, Salzberg SL, Rinn JL, Pachter L: TopHat和Cufflinks RNA-seq实验的差异基因和转录表达分析。光子学报。2012,37(3):562-578。10.1038 / nprot.2012.016。
- 27.
Anders S,Pyl Pt,Huber W:Htseq-A Python框架,用于使用高吞吐量排序数据。生物信息学2014年。。,http://www.biorxiv.org/content/biorxiv/early/2014/02/20/002824.full.pdf]
- 28.
Zeng J, He X, Wu D, Zhu B, Cai S, Nadira UA, Jabeen Z, Zhang G:两种西藏野生大麦基因型对低钾胁迫响应的转录组分析。公共科学图书馆。2014,9 (6):e100567-10.1371/journal. pon.0100567。
- 29.
Kopecky D, Martis M, Cihalikova J, Hribova E, Vrana J, Bartos J, Kopecka J, Cattonaro F,获得stoc年代,诺瓦克P,诺伊曼P吗咖能够J, Simkova H,瑞士思德利公司B, Asp T, Baird JH, Navratil P, Karafiatova M, Kubalakova M,回历2月J, K Mayer, Dolezel J:流排序和测序草地羊茅染色体4 F。植物营养与肥料学报。2013,27(3):423 - 427。10.1104 / pp.113.224105。
- 30。
Pfeifer M, Martis M, Asp T, Mayer KF, Lubberstedt T, Byrne S, Frei U, Studer B:多年生黑麦草GenomeZipper:有针对性地利用基因组资源进行比较草基因组学研究。植物营养与肥料学报。2013,32(2):531 - 534。10.1104 / pp.112.207282。
- 31。
Hernandez P,Martis M,Dorado G,Pfeifer M,Galvez S,Schaaf S,Jouve N,Simkova H,Valarik M,Dolezel J,Mayer Kf:小麦染色体4A流量排序臂的下一代测序和同步整合染色体结构和基因含量。工厂J. 2012,69(3):377-386。10.1111 / J.1365-313x.2011.04808.x。
- 32。
关键词:小麦,镰刀菌病,共表达网络,数量性状,基因座依赖中国生物医学工程杂志。2013,14:728-10.1186/1471-2164-14-728。
- 33。
Langfelder P, Horvath:加权相关网络分析的R包。生物信息学。2008,9:559-10.1186/1471-2105-9-559。
- 34。
Brenchley R, Spannagl M, Pfeifer M,巴克GL,爱与R,艾伦,麦肯齐N,克雷默M, Kerhornou,赤身裸体D,凯,韦特D,技巧,班克罗夫特,顾Y,霍N,罗MC, Sehgal年代,吉尔B, Kianian年代,安德森O,凯西P,德沃夏克J, McCombie WR,大厅,梅耶尔KF,爱德华兹KJ,贝文MW,大厅N:使用全基因组霰弹枪测序分析面包小麦基因组。自然科学进展。2012,421(4):427 - 434。10.1038 / nature11650。
- 35。
关键词:小麦,枯萎病,抗枯萎病基因,Fhb1,基因定位acta photonica sinica, 2006, 40(8): 1465-1472。10.1007 / s00122 - 006 - 0249 - 7。
- 36。
Schweiger W, Pasquet JC, Nussbaumer T, Paris MP, Wiesenberger G, Macadre C, Ametz C, Berthiller F, Lemmens M, Saindrenan P, Mewes HW, Mayer KF, Dufresne M, Adam G: Brachypodium二achyon udp -糖基转移酶两簇编码脱氧雪腐镰刀菌醇解毒基因的功能特征。植物营养与肥料学报,2013,26(7):781-792。10.1094 / mpmi - 08 - 12 - 0205 - r。
- 37.
德斯贾丁斯AE:镰刀菌素真菌毒素:化学、遗传学和生物学。2006年,APS出版社,ᅟ
- 38.
Lemmens男,肖尔茨U,Berthiller楼Dall'Asta C,Koutnik A,SCHUHMACHER R,亚当G,Buerstmayr H,Mesterhazy A,Krska R,Ruckenbauer,P:解毒霉菌毒素呕吐毒素的能力与主要数量性状基因座为共定位赤霉病小麦白叶枯病。Mol植物微生物相互作用。2005年,18(12):1318至1324年。10.1094 / MPMI-18-1318。
- 39.
陈志强,陈志强,陈志强:一种基于网络的多基因组同步数据查看器。生物信息学。2012,13:190-10.1186/1471-2105-13-190。
- 40。
Soderlund C,Bomhoff M,Nelson WM:Symap V3.4:具有应用于植物基因组的交钥匙同轴系统。核酸RES。2011,39(10):E68-10.1093 / NAR / GKR123。
确认
我们衷心由德意志研究联合会(DFG)SFB 924 KFXM和奥地利科学基金(FWF)专项研究项目F37(F3705,F3711)确认资金。
作者信息
隶属关系
通讯作者
附加信息
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
作者的贡献
HG,TN和KFXM启动该软件的第一个版本。TN,KGK和KCB实现的软件。KGK,TN,WS,NP,MS,MP和KFXM起草并设计了用例。TN,KGK,WS和KFXM起草和撰写文章。所有作者批准稿件的最终版本。
电子辅料
用例1的基因列表。
附件1:19倍的基因名单从特定基因组彬拍摄Brachypodium并用于演示的基本功能chromoWIZ.(CSV 411字节)
用例2大麦低钾反应基因。
附加文件2:从低钾下的藏野生大麦基因型的研究中匹配成绩单的大麦基因列表[28.用于集成到数据管理器中。(CSV 6 KB)
用于用例4的真菌病原体应答基因列表。
附加文件3:基因的列表聚集在一起镰刀菌素graminearum响应式网络模块载于[32].(CSV 930 KB)
UDP-糖基转移酶如用案例5所报告的同源物。
附加文件4:Brachypodiumudp -糖基转移酶家族的基因及其与水稻、大麦和面包小麦的同源性。(XLS 42 KB)
作者为图像提交的原始文件
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。
本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。
如欲查阅本许可证副本,请浏览https://creativecommons.org/licenses/by/4.0/.
创作共用及公共领域专用豁免书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
NUSSBAUMER,T.,库格勒,K.G.,施魏格尔,W.et al。Chromowiz:从谷物和模型基因组查询和可视化染色体锚定基因的网络工具。BMC植物杂志14,348(2014)。https://doi.org/10.1186/s12870-014-0348-6
已收到:
接受:
发表:
关键字
- 谷物
- 面包小麦
- 大麦
- Brachypodium
- 大米
- 比较基因组学