抽象的
背景
GRAS蛋白属于植物转录因子家族,这些家庭参与植物中的多种作用。虽然已经报道了以前对该蛋白质家族进行了研究拟南芥,大米,大白菜和其他物种,在不同物种的比较基因组学的基础上调查扩张模式和进化率仍然不足。
结果
共鉴定了总共289种GRAS基因拟南芥,b . distachyon,米饭,大豆,S. Moellendorffii.,P. Paten.并根据其外显子-内含子模式和结构基序的相似性将其分为7个亚家族。除一簇水稻外,在组II中发现了所有的串接复制基因,表明串接复制极大地促进了组II的扩展。此外,片段重复主要存在于大豆基因组中,而在其他植物中没有单一的扩增模式,表明这5种植物的GRAS基因可能具有更复杂的进化机制。有趣的是,正选择的分支-位点模型分析显示,前景分支I和v下有大量位点正选择。这些结果强烈表明,这些群体正选择压力更高。同时,位点特异性模型显示GRAS基因在P. Paten.s. DIVERGE v2.0检测关键氨基酸位点,结果显示,进化速率的转移主要归因于两组GRAS基因之间的功能差异。此外,结果还表明GRAS复制基因在进化过程中存在表达差异。总之,以上结果为进一步对GRAS基因超家族进行功能解剖奠定了坚实的基础。
结论
在这项工作中,预测了GRAS基因家族在6个物种中的差异表达、进化速率和扩展模式。特别是串联重复事件在第二组扩展中发挥了重要作用。这些结果有助于进一步的功能分析和GRAS基因超家族的分子进化。
背景
基因表达的转录调节是植物中最重要的调节机制之一。转录因子促进转录调节以应对发育和环境变化。通常,转录因子可以基于其共同的结构特征来分组到特定家庭。GRAS蛋白属于植物的转录因子家族,并以三个创始成员命名:赤霉酸不敏感(G人工智能),Ga1的阻遏物(RG一个)和稻草人(年代CR)[1] - [5].最近,在细菌中也鉴定了GRAS蛋白[6].通常,GRAS蛋白质长度为400-700个氨基酸。它们有一个可变的n端和一个高度保守的c端,c端包含五个可识别的基序,顺序如下:亮氨酸七肽重复序列I (LHR I)、VHIID、亮氨酸七肽重复序列II (LHR II)、PFYRE和SAW [7].其中,PFYRE主题由三个单位组成:P,FY和RE,SAW主题的特点是三对保守的残留物:R-E,W-G和W-W [5].显着地,vhiid,pfyre和Saw域名在SLR1蛋白中作为抑制域[8].GRAS蛋白的区分区域是位于VHIID基元两侧的两个富含亮氨酸的区域,这可能作为一个dna结合区域,类似于bZIP蛋白- dna相互作用区域[4].此外,除PAT1和SCL13外,大多数GRAS蛋白都是核定位的,而PAT1和SCL13是双定位于细胞质和细胞核的[9].
作为转录因子,已显示GRAS蛋白在许多与胃肠杆菌素信号转导相关的特定生物学过程中发挥关键作用[3.],[10.],[11.,腋生分生组织起始[12.] - [14.],拍摄公司保养[15.],根径向图案[1],[16.],Phytochrome一个信号转导[9和男性配子发生[17.].例如,在拟南芥,五个DELLA蛋白-盖,RGA.,RGL1,RGL2,RGL3- 作为胃肠杆菌素响应植物生长的阻遏物。在米饭,osmoci.已被证明可以控制分蘖[14.].在矮牵牛,佛兰对于维护拍摄顶端公司来说是必不可少的[15.].最近,由于生物信息化学和新型分子生物学技术的发展,通过逆转录-PCR(RT-PCR),cDNA或寡核苷酸微阵列和基因组水平的cDNA实时PCR进行了综合表达分析。这些分析有助于我们对GRAS家族的函数的理解[18.].
在GRAS蛋白的第一个成员诞生后,稻草人,被隔绝拟南芥[1已鉴定出不同类群的GRAS蛋白,包括番茄、矮牵牛花、百合、水稻、葡萄、松树、玉米和大麦。GRAS基因存在着很大的多样性,这取决于物种。到目前为止,各种硅分析预测有33个、60个和48个GRAS基因拟南芥,米饭和大白菜[7],[19.),分别。与此同时,大规模基因组测序和比较基因组学的快速发展,可能会导致在其他植物中发现GRAS蛋白。虽然物种之间在基因组大小、倍性水平和染色体数目方面存在很大的差异,但人们试图在比较基因组学的基础上揭示存在的共线性和共同性。
最近完成的排序和装配工作提供了在全基因组水平上更好地了解GRAS Superfamily的演变。在目前的工作中,我们在六种植物种类中鉴定了GRAS基因家族:拟南芥,b . distachyon,米饭,大豆,S. Moellendorffii.,P. Paten..然后我们构建了一种系统发育树,以评估六种植物物种的GRAS基因之间的进化关系,并计算了与复制事件日期的同义替代率(KS)。然后,我们分析了不同组织中GRAS基因的表达谱,这表明该家庭内的巨大功能性分歧。为了检查功能演化的驱动力,我们进一步分析了氨基酸水平的功能性分歧和适应性演化。我们的系统分析为进一步的功能剖析和植物中GRAS基因的分子演化提供了坚实的基础。
结果
GRAS基因家族的基因组鉴定
在硅分析中预测有33、44、47、106、21和38个GRAS基因存在于拟南芥,b . distachyon,米饭,大豆,S. Moellendorffii.,P. Paten.. s(附加文件1和2).在附加文件中提供了GRAS基因,基因座基因,染色体和位置,氨基酸序列的长度,等值(PI)和分子量(MW)。3.,4,5,6,7和8.大多数推导的GRAs氨基酸序列长度从400〜700个氨基酸变化,而超过一半的蛋白质P. Paten.含有700多种氨基酸大部分GRAS蛋白的pI值在4.68 ~ 6.92(弱酸性)之间变化,少数GRAS蛋白呈碱性。所有GRAS蛋白中,那些来自拟南芥和P. Paten.均为弱酸性,而GRAS蛋白的pI最高为9.57,在b . distachyon.GRAS蛋白的分子量范围为39.2 ~ 111.4 kD。这些结果表明,GRAS蛋白的氨基酸序列长度和理化性质可能发生了变化,以满足不同的功能。
所有GRAS蛋白均被定位到相应的染色体上S. Moellendorffii.和P. Paten.(附加文件9).在拟南芥,预测的33 atgras(拟南芥GRAS蛋白)基因分布在五种染色体中。染色体1和3分别具有九个和七个Atgras基因,而在每种染色体2和5中发现六个Atgras基因。在b . distachyon,预测的44 bdgras(b . distachyonGRAS蛋白)基因也分布在五条染色体中。染色体1和4分别具有最多17和14个BDGRAS基因,而染色体5具有至少两个BDGRAS基因。在米饭中,推定的47 osgras(奥雅萨苜蓿Gras蛋白)基因在12个染色体中的10个中组织。染色体11具有最多九种OSGRAS基因,而染色体10具有至少两种OSGRAS基因。染色体1,5和7含有五种OSGRAS基因,每种OSGRAS基因和染色体2,4和12含有四种OSGRAS基因。在大豆,106 Gmgras(甘氨酸最大GmGRAS蛋白)基因分散在20条染色体上,其中14个成员分布在11号染色体上,是GmGRAS基因密度最高的。在1、2、5、9、10、16、17、18号染色体上各发现5个GmGRAS基因,在3、4、6、7号染色体上各发现4个,在8、14、20号染色体上各发现3个。
GRAS蛋白的系统发育关系
GRAS家族成员之间的保守基板的比较暗示,它们可以分为不同的群体和亚组。为了更好地分开群体并调查GRAS蛋白中的进化关系拟南芥,b . distachyon,米饭,大豆,S. Moellendorffii.,P. Paten.,采用邻接(neighbor-joining, NJ)算法从289条全长氨基酸序列构建了无根系统发育树(图1和附加文件10.).为了确认树的拓扑结构,我们还构建了一个ML(最大似然)系统发生树,它显示了与NJ树相似的拓扑结构,只是进行了轻微的修改(附加文件)11.).还构造了ME(最小evolution)系统发育树,其显示了NJ树的拓扑相同(附加文件12.).虽然NJ树通常与ME树相同,但当类群数量较小时,NJ树和ME树之间的差异可能很大[20.].在这种情况下,如果使用长的DNA或氨基酸序列,ME树是更好的。当使用的核苷酸或氨基酸数量相对较少时,NJ方法比ME方法更容易生成正确的拓扑结构[21.],[22.].在这项研究中,289克拉蛋白的平均氨基酸长度为约580,所以我的树是可信的。连胜,采用NJ系统发育树进行进一步分析。根据以前分析的信息和从树木的拓扑和保守的主题的位置,我们将所有GRAS基因分组为七个主要集群,I-VII组[7],[18.].第五组进一步分为两个子组,VA和VB。不同组中的GRAS蛋白的数量在附加文件中显示1.在本集团中,第二组构成了最大的思工。它包含67名成员,占总GRAS基因的23.2%。同时,与其他亚组相比,Angiosperm的II组基因的数量也达到了最大值,其强烈表明这些GRAS基因更容易保留在II族中。相反,成员S. Moellendorffii.和P. Paten.更多的聚集在v组。此外,鉴定出的DELLA蛋白:盖,RGA.,RGL1,RGL2,RGL3,SLR1.(loc_os03g49990.)均出现在IV组[8],[18.].我们还推导出12个DELLA蛋白(BRADI1G11090.,Glyma10g33380,glyma08g10140,glyma06g23940.,glyma04g21340,glyma05g27190,Glyma11g33720,glyma18g04500.,139506,122441.,PP1S12_244V6,pp1s175_16v6),基于DELLA蛋白n端含有保守的DELLA和VHYNP基序,属于IV类的特征1)还展示了许多推定的直脑(例如,Bradi4g03867/loc_os12g38490.,BRADI4G43680/LOC_Os03g48450)由高引导值支持。
GRAS蛋白的完整氨基酸序列的对比分析与所呈现的系统发育分析一致,并且表明可以针对每个定义的基团确定几个家庭和亚家族特异性保守的基序。GRAS蛋白共享一个高度保守的C末端区域,含有vhiid基序侧翼的两个亮氨酸七肽(LHRI和LHRII),然后是PFYRE主题,最后是SAW主题。在以前的研究中已经多次报道了五个图案的特征[4],[5],[23.].例如,LHR I和LHR II似乎由两个重复单元(A和B)组成。由于其P-N-H-D-Q-L残基,VHIID基序在所有成员中都很容易识别。值得注意的是,我们的结果与他们的陈述非常相似,并且在附加文件中列出了6种植物的GRAS结构域的多重序列比对13.和14..总之,GRAS蛋白之间存在大量的c端同源性,表明这些保守的残基是使GRAS基因产物活性得以实现所必需的。此外,通过模因搜索GRAS结构域外的保守蛋白基序,确定GARS基因结构进化的可能机制。作为一些SmGRAS (S. Moellendorffii.GRAS蛋白)和PpGRAS (P.Patens Gras蛋白)基因与四个其他物种共享相同的主题,只有AgeniSperms的主题数据在附加文件中呈现15..其中,仅在II族中检测到五个基序组分(图案1,2,3,5和6)。有趣的是,MOTIF 5只发现在单码(b . distachyon这表明这些基因在单子叶和双子叶分裂后发生了分化。ⅳ组的DELLA蛋白具有相同的两个基序成分(DELLA和VHYNP基序),与其他各组差异显著。第一组的大部分成员都包含了主题4。GRAS蛋白基序示意图见附加文件16..简而言之,不同群体或克拉斯基因亚组中的基序分布的差异显示,GRAS基因的功能可能在进化中分化。
内含子的分布也为支持一个基因家族的系统发育关系提供了重要的证据。应用基因结构显示服务器(gene structure Display Server)分析了289个GRAS基因的基因结构演变。推测的GRAS基因家族的基因结构见附加文件3.,4,5,6,7和8.289个GRAS基因中,53个含有内含子,236个没有内含子。在这其中,loc_os10g40390.似乎具有九个内含子的复杂基因结构。简而言之,来自Angiosperm的大多数GRAS基因S. Moellendorffii.(243 of 251; 96.8%)缺少内含子或只有一个内含子,这表明这些GRAS基因被保守。但是,GRAS基因来自P. Paten.与其他物种的那些完全不同,36.8%(38个)基因具有多于一种内含子,其中包括六个内含子的三种Ppgras基因,一种具有五个内含子的PPGras基因,具有四个内含子的七个Ppgras基因,以及三个PPGRAS基因和三个PPGRAS基因和三种PPGRAS基因三个内含子。这些结果表明,GRAS基因的内含子进化可能具有更高的可变性P. Paten..此外,63.2%(24个中的38个)ppgras基因有一个或零内含子,类似于Angiosperm和S. Moellendorffii..这种现象表明,古代ppgras基因可能具有多个内含子,但逐渐失去进化中的一些内含子。最后,大多数ppgras基因都丢失了所有内含子,或者只保留了一个内含子。
这些结果表明,GRAS蛋白可分为7大组(I-VII组),这一分类得到了保守基序位置的支持。大多数GRAS蛋白具有相似的外显子-内含子结构P. Paten.,表明这些保守的内含子结构是对GRAS基因表达调控所必需的。
GRAS基因家族的复制事件
众所周知,基因复制为功能多样化提供了原料。基因家族可以通过串联扩增,导致聚集性的发生,或者通过染色体区域的节段复制,导致家族成员的分散发生。在这个分析中,我们着重于串联和节段复制模式。为了确定GRAS基因家族的扩增模式,我们首先确定了串联重复的存在。289个GRAS基因中,36个(12.5%)聚类在一起,最多有10个额外的基因在一起,可以认为是串联重复基因[24.].表中列出了6种植物的串联重复基因的成员1,包括4,6,7,17,0和2genes拟南芥,b . distachyon,米饭,大豆,S. Moellendorffii.,P. Paten.分别。有趣的是,所有假定的串联复制基因都在第二组中发现了,除了loc_os02g44360和loc_os02g44370,提示串联复制对II组GRAS基因家族的扩展可能比其他组贡献更大。检测节段复制事件的一种有效和高效的方法是在每个GRAS基因附近确定额外的副同源蛋白对[25.].如表所示2,检测107对(43.9%; 289种,共289个)的副基因基因,由系统发育树中的高自举值和类似的外显子系统结构负载,这表明细分重复有助于扩增GRAS基因家族.除了大豆(82对)外,在GRAS基因系列中,细分重复事件似乎在罕见中似乎是罕见的,其中6,4,10,0和4对拟南芥,b . distachyon, 白饭,S. Moellendorffii.,P. Paten.分别。106个GmGRAS基因中有84个存在节段重复,这表明节段重复事件主要发生在大豆基因组中。总之,节段复制和串联复制事件都参与了GRAS超家族在所有物种中的扩展S. Moellendorffii..其中,串联复制显著扩增II组,节段复制是GmGRAS基因进化的主要模式。然而,在拟南芥,b . distachyon, 白饭,S. Moellendorffii.,P. Paten.,没有单一的扩展模式表现出优势,表明来自这些物种的GRAS基因可能已经受到更复杂的进化机制。
以前的研究已经报道了几轮全基因组复制(WGD)拟南芥,b . distachyon,大米,大豆,和P. Paten..因此,利用k估计了片段重复事件的大致日期。表中列出了与GRAS基因相关的所有片段重复事件的平均Ks值、标准差和估计日期2.在拟南芥,六对Atgras副基因基因起源于23.8 mya(百万年前)至27.9 mya,这与最近发生在24-40 mya的大规模重复的日期26.].在b . distachyon,三对Bdgras副基因基因对应于迄今为止发生的WGD事件发生在56-73 mya [27.].另外两对可能是发生在约40米亚的单重复事件的结果。在水稻中,9对OsGRAS副同源基因似乎来自于发生在40-50 Mya的WGD [28.].一对(LOC_Os11g03110和loc_os12g02870.)估计节段性重复率源于7个MyA,其与在染色体11和12的末端发生的节段重复相容,估计在5-10 mya的演变中被分离[7].在大豆,Schmutz等。发现,两个大规模复制事件分别发生在大约59和13 mya [29.].我们的结果集中在两个时期,9-16 Mya和40-70 Mya,这与两个重复事件的年龄大致一致。在之前的研究中,Du等人[30.]已经在大豆基因组中鉴定出起源于WGD复制和独立复制的基因。为了进一步验证结果,我们将研究中发现的84个节段复制的GmGRAS基因与Du等人的结果进行了比较[30.].我们得出结论,84个GmGRAS基因中有70个(83.3%)来自于WGDs,而84个GmGRAS基因中有10个(11.6%)来自于独立的重复事件(数据未显示)。在P. Paten., Rensing等人发现了一个古老的基因组复制事件,该事件被认为发生在30到60年之间[31.].Later, they reported that the Ks distribution plot (i.e., the frequency classes of synonymous substitutions) among paralogs showed a clear peak at around 0.5 to 0.9 in 2008, which suggests that a large-scale duplication, possibly involving the whole genome, has occurred [32.].我们的结果表明KS.四对ppgras副偶胆基基因的值范围为0.48至0.78,与先前的研究相容。在S. Moellendorffii.,未检测到节段和串联重复事件,这一结果可能与Selaginella.基因组缺乏古代全基因组复制或多倍性的证据[33.].此外,这些结果与Edger等人的分析一致。转录因子优先保留在WGDS后[34.].我们还将所有推断的串联重复基因提交到植物基因组重复数据库中,以获得6个物种的串联重复对。但在物种间未发现同源基因,说明我们研究的6个物种形成后,这些串联重复的基因被保留了下来。
简而言之,串联重复事件在群体扩张中发挥了重要作用。在大豆的GRAS基因中,分段重复呈现。此外,WGD后,将在段重复的大多数参与分段重复中的基因。
GRAS系列功能分析
使用分歧2推断出Gras群基因簇之间的功能分歧的两种类型(II型和II型),其估计进化率(I型)的场地特异性偏移或特定位置转变的显着变化氨基酸特性(II型)出现两种副骨序列[35.].这些方法的优点是它们使用氨基酸序列,因此对同义位点的饱和不敏感[36.].估计是基于GRAS蛋白NJ树,其中8个主要亚家族清晰地呈现出来,bootstrap值高度显著支持。结果表明,I型泛函散度系数(θ我)之间的差异显著大于0 (p < 0.05, Table3.)表示它们之间的高度不同的位点特异性改变的选择性约束。II型功能分歧的系数(θ2)只有重要意义(p < 0.05) between I/III, III/IV, and III/V, particularly III/V. The coefficient of Type II functional divergence (θ2)小于0,而标准误差相对较高。这些结果表明,GRAS基因家族亚家族的功能进化可能不同程度地采用I型和II型功能差异。
为了确定可能导致GRAS亚群之间功能差异的关键氨基酸位点(CAASs),后验概率(Qk)的散度,使用与函数散度相关的残基确定[35.].一个大问k值表明位点的功能约束或氨基酸理化特性在两个簇之间不同的可能性。在这项研究中,qk> 0.95作为基因簇间CAASs的截断点。我们的结果显示,在每对基因中,预测功能差异的位点数量存在显著差异。共66个CAASs(氨基酸参照AT3G54220序列通过I型功能分解分析预测。其中24,24,23和20型相关的CAAS分别用于I / VII,II / IV,I / II和I / III对,这表明这些网站可能是一个主要的推动I / VII,II / IV,I / II和I / III的分歧的进化力。同时,鉴定了87型相关的CAAS,用于I / II,I / V,I / VI,I / VII,III / IV和III / VII对。与I / VA之间的I型功能分歧仅有三个CAASS相比,II型功能分歧的57个预测部位,表明氨基酸理化特性的快速变化主要归因于两组基因之间的功能性分歧,并将其归因于进化率的转变。案例类似于I / II和I / VII对。然而,大部分对没有遵循上述模型,表明进化率的特异性差异和氨基酸性能的变化不会在进化时间上均匀地作用于GRAS亚家族成员。最后,鉴定了44种氨基酸作为I型和II型功能分歧的共同发生的氨基酸(附加文件17.),提示这些位点对GRAS基因的亚群特异性功能进化具有重要意义。
GRAS基因家族中的正向选择
阳性选择是基因重复后新的基序和蛋白质中的新的主题和功能的主要力量之一。在本研究中,在PAML V4.4软件包中实施了似然比测试[37.]使用特异性模型测试GRAS基因家族中阳性选择的假设。首先,我们使用来自六种不同物种的全长蛋白质GRAS序列进行独立分析。结果(附加文件18.,19.,20.,21.,22.和23.),未发现阳性选择的caas拟南芥,大米或大豆,b . distachyon,S. Moellendorfii.,而30(其中11个是在0.05分显性水平和19个中的0.01分显性水平)中,鉴定了阳性选择位点P. Paten.基于贝叶斯经验贝叶斯(BEB)估计方法。这些结果表明,PpGRAS基因处于较高的正选择压力下,而其他5个物种表现出更保守的倾向。对组合的六种物种进行分析,每个模型的参数估计和对数似然值如表所示4.M3与M0比较的LRT统计值为2Δℓ= 3508.354,远远大于a的临界值2d.f. = 4的分布,表明单类ω不足以描述氨基酸位点选择压力的变异性。然而,当比较M7/M8时,没有一个CAASs被确定为阳性选择位点。这一结果表明GRAS基因超家族在进化过程中相对保守。总之,不论是种内基因还是种间基因,GRAS基因都受到不同程度的正向选择压力。
为研究GRAS亚属植物的自适应演化,我们进一步分析了分支机构模型。在GRAS基因树上(图1),七个分支(I,II,III,IV,V,VI和VII)被独立定义为前景分支。桌子5列出了分支站点模型下的参数估计和对数似然值。在x下没有发现或只有少数具有显著意义的地点2测试 (p< 0.05)。然而,当I组和V组定义为前景分支时,检测到显著的阳性选择。其中,以分支I为前景分支时,确定16个位点为阳性选择位点,其中4个位点(415P、453 F、476E、505 T)根据x显著性2测试 (p < 0.01). 11 sites were identified as positively selected sites when branch V was considered the foreground branch. Of these sites, one positive selection site (418 F) was at the 0.05 significance level, while ten sites (296Q, 303A, 412 L, 453 F, 490 W, 497D, 508 L, 511R, 513A, and 518 T) were at the 0.01 significance level. These results suggested that groups I and V were confronted with strong positive selection pressure, as many highly significant positive sites were present, whereas the other groups were likely experiencing strong purifying or neutral selection pressure.
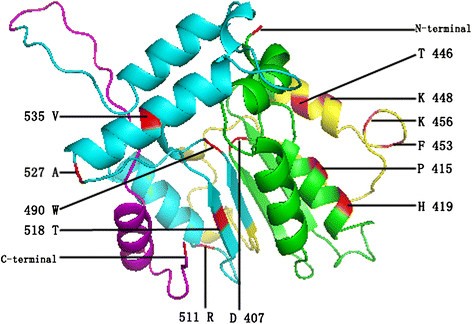
最后,我们观察到阳性选择和功能性分歧的氨基酸位点之间的关系,14个临界氨基酸位点在阳性选择以及I型和II型功能分歧(附加文件17.).我们位于三维GRAS结构上并进行了多个序列对齐,以进一步研究其功能。随着CPHModels的显示序列产生了无能的[38.[在三维结构上仅标记12位点,在多个序列对准中标记其他氨基酸位点(图2和附加文件13.).其中,两个氨基酸(296Q和368S)位于LHRI基序,三个氨基酸(407d,415p和419h)位于vhiid基序和四个氨基酸(446吨,448k,453f,456 K)位于LHRII基序,5个氨基酸(490W,511R,518 T,527A和535 V)位于PFYRE主题上。简而言之,大部分氨基酸位于α - 螺旋上。这些结果表明,这些氨基酸可以充当驱动GRAS保守基序的发散的主要进化力,并且可以进一步影响GRAS子组功能的差异。需要更多的实验证据来了解所确定的金星的功能重要性。此外,张等人。在细菌GRAS蛋白中回收到几个Rossmann折叠甲基转移酶结构域的显着命中[6].令人惊讶的是,我们还在我们的蛋白质中发现了Rossmann折叠(βαβαβ)(AT3G54220)。这些结果还表明,克拉斯蛋白的结构在较低和更高的生物体中被保守。
GRAS基因表达分析
为了研究植物生长和发育中涉及的亚组中同源GRAS基因的表达模式,我们使用基因图案计划构建了热图。作为微阵列数据和RNA-SEQ图谱b . distachyon是不完整的,我们专注于其他三种物种(拟南芥本文研究了大豆和米饭。在拟南芥的探测集IDAT2G29060在ATH1数据源中找不到。因此,在我们的差异表达分析中,只考虑了32个AtGRAS基因。在水稻中,八个基因(LOC_Os11g47890,loc_os11g47910.,loc_os11g47920,loc_os12g04200.,loc_os05g4071.0,loc_os12g02870.,LOC_Os12g04380,LOC_Os06g40780)不能在其相应的探测集中找到。共找到39个探针集,对应47个unigenes中的39个(83%)。在大豆中,表示值glyma02g01530,glyma03g06530,glyma10g35920,glyma11g20980.,Glyma12g16750,glyma15g28410.,glyma17g13680.,glyma19g40440,glyma20g31680为零,表明在一些特殊的组织中表达了这9个基因,或者器官被诱导(即,诱导基因)。而且,Glyma01g18040缺乏大豆表达信息。因此,在RNA-SEQ ATLAS数据集中仅易于识别仅96个GMGRAS基因的不同转录物丰度模式。
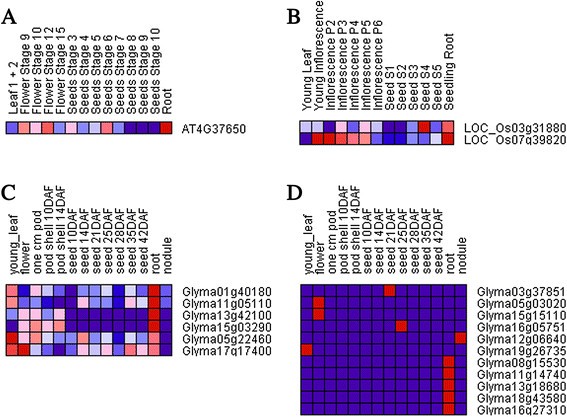
根据其他文件中的表达式配置文件24.,25.和26.广泛地,我们的结果表明,大多数草在不同组织或器官中具有不同的表达水平。此外,一些GRAS基因在营养生长期和生殖的生长阶段明显表达,表明这些GRAS基因可能调节对应于植物生长和发育的不同阶段的特定功能。同时,通过多种基因调节相同的组织和器官,表达水平在不同的GRAS基因中不同,这表明多种GRAS基因参与调节同一组织或器官的生长和发育。GRAS基因在不同物种中显示出不同的优先表达,并且大多数GRAS基因仅在单个组织类型中表现出具有标记峰的表达曲线。特别是大豆中有许多组织特异性基因(图3.).例如,仅在根中仅表达五个基因,并且仅在种子中表达两个基因。这些结果表明,那些GRAS蛋白质用作组织特异性调节剂,或者限于单个器官或细胞类型。此外,李等人。已经描述了一些GRAS基因的表达分析拟南芥[18.].虽然加工时间不同,但我们的结果显示许多ATGRAS基因具有相似的表达水平。例如,SCL23在叶、花和种子中的表达水平高于在根中的表达水平,这证实了之前的观点,即SCL23在地上部分发挥作用。其他许多SCL基因在根中也有表达,包括SCL4、SCL9、SCL11、SCL28、SCL30、SCL31等。此外,在同一物种中还存在表达谱相似但系统发育差异较大的基因亚群。然而,多个系统发育分支在很大程度上具有相同的转录丰富度。在组III中,一个系统发育分支包括来自三个物种的9个GRAS基因(图)3.),它们优先在根中表达。显然,我们所研究的三个物种中同源基因亚群的表达模式有不同程度的保守。
众所周知,基因复制增加了表达的多样性,使组织或发育专门化得以进化。大野经典模型[39.]关于重复基因的命运和duplication-degeneration-complementation (DDC)模型,预测每一个副本获得的一个新函数(neofunctionalization),其损失(pseudogenization)或重叠冗余功能的发展和表达模式(subfunctionalization) [40],[41].为了追踪GRAS重复基因的表达多样性和功能,拟南芥表示已经广泛研究了基因组结构和基因表达模式的模型系统。如附加文件19所示,一对重复的基因(AT2G45160和AT3G60630)有相同的表达模式。然而,AT3G4660和AT5G17490在叶和根中,它们分别促进/抑制萌发,并发挥相反的调控作用。这种效果与新功能化的情况更相关。在剩余的重复基因中也发现了类似的病例。此外,另一对复制基因(AT1G07520和AT2G29065).前者似乎具有明显较弱的表达比后者在种子中。但是,事实上AT1G07520在种子中具有一定程度的表达可能意味着伪遗传化尚未完成。
总之,在不同器官和物种中,GRAS亚群成员的表达谱是不同的,说明GRAS基因在不同的组织和物种中表达存在差异,GRAS基因的调控区域可能存在差异。值得注意的是,该结果也证明了GRAS复制基因在进化过程中的表达差异。
讨论
GRAS基因家族的比较基因组分析
在本研究中,我们确定了来自六种植物物种的289个Gras基因,并构建了一个系统发育树(图1)将所有GRAS基因分为七个主要集群,由保守的主题职位支持的I-VII组。对于树中的许多已定义的组和子组存在相当大的引导值支持,但剩余的支持值差别为几个集群。这是表演研究的预期后果,如本发明,平均约为580个氨基酸 - 长度序列,这是通过GRAS保守的基序中大量可选残留物施加的约束。值得一提的是,大多数组的定义都是通过GRAS结构域外的常见蛋白质基序的存在来支持。在每组中,大豆中的GRAS基因的数量是其他物种的GRAS基因数量的两三倍,并且在大豆中的串联和节段性重复事件比其他物种更多。主要原因可能是大豆具有高度重复的基因组(1,115 MB),比更重复拟南芥(145 Mb) [42],b . distachyon(272 MB)[27.],米饭(430 MB)[43],S. Moellendorffii.(212.6 Mb) [33.] 要么P. Paten.(511 MB)[44]近75%的基因显示出现多个副本[29.].
另一方面,系统发育树中的大多数密切相关的成员具有常见的基序组合物,表明在同一亚家族内的GRAS蛋白质中存在功能性相似性,因此系统发育分析还将促进功能性基因组学研究。例如,推导的12个Della蛋白与所确定的六个Della蛋白聚集良好(盖,RGA.,RGL1,RGL2,RGL3,SLR1.),其介于赤霉素调节基因表达的调节[45].在树上(图1),一组两种Gmgras蛋白(glyma02g47640.和glyma14g01020)与Pat1.它在对远红光的反应中起作用,似乎在光敏色素a信号通路中起早期作用。因此,这两个GmGRAS蛋白的作用机制可能与Pat1.蛋白质。在包含的集群中发现了类似的病例SHR.(AT4G37650/glyma01g40180./glyma11g05110/Glyma05g22460/glyma17g17400.,SCL13(AT4G17230/glyma17g01150/Glyma07g39650/Glyma09g01440/glyma15g12320,SCL3.(AT1G50420/glyma01g43620/Glyma11g01850/glyma11g10170/glyma12g02490.,可控硅(At3g54220/glyma18g45220/glyma09g40620/LOC_Os11g03110/loc_os12g02870..在这其中,SHR.参与根的径向组织,射击轴向器官[5],SCL13是Phytochrome依赖性红光信号的正调节因子[46],SCL3.通过拮抗母体生长抑制器Della促进胃肠杆菌素信号传导拟南芥[47],和可控硅基因调控不对称细胞分裂[1].
内含子进化是基因组进化的重要组成部分,以及品种的自适应过程。我们的结果表明,大多数GRAS蛋白有很少的内含子(零或一个内含子),只有几种GRAS基因有两个内含子。高度保守内含子的类似基因结构对于GRAS系列的分子演变是重要的。然而,大多数GRAS基因来自P. Paten.内含子的数量与其他物种不同,其中近一半的内含子在GRAS结构域外的序列较长,表明PpGRAS基因内含子的进化是一个多样化而复杂的过程。
GRAS基因家族的扩展模式
Edger等人。[34.表示,在WGDS后,优先保留包括转录因子的剂量敏感基因。最近,验证了一些转录因子家族,如Wrky和Dof,通过分段重复事件扩展,并且在WGDS之后保留了大部分[48],[49].一些大型多曲线复合物也遵循相同的模式。例如,朱等人。证明大多数分段重叠的大豆扩展基因已从WGDS保留[50].本研究表明,在6个物种中,WGD也保留了大部分已鉴定的节段复制基因,这支持了Edger等人的研究结果。另一方面,从组来看,组II(67个基因,23.2%)是GRAS基因组中最大的分支,推测的串联重复基因大部分都在该组中。这一结果表明,串联重复极大地促进了II组的扩展。然而,造成这一结果的原因尚不清楚,需要进一步研究。就物种而言,在我们研究的6个物种中,大豆具有最多的GRAS基因成员,这可能是由多种因素造成的。一个原因是大豆是一种古老的多倍体,它的基因组比许多其他物种都大。另一个原因是在大约59和13 Mya发生的两次大规模WGDs导致基因组高度重复,近75%的基因存在于多个拷贝中,并且大多数涉及节段复制的基因在WGDs后被保留[29;34)。其中,片段复制是大豆GRAS基因扩增的主要方式。 Among these, four pairs of GmGRAS genes (Glyma11g14670/Glyma11g14700,glyma12g06630./glyma12g06640.,Glyma13g41220/Glyma13g41240,glyma15g04160./glyma15g04173.)在串联和节段性重复事件中检测到,证明了四对Gmgras基因经历了两种不同类型的扩展。然而,来自节段性重复事件的这些基因的估计日期显然是不同的,揭示这些Gmgras基因首先进行了串联重复和第二节分段重复。简而言之,GRAS基因家族在不同物种中显示出不同的膨胀模式。这些不同物种的GRAS基因家族的不同进化模式将有助于促进进一步的基因功能分析。
作为表2所示,所有推导的寄生基因对的估计日期范围为7.9至67.9 mIRA,并且所有推导出的串联重复的基因可能在其各自物种的形态之后起源。结合在一起,结果清楚地表明这些GRAS复杂的基因,其中包括42%(14个),32%(14个),47%(22个,共47个),89%(106个)和26%(第38个中的10个)基因拟南芥,b . distachyon,大米,大豆,和P. Paten.分别在大约200 mya逐渐分开后延迟单子叶 - Dicot [51].但是,有些存在P. Paten.7个亚科的序列表明GRAS基因家族形成于苔藓和种子植物祖先分化之前。Engstrom(2011)发现主要的GRAS蛋白亚家族是古老的,这与Nishiyama等人的结果一致,即GRAS基因家族出现在4亿多年前陆生植物出现之前[52],[53].以上分析表明,GRAS基因家族可能起源于一个共同的祖先,在其进化过程中,每个谱系和物种会发生谱系特异性的扩展和分化。此外,内含子数量的变化也揭示了GRAS基因家族内含子的进化过程。大多数GRAS基因来自被子植物和植物S. Moellendorffii.要么缺少内含子,要么只有一个内含子,而36.8%的PPGRAS基因有多个内含子,这表明GRAS基因家族最初可以含有多个内含子,然后丢失所有内含子或仅保留单个内含子。此外,Tian等人。推导出两对Osgras古代复制品,基于对的并置loc_os05g42130./loc_os07g40020与AT3G49950和LOC_Os03g31880/loc_os07g39820与AT4G37650在系统发育树中,和AT3G49950和AT4G37650是古代复制品,似乎是从动物复制事件中衍生出来的,预测单子叶二十滴分歧[7].在本研究中使用了相同的方法,我们推导出有三个古老的BDGRAS基因,Bradi1g22907,BRADI2G20760,BRADI1G23060.此外,所有推导的古老GRAS基因来自III组,含有没有节段性或串联复制事件,这意味着这些古老的GRAS基因来自三种物种,在进化过程中,经历了很少或没有扩增。
阳性选择和功能分析分析
在基因家族中,通过重复产生的新基因在进化新功能,并且由于阳性选择或在进化过程中丢失[54].通常,在重复基因的进化的早期阶段中,基因不受选择压力(Ka /ks≈1)或将它们进行阳性选择的显示特性(Ka / ks> 1)。在特定的功能演化中,每个基因具有固定功能,并且选择压力趋于净化选择(Ka / ks <1)[55],[56].因此,当复制的基因非常老时,很难观察到积极的选择压力。在本研究中,无论采用位点特异性模型还是分支位点模型,除I组和V组外,GRAS亚科中均未发现或仅有少量显著位点(见表)4和5).有些古代GRAS蛋白可能受到净化选择的主要进化型,这将部分解释上述结果。尽管如此,我们检测到几个在正选择压力下的卡斯。相比之下,PPGRAS基因经历了相对较高的阳性选择压力,因为它们30阳性选择位点,而研究的其他五种物种似乎是更保守的,并且没有检测到阳性选择位点。此外,P. Paten.在GRAS结构域外的各种外显子内部结构和较长的序列比其他物种强烈支持这种观点。另一方面,我们在I组中检测到16个重要部位,表明这些氨基酸位点可以作为I组中的主要进化力。此外,功能分歧的分析也支持了这一假设。将CAAS始终鉴定在I型中的其他组(进化率换档)的组中,强烈建议发生GRAS基因的群体群体功能演进。同时,在V组中检测到11个CAAS。它与来自的v基因数量相当显着S. Moellendorffii.和P. Paten.与其他子组相比,s达到了最大值。此外,与I / VA和I / VB对鉴定出I / VA和I / VB对的只有三种CAASS相比,只有三种CAASS用于I型功能分歧,有57型相关的CAASS,其强烈表明了一些古代氨基的物理化学性质酸可能在进化中发生变化,进一步推动I族和群体的功能分歧。此外,我们确定了12位点,该遗址负责功能性分歧和阳性选择。通常,氨基酸残基在一种重复基因中高度保守,但另一个在另一个重复基因中得到高度变化[57].因此,这些CAAS部分反映了GRAS基因家族的编码区可能已经分散,并且这些CAASS可以充当推动GRAS基因家族功能分歧的主要进化力。另一方面,功能分歧可能反映了长期选择性压力的存在。特别地,亚家族对之间的I型功能分歧的显着差异表明,可能已经发生了进化率的不同场地特定偏移。通过长期选择的短暂,通过长期选择的重复基因导致GRAS基因家族的基因簇之间的功能性约束改变。
Della蛋白的表达分析
DELLA蛋白是植物特异性蛋白GRAS家族的一个亚群。在本文中,我们预测了14个通过赤霉素介导基因表达调控的DELLA蛋白的存在,这些蛋白参与了从营养生长到生殖生长的过渡[58].以前的研究表明,它们促进种子萌发,叶膨胀,开花,干伸长率和花卉发育。在我们的表达配置文件中,RGL1,RGL2,RGA.,盖在花中优先表达,这与Cao等人的研究结果一致,即赤霉素动员不同的della依赖转录组调控拟南芥[59].同时,其他Della蛋白(loc_os03g4990.,glyma08g10140,glyma05g27190)来自水稻和大豆也在花中表现出高表达水平(附加文件24.,25.和26.).此外,RGL3优先以种子表达,如Glyma10g33380,glyma06g23940.,glyma04g21340,glyma18g04500..然而,Glyma11g33720优先于结节表达。这些结果表明,Della蛋白的功能相对节省,而且仍然存在功能性偏差,以满足不同物种的特殊要求。Gallego-Bartolome等。报道了不同Della蛋白的功能多样化拟南芥是亚功能化的结果,可能是由于蛋白质调节序列的变化[60].需要更多的实验来揭示不同物种中的Della蛋白的不同转录机制。
结论
本研究提供了GRAS基因家族的比较基因组分析拟南芥,b . distachyon,米饭,大豆,S. Moellendorffii.,P. Paten.,将GRAS基因分配给七个主要集群。复制的GRAs基因差异表达的结果表明,蛋白质的功能可能已经发散,以满足不同物种的特殊要求。GRAS系列基因在不同物种和群体中显示出不同的膨胀模式。节段性重复是大豆GRAS基因家族的主要膨胀模式,而串联复制事件在II组基因的扩展中发挥着重要作用。所有推定的重复基因被鉴定过透明单子叶蛋白分裂。此外,来自I族和组V组的这些基因在较高的阳性选择压力下,该压力由分支部位模型揭示。此外,特定于网站特异性模型显示GRAS基因在较高的阳性选择压力中P. Paten.S比其他五种保守的物种。功能分解的分析表明,当与I类型中的其他组进行比较时,始终鉴定了CAASS,强烈表明移位的进化率可能主要归因于I族特异性功能演进。最后,尽管预测的18个Della蛋白相对节省了,但它们的功能根据GRAS系列的表达谱发散。简而言之,我们的分析为植物中GRAS基因的进一步功能解剖进行了实心的基础。
方法
在四种植物物种中鉴定GRAS家族成员
在植物中,模型生物体拟南芥通常用于预测新测序或部分测序生物体中某个基因的功能。Lee等人确定了33名GRAS成员拟南芥,其中我们排除了一个假基因,AT5G67411,来自我们的分析[18.].来自32个非冗余GRAS基因序列拟南芥信息资源(TAIR)用于爆炸植物血统数据库http://www.phytozome.net..从上述网站上收集了包含目标基因的所有信息的数据文件,包括染色体位置、基因组序列、完整编码序列和蛋白质序列。选取E值≤1e-5的序列作为候选蛋白。通过去除多余基因和不完整的开放阅读框序列,鉴定出独特的GRAS基因。通过在SMART数据库(http://smart.emblheidelberg.de/).然后,没有典型的GRAS结构域(五个可识别的基序,LHR I,VHIID,LHR II,PFYRE和SAW)的基因被删除。此外,还排除了含有多于一种涂布结构域的推定的GRAS蛋白。最后,将GRAS蛋白提交到扩展数据库以确定MW和PI。
为了避免伪原的干扰,我们用以下步骤排除伪原。首先,没有排除没有完整域的基因。其次,为了鉴定EST或全长cDNA,对GENBANK的非小鼠和非人类EST数据库进行了爆破的克拉斯基因的编码区。第三,我们试图找出这些基因是否拥有Plantcare数据库的真正启动子(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/).对所有GRAS基因的翻译起始密码子上游总共1500bp核苷酸序列进行了用于硅基分析。含有一般的CIS作用元素的那些基因,例如TATA盒,CAAT-BOX,不被视为假生素。
对准,系统发育分析和基因结构预测
使用MUSCLE程序对鉴定出的GRAS蛋白进行比对[61]带有默认参数。通过使用Mega5.0推断出直接分析(邻接,最大可能性和最小展会)的三种不同的分析(邻近的,最大可能性和最小演变),并评估内部分支的可靠性,用1000-举自动启动重采样进行评估[62],[63].除GRAS域外,GRAS家族中的其他基序使用具有默认设置的模因进行统计识别。找到的最大图案数是7个。利用gene structure Display Server对预测GRAS基因的外显子-内含子基因结构进行分析,并与Phytozome中相应基因组DNA序列的编码序列进行比较[64].
计算到GRAS基因家族复制事件的日期的Ks
GRAS基因在染色体上显示出散射的分布模式。基于其基因座,几个基因清楚地彼此邻近。因此,我们专注于分段和串联重复的过程。根据Schauser等。,检测节段性重复事件的有效方法是识别每个家庭成员的附近的额外的递质蛋白对[25.].植物基因组复制数据库(PGDD;http://chibba.agtec.uga.edu/duplication),我们选择200 KB的参数模型来运行查询。
预计ks的重复基因会随着时间的推移相似,因此为了迄今为止进行分段复制事件,我们将KS作为代理估算了分段复制事件的日期,我们选择了0到1的ks值。使用均值k计算重复事件的近似日期年代从T = K年代/2λ,假设同义替换为6.5×10的时钟速率(λ)−9每年每年的单码遗址的替换[65, 1.5 × 10−8为了拟南芥[26., 6.1 × 10−9大豆(54].然而,一个精确的λP. Paten.没有被发现。
估计功能性分歧
在顾之后进行了功能分歧的最大可能性测试66],使用DIVERGE v2.0包[67],其估计在出现两次寄生序列后进化率(I型)或氨基酸特性(II型)的特异性特异性变化的显着变化[35.].I型表示在基因1中高度保守但基因2中高度可变的氨基酸配置,反之亦然,反之亦然,这意味着这些残留物具有变化的功能约束[35.],[66].II型表示在两个基因中高度保守的氨基酸配置,但其生化特性非常不同,这意味着这些残留物可能对功能规范负责[35.].I型和II型功能分歧的系数(θ我和θ.2)之间的关系。θ的值我或θ.2that was significantly greater than 0 indicates that site-specific altered selective constraints were present (i.e., the types are experiencing different evolutionary rates) or a radical shift in amino acid physiochemical properties had occurred (e.g., positive versus negative charge) after gene duplication and/or speciation [35.],[66].
自适应演进分析
使用PAML V4.4软件包中包含的Codeml程序来识别正面选择[37.],使用站点特定模型和分支站点模型。在位点特异性模型中,非同义替换率(dN)高于同义替换率(dS);当ω (dN/dS)大于1时,表明在分子水平上存在正向选择。在分析中,选择了两对模型,使用BEB确定积极选择的地点[68估计方法。模型M0假设所有站点的单个Ω比例。型号M3允许三个不受约束的Ω类别(ω<1,净化选择;Ω= 1个中性或正选择)。比较M0(一个比率)和M3(离散),使用DN / DS比率值ω的密码子位点之间的异质性测试。模型M7是针对正面选择的空测试,假设在0和1之间的Ω之间具有Ω的β分布。M8的型号M8添加了具有相同比率ω的额外类[69].M7(Beta)与M8(β+ω> 1)的比较是阳性选择的最严格测试[70].最后,使用似然比测试来确定仪表之间的ω比是否不同;也就是说,当允许选择的模型(M3和M8)明显优于NULL模型时(无选择),指示正选择。
分支部位方法假定系统发育树的分支被划分为一个先验以及密码子位点之间的ω比不同。序列中有四个站点类。第一类位点在整个树中高度保守,为0 < ω0 < 1. The second class includes codons that are evolving neutrally throughout the tree with ω1 = 1. In the third and fourth classes, the background lineages are conserved or neutral, but positive selection on the foreground branches with ω2 > 1; that is, only foreground lineages experience positive selection. The likelihood ratio test was calculated using the BEB estimation method [68].
提取微阵列数据或RNA-Seq图谱
在不同发育背景下研究的基因的表达芯片数据拟南芥是从TAIR获得的(http://www.arabidopsis.org/)实验基因表达图谱拟南芥发展 [71].表达值是根据以下组织测定的:幼叶,花阶段(9,10,12,15),种子阶段3与单粒,种子阶段4与单粒,种子5与单片机,种子阶段6,没有分层,种子第7阶段没有单片机,种子阶段8没有分层,种子阶段9没有分层,种子阶段10没有分层,根。通过GCOS方法,TGT值为100标准化数据。
Rice eFP浏览器(http://www.bar.utoronto.ca/efprice/cgi-bin/efpWeb.cgi)工具用于搜索米的微阵列数据。我们还使用实验GSE6893,用于分析各种组织中的空间和颞型基因表达和稻米生殖发育的各个阶段[72].在不同组织和发育阶段的表达量分别为:幼叶、穗部各阶段(P1-P6)、苗根和种子(S1-S5)。数据采用MAS.5.0和RMA方法归一化。TGT值为100,所有组织取样3个重复。
引入了RNA-SEQ数据以分析Gmgras基因的表达。使用读取的S / KB /百万方法的变型进行标准化数据,并从大豆(http://soybase.org/soyseq/).在几个器官中进行表达分析:幼叶,花,一厘米豆荚,荚壳(开花后10天和14天),种子(10,14,21,25,28,35和42天后),根和结节。同时,使用基因图案程序生成所有热图(http://www.broadinstitute.org/cancer/software/genepattern/).
附加文件
参考
- 1。
dilaurenzio L, Wysocka-Diller J, Malamy JE, Pysh L, Helariutta Y, Freshour G, Hahn MG, Feldman KA, Benfey PN:稻草人基因调控拟南芥根径向组织产生的不对称细胞分裂。细胞。1996,86:423-433。10.1016 / s0092 - 8674(00) 80115 - 4。
- 2。
Peng J,Carol P,Richards de,King Ke,Cowling RJ,Murphy GP,Harberd NP:拟南芥GAI基因定义了一种负调节胃肠杆菌素反应的信号通路。基因开发。1997,11:3194-3205。10.1101 / gad.11.23.3194。
- 3.
Silverstone Al,Ciampaglio CN,Sun TP:Arabidopsis RGA基因编码转录调节剂压制胃肠杆菌素信号转导途径。植物细胞。1998年,10:155-169。10.1105 / TPC.10.2.155。
- 4.
PYSH LD,Wysocka-Diller J,Camilleri C,Bouchez D,Benfey PN:拟南芥GRAS基因家族:恐慌 - 乌鸦类基因的序列表征和基本表达分析。工厂J. 1999,18:111-119。10.1046 / J.1365-313x.1999.00431.x。
- 5.
Bolle C: GRAS蛋白在植物信号转导和发育中的作用。植物营养学报。2004,32(6):692 -692。10.1007 / s00425 - 004 - 1203 - z。
- 6.
张D,Iyer LM,Aravind L:细菌Gras结构域蛋白质在凝胶酸反应机制上投掷新灯。生物信息学。2012,28(19):2407-2411。10.1093 / Bioinformatics / BTS464。
- 7.
田超,万平,孙绍华,李军,陈敏:水稻和拟南芥GRAS基因家族的全基因组分析。acta botanica sinica(云南植物学报),2004,29(4):531 - 534。10.1023 / B: PLAN.0000038256.89809.57。
- 8.
itoh H,Ueguchi-Tanaka M,Sato Y,Ashikari M,Matsuoka M:吉伯雷林信号通路是由细胞核中细长米的外观和消失的调节。植物细胞。2002,14:57-70。10.1105 / TPC.010319。
- 9。
Bolle C, Koncz C, Chua NH: PAT1是GRAS家族的新成员,参与光敏色素a信号转导。基因工程学报,2000,14(4):469 - 478。
- 10.
Peng J, Richards DE, Hartley NM, Murphy GP, Devos KM, Flintham JE, Beales J, Fish LJ, Worland AJ, Pelica F, Su-dhakar D, Christou P, Snape JW, Gale MD, Harberd NP: ‘Green revolution’ genes encode mutant gibberellin response modulators. Nature. 1999, 400: 256-261. 10.1038/22307.
- 11.
Ikeda A, Ueguchi-Tanaka M, Sonoda Y, Kitano H, Koshioka M, Futsuhara Y, Matsuoka M, Yamaguchi J:纤细水稻,一种构成型赤霉素反应突变体,是由SLR1基因的空突变引起的,SLR1基因是高度调节基因GAI/RGA/RHT/D8的同源基因。acta botanica sinica(云南植物学报). 2001,13(4):489 - 494。10.1105 / tpc.13.5.999。
- 12.
番茄侧向抑制基因(Ls)编码一个新的VHIID蛋白家族成员。台湾大学学报(自然科学版),1998,16(4):429 - 434。10.1073 / pnas.96.1.290。
- 13。
Greb T, Clarenz O, Schafer E, Muller D, Herrero R, Schmitz G,’s K:拟南芥侧抑制基因的分子分析揭示了腋生分生组织形成的保守控制机制。基因工程学报,2003,17(4):417 - 417。10.1101 / gad.260703。
- 14。
李X,钱Q,傅Z,王y,熊g,zeng d,王x,刘x,腾s,hiroshi f,元m,罗d,汉b,李j:稻米分蘖控制。自然。2003,422:618-621。10.1038 / Nature01518。
- 15.
斯金曼J,Jaggi F,Kuhlemeier C:拍摄分发维护由来自差分细胞的GRAS-基因介导的信号控制。基因开发。2002,16:2213-2218。10.1101 / gad.230702。
- 16.
Helariutta Y,Fukaki H,Wysocka-Diller J,Nakajima K,Jung J,Sena G,Hauser Mt,Benfey Pn:短根基因通过径向信号传导控制拟南芥根系的径向图案化。细胞。2000,101:555-567。10.1016 / s0092-8674(00)80865-x。
- 17.
Morohashi K, Minami M, Takase H, Hotta Y, Hiratsuka K:一种新的调控减数分裂相关基因表达的GRAS基因的分离和特性。中国生物医学工程学报。2003,27(4):457 - 461。10.1074 / jbc.M301712200。
- 18.
Lee Mh,Kim B,Song Sk,Heo jo,yu ni,lee sa,金m,金dg,Sohn So,Lim Ce,Chang Ks,Lee mm,Lim J:拟南芥GRAS基因家族的大规模分析蒂利亚纳。植物mol biol。2008,67:659-670。10.1007 / s11103-008-9345-1。
- 19。
宋晓敏,刘克天,段文坤,马庆华,任静,王智,李勇,侯小龙:大白菜GRAS基因家族的全基因组分析。学报)。中国生物医学工程学报,2014,35(1):1 - 8。10.1016 / j.ygeno.2013.12.004。
- 20.
王志强:分子进化与系统发育。纽约:牛津大学出版社;2000年。
- 21。
Nei M, Kumar S, Takahashi K:在系统发育分析中,当使用的核苷酸或氨基酸数量很少时,优化原则往往会给出错误的拓扑结构。中国科学(d辑:地球科学),1998,19(5):593 - 598。10.1073 / pnas.95.21.12390。
- 22。
Takahashi K,Nei M:在使用大量序列时,在最大规定的标准,最小序列和最大可能性下,系统发育的快速算法的效率。mol Biol Evol。2000,17:1251-1258。10.1093 / oxfordjournals.molbev.a026408。
- 23。
Heery DM,Kalkhoven E,Hoare S,Parker Mg:转录共激活剂中的签名基质介导与核受体的结合。自然。1997,387:733-736。10.1038 / 42750。
- 24。
Ramamoorthy R,Jiang Sy,Kumar N,Venkatesh Pn,Ramachandran S:在各种非生物和植物甾族治疗中的水稻中的Wrkygene家族的综合转录分析。植物细胞物理。2008,49:865-879。10.1093 / PCP / PCN061。
- 25.
Schauser L,Wieloch W,Stougaard J:拟南芥,米和莲花粳稻类似萘蛋白的演变。J Mol Evol。2005,60:229-237。10.1007 / s00239-004-0144-2。
- 26.
Blanc G,Hokamp K,Wolfe KH:最近的多倍体叠加在拟南芥基因组中的较老的大规模重复上。Genome Res。2003,3(2):137-144。10.1101 / GR.751803。
- 27.
国际刷型倡议:模型草原桥型探矿的基因组测序和分析。自然。2010年,463:763-768。10.1038 / Nature08747。
- 28。
帕特森啊,鲍德·杰,查普曼坝:古代多倍化谷物的差异性,以及对比较基因组学的后果。PROC NATL ACAD SCI。2004,101:9903-9908。10.1073 / pnas.0307901101。
- 29。
污物J,大炮某人,马Chlueter J, J, Mitros T,纳尔逊W,海顿耐DL,歌Q, JJ,泰伦程J,徐D, Hellsten U, GD,于Y,樱井T, Umezawa T, Bhattacharyya可,Sandhu D, Valliyodan B,林奎斯特E,皮托,格兰特D,蜀年代,古德斯坦D,巴里·K Futrell-Griggs M B,阿伯纳西Du J,田Z,朱李:古多倍体大豆的基因组序列。自然科学学报。2010,463(7278):178-183。10.1038 / nature08670。
- 30.
Du J,Tian Z,Sui Y,Zhao M,Song Q,Cannon Sb,Cregan P,MA J:蠕动术效果造成古代多倍醇大豆中复制基因的分歧,保留和表达模式。植物细胞。2012,24:21-32。10.1105 / TPC.111.092759。
- 31.
Rensing SA, Ick J, Fawcett JA, Lang D, Zimmer A, Van de Peer Y, Reski R:一个古老的基因组复制导致了苔藓小Physcomitrella patens代谢基因的丰富。生物谷bioon . 2007, 7: 130-10.1186/1471-2148-7-130。
- 32.
仁瑟SA,朗D,齐默广告,特里,Salamov,夏皮罗H,西山T, Perroud PF,林奎斯特EA Kamisugi Y, Tanahashi T,神K, Fujita T, Oishi K, Shin-I T, Kuroki Y,丰田章男,铃木Y,桥本年代,山口K, Sugano年代,Kohara Y,富士山,Anterola,青木年代,阿什顿N, Barbazuk WB,巴克E, Bennetzen杰,布兰肯希普R:小Physcomitrella的基因组揭示了植物征服土地的进化过程。中国科学(d辑:地球科学),2008,32(6):591 - 598。10.1126 / science.1150646。
- 33.
Banks Ja,Nishiyama T,Hasebe M,Bowman JL,Gribskov M,De Pamphilis C,Albert VA,Aono N,Aoyama T,Ambrose Ba,Ashton NW,Axtell MJ,Barker E,Barker Ms,Bennetzen JL,Bonawitz Nd,ChappleC,Cheng C,Correa Lg,Dacre M,Debarry J,Dreyer I,Elias M,Engstrom Em,Estelle M,Feng L,Finet C,Floyd Sk,Frommer WB,Fujita T:Selaginella Genome识别与之相关的遗传变化血管植物的演变。科学。2011,332(6032):960-963。10.1126 / Science.1203810。
- 34.
edger pp,piresjc:基因和基因组重复:剂量敏感性对核基因命运的影响。染色体res。2009,17:699-717。10.1007 / S10577-009-9055-9。
- 35。
谷旭:蛋白质(家族)序列进化中的功能差异。中国生物医学工程学报。10.1023 /: 1024197424306。
- 36。
Liu Q, Wang H, Zhang Z, Wu J, Feng Y, Zhu Z: NOD26-like intrinsic的功能和表达。中国生物医学工程学报。2009,10:313-10.1186/1471-2164-10-313。
- 37。
PAML4:最大似然系统发育分析。生物科学进展。2007,24(4):591 - 598。10.1093 / molbev / msm088。
- 38。
Nielsen M,Lundegaard C,Lund O,Petersen TN:CPHModels-3.0 - 使用结构引导序列配置文件的远程同源性建模。核酸RES。2010,38:W576-W581。10.1093 / nar / gkq535。
- 39.
OHNO S:基因复制的演变。柏林海德堡纽约:斯普林克;1970年。
- 40.
Force A, Lynch M, Pickett FB, Amores A, Yan YL, Postlethwait J:通过互补和退化突变保存重复基因。遗传学报。1999,31(4):591 - 598。
- 41.
林奇M,强制答:子官能化重复基因保存的概率。遗传学。2000,154:459-473。
- 42.
Arumuganathan K,Earle Ed:一些重要植物物种的核DNA含量。Genet Res。1991,9(3):208-218。
- 43.
Chen M,Presting G,Barbazuk WB,Goicoechea JL,Blackmon B,方G,金H,Frisch D,Yu Y,Sun S,Higingbottom S,Phimphilai J,Phimphilai D,Thurmond S,Gaudette B,Li P,Liu J.,Hatfield J, Main D, Farrar K, Henderson C, Barnett L, Costa R, Williams B, Walser S, Atkins M, Hall C, Budiman MA, Tomkins JP, Luo M: An Integrated Physical and Genetic Map of the Rice Genome. Plant Cell. 2002, 14 (3): 537-545. 10.1105/tpc.010485.
- 44.
Victoria FC,Da Maia LC,De Oliveira AC:在植物中SSR标记的SILICO比较分析。BMC植物BIOL。2011,11:15-10.1186 / 1471-2229-11-15。
- 45。
Cao D,Hussain A,Cheng H,Peng J:四个Della基因的功能丧失导致光线和吉布林素无关的种子萌发拟南芥.植物营养学报。2005,22(3):427 - 434。10.1007 / s00425 - 005 - 0057 - 3。
- 46。
Torres-Galea P,Huang Lf,Chua NH,泡杆菌C:GRAS蛋白SCL13是植物依赖性红光信号的阳性调节器,但也可以调节植物的反应。Mol Genet基因组学。2006,276:13-30。10.1007 / s00438-006-0123-y。
- 47。
Zhang ZL, Ogawa M, Fleet CM, Zentella R, Hu J, Heo JO, Lim J, Kamiya Y, Yamaguchi S, Sun TP:稻草人样3通过拮抗生长抑制因子DELLA促进拟南芥赤霉素信号转导。中国科学(d辑),2011,32(5):531 - 534。10.1073 / pnas.1012232108。
- 48。
Guo Y,QIU LJ:对DOF转录因子基因系列的基因组分析显示大豆特异性重复和功能特性。Plos一个。2013,8(9):E76809-10.1371 / journal.pone.0076809。
- 49.
尹g,xu h,xiao s,qin y,李y,yan y,hu y:大豆(甘氨酸最大)Wrky Tf家族扩大了分段重复事件,随后在亚组之间进行了不同选择。BMC植物BIOL。2013,13:148-10.1186 / 1471-2229-13-148。
- 50。
朱y,吴,宋,尹g,qin y,yan y,hu y:大豆(甘氨酸max)扩展蛋白基因超级家族起源:分段和串联复制事件,然后在亚属之间进行分歧选择。BMC植物BIOL。2014,14(1):93-10.1186 / 1471-2229-14-93。
- 51。
Wolfe Kh,Gouy M,Yang Yw,Sharp PM,Li WH:单子叶菌差异的日期叶绿体DNA序列数据估算。Procnat Acad Sci U S A. 1989,86(16):6201-6205。10.1073 / pnas.86.16.6201。
- 52。
Engstrom EM:对苔藓、石松和维管植物谱系中GRAS蛋白的系统发育分析表明,GRAS基因起源于苔藓和维管植物共同的祖先谱系,并经历了大量的多样化。植物信号学报,2011,6(6):850-854。10.4161 / psb.6.6.15203。
- 53。
Nishiyama T,Fujita T,Shin-I T,Seki M,Nishide H,Uchiyama I,Kamiya A,Carninci P,Hayashizaki Y,Shinozaki K,Kohara Y,Hasebe M:Physcomitrella的比较基因组学Patensophytic转录组和拟南芥:含义土地植物演化。Procnat Acad Sci U S A. 2003,100(13):8007-8012。10.1073 / PNAS.0932694100。
- 54。
Lynch M, Conery JS:重复基因的进化命运和后果。科学通报。2000,32(4):457 - 461。10.1126 / science.290.5494.1151。
- 55.
Kondrashov Fa,Rogozin IB,Wolf Yi,Koonin EV:在基因重复的演变中的选择。基因组Biol。2002,3(2):0008.1-0008.9。10.1186 / GB-2002-3-2-Research0008。
- 56.
张杰,迪恩am,brunet f,Long My:在果蝇新的新基因中不断发展的蛋白质功能多样性。Procnat Acad Sci U S A. 2004,101:16246-16250。10.1073 / PNAS.04076101。
- 57.
郑y,徐d,ug x:基因复制和序列结构关系后的功能性分歧:一种G蛋白α亚基的案例研究。j exp zool b mol dev evol。2007,308(1):85-96。10.1002 / JEZ.B.21140。
- 58.
DELLA基序在赤霉素诱导的RGA降解过程中起重要作用。中国科学(d辑:地球科学),2001,19(4):531 - 534。10.1073 / pnas.251534098。
- 59。
Cao D,Cheng H,Wu W,Soo HM,Peng J:Gibberellin调动不同的Della依赖的转录om,以调节拟南芥的种子萌发和花卉发育。植物理性。2006,142(2):509-525。10.1104 / pp.106.082289。
- 60。
Gallego-BartoloméJ,Minuet Eg,MarínJA,PRAT S,BlázquezMa,alabadíd:拟南芥中Della蛋白之间的转录多样化和功能保护。mol Biol Evol。2010,27:1247-1256。10.1093 / molbev / msq012。
- 61。
Edgar RC:肌肉:具有高精度和高吞吐量的多个序列对齐。核酸RES。2004,32(5):1792-1797。10.1093 / nar / gkh340。
- 62.
王志强,王志强:系统发育树重建的新方法——邻域连接法。中国生物化学学报,2017,32(4):457 - 461。
- 63.
Tamura K,Peterson D,Peterson N,Stecher G,Nei M,Kumar S:Mega5:使用最大可能性,进化距离和最大判定方法的分子进化遗传学分析。mol Biol Evol。2011,28(10):2731-2739。10.1093 / Molbev / MSR121。
- 64.
郭艾,朱庆华,陈旭,罗继成:GSDS:基因结构显示服务器。易川。2007,29(8):1023-1029。10.1360 / yc - 007 - 1023。
- 65.
Nei M,Kumar S:分子进化和系统发育。牛津:牛津大学出版社;2000年。
- 66.
谷旭:基因复制后功能差异检测的统计方法。中国生物医学工程学报。1999,16(4):593 - 598。10.1093 / oxfordjournals.molbev.a026080。
- 67.
基于系统发育学的蛋白质家族功能-结构差异分析。生物信息学。2002,18:500-501。10.1093 /生物信息学/ 18.3.500。
- 68.
Yang Z,WONG WS:尼尔森R. Bayes经验贝叶斯阳性选择下的氨基酸部位推断。mol Biol Evol。2005,22:1107-1118。10.1093 / molbev / msi097。
- 69。
杨卓:PAML 4:最大似然法的系统发育分析。中国生物化学学报,2007,24(8):1586-1591。10.1093 / molbev / msm088。
- 70。
杨铮:似然比检验在适应性分子进化检测中的应用。生物科学进展。2001,18:1585-1592。10.1093 / oxfordjournals.molbev.a003945。
- 71。
施密米,达维森Ts,Heenz SR,Pape Uj,Demar M,Vingron M,Schölkopfb,Weigel d,Lohmann Ju:Arabidopsis Thaliana开发的基因表达图。NAT Genet。2005,37(5):501-506。10.1038 / ng1543。
- 72。
Jain M, Nijhawan A, Arora R, Agarwal P, Ray S, Sharma P, Kapoor S, Tyagi AK, Khurana JP:大米中的F-box蛋白。全基因组分析、分类、穗和种子发育过程中基因的时空表达及光和非生物胁迫的调控。植物营养与肥料学报。2007,27(4):457 - 461。10.1104 / pp.106.091900。
致谢
作者要感谢中国的国家自然科学基金(30971783)和中国北京的自然科学基金(5132005)进行财政支持。
作者信息
从属关系
相应的作者
额外的信息
利益争夺
两位作者宣称他们没有相互竞争的利益。
作者的贡献
NW进行了生物信息分析并起草了稿件。YH设计了研究并为整个研究提供指导。YZ和WS参加了该研究并帮助起草了稿件。YL,YY协调研究并在手稿上阐述。所有作者阅读并认可的终稿。
电子补充材料
GRAS基因亚家族的数量
额外的文件1:拟南芥,Broachypodium distachyon.,米饭,大豆,卷柏moellendorffii,Physcomitrella patens..(Doc 36 KB)
GRAS基因亚家族的蛋白质序列数据
附加文件2:拟南芥,Broachypodium distachyon.,米饭,大豆,卷柏moellendorffii,Physcomitrella patens..(医生234 KB)
12870 _2014_373_moesm3_esm.doc
附加文件3:预测的Atgras基因和相关信息。A.aa =氨基酸;b. pI =推导多肽的等电点;c.Mw =分子量;红色方框表示内含子的相对位置。(Doc 90 KB)
12870_2014_373_moesm4_esm.doc.
附加文件4:预测的BdGRAS基因和相关信息。A.aa =氨基酸;b. pI =推导多肽的等电点;c.Mw =分子量;红色方框表示内含子的相对位置。(医生110 KB)
12870_2014_373_moesm5_esm.doc
附加文件5:预测OSGRAS基因和相关信息。A.aa =氨基酸;b. pI =推导多肽的等电点;c.Mw =分子量;红色方框表示内含子的相对位置。(Doc 116 KB)
12870_2014_373_moesm6_esm.doc
附加文件6:预测的GmGRAS基因和相关信息。A.aa =氨基酸;b. pI =推导多肽的等电点;c.Mw =分子量;红色方框表示内含子的相对位置。(医生234 KB)
12870_2014_373_moesm7_esm.doc.
附加文件7:预测的SmGRAS基因和相关信息。A.aa =氨基酸;b. pI =推导多肽的等电点;c.Mw =分子量;红色方框表示内含子的相对位置。(医生728 KB)
12870_2014_373_moesm8_esm.doc
附加文件8:预测的PpGRAS基因和相关信息。A.aa =氨基酸;b. pI =推导多肽的等电点;c.Mw =分子量;红色方框表示内含子的相对位置。(医生1 MB)
GRAS基因的染色体分布来自
附加文件9:拟南芥,Broachypodium distachyon.,米饭和大豆。染色体的大小由它的相对长度来表示。红色的基因表示串联复制基因,绿色的圆圈表示分段复制基因。定位信息和染色体信息来源于植物区系。图是用MapInspector程序生成的。(PNG 200 KB)
肝细胞GRAS基因亚家族的系统发育树数据
附加文件10:拟南芥,Broachypodium distachyon.,米饭,大豆,卷柏moellendorffii,Physcomitrella patens..(DOC 32 KB)
GRAS蛋白的ml系统发育树
附加文件11:拟南芥,Broachypodium distachyon.,米饭,大豆,卷柏moellendorffii,Physcomitrella patens..正交基因的主要簇显示在不同的颜色:I族=紫色,II组=深蓝色,III族=黄色,组IV =浅绿色,组v =粉红色,组VI =暗绿色,七级=光组蓝色的。秤条对应于每位位点0.1估计氨基酸取代。(PNG 1 MB)
GRAS蛋白的ME系统发育树
额外的文件12:拟南芥,Broachypodium distachyon.,米饭,大豆,卷柏moellendorffii,Physcomitrella patens..正交基因的主要簇显示在不同的颜色:I族=紫色,II组=深蓝色,III族=黄色,组IV =浅绿色,组v =粉红色,组VI =暗绿色,七级=光组蓝色的。秤条对应于每位位点0.1估计氨基酸取代。(PNG 1 MB)
12870_2014_373_moesm13_esm.png
附加文件13:七组GRAS蛋白的多序列比对。采用多重序列比对(Corpet 1988)对保守的GRAS结构域残基进行完全比对。GRAS蛋白共有5个保守基序:LHRI、VHIID、LHRII、PFYRE和SAW基序。绿色箭头代表16个负责积极选择的关键氨基酸残基和两种类型的功能分歧。(PNG 5 MB)
12870 _2014_373_moesm14_esm.png
附加文件14:六种物种中GRAS蛋白的多序列对准。采用多重序列比对(Corpet 1988)对保守的GRAS结构域残基进行完全比对。GRAS蛋白共有5个保守基序:LHRI、VHIID、LHRII、PFYRE和SAW基序。(PNG 7 MB)
12870 _2014_373_moesm15_esm.doc
附加文件15:通过定义的基因集群中的MEME软件鉴定的保守基序的示意性分布。在括号中代表的所有GRAS蛋白中的每个识别的主题的位置。用蓝色和红色突出显示,分别代表保守的GRAS和Della域。(DOC 260 KB)
12870 _2014_373_moesm16_esm.doc
附加文件16:来自拟南芥,Brocapodium distachyon,大米和大豆不同GRAS蛋白观察到的MEME定义基序的多级共识序列。(Doc 34 KB)
12870_2014_373_moesm17_esm.png
附加文件17:阳性选择下氨基酸位点与两种类型功能差异之间的关系。(PNG 446 KB)
拟南芥位点特异模型的参数估计和似然比检验。
附加文件18:注意:* P <0.05和** P <0.01(X2测试)。在M0、M3、M7、M8模型下估计了a ω;P和q是分布的参数。b估计已经过积极选择的氨基酸位点数目。(Doc 33 KB)
参数估计和似然比测试在特定于站点的模型中
附加文件19:Broachypodium distachyon..注:*p < 0.05及**p < 0.01 (X2测试)。在M0、M3、M7、M8模型下估计了a ω;P和q是分布的参数。b估计已经过积极选择的氨基酸位点数目。(DOC 37 KB)
大米地点特定模型的参数估计和似然比测试。
附加文件20:注意:* P <0.05和** P <0.01(X2测试)。在M0、M3、M7、M8模型下估计了a ω;P和q是分布的参数。b估计已经过积极选择的氨基酸位点数目。(Doc 33 KB)
大豆网站特定模型的参数估计和似然比测试。
附加文件21:注意:* P <0.05和** P <0.01(X2测试)。在M0、M3、M7、M8模型下估计了a ω;P和q是分布的参数。b估计已经过积极选择的氨基酸位点数目。(Doc 36 KB)
参数估计和似然比测试在特定于站点的模型中
附加文件22:卷柏moellendorffii.注:*p < 0.05及**p < 0.01 (X2测试)。在M0、M3、M7、M8模型下估计了a ω;P和q是分布的参数。b估计已经过积极选择的氨基酸位点数目。(Doc 33 KB)
参数估计和似然比测试在特定于站点的模型中
附加文件23:Physcomitrella patens..注:*p < 0.05及**p < 0.01 (X2测试)。在M0、M3、M7、M8模型下估计了a ω;P和q是分布的参数。B估计的氨基酸位点的数量估计是阳性选择,氨基酸是指PP1S84_112V6序列。(Doc 34 KB)
12870_2014_373_moesm24_esm.png
附加文件24:拟南芥各种器官中GRAS基因的表达。基因名称显示在每行的右侧。用于表示表达级别的颜色方案是红色/蓝色:蓝色框表示低表达式,红色框表示高表达式。(PNG 9 KB)
12870_2014_373_moesm25_esm.png
附加文件25:克拉斯基因在稻米各种器官中的表达。基因名称显示在每行的右侧。用于表示表达级别的颜色方案是红色/蓝色:蓝色框表示低表达式,红色框表示高表达式。(PNG 12 KB)
12870_2014_373_moesm26_esm.png.
附加文件26:大豆各种器官中GRAS基因的表达。基因名称显示在每行的右侧。用于表示表达级别的颜色方案是红色/蓝色:蓝色框表示低表达式,红色框表示高表达式。(PNG 23 KB)
作者为图像提交的原始文件
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。
除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。
要查看本许可证的副本,请访问https://creativecommons.org/licenses/by/4.0/.
Creative Commons公共领域奉献豁免(https://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
吴,N.,朱,Y.,宋,W.等等。植物GRAS转录因子超家族亚组的异常串联扩增和阳性选择。BMC植物杂志14,373(2014)。https://doi.org/10.1186/s12870-014-0373-5
收到了:
接受:
发表:
关键词
- ᅟ