摘要
背景
西部白松(wp,松果体monticolaDouglas ex . Don)在森林育种和保护中具有很高的兴趣,因为它对由真菌引起的入侵性疾病白松水泡锈病(WPBR)具有很高的敏感性柱锈菌属ribicolaJ. C. Fisch)。然而,WWP缺乏基因组资源开发,在进化上与已有基因组草图序列的植物距离较远。在这里,我们报告了一项单核苷酸多态性(SNP)的研究,该研究基于散装分离RNA-Seq分析。
结果
利用抗性种质资源构建cDNA文库并进行SNP基因分型。每个库获得约3600 - 8900万份2 × 100-bp的阅读新创组装产生了第一个包含54,661个独特转录本的茎尖参考转录组。生物信息学SNP检测在松树高度保守基因(hcg)、植物防御反应差异表达基因(DEGs)和抗性基因类似物(RGAs)三个候选基因组中鉴定出>10万个高质量SNP。估计效率in-silico使用Sequenom iPlex开发了SNP发现,基因分型检测,SNP成功率从40.1%到61.1%。SNP聚类分析一致显示不同的群体,每个群体由多个全兄弟姐妹种子科组成,在WWP种质资源收集中通过亲代分配。连锁不平衡(LD)分析发现6个基因与主基因(Cr2)抗性,包括三个RGAs(两个NBS-LRR基因和一个受体样蛋白激酶-RLK基因),两个hcg和一个DEG。至少一个SNP位点提供了一个很好的标记Cr2选择在p . monticola人群。
结论
WWP茎尖转录组和这些已验证的SNP标记为遗传、进化和生态研究提供了新的基因组资源。这些与抗性表型相关的候选基因的SNP位点可作为进一步鉴定WWP主基因抗性的位置和功能变异位点c . ribicola.我们的研究结果表明,整合基于rna序列的转录组分析和高通量基因分型是发现大量核苷酸变异和鉴定与非模式物种适应性性状相关的功能基因变异的有效方法。
背景
西部白松(wp,松果体monticola道格拉斯(Douglas ex D. Don)是一种重要的经济和生态树种,广泛分布在北美西部。由于对外来真菌引起的白松水泡锈病(WPBR)的易感,世界自然保护区面临着严重的保护挑战柱锈菌属ribicolaJ.C.费。,its high vulnerability to other disturbance agents including the mountain pine beetle (Dendroctonus ponderosae)和火灾,两者都因气候变化而加剧[1].今天,主要由于WPBR,p . monticola存在于碎片化的种群中,占该物种历史景观的不到10% [2]、[3.].其他五针松,如白皮松(p . albicaulis恩格尔姆)和软松木(p . flexilisE.James),亦面临类似的保育挑战[4].虽然在若干操作项目中正在开发水蚤及其他相关物种对水蚤br的遗传抗性[5],更好地了解五针松的遗传多样性、群体结构、基因流和抗病虫害能力,对其合理管理、保护和恢复至关重要。
在过去十年左右的时间里,分子标记已被开发出来,并用于促进保护和WPBR抗性育种计划[6].对扩增片段长度多态性(AFLP)标记的分析表明,不同地理区域WPBR的疾病压力和由不同气候导向的选择影响了WPBR群体的遗传多样性[7] - [9].几种AFLP标记已被证明与WWP主基因紧密相连(Cr2)抗WPBR [10].最近,通过对适应宿主防御反应的候选基因进行pcr测序,研究了核苷酸多样性[11].关联遗传学的进展已使一些与定量抗病性状相关的候选基因的单核苷酸多态性(SNP)和简单序列重复(SSR)标记得到鉴定[12]、[13].尽管取得了这些进展,但对于wpp和其他五针松来说,基因组资源的应用,如高通量标记(SNPs和SSRs)和基因分型阵列,仍然非常稀缺,因为这些物种在进化上与少数具有可用的草图基因组序列和相关基因组信息的针叶松相当遥远[14]、[15].
为制定有效的、长期的水自然保护区和水生物群落管理战略,需要进行持续的研究,以增进对气候和环境因素在变化和形成过程中所产生的影响的了解p . monticola人群。为了实现这一目标,并使用廉价的高通量技术对个体基因型进行现实的评分,需要大量易于在大量WWP群体中评分的分子标记。虽然SNP标记在基因组中丰富,并有潜力成为这些研究目标的优秀工具,但迄今为止还没有用于WWP的SNP数据库或SNP阵列。
用于高通量SNP发现和基因分型的下一代测序(NGS)策略包括限制性位点相关DNA标签- RAD [16],基因分型- GBS [17],以及多重霰弹枪基因分型- MSG [18].RNA-seq也是发现大量DNA标记的重要基因组技术,包括转录组水平的SNP和SSR。由于RNA-seq产生针对外显子的短cDNA序列,主要是在蛋白质编码区域,与表型性状相关的DNA变异比使用基于基因组DNA的方法更容易与候选基因的功能表征的生物学作用联系起来。RNA-seq在生态和进化研究中有着广泛的应用,它非常适合于通过揭示种群间基因表达模式的差异来理解物种形成和生态类型特异性适应[19]
本研究的目的是:1)研究抗性种质树芽组织的转录组特征;2)基于候选基因方法开发SNP标记;3)将高通量SNP基因分型应用于水野生动物保护和育种项目的谱系重建和抗性筛选。我们使用RNA-seq在转录组中发现SNP新创由茎尖组织根据主基因抗性的散装分离组装而成(Cr2 / -)和易感性(cr2 / cr2)c . ribicola.基于松树抗病相关候选基因和高度保守基因(hcg)设计了SNP检测方法。通过对抗性种质资源的高通量基因分型,这些snp得到了验证,改进了用于WWP和其他五针松的基因组工具。
结果
新创茎尖转录组的组装
从代表WPBR抗性和敏感基因型的聚合RNA样本中构建6个cDNA文库,使我们能够生成并获得在茎尖组织中的转录组的整体视图p . monticola.从6个cDNA文库中共收集到3.486亿个100 bp对端reads,每个文库大约有3320 - 8980万个对端(PE) reads。共生产了95,727个独特的contigs, N50为920 bp,平均长度为630 bp新创装配1.23亿RNA-seq 100-bp PE从三个cDNA文库构建的抗性组织(附加文件1:表S1)。
在Pinus基因索引(PGI)数据库中,从读计数≥50 / contig或读计数< 50 / contig但BLASTn E < 10 E -10的装配中提取了54,661个转录本(附加文件)1:表S1)。所有这些contigs均作为茎尖参考转录组进行进一步分析,其总长度为46 Mb, N50为1376 -bp,平均长度为843-bp(附加文件1:表S1)。对茎尖参考转录组的BLASTn分析显示,它含有21,930个contigs(占总数的40.10%),因为它们在PGI数据库中显示了相同的命中值(E值< 10 E -100)。从这个参考转录组中,TransDecoder共预测了41460个蛋白质,最小蛋白质长度为50。在所有假定的蛋白质中,14,287个(占总数的30.7%)是假定的完整蛋白质序列(附加文件)1:表S2)。含有54,661个contigs的WWP茎尖参考转录组已存入DDBJ/EMBL/GenBank,编号GBQX01000000。
在从WWP茎尖参考转录组中推测的41460个蛋白质中,79.4%和61.5%的蛋白质与PGI数据库和火炬松(p . taeda)基因组数据库(E < 10e-6的tBLASTn或BLASTp)。tBLASTn搜索p . taeda蛋白质数据库(包括64809个假定的蛋白质序列)对WWP序列进行了比对,结果显示92.9%的序列在WWP的茎尖参考转录组中有显著的同源命中(E < 10e-6)1:表S3)。相比之下,只有830份WWP茎尖转录本(占总数的1.5%)在杨树叶锈病真菌(Melampsora laricis-populina)基因组(BLASTx E值< 10 E -100),提示耐药组织中存在罕见的真菌感染(附加文件)1:表S3)。
基因注释
利用BLAST2GO对WWP茎尖参考转录组中的54,661个转录本进行了基因本体分析,其中56.7%的转录本在NCBI nr数据库中显示了显著的BLASTx命中。除一种真菌外,所有BLAST高发物种均为植物葡萄孢菌,云杉sitchensis占总contigs的24.9%b .灰质仅占总contigs的0.2%(附加文件2:图S1),表明污染在WWP茎尖参考转录组的数据集中不是一个严重的问题。26,831个contigs(占总数的49.1%)被分配到至少一个GO项,其中6,327个编码了假定的酶。与WWP主要针组参考转录组相比[15],发现一系列GO术语类别显著富集,一般来说,这些类别下的序列在茎尖组织中显著过代表(附加文件3.:图S2)。例如,在“响应生物刺激”类别中,茎尖组织中表达了1170个基因,而初生针叶组织中仅表达了465个基因,表明这两种类型的水磷组织在基础防御方面存在差异。
通过BLASTx比对之前克隆的128个wpp RGAs,在茎尖参考转录组中鉴定出745个contigs为抗性基因类似物(RGAs),编码具有核苷酸结合位点和富亮氨酸重复序列(NBS-LRR)结构域的蛋白质。10].一组差异表达基因(DEGs)在p . monticola针状组织在宿主防御中的反应c . ribicola早期感染[15],在茎尖中检测到740个表达。我们选择hcg、RGAs和DEGs这三组基因作为参考序列进行RNA-seq reads的作图,以进一步发现SNP。
SNP的发现和表征
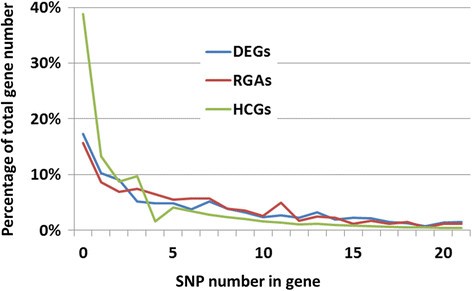
使用CLC Genomics Workbench 5.1将6个cDNA文库的PE reads映射到参考序列,将2043个indels, 2857个多核苷酸变体(MNV)和104452个双等位基因SNP映射到41460个推测的蛋白质编码区域,57,139个SNP(54.7%)导致氨基酸变化(非同义SNP)。我们还在HCG组中检测到97,063个SNPs,在DEG组中检测到7,248个,在RGA组中检测到6,078个1:表S4)。这些SNP共106,399个,分布在14,730个contigs中,平均每263-bp有一个SNP。hcg的SNP密度最低,为1个SNP / 285 bp(0.35%)。DEGs和RGAs具有中高SNP密度,分别为每126 bp和81 bp 1个SNP(0.79%和1.23%)。在hcg、DEGs和rga候选组中,多态性基因分别占总基因的61%、83%和84%(图1,和附加文件1:表S4)。
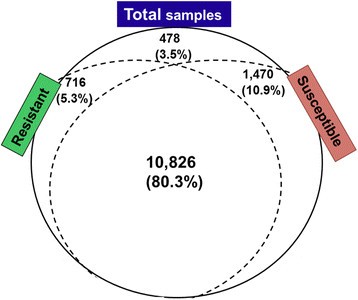
共13490个hcg有多态性。结果表明,80.3%(10826个)的hcg在抗性和敏感样品中都有多态性,10.9%(1470个)的hcg只在敏感幼苗中有多态性,5.3%(716个)的hcg只在抗性幼苗中有多态性。其余3.5%(478)的多态hcg是纯合的,但它们的等位基因在抗性和敏感样本之间是不同的(图2).仅在抗性或易感苗中存在的SNP位点被认为是基因分型鉴定耐药性状相关DNA标记的最高优先级SNP位点。
SNP基因分型
两种不同的基因分型检测共检测了432个in-silico利用Sequenom iPlex技术检测376株幼苗的SNP位点。在第一个(第一个)SNP阵列中,由于缺失数据太多,从基因分型分析中删除了9个基因组DNA样本,导致样本量n = 179。对三种基因型(A/B/H)的每个SNP位点进行分析发现,有301个SNP(69.7%)被成功分型,而剩余的131个SNP(30.3%)由于在超过20%的样本中缺失基因型数据而被评分为“失败”;PCR扩增效果差,信号强度低,导致数据缺失。
如表所示1,在进行信号基因分型的301个SNP位点中,74个(24.6%)为单型SNP,另外227个(占301个基因分型SNP的75.4%)在基因分型样本中被验证为多态性(其引物和探针序列列在附加文件中4:表S5)。对于每个SNP位点,在Hardy-Weinberg平衡(HWE)下观察到的(Ho)和期望的杂合度(He)水平,以及偏离HWE检验的显著性水平显示在附加文件中4:表S6。在第1组中有45个SNP标记,在第2组中有33个SNP标记被鉴定出明显偏离HWEPbonferroni校正< 0.05,可能是由于从自然居群中育种选择抗性种质所致。
在两个基因分型阵列中,多态位点的小等位基因频率(MAF)和Ho分布相似(附加文件)5:图S3和附加文件6:图S3)。所有227个多态性SNP标记的平均Hos分别为0.529±0.2414,第一组和第二组的平均Hos分别为0.446±0.178。候选组hcg的转化成功率最高in-silicoSNP位点转化为SNP标记(61.1%),而rga候选组的这一比例仅为40.1%。共有215个SNPs的MAF值为> 0.05。排除Ho水平最高的12个snp进行群体遗传学分析。因此,使用203个SNP位点组成的最终基因型数据集进行谱系重建和LD分析。
种群结构与全兄弟姐妹关系重建
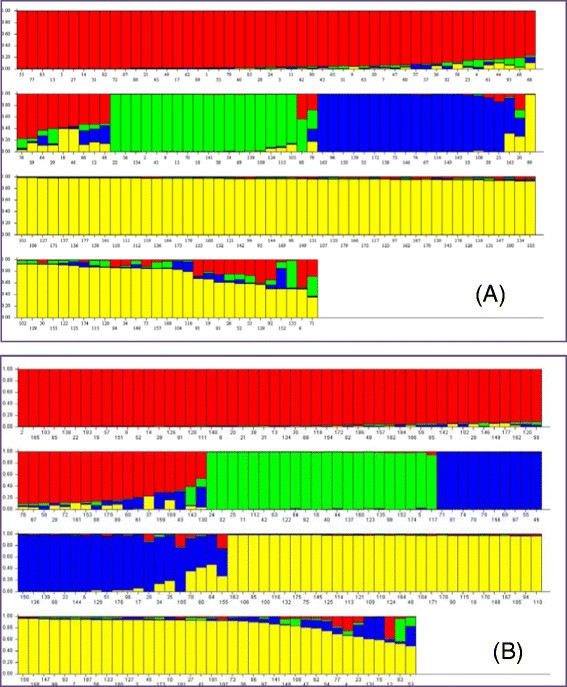
主成分分析(PCA)表明,前三个主成分解释了大约60%的总变异和明确的祖先集群显示在收集的样本(附加文件7:图S4)。structure中基于模型的贝叶斯聚类方法对种群结构的调查表明,使用ΔK计算,最可能的聚类数量(K)为4(附加文件)8:图S5)。在第1和第2个SNP阵列中,通过两次不同的采样,在抗性种质中一致发现了四个遗传簇(图3.).
采用最精确的全似然方法,利用COLONY重建兄弟姐妹关系和亲缘关系,我们发现第1个SNP序列中的179株幼苗和第2个SNP序列中的188株幼苗分别被分配到35个和36个全兄弟姐妹种子家族。两种SNP检测均显示出三个最丰富的种子家族,每个家族占总基因分型样本的>10%(附加文件)2:表S7)。这些结果在很大程度上得到了从育种项目中收集的抗性种质中这些幼苗的已知谱系和来源的支持。成员最少的种子家族只分配一株幼苗。
连杆不平衡分析
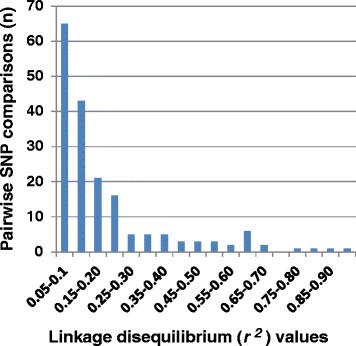
共比较了11,139个SNP对的LD估计。卡方检验(在P< 0.05)显示有962对SNP(占总数的8.6%)有显著的LD估计,但这对SNP数量减少到183对(占总数的1.6%),平均LD估计在r2多次试验高度保守Bonferroni校正后= 0.2(图4).当主要基因抗性基因型(Cr2 / -vs。cr2 / cr2)在LD分析中被考虑,我们在显著的LD中检测到21个SNPs(每个来自一个独特的基因)Cr2.经Bonferroni校正后,6个基因仍表现出显著的LDsCr2包括3个RGAs(2个NBS-LRR基因和1个RLK基因),2个hcg和1个DEG(表2).尽管不知道它们的遗传距离,但具有显著LD的SNP位点可能在同一染色体上共享位置。DEG A05_contig_4105的SNP位点与主基因(Cr2)阻力(r2= 0.81,P= 2.6 e-39)。其中CC、GC和GG基因型分别占抗性苗总数的26.1%、68.5%和5.4%;敏感苗分别占总敏感苗的0%、4.6%和95.4%。因此,这个SNP位点是一个很好的标记Cr2- WWP种质中四个居群的抗性选择。
讨论
通过改进的RNA-seq方法发现SNP
由于不需要预先存在的基因组序列数据,RNA-seq已被证明在发现非模式物种中的新基因、转录本、rna、可选剪接连接、融合序列和核苷酸变异(如SNP和SSR)方面具有越来越广泛的应用[20.] - [23].通过将常规RNA-seq与散装分离分析相结合,我们证明了这种方法是选择具有高潜力的snp的有效策略,可以在WWP转录组水平上识别与适应性性状相关的DNA变异。最近的一项研究发现,通过RNA-seq方法准确预测等位基因频率需要15个个体[24].巧合的是,我们的工作使用了6个bulk样本(每个样本来自15个个体),在一系列严格的检测标准下,通过对三组候选基因进行3.486亿RNA-seq reads的映射,总共恢复了约10万个高质量snp。这些小说的可用性in-silicosnp将为这一重要针叶树种的育种和保护计划提供大量的DNA标记。
Sequenom iPlex被报道为高度可靠的高通量SNP基因分型平台之一,具有广泛的应用[25]、[26].由于这项技术更具成本效益和灵活性,我们将其用于WWP SNP基因分型。SNP标记的转化率从in-silico已报道了海松验证位点的snp (p .松树)(42.5%)、黑松(大榄)(30.0%)、阿勒颇松(p . halepensis(76.6%)及花旗松(72.5%)[27] - [30.].目前的研究显示,平均转化率为52.5%p . monticola.HCG组的转换率高得多,为61.1%;这一速率与其他树种的基因组重测序所挖掘的SNPs相当[31]、[32].SNP标记转换率的变化建议标准in-silicoSNP的选择和基因分型设计,以及基因分型平台的类型是重要的。例如,in-silico严格质量标准的snp挖掘过程可以区分序列变异和人工测序。这是可能的转化率in-silico通过优化引物设计和PCR扩增条件,我们发现一些iPlex失败的SNPs可以通过TaqMan和HRM等qPCR基因分型方法进行正确的基因分型(Liu,未发表数据)。通过外显子组测序识别外显子-内含子边界将改进SNP基因分型阵列的设计。此外,与样本池策略相比,NGS对单个样本,特别是对针叶树单倍体大配子体样本的SNP检测有可能提高整体的置信度in-silicoSNP检测。
我们的工作展示了如何将基于隔离的大体积RNA-seq与高通量SNP阵列相结合,能够快速、经济且可靠地识别数十万个基因中最具信息量(人群特异性)的标记in-silico单核苷酸多态性。我们相信,这种检测信息量最大的SNPs的经济有效的方法可以很容易地适应并应用于其他非模式针叶松,包括五针松物种(例如,p . albicaulisEngelm和p . flexilisE.James),
候选基因SNP阵列
在本研究中,我们展示了基于候选的方法的效用,以选择可用的子集in-silicosnp:首先,基于rna序列的转录组分析确定了WWP候选基因(如RGAs和DEGs),它们在遗传抗性和宿主对害虫、病原体和环境胁迫的防御方面具有潜在的生物学功能;第二,转录组分析也揭示了针叶树物种中高度保守的基因,甚至是同源基因[15].由于rga和DEGs是研究植物-微生物-环境相互作用的极好靶点,而hcg是跨相关类群比较基因组学研究的最有利选择,因此我们选择这些候选类群的snp进行高通量基因分型分析。虽然SNPs在最好的水平上代表了个体的遗传变异性,但如果有大量的SNPs可用,则没有必要在整个基因组中对所有可用的SNPs进行基因分型。选择一个信息量足够大,但仍足够小的snp子集,以实现可负担的成本和研究目标的最佳平衡,是迈向有效关联研究和基因组选择的重要一步[33].
一些基于基因的候选案例研究发现了针叶树中与数量性状相关的snp和单倍型[11]、[34]和其他植物[35] - [40].利用LD分析,在本研究中,我们发现了一个防御反应性基因A05_contig_4105与肿瘤相关Cr2基因(表2).A05_contig_4105编码一个F-box蛋白p . taeda蛋白AEW08082及其表达的转录本在抗性苗初叶中特异性上调c . ribicola感染(15].F-box蛋白包含至少一个F-box结构域,该结构域通常与其他基序相连,如lrr和色氨酸-天冬氨酸(WD)重复序列,用于与信号转导网络和其他细胞功能相关的蛋白质-蛋白质相互作用[41].
尽管有读映射覆盖率相对较低和多态级别较高的缺点,我们还是包含了in-silicoSequenom iPlex检测152个RGAs的snp。RGA SNPs的基因分型更有可能识别与抗病性性状的遗传关联,因为它们在植物先天免疫系统中具有假定的功能。植物NBS-LRR和RLK蛋白主要通过与病原体效应物的特异性相互作用在寄主抗性中发挥作用,从而触发植物防御反应,抑制病原体生长和在受感染组织内扩散[42].我们之前在NBS-LRR和RLKs中鉴定了100多个rgap . monticola通过基因组PCR克隆和几个rga相关的AFLP标记连接Cr2在基因定位人群中[10].我们发现在茎尖组织中表达了175个独特的RGA转录本,约2000个in-silico在其序列中鉴定出snp。在成功分型的96个RGAs中,有61个存在多态性1).3个多态RGAs与主基因(Cr2)在基因分型人群中进行LD分析(表2).RGA F0_contig_3186编码了一个推测的RLK蛋白,该蛋白与RLK蛋白同源性最高云杉glauca蛋白ABF73316.1(期望E = 0.0)和另外两个RGAs, F0_contig_48562和F0_contig_9161编码NBS-LRR蛋白。额外的snp,特别是上述三种rga中的非同义snp,将为进一步鉴定主要基因抗性提供位置和功能变异位点c . ribicola.大量的SNP标记,特别是候选基因中的SNP标记,对于研究北美本土白松群体在气候变化和WPBR选择压力下抗性机制的进化和适应具有重要意义。在未来,我们将对相关NBS-LRR和RLK基因的耐药和易感单倍型进行序列比较和随后的功能表征,以确定这些RGAs中是否有任何一种负责c . ribicola为表现型。
利用LD分析鉴定SNP标记进行抗性筛选
利用NGS在基因组上发现大量snp,然后对一组具有可用表型的样本进行基因分型,已经成为复杂性状精细遗传定位的标准实践。在本研究中,我们利用抗水枯病种质资源来研究基因型-表型关系。LD是等位基因在两个位点上的非随机共分离,可由许多因素引起,包括有效群体大小和结构、重组率、遗传漂变、交配系统和选择[43].同源染色体之间的重组导致LD在减数分裂期间随着两个位点之间距离的增加而衰减。一般来说,在开放授粉的植物和同一物种的更多样化的种群中,LD的衰变速度更快,但在同一物种的不同基因和基因组区域中,LD的衰变速度可能有很大差异[44].因此,LD含量的信息是任何全基因组关联研究的关键前提,以微调目标基因组区域和候选基因。
作为雌雄同株的裸子植物,松树的LD衰减速率为~500 ~ 2000 bp [45].由于针叶树的这种LD快速衰减模式,snp揭示的遗传关联可能位于致病多态性的附近[34].我们之前的研究表明LD在基因内衰变到r2在600 ~ 700 bp内估计0.3p . monticola度(11] - [13],表明相关候选基因可能具有较高的关联研究分辨率。在目前的研究中,至少有一个SNP标记被发现与Cr2在多达35个全兄弟家系的4个群体中,所测试的种质资源具有高LD(表2)2,图3.,和附加文件2:表S7)。我们认为,这些核苷酸变异可能被用作育种具有主基因抗性的WWP的选择性标记c . ribicola.其他具有显著LDs的rga和deg的SNP标记(表2)也可能非常接近或在影响抗性性状的基因内。为了证实这一假设,我们将进行一项连续的研究,以确定使用WWP遗传定位人群的染色体间和染色体内LD的程度。利用全基因组方法进行关联作图仍然需要在五株针松中积累足够的基因组资源。
抗水枯病种质的群体结构
缺乏遗传多样性和生态挑战(如栖息地破坏和环境变化)是导致种群减少和物种灭绝的两个原因。针叶树种子园通常用于生产稳定、丰富和基因改良的种子,具有良好的适应环境的性能。这些果园种子地段用于重新造林和恢复活动的物种,如WWP。不幸的是,果园种子地块通常由许多亲本通过异交和开放授粉贡献的种子组成。此外,保持适当水平的遗传多样性是至关重要的,以避免近亲繁殖和在森林种子园或种子收集中因遗传漂变而导致稀有等位基因的丢失。虽然优良种子园可以通过有利等位基因的金字塔化来发展,但有利等位基因可能分散在不同的林分/祖先中。因此,在现代保护育种中,完整的谱系信息是选择和部署精英基因型的必要前提[46].基于分子的亲本分析已被用于量化遗传多样性,并有助于防止再造林种群的近亲繁殖[47]、[48].维持像WWP这样长寿树种的再造林种群的遗传多样性是帮助确保该树种在森林和森林生态系统中继续存在的关键。
本研究的亲缘关系重建为wpp种质资源的种子科结构提供了最清晰的证据。亲子分析的准确性随着遗传位点的数量和多样性的增加而增加。流行的亲代推断方法(例如,Colony)可以有信心地应用于具有高度多态位点的自然种群[49].SNPs对于亲子推断非常有效,之前的一项研究表明,60-100个SNPs可以在大型管理和/或自然种群中实现精确的谱系重建[50].我们仔细考虑了SNP标记的数量和质量,以提高亲子关系分配的准确性。wpp亲本分配和系谱重建显示,在我们测试的复合种子批中出现了35-36个全兄弟姐妹家系。此外,在使用108个SNPs的第一个SNP试验和使用95个SNPs的第2个试验中,通过单独采样获得了一致的结果(图3.,附加文件2:表S7)。wpp育种种质资源由野生生态系统中选择的种子科组成,被证实具有强结构的复杂群体。目前这种全面的遗传特征有助于了解遗传多样性的水平和分布,并获得对现有水干旱抗性资源的遗传细分的新见解。我们的研究结果阐明了收集到的基因构成的知识p . monticola在保护计划中,我们可以根据保护价值对个体进行优先排序,以最大限度地减少遗传变异的损失,并通过确定主要亲本,在最大限度地提高基因多样性和最大限度地减少近亲繁殖之间取得平衡,从而制定育种建议。本研究的基因型数据可以有效地指导该树种在北美西部的长期管理和再造林中的进一步应用。
结论
本研究是首次在五针松中使用聚合RNA-seq方法结合散装分离分析发现候选基因SNP的研究。我们从茎尖组织中生成了新的转录组和SNP数据c . ribicola来自一个复合种子批的抗、敏感wpp种质资源。通过高通量基因分型验证了432个SNP位点,其中52.5%为多态性。利用这些SNP标记的基因型数据,在采集的wp种质资源中确定亲缘关系和遗传多样性,并鉴定SNP标记,用于wp群体抗WPBR的育种筛选。这些经过验证的SNP资源可能为五针松物种的生态基因组学和比较遗传定位开辟新的途径。
方法
植物材料
一个复合p . monticola带有'主基因(Cr2)对WPBR的hr样耐药进行了研究。该地块主要来自俄勒冈州乌姆普夸国家森林的Cottage Grove Ranger区冠军矿区的母树。这些母树组成的育种杂交是在Dorena遗传资源中心(DGRC, Cottage Grove, Oregon)建立的早期领域;在1960年代的移植中,父母对冠军矿的父母有很大的权重(Cr2 / -)和贝尔帕斯的父母(很多都有Cr2 / -)在威拉米特国家森林的熊山口种植。在繁殖植物园里也有一些来自俄勒冈州和华盛顿州其他地区的克隆。
育苗时,人工接种用c . ribicola,表型评估均在DGRC进行,如Danchok等人所述。[51].简而言之,经过4个月的分层,于2010年6月播种。幼苗在温室中生长并接种c . ribicola在2010年9月,利用受感染的Ribes spp.叶进行了研究c . ribicola),从该毒株(vcr2)。接种时,接种量平均为6000个担孢子/厘米2且孢子发芽率为89%。2011年,当针和茎感染症状明显时,定期对表型性状进行评估。每棵幼苗都被确定为抗性(Cr2 / -)或易受影响(cr2 / cr2)基于他们的针斑类型(即,都是hr样;所有的敏感;混合;未确定的疾病斑点)及其茎症状(即存在或不存在溃疡病)。针样采集于2011年7月,保存于- 20°C,用于基因组DNA提取。2011年10月(约13个月后c . ribicola使用液氮从每个基因型的幼苗亚组中收集分枝和茎组织,并在−80°C保存,直到提取RNA。
基于堆积分离的RNA-Seq分析
分别采集45株抗性和45株易感苗的茎尖,按照前面描述的方法提取总RNA [52].RNA-seq分析采用体积分离分析。总RNA样本被汇集到总共6个样本中(每个RNA样本从15个幼苗中均等地汇集):3个具有抗性(Cr2 / -)表型和三种易感(cr2 / cr2)表型。DNase (RNase-free)在37°C处理30分钟后,使用RNA-Seq样品制备试剂盒(Illumina)分离mRNA,并如前所述用于cDNA文库的构建[15]除了每个库包含样本特异性的6-bp核苷酸条形码标签。6个标记的cDNA文库以等比例汇集,在加拿大国家研究委员会(Saskaton, Canada)的Illumina HiSeq2000测序仪的一个通道上进行2 × 100 bp测序。Illumina RNA-seq 100 bp PE原始序列保存在NCBI SRA中,登录号为SRR1574690-1574692。
RNA-seq数据分析使用CLC Genomics Workbench 5.1 (CLC Bio, Cambridge, Mass, USA)进行。原始读数之前被修剪过新创转录本组装,默认设置为质量限制= 0.05,模糊限制= 2,读取的最小核苷酸数= 15。耐腐蚀的枪头(Cr2 / -)幼苗被认为没有c . ribicola这样修剪过的菌丝从三个抗(Cr2 / -)新创装配用于生成WWP茎尖转录组,图参数为自动字长和自动气泡大小,映射参数为错配代价= 2,长度分数= 0.5,相似分数= 0.8,删除或插入代价= 3,最小contig长度= 200。
来验证新创汇编质量,转录本序列中的假定开放阅读帧(orf)由TransDecoder (http://transdecoder.sourceforge.net/)蛋白质长度最少为50。假定的WWP蛋白序列与PGI数据库(77,326 contigs, Release 9.0, March 26, 2011)进行比较。http://compbio.dfci.harvard.edu/tgi/),火炬松基因组数据库(assembly v1.01, 2013年11月20日,http://pinegenome.org/).来估计被感染者的转录本c . ribicola,对WWP茎尖转录组进行了对照研究m . laricis-populina蛋白质资料库(http://genome.jgi-psf.org/Mellp1/Mellp1.download.ftp.html).
叠连群注释
如先前研究所述[15],对NCBI nr、PIR (http://pir.georgetown.edu/pirwww/),去(http://www.geneontology.org/)、UniProts (http://www.ebi.ac.uk/UniProt/),及KEGG (http://www.genome.jp/kegg/)使用BLAST2GO程序(Biobam生物信息学S.L,巴伦西亚,西班牙,http://www.blast2go.com/).WWP主针标注差异[15]和茎尖参考转录组通过Fisher精确测试评估,并使用BLAST2GO进行多次测试校正。利用BLASTn在PGI数据库中预测wpp茎尖转录组中的hcg,同源性E值≤10e-100。一套水务系统rga [10]和DEGs在宿主防御反应中的作用c . ribicola感染(15]用于预测茎尖组织中表达的候选基因c . ribicolaBLAST搜索和序列比对分析。
通过高通量基因分型发现和验证SNP
In-silico使用CLC Genomics Workbench 5.1将6个cDNA文库的RNA-seq PE reads映射回功能基因组的目标集,并在以下参数下进行基于质量的变异检测,从而进行SNP检测:窗长= 11,最大间隙和错配数= 1,最小平均质量= 20,最小中心质量= 20,最小覆盖率= 20,最小变异频率(MVF) = 30%,最大期望变异(倍性)= 2,正向和反向读均存在。只使用映射到参考序列上唯一位置的读取。为了预测每个SNP背后的突变在氨基酸水平上的影响,使用TransDecoder预测的最佳orf作为参考序列,使用CLC Genomics Workbench进行SNP检测,然后确定每个SNP为同义或非同义突变。在那些WWP ORF序列中显示出最佳匹配的snpp . taeda在蛋白质水平上通过BLAST搜索和PGI数据库进行SNP基因分型验证。
由于松树基因家族中内含子-外显子边界未知,且谬误子比例较高[53],另外,当选择snp进行基因分型阵列设计时,还考虑了严格的标准。标准包括contig SNP频率,SNP位置和3 ' -和5 ' -端的侧翼序列。在候选基因方法中发现SNP,in-silicoSNP数据使用hcg、deg和rga作为单独的映射参考。
对于SNP基因分型,基因组DNA是从属于RNA-seq分析所用的同一复合种子批次的单个幼苗的针状组织中提取的。使用FastPrep®-24仪器(MP Biomedicals, Santa Ana, CA, USA)将约100mg针头组织切成小碎片,并在液氮中均质。基因组DNA提取使用DNeasy Plant Mini试剂盒(Qiagen, Mississauga, ON, Canada)。
使用Sequenom iPlex MassARRAY平台进行高通量基因分型(Sequenom, San Diego, CA, USA) [54)在麦吉尔大学Génome Québec创新中心。分别设计了两个SNP检测,每个检测由216个SNP位点组成,并对188个幼苗(~50%抗性和~50%敏感样本)进行基因分型。几乎每个SNP都是从一个唯一的功能基因中选择的,除了第二个SNP阵列包含6个基因中的20个SNP,在同一个contig中有2到4个SNP。使用MASSARRAY®Assay Design软件设计多路分析,在6个多路分析面板中各有36个snp,设置如下参数:扩增子长度(bp):最小:80,最佳:120,最大:320;引物长度(bp):最小:16,最优:20,最大:25;延伸底漆长度(bp): min: 16, max: 31;Tm(°C): min: 45, max:100。采用Sequenom iPlex Gold试剂盒按照标准程序进行PCR反应。使用36对PCR引物池,在95°C循环15分钟,45 ×(95°C 20秒,56°C 30秒,72°C 60秒),72°C最终延伸3分钟的条件下,扩增出约20 ng的基因组DNA。使用虾碱性磷酸酶去除所有未结合的dNTPs。在探针的单碱基扩展后,使用纳米分配器在Sequenom 384孔芯片上发现了产物,并用质谱仪读取了芯片。 Genotypes for each SNP marker in each sample were analyzed by the MassARRAY Analyzer 4 System. Sequence and nucleotide variation of verified SNP markers have been submitted to GenBank dbSNP databases (GenBank: ss#947846159 – ss#947846384).
SNP基因型数据分析
手工评估样本收集中每个SNP位点的SNP基因分型质量。利用GenAlex 6.41计算MAF、Ho、He等snp的群体特征及其与HWE的偏差[55].排除调用率低于总样本80%、MAF低于0.05、杂合度低于5%的snp进行进一步分析。
采用主成分分析(PCA)和贝叶斯系统发育方法(Bayesian)鉴定复合种子批中是否存在群体结构/分组。根据每个幼苗的SNP基因型将SNP数据转换为等位基因频率,并使用TASSEL中SNP等位基因频率的方差-协方差矩阵进行主成分分析[56].在软件包中,假设种群内Hardy-Weinberg平衡和连锁平衡,使用基于贝叶斯模型的聚类算法将幼苗分配到祖先聚类中。57].无混合模型假设每个个体只来自一个聚类,用于SNP单倍型分析,有50,000个烧伤长度和500,000个重复。进行了20次模拟,K值设置为1到10,以估计聚类数量(K)。然后使用DeltaK方法确定最可能的聚类数量[58].
结构分析的个体分配可以将不同的种子科归为一个群体。利用COLONY程序中最精确的全系谱似然方法,对WWP种质进行了亲本分析和亲本重建[59].
卡方检验用于评价所有snp成对组合之间的LD。SNP位点与主基因表型性状的关系(Cr2)通过观察LD值(r2)与抗性表型(Cr2 / -vs。cr2 / cr2)使用上文所述的流苏[11].对于多个snp,加权平均值为r2在每个SNP对之间计算[60].P值为r2均采用Bonferroni校正[61].
支持数据的可用性
支持本文结果的数据集包含在本文及其附加的支持信息文件中。Illumina原始序列存放在NCBI GenBank SRA中,登录号为SRR1574690-1574692。含有54,661个contigs的WWP茎尖参考转录组保存在GenBank,编号为GBQX01000000。SNP标记存放在GenBank,登录号为ss#947846159 - ss#947846384。
附加文件
缩写
- 妊娠:
-
扩增片段长度多态性
- 度:
-
植物防御反应中的差异表达基因
- 他:
-
期望杂合度水平
- 砍:
-
哈迪温伯格平衡
- 何:
-
观察杂合度水平
- LD:
-
连锁不平衡
- 加:
-
小等位基因频率
- NBS-LRR:
-
核苷酸结合位点和富亮氨酸重复序列
- 主成分分析:
-
主成分分析
- 促:
-
松高度保守基因
- RGA:
-
抗性基因模拟物
- RLK:
-
受体样蛋白激酶
- SNP:
-
单核苷酸多态性
- 苏维埃社会主义共和国:
-
简单序列重复
- WPBR:
-
白松锈病
- WWP:
-
西洋白松
参考文献
- 1.
Loehman RA, Clark JA, Keane RE:气候变化和火灾管理对西部白松的影响(松果体monticola)在美国落基山脉北部。森林科学,2011,2:832-860。10.3390 / f2040832。
- 2.
Jain T, Graham R, Morgan P:西部白松生长相对于森林开口。中华民国宪法,2004,34:2187-2198。10.1139 / x04 - 094。
- 3.
Harvey AE, Byler JW, McDonald GI, Neuenschwander LF, Tonn JR:《生态系统的死亡:二十世纪末北美西部白松生态系统的展望》。美国柯林斯堡:Gen TechRep RMRS-GTR-208。美国农业部、林业局、落基山研究站;2008:10。
- 4.
Tomback DF, Achuff P:水泡锈病与西部森林生物多样性:白松的生态学、价值与展望。路径学报,2010,40:186-225。10.1111 / j.1439-0329.2010.00655.x。
- 5.
Fins L, Byler JW, Ferguson D, Harvey A, Mahalovich MF, McDonald G, Miller D, Schwandt J, Zack A:巨人的回归:恢复西部白松到内陆西北。林学学报,2002,100:20-26。
- 6.
Richardson BA, Ekramoddoulah AK, Liu J-J, Kim M-S, Klopfenstein NB:白松泡锈病病理系统的分子研究现状和未来。生物工程学报。2010,40:314-331。10.1111 / j.1439-0329.2010.00660.x。
- 7.
Kim M-S, Brunsfeld SJ, McDonald GI, Klopfenstein NB:白松水泡锈病的影响(柱锈菌属ribicola)和西部白松遗传变异的防锈育种(松果体monticola).应用理论与实践,2003,26(3):344 - 344。
- 8.
Kim M-S, Richardson BA, McDonald GI, Klopfenstein NB:西部白松遗传多样性与结构(松果体monticola):一项关于保护、恢复和应对气候变化影响的基线研究。植物基因组学报,2011,7:11-21。10.1007 / s11295 - 010 - 0311 - 0。
- 9.
Richardson BA, Rehfeldt GE, Kim M-S:基于分子标记和数量性状的西部白松气候相关基因生态响应(松果体monticola).植物学报,2009,30(1):1 - 4。10.1086/605870。
- 10.
刘金杰,Ekramoddoullah AK:富亮氨酸重复序列多态性、扩增片段长度多态性和序列特征扩增区域标记的研究进展柱锈菌属ribicola西部白松抗性基因Cr2 (松果体monticola).植物基因组学报,2008,4:601-610。10.1007 / s11295 - 008 - 0135 - 3。
- 11.
刘建杰,斯涅兹科,艾克拉莫杜拉:小说的联想松果体monticola几丁质酶基因(PmCh4B)对柱锈菌属ribicola.植物病理学杂志,2011,30(4):344 - 344。10.1094 /发朵- 10 - 10 - 0282。
- 12.
刘剑杰,Hammett C, Sniezko RA:松果体monticolapathogenesis-related基因PmPR10-2白松泡锈病茎秆定量抗病等位基因(柱锈菌属ribicola).植物基因组学报,2013,9:397-408。10.1007 / s11295 - 012 - 0561 - 0。
- 13.
刘俊杰,Zamany A, Sniezko RA:抗微生物肽(AMP)的核苷酸变异、表达及其与白松泡锈病抗性的关系。植物学报,2013,37(3):344 - 344。10.1007 / s00425 - 012 - 1747 - 2。
- 14.
Neale DB, Langley CH, Salzberg SL, Wegrzyn JL:树木基因组的开放获取:通往更好森林的道路。中国生物医学工程学报,2013,14:1-8。10.1186 / gb - 2013 - 14 - 1 - r1。
- 15.
刘金杰,Sturrock RN, Benton R:转录组分析松果体monticola通过RNA-seq分析初生针对宿主的抗性提供了新的见解柱锈菌属ribicola.中国生物医学工程学报,2013,30(4):344 - 344。
- 16.
Baird NA, Etter PD, Atwood TS, Currey MC, Shiver AL, Lewis ZA, Selker EU, Cresko WA, Johnson EA:使用测序RAD标记快速发现SNP和遗传图谱。公共科学学报,2008,3:e3376-10.1371/journal. pone.net .0003376。
- 17.
Elshire RJ, Glaubitz JC, Sun Q, Poland JA, Kawamoto K, Buckler ES, Mitchell SE:高多样性物种的基因分型方法。公共科学学报,2011,6:e19379-10.1371/journal.pone.0019379。
- 18.
杨晓明,王晓明,王晓明,王晓明,王晓明。基于基因分型的基因定位方法研究。中国生物医学工程学报,2011,29(4):344 - 344。10.1101 / gr.115402.110。
- 19.
Wolf JBW:转录组分析和基因表达定量的原理:RNA-seq教程。生物化学学报,2013,13:559-572。10.1111 / 1755 - 0998.12109。
- 20.
王志强,王志强,王志强:RNA-Seq:转录组学的革命性工具。中国科学,2009,10:57-63。10.1038 / nrg2484。
- 21.
杨世生,涂志军,徐文文,兰文杰,郑海杰,万斯CP, Gronwald JW:利用RNA-Seq对两种具有不同细胞壁组成的紫花苜蓿基因型进行基因鉴定、多态性检测和转录谱分析。中国生物医学工程学报,2011,29(4):344 - 344。
- 22.
王丽娟,王丽娟,王丽娟,王丽娟,王丽娟:虹鳟鱼生长性状的SNP标记。公共科学学报,2012,7:e36264-10.1371/journal.pone.0036264。
- 23.
Quinn EM, Cormican P, Kenny EM, Hill M, Anney R, Gill M, Corvin AP, Morris DW: RNA-Seq数据中SNP检测策略的开发:应用于淋巴母细胞系和使用1000个基因组数据进行评估。公共科学学报,2013,8:e58815-10.1371/journal.pone.0058815。
- 24.
Schunter C, Garza JC, Macpherson E, Pascual M:非模式鱼中RNA-seq数据的SNP发育:精确的等位基因频率预测需要多少个个体?生物化学学报,2014,14:157-165。10.1111 / 1755 - 0998.12155。
- 25.
Apinjoh TO, Anchang-Kimbi JK, Njua-Yafi C, Mugri RN, Ngwai AN, Rockett KA, Mbunwe E, Besingi RN, Clark TG, Kwiatkowski DP, Achidi EA, MalariaGEN Consortium:细胞因子和toll样受体基因多态性与喀麦隆三个地区严重疟疾的关联。公共科学学报,2013,8 (11):e81071-10.1371/journal.pone.0081071。
- 26.
Mitchell AL, Macarthur KD, Gan EH, Baggott LE, Wolff AS, Skinningsrud B, Platt H, Short A, Lobell A, Kämpe O, Bensing S, Betterle C, kasperli - zaluska A, Zurawek M, Fichna M, Kockum I, Nordling Eriksson G, Ekwall O, Wahlberg J, Dahlqvist P, Hulting AL, Penna-Martinez M, Meyer G, Kahles H, Badenhoop K, Hahner S, Quinkler M, Falorni A, phips - green A, Merriman TR,等:欧洲队列中自身免疫性艾迪森病与STAT4和GATA3等位基因的关联。公共科学学报,2014,9 (3):e88991-10.1371/journal.pone.0088991。
- 27.
Lepoittevin C, Frigerio JM, Garnier-Géré P, Salin F, Cervera MT, Vornam B, Harvengt L, Plomion C:在体外vs在网上为基因分型阵列的发展检测snp:我们能从非模式物种中学到什么?公共科学学报,2010,5:e11034-10.1371/journal.pone.0011034。
- 28.
Cullingham CI, Cooke JEK, Coltman DW:渗透对两种重要生态和经济针叶树种遗传群体结构的影响:黑松(松果体contortavar。latifolia)和杰克·派恩(松果体banksiana).中国生物医学工程学报,2013,29(3):344 - 344。10.1139 / gen - 2013 - 0071。
- 29.
Howe GT, Yu J, Knaus B, Cronn R, Kolpak S, Dolan P, Lorenz WW, Dean JF:杉木的SNP资源:新生转录组组装和SNP检测与验证。中国生物医学工程学报,2013,30(4):357 - 357。
- 30.
Pinosio S, González-Martínez SC, Bagnoli F, Cattonaro F, Grivet D, Marroni F, Lorenzo Z, Pausas JG, Verdú M, Vendramin GG:一种分布广泛的环地中海树种的转录组和新基因组工具的开发,松果体halepensis轧机。分子生态学报,2014,14(4):846-856。10.1111 / 1755 - 0998.12232。
- 31.
Pavy N, Pelgas B, Beauseigle S, Blais S, Gagnon F, Gosselin I, Lamothe M, Isabel N, Bousquet J:通过设计高多重SNP阵列增强复杂基因组的遗传定位:应用于白云杉和黑云杉的大型和未测序基因组。中国生物医学工程学报,2008,9:21-10.1186/1471-2164-9-21。
- 32.
Eckert AJ, Pande B, Ersoz ES, Mark H, Wright MH, Rashbrook VK, Nicolet CM, Neale DB:火火松单核苷酸多态性的高通量基因分型与定位(松果体taedal .)。植物基因组学报,2009,5:225-234。10.1007 / s11295 - 008 - 0183 - 8。
- 33.
Halperin E, Kimmel G, Shamir R:基因型数据中标签SNP选择以最大限度地提高SNP预测精度。生物信息学,2005,21:195-203。10.1093 /生物信息学/ bti1021。
- 34.
González-Martínez SC, Wheeler NC, Ersoz E, Nelson CD, Neale DB:关联遗传学在松果体taedaL. I.木材特性。遗传学报,2007,34(3):349 - 349。10.1534 / genetics.106.061127。
- 35.
Bundock PC, Eliott FG, Ablett G, Benson AD, Casu RE, Aitken KS, Henry RJ:利用454测序在高多倍体植物物种中发现靶向单核苷酸多态性(SNP)。中国生物工程学报,2009,7:347-354。10.1111 / j.1467-7652.2009.00401.x。
- 36.
Hendre PS, Kamalakannan R, Varghese M:在筛选的候选基因中高通量和平行SNP的发现桉树camaldulensis使用Illumina NGS平台。中国生物工程学报,2012,30(4):344 - 344。10.1111 / j.1467-7652.2012.00699.x。
- 37.
Kharabian-Masouleh A, Waters DLE, Reinke RF, Henry RJ:利用聚合DNA扩增和深度并行测序发现水稻种质淀粉相关基因的多态性。中国生物工程学报,2011,29(4):344 - 344。10.1111 / j.1467-7652.2011.00629.x。
- 38.
杨晓明,陈晓明,陈晓明,杨晓明,杨晓明。桉树次生代谢产物生物合成途径基因的SNP多样性研究。中国生物医学工程学报,2009,10:452-10.1186/1471-2164- 10452。
- 39.
Schneeberger K, Weigel D:新测序技术使遗传学快速发展。植物科学进展,2011,16:282-288。10.1016 / j.tplants.2011.02.006。
- 40.
Tollenaere R, Hayward A, Dalton-Morgan J, Campbell E, Lee JRM, Lorenc MT, Manoli S, Stiller J, Raman R, Raman H, Edwards D, Batley J: Rlm4黑腿抗性候选基因的鉴定与鉴定芸苔属植物显著使用下一代测序技术。中国生物工程学报,2012,30(4):357 - 357。10.1111 / j.1467-7652.2012.00716.x。
- 41.
Kipreos ET, Pagano M: F-box蛋白家族。基因组生物学,2000,1:reviews 3002-10.1186/gb-2000-1-5-reviews3002。
- 42.
Jones JD, Dangl JL:植物免疫系统。自然科学,2006,44(4):323-329。10.1038 / nature05286。
- 43.
Rafalski A, Morgante M:玉米和人类:两个相似大小基因组的重组和连锁不平衡。植物学报,2004,20:103-111。10.1016 / j.tig.2003.12.002。
- 44.
Khan MA, Korban SS:森林树木和水果作物的关联映射。应用物理学报,2012,32(3):344 - 344。10.1093 / jxb / ers105。
- 45.
Neale DB, Ingvarsson PK:森林树木适应的种群、数量和比较基因组学。植物学报,2008,11:149-155。10.1016 / j.pbi.2007.12.004。
- 46.
El-Kassaby YA, Lstiburek M:没有繁殖的繁殖。植物学报,2009,29(3):344 - 344。10.1017 / S001667230900007X。
- 47.
黄杉选择和生产居群成员间的系谱关系(Callitropsis nootkatensis)在没有父母资料的情况下。中华医学杂志,2010,30(4):344 - 344。10.1093 / jhered / esp102。
- 48.
Klápště J,李志强,王志强,王志强,等。基于标记关系的西部落叶松开放授粉家系遗传参数和育种价值的估算。植物基因组学报,2013,9:1-9。10.1007 / s11295 - 012 - 0521 - 8。
- 49.
Harrison HB, Saenz-Agudelo P, Planes S, Jones GP, Berumen ML:自然群体亲本分析三种常用方法的相对准确性。分子生态学报,2013,22:1158-1170。10.1111 / mec.12138。
- 50.
Anderson EC, Garza JC:单核苷酸多态性在大规模亲子推断中的作用。中国生物医学工程学报,2006,27(3):344 - 344。10.1534 / genetics.105.048074。
- 51.
Danchok RS, Sniezko RA, Long S, Kegley A, Savin D, Mayo JB, Liu J-J, Hill J:一种高敏感样反应和茎部感染的大兄妹家族松果体monticola.第四届林业宿主-寄生虫相互作用遗传学国际研讨会论文集,2011年7月31日至8月5日,俄勒冈州尤金。编辑:Sniezko RA, Yanchuk AD, Kliejunas JT。2012,美国农业部林业局通用技术代表PSW-GTR-240,太平洋西南研究站,281-285。
- 52.
刘俊杰:异位表达截断松同族体(PrAG1)引起花序结构的改变和雄性不育烟草.动物遗传学报,2012,30:453-467。10.1007 / s11032 - 011 - 9635 - 1。
- 53.
Cui L, Wall PK, Leebens-Mack JH, Lindsay BG, Soltis DE, Doyle JJ, Soltis PS, Carlson JE, Arumuganathan K, Barakat A, Albert VA, Ma H, DE Pamphilis CW:开花植物历史上广泛的基因组复制。中国生物工程学报,2006,29(4):344 - 344。10.1101 / gr.4825606。
- 54.
Gabriel S, Ziaugra L, Tabbaa D:基于Sequenom MassARRAY iPLEX平台的SNP基因分型。2009,第2章,第2.12单元。
- 55.
Peakall R, Smouse PE: GenAlEx 6.5: Excel中的遗传分析。种群遗传教学与研究软件更新。生物信息学,2012,28:2537-2539。10.1093 /生物信息学/ bts460。
- 56.
张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚。生物信息学,2007,23:2633-2635。10.1093 /生物信息学/ btm308。
- 57.
李志强,李志强,李志强,等。基于多位点基因型数据的种群结构推断。遗传学报,2000,37(3):344 - 344。
- 58.
王晓明,王晓明,王晓明。基于结构的个体聚类数量检测方法研究。分子生物学学报,2005,14:2611-2620。10.1111 / j.1365 - 294 x.2005.02553.x。
- 59.
王娟,王俊杰:COLONY:一种基于多位点基因型数据的亲子关系和兄弟姐妹关系推断程序。生物化学学报,2010,10:551-555。10.1111 / j.1755-0998.2009.02787.x。
- 60.
Farnir F, Coppieters W, Arranz JJ, Berzi P, Cambisano N, Grisart B, Karim L, Marcq F, Moreau L, Mni M, Nezer C, Simon P, Vanmanshoven P, Wagenaar D, Georges M:牛广泛的全基因组连锁不平衡。中国生物工程学报,2000,30(4):344 - 344。10.1101 / gr.10.2.220。
- 61.
Weir BS:遗传数据分析2。马萨诸塞州桑德兰:Sinauer Associates;1996.
确认
我们感谢Aimin Guan和Gary Zhang的生物信息学编程,Kristian Arndt的数据库管理,Arezoo Zamany的样本收集,Angelia Kegley和DGRC的同事进行抗性性状评估,以及Grace Sumampong和Danelle Chan的基因组DNA提取。这项研究得到了CFS和J-JL的CFS- grdi基金的部分支持。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
HC构建生物信息学服务器,改进数据分析;RNS构想并构建了生物信息学平台;RAS管理抗性育种项目,提供植株材料和表型数据;J-JL设计了基因组学项目,分析了数据并撰写了手稿。所有作者均已阅读并批准最终稿。
电子辅助材料
表S2。
附加文件1:表S1。:的特点the shoot-tip transcriptome新创从抗性幼苗构建的三个cDNA文库的RNA-seq reads中组装而成。表S2。假定的松果体monticola由TansDecoder预测的蛋白质(最小长度为50)。表S3。西部白松茎尖参考转录组的BLAST分析。表S4。通过高度严格的阈值绘制RNA-seq reads,总结了在茎尖参考转录组中三组候选基因中检测到的snp。(xlsx24kb)
12870 _2014_380_moesm3_esm.pptx
附加文件3:图S2。:Differential GO-term distribution between reference transcriptomes of western white pine shoot-tip and primary needle. Fisher’s exact test was performed using program BLAST2GO with term filter value at 0.05 and term filter mode of corrected p-value by false discovery rate (FDR) control. Categories of GO terms include cell components C1, plasma membrane; C2, cytosol; C3, extracellular region; C4, cell wall; C5, ribosome; C6, endoplasmic reticulum, C7, thylakoid; C8, nucleolus; C9, endosome; C10, cytoskeleton; C11, plasmodesma; C12, nucleoplasm; C13, vacuolar membrane; C14, trans-Golgi network; C15, viral nucleocapsid; F1, RNA binding; F2, chromatin binding; F3, oxidoreductase activity, acting on paired donors, with incorporation or reduction of molecular oxygen; F4, carbohydrate binding; F5, receptor binding; F6, ADP binding; F7, hydroquinone:oxygen oxidoreductase activity; F8, protein kinase binding; F9, RNA polymerase II transcription cofactor activity; F10, catechol O-methyltransferase activity; F11, aminoacyl-tRNA editing activity; P1, lipid metabolic process; P2, response to endogenous stimulus; P3, anatomical structure morphogenesis; P4, response to biotic stimulus; P5, reproduction, P6, DNA metabolic process; P7, translation; P8, cell differentiation; P9, cell cycle; P10, flower development; P11, generation of precursor metabolites and energy; P12, embryo development; P13, cell growth; P14, secondary metabolic process; P15, regulation of gene expression, epigenetic; P16, photosynthesis; P17, response to extracellular stimulus; P18, pollination; P19, tropism; P20, methylation; P21, ATP catabolic process; P22, cell-cell signaling; P23, response to karrikin; P24, pectin catabolic process; P25, regulation of plant-type hypersensitive response; P26, membrane fusion; P27, MAPK cascade; P28, Golgi organization; P29, cellular response to iron ion; P30, protein peptidyl-prolyl isomerization; P31, carbohydrate transmembrane transport; P32, amino acid transmembrane transport; P33, response to cyclopentenone; P34, monocarboxylic acid transport. (PPTX 101 KB)
表S6。
附加文件4:表S5。:Sequences of primers and probes used for Sequenom iPlex genotyping on the SNPs with successfully verified genotypes in western white pine populations. Population genetic parameters of the SNPs with successfully verified genotypes.表S7。利用Colony软件包进行亲本分析,以分配抗病种质中最好的全兄弟亲本。(pptx119kb)
12870 _2014_380_moesm5_esm.pptx
附加文件5:图S3A。:Distributions of minor allele frequency (MAF) and observed heterozygosities (Ho) of the SNP loci genotyped successfully with polymorphism and call rate > 80% in the whole array set. (A) Distributions of minor allele frequency (MAF). (PPTX 75 KB)
12870 _2014_380_moesm6_esm.pptx
附加文件6:图S3B。:Distributions of minor allele frequency (MAF) and observed heterozygosities (Ho) of the SNP loci genotyped successfully with polymorphism and call rate > 80% in the whole array set. (B) Distributions of observed heterozygosities (Ho). (PPTX 65 KB)
12870 _2014_380_moesm7_esm.pptx
附加文件7:图S4。:Graph of the first three principal components based on marker frequencies. Principle component analysis (PCA) was based on SNP genotypic data showing genetic diversity of a composite seed lot from a western white pine breeding program. (PPTX 193 KB)
12870 _2014_380_moesm8_esm.pptx
附加文件8:图S5。:Estimated number of clusters obtained with STRUCTURE for K values from 1 to 10 using SNP data. Graphical representations are shown the statistics ΔK in two SNP arrays separately by STRUCTURE simulations. (A) The ΔK was calculated based on genotypic data of 108 SNP markers in 179 samples of the 1st SNP array; and (B) The ΔK was calculated based on genotypic data of 95 SNP markers in 188 samples of the 2nd SNP array. (PPTX 107 KB)
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/4.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
刘,JJ。,Sniezko, R.A., Sturrock, R.N.et al。西部白松SNP的发现和高通量基因分型在育种和保护中的应用。BMC植物生物学14日,380(2014)。https://doi.org/10.1186/s12870-014-0380-6
收到了:
接受:
发表:
关键字
- Five-needle松
- 基因分型数组
- 连锁不平衡
- 概略介绍选择
- 血统重建