摘要
背景
单独的着丝粒DNA序列既不是规定着丝粒的必要条件,也不是规定着丝粒的充分条件。着丝粒特异性组蛋白,Cenh3.这可能是对快速进化的着丝粒DNA的共同进化反应。深入了解Cenh3.进化,我们描述了三种不同被子植物属的二倍体和异源多倍体之间的核苷酸和蛋白质多样性模式,芸苔,oryza., 和Gossypium(棉花),重点研究在不同领域的多样化选择的证据Cenh3.基因。此外,我们还比较了表达式配置文件和可选的拼接模式Cenh3.在每个属的代表。
结果
所有三个世代都保留了两项重复Cenh3.副本,而芸苔和Gossypium表现出明显的同源表达水平偏差。属之间的比较揭示了共同和独特的方面Cenh3.进化,不同层次的多样化选择在不同Cenh3.结构域,可变剪接对Cenh3.多样性。
结论
由于N末端受到多样化的选择,但是DNA结合域不似乎,快速发展的CENTROMERE序列不太可能是主要驱动器Cenh3.序列多样化。目前,在N个终端域的常规蛋白质演化中产生的多样性的功能解释,以及替代剪接仍然是未解释的。
背景
着丝粒是真核染色体上的一个特定区域,它是着丝粒的集合点,着丝粒是一组蛋白质,在细胞分裂期间充当微管的系绳。虽然真核生物着丝粒具有高度保守的染色体分离机制,但着丝粒序列和特定于着丝粒染色质的结合蛋白是高度可变的,即使在密切相关的类群中[1],[2].真核生物着丝粒的DNA中常见的是特定的反转录因子和高度均质的串联重复序列[3.],[4],尽管这些差异在近亲物种中也很明显地迅速分化[2],[5],[6].适当的招募这些潜在的共同进化的分子成分到同一地点是谜一样的,因为是它们在染色体之间传播和同质化的机制。对于功能守恒与序列变异之间的明显悖论的解释,通常援引着丝粒功能、着丝粒序列和表观遗传因素(如DNA序列)之间的相互作用本身变得不那么受功能约束[2],[6],[7].因此,人们普遍认为着丝粒是表观遗传上特定的[4].
演变Cenh3.是特别有趣的,因为它在着丝粒的规格和功能中的中心地位[8].与高度保守的对应蛋白(组蛋白H3)不同,Cenh3.具有广泛的序列变异性,特别是在两个区域:(1)非典型的NH2末端,(2)较长的环1区域[9],[10.].DNA序列多样性和可变剪接都可以在产生多样性中发挥作用,这被认为是对快速进化的着丝粒DNA的补偿,以确保一致的着丝粒功能,尽管着丝粒序列对着丝粒组装既不是必要的,也不是充分的[2],[11.].
考虑到在物种内染色体中的碳体重复的快速序列均质化的双序观察,但在物种之间的重复之间的迅速发散,问题出现为焦化重复的命运,Cenh3.异源多倍体进化。多倍体在许多生物的进化中起着不可或缺的作用,尤其是植物[12.] - [14.].多倍体的后果通常很广泛[15.] - [23.,由此产生的复制基因有无数种可能的命运[18.],[24.,包括基因丢失。基因组复制后的基因保留和丢失模式已就其广泛的分类进行了广泛的研究[25.] - [29.].由于在异源多倍体形成时,两个不同的着丝粒序列组合成为共同的细胞核,因此研究着丝粒重复序列以及现在重复的着丝粒序列的后续进化动态是一件有趣的事情Cenh3.序列。在大多数现代二倍体中,Cenh3.似乎在古多倍性事件之后回到了单拷贝状态[30.],但有几个明显的例外[31.].相反,新近的异源多倍体通常有多个Cenh3.基因复制(30.] - [32.].
在这里,我们评估Cenh3.在三个发散的属于三种分歧(芸苔,oryza., 和Gossypium)来解决是否进化的问题Cenh3.在许多被子植物类群中都是相似的。所选的属包含异源多倍体种的多样性,既有单系起源,也有多系起源。的芸苔该属包含三个多样化和广泛栽培的二倍体物种(基因组指定为A - C)和三个由独立多倍体事件产生的异源多倍体物种(BBCC, AACC, AABB) [33.),而oryza.含有多个二倍体基因组(指定A - G)和各种基因组源的异种多倍体(BBCC,CCDD和HHJJ)[34.].Gossypium包括45个二倍体物种,分为A - G和K基因组组[19.],以及单一的,单系的[35.多倍体分支(AD基因组),包含6个物种。先前的工作Cenh3.在芸苔和oryza.异源多倍体主要关注基因特定区域的选择和保留同源基因的相对表达[30.],[32.].很少有人知道Cenh3.和各种多百倍的焦化进化Gossypium.先前的一项研究报道了在两种异源四倍体的所有着丝粒中都存在着一个着丝粒吉普赛样反转录因子(CRG)G. Hirsutum.(ad -基因组)和模型祖d -基因组二倍体(g . raimondii就),但a -基因组物种没有[36.].序列都不是重复的表达Cenh3.已被评估。
我们描述Cenh3.含有二倍体和异聚倍数的三种系统源性脓毒症属的序列演化,评估分子演化的模式。我们满足了所有多元层是否保留了所有多元层组的复制副本;重复,新共同居民序列序列演化的动态;同优拷贝的相对表达水平。除了报告Cenh3.序列在属内和属间的进化,我们描述了可变剪接的新模式Cenh3..
结果
我们克隆并测量了Cenh3.来自7类二倍体和5个同种异体一体化的基因Gossypium物种。基因组序列的长度在二倍体中为2565 ~ 2673 bp,在多倍体中为2654 ~ 2673 bp1),但所有物种的蛋白编码长度一致,为492 bp。虽然cdna之间没有检测到长度变化,但我们不能解释在我们的外部引物之外的第一个和最后一个外显子中发生的长度变化。的结构Cenh3.在所有多倍体物种中均存在7个外显子和6个内含子,(从基因组序列推断为二倍体cDNA序列)。正如所料,Cenh3.在整个属中是保守的,大多数多态性发生在内含子中。Gossypium exiguum在二倍体中差异最大(内含子6缺失93 bp);其余的多态性较小(<10 bp),其中5个具有系统发育信息(附加文件)2).
由于CENH3被认为与快速变化的焦化DNA共同发展,因此我们将非唯一的取代映射到三属的二次蛋白质结构(芸苔,oryza., 和Gossypium)(图1),以便沿着蛋白质定位进化。N端是三个属中唯一具有高水平非同义替换的结构域。芸苔有最多的非同义替换,然后是oryza.然后Gossypium.该观察结果与先前的分析一致,表明草本植物中的分子演化率更快(oryza.和芸苔)而不是树木和灌木(Gossypium)[37.].
每个属中单个物种的CenH3蛋白的三维结构。一个.芸苔属植物显著(AC8).b.澳大利亚稻属(EE).c.Gossypium raimondii就(D5)。二级蛋白结构如下图所示,每个3D模型包含不同种类的SNPs,这些SNPs与CenH3蛋白的二级结构相对应。不同深浅的蓝色表示每个蛋白质域的边界;α表示α螺旋,L表示循环。黑色矩形表示非同义SNPs,黄色矩形表示正选择下的非同义SNPs(图3.).N端是三属间长度变异的主要位点,N端长黑色矩形是三属间长度变异的主要位点芸苔属植物CenH3在对齐中表示差距芸苔物种。
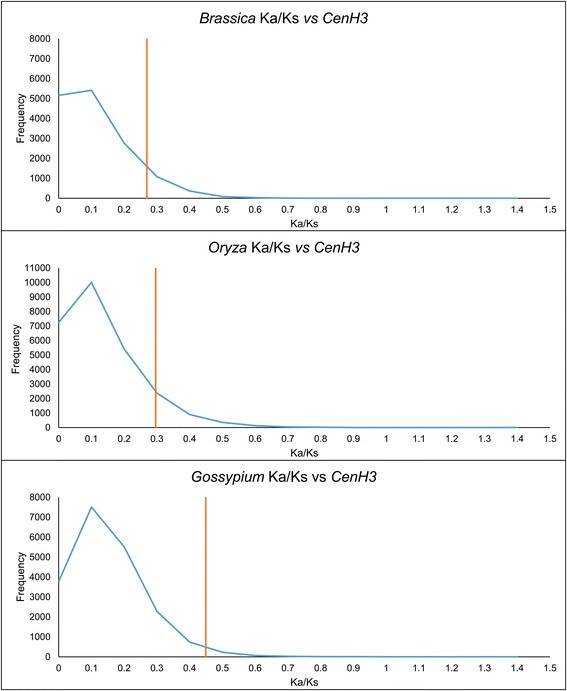
确定…的程度Cenh3.在进化过程中,我们比较了Ka/Ks(非同义/同义替换)比值,每个属的物种相对于外群的所有cds。Cenh3.确实是一个进化相对较快的基因,与芸苔,oryza., 和Gossypium基因分别位于这些基因组中所有基因的第81、85和97百分位(图)2).
量化Cenh3.我们估算了各属的Ka/Ks平均值Cenh3.使用dnasp(表1)[38.].各属内、属与外群间Ka/Ks比值的总体平均值均表明,该属没有多样化选择Cenh3.基因作为一个整体。为了确定这一发现在更精细的尺度上是否一致,我们使用了进化软件(MEME)的混合效应模型[39.]评估每个属的密码子水平的选择(表2,图3.).在芸苔和oryza.,仅有N端有多样化选择的证据,而N端没有多样化选择的证据Gossypium.中受选择的推断密码子oryza.和芸苔对于每个密码子,系统发育上是偶发性的,并且只针对少数物种(图3.).
Cenh3.在异源多倍体进化
鉴于到目前为止在大多数二倍体被子植物中观察到的单拷贝状态,尽管有一个包含多个多倍体片段的进化历史,我们希望解决是否Cenh3.在最近的全多大宽度事件发生后一份重复保留。在Gossypium,我们发现每个二倍体代表有一个单倍体Cenh3.基因,所有的所有国内单倍异物种都有两种,表明据报道,表明父母副本的保留芸苔和oryza.[30.],[32.],[40.].
处理是否重复Cenh3.序列在异源多倍体形成后彼此独立进化,或者如果它们受到某种形式的序列相互作用或均质化,我们手动分析Cenh3.所有三个属的序列。这种独立性的缺乏已经在异源多倍体的其他同源单拷贝基因中得到证实,尤其是在Gossypium第一次描述这种现象的地方[41.],[42.].Cenh3.在所有三个属中都没有基因转换,这与之前的报道一致芸苔和oryza.[30.],[32.].
基因组DNA序列Cenh3.从Gossypium用于构建最大似然树,这种似乎与当前接受的属植物发育(附加文件)3.).我们解决了一个a的单胞水工T和DT同源物及其各自的模型二倍体祖先。同源性中未检测到基因转换,进一步证实了独立进化Cenh3.同源基因在1-2 MYD多倍体分支中发生。
Cenh3.基因表达
如上所述,通过多倍体复制的基因有多种可能的命运。而父母的副本Cenh3.在所有研究的异源多倍体种oryza.,芸苔, 和Gossypium,每个父母副本的转录用法可能因相同而异,以完全沉默一个父母副本。评估表达Cenh3.在Gossypium,我们使用了三种独立的方法来评估同源性的相对表达。相同的组织来源用于克隆计数和色谱测量,而不同来源的RNA-seq数据用于研究CenH3表达的其他方面。RNA-seq数据来源包括(A2 vs D5)以确定相对表达Cenh3.在棉花多倍体的模式二倍体祖中,两个二倍体(A2XD5F1)、一个合成多倍体(2_A2D1)和一个驯化的和野生的AD1杂种(maxxa和yuc)。
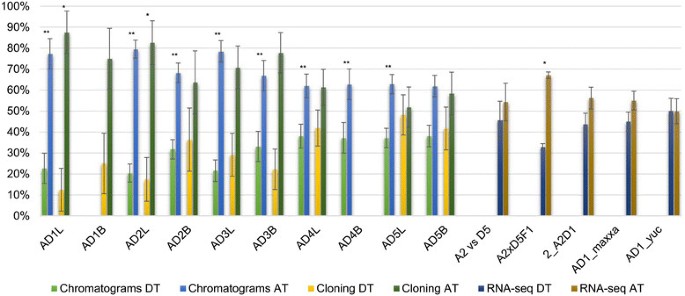
三种分析表达的方法导致了不同程度的同源性偏倚(AT和DT用于表示两种同源物质,其在RNA-SEQ数据中等,更极端的其他方法(图4).一个T同系物,表达在每个物种,组织和试验中都很受青睐(图)4).通过RNA-SEQ数据,我们将模型祖素二倍体(A2 VS D5)的总表达水平进行比较,合成多倍体(2(A2D1))和AD1(尤卡坦义和MAXXA)的野生和驯化过程,所有这些表达缺乏显着差异。具有显着表达偏压的唯一样品是F1(A2D5),偏置为87.5%(P≤0.05)。除了一个外,同源表情表达的差异在叶子和芽组织之间没有显着差异(t试验)T叶片中同源基因的表达量显著增加g .取得(p = 1.1024 × 10−10).
Homoeolog表达Cenh3.在Gossypium物种。AD1-AD5表示陆地革兰,巴巴多斯革兰,绒毛革兰,野马革兰,和g . darwinii,分别。L和B后缀分别表示叶片和芽组织。A2 VS D5是模型二倍体祖细胞物种中表达的总水平的比较,G. Arboreum.(一)和G. Raimondii.(D). A2xD5F1是一个F1杂种之间G. Arboreum.和G. Raimondii..2_A2D1为秋水仙碱双杂交一代G. Arboreum.(一)和g . thurberi(d)。AD1_MAXXA和AD1_YUC是驯化和野生的G. Hirsutum.,分别。AD1B和AD4B只生成了部分数据。标准差用误差棒表示。单个星号表示样本在0.05以下具有统计学意义,两个星号表示样本在0.01以下具有统计学意义。
由于同源物质之间的序列相似性,因此不能设计特异性引物用于定量PCR,因此我们用三种单独的方法测量同种型表达偏压,伴随着不同的警察。由于同种型在同源物之间的少量的SNP,因此不能将大量RNA-SEQ读取分配给特定的同源性。我们也克隆了Cenh3.虽然样本量较小,但用于计算同源物相对表达的cdna(附加文件)4).
可变剪接
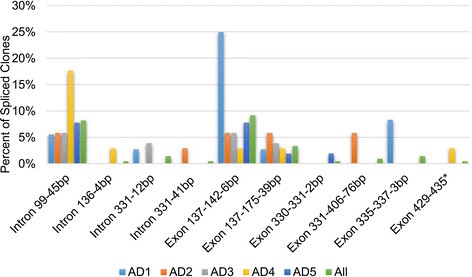
转录本的选择性剪接是一种创造新蛋白质的机制,可以想象,它可能为快速进化的伙伴(如CenH3和着丝粒DNA)之间的相互作用提供多样性。来解决可变剪接的影响Cenh3.,我们评估了每个同系物的可变剪接水平Cenh3.在Allopolyploid.Gossypium通过克隆和测序扩增Cenh3.来自叶片和叶芽组织的cdna。所有异源多倍体的交替剪接转录本的总频率为26.7%(55/206转录本),包括33个外显子缺失和22个内含子保留(相对于主要变体)。在所有被评估的物种中发现了三种剪接变异(图5):(1)99磅内含子保留99(成绩单的8.3%);(2)从137-142的位置(9.2%的转录物)的6bp外显子缺失;(3)A 39 BP外显子缺失T来自137-175的同源基因(3.4%的全部转录本或4.9%的AT成绩单;数字5).一个剪接变体,一个12 bp的内含子保留,只在两者之间共享G. Hirsutum.(AD1)和g . tomentosum(ad3)(图5).最常见的剪接变异导致N端轻微的缺失或扩展,从而增加了一个已经快速进化的区域的多样性Cenh3.,而最不常见的剪接变异导致非功能性蛋白质预测。
G. Hirsutum.(AD1)在44.4%的剪接成绩单的频率最高(附加档案5).每个物种具有不同比例的每个拼接变体;例如,外显子137-142存在于25中存在%的G. Hirsutum.(AD1)克隆,虽然其其他物种的2.9-7.8%。17.6%g . mustelinum(AD4)克隆在99位有45 bp的内含子插入,在其他物种中仅存在5.6-7.8%(图4)5).
讨论
尽管它在着丝粒规范中起着至关重要的作用,但在许多方面Cenh3.人们对进化知之甚少。近年来,我们对着丝粒表观遗传学和进化的认识有了长足的进展。例如,许多物种的串联重复序列与CenH3蛋白有进化关系,以降低核小体弯曲能[43.]多种蛋白质与焦化DNA相互作用,诱导厘米浓度的焦化DNA [44.]和CenPA(植物中的CenH3)提供了结合其他着丝粒蛋白的基础[45.].
Cenh3.AgeniSperms的序列演进
目前的研究扩展了我们的知识的步伐和过程Cenh3.三种多样性贪眼属于基因组和表达变化的进化。以前的研究芸苔属植物和选用表明了这一点Cenh3.n末端和CATD(环1和α2螺旋域)序列在谱系上的不同选择下进行了多样化[30.],[32.].评估在Angiosperms中这些发现的一般性,我们重新分析了oryza.和芸苔序列在更细的尺度上识别出特定的区域Cenh3.他们积极地进行多元化经营,并取得了第一的成绩Cenh3.序列分析Gossypium.在所有三个属中,多样化通常发生在N端,这一结果与之前关于该域快速进化的报道一致,但与CenPA Targeting域多样化的报道相反[10.],[46.] - [50.].
保留与表达Cenh3.Allopolyproids的等位基因
在所有三个属中,在基因组倍增后保留了同源副本,证明在现代“二倍体”植物中广泛观察到的单一拷贝身份的恢复,不需要在WGD事件之后快速发生。有趣的是,表达同种型Cenh3.册Gossypium在所有的样本中显示出方向偏差,尽管差异并不总是具有统计学意义(图4);这一结果与来自芸苔和oryza.异源多倍体(30.],[32.].在芸苔异源四倍体,各种Cenh3.发现表达模式用于同种型,从加入中的2:1比例b . juncea完成B-genomeCenh3.对加入的压制B. Carinata.[32.].在异源四倍体o . minuta和o·阿尔塔,Cenh3.表达是公正的[30.].这三个属中异源多倍体的表达谱差异是显著的,但没有一个明显的解释,尽管同源表达水平似乎反映了每个属中杂交和基因组加倍的独特基因组和进化特性。
可变剪接的Cenh3.
除了非同义的进化,蛋白质多样性也可能由选择性剪接产生。从三个属的整体来看,选择性剪接经常修饰N端Cenh3.在芸苔和Gossypium,这是唯一修改的域oryza.[30.],[32.].有趣的是,N端在亲缘关系较近的属之间往往不能排列,但它对植物的着丝粒沉积是必要的Cenh3.在减数分裂期间A. Thaliana.[51.],[52.].N端也与体内的着丝粒蛋白相互作用美国cerivisiae[53.].也许替代剪接代表了另一种方法,以产生CENH3在减数分裂中靶向偏摩尔所需的序列的分集,或者响应于升高的厘米尺寸引入新的CENH3蛋白的快速防御。另一种替代方案是替代剪接还可以导致细胞和组织之间的不同替代转录物,其在焦化DNA中具有含义Cenh3.和着丝粒DNA确实是共同进化的。
结论
通过对比Cenh3.序列来自三个不同的被子植物属,我们已经了解了这个重要的蛋白质的进化速度和区域。最常见的突变结构域是N端,它也受选择性剪接的影响,对N端多样性有重要贡献芸苔和GossypiumCenh3s..尽管环1和α2螺旋结构域结合着着丝粒DNA,因此可能受到长度的限制,但在组蛋白折叠结构域中基本没有选择性剪接。
虽然CENH3被认为是一种快速发展的蛋白质,但是N末端是唯一一个在密切相关的属中不可识别的域,并且是最多样化的域名。角色已归因于N末端Cenh3.,例如泛素介导的蛋白水解[53.],对CenH3的减数分裂定位不可或缺,但在有丝分裂中该结构域是可消耗的[51.],[52.].在人类中,N端是翻译后修饰的热点,与其他着丝粒蛋白相互作用[54.,也许植物也是如此。目前,无论是传统的蛋白质在N端结构域的进化,还是选择性剪接,都无法从功能上解释这种多样性。
CENH3兴趣的基础是它能够与快速发展的焦化重量结合,但仍然与KINETOCHORE中的保守元素相互作用。由于重复,多倍形规模的这种关系的动态越来越复杂Cenh3.和暴露于新的政权符号重复。多倍体的单体和多晶体起源似乎影响了表达偏差的方向和水平Cenh3.homoeologs(图4)在杂交之后最明显,如F1(A2D5)所见Gossypium,但没有遵循多倍体2(A2D1)。另一个引人注目的问题是在这些成绩单中获得多样性以响应Centromere中的重复演进,这在我们的选择测试中稀疏。替代的拼接可以是获得这种分集的另一个方法,尽管既不选择也不是替代剪接修改DNA结合域Cenh3..由于N端是唯一被选择性剪接修饰的结构域,并受到多样化的选择,但DNA结合结构域没有被修饰,所以快速进化的着丝粒序列不太可能是主要的驱动因素Cenh3.序列多样化。
方法
植物材料
从八个二倍体棉花收集叶(2-4厘米)和叶芽:G. Arboreum.简历。101 (A2) (ISC427583),G.异常(B1) (ISC447893), g . robinsonii(C2) ISC451818), g . raimondii就[D5] (ISC429440), g . stocksii(E1) (ISC447876),G. longicalyx.(F1) (ISC418550),G. Bickii.[G1](ISC414834), g . exiguum[k](ISC416400),以及五个Allopolyploid棉花(G. Hirsutum.简历。TM1 [AD1](ISC451819),g .取得简历。Pima S6 [AD2] (ISC451820),g . tomentosum95 (AD3粉)(ISC451821),g . mustelinum本地实验室加入[AD4](ISC429442),和g . darwiniiPW45 [AD5] (ISC429431)。为了进行系统发育重建,我们将群外种纳入其中Gossypioides Kirkii.(ISC 418555)[55.].所有线条在爱荷华州立大学的Pohl音乐学会中生长,用于DNA和RNA提取。在制造商推荐的协议之后,使用QIAGEN DNEASY植物套件提取DNA。使用Sigma谱RNA提取试剂盒提取RNA推荐方案,推荐方案具有以下修改:在步骤4进行方案A,对洗涤溶液I和II进行一次洗涤,并用柱DNA消化进行。Sigma在列柱DNASE I DIGEST集合。
Cenh3.测序
Cenh3.基因序列数据G. Raimondii.是从植物血红素中获得的[56.],[57.引物的设计(附加文件6)进行PCR扩增和测序。PCR扩增采用制造商推荐的反应混合物/循环条件,熔化温度为57℃。PCR扩增来自所有二倍体材料(除G. exiguum.),通过Qiaquick PCR柱cleanup (Qiagen)对其进行清洗,并利用扩增引物和一套内部测序引物进行测序(附加文件)6).
PCR产品G. exiguum.所有多倍体物种在Invitrogen E-gel上可视化,分离条带,并根据推荐方案,用P-GEM-T Easy Vector ligkit (Promega)和Top10 Competent Cells (Invitrogen)进行克隆。克隆在爱荷华州DNA测序设施使用M13引物和内部引物进行测序(附加文件)6).
Cenh3.cDNA序列oryza.和芸苔已从GenBank下载[58.]及Phytozome [57.)(附加文件7).
评估选择
Jukes Cantor校正的Ka/Ks估计使用DNAsp测量。MEME软件包[39.]透过(http://www.datamonkey.org) 服务器 [59.,用于检测密码子水平上的选择。利用自动选择工具确定正确的替代模型芸苔(F81),Gossypium(F81),oryza.(HKY85)。显著性水平临界值设为P = 0.05。我们使用拟南芥蒂利亚纳(2400万年辐散(MYD);Lysak等人[60.]),Gossypioides kirki(13.6 MYD;Cronn等人[61.)),Brachypodium distachyon(46 MYD;桑德森(62.)作为外群体芸苔(7.9 MYD;Jacquemin等[63.]),Gossypium(5 - 10 MYD;Senchina等人[64.)),oryza.(15 MYD;桑德森(62.]), 分别。
使用CoGe的SynMap工具(http://genomeevolution.org/coge.)[65.],[66.]鉴定同同源基因块,评估每个属中一个代表物种的全基因组Ka/Ks (甘蓝,水稻,棉花).使用以下参数:BlastN,相对基因顺序,−D 50,−A 10, quota align merge -Dm 80,按覆盖深度3:1比例对(b·拉伯:A. Thaliana.), 1:1为(o.苜蓿:B. Distachyon.)和6:1为(G. Raimondii.:Theobroma可可),重叠距离40。每个物种都与之前描述的外群体进行了比较,除了t .可可(60 myd)[67是外群体g . raimondii就自从Gossypioides kirki缺乏测序的基因组。
系统发育分析
基因组DNA序列使用CLUSTALW对齐[68]在BioEdit [69],并使用readal (http://trimal.cgenomics.org).利用AIC和BIC确定DNA序列进化的最佳拟合模型,由jModelTest计算[70],[71].由于GTR + Γ模型和HKY + Γ模型分别受到AIC和BIC的青睐,因此使用MEGA6在两种模型下构建了100个重复的bootstrap最大似然树[72],[73].GTR + Γ树的日志可能性略高,并在这里报告;然而,这两棵树呈现出相似的拓扑结构。
蛋白质结构预测
二级和三级蛋白质结构采用RaptorX建模[74] - [77].该软件将样品蛋白的对准与已知结构信息的其他蛋白质进行比较,以确定使用统计数据的可能结构。一个代表Cenh3.来自每个属的序列是模拟的(G.raimondii(D5),Oryza Australiensis.(ee),和芸苔属植物显著(AC8))(图1).
cDNA生成和排序Cenh3.成绩单
逆转录使用Invitrogen SuperScript III First-Strand Synthesis System Kit进行,使用oligo dT引物Cenh3.从cDNA文库中扩增,引物由5 '和3 '外显子设计(附加文件6).PCR产物经Qiaquick PCR Cleanup柱处理,扩增引物测序(附文件)6).
以色谱为基础的表达估计如前所述计算[78].每个组织至少使用三个重复,以允许标准误差计算,并使用配对的双尾T检验检验显著性。通过RNA-seq数据和克隆cDNA扩增子(如上所述),对多倍体材料的表达水平进行了二级估计。从每个样本中随机选择克隆,测序,然后按其亚基因组来源“AT”和“维T”。由于样本应遵循二项分布,每个CenH3同源副本的克隆率的零假设应为0.5。为了控制α = 0.05的FWER(家族错误率),使用Bonferroni校正确定显著性。
通过RNA-SEQ的表达估计
评估Cenh3.基因表达,我们分析了先前产生的叶片转录组数据(SRA Bioproject Prjna171342)[79]对于两个模型二倍体父母,一个F1杂交G. Arboreum.(A2基因组)和G. Raimondii.(D5基因组),秋水仙碱双F1杂种G. Arboreum.和g . thurberi(D1基因组),和Allopolyploid(AD Genome)的两种含义G. Hirsutum.(G. Hirsutum.var yucatanense,野生加入;G. Hirsutum.CV Maxxa,驯化的加入)。原始读数被镰刀修剪(https://github.com/najoshi/sickle),并映射到生成的Cenh3.使用GSNAP的序列(批量4,新颖的拼接)[80与…结合Cenh3.-specific SNP index可以有效地绘制不同物种和亚基因组的序列。SNP指数由Sanger测序的cDNA 's和基因序列手工整理。对于杂交多倍体的序列,PolyCat [81用来分配A-和D-基因组衍生读数。使用配对的学生的T-Test计算实质偏置的重要性,使用LOG2转换进行计算,以确保表达式值中的正常性。
可获得的支持数据
CenH3基因序列数据已提交至GenBank。登录号可在附加文件中找到1.
附加文件
参考文献
- 1。
“着丝粒的序列、结构和生物多样性”。植物基因组的多样性。编辑:Wendel JF, Greilhuber J, Dolezel J, Leitch IJ。2012年,施普林格,维也纳,59-70。10.1007 / 978 - 3 - 7091 - 1130 - 7 - _4。
- 2。
Henikoff S,Ahmad K,Malik Hs:Centromere Paradox:稳定的遗传伴快速不断发展的DNA。科学。2001,293(5532):1098-1102。10.1126 / Science1062939。
- 3。
人类α卫星DNA的染色体特异性亚群:染色体亚群内部和之间序列差异的分析和祖先五聚体重复的证据。acta photonica sinica, 2011, 40(3): 441 - 446。10.1007 / BF02100014。
- 4.
蒋建平,王志强,王志强:植物着丝粒的分子生物学研究。植物科学进展,2003,8(12):570-575。10.1016 / j.tplants.2003.10.011。
- 5。
Melters DP, Bradnam KR, Young HA, Telis N, May MR, Ruby JG, Sebra R, Peluso P, Eid J, Rank D:对数百个物种串联重复序列的比较分析揭示了对着丝粒进化的独特见解。中国生物医学工程学报。2013,34(1):1- 4。
- 6。
着丝粒DNA和着丝粒蛋白的系统发育和结构分析。中国生物医学工程学报。2006,27(3):353 - 356。
- 7.
著丝粒和着丝粒:从表观遗传学到有丝分裂检查点信号。细胞。2003,112(4):407-421。10.1016 / s0092 - 8674(03) 00115 - 6。
- 8.
Malik HS,Vermaak D,Henikoff S:果蝇焦化组蛋白中DNA结合基序的复发演变。Proc Natl Acad SCI U S A. 2002,99(3):1449-1454。10.1073 / pnas.032664299。
- 9.
Talbert PB,Masuelli R,Tyagi AP,Comai L,Henikoff S:封送型和自适应演变拟南芥组蛋白H3变体。植物学报。2002,14(5):1053-1066。10.1105 / tpc.010425。
- 10。
CID是一种中心体特异性组蛋白果蝇.中国生物医学工程学报。2001,27(3):453 - 457。
- 11.
着丝粒染色质的表观遗传调控:老狗,新把戏?中国科学院大学学报(自然科学版)。10.1038 / nrg2466。
- 12.
宋超,刘胜,肖军,何伟,周勇,秦强,张超,刘勇:多倍体生物。中国科学:生命科学,2012,55(4):301-311。10.1007 / s11427 - 012 - 4310 - 2。
- 13。
Heslop-Harrison JS:基因组进化:灭绝,延续或爆炸?CurrOp植物BIOL。2012,15(2):115-121。10.1016 / J.PBI.2012.03.006。
- 14。
梵德·梅耶,Meyer A:古代基因组重复的进化意义。NAT Rev Genet。2009,10(10):725-732。10.1038 / NRG2600。
- 15.
多倍体的基因组进化。植物学报。2000,42(1):225-249。10.1023 /: 1006392424384。
- 16。
Soltis DE, Albert VA, Leebens-Mack J, Bell CD, Paterson AH, Zheng C, Sankoff D, DE Pamphilis CW, Wall PK, Soltis PS:多倍体与被子植物多样化。中国生物医学工程学报。2009,27(1):136 - 142。10.3732 / ajb.0800079。
- 17.
Adams KL,Wendel JF:植物中的多倍体和基因组进化。CurrOp植物BIOL。2005,8(2):135-141。10.1016 / j.pbi.2005.01.001。
- 18.
通过多倍性复制的基因在转录组和器官特异性的相互沉默中贡献不平等。中国科学(d辑:地球科学),2003,30(6):591 - 598。10.1073 / pnas.0630618100。
- 19.
牛仔裤、基因和基因组:棉花作为研究多倍体的模型。多倍性与基因组进化。编辑:Soltis PS, Soltis DE. 2012,施普林格,纽约,181-207。10.1007 / 978 - 3 - 642 - 31442 - 1 - _10。
- 20.
多倍性与植物的进化。植物营养与肥料学报。2005,8:135-141。10.1016 / j.pbi.2005.01.001。
- 21.
多倍性:周期性形成和基因组进化。生态学报,1999,14(9):348-352。10.1016 / s0169 - 5347(99) 01638 - 9。
- 22.
陈志军,倪智:植物多倍体基因组重排与基因表达变化的机制。生物学报。2006,28(3):240-252。10.1002 / bies.20374。
- 23.
陈祖:遗传和表观遗传机制,基因表达和植物多倍体中的表型变异。Annu Rev植物Biol。2007,58:377-406。10.1146 / annurev.arplant.58.032806.103835。
- 24.
黄志强,王志强:多倍体植物基因表达的新模式。电子学报。2005,21(10):539-543。10.1016 / j.tig.2005.07.009。
- 25.
在多倍体酵母中,多轮物种形成与互易基因丢失相关。自然科学学报。2006,40(4):441 - 445。10.1038 / nature04562。
- 26.
镇CD,张F, Maiti R, Crabtree J, Haas BJ, Wortman JR, Hine EE, Althoff R, Arbogast TS, Tallon LJ:比较基因组学芸苔属植物oleracea和拟南芥蒂利亚纳揭示多倍体后的基因丢失、碎片化和分散。植物学报,2006,18(6):1348-1359。10.1105 / tpc.106.041665。
- 27.
关键词:番茄,拟南芥,基因组测序,基因复制,共同网络中国科学(d辑:地球科学),2000,19(4):531 - 534。10.1073 / pnas.160271297。
- 28.
Doyle JJ, Flagel LE, Paterson AH, Rapp RA, Soltis DE, Soltis PS, Wendel JF:植物基因组合并和加倍的进化遗传学。王志强。基于遗传算法的生物信息学研究[j] .中国生物医学工程学报。2008,42(1):443-461。10.1146 / annurev.genet.42.110807.091524。
- 29.
Barker MS, Kane NC, Matvienko M, Kozik A, Michelmore RW, Knapp SJ, Rieseberg LH:菊科进化过程中的多重多倍化揭示了数百万年后重复基因保留的平行模式。中国生物防治学报。2008,25(11):2445-2455。10.1093 / molbev / msn187。
- 30.
严华,姜建平,吴玉梅,等:CenH3中心体蛋白在二倍体和异源四倍体中的谱系特异性适应进化oryza.物种。中国生物化学学报,2009,26(12):2877-2885。10.1093 / molbev / msp208。
- 31。
Lermontova I, Schubert I: CenH3用于建立和维持着丝粒。:植物厘米生物学。牛津,英国:Wiley-Blackwell;2013:67-82。
- 32。
王刚,何强,刘芳,程志平,金伟:二倍体和异源四倍体中CenH3蛋白和着丝粒相关DNA序列的分析芸苔物种。中国生物医学工程学报,2011,30(4):353-365。10.1007 / s00412 - 011 - 0315 - z。
- 33。
联合国:基因组分析芸苔特别参考实验形成显著和特殊的施肥方式。日本J机器人。1935,7:389-452。
- 34。
葛松,桑涛,吕宝荣,洪德勇:水稻基因组系统发育及其异源四倍体起源研究。中国科学(d辑:地球科学),1998,16(4):531 - 534。10.1073 / pnas.96.25.14400。
- 35。
李志强,李志强,李志强,等:多倍体单倍体的鉴定Gossypium物种。acta botanica sinica, 2017, 36(6): 1177-1183。10.1007 / s00606 - 012 - 0615 - 7。
- 36。
Luo S, Mach J, Abramson B, Ramirez R, Schurr R, Barone P, Copenhaver G, Folkerts O:棉花着丝粒中含有aty3-gypsy-像LTR retroelement。公共科学图书馆。2012,7 (4):e35261-10.1371/journal. pon.0035261。
- 37。
Smith SA, Donoghue MJ:分子进化速率与开花植物的生活史有关。中国科学(d辑:地球科学),2008,32(6):591 - 598。10.1126 / science.1163197。
- 38。
Librado P, Rozas J: DnaSP v5:一个全面分析DNA多态性数据的软件。生物信息学。2009,25(11):1451-1452。10.1093 /生物信息学/ btp187。
- 39。
Murrell B, Wertheim JO, Moola S, Weighill T, Scheffler K, Pond SLK:检测受偶发性多样化选择影响的个体位点。acta photonica sinica, 2012, 38(6): 657 - 662。
- 40.
慧玲,卢玲,衡宇,秦锐,邢颖,金伟:语感的表达CENH3合成的Allopolyploid等等位基因oryza.物种。j遗传学基因组学。2010,37(10):703-711。10.1016 / s1673-8527(09)60088-6。
- 41.
关键词:棉花,多倍体,同源非互反重组。植物学报,2010,42(1):1 - 8。10.1111 / j.1469-8137.2009.03093.x。
- 42.
关键词:棉花,复制基因,同源重组,转录组特性中国生物医学工程学报。2012,13(1):302-10.1186/1471-2164-13-302。
- 43.
Zhang T ., Talbert PB, Zhang W ., Wu Y ., Yang Z ., Henikoff JG, Henikoff S ., Jiang J: CentO卫星在水稻着丝粒CenH3核小体上的翻译和旋转相位。中国科学(d辑:地球科学),2013,40(5):591 - 598。10.1073 / pnas.1319548110。
- 44.
Takeuchi K, Nishino T, Mayanagi K, Horikoshi N, Osakabe A, Tachiwana H, Hori T, Kurumizaka H, Fukagawa T:中心点核小体样CENP-T-W-S-X复合物诱导阳性超螺旋进入DNA。核酸学报,2014,42(3):1644-1655。10.1093 / nar / gkt1124。
- 45.
Carroll CW, Milks KJ, Straight AF: CENP-A核小体的双重识别对于着丝粒组装是必需的。中国生物医学工程学报。2010,31(4):457 - 461。10.1083 / jcb.201001013。
- 46.
Vermaak D, Hayden HS, Henikoff S: Cid组蛋白折叠域内的着丝粒靶向元件。中国生物医学工程学报。2002,22(21):7553-7561。10.1128 / mcb.22.21.7553 - 7561.2002。
- 47.
中心性组蛋白中组蛋白折叠结构域的适应性进化。中国生物医学工程学报。2004,21(9):1712-1718。10.1093 / molbev / msh179。
- 48.
Shelby RD, Vafa O, Sullivan KF: CENP-A组装到着丝粒染色质需要一个核小体DNA接触位点的合作阵列。中国生物医学工程学报。1997,13(3):501-513。10.1083 / jcb.136.3.501。
- 49.
Lermontova I, Schubert V, Fuchs J, Klatte S, Macas J, Schubert I:拟南芥着丝粒组蛋白CENH3的加载主要发生在G2期,需要组蛋白折叠结构域的存在。植物学报,2006,18(10):2443-2451。10.1105 / tpc.106.043174。
- 50。
Black BE, Foltz DR, Chakravarthy S, Luger K, Woods VL, Cleveland DW:形成着丝粒染色质的结构决定因素。自然科学学报。2004,30(6):578-582。10.1038 / nature02766。
- 51。
Lermontova I,KoroLeva O,Rutten T,Fuchs J,Schubert V,Moraes I,Koszegi D,Schubert I:敲低Cenh3.在拟南芥中通过干扰减数分裂染色体分离减少有丝分裂并引起不育。植物学报,2011,68(1):40-50。10.1111 / j.1365 - 313 x.2011.04664.x。
- 52。
Chen C, Murata M, Chan SW:减数分裂特异的着丝粒特异性组蛋白CENH3的负载拟南芥蒂利亚纳.Plos Genet。2011,7(6):E1002121-10.1371 / journal.pgen.1002121。
- 53。
Chen Y, Baker RE, Keith KC, Harris K, Stoler S, Fitzgerald-Hayes M:着丝粒h3样蛋白Cse4p的N端具有与组蛋白折叠结构域不同的基本功能。中国生物医学工程学报。2000,20(18):7037-7048。10.1128 / mcb.20.18.7037 - 7048.2000。
- 54。
Bailey AO, Panchenko T, Sathyan KM, Petkowski JJ, Pai P-J, Bai DL, Russell DH, Macara IG, Shabanowitz J, Hunt DF, Black BE, Foltz DR: CENP-A的翻译后修饰影响着着丝粒染色质的构像。Proc Natl Acad Sci U S a2013,110(29):11827-11832。
- 55。
《棉花部落的一致性与共识》。系统机器人。1997,22:259-290。10.2307 / 2419457。
- 56。
王k,王z,李f,ye w,王j,歌,yue z,cong l,尚h,zhu s:二倍棉草案的基因组草案Gossypium raimondii..acta photonica sinica, 2012, 44(10): 1098-1103。10.1038 / ng.2371。
- 57。
Goodstein DM, Shu S, Howson R, Neupane R, Hayes RD, Fazo J, Mitros T, Dirks W, Hellsten U, Putnam N: Phytozome:绿色植物基因组学的比较平台。核酸学报2012,40(1):1 - 4。10.1093 / nar / gkr944。
- 58。
Benson DA, Karsch-Mizrachi I, Lipman DJ, Ostell J, Wheeler DL: GenBank。核酸学报2008,36(增刊1):D25-D30。
- 59。
Delport W, Poon AF, Frost SD, Pond SLK: Datamonkey 2010:进化生物学的一套系统发育分析工具。生物信息学。2010,26(19):2455-2457。10.1093 /生物信息学/ btq429。
- 60.
Lysak MA, Koch MA, Pecinka A, Schubert I:在芸薹属部落中发现的染色体三倍性。中国生物医学工程学报,2015,30(4):516-525。10.1101 / gr.3531105。
- 61.
棉花属植物的快速多样化(Gossypium:锦葵科),分析了16个核和叶绿体基因。acta botanica sinica, 2010, 32(4): 489 - 494。10.3732 / ajb.89.4.707。
- 62.
Sanderson M:在没有速率常数的情况下估计发散时间的非参数方法。中国生物医学工程学报。1997,14(12):1218-10.1093/牛津学报。molbev.a025731。
- 63.
Jacquemin J, Ammiraju JSS, Haberer G, Billheimer DD, Yu Y, Liu LC, Rivera LF, Mayer K, Chen M, Wing RA: 1500万年的演化oryza.属显示广泛的基因家族扩展。摩尔的植物2013年,7(4):642 - 656。
- 64.
刘斌,刘斌,荣杰,Noyes RD, Paterson AH, Wing RA, Wilkins TA, Wendel JF:多倍体核基因的速率变异与年龄Gossypium.中国生物化学学报,2003,20(4):633-643。10.1093 / molbev / msg065。
- 65.
Lyons E,Freeling M:如何使用与DNA序列相比将同源植物基因和染色体进行比较。工厂J. 2008,53(4):661-673。10.1111 / J.1365-313x.2007.03326.x。
- 66.
李志强,王旭东,唐洪涛,王旭东:拟南芥与番木瓜、杨树、葡萄等外类群的同源区域的发现与比较:拟南芥与蔷薇。植物营养与肥料学报。2008,29(4):441 - 446。10.1104 / pp.108.124867。
- 67.
陈志刚,陈志刚:南美北部古新世马尔vaceae及其生物地理意义。植物学报。2011,98(8):1337-1355。10.3732 / ajb.1000539。
- 68.
Thompson JD, Higgins DG, Gibson TJ: CLUSTAL W:通过序列加权、特定位置间隙惩罚和权重矩阵选择来提高累进式多序列比对的灵敏度。核酸学报1994,22(22):4673-4680。10.1093 / nar / 22.22.4673。
- 69.
大厅TA:BioEdit:适用于Windows 95/98 / NT的用户友好的生物序列对齐编辑器和分析程序。核酸研讨会系列。1999,95-98。
- 70。
jModelTest 2:更多的模型,新的启发式和并行计算。中国生物医学工程学报。2012,9 (8):772-10.1038/nmeth.2109。
- 71。
Guindon S,Gascuel O:一种简单,快速,准确的算法,可以通过最大可能性来估计大的系统发育。系统中的生物学。2003,52(5):696-704。10.1080 / 10635150390235520。
- 72。
Kumar S,Nei M,Dudley J,Tamura K:Mega:一种以生物学家为中心的软件,用于DNA和蛋白质序列的进化分析。简短生物形式。2008,9(4):299-306。10.1093 / bib / bbn017。
- 73。
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S: MEGA6:分子进化遗传学分析版本6.0。中国生物医学工程学报,2013,30(12):2725-2729。10.1093 / molbev / mst197。
- 74。
马建军,王志强,王志强:植物着丝粒结构与功能的动态关系。电子学报。2007,23(3):134-139。10.1016 / j.tig.2007.01.004。
- 75。
Källberg M ., Wang H ., Wang S ., Peng J ., Wang Z ., Lu H ., Xu J:基于RaptorX web服务器的蛋白质结构建模。网络协议。2012,7(8):1511-1522。10.1038 / nprot.2012.085。
- 76。
彭建军,徐建军:一种基于多模板的蛋白质丝线构建方法。蛋白质。2011,79(6):1930-1939。10.1002 / prot.23016。
- 77。
彭建军,徐建军:Raptorx:利用结构信息进行蛋白质比对的统计推断。蛋白质。2011,79 (S10): 161-171。10.1002 / prot.23175。
- 78。
一种新合成的棉花异源四倍体基因的器官特异性沉默。中国生物医学工程学报。2004,27(4):427 - 434。10.1534 / genetics.104.033522。
- 79。
Yoo MJ,Szadkowski E,Wendel JF:各种多联块棉花的同源表情偏见和表达水平优势。遗传。2013,110(2):171-180。10.1038 / hdy.2012.94。
- 80.
Wu Td,NaCu S:快速和SNP耐受复杂变种和短读拼接。生物信息学。2010,26(7):873-881。10.1093 / Bioinformatics / BTQ057。
- 81.
Page JT, Gingle AR, Udall JA: PolyCat:来自异源多倍体生物体的基因组测序阅读分类资源。G3。2013, 3(3): 517-525。10.1534 / g3.112.005298。
致谢
作者感谢Patrick Edger的意见和建议芸苔,胡冠景求助于世Gossypium马特·赫福德(Matt huffford)帮助进行进化分析。这项研究得到了美国国家科学基金会植物基因组计划和棉花公司的支持。
作者信息
从属关系
通讯作者
额外的信息
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
作者的贡献
RM参与了实验设计,基因测序,克隆,表达分析,并起草了手稿。JG参与了进化分析和统计的概念,进行了系统发育分析,并参与了手稿的起草。JJ克隆和测序了许多CenH3基因,并参与了手稿的起草。SRB参与了CoGE和模因分析的实验设计和监督,并参与了手稿的起草。CG参与了实验设计,并帮助起草了手稿。LG参与了部分克隆工作,进行了所有的统计分析,并编写了分析方法。JW参与了实验设计,进化分析,并帮助起草了手稿。所有作者阅读并批准了最终的手稿。
电子辅料
CENH3中的基因组序列长度
附加文件1:Gossypium和酸脂酸。Gossypium cenh3登录数和基因长度。(多克斯13 KB)
系统源信息替代拼接变体
附加文件2:Gossypium异源多倍体。剪接变异体的Gossypium分组来佐证。(多克斯12 KB)
CenH3发展史。
附加文件3:GossypiumCenH3发展史。CenH3系统发育Gossypium二倍体和多倍体植株。的发展史Gossypium使用基因Cenh3.序列。的Gossypiumphylogyy与先前公布的文学系统有关[19.排除了在AT子树中看到的多切分,这是CenH3基因中信息性snp太少的结果。一个是G. Arboreum.,b是G.异常C是G. robinson, D是G. raimondii, E是g . stocksii,f是g . longicalyxG是g . bickii,k是G. exiguum.[K], AD1G. Hirsutum., AD2g .取得, AD3粉g . tomentosum, AD4G. Mustelinum,AD5是G. Darwinii, Gk是Gossypioides Kirkii..底部有一个比例尺;每个节点上的数字是引导支持值。(TIFF 366 KB)
各种子聚合物中每个亚基因组的克隆数量
附加文件4:Gossypium.各亚基因组中CenH3克隆的数量Gossypium异源多倍体。(多克斯13 KB)
每个多倍体基因组中具有剪接变异的克隆的百分比。
附加文件5:每个多倍体基因组中包含剪接变异的克隆总数的百分比。在每个物种的克隆体中发现的剪接变异的百分比。每个物种的无性系总数为芽组织和叶组织的无性系总数,如果两者都可用的话。AD1-AD5表示陆地革兰,巴巴多斯革兰,绒毛革兰,野马革兰,和g . darwinii,分别。(JPEG 415 KB)
从GenBank和Phytozome下载。
附加文件7:物种名称和登录号芸苔和oryza.CenH3序列。(多克斯18 KB)
作者的原始提交的图像文件
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。
除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。
要查看本许可证的副本,请访问https://creativecommons.org/licenses/by/4.0/.
创作共用及公共领域专用豁免书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
梅森布林克,r.e.,加拉格尔,j.p.,亚雷切克,J.J.et al。CENH3在三个Agiosperm属的二倍体和多倍体中的演变。BMC植物BIOL.14,383(2014)。https://doi.org/10.1186/s12870-014-0383-3
已收到:
接受:
发表:
关键字
- 多元化的选择
- CenH3进化
- 可变剪接
- 着丝粒
- Gossypium
- 芸苔
- oryza.