摘要
背景
钙离子是植物中功能最广泛的二级信使,参与调控植物生长发育的不同阵列,以及生物和非生物胁迫反应。钙调磷酸酶b样蛋白是钙传感器中最重要的基因之一。
结果
在这项研究中,我们发现了38种不同的植物物种的钙素素B样基因家族成员,并为每个植物物种分配了独特的术语。序列分析表明,CBL蛋白含有三种结合的EF手结构域,其含有几个保守的ASP和Glu氨基酸残基。发现CBL蛋白的第三次EF手用于占有D / E-X-D钙结合传感器基序。系统发育分析表明,CBL基因落入六种不同的基团。另外,除B CBL之外,发现所有CBL蛋白含有N-末端棕榈酰基和肌苷蛋白。一种进化研究表明,CBL基因由共同的祖先演变,随后在陆地植物的演变过程中分歧。Tajima的中性测试表明,由于均衡选择,CBL基因是高度多态性的,并且通过降低群体大小而通过降低。冷热应激处理的差异表达分析导致偏振基因的差异调节。
结论
植物CBL基因的基本结构在整个植物王国中受到保守。进化分析表明,这些基因由低真核生物谱系的共同祖先演变,并导致较高真核生物中的钙信号传导事件的扩大。
背景
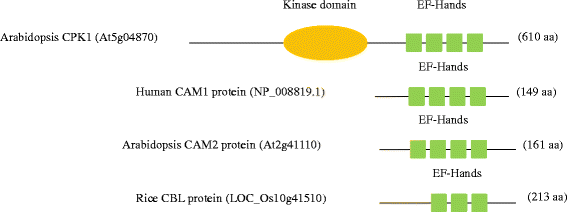
在各种生物过程中,钙信号由于其强大的稳态机制而作为细胞内二次信使的重要作用,其维持了细胞内的CA2+浓度(1].钙离子的浓度在静息细胞和细胞器中的毫米范围内变化在30至400nm。2- - - - - -4].为胞质钙2+离子从细胞质运输到细胞的其他部分时,需要维持低水平的细胞水平。这可以通过Ca的作用来实现2+- atp酶泵,输送Ca2+离子从血浆膜中的细胞外,以及莎草内质网Ca2+-泵Ca的atp酶2+进入内质网管腔[3.].有报道称,一旦细胞开始使用高效磷酸盐化合物作为代谢货币,维持细胞内Ca的低水平将面临巨大挑战2+[5]防止钙和磷酸盐在细胞溶胶中沉淀,最终形成固体,骨状结构。自加州以来2+离子是一种多功能的信号离子,它在植物的信号级联中发挥不同的作用来调节基因表达[6].事实上,Ca2+信号是植物生长、发育以及生物和非生物胁迫的重要调节因子[7].由Ca编码的信令信息2+离子通过钙离子传感器进行解码和传输2+结合蛋白(8,9].这种传感器绑定CA.2+并改变它们在Ca中的构象2+在高水平Mg存在的情况下2+和单价阳离子[1,10].一些钙传感器包括(i)钙依赖性蛋白激酶(CPK),(II)钙调节液(凸轮)和(III)钙素B样蛋白(CBL)[7,11].CPK是单体蛋白,具有独特的结构,其含有五个结构域,(i)n-末端可变结构域,(ii)激酶结构域,(iii)一种自动抑制域,(iv)调节结构域和(v)c-终端域。CPK的监管领域的特点是存在四个CA2+拉伸因子-手。ef -手是一种钙传感器,其特征是存在保守的Asp (D)或Glu (E)残基[7].EF-HAND MOTIF在每次EF手中具有36个氨基酸残基的螺旋环 - 螺旋结构。与CPKS不同,CAM和CBL是缺少效应激酶结构域的小蛋白质(图。1).凸轮包含四个Ca2+而CBL包含三个ef -hand(图。1)[12].将Ca2+信号,CPKs, CBLs和cam与它们的靶蛋白相互作用,并调节它们的基因表达[13].这些靶蛋白可能是蛋白激酶、代谢酶或细胞-骨骼相关蛋白。CIPKs (CBLs - interaction protein kinase)是CBLs的重要靶蛋白[14].
不同钙结合传感器蛋白的一般结构。(一个)一个拟南芥CPK蛋白含有激酶结构域和四个钙结合ef手,(b)人钙调蛋白(CAM)蛋白含有四只EF手,(c)拟南芥蒂利亚纳CaM2蛋白含有四个ef手,(d)大米CBL蛋白含有三个ef手。从图中可以清楚地看出,CaM蛋白包含四个钙结合EF手,而CBL蛋白包含三个。这里展示了人类CaM蛋白,以确定人类CaM蛋白与植物CaM蛋白的确切相似性和植物CaM蛋白与CBL蛋白的区别。在SCAN PROSITE (http://prosite.expasy.org/scanprosite/)软件检查是否存在钙绑定的EF手
虽然对CBL基因的作用进行了大量的研究,但对确定这些基因的确切特征所做的努力却很少。因此,在本研究中,我们从38种不同的植物中鉴定了CBL基因家族成员,并对其进行了独特的命名系统。此外,我们还对这些CBL基因的基因表达、基因组学、系统发育和进化方面进行了研究。
结果与讨论
CBL基因的命名
迄今为止,根据其识别的序列号已经命名了特定基因家族的不同成员。如果迄今为止未鉴定给定植物物种的CBL基因,则不管与已知对应基因的正交序列相似如何,所鉴定的第一个鉴定为CBL1,下一个作为CBL2等。基因组序列数据的体积每天增加,为基因组学研究提供优异的平台。然而,对于特定基因或基因家族的缺乏系统性命名系统导致了解越来越多的基因组信息,导致了混淆和困难。例如,ATCBL1基因差异地调节盐,干旱和冷响应拟南芥[15],但尚不清楚OsCBL1基因是否也具有相同的功能。原则上,序列相似性决定基因的结构相似性,结构相似性决定基因的功能相似性[16,17].因此,AtCBL1和OsCBL1可能赋予或多或少类似的功能。然而,由于缺乏一个合适的命名系统,很难正确理解它的功能。正交理论使功能信息从实验特征蛋白转移到非特征蛋白具有合法性[18,19].因此,本研究中发现的所有CBL基因均采用基于同源学的命名系统,由不同的研究者提出[7,20.- - - - - -23].在此系统中,拟南芥蒂利亚纳和奥雅萨苜蓿CBL蛋白序列被视为直晶类查询基因。在命名系统中,属的第一个字母保持大写,并且物种的第一个字母保持小写,然后是CBL然后A. Thaliana.或奥雅萨苜蓿基于CBL基因数。单子叶植物是根据o .漂白亚麻纤维卷,而双子叶植物和其他植物则是根据A. Thaliana.。在单焦植物的情况下,根据正交基因分配CBL基因数栽培稻。如果在特定物种中发现了多于一个正牙,则使用额外的数量,然后用连字符分辨率。当生物体属的第一个字母与另一个生物吻合时,属的第一个字母保持恒定,第一个,第二,第三或第四个字母或包括物种的第一,第二,第三和第四个字母被考虑在考虑。例如,CBL基因危害风疹被命名为CrCBL,而Chlamydomonas Reinhardtii.命名为CreinCBL。在这种情况下,属和种的名称的字母是互相重合的。因此,CBL基因c . reinhardtii被称为creincbl。该命名系统还可以提供有关相关的完全物种的信息。一种物种的独特外置基因可以类似于另一种物种的正交对应基因,并且经过相似的细胞功能。相同的方法通常用于预测新测序基因及其蛋白质产品的潜在功能。难以研究所有CBL基因在具有不同功能方面的所有植物种类中的作用。因此,CBL基因的基于局部的术语命令系统将有助于提供对应正交基因所需的基本信息。
CBL基因基因组学
一个物种的基因组被认为是一个包含所有必要信息的基因包,以弥补基因型和表现型之间的差距[24].在未来十年,几乎所有被子植物以及重要的绿藻、苔藓植物、蕨类植物和裸子植物的基因组序列将完成。这些基因组序列将成为有价值的工具,可以提供一个强大的框架,将基因组水平的事件联系起来,以破译导致从水生生境到陆地生境的形态和生理变化。对38种不同植物CBL基因的全基因组分析显示,共有328个CBL基因(见表)1).在这其中,g . raimondii就发现在高地植物中含有最高数量的CBL基因(13)。较低的藻类喜欢衣藻和Micromonas在其基因组中分别仅含有2和3个CBL基因。苔藓植物,Physcomitrella金属盘,和蕨类植物,Selagnella moellendorffii,仅编码四种CBL基因。发现的CBL基因的数量p .金属盘符合Kleist的研究et al。[25].型号裸子植物植物,Picea amies,编码13个CBL基因。生物体的基因组大小因物种而异(见表)1).在单码子植物中,玉米拥有最大的基因组(2500 Mbs),编码9个CBL基因,而在双子叶植物中,大豆拥有最大的基因组(975 Mbs),编码9个CBL基因。裸子植物的基因组大小Picea amies为1960 Mbs,编码13个CBL基因。类似地,双子叶植物危害风疹具有最小的基因组(134.8 MBS),仍然在其基因组中含有9个CBL基因。从本研究开始,显然,植物中CBL基因的基因组大小和数量之间没有相关性。在蓝绿藻的情况下micromonas pusila.,其基因组大小为22 MBS,仍然含有3个CBL基因,而基因组大小Chlamydomonas Reinhardtii.是118.8 MBS,只含有2个CBL基因。其基因组中特定数量的CBL基因的存在与基因组大小无关,并且可能与植物的功能进化要求相关。本研究期间鉴定的所有CBL基因仅含有三种钙结合EF手。在我们的调查中,我们没有发现绿藻种类的任何CBL基因Coccomyxa subellipsoidea,Ostreococcus lucimarinus或Volvox Carteri.。CBL基因在其基因中最多包含6、7、8或9个内含子;而只有少数CBL基因是无内含子的(附加文件1).CBL基因Picea amiesintronless。本研究中发现的其他低真核生物无内含子CBL基因来自m . pusila(mpcbl2),p .金属盘(PpCBL3-3)和美国moellendorffii(SmCBL5),而更高的真核无内含子CBL基因S. lycopersicum.(SLCBL3-3)和美国tuberosum(stcbl3-3)(附加文件1).CBL基因F. VESCA.FvCBL4是最大的CBL基因,其开放阅读框长度为3048个核苷酸,编码1015个氨基酸。同样的,CBL基因M. Domestica.MDCBL5编码最小的CBL基因,并且仅包含426个核苷酸ORF,其编码141个氨基酸。基因组z梅斯是最大的一个,只包含9个CBL基因,而m . pusila最小的一个,只有两个CBL基因。然而,如表所示1在美国,更大的基因组大小并不与更多的CBL基因数量成正比。CBL蛋白分子量在12.774 (PaCBL10) ~ 115.266 (FvCBL4) kDa之间,等电点(pI)在4.02 ~ 9.61之间。大多数CBL蛋白是酸性的(附加文件2).基于CBL蛋白的平均氨基酸组成,发现最重要的钙感测氨基酸,ASP(D)和Glu(E)分别为8.07和8.94(表2).CBL蛋白中色氨酸和胱氨酸的平均丰度分别为0.62和1.27。
植物的基因组尺寸非常多样化,不同于物种的种类,尺寸范围为63(Genlisea Aurea)至149,000 mb (巴黎japonica),分为n= 2 to约n= 600条染色体,在一个物种内保持不变[26].在本研究中,我们发现双子叶植物拟南芥蒂利亚纳和番木瓜(135 MBS)具有最小的基因组大小,而在单子叶植物中玉米(2500 MBS)具有更高植物中最大的基因组大小。较低的真核生物藻类,micromonas pusila.(22 MBS),含有所研究的物种中最小的基因组。裸子植物的特征在于存在非常大的基因组(高达35,000 MB),Picea amies含有1960 MBS基因组[27].尽管裸子植物的基因组更大,但它们的染色体数目并不多,在2n = 2x = 14-28之间。拟南芥基因组测序是基于基因和基因序列拟南芥与其他植物的相似,后来发现这是真的;然而,蛋白质编码基因的数量差异显著。在这项研究中也发现这是正确的,因为特定植物的特定基因家族的蛋白质编码基因的数量不同。植物的核DNA由编码序列、内含子、启动子和调控DNA序列的低拷贝数组成[26].在本研究中,我们发现大多数CBL基因内存在6、7或8个内含子,这表明一个特定基因家族内的内含子数量因物种而异,与其对应的同源基因也不同。
众所周知,单个基因和整个基因组的核苷酸组成可能存在显著差异[28,29].蛋白质的突变过程和蛋白质的功能与功能的关系被认为是氨基酸组成的主要决定因素和蛋白质进化速率[30.].自然选择事件通常通过偏爱编码特定基因区域特定氨基酸的密码子来增强蛋白质的特异性和稳定性[31].然而,蛋白质结构和组成的代谢限制可能包括氨基酸生物合成的能量成本。像色氨酸这样的芳香族氨基酸的生物合成需要更高的能量(74.3单位),因此每个CBL基因色氨酸的平均丰度只有0.62个氨基酸[30.].合成TRP氨基酸需要高能量,因此植物每CBL蛋白仅编码0.6个氨基酸,以避免额外的能量费用。类似地,12.7和15.3单位能量分别需要ASP和Glu氨基酸的生物合成。ASP和Glu氨基酸的生物合成成本相对较低;因此,每个CBL蛋白分别编码8.07和8.94个氨基酸的植物。由于植物使用大量的氨基酸生物合成的能量,因此有利于在其蛋白中编码较低的昂贵的氨基酸[30.].
保守EF手
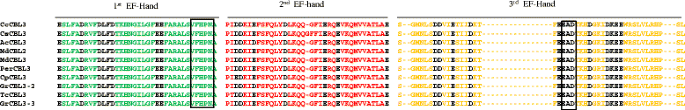
CBL蛋白的多序列对准显示出几种新的保守结构域和基序的存在。植物王国的CBL蛋白仅含有三个EF手域并节省保守。总的来说,每个EF手段的长度为36个氨基酸,并且具有螺旋环螺旋结构[32].每个螺旋环包含12个氨基酸;因此,每个EF-hand含有36个氨基酸。多序列比对显示,Asp (D)氨基酸在第一ef手第7和11位的保守性不显著,而在第14位的保守性最显著(图2)。2额外的文件3.).此外,Asp (D)/Glu (E)氨基酸在22和25位保持保守。其他几种氨基酸也在第一ef手中保存。而钙敏感氨基酸Asp (D)和Glu (E)则是研究的重点。如果我们考虑CBL蛋白中存在保守结构域,则在第一ef手末端有一个保守的V-F-H-P-N结构域(图1)。2).在第二次EF手中,ASP / Glu氨基酸在3,4和7位置略微保守,但ASP在14位显着保守(图。2).Glu氨基酸在22位的保守性最显著,在25位的保守性不显著。谷氨酸在36位也显著保守。在第三层EF-hand中,氨基酸在7、8、14位保守;而Glu在11、19、20、21和22位点保持保守(图5)。2).Asp和Glu氨基酸以D/E-x-D基序出现在第三手的20、21和22位。另一个基序D-x-E-E出现在第三只EF-hand的30、31、32和33位置。综上所述,第三ef手氨基酸中含有最多的Asp和Glu氨基酸。在EF-hand环中,钙离子呈五角形双锥体结构。前期对CPK EF-hand的研究发现,每个EF-hand中有6个氨基酸残基参与钙离子的结合,分别位于1、3、5、7、9和12位[7].这些留数用X, Y, Z, - Y, - X和-Z表示。位于第12位的不变的谷氨酸或Asp氨基酸为配体Ca提供了两个氧分子2+(二齿配体)离子[7].位置1(x),3(y)和12(-Z)是最保守的并且在钙结合中起着关键作用。在CBL的情况下,位于7,14和22的ASP或Glu氨基酸的存在对于结合钙离子非常关键,而其他保守的ASP和Glu氨基酸可以为辅助亲和力部位提供强钙结合。
图中显示CBL蛋白中存在三个ef手。其中绿色为第一ef手,红色为第二ef手,橙色为第三ef手。CBL蛋白ef -hand中保守的Asp (D)和Glu (E)氨基酸的存在使钙离子结合。CBL蛋白的三个EF-hand中,第3个EF-hand包含E-E-x- d和D-x-D/E钙结合基序。EF-hands中存在的所有保守氨基酸(D和E)和基序均以黑色标记。在第一个EF-hand氨基酸中,Glu (E)氨基酸被保存在1,23和24位,Asp (D)氨基酸被保存在6,10和13位。在二手ef中,Asp/Glu氨基酸在3、4和7位保守,而Asp氨基酸在14位显著保守。谷氨酸在22位点的保守性最显著,在25位点的保守性不显著。在第36位,Glu氨基酸明显保守。第3个EF-hand D-D-x-x-E motif位于7、8、9、10和11位。 Asp (D) amino acid is conserved at 15 and 26 position. The E-E-x-D motif is present at 19, 20, and 21 and 22 and D-x-E-E motif is present at 30, 31, 32 and 33 position respectively. The abundance of Asp and Glu amino acids are much more in 3 EF-hand when compared to 1 and 2 EF-hand
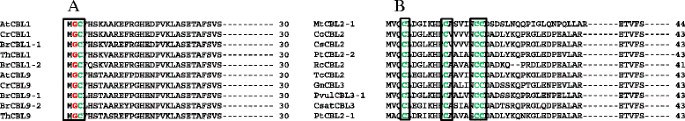
在CBL蛋白的第一个ef手附近有一个上游区域(图)。3.).该上游区域没有明显的保守性,但包含一些钙结合的Asp和Glu氨基酸(附加文件)3.).发现D组CBL蛋白在n端16、17和18位(E-E/D- p)含有保守的Asp和Glu(图)。3.一种)。在CBL蛋白组中,存在存在于第一EF手(n末端区域)的上流的D / E-X-E / D基序(图。3.b).一个保守度较低的域E/D-D-P-E-X4-前任6-e存在于CBL蛋白的N末端区域(附加文件3.).在C末端区域中,存在存在于第三EF手下下游的保守的P-S-F-V-F-X-S-E-V-D-e-e域(图。4).
CBL蛋白c端存在保守的P-S-F-V-F-x-S-E-V-D-E结构域。有关保守序列的更多细节,请参阅附加文件3.
这些生物能够识别感官并对环境做出反应以生存。在植物中,感知机制是对激素和环境信号作出反应而进化出来的[33].要引起细胞反应,所感知的信号必须传递到细胞机制。Ca是最重要的次级信使之一2+,感知刺激,并将其转导给下游蛋白,启动Ca2+介导的反应。Ca2+介导性刺激使植物对激素和外部刺激作出反应,这些激素和外部刺激介导和调节各种基本的细胞过程,如细胞分裂、细胞伸长、细胞分化、细胞极性、光形态发生、植物防御和应激反应[31].CBL蛋白是钙敏感蛋白家族之一,包括钙依赖蛋白激酶(CPK)和钙调蛋白。CPK蛋白包含一个激酶结构域和一个调节结构域,该结构域具有四个钙感知ef手。ef手上的酸性氨基酸Asp (D)和Glu (E)是重要的钙传感器[34].CBL蛋白缺乏激酶结构域,只含有三个钙结合ef手。的CBL蛋白拟南芥蒂利亚纳和奥雅萨苜蓿先前据报道,含有四个钙结合的EF手[35- - - - - -37].然而,扫描蛋白酶软件研究发现,所有植物的CBL蛋白仅含有3个钙结合EF-hand结构域(fig .)。1和5)[38].库德拉CBL蛋白的研究et al。[35],分手和Kudla [39)和顾et al。[37研究发现,所有被报道有四个ef手的CBL蛋白实际上只包含三个ef手。他们报告说,在某些情况下,CBL蛋白含有四个ef -hand,而在其他情况下,它们含有不完整的四个ef -hand。Weinl和Kudla提供的数据的原石分析[40)表明,O. TAURI.蛋白质中含有清晰的四撇子美国moellendorfii蛋白质只有三个ef手。一个CBL蛋白含有四个ef手,而其他蛋白含有三个ef手。这是非常矛盾的。这证明,Weinl和Kudla提供的数据是矛盾的。本手稿提供的其他资料均属属Physcomitrella金属盘(FJ901251, FJ901252, FJ901253和FJ901254)。在这里,p .金属盘FJ901254蛋白含有4个ef -hand,而其他蛋白只有3个ef -hand。CBL基因来自单细胞衣藻到现代土地植物。这衣藻被认为是早于陆生植物的3500万年前进化而来的光合绿色植物的基本进化谱系。因此,在超过3500万年的时间里,基因组不太可能对功能不完整的ef手进行编码。基因组在本质上是非常特定的。他们要么为完整的ef手编码,要么删除不完整的ef手。但是什么也没有发生;因为在cbl中不存在这样不完整的ef手。进化压力不可能让不完整和无功能的ef手在数百万年里转移。这证明了CBLs蛋白只含有三个钙结合ef手,而不是四个或不完整的四个。
虽然我们对CBL蛋白的理解具有显着进展,但没有关于其保守域和主题的研究。在这项研究中,我们发现钙结合EF手高度保守并含有第三EF手中的E / D-X-D基序(图。2).除了这个基序,CBLs还包含几个c端下游的保守基序,特别是保守的Asp和Glu氨基酸(图)。3.a和b). CBLs中高比例的Asp和Glu氨基酸为Ca的调节提供了机会2+离子。
豆蔻酰化和棕榈酰化位点
蛋白质豆蔻酰化和棕榈酰化是蛋白质运输、稳定和聚集所必需的两个重要事件[41].向N-末端Gly氨基酸添加肉豆蔻酸导致蛋白质myRistoylation,同时向N-末端Cys氨基酸加入棕榈酸导致蛋白棕榈酰化[42].在大多数研究的CBLs中,n端甘氨酸是蛋白质肉豆芽酰化所必需的,并且在第二位置被保守(图2)。3.).已经发现在一些其他CBL蛋白中的N-末端Gly氨基酸在第七位置被保守。类似地,N-末端Cys氨基酸是蛋白质棕榈酰化所必需的,并且在D CBL蛋白组中的第三位保守(图。3.a)和在CBL蛋白组中的第四位置(图。3.b)。B组大多数BBLS不含N-末端CYS氨基酸。
蛋白质棕榈酰基化是在膜结合蛋白中发现的广泛修饰,其包括在可溶性核糖体中合成的跨膜 - 跨度蛋白质[43].通常,蛋白质棕榈酰基化增加蛋白质对膜附着的亲和力,因此影响蛋白质定位和功能。经过棕榈酰化的蛋白质包括Rasgtpase [44],rho gtpase [45]及CDPKs [7].RasGTPase, Rho GTPase和CDPKs在第三、第四或第五位包含n端Cys残基[46].所有的24拟南芥CPKs被预测具有豆蔻酰化共识序列,并在第4、5或6位包含至少一个Cys残基[47].该研究显示在几个CBL中的第三,第四,第五或第六位存在n末端Cys残基(图。3.和5A和B)。除B CBL(CBL10)组外,所有其他CBL蛋白(A,C和D)含有N-末端Cys残基。这些发现清楚地表明,B组CBL蛋白不能经历蛋白质棕榈酰基,并且只有选择性CBL蛋白质POSSE的蛋白棕榈酰基化活性。
肉豆蔻酰化通过酰胺键将肉豆蔻酸酯加到n端甘氨酸氨基酸上称为肉豆蔻酰化[42].除B组CBLs外,其他CBLs均在第2位含有n端甘氨酸残基(图2)。5一种)。另外,含有N-末端半胱氨酸氨基酸的所有CBL(B组CBL)在第二位置同时具有N-末端Gly氨基酸(图。3.和5).n端肉豆蔻酰化促进蛋白质-膜的附着和蛋白质-蛋白质的相互作用。n端gly突变可消除脂质修饰,从而阻止膜结合[48].二十四个拟南芥钙敏感CDPK蛋白预测有n端肉豆蔻酰基序用于膜结合。其中,AtCPK2已被实验证实在n端甘氨酸残基上肉豆素化,CPK蛋白的前10个氨基酸对内质网膜定位至关重要[49].在大多数病例中,N末端肌肉和棕榈酰胺事件彼此补充。在第2位的甘氨酸氨基酸中的N-末端MyRistoylation在第2位和Cys氨基酸中的棕榈酸盐在第4和5位,已经通过实验验证了膜结合的OSCPK2 [48].当通过甘氨酸氨基酸的突变废除N-末端MyRistoylation时,蛋白质不能再棕榈酰化,表明N-末端MyRistoylation是棕榈酰基的先决条件。只有蛋白质myRistoylation为膜附着提供弱亲和力,而棕榈酰基和肌脲提供非常高的亲和相互作用[48].
系统与进化
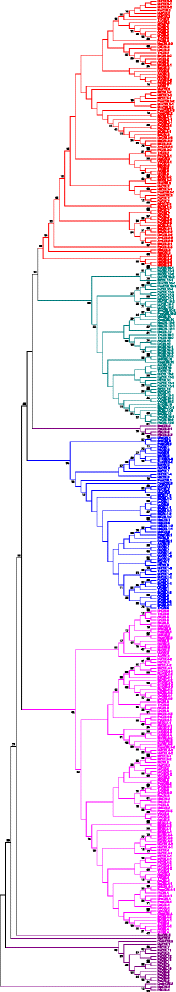
蛋白质家族定义为具有超过50%氨基酸序列相似性的蛋白质组[50.].通常在基因的单个基因或家族的水平下研究分子演化[51.].然而,仍然没有模型可以推断基因家族的进化,以使估计祖先的状态。系统发育分析是一种强大的工具,可以推断基因之间的关系和分析它们的进化事件[52.].对所有CBL基因的系统发育分析显示,它们可分为6个不同的组(图)。6).一些较低的真核特异性CBL基因如SmCBL9、PpCBL3-1、PpCBL9和PpCBL3-2作为一个簇(F组)存在于系统发育树的中心,而E组CBLs存在于系统发育树的远端。高等真核植物其他CBL基因簇(A、B、C、D组)与F组CBL基因簇直接相连(图1)。6额外的文件5).这些发现表明,较高真核植物的CBL基因系列来自下真核植物的共同祖先(图。6).下部真核植物非常简单,具有单细胞到多蜂窝架构。随着有机体的复杂性增加,需要使水生栖息地的复杂性栖息地适应复杂的陆地栖息地,因此每个基因组的CBL基因数量增加[53.].这表明这些CBL基因可能已经演变,用于一些独特和特定的功能,负责适应复杂的生活方式。低真核植物如藻类的CBL基因,Physcomatrella.,Selaginella.和松果体在E和F组中属于e。这些基因可能在进化期间独立进化。下真核植物的一些CBL基因(SMCBL9,PPCBL3-1,PPCBL9和PPCBL3-2)落在系统发育树的中间,而高性植物植物的CBL基因是与下真核生物的CBL基因簇的系统饱和连接植物。这些发现表明,现代植物的CBL基因可能来自下真核生物植物的共同祖先[54.].系统发育分析显示,CBL2,CBL3,CBL6和CBL7落入A组,CBL10落入B组,CBL1和CBL9落入C组,CBL4,CBL5和CBL8落入D组D.下核心CBL基因Selaginella.(SmCBL5),Micromonas(MpCBL2 MpCBL6),衣藻(CreinCBL8, CreinCBL9)和CBL基因Picea amies属于E组和F组。
CBL蛋白的系统发育树。系统发育分析表明,CBL蛋白可分为5个不同的分支。根据CBLs在系统发育树中从上到下的存在情况进行分类,并用颜色标记;A组(红色)、B组(绿色)、C组(蓝色)、D组(玫红色)、E、F组(紫色)。不同的CBL蛋白分布在不同的组;A组(CBL2、CBL3、CBL6、CBL7)、B组(CBL10)、C组(CBL1、CBL9)、D组(CBL4、CBL5、CBL8)、E、F组为低真核特异性CBLs。利用MEGA5软件构建系统发育树。构建系统发生树的统计参数如下:系统发生检验、bootstrap法;靴带复制数量,2000;模型/方法,Jones-Taylor Thornton (JTT); missing data treatment, partial deletion; ML heuristic method, nearest neighbor-interchange (NNI) and branch swap filter, very strong. Detailed data of CBLs can be found in Treebase (Additional file5),系统发育知识数据库(http://purl.org/phylo/treebase/phylows/study/tb2:s17414?cax-access-code=1b88565e08ce238f8fc7928d2fa11a12&format=html.)
CBL基因序列之间的显著相似性表明它们是最近通过基因复制产生的,可能具有相似或重叠的功能。副同源基因由于新功能的发展而进化,并为适应提供了最有可能的作用。基因复制和多样化被认为是进化生物学中最重要的事件。如果一个基因从它的原始基因复制而来,对额外拷贝的选择限制就会大大降低,它可以进化出稍微不同的功能,而该基因的原始功能保留在另一个拷贝中。因此,基因复制和随后的多样化是获得新功能的最简单的方法之一。因为CBL基因对钙感知非常重要,还有其他几个钙感知基因家族(CPK, CaM,等等。)存在于植物界,CBL基因的复制基因仍在被发现。这可能是由于倍性水平,以及不同基因组的一些其他方面。一些在进化过程中经历复制的植物基因组包含少量复制的CBL基因,包括甘蓝,巨桉,甘氨酸,长舌妇和Medicago truncatula。
塔吉玛的统计数据
田岛的分子测试假说解释了进化的重要性和速度[55.].随机分析CBL序列在田岛的相对率检验和P-价值和X2-检验被发现是显著的(表3.).进行了三种随机复制分析。在每种分析中,通过使它们作为组A,B和C进行研究,考虑三个序列。第一次分析含有MDCBL3(A),CSCBL3(B组)和Percbl3(C组)的序列。第二分析含有Mecbl3(A组),BRCBL2-2(B组)和PTCBL3(C组);并且第三分析含有FVCBL10-1(A组),BRCBL2-2(B)和MGCBL5(C组)。在统计分析中,P-值分别为0.00666、0.00284和0.005553.).同样,单自由度的第一次、第二次和第三次分析的卡方值分别为7.36、8.91和7.69(表1)3.).这些结果表明,本文的结果具有统计学意义。在田岛中立性试验中,发现田岛对CBLs的D值为4.413697(表2)4).在Tajima的D-Test中,当D = 0时,群体的平均杂合子变得等于隔离位点的数量。发生这种情况,因为预期的变化类似于观察到的变化[55.,56.].因此,人口的演变可能是由于突变漂移的平衡,并且没有选择的证据。当D <0时,平均杂合子低于隔离位点的数量[55.,56.].这表明,稀有等位基因的出现频率非常低,最近的选择性扫描导致了种群规模在最近瓶颈后的扩大。当D > 0时,平均杂合度大于分离位点,可以认为是高频率存在多个等位基因[55.,56.].由于人口大小突然收缩,这导致均衡的选择。Tajima的阴性D值表示相对于期望的多态性的非常低的频率,通过选择性扫描或净化选择,通过尺寸表示群体的扩张。Tajima的阳性D值表示多态性的高频,表明通过平衡选择降低人口大小。Tajima的D值大于2或小于-2被认为是显着的[55.,56.].在本研究中,田岛的D值为4.413697(表4),表示CBL基因由于均衡选择而通过降低群体大小而经历了高频态的多态性。因此,CBL的杂合性大于分离位点的数量,并作为多个等位基因存在。
oscbl基因的差异表达
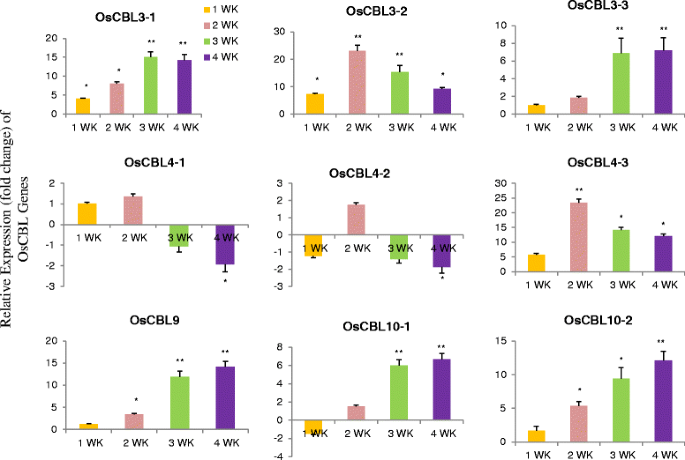
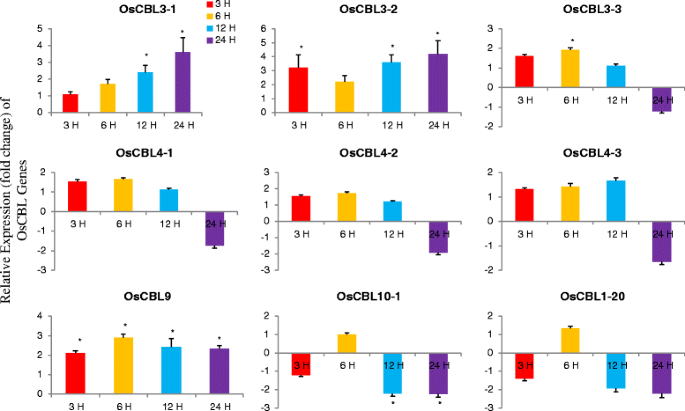
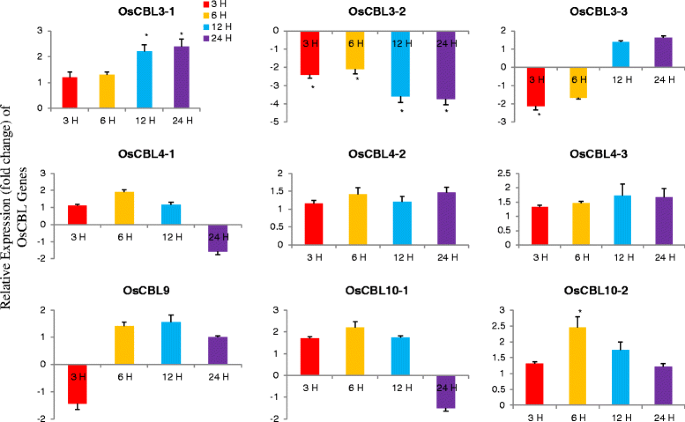
植物已成为遗传调控的重要靶点,并为研究调控发育的不同生物过程提供了良好的平台。在分子水平上分析这些发育过程需要分离和鉴定重要的调控基因,包括那些有差异表达的基因。基因表达在不同的发育阶段和特定的组织是极大的兴趣。其中一个主要的兴趣是基因在特定发育阶段的特定细胞或组织类型中的特定表达模式是否可以作为研究发育的标记。因此,我们研究了OsCBL基因在不同发育阶段的相对表达模式(图2)。7).OsCBL基因在叶片组织中的相对表达量显示,OsCBL3-1、OsCBL3-2、OsCBL3-3、OsCBL4-3、OsCBL9和OsCBL10-2在四个时间点均上调(图2)。7).OsCBL4-1在第3、4周表达下调,OsCBL4-2在第1、3、4周表达下调。在第3周和第4周观察到OsCBL基因表达的主要变化。为了更好地了解CBL基因在胁迫反应中的作用,我们对OsCBL基因进行了不同时间点的冷热胁迫差异表达分析(图)。8).OsCBL3-1、OsCBL3-2、OsCBL9在各时间点的相对表达量均增加,而OsCBL4-1、OsCBL4-2、OsCBL4-3、OsCBL10-1、OsCBL10-2在24 h表达量下降(图3)。8).在热处理的植物中,OSCBL3-1,OSCBL4-2,OSCB14-3和OSCBL10-2在所有四个时间点都经过上调(图。9).在所有四个时间点下调Oscbl3-2的表达(图。9).OsCBL3-3在第3、6 h表达下调,在第12、24 h表达逐渐上调。OsCBL9在第3 h表达下调,在第6、12、24 h表达逐渐上调。CBL基因对冷热胁迫均有响应,且在不同胁迫下表达差异较大。
结论
研究表明,CBL基因的基本结构在绿藻、苔藓植物、蕨类植物、裸子植物和被子植物等所有植物中都是保守的。绿藻和松树等低等真核生物的CBL基因似乎是独立进化的。由此可见,绿藻门(绿藻)和胚胎门(高等植物)的分裂在CBL基因的进化中发挥了重要作用。在进化过程中,陆生植物的CBL信号事件通过基因复制显著扩展。表达分析表明,OsCBL3-1、OsBL3-2、OsCBL3-3和OsCBL10-2在不同发育阶段均有显著的调控o .漂白亚麻纤维卷。OsCBL3-1的差异表达在冷和热胁迫中被显著调节,表明其在这些事件中发挥重要作用。
方法
钙调磷酸酶b样(CBL)基因家族拟南芥蒂利亚纳和奥雅萨苜蓿已从拟南芥信息资源(TAIR)数据库和TIGR RIS基因组注释项目。鉴定的蛋白质序列A. Thaliana.和o .漂白亚麻纤维卷然后用于识别植物群落和云杉基因组数据库中其他植物物种的CBL基因家族成员[57.].使用BLASTP程序(默认)识别其他植物物种的CBL基因家族成员。在BLASTP分析中使用的默认统计参数为:蛋白数据库的BLASTP-蛋白查询;预期阈值(E):−1,比较矩阵:BLOSUM62;不。排列显示:100。e值为7.0e-200的序列被纳入进一步分析,以覆盖最大数量的基因。然后将收集的蛋白序列用prosite软件扫描,分析EF-hand结构域的存在。只有三个钙结合ef手的序列被认为是钙调磷酸酶b样基因,因为所有的CBL蛋白A. Thaliana.只含有三种钙结合ef手。再次对这些CBL蛋白进行BLASTP分析拟南芥基因组数据库使用默认参数重新确认它们为CBL蛋白。植物物种的序列,使BLASTP攻击A. Thaliana.CBL蛋白被认为是CBL蛋白。CBL基因按拟南芥他们在BLAST搜索中匹配CBL基因,以确保正确的同源编号。
CBL的多序列比对和分子建模
使用多环软件进行所有物种的CBL基因的多序列对准(http://multalin.toulouse.inra.fr/multalin/).用于运行程序的统计参数如下:蛋白质重量矩阵:展开62,开幕式差距:违约,差距处罚:默认,四肢差距:无,仅限迭代:没有,高共识:90%和低共识值:50%。
系统发育树的构建
为了构建系统发生树,在CLUSTALW软件中使用所有CBL基因的蛋白序列生成簇文件[58.].运行CLUSTALW程序的参数如下:蛋白质量矩阵:blossom 62,间隙开度:10,间隙扩展:0.2,迭代:无。使用MEGA5软件将生成的簇文件下载并转换为MEGA文件格式[52.].生成的MEGA文件在MEGA5软件中运行,构建系统发生树。构建系统发生树的统计参数如下:分析:系统发生重建;统计方法:最大似然法;系统发生检验:bootstrap法;bootstrap复制:3000个,替代类型:氨基酸,模型/方法:琼斯-泰勒-桑顿(JTT)模型,站点之间的比率:统一率,差距/缺失数据处理:部分删除,站点覆盖截止:95%和分支交换过滤:非常强。
统计分析
采用田岛相对速率试验,研究植物CBL基因的显著性和进化速率。生成用于构建系统发生树的MEGA文件,采用MEGA5进行Tajima的相对率检验和Tajima的中立检验。田岛相对率检验采用的统计参数如下:田岛相对率检验范围:对选择的三个序列,替代类型:氨基酸,缺失/缺失数据处理:完全缺失。进行田岛中立检验所用的统计参数如下:分析:田岛中立检验,范围:所有选定的类群,替代类型:氨基酸,空白/缺失数据处理:完全缺失。
植物处理和定量实时PCR
野生类型奥雅萨苜蓿japonica var。Nipponbare在A16 H光下的温室中在土壤中生长:8小时黑暗循环,22-25°C为20天。冷处理由4°C组成,而干旱/热处理由40°C组成。将叶子在0,3,6,12和24小时中取样,并立即转移到液氮中以进行随后的分析。未经处理的植物用作对照样品。在本研究期间使用了三种生物学重复。使用Trizol试剂从处理和对照样品中分离出总RNA。使用Nanodrop1000量化RNA,并通过电泳在1.5%(w / v)琼脂糖凝胶中通过电泳检查其完整性。使用FERMENS REDAID第一链cDNA合成试剂盒对高质量的RNA进行CDNA的制备。通过加入1μg总RNA,2μl10×RT缓冲液,2μl10mmdntps混合物,2μl寡核苷酸(DT)来制备反应。18引物,1 μl的逆转录酶,1 μl的核糖核酸酶抑制剂和核酸酶游离无菌水高达20 μl。将反应混合物在42℃下热孵育60 min,在70℃下终止反应5 min,将生成的cDNA稀释10倍保存备用。的引物o .漂白亚麻纤维卷利用引物3软件设计CBL基因,分别针对不保守的5 '端和3 ' UTR区域,扩增子大小在120 ~ 200 bp之间(引物长度在20 ~ 24 bp之间)4).通过常规PCR扩增、琼脂糖凝胶电泳和Mx3000P实时荧光定量PCR仪检测引物的熔化曲线,检测引物的特异性。引物的熔化曲线分析在60-95°C进行,每次运行结束时升温步骤为0.06°C/s(每摄氏度5次)。采用Mx3000P实时荧光定量PCR系统,SYBR green master mix (2x) (Fermentas)和ROX作为被动参考标准,对SYBR荧光信号进行归一化。以1 μl cDNA为模板,12.5 μl SYBR绿主混合(2X),正反引物各1 μl,核酸酶游离水最高25 μl为反应条件,进行PCR扩增。实时定量PCR的热谱为:95℃初始激活10 min,然后依次为95℃30 s、60℃30 s、72℃30 s 40个循环。使用3个生物重复进行3个重复的分析。引物的效率为90 ~ 105%,为显著性引物。OsCBL基因的相对表达量用2——ΔΔCt方法 [59.].
缩写
- CBL:
-
钙调磷酸酶b
- 类ef - hand:
-
延长因子的手
- 肌酸磷酸激酶:
-
钙依赖性蛋白激酶
- 摄像头:
-
钙调蛋白
- CIPK:
-
CBL相互作用蛋白激酶
参考文献
- 1.
陈志强,陈志强,陈志强,等。绿色植物CDPKs的基因组学研究进展。植物科学进展。2014;19:79-89。
- 2.
Tuteja n,Mahajan S.植物中的钙信号网络。植物信号表现。2007; 2(2):79-85。
- 3.
Hogan PG,Lewis Rs,Rao A.淋巴细胞中钙信号传导的分子基础:味道和奥莱。Annu Rem Immunol。2010; 28:491-533。
- 4.
Mohanta TK, Sinha AK。钙依赖蛋白激酶在非生物胁迫耐受中的作用。在:Tuteja N, Gill S,编辑。非生物胁迫反应植物。2015版。魏海姆,德国:Wiley-VCH Verlag GmbH & Co;2015.p . 185 - 208。
- 5。
韦伯AR。焦点问题:钙信令。植物理性。2013; 163:457-8。
- 6。
Eprintsev AT, Fedorin DN, Igamberdiev AU。Ca2+参与拟南芥光敏色素a依赖性琥珀酸脱氢酶基因sdh1-2的调控。acta botanica yunnanica(云南植物研究),2013;
- 7.
Kanchiswamy CN,Mohanta TK,Capuzzo A,Occhipinti A,Verrillo F,Maffei Me,等。CPKS和细胞溶质CA2 +变异抗性和易感苹果品种(Malus X Dibustoma)的差异表达,响应于埃尔维亚氨基甲酰胺和机械伤口。BMC基因组学。2013; 14:760。
- 8.
一天是,reddy vs,shad ali g,reddy a。拟南芥含有EF手的蛋白质分析。基因组Biol。2002; 3:Research0056。
- 9。
钙调素和钙调神经蛋白B -样蛋白:植物钙传感器的研究。植物细胞。2002;14:389 - 400。
- 10.
Sulmann S, Dell 'orco D, Marino V, Behnen P, Koch K-W。分子拥挤条件下钙敏感蛋白的构象变化。化学(伊斯顿)。2014; 20:6756 - 62。
- 11.
拟南芥CDPK-SnRK蛋白激酶超家族。植物杂志。2003;132:666 - 80。
- 12.
lewitt - bentley A, Réty S. EF-hand钙结合蛋白。《结构生物学》2000;10:637-43。
- 13.
Popescu Sc,Popescu GV,Bachan S,Zhang Z,Seay M,Gerstein M等。钙调蛋白相关蛋白与高密度拟南芥蛋白微阵列揭示的靶向差异结合。Proc Natl Acad Sci U S A. 2007; 104:4730-5。
- 14.
Nozawa A,Koizumi N,Sano H.拟南芥SNF1相关的蛋白激酶ATSR1与钙结合蛋白,ATCBL2相互作用,其中转录物反应光。植物细胞生理。2001; 42:976-81。
- 15.
在拟南芥中调控盐、干旱和寒冷反应的钙离子传感器。植物细胞。2003;15(8):1833 - 45。
- 16。
Schlicker A,Domingue FS,RahnenführerJ,Lengauer T.基于基因本体学基因产品功能相似性的新措施。BMC生物信息学。2006; 7:302。
- 17.
Aravind L, Mazumder R, Vasudevan S, Koonin EV。从序列和结构分析推断的蛋白质进化趋势。《当代结构生物学》2002;12:392-9。
- 18.
Illergård K, Ardell DH, Elofsson A.结构比序列保守3到10倍-蛋白质核心结构反应的研究。蛋白质。2009;77:499 - 508。
- 19.
Choi I-G,Kim S-H。蛋白质结构类和蛋白质序列系列的演变。Proc Natl Acad Sci U S A. 2006; 103:14056-61。
- 20。
等。古老的信号:植物MAPK和MAPKK基因家族的比较基因组学。植物科学进展。2006;11:192-8。
- 21。
Mohanta TK,Arora PK,Mohanta N,Parida P,Bae H.植物中MAPK基因家族的新成员的识别显示不同的保守结构域和新型激活回路变体。BMC基因组学。2015; 16:58。
- 22.
Tapan KM, Mickael M, Nibedita M, Chidananda NK。非洲狗尾草生长素外排载体基因家族的in - silicon鉴定及系统发育分析[J]。
- 23.
Mohanta TK,Mohanta N. Genome在Physcomitrella含有植物素流出载体基因家族的范围广泛鉴定。j biotechnol sci。2013; 1:54-64。
- 24.
黄志强,陈志强。基因组进化的测量方法。中国科学(d辑:地球科学)1998;95:5849-56。
- 25.
关键词:小立碗藓,拟南芥,CBL-CIPK,钙解码网络,系统基因组学植物科学学报。2014;5(5):187。
- 26.
植物基因组在染色体中的组织。植物j . 2011; 66:18-33。
- 27.
Nystedt B, Street NR, Wetterbom A, Zuccolo A, Lin Y-C, Scofield DG,等。挪威云杉基因组序列和针叶树基因组进化。大自然。2013;497:579 - 84。
- 28。
核苷酸偏差导致蛋白质氨基酸组成的全基因组偏差。Mol Biol Evol. 2000; 17:1581-8。
- 29.
Foster PG, Jermiin LS, Hickey D a.核苷酸组成偏差影响动物线粒体编码蛋白质的氨基酸含量。中华民族大学学报(自然科学版)1997;44:282-8。
- 30.
关键词:枯草芽孢杆菌,蛋白质组,代谢效率,氨基酸组成美国国家科学研究院2002;99:3695-700。
- 31。
Akashi H.翻译选择和酵母蛋白质组进化。遗传学。2003; 164:1291-303。
- 32。
rett SP, King G, Shabanowitz J, Hunt DF, Hartman KL, Laue TM,等。银hake parvalbumin isoform b的螺旋-环-螺旋(EF hand)基序的特征。
- 33。
徐涛,戴宁,陈建军,曹敏,李华,等。细胞表面ABP1-TMK生长素感应复合物激活ROP GTPase信号。科学(80 -)。2014; 343:1025-8。
- 34.
Poovaiah BW,Du L,Wang H,杨T.钙/钙调蛋白介导的信号传导的最新进展,重点对植物微生物相互作用。植物理性。2013; 163:531-42。
- 35.
拟南芥钙调磷酸酶b样蛋白基因的表达受胁迫信号的调控。美国国立科学学院学报1999;96:4718-23。
- 36.
钙调素和钙调神经蛋白b -样蛋白:植物钙传感器的研究。植物细胞。2002;S389-S400。
- 37.
顾志强,马斌,姜勇,陈志强,苏旭,张华。钙调磷酸酶B样基因家族在水稻逆境胁迫下的表达分析。基因。2008;415:1-12。
- 38.
De Castro E, Sigrist CJA, Gattiker A, Bulliard V, langendick - genevaux PS, Gasteiger E, et al.;ScanProsite:检测蛋白质中PROSITE特征匹配和prorule相关的功能和结构残基。核酸Res. 2006;34(Web服务器版):W362-5。
- 39.
植物钙调磷酸酶b样蛋白及其相互作用蛋白激酶。Acta botanica sinica; 2009; 33(4): 593 - 598。
- 40。
CBL - CIPK Ca 2+−解码信令网络:功能与展望。新植醇。2009;184:517-28。
- 41。
Linder ME, Deschenes RJ。棕榈酰化:控制蛋白质的稳定性和流量。《细胞生物学》2007;8:74-84。
- 42.
我,我。细胞内信号蛋白棕榈酰化:调节和功能。生物化学学报2004;73:559-87。
- 43。
Blaskovic S, Blanc M, van der Goot FG。s -棕榈酰化对膜蛋白有什么作用?2月j . 2013; 280:2766 - 74。
- 44。
Singaraja RR, Hadano S, Metzler M, Givan S, Wellington CL, Warby S,等。HIP14是一种新型的含有锚蛋白结构域的蛋白,将杭丁顿蛋白连接到细胞内运输和内吞作用。Hum Mol Genet, 2002; 11:2815-28。
- 45。
Michaelson D,Silletti J,Murphy G,D'Eustachio P,Rush M,Philips Mr。活细胞中Rho GTP酶的差分定位:通过高变区和rhogdi结合调节。J细胞BIOL。2001; 152:111-26。
- 46.
robert PJ, Mitin N, Keller PJ, Chenette EJ, Madigan JP, Currin RO, et al.;Rho家族GTPase修饰和对CAAX基序信号翻译后修饰的依赖。中国生物化学杂志。2008;283:25150-63。
- 47.
Cheng S,Willmann M,Chen H,Sheen J.通过蛋白激酶的钙信号传导。拟南芥钙依赖性蛋白激酶基因家族。植物理性。2002; 129:469-85。
- 48.
Martín ML, Busconi L.水稻膜结合钙依赖蛋白激酶在响应低温时被激活。植物杂志。2001;125:1442-9。
- 49.
拟南芥钙依赖蛋白激酶与内质网相关1。植物杂志。2002;128:1008-21。
- 50.
黄益平,裴建平,黄益平等。蛋白质超家族中结构保守区域的定义和预测。生物信息学。2013;29:175 - 81。
- 51.
Peñami,班奈特先生van Itallie E. Shamoo Y.一个基因的演变强调了适应性的分子描述的复杂性。混乱。2010; 20:026107。
- 52。
Tamura K,Peterson D,Peterson N,SteCher G,Nei M,Kumar S. Mega5:使用最大可能性,进化距离和最大判定方法的分子进化遗传学分析。mol Biol Evol。2011; 28:2731-9。
- 53。
等。多细胞绿藻团藻生物复杂性的基因组分析。科学(80 -)。2010; 329:223-6。
- 54.
Galperin MY, Koonin EV。酶进化中的分化与趋同。J Biol Chem. 2012; 287:21-8。
- 55.
分子进化时钟假说的简单检验方法。遗传学。1993;135:599 - 607。
- 56。
利用DNA多态性检验中性突变假说的统计方法。遗传学。1989;595:585 - 95。
- 57。
Goodstein DM,Shu S,Howson R,Neupane R,Hayes Rd,Fazo J等人。植物血统:绿色植物基因组学的比较平台。核酸RES。2012; 40:D1178-86。
- 58.
陈志强,陈志强,陈志强,等。clusteral W和clusteral X 2.0版本。生物信息学。2007;23:2947-8。
- 59.
Schmittgen TD, Livak KJ。采用对比CT方法分析实时荧光定量PCR数据。Nat Protoc。2008; 3:1101-8。
确认
这项研究得到了埃文国大学研究补助金的支持214A367010。
作者信息
隶属关系
相应的作者
附加信息
竞争利益
作者声明本文的发表不存在利益冲突。
作者的贡献
TKM:实验的构想和设计进行了实验,分析和解释了数据,起草了稿件,NM:分析了数据,起草稿件,YM:进行了实验,PP:分析了数据,HB:给予批准出版物。所有作者阅读并认可的终稿。
附加文件
附加文件1:
本研究中鉴定的38种不同植物中不同CBL基因的详细基因组信息。
额外的文件2:
本研究中鉴定的38种植物不同CBL基因的分子量(kDa)和等电点表。
额外的文件3:
本研究分析了所有CBL基因的多序列比对。序列比对使用在线可用的Multalin软件(http://multalin.toulouse.inra.fr/multalin/multalin.html)使用默认程序。多个对准显示CBL基因中不同保守的结构域和基序。
附加文件4:
表显示用于qRT-PCR分析的OsCBL引物列表。引物采用primer3软件设计(http://primer3.ut.ee/).
附加文件5:
系统发育数据的细节被提交到TreeBase数据库,可以在以下链接中使用http://purl.org/phylo/treebase/phylows/study/tb2:s17414?cax-access-code=1b88565e08ce238f8fc7928d2fa11a12&format=html.。
权利和权限
开放获取本文是在知识共享署名许可协议(http://creativecommons.org/licenses/by/4.0.),允许在任何媒介上不受限制地使用、分发和复制,但须注明原作的出处。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
Mohanta, t.k., Mohanta, n.k., Mohanta, Y.K.et al。植物钙调磷酸酶b样(CBL)基因家族的全基因组鉴定揭示了钙信号事件中新的保守基序和进化方面。BMC植物BIOL.15日,189(2015)。https://doi.org/10.1186/s12870-015-0543-0
已收到:
接受:
发表:
关键字
- CBL.
- 肌酸磷酸激酶
- 棕榈酰化
- Myristoylation
- 进化