抽象的
背景
Lamiaceae家族成员Krishna Tulsi是一种以其在印度的精神,宗教和药用意义的草本植物。这种植物的共同名称是'tulsi'(或'tulasi'或'thulasi'),被印度教徒被认为是神圣的。我们介绍了基因组草案OCimum Tenuiflurum.L (Krishna Tulsi亚型)。利用Illumina Hiseq 1000进行全基因组测序,生成了配对端和配对序列库,组装的基因组为374 Mb,基因组覆盖率为61%(估计基因组大小为612 Mb)。我们还研究了两个亚型的转录组(RNA-Seq)o . tenuiflorumKrishna和Rama Tulsi,并报道了这两个品种基因的相对表达。
结果
已经详细研究了导致医学上重要的专门化代谢物产生的途径,并与相关的途径拟南芥蒂利亚纳和其他植物。观察到Krishna Tulsi叶样品中花青素生物合成相关基因的表达水平相对较高,解释克里希纳·塔尔西叶的紫色着色。通过在五种不同种类的不同组织中进行Q-RT-PCR来验证从基因组数据中鉴定的六种重要基因的表达,其显示了RAMA亚型的幼叶中的尿溶胶产生基因的高度.此外,质谱证实了丁香酚和熊果酸的存在,暗示它们是治疗包括癌症在内的许多疾病的潜在药物。
结论
整个基因组的可用性O.tenuiflorum我们的序列分析表明,参与代谢产物合成途径的基因功能位点的微小氨基酸变化赋予了这种草药特殊的药用特性。
背景
植物的属罗勒属属于家庭Lamiaceae(命令Lamiales),广泛分布在世界的热带,亚热带和温暖的温带地区[1].众所周知,这些植物可以产生由许多芳香族化合物组成的精油,图丝正是因为这个原因而被称为“草药女王”。在印度,这些植物大多是在家里种植的,用于祭祀和寺庙里的供品。在具有药用价值的植物中,属植物罗勒属是非常重要的芳香草药或灌木。
属罗勒属在种内和种间水平上具有高度的变异性和广泛的遗传多样性。九种罗勒属viz。,o . teniuflorumlo. basilicum.lO。格雷西姆lO. kilimandscharcum,o. micranthuml,O. Campechianum.lO. Americanum.lO。最低限度土地o. citriodorum.L.在印度发现,其中三种(O. Americanum.lO。最低限度土地o. citriodorum.L.)是异国情调的[2]. 仅根据叶片形态很难区分所有这些物种(图。1).属的代谢产物(精油)罗勒属据报道,据报道具有抗氧化剂和抗真菌性能,并治愈许多疾病,包括Ayurveda的支气管炎,印度医学系统[3.].植物生产专门的代谢物,作为其防御机制的一部分,这些代谢物具有显着的药物性能,治愈了几种人类疾病。它们可以与植物的各个部分隔离,包括叶子,鲜花,根,树皮,种子和茎[4].植物代谢物的药理筛选和化学成分的系统研究为开发新药提供了基础。一些重要的代谢物报道罗勒属品种包括芳樟醇、芳樟醇、香叶醇、柠檬醛、樟脑、丁香酚、甲基丁香酚、甲基茶酚、肉桂酸甲酯、百里酚、番红花、紫杉醇、尿溶胶酸等[4].这些代谢物在制药,香水和化妆品产业中具有巨大的价值。代谢物衍生自罗勒属植物被发现含有许多药物相关的特性,包括抗癌,抗氧化,抗真菌和抗炎的优点,也被推荐用于治疗疟疾,支气管炎,腹泻,痢疾等[5].在植物的叶子、种子、花和根中发现的特殊代谢物产生的精油罗勒属物种用于药物和许多传统印度医学系统[3.,4].药用植物基因组和转录组测序是基因发现和重要药用代谢物下游生化途径发现的有力工具[6].最近,萜类化合物生物合成的丰富转录物O。圣殿和苯丙素o. basilicum.[7是在试图比较两个物种的转录组时报道的罗勒属.尽管克里希纳图尔西在印度传统医学中发挥着重要作用,其生物活性化合物令人印象深刻,但我们对克里希纳图尔西的生物学了解有限。本文给出了该非模式植物的基因组草图O. Tenuiflorum.(亚型Krishna),以及来自叶样品的两种亚型,克里希纳和拉玛Tulsi的转录组。我们已经鉴定了一大组基因,参与了药用兴趣的专用代谢物,如Apigenin,葡萄呤,罗马辛酸途径,丁醇和熊果酸。
结果
非模型植物的基因组测序和组装O. Tenuiflorum.克里希纳亚型
paired-end (PE;2 × 100-bp)和配对(MP;2 × 50 bp),利用Illumina协议生成Krishna Tulsi亚型的DNA库。我们总共获得了Krishna Tulsi的3.73亿读PE和1.66亿读MP数据。低质量(LQ)序列读取被修剪(附加文件1:图S1和附加文件2:图S2)和质量分数小于Q30的reads被删除。高质量的测序结果用于基因组的重组。PE数据中位数插入尺寸为335(中位数绝对偏差为21),MP数据中位数插入尺寸为2473(中位数绝对偏差为704)。通过对不同K-mer的统计分析,选择K-mer 43作为最佳组合。我们得到支架的最大长度为184.7 Kb(见表)1)具有27.1 Kb的N50长度。该组件共产生78224个脚手架,包括等于或大于100 bp的脚手架。克里希纳·图尔西(Krishna Tulsi)目前的基因组草图长度为374.8 Mb。克里希纳图尔西的基因组含量为0.72 pg/2C,相当于704.6 Mb[8],但k-mer法估算的基因组大小为612 Mb,其中61%的基因组已组装完成。文献中报道的基因组大小[8,可能属于不同的品种。这种较低的基因组覆盖率可能是由于测序数据有限(测序时只使用了两个文库)或重复率高(42.9%)。在测序深度方面,我们用配对端(100 bp)和配对端(50 bp)文库对59×基因组进行了测序(因为即使读取覆盖了估计的612 Mb基因组大小,一条lane也可以产生大约30Gb的数据)。罗勒属物种的特征是不同的基本染色体数x = 8、10、12或16 [9,10].的情况下O. Tenuiflorum.已经记录了2N = 32,2n = 36和2n = 76的个体,并且染色体数量O. Tenuiflorum.为2n = 36 [8].
对单独使用PE数据和PE和MP数据生成的组装体进行比较分析表明,包含MP数据后,单独使用PE数据组装的基因组的大小和质量显著提高(附加文件)3.:数字S3和附加文件4:图S4,附加文件5:表S1和附加文件6:表S2)。
的验证新创基因组组装、注释和重复内容圣罗勒克里希纳亚型基因
的新创基因组装配通过映射原始读取到装配的基因组来验证。平均来说,74%的reads被映射回组装后的基因组。几乎83.3%的RNA-seq序列被映射到组装的基因组上。的完整性新创基因组组装和注释也通过其他两种方法进行检查,即使用CEGMA (Core Eukaryotic Genes Mapping approach) [11]和DEG(基本基因数据库)[12](详情请参阅方法)。首先,我们寻找真核生物的基本基因O. Tenuiflorum.集会。这导致了85.1%的完全核心蛋白(CEGMA)和95%以上,包括针对我们基因组大会的部分基因(附加档案7:表S3)。其次,我们从Deg数据库中记录的必要基因的最终组合搜索预测基因。我们观察到,大会内包括约89%的基因。还使用PFAM域注释验证这些基因,并且与该家庭的经典成员相当的域长度(附加文件)8:表S4)。高度保守的基本基因如甘油醛3-磷酸脱氢酶的系统发育树(附加文件9:图S5),细胞色素P450(附加文件10:图S6)和actin(附加文件11:图S7)对Krishna Tulsi及其各自的同源物进行了分析,并与其他植物物种进行了比较。Krishna Tulsi基因被发现与属于相关物种的基因聚集在一起,即,番茄茄甚至是与远亲拟南芥,这表明植物生长和功能所必需的高度保守基因已经被检测到O. Tenuiflorum.程序集。这些趋势进一步支持了基因组组装的质量。
关于基因组的重复内容,我们确定了78224个重复区域,GC含量为36.1%,再加上160889218 bp(160 Mb),占组装基因组的42.9%,组装基因组长度为374806882 bp(374 Mb)(附加文件12:表S5)。在植物基因组中大量存在长终端重复(LTR)(Schmidt T.,1999),在图尔西基因组的重复序列中也发现了类似的趋势。
基因组注释
我们在最初的基因组草案(1.2版)中鉴定了36768个推测的基因模型O. Tenuiflorum.基因组。在10012个支架中,每个支架至少观察到一个基因,平均每个支架有3到4个基因。在基因精细预测的过程中,观察到16384个基因模型有表达证据(Tulsi (Krishna and Rama)叶片的RNA-Seq数据)。共鉴定了19384个基因模型从头开始手段(没有任何RNA或蛋白质证据)(表2).
所有的基因预测,无论是否有RNA/蛋白证据,都是基于长度(>100 bp)进行筛选。在不同基因模型之间序列重叠的情况下,具有RNA或蛋白质证据的长度较长的基因模型优于无证据的基因模型。
NRDB中含有至少一个同源基因的基因有31,020个,含有至少一个Pfam结构域的基因有24,607个。在图西的所有预测基因中,共鉴定出3929个独特的Pfam域(请参阅URL:http://caps.ncbs.res.in / sote.完整的预测基因列表)。大多数识别的结构域是蛋白激酶或含有lrr的结构域(附加文件13:图S8)。进一步将Pfam结果与大小相近的组装植物基因组进行比较,发现预测的基因模型数量在数量和基因边界上总体一致。
Tulsi基因的矫形局
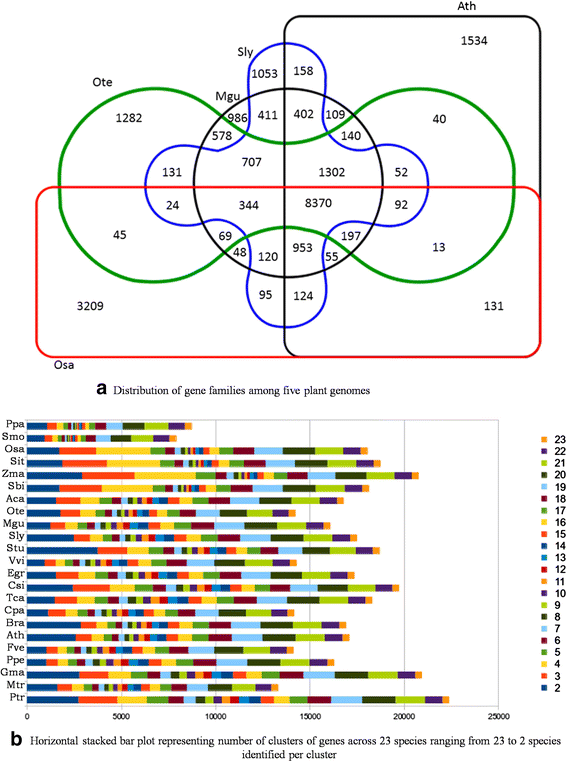
Krishna Tulsi (O. Tenuiflorum.;(Ote)和其他四种物种。拟南芥蒂利亚纳(ATH),Mimulus guttatus(开战),茄属植物lycopersicum(狡猾)和水稻(OSA)(请参阅详细信息)。我们观察到8370个群集,其中包含来自五种植物物种的8922种基因产物(图。2A).m . guttatus和O. Tenuiflorum.有相同的目(唇形科),但属于不同的科(分别为水芹科和唇形科),这从它们之间存在最多的共同基因家族(11707)就可以看出。随后是茄属植物lycopersicum(11022),拟南芥蒂利亚纳(10206)和水稻(9154)从分类层次中预期(图9154)。2A).我们发现了17584个基因与上述四个物种中的任何一个同源。在36768个Ote基因中,有1282个组仅包含Ote Krishna Tulsi基因(3302)。我们获得了16个Ote基因,这些基因与其他22种植物缺乏同源性和同源关系(这些基因的列表可在数据库中找到)。这些独特的Ote基因很少是转座子。
图尔西基因组同源基因在其他相关植物基因组中的分布与聚类分析。一个. 基因家族在五种植物基因组中的分布。圣罗勒(Ote -绿色),拟南芥蒂利亚纳(Ath-Black Rectangle),水稻(Osa -红色),茄属植物lycopersicum(狡猾 - 蓝色)和Mimulus guttatus(Mgu -黑色圆圈)。维恩图中的数字代表了由OrthoMCL获得的这5个物种共有的和独特的基因家族。b.所有基因的水平堆积条形图23种不同的基因组。该图显示了所有23种植物种类的正交组分布,包括Tulsi。每一行代表一种植物物种 -Physcomitrella patens.(Ppa),卷柏moellendorffii(Smo),栽培稻(阻塞性睡眠呼吸暂停综合症), Setaria斜体(坐),Zea mays(ZMA),高粱双色(印度国家银行),耧斗菜caerulea(aca),Ocimum Tenuiflorum.(注), Mimulus guttatus(开战),番茄茄(狡猾)茄属植物,tuberosum(斯图),葡萄(Vvi),桉树祖母(Egr),柑橘类sinensis.(Csi),可可树(TCA),番木瓜(Cpa),芸苔属(胸罩),rapidopsis thaliana(Ath),维斯卡草莓(FVE),碧桃(PPE),大豆(Gma),Medicago Truncatula.(地铁),杨树trichocarpa(Ptr)。条形图表示该物种的直系同源蛋白质组,根据与其他22种植物物种的共享程度,将其细分为22个类别。例如,类别2表示代表感兴趣物种的直系同源蛋白质组的数量,以及23种选择用于研究的物种中的一个或多个物种的直系同源蛋白质组的数量
为了详细检查局部关系的分布罗勒属22个全序列植物基因组(附加文件14:表S6)。根据聚类结果,对23种植物的同源类群进行了分类。研究中选择的23个物种共有334个基因簇。所有物种的共同基因,包括它们各自的同源组,被绘制成一个水平堆叠条形图(图。2B.).共享同源群的模式是原始植物基因组(如石松和苔藓植物)和单子叶植物所特有的。然而,在图尔西基因组中观察到的模式与m . guttatus(MGU)。有趣的是,这种图案对于两个菌根的成员有些不同,其具有更多的基因仅在23个基因组中仅共享,也许是由于多倍体等其他特征。
参与合成药物专用代谢物的基因:比较分析O. Tenuiflorum.(otte,krishna tulsi)和其他植物基因组
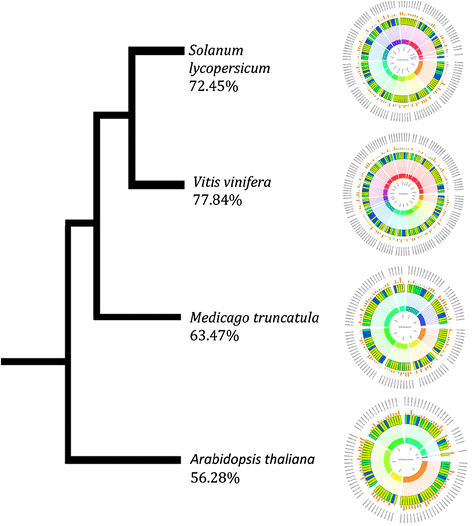
接下来,我们对Ote中涉及代谢物产生的基因进行了限制性分析,并对少数亲缘关系密切的植物(S. lycopersicum.,V.Vinifera)或特征明显(M. Truncatula.,A. Thaliana.).我们观察了121(72.45%)、130(77.84%)、106(63.47%)和94(56.28%)四个具有代表性的基因组与167个代谢相关的支架和contigs在Ote Krishna Tulsi(图。3.)分别.就与蛋黄代谢物基因相关的该选定的植物基因组的正交基因数量,我们观察到与601,620,570和556基因相似的关联趋势S. lycopersicum.,葡萄属,截齿属,A. Thaliana.分别地这些数字与分类学的系统发育和层次结构相一致,表明参与代谢途径的基因进化不是最近扩张或突然漂移的原因。
五种选定植物基因组的系统发育表示。,茄属植物lycopercicum(72.45 %),vitis Vinifera(77.84%),Medicago trucatula(63.47%),和拟南芥蒂利亚纳(56.28%)。这些数字表示这些基因组与代谢物基因关联的百分比罗勒属基因组。这些百分比与分类学系统发育和层次结构一致,表明参与代谢途径的基因的进化不是最近基因组扩张或突然基因组漂移的原因。内圈代表来自各自同源基因组的染色体。每个支架组织在中间的圆圈中,并按照染色体位置的时间顺序表示。这条线表示每个支架在各自染色体上的位置。颜色显示 = < 2个基因,
= < 2个基因, = 2基因,
= 2基因, = > 2基因,
= > 2基因, =代谢相关基因。最外圆橙色列的高度表示对应支架的重复次数
=代谢相关基因。最外圆橙色列的高度表示对应支架的重复次数
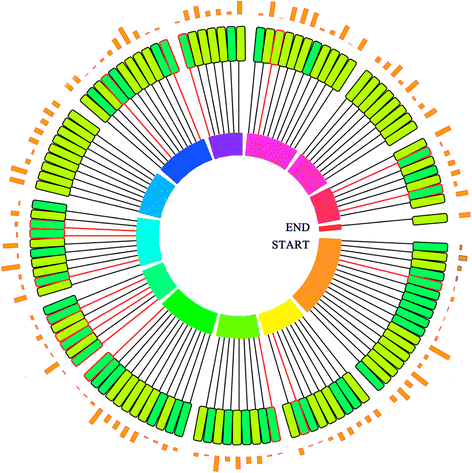
与OCTE的11,389个支架(大小大于10kb)进行比较,发现10032,9997,8648和8277支架与四个参考植物基因组相关(附加文件15:图S9,附加文件16:图S10和“附加”文件17:图S11为三个基因组和附加文件18:四个基因组的表S7)。此外,OTTe krishna Tulsi中的大多数与磷酸盐相关的支架与番茄中的染色体1,6,8和10有关(图。4).特别是,木犀草素合成途径相关的基因产物被观察到聚集在支架中,类似于番茄基因组第3、5、6、8和10染色体上的核苷酸延伸(图)。4).
圆形的代表O.tenuiflorum代谢物相关基因映射到染色体上茄属植物lycopersicum基因组。橙色柱高 外圆表示各支架中出现的重复次数。内圈代表来自番茄基因组的染色体。内圆的矩形表示支架,每个支架组织在中间圆,并按照染色体位置的时间顺序表示。每个支架的颜色表示以下信息:
外圆表示各支架中出现的重复次数。内圈代表来自番茄基因组的染色体。内圆的矩形表示支架,每个支架组织在中间圆,并按照染色体位置的时间顺序表示。每个支架的颜色表示以下信息: = 2基因,
= 2基因, = > 2基因,
= > 2基因, =代谢相关基因。支架与染色体之间的连接线表示支架在基因组中的位置。连接线的红色表示代谢物相关基因的存在。脚手架数字在附加文件中提到24.:文本A.
=代谢相关基因。支架与染色体之间的连接线表示支架在基因组中的位置。连接线的红色表示代谢物相关基因的存在。脚手架数字在附加文件中提到24.:文本A.
转录组新创克里希纳和拉玛特丽成熟叶样品的组装
德诺维对Krishna Tulsi亚型成熟叶片样品进行转录组组装。最佳组合为109291个contigs, N50为893 bp,最长序列为12.1 Kb。所有这些contigs加起来为49.5 Mb, GC含量为42.9%。结果共得到89878个支架,N50为1597 bp,最长序列为12.7 Kb。所有支架加起来为56.3 Mb, GC含量为42.9%(见表)3.).同样,对亚型Rama Tulsi和组合读数(Krishna和Rama Tulsi)进行组装(表3.).
转录本的差异表达
对两种Tulsi亚型转录组中发现的差异表达基因进行了分析。与罗摩图尔西相比,我们观察到克里希纳图尔西中有大量基因上调和下调。一些高表达基因也通过q-RT-PCR技术在茎、叶和花等不同组织样本中以及在五个物种(即。O. Tenuiflorum.奎师那和罗摩的亚型,O。格雷西姆,o. basilicum.,o . kilmand。
为了比较,我们生成了Krishna Tulsi样品中差异更丰富的前50个基因的热图(图2)。5a).类似地,还绘制了克里希纳样品的RAMA中的前50个差异更丰富的基因(图。5b). γ-卡丁烯合酶是克里希纳和拉玛图尔西样本中RPKM值分别为577.0和31.7的前50个差异表达转录物之一(详情请参见下文)。Krishna Tulsi样本中的其他高表达转录物是热休克同源蛋白80、纤维素合成酶A催化亚基6(UDP形成)、果糖二磷酸醛缩酶(氯偏酸)、光致变色素-2和Rubisco活化酶1(氯偏酸)。查耳酮合酶或柚皮素查耳酮合酶(CHS)是植物部分着色的重要酶之一,被观察到高度表达。所有转录本的丰度值,以及NCBI BLAST结果的功能注释和相应的Krishna Tulsi基因组支架,表明参与合成具有药用价值的特殊代谢物的几个基因(附加文件19:表S8)。
Tulsi Krishna和Rama亚型的转录本表达为RPKM值。高度显著差异丰富的RNA支架/转录本被定义为两种亚型的RPKM至少为5,两种亚型之间的fold change差异至少为8倍。只有那些95%的较丰富亚型的低置信限和95%的较不丰富亚型的高置信限的转录本保留了至少8倍的差异。在这些差异丰富的转录本中,Krishna和Rama亚型的前50名以热图的形式绘制出来。一个.克里希纳的差异更丰富的成绩单。b.在Rama中差异更丰富的转录本。(请参阅附加档案24.文本B和C是a和B的成绩单id
Krishna Tulsi亚型的叶和茎呈深紫色是其特征表型之一,是其区别于其他亚型和属种的重要标志罗勒属。查尔酮合成酶(CHS)属于聚酮合成酶家族,对类黄酮生物合成的起始步骤进行催化。黄酮类化合物是重要的植物特异性代谢产物,具有色素沉着、抗真菌防御等多种功能等. 从UniProt(通用蛋白质资源)数据库中审查CHS的蛋白质序列[13]被用来搜索Krishna Tulsi基因组的注释蛋白质序列,并获得六个可能的转录本。最好的命中率可以通过95%的查询覆盖率和99%的序列标识来识别。该hit(蛋白质序列)的丰度在Tulsi亚型即。奎师那与罗摩。六个转录本的丰度(以RPKM计算)平均是奎师那的两倍于罗摩(见图)。5),可能与Krishna亚型植物的着色表型有关[14].为了进一步确认这些转录物的表达,进行Q-RT-PCR。如预期的那样,观察到克里希纳幼叶样品和成熟叶样品(用作对照)的克里希诺素产生基因更丰富(图。6a和b). 相比之下,克里希纳成熟叶片样本中的叶绿素结合蛋白更为丰富。此外,我们还检测了负责香气的γ-卡丁烯合酶基因的存在[15].发现该基因在Rama根样品和幼叶样品中更丰富o .蔗糖,但在O. Kilmund..
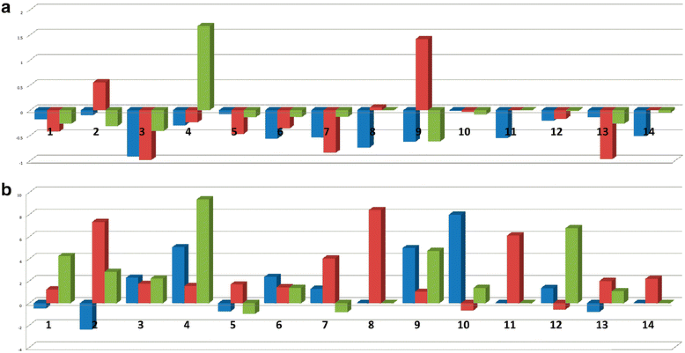
用q-RT-PCR方法对所选基因进行表达定量。一个.通过Q-RT PCR获得颜色生产中涉及的基因的变化。蓝色水平杆适用于叶绿素A-B结合蛋白,RED表示γ-钙氨酸合成酶和绿色以表示花青素。以克利须那亚型成熟叶片为对照。可以看出,与成熟的Krishna叶相比,下调负责诸如叶绿素A-B结合蛋白和基因的叶绿素A-B结合蛋白和基因负责的基因,这与表型特征相当。b.通过qRT-PCR获得5个不同图尔西亚型熊果酸生物合成途径相关基因的Fold changes。蓝色横杠为角鲨烯环氧化酶,红色为α -淀粉酶,绿色为细胞色素P450单加氧酶。以克利须那亚型成熟叶片为对照。Rama亚型成熟叶片基因表达量高,表达量大最大的Kilmund.很低。这些基因在小的、发育中的植物中表达量一致很高。样品如下:1)O. Tenuiflorum.(Rama) -采样叶子2)O. Tenuiflorum.(Rama) -抽样根。O. Tenuiflorum.(Rama) -成熟的叶子O. Tenuiflorum.(克里希纳)-采样树叶O. Tenuiflorum.(奎师那)-取样根O。格雷西姆-采样树叶O。格雷西姆-采样根。O。格雷西姆——成熟的叶子o . sacharicum-采样树叶o . sacharicum-取样根o . sacharicum——成熟的叶子O. Kilmund.-采样树叶O. Kilmund.- 抽样root。14)O. Kilmund.——成熟叶
专门的代谢物检测和验证
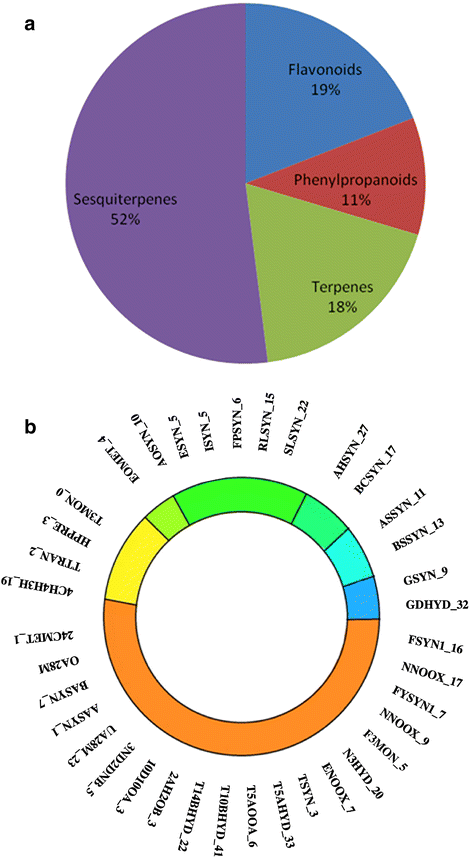
近30个专门的代谢物(图。7a)罗勒属发现有药物价值或性质[4].其中,在PlantCyc数据库(http://www.plantcyc.org/) [16)(附加文件20.:图S12)。因此,选择参与这些途径的基因用于进一步分析并搜索组装的基因组O. Tenuiflorum..数字7b强调了在与疾病相关的各种代谢产物中鉴定的基因的分布(即,这些代谢产物被认为是治疗人类疾病的药物)。
塔尔西基因组中涉及专门代谢物合成的基因数。一个.有四类代谢物存在于罗勒属基因组Qiz。,辛伯萜(52%),黄酮类化合物(19%),Terpenes(18%)和苯丙醇(11%)。支架中的数量是基因组中存在的次微征化代谢物的百分比。将458个基因鉴定为参与专用代谢物的合成中酶的酶的编码。b.提出的疾病相关性专门的代谢途径OCimum Tenuiflorum。研究的主要疾病类别用不同的颜色表示:抗癌 , anticancer-antioxidant
, anticancer-antioxidant ,抗真菌
,抗真菌 ,防腐剂
,防腐剂 ,抗感染
,抗感染 ,抗氧化剂
,抗氧化剂 和抗炎剂
和抗炎剂 .为方便起见,酶已用5-7个字母标记。酶标签中'_'之后的数字代表了在基因组组件中为给定酶的推定命令的数量O. Tenuiflorum..参与疾病相关性和参与这些代谢物合成的酶的代谢产物如下:Apigenin(Flavone-Inflicalsi-Fsyn1,Naringenin-Nadph-氧 - 氧化还原酶-NNOOX),叶氏菌素(黄酮 - 合成酶-FSYN1,鼻疽蛋白 -NADPH-氧 - 氧化还原酶-NNOOX,黄酮-3-单氧基酶-F3),紫杉醇(税偶氮合酶-TSYN,税前-5-alpha-羟基酶-T5AHYD,税前-5-alpha-OL-乙酰转移酶-T5AOOA,紫杉烷-10-β-羟基化酶-T10BHYD,番茄 - 14-β-羟基化酶-T14BHYD,2-α-羟基氮烷-2-o-苯甲酰基转移酶-2ah20Ob,10-deacetylbaccatin-III-10-O-乙酰转移酶-10d10a,3-n-debenzoyl-2-脱氧氧基醇-n-苯甲酰基转移酶-3ND2DNB,熊磷酸(Ursolic-醛-28-单氧基酶-UA28M,α-氨林 - 合成酶-Aasyn),OleAlic acid(β-氨基 - 合成酶-Aasyn,OleAnolic-醛 -28-单氧化酶-OA28M),谷甾醇(24℃-甲基转移酶-24Cmet),罗马啉酸I(4-香豆酰-4-羟基苯甲酸酯-3-羟基酶-4C4H3H,酪氨酸 - 转氨酶 - TTRAN),ROMARINIC ACID II (Hydroxyphenylpyruvate-reductase-HPPRE, Tyrosine-3-monooxygenase-TTRAN), METHYL CAHVICOL (Eugenol-o-methyltransferase-EOMET), EUGENOL (Alcohol-o-acetyltransferase-AOACE, Eugenol-synthase-ESYN, Isoeugenol-synthase-ISYN), LINALOOL (Farnesyl-pyrophosphate-synthase-FPSYN, R-linool-synthase-RLSYN, S-linool-synthase-SLSYN), CARYOPHYLENE (Alpha-humulene-synthase-AHSYN, Beta-caryophyllene-synthase-BCSYN), SELINENE (Alpha-selinene-synthase-ASSYN, Beta-selinene-synthase-BSSYN), CITRAL (Geraniol-synthase-GSYN, Geraniol-dehdrogenase-GDHYD)
.为方便起见,酶已用5-7个字母标记。酶标签中'_'之后的数字代表了在基因组组件中为给定酶的推定命令的数量O. Tenuiflorum..参与疾病相关性和参与这些代谢物合成的酶的代谢产物如下:Apigenin(Flavone-Inflicalsi-Fsyn1,Naringenin-Nadph-氧 - 氧化还原酶-NNOOX),叶氏菌素(黄酮 - 合成酶-FSYN1,鼻疽蛋白 -NADPH-氧 - 氧化还原酶-NNOOX,黄酮-3-单氧基酶-F3),紫杉醇(税偶氮合酶-TSYN,税前-5-alpha-羟基酶-T5AHYD,税前-5-alpha-OL-乙酰转移酶-T5AOOA,紫杉烷-10-β-羟基化酶-T10BHYD,番茄 - 14-β-羟基化酶-T14BHYD,2-α-羟基氮烷-2-o-苯甲酰基转移酶-2ah20Ob,10-deacetylbaccatin-III-10-O-乙酰转移酶-10d10a,3-n-debenzoyl-2-脱氧氧基醇-n-苯甲酰基转移酶-3ND2DNB,熊磷酸(Ursolic-醛-28-单氧基酶-UA28M,α-氨林 - 合成酶-Aasyn),OleAlic acid(β-氨基 - 合成酶-Aasyn,OleAnolic-醛 -28-单氧化酶-OA28M),谷甾醇(24℃-甲基转移酶-24Cmet),罗马啉酸I(4-香豆酰-4-羟基苯甲酸酯-3-羟基酶-4C4H3H,酪氨酸 - 转氨酶 - TTRAN),ROMARINIC ACID II (Hydroxyphenylpyruvate-reductase-HPPRE, Tyrosine-3-monooxygenase-TTRAN), METHYL CAHVICOL (Eugenol-o-methyltransferase-EOMET), EUGENOL (Alcohol-o-acetyltransferase-AOACE, Eugenol-synthase-ESYN, Isoeugenol-synthase-ISYN), LINALOOL (Farnesyl-pyrophosphate-synthase-FPSYN, R-linool-synthase-RLSYN, S-linool-synthase-SLSYN), CARYOPHYLENE (Alpha-humulene-synthase-AHSYN, Beta-caryophyllene-synthase-BCSYN), SELINENE (Alpha-selinene-synthase-ASSYN, Beta-selinene-synthase-BSSYN), CITRAL (Geraniol-synthase-GSYN, Geraniol-dehdrogenase-GDHYD)
在遗料基因组中鉴定了共458个基因,其是同源的或直接编写参与专用代谢物合成的酶(图。8)(这些蛋白质的基因id详情见表4和附加文件21.:表S9)。二十八O. Tenuiflorum.BLAST序列搜索结果为推测的萜烯合成酶,e值为10−4查询覆盖率过滤器>75%(附加文件)22.:表S10)。
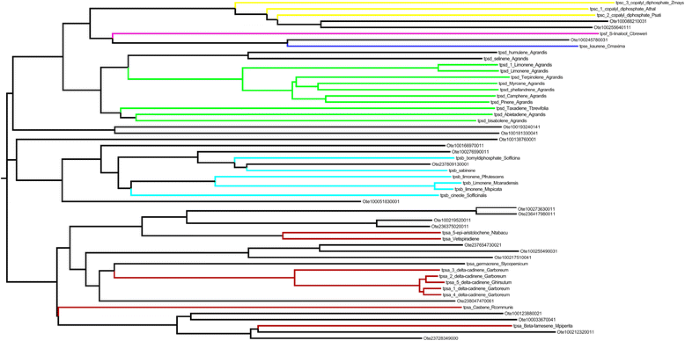
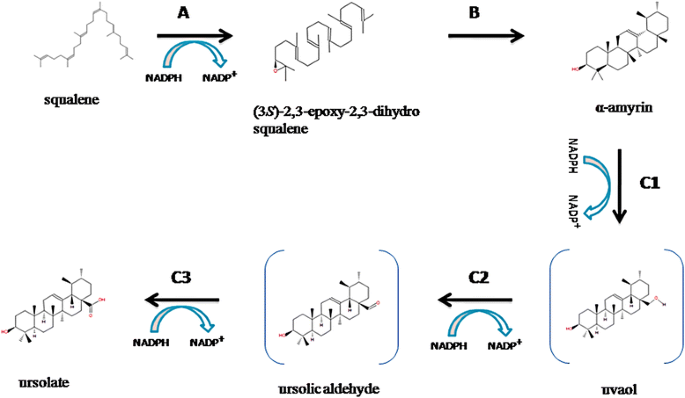
在这些专业的代谢物中,我们专注于属于筛窦的熊糖酸,因为已知具有抗炎,抗微生物,抗肿瘤和抗癌性质。来自Squalene的熊磷酸的合成是从角鲨烯开始的三步方法(图。9). α-胰淀素由环氧化物角鲨烯协同环化形成,而熊果酸最终由多功能细胞色素P450催化合成。因此,所涉及的酶是角鲨烯环氧化酶、α-胰淀素合酶和α-胰淀素2,8单加氧酶。序列搜索算法用于搜索Tulsi基因组中该途径的三种酶,从PlantCyc数据库中每种酶的蛋白质序列开始作为查询。利用Tulsi中角鲨烯环氧化酶的序列在栽培稻粳稻(LOC_Os02g04710.2)作为查询,产生一个hit (C3776143),其中50%的序列标识覆盖80%的查询长度(附加文件23.:图向)。利用来自A. Thaliana.(Q8RWT0)和其他13个被广泛接受的α / β amyrin合成酶作为查询,在Tulsi基因组中鉴定出4个命中的序列(scaffold16333, scaffold20801, scaffold12312和maker-C3776143)。在经典的amyrin合酶中,QW结构基序在整个序列中重复6次[17,18,而有两个功能主题,即。,一间保存完好的SDTAE [19]基序,该基序被认为是形成催化囊和多壁循环磷酸酶[20.基序在催化中起关键作用。这些motif被观察到在图尔西基因组的四个命中(附加文件24.进一步,使用16个查询序列和这4个查询结果构建了系统发育树(图4)。10).其中一个Tulsi命中,(scaffold 16333_mrnal)簇具有来自c . roseus也叫(H2ER439)表明这个特殊的支架可能确实保留了α - amyrin合成酶。
有趣的是,许多参与与疾病治疗相关的特殊代谢物合成的基因也更丰富,正如在组装的转录组中观察到的那样(附加文件)21.:表S9)。类似地,参与16种其他专门化代谢物合成的基因(附加文件25.:表S11),也同样有趣。然而,这需要详细了解合成机制和参与途径的酶。我们分析了两个叶片样品的RNA-Seq数据,以比较与重要代谢途径和特殊表型相关的基因O. Tenuiflorum.克里希纳亚型与罗摩图西亚型。有104个转录本,其表达的fold change在Krishna Tulsi中被观察到比Rama Tulsi多8倍。同样,在Rama Tulsi中有229份转录本的fold change表达是Krishna Tulsi的8倍。这些可以在- (caps.ncbs.res.)下载。在/下载/ tdat_data / Supplementary_tables /补充表8. txt)。
在多功能细胞影脉P450的情况下(该催化铀酸合成的最后三步骤)。9),从scaffold2032中获得了一个预测基因,当从UniProt中审查了F1T282v葡萄被认为是查询,并在塔尔西基因组组装中使用爆炸搜查。此命令保留了61%的序列标识,并且对齐覆盖查询长度的90%(对齐在附加文件中显示23.:图向)。该支架共包含三个预测基因,即Ote100020320011, Ote100020320001(类似于uhrf1结合蛋白)和Ote100020320031(感兴趣基因)。
从可用的转录组组件中,分析了鉴定的这些基因,其鉴定在尿囊酸的合成中鉴定为它们的表达水平。这三个基因的RPKM值也很高(请参阅附加文件21.:表S9)。为了进一步验证这些基因的表达水平,使用序列特异性引物进行q-RT-PCR。这三种酶在所有成熟叶片样品中的含量通常较高,在Rama亚型中最高(使用克里希纳亚型作为对照)。α-胰淀素合酶在小麦成熟叶片样品中更为丰富O。格雷西姆和o . sacharicum物种。然而,有趣的是,这三种酶在Rama亚型的幼叶样品中更丰富;相比之下,克里希纳树叶样本和所有根样本中至少有一个基因较少。在尿糖酸合成的样本中,三个基因的表达均较低o . kilmund。
接下来,为了关联基因表达并量化熊果酸和丁香酚的存在,使用lc -质谱对不同组织和样品进行化学分析。Rama亚型成熟叶样品中丁香酚和熊果酸含量最高,而Rama亚型成熟叶样品中丁香酚和熊果酸含量相对较低O. Kilmund.. 植物叶片样品中丁香酚的含量O. Tenuiflorum.Rama亚型(2235 ng/mg)相当高,其次是O. Kilmund.(1472 ng /毫克),o . sacharicum(651 ng / mg)和最低的O。格雷西姆(73纳克/毫克)。在所有的茎样本中,丁香酚的含量始终较低,最高的是O. Tenuiflorum.Rama亚型(24 ng/mg),O. Tenuiflorum.亚型krishna(17 ng / mg),O. Kilmund.(15 ng/mg)及以下的定量限值O。格雷西姆和o . sacharicum.与成熟叶样品相比,在RAMA亚型(2869ng / mg)的干燥样品中,在RAMA亚型(2869 ng / mg)和Krishna亚型(1088ng / mg)中的存在性,oleaHolic酸的存在(1088 ng / mg)(7556 Ng / mg,RaMa和4630 ng / mg对于克里希纳)。与成熟叶样品(4597)相比,在RAMA亚型(2883ng / mg)的干燥样品中存在铀酸的存在少50%,而与叶片样品相比,其它物种的茎样品中的茎样品中较低得多.Krishna亚型(746ng / mg)的茎样品中熊胆酸的量比成熟叶样品的4.6倍(3471 ng / mg)(请参阅表格5).
讨论
O. Tenuiflorum.克里希纳图尔西亚型是一种具有巨大药用价值的非模式植物,迄今为止还没有可用的基因组信息。我们已经进行了基因测序O. Tenuiflorum.成对末端克里希纳亚型(PE;2x100 bp)和配对(MP;Illumina Hiseq 1000的2x50 bp)DNA文库。最好的新创由真核生物soapdenvo2在k-mer 43获得新创基因组汇编。利用基因组学、转录组学和EST数据,利用MAKER注释管道进行基因预测和注释。最近的基因组已被测序的物种是猴花(m . guttatus),它与O. Tenuiflorum.(OTE)但落在不同的家庭(虾)。四种基因组QIZ中的OTTO krishna tulsi基因的正轨。A. Thaliana.(ATH),m . guttatus(开战),S. lycopersicum.(狡猾)和o.苜蓿(Osa)也证实了克里希纳·图西和m . guttatus(MGU),就常见基因家族的数量,即2488个总基因中的578种。当我们考虑来自克里希纳廷西基因组的所有36,768个预测的基因时,我们发现1282个ortholog组有罗勒属- - - - - -只有基因。这1282组包含13306个罗勒属因此,OrthoMCL把它们称为谬误组。在剩余的Ote基因中,发现有17584个基因与本案例中研究的其他四个物种中的任何一个同源。我们分析了与代谢产物产生有关的基因注意以及其他一些相关植物的基因组。根据直接证据或同源性,在Ote基因组中共鉴定出458个基因,这些基因参与特定代谢物合成中暗含的酶的编码。不同基因转录组的比较分析O. Tenuiflorum.进行亚型Krishna和Rama以检测潜在的差异调节基因及其参与代谢物合成。在比较转录ome,观察到差异表达的基因,在大量的基因中观察到更丰富,其他亚型在任何亚型中都不那么丰富。与RAMA样品相比(RPKM值31.73)相比,Krishna样品(RPKM值577.047)中γ-起角花合酶更丰富。为了确认一些更丰富的基因以及γ-起角花合酶,我们在不同的组织样品中进行Q-RT-PCR,即茎和叶,也以五种viz。O. Tenuiflorum.奎师那和罗摩的亚型,O。格雷西姆,o. basilicum.,o . kilmand。通过q-RT-PCR,在Krishna样品中发现γ -cadinene合酶的表达量高于Rama。同样,查尔酮合酶(Chalcone synthase, CHS)是一种产生花青素的基因,在转录组数据中发现,该基因在Krishna的幼叶样本和成熟叶样本中含量更高。随后,q-RT-PCR和来自不同组织样本和不同物种的熊果酸和丁香酚的质谱读数证实了这一点。
结论
我们提出了一个草图的基因组O. Tenuiflorum.克里希纳图西亚型克里希纳图西。属的栖息地罗勒属热带气候是亚洲,非洲,中美洲和南美洲广泛传播。观察到与RAMA亚型相比,负责克里希纳亚型的植物部件紫色着色的基因的高RNA-SEQ表达值。我们还确定了一种蛋酒的独特基因(16),其缺乏来自本研究中使用的所有22种的可追踪原子和同源性关系。
奎师那图西在《吠陀经》和《往世书》(印度古籍)中有描述,有悠久的栽培历史,大约有3000年,因此被认为是印度裔[21.].在文学上,它也被称为“药草女王”。尽管在测序和覆盖方面的数据有限,但参与植物中重要药用专门代谢物合成的主要基因可能被解开[22.].通过互补RNA-SEQ数据和Q-RT-PCR方法来确认这些基因的表达。我们还通过质谱和Q-RT-PCR方法研究了涉及熊酸生产的重要代谢途径之一。专门的代谢物或其前体的合成似乎开始于Tulsi的幼叶。随后,成熟的叶子保留了药物相关的代谢物。O. Tenuiflorum.RAMA亚型保留了丁香酚和熊果酸等键的主要药物相关代谢物,如转录组,代谢物量化和Q-RT-PCR表达值所观察到的,其与其高药物值一致。我们的主要重点是通过使用基因组和转录组数据来解开重要的代谢物基因,尽管测序信息有限。
方法
基因组DNA的分离O. Tenuiflorum.亚型克里希纳·塔尔西
Tulsi亚型Krishna和Rama的年轻叶子用于基因组DNA分离。加入约一克叶子用液氮和DNA提取缓冲液(200mM TrishCl [pH-8.0],200mM NaCl,25mM EDTA和1%PVP)粉碎[23.].将研磨材料一起在65℃下在65℃下温育30%SDS溶液30分钟。将管在室温下以14,000rpm离心10分钟以除去碎片。将上清液转移到新鲜管中并用相等体积的苯酚处理:氯仿:异戊醇(25:24:1)并轻轻混合5分钟。将混合物以12,000rpm离心10分钟以分离相。将来自离心管的水相转移到新鲜的管中,用1/5体积的2M NaCl和2体积的冰冷乙醇沉淀DNA。通过以12,000rpm离心10分钟,将DNA沉淀。使用Sigma基因株植物DNA分离试剂盒(G2N70,Sigma)作为纯化用于纯化的起始材料(G2N70,Sigma),将沉淀的DNA颗粒作为原料。在1%琼脂糖凝胶上运行DNA以评估质量。使用纳米玻璃测定A260 / 280的比率和数量。
基因组测序,装配和注释
通过在细胞和分子平台中心(C-CAMP)的下一代基因组学设施中使用Illumina Hiseq 1000技术进行基因组测序。基因组DNA配对和凝胶游离伴侣对库制剂用于使用Truseq DNA样品制备试剂盒(Fc-121-2001)和来自Illumina(www.illumina.com.).FASTX-Toolkit [24.]和FASTQC工具[25.]用于原始reads的预处理和reads的质量检查。用soapdenvo2, a新创基因组汇编程序草案[26.]. 根据k-mers从21到63进行初步组装,间隔为两次。使用MAKER注释管道进行基因预测和注释[27.]使用AUGUSTUS预测基因模型[28.),A. Thaliana.基因作为初始预测的参考。通过对所有蛋白质序列的同源性搜索,改进了基因模型Viridaeplantae王国。
基因组组装和注释的验证
为了验证基因组组装,我们将原始数据映射到新创利用REAPR(SMALT)组装基因组[29.],samtools [30.]及Picard工具(http://broadinstitute.github.io/picard/).最大插入长度为500 bp,最小插入长度为0 bp。我们报告了使用标准Smith-Waterman评分的最佳评分对齐配对。所使用的阈值最小值的计算公式为:<最小值> = <世界长度> +步长- 1。这里的单词长度为13,步长为6。图尔西基因组基因组大小的估算是通过水母的k-mer分布分析完成的[31.].在克利须那图尔西组装基因组中,通过双向方法确定了涉及植物细胞调控、组装和功能的基本基因。首先,使用源自KOG数据库的CEGMA [32.](对于真核基因组)和任何真核基因组的核心蛋白(包括处于草拟阶段的),必需基因被注释。第二,子集A. Thaliana.从特征的基因数据库(DEG)数据库中提取基因,并与Krishna Tulsi组件进行比较。通过PFAM结构域注释方法进行提取的基因的验证。使用BLASTP进一步搜索来自Krishna Tulsi DataSet的推定的必要基因[33.],对NCBI (NR)数据库中的亲缘关系进行了比对,并构建了系统发育树。
重复鉴定
使用RepeatScout (version 1.0.5)鉴定组装的基因组中的重复元素[34.和RepeatMasker(版本4.0.3)[35.].图书馆从头开始repeatscout生成的重复被分类为使用重复repscout的重复分组模块(附加文件)分类为已知的重复类别(附加文件12:表S5)。的RepBase库和非冗余库ab-initio.然后用分类的重复序列来掩盖组装的基因组中的重复元素。然后用重复掩码的基因组组装进行基因组注释。
基因组注释
通过制造商注释管道处理克里希纳·塔尔西的重复掩蔽组装基因组[27.].奥古斯都(28.]用于基因预测,训练于A. Thaliana.基因模型。从叶样品获得的RNA-SEQ数据被用作EST证据来细化基因模型。属于蛋白质序列的初始基因模型Viridaeplantae以NCBI数据库中获得的蛋白为依据,进一步完善基因预测。EST和蛋白证据均使用EXONERATE制备[36.]并通过AUGUSTUS用于基因预测细化。这些基因模型的所有蛋白质序列均通过E值截止值为10时针对NRDB的BLASTP搜索鉴定同源物进行验证−3.咨询PFAM版本27,用于所有域预测,E-Value截止值10−5使用HMMER3包[37.].
Orthology检测
所有来自Krishna Tulsi的预测基因模型均使用OrthoMCL工具[38.]来识别选定的物种之间的群集A. Thaliana.(ATH),o.苜蓿(Osa),S. lycopersicum.(狡猾),m . guttatus(MGU)。为了检查局部关系的分布罗勒属不同物种和分类水平的基因,ProteinOrtho工具[39.在克里希纳图尔西(Ote)基因模型以及22个不同物种上实施:Aquilegia caerulea(Aca),大豆(Gma),Setaria斜体(坐),Mimulus guttatus(开战),茄属植物lycopersicum(狡猾),拟南芥蒂利亚纳(ATH),Medicago truncatula(地铁),卷柏moellendorffii(smo),芸苔属植物拉伯(内衣),水稻(Osa),Solanum Tuberosum.(斯图),卡里卡番木瓜(CPA),Physcomitrella patens.(Ppa),Theobroma可可(柠檬酸),茶树(Csi),Prunus Persica(个人防护装备),vitis Vinifera(VVI),巨桉(Egr),杨树trichocarpa(PTR),玉米(ZMA),草莓属vesca(FVE),二色高粱(印度国家银行)。所有完整的蛋白质组均来自Phytozome资源[40].利用“RbcS”(Rubisco小亚基)编码序列对23个物种进行系统发育树重建。CLUSTALW [41.]及philip package [42.]分别进行多序列比对(MSA)和相邻连接(NJ)聚类。通过PSI-BLAST验证了远缘同源关系[33.]在不同的e值临界值集。我们无法建立任何同源或同源关系,但由Pfam结构域组成的基因产物被认为是Ote特有的基因。
图尔西与其他植物基因组的比较分析
最新版本的全基因组序列S. Lycopersicum,V.Vinefera,M. Tranculata和A. Thaliana.下载自NCBI (ftp://ftp.ncbi.nlm.nih.gov/genomes/).blat [43.使用序列搜索使用S. Lycopersicum,V.Vinefera,M. Tranculata和A. Thaliana.两组Tulsi基因组数据:一组包含11389个支架(大于10000 bp),另一组包含167个支架和带有代谢相关基因的contigs(在Krishna Tulsi基因组中较早发现)。这些数字是使用为此目的编写的内部软件编制的。
Tulsi亚型、Krishna亚型和Rama亚型RNA的分离及RNA-seq文库的制备
使用Sigma谱植物总RNA试剂盒(STRN50, Sigma)对100 mg的叶片组织(Rama和Krishna)进行RNA分离。使用无DNA™试剂盒(AM1906, Ambion)通过DNAse处理去除DNA污染。采用安捷伦生物分析仪检测dna酶游离RNA质量。所有样品的RNA Integrity Number (RIN)均大于6。利用纳米滴确定了A260/280的比例和用量。按照Illumina公司的TruSeq RNA样品(RS-122-2001),用1 μg总RNA制备RNA-seq文库。
转录组测序和组装
我们分别组装了Krishna和Rama亚型中所有超过20个碱基的HQ分数的所有mRNA reads,并通过使用soapdenvo -trans将这两个亚型的reads结合起来[26.]在不同的K-mers,从19到63,间隔为2。插入大小为350的片段用于转录组的组装。RNA seq读取通过Tophat2映射到组装的基因组[44.],使用Bowtie2 [45.]作为映射工具。我们使用的最小和最大内含子长度分别为50和50000bp。最大多重命中数(指定给定读取与参考比对次数的参数)指定为20,转录组最大命中数(与转录组比对时允许读取的最大映射数)指定为60。
转录差异表达比较
以每千碱基每百万reads (RPKM)来量化表达,非冗余组合组装的转录本序列(CD-hit EST序列相似性为90%)[46.)作为参考。该非冗余转录组作为参考转录组,计算两个样本中转录本的差异表达[6,47.].通过使用SeqMap (version - 1.0.12), Krishna和Rama亚型的RNA-seq实验reads被映射回参考转录组[48.]使用rSeq:RNA-seq分析仪(版本0.1.1)测定RPKM和RPKM值[49.].
专门的代谢物检测和验证
通过对组装后的基因组进行基因预测获得的数据集,用于搜索与次生代谢产物生产相关的酶。有14种代谢物(类黄酮(2),苯丙素(4),萜类(2),倍半萜类(5)和甾醇(1))被报道存在于罗勒属已知PlantCyc (http://www.plantcyc.org/) [16].来自UniProt数据库的审查条目和来自其他具有这些酶的物种的所有已知的酶序列被用作查询,在scaffold和contigs的完整数据集中进行搜索,使用pci - blast, e值为10−5和三个迭代。在我们的数据集中获得的蛋白质点击率进一步使用75%的查询覆盖率过滤器进行验证。
为了研究参与特殊代谢物合成的基因的表达,组装了两者的转录组罗勒属搜索物种,采用UNIPROT数据库中对应于每种酶的审查条目。这些搜索是使用Tblastn执行10的tblastn来执行−3,在我们的数据集中,最好的击中是根据最小的e值选择的。如果任何一种酶的审查条目不存在,则使用PlantCyc数据库中的未审查条目。
UHPLC-MS/SRM法测定丁香酚和熊果酸的含量
使用Vantage TSQ三级四极杆质谱仪(Thermo Fisher Scientific, San Jose, CA, USA),配备加热电喷雾电离(HESI)源分析丁香酚,使用APCI探针分析熊果酸。质谱仪与Agilent 1290无限UHPLC系统(Agilent Technologies India Pvt. Ltd., India)连接,配有柱箱(设置在40°C)、自动进样器和温度控制器(设置在4°C)。每次注射前用乙腈(0.1%甲酸)从外部清洗针头,以避免任何潜在的遗留问题。采用垫片包装XR-ODSIII色谱柱(2 × 150 mm, 2 μm)进行分离。丁香酚:流动相A为含0.1%甲酸的水(10 mM醋酸铵),流动相B为含0.1%甲酸的乙腈。熊果酸:流动相A为水(10 mM乙酸铵),流动相B为乙腈:甲醇(3:1)。10 μL采用针流注射
- (一种)
丁香酚:
以雌酮-d4为内标,与吡啶磺酰氯衍生后对丁香酚进行定量分析。用甲醇从鲜叶(2 mg/mL)和干茎粉(20 mg/mL)中提取丁香酚。将提取液10 μL和内标液10 μL (2.5 μg/mL)加入缓冲液[丙酮:NaHCO3 (1:1)] 200 μL中。这10μL吡啶磺酰氯(10毫克/毫升)添加和孵化,孵化后60°C,持续15分钟。800μL的导数提取MTBE和有机层是干和重组50μL甲醇紧随其后10μL注入分析。以200 μL/min的流速启动梯度(0-2 min: 30% B, 2-5 min:30 - 90% B, 5-7 min:90 - 100% B, 7-10 min: 100% B, 10-10.1 min:100 - 30% B, 10.1-15 min:30)。操作条件为:喷淋电压3000 V;离子转移毛细管温度270°C;源温度100°C;鞘气20,辅助气5(任意单位);碰撞气体氩; S-lens voltage was optimized for individual metabolites; scan time of 50 millisec/transition; and ion polarity positive. A standard curve was constructed from 0.078 to 5ngon column to quantify eugenol. The SRM transition used for the analysis of eugenol is (306.1 → 79) and for estrone-d4 (416.3 → 274.1).
- (b)
熊果酸:
以雌酮-d4为内标,定量测定熊果酸。从2 mg/mL的干粉中使用1 mL的甲醇进行简短的提取(超声-3分钟,离心- 5分钟)。提取液在甲醇中进一步稀释至0.2 mg/mL。提取液10 μL,加入10 μL内标液(0.1 ug/mL)和30 μL甲醇,注射10 μL进行分析。以200 μL/min的流速启动梯度(0-2 min: 20% B, 2-8 min:20 - 100% B, 8-14.5 min: 100% B, 14.5-14.6 min:100 - 20% B, 14.6-20 min: 20% B)。实验条件为:放电电流4 μA;离子转移毛细管温度270°C;源温度300°C;鞘气20,辅助气5(任意单位);碰撞气体氩; S-lens voltage was optimized for individual metabolites; scan time of 50 millisec/transition; and ion polarity positive. A standard curve was constructed from 0.034 to 2.5 ng on column to quantify ursolic acid. The same standard curve was used for the analysis of oleanolic acid. The SRM transition used for the analysis of both ursolic and oleanolic acid is (439.4 → 119) and for estrone-d4 (275.3 → 257.1).
支持数据部分的可用性
图西基因的信息,以及支架的编号,都提供在http://caps.ncbs.res.in / sote..
Bioproject:prjna251328
SRA ID:SRP051184
加入的数量O. Tenuiflorum.:JQCZ00000000
DateRad Digital存储库提供的数据:http://dx.doi.org/10.5061/dryad.6f1r2.
缩写
- 体育:
-
成对的结束
- 议员:
-
配对
- 塞加尔:
-
核心真核基因映射方法
- 度:
-
基本基因数据库
- LTR:
-
长末端重复
- 投票:
-
圣罗勒
- 重点注意:
-
拟南芥蒂利亚纳
- 一开战:
-
Mimulus guttatus
- 狡猾:
-
茄属植物lycopersicum
- 阻塞性睡眠呼吸暂停综合症:
-
水稻(阻塞性睡眠呼吸暂停综合症)
- 苏维埃社会主义共和国:
-
简单序列重复
- CHS:
-
查耳酮合酶
- 红细胞表面:
-
二磷酸核酮糖羧化酶小亚基
- MSA:
-
多序列比对
- NJ:
-
邻居加入
参考文献
- 1.
帕顿A、哈雷RM和哈雷MM。Ocimum概述了关系和分类。罗勒芳香植物工业概况。阿姆斯特丹:哈伍德学院(1999年)
- 2。
威利斯JC。开花植物和蕨类植物的字典,由J. C. Willis。剑桥:大学出版社;1919年。
- 3.
丁香酚和它的药理作用的注释:一个简短的回顾。acta physol Pharmacol. 2005; 49:125-31。
- 4.
《印度药用植物:有插图的字典》。施普林格科学与商业媒体;2007.
- 5。
Rao PS,卫星A, Moridani M, Jenkins M, Rao US。木犀草素在不影响药物转运体功能的情况下诱导多药耐药癌细胞凋亡:涉及细胞系特异性凋亡机制。中华结核和呼吸杂志。2012;130:2703-14。
- 6。
góngora-castillo e,fedewa g,yeo y,chappell j,dellapenna d,buell cr。用于询问药用植物物种生物化学的基因组方法。方法酶。2012; 517:139-59。
- 7.
Rastogi S, Meena S, Bhattacharya A, Ghosh S, Shukla RK, Sangwan NS,等。圣罗勒和甜罗勒转录组的从头测序和比较分析。BMC基因组学。2014;15:588。
- 8.
Carović-Stanko K, Liber Z, beendorfer V, Javornik B, Bohanec B, Kolak I, et al.;基于分子标记、核DNA含量和染色体数目分析罗勒类群的遗传关系。acta botanica sinica(云南植物研究),2009;
- 9
莫顿JK。西非唇形科的细胞分类学研究。林恩,伦敦,博特。1962;58:231–83.
- 10.
科斯拉M,索布蒂SN。罗勒属植物核型形态学研究。圣殿集团。细胞学50:253-2631935。
- 11.
CEGMA:在真核生物基因组中准确注释核心基因的途径。生物信息学。2007;23:1061-7。
- 12.
张锐,欧宏宇,张春涛。基本基因数据库。核酸Res. 2004;32(数据库版):D271-2。
- 13。
Apweiler R,Bairoch A,Wu Ch,Barker Wc,Boeckmann B,Ferro S等人。UNIPROT:通用蛋白质知识库。核酸RES。2004; 32(数据库问题):D115-9。
- 14。
Ferrer JL,JEZ JM,Bowman Me,Dixon Ra,Noel JP。Chalcone合成酶的结构与植物聚酮化合物生物合成的分子基础。NAT STRUCT BIOL。1999年; 6:775-84。
- 15.
波特诺伊V, Benyamini Y,巴尔E, hael - beja R, Gepstein S, Giovannoni JJ,等。甜瓜(Cucumis melo L.)皮倍半萜含量变异的分子生化基础。植物学报。2008;66:647-61。
- 16。
植物代谢网络(PMN),http://www.plantcyc.org/tools/tools_overview.faceswww.plantcyc.org,2008年2月28日[http://www.plantcyc.org/about/citing_pmn.faces]. 日期:2014年8月5日。
- 17
王敏,王敏,陈志强,等。一种罕见的植物三萜合酶,具有显著的α-淀粉酶活性,通过对来自苹果的氧化角状烯环化酶的特征鉴定。2月j . 2011; 278:2485 - 99。
- 18
Poralla K,Hewelt A,Prestwich Gd,Abe I,Reipen I,Sprenger G.在Squalene和氧化喹啉环酶中的特定氨基酸重复。趋势Biochem SCI。1994年; 19:157-8。
- 19
Abe I, Prestwich GD。脊椎动物氧化角鲨烯环化酶活性位点的鉴定。脂质。1995;30:231-4。
- 20.
陈志强,陈志强,陈志强,等。三萜合成酶的突变研究:合成-戊苷合成酶。2000:6816 - 6824。
- 21.
巴斯特F,拉尼P,米娜D。印度次大陆罗勒叶绿体DNA系统地理学。科学世界杂志。2014;2014:847482.
- 22.
王伟,冯波,肖建军,夏志,周旭,李鹏,等。从野生祖先到栽培品种的木薯基因组。Nat Commun。2014;5:5110。
- 23.
Ghosh P,Chattopadhyay Sk,Adhikari S,Saha S,Mondal S.一种高通量DNA提取方法,来自化学上的异质植物。P. 2013; 2603。
- 24。
Lab H. FastX Toolkit。http://hannonlab.cshl.edu/fastx_toolkit/index.html.
- 25。
安德鲁斯S。FastQC是一种用于高通量序列数据的质量控制工具。http://www.bioinformatics.babraham.ac.uk/projects/fastqc/.
- 26。
谢勇,吴刚,唐建军,罗荣杰,刘胜,等。SOAPdenovo-Trans:与短rna序列的从头转录组组装。生物信息学。2014;30:1660-6。
- 27.
Cantarel BL, Korf I, Robb SMC, Parra G, Ross E, Moore B,等。MAKER:为新兴模式生物基因组设计的易于使用的注释管道。基因组研究》2008;18:188 - 96。
- 28.
STANKE M,Morgenstern B. Augustus:用于基因预测的Web服务器,用于允许用户定义的约束。核酸RES。2005; 33(Web服务器问题):W465-7。
- 29.
Hunt M, Kikuchi T, Sanders M, Newbold C, Berriman M, Otto TD。REAPR:用于基因组组装评估的通用工具。基因组医学杂志。2013;14:R47。
- 30.
陈志强,王志强,王志强,等。序列对齐/映射格式和SAMtools.pdf。生物信息学。2009;25:2078-9。
- 31。
Marçais G, Kingsford C.一种快速、无锁的k-mers并行计数方法。生物信息学。2011;27:764 - 70。
- 32.
Tatusov RL, Fedorova ND, Jackson JD, Jacobs AR, Kiryutin B, Koonin EV等。COG数据库:更新版本包括真核生物。BMC生物信息学。2003;4:41。
- 33.
Altschul SF,Madden TL,Schaffer AA,张J,张Z,米勒W等人。Papped Blast和Psi-Blast:新一代蛋白质数据库搜索程序。核酸RES。1997年; 25:3389-402。
- 34.
Price AL, Jones NC, Pevzner PA。大基因组中重复家族的从头鉴定。生物信息学。2005;21增刊1:i351-8。
- 35.
重复掩蔽器开-3.0。;1996–2010.http://www.repeatmasker.org.[http://www.repeatmasker.org/faq.html#faq3]
- 36。
斯莱特GSC,伯尼E。自动生成用于生物序列比较的启发式算法。生物信息学。2005;6:31.
- 37。
Mistry J,Finn Rd,Eddy SR,Bateman A,Punta M.在同源性中的挑战:HMMER3和卷绕式线圈区域的收敛演变。核酸RES。2013; 41:E121。
- 38。
Li L, Stoeckert CJ, Roos DS。OrthoMCL:真核生物基因组同源组的鉴定。基因组研究》2003;13:2178 - 89。
- 39。
Lechner M,Findeiss S,Steiner L,Marz M,Stadler PF,Prohaska SJ。蛋白质替代:在大规模分析中检测(CO-)矫正器。BMC生物信息学。2011; 12:124。
- 40.
等。植物群落:绿色植物基因组学的比较平台。核酸Res. 2012;40(数据库版):D1178-86。
- 41.
Larkin Ma,Blackshield G,Brown NP,Chenna R,McGettigan Pa,McWilliam H,等。Clustal W和Clustal X 2.0版。生物信息学。2007; 23:2947-8。
- 42.
费尔森斯坦J。PHYLIP-系统发育推断包(3.2版)。分支学。1989;5:164–6.
- 43.
肯特WJ。BLAT——类似blast的对准工具。基因组研究》2002;12:656 - 64。
- 44.
Kim D, Pertea G, Trapnell C, Pimentel H, Kelley R, Salzberg SL. TopHat2:在插入、缺失和基因融合中转录组的精确比对。基因组医学杂志。2013;14:R36。
- 45.
Langmead B, Salzberg SL.快速间隙读对齐与领结2。Nat方法。2012;9:357-9。
- 46.
Li W,Godzik A. CD-his:用于聚类和比较大套蛋白质或核苷酸序列的快速计划。生物信息学。2006; 22:1658-659。
- 47.
góngora-castillo e,buell cr。在没有参考基因组序列的情况下使用短读取序列在De Novo转录组组件中挑战的生物信息学挑战。Nat Prod Rep。2013; 30:490-500。
- 48。
江华,王华。SeqMap:将大量寡核苷酸映射到基因组。生物信息学。2008;24:2395–6.
- 49。
Salzman J,Jiang H,Wong Wh。RNA-SEQ数据的统计建模。stat sci。2011; 26:62-83。
确认
我们感谢R. Savithri女士和Anna Spudich博士进行有用的讨论。我们感谢NCBS和串行的基础设施和其他设施。测序在C-CAMP(BT / PR3481 / INF / 22/140 / 2011)进行。我们感谢Kannan先生在C-Camp完成的代谢物确认。我们承认NCBS奖学金对AU和RS,GLUE GRANT(BT / PR15352 / MED / 15/15/15 / 2011)的财务支持由印度生物技术部门提供给AC。,卓越奖Grant(BT / 01 /Coe / 09/01)由Biotechnology,印度,okm,AS,Sn,SNP和MS资助。,大学拨款委员会,印度,印度为PG和Kothari奖学金到MM,桥梁博士后研究员,NCBS和Instem印度到KH,APJ,MN,PS和MSS。,印度生物技术部资助的印度至KH的BT / IC /日本(BI)/ 01/2010。,印度科学和工业研究理事会到AGJ,SDK,JM,AS和USR,Biotechnology奖学金系,印度SK,EM,PS和SM,由印度科学和工业研究理事会资助的越来补助金(37/1606 / 13 / EMR-II)SN。,印度政府生物技术系(Ramalingaswami奖学金授予; BT / HRD / 35/02 / 2006)到MG。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明他们没有相互竞争的利益。
作者的贡献
构思和设计的实验:Rs,SR,MG。数据生成:NP,CS,RM,SS,MS,AR,SN,Mn。数据分析和演示:Aku,AS,MS,KH,EM,APJ,AGJ,OKM,PNS,USR,SM,SK,AG,NK,HRS,ARC,SDK,JM,PG,MM,MSS,HRS,SN,SNP。写作稿件:Aku,As,Ms,Kh,Em,APJ,AGJ,OKM,PNS,USR,SM,SK,AG,NK,HRS,ARC,SDK,JM,PG,MSS,HRS,SN,SNP.提供资源和工具和批判综述稿件:Rs,Mg。所有作者阅读并认可的终稿。
附加文件
附加文件1:图S1。
用于最终基因组组装的PE序列R1 reads的每碱基序列质量。
附加文件2:图S2。
用于最终基因组组装的PE序列R2 reads的每碱基序列质量。
附加文件3:图S3。
组装支架的长度分布。
附加文件4:图S4。
配对端与配对对端组件之间支架长度差的分布。
附加文件5:表S1。
MP + PE和PE组件中的脚手架长度分布。
附加文件6:表S2。
统计PE和MP + PE组合支架长度比较。
附加文件7:表S3。
CEGMA结果组装的完整性和基本基因的存在O. Tenuiflorum.从两个层面;(a)只在PE装配中(b)在PE + PM装配中。
附加文件8:表S4。
必需基因的存在O.tenuiflorum(Tulsi)在三个层面;一个)in only paired end assembly (ab-initio gene prediction), b) in paired end and mate-pair assembly’s Level 2 [evidence from RNAseq, EST and known tulsi genes], c) in paired end and mate-paired assembly’s Level 1 (gene prediction).
附加文件9:图S5。
来源细胞色素P450基本基因的系统发育树O.tenuiflorum以及它们各自的同系物。
附加文件10:图S6。
蛋白质甘油醛磷酸脱氢酶的NJ树O. Tenuiflorum.(图西,红色标记)及其最近的同系物。
附加文件11:图S7。
基因的系统发育树,肌动蛋白O.tenuiflorum以及它们各自的同系物。
附加文件12:表S5。
在图西基因组组装中鉴定出重复元件,并在不同的重复组中进行分类。
附加文件13:图S8。
所有预测基因的蛋白结构域(Pfam)的饼状图O. Tenuiflorum.亚型Krishna Genome。
附加文件14:表S6。
用于系统发育分析的物种列表,连同罗勒属描述本种在植物界的分类学分布。
附加文件15:图S9。
圆形的代表O. Tenuiflorum.代谢相关基因的图谱vitis vinefera.植物基因组。颜色表示蓝色=2个基因,绿色=2个基因,黄绿色= > 2个基因,红色=代谢物相关基因。支架与染色体之间的连接线表示支架在基因组中的位置。连接线的红色表示代谢物相关基因的存在。
附加文件16:图S10。
圆形的代表O. Tenuiflorum.代谢相关基因的图谱Medicago tranculata植物基因组。颜色表示蓝色= < 2个基因,绿色=2个基因,黄绿色= > 2个基因,红色=代谢相关基因。支架与染色体之间的连接线表示支架在基因组中的位置。连接线的红色表示代谢物相关基因的存在。
附加文件17:图S11。
圆形的代表O. Tenuiflorum.代谢相关基因的图谱拟南芥蒂利亚纳植物基因组。颜色表示蓝色= < 2个基因,绿色=2个基因,黄绿色= > 2个基因,红色=代谢相关基因。支架与染色体之间的连接线表示支架在基因组中的位置。连接线的红色表示存在代谢相关基因。
附加文件18:表S7。
联想O. Tenuiflorum.a)与代谢物相关基因相关的支架,b)长度的支架(大小大于10kb)至四种不同的植物基因组。
附加文件19:表S8。
所有转录本在不同水平上的表达和验证,如基因组命中和blast命中NCBI非冗余数据库的结果。
附加文件20:图S12。
详细研究了图尔西基因组14种重要药物代谢物的代谢途径。
附加文件21:表S9。
用于每个具有已知途径的代谢物的专门代谢产物产生的基因ID。
附加文件22:表S10。
推定萜烯合成序列O. Tenuiflorum.基因组。
附加文件23:图S13。
A.支架14352预测的代谢产物蛋白质序列比对罗勒属和O65402蛋白质序列拟南芥。B来自scaffold16333中预测的蛋白序列比对罗勒属基因组和Q8RWT0蛋白质序列拟南芥。c。脚手架2032中预测的蛋白质序列的序列对准罗勒属基因组和F1T282蛋白序列葡萄蛋白质组。
附加文件25:表S11。
患有未知途径的代谢物,疾病的影响。有15个药用相关代谢物罗勒属sp。与未知的途径。
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
阿帕德海耶,a.k.,查科,a.r.,甘地。等等。图丝草基因组测序(圣罗勒)无解释其强大药用特性背后的关键基因。BMC植物BIOL.15,212(2015)。https://doi.org/10.1186/s12870-015-0562-x
收到了:
接受:
发表:
关键词
- O. Tenuiflorum.
- 罗勒
- Thulasi
- 基因组
- 转录组