抽象的
背景
分析了系统育孕组分析了18种PACMAD草(Subfamilies Panicideae,Aristideae,Chloridoideae,MicraIroideae),Danthoniodeae)的塑料序列。下一代测序方法用于为12种提供完整的塑性序列。进行Sanger测序以确定一个物种的塑料,Hakonechloa Macra,提供注释的参考。进行这些分析以解决思工内的深层关系。评估了分歧估计,以确定导致这种谱系快速辐射的潜在因素及其对暖栖栖息地的优势。
结果
对13个PACMAD物种的新细胞体进行了测序和特征分析。发现一个~1140 bp的自变性缺失Hakonechloa Macra令人挑剔的假发化rpl14.并消除rpl16.从这个塑料。作为姐妹组的系统核发生物分析支持Acmad Clade。完整的塑料序列在Pacmad Clade内的深部节点提供更大的载体。估计Pacmad Subfamilies的初始多样化估计在32.4 mya。
结论
完全塑性的系统核糖分析为PACMAD草的深层关系提供了分辨率。Pacmad Clade在Pacmad Clock节点处的32.4 mya的分歧估计与何寡烯转化(EOT)一致。农民是全球冷却和干燥的时期,导致森林碎片和开放栖息地的扩张现在由这些草座主导。了解这些草如何相关并确定它们的快速辐射的原因允许在不断变化的全球气候中对草地分布的未来预测。
背景
由于其经济和生态重要性,Poaceae是许多系统发育研究的主题,以及主要陆地生物群系的优势[1- - - - - -5].Poaceae目前的系统发育分类包括深度的三种谱系:Anomochloideae,Pharoideae和Puelioideae,以及由BEP(Bambusoideae,Ehrhartoideae,Poooideae)代表的皇冠草[2]和Pacmad(Panicoideae,Aristidoideae,氯缺水,MicraIroideae,Arundinoideae,Danthonioideae)[3.演化支。本研究特别关注PACMAD分支,因为尽管其在经济和生态上具有重要意义,但其主要谱系之间的系统发育关系仍不确定。
BEP分支的姐妹组被不同地定义为具有不同组成亚家族的PACC、PACCAD、PACMAD或PACMAD分支。先前的研究[1利用质体基因序列ndhF,支持单味菌 - PACC(胰腺炎,Arundinoideae,Chloridoideae,CentoTheCoideae)的思考,以及表明Arundinoideae的多淋巴性质。随后由草来源发生工作组(GPWG)的工作[2通过使用七个分子数据集中的信息特征以及形态数据集来解决弱者内容的弱势支持。出于比较目的,我们将参考三种塑性序列的结果(ndhF,rbcl.,trnK / matK),而不是它们的八个数据集分析,因为这些分类并没有不同的子组织排列,或提供进一步的分辨率。
GPWG还增加了以前的系统发育研究的分类群,包括62个属的代表,其中30个,其中30个属于新成立的PACCAD(Panicoideae,Arundinoideae,Chloridoideae,CentoTheaideae,Aristidoideae,Danthonioideae)的植物中落下的2].嵌套在Paccad Clade内的三个分类群(Eriachne.,Gynerium, 和Micraira)被归类为Incertae Sedis.(不确定的安置)。还发现Arundinoideae缺乏统一形态或分子突录形态,以确定它为单晶素。本身归类为Incertae Sedis.在与其他代表的单独研究中进一步分析Eriachne.和Micraira通过使用69个结构性格以及ndhF和rpl16.质体序列(6].他们恢复微型射游作为独特的亚家族,改变了PACCAD首字母缩略词。随后研究中,随着胰岛胰腺炎和CentoTheCoideae的增加[7[是否得出结论,CentoThoideae与胰腺炎“......和名称不应具有系统发育意义”(第1738页)。该研究定义了PACMAD疏水板的组成亚颗粒,并建立了骨干系统发育假设,以探讨更深层的系统发育关系。
第二个GPWG构建了迄今为止最详细的草系统发育[4].其中一个主要目标是确定C的数量4光合作用在Pacmad Clade跨越的起源。他们分析了452种PACMAD物种,包括使用前一个GPWG研究的相同的塑体标志物在思克内三分之二(rbcl.,ndhF,trnK/mat).多种系统发育分析和分类样品的增加为Aristidoideae提供了诸如亚家族的姐妹对地区的其他地区的支持。然而,胰腺炎和CMAD(氯离样,MicraIroideae,Arundinoideae,Danthoniodeae)的关系仅是弱支持的(自举(BS)值:61%,后概率(PP):0.99),以及MA之间的关系(MicraIroideae,Arundinoideae)和DC(Danthonioideae,氯离样)疏水(BS值:51%,PP:0.98)。此外,雄树脂蛋白酶仅被弱支持单晶状体。
Pacmad草的深度分歧时间估计相对较少。部分原因是因为自信地日期的草化石的缺乏用作特定节点的校准点[8,9].用于校准的化石包括花粉、植物岩和小穗[8,10.,11.].另一个贡献因素是亚颗粒水平缺乏支持良好的拓扑,特别是对于Pacmad疏水板内的深层关系,这需要足够的分子序列。PACMAD疏水板的先前分歧估计是高度变化的,并且已经在茎aristidoideae(28.8至61.1 mya)中检查,皇冠pacmad(38至61.1 mya),茎胰腺(26至42.1 mya)[8,10.- - - - - -12.].这四项研究在系统发育分析中使用了相对少量的分子标记,并且可能导致拓扑的缺乏不变。
使用Poaceae的完全塑料的系统核发科学研究已经为与其他亚类内部和其他亚类的关系提供了强烈的支持[13.- - - - - -15.].本研究通过利用完整的塑料解决了PACMAD疏水板在PACMAD疏水区的深层节点的弱者的弱势支持。来自一种雄树脂蛋白物种的塑料(Hakonechloa Macra使用Sanger技术测序,以提供注释的参考,并通过Pacmad Taxa的下一代测序(NGS)方法确定12种完整的塑料。分析完全的塑料,分析了系统托儿或估计的发散日期,以寻求PACMAD辐射的潜在选择性原因。这里呈现的分析利用更多的系统源性信息性质,并且通过使用完全的塑料提供了更大的发散估计的准确性。
线粒体和质体序列可能产生不一致的基因树,因为不完全的谱系分类,重组事件,或潜在的替代率升高[16.,17.].这里探讨了线粒体序列,目的是提高代表性分类群之间的潜在遗传性的性格抽样。提取和分析线粒体序列数据,作为潜在保守的特征的来源,其在与塑性序列组合确定亚组合关系中有用[18.- - - - - -20.].
结果
外围集团选择、质体基因组
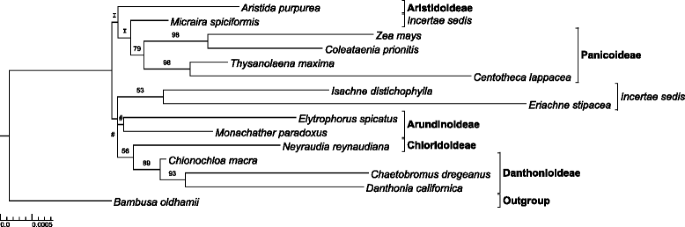
基于质体数据的PACMAD拓扑结构在不同外群体的可能性分析中基本保持一致。在除一个外群分类单元的分析中,Panicoideae与其余的PACMAD分类单元是姐妹类。当单一分类单元奥雅萨苜蓿被选为小组,Aristidoideae是剩下的Pacmad分类群,BS值为56%。请注意使用由作为一个外群不会以这种方式改变拓扑。
小组选择极大地影响了Aristidoideae的位置的支持值(附加文件1:图。S1)。只考虑单个分类小组,选择Puelia oloriformis.为PACMAD节点生成了67%的引导支持(bs)值。使用更接近的ehrhartoid物种,选用高产稻,将bs的价值提高到76%。当Bambusa Oldhamii.被用作除OffGroup这个节点的BS值为80%,但是Rhynchoryza subulata提供最大的支持,BS值为99%。Bambusa Oldhamii.由于这种物种可获得线粒体数据,因此选择了线粒体分析的小组。
质体系特征
这13个新细胞体在基因含量和组织结构上基本保守。短单拷贝区(SSC)长度为11771 ~ 14756 bp,长单拷贝区(LSC)长度为78798 ~ 82525 bp,反向重复区(IR)长度为20103 ~ 22730 bp1).在大肠杆菌中发现一个约1140 bp的独特缺失rpl14.和rpl16.地区Hakonechloa Macra.此删除消除了所有rpl16.以及前70 bp的rpl14.它们之间的非编码序列。删除在下游〜450 bprpl8和〜100 bp上游rps3..
质体系phylogenomic分析
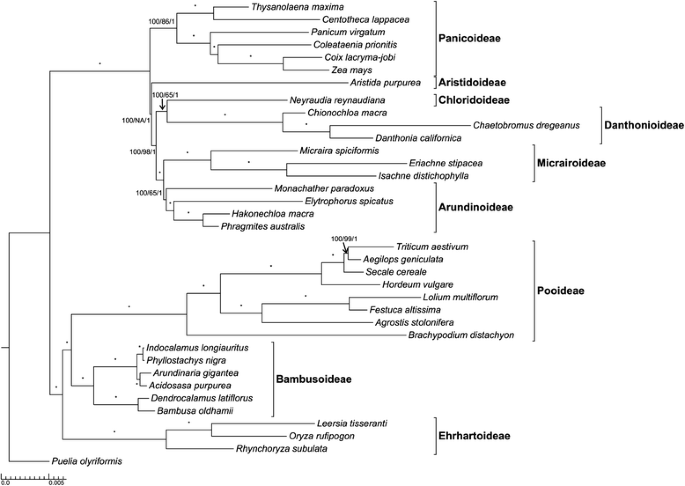
最大可能性(ML)分析产生了一棵树 - LNL = 274737.67。树的平均末端分支长度(0.009)大于内部分支长度的平均值超过2.5倍(0.0035)。在从M1分析产生的拓扑中,胰腺炎(六种)被欺骗亚家族分离到帕米德·疏地的其余部分(图。1).下一个亚家族分歧是Aristidoideae,它与CMAD疏水板合法,BS值为77%。将氯微脂作为Danthonioideae的姐妹,BS值为100%。MicraIroideae和Arundinoideae之间的姐妹关系也得到了100%的BS值。Elytrophorus spicatus嵌入在Arundinoideae中,并作为Hakonechloa Macra和芦苇澳大利亚人最大BS支持。将氯化物/丁二氨酸疏水疏水湿润为100的BS值作为孤立管/微单的姐妹。虽然此处检索到的ML拓扑得到良好的支持,但是Shimodaira-habegawa(SH)测试未能拒绝Aristidoideae姐妹姐妹到PCMAD Clade的替代假设(P <0.151)。贝叶斯推理(BI)拓扑与ML相同拓扑。aristidoideae的位置和拓扑中的所有其他节点的支持概率(PP)值为1.0。在Parsimony分析下的相同数据矩阵具有10,779个定义信息网站。最大分析(MP)分析产生了一棵长度为33,593步。MP分析具有0.5764的集合一致性指数,保留指数为0.7911。MP分析的发散顺序从ML和BI与Aristidoideae姐妹分析到Pacmad Subcamilies的其余部分,然后是胰岛素的次数不同。除了:冠状狂欢节(BS值= 65%),Crown DC(BS值= 99%),Crown Cmad(BS值= 98%),以及冠胰腺(BS值)(BS值 = 86 %). The sister relationship of two outgroup taxa,Triticum Aestivum.和aegilops geniculata.在MP分析中,bs值为99%。分区分析仅使用76个蛋白质编码序列,产生了相同的拓扑结果和类似的引导支持,这里不再进一步考虑。
线粒体分析
线粒体分析生成的树-lnL = 17041.15(图。2).胰腺炎是单噬细胞,但备受较弱的载体(BS值= 79%)。姐妹的关系t .最大值和Centotheca lappacea也z梅斯和Coleataenia prionitis均以98%的bs值支持。89%的bs值支持石竹科为单系。白头草亚科和chloridoidae的姊妹关系仍保持,但支持度较低(bs值:56%)。在这里取样的青苇属植物,其特征为单系,但bs值< 50%。
对15个物种的线粒体基质和7个内含子序列进行组装和排列分析得到的最大似然系统图。分支长度与沿分支的取代率成正比。Bambusa Oldhamii.被选为小组。注意到BS值> 50和<100。用#标记的节点表示BS值<50。标记为“I”的每个节点都与我们的ML塑性分析拓扑进行了不一致(图。1)
深层线粒体拓扑从塑性分析的差异很大(图。2).Arundinoideae是氯葡萄球菌/黄哚诺伊糖的妹妹,与BPWG II中的关系不同<50%的BS值<50%[4或这张纸(图。1).MicraIroideae是多噬细胞的,并且将这三种物种的所有BS值均低于Plastid Ml树。线粒体拓扑的深层节点基本上不统一塑料分析的那些,并且在此进一步使用深胶质节点的进一步分析。
发散日期估计
在两种校准情景下估计发散日期(表2).Crown Pacmad节点的估计发散日期为32.44 [11.9,50.6] Mya和32.74 [17.0,45.2] Mya分别设定一个和两个,表明在小组中使用有争议的植物植物对估计影响很小这个节点的年龄。第一个集合和20.46 [12.8,46.6] Mya的Aristidoideae散系Mya和第二组的20.46 [10.6,25.6] Mya是两者之间最可变的。在SET 2中,添加另一个校准点导致茎和冠状植物曲线曲线分歧,同时冠状微单,甘蓝醛,胰腺和茎氯化晶体谱系的发散日期增加(表2).组1的冠状花序辐散期为20.24 [7.9,36.8]mya,组2的冠状花序辐散期为23.61[8.2,36.8]mya,也值得注意。我们对圆锥花序科冠的估计日期与近期对Andropogoneae的分歧估计一致[21.].
讨论
在我们的分析中,Pacmad Clade的短暂的深枝与本集团早期的快速辐射符合。无论原因如何,Pacmad草中的深枝都很难解决。Pacmad Clade的全塑性塑造与数百个信息特征的塑料分析为理解本集团的深层分歧提供了明显的优势。全塑性分析和分歧时间的估算允许对该加速多样化的原因进行猜测。
Phylogenomic分析
植物谱系中快速辐射的系统发育分析往往是挑战性的,因为长的InGroup分支只能通过具有相对较少的系统发育信息的短深枝连接,这可能阻碍了深层关系的鲁棒解决[22.].当除了相对较长的分支时,它们可以在逐步吸引长的Ingroup分支并建议错误的关系。我们的ML和MP分析之间的冲突也暗示了长分支吸引力,这一限制甚至更容易受到影响[23.].
只有一个例外,每个ML分析产生了相同的INGROUP拓扑。长分支景点往往表明均质取代或可能的升高率升高,并且可能负责弱支持的结果ativa。与所有其他OutGroup组合相比,这是一个独特的例外。几个小组的分类群对茎aristidoideae的支持较少。除了与Pacmad草的相对密切的关系中选择了小组的分类群。Bambusa Oldhamii,Oryza Rufipogon, 和Rhynchoryza subulata对这个节点的支持更强大,而不是更恒定相关普利亚oloriformis。B. Oldhamii.呈现较短的终端分支长度r. subulata.和由[15.],这与阿里斯蒂亚科更大的支持度(80%)相关,介于这些值之间r. subulata.(99%)和由(76%)被选为小组。包含18个外群的含税导致添加更多的系统发育信息。虽然对胰岛素的姐妹关系的支持仍然小于与单一的小组分类(77%)的分析,但较大的小组允许对Ingroup拓扑的更大信心。
禾本科植物系统学的重要研究[4]为胰腺炎患者与CMAD CLADE(BS值:61%)以及微单/雄蛋白和氯化物/氯化物片材(BS值:51%)之间提供了弱胰岛素关系的弱者。虽然GPWG II的分类群采样包括452种PACMAD物种,但分析了每种600-800个碱基对(BP)的三种遗传标记。这里的系统核糖方法允许通过几个数量级来增加分子标记的数量,以提供额外的信息位点并提高系统发育的支持值。
在我们最良好支持的可能性拓扑中(图。1), Panicoideae与其他PACMAD草类是姊妹,Aristidoideae与CMAD枝是姊妹,但这一关系经SH检验与GPWG II拓扑没有统计学差异。区分这两种不同的拓扑结构的困难可能是由于PACMAD草类的快速辐射。所有两个或两个以上物种的亚科被恢复为单系。在panicoidae内,两个andropogonae (Coix Lacryma-Jobi和玉米)是姊妹,就像两个以前被认为是百齿目动物的物种一样(Thysanolaena Maxima和Centotheca lappacea)(BS值,100%)。因此,完全的塑料序列分析能够为系统发育关系提供更大的支持,并表明来自Pacmad Taxa的完全塑料的进一步取样可能有助于解决较低的分类水平的关系。由于GPWG II相比,该分析可能是由于适度的分类群体抽样而产生艺术组织[4]但大大改善了字符抽样。
线粒体数据的ML拓扑基本上与我们的整个塑性的系统发育基本上不一致(图。12)和GPWGII分析中的关系[4],尤其是最深节点。这可能是由于植物线粒体基因组中观察到的替代速率相对较低,或者它们与其他基因组/生物的碎片重新组合的倾向,可以将基因座与不同的进化历史联系起来[24.].通过中等载体检索一些亚属(胰腺炎,甘氨酸))。线粒体序列可以更适当地用作Pacmad草的亚壳内的系统发育工具。
发散估计
以前的研究已经开始使用许多类群来确定PACMAD草的分化日期,但分子标记的数量相对较少。本研究中分子数据的增加,由于存在更多的系统遗传信息位点,使得对深度节点的分歧日期的评估更加准确。最近估计BEP/PACMAD分支的年龄为54.9(±7.0)mya [9].这一估计表明,BEP和PACMAD演化支在古新世-始新世热最大值(PETM) (55-56 mya)和始新世过渡时期发生了分化。始新世的特征是一个冷却和干燥的时期,导致森林破碎,为开放的栖息地和森林边缘物种创造了新的或更广泛的栖息地[25.].
Pacmad Subfamilies的分歧,已估计在38 Mya之间陷入困境[12.]和45 mya [8],在32.4 [11.9,50.6](95% HPD下位和上位,分别)mya分析。根据这一较年轻的估计,PACMAD演化支的快速辐射发生在始新世-渐新世过渡时期。在始新世期间,随着南极冰川事件的发生,全球出现了降温趋势[26.以及大气中CO的减少2[12.].这些气候变化影响了全球各地的栖息地多样化,增加了EOT之后植物殖民的开放栖息地和森林边缘。
Aristidoideae几乎完全开放栖息地草,对亚家族的祖先条件最具典范的解释是它也是一个开放的栖息地谱系[2].如果是GPWG II的建议,Aristidoideae是其他Pacmad草的姐妹,那么开放栖息地的开采长期以来,在Pacmad Clade的辐射之前没有相应的解释假设[4].在整体草地区的背景下,如果姐妹组给其他PACMAD物种具有祖先栖息地,Pacmad栖息地的栖息地转变更加混乱地解释。注意,深深分歧的谱系普利奥基氏菌,Pharoideae和Anomochloideae专门在森林地板上发现[2].panicoidae包括出现在阴生生境、开放生境和混合生境的属[2].如果气候变化在EOT时促进了开放栖息地物种的辐射和多样化,那种亚家族妹妹种类的这种栖息地的栖息地将预期在Pacmad Clade的其余部分之间的多样性。这些后代的物种组成,这些后代被作为姐妹组在其余的Pacmad谱系中得到了解,预计将填补在当代胰腺炎中看到的宽容和开放的栖息地利基[4].值得注意的是,在我们的分析中,疯子姐姐帕尼西亚佩的其余部分是森林边际(Thysanolaena.)和宽容耐受性(Centotheca) 物种 [27.,这与Panicoideae最初占据森林和森林边缘,然后辐射,可能多次,进入开放栖息地的假设是一致的。
结论
通过使用系统基因组学方法,本文检索到的深层PACMAD关系得到了比以往更大的支持。我们的结果支持PACMAD拓扑,其中Panicoideae是ACMAD分支的姐妹,允许进一步探索终端关系。它也为快速研究辐射植物谱系提供了一般的系统基因组学方法。
Pacmad Clade的分歧估计提供了深入了解气候变化导致栖息地多样化的推定作用,这可能引发了这些草的快速辐射。初始辐射的32.4 myA的日期与EOT相关。冰川事件和整个农部整体的全球冷却趋势导致了通过森林碎片化和开放栖息地扩张的环境多元化。这些变化可能使草迅速调整,杂交,最终主导新开发的栖息地。
方法
超级选择
由于Aristidoideae和胰腺炎作为姐妹组给其他Pacmad谱系的姐妹组,超出了分类群选择和数量的采样构成了挑战。最初,选用高产稻(NC_022668)被选为除群,但对CMAD疏水的姐妹的支持是低(BS值76%)。另一个BEP代表,Bambusa Oldhamii.(NC_012927)作为外群,拓扑结构保持一致,但阿里斯蒂亚科茎节仍存在弱支撑(bs值80%)。多达17个具有代表性的BEP分类群,以及基级分类群(Anomochloa marantoidea,nc_014062;Pharus Latifolius.nc_021372;Puelia olyriformis,NC_023449)在九个子集中组合,以确定OffGroup选择对InGroup拓扑和支持的影响.Ingroup拓扑证明在这些分析中稳定,尽管对短深枝的支持是可变的。最终,单个小组种类Rhynchoryza subulata(NC_016718; EHRHARTOIDEAE)被发现产生结果与此处分析的最大的小群种类集的成果一致。
分类抽样
根据亚家族成员身份对类群进行抽样,以获得所有感兴趣的主要类群的代表性。Panicoideae分类群包括五个主要部落的代表,黍virgatum(Paniceae) (NC_015990),玉米(andropogoneae)(NC_001666),和Coleataenia prionitis(Paspaleae)。Centotheca lappacea(Centhotheceae)和Thysanolaena Maxima(Thysanolaeneae)也包括以前公认的CentoThecoideae [1,2,6), (7归类为胰腺炎。三个代表性的arundinoideae,Hakonechloa Macra,Monachather脉, 和芦苇南极光,包括了GPWG II检索到的Arundinoideae的三个主要分支[4].Arundinoid属的一个成员分类为Incertae Sedis.,Elytrophorus spicatus,也包括在试图解决这种arundinoid关系[28.].本文还分析了微线亚科的三个分类单元,为微线/苇状线分支提供了代表。micraira spiciformis.(micrairea),Eriachne Stipacea.(Eriachneae),isachne distichophylla.(以萨尼)被选为代表四个微型机器人部落中的三个。此外,还纳入了白头翁亚科的4个属,作为白头翁谱系的代表,以及aristtidoideae的代表种,aristida purpurea(aristideae).这是唯一发表过的类氯化物的质体,Neyraudia reynaudiana,也包含在我们的分析中[29.].
使用NGS方法测序的质体分析中包含的每个分类单元的线粒体序列数据从Illumina read文件中检索。从NCBI数据库中检索其他类群的序列。因此,我们的线粒体样本仅限于使用NGS测序的物种和Genbank中存在的物种。在这部分的研究中,分类群限于15种B. Oldhamii.作为外群分类单元。
DNA提取和测序
为每种兴趣物种获得叶组织样品(表中列出的来源3.)用硅胶干燥剂干燥。在植物组织的手动均质期间使用液氮来溶解细胞,以最大化DNA产率。在制造商的协议之后,使用DNeasy植物迷你套件(Qiagen,Valencia,CA)进行提取。
一个物种的质体,Hakonechloa Macra,利用草类特异性引物进行了扩增和桑格测序[30.[[31].仅测序IR(反转重复)的一个副本以及所有四个红外边界。[的替代方法32遵循扩增失败,包括设计和使用底漆定制h . macra(表4).使用巫师SV PCR清理系统(Promega,Madison,Wi,USA)来制备扩增用于测序。Sanger测序是在宏观原,首尔,韩国。使用葛益序列7.0.1(BioMatters,New Zealand)手动组装完整的塑料。未通过NCBI核苷酸数据库获取的其他12个塑料使用NGS测序。从三个DNA提取物中开始总基因组DNA(加利福尼亚丹顶虫,细鳞鞘翅虫,似是而非的蝴蝶)用纳米玻璃1000(Thermofisher Scientific,Wilmington,De,USA)测定,每次为1.5μg。将DNA稀释至2ng /μl并使用Bioruptor®Songator(Diabode,Denville,NJ,USA)剪成300个碱基对(BP)片段,在两个12分钟内,它们之间的反转。然后使用小烯醇萃取试剂盒(Qiagen Inc.,Valencia,Ca,USA)纯化DNA制剂并浓缩。根据制造商说明书(Illumina,San Diego,CA,USA),使用Truseq低通量协议(GEL方法)编写单个读取库。在爱荷华州立大学,美国阿梅省的Hiseq 2000仪器(Illumina,San Diego,CA)上进行了单端测序。Illumina读数为99英镑。
改进的NGS方法用于剩余的九种物种。将剩余的含税的总基因组DNA提取物稀释至20μl水中的2.5ng /μl。Nextera Illumina文库制备试剂盒(Illumina,San Diego,CA,USA)用于制备用于测序的文库和DNA清洁和浓缩器试剂盒(Zymo Research,Irvine,CA,USA)用于图书馆样品纯化。在IOWA州立大学测序设施中使用Hiseq 2000仪器进行单端测序,如上所述。
读取是使用SolexaQA软件包的DyandicTRIM V2.1过滤的第一个质量[33]默认设置,然后在同一包装中使用LorightSort V2.1删除少于25bp的序列(默认设置)。然后使用FASTQC V0.10.1评估读取的质量(www.bioinformatics.babraham.ac.uk/projects/fastqc/).使用完整的质量修剪的读取集进行装配。
塑料组装和注释
完全进行塑料组件新创方法。天鹅绒软件包[34]是按照先前建立的方法迭代运行的[26.将读取的数据组合成连续序列(contigs)。使用锚定保守区域扩展(ACRE)方法对Contigs进行支架搭建[29.].利用草科范围内的质体比对确定了保守区域。利用contigs或通过定位重叠区域来解决质体中的任何剩余缺口,直到圆形图谱完成。组装的质体在geneipro中进行注释,方法是将它们对准一个密切相关的、之前注释过的参考质体,当注释的相似性达到70%或以上时,将注释从参考质体转移到组装的质体。然后对每个编码序列进行检查,并进行必要的调整,以保存内含子边界、阅读框、终止密码子或识别伪基因。红外线探测边界的定位方法为[13.].
塑料分析
使用MAFFT插件在genepro中对完全组装的质体进行对齐[35].除去一个或多个序列中引入间隙的特征以减少模糊同源性的区域,除去了一种红外序列以减少重复序列的夸张。在IR和间隙去除后,这种对准的长度为96,493bp。使用XsEDE上的raxml-hpc2进行m1分析[36]并使用CIPRES Science Gateway访问[37].获得了最高可能性的树。在GTR模型下使用默认参数,估计每站点25个伽玛率类别和所有其他免费型号参数。使用1,000个假序进行快速引导分析。指定的小组包括18个BEP分类群(附加档案1:图。S1)。相同的方法用于除数分类群的其他组合。使用Phylip软件包的满足功能生成共识引导树[38)(图。1).使用FigTree v1.4.0对树文件进行可视化和编辑。[39].使用独立的外群分类群进行了其他几个可能性分析,Puelia oloriformis.,Bambusa Oldhamii., 和选用高产稻,但这些分析提供了茎aristidoideae的低自举支持(BS)值。sh test [40进行以确定在此获得的ML拓扑是否从GPWG II假设中显着不同。使用PAUP * V4.0B10进行SH测试[41],以确定Aristidoideae作为其他PACMAD亚科姐妹群的拓扑结构[4可以被这里分析的数据拒绝。
使用MRBAYES 3.2.2在XSEDE上进行BI分析[42],通过CIPRES科学网关访问。马尔可夫链蒙特卡罗(MCMC)分析在每10,000,000代运行两次。这是运行了两次,有500万代的老化。分析一直运行到完成,分裂频率的平均标准偏差< 0.00001。
使用PAUP * V4.0B10进行分支和绑定的最大定期分析(MP)分析[41].采用MP自举法分析1000个伪重复,每个伪重复10个随机添加序列。
将每个完整质体中的76个蛋白编码序列串联进行分区分析。用与全质体分析相同的方法对分块的基质进行分析。
线粒体分析
线粒体序列由相同的NGS读取文件组装并使用与塑料分析相同的公用事业的似然框架分析。这马基因序列的选择是基于系统发育分析中先前的应用[18.].六个相对可变的内含子序列[19.]也被选中:nad1基因内区2,nad4内含子1,和nad7内含子1 2 3 4。比对时,每个内含子序列的配对一致性小于99%5).来自的线粒体序列玉米和Bambusa Oldhamii.直接从GenBank获得。对NGS库中包含的每个物种进行参考mapping分析,将读文件映射到感兴趣的完整线粒体序列中的每个编码序列玉米.检查来自每个参考映射的共有序列,用于缺少核苷酸位点和适用的帧突变突变,以检测错误的组件。相应的马和内含子序列数据(表5)被组装、连接和排列为每个物种。与以前一样,对这些数据进行ML分析,但与B. Oldhamii.作为超组,生成ml树(图。2).
发散估计
利用野兽V2.1.2估计分歧日期[43],参数在beauty中设置。估计了不同校正组合和不同先验分布下的初步发散日期。对PACMAD草类的校准有用的化石很少,而且日期有时是矛盾的。本文选取了两种差异分析,以显示估计日期的最大范围。两种估算都给出了两种不同的种子价值和1000万的链长,每一种都有2000万代。先验设置限制关系,以保存由ML质体分析生成的拓扑Rhynchoryza subulata外围集团。所采用的替代模型为GTR + G + I在不相关松弛log-normal时钟下。对于每一个都选择了对数正态分布。Parametersolver v3.0 [44用于计算这些日志正常的先前分布的平均值和方差。每次运行校准的化石估计不同,以评估潜在争议的化石在超组中的效果。共使用六种化石来校准分析。使用14 mya的茎氯离样节点的下限评估第一次运行,其校准用嵌入作为氯微藻的成员的化石,由于气孔辅细胞和硅胶体的形状[45].然而,关于这个化石的分类物理到属的分类标识存在一些辩论Distichlis进一步的显微解剖评估[46].然而,我们在这里使用这种化石是皇冠的校准点。使用氯化物植物植物化石溶液将19μA的上限置于干氯微藻节点[47].核心胰腺炎(泛岩)[28.]也限制了第一次运行,使用七个mya的下限奥特拉里亚化石和八个mya的上限,其中化石分配给Dichanthelium.sp。[48].使用与第一次运行相同的校准进行第二野兽运行,但是在65 mya的冠BEP / PACMAD节点上添加下限,使用Oryzeae Phytolith Fossils的67 mya的上限[10.].使用植物岩作为估算禾本科植物分化时间的校准是有争议的[49].比较有和没有植物岩的两组分析,可以评估这种校准对差异估计的影响。这种校准策略包括了可获得的PACMAD草类的分类鉴定化石。它还评估了外群使用米草科植物岩和相关角质层化石的效果,这些化石已被证明产生了与其他数据不兼容的日期估计[9].从野兽分析产生的树和日志文件与LogCombiner v2.1.2组合,并使用示踪剂v1.6评估的收敛性[50].Figtree v1.4.2 [39用于查看和编辑为每次野兽生成的组合树文件,刻录为20%。给出了与Pacmad草的深层关系相关的节点的分歧估计(表2).
提供支持数据的可用性
支持本文结果的数据集可在TreeBase存储库中获得,http://purl.org/phylo/treebase/phylows/study/TB2:S17812.将所有核苷酸序列沉积在NCBIGegank储存库中。登录号码可以在表中找到3..
缩写
- 英亩:
-
锚定节约区延期
- 野兽:
-
贝叶斯进化分析采样树木
- Beauti:
-
贝叶斯进化分析效用
- cep:
-
Bambusoideae Ehrhartoideae poo eae
- Cipres:
-
系统发育研究的网络基础设施
- CMAD:
-
Chloridoideae micrairoideae arundinoideae danthonioideae
- DC:
-
Danthonioideae氯多水陶膜
- 测试结束:
-
藿香 - 寡烯转型
- GPWG(ii):
-
草地发育工作组(II)
- gtr + g + i:
-
一般时间可逆加上伽玛分配加上不变网站的比例
- 红外光谱:
-
倒置重复
- LSC:
-
长单副本
- 马:
-
Micrairoideae Arundinoideae
- Mafft:
-
使用快速傅里叶变换多次对齐
- 密度:
-
马尔可夫链蒙特卡洛
- ML:
-
最大似然
- MP:
-
最大限定
- ngs:
-
新一代测序
- Paup *:
-
使用Parsimony *等方法进行系统发育分析
- PACC:
-
板栗亚科、芦笋亚科、绿豆亚科、蜈蚣亚科
- PACCAD:
-
青藤亚科芦笋亚科绿藤亚科芦笋亚科石竹亚科
- PACCMAD:
-
青藤亚科、芦笋亚科、绿苔亚科、蜈蚣亚科、小鞭亚科、芦笋亚科
- Pacmad:
-
青藤亚科、青藤亚科、绿藤亚科、小藤亚科、青藤亚科
- PETM:
-
Paleocene-Eocene热最大
- SSC:
-
短单副本
参考
- 1。
张伟,张文德。草科(禾本科)的系统发育ndhF序列数据。SYST BOT。1995年; 436-460。
- 2。
草地下发育工作组I,Barker NP,Clark Lg,Davis Ji,Duvall Mr,Guala GF等。地发育和草地(Poaceae)的亚细胞分类。安莫僵尸加德。2001; 88:373-457。
- 3.
Duvall Mr,Davis JD,Noll JD,Goldman Dh,Sánchez-ken JG。重新审视草(Poaceae)的系统发育。Aliso:系统和进化植物学的杂志。2007; 23(1):237-47。
- 4.
草地下发育工作组II,Aliscioni S,Bell HL,Besnard G,Christin Pa,Columbus JT等。新的草地系统解析了深入的进化关系并发现了c4起源。新植物。2012; 193:304-12。DOI:10.1111 / J.1469-8137.2011.03972.x.
- 5.
Saarela JM,Graham Sw。基于体积基因组的中学尺度示例的基于中学样的基于中的基于中学尺度的基于族级的基于模拟的基于体积级的血管发育关系的推理。植物学。2010; 88(1):65-84。
- 6。
Sánchez-Ken JG, Clark LG。CentoToideae +胰腺发生的系统发育关系基于ndhF和rpl16.内含子序列和结构数据。Aliso:系统和进化植物学的杂志。2007; 23(1):487-502。
- 7。
Sánchez-Ken JG, Clark LG。基于质体和核序列数据和结构数据,建立了板栗科植物的系统发育和新的部落分类。acta botanica sinica(云南植物学报),2012;
- 8。
Bouchenak-Khelladi Y,Verboom Ga,Savolainen V,Hodkinson Tr。草地的生物地理造影(POACEAE):一种揭示地理空间和地质时间进化史的系统发育方法。Bot J Linnean Soc。2010; 162(4):543-57。
- 9。
Christin Pa,Spriggs E,Osborne CP,Stromberg Ca,Salamin N,Edwards EJ。分子约会,进化率和草的年龄。系统中的生物学。2013; 63(2):153-65。SYT072。
- 10。
Prasad V,Steromberg Ca,Leache Ad,Samant B,Patnaik R,Tang L等。稻米部落的后期白垩纪起源为淘汰淘汰的初期多样化提供了证据。NAT Communce。2011; 2:480。
- 11.
Vicentini A,Barber Jc,Aliscioni SS,Giussani LM,Kellogg EA。草的年龄和c的簇4光合作用。中国科学(d辑:地球科学)2008,14(12):2963-77。
- 12.
作者单位:国家自然科学基金青年科学基金,国家自然科学基金青年科学基金。渐新世有限公司2下降促进了C.4草丛中的光合作用。目前的BIOL。2008; 18(1):37-43。
- 13。
Burke SV,Grannan CP,Duvall Mr。两个新世界竹子的塑料序列 -arundinaria gigantea和Cryptochloa strictiflora.(POACEAE) - 延长对Bambusoideae的文学理解。我是J机器人。2012; 99(12):1951-61。DOI:10.3732 / AJB.1200365.
- 14.
从禾本科的系统发育学、分子进化和谱系年龄的估计。acta botanica sinica(云南植物学报). 2014;40(6):1421 - 1436。
- 15.
吴ZQ,GE S.草地上的BEP思工的系统发育重新判断:来自叶绿体的全基因组序列的证据。mol phylocyet evol。2012; 62(1):573-8。
- 16。
纽约州米德尔R.仙人掌最接近的亲属:叶绿体和线粒体序列的洞察力,特别强调部落Anacampseroteae的关系。我是J机器人。2007; 94(1):89-101。
- 17。
沙琳,邢峰,张海清,康海英,王勇,王小林,等。羊草(小麦科)的系统发育关系来自叶绿体trnH‐psbA和线粒体coxII内含子序列的证据。地球科学进展。2014;52(6):722-34。
- 18。
Clifton SW,Minx P,Fauron Cm,Gibson M,Allen Jo,Sun H等人。玉米Nb线粒体基因组的序列与对比分析。植物理性。2004; 136(3):3486-503。
- 19。
郭玉玲,葛胜。基于叶绿体、线粒体和核基因组DNA序列的水稻科分子系统发育研究。植物学报。2005;92(9):1548-58。
- 20。
Freudenstein JV,Chase MW。线粒体分析nad1orchidaceae中的B-C内含子序列:效用和长度变化字符的编码。SYST BOT。2001; 26(3):643-57。
- 21。
Estep MC, McKain MR, Diaz DV, Zhong J, Hodge JG, Hodkinson TR, et al.;异源多倍体、多样化和中新世草原扩张。《美国国家科学院学报》上。2014年,111年(42):15149 - 54。
- 22。
Rothfels CJ,Larsson A,Kuo Ly,Korall P,Chiou WL,Pryer Km。克服深根,快速率和短的间,可以解决Eupolypod II蕨类植物的古老快速辐射。系统中的生物学。2012; 61(3):490-509。
- 23。
斯沃福德DL, Waddell PJ, Huelsenbeck JP, Foster PG, Lewis PO, Rogers JS。系统发育估计的偏倚及其与简约和似然方法选择的相关性。系统医学杂志。2001;(4):525 - 39。
- 24。
两种杂交针叶树接触带的线粒体基因组重组。遗传学。2005;171(4):1951 - 62。
- 25。
贝洛西,克劳斯。中始新世全球变冷和巴塔哥尼亚中部开阔植被栖息地扩张的开始。安第斯地质师。2013;41(1):29-48。
- 26.
王志强,王志强,王志强,等。新生代海洋底栖生物δ18O记录的时间序列分析。“牧师。2014;52(3):333 - 74。
- 27.
Watson L,Dallwitz MJ:1992年。世界的草属:描述,插图,识别和信息检索;包括同义词,形态,解剖学,生理学,植物化学,细胞学,分类,病原体,世界和地方分布以及参考。版本:2014年8月12日。http://delta-intey.com.
- 28。
中国禾本科(Gramineae)科的系统发育分类。Tropicos》2014。等。http://www.tropicos.org/Project/CNWG访问:2015-01-07。
- 29。
Wysocki WP, Clark LG, Kelchner SA, Burke SV, Pires JC, Edger PP,等。草中短读全质体序列组装方法的多步骤比较。分类单元。2014;63(4):899 - 910。
- 30.
莱斯伯格CH,Duvall Mr。完全叶绿体基因组Coix Lacryma-Jobi谷物中质体的比较分子进化分析。中国科学(d辑:地球科学)2009;
- 31.
Dhingra, Folta KM。ASAP:质体的扩增、测序和注释。BMC基因组学。2005;6(1):176。
- 32。
Morris LM, Duvall先生的叶绿体基因组Anomochloa marantoidea(Anomochloideae; Poaceae)包括草状和独特的特征的混合物。我是J机器人。2010; 97(4):620-7。
- 33。
Cox MP,Peterson Da,Biggs PJ。SOLEXAQA:IL-A-GLANCAL METILININ第二代排序数据的质量评估。BMC生物信息学。2010; 11(1):485。
- 34。
Zerbino Dr,Birney E. Velvet:De Bruijn图表的De Novo短读组装的算法。Genome Res。2008; 18(5):821-9。
- 35。
Katoh K,Kuma Ki,Toh H,Miyata T. Mafft版本5:提高多序列对齐的准确性。核酸RES。2005; 33(2):511-8。DOI:10.1093 / nar / gki198.
- 36。
Stamatakis A. Raxml-VI-HPC:最大基于似然的系统发育分析,有数千个分类群和混合模型。生物信息学。2006; 22(21):2688-90。
- 37。
Miller Ma,Pfeiffer W,Schwartz T.创建Cipres科学门户,了解大型系统发育树的推断。GCE。2010; 1-8。
- 38.
系统发育推理软件包。西雅图:华盛顿大学基因组科学系;2005.
- 39.
Rambaut A: FigTree v1.4.2。2014.可用http://tree.bio.ed.ac.uk/software/figtree/
- 40.
Shimodaira H,Haegawa M.对系统发育推理的应用对数值的多重比较。mol Biol Evol。1999; 16:1114-6。
- 41.
Swofford DL。Paup *。使用规定(*和其他方法)的系统发育分析。版本4. Sinauer Associates。马萨诸塞州,美国:桑德兰;2003年。
- 42.
Ronquist F, Teslenko M, van der Mark P, Ayres DL, Darling A, Höhna S, et al.;MrBayes 3.2:跨大模型空间的高效贝叶斯系统发育推理和模型选择。系统医学杂志。2012;61(3):539 - 42。
- 43.
Bouckaert R,Heled J,Kühnertd,沃恩T,Wu Ch,谢D等人。野兽2:贝叶斯进化分析的软件平台。PLOS计算BIOL。2014; 10(4),E1003537。DOI:10.1371 / journal.pcbi.1003537..
- 44。
库克j,wathen kj,nguyen h:partagesolver v3.0。2013年,可从https://biostatistics.mdanderson.org/SoftwareDownload/SingleSoftware.aspx?Software_Id=6
- 45。
Dugas DP,Retallack GJ。从德尔瑙,肯尼亚堡垒的中间内肾化石草。J Paleontol。1993年; 67:113-28。
- 46。
贝尔HL,哥伦布JT。建议扩大Distichlis(POACEAE,氯磷酸):来自分子,形态学和解剖结构的支持。SYST BOT。2008; 33(3):536-51。
- 47。
Strömberg加利福尼亚州。北美新生代露天栖息草脱耦分类辐射与生态扩张。Proc Natl Acad Sci U S A. 2005; 102(34):11980-4。
- 48。
Thomasson JR。关于晚年血统草地的引理特征的观察黍线虫.Am J Bot. 1978; 65:34-9。
- 49。
Ille Wd,Smith Sy,Gandolfo Ma,Graham SW。单子叶化石适用于分子约会分析。Bot J Linn Soc。2015; 178(3):346-74。
- 50。
rambaut a,suchar ma,谢d,drummond aj:追踪v1.6。2014.可用http://beast.bio.ed.ac.uk/tracer.
确认
我们感谢A. Murrell,K. Zeno,以及C.Jäeger的技术援助和Y. Yin,允许获得计算资源。我们还感谢L. Attigala向ISU DNA设施提交订单。感谢那些在本研究中提供样品的人感谢;J. Saarela,G. Sanchez-Ken,O. Morrone(1957-2011),T.Gallaher,S. Jacobs(1946-2009),C.Morden和N. Barker。这项工作是由伊利诺伊州北部大学生物科学系的植物分子生物学中心和国家科学基金会,授予LGC,DEB-1120856至LGC,DEB-1120761至MRD。在本材料中表达的任何意见,调查结果和结论以及提交人的建议,并不一定反映国家科学基金会的意见。
作者信息
隶属关系
通讯作者
额外的信息
相互竞争的利益
提交人声明在此处提供的工作方面没有竞争利益。
作者的贡献
MRD对研究的设计、手稿的起草和促进合著者之间的互动做出了贡献。JLC进行了数据采集和分析,并起草了手稿。WPW协助起草稿件、数据分析、序列组装。CJP、PPE、DMJ协助TruSeq库的准备工作。LGC和SAK也对项目的设计和一般方法做出了贡献。所有作者阅读并贡献了最终手稿的书面部分。
Joseph L. Cotton, William P. Wysocki和Melvin R. Duvall对这项工作做出了同样的贡献。
附加文件
附加文件1:
概述外谷组选择对Pacmad Clade内的亚组合关系的影响。每一个估计所包括的外群都显示了与其余PACMAD分支的姐妹亚科。此外,还包含了对这种姐妹关系的ML引导支持。图S1。系统基因组学分析所选择的外群分类单元子集,包括外群种的总数和身份。显示了联合所有类群姐妹到最深处分叉枝,即Panicoideae或Aristidoideae的Bootstrap值。以上给出了禾本科植物系统发育关系的概化树。
权利和权限
这是一篇基于知识共享署名许可协议(http://creativecommons.org/licenses/by/4.0.),允许在任何媒介上不受限制地使用、分发和复制,但须注明原作的出处。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
棉花,J.L.,Wysocki,W.P.,Clark,L.G.等等。解决Pacmad草的深层关系:一种系统染色方法。BMC植物杂志15日,178(2015)。https://doi.org/10.1186/s12870-015-0563-9
已收到:
接受:
发表:
关键词
- 完整的塑料
- 发散估计
- PACMAD进化枝
- 胰腺炎
- 系统核糖学
- 快速辐射