摘要
背景
温度变化的感知和转导导致生长的改变,使植物适应环境温度的升高。虽然植物色素相互作用因子4 (PIF4)已被确定为一个主要的环境温度信号中枢,但其上游调控似乎很复杂,而且人们对其知之甚少。在这里,我们利用自然变化的热响应生长拟南芥采用数量性状位点(QTL)分析。
结果
我们确定了GIRAFFE2.1这个QTL解释了Bay-0 × Sha重组自交系群体中约18%的温度诱导下胚轴伸长表型变异。转基因互补证明了昼夜节律调节因子的等位基因变异早期FLOWERING3(ELF3)是这个QTL的基础。变异的来源可以分配到单核苷酸多态性ELF3编码区,导致的差异表达PIF4和它的靶基因,很可能导致观察到的热响应性生长的自然变化。
结论
结合最近的其他研究,本工作确定了ELF3在环境温度信号网络中的作用。elf3介导的门控的自然变异PIF4夜间生长期间的表达似乎受到编码序列数量性状核苷酸的影响,该序列在某些环境中具有选择优势。此外,天然的ELF3等位基因似乎可以不同地整合温度和光周期信息来诱导建筑变化。因此,ELF3在应对各种环境因素时作为生长和发育的重要协调者而出现,并意味着ELF3是适应的重要目标。
背景
与光形态发生类似,术语thermomorphogenesis描述温度对形态发生的影响[1]。下胚轴伸长[2]和叶片发育不全[3.]属于植物发育过程中最敏感的产热变化。从生理上讲,这些协调的反应可能会增强叶片的蒸发冷却[4,5从而使植物能够适应温暖。在全球环境温度升高的背景下,提高我们对植物对这种环境扰动作出反应的基本过程的理解是势在必行的。
环境温度信号网络中的一个主要枢纽是基本螺旋-环-螺旋(bHLH)转录因子PHYTOCHROME-INTERACTING FACTOR4 (PIF4)。PIF4蛋白结合生长素生物合成启动子及应答基因[j]6- - - - - -9]。因此,它转录激活生长素反应,导致伸长生长。PIF4其本身似乎受bZIP转录因子伸长下胚轴l5 (HY5)以温度依赖的方式进行转录调节[10]。从光信号、光形态发生和生物钟等方面积累关于PIF4调控的数据[11- - - - - -13表明PIF4活性在几个水平上有更复杂的调控。
本研究的目的是利用自然变异的基因库中的拟南芥确定植物用来适应环境温度变化的复杂信号网络的其他组成部分。基于定量遗传方法,我们在此展示了两个自然发生的等位基因早期FLOWERING3(ELF3)在热形态发生中引起不同的反应。
结果
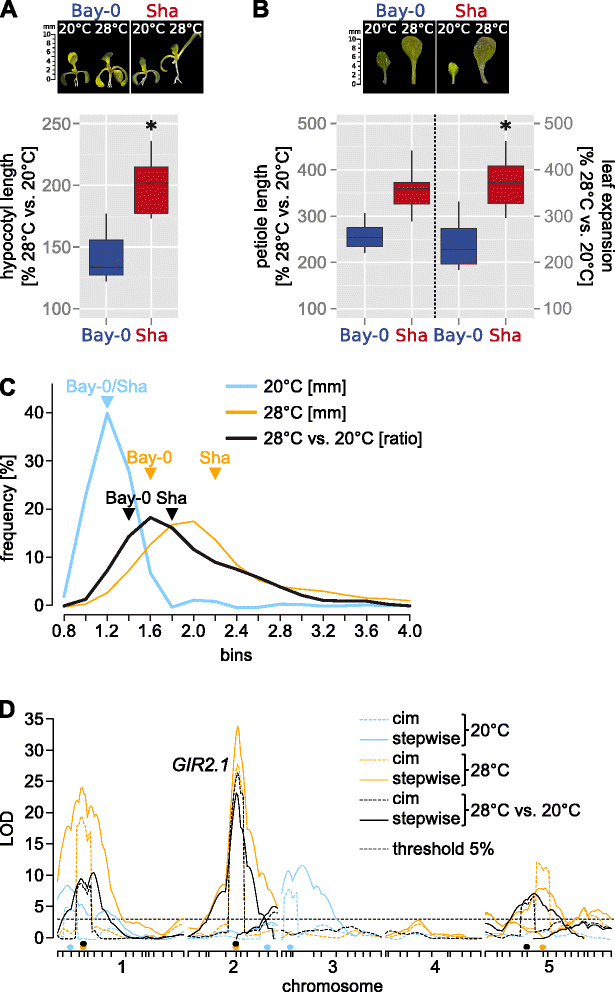
我们以前观察到广泛的自然变化的热形态特征表型,我们称之为温度诱导下胚轴伸长(TIHE;[10,14])。为了确定潜在的遗传变异,我们基于两个地理位置较远的自然加入进行了数量性状位点(QTL)分析。在比较20°C和28°C下的生长情况时,所选材料Bay-0(德国)和Sha(塔吉克斯坦)在几个产热响应方面存在显著差异(图2)。1,b;额外的文件1).识别潜在的遗传变异将有助于提高我们对环境温度变化如何转化为生长反应的理解。
植物温度诱导生长响应的数量性状位点分析拟南芥.一个相对下胚轴长度(28°C/20°C, %;n= 15) 10天大答:芥加入Bay-0和Sha。b12日龄幼苗的相对叶柄长度和叶片膨大。a、b箱形图显示了中位数(水平线)、四分位数范围(IQR,方框)和数据范围(晶须)。黑点表示异常值(定义为> 1.5 × IQR)。星号表示温度反应的显著差异(P< 0.05),通过双因素方差分析(即基因型x治疗效果)对附加文件中提供的绝对数据进行评估1.cBay-0 x sha衍生重组自交系群体在20°C下生长10日龄幼苗下胚轴长度的表型分类频率图(n= 400)或28°C (n= 395),对于28°C与20°C的比值(n= 387)表示。表明亲本表型等级是为了说明种群内的越界效应。d从复合区间映射(cim)和多个QTL映射(stepwiseqtl)得到的LOD分数(y轴)被绘制在所有染色体上(x轴)。x轴上的标记对应于基因图谱中的分子标记。x轴上的彩色点表示cim的协变量集。阈值基于1000种排列,alpha值为0.05。28°C vs. 20°CGIR2.1QTL是本研究的主题
温度诱导下胚轴伸长的QTL分析
我们对Bay-0 x Sha重组自交系群体进行表型分析[15]以获取TIHE的响应。我们在不同的环境温度(10天20°C vs 10天28°C)下进行了长日周期的幼苗生长。利用R/ QTL对温度单独下胚轴长度或28°C/20°C下胚轴长度之比(28°C/20°C)进行基于复合区间和多QTL定位的QTL分析。我们总共鉴定出14个不同的qtl(图2)。1 c,d、附加文件2和3.).关注高低温(28°C/20°C比)生长差异,鉴定出5个qtl,命名GIRAFFE1/2.1/2.2/5.1/5.2(GIR1,GIR2.1, GIR2.2,GIR5.1, GIR5.2),根据他们各自的染色体位置。这5个qtl加起来解释了作图群体中约43%的表型变异。最强QTL,GIR2.1(LOD评分为23,stepwiseqtl程序),解释了~ 18%的这种变异(附加文件2和3.),表明Bay-0和Sha之间的很大一部分自然变异可归因于该基因座。
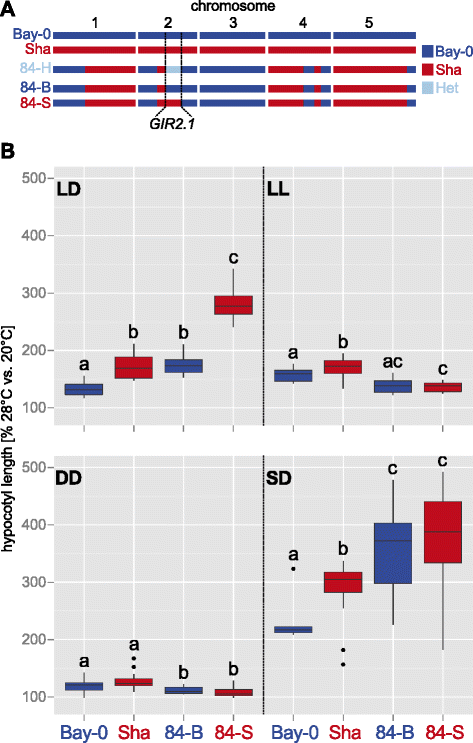
以方便基于地图的克隆GIR2.1,我们首先用异种近交家族(hif;[16];无花果。2、附加文件4).在目标区域携带亲本等位基因的两个HIF系之间的表型差异,虽然在其他方面遗传相同,可归因于目标QTL区间的遗传变异。除了…之外GIR2.1,我们还包括GIR1和GIR5.1在这个分析中。我们无法验证GIR5.1但在分离两个亲本等位基因的HIF系中,观察到TIHE有显著差异GIR1(194-B和194-S)和GIR2.1(84-B和84-S)。2 b、附加文件4和5).由于对表型变异的高影响,进一步的分析集中在GIR2.1.在这里,Sha等位基因赋予长下胚轴,而Bay-0等位基因赋予短下胚轴。2 b;有趣的是,这种情况正好相反GIR1(额外的文件4])。我们发现两者之间的差异GIR2.1在连续光照、黑暗或短日照条件下,HIF细胞系不能持续存在(图2)。2 b、附加文件5).因此,昼夜循环与广泛的光相似乎是必要的自然变化引起的TIHEGIR2.1.此外,双亲在单色光下的差异似乎不依赖于GIR2.1(附加文件5).
GIR2.1验证和光周期特异性。一个分离bay0和Sha等位基因的异种近交系(HIF) 84的单倍型研究GIR2.1时间间隔。b箱形图显示下胚轴的相对长度(28°C/20°C, %;n在长日照(LD)、连续光照(LL)、黑暗光照(DD)或短日照(SD)光周期下生长8 d的幼苗中,> 15)。水平条形图、框状图和须状图分别表示中位数、四分位数范围和数据范围。黑点表示异常值(定义为> 1.5 × IQR)。不同字母表示通过双向方差分析(即基因型x处理效果)评估的温度反应(在一个光周期内)的统计差异。P< 0.05)的绝对数据5
GIR2.1可能是一个幽灵QTL
由84-B与84-S杂交得到的F1表明,Sha等位基因遗传的长下胚轴表型比Bay-0短下胚轴表型显性(附加文件)5).在精细映射的过程中GIR2.1在28℃条件下,对长下胚轴(大于84-B)的F2和F3重组体进行基因分型,发现长下胚轴表型可归因于不同的、不重叠的Sha区间GIR2.1地区。这表明,确切的定位GIR2.1LOD评分峰值可能由两个或多个贡献位点引起。这种现象在QTL分析中经常观察到,被称为鬼QTL (17]。使用其他协变量重新计算QTL分析,将该峰值分离为两个相邻峰,从而支持该场景(附加文件)6).有趣的是,Jimenez-Gomezet al。[18[j]在同一海湾-0 ×沙种群中报道了该地区遮荫躲避反应的类似现象,这反映了高温反应。然而,由于我们只可重复地观察到长下胚轴在两个Sha间隔(chr)中的一个。2: 9,199,751-10,426,485 bp),我们将后续分析重点放在该稳健区间上。
的单核苷酸多态性早期FLOWERING3隐藏在GIR2.1QTL
了解昼夜光周期是Bay-0和Sha之间TIHE差异的先决条件。2 b),我们确定早期FLOWERING3(ELF3)作为位于GIR2.1目标区间。ELF3是生物钟的一个组成部分[19在夜间发挥抑制生长的作用[20.]并且先前已被证明可以调节下胚轴伸长以应对遮荫[18]。使用转基因方法,答案et al。[11最近的研究表明ELF3Bay-0和ELF3沙等位基因以不同的方式调节生物钟的周期长度。
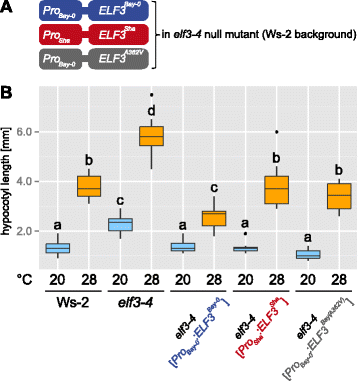
与ELF3在热形态发生中的作用一致,我们发现elf3-4与Ws-2野生型相比,零突变体具有较长的下胚轴(图2)。3.).为了研究Bay-0和Sha HIF系之间的TIHE差异是否确实归因于ELF3,我们采用了转基因互补方法。我们利用了answer产生的同样的转基因品系et al。[11],其中包含ELF3Bay-0或者是ELF3沙等位基因elf3-4突变体基因组(图2)3),这使我们能够在一个独立的环境中研究等位基因特异性的TIHE效应elf3功能丧失的背景。数字3 b结果表明,携带两种亲本等位基因的转基因株系与基因的互补elf3-4在20°C时表型。然而,在28°C时,转基因携带ELF3Bay-0(elf3-4[箴Bay-0:ELF3Bay-0[3])对下胚轴伸长的抑制显著高于携带ELF3沙等位基因(elf3-4[箴沙:ELF3沙])。这表明等位基因变异在ELF3影响xsha湾人群的TIHE。
Bay-0和ShaELF3变体显示几个启动子多态性[11]并且不同于(i)在一个非同义SNP上导致362位置的氨基酸变化,编码丙氨酸到缬氨酸的转变(A362V),以及(ii) c端谷氨酰胺延伸的长度[21,22]。尽管这可能取决于遗传背景[23),日本田岛et al。[22提示聚谷氨酰胺区长度与下胚轴伸长之间无明显相关性。因此,我们将重点放在A362V多态性上,并研究其在赋予温度形态发生等位基因差异中的潜在作用。再一次,我们在elf3-4由answer产生的突变背景et al。[11]。我们对其进行了转基因检验elf3-4行中包含箴Bay-0:ELF3Bay-0等位基因箴Bay-0:ELF3湾(A362V)等位基因,仅在A362V SNP上不同[11]。我们发现,在362位携带缬氨酸的转基因株系(如沙)中,温度诱导的下胚轴与沙的下胚轴相似ELF3沙重要的是,它比在相同位置具有丙氨酸对偶体的等位基因明显更长(图2)。3 b).从理论上讲,不同物种在热形态发生方面的差异elf3-4[箴沙:ELF3沙),elf3-4[箴湾:ELF3湾也可能受到昼夜节律或其他生理差异的影响ELF3表达式。之间观察到的显著差异elf3-4植物表达箴湾:ELF3湾和箴Bay-0:ELF3湾(A362V),然而,我们证明了A362V背后的SNP在ELF3导致TIHE表型变异,并确定ELF3是热形态发生的负调节因子。
差异转录反应引起的自然ELF3变体
最近有研究表明,由ELF3、ELF4和LUX组成的生物钟夜间复合体是下胚轴生长昼夜节律门控的分子基础,它直接下调了ELF3、ELF4和LUX的表达PIF4在傍晚[20.]。因此,在昼夜周期下,下胚轴伸长在黎明达到峰值。因此,我们测试了采用该模型的环境温度信号的假设,并研究了是否PIF4介导细胞伸长的pif4调控基因的表达和转录水平可能也受到温度升高的影响。
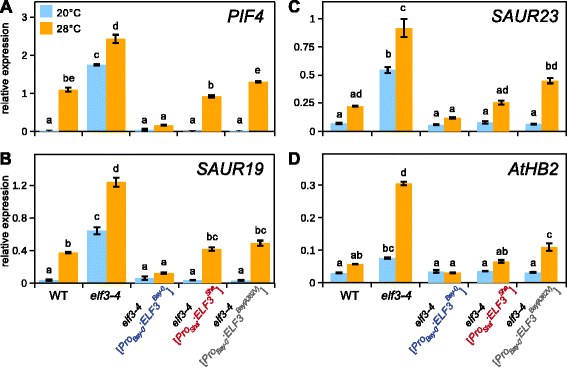
为了评估温度响应性,我们将幼苗在20°C的长日光周期下生长7天,将对照板保持在20°C,并将剩余的幼苗在关灯时转移到28°C (t = 16)。对照组和28°C的幼苗在移栽4小时后收获。我们发现损失了ELF3在elf3-4导致PIF4两种温度下的转录物水平(图2)。4).在20°C时,我们观察到elf3-4有一个转基因等位基因(箴Bay-0:ELF3Bay-0;箴沙:ELF3沙;箴Bay-0:ELF3湾(A362V))恢复野生型PIF4水平(无花果。4),证明了这些结构的功能性,但表明在20°C时不存在等位基因差异。这一观察结果与20°C下胚轴长度相似的三个系一致(图2)。3 b).然而,在28℃时,PIF4的表达水平elf3-4[箴沙:ELF3沙),elf3-4[箴Bay-0:ELF3湾(A362V)的比例明显高于elf3-4[箴Bay-0:ELF3Bay-0(图。4),再次反映了下胚轴表型(图2)。3 b).重要的是,这种表达行为在20°C和28°C下解释了GIR2.1QTL(=28°C/20°C比率),28°C和20°C时缺失QTL(图2)。1 d、附加文件3.).总之,这表明观察到的TIHE的自然变化可归因于温度依赖性的差异PIF4A362V SNP引起的表达水平ELF3沙.
的影响ELF3表达的等位基因变异PIF4生长素反应基因。qRT-PCR分析一个)PIF4,(b)SAUR19,(c)SAUR23,和(d)AtHB2在WT (Ws-2)和转基因互补系中的表达(见图2)。3).幼苗在20℃下生长7天,然后转移到28℃或20℃保存(对照)。幼苗在8小时黑暗期中间的4小时后收获。每个处理的三个生物重复的相对表达水平评估At1g13320作为控制基因。条形图显示均值和扫描电镜。不同字母表示样本之间的统计差异,经单因素方差分析和Tukey HSD检验(P< 0.05)
然后,我们将这种情况与负责细胞伸长的PIF4靶基因水平联系起来。参与细胞伸长的基因,如小生长素上调RNA(阿富汗二月)家庭或拟南芥同源盒蛋白2(AtHB2)已被证明以温度依赖的方式被PIF4激活[6,24,25]。无花果。4 b- - - - - -d结果表明,我们发现温度诱导的被测基因的表达水平elf3-4[箴Bay-0:ELF3湾(A362V)]明显高于elf3-4[箴Bay-0:ELF3Bay-0]。这强烈提示A362V SNP引起温度诱导的自然变异PIF4表达水平直接导致生长素介导的细胞伸长的改变。
讨论
我们在这里的发现说明了自然变异方法和支持的力量ELF3作为环境温度信号的负调节因子。生理和基因表达数据表明,ELF3蛋白可能参与下调主要环境温度信号中枢的转录物水平PIF4,从而影响热响应性生长。转基因互补实验进一步证明,天然植株Bay-0和Sha之间的非同义SNP显著影响ELF3调节温度诱导的能力PIF4转录水平、靶基因和下胚轴伸长。
一般来说,不同类型的多态性,如非同义snp或表达水平多态性,可以促进特定性状的表达[26]。与这一现象相一致,不同类型的自然发生ELF3多态性似乎有助于下胚轴伸长对不同温度的响应。盒子et al。[27]最近使用了一种不同的基于MAGIC系的定量遗传方法[28],并优雅地展示了蛋白质编码和表达水平的多态性ELF3可能是造成自然资源的差异的原因。作者提出了令人信服的证据,证明温暖减轻了夜间ELF3的生长门控。具体来说,转录靶标的ELF3门控通过ELF3与靶启动子的温度依赖性结合迅速响应温度变化,包括PIF4.连同盒子et al。的(27在非转基因定量互补分析中,我们的转基因互补明确地确立了ELF3在热形态发生信号传导中的作用。
有趣的是,ELF3两项研究的qtl在不同的光周期中得到鉴定。ELF3在短日照条件下,确定了MAGIC群体中引起变异的多态性。相比之下,我们的研究发现ELF3长日光周期下的多态性,随后对HIF系的分析显示出长日特异性(图2)。2 b).此外,还将Bay-0和Sha与Box研究中使用的两个亲本系进行了直接比较et al。[27]揭示了Sf-2和Zu-0等位基因在促进下胚轴伸长方面的短日特异性7).不同天然等位基因的光周期特异性本身就是一个有趣的观察结果,需要进一步的研究。这两项研究的另一个意想不到的差异与生长温度有关ELF3QTL检测。而我们确定了ELF3下胚轴生长的QTL在28°C时达到峰值,而在20°C时达到峰值。1 d),盒et al。[27没有发现ELF3在高温下,而不是在22℃的标准生长条件下。这种差异可归因于自然温度和光周期的微分积分ELF3等位基因。或者,遗传背景和与其他贡献位点的相互作用可能涉及。为了支持这一点,我们知道ELF3调节生长取决于基因组的环境[23]。因此,盒子et al。[27],本研究提供了补充证据,证明ELF3在环境温度信号网络中作为PIF4上游的主要信号枢纽发挥核心作用,并为其复杂的调控增加了另一层(图2)。5).
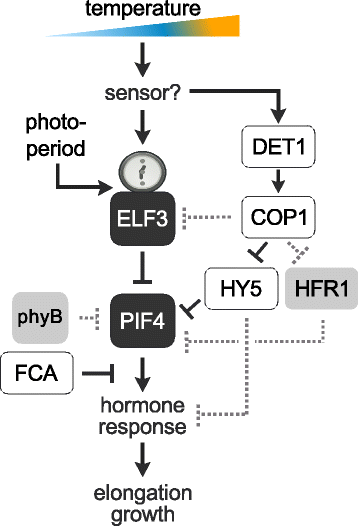
环境温度信号的简化模型。ELF3作为PIF4的转录抑制因子,整合温度和光周期信息调控热形态发生。此外,PIF4温度响应的调节涉及光形态发生途径的调节成分DET1, COP1和HY5 [10]。PIF4介导的靶基因转录调控可被RNA结合蛋白FCA终止,导致PIF4与靶基因启动子分离[33]。先前显示的其他机制有助于PIF4调控,例如,通过转录因子的BAP/HLH/bHLH模块对靶基因的转录调控[34]。也可能有助于调节温度反应。从其他生物学背景中已知的PIF4活性的其他推定参与的调节剂用灰色虚线表示。这些包括DET1与PIF4的直接结合[12], PIF4和其他转录因子对类似结合位点的竞争[35], HFR1对PIF4蛋白的隔离作用[36]和phyb介导的PIF4的磷酸化和降解[37]
出全新et al。[11最近确定的ELF3作为海湾-0 ×沙种群生物钟调控的QTL。事实上,他们证明了ELF3沙蛋白质变异不能正确定位到细胞核,其在黑暗中积累的能力受到损害。如果同样的情况适用于环境温度响应,则ELF3沙可能无法在夜间下胚轴生长高峰时积聚在细胞核内。由于ELF3的电位较弱沙镇压PIF4因此,温度升高可能导致pif4介导的信号传导过程在夜间更早地激活。事实上,盒子et al。[27研究表明,在高温下下胚轴的生长动态与标准条件下的生长动态有很大不同,并且在第一个晚上的生长时间延长,在随后的夜晚的黑暗时期开始时出现额外的生长高峰。然而,目前尚不清楚温度如何影响ELF3蛋白的定位。
结论
综上所述,在了解ELF3的功能方面取得了显著进展。结合最近关于ELF3在避荫反应中的作用的研究[18,21]和生物钟[11,13,29],这项工作有助于理解其在协调调节植物生长和发育的各种环境刺激的微调整合中的作用(图2)。5).引起热形态发生的自然变化ELF3变异可以在几个层次上进行调节。第一,转录调控ELF3本身由表达水平引起的多态性可导致不同数量的PIF4-抑制ELF3蛋白[27]。此外,编码序列多态性可能影响ELF3蛋白与PIF4蛋白相互作用的能力,从而抑制其转录活性,如Nieto所示et al。[13]。然而,目前尚不清楚这种蛋白质-蛋白质相互作用是否依赖于温度,并且这种机制是否存在自然变化尚未报道。最后,非同义snp可能影响ELF3的核积累,这与上述表达水平多态性一样,会导致核ELF3数量的变化[11能够抑制转录的PIF4,以及其他目标。可能后一种机制是本文报道的Bay-0和Sha热形态发生自然变化的原因。
我们的工作结合了Box的研究et al。[27]进一步深入了解了ELF3在集成多个信号以促进架构变化中的重要作用。天然ELF3等位基因的光周期特异性功能可以为阐明时钟介导的生长调控和ELF3的具体作用方式提供新的途径。
方法
植物材料
用于QTL分析的植物材料来自凡尔赛拟南芥库存中心:Bay-0(登记号41AV), Sha (236AV),异种近交系(hfs, 33HV84, 33HV194), Bay-0 × Sha群体(33RV)。用于互补测定的行已在回答中描述et al。[11]。自然材料Sf-2 (N6857)和(Zu-0 N6902)来自诺丁汉拟南芥资源中心。
热响应生长试验
对种子进行表面灭菌,保存在去离子化的H中2播种前在4°C下放置3天。幼苗在无菌条件和指定温度下发芽和生长拟南芥溶液(ATS)介质[30.]。在250 μmol m下培养8 ~ 10 d,定量测定下胚轴的生长情况−2年代−1白光和长日照周期(16/8),除非另有说明。下胚轴长度采用RootDetection软件包(http://www.labutils.de/).利用ImageJ软件对12 d幼苗的叶柄长度和叶片展开进行了测定。对Bay-0 × Sha群体进行了包括表型在内的所有生长试验,至少重复了三次,结果相似,其中显示了一个代表性数据集。
QTL定位
描述QTL定位采用Haley-Knott回归[31用R/qtl包以1 cM的速度[32]。概率对数(LOD)评分阈值基于1000个排列和α错误率0.05。QTL映射程序的详细说明见附加文件8.对Bay-0 × Sha定位群体的表型进行了三次独立的QTL定位,结果相似。给出了一个代表性数据集的QTL映射数据。用于QTL定位的表型数据作为附加文件提供9.
光响应试验
将种子表面消毒,在4°C下分层2天,分散在0.8%琼脂(w/v) Murashige和Skoog培养基上。单色红(670 nm峰波长,0.89 μmol m)−2年代−1),远红(峰值波长735 nm, 0.024 μmol m)−2年代−1)或蓝光(峰值波长470 nm, 4.93 μmol m)−2年代−1)用LED SNAP-LITE (Quantum Devices, Barnereld, WI)生成。绿光(峰值波长540 nm, 0.32 μmol m)−2年代−1)是由过滤过的荧光灯产生的。峰值波长验证和光强测量使用SPEC-UV/PAR光谱仪(Apogee Instruments, Logan, UT)完成。植物在黑暗或光照下孵育指定时间后,拍摄植物图像。采用ImageJ软件测量下胚轴长度。
中存在
将表面灭菌的种子置于ATS培养基上,在长日照(16/8)和100 μmol m条件下培养7 d−2年代−120℃白光。温度诱导的样品在黄昏时转移到28°C,而对照植物保持在20°C。用于qRT-PCR分析的样本在主观黎明前4小时的半夜采集。如前所述进行样品制备和qRT-PCR(包括引物序列)[10]。
统计分析
绝对长度或表达值之间的统计差异采用单因素方差分析,使用默认参数R(www.r-project.org).为了评估不同基因型之间治疗反应的差异,我们使用了“基因型x治疗”相互作用结果的双向方差分析,对其中的绝对值进行了分析R.在显示相对长度数据的箱形图中突出显示了统计学上显著的差异。
缩写
- 美国胸科协会:
-
拟南芥解决方案
- AtHB2:
-
拟南芥同源盒蛋白2
- 软面包卷:
-
BZR-ARF-PIF
- bHLH:
-
基本helix-loop-helix
- COP1:
-
本构PHOTOMORPHOGENIC1
- 弟弟:
-
黑暗
- DET1:
-
DE-ETIOLATED1
- ELF3:
-
早期FLOWERING3
- FCA:
-
花期控制蛋白
- 吉尔:
-
长颈鹿
- HFR1:
-
在远红色的长下胚轴1
- 低氧诱导因子:
-
异种近交系
- 通过:
-
Helix-loop-helix
- HY5:
-
细长HYPOCOTYL5
- 差:
-
四分位范围
- LD:
-
漫长的一天
- 噢,
-
连续光
- LOD:
-
概率对数
- 勒克斯:
-
勒克斯ARRHYTHMO
- PIF4:
-
PHYTOCHROME-INTERACTING FACTOR4
- QTL:
-
数量性状位点
- 阿富汗二月:
-
小生长素上调RNA
- SD:
-
短暂的一天
- SNP:
-
单核苷酸多态性
- TIHE:
-
温度诱导下胚轴伸长
参考文献
Erwin JE, Heins RD, Karlsson MG。长花百合的热形态发生。[J] .中华医学会杂志。1989;76:47-52。
黄晓明,黄晓明,黄晓明,等。高温对拟南芥下胚轴伸长的影响。科学通报,1998;35(5):397 - 397。
范赞腾M, Voesenek LACJ, Peeters AJM, Millenaar FF。激素和光介导的拟南芥热诱导叶柄差异生长调控。植物生理学报。2009;51(1):444 - 444。
Crawford AJ, McLachlan DH, Hetherington AM, Franklin KA。高温暴露增加了电厂的冷却能力。中国生物医学工程学报,2012;22:396 - 397。
桥LJ,富兰克林KA,荷马ME。植物茎部结构对叶片冷却的影响:一个耦合传热传质模型。[J] . Soc接口。2013;10:20130326。
Franklin KA, Lee SH, Patel D, Kumar SV, Spartz AK, Gu C,等。光敏色素相互作用因子4 (PIF4)调控生长素在高温下的生物合成。科学通报,2011;38(2):391 - 391。
孙洁,祁玲,李莹,褚军,李超。pif4介导的YUCCA8表达激活将温度整合到生长素通路中调控拟南芥下胚轴生长。科学通报,2012;8:1002594。
王晓明,王晓明,王晓明,等。高温胁迫下PIF4信号通路的研究进展。植物学报,2013;18:59-64。
王晓明,王晓明,王晓明,等。植物生长素对植物生长的影响。植物物理学报,2014;51(1):13 - 24。
Delker C, Sonntag L, James GV, Janitza P, Ibañez C, Ziermann H,等。DET1-COP1-HY5通路是调控植物光形态发生和热形态发生的多用途信号模块。Cell Cell . 2014; 9:1983-89。
Anwer MU, Boikoglou E, Herrero E, Hallstein M, Davis AM, James GV等。自然变异揭示了ELF3蛋白在细胞内的分布与生物钟的功能有关。eLife。2014; 3: e02206。
董军,唐东,高志,于锐,李坤,何红,等。拟南芥DE-ETIOLATED1通过积极调节光敏色素相互作用因子在黑暗中抑制光形态发生。植物学报。2014;26:3630-45。
Nieto C, López-Salmerón V, david j . m。ELF3-PIF4相互作用独立于Evening Complex调控植物生长。中华生物医学杂志,2015;25:187-93。
Delker C, Pöschl Y, Raschke A, Ullrich K, Ettingshausen S, Hauptmann V,等。拟南芥中生长素转录反应网络的自然变异。植物学报。2010;22:2184-200。
刘建军,张建军,张建军,等。拟南芥的遗传分析及其在遗传分析中的应用。理论与应用学报。2002;104:1173-84。
图斯特拉先生,Ejeta G, Goldsbrough PB。异种自交系(HIF)分析:一种在数量性状位点上存在差异的近等基因系的开发方法。理论与应用。1997;95:1005-11。
Martínez O, Curnow RN。利用侧翼标记估计数量性状位点的位置和效应大小。理论与应用。1992;85:480-8。
Jiménez-Gómez JM, Wallace AD, Maloof JN。网络分析表明,ELF3是拟南芥遮荫躲避反应的QTL。科学通报,2010;6:e1001100。
Herrero E, Kolmos E, Bujdoso N, Yuan Y, Wang M, Berns MC,等。细胞核中EARLY FLOWERING3的募集维持着拟南芥的生物钟。植物学报。2012;24(4):428 - 43。
张建军,张建军,张建军,等。elf4 - elf4 - lux复合体将生物钟与下胚轴生长的昼夜控制联系起来。大自然。2011;475:398 - 402。
Coluccio MP, Sanchez SE, Kasulin L, Yanovsky MJ, Botto JF。避荫反应自然变异的遗传定位:ELF3是下胚轴生长调控QTL的候选基因。[J] .中国生物医学工程学报,2011;32(2):557 - 557。
田岛T,田田A,中川M, Kamada H, Mizoguchi T.拟南芥生物钟基因ELF3重复序列的自然变异。植物学报。2007;24(4):344 - 344。
Undurraga SF, Press MO, Legendre M, Bujdoso N, Bale J, Wang H,等。拟南芥ELF3基因中多谷氨酰胺变异的背景依赖效应科学通报,2012;19(3):363 - 367。
Kunihiro A, Yamashino T, Nakamichi N, Niwa Y, Nakanishi H, Mizuno T.光敏色素相互作用因子4和5 (PIF4和PIF5)激活同源体ATHB2和生长素诱导的IAA29基因在拟南芥光周期控制植物生长中的一致性机制。植物物理学报,2011;32(2):1315 - 1329。
Spartz AK, Lee SH, Wenger JP, Gonzalez N, Itoh H, Inze D,等。SMALL生长素UP RNA基因的SAUR19亚家族促进细胞扩增。植物科学学报,2012;37(1):391 - 391。
王晓明,王晓明。基因表达水平多态性:遗传性状对自然变异的影响。植物学报,2011;16(4):481 - 888。
Box MS, Huang BE, Domijan M, Jaeger KE, Khattak AK, Yoo SJ,等。ELF3控制拟南芥的热响应性生长。中华生物医学杂志,2015;25(1):94 - 94。
Kover PX, Valdar W, Trakalo J, Scarcelli N, Ehrenreich IM, Purugganan MD,等。拟南芥多亲本高级代间杂交精细定位数量性状。生物医学工程学报,2009;5:e1000551。
水野,野本,冈,北山M,竹内A, Tsubouchi M等。在拟南芥中,环境温度信号通过EC夜间抑制因子进入生物钟转录回路。植物生理学报,2014;55(5):958 - 976。
李建军,李建军,李建军,等。拟南芥基因突变体axr1的生长发育。植物细胞。1990;2:1071-80。
哈利·CS,诺特SA。利用侧翼标记定位单系杂交数量性状位点的简单回归方法。遗传。1992;69:315-24。
Broman KW, Wu H, Sen S, Churchill GA。R/qtl:实验杂交的qtl定位。生物信息学。2003;19:889 - 90。
李宏杰,郑建辉,cortsams Llorca L, Kim S- g, Lee S, Baldwin IT,等。FCA通过减弱拟南芥的生长素作用介导茎生长的热适应。自然科学学报。2014;5:5473。
吴娥,朱建勇,白明勇,Arenhart RA,孙勇,王志勇。细胞伸长是通过拟南芥下胚轴中相互作用的转录因子的中央回路调节的。eLife。2014; 3: e03031。
李建军,李建军,李建军,李建军。HY5-PIF调节模块协调光合基因转录的光和温度控制。科学通报,2014;10:e1004416。
李建军,刘建军,张建军,等。植物抗光性的研究进展。中国生物医学工程学报,2009;28(3):391 - 391。
刘建军,张建军,张建军,等。植物光合色素对植物生长的抑制作用。植物学报,2008;33(3):391 - 391。
致谢
我们感谢Christine Camilleri和INRA Versailles提供的HIF生产线。这项工作得到了德意志研究基金会(german Forschungsgemeinschaft)对MQ (Qu 141/3-1)和莱布尼茨协会的资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们没有竞争利益。
作者的贡献
MQ设计了这项研究。AR、CI、SB、AG、JT、KD和CD进行热响应生长试验。KKU和BS进行QTL分析。AR和CI精细映射了GIR2.1QTL。CI和CD进行qRT-PCR分析。XS和MN进行光响应试验。MUA和SJD提供了材料并编辑了手稿。AR, CI, CD和MQ撰写了手稿。所有作者都阅读并认可了稿件。
作者的信息
Anja Raschke和Carla Ibañez共同第一作者。
附加文件
附加文件1:图S1。
海湾-0和沙的温度诱导生长响应。(A)温度诱导下胚轴伸长(TIHE)、(B)温度诱导叶柄伸长(TIPE)和(C)温度诱导叶片伸长(TILE)的绝对值。数据对应图中所示的相关数据。1.箱形图显示了中位数(水平线)、四分位数范围(IQR,方框)和数据范围(晶须)。黑点表示异常值(定义为> 1.5 × IQR)。统计学差异采用单因素方差分析和Tukey HSD (P< 0.05),分别用不同的小写字母和大写字母表示20℃和28℃的值。
附加文件2:表S1。
海湾-0 ×沙种群表型描述统计分析。
附加文件3:表S2。
QTL汇总统计。
附加文件4:图S2。
验证GIR1QTL。(A)海湾-0和沙分离的异种近交系(HIF) 194的单倍型综述GIR1的验证和映射GIR1QTL。(B)箱形图显示了10日龄幼苗的相对(28°C/20°C, %)下胚轴长度,该数据来自(C)中的绝对下胚轴长度数据。(D)海湾-0和沙的杂种自交系(HIF) 214的单倍型概述GIR5.1间隔,用于该QTL的验证。(E)箱形图显示10日龄幼苗的相对(28°C/20°C, %)下胚轴长度,来源于(F)中的绝对下胚轴长度数据。(B,C,E,F)水平条、箱形图和须形图分别表示中位数、四分位数范围(IQR)和数据范围。黑点表示异常值(定义为> 1.5 × IQR)。(B+E)不同字母表示经双向方差分析评估的温度反应统计差异(P< 0.05)的绝对下胚轴长度数据。(C+F)统计学差异采用单因素方差分析和Tukey HSD (P< 0.05),分别用不同的小写字母和大写字母表示20℃和28℃的值。在亲本系Bay-0和Sha中观察到的TIHE的显著差异并没有反映在携带Bay-0或Sha等位基因的214-B和214-S中GIR5.1分别间隔。因此,GIR5.1QTL无法用现有遗传物质进行验证。
附加文件5:图S3
改变光照条件对GIR2.1介导的下胚轴伸长。(A)箱形图显示了Bay-0、Sha和HIF系10日龄幼苗的相对(28°C/20°C %)和绝对下胚轴长度,Bay-0 (84-B)和Sha (84-S)均为纯合子GIR2.1时间间隔。水平条形图、框状图和须状图分别表示中位数、四分位数范围和数据范围。黑点表示异常值(定义为> 1.5 × IQR)。由84-B和84-S杂交而成的F1植株对应图中84-H单倍型。2并说明了Sha对Bay-0等位基因的优势。不同字母表示通过双向方差分析评估的温度反应的统计差异(即基因型x治疗效果,P< 0.05)的绝对下胚轴长度数据。(B)图中相对数据对应的下胚轴绝对长度。2 b.统计学差异采用单因素方差分析和Tukey HSD (P< 0.05),分别用不同的小写字母和大写字母表示20℃和28℃的值。(C)单色光条件下下胚轴长度。在20℃恒蓝(4.93 μmol m)培养4 d的幼苗中,Bay-0和Sha的差异显著−2证券交易委员会−1),绿色(0.32 μmol m)−2证券交易委员会−1红色(0.89 μmol m)−2证券交易委员会−1)光。这些差异似乎是独立于GIR2.1因为84-B和84-S的生长反应没有差异。在远红(0.024 μmol m)环境中生长的幼苗基因型间无差异−2证券交易委员会−1)光。统计学差异采用单因素方差分析和Tukey HSD (P< 0.05),分别用不同的小写字母和大写字母表示20℃和28℃的值。
附加文件6:图S4。
GIR2构成幽灵QTL。中设置其他协变量GIR2.1目标区域将单峰分隔成两个相连的峰(如图2所示)。1 d),表明可能存在两个连锁位点。x轴上的标记对应于Bay-0和Sha作图群体遗传图谱中的分子标记。x轴上的圆圈表示复合区间映射的协变量集。
附加文件7:图S5。
光周期和等位基因对elf3介导的TIHE的影响。在短日照(SD)和长日照(LD)下生长的7日龄幼苗的he比较。箱形图显示Bay-0、Sha和MAGIC群体亲本系Sf-2和Zu-0的相对(28/20°C %)和绝对下胚轴长度ELF3.虽然Sf-2和Zu-0在SD中表现出强烈的TIHE响应,但Sf-2和Zu-0在LD下的响应要弱得多。水平条形、箱形和须形分别显示了中位数、四分位间距(IQR)和数据范围。黑点表示异常值(定义为> 1.5 × IQR)。(A)不同字母表示通过双向方差分析评估的温度反应的统计差异(即基因型x处理效果,P< 0.05)的绝对下胚轴长度数据。(B)统计学差异采用单因素方差分析和Tukey HSD (P< 0.05),分别用不同的小写字母和大写字母表示20℃和28℃的值。
附加文件8:
方法S1。遗传力估计及QTL分析程序。
附加文件9:
补充数据集表型数据用于QTL定位。该数据集已上传到figshare,可通过http://dx.doi.org/访问10.6084 / m9.figshare.1339892.
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
Raschke, A., Ibañez, C., Ullrich, K.K.et al。的自然变种ELF3通过转录调节影响热形态发生PIF4-依赖性生长素反应基因。BMC Plant Biol15日,197(2015)。https://doi.org/10.1186/s12870-015-0566-6
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-015-0566-6
关键字
- 环境温度信号
- 生长素
- ELF3
- PIF4
- Thermomorphogenesis