摘要
背景
菠萝(ananas comosus.var。comosus)因其皇冠而被誉为水果之王,是仅次于香蕉和柑橘的第三重要热带水果。这种植物原产于南美洲,是凤梨科中最重要的物种,主要用于新鲜水果的消费。本文报道了利用PacBio测序技术对MD-2菠萝叶绿体的完整序列进行测序的结果。
结果
在这项研究中,PACBIO长序列读取的高误差率A. comosus通过使用Novocraft最新的纠错模块,利用Illumina的高精度但短的纠错读取,改进了总基因组DNA。通过使用一个单一的工具来生成代表菠萝叶绿体基因组的contig,长PacBio读取错误被纠正。基因组长度是159636个基点的特色与叶绿体的守恒的由四部分组成的结构包含一个大的单一副本地区87482个基点(LSC)的大小,一个小单副本区域(SSC)与18622个基点的大小和两个反向重复区域(爱尔兰共和军和IRB)每26766个基点的大小。总体而言,该基因组包含117个独特编码区,其中30个在IR区域重复,其基因内容、结构和排列与其姐妹分类单元相似,香蒲latifolia.总共35个重复结构的两个与大多数是串联重复的编码和非编码区进行检测。此外,在具有六个蛋白质编码基因的基因组中检测到的SSR 205包含两个以上的固态继电器。从子类比较基因组叶绿体揭示鸭跖草亚纲保守蛋白编码基因尽管位于在高度发散的区域。使用嘉/ Ks率蛋白质编码基因的选择压力的分析表明施加在显著正选择RPS7P < 0.05。系统发育分析证实了两种属间的新分类关系,支持了槟榔科(arerecales)与大枣科(Dasypogonaceae)以及姜科(Zingiberales)与Poales的单系关系A. comosus.
结论
菠萝的叶绿体的完整序列提供见解从子类鸭跖草亚纲的成员核不叶绿体序列的发散。完整的菠萝叶绿体将作为在凤梨家族的深入研究,分类学的参考,当家人在更加物种在未来的测序。基因序列信息,也会使菠萝叶绿体的植物遗传改良是可行的其它分子的应用。
背景
菠萝是一种古老的果实,从此感觉到它具有悠久的人类消费历史。在1519年,Pigafetta被描述为“存在最精致的果实”([1),时至今日,它的甜味和酸味仍备受推崇。这种水果大多是新鲜食用的,不仅被认为是一种美食的乐趣,而且在两餐之间还被用作助消化,由于它含有一种被称为菠萝蛋白酶的强蛋白酶,它还可以作为肉的嫩化剂[2].传统上,不同的品种菠萝这种植物被种植用于制造酒精饮料和毒药,它的高纤维叶子被用作衣服的织物[3.].ananas comosus.从凤梨家族禾本目秩序,鸭跖草亚纲的子类。该系列产品由56个属和超过2885种[4].然而,截至2015年2月11日,在NCBI Organelle Genome Resources数据库中,该家族的任何物种都没有完整的质体序列输入。尽管如此,已经进行了多项研究,利用叶绿体衍生的多个短序列来研究家族成员之间的关系[5- - - - - -8].叶绿体序列为植物分子系统学研究提供了重要信息。利用分子数据推断植物之间的系统发育关系,发生了许多重分类,新的目被确认[9].这包括对凤梨科新进化枝的识别。
最初,叶绿体DNA序列的发现始于物理图谱的绘制玉米由多种限制性内切酶消化产生的叶绿体[10.].后来,第一个完整的核苷酸序列尼科尼亚塔哈瓦姆被解开通过乏味克隆由所述质粒和粘粒文库的克隆测序11.].接下来是几种植物的叶绿体序列[12.- - - - - -14.].率先对松树家族的叶绿体基因组进行测序[15.,使用诸如大规模的平行测序和焦肉测序等下一代测序(NGS)技术进行了更多的叶绿体基因组。但是,这是新创事实证明,组装这数百万个NGS读取数据是一个繁琐的过程。该技术通常调用参考引导程序集,然后调用从头短阅读的集合,因为它不能相互独立地重叠[16.- - - - - -18.].然而,通过读取末端的迭代映射成功地利用焦点测序技术读取以形成单个折叠,但是该草案容易达到均聚物误差,这需要额外的低通量Sanger测序进行装配校正[19.].最近,利用PacBio的长读测序技术发表了几个叶绿体基因组[20.- - - - - -23.].在这些研究中,质体基因组序列被组装成最终的单个序列。这些成就突出了长读序列的用处新创通常,许多基因组已经通过PacBio成功组装[24.- - - - - -26.].
叶绿体基因经常用于在不同的分类令中推断植物系统发育。最值得注意的是识别Commelinids Clade,其由Angiosperm Phylogy基团认识到的分子和形态学数据支持[9].进化枝被分组为五个数量基于分子数据的均匀性[27.]和UV-荧光阿魏酸结合的细胞壁的共存[28.].在最近的研究中,巴雷特等人。[29.结果表明,槟榔属和拟南芥属(Dasypogonalesas)与姜属(Zingiberales)、拟南芥属(Commelinales)和Poales是姐妹关系。本研究旨在描述菠萝叶绿体基因组的特征,并利用PacBio错误校正读取功能将菠萝叶绿体与commelinids分支的其他成员进行比较新创组件中的叶绿体基因组的。我们也希望分享与叶绿体的大反向重复区域打交道时的优势和面临的挑战读者。
材料和方法
样品材料
世界著名的MD-2菠萝杂交品种由于其高品质的果实和世界范围的需求而被选择进行测序。这些菠萝是由马来西亚菠萝工业委员会提供的。MD-2最初是通过菠萝研究所(PRI)在20世纪60年代的育种计划开发的。这条生产线后来在20世纪80年代交给毛伊菠萝公司和德尔蒙特公司进行进一步评估。MD-2是由两个PRI杂交种杂交而成,由几个菠萝品种混合而成,但其中有很高比例的光滑辣椒,这是当时的优势菠萝品种。
Illumina图书馆建设和测序
总基因组DNA按照CARLIER等人的协议从新鲜菠萝叶提取。[30.略作修改。简单地说,菠萝叶被液氮研磨成粉末。然后将粉末混合到预热的5 mL萃取缓冲液中,缓冲液中含有200 mM Tris-HCl pH 8;25毫米EDTA;250毫米氯化钠;1% SDS和2% PVP。然后混合物在65°C孵育30分钟,10分钟的混合间隔。然后加入10 μL RNAse (100 mg/mL), 37℃继续孵育1小时。随后,加入5 μL蛋白酶K (20 mg/mL),在55℃下再孵育30分钟。用苯酚:氯仿:异戊醇有机溶剂连续萃取,净化混合物以去除蛋白质污染,然后在水溶液中加入1/10体积的2 M乙酸钾和等体积的异丙醇沉淀DNA。 Finally the crude DNA sample was washed with two rounds of freshly prepared 70 % alcohol and diluted in 100 μL TE buffer.
使用1%琼脂糖凝胶电泳检查DNA样品的完整性,同时通过使用分光光度计(NanoDrop 2000,Thermo Sciencific),在A260 / A A280和A260 / A230的比率下测量其纯度。使用荧光计(Qubit 2.0,寿命技术)测量DNA的浓度,并在100bp成对端格式的一条车道上使用Illumina Hiseq测序10μg总DNA。测序服务由韩国宏观原提供。对于图书馆制备,根据制造商的协议使用Truseq PCR Free(Illumina,USA)图书馆制剂。然后,收到的短读取使用FQTRIM软件(https://ccb.jhu.edu/software/fqtrim/index.shtml),最低质量为Q20,长度为50 bp或以上,并结合测序适配器的移除。总大小为38 GB的高质量读只产生了总数据的8.87%。
PACBIO图书馆建筑和测序
在使用PacBio RSII进行实时单分子测序时,DNA纯度高是至关重要的,以保证良好的测序性能。传统上,在DNA提取过程中使用酚、氯仿和异戊醇的组合来变性蛋白质污染,正如前面所描述的Carlier等人的方法[30.].然而,使用强溶剂可能会导致有机物质携带,从而降低提取的DNA的纯度。因此,如Dellapota等人所述的替代方法,例如盐析出方案。[31.被采用。同样,新鲜的叶子被压在液氮中。然后,将提取缓冲液(1% β-巯基乙醇,100 mM Tris pH 8, 50 mM EDTA pH 8, 500 mM氯化钠)和330ml 20% SDS加入叶粉中。然后将混合物在60°C的温度下以950 rpm混合10分钟(美国Eppendorf)。然后,加入5 μl RNase A (100 mg/mL),在37℃恒温箱中500 rpm搅拌1小时。然后加入1.6 mL 5 M醋酸钾,在冰上继续孵育半小时。然后在13000 rpm下离心20分钟,只收集上清液到含3.30 mL异丙醇的新鲜管中。然后将混合物倒置并在−20°C孵育过夜。DNA样品10000 rpm离心30分钟成球,500 μl TE缓冲液重新悬浮。待微丸完全溶解后,加入3 M醋酸钠75 μl和异丙醇500 μl,倒置多次混匀。 The tube was then incubated in −80 °C for 20 mins and was centrifuged at 10,000 rpm for 30 mins. The crude DNA was then washed with 70 % alcohol twice before it was re-dissolved in 50 μl TE buffer.
使用1.0%琼脂糖凝胶电泳检查DNA的完整性,并且使用Nanodrop 2000分光光度计测量纯度指数,用于A260 / A280和A260 / A230比率。两个指数分别在1.9和1.8点测量。SMRTBELL DNA模板预备Kit V.1(太平洋Biosciences,USA)与P4-C2和P5-C3测序化学结合使用,根据PACBIO示例净共享协议构建文库,可提供http://pacificbiosciences.com/.使用PACBIO RSII测序文库,共测量总共6655 MB。PacBio测序读数的平均读取长度为5306 BP,最长读数为37,591 BP。然后使用NovocleAverlR从Novocraft中处理PACBIO读取,该Novocraft是对准的并且随后去除PACBIO的过滤的底亚的任何存在SMRTbell适配器序列的存在。这产生了873,181个高质量读数为4.6 GB的总体大小。然后使用NovoAlign程序使用350bp和750bp Illumina高质量短读取来校正清洁的读取。然后将novoxorrector应用于生成的对齐文件以执行变体调用并产生错误纠正的PACBIO读取。
基因组组装
四百七可用的叶绿体基因组(附加文件1)从NCBI数据库下载,并且这些序列被用于找到其类似于菠萝叶绿体通过使用GMAP对准在默认设定阅读序列[32.].以叶绿体为数据库,通过GMAP比对器查询了818,142条PacBio长错误修正序列。使用内部脚本处理来自GMAP输出的psl对齐文件,以生成百分比标识分数和对齐长度。总的来说,有58126个独特的读本进入了叶绿体数据库。在此基础上,以100 bp以上的比对长度选取41188条reads,并以1000 bp的最小读长作为最小读长新创菠萝叶绿体基因组的组装。米拉v4.0.2 [33.用于从41,188长的叶绿体误差纠正PacBio读取的折叠,从而使用重叠图算法和自动基因组精加工。使用Mira V4.0.2的41,188次错误校正的PACBIO的初始组装读取(总大小为182 MB),产生246个COLDIG,N50为14,757 BP。尺寸为95,865和64,927bp的两个最大的凸点具有相似的GC含量(36%),与其他Contigs相比,两者均具有大致相同的读取覆盖率(108-118×)。从组装的Contigs,使用类似GC百分比的两个最大的COLIG百分比再次捕获属于氯化体序列的读取,从完全误差校正使用GMAPigner读取,设置为默认值。对于这一轮,选择百分比百分比的百分比和匹配中的比赛长度的命中率仅在1000年以上的匹配中获得,并且这给了19,260个读取。然后使用相同的参数再次由Mira处理这些读取新创组装,生产的单个contig尺寸为164,813 bp。对准香蒲latifolia完成叶绿体基因组(GenBank登录号:NC_013823.1),重叠群然后手动重排以形成所述保守四方结构。详细地,重叠群被截断成两个片段,并使用强强联合(合并http://emboss.bioinformatics.nl/cgi-bin/emboss/megamerger),找出重叠区域,并进一步合并成序列。合并后的片段随后被加载到SMRTPortal中作为参考,PacBio的原始读取被映射回组装的叶绿体,使用bridgeemapper_rs和Quiver的碱基校正进行验证。
对于评估目的,来自未校正过滤的PACBIO的所有读数总计4.6GBP,总共校正的PAGBIO读取(821,079读数)使用Blasr长距离对准映射到最终叶绿体基因组中的总共3.14 gbp(-bestn 5-minpctifidity 90-placerepeatsrandomly)[34.].
基因组注释
组装的叶绿体序列用Dual Organellar Genome Annotator (DOGMA)注释[35.],使用NCBI的Sequin工具手动启动和停止密码子验证(http://www.ncbi.nlm.nih.gov/sequin.).接着,tRNAScan-SE [36.],采用细胞器搜索模式注释tRNA, Cove cut-off评分为15。使用REPuter识别正向重复和反向重复[37.最小重复大小为30 bp,汉明距离为3,同一性不小于90%。核苷酸频率和相对同义词密码子用法[38.]用DAMBE分析[39.]在IRA 83蛋白编码基因和仅基因用于表示重复基因。圆形的基因组是使用OGDRAW程序[绘制40].
简单序列重复(SSRs)鉴定
采用MISA (http://pgrc.ipk-gatersleben.de/misa/misa.html.),其参数如下:最小SSR基序长度为8 bp,单-8、二-4、三-4、四-3、五-3和六-3的重复长度。MISA还鉴定出了相邻的、被一定长度的序列分隔的复合SSR。在本分析中,在一个复合SSR中,两个不同SSR之间允许的最大中断长度为100 bp。
基因组比较
除Poales (BEP、PACMAD、Pueliodeae、Pharoideae、Anomochlooideae和Typhaceae)各选一种外,其余亚纲各选一种。在波尔的命令下,Olyra latifolia,紫三芒草,Puelia olyriformis,Pharus lappulaceus和Anomochloa marantoidea被选择来表示graminids(禾本科)。还包括禾本目大小顺序为:t . latifolia代表毛骨悚然的家族,众所周知,这是伯罗西亚群岛的姐妹的思考[29.因此,它与菠萝叶绿体基因组最接近。包括其他叶绿体基因组Ravenala Madagascariensis.代表Zingiberales,菖蒲caryotoides槟榔和D. Bromeliifolius.为家庭多须草科。所有九个叶绿体基因组进行了比较,在默认设定下通过BLAST环图像生成器(BRIG)使用NCBI BLASTX工具菠萝叶绿体基因组[41.].通过Artemis比较工具(ACT)辅助对菠萝叶绿体基因组的成对比较特定序列的紧密检测42.],该工具所需的比较文件由Double ACT v2 (http://www.hpa-bioinfotools.org.uk/pise/double_act.html).
系统发育分析
截至2015年2月,NCBI细胞器基因组资源(Organelle Genome Resources)收录的Commelinidae亚纲质体基因组共有108个。从中选择100个类群进行系统发育分析;其他8个被排除在外,因为他们有超过10个基因缺失或未注释。对所有100个类群和菠萝的80个蛋白编码基因序列进行了比对;orf48,ORF56.和ycf15被排除在外,因为它们不存在于任何其它鸭跖草类植物叶绿体基因组。对于每个种类,80蛋白编码基因序列翻译成氨基酸,使用MAFFT对准工具对准。43.最后,各自的核苷酸序列基于来自TreeBest的后翻版物工具的翻译氨基酸对准对准(http://treesoft.sourceforge.net/treebest.shtml).使用Kimura双参数(K2P)模型,利用Mega 6计算每个基因的平均两两序列差异[44.].每个基因的Ka/Ks值使用默认设置(−c 11)的KaKs_calculator计算[45.].另外,测试积极选择的重要性RPS7基因,从100个类群中获得的所有序列按上述方法进行比对,并使用Mega 6的基于密码子的阳性选择z检验[44.].为系统发育分析,56个蛋白质编码基因序列共同所有100类群依次连接起来并进行比对如上。的一个ligned nucleotide was then tested in DAMBE for saturation of substitution and this resulted in Iss < Iss.c atp小于0.05,说明这些序列中没有饱和位点。然后使用jModelTest 2.1.7分析序列[46.]以找到具有最低值AICC和所选择的模型最优化的模型是与网站和不变的站点之间的变化率的一般时间可逆(GTR)模型。系统发生树,通过使用最大似然法具有1000次重复的自举快速和二进制的模型,其中GTRCAT使用的伽马分布的下RAxML版本8工具的离散近似[推断47.].最后使用的核酸比对显示在附加文件中2.使用CIPRES Science Gateway V3.3进行对准和最大似然分析48.].
结果与讨论
基因组组装
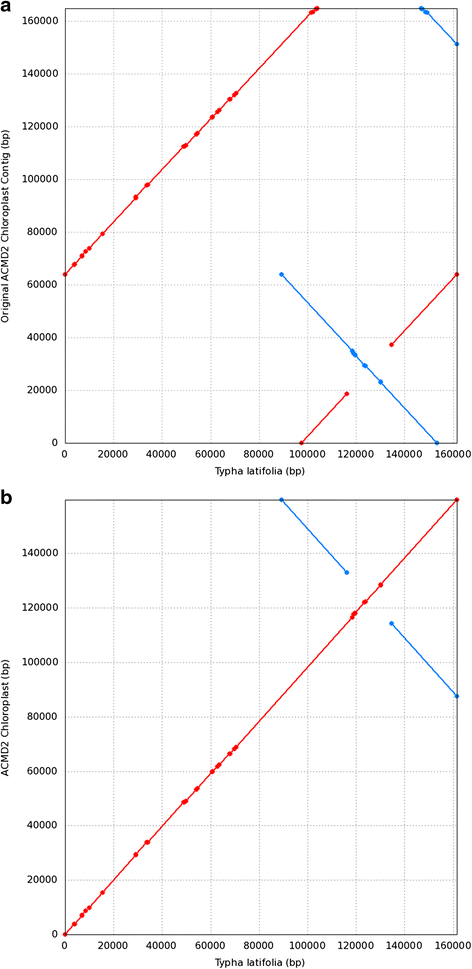
通过Mira读取叶绿体长序列的初始组装,其尺寸为164,814 bp的单个角色。然而,当它映射到其最近的相对时,我们认识到Contig的两个问题,t . latifolia(图1).首先,发现contig的起始和结束与参考基因组(即基因组)不同。t . latifolia叶绿体基因组)。但是,这个问题不是主要问题,因为它很容易通过根据参考文献进行人工重新安排而得到解决。其次,与参考序列相比,短单拷贝(SSC)处于反方向,其两侧有大量的反向重复序列。虽然在某种意义上,contig的排列是正确的,即SSC的两侧有两个相同的序列(即反向重复副本),但SSC的方向不能明确确定。反向重复序列彼此完全相同,大小均在26 kb以上,仅由一个长度为18 kb的短序列分隔。因此,为了解决这个问题,一个组装者需要读遍重复,只在叶绿体的唯一副本(即LSC/SSC)开始和停止。任何从SSC开始并在重复区域结束的读取都将被模糊地放置在任何一个重复副本中,并将导致重复位置的模糊,从而导致SSC的方向。对于包含重复描述的读,它应该有超过30 kb的长度,但是在装配中使用的错误纠正序列读的最大长度只有24,950 bp。因此,为了解决这个问题,Megamerger对contig进行了分解和重组,并根据已发表的陆生植物叶绿体基因组的保守结构进行人工重组。
由MUMmer生成的Syntenic dotplot [96.基于由MIRA(产生的重叠群之间nucmer对准]一个),然后重新排列和(b)重排后t . latifolia叶绿体基因组作为参考。MIRA产生的最初的contig与保守的叶绿体结构如t . latifolia它和它的末端是重叠的也就是反向重复区域。此外,由于将重复序列的第一个副本置于SSC侧翼的不确定性,SSC与参考文献相比处于相反的位置
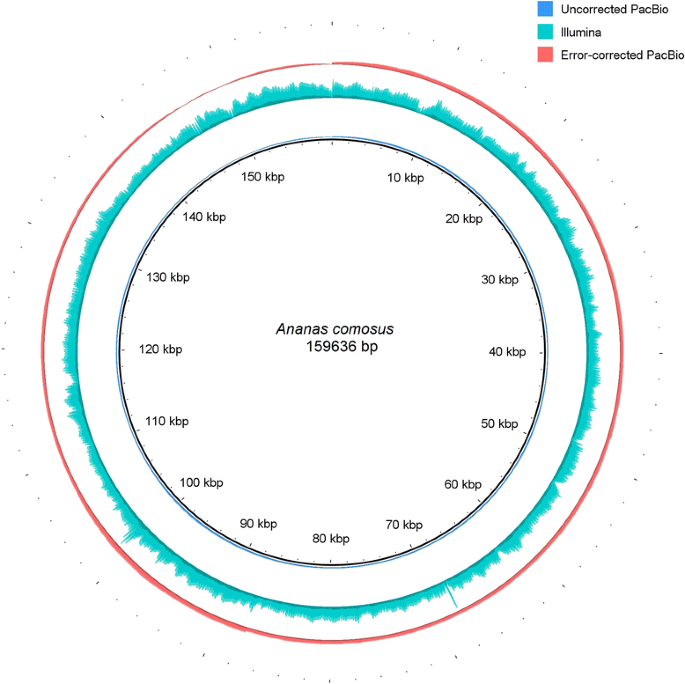
Blasr [34.]用于映射校正和未校正PacBio读取的数据,得到的平均深度覆盖分别为544×和142×(图2)。2)和Illumina公司的取向短读取到用Novoalign其显示的1788的平均深度×执行的叶绿体。虽然短期的对准读取显示更高的平均深度,覆盖整个波动的叶绿体。与此相反,在读取PacBio覆盖更均匀的IR区域,在那里读取优选映射到相对于其他(即使当-placeRepeatsRandomly blasr [红外线中的一个的异常34.使用参数)。与PACBIO读数相比,Illumina读取的类似映射行为在Ferrarini等人中也观察到。[23.].
回映射到错误后的菠萝叶绿体的覆盖面轮廓校正,裸眼和Illumina的快速读取。每三个环表示从背面映射深度覆盖在菠萝叶绿体基因组的组装读取使用。从最外到最内的环表示校正PacBio,Illumina的短读取和未校正PacBio映射到菠萝的叶绿体基因组。每个环的高度成比例的跨过叶绿体基因组读取映射数。图使用BRIG [示出41.]
三个地区ndhB位被Quiver纠正的基因,被选择使用基于pcr的Sanger测序进行验证(附加文件3.).序列验证结果相似度为100%。使用错误校正PacBio读取的菠萝叶绿体的单个contig组装是其潜在的直接证据新创基因组组装。此前,FERRARINI等。[23.]组装成功的叶绿体蕨麻micrantha使用纠错PacBio读取其通过塞莱拉公司装配成重叠群97,并最终到脚手架使用minimus2和Seqman单个序列。在另一项研究中,吴等人。[22.]用于LASTZ工具来组装4塞莱拉公司预组装重叠群为一个最终莲属椰子叶绿体。MIRA的菠萝叶绿体组装成功产生了一个单一序列,不需要任何额外的脚手架工具。然而,在保守的叶绿体结构中普遍存在的反向重复的方向错位,与已发表的其他陆生植物叶绿体基因组的四部分结构不一致。
基因组特征
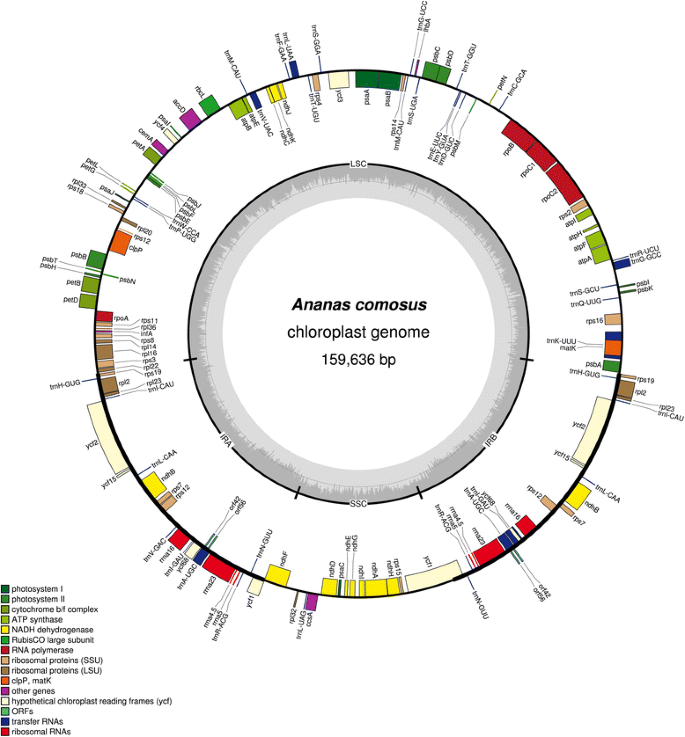
的完整基因组choloroplastA. comosus全长159,636 bp (Genbank: KR336549)。菠萝叶绿体基因组具有陆地植物叶绿体基因组中常见的保守的四部分结构。基因组由大单拷贝(LSC, 87,482 bp)、倒位区a (IRA, 26,766 bp)、短单拷贝(SSC, 18,622 bp)和倒位区B (IRB, 26,766 bp)组成(图)。3.).菠萝叶绿体基因组GC含量为37.37%。IRB和IRB区域的GC含量均较高,分别为42.74%,LSC和SSC的GC含量分别为35.36%和31.41%。结果表明,菠萝叶绿体的基因含量与凤梨叶绿体的基因含量最接近t . latifolia这是凤梨科的妹妹分支[49.].该基因组共有141个编码区,其中117个为唯一编码区,24个为反向重复编码区。其中30个独特的trna有8个重复,83个不同的肽编码基因;12个重复,4个rrna在IR区域均重复(表)1).正如在许多其他陆地植物通常会发现,18个基因,用内含子,从12蛋白编码基因是和6来自tRNA的。一个基因,rps12编码为trans-剪接,LSC区域有一个5 '端,两个IR区域都有一个重复的3 '端[50.- - - - - -52.].
的A. comosus叶绿体基因组。菠萝叶绿体基因组的图所示的圆形表示与这是根据它的功能类别颜色编码的基因含量的环结构组织。最里面的圈表示在整个基因组的GC含量。该被转录逆时针和顺时针旋转的基因分别为在所述外环和内环,。叶绿体插图用OGDRAW [绘制40]
菠萝叶绿体基因发生了1个碱基突变和5个碱基缺失ycf15这引起了基因的3'末端区域的过早止斜墩和帧间偏移。也观察到类似的案例t . latifolia叶绿体基因组,尽管该基因是在当前记录未注释[53.还在其他单焦叶类别的基因组中如凤凰dactylifera(基因库:NC013991.2) (54.),而D. Bromeliifolius.(基因库:NC020367.1)。95个蛋白编码基因包含重复区和伪基因,共计82,389 bp,包含27,361个密码子,不包括终止密码子。同时,编码最多和最少的氨基酸分别是亮氨酸(10.69%)和半胱氨酸(1.30%)(见表)2).这与陆地植物叶绿体中的许多其他蛋白质编码基因相似[55.,56.].
菠萝叶绿体蛋白编码基因的蛋白质合成所需的trna全部被鉴定出来,其数量和种类与其他特征良好的叶绿体基因组相似。RSCU分析显示,包括终止密码子在内的所有氨基酸,与G或C相比,第三密码子位置的a或T存在较强的偏置(表)2).例如,氨基酸丙氨酸以A或T结尾的密码子分别为28%和45%,而G或C的密码子分别为10%和15%。在许多其他叶绿体基因组中也观察到类似的现象[57.- - - - - -59.].
重复分析
菠萝叶绿体基因组共鉴定出35个重复结构,包括3个分散重复、8个回文重复和24个前向串联重复,最小长度为30 bp3.).根据Zhang等人的研究对重复序列进行了表征[60.],其中正向重复被分配成任一分散或串联取决于重复单元的位置。大多数重复了串联和LSC区域(特别是在基因间隔区)中发现而几分别在编码序列。其中包含编码区的重复是的ycf2,诗篇,PSAA,rpoC2,ycf1和rps11.尽管叶绿体基因在许多陆生植物中都是保守的,但编码区重复出现的情况可能有所不同。例如,包含重复的编码序列山茶花cp基因是infA,rps18,RPS3和rpoC2[61.而在曼陀罗stratomoniumCP基因,编码序列重复发生ndhF,ycf1,rps18,ycf2和PSAA[52.].
一个重复,大小39 bp,位于内含子ycf3在LSC和IRA区域有2倍回文重复,在IRB区域有1次分散的前向重复。最大的重复序列为基因间隔区分散重复序列,大小为72 bpPSBI./trnG-GCC和Psbe./petL这构成36碱基对的重复单元的大小发生两次。与此同时,rpoC2基因,该基因被称为是在许多叶绿体基因组高度发散区域[60.,62.],后来发现只含有菠萝CP基因组的单一重复。在相反,从禾本科多个叶绿体基因组已证明携带几套重复的,除了这个编码区大麦芽[63.].
简单重复序列(SSR)
简单序列重复(SSR)或微卫星是DNA小重复单元的延伸,同时存在于编码区和非编码区。由于其多态性和共显性遗传模式,这些DNA片段已被作为DNA标记用于群体遗传学研究和更多的研究。特别是,叶绿体SSRs被广泛用于计算植物基因型之间的遗传变异[64.- - - - - -67.].在菠萝叶绿体中鉴定出205个SSRsin-silico其中单核苷酸129例,二核苷酸59例,三核苷酸5例,四核苷酸12例。51个SSRs发生在复合地层中,该地层由多个SSRs组合组成,最大中断距离为100 bp [68.].最丰富的基序的单核苷酸A / T的运行时,由约61%。数略低于报道在以前的研究上菊类植物(68%)和单子叶(76%)[50.,69.].
在菠萝ssr编码区中发现48个,其中6个基因至少含有2个ssr。这些包括psbC, accD, cemA, petA, ycf2和ycf1.尽管叶绿体包含保守基因,但它们所携带的SSRs的数量和包含SSRs的编码序列是不同的。例如,在叶绿体中d .曼陀罗,只有五个基因(atpA,ycf3,accD,rbcL基因和clpP)所载SSRs [52.]而这些人在菠萝中发现的不同。SSR标记的编码区的存在(II型SSRS)引起了关注,因为它们容易发生变异,并在编码区的任何变化可导致帧偏移发生,并且使基因无功能[70].相比于IR区域,固态继电器分别在LSC和SSC区域更普遍,这是相干与其他基因组叶绿体[56.,69.,71.].此外,与其他叶绿体基因组比较,LSC和SSC还包含与其他叶绿体基因组序列差异较大的区域。菠萝叶绿体中所鉴定的SSRs的完整列表载于附加文件4.
品系分支叶绿体基因组的比较
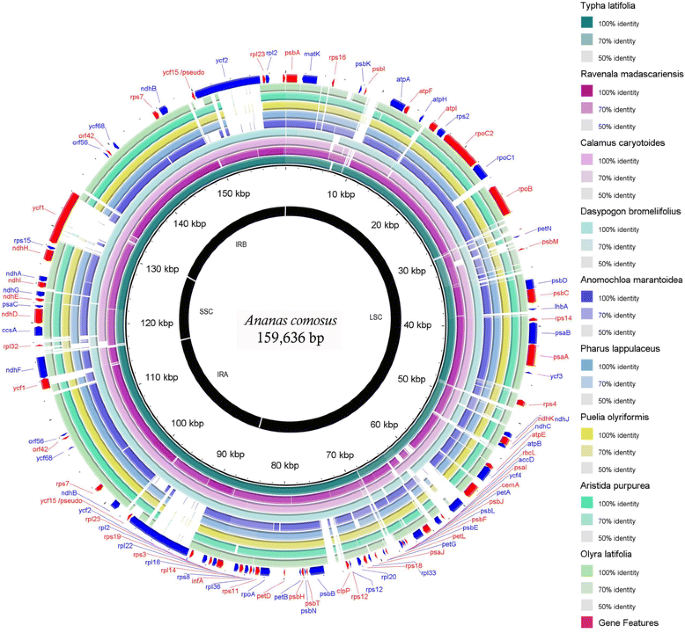
与叶片相比,九个叶绿体基因组代表亚类Commelinidae下的每个订单进行比较A. comosus(图。4).9个基因组的大小在136,785 ~ 166,170 bp之间O. Latifolia.是最小的r . madagascariensis分别最大的大小。与这一点相比A. comosus叶绿体基因组(159636碱基),所有其它的禾本目的是通过22,21,19,17和21 KB的小得多O. Latifolia.,答:紫竹,P. Olyriformis.,p . lappulaceus和答:marantoidea分别。同样地,D. bromoliifolius和c . caryotoides也分别比菠萝叶绿体更小1.7和2.3 kb。只有叶绿体基因组t . latifolia和r . madagascariensis分别比菠萝叶绿体大1.9 KB和6.5 KB。
菊苣属亚纲9个叶绿体基因组与菠萝叶绿体基因组的基因组比较。从第三圈开始:香蒲latifolia(绿色),Ravenala Madagascariensis.(紫色),菖蒲caryotoides(浅紫色),Dasypogon bromelifolius(土耳其石),Anomochloa marantoidea(蓝色),Pharus lappulaceus(浅蓝色),puelia oloriformis.(黄色),紫三芒草(绿色)和Olyra latifolia(浅绿色)。利用blast n对菠萝叶绿体基因组和叶绿体基因组进行两两比较,并根据相似度评分进行颜色编码:深色、浅色和灰色分别表示相似度评分在90%以上、80%以上和80%以下。第一个外环是基于菠萝叶绿体基因组定位的蛋白质编码基因特征
在菠萝叶绿体和鸭跖草类植物的其他代表之间的序列同一性的分析,结果表明叶绿体基因组这是丢失或被发散(小于80%的序列同一性)的一致的区域。此前的纪录表明,该Poacea家庭的几名成员都的部分缺失ycf1,accD和ycf2基因[53.[这使得其叶绿体基因组遭受较长的减少,而与致癌的其他成员相比。在与菠萝叶绿体相对比较中,部分基因丧失ycf1,accD和ycf2基因扩展到禾草类分支的所有成员(即不包括T.latifolia).对于非诗节的所有其他成员,该区域仍保持高于90%的均匀性(即,通过图3中的环颜色的亮度表示。4),除了r . madascariensis.该物种在与禾草属相同的基因丢失位点上表现出最小的基因间间隔缺失。然而,损失是最小的,所有的三个基因仍然完整。
总体而言,与菠萝相比,在IR区域中,在所有九个叶绿体基因组中具有最高相似性的区域,特别是在基因之间ycf1和ycf15,与外D. bromoliifolius.与他人的区域的前缀类似D. bromoliifolius具有高于90%的高序列相似性,并且仅在达到代际间隔物时RPS7-ycf68该值下降至90%以下。有趣的是,序列相似性改变D. bromoliifolius,答:marantoidea过小的缺失在那个特定的位点,但仍保持过这个红外区的高均匀性。在叶绿体中红外区的这种进化保守特性报道在其他一些研究[72.,73.]及Palmer [73.]提出,这是由于仅位于在IR区域中的rRNA基因的保守性质。在这项研究中,rRNA基因的保守性质也是鸭跖草亚纲的子类的所有成员中突出如图所示,在该区域附近的基因rpl2和rpl23.
与其他叶绿体基因组平行[74.]中,LSC和SSC地区忍受更高的分歧。最少保守区域是在基因的基因间隔之间rpoB和PSB.D.该地区有身份一直很低%归A. comosus叶绿体基因组的全部的九种比较。然而,所有的人仍抱有的PETN和PETM该区域包含的基因。相似的分化格局之间的区域rpoB和PSBD.还观察到浮萍的四个叶绿体中[75.].在一个不同的说明,该区域两侧由matK基因和atpA整个区域的基因相似性评分也很低。这对所有Poales家族来说都很重要D. bromoliifolius结果显示整个区域的序列一致性低于80%,而叶绿体c . caryotoides,r . madascariensis,和T. latofolia与叶绿体的相似性仍保持在80%以上A. comosus但随着基因间隔几个插入缺失。显而易见的是,热点发散跨物种在鸭跖草类植物的位置是相同的。显然,在鸭跖草类植物发散的图案,随后跨越顺序相同的图案,特别是在基因间间隔区,其中基因的损失是不损害细胞器的功能。这些高度不同的位点提供为生物学家解剖跨类群叶绿体发展变化的重要线索。
蛋白质编码鸭跖草类植物的基因序列多样性
为了进一步研究菠萝叶绿体基因组的分化模式,比较了80个蛋白编码基因序列,得出100个成员之间的平均两两序列距离(附加文件)5).结果表明,大多数基因的平均序列距离维持在较低水平。92%以上的基因在0.1以下,只有6个基因的平均序列距离值在0.1以上。这六个基因是ycf1,accD,rpl32.,rpl22.,matK基因和infA,按照发散性最大到最小的顺序排列。如表所示,所有这些基因都位于LSC或SSC的单拷贝区。这6个基因也被认为是小行星进化支中最分散的10个基因之一[50.)和其中的五个(除了rpl32.)17种维管束植物中被记录为最发散的,三七周效磺胺-乙胺嘧啶(56.].nie等。[57.表明基因的平均序列距离较高ycf1主要是由于菊类植物进化枝的成员之间的各种插入缺失的次数。从对齐的缺失/插入多态性(DIP封装)分析ycf1基因,197年indel事件录得100个征集。其他五个最分散的基因仅包含少于31的indel事件(数据未显示)。即使是ycf1已在禾草类中丢失,且含有许多可使基因无功能的吲哚,该基因已被证明在烟草中必不可少[76.].参与光系统I或II基因的多数人的低于0.05的低序列差异与例外PSBK,PSHH.和PSBM..这说明叶绿体维持了较低的序列分化率,以保持其基本功能。其他已经失去光合作用能力的寄生植物已经显示出失去了功能光合基因[77.,78.].然而,这是不正确的假设,高水平的序列差异中的任何基因将很快失去其功能,变成假。例如,matK基因众所周知,不仅具有高序列分歧,不仅在致癌中,而且在许多其他曲囊中。尽管它的分歧程度,产品的差异matK基因是至关重要的,因为它是叶绿体中唯一可以在含内含子基因中执行RNA剪接的II组内含子成熟酶[79.].该基因以某种方式能够适应高取代率,但仍保持保守的二级结构,这对其作为成熟酶的功能很重要[80].与此同时,PETN和PSBM.基因位于在其最相比较,菠萝CP基因组九个叶绿体基因组之间的分歧地区。然而,这两种基因的分别具有0.008和0.015,相对低的序列的距离。这表明,尽管周围的基因重排的许多事件中,细胞器仍然坚持认为,被视为重要基因的完整性。
在选择压力A. comosus叶绿体基因组
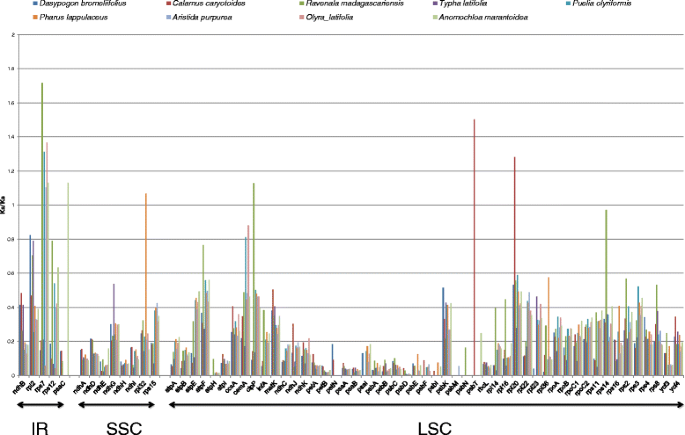
非同义(KA)同义速率比(由嘉/ Ks的表示)被用来评估基因序列之间并依次偏离率来确定正,中性或纯化的选择及其相对效果。的多于一个KA / Ks率表示阳性选择,而值小于一个指示纯化选择。的值0指示选择中性的存在。该比例在许多研究中用来检测的进化力量正在对某些组基因[中81.- - - - - -83.].
在本研究中,计算了所有9个叶绿体基因组中共有的64个蛋白编码基因的Ka/Ks比值。结果如图所示。5.计算值时,存在具有50和Na的Ka / Ks值的基因。当KS极低或者分别在对齐(即100%匹配)中没有替换时发生这种情况发生。对于这两种情况,50和Na的值改变为0.总的来说,在IR区域内的蛋白质编码基因的Ka / Ks的蛋白质编码基因的比例较高,并且SSC区域中的基因降低。然而,对于参与细胞色素B / F复杂生物发生的基因,观察到最低的Ka / ks值(PETA,PETB,和PETN),照相我(PSAA和诗篇)及光系统II (PSII) (psbA的,psbB,邮储银行,PSBD,psbE,PSBF,PSBI,PSBM和PSBT(除了。c . caryotoides)))。这种对Commelinids的所有100个叶绿体的Kimura校正的平均成对距离正确(如上述部分)作为对叶绿体功能至关重要的基因,具有最低的遗传分歧。在涉及照片系统II的基因中PSBK.与相同功能组的基因成员相比,具有较高的Ka/Ks比值。对该基因的功能研究表明PSBK.PSII的功能并不直接需要基因,但需要将其他对类囊体膜稳定重要的基因联系起来[84.].
9个叶绿体基因组中,蛋白质编码基因的Ka/Ks值最高RPS7尽管其平均两两序列散度(K2P)值很低。与菠萝相比,9个基因组中有5个的Ka/Ks值大于1,表明菠萝叶绿体中的基因发生了正向选择。为了进一步研究,将Ka/Ks比值的计算扩展到本系统发育研究中使用的所有100个品系(附加文件)6),并简化100个序列的计算RPS7基因,采用基于密码子的Z选择检验进行计算,检验正选择假设(无效假设:严格中性(dN = dS)) (dN/dS相当于Ka/Ks)。结果显示,有12个物种的非同义替换数显著增加(用dN)为P小于0.05。这些物种是Triticum aestivum.,Olyra latifolia,水稻粳稻,o . australiensis,非洲栽培稻,o .南方猛犸,o . nivira,白纹莲,黑斑莲,Danthonia加州,Ravenala Madagascariensis.,和Rhynchoryza subulata.总之,RPS7基因产物为小核糖体蛋白编码,它对核糖体的组装和稳定性至关重要。这种基因不仅存在于叶绿体中,也存在于细胞核和线粒体基因组中。该基因在叶绿体中的具体功能仍然有限,但对其翻译及其蛋白质相互作用的研究支持了其作为叶绿体翻译启动器的功能[85.- - - - - -87.].在许多叶绿体蛋白编码基因的比较研究中,距离值为RPS7基因序列以小为主,表明序列差异程度较低[88.,89.].尽管如此,高dN/d年代在[53.]它类似于我们的研究结果。结果提供了施加的选择性压力的指示RPS7在菠萝的叶绿体中。这提示在其他基因的整体基因组保存中,单个基因的非同义替换明显高于其他12个物种。
红外收缩与膨胀
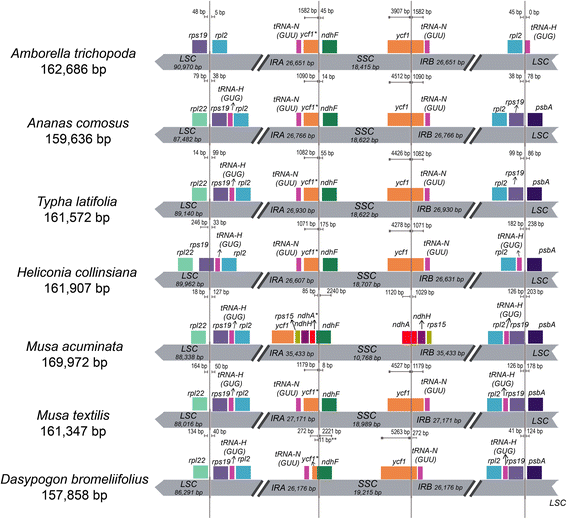
许多证据表明Amborella trichopoda是被子植物唯一幸存的姐妹系[90.].尽管有些人已经反驳了它在被子植物基部的地位[91.],物种仍充当自被子植物的发散的结构变化的研究的通用标准。在这项研究中,代表子鸭跖草亚纲的相同的9个叶绿体基因组相比,叶绿体A. trichopoda来研究反向重复区域边界的扩展(图。6).与…相比A. trichopoda的叶绿体序列A. comosus和t . latifolia没有显示,只是包含的任何重大的扩张rps19基因和trnH-GUGtRNA在爱尔兰共和军/LSC边境。Martin等人也对其他类群成员进行了类似的观察[92.].爱尔兰共和军地区没有独特的扩张或收缩D. Bromeliifolius.,这是基础大部分鸭跖草类植物的进化枝(基于[29.和本研究中观察到的一样)。然而,部分纳入ycf1基因进入IRB区域的大小只有285 bp,使其明显小于T. Latifolius.和A. comosus其中包括1082 bp和1146 bpycf1基因分别进入同一区域。
在香蕉的叶绿体中Musa Acuminata.),IR的主要扩展报道[92.]和IR扩展到包括三个另外的基因(RPS15,NDHH.和ycf1)和的部分序列ndhA.这种扩展在任何其他单子叶植物中都没有观察到,即使是在同一属内,穆萨textilis只有一个扩展类似于在A. comosus.同样,它的姐妹类群,海里康属植物collinsiana(属于同一Zingiberales科)在IR/SSC边界也没有重大的扩展。主要的扩展看到M. accuminata似乎是该物种独有的。然而,在一定程度上,草科也表现出适度的IR边界扩展ycf1和rps15基因和部分序列ndhH其他地方报道的基因[63.,93.,94.].一般来说,在被子植物中观察到多个IR扩张事件,这些事件被认为是在多个步骤中发生的,并且彼此独立[92.,95.].总体而言,它可以推断出,IR区域的膨胀和收缩A. comosus和其他鸭跖草类植物还是因为被子植物的发散稳定,与例外的单一品种姜目.
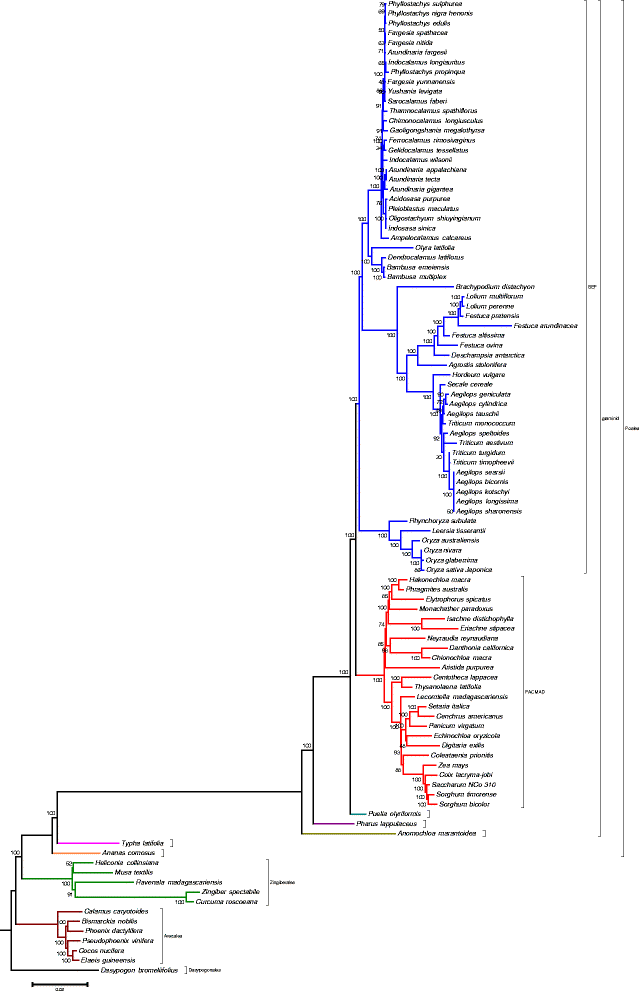
系统发育分析
利用NCBI细胞器基因组资源中包括菠萝叶绿体在内的100个品系叶绿体共有的56个蛋白编码基因进行系统发育分析。RAxML的最大似然(ML)分析产生了-lnL为400419.797049的树。1000次重复的Bootstrap分析显示,100个节点中有55个节点的值为100%,只有5个节点的值低于50%(图5)。7).树建议获得正确的系统发育推断作为树的拓扑结构遵循了最新的被子植物种系发生学组III [中9].A. comosus是凤梨科的唯一代表,是形成最早的分支,导致Poales从其他成员的凤梨科分裂。该树还支持槟榔属(arerecales)和姜科(Zingiberales)的系统发育,如Barret等人所描述的那样,它们是Dasypogonaceae的单系植物,而姜科(Zingiberales)是Poales的单系植物,具有100%的bootstrap值。29.].在这项研究中,只有56通用于所有100类群蛋白质编码基因在分析中使用,相对于83组中蛋白质编码基因的[29.].尽管在分析中使用的基因集较少,但生成的树能够验证先前研究中获得的commelinid亚类的家族之间的关系,具有鲁棒性支持。在树中,树枝的长度为t . latifolia是超过A. comosus,表明在t . latifolia.这用于为表明如菠萝表示的凤梨科家族是在基底的禾本目次序的最。与其他研究一致,该Poacea家庭显示支长,因为它从其他禾本目成员,但具有较短的内部分支分歧[29.,49.].在禾本目的长枝表示,禾本目的graminids经历更快质序列进化的速率相比,子鸭跖草亚纲的其他成员。
结论
我们成功地组装、注释和分析了MD-2杂交菠萝(A. comosus).叶绿体基因组是一个很有价值的例子,说明了大规模重复的存在是如何导致装配错误的,并将对装配过程造成相当大的挑战新创组件的基因组。龙读取,类似于由PacBio产生将有助于在装配复杂的基因组中,只要读取比重复更长。但是,与错误率的限制,所要求的误差校正模块将减少长序列的长度读取并因此将限制序列的电位读来解决长重复。也许,用于纠错的新算法可以调解面临在这项研究中长度缩短的问题。相对于早期的被子植物a . trichopoda菠萝叶绿体基因组仍然高度保守,被发现与它的姐妹分类单元非常相似,t . latifolia.通过对56个叶绿体基因数据集的分析,证实了其系统发育为棕榈目,Dasypogonaceae,姜目,鸭跖草目和禾本目坚固的支持。菠萝叶绿体基因组的可用性将作为溴脲对其他单胶质或高管的比较研究的有价值的参考,最重要的是它将使其他分子生物学应用如叶绿体基因转化可行。
可用性支持数据
MD-2菠萝叶绿体全基因组已提交到GenBank (accession KR336549)。用于系统发育分析的核酸矩阵和树可在TreeBASE网站的此链接找到http://purl.org/phylo/treebase/phylows/study/TB2:S18029?x-accesscode=2a8e85af14b2eb2378b3d6c838f1fa90&format=html.分析中使用的其他数据包括在文章及其附加文件中。
缩写
- cp:
-
叶绿体
- LSC:
-
大单拷贝
- SSC:
-
小单副本
- 红外光谱:
-
反向重复
- 伊拉:
-
倒置重复A.
- IRB:
-
倒置重复B.
- RSCU:
-
相对同义词密码子用法
- 苏维埃社会主义共和国:
-
简单序列重复
- 门店:
-
新一代测序
参考文献
- 1。
莱亚尔楼Coppens的D'Eeckenbrugge G.菠萝。在:果树育种,第1卷树TROP水果,第一卷。1. 1996年第515-57。
- 2。
营养和健康的百科全书。纽约州:Facts On File, Inc;1981.p。98。
- 3.
莫顿摩根富林明。菠萝。适合:气候温暖的水果。佛罗里达州迈阿密:Creative Resource Systems, Inc;1987.18-28页。
- 4。
路德他。凤梨二项式的字母列表。2010.
- 5。
Hamdan N,Samad Aa,Haydat T,Salleh FM。使用氯塑料标记物(RBCL基因)八个马来西亚菠萝品种的系统发育分析。J Teknol。2013; 2:29-33。
- 6。
杨志强,王志强,王志强,等。凤梨科凤梨亚属植物叶绿体和核序列的系统发育与进化。植物生态学报。2014;30:1591 - 614。
- 7。
排名TA, Soltis DE, Soltis PS, Gilmartin AJ。凤梨科亚家族系统发育关系:来自叶绿体DNA限制位点变异的证据。系统机器人。1990;15:425-34。
- 8。
王志强,王志强,王志强,等。褐飞虱的系统发育分析。(凤梨科皮特凯恩亚科)基于四个叶绿体DNA区域。分子系统学进展。2009;51:472-85。
- 9。
Bremer B, Bremer K, Chase MW, Fay MF, Reveal JL, Bailey LH等。开花植物目和科被子植物系统发育类群的更新:APG III。中国科学(d辑:地球科学)2009;
- 10。
Bedbrook JR,Bogorad L.核酸内切酶识别位点映射到玉米叶绿体DNA。国家科学院院刊美A. 1976; 73:4309-13。
- 11.
Kumano M.烟草克隆群(烟草竹岩)叶绿体基因组作为一组重叠限制性内切核酸酶片段:11核糖结构蛋白基因的映射。植物SCI。1986; 44:211-6。
- 12.
张志强,张志强,张志强,等。浅绿藻叶绿体DNA的完整序列。核酸Res. 1993; 21:3537-44。
- 13。
等。水稻(Oryza sativa)叶绿体基因组的完整序列:不同tRNA基因之间的分子间重组解释了谷物进化过程中主要的质体DNA倒置。MGG Mol Gen Genet, 1989; 217:185-94。
- 14。
Ohyama K,Yamano Y,Fukuzawa H,Komano T,Yamagishi H.来自Liverwort Markantia多晶型L.细胞悬浮培养物的叶绿体DNA的物理映射。Mol Gen Genet。1983; 189:1-9。
- 15.
Cronn R,Liston A,Parks M,Gernandt DS,Shen R,Mockler T.使用SOLEXA测序技术的植物叶绿体基因组的多重测序。核酸RES。2008; 36。
- 16。
凯恩N,Sveinsson的S,Dempewolf H,羊JY,张d,恩格斯JMM,等。使用全基因组叶绿体和核核糖体DNA超条形码在可可(可可属;锦葵科)。牛J博特。2012; 99:320-9。
- 17。
McPherson H, van der Merwe M, Delaney SK, Edwards MA, Henry RJ, McIntosh E, et al.;为分子生态学研究捕捉叶绿体变异:应用于雨林树的下一代简单测序方法。BMC生态。2013;13:。
- 18。
Whittall JB,Syring J,Parks M,Buenrostro J,Dick C,Liston A等人。在大海捞针中找到(松)针:罕见和广泛的松树中的叶绿体基因组序列分歧。Mol Ecol。2010; 19:100-14。
- 19。
刘艳,霍宁,董丽,王艳,张树华,杨夏,等。蒙药冷蒿叶绿体全基因组序列及其与其他植物的系统发育关系。《公共科学图书馆•综合》。2013;8:e57533。
- 20.
陈X,李琦,李义,钱j,汉族。Aconitum barbatum var的叶绿体基因组。Puberulum(ranunculaceae)从CCS派生使用PACBIO RS平台读取。前植物SCI。2015; 6:42。
- 21.
李强,李勇,宋军,徐华,徐军,朱勇,等。利用SMRT循环共识测序策略实现叶绿体基因组的高精度从头组装和SNP检测。新植醇。2014;9:1041-9。
- 22.
吴志,桂胜,全志,潘磊,王胜,柯伟,等。利用Sanger、Illumina MiSeq和PacBio RS II测序平台对莲藕(Nelumbo nucifera, Nelumbonaceae)叶绿体基因组进行了精确的评估:深入了解基础真核生物的质体进化。BMC Plant Biol. 2014;14:289。
- 23.
等。PacBio RS平台对叶绿体基因组测序和从头组装的评估。BMC基因组学。2013;14:670。
- 24.
巴希尔A,Klammer AA,罗宾斯WP,展C-S,韦伯斯特d,Paxinos E,等人。用于细菌基因组的自动整理混合方法。NAT BIOTECHNOL。2012; 30:701-7。
- 25.
Chin C-S,Alexander DH,Marks P,Klammer AA,Drake J,Heiner C等人。非血小,完成的微生物基因组组件来自长读SMRT测序数据。NAT方法。2013; 10(6):563-9。
- 26.
Hoefler BC,Konganti K,直PD。De Novo组装streptomyces sp。使用PACBIO单分子测序的菌株MG1基因组。基因组公告。2013; 1:1-2。
- 27。
Chase MW,Duvall Mr,Hill,Conran JG,Cox Av,Eguiarte Le等。Lilianae的分子母灌注。在:rudall pj,cribb pj,cutler df,humphries cj,编辑。单圈子系统系统中的evol。凯威:皇家植物园;1995. p。109-37。
- 28。
Dahlgren RMT,Clifford HT,Yeo P.单子圈的家庭。柏林海德堡:斯普林克;1985. p。550。
- 29。
Barrett CF,Davis Ji,Leebens-Mack J,Conran JG,Stevenson DW。血肿单子宫植物植物血浆基因组和深层关系。克莱斯。2013; 29:65-87。
- 30.
Carlier JD, Reis A, Duval MF, D 'Eeckenbrugge GC, Leitao JM。利用拟试验杂交的方法绘制了小苞蕨和山梨的RAPD、AFLP和ISSR标记的遗传图谱。植物繁殖。2004;192:186 - 92。
- 31。
delaporta SL, Wood J, Hicks JB。植物DNA迷你制剂:版本二。Exp Protoc。1983;1:19-21。
- 32。
吴泰,瓦丹甲凯克。GMAP:MRNA和EST序列的基因组映射和对准程序。生物信息学。2005; 21:1859-75。
- 33。
Chevreux B,PFISTERER T,Drescher的B,Driesel AJ,穆勒WEG,湿润剂T,等人。使用miraEST汇编可靠和自动化的mRNA转录组装和SNP检测测序的EST。Genome Res。2004; 14:1147年至1159年。
- 34。
使用基本局部序列与逐次细化(BLASR)绘制单分子测序reads:应用和理论。BMC生物信息学。2012;13。
- 35。
黄伟文SK,扬森RK,Boore JL。与教条细胞器基因组的自动标注。生物信息学。2004; 20:3252-5。
- 36。
Schattner P, Brooks AN, Lowe TM。tRNAscan-SE、snoscan和snoGPS网络服务器用于检测tRNAs和snoRNAs。核酸Res. 2005;33(Web服务器版):W686-9。
- 37。
Kurtz S,Choudhuri JV,Ohlebusch E,Schleiermacher C,Stoye J,Giegerich R.声誉换句者:重复分析对基因组规模的歧管应用。核酸RES。2001; 29:4633-42。
- 38。
酵母密码子的使用:聚类分析清楚地区分高表达和低表达基因。核酸Res. 1986; 14:5125-43。
- 39。
DAMBE5:一个用于分子生物学和进化数据分析的综合性软件包。生物学杂志。2013;30:1720-8。
- 40.
Lohse M, Drechsel O, Kahlau S, Bock R. OrganellarGenomeDRAW——一套用于生成质体和线粒体基因组物理图和可视化表达数据集的工具。核酸Res. 2013;41(Web Server issue): W575-81。
- 41.
Alikhan N-F,Petty NK,Ben Zakour Nl,Beatson SA。爆炸环图像发生器(BRIG):简单的原基基因组比较。BMC基因组学。2011; 12:402。
- 42.
Carver TJ, Rutherford KM, Berriman M, Rajandream MA, Barrell BG, Parkhill J. ACT: Artemis比较工具。生物信息学。2005;21:3422-3。
- 43.
加藤K,斯坦德利DM。MAFFT多序列比对软件版本7:在性能和可用性方面的改进。mol Biol Evol。2013; 30:772-80。
- 44.
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析版本6.0。生物学杂志。2013;30:2725-9。
- 45.
王东,张勇,张震,朱建军,于建军。kachs_calculator 2.0:一个集成了伽玛级数方法和滑动窗口策略的工具箱。基因组学,蛋白质组学,生物信息学。2010;8:77-80。
- 46.
jModelTest 2:更多的模型,新的启发式和并行计算。Nat方法。2012;9:772。
- 47.
Stamatakis a . RAxML版本8:系统发育分析和大系统发育后期分析的工具。生物信息学。2014;30:1312-3。
- 48.
基于CIPRES的大型系统发育树推断的科学门户。参见:2010 Gatew computing Environ Work GCE 2010。2010.
- 49.
Givnish TJ, Ames M, McNeal JR, McKain MR, Steele PR, dePamphilis CW等。单子叶树的组装:Poales的质体序列系统发育与进化1。安密苏里Bot Gard. 2010; 97:584-616。
- 50。
钱建军,宋建军,高华,朱勇,徐建军,庞旭,等。药用植物丹参叶绿体基因组的完整序列。《公共科学图书馆•综合》。2013;8。
- 51。
杨建斌,唐敏,李海涛,张志荣,李德志。大花蕙兰属的完整叶绿体基因组:物种鉴定、系统发育和种群遗传分析。BMC Evol Biol. 2013;13:84。
- 52。
杨Y,原野d,清L,金健L,陈锡文L,有毒药用植物曼陀罗溢滔W.完整的叶绿体基因组序列:组织和基因工程的影响。Plos一个。2014; 9。
- 53。
Guisinger MM, Chumley TW, Kuehl JV, Boore JL, Jansen RK。typha (typhaceae, poales)质体基因组序列对了解类人猿基因组进化的意义。J Mol Evol. 2010; 70:149-66。
- 54。
杨敏,张旭,刘刚,尹勇,陈坤,云强,等。枣树叶绿体全基因组序列。《公共科学图书馆•综合》。2010;5:1-14。
- 55。
引用本文:Kato T, Kaneko T, Sato S, Nakamura Y, Tabata S.豆科植物Lotus japonicus叶绿体基因组的完整结构。DNA研究》2000;7:323-30。
- 56。
金志强,李志强。人参叶绿体全基因组序列及17种维管植物序列进化的比较分析。DNA研究》2004;261:247 - 61。
- 57。
聂旭,吕胜,张勇,杜欣,王磊,等。主要入侵种紫茎泽兰的叶绿体全基因组序列。《公共科学图书馆•综合》。2012;7。
- 58。
唐法特sornruang S, Sangsrakru D, Chanprasert J, Uthaipaisanwong P, Yoocha T, Jomchai N, et al.;绿豆叶绿体基因组序列(豇豆属辐射动物)的高通量焦磷酸测序:结构组织和系统发育关系。DNA Res. 2010;17(December 2009): 11-22。
- 59。
Yi DK, Kim KJ。3 .种子作物种子(Sesamum indicum L. PLoS One)叶绿体全基因组序列分析。
- 60.
张永军,马鹏飞,李德志。6个竹子叶绿体基因组的高通量测序:温带木本竹子(pousoideae)的系统发育意义。《公共科学图书馆•综合》。2011;6。
- 61.
杨杰,杨谢,李HT,杨杰,李迪兹。山茶花种类的比较叶绿体基因组。Plos一个。2013; 8:1-12。
- 62.
Samson N, Bausher MG, Lee S, Jansen RK。咖啡(Coffea arabica L.)叶绿体基因组的完整核苷酸序列:生物技术和被子植物中系统发育关系的组织和意义。acta botanica yunnanica(云南植物研究);2012;
- 63.
李淑娟,李淑娟,李淑娟,等。普通大麦、高粱和匍匐茎Agrostis的叶绿体全基因组序列,以及与其他禾草基因组的比较分析。Theor Appl Genet. 2007; 119:571 - 90。
- 64.
Deguilloux M-F,Pemonge M-H,佩蒂特RJ。使用叶绿体微区分橡木人群。安脊髓损伤。2004; 61:825-30。
- 65.
Vendramin GG, Degen B, Petit RJ, Anzidei M, Madaghiele A, Ziegenhagen B.欧洲冷杉叶绿体微卫星位点的高水平变异。摩尔生态。1999;8:1117-26。
- 66.
高杜·M,吉拉德·T, Kiss L, Shykoff JA。核微卫星和叶绿体微卫星显示,豚草(豚草)在世界范围内的入侵历史中多次引入。《公共科学图书馆•综合》。2011;6 (3):e17658。
- 67.
尼泊尔议员Piya S核和叶绿体微卫星标记的特征Falcaria寻常(伞形科)。植物学报,2013;04(3):590-5。
- 68.
von Stackelberg M,Rensing SA,Reski R.鉴定基因莫斯SSR标记的鉴定和二十四个藻类和植物基因指数的比较分析揭示了物种特异性而不是微卫星的群体特征。BMC植物BIOL。2006; 6:9。
- 69.
Huotari T,Korpelainen H.完全叶绿体基因组序列伊乐藻属植物黄花和比较分析与其它单子叶质体基因组。基因。2012; 508:96-105。
- 70.
王美利,巴克利NA,詹金斯TM。植物和昆虫中的微卫星标记。第一部分:生物技术的应用。基因。基因组基因。2009;3:54 - 67。
- 71.
易d-K,李H-L,阳光B-Y,钟MY,K金-J。的完整的叶绿体DNA序列Eleutherococcus senticosus.(五茄科);与其他三颗小行星的比较进化分析。摩尔细胞。2012;33:497 - 508。
- 72.
Curtis SE,CLEGG MT。叶绿体DNA序列的分子演变。mol Biol Evol。1984; 1:291-301。
- 73。
帕尔默JD。叶绿体基因组的比较组织。Annu启遗传学。1985; 19:325-54。
- 74。
山根K,矢野K,川原T.模式和甘蔗,玉米和大米从整个叶绿体基因间区域推断插入缺失的进化速度。DNA水库。2006; 13:197-204。
- 75。
王伟,王文杰。浮萍亚科叶绿体基因组的高通量测序。《公共科学图书馆•综合》。2011;6。
- 76。
Drescher的A,斯蒂芬妮R,Calsa T,CARRER H,博克R的高等植物的两个最大的叶绿体基因组编码的开放阅读框是必需基因。植物杂志2000; 22:97-104。
- 77。
张永强,张永强,张永强,等。帚形油菜科光合和非光合寄生植物功能性和物理基因组减少的机制。植物细胞。2013;25(10):3711 - 25。
- 78。
质体基因在被寄生的过程中丢失。植物细胞。2013;25(10):3636。
- 79。
Hausner G, Olson R, Simon D, Johnson I, Sanders ER, Karol KG,等。叶绿体trnK (matK)内含子的起源与进化:II组内含子RNA结构进化的模型。生物学报2006;23:38 - 91。
- 80.
Barthet MM,Hilu KW。上迅速变化的基因评估进化限制使用的蛋白质组合物MATK。Ĵ分子EVOL。2008; 66:85-97。
- 81.
张鹏,顾震,李卫华。人类基因组中年轻复制基因之间的不同进化模式。基因组医学杂志。2003;4:R56。
- 82.
Gimelbrant AA,Skaletsky H,对由于人类黑猩猩差异的嗅觉受体库象棋A.选择压力。国家科学院院刊美A. 2004; 101:9019-22。
- 83.
Brunet FG, Crollius HR, Paris M, Aury JM, Gibert P, Jaillon O, et al.;硬骨鱼全基因组复制后的基因丢失和进化率。生物学报2006;23:1808-16。
- 84.
Iwai M,Suzuki T,Kamiyama A,Sakurai I,Dohmae N,Inoue Y等。PSBK亚基需要在嗜热性蓝杆菌热梭中PSII复合物中的其他小亚基的稳定组装和稳定性所必需的。植物细胞生理。2010; 51:554-60。
- 85.
罗伯逊D,博因顿JE,吉勒姆西北。编码CF1/CF0 epsilon亚基的野生型叶绿体atpE基因与rps7基因的3 '半位点共转录莱茵衣藻atpE的移码突变特征。MGG Mol Gen Genet, 1990; 221:155-63。
- 86.
Fargo DC,Zhang M,Gillham NW,Boynton Je。叶绿体MRNA的翻译不需要Shine-Dalgarno样序列莱茵衣藻叶绿体或大肠杆菌。孟根。1998;257:271-82。
- 87.
Fargo DC,Boynton Je,Gillham NW。衣原体的叶绿体核糖体蛋白S7与叶绿体mRNA领导序列结合,并且可以参与翻译引发。植物细胞。2001; 13(1月):207-18。
- 88.
ku c,胡锦涛,kuo ch。完全塑体基因组序列的基础Asterid管理polysticta进行筛选。小行星质体基因组的比较分析。《公共科学图书馆•综合》。2013;8 (4):e62548。
- 89.
罗Ĵ,侯BW,牛ZT,刘伟,薛QY,丁XY。光合兰花的比较基因组叶绿体:分析上市公司兰科植物进化和系统发育应用分子标记的开发。Plos一个。2014; 9。
- 90.
Jansen RK, Cai Z, Raubeson LA, Daniell H, Claude W, Leebens-mack J,等。通过对64个质体基因组中的81个基因的分析,揭示了被子植物的亲缘关系,并确定了基因组规模的进化模式。中国科学院院刊2007;104:19369-74。
- 91.
Goremykin VV,赫希恩斯特KI,WölflS,海尔威格FH。分析对Amborella trichopoda叶绿体基因组序列表明无伞草不是基部被子植物。生物化学学报2003;20:1499-505。
- 92.
马丁·G, Baurens FC, Cardi C, D 'Hont A, Aury JM。香蕉的完整叶绿体基因组(Musa Acuminata.,姜类):洞察质单子叶植物进化。Plos一个。2013; 8,e67350。
- 93.
荻原Y,矶野K,小岛T,远藤A,花冈男,椎名T,等人。通过结构叶绿体DNA的完整测序揭示了小麦质体的功能。分子遗传学基因组学。2001; 266:740-6。
- 94.
Bortiri E,Cooleman-Derr D,Lazo Gr,安德森od,顾yq。完全叶绿体基因组序列Brachypodium distachyon: 8个草质体的序列比较和系统发育分析。BMC Res Notes. 2008;1:61。
- 95.
Mardanov AV, Ravin NV, Kuznetsov BB, Samigullin TH, Antonov AS, Kolganova TV等。浮萍的完整序列(浮萍属小)叶绿体基因组:与其他被子植物的结构组成和系统发育关系。中国科学(d辑:地球科学)2008;
- 96.
Kurtz S, Phillippy A, Delcher AL, Smoot M, Shumway M, Antonescu C,等。通用和开放的软件比较大基因组。基因组医学杂志。2004;5。
致谢
我们感谢Hydayaty Yusoff和马来西亚菠萝工业委员会提供的菠萝样本,太平洋生物科学(亚太)的Caroline Chan和TreeCode Sdn Bhd的Dana Chow对太平洋生物科学RSII的帮助。本项目由马来西亚教育部和马来西亚科学技术创新部资助,分别获得基础研究资助计划(FRG0319-SG-2013)和科学基金(SCF0087-BIO-2013)资助。
作者信息
从属关系
通讯作者
额外的信息
相互竞争的利益
提交人声明他们没有竞争利益。
作者的贡献
SVK发起并构思了这项研究,并参与了协调工作。RMR设计并执行了大部分实验,并起草了手稿。RMR和AS执行和分析数据,并帮助起草和修改手稿。SVK, RMR, AS对手稿进行了修改。所有作者阅读并批准了最终的手稿。
这篇文章的勘误表载于http://dx.doi.org/10.1186/s12870-015-0619-x.
附加文件
附加文件1:
绿色植物叶绿体的登录数。绿色植物(Viridiplantae)叶绿体的接入数,用于引诱菠萝总基因组DNA PacBio测序读取叶绿体。(DOCX 35 KB)
附加文件2:
最终核酸对准在系统发育分析使用。从子鸭跖草类植物所有可用的叶绿体基因八十使用MAFFT对准工具对准并用于推断其系统发育。提供的文件以Fasta格式为单位。(TXT 2634 KB)
附加文件3:
菠萝叶绿体基因组的PCR验证。使用PacBio序列读取的Quiver工具校正的三个区域使用基于pcr的测序进行了验证。验证读取序列到叶绿体的比对文件以FASTA格式提供。破折号表示Sanger序列读取到引用之间的间隙ndhB位基因。(TXT 6 KB)
附加文件4:
菠萝叶绿体中所鉴定的SSRs的完整列表。SSRS识别使用MISAA. comosus叶绿体基因组。(DOCX 38 KB)
附加文件5:
在从子类鸭跖草类植物80蛋白编码基因的序列平均进化分歧的估计。标准误差估计(s)显示在第二列,并通过bootstrap程序获得(1000次重复)。使用Kimura 2参数模型进行分析[1].分析涉及100个核苷酸序列。密码子位置为1 + 2 + 3 +非编码。所有现场覆盖率低于95%的职位均被淘汰。也就是说,在任何位置都允许少于5%的对齐间隙、缺失的数据和不明确的基底。最终数据集中总共有465个职位。在MEGA6中进行了进化分析。(多克斯29 kb)
额外的文件6:
为阳性选择的密码子Z检验RPS7从100种亚类鸭跖草类植物的包括基因A. comosus.的probability of accepting alternative hypothesis of positive selection (dN > dS) is shown (below diagonal). Values of P less than 0.05 are considered significant at the 5 % level and are highlighted. The test statistic (dN - dS) is shown above the diagonal. dS and dN are the numbers of synonymous and nonsynonymous substitutions per site, respectively. The variance of the difference was computed using the analytical method. Analyses were conducted using the Nei-Gojobori method. The analysis involved 100 nucleotide sequences. All ambiguous positions were removed for each sequence pair. There were a total of 196 positions in the final dataset. Evolutionary analyses were conducted in MEGA6. (XLS 166 kb)
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
MD-2菠萝叶绿体全基因组序列及其与其他9种植物的比较分析。BMC植物BIOL.15,196(2015)。https://doi.org/10.1186/s12870-015-0587-1
收到:
接受:
发表:
关键字
- 叶绿体基因组
- 相对同义密码子使用
- 大单拷贝
- 反向重复区域
- 完整的叶绿体基因组