摘要
背景
苄基异喹啉生物碱(BIAs)是一类具有共同生物合成起源的植物特化代谢物,从酪氨酸开始。许多偏丁酸具有强大的药理活性,积累了它们的植物在传统医学和文化实践中有着悠久的使用历史。几十年来,人们一直专注于选择一些植物物种作为模型系统,这使得人们能够接近或完全阐明主要的BIA通路,包括吗啡、血碱和小檗碱的通路。然而,这种关注导致了对非模式物种的知识缺乏,这些物种也被认为积累了广泛的BIAs,但迄今为止其生物合成完全未被探索。此外,这些非模式物种代表了丰富的催化剂多样性来源,对植物生物化学家和新兴的合成生物学工作有价值。
结果
为了研究非模式植物积累偏倚的遗传多样性,我们选取了毛茛科4科20种。从每个物种中提取的RNA通过1)Roche GS-FLX Titanium和2)Illumina GA/HiSeq平台进行处理分析,共生成40个深度测序转录组文库。新创组装、注释和随后的全长编码序列(CDS)预测表明,使用illumina平台的大多数物种都取得了更大的成功。每个转录组的组装数据被存入基于web的BLAST门户网站(www.phytometasyn.ca)供公众查阅。利用bia -生物合成酶作为查询,基于同源性的文库挖掘获得了大约850个可能涉及生物碱生物合成的候选基因。使用库间FPKM归一化方法对这些候选基因进行表达分析。这些表达数据为合理选择候选基因提供了依据,并提示了BIA代谢中可能存在的代谢瓶颈。对15种不同的酶/蛋白质组进行了系统发育分析,突出了许多可能参与一种或多种生物碱类型形成的新基因,包括morphinan, aporphine和phthalideiso喹啉生物碱。转录组资源用于设计和执行候选案例研究N-甲基转移酶(NMTs)Glaucium flavum,揭示了预测的和新的酶活性。
结论
这项研究为分离和发现BIA代谢中的1)功能同源物和2)全新催化剂提供了必要的资源。功能分析g . flavumNMTs证明了这一资源的效用,并强调了经验确定拟议的酶功能的重要性。尽管在这些植物中发现了丰富的生物活性生物碱及其对传统医学的重要性,但本报告中包括的大多数物种之前都没有公开可获得的、完全注释的、blast可访问的转录组。本文的结果提供了必要的序列信息,并为进一步阐明BIA代谢的实验设计提供了依据。
背景
苄基异喹啉生物碱(BIAs)是一类不同种类的植物特化代谢物,包括大约2500种已知化合物。虽然BIAs呈现出广泛的结构主干排列,但它们在共同的生物合成起源上是统一的,这始于两个酪氨酸衍生物的缩合,形成第一个专用的BIA, (年代) -norcoclaurine(无花果。1).人类最古老的几种药物、毒药、狩猎辅助工具和仪式制剂都来自于植物积累的偏见,在旧世界和新世界的文化中都有发现[17].值得注意的积累bia的植物包括吗啡、可待因和积累noscapine的罂粟(果实)、积聚小檗素的小檗(小檗属植物)属,日本金线线(黄连粳稻)、meadowrue (Thalictrum flavum),以及产生抗微生物物质血素的品种,例如墨西哥花椒(Argemone墨西哥)及加州罂粟(Eschscholzia californica).这些植物形成了过去几十年广泛研究的模式物种的核心群,导致在生化和分子遗传水平上的主要途径几乎完全阐明。大多数或所有负责罂粟碱、吗啡、血碱、小檗碱和诺卡平生物合成的酶已被克隆和鉴定(图。1) [6,17].有限数量的酶家族与BIA代谢有关,这可能反映了该途径的单系起源[34].这一特性使基于同源性的酶发现策略成为可能,其中预测作用于BIA代谢网络中未解决点的酶类型。例如,碳碳或切断偶联反应几乎完全由与CYP80、CYP82或CYP719家族或2-氧戊二酸盐/Fe同源的细胞色素P450催化2 +端依赖加双氧酶。通过基于同源性的转录组资源查询以及靶向代谢物分析,血碱和诺scapine代谢中先前未被描述的步骤得以解决[1,6,7].这种方法最近被用于发现二氢血红素二苯甲苯胺氧化酶(DBOX),这是一种fad依赖的氧化酶,与小檗碱桥酶(BBE)同源[15].在BIA代谢中反复发现的其他酶类型包括O- - -N-甲基转移酶,BAHD酰化酶[5]和属于醛酮(AKR)的还原酶[39]或短链脱氢酶/还原酶[23总科。只有BIA生物合成的第一步是由一个独特的蛋白质家族,与致病相关的10 (PR10)/Bet v1过敏原催化的,否则在生物碱代谢中不存在(即。nc;(年代) -norcoclaurine合成酶)。尽管如此,NCS的同源物似乎在不同的植物类群中发挥了关键的入口作用[27].
BIA生物合成的主要途径为(年代)-网纹碱(淡粉色),罂粟碱(黄色),吗啡(绿色),血碱(橙色),小檗碱(蓝色)和诺卡平(紫色)。切断而且碳碳偶联反应分别为贝巴木宁(橄榄色)和棒状结核素(深粉色)。每个生物碱内的红色突出了酶催化的结构变化。实心箭头和虚线箭头分别代表单酶和多酶催化的反应。蓝色字体的酶已经在分子水平上被鉴定,而黑色字体的酶还没有被克隆。缩写:3'-OHase, 3'-羟化酶;3 'omt 3 ' -O甲基转移酶;3 ohase, 3-hydroxylase;4HPPDC, 4-羟基苯丙酮酸脱羧酶;4 'omt, 3 '羟基-N-methylcoclaurine 4 ' -O甲基转移酶;6OMT,去甲磺酸6-O甲基转移酶;AT1 1 13-dihydroxy -N-methylcanadine 13 -O乙酰转移酶;BBE,小檗碱桥酶;BS,贝巴木宁合成酶;CAS,加拿大碱合成酶;CFS,雪兰花啉合成酶;CNMT, coclaurineN -甲基转移酶;CODM,可待因O-demethylase;CoOMT, columbamineO甲基转移酶;COR,可待因酮还原酶;CTS,棒状结核合酶;CYP82X1 1-hydroxy-13 -O乙酰-N-methylcanadine 8-hydroxylase;CYP82X2 1-hydroxy -N-methylcanadine 13-hydroxylase;CYP82Y1,N-methylcanadine 1-hydroxylase;CDBOX,二氢苯并苯胺氧化酶;CXE1, 3 -O-acetylpapaveroxine羧酸酯酶;MSH,N -methylstylopine羟化酶;N7OMT,去甲网状7-O甲基转移酶;NCS,去甲磺酸合酶;NMCanH,N -methylcanadine 1-hydroxylase;NMCH,N-methylcoclaurine 3 ' -羟化酶;NOS,诺scapine合成酶;P6H,原氨酸6-羟化酶;REPI,网状外素酶;SalAT, salutaridinool 7-O -乙酰转移酶;SalR,有益马碱还原酶;SalSyn,敬礼碱合成酶;血氨酸还原酶(SanR);some, scoulerine 9-O甲基转移酶;SPS, stylopine合成酶;STOX (年代) -tetrahydroprotoberberine氧化酶;T6ODM, thebaine 6-O-demethylase;TNMT, tetrahydroprotoberberineN -甲基转移酶;TYDC,酪氨酸脱羧酶;TyrAT,酪氨酸转氨酶
除了模式物种之外,已知有无数其他植物也会积累BIAs。这些生物碱的结构多样性是显著的,但它们的生物合成在很大程度上或完全未被探索。这些化合物中有许多具有强大的药理活性,积累这些化合物的植物在传统医学中有着悠久的使用历史。委员会成员Cissampelos属,积聚新的双苯基异喹啉,阿吗啡,和前吗啡型生物碱(附加文件1)几个世纪以来一直被用作狩猎毒药和草药,尤其是在南美洲和撒哈拉以南非洲[45].三叶碱是一种高度交联的非典型双苄基异喹啉生物碱,被认为具有抗阿米巴活性Cocculus治疗婴儿腹泻的制剂[41].许多罂粟科植物产生的生物碱具有独特的变化基本的原小檗碱和二苯甲苯乙烯基,和一些属,如紫堇属之植物积累了令人惊讶的多种BIA类型,包括原碱,pthalideisoquinoline, spirrobenzylisoquinoline和morphinan生物碱[21].这些生物碱是如何形成的尚不清楚,而且能够产生这些生物碱的非模式植物资源稀缺。为了阐明途径和发现新酶,我们使用Roche 454和Illumina测序平台为20种bia积累植物生成了广泛的数据集。基于对公共数据库的直接搜索,使用多种注释方法构建了数据挖掘框架,并收集和总结了每个单基因的相关信息,包括京都基因和基因组百科全书(KEGG)路径图、基因本体论(GO)和酶委员会(EC)注释。在相同的植物组织上,结合本文所述的转录组分析,进行了全面、广泛的代谢物调查[18].结合使用,这些前所未有的资源将允许在大部分未开发的系统中组装代表BIA代谢的生化快照,指导途径阐明和新催化剂的搜索工作。此外,从不同植物物种中提取的酶变体的可用性将极大地扩大合成生物学工作所必需的“工具箱”。
结果与讨论
生物合成基因富集的物种和组织选择
根据相关文献来源和我们对候选物种代谢物含量的研究确定的生物碱积累谱,选择了20个植物物种进行转录组分析[18].其他考虑因素包括分类分布、在传统医学或文化实践中的使用(表明可能存在具有药理活性的BIAs)和组织可用性。优先级被分配给序列信息不可用或缺乏的物种。我们的目标是毛茛科的四个植物科:罂粟科(8种),毛茛科(4种),Berberidaceae(4种)和Menispermaceae(4种)(表2)1).尽管BIAs在不同被子植物类群中都有报道,但它们最常发生在这些科[17].强有力的证据支持毛茛属的单系起源,在这一目中,罂粟科家族似乎早从“核心”毛茛属群中分离出来(补充文件2) [50].进一步的证据支持BIA生物合成在eudicots出现之前的早期单系起源[34表明毛茛属物种的最后一个共同祖先已经在制造生物碱了。为了富集BIA生物合成转录本,分析仅限于富含生物碱的器官(茎、根茎或根)或愈伤组织培养(表2)1).作为完整植物的替代品,细胞培养已经作为生物合成模型和生物碱生产系统使用了三十多年[54].体外植物细胞培养有助于发现血碱、小檗碱、诺scapine和吗啡生物合成中的几种关键酶和调节过程[17,44].最近,建立了18种产生生物碱的培养物的少量文库(约3500个unigenes),其中包括3种Menispermaceae物种的愈伤组织[10].为了建立在这些资源之上,愈伤组织Cocculus trilobus,金果榄等而且Cissampelos mucronata被选中进行深度测序。
Roche vs Illumina平台:增强读取深度的好处
通过Roche GS-FLX Titanium或Illumina GA/HiSeq平台进行深度测序之前,对RNA进行足够质量和数量的筛选。对于基于illumina的测序,GA (Genome Analyzer)和HiSeq仪器被用于生成质量基本相同的数据,允许随后的数据池。表格2总结了这两种技术的结果,而附加文件3.而且4分别列出罗氏和illumina平台的进一步细节。20个物种中6个物种的数据1),虽然有一些小错误(如表1 b(53])。本文提供了校正值,包括14个新植物物种的数据,以供比较。多平台研究强调了基于illumina的测序相对于其他技术的某些优势,包括更低的成本(0.06美元/Mb),较高的准确性(< 2%的错误率)和良好的读取深度,允许稳健的转录本量化[32,40,46].良好的读深度体现在每碱基对的平均读(69.6;额外的文件4)允许每个库的平均unigenes数量几乎是Roche技术的两倍(分别为34,368对63,886;表格2).相反,Roche 454 GS flx测序的优点包括更长的平均读取长度(例如>比Illumina HiSeq平台长12倍;[32]),从而可靠地检测剪接变体。尽管读取时间更长,但与基于illumina的测序相比,基于roche的测序结果预测的全长编码序列(CDSs)更少3.而且4).尽管如此,使用两种不同的平台具有增强整体转录组覆盖的固有优势。Roche和Illumina文库平均分别为~ 14000和~ 24500个全长CDS,根据保守的Mega BLAST估计,两个文库之间平均有~7700个CDS交叉e -0 ([56];额外的文件3.).低数量的CDS交叉可能反映了严格的BLAST参数的使用,而不是两个库之间的固有差异,并增加了e -价值截点将显示出更大的一致性。
库比较揭示了低交集的孤立案例
不同来源组织(如茎与根、愈伤组织)的库质量差异不明显。对于质量控制措施,基于illumina的测序对茎和根进行Chelidonium majus产生类似的结果(附加文件5).然而,在个别情况下,图书馆的质量出现了下降。例如,illumina公司Cocculus trilobus库包含大量的读取,但产生了高于平均数量的未组装contig和少量的完整长度CDSs(附加文件4).相反,Roche-basedc . trilobus测序似乎相对成功(附加文件3.).由于基于Illumina和roche的库是使用相同的源材料构建的,我们排除了这种可能性c . trilobus组织受损,因为糟糕的组织质量会影响两个转录组,而不仅仅是Illumina的数据。另一个Illumina库具有更少的全长cds(与原始读取相比)和与Roche数据的低交集十大功劳aquifolium.在这些案例中,可能发生了与来自其他植物的样品的交叉污染,从而妨碍了在后期对外来或本土序列进行适当的组装和分离。
建立完全注释的BLAST-可达转录组
平均而言,所有unigenes中79% (Roche)和69% (Illumina)获得了功能注释,基于更严格标准的高水平注释分配给57% (Roche)和50% (Illumina)(表2)2).酶委员会(EC)编号分配被纳入分析,以获得每个文库中所代表的酶的数量,并实现相应的KEGG通路图链接(www.genome.jp kegg /通路).对于酶的发现来说,更重要的是,EC赋值可以促进基于酶功能的单词搜索。平均而言,Roche和Illumina库中大约12%的注释对应于EC编号。EC编号分配的成功率较低c . trilobus而且m . aquifoliumIllumina库,可能是由于完整长度cds组装不良。每个unigene的结果,包括成分读取、表达数据、BLAST结果、注释证据和相关链接都汇总在通过MAGPIE可用的单个页面上。先前建立的基于magpie的BLAST门户[53]供公众查阅本文所报道的每个转录组的汇编数据(www.phytometasyn.ca).
基于同源性的BIA生物合成基因挖掘
基于Illumina和Roche 454的转录组被挖掘,以寻找可能参与BIA代谢的候选基因。tBLASTn搜索是基于与完全特征的生物碱生物合成酶的同源性进行的,在大多数情况下使用40%的序列同源性截断。例外包括o -乙酰转移酶(OATs)和羧酸酯酶(CXEs),通常使用30%的搜索截止值。对于oat和cx,分类组之间的序列差异较大,因此搜索标准更灵活。在某些情况下不需要预定义的截止点,因为tBLASTn产生了少量具有相对较高身份的命中。例如,搜索使用小檗碱桥酶从花菱草californica,果实而且小檗属植物多茎目(分别为EsBBE, PsBBE和BsBBE)共产生了18个具有大量(> 60%)身份的命中。类似的结果得到了二氢苯并苯胺氧化酶(DBOX)样fad依赖氧化酶(FADOX)。共从40个深度测序文库中筛选出约850个候选unigenes,代表20个植物物种。额外的文件6以FASTA格式列出这些候选氨基酸序列。
候选选择和瓶颈识别的基因表达
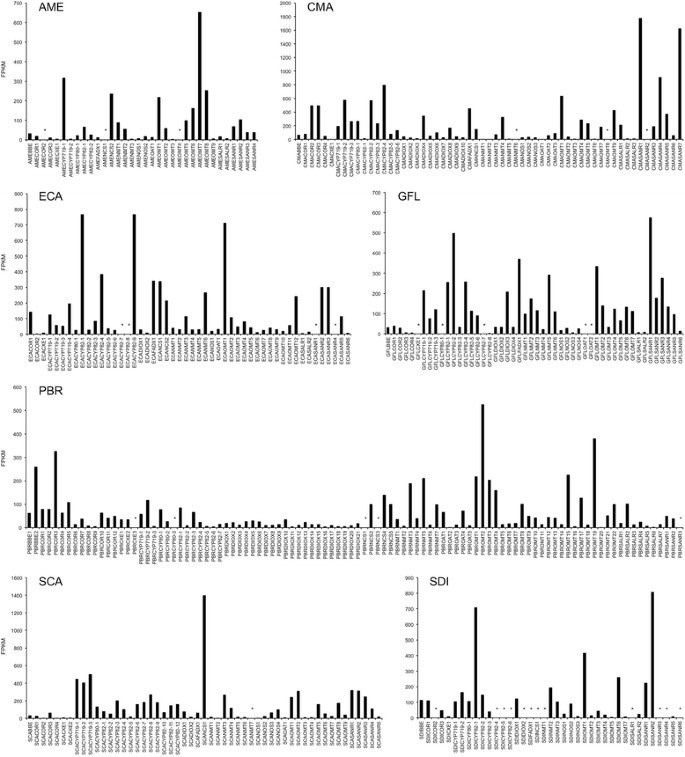
表达数据以从Illumina库中提取的FPKM (Fragments Per Kilobase of exon model Per Million mapped reads)的形式记录。数字2总结了罂粟科(Papaveroideae)部落成员的结果。的表达式结果chelanthifolia延胡索(Fumarioideae tribe, Papaveraceae),小檗科和毛茛科物种在附加文件中被发现7, Menispermaceae物种的结果在附加文件中找到8.未进行表达分析m . aquifolium而且c . trilobus因为全长信用违约掉期的数量减少了。表达值在所有Illumina库中被标准化,允许跨物种比较(见方法).FPKM和相关RNA-seq工具是可靠的表达指标;事实上,最近Illumina和基于微阵列的数据的直接比较表明,RNA-seq在识别差异表达基因方面显著优于微阵列[49].为了发现新的催化剂,基因表达数据可以用来对进一步分析的候选物进行优先排序。在bia合成组织中高表达的基因可以从表达水平非常低的候选基因中选择出来。例如,虽然在罂粟科文库中鉴定出了17个假定的(S)-去甲葡萄醛酸合成酶(NCS)候选基因,但其中一些unigenes仅作为低读的Roche contigs被观察到,而在Illumina数据中完全不存在(图2)。2,附加文件7).缺少Illumina数据可能反映了平台偏差或处理错误,尽管这可能是非常低的基因表达的结果。表达比较可以在不同的基因家族之间进行,以获得关于假定的代谢瓶颈的见解。罂粟花bracteatum可累积大量的thebaine,但下游生物碱可待因和oripavine只有微量[24],表明代谢阻滞在thebaine 6-O-去甲基酶(T6ODM)和可待因O-去甲基化酶(CODM)1).T6ODM和CODM已在罂粟中发现,属于Fe2 +/2-羟戊二酸依赖双加氧酶家族[16].与其他生物合成基因相比p . bracteatum, DIOX同源物的表达水平非常低,可能有助于观察到通路限制。
罂粟科(罂粟科)中可能参与BIA生物合成的候选基因的归一化表达分析。每个候选物种都被标记为各自的物种缩写(例如AME,Argemone墨西哥)和可能由基因编码的酶的类型(例如BBE,小檗碱桥酶)。仅在roche转录组中出现的候选对象不能被分配FPKM值,并被标记为星号。参考表格1用于物种缩写。酶/蛋白家族缩写:BBE,小檗碱桥酶;COR,可待因酮还原酶;CXE,羧酸酯酶;CYP,细胞色素P450单加氧酶;DIOX加双氧酶;FAD, FAD依赖氧化酶;NCS,去甲磺酸合酶;NMT N-methyltransferase;NOS,诺scapine合成酶; OAT,O乙酰转移酶;OMT,O甲基转移酶;敬礼碱还原酶(SALR);血氨酸还原酶
系统发育分析作为基因功能的预测工具:NMT案例研究
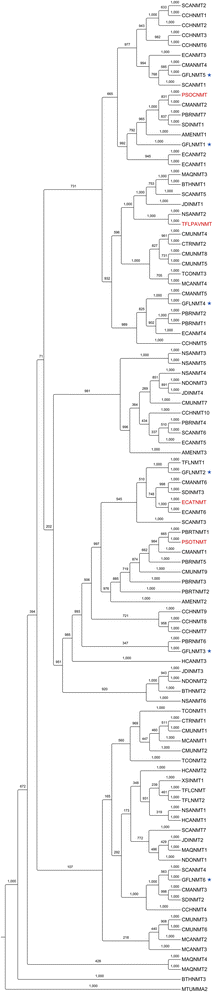
对15类蛋白质/酶进行了氨基酸比对和系统发育树的组装,共代表约850个候选基因。数据3.而且4说明了使用CYP719和构建的树N-甲基转移酶候选。其余的树在附加文件中找到9,10,11,12,13,14,15,16,17,18,19,20.而且21.与相应的FPKM数据和特定物种的生物碱剖面一起使用[18这些结果为发现新的酶提供了重要的资源,这些酶催化(i)以前描述过的反应(即功能同源物)和(ii)在生化和分子水平上未描述的反应。为了检验我们的假设,即系统发育的考虑可以用来预测酶的功能,我们设计了一个实证案例研究使用黄Glaucium yellow N-甲基转移酶(NMT)候选基因。基于同源的挖掘在Roche和illumina中都发现了6个全长的候选NMTg . flavum转录组(无花果。2).系统发育分析显示,某些g . flavum候选特征酶与其他酶比较。例如,GFLNMT1与PSOCNMT形成了一个六人分支,PSOCNMT是一个已建立的共子N -甲基转移酶(CNMT)果实[19(图。4).相比之下,GFLNMT2形成了一个由6个成员组成的分支,包括(年代) -tetrahydroprotoberberineN-甲基转移酶(TNMT)Eschscholzia californica(ECATNMT) [35].基于这些结果,我们预测GFLNMT1和GFLNMT2酶将分别表现出CNMT和TNMT活性。尽管其余的GFLNMTs与已知酶没有形成类似的小分支,或表现出如此高的同一性(> 70%),但由于>与查询序列具有40%的同一性,因此与BIA底物的活性是预期的。所有六个g . flavum候选人在大肠杆菌作为his标记的重组蛋白,每个蛋白都显示出通过与分子量标准比较确定的预测分子量(附加文件)22).使用六种关键生物碱底物测试每种蛋白质的NMT活性(表2)3.).事实上,GFLNMT1和GFLNMT2分别在使用椰磺酸和原小檗碱底物时表现出CNMT和TNMT活性。进一步,我们的预测认为g . flavum酶能接受BIA底物,证明正确。GFLNMT3作为TNMT使用(年代)-stylopine基质,但没想到也N甲基化(年代) -reticuline。(年代) -ReticulineN-甲基转移酶活性也观察到GFLNMT5。GFLNMT4作为CNMT,具有显著的执行后续任务的特点N,N-二甲基化反应生成季胺。尽管GFLNMT6与特征CNMT的聚类不紧密(图2)。4),它接受coclaine底物。这些结果证明了系统发育分析作为一种预测工具的一般效用,但强调了为了基因发现的目的需要经验分析数据。
的系统发育分析N-甲基转移酶(NMT)候选基因的研究。红色文本表示用于转录组挖掘的tBLASTn查询的特征基因或酶。黑色文本表示通过挖掘识别出的未特征的候选基因(> 40%与查询相同)。每个分支的引导值基于1000次迭代。每个候选物种都被标记为各自的物种缩写(例如AME,Argemone墨西哥;见表1)和候选编号(例如NMT1)。每个查询都根据物种进行标记(其他物种:PSO,果实)和特定的NMT功能(CNMT, coclaurineN甲基转移酶;PAVNMT, pavineN -甲基转移酶;TNMT, tetrahydroprotoberberineN甲基转移酶;见图。1).外基是霉菌酸合成酶结核分枝杆菌(MTUMMA2)。NMT考生来自Glaucium flavum催化活性测试用星号表示。候选、查询和外组的氨基酸序列可在附加文件中找到6
合成生物学功能同源资源
为了新兴的合成生物学计划的目的,功能同源物——通常被称为酶的“变体”——是必不可少的工程工具。使用异质表达的植物酶在微生物中组装生物碱途径充满了问题——包括蛋白质表达差、不可预测/脱靶活性、与其他途径酶相互作用差以及催化效率低[28-在某些情况下可以通过变量替换来缓解。例如,测试来自果实而且Thalictrum flavum揭示了特定的变异和变异的组合,改善(年代)-酵母中网状蛋白的产生[19].我们收藏的N -而且O-甲基转移酶候选物来源于各种各样的植物(图。4,附加文件18)将使单细胞系统中的生物碱生物合成进一步细化。
在morphinan和aporphine生物碱的形成中具有假定作用的候选者
鉴定在吗啡生物碱生物合成中起作用的功能同源物是一个重要的目标,因为在微生物中重建这一途径是一个新兴的目标[48].产生吗啡生物碱的Illumina转录组p . bracteatum含有三个CYP719候选基因,它们与罂粟(果实)敬礼碱合成酶(SalSyn, PSOC719B1;无花果。3.).此外,六p . bracteatum已鉴定出与鸦片罂粟碱还原酶(SalR)具有大量同源性(高达92%氨基酸同源性)的unigenes(图。2,附加文件14).我们的研究包括已知产生不太为人所知的吗啡类生物碱的植物属,例如紫堇属之植物,南天竹属而且Thalictrum,它们分别产生(+)-pallidine, sinoacutine和(-)-pallidine [21,22,47].值得注意的是,这些植物还会产生多种阿啡碱生物碱,如南藤碱(南天竹属;[22])、异莨菪碱(紫堇属之植物;[14])及紫堇(Thalictrum;[47])。这些morphinan和aporphine生物碱的生物合成途径尚不清楚,但可能依赖于cyp介导C-C耦合(年代)-或(R) -reticuline。在这些物种中发现了相对较少(<10)的CYP80、CYP719和CYP82候选者(图2)。3.,其他文件16而且17)可以测试网板氧化酶活性,并评估参与吗啡和/或阿orphine途径。
邻苯二代异喹啉生物碱生物合成的潜在新催化剂
以最近阐明的罂粟中noscapine生物合成为指导[6,51],苯酞代异喹啉积累物种的转录组被用于新的催化剂。黄连碱黄花产生水螅氨酸、水螅氨酸和其他次要成分[26),而紫堇属之植物物种积累了各种各样的邻苯二甲酸异喹啉生物碱[2].大量的乙酰转移酶,羧酸酯酶和CYP82候选者可能参与邻苯二代异喹啉生物合成h .黄花而且c . chelanthifolia转录组。紫堇属之植物种累积半缩醛[52],这可能需要一种noscapine合成酶(NOS)样酶来假设转化为双头素[12].年选出了六名候选人c . chelanthifolia具有高达52%的同一性p .果实NOS,尽管在某些情况下表达非常低(附加文件7).3个nos样基因候选,可能作为肼合酶的作用h .黄花(附加文件7而且21).
结论
为20种积累bia的植物建立了完全注释的、深度测序的转录组,为新型催化剂的发现提供了巨大的资源。尽管在这些物种中发现了丰富的生物活性生物碱及其在传统医学中的重要性,但在此之前,本报告中所包括的大多数植物都没有blast可访问的转录组。本文提出的结果,以及伴随的代谢物概况[18]和相关文献,旨在提供必要的工具(即。基因序列),也为继续阐明BIA代谢的实验设计提供信息。
方法
生物碱
用作底物或标准物的生物碱来源如下:(年代-网状草酸盐是Tasmanian Alkaloids(塔斯马尼亚,澳大利亚)的礼物;((R, S))-canadine购自Latoxan (Valence, France);(±)-pavine购自Sigma-Aldrich(圣路易斯,密苏里州),(年代)-可可碱购自Toronto Research Chemicals (Toronto, ON);((R, S))-stylopine的合成方法如前所述[33].
植物材料
所选组织取自黄连碱黄花,Sanguinaria黄花,黑种草,十大功劳aquifolium,Menispermum canadense,Stylophorum diphyllum,Xanthoriza simplicissima在植物园(Jardin Botanique de Montréal)种植的户外植物(Montréal, Québec;http://espacepourlavie.ca).Jeffersonia diphylla而且小檗属植物thunbergii植物购买自Plant Delights Nursery(北卡罗来纳州罗利;www.plantdelights.com)和Sunnyside温室(阿尔伯塔省卡尔加里;www.sunnysidehomeandgarden.com),分别。Chelidonium majus,罂粟花bracteatum,Argemone墨西哥,花菱草californica,南天竹属有,Glaucium flavum,Thalictrum flavum而且chelanthifolia延胡索在卡尔加里大学(卡尔加里,阿尔伯塔)的标准露天温室条件下,在盆栽土壤中发芽的种子生长而来。种子均来自B和T World Seeds (B -and- T - worldseeds.com)t . flavum而且p . bracteatum,由Staudensamen (www.jelitto.com)及玫瑰花园人生(www.lavieenrosegardens.com),分别。愈伤组织培养Cissampelos mucronata,Cocculus trilobus,金果榄等购买自Deutsche Sammlung von Mikroorganismen und Zellkulturen (DSMZ, Braunschweig, Germany;www.dsmz.de),并按所述的方法维修[10].所有组织在液氮中快速冷冻,并在−80°C保存,直到分析。
Poly(A) + RNA纯化,cDNA文库制备及下一代测序
使用改良的CTAB方法从茎、根茎、根或愈伤组织中提取总RNA [38].RNA质量基于紫外吸收比,仅使用比值高于2.0 (260/280 nm)和2.2 (260/230 nm)的样品。Poly(A) + RNA纯化,cDNA文库合成,乳化PCR (emPCR)和NGS在麦吉尔大学和Génome Québec创新中心(Montréal, Québec)进行,如所述[53].简单地说,RNA质量和数量使用NanoDrop nt -1000 (Thermo Scientific, Waltham, Massachusetts)和BioAnalyzer 2100 (Agilent Technologies, Santa Clara, California)仪器进行评估,Poly(A) + RNA纯化使用Dynabeads mRNA纯化试剂盒(Invitrogen)或TrueSeq链mRNA样品准备试剂盒(Illumina, San Diego, California)进行。根据下游NGS方法,使用cDNA Rapid Library试剂盒(Roche, Basel, Switzerland)或TruSeq搁浅mRNA Sample Prep试剂盒(Illumina)进行cDNA合成。Roche-454 GS- flx Titanium pyrosequencing采用GS Run Processor (Roche)进行数据处理,生成标准流图格式(Standard Flowgram Format, SFF)文件。对于Illumina GA和HiSeq测序,使用HCS 1.4和CASAVA 1.6-1.8软件套件(Illumina)生成原始fastq reads。
从头转录组组装、功能注释和GO分析
序列质量控制和筛选如所述[53].剪切适配体/引物序列,根据Phred质量评分修剪所有序列,掩盖低复杂度区域,使用Paracel过滤包(PFP) (Paracel Inc., California)的Scylla程序从每个454数据库中删除核糖体RNA (rRNA)序列。使用Fast QC对Illumina reads进行质量评估和清洗(www.bioinformatics.babraham.ac.uk项目/ fastqc /)及Cutadapt [37].使用MIRA (v. 3.2)组装清洗后的454个序列数据[4],与Paracel Transcript Assembler (Paracel Inc.)或Newbler v. 2.3相比,该方法产生更长的contigs (>1000 bp) [36)平台。过滤后的Illumina读数使用Velvet-Oases v. 0.1.16组装[55].CD-HIT-EST [30.]用于通过聚类几乎相同的转录本(> 99%)来减少冗余,并使用CAP3 [20.].喜鹊(喜鹊自动化基因组计划调查环境)[11]用于注释每个数据集,这些数据集基于对公共和内部数据库的序列相似性搜索,包括NCBI和RefSeq的viridiplantae子集。加速隐马尔可夫模型(HMM)搜索也被执行。全长编码序列预测按所述进行[53].基于与数据库中已经标注的序列进行比较的功能描述,以及域级内容,根据所有搜索结果的加权摘要分配给每个contig。GO(基因本体)注释和EC编号被指定为每个contig,如前所述[53].朝着整合转录组学与相应代谢组学数据的目标[18]转录数据通过EC数映射到KEGG代谢途径。
基因表达分析
作为第一轮,基于原始reads丰度获得所有40个文库(20个基于454,20个基于illumina)中每个contig的相对基因表达信息。对于454个库,从contig程序集文件中提取原始读计数。对于Illumina库,使用Bowtie [25]将原始读取重映射到已组装的contigs,以及RSEM [29]用于最终量化。相对归一化(即。通过计算每个contig的FPKM(每百万外显子模型的千碱基片段数)来实现。为了实现跨不同文库的基因表达比较,使用Illumina数据进行了第二轮归一化。首先,将来自所有20个Illumina库的contigs编译在一起,并根据序列相似性将其分组。数据的聚类使用OrthoMCL进行,OrthoMCL是一种设计用于跨多个真核生物类群的同源组的可伸缩构建的程序[31].不同文库之间RNA数量的差异(即。RNA组成偏差)通过计算每个文库的组合比例因子来解释。这一步是使用edgeR v.3的calcNormFactors函数执行的(www.bioconductor.org),它决定了一组因子,然后组合成一个单一的“比例因子”,每个文库都唯一,最大限度地减少了大多数基因样本之间的对数倍变化[3.].通过将第一组特定于库的FPKM值乘以相应的缩放因子,生成了第二组支持跨物种比较的FPKM值。
候选基因的比对和系统发育分析
使用geneous (Biomatters, Aukland, New Zealand)的内置肌肉校准功能对属于单个酶类的候选氨基酸进行校准。对齐是作为自由端间隙执行的,计算对齐之后是手工排序。最大似然系统发育分析使用了genous [13].每个分支的引导值基于1000次迭代。对于P450和NMT树,智人而细菌(结核分枝杆菌)序列分别作为外群。来自远亲类群的序列通常不作为外群使用,因为系统发育距离会导致排列退化[43].然而,没有观察到排列退化,这与使用来自遥远类群的外群进行CYP分析的类似报道一致[42].
n -甲基转移酶候选基因功能分析
6个序列大于40%的候选基因与4个已知编码的查询序列中的一个或多个相同N-甲基转移酶(NMTs)在BIA生物合成中具有既定的作用(图;4;额外的文件6)已在Glaucium flavumIllumina-based转录组。使用Q5 HiFi DNA聚合酶(New England Biolabs)和含有attB位点的基因特异性引物扩增编码序列g . flavum根的互补。利用BP和LR Clonase II (Thermo Scientific)进行重组反应,生成pDONR221-GfMMT入口质粒和pHGWA-GfNMT表达质粒。在16°C下用大肠杆菌ArcticExpress(安捷伦科技)生长在Studier的自诱导介质(ZYP-5052) (Amresco, Solon, Ohio)。从每个培养物中提取总可溶性蛋白,并根据制造商的说明(SuperSignal West Pico Chemiluminescent Substrate kit, Thermo Scientific)通过免疫印迹程序验证his标记重组蛋白的存在。筛选了五种生物碱(加拿大碱、椰碱、stylopine、网碱、白藜芦醇)作为潜在底物g . flavum在线性产物形成条件下(30 μg总蛋白,100 μM生物碱,200 μM年代-腺苷蛋氨酸,100 mM磷酸钠,pH 7)。总测定量为100 μL,测定在30℃下进行5或30 min,测定时间取决于每种酶预先确定的线性范围。采用LC-MS/MS进行分析,如前所述[9].大多数产品通过与原品的保留时间和CID光谱比较进行了鉴别。N,N通过将反应产物CID光谱与先前报道的数据进行比较,鉴定了-二甲基coclaurine [8].相对于空载体对照,对产物形成进行监测。对于每种酶,相对于观察到底物转化率最高的测定法(即后一种测定法设置为100%)计算活性。
支持数据的可用性
本文所讨论的所有序列数据均已存入国家生物技术信息中心序列阅读档案(http://www.ncbi.nlm.nih.gov/sra)在附加文件中列出的注册编号下3.而且4.所有的系统发育数据可在Dryad (http://dx.doi.org/)中获得10.5061 / dryad.bh276).
缩写
- AKR:
-
Aldo-keto还原酶
- 被:
-
小檗碱桥酶
- BIA:
-
Benzylisoquinoline生物碱
- cd:
-
编码序列
- 林后:
-
Codeinone还原酶
- CXE:
-
羧酸酯酶
- CYP:
-
细胞色素P450单加氧酶
- DIOX:
-
2-oxoglutarate /铁2 +端依赖加双氧酶
- FADX:
-
fad依赖的氧化还原酶,FPKM,每千碱基的外显子模型每百万映射读取片段
- 燕麦:
-
O乙酰转移酶
- OMT:
-
O甲基转移酶
- nc:
-
Norcoclaurine合酶
- 号:
-
诺司卡品合酶
- SalR:
-
Salutaridine还原酶
- SanR:
-
血根碱还原酶
参考文献
Beaudoin GAW, Facchini PJ。cDNA编码的分离与鉴定(年代) - - -独联体-N-甲基苯乙烯平14-羟基化酶,是血碱生物合成的关键酶。生物化学学报,2013;
Blaskó G, Gula DJ, Shamma M.邻苯二甲酸异喹啉类生物碱。王志强。2002;26(5):1 - 5。
陈勇,McCarthy D, Robinson M, Smyth GK。edgeR:数字基因表达数据的差异表达分析。生物导体用户指南。2014;1-78。
Chevreux B, Pfisterer T, Drescher B, Driesel AJ, Müller WE, Wetter T,等。使用miraEST组装程序进行可靠和自动化的mRNA转录本组装和测序est中的SNP检测。基因组资源2004;14:1147-59。
D 'Auria JC。植物中的酰基转移酶:BAHD的好时机。植物学报,2006;9:331-40。
Dang TTT,陈旭,Facchini PJ。乙酰化是罂粟诺scapine生物合成过程中的一个保护基团。生物化学学报2015;11:104-6。
Dang TTT, Facchini PJ。CYP82Y1是N-甲基加拿大碱1-羟化酶,罂粟中的一种关键的诺scapine生物合成酶。中国生物化学杂志,2014;29(3):329 - 329。
Desgagné-Penix我,Facchini PJ。系统沉默苄基异喹啉类生物碱合成基因揭示了罂粟碱的主要途径。植物学报,2012;
法罗SC,法奇尼PJ。加双氧酶催化O脱甲基,O,O-脱甲基作用在罂粟苯基异喹啉类生物碱代谢中有广泛作用。中国生物医学工程学报,2013;
法罗SC,哈格尔JM,法奇尼PJ。18种产生苄基异喹啉生物碱的植物细胞培养的转录本和代谢物分析。植物化学,2012;77:79 - 88。
Gaasterland T, Sensen CW。MAGPIE:自动基因组解读。《趋势》1996;12:76-8。
Gözler B, Gözler T,沙玛M.甘油三酯:一种可能的邻苯二代异喹啉生物发生中间体。四面体。1983;39:577 - 80。
金登S, Gascuel O.一个简单,快速和准确的算法估计大系统发育的最大似然。中国生物医学工程学报。2003;52:696-704。
郭震,蔡锐,苏华,李勇。根茎加工中的生物碱紫堇属之植物生根茎紫堇属之植物GC-MS分析。《肛管化学》2014;2014:1 - 6。
哈格尔JM,博多因GAW,福萨提E, Ekins A,马丁VJJ,法奇尼PJ。罂粟黄蛋白氧化酶催化血碱和罂粟碱生物合成的最后步骤的特性。中国生物医学工程学报。2012;
哈格尔JM,法奇尼PJ。双加氧酶催化O罂粟中吗啡生物合成的去甲基化步骤。生物化学学报。2010;6:273-5。
哈格尔JM,法奇尼PJ。苄基异喹啉生物碱代谢:一个世纪的发现和一个美丽的新世界。中国生物医学工程学报,2013;
哈格尔JM,曼达尔,R,韩bs,韩j, Dinsmore DR, Borchers CH,等。20种有分类关系的产苄基异喹啉生物碱植物的代谢组分析。BMC植物生物学。2014。doi:10.1186 / s12870 - 015 - 0594 - 2.
霍金斯KM,斯默克CD。苯基异喹啉生物碱的生产酿酒酵母.生物化学学报,2008;4:564-73。
黄霞,马丹。CAP3: DNA序列组装程序。Genome Res. 1999; 9:868-77。
Iranshahy M, Quinn RJ, Iranshahi M.属具有药物样性质的生物活性异喹啉生物碱紫堇属之植物.RSC Adv. 2014; 4:15900-13。
Iwasa K,高桥T,西山Y,森康M,杉浦M,竹内A,等。黄芪粗提物和培养细胞中生物碱等成分的在线结构信息南天竹属有结合LC-MS/MS, LC-NMR和LC-CD分析。中国科学(d辑),2008;
Kavanagh KL, Jörnvall H, Persson B, Oppermann U. SDR超家族:代谢和调节酶家族中的功能和结构多样性。中国生物医学工程学报,2008;
Küppers FJEM, Salemink CA, Bastart M, Paris M罂粟花bracteatum:有可待因、新安平和高平。植物化学。1976;15:444-5。
Langmead B, Trapnell C, Pop M, Salzberg SL.人类基因组短DNA序列的超快和内存高效对齐。中国生物工程学报。2009;10:R25。
李丽娟,李丽娟,李丽娟。超高效液相色谱- qtof -MS在质谱分析中的应用E黄海豹中生物碱的快速准确鉴定方法(黄连碱黄花).《肛肠生物化学》2014;406:1739-49。
李永杰,法奇尼。去甲钴酸合成酶是与发病相关的10/Bet v1蛋白家族成员。《植物科学》2010;22:389 - 389。
李继伟,罗东,朴建民,李俊,崔松,李世勇。微生物对天然和非天然化学物质的系统代谢工程。生物化学学报。2012;8:536-46。
李B,杜威CN。RSEM:从RNASeq数据中精确的转录物定量,有或没有参考基因组。生物信息学。2011;12:323。
Li W, Godzik a . Cd-hit:用于聚类和比较大组蛋白质或核苷酸序列的快速程序。生物信息学。2006;22:1658-9。
李,施托克特CJ,罗斯DS。真核生物基因组正正交基的鉴定。基因组res 2003; 13:2178-89。
李淑娟,李志强,李志强,李志强,等。在ABRF下一代测序研究中使用RNA-seq进行转录组分析的多平台评估。生物工程学报。2014;32:915-25。
Liscombe DK, Facchini PJ。四氢原小檗碱的分子克隆与表征独联体-N-甲基转移酶,一种参与罂粟生物碱生物合成的酶。中国生物化学杂志,2007;
Liscombe DK, MacLeod BP, Loukanina N, Nandi OI, Facchini PJ。被子植物中苄基异喹啉生物碱合成单系进化的证据。植物化学。2005;66:2501-20。
李斯科姆DK,齐格勒J,施密特J, Ammer C,法奇尼PJ。用于阐明酶功能的靶向代谢物和转录谱分析:新型的分离N-甲基转移酶来自三种产生苄基异喹啉生物碱的物种。植物J. 2009; 60:729-43。
马格丽斯,艾霍尔姆,阿尔特曼,阿提亚,杰德,本本,等。微制造高密度皮升反应器中的基因组测序。大自然。2005;437:376 - 80。
王哲。下一代转录组组装。《中国日报》,2011;12:671-82。
Meisel L, Fonseca B, Gonzalez S, Baeza-Yates R, Cambiazo V, Campos R,等。一种快速高效的桃总RNA纯化方法(碧桃)进行功能基因组学分析。生物学决议2005;38:83-8。
minnich RD, Penning TM。醛酮还原酶(AKR)超家族:基因组学和注释。胡姆基因组学。2009;3:362-70。
Nagarajan N, Pop M.序列组装去神秘化。Nat Rev Genet, 2013; 14:157-67。
Natajaran B, Paulsen BS。来自印度马哈拉施特拉邦塔那地区的民族药理学研究:传统知识与现代生物科学的比较。《医药生物学》2000;38:139-51。
植物细胞色素P450s从苔藓到杨树。植物化学Rev. 2006; 5:193-204。
Retief JD。使用PHYLIP进行系统发育分析。入:Misener S, Krawetz SA,编辑。生物信息学:方法和协议。纽约:Humana出版社;1999.243 - 58页。
利用培养植物细胞对植物功能的表征,以及生物技术应用。生物化学学报,2013;
Semwal DK, Semwal RB, Vermaak I, Viljoen A.从箭毒到草药——从民族植物学,植物化学和药理意义Cissampelos(防己科)。中华民族药典杂志,2014;
SEQC / MAQC-III财团。由测序质量控制联盟对RNA-seq准确性、可重复性和信息含量的全面评估。生物工程学报。2014;32:903-14。
沙玛M,萨尔加SS。Pallidine和corydine从Thalictrum dioicum.植物化学。1973;12:1505-6。
王志强,王志强,王志强,等。天然和半合成阿片类药物的微生物生物制造平台。中国生物化学杂志,2014;10:837-44。
王超,龚波,Bushel PR, Thierry-Mieg J, Thierry-Mieg D,薛娟,等。RNA-seq和微阵列数据之间的一致性取决于化学处理和转录丰度。生物工程学报。2014;32:926-32。
王伟,陆亚敏,任勇,Endress ME,陈志东。毛茛属植物的系统发育与分类:来自4个分子位点和形态学数据的证据。植物生态学报。2009;11:81-110。
温泽尔T, Gazda V,何z, Kaminski F, Kern M, Larson TR,等。一个果实用于合成抗癌生物碱诺scapine的10基因簇。科学。2012;336:1704-8。
吴超,闫荣,张荣,白峰,杨勇,吴震,等。比较四种生物碱在不同配方中的药代动力学和生物利用度decumbens延胡索.中华民族药典杂志,2013;149:55-61。
肖敏,张勇,陈霞,李艳杰,Barber CJS, Chakrabarty R,等。基于非模式植物的下一代测序的转录组分析,产生生物技术兴趣的专门代谢物。中国生物工程学报,2013;
岳伟,明庆林,林波,拉赫曼,郑长杰,韩涛,等。药用植物细胞悬浮培养:所需次生代谢产物的药物应用和高产策略。生物技术,2014。早期在线编辑:1-18。
Zerbino DR, Birney E. Velvet:使用de Bruijn图的从头短读组装算法。基因组决议2008;18:821-9。
张志强,张志强,张志强。一种基于贪心算法的DNA序列匹配算法。中华计算机科学,2000;7:203-14。
致谢
我们非常感谢Stéphane Bailleul和Renée Gaudette来自Jardin Botanique de Montréal的宝贵帮助和植物收藏。这项工作由加拿大基因组、阿尔伯塔基因组和阿尔伯塔政府资助。国开行和SCF获加拿大自然科学与工程研究理事会研究生奖学金。SCF还获得了阿尔伯塔独创性技术期货研究生奖学金。PJF担任加拿大植物代谢过程生物技术研究主席。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
PJF已经提交了与这项工作相关的专利申请。
作者的贡献
JMH解读数据并撰写手稿;JSM进行NMT测定;EJL和IDP提取RNA,协调DNA测序并进行初始数据库分析;CDB、LC、XC和SCF进行BLAST分析,建立系统发育树;YZ、JS和CWS进行了生物信息学分析;PJF构思了这项研究,采购了植物,准备了数据,并编辑了手稿。所有作者都阅读并批准了最终的手稿。
附加文件
附加文件1:
由碱性苄基异喹啉亚基衍生的BIA结构亚基的选定示例。(PDF 854kb)
附加文件3:
基于roche深度测序平台的结果总结。在roche和illumina转录组之间预测的全长CDS交叉是为了进行比较(见结果和讨论)。(PDF 73kb)
附加文件4:
基于illumina深度测序平台的结果总结。(PDF 80kb)
附加文件5:
基于illumina深度测序平台的结果总结。为了便于比较,茎和根Chelidonium majus进行转录组分析。(PDF 55kb)
附加文件6:
所有候选、查询和外组的FASTA文件。(PDF 253kb)
附加文件10:
20种植物小檗碱桥酶(BBE)候选基因的系统发育分析。红色文本表示用于转录组挖掘的tBLASTn查询的特征基因或酶。黑色文本表示通过挖掘识别出的未特征的候选基因(> 40%与查询相同)。tBLASTn查询没有产生命中的物种不在树中表示。每个分支的引导值基于1000次迭代。每个候选物种都被标记为各自的物种缩写(例如AME,Argemone墨西哥;见表1)和候选编号(如BBE1),即识别出多个命中。每个查询都根据物种进行标记(其他物种:PSO,果实;BST,小檗属植物多茎目).外基为四氢大麻酚酸合成酶(THCAS)大麻(CSA)。候选、查询和外组的氨基酸序列可在附加文件中找到6.(PDF 185kb)
附加文件16:
20种植物CYP80候选基因的系统发育分析。红色文本表示用于转录组挖掘的tBLASTn查询的特征基因或酶。黑色文本表示通过挖掘识别出的未特征的候选基因(> 40%与查询相同)。每个分支的引导值基于1000次迭代。每个候选物种都被标记为各自的物种缩写(例如AME,Argemone墨西哥;见表1)和候选编号(例如CYP80-1)。每个查询都根据物种进行标记(其他物种:HNI,Hyoscymus尼日尔;CJA,黄连粳稻;BST,小檗属植物多茎目;算法,果实),表明CYP80亚家族和基因数量(如CYP80A1,棒状结核合酶;见图。1).外群是CYP1B1来自智人(HSA)。候选、查询和外组的氨基酸序列可在附加文件中找到6.(PDF 13027kb)
附加文件18:
的系统发育分析O-甲基转移酶(OMT)候选基因的研究。红色文本表示用于转录组挖掘的tBLASTn查询的特征基因或酶。黑色文本表示通过挖掘识别出的未特征的候选基因(> 40%与查询相同)。每个分支的引导值基于1000次迭代。每个候选物种都被标记为各自的物种缩写(例如AME,Argemone墨西哥;见表1)和候选编号(例如OMT1)。每个查询都根据物种进行标记(其他物种:PSO,果实;CJA,黄连粳稻)和特定的OMT函数(SOMT, scoulerineO甲基转移酶;CbOMT, columbamineO甲基转移酶;N7OMT,去甲网状7-O甲基转移酶;7 omt;6 omt;4 'omt;见图。1).外基是异黄酮O甲基转移酶的紫花苜蓿(MSA)。候选、查询和外组的氨基酸序列可在附加文件中找到6.(PDF 13001 kb)
附加文件19:
bhd型的系统发育分析O乙酰转移酶(燕麦;D’aura 2006)来自20种积累bia的植物物种的基因候选。红色文本表示表征的AT1(1,13-二羟基-)N-methylcanadineO乙酰转移酶;[6])和SAT(有益菌素合成酶)果实(PSO)用于转录组挖掘的tBLASTn查询。黑色文本表示通过挖掘识别出的未特征的候选基因(> 30%同一性查询)。每个分支的引导值基于1000次迭代。每个候选物种都被标记为各自的物种缩写(例如AME,Argemone墨西哥;见表1)和候选编号(例如AT1)。外基是长春碱合成酶Rauvolfia serpentina(RSEVS)。候选、查询和外组的氨基酸序列可在附加文件中找到6.(PDF 5574 kb)
附加文件22:
免疫印迹分析显示,在可溶性提取物中存在his标记的重组蛋白大肠杆菌表达六种中的一种的菌株N-甲基转移酶候选物(GFLNMT1-6)Glaucium flavum.(PDF 203kb)
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
哈格尔,j.m.,莫里斯,j.s.,李,EJ。et al。20种有分类亲缘关系的产苄基异喹啉生物碱植物的转录组分析。BMC植物生物学15日,227(2015)。https://doi.org/10.1186/s12870-015-0596-0
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-015-0596-0
关键字
- 血根碱
- 罂粟
- 酶委员会编号
- 诺司卡品
- Illumina公司库