摘要
背景
长链非编码rna (Long non-coding RNAs, LncRNAs)已被确定为影响其邻近蛋白质编码基因转录的基因调控元件。在动物中发现lncrna刺激了对植物基因组中这些元素的全基因组扫描。最近,有6480个lincrna在拟南芥(芸苔科),但关于其保存的资料有限。
结果
利用系统基因组学方法,我们通过分析基础芸苔科物种的基因组,评估了这些lncrna的位置和序列守恒Aethionema arabicum而且Tarenaya hasslerianaCleomaceae的姐妹科植物。此外,我们生成了另外三个的转录组Aethionema和另一种Cleomaceae物种来验证它们的转录活性。我们发现lncrna的一个子集在核苷酸水平上高度分化,但在位置上保守(共位)。位置保守的LncRNAs表达与重要的发育和生理基因相邻。有趣的是,65%的位置保守的LncRNAs位于端粒的2.5 Mb内拟南芥染色体。
结论
这些结果强调了分析植物非编码遗传元件(包括LncRNAs)序列守恒和位置守恒的重要性。
背景
基因调控转录本在表达或抑制蛋白质编码基因中起着至关重要的作用。例如,植物中的基因抑制可以通过microRNAs (miRNAs, 19-22 nt长)和小干扰rna (siRNAs, 23-24 nt长)来维持。虽然miRNAs主要参与转录后基因抑制,但siRNAs也参与转录前基因抑制新创染色质标记的沉积[1].一类新的RNA依赖基因调控因子是长链非编码RNA (LncRNAs,长度大于200 nt, ORF小于100个氨基酸),它们可以在转录前抑制基因表达的过程中发挥作用[2- - - - - -4].
长链非编码rna可以通过作为染色质的序列特异性模板或与下游蛋白质相关联来沉默基因[3.并从基因间(长基因间非编码rna = LincRNAs)、内含子或反义区域转录[5,6].最近在COOLAIR中的LncRNAs中也发现了这种现象拟南芥[7,8]对于水稻LncRNA LDMAR [9,10它们如何影响表型上重要的调节基因的表达。COOLAIR(冷诱导长反义基因内RNA)转录自开花位点C (FLC)并加速转录抑制方法通过减少基因激活染色质标记H3K36me3 [7].与此同时,基因沉默染色质标记H3K27me3在基因内积累方法polycomb定向成核位点[7].因此,LncRNAs COOLAIR有助于诱导春化后的开花。突变水稻58S在长日照条件下花粉不育,而在短日照条件下花粉变异可育。丁等人。[9]研究发现,58S中LncRNA LDMAR过表达后,水稻在长日育下恢复了育性。58S中LDMAR的转录是由一个叫做Psi-LDMAR的siRNA的负反馈环控制的。Psi-LDMAR是从LDMAR的启动子区转录而来。Psi-LDMAR诱导RNA依赖的DNA甲基化;这导致LDMAR的转录减少,从而降低58S在长时间下的育性[10].这些最近发现的植物LncRNAs强调了它们对重要的适应性性状的影响,如雄性不育(LDMAR)和开花时间(COLDAIR, COOLAIR, IPS1) [8,9].lncrna对染色质结构调节的影响表明它们参与了允许植物对环境线索做出反应[3.].
lncrna也在其他植物中被鉴定和研究,包括玉米,小麦而且栽培稻[11- - - - - -13].这些lncrna的全基因组鉴定是使用现有的EST序列、全长cDNA数据库和/或全基因组平铺微阵列完成的[11- - - - - -13].李等人。[11]在水稻中发现了超过2万个假定的LncRNAs;尽管> 90%被认为是小RNA前体。相似的结果在玉米其中~ 60%的lncrna可能是小rna前体[14].大约40%的水稻非外显子转录活性区域似乎是非编码rna [11].刘等。[5]在模型植物中发现了6480个LincRNAs拟南芥(十字花科)。其中一些假定的L(i) ncrna通过表达模式分析、定制微阵列和RNA-seq进一步验证[5,11- - - - - -13].然而,迄今为止,所有这些研究都依赖于对单一物种的分析。
跨物种的全基因组比较表明,蛋白质编码基因不仅因序列而保守,而且因其在基因组中的位置而保守(例如synteny) [15].在不同的系统发育尺度上,基因组位置的守恒可以表明给定基因的位置处于强烈的纯化选择之下[16].的全基因组复制历史拟南芥(芸苔科)是通过鉴定和分析由多个古代全基因组重复产生的共线复制块而发现的[17].最近,基因组Aethionema arabicum是芸苔科最早分化谱系中Aethionemeae族的一员,经测序[18的基因组Tarenaya hassleriana芸苔科的姐妹科[19].通过对这三个基因组的比较,我们可以深入了解哪些基因和基因间区域可能在芸苔科和cleomaceae之间的位置上是保守的。然而,基因组序列还不足以理解其潜在的功能意义。因此,获得转录组数据来补充物种进化重要位置的基因组数据,以推断包括LncRNAs在内的调控转录本的位置守恒也是很有价值的。
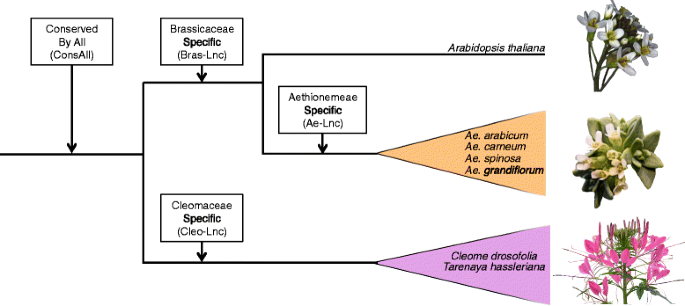
这里我们用的是Ae。arabicum,t . hassleriana而且答:芥除了我们新生成的四种Aethionemeae和两种Cleomaceae物种的转录组数据外,还可以了解LncRNAs在系统基因组环境中的保存(图2)。1).我们不仅分析了lncrna的核苷酸保守性,还分析了它们是否因基因组位置而保守。我们发现,在Cleomaceae、Brassicaceae或Aethionemeae中似乎具有序列特异性(例如谱系特异性)的LncRNAs中,> 25%是按位置保守的。这种位置保护可以告诉我们更多关于这些lncrna的假定功能,以及这些基因组特征位置保护的进化重要性。
结果
序列保护
我们从四种Aethionemeae和两种Cleomaceae物种的转录组数据中鉴定出LncRNAs。为了评估这些lncrna的序列守恒性,我们使用了OrhtoMCL [20.].对于位置守恒,我们使用CoGe工具SynFind和GeVo [21].
我们使用了之前的lncrna分类拟南芥[5]: 1)如果转录单位(TU)距离最近的蛋白质编码基因≥500 bp,无论在正反意义链上。2)基因相关转录单位(GATU),如果TU在蛋白编码基因的500bp范围内。3)“TU编码NAT”,如果该TU是从蛋白质编码基因的意义链的相反链转录而来。4) miRNA前体,可以有很长的转录本作为前体。
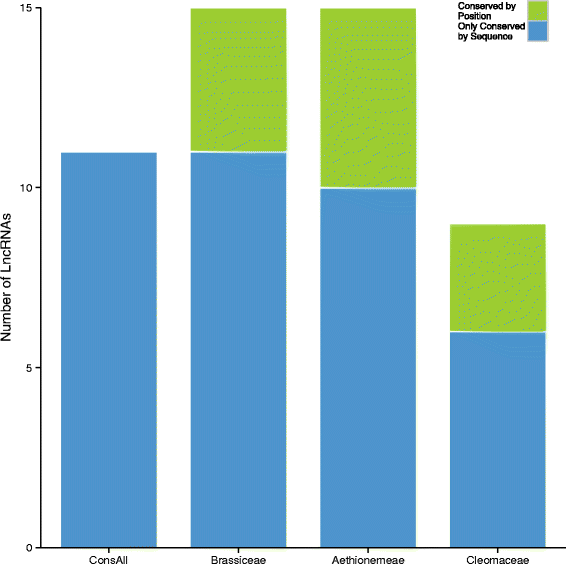
我们评估了6480答:芥LincRNAs (Ath-Linc)由[5OrthoMCL分析显示,十字花科(Brassicaceae)和Cleomaceae (All-Lnc)均为保守;基于倒数最佳爆炸命中的聚类算法[20.].分析包括Ath-Linc和Aethionema arabicum而且Tarenaya hassleriana(见方法和附加文件1:详情见图S1).因为lncrna的突变率高于蛋白质编码序列[14,22],采用递增序列相似性截断值≥10%、≥20%和≥50%进行分析。在6480个Ath-Lincs中,只有11个在基因组水平上被所有三个物种所保留。在这11个保守的Ath-Lincs中,根据我们的RNA-seq数据(见下文)和[的RNA-seq数据],只有9个在所有三个物种中转录。5](附加文件2:表S1为这些lncrna的平均转录本和ORF长度)。将保守的Ath-Lincs(局部BlastN)与ncbi数据库进行比对,以评估该序列在其他生物中是否保守。At3NC056191,序列相似度≤20%Ae。arabicum而且t . hassleriana转录组和基因组序列均与卵菌的5.8S核糖体RNA基因和内部转录间隔2序列同源角膜白斑laibachii。基因组保守的At2NC003370、At4NC004390和At4NC004390在包括苔藓在内的大多数陆生植物中都是保守的Physcomitrella金属盘(附加文件3.).
我们定义了一个谱系特异性的LncRNA,在核苷酸水平上由我们的焦点谱系中的多个物种共享(例如芸苔科,Aethionemeae或Cleomaceae),但在其他谱系中没有发现。有15个Ath-Lincs是芸苔科特有的(Bras-Lnc,见图。1).来确定Ath-Lincs和它们对应的Ae。arabicum转录本仅限于芸苔科,我们使用BlastN, BlastX和TblastX将其与NCBI和Phytozome数据库进行了比较方法和附加文件1详情及分界值见图S1)。在15个ras- lncs中,9个是由Ae。arabicum和/或答:芥(附加文件4的平均记录和ORF长度见表S3Ae。arabicum成绩单)。
为了测试Aethionemeae特定的lncrna (Ae-Lnc),我们生成了四种Aethionemeae物种的RNA-seq数据:Ae。arabicum, Ae。carneum, Ae。羊藿而且Ae。spinosa。我们鉴定了15个在这四个Aethionemeae物种之间序列相似度≥50%的LncRNAs Ae-Lncs(见方法和附加文件5:管道为图S2)。这15个Ae-Lncs分别对应15、15、16和20个转录本Ae。arabicum, Ae。carneum, Ae。羊藿而且Ae。spinosa分别来自19,037、18,305、48,609和60,772个预测转录本。四个物种中假定的lncrna的平均ORF长度(±SD)为145.89 bp(±10.00 bp),平均转录本长度为546.83 bp(±28.63 bp SD)(附加文件)6:物种特定平均值表S4)。Ae-Lnc由2个gau、4个编码nat的tu和9个lincrna组成(附加文件)3.和附加文件7:表S2)。两种Ae-LncRNAs是ath-MIR403和aly-MIR408的微rna前体(MFE分别为−71.8和−74.2千卡/摩尔)。虽然ath-MIR403不是组织特异性表达,但在缺氧条件下,它在叶片和整株植物中比在根中更多地存在[23,24].ally - mir408的功能和组织特异性尚不清楚[25].
Cleomaceae-specific LncRNA (Cleo-Lnc)的RNA-seq数据Tarenaya hassleriana而且醉蝶花属droserifolia如上所述,对Ae-Lnc进行了相同的分析(附加文件5:图S2)。我们根据84,967份cleomaceae基因转录本鉴定出9个cleomaceae特异性LncRNAt . hassleriana54,332份成绩单c . droserifolia序列相似性≥50%.这9个转录本的平均ORF和转录本长度(±SD)分别为181.5 bp(±7.78 bp)和675.71 bp(±201.53 bp)(附加文件)4:表S3为物种特定长度)。根据上述分类,这9个lncrna包括2个GATUs, 4个编码NATs的TUs和3个假定的LincRNas。我们没有发现任何假定的microRNA前体。
通过转录lncrna的位置进行保存
为了排除保守的非编码序列(CNSs)并支持功能,我们只考虑了我们检测到至少由一个物种转录的lncrna。
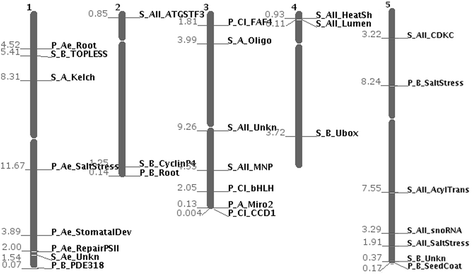
我们分析了每个分支转录的谱系特异性lncrna,以及它们在另一个谱系的基因组中的位置是否保守。使用CoGe-tools CoGeBlast、SynFind和GeVo ([21),看方法详情)。在核苷酸水平上似乎具有谱系特异性的39个lncrna中(例如,在支之间高度分化;15个Bras-Lncs, 15个Ae-Lncs和9个Cleo-Lncs) 12个在至少一个其他谱系中位置保守(见图。2以获取示例和附加文件8:其他图S3-S9)。根据不同的分支(Aethionemeae特异的,Cleomaceae特异的或Brassicaceae特异的),在另一个分支中不按序列保守而是按位置保守的lncrna的百分比在26% - 33%之间变化(图2)。3.和附加文件7:表S2)。数字4的位置保守的lncrna分布答:芥基因组。值得注意的是,66.66%(12个中有8个)的位置保守的lncrna位于距离染色体末端2.5 MB以内,包括在端粒下区域。4和附加文件7:表S2)。这与其他人的发现相一致,即端粒和端粒下区域的基因密度高于基因组平均水平[26].这可能表明基因调控元件的数量较高。
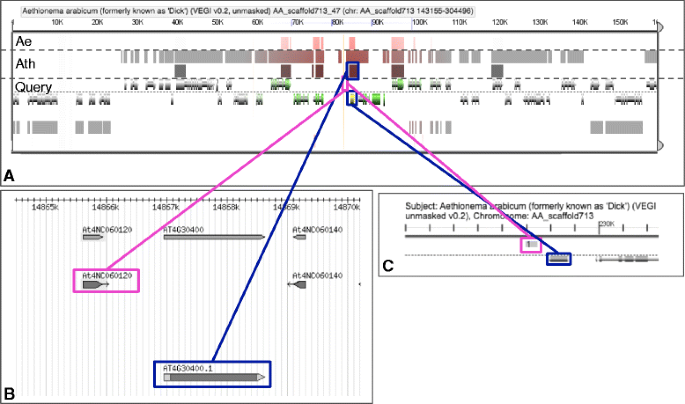
共线性的例子和长链非编码RNA (LncRNA)的位置守恒分析。一个截图来自GeVo。GeVo计算查询序列与主题生物体基因组的共线性。这里的查询是最近的蛋白质编码基因Ae。arabicum显示于(c),科目为Ae。arabicum而且答:芥。这里有两个共线区域答:芥。位置保守的LncRNA的位置如图所示粉红色的而蛋白质编码基因的答:芥而且Ae。arabicum显示为蓝色的盒子。bPLncDB网站截图,显示的是拟南芥LncRNA (粉红色的)和最接近的蛋白质编码基因(蓝色的).c截图来自CoGe Blast HSP。粉红色的是Aethionema arabicum抄本沿着Ae。arabicum基因组。蓝色的最近的是Ae。arabicum蛋白质编码基因。这个SynFind和GeVo分析可以通过以下链接重新完成:https://genomevolution.org/r/fmnf
表格1显示了相邻基因对位置保守的lncrna的功能。BrassLnc和Ae-Lnc相邻基因(AT5G62420、AT5G24270和AT1G50640)与盐胁迫反应相关。的答:芥位置保守的黄铜- lnc和Ae-Lnc相邻基因参与了不同的形态和生理发育水平。从影响根的生长,到气孔的发育,到修复光系统II,到胚胎发生和线粒体形态发生(表2)1).
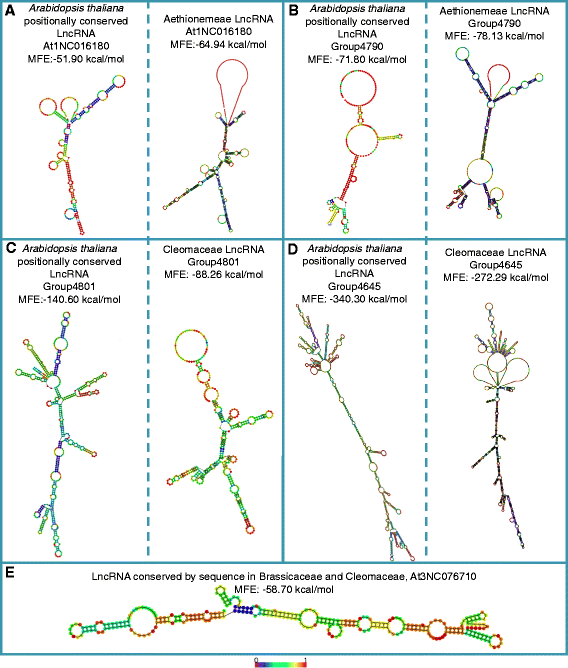
一些lncrna已被证明具有茎环二级结构[9,27,28].我们研究了位置保守的LncRNA是否具有假定稳定的二级结构,以及位置保守的LncRNA之间是否存在共同特征(图2)。5和附加文件9:图S10)。二级结构的稳定性由其最小自由能(Minimum Free Energy, MFE)决定,假设能量越低,结构越稳定[29].因此,我们认为MFE≥- 80 kcal/mol的结构是不稳定的。Ae-Lnc和Ath-Linc的二级结构因此是不稳定的(图。5).两个Cleo-Linc和Bras-Linc更稳定(图。5).根据其他lncrna的二级结构[9,27,28)所有稳定的结构都有长茎和一边的大环(图。5).
序列和/或位置保守的长链非编码rna (LncRNAs)的二级结构和最小自由能(MFE)一个拟南芥之间既有序列对话又有位置守恒的lncrna (左)和Aethionema (正确的) (b)在拟南芥之间只存在位置守恒的lncrna (左)和Aethionema (正确的) (c拟南芥之间既有序列对话又有位置守恒的lncrna (左)和Tarenaya (正确的) (d)在拟南芥之间只存在位置守恒的lncrna (左)和Tarenaya (正确的) (e) A. thaliana中LncRNA序列和位置保守,Aethionemea arabicum和Cleomaceae.下面的彩色条显示了每个结构的基概率
讨论
随着更完整的基因组变得可用,除了序列相似性外,还可以使用遗传共线性来解决系统基因组环境中非编码序列的保守性问题。通过与姐妹科芸苔科(Brassicaceae)和Cleomaceae的比较,我们发现LncRNAs在位置上是保守和表达的,但在核苷酸水平上高度分化。因此在这里我们发现植物的lncrna是位置保守而不是序列保守的,而序列保守的lncrna是位置不保守的。虽然这一结果已在较早的比较动物研究中得到描述[30.],据我们所知,我们的工作代表了这一趋势在植物中的第一个例子。
长(基因间)非编码rna已被证明影响其邻近基因的表达[30.],由此可见,位置保护在正确调控编码各种性状的相邻基因中具有重要意义。例如,在这里发现的位置保守的lncrna与相关基因相邻:对盐胁迫的响应,影响重要的生理功能(如光系统II修复机制)或影响形态结构(如根的生长)。
我们的位置守恒分析基于最新的可用基因组arabicum, Tarenaya hassleriana而且拟南芥。最新出版的Aethionema arabicum基因组占其总基因组大小的85% [18]和最新公布的基因组Tarenaya hassleriana是其总基因组大小的94% [19].虽然这些基因组已经发表,但我们的分析总是受到基因组组装质量的限制。
长链非编码rna是真核生物基因调控和基因组的一个潜在的重要特征。迄今为止,在脊椎动物中对lncrna的研究比在植物中更广泛。在48个功能验证的脊椎动物lncrna中,有25个在人和小鼠之间的序列相似性为50% [31].刘等。[5],他们的数据已经在这里进行了探索,发现在他们发现的所有假定的lncrna中,< 2%答:芥在整个植物王国都是保守的。通过比较玉米(单子叶)lncrna和答:芥(eudicot) [12].豆科植物的LncRNAs在非豆科植物中仅显示5%的序列保守[32].比例要高得多玉米LncRNAs < 25%,在近缘种高粱中保守[12].在这里,我们发现在总共39个在核苷酸水平上分化的转录lncrna中,有12个在位置上是保守的。这是我们在Aethionemeae和Cleomaceae的转录组中发现的30%以上的LncRNAs。
考虑lncrna位置的研究主要假设序列守恒,并额外分析这些lncrna是否也由位置守恒。然而,在斑马鱼和人类的比较中,Batista和Chang (2013) [30.]发现弱序列守恒的lncrna仍然可以具有完整的功能,因为它们在结构和位置上仍然是保守的。在这里,我们在植物中显示了类似的结果:远亲物种之间LncRNAs的位置守恒和弱序列相似性。
缺乏序列守恒,但存在位置守恒可能是由于这些调控元件的突变率增加。庞等人已经指出了这一点。[31],他假设,对于mirna和更长的非编码rna,调控网络内的相互作用类型可能受到选择压力,而不是调控元件本身的序列。这一假设与lncrna的调控功能和位置非常吻合。当lncrna调节其邻近蛋白质编码基因的表达时,它们与该基因的相互作用,因此它们的位置而不是它们的序列可以被选择。
我们比较了位置保守的lncrna的二级结构(图2)。5).除了lncrna的位置守恒外,其二级结构也可能守恒。Aethionemeae位置保守的lncrna比Cleomaceae位置保守的lncrna更不稳定(MFE更高)。在它们的位置守恒中也可以看到类似的稳定性差异拟南芥。LncRNA二级结构的稳定性可能是细分LncRNA大群体的一个步骤。
不同物种的基因组区域可以在序列上相似,也可以完全共线。然而,这些序列并不是必须转录的(见附加文件)7:表S2)。在这里,我们使用聚腺苷化mrna来评估lncrna在不同物种之间的保守性。已有研究表明,尽管lncrna可以被聚腺苷化,但它们并不总是被聚腺苷化[33].因此,位置保守只显示了植物可能转录本的一个子集。此外,我们应用了严格的规则,即每个LncRNA必须在同一谱系的至少两个物种中转录。因此,这些结果是一组高度自信的位置保守的lncrna,仅代表了冰山一角。
这里发现的少量保守的lncrna与上面讨论的其他系统的发现相一致[5,12,32].低核苷酸守恒的一致性发现提出了lncrna突变率的新问题。研究表明lncrna的突变率与内含子的突变率相似[12,30.,31,34],这可能部分解释了lncrna在深度进化过程中缺乏序列相似性。然而,这种序列相似性的缺乏并没有导致位置守恒的缺乏,这可能表明了函数守恒,因为前面已经表明,位置守恒也解释了函数守恒[15,30.].
超过65%的位置保守的lncrna仅在2.5 Mb染色体臂内的存在是显著和意外的。在许多植物的次端粒区域由重复序列组成,称为卫星,尽管这些在植物中是不存在的答:芥[26].它们的存在因物种而异,甚至同一物种内的个体也各不相同[26].亚端粒区域的卫星通常由大量富含A-T的重复延伸组成,这使得DNA更容易弯曲,异染色质形成更紧密,这可以通过致密异染色质块的存在来证明[26,35].这些卫星阵列的存在的一个被认为的功能是它们支持染色体亚端粒区域的染色质状态[26].然而,卫星阵列的缺席答:芥可能由调节亚端粒区蛋白质编码基因染色质特征的lncrna的存在来补偿。我们不知道为什么位置保守的lncrna只存在于染色体末端的具体原因。当然,需要更多的研究来解决这一发现和上述假设。
最好我们已经测试了位置保守的lncrna是否也在染色体臂的2.5 Mb内Aethionema arabicum和/或Tarenaya hassleriana.然而,这些物种的染色体水平的基因组组装还没有得到。然而,我们正在研究这些基因组组合,以便在不久的将来解决这些问题。
通过研究敲除这些保守序列对各种性状(如开花时间、生育力等)的有害影响,已鉴定出长(基因间)非编码rna。6,8,9].这些湿实验室实验对于理解任何假定途径的功能(从基因和转录到适应性效应)至关重要。他们可以确认LncRNA中缺乏小orf,并了解LncRNA影响的完整途径,无论是对邻近基因还是跨染色体[30.,36].
结论
综上所述,我们在芸苔科和Cleomaceae系统基因组系统中发现,在序列水平上似乎只在一个世系中保守的植物长链非编码rna (Long non-coding RNAs, LncRNAs)在其他世系中相同的基因组位置上是保守的。位置守恒也可以是函数的守恒,而不是序列的发散。此外,65%的位置保守的lncrna位于端粒区域2.5 Mb内。这强调了LncRNAs可以发挥的基因调节作用。这些结果表明,谱系特异性不仅应在核苷酸水平上考虑,还应在位置水平上考虑。
方法
转录组分离、文库制备和组装
arabicum Aethionema;carneum, Ae。、Ae。大花莲,大花莲而且醉蝶花属droserifolia种子在播种土壤中发芽,在阿姆斯特丹大学温室(夜间18°C,白天20°C,光照12小时,黑暗12小时)中生长。表格2显示用于RNA分离的组织。为了减少RNA降解,将组织在液氮中磨碎,立即使用PureLink™RNA迷你试剂盒(Ambion, Life Technologies Corporation, Carlsbad, CA, USA)分离RNA,然后根据制造商的协议,使用TURBO无dna™试剂盒(Ambion)进行DNase处理。RNA的质量和数量在1%琼脂糖凝胶上用溴化乙胺染色,在1x TBE缓冲液和NanoDrop 1000©分光光度计上进行检查(赛默飞世尔科学公司,威尔明顿,德州,美国)。样品用GenTegraTM (GenVault, Carlsbad, CA, USA)干燥,然后运往密苏里-哥伦比亚大学的测序中心。ds-cDNA文库按照TruSeq-RNATM试剂盒(Illumina, San Diego, CA, USA)的制造商协议构建。这里使用的六个新的转录组是在cDNA合成过程中为mRNA选择的。因此,所有非聚腺苷化lncrna均未测序。Aethionema羊藿而且答:spinosa用Illumina Hiseq2000测序仪在1x100bp的车道上配对端测序,每车道3行。的Ae.arabicum转录组是新创用三位一体组装[37].的Ae。carneum, Ae。羊藿而且Ae。spinosa转录组被组装Ae。arabicum使用NextGene V2.17®(SoftGenetics, State College, PA, USA)进行contigs,匹配要求≥40 bp,相似性≥90%,且≤20%存在突变。对于每一行,按照以下参数设置构建一致序列:90%的最小纯合子序列,25%的最小杂合子序列,85%的最小纯合子序列。
基因组、CDSs和LncRNA
的Athionema arabicum而且Tarenaya hassleriana基因组已从CoGe网站下载[19].信用违约掉期油菜,拟南芥而且Eutrema halophila来自PlantGDB网站[38]和拟南芥(Ath) CDS v10 from TAIR [39].蛋白质组玉蜀黍属梅斯,水稻,短柄草,双色高粱而且高粱italica已从Phytozome下载[40].这些后一种CDS和蛋白质组用于OrthoMCL分析(见附加文件)5:图S2)以确定lncrna是谱系特异性的。Ath lncrna (Ath- lnc)的位置可从PLncDB网站下载[41],并用于提取序列答:芥染色体(39]使用内部python脚本。Phytozome中2013年11月出现的所有基因组[40],以供日后分析。
OrthoMCL,爆破和位置守恒分析
OrthoMCL [20.,该算法基于互反最佳爆炸命中(RBH),并使用聚类算法(MCL)对RBH进行聚类。根据所执行的Blast,可以将OrthoMCL用于核苷酸或蛋白质序列。我们使用OrthoMCL和BlastN,查询identity = 50%, value = 1e-10,用于将同源组分配给感兴趣的谱系(附加文件1:图S1和附加文件10:图S11)。所有的爆炸都是用命令行爆炸[42]对Phytozome内部制作的基因组数据库和/或对NCBI数据库使用' -remote '命令。ORF大小通过VirtualRibosome网站评估[43]并使用严格的开始密码子。的位置Ae。arabicum成绩单和t . hassleriana用CoGeBlast评估其自身基因组上最近基因的转录本[21].的Ae。arabicum揭开基因组v2.5和t . hassleriana使用未蒙面基因组V4。只有当转录本的查询命中率≥50%且HSP = 1时,才假定它们命中了基因组上的正确位置。这一假设排除了选择性剪接,冗余基因组命中也是如此。SynFind及GeVo [21]用于评估LncRNA蛋白编码基因最近区域与答:芥,Ae。arbicum和/或t . hassleriana基因组(s)。例如:Ae-Lnc的一个蛋白编码基因与中的一个区域共线答:芥PLncDB的“GenomeBrowse”实用程序[41]来评估是否存在与Ae-Lnc位置相对应的同一方向(上游、下游或天然反义)的LncRNA(见图。2).因此,这些lncrna在序列水平上不同,但在位置上相似(另见附加文件)10:反例如图S11)。
所有转录本都进行了测试,以确定它们是否可能是微rna前体。为此,他们被炸(BlastN)对mirBase数据库[44].我们使用RNAfold服务器[45]来观察转录本是否具有稳定的二级结构作为microRNA。当吉布斯自由能在- 30到- 80千卡/摩尔之间时,认为该结构是稳定的。
保守的LncRNA和二级结构
根据附加文件中描述的管道测试LncRNA的守恒1:图S1。这是在管道的开始,用10%,20%和50%的OrthoMCL分析查询标识完成的。
为了评估位置保守的lncrna是否具有稳定的二级结构,研究了RNAalifold和RNAfold服务器[45]被使用。RNAalifold使用两个以上物种的排列序列,而RNAfold基于单个RNA序列计算二级结构。Ae-Lncs我们用的是Ae。arabicum, Ae。羊藿Ae。carneum而且Ae。spinosa。来自同一物种的转录本(如果在OrthoMCL分析中存在,见上文)用于芸苔科特定的LincRNAs。对于Cleomaceae的特定LincRNAst . hassleriana而且c . drosofolia被使用。为了比较位置保守的lncrna,还计算了Ath-Linc的二级结构。
缩写
- LncRNA:
-
长链非编码RNA
- LincRNA:
-
长基因间非编码RNA
- Ath-Linc:
-
长基因间非编码RNA来自[5]
- Ae-Lnc:
-
4种aeethionemeae种在核苷酸水平上保守的长链非编码RNA
- Brass-Lnc:
-
长链非编码RNA在核苷酸水平上被保守拟南芥而且Aethionema arabicum
- Cleo-Lnc:
-
两个Cleomaceae物种在序列水平上保守的长非编码RNA
- All-Lnc:
-
芸苔科和Cleomaceae在核苷酸水平上保守的长链非编码RNA
参考文献
- 1.
Axtell乔丹。植物小分子rna的分类与比较。植物学报。2013;34(4):529 - 529。
- 2.
张娟,Mujahid H,侯勇,Nallamilli BR,彭哲。植物长链ncRNAs:基因调控的新前沿。植物科学学报,2013;
- 3.
Wierzbicki。长链非编码RNA在转录基因沉默中的作用。植物学报,2012;15:517-22。
- 4.
梅丁杰,彭建昌,莫瑟TR,马蒂克JS。区分蛋白质编码和非编码RNA:挑战和歧义。3 .《公共科学图书馆·计算生物学》,2008;
- 5.
刘娟,荣昌,徐娟,王辉,邓松,Bernad L,等。全基因组分析揭示了拟南芥长基因间非编码rna的调控。《植物科学》2012;24:433 - 45。
- 6.
张永春,陈永强。长链非编码rna:植物发育中的新调控因子。生物化学学报。2013;436:111-4。
- 7.
Csorba T, Questa JI,孙Q, Dean C.反义COOLAIR介导春化过程中FLC染色质状态的协调切换。中国科学院学报,2014。
- 8.
刘芳,刘志刚,刘志刚,刘志刚。拟南芥Polycomb基因冷诱导的长反义转录本沉默。大自然。2009;462:799 - 802。
- 9.
丁俊,卢强,欧阳阳,毛华,张鹏,姚杰,等。一种长链非编码RNA调节光周期敏感雄性不育,这是杂交水稻的基本组成部分。《美国国家科学院学报》上。2012, 317784(农垦58)。
- 10.
丁娟,沈娟,毛红,谢伟,李霞,张强。rna介导的DNA甲基化参与调控水稻光周期敏感雄性不育。植物学报。2012;5:1210-6。
- 11.
李玲,王旭,Sasidharan R, Stolc V,邓伟,何华,等。水稻基因组中转录活性区域的整体鉴定和特征。PLoS ONE。2007; 2: e294。
- 12.
李玲,刘志强,刘志强,刘志强,等。玉米长链非编码rna的全基因组发现和表征。中国生物工程学报。2014;15:R40。
- 13.
辛敏,王勇,姚勇,宋楠,胡震,秦东,等。利用微阵列分析和SBS测序鉴定小麦长链非蛋白编码rna对白粉病感染和热胁迫的响应。BMC植物生物学2011;11:61。
- 14.
Boerner S, McGinnis KM。玉米长链非编码RNA的计算鉴定与功能预测。PLoS ONE。2012; 7: e43047。
- 15.
唐华,Bowers JE,王旭,Ming R, Alam M, Paterson AH。植物基因组的共线性和共线性。科学(纽约,纽约州)。2008; 320:486-8。
- 16.
李志刚,张志刚,张志刚,等。番茄、马铃薯和拟南芥共联转化酶基因家族的功能分化。植物物理学报2003;131(2):603-9。
- 17.
杨晓明,王晓明,陈晓明,等。基于染色体复制事件的被子植物基因组进化研究。大自然。2003;422(3):433 - 8。
- 18.
杜德华,王志强,王志强,等。超过90,000个保守非编码序列的图谱提供了对十字花科调控区域的深入了解。Nat Genet, 2013; 45:891-8。
- 19.
程松,van den Bergh E,曾鹏,钟霞,徐娟,刘霞,等。研究了十字花科植物Tarenaya hassleriana基因组,为研究十字花科植物的生殖特性和基因组进化提供了新的思路。《植物科学》2013;
- 20.
李磊,Jr CJS, Roos DS。真核生物基因组正交基的鉴定。基因组研究。2003:2178 - 89。
- 21.
Lyons E, Freeling M.如何有效地比较同源植物基因和染色体作为DNA序列。植物J. 2008; 53:661-73。
- 22.
Guttman M, Amit I, Garber M, French C, Lin MF, Huarte M,等。染色质特征揭示了哺乳动物中超过一千种高度保守的大型非编码rna。大自然。2009;458:223-7。
- 23.
孙晓峰,朱军。拟南芥中新型的胁迫调控MicroRNAs和其他小rna。植物细胞。2004;16(8):2001-19。
- 24.
Moldovan D, Spriggs A, Yang J, Pogson BJ, Dennis ES, Wilson IW。拟南芥中缺氧响应microrna和反式作用小干扰rna。中国科学(d辑),2010;
- 25.
马Z,科鲁C, Axtell MJ。拟南芥小RNA:拟南芥属内的瞬时MIRNA和小干扰RNA位点。《植物科学》2010;22:1090-103。
- 26.
Bass HW, Birchler JA。植物细胞遗传学。纽约,纽约:施普林格纽约;2012.
- 27.
flintft L.非编码RNA: lncrna的结构和功能。Nat Rev Genet. 2013;14:598。
- 28.
Novikova IV, Hennelly SP, Sanbonmatsu KY, Rna K. lncrna有二级和三级结构吗?评估长非编码rna。BioArchitecture。2012; 2:189 - 99。
- 29.
朱克M,施蒂格勒P.优化计算机折叠大RNA序列使用热力学和辅助信息。核酸决议1981;9:133-48。
- 30.
张海燕。长链非编码rna:发育与疾病中的细胞地址码。细胞。2013;152:1298 - 307。
- 31.
庞kc, Frith MC, Mattick JS。非编码rna的快速进化:缺乏保守性并不意味着缺乏功能。《趋势》,2006;22:1-5。
- 32.
温军,帕克,王杰,魏勒。截断紫花苜蓿mrna样非编码转录本的硅片鉴定与鉴定。《生物科学》2007;7:485-505。
- 33.
狄超,袁杰,吴勇,李军,林华,胡林,等。拟南芥胁迫响应型lncrna的表达、表观遗传和结构特征分析植物J. 2014; 80:848-61。
- 34.
马蒂克JS,加根MJ。回顾文章受控多任务基因网络的进化:内含子和其他非编码rna在复杂生物发育中的作用。分子生物学杂志2001;18(9):1611-30。
- 35.
马卡斯,梅萨洛斯,卢佐瓦。植物卫星:植物卫星重复序列的专门数据库。生物信息学(牛津,英国)。2002; 18:28-35。
- 36.
韩田,张旭,赵柏维,李文华,邵世华。拟南芥基因组的基因间区有大量新的编码小开放阅读框被转录和/或进行纯化选择。Genome res 2007; 17:632-40。
- 37.
Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson JD, Amit I,等。三位一体:从RNA-Seq数据中重建没有基因组的全长转录组。中国生物工程学报,2011;29(4):644 - 52。
- 38.
Duvick J, Fu A, Muppirala U, Sabharwal M, Wilkerson MD, Lawrence CJ,等。PlantGDB:比较植物基因组学的资源。核酸决议2008;36(数据库issue): D959-65。
- 39.
胡华拉,吴志强,刘志强,等。拟南芥信息资源(TAIR):模型植物的综合数据库和基于网络的信息检索、分析和可视化系统。核酸决议2001;29:102-5。
- 40.
郭德华,陈志强,张志强,等。Phytozome:绿色植物基因组学的比较平台。核酸决议2012;40(数据库issue): D1178-86。
- 41.
金俊,刘俊,王华,王玲,蔡恩华。PLncDB:植物长非编码RNA数据库。生物信息学(牛津,英国)。2013; 29:1068 - 71。
- 42.
卡马乔C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K,等。BLAST+:架构和应用程序。生物信息学。2009;10:421。
- 43.
虚拟核糖体-一个全面的DNA翻译工具,支持整合序列特征注释。Nucleic Acids Res. 2006;34(Web Server issue): W385-8。
- 44.
格里菲思-琼斯S, Saini HK, van Dongen S, Enright AJ。miRBase: microRNA基因组学工具。核酸决议2008;36(数据库issue): D154-8。
- 45.
格鲁伯AR,洛伦兹R,伯恩哈特SH, Neuböck R,霍法克IL.维也纳RNA网络套件。核酸决议2008;36(Web服务器问题):W70-4。
- 46.
黄娟,DeBowles D, Esfandiari E, Dean G, Carpita NC, Haughn G.拟南芥转录因子LUH/MUM1对种皮粘液的挤压作用植物科学进展。2011;
- 47.
宋国平,加尔布瑞斯DW。AtSAP18是人类SAP18的同源基因,在拟南芥中参与盐胁迫的调节并介导转录抑制。植物科学进展。2006;30(4):344 - 344。
- 48.
小山T, Nii H, Mitsuda N, Ohta M,北岛S, Ohme-Takagi M,等。涉及II类乙烯反应因子转录抑制因子的级联调控在叶片衰老过程中起作用。中国生物医学工程学报,2013;
- 49.
工程师CB, Ghassemian M, Anderson JC, Peck SC, Hu H, Schroeder JI。碳酸酐酶、EPF2和一种新型蛋白酶介导CO2对气孔发育的控制。大自然。2014;513:246 - 250。
- 50.
金华,刘波,罗林,冯东,王鹏,刘杰,等。在拟南芥中,高光敏感基因1与PHOTOSYSTEM II1的低量子产量相互作用,保护PHOTOSYSTEM II免受光损伤。植物细胞。2014;26:1213-29。
- 51.
山冈S, Leaver CJ。EMB2473/MIRO1是一个拟南芥Miro GTPase,是胚胎发生所必需的,并影响花粉中的线粒体形态。植物细胞,2008;20:589-601。
- 52.
tolido - ortiz G, Huq E, Quail PH.拟南芥基本/螺旋-环-螺旋转录因子家族。植物细胞。2003;15:1749-70。
- 53.
郭永林,郭永林,郭永林。FANTASTIC FOUR蛋白对拟南芥芽分生组织大小的影响。中国生物医学工程学报。2010;
- 54.
梁玉生,全永阿,林世华,金桂桂,李建勇,金永明,等。拟南芥类胡萝卜素切割双加氧酶7基因启动子的血管特异性活性。植物科学进展。2011;30:973-80。
确认
本研究由NWO Vernieuwings impulse VIDI资助(授权号:864.10.001)。我们也感谢Frank Becker和Lars Chatrou提供的图片Tarenaya hassleriana而且拟南芥无花果。1.
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
所有的分析和图表都是由SM完成的。MES和JCP对稿件进行了修改。PPE对手稿进行了修改,并整理了抄本。MES和SM撰写了手稿。所有作者都已阅读并批准了论文的最终版本。
附加文件
附加文件1:图S1。
评估转录的(上)和基因组的(下)长链非编码rna (LncRNA),这些rna在整个芸苔科和Cleomaceae中在核苷酸水平上是保守的,或者是芸苔科特有的。(PDF 42kb)
附加文件2:表S1。
抄本和ORF的长度Tarenaya hasslerianaAethionemeae转录本按顺序保存。序列相似度百分比是OrthoMCL中序列相似度的截断点。(docx55kb)
附加文件3:
十字花科的长链非编码rna,基因组。(xlsx41 kb)
附加文件4:表S3。
aeethionemeae和Cleomaceae特异性Long非编码rna的转录本和ORF长度。(docx39kb)
附加文件5:图S2。
用于评估特定的lncrna的管道Aethionemeae或Cleomaceae。(PDF 51kb)
附加文件6:表S4。
芸苔科特异的Aethionemeae转录本和ORF长度。序列相似度百分比是OrthoMCL中序列相似度的截断点。(docx56 kb)
附加文件7:表S2。
表1显示的是arabicum, Tarenaya hassleriana而且拟南芥以及它们是由位置守恒还是仅由不同谱系的序列守恒。表2显示了位置保守的lncrna到的最近一端的距离答:芥染色体。(xlsx22 kb)
附加文件8:图S3-S6。
序列多样性Aethionemeae lncrna的共线性和位置守恒分析。(A)来自GeVo的截图。GeVo计算查询序列与主题生物体基因组的共线性。这里查询的是最近的蛋白质编码基因Ae。arabicum如图B所示,受试者为Ae。arabicum而且答:芥。位置保守的LncRNA的位置用粉色方框显示,编码的蛋白基因为答:芥而且Ae。arabicum用蓝色方框显示。(B) PLncDB网站截图,显示的是拟南芥LncRNA(粉色)和它最近的蛋白质编码基因(蓝色)。(C) CoGe Blast HSP的截图。粉色是Aethionema arabicum抄本沿着Ae。arabicum基因组。蓝色是最接近的Ae。arabicum蛋白质编码基因。图S7-S9。Cleomaceae序列多样性lncrna的共线性和位置守恒分析。(A)来自GeVo的截图。GeVo计算查询序列与主题生物体基因组的共线性。这里查询的是最近的蛋白质编码基因Taranaya hassleriana,研究对象为t . hassleriana而且答:芥。位置保守的LncRNA的位置用粉色方框显示,编码的蛋白基因为答:芥而且t . hassleriana用蓝色方框显示。(B) PLncDB网站截图,显示的是拟南芥LncRNA(粉色)和它最近的蛋白质编码基因(蓝色)。(ZIP 716kb)
附加文件9:图S10。
序列和/或位置保守的LncRNAs的二级结构和最小自由能(MFE)。(A)拟南芥(左)和Aethionema(右)之间既有序列对话又有位置守恒的lncrna (B)仅在拟南芥(左)和Aethionema(右)之间既有序列对话又有位置守恒的lncrna (C)拟南芥(左)和Tarenaya(右)之间既有序列对话又有位置守恒的lncrna (E)。下面的彩色条显示了基基每个结构的概率。(PDF 667 kb)
附加文件10:图S11。
序列保守的LncRNA共线性分析和无位置守恒的例子。例如,在芸苔科中,序列水平保守的LncRNA。A) PLncDB网站截图,显示的是拟南芥LncRNA(绿色)及其最近的蛋白质编码基因(蓝色)。B) CoGe Blast HSP的截图。绿色是Aethionema arabicum抄本沿着Ae。arabicum基因组。蓝色是最接近的Ae。arabicum蛋白质编码基因。C)截图来自GeVo。GeVo计算查询序列与主题生物体基因组的共线性。这里查询的是最近的蛋白质编码基因Ae。arabicum如图B所示,受试者为Ae。arabicum而且答:芥。这里的查询显示了中的两个共线区域答:芥。LncRNA的位置为绿色方框,蛋白编码基因为答:芥而且Ae。arabicum用蓝色方框显示。D)放大答:芥与共线的区域Ae。arabicum并与最近的相对应答:芥a中所示的最接近的蛋白质编码基因。这些SynFind和GeVo分析可以通过以下链接重新进行:https://genomevolution.org/r/fmqj.(PDF 146kb)
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
默罕默丁,S.,埃杰,p.p.,皮雷,J.C.et al。位置保守但序列发散:芸苔科和Cleomaceae中长链非编码rna的鉴定。BMC植物生物学15日,217(2015)。https://doi.org/10.1186/s12870-015-0603-5
收到了:
接受:
发表:
关键字
- 蛋白质编码基因
- 最小自由能
- Spinosa
- 位置的保护
- 稳定二级结构