摘要
背景
DNA复制和转录是调节植物发育的动态过程,依赖于染色质的可及性。属于Agenet/Tudor结构域家族的蛋白质被称为组蛋白修饰“阅读器”,并被分类为染色质重塑蛋白。组蛋白修饰和染色质重塑对基因表达和DNA复制有深远的影响,但这些过程是如何整合的还没有完全阐明。很明显,Agenet/Tudor家族的成员是重要的发育调节因子,在植物中发挥着不为人所知的作用。
方法
利用现有全基因组数据库中的序列,对植物王国中Agenet/Tudor家族结构域进行了生物信息学和系统发育分析。通过I-TASSER服务器计算Agenet/Tudor结构域的三维结构预测。通过双杂交、GST下拉、半体内下拉和串联亲和纯化试验测试蛋白质相互作用。研究了T-DNA插入gabi系的基因功能。
结果
在目前的工作中,我们分析了植物王国中Agenet/Tudor结构域蛋白家族,并绘制了该家族在植物进化过程中的组织。此外,我们从拟南芥命名为AIP1,包含Agenet/Tudor和DUF724域。AIP1与DNA复制许可和基因转录的植物调节因子ABAP1相互作用,与植物组蛋白修饰“阅读器”(LHP1)和未修饰的组蛋白相互作用。AIP1在生殖组织中表达,其下调延迟花的发育时间。AIP1水平降低的植物花蕾中ABAP1和LHP1靶基因的表达也受到抑制。
结论
AIP1是植物中一种新型的Agenet/Tudor结构域蛋白,可能在花发育过程中发挥DNA复制、转录和染色质重塑之间的链接作用。
背景
染色质是一种高度调控的动态结构,在发育过程中不断被重塑,以便将基因转录事件与细胞分裂和分化等细胞过程耦合起来。组蛋白修饰是调节染色质重塑的重要机制,它们由特定的酶进行,然后由所谓的“组蛋白阅读器”蛋白质识别[1].Agenet和Tudor结构域,以及染色质结合(Chromo), Bromo, Bromo-毗邻同源(BAH), PWWP(保守脯氨酸和色氨酸)和恶性脑肿瘤(MBT)结构域被称为组蛋白修饰“阅读器”,存在于许多被归类为染色质重构的蛋白质中[2,3.].Agenet结构域最初被描述为更大的皇家结构域家族的植物特异性成员,因为它与脆性X智力迟钝蛋白(FMRP)的动物Tudor结构域相似[3.].后来,Agenet结构域在人蛋白中也有报道[4,5],因此该蛋白家族现在被称为Agenet/Tudor结构域家族。在过去的几年里,更多关于Agenet/Tudor蛋白功能的见解被揭示出来[5- - - - - -9],包括在人类FMRP的Agenet/Tudor结构域附近鉴定出一个负责rna结合功能的rna结合域(KH) [5].然而,对于Agenet/Tudor结构域在植物中的作用及其在植物发育中的重要性,人们知之甚少。
Agenet/Tudor结构域蛋白广泛存在于植物王国中,共鉴定出28个基因拟南芥基因组(2].emsy样n -末端(ENT)、BAH、植物同源结构域(PHD)和DUF724结构域被报道经常与植物Agenet/Tudor结构域共同出现,可能赋予这些蛋白不同的功能[3.].植物的ENT结构域类似于人类癌蛋白EMSY的结构域,据报道,EMSY是肿瘤抑制因子BRCA2转录激活因子功能的抑制因子[2].BAH结构域参与了芳香族笼的形成,该笼结合核小体的组蛋白H3赖氨酸9二甲基化(H3K9Me2),相互作用DNA甲基化和组蛋白修饰[10].PHD结构域是一类锌指(ZnF)基序,在多蛋白复合物中促进蛋白-蛋白相互作用,并参与染色质重塑和泛素化过程[11].DUF724结构域被报道参与介导蛋白-蛋白相互作用[4].到目前为止,在植物中只有包含ENT结构域的Agenet/Tudor具有功能特征。在拟南芥中,AtEMSY-like 1 (AtEML1)和AtEMSY-like 2 (AtEML2)被描述为与转录因子增强型霜霉病2 (EDM2)相互作用,负责抑制霜霉病的表达开花位点C(方法),从而影响花期的控制[12].另一种在玉米中报道的ENT/Agenet/Tudor蛋白被命名为R相互作用因子1 (RIF 1),它是一个复合物的一部分,该复合物固定在启动子区域的染色质中,增加组蛋白3赖氨酸9的乙酰化(H3K9/K14ac),以激活花青素生物合成途径中参与选择的基因的表达[13].此外,拟南芥Coilin蛋白具有c端Agenet/ tudor样结构,不含任何其他分类结构域,能够以非特异性的方式与RNA结合,随后进行多聚化,这可能有助于其作为脚手架蛋白的功能[14].
组蛋白修饰对基因表达以及DNA复制有深远的影响,但尚未完全阐明这些过程是如何整合的。在动物中,Agenet/Tudor结构域蛋白已被报道在DNA修复过程中染色质修饰中起作用,将其与细胞周期检查点连接起来。含有肿瘤抑制因子p53结合蛋白1 (53BP1)的串联都铎结构域可以与组蛋白修饰结合,标记双链DNA断裂(DSB) [7],并与甲基化视网膜母细胞瘤(RB)相互作用;通过这种方式,它将RB的细胞周期控制与DNA损伤反应和染色质重塑过程联系起来[7].Spindilin是一种来自人类的都铎结构域蛋白,与甲基化组蛋白结合[15],也已知与有丝分裂纺锤体结合并对DSB作出反应[16].都铎结构域FMRP已经通过特异性结合甲基化组蛋白参与DNA修复,甲基化组蛋白在复制应激期间标记人类细胞中的DNA损伤[6].此外,UHFR1蛋白(泛素样蛋白,包含PHD和RING finger结构域1),在人类中也被称为ICBP90,是一个都铎结构域,在组蛋白甲基化、DNA甲基化、DNA修复和细胞周期调节过程中起核心作用[9].UHFR1是E3连接酶家族成员,具有RING结构域,可招募DNA甲基转移酶,并调节G1到S过渡阶段重要基因的表达,包括RB [9].
在植物中,Armadillo BTB拟南芥蛋白1 (ABAP1)被描述为一种植物调节蛋白,参与控制基因表达和DNA复制[17].ABAP1与Pre-Replication Complex (pre-RC)的成员结合,也与转录因子结合以负调控关键pre-RC基因的转录[17].它通过将植物发育信号与DNA复制和转录控制相结合,参与一个控制细胞周期从G1期到S期进展的信号网络[17].DNA复制和转录是依赖于染色质可及性的动态过程。目前还不清楚组蛋白修饰在协调复制和转录中的作用,以及它们如何与发育结合。
在这里,我们报道了一种新的Agenet/Tudor/DUF724结构域蛋白的鉴定和鉴定,该结构域蛋白与ABAP1相互作用,命名为ABAP1相互作用蛋白1 (AIP1)。首先,对植物王国中Agenet/Tudor家族区域进行了一般的生物信息学和系统发育分析。这表明这个家族在动植物中有第三种结构。此外,对完整植物基因组的研究表明,Agenet/Tudor随着植物进化而扩展。拟南芥中鉴定出该家族成员30个,按系统发育可分为4个类群。不同家族成员的表达模式在生殖组织中有显著的发生率。拟南芥Agenet/Tudor结构域蛋白AIP1先前被报道为DUF724结构域蛋白DUF7 [6],并在本文中记为AIP1。除了与DNA复制和转录的负调控因子ABAP1相互作用外,我们还发现AIP1在体内与植物组蛋白修饰的“读本”LHP1和未修饰的组蛋白相互作用。AIP1在生殖组织中表达,其下调延迟花的发育时间。在AIP1水平降低的植物花蕾中,ABAP1和LHP1靶基因mRNA水平下调。这是首个具有Agenet/Tudor和DUF724结构域的植物蛋白,并进行了功能鉴定。这些数据可能表明,AIP1可能是花发育过程中DNA复制、转录和染色质重塑之间的纽带。
方法
在网上含有Agenet/Tudor结构域的蛋白质分析
TBLASTN使用以下数据库搜索Agenet/Tudor家族蛋白:18],国家生物技术信息中心(NCBI)数据库[19],拟南芥信息资源(TAIR)数据库[20.]和Congenie数据库[21].的部分长度(Agenet/Tudor域)序列At2g17950(FSSGTVVEVSSDEEGFQGCWFAAKVVEPVGEDKFLVEYRDLREKDGIEPLKEETDFLHIRPPPPR)被用作TBLASTN的查询序列。的e -所选序列的值均小于1e−5。在所有序列中,保守域的存在都是使用Pfam [22]、SMART [23]和NCBI数据库[19], e值小于1e−3。
使用MUSCLE 3.6 (http://www.ebi.ac.uk/Tools/msa/muscle/)使用默认参数。将Agenet/Tudor蛋白家族成员序列用MEGA (version 3.0;)比对,利用邻居连接法构建系统发育树[24].使用以下参数进行NJ分析:泊松校正法、间隙成对删除和bootstrap(1000个重复;随机种子)。对于Domain assiniture,我们使用WebLogo(基于web的序列logo生成应用程序;Weblogo.berkeley.edu) (25].参见附加文件13用于通过WebLogo在植物中构建Agenet/Tudor签名的序列。
的在网上利用iPSORT on line软件分析AIP1氨基酸序列中细胞定位的肽信号,根据[26].
蛋白质结构建模
利用I-TASSER服务器进行蛋白质三维结构预测,对Agenet/Tudor结构域进行结构建模和可视化[27].生成的三个模型使用PyMol包进行可视化和处理[28].使用PyMol对Agenet/Tudor结构域的结构进行比对,并使用Multalin服务器计算其主多序列比对[29].调整了最重要模型二级结构的对准图像,使用ESPript [30.].PDBeFOLD [31]被用来评估Agenet结构域的折叠,并识别PDB中的结构同源性。利用ProFunc预测蛋白质的可能功能[32].
植物材料及表达分析
拟南芥植株在标准温室条件下,23°C的长日照条件下(光照16小时,暗8小时),在琼脂板或土壤上生长。所有的分析在足底以拟南芥加入Columbia-0为背景。在附加文件中描述了使用qRT-PCR进行表达分析13.引物序列可在附加文件中找到12.
35S::RFP-AIP1和35S::GFP-ABAP1分析
中的瞬态表达式烟草benthamiana对于亚细胞定位,根据[33].简单地说,质粒被引入农(GV3101)。将隔夜培养的细菌离心,并将颗粒悬浮在10 mMMgCl2中,在600 nm处的光密度为0.5,并用200 mM乙酰丁香酮诱导。叶龄4-5周n benthamiana植物与两种待测结构的等摩尔细菌悬浮液共同浸润。在浸润后2天记录蛋白质共定位的共聚焦激光扫描图像(LSM-700, Carl Zeiss)。
酵母双杂交试验
酵母双杂交试验根据[17].简单地说,年代accharomyces酵母将PJ694菌株与1 μg聚乙二醇/LiAc法共转化,并将其镀在不含亮氨酸/色氨酸(-leu/-trp)的合成掉液培养基上(以测试转化效率);或亮氨酸、色氨酸和组氨酸(-leu/-trp/-his)(低严格条件);或亮氨酸、色氨酸、组氨酸和腺嘌呤(-leu/-trp/-his/-ade)(高严格条件),30°C孵育3天。
体外和半体内蛋白质相互作用试验
AIP1-GST, ABAP1-HIS, ARIA-HIS和LHP1-HIS在细胞中产生大肠杆菌菌株BL21(附加文件13).体外GST下拉分析根据[34].根据附加文件中描述的协议,采用标准技术进行植物蛋白提取和蛋白质凝胶印迹13.半体内GST下拉在附加文件中描述13.
串联亲和纯化(TAP)
在构成型花椰菜花叶病毒35S启动子的控制下,克隆AIP1 CDS,将其n端融合到TAP标记系统中,导入NGSrhino载体。然后对拟南芥细胞悬浮培养物进行转化,如[35].利用蛋白质G和链霉亲和素结合肽标签进行蛋白复合物的串联亲和纯化,然后进行蛋白沉淀和分离,根据[36].蛋白质水解和肽分离、4800蛋白质组学分析仪(Applied Biosystems)质谱采集以及基于拟南芥信息资源8.0基因组数据库的基于ms的蛋白质同源性鉴定,根据[37].基于大约40个野生型培养物和表达TAP标记模拟蛋白β -葡萄糖醛酸酶、红色荧光蛋白和绿色荧光蛋白的培养物的TAP实验,减去实验背景蛋白[38].
AIP1突变植物的分析
GABI_645B06的T-DNA插入线(https://www.gabi-kat.de/)经GABI T-DNA插入特异性引物PCR基因分型鉴定AIP1.有关AIP1突变体的分子和表型分析的详细信息,请参阅附加文件13.
结果
都铎家族成员随着植物的进化而扩大
大多数含有Agenet/Tudor结构域的蛋白质在植物中仍未得到很好的表征。为了更深入地了解这些蛋白质的进化和可能的生物学作用,作者在网上对植物王国的Agenet/Tudor结构域进行了分析。为了在植物中寻找属于Agenet/Tudor结构域家族的蛋白质,我们使用了该基因的Agenet/Tudor序列At1g09320对Phytozome、NCBI、TAIR和Congenie数据库中可用的基因组序列进行TBLASTN查询[18- - - - - -21].研究对象包括单细胞绿藻(4种)、无维管植物(苔藓植物- 1种)、无籽植物(石松植物- 1种)和有籽植物(裸子植物- 1种;针叶树- 1种;银杏属1种;被子植物22种。手动删除冗余序列。此外,在拟南芥中,每个含有Agenet/Tudor结构域的蛋白质的假定同源物都被TAIR(附加文件)中的TBLASTN识别出来7).
共研究了31个物种,从绿藻到被子植物,如附加文件所述1.分析显示,与高等植物相比,绿藻和苔藓等低等植物没有或更少的Agenet/Tudor基因。只发现了一位都铎家族的成员Coccomyxa,四名成员被发现Physcomitrella金属盘,在大多数高等植物中发现了10个以上的成员。这些数据表明,Agenet/Tudor家族成员在植物基因组中的数量随着植物的进化而增加。
植物王国中含有Agenet/Tudor结构域的蛋白质的系统发育分析显示了高等植物的关键分支
为了研究含有Agenet/Tudor结构域的蛋白质的进化变化,利用从绿藻到被子植物的30个物种的386个结构域的全长序列进行了系统发育分析。内部分支的一些自举值很低,因为包含了大量的序列[39].在去除所有拟南芥蛋白质后,可以构建一个相对支持良好的系统发育树,这可能是由于这些非常不同的序列在解析分析时引起的大量噪声(图2)。1).Agenet/Tudor家族的成员被分为三个主要的分支,除了Agenet/Tudor之外,它们被各自的保守领域分开。这3个演化支分别为:a)衍生演化支,包含26个物种的279条序列;B)中间进化支,包含25种111条序列;C)古(基)演化支,包含20个物种的29个序列。
植物王国中Agenet/Tudor蛋白家族的系统发育分析。一个系统发育分析以邻居连接(NJ)树的简化版本表示,包含31个物种的416个蛋白质序列,从绿藻到被子植物。该树分为3个分支:派生分支,包含26个种的279条序列,包含Agenet/Tudor结构域和BAH、DUF724、F-box等结构域;b)中间分支,包含来自25个物种的111条序列,在n项或中心存在Agenet/Tudor结构域的重复,与ENT结构域合并或不合并;c)古(基)支,包含20种29条序列,c项中包含一个Agenet/Tudor结构域。bAgenet/Tudor家族成员的分布示意图,共发生结构域的多样性及其系统发育关系。从绿藻到被子植物共24科33种442条序列。正方形表示蛋白质中存在的结构域,颜色根据图例指定结构域。一些罕见的域没有被表示出来。物种在附加文件中列出7
为了进一步研究三个演化支的Agenet/Tudor成员之间的进化关系,还对所有序列进行了Pfam [22]及SMART [23)与e-value >的截断值e-5用于域识别。每个序列都标注了Agenet/Tudor结构域的数量及其位置(n端、中心端或c端),以及可能与Agenet/Tudor结构域同时出现的其他结构域(图2)。1 b和附加文件7).分析表明,在同一蛋白中,第一个基础Agenet/Tudor结构域不与其他结构域共存。然而,在苔藓植物和石松植物中,Agenet/Tudor与BAH结构域结合。在裸子植物中,它与耳鼻喉科共存。最后,在被子植物中,该家族富含Agenet/Tudor重复,并且在相同的蛋白质结构中存在其他类别的结构域。
植物代理/都铎结构域在结构上与动物都铎结构域非常相似
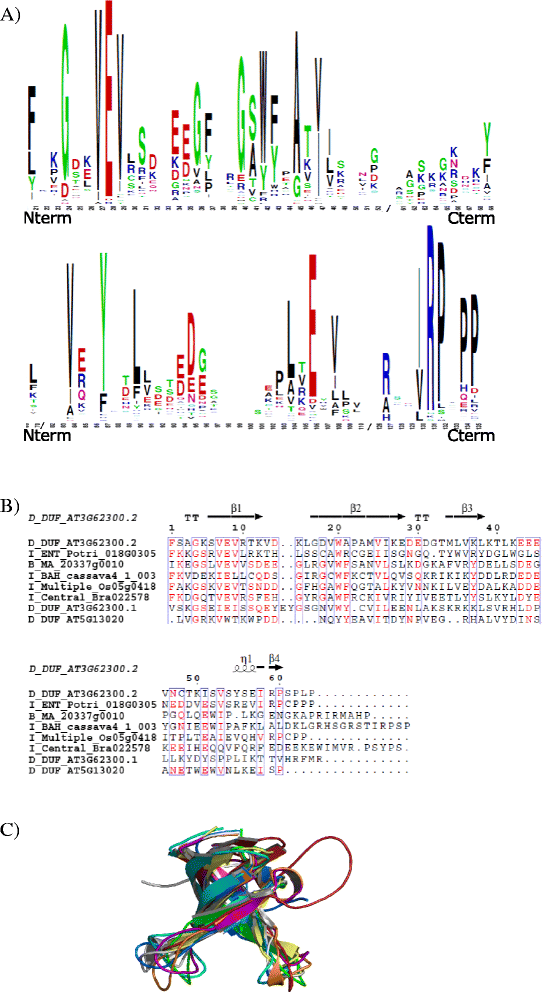
Agenet/Tudor域以前被归类为皇家家族域的一员,Agenet/Tudor被描述为都铎式植物域[3.].此前已有报道称,拟南芥蛋白质的Agenet/Tudor结构域平均含有60个氨基酸,且在少数保守位置上,与王室结构域的序列排列具有较远的关系[3.].为了构建植物王国中Agenet/Tudor结构域的通用签名,对植物中发现的54个最显著的Agenet/Tudor序列进行了多序列比对,以确定WebLogo(基于web的序列标识生成应用程序;Weblogo.berkeley.edu)(图2).来自植物的Agenet/Tudor结构域签名在51 ~ 101 aa的结构域序列中有少量保守的氨基酸(至少16 aa),与仅基于拟南芥构建的Logo非常相似”s来自动物的Agenet/Tudor结构域和FRMPs。Agenet/Tudor结构域的特征表明,该结构域的主要序列在不同蛋白质之间变化很大。为了研究来自植物蛋白的Agenet/Tudor结构域的结构同源性,首先利用Multalin对来自不同植物的不同Agenet/Tudor蛋白进行比对,建立二级结构的特征。29]和ESPript [30.].二级结构的特征是严格的β-弯,4个β-片和一个310螺旋(无花果。2 b-请参见附加文件中的参数数据8),(类似文献3中二级结构的信息)。接下来,同一Agenet/Tudor序列之间的结构同源性通过I-TASSER评估[27].所有的Agenet/Tudor模型都显示了显著的c评分和tm评分参数(见附加文件)9),在本研究提出的植物Agenet/Tudor模型中,Tudor -like Beta-barrel折叠的特征结构被认为是保守的(图。2摄氏度).各个结构在附加文件中表示2.综上所述,本研究中预测的二级结构模型表明,植物Agenet/Tudor结构域之间可能在总体上非常相似,这表明它们可能属于一个一致的蛋白质结构域家族,尽管它们在氨基酸序列上的相似性较低。
拟南芥蛋白Agenet/Tudor结构域的特征及预测结构。一个拟南芥Agenet/Tudor序列的校准显示WebLogo分析的典型保守残基。高度保守的残基在序列中用较大的字母表示。虽然非常多样化,但一些关键位置含有保守的氨基酸,并可能保持所观察到的保守的二级结构。b植物蛋白Agenet/Tudor结构域序列多序列比对:采用Multalin比对,比对结果提交至ESPript服务器,绘制保守结构域在主序列上的二级结构信息。在二级结构上显示,310-螺旋表示为小波浪(Ƞ), β-链表示为箭头,严格的β-转弯(TT)。在一次序列比对中,红色表示同一列同组氨基酸残基的相似性,蓝色框表示组间相似性。用于结构分析和计算机建模的序列被选择来代表所有的植物分支:裸子植物挪威云杉MA_20337g0010;被子植物单子叶植物栽培稻Os05g04180;被子植物Eudicot杨树trichocarpaPotri_018G030500_5,芸苔属植物拉伯Bra022578,木薯耐cassava4_1_003152,答:芥AT3G62300 AT5G13020。使用AIP1的两个Agenet/Tudor重复序列(AT3G62300.1和AT3G62300.2)。c在I-TASSER服务器中生成的重叠Agenet/Tudor模型。结构颜色为白色(B_MA_20337g0010),紫色(I_ENT_Potri_018G030500_5),耐火砖(I_Central_Bra022578),橙色(I_Multiple_Os05g04180),蓝色(I_BAH_cassava4_1_003152),青色(D_DUF_AT3G62300.1),黄色(D_DUF_AT5G13020),绿色(D_DUF_AT3G62300.2)
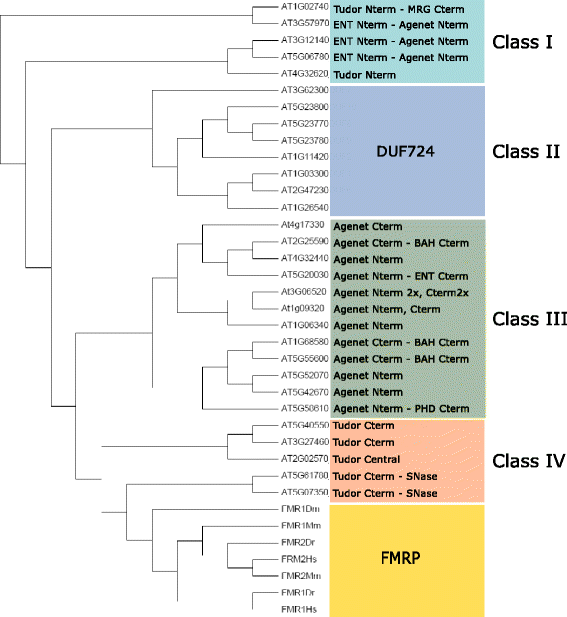
拟南芥中的Agenet/Tudor家族根据结构域的不同分为四个不同的类
为了更好地了解来自拟南芥的Agenet/Tudor含蛋白的系统发育,利用来自家族成员的30条序列在Mega 6.0程序中构建树[24].人类、小鼠、苍蝇和斑马鱼的FMRPs序列来自NCBI [19]也被使用。我们构建了一个专注于功能特征的拟aphytic树,并允许将蛋白质根据其结构域的组织进行分类的不同分支可视化。Agenet/Tudor类I有N个终端Agenet/Tudor域,一些成员还包含ENT域。II类蛋白与DUF724结构域共同出现在c端。III类具有更多样化的成员,其Agenet/Tudor结构域位于N和/或C端位置,多个Agenet/Tudor重复或与BAH或PHD共存。IV类蛋白与动物fmrp最为相似(图2)。3.).
中国Agenet/Tudor家族的系统发育分类拟南芥。利用拟南芥中发现的成员和拟南芥的FMR1和FMR2蛋白,用MEGA6构建系统发育树(NJ)D.黑腹动物,M. musculus, D. rerio而且智人作为根(附加文件7)
为了研究拟南芥中不同类型的Agenet/Tudor基因可能参与的发育过程,研究了它们的表达模式在网上透过genevvestiator资料库[40].一般来说,Agenet/Tudor家族成员在种子和胚胎等生殖组织中高度表达(图2)。4).不同的Agenet/Tudor家族在其成员的表达谱上表现出一些特殊性(图2)。4).I类基因在种子和胚组织中高度表达。II类同样存在于种子和胚中,但在茎尖和花雌组织(心皮和胚珠)中也有高表达。III类成员的表达分布在不同的植物器官和组织中,部分基因在花粉和种子中表达较多。在IV类的五个成员中,有两个基因没有在微阵列数据实验中表示,使模式分析无效。分析了发育过程中Agenet/Tudor结构域蛋白的表达情况在网上通过genevvestiator数据库(附加文件3.).I类成员在发育过程中表达量适中,几乎没有变化,在种子的晚期成熟和叶片的衰老过程中观察到mRNA水平的升高。II类成员也表现出中等表达水平,在抽苔期和胚胎成熟期达到峰值。III类成员的表达谱也非常多样化。
拟南芥Agenet/Tudor家族各类成员的表达谱。表达模式显示在不同的植物组织和器官中,作为表达值的平均值的热图表示,这些平均值发表在genevarcheator (https://genevestigator.com) [40].IV类基因AT5G07350和AT3G27460不在分析范围内,因为可用的微阵列数据中没有探针
有趣的是,拟南芥Agenet/Tudor基因在生殖组织中高度表达,进化分析显示,开花植物中Agenet/Tudor家族的成员和结构域多样性显著增加(图2)。1 b).综上所述,这些数据表明Agenet/Tudor结构域蛋白可能在花发育和胚形成过程中发挥作用。
鉴定AIP1为Agenet/Tudor/DUF724结构域蛋白,可与ABAP1相互作用
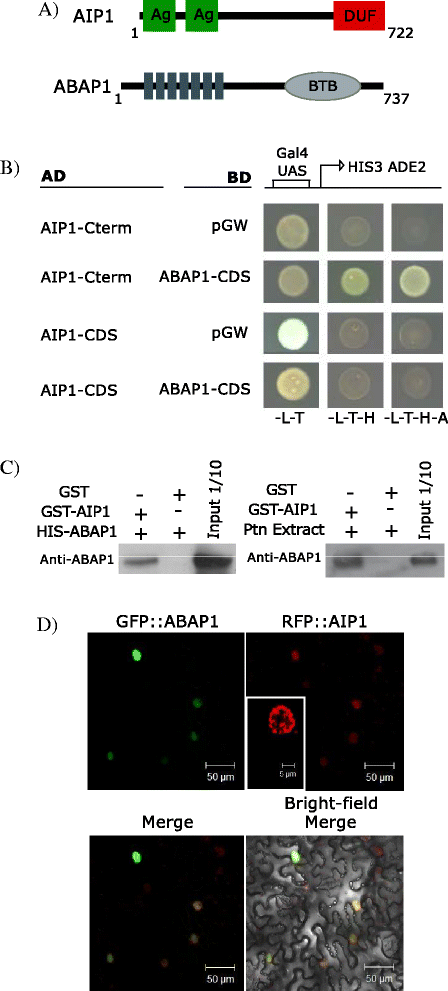
为了寻找能够参与ABAP1控制DNA复制和转录的蛋白,以ABAP1为诱饵,利用拟南芥cDNA文库进行酵母双杂交筛选[17].鉴定了转录因子家族成员,如TCP24,它与ABAP1一起调节叶片中的细胞分裂[17].在鉴定的abap1相互作用蛋白(AIPs)中,有一种未知蛋白AIP1 (At3G62300),预测其含有722个氨基酸,约809 kDa。它在n端有两个重复的Agenet/Tudor结构域(氨基酸13-84和161-224),在c端有一个DUF724结构域(氨基酸540-722)(图。5).Agenet/Tudors结构域有63和71个氨基酸长,DUF724结构域有182个氨基酸长。前人对拟南芥DUF724基因家族的研究,基于Agenet/Tudor与FMRP的动物Tudor结构域的相似性,将其描述为一个rna结合结构域,并将AIP1命名为DUF7 [4].AIP1与其他Agenet/Tudor/DUF724蛋白一起属于拟南芥Agenet/Tudor家族的II类蛋白(图。3.).
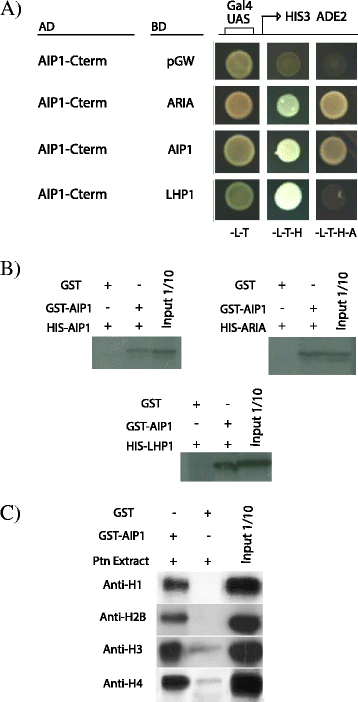
AIP1蛋白相互作用和亚细胞定位的特征。一个AIP1和ABAP1蛋白的结构示意图。AIP1在n端有两个重复的Agenet/Tudor (Ag)结构域,在c端有一个DUF724;ABAP1 n端含有8个β -catenin-type Armadillo (ARM)基因,c端含有1个BTB/ POZ (BTB / POZ)结构域。b用AIP1的c端区域(aa 540-723)或与GAL4 DAD融合的AIP1完整CDS(分别为AIP1- c -term AD和AIP1-CDS AD)与与GAL4 DBD融合的全长ABAP1进行两次杂交试验。阴性对照为GAL4 DBD空载体。构造的详细信息可以在附加文件中找到13.酵母转化在-L-T(缺乏亮氨酸和色氨酸的SD培养基)中选择,蛋白质相互作用在-L-T- h或-L-T- h - a(缺乏亮氨酸、色氨酸和组氨酸或组氨酸和腺嘌呤的SD培养基中选择。c左图:细菌表达重组GST- aip1和HIS-ABAP1的GST下拉。右图:拟南芥10日生植株细菌表达重组GST-AIP1和蛋白裂解物的半体内下拉试验。用抗ABAP1抗体免疫印迹法检测ABAP1相互作用蛋白。dGFP::ABAP1和RFP::AIP1在植物后表皮的亚细胞定位n benthamiana共聚焦显微镜观察14天大的叶子。RFP::AIP1插图显示细胞核内的斑点图案
酵母双杂交实验中AIP1-ABAP1之间的相互作用被定位在AIP1的c端区域(氨基酸532-723)和ABAP1的N端区域(氨基酸1-350),后者包含DUF724结构域(ARM重复序列)(图2)。5 b和附加文件4).令人惊讶的是,在酵母双杂交实验中,全长AIP1没有与ABAP1相互作用(图2)。5 b).尽管如此,在HIS::ABAP1和GST::AIP1的GST下拉实验中,ABAP1和全长AIP1之间的联系得到了证实。5度),并且用GST::AIP1和10日龄拟南芥的蛋白提取物进行半体内拉下实验进一步证实了这一点(图。5度).
AIP1没有任何明显的dna结合特征,iPSORT搜索也没有信号肽预测。RFP::AIP1和GFP::ABAP1共转染实验烟草benthamiana叶背面表皮证实了AIP1的核定位[4],并与ABAP1共定位(图;5 d).共聚焦显微镜图像显示AIP1仅位于细胞核中,并富集于核畴(图2)。5 d).值得注意的是,也有报道称ABAP1仅位于细胞核中,以散斑的形式均匀分布或富集在核结构域[17].
综上所述,这些数据表明AIP1可以与ABAP1共同参与调控复合物。需要解决的一个重要问题是,在ABAP1调控DNA复制和/或基因表达过程中,AIP1是否在染色质重塑中发挥作用。
AIP1 Agenet/Tudor结构域在结构上与作为组蛋白修饰阅读器的Tudor结构域最为相似
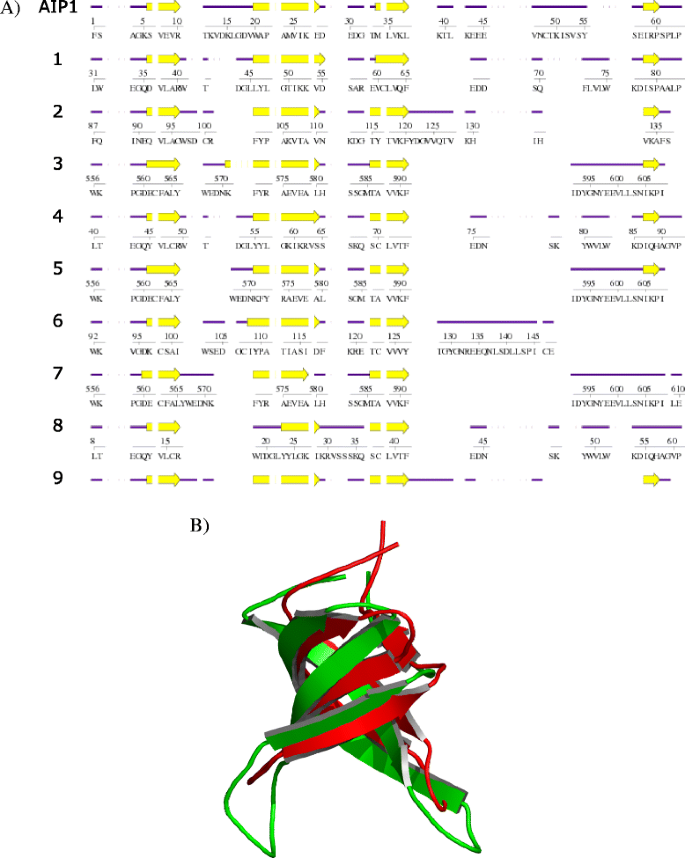
为了深入了解AIP1的Agenet/Tudor结构域的功能,我们首先通过执行计算机结构建模解决了AIP1结构域与动物Tudor结构域的接近程度。对于ProFunc分析[32],使用AIP1在N端Agenet/Tudor域的第二次重复作为查询。分析表明,蛋白质数据库(PDB)中得分较高的所有结构都存在于动物都铎结构域(图2)。6).由于所有的Agenet/Tudor模型域都可能具有保守的Tudor-like Beta-barrel折叠,因此Agenet/Tudor和Tudor域可能具有类似的折叠和结构。c - α原子与比较结构的统计相关z分数之间的均方根偏差(RMSD)(见附加文件)8)确保发现的命中的重要性。最佳结果(红色漫画表示)与AIP1 N端Agenet/Tudor结构域(绿色漫画表示)叠加,可以观察到直接的相似性(图2)。6 b).最相似的Agenet/Tudor结构域(如图1所示)。6)存在于PHD手指蛋白1 (PHF1)中polycomb集团(PcG)基因,该基因是一种组蛋白修饰阅读器,已知在人类中特异性结合组蛋白H3K36me3并招募Polycomb repression Complex 2 (PRC2) [41].pcg已知沉默表达主要认为是染色质结构的调节,部分是通过组蛋白的翻译后修饰[42].
不同Tudor结构域的AIP1预测Agenet/Tudor结构域的二级结构匹配。一个ProFunc使用AIP1的Agenet/Tudor序列作为查询执行二级结构的比对。所示蛋白质为:1;PHF1与H3K36me3配合物的x射线衍射研究2.人体PHF20都铎结构域2的x射线衍射研究3.人TDRD3都铎结构域(都铎结构域蛋白3)的x射线衍射分析4.人PHF19连接H3K36me的核磁共振溶液谱;5. Tudor domain of human TDRD3 by X-Ray Diffraction; 6. Human Tudor domain of SMN1 in complex with aa organic molecule by X-Ray Diffraction; 7. Human TDRD3 complex with asymmetric dimethylarginine mark in histone by Solution NMR; 8. Solution NMR structure of the human Tudor domain of PHF19, isoform b; 9. The second Tudor domain of human PHF20 by X-Ray Diffraction.bAIP1 (Green)的Agenet/Tudor域的建模结构叠加到PHF1 (Hit 1)的Tudor域。PDB id: 4HCZ)(红色)
AIP1在生殖组织中高度表达
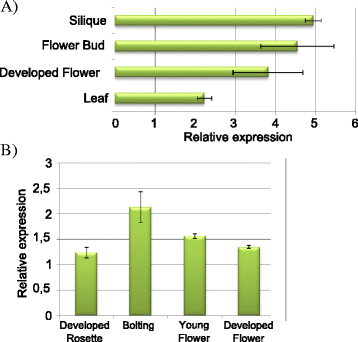
为了确定AIP1在植物发育过程中可能的功能,对其基因表达谱进行了分析在网上在开放获取的微阵列数据库和qRT-PCR分析(图。7).高AIP1在各种生殖组织,如心皮,以及球状和心形胚胎的合点处种皮等种子组织中均有表达[43]在胚柄中,其表达量约为其他组织的4、5倍[44(图。4).qRT-PCR证实高表达AIP1与发达的花和叶相比,在角果和花蕾中较少(图2)。7一个).茎尖分生组织也较高AIP1mRNA水平[45(图。4).的高峰AIP1在螺栓连接过程中观察到表达(图;7 b),标志着从营养阶段过渡到生殖阶段的发育时间,以及在种子发育期间观察到的第二个表达峰值。总之,数据显示了一个峰值的表达AIP1其主要表达发生在花的早期发育阶段,特别是在雌性器官中,这表明AIP1可能在植物的生殖阶段发挥作用。
AIP1与拟南芥中ABAP1同源物ARIA和染色质重塑蛋白LHP1相互作用
为了进一步了解AIP1的功能,我们使用酵母双杂交试验寻找了AIP1参与的其他相互作用蛋白和复合物。由于在酵母双杂交实验中全长AIP1不能建立蛋白-蛋白相互作用,因此使用含有DUF724结构域的AIP1 C端区域检测AIP1与其他与ABAP1网络、前rc成员和染色质重塑蛋白相关的蛋白相互作用。酵母- 2杂交测试的对相互作用的完整列表列在附加文件中10.与ABAP1不同,AIP1没有表现出与pre-RC部分蛋白质的正向相互作用。有趣的是,在酵母-两种杂交实验中,AIP1 C-term能够与AIP1全长蛋白形成同型二聚体(图2)。8),在GST拉下实验中观察到全长AIP1蛋白与GST::AIP1和HIS::AIP1的关联(图。8 b).AIP1也与ARIA - ABAP1同源物相互作用(图。8,附加文件4),通过酵母-双杂交鉴定AIP1 DUF724结构域与LHP1全长蛋白之间存在弱相互作用(图2)。8).这些相互作用通过融合CDS序列的GST下拉实验进一步证实:GST::AIP1和HIS::ARIA或HIS::LHP1(图2)。7 b).与LHP1的关联支持了AIP1蛋白在染色质重塑中的可能作用,因为LHP1是一种基因表达的调节因子,它通过依赖其组蛋白的甲基化状态来控制染色质的包装[46].
AIP1蛋白相互作用。一个用AIP1 c端(aa 540-723)与GAL4 DAD (AIP1- c -term AD;)融合的酵母双杂交检测ARIA, AIP1和LHP1全长CDSs与GAL4 DBD融合。阴性对照为GAL4 DBD空载体。构造的详细信息可以在附加文件中找到13.酵母转化在-L-T(缺乏亮氨酸和色氨酸的SD培养基)中选择,蛋白质相互作用在-L-T- h或-L-T- h - a(缺乏亮氨酸、色氨酸和组氨酸或组氨酸和腺嘌呤的SD培养基中选择。b细菌表达的重组GST- aip1与HIS-AIP1,或HIS-ARIA,或HIS-LHP1的GST下拉。用免疫印迹法检测HIS-tag相互作用蛋白。c拟南芥10日生植株细菌表达重组GST-AIP1和蛋白裂解物的半体内下拉试验用Anti- h1、Anti- H2B、Anti- h3、Anti- h4抗体免疫印迹法检测组蛋白相互作用蛋白
AIP1与未修饰的组蛋白相互作用
接下来,使用AIP1作为诱饵进行串联亲和纯化(TAP)试验,以鉴定在体内形成的蛋白质复合物(见方法).然而,由于在细胞培养中难以很好地表达融合亲和标记的全长AIP1, TAP受到阻碍。然而,TAP纯化分析的输出是组蛋白超家族保守序列的一小部分(附加文件)11).由于AIP1是与染色质重塑因子LHP1和DNA复制和转录调控因子ABAP1相互作用的皇家结构域家族的成员,因此可以合理地预期AIP1可以与组蛋白结合,无论它们是通过甲基化或乙酰化修饰的,还是没有。为了进一步研究AIP1和组蛋白之间的可能联系,我们使用全长GST::AIP1和拟南芥10日龄植株的蛋白提取物进行了半体内下拉试验,并用针对特定组蛋白的抗体检测AIP1相互作用蛋白(图2)。8 c).结果表明,AIP1可以与未修饰的组蛋白H1、H2B、H3和H4结合。然而,它不能与两种乙酰化组蛋白H3K9ac和H3K14a结合(附加文件)5),它们在玉米中被Agenet/Tudor/ENT结构域蛋白RIF1识别[13],表明这两个Agenet/Tudor成员可能在植物发育中发挥了不同的作用。
下调AIP1会延缓花的成熟
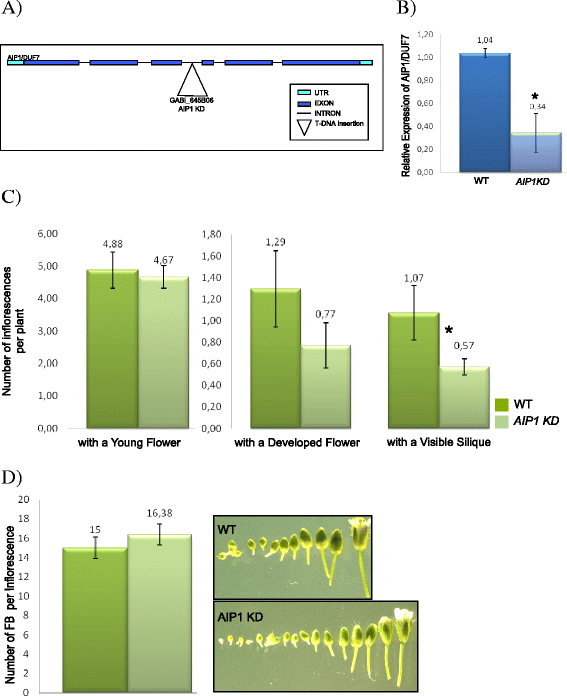
为了了解AIP1在拟南芥发育过程中的功能,研究了AIP1表达水平降低或沉默的植物AIP1在T-DNA插入突变体的集合中进行搜索。没有发现SALK T-DNA插入AIP1.一个T-DNA插入第三个内含子的纯合子GABI系突变体(GABI_465B06)被表征(图。9).AIP1GABI_465B06纯合子植株(这里记为AIP1)的表达水平下降了约3倍KD)(图。9 b).
AIP1的分子和表型分析KD行。一个AIP1的示意图KD(GABI_645B06)表示T-DNA插入第三个内含子。b相对mRNA水平AIP1在21日龄WT和AIP1KD采用qRT-PCR方法检测纯合子植株。数据用AtUBI14作为内参基因。所示数据为独立扩增反应所得的平均值(n= 3)和生物重复(n= 2)。每次生物复制都使用从至少六株植物池中收集的材料进行。柱状图表示生物重复的平均值±标准误差。进行统计分析t以及(p值< 0.05)。星号(*)表示样本之间有显著变化。c30日龄生殖结构数量的比较分析AIP1KD和WT植物。该图显示了每株含有幼花的花序数量(左),每株含有已发育花的花序数量(中),每株含有可见角锥的花序数量(右)。数据已在10个工厂进行了量化。所示数值为两个独立实验的平均值。柱状图表示生物重复的平均值±标准误差。进行统计分析t以及(p值< 0.05)。星号(*)表示对照(野生型)和样本之间有显著变化。d(左图)30日龄花发育时间的比较分析AIP1KD和WT植物。所示数值为两个独立实验的平均值。柱状图表示生物重复的平均值±标准误差。进行统计分析t以及(p-value为0.06)。(右图)WT和AIP1的代表性花序解剖照片KD植物
的表型AIP1KD在整个发育过程中对植株进行分析,并与野生型对照系进行比较。在营养发育过程中,AIP1KD低水平的植物AIP1发育正常,叶面积、细胞数、倍性及根生长均无显著差异(附加文件)6).在生殖期,两种生殖器官的发育时间略有差异AIP1KD在30天的植株中,每株成熟花和角果的花序数量减少(图2)。9 c).然而,如果植物保持浇水,这些差异在植物衰老过程中消失(数据未显示)。试图测量30天大的花的发育时间AIP1KD突变体,在第6-12阶段,每个花序含有一朵开放花的可见花芽数量,根据[47),在双筒镜下计数(图;9 d).虽然花的成熟有延迟的趋势AIP1KD突变体,没有统计学上的不同。
虽然植物完全沉默了AIP1无法获得的,植物的表型水平降低了AIP1提示该蛋白在开花和繁殖中有一定的作用,并与高表达水平的AIP1在植物发育的这个阶段。此外,AIP1可能的同源物仅在被子植物中发现(在附加文件中以黄色突出显示)7).
下调AIP1会增加ABAP1和LHP1靶基因的表达水平
为了研究AIP1是否能与ABAP1和LHP1共同作用于花的发育过程,我们分析了AIP1水平降低的植物花芽中ABAP1和LHP1靶基因的mRNA水平KD)与WT对照植物进行qPCR分析。
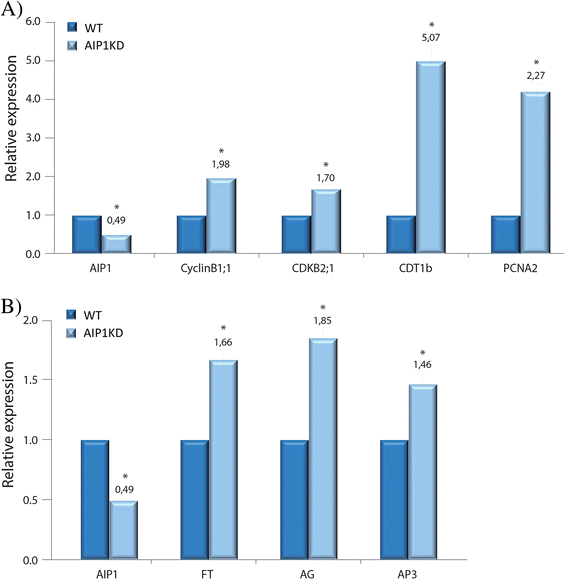
为了验证AIP1在DNA复制中的可能作用,研究了Cdt1b的表达,Cdt1b是pre-RC的一个成分,是ABAP1转录抑制的靶标(Masuda等人,[17)进行了调查。除了受到转录调控外,Cdt1b也是一种DNA复制标记增殖细胞核抗原2(PCNA2),另一个S期标记基因,负责在DNA受损时恢复复制叉进展[48].分析G2-M转化和细胞分裂标志物CyclinB1;1和CDKB2;1的表达[49].如图所示。10, AIP1的表达水平降低了约50%KD花蕾与对照植物比较。另一方面,AIP1中Cdt1b和PCNA2 mRNA水平显著升高KD这表明AIP1可能与ABAP1一起负调控DNA复制。在AIP1中也观察到CyclinB1;1和CDKB2;1 mRNA水平的升高KD花蕾(Fig。10),表明在AIP1中,DNA复制刺激可能导致细胞增殖率增加和/或细胞分化延迟KD突变体。
为了验证AIP1和LHP1在花发育中的作用,研究了被LHP1表观遗传抑制的花发育靶基因,如flower Locus T (FT)、Agamous (AG)和APETALLA 3 (AP3)的表达[50],进行了分析。如图所示。10 bAIP1中FT、AG、AP3 mRNA水平升高KD花蕾,比起控制。综上所述,这些数据表明AIP1与ABAP1和LHP1相互作用,并可能参与了花发育过程中抑制其部分靶基因的表达。
讨论
染色质修饰和可及性的动态可能在调节DNA复制和转录中发挥关键作用,这是调节植物生长和发育的关键细胞过程。然而,这些过程如何与发育信号协调和整合还没有完全阐明。在这项工作中,我们研究了植物中染色质重塑蛋白家族,包含Agenet/Tudor结构域,被称为组蛋白修饰“读者”。这类蛋白质在植物界的特征仍然很模糊。我们发现并描述了一个新的成员,命名为AIP1,之前称为DUF7 [6],可以进一步揭示DNA复制、基因转录和染色质重塑之间的联系机制。
具有Agenet/Tudor结构域的植物蛋白可能在植物繁殖阶段的发育过程中发挥作用
在这项研究中,描述了416个植物中含有Agenet/Tudor结构域的蛋白质,包括至少380个以前未定义的蛋白质。我们的分析显示,在四分之一所分析的绿藻物种中,只有一种蛋白质含有Agenet/Tudor结构域,这表明这个家族是从那时开始出现的。对来自30种植物的Agenet/Tudor家族386个成员的系统发育分析支持Agenet/Tudor家族具有单系起源。以前对DUF724蛋白家族的研究,包括一些Agenet/Tudor结构域蛋白答:芥,水稻,杨树和葡萄,也支持了这个家族的共同祖先来源[4].
Agenet/Tudor域以前被归类为皇家家族域的一员,Agenet/Tudor被描述为都铎式植物域[3.].多年来,Tudor被描述为RNA结合域[51,52],在一定程度上参与了与修饰的组蛋白尾的相互作用,以及DNA修复和细胞周期,但对其功能尚不清楚[53- - - - - -55].它来自于对植物Agenet/Tudor结构域的观察[4,13,14,56,57]最近才有可能完全理解人类都铎王朝的结构和功能[5,7- - - - - -10].据阐明,都铎结构域并不负责与核酸的相互作用,而是存在于一些都铎蛋白中的一个称为KH的相邻结构域[5].尽管Agenet/Tudor结构域被描述为RNA结合结构域[4],这种活性还没有实验证明。此外,目前尚未在植物Agenet/Tudor蛋白中发现KH结构域;然而,这一结构域的预测依赖于更复杂的蛋白质结构工具的使用,因此可能被忽视。一个例外是AtCoilin蛋白,它有三个与RNA结合的位点,但没有一个位于Agenet/Tudor结构域[14].KH结构域的预测依赖于更复杂的蛋白质结构工具的使用,因此我们有理由期望其他植物Agenet/Tudor蛋白也含有KH结构域,并且能够结合组蛋白和rna。
我们发现的证据表明,含有Agenet/Tudor结构域的植物蛋白单独或与BAH结构域共存是进化尺度上最古老的成员。绿藻只含有一个Agenet/Tudor结构域的单一蛋白质,而在苔藓植物和番茄植物中,Agenet/Tudor结构域与BAH结构域同时出现。在进化的后期,在裸子植物中,ENT结构域可以与Agenet/Tudor结构域共存。最后,被子植物中出现了Agenet/Tudor蛋白形态和序列多样性的“繁荣”,这可能与Agenet/Tudor蛋白在植物繁殖和花发育过程中的功能多样化有关。拟南芥中Agenet/Tudor家族的表达主要在生殖组织和胚胎中表达,证实了该家族成员在植物生殖阶段的作用。
一些动物Agenet/Tudor结构域蛋白被认为在配子生过程中起作用。果蝇的Tudor-SN (TSN)蛋白参与卵发生[51]和11个都铎结构域中的5个足以形成生殖细胞[58].尽管TSN的作用机制尚不清楚,但它是调控RNA沉默的RISC复合体的一部分[52].FMRP的Tudor结构域可以通过H3K79me与染色质结合,通过调节组蛋白yH2A变体的沉积来响应DSB。X,该基因突变导致配子体发生缺陷[6].研究表明,在拟南芥中,以AtEMLs命名的ENT/Agenet/Tudor蛋白以及EDM2可能通过染色质重塑过程将种族特异性病原体识别与一般防御机制联系起来[12].此外,EDM2通过抑制花的转变而积极地影响花的转变方法表达式[12),而EML1而且EML2突变体具有和早花表型[12].另一个描述得很好的调节器方法为LHP1,是拟南芥顶端分生组织中染色质调控花期的一个染色质结构域。LHP1识别并结合H3K27Me3,表观遗传调控方法春化期前后[59],以及其他MADS盒基因[60].LHP1的同源物仅在被子植物中发现,这表明LHP1在开花植物物种中发挥的作用可能是广泛存在的[46].
我们还发现AIP1的同源物只存在于被子植物支系中,这支持了其在花期和/或发育中发挥作用的观点。此外,AIP1与LHP1相互作用,两者都在茎尖分生组织和生殖组织中表达,这表明它们可能在这些器官中共同发挥作用。此外,我们的研究表明,基因表达降低的突变植物AIP1LHP1调控的花发育基因表达水平较高,且与野生型相比,这些植物的花发育时间也略有延迟,形成的花序至少多一个花蕾。表型是微妙的,发生在一个短暂的时间窗口,可能是因为大约40%AIP1mRNA水平在这些植物中仍然表达和/或在调控过程中存在冗余。基因突变CORYMBOSA2(CRM2)编码miRNAs和siRNAs的甲基转移酶,其表型对花诱导的时间影响不大,但在花的发育中表现出明显的延迟[61].结果,crm2突变体在花序中花蕾数量增加[61],与中观察到的表型相似AIP1KD突变体。尽管其作用机制尚不清楚,但似乎有理由假设基因表达的表观遗传调控正在影响这两个突变体的花发育过程。
AIP1是一种Agenet/Tudor蛋白,可能与细胞周期和染色质重塑过程有关
含有Agenet/Tudor结构域的蛋白质在染色质重塑和细胞周期调节中的作用仍不清楚。在植物中,该蛋白家族的成员尚未被报道在细胞周期中直接参与染色质动力学。这项工作更详细地描述了Agenet/Tudor蛋白AIP1,它与组蛋白和ABAP1结合,ABAP1是DNA转录和许可DNA在拟南芥中复制的调节因子[17].全长的GST::AIP1在半体内的GST下拉实验中能够同时拉下ABAP1和组蛋白,这表明AIP1、ABAP1和组蛋白可能存在于同一个复合体中。qRT-PCR数据显示,ABAP1抑制的preRC基因Cdt1在AIP1中表达增加KD;以及其他细胞分裂标志物的表达。这些结果支持AIP1可能与ABAP1共同调控拟南芥细胞周期和/或基因表达,控制G1到S阶段的细胞事件。一个重要的问题是解开Agenet/Tudor整合这些细胞过程的机制。可能与组蛋白和染色质重塑蛋白相互作用有关。
动物中的Agenet/Tudor蛋白被描述为各种组蛋白修饰的读者。都铎结构域FMRP通过特异性结合H3K79me参与DNA修复[6].53BP1可通过与K810位点甲基化的RB蛋白相互作用,维持其低甲基化状态来控制S期持续时间[7],并能与DSB标记H4K20me2结合[7].来自人类的串联都铎结构域蛋白Spindlin1识别H3K4甲基化[15],可与有丝分裂纺锤体结合并对DSB反应[16].
在植物中,Agenet/Tudor结构域的一级结构变化很大。如WebLogo签名所示,它包含大约50到100个碱基长度,初级结构中很少(至少16个)保守氨基酸。然而,结构建模表明,植物的Agenet/Tudor结构域之间可能是相似的,这表明它们属于一个一致的蛋白质结构域家族。此外,本研究模拟的所有植物Agenet/Tudor结构域和动物Tudor结构域都对保守的Tudor-like Beta-barrel折叠给出了良好的结果,与之前对动物Tudor折叠的描述相似[28],这表明这些结构域可能是单独进化的,但收敛到类似的结构及其相关折叠。尽管植物Agenet/Tudor结构域蛋白与染色质重塑过程有关[6- - - - - -8],与组蛋白直接结合尚未见报道。在这项工作中,观察到AIP1与未修饰的组蛋白H1, H2B, H3和H4有很强的相互作用。然而,AIP1与半体内拉下试验中测试的两种乙酰化组蛋白H3K9ac和H3K14ac(附加文件)无关5),它们可能被来自玉米的Agenet/Tudor/ENT RIF1蛋白识别[13].可能,AIP1可以识别不同的组蛋白修饰,在植物发育中发挥不同的作用。此外,我们发现AIP1还与一种识别并结合H3K27Me3的色域蛋白LHP1相互作用[60].此外,AIP1 Agenet/Tudor结构域的预测结构与PHF1中存在的结构最相似,已知可特异性结合组蛋白H3K36me3 [41].虽然在这项工作中,我们发现AIP1与未修饰的组蛋白相互作用,但它可以识别不同于测试的组蛋白修饰,需要进一步的蛋白质结构和相互作用工作来解决AIP1在染色质重塑中的作用机制。
最近,有报道称拟南芥SAWADEE Homeodomain Homolog 1 (SHH1)含有一个与Agenet/Tudor结构非常相似的结构域,即SAWADEE结构域[57].它优先结合二甲基化或三甲基化的H3K9,但也可以结合未修饰的H3K4 [57].有人提出,即使存在沉默标记(H3K9me2/3), SHH1 Agenet/Tudor结构域也可能允许由H3K4信号的转录[57].
都铎蛋白UHFR1具有变构调控,在基态时,蛋白的c端多基区被折叠回都铎结构域重复位点,而PHD结构域与未修饰的H3结合。在活性状态下,一个辅因子(磷脂酰肌醇磷酸- PI5P)连接到多基区,稳定了都铎结构域的取向,使其能够与修饰的组蛋白(H3K9me3)结合[8].AIP1只能结合未修饰的组蛋白,或者很有可能,蛋白质的内部折叠和/或与外部分子的结合可以调节不同的活性状态,其中AIP1可以选择性地结合组蛋白。在酵母双杂交实验中观察到的结果支持通过内部折叠状态调节AIP1蛋白相互作用。虽然体外和半体内GST拉降实验显示全长AIP1与ABAP1、ARIA、LHP1及其自身相互作用,但在酵母双杂交实验中只能观察到AIP1的同二聚体。可能,AIP1的原生结构需要进行结构修饰,以允许DUF724与这些蛋白质结合。我们可以推测,这种修饰可能涉及AIP1 N端区与组蛋白的结合,以允许DUF724与其他蛋白结合。
尽管Polycomb动物和植物发育中的基因及其在植物中的作用机制仍然知之甚少[62].在这项工作中,我们发现AIP1可以结合LHP1,一种植物POLYCOMB REPRESSOR COMPLEX1-like (PRC1-like)功能的主要蛋白[63].此外,AIP1的Agenet/Tudor结构域的预测结构非常接近哺乳动物PHF1蛋白的Tudor结构域,后者与甲基化组蛋白结合,必要地将PRC2招募到染色质[31].此外,已有报道称LHP1与PRC2的一个成员结合,促进其招募到携带H3K27me3的染色质区域[64].LHP1与H3K27me3的结合是其功能和抑制几种PcG蛋白靶点如FT、AG和AP3所必需的[63],这项工作表明这三个基因在AIP1中上调KD植物。研究表明,植物PRC2-like复合物和PRC1-like复合物之间的相互作用有助于DNA复制期间H3K27me3的遗传,并有助于间期期间H3K27me3水平的维持[64].因此,我们可以推测,AIP1通过与LHP1和ABAP1结合,参与蛋白复合物,通过组蛋白修饰和染色质重塑来调控花发育过程中的基因表达。最后,在AIP1中观察到花成熟的延迟KD可能是由于DNA复制、细胞分裂和花发育基因的表达上调,导致发育中的花细胞分裂和细胞分化率不平衡。
结论
对含有Agenet/Tudor结构域的植物蛋白的系统发育和表达分析表明,它们可能在植物繁殖过程中发挥了作用。我们认为AIP1是Agenet/Tudor家族的一个新成员,可能在DNA复制和/或DNA转录过程与染色质可及性动态之间相互连接,以调节拟南芥花的发育.这一假设得到了AIP1与组蛋白、ABAP1、ARIA和LHP1相互作用的发现的支持;此外,AIP1、ABAP1和组蛋白似乎存在于同一复合物中。此外,AIP1的Agenet/Tudor结构域的预测结构与哺乳动物中招募PRC2到染色质的PHF1蛋白最相似;LHP1与植物中PRC2的一个成员结合,促进其募集到染色质,以调节与开花有关的基因的表达。此外,假定的AIP1同源体只存在于被子植物支系中。此外,AIP1主要在生殖组织和植物中表达,AIP1显示花发育时间的延迟。最后,在AIP1水平降低的植物花蕾中,ABAP1或LHP1转录抑制靶基因的mRNA水平下调。进一步对含有AIP1/DUF71的蛋白质复合物进行生化和结构分析,对于揭示Agenet/Tudor整合这些细胞过程的机制非常重要。
参考文献
穆塞尔曼,Avvakumov N, Watanabe R, Christopher G, Lalonde M, Hong Z,等。分子生物学杂志,2013;19(12):1266-72。
Hughes-davies L, Huntsman D, Ruas M, Fuks F, Bye J, Chin S,等。EMSY连接BRCA2通路。细胞。2003;115:523-35。
毛勒-stroh S, Dickens NJ, Hughes-davies L, Kouzarides T, Eisenhaber F, Ponting CP.都铎域“皇室”:趋势生物化学,2003;28(2):69-74。
Alpatov R, Lesch BJ, Nakamoto-Kinoshita M, Blanco A, Chen S, Stützer A,等。脆性X智力迟钝蛋白FMRP在DNA损伤反应中的染色质依赖作用。细胞。2014;157(4):869 - 81。
Myrick LK, Hashimoto H, Cheng X, Warren ST.人类FMRP含有一个完整的串联Agenet (Tudor)和KH基序在氨基端结构域。Hum Mol Genet, 2014; 1-8。
曹旭,杨克忠,夏超,张学强,陈立强,叶丹。拟南芥DUF724基因家族的鉴定。植物分子生物学杂志,2010;32(1):344 - 344。
张文杰,张文杰,张文杰,等。赖氨酸甲基化依赖53BP1结合到pRb肿瘤抑制因子。中国科学(d辑),2014,26(3):344 - 344。
Gelato KA, Tauber M, Ong MS, Winter S, Hiragami-Hamada K, Sindlinger J,等。UHRF1不同组蛋白h3结合域的可达性受磷脂酰肌醇5-磷酸的变构调控。中国生物医学工程学报。2014;54(6):905-19。
Mudbhary R, Hoshida Y, Chernyavskaya Y, Jacob V, Villanueva A, Fiel MI,等。UHRF1过表达导致DNA低甲基化和肝细胞癌中国癌症杂志。2014;25:196 - 9。
杜俊,钟旭,Bernatavichute YV, Stroud H,冯s, Caro E,等。色素甲基化酶域与含h3k9me2的核小体的双结合指导植物DNA甲基化。细胞。2012;151(1):167 - 80。
桑切斯R,周敏敏。博士手指:多功能表观基因组阅读器。生物化学学报,2012;36(7):364-72。
拟南芥防御成分EDM2以flc依赖的方式影响花的转变。植物学报,2010;36(3):344 - 344。
Hernandez JM, Feller A, Morohashi K, Frame K, Grotewold E.玉米R的基本螺旋-环-螺旋结构域通过招募emsy相关因子连接转录调控和组蛋白修饰。自然科学进展,2007;26(3):344 - 344。
Makarov V, Rakitina D, Protopopova A, Yaminsky I, Arutiunian A, Love AJ,等。植物线圈的结构特征和rna结合特性。科学通报。2013;18 (1),e53571。
王伟,陈震,毛震,张宏,丁霞,陈松,等。核仁蛋白Spindlin1识别H3K4甲基化并刺激rRNA基因的表达。环境科学学报,2011;12(11):1161-7。
赵强,奎林,姜峰,吴波,岳伟,徐峰,等。人类纺锤蛋白1的结构。细胞周期调控的串联都铎类结构域。中国生物化学杂志,2007;
Masuda HP, Cabral LM, De Veylder L, Tanurdzic M, De Almeida EJ, Geelen D,等。ABAP1是一种新型的犰狳BTB蛋白,参与DNA复制和转录。中国生物工程学报。2008;27(20):2746-56。
郭德华,陈志强,张志强,等。Phytozome:绿色植物基因组学的比较平台。核酸研究,2012;1178-86。
协调员NR.国家生物技术中心数据库资源。信息。2013;41:8-20。
李建平,李志强,李志强,等。拟南芥信息资源(TAIR):改进的基因注释和新工具。2012; 40:1202-10。
nestedt B, Street NR, Wetterbom A, Zuccolo A, Lin Y-C, Scofield DG,等。挪威云杉基因组序列与针叶树基因组进化。自然。2013;497(7451):579 - 84。
李志强,李志强,李志强,等。Pfam:蛋白质家族数据库。2014; 42:222-30。
Letunic I, Doerks T, Bork P. SMART 6:最新更新和新发展。核酸决议2009;37:229-32。
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6 :分子进化遗传学分析版本6.0。MBE。2013; 1 - 12。
克鲁克斯GE,汉G,夏冬尼亚J,布伦纳SE。WebLogo:一个序列Logo生成器。基因组研究,2004;1188-90。
Bannai H, Tamada Y, Maruyama O, Nakai K, Miyano S. n端蛋白分选信号的广泛特征检测。生物信息学,2002;18(2):298 - 305。
张艳,张志刚,张志刚,张志刚。一种基于tasser的蛋白质结构和功能预测系统。Nat协议。2010;5:725-38。
德拉诺王。PyMOL分子图形系统。蛋白质晶体。2002;40:44-53。
基于层次聚类的多序列比对。中国生物医学工程学报。1988;16(22):10881-90。
Gouet P, Courcelle E, Stuart DI, Métoz F. ESPript: PostScript中多序列对齐的分析。生物信息学,2000;15(4):305 - 8。
二级结构匹配(SSM)是一种快速三维蛋白质结构比对的新工具。国际结晶学联合会;2004; 2256 - 68。
拉斯科夫斯基RA,沃森JD,桑顿JM。ProFunc:从3D结构预测蛋白质功能的服务器。核酸,2005;33:89-93。
Sparkes IA, runion J, Kearns A, Hawes C.荧光融合蛋白在烟草植物中的快速瞬时表达和稳定转化植物的生成。Nat Protocols. 2006;1(4): 2019-25。
Tarun SZ, Sachs AB.酵母聚(A)尾结合蛋白与翻译起始因子elF-4G的关系。中国科学d辑。1996;15(24):7168-77。
Gietz D, St A, Robin J, Schiestl RH。完整酵母细胞高效转化的改进方法。核酸研究,1992;20(6):1425。
Van Leene J, Stals H, Eeckhout D, Persiau G, Van De Slijke E, Van Isterdael G,等。拟南芥细胞周期相互作用组研究的串联亲和纯化技术平台。Mol细胞蛋白质组学。2007;1226-38。
范丽娟,杨晓娟,杨晓娟,Inzé D。植物蛋白复合物的分离纯化。植物科学进展,2008;13:517-20。
Van Leene J, Hollunder J, Eeckhout D, Persiau G, Van De Slijke E, Stals H,等。目标相互作用组学揭示了拟南芥复杂的核心细胞周期机制。中国生物医学工程学报,2010;6(397):397。
徐玲,苏智,顾志,顾旭。叶猴rnase的分子系统发育与进化:基因平行复制或基因平行转换是分子系统发育的一个问题。分子系统进化学报,2009;50(2):397-400。
赫鲁兹,劳勒O, Szabo G, Wessendorp F, Bleuler S, Oertle L,等。基因研究者V3:转录组元分析的参考表达数据库。Adv In Bioinform. 2008;2008:420747 - 52。
穆塞尔曼,Avvakumov N, Watanabe R, Christopher G, Lalonde M, Hong Z,等。PHF1 Nat结构的Tudor结构域识别H3K36me3的分子基础。Mol生物学,2013;19(12):1266-72。
玛格丽特R,莱茵伯格D.多梳复合体PRC2及其在生命中的印记。大自然。2011;469(7330):343 - 9。
李志强,李志强,李志强,等。拟南芥种子发育过程中基因活性的全局分析及种子特异性转录因子的鉴定。自然科学进展,2010;26(3):344 - 344。
Belmonte MF, Kirkbride RC, Stone SL, Pelletier JM, Bui AQ, yang EC。拟南芥种子区域和亚区域基因活性的综合发育特征。自然科学进展,2013;29(3):344 - 344。
石田Y,林林K,苏野K,麻美T,中村S,铃木M,等。拟南芥生长素生物合成抑制剂吲哚-3-氧乙基膦酸的分析。中国生物工程学报。2014;37(1):344 - 344。
莫里托A,沈文华。多梳复合体PRC1在植物中的组成与功能。中国基因工程学报。2013;40(5):329 - 329。
张志刚,张志刚,张志刚。植物早期花发育。《植物科学》1990;2:755-67。
李志强,李志强,李志强,等。GFP-PCNA在胚胎中s期标记的研究进展。生物细胞。2005;97:221-9。
李志强,李志强,李志强,等。一种不稳定的细胞周期蛋白- gus融合蛋白的有丝分裂活性时空分析。植物学报1999;20:503-8。
Germann S, Juul-Jensen T, Letarnec B, Gaudin V. DamID,一种研究植物体内染色质谱的新工具,及其用于确定LHP1的目标位点。植物学报,2006;48:153-63。
Gillespie DE, Berg CA。无家可归的人是RNA定位所必需的Drosovhiladna依赖atp酶的DE-H家族的一个新成员编码。基因发展。1995;9:2495-508。
RISC亚基Tudor-SN与超编辑双链RNA结合并促进其裂解。中国生物工程学报。2005;12:489-96。
DC, Ayyanathan K, Negorev D, Maul GG, Iii FJR。SETDB1:一种新的kap -1相关组蛋白H3,赖氨酸9特异性甲基转移酶,有助于hp1介导的KRAB锌指蛋白对纯色基因的沉默。基因发展。2002;919-32。
黄勇,方杰,Bedford MT,张勇,徐瑞敏。JMJD2A双都铎结构域识别组蛋白H3赖氨酸-4甲基化。科学。2006;312:748-51。
都铎域。中国生物医学杂志,2010;20(16):666-7。
拟南芥EMSY - Like基因在RPP7介导的种族特异性免疫和基础防御中的作用植物微生物相互作用。2011;24(12):1573-81。
罗佳,杜j,黑尔CJ,冯S, Krajewski K, Palanca AMS,等。聚合酶IV占据rna定向的DNA甲基化位点需要SHH1。大自然。2013;498(7454):385 - 9。
王俊杰,王俊杰,王志强,等。都铎结构域在植物种系发育中的作用。发展。2006;4062:4053 - 62。
拟南芥TERMINAL FLOWER 2基因编码异染色质蛋白1同源物并抑制开花位点T调控开花时间和若干花同源异型基因植物生理学报。2003;44(6):555-64。
陈琳,宋志荣,陈伟杰,陈伟杰。胚花1参与拟南芥多梳群介导的AG基因沉默。《植物细胞》2008;20:277-91。
Suzuki M, Takahashi T, Komeda Y.拟南芥corymbosa2突变体因抽苔和花发育延迟而形成的伞房花序。植物细胞生理学杂志,2002;43(3):298-306。
濮林,宋志荣。PcG和trxG在植物-朋友或敌人。植物学报,2015;31(5):252-62。
李志强,李志强,李志强,等。LIKE异染色质蛋白1的染色体结构域在拟南芥发育过程中与H3K27me3结合并发挥作用。科学通报。2009;4(4):1-10。
陈晓华,杨晓华,杨晓华,等。Mozgová拟南芥MSI1连接LHP1和PRC2复合物。中国生物医学工程学报。2013;32(14):2073-85。
致谢
这项研究得到了Fundação de Amparo à Pesquisa do Estado do里约热内卢de Janeiro (FAPERJ), Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES), Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq)和Instituto Nacional de Ciência de Tecnologia (INCT)生物固氮的支持。JNB得到了CNPq和国际交流项目科学无国界(CNPq)的博士奖学金支持。LPPG由CAPES硕士奖学金支持。ASH和PCGF得到CNPq研究基金的支持。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
JNB进行了分子和细胞研究在网上分析并起草稿件。LMC进行生化分析。ILBN执行在网上建模。NBE参与了分子研究。LFP参与了分子和酵母-两种杂交实验。LPPG参与基因表达分析。PCGF、NG和DI参与了结果讨论。ASH构思了这项研究,并参与了其设计和协调,并帮助起草了手稿。所有作者都阅读并批准了最终的手稿。
附加文件
附加文件1:
Agenet/Tudor结构域蛋白在植物王国中所研究的基因组及其系统发育关系的示意图。(PDF 552 kb)
附加文件2:
植物蛋白Agenet/Tudor结构域的建模结构。(PDF 925kb)
附加文件3:
来自Agenet/Tudor家族四个不同类别的每个基因的时间表达模式。(PDF 829kb)
附加文件4:
酵母双杂交实验中AIP1与ABAP1结构域的相互作用。(PDF 204kb)
附加文件5:
下拉实验中AIP1与H3K9ac和H3K14ac相互作用的分析(PDF 60kb)
附加文件6:
的表型分析AIP1KD营养发育时期的线。(PDF 640kb)
附加文件7:
都铎家族植物的所有成员。(PDF 417kb)
附加文件8:
利用AIP1蛋白的第二个Agenet/Tudor结构域作为查询,在ESPrint中获得结构相似性参数数据。(PDF 178kb)
附加文件9:
参数数据来自I-TASSER中的结构建模。(PDF 7kb)
附加文件10:
使用含有DUF724结构域的AIP1 c端部分作为诱饵,在酵母二杂交筛选中测试的蛋白质列表。(PDF 11kb)
附加文件11:
以AIP1为诱饵的串联亲和纯化蛋白。(PDF 100kb)
附加文件12:
用于PCR克隆和qRT-PCR扩增的引物。(PDF 10kb)
附加文件13:
补充方法。(docx26kb)
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
巴西,j.n.,卡布拉尔,l.m.,埃洛伊,N.B.et al。AIP1是一种来自拟南芥的新型Agenet/Tudor结构域蛋白,与DNA复制、转录和染色质重塑的调控因子相互作用。BMC植物生物学15日,270(2015)。https://doi.org/10.1186/s12870-015-0641-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-015-0641-z
关键字
- Agenet /都铎
- 都铎王朝
- DUF7
- DUF724
- ABAP1
- 染色质重塑
- 细胞周期
- 拟南芥