抽象的
背景
抽穗期的变异可能有助于小麦适应当地环境。因此,发现新的抽穗期决定因素对谷物改良具有重要意义。本研究以普通小麦品种中国春(CS)为材料,以具有5B染色体的中国春(CS)代换系为材料T. dicoccoides.(CS-5BDIC),在他们的标题日期与两周不同,以检测5B染色体上标题日期的决定因素。
结果
可能的影响VRN-B1结果表明,CS- 5bdic与CS- 5bdic在抽穗时间上存在差异。对CS-5Bdic基因的测序表明,插入一个核苷酸三联体会在相应的蛋白质中产生额外的氨基酸。每个同源体的转录水平没有变化VRN-1与CS比较,CS- 5bdic中存在基因座。为确定决定抽穗期差异的基因座,利用CS- 5bdic与CS- 5bdic杂交,建立了116个重组自交系5В,并对其抽穗期进行了估算。利用Illumina Infinium 15 k Wheat平台,共检测到379个5b特异性多态性标记,构建了包含82个骨骼标记的遗传图谱。表型(抽穗期)-基因型关联分析显示,5B染色体中有78个标记与抽穗期变异显著相关。基于这种估计和与模式作物基因组的一致性,我们确定了三个最佳候选基因:怀疑那小块土地/ AP2和FHY3 / FAR1.
结论
我们认为活动的差异怀疑那小块土地/ AP2和/或FHY3 / FAR1CS和CS-5BDIC之间的转录因子是观察到期限日期差异的可能原因。本研究中获得的数据为随后调查小麦的前线时间途径提供了良好的基础。
背景
谷物作物最重要的性状之一是抽穗期[1].从营养生长到生殖生长的过渡是一个关键的发育阶段,是作物和野生谷物物种的主要适应特性[2].小麦品种与目标环境的适当出头时间和生命周期持续时间将有助于最大化任何环境的产量潜力[3.那4.].小麦抽穗期由3个主要遗传因子决定:春化要求、光周期敏感性和狭感早度[5.那6.].5号染色体的小麦组携带影响抽穗期的基因,例如VRN-1[7.-9.],PHY-C[10-12].例如,VRN-1基因编码MADS-box转录因子已被报道是决定春化要求和抽穗期的关键调控因子[13-16].估计VRN-1转录本水平表明,它们的浓度必须达到一个阈值,才能触发从营养发育到生殖发育的过渡[17].在需要春化(冬季)的谷类植物中,VRN-1在低水平表达,并由春化诱导,表达水平依赖于冷暴露的长度[18-21].调控区域(启动子或内含子1)发生显性突变,导致冬型向不需要春化的春型的生长习性发生变化VRN-1.利用近等基因系,研究了基因第一个内含子的结构变异VRN-B1是导致两线之间的高差异的原因VRN-B1转录及花期[22].
影响标题日期的其他一些基因座在5B染色体上位于5B染色体上[9.那23-26].使用基于遗传联系地图的QTL分析来识别这些基因座。基因图是基因组研究的重要工具,标记辅助选择,QTL分析和基于地图的克隆,物理映射和进化基因组学[27那28].有一些地图t .硬质xT. dicoccoides.基于SSR、RFLP、AFLP、RAPD和DArT标记[29].这些标记不容易进行高通量基因分型[30.].与这些类型的分子标记相比,snp非常适合于高分辨率遗传图谱的构建和标记-性状关联的发现[31那32].
在我们的研究中,我们使用了CV。中国春天(CS,单倍型vrn-A1vrn-B1Vrn-D1)和CS- 5bdic,由5B染色体取代的CS系T. dicoccoides.检测5B染色体上标题日的决定因素。CS早于CS-5BDIC 15天。我们检查了(i)可以通过转录的差异充分解释这些线之间的标题日期的差异VRN-1或(ii)可以通过映射到5b的一些其他基因座(qtls)的差异来解释。为了识别出头时间的基因座确定差异,我们开发了源自CS-5BDIC的CS的十字谱的重组近亲染色体线(RICL)的映射群。使用Illumina Infinium 15 K小麦平台进行基因分型进行映射群。QTL分析允许我们识别在5B染色体的泌乳术区中覆盖78个SNP标记的基因座(p-Value = 0.001)与标题日期变化相关联。基于Synteny与模型作物基因组,我们确定了三种最佳候选基因:怀疑那小块土地/ AP2和FHY3 / FAR1.
方法
植物材料
中国春天(CS),CS替代线,5B染色体T. dicoccoides.(CS- 5bdic)和CS- 5bdic F1种子由美国堪萨斯州立大学b.s. Gill教授和美国法戈大学j.d. Faris教授提供。通过7代自花授粉,获得了116个重组自交系(RICLs)群体。这些行预计包含的区域为T. dicoccoides.5B染色体向5B染色体渐渗。
DNA提取和纯化
使用亚硫酸氢钠方案提取DNA。新鲜的叶片组织(100-150mg)具有200μl的温热(60℃)萃取缓冲液(0.1M Tris,0.5M NaCl,0.05M EDTA,0.38%NaHSO3.(1.25% SDS)用MP FastPrep-24均质。然后加入温提取缓冲液(60°C) 500 ml,混合,60°孵育30 min。加入氯仿/异戊醇700 μl (24:1 vol:vol)。离心后(15min, 12000 rpm, Eppendorf 5415 R),收集上清于新管中,加入1.4 ml 96%冷(−20℃)乙醇,轻轻混合。离心15 min, 12000 rpm后,取上清,70%乙醇洗涤DNA颗粒,干燥后用50 μl TE缓冲液重悬。SNP基因分型的DNA纯化使用“Bio-Silica试剂盒从反应混合物中纯化DNA”,根据制造商的协议进行。用NanoDrop M2000 (Thermo Scientific)进行DNA浓度测定。
隔离与分析Vrn-B1序列
的相邻外显子1的启动子的分离Vrn-B1我们使用引物P1 (5'-TACCCCTGCTACCAGTGCCT-3')和Int1R (5'-GCAGGAAATCGAAATCGAAG-3')33] 和 [34], 分别。我们孤立第一个内含子的最关键部分Vrn-B1使用引物对ex1 / c / f // intor1 / b / r4报告[35].外显子2和外显子8之间的区域(ex2-ex8)被拆分为两个亚区,并分别扩增,如[22].扩增的DNA片段从1%琼脂糖凝胶中回收,使用QIAGEN MinElute Gel Extraction Kit (QIAGEN,德国)纯化,使用ABI PRISM Dye Terminator Cycle Sequencing ready reaction Kit (Perkin Elmer Cetus, USA)和相应的特异性引物直接测序。使用ABI PRISM 310基因分析仪(Perkin Elmer Cetus)对每个片段进行双向测序。报道的核苷酸序列Vrn-B1从简历的等位基因。Chinese Spring和取代系CS-5Bdic分别以登录号[GenBank:KT750252]和[GenBank:KT750253]保存在EMBL、GenBank和DDBJ核苷酸序列数据库中。
RNA提取和逆转录
本研究中使用的植物的生长条件、RNA提取和逆转录如Shcherban等人所述进行[22].植物在温室(20℃-25℃)的温室(20℃-25℃)下,每天12小时,没有vernalization。将单个植物的叶片切割并在不同的发育阶段中汇集,由完全出现的叶子的数量定义:第三(第3),第四(第4)和第五(第5)叶阶段。我们使用每次重复5株植物的叶形端子。在每个发育阶段进行两次重复。
使用RNEasy植物迷你RNA提取试剂盒(德国)从植物的叶子顶部的叶子顶部提取RNA提取RNA:三叶,四叶和五叶阶段。使用特异于泛素基因(UBC)的引物检查RNA样品进行基因组DNA污染物[36].使用(dt)从1μg的总RNA合成单链cDNA15引物与QIAGEN全基因逆转录试剂盒在20 μl反应混合物中。
半定量rt - pcr
我们使用了特定的底漆Vrn-A1那Vrn-B1和Vrn-D1由Shcherban等人开发的编码序列。[22].PCR混合物(20μL)含有50ng cDNA,每种引物,0.25mm,0.2mM DnTPS,1x反应缓冲液(67mM Tris HCl pH 8.8; 1.8 mm MgCl2;18毫米(NH4.)2所以4.;0.01%吐温20)和1单位TaqDNA聚合酶。使用触地相PCR(30℃的13个循环为30℃,65℃(-0.5℃/循环),30 s,72℃,24个循环为94℃,60℃,60℃,72℃为45℃。PCR产物分离1.5%琼脂糖凝胶电泳。
表现型的人口
在受控条件下的温室中进行了线性的表型分析。如果必须满足春化要求,则在黑暗中+ 3°C致春季春季化的种子在+ 3°C中富含春季。随着从发芽到标题的期限的持续时间计算标题日期的评估。随后为所有植物记录出标题的日期,作为从旗叶中出现的1/3秒的日期。向航行的日子被录制为从萌芽到标题日期的天数。计算每条线的标题日期的平均值为10个植物。
SNP基因分型
利用116个RICLs,亲本,DT5BL和N5BT5D SNP基因分型,使用由tritgenetics GmbH [37].分析的基因座的总量为13007.该组中包含DT5BL和N5BT5D,用于将标记物的进一步定位成短(5B)或5B染色体的长(5BL)臂。
筛选RICLSVrn-B1特定的引物
为了筛选重组自交系,利用特异性引物进行区分Vrn-B1来自的等位基因T. Aestivum.,简历。中国春(CS等位基因)、CS- 5bdic系(CS - 5bdic等位基因)T. dicoccoides.).正向引物DICCSINTR5(5'-CCTTGCATACCTGAACCG-3')和CSINTR5(5'-ACCTTGCATACCTGAACCA-3')的序列以这样的方式设计了,即3'末端的最后位置与Intron 5中的SNP匹配Vrn-B1.这些引物与常见的反向引物EX8 / 7(5'-GCCCTTCAGCGTTTGATGGGGGCTA-3'组合使用。使用触控方案进行扩增,以0.5℃/循环在62℃至56℃下降低退火温度。引物对CSINTR5 // EX8 / 7在CS等位基因的情况下给出了约700bp的产物,而在此情况下没有获得产品T. dicoccoides.等位基因。相反,用对DICCSINTR5 // ex8 / 7的扩增在其中提供了700磅的产品T. dicoccoides.等位基因。这种方法使我们能够检测杂合等位基因。
遗传联系地图建设
利用MultiPoint version«UltraDense»软件构建5B染色体遗传连锁图谱[38].具有超过8个缺失的数据点和具有大分离失真的标记的标记(χ2 > 21) were removed. Coefficients of priority were 0.9 for missing data and 0.1 for segregation since we expected only one linkage group. The minimum size of group of co-segregating markers was 2. Co-segregating markers are tightly linked markers located in the same position. Ordering of skeletal markers was performed using guided evolutionary strategy (GES) algorithm [39进行了10次折刀取样。为了获得稳定的骨架图,去除引起不稳定邻域和干扰重组变化单调性的标记。单调性是一个标记和它的相邻标记之间的一个逐步的rf(重组分数)增量。然后利用“延伸连锁群”功能将单个标记添加到图谱中,扩大系数由1.0逐步增加到1.4。对标记顺序进行了单调失真和地图尺寸放大检查。标记,不涉及稳定的地图构建,但对应于任何确定的间隔被称为“附加”。由于多点映射的标记分布在附加文件中有详细的报告1:表S1和由于N5BT5D / DT5BL分析和与先前报告的5B映射数据相比,标记的定位在附加文件中2:表S2。
地图的可视化由MapChart 2.2完成[40].要将所获得的地图与现有的地图进行比较,我们使用生物钻头v4.2软件[41那42].
QTL分析
在2014年和2015年的两种不同的条件下,RICL在两种不同的条件下生长。对于分析,通过降低标记数量来修饰遗传联系地图,以便在标记物2-4cm之间的距离。对于QTL分析,MultiQTL软件[43]用于执行区间映射(一个特征)。为确定QTL阈值LOD值,采用“比较假设H1→H0”选项进行1000次排列。为估计主要参数的标准差,采用bootstrap分析。标题日期性状的区间分析LOD值见附加文件3.:表S3。
确定标题日期QTL的候选基因
鉴定标题日期的候选基因,SNP标记的序列[44[QTL区域是否使用甲克底伤分析[45] 反对Brachypodium distachyon那栽培稻(indica),Hordeum Vulgare.那Triticum Urartu.那Triticum Aestivum.那Aegilops Tauschii..从UniProt数据库获得的序列结果[46)进行了综述。提供了推定蛋白质的参考资料。QTL区域及其相关基因所覆盖的SNPs见附加文件4.:表S4(参见家族/领域注释和根据InterPro实现的描述)。然后分析了这些蛋白参与开花途径的可能性。
结果
序列的分析VRN-B1来自CS和CS-5BDIC的基因
研究的长度VRN-B1来自CS和CS-5BDIC的等位基因,包括启动子和编码区,分别为4780 bp和4783 bp。两种情况下的测序区域分为两部分:1)启动子与外显子1和内含子1的上游序列;2)外显子内含物的外显子。除了Intron 1中的一些替换,VRN-B1CS- 5bdic序列与CS相比只有一个显著变化VRN-B1序列,在外显子7中插入三核苷酸TCT,导致在蛋白质的C-末端区域中添加氨基酸丝氨酸。有趣的是,在该位置,三联体分别在Cs和Cs-5bdic序列中串联重复(分别为6和7次)。
分析VRN-1CS和CS- 5bdic的转录
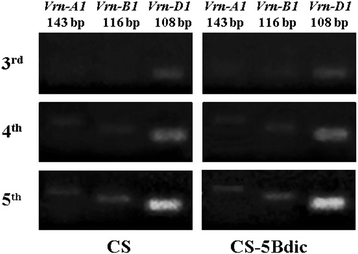
学习Vrn-1CS和CS-5BDIC中的转录水平,我们进行RT-PCR,在标题前的不同阶段的来自无比植物的叶子的RNA样品的RNA样品:第三(3理查德·道金斯)、第四(4th)和第五(5th)叶阶段。在转录本积累的动力学上,两系之间没有差异。的Vrn-D1基因首先被转录,并与该基因在CS基因组中的显性地位相对应。该基因的转录水平从第3叶期到第5叶期逐渐升高,在第5叶期达到最大值(图2)。1).其他两个同源基因的转录本Vrn-1从第4叶阶段开始检测到基因,其强度几乎没有差异。
表达式VRN-1CS和CS- 5bdic的等位基因。RT-PCR产物用针对每个等位基因的特异性引物进行扩增(见方法).PCR产物的长度在碱基对的图像上方给出。叶阶段在面板左侧表示
RICLs表型和SNP基因分型
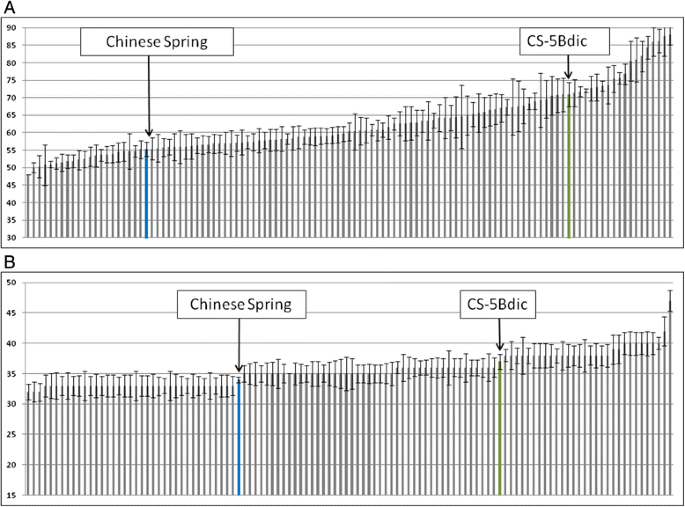
而不是表达水平没有差异VRN-1CS和CS- 5bdic的同源基因CS- 5bdic在重组自交系(RICLs)发育过程中抽穗期延迟,CS和CS- 5bdic的抽穗期相差约15天(图)。2).
为了鉴定Cs和CS-5Bdic的前线时间的基因座和CS-5BDIC,116重组近亲染色体线(RICl)使用Illumina Infinium 15 K小麦平台进行基因分型,并为几个标题特征表现出来。在温室的受控条件下进行了vernalized和非vernalized RICL的航向日期。非常规RICL的航行日期变化为48-88天,分别为父母系CS和CS-5BDIC - 55和71天(图。2).春化植株的抽穗期在32 ~ 47天之间变化,且无显著差异(图2)。2 b).
利用Illumina Infinium 15k小麦平台上的1307个SNP标记对RICLs进行基因分型。其中418个标记在亲本CS和CS- 5bdic之间具有多态性。大多数标记被证明是共显性的(两个等位基因都被检测到),而10个标记仅检测到一个亲本。此外,利用N5BT5D和DT5BL基因分型数据,在5BS或5BL染色体臂上进行多态标记定位。
5B染色体遗传连锁图谱的构建
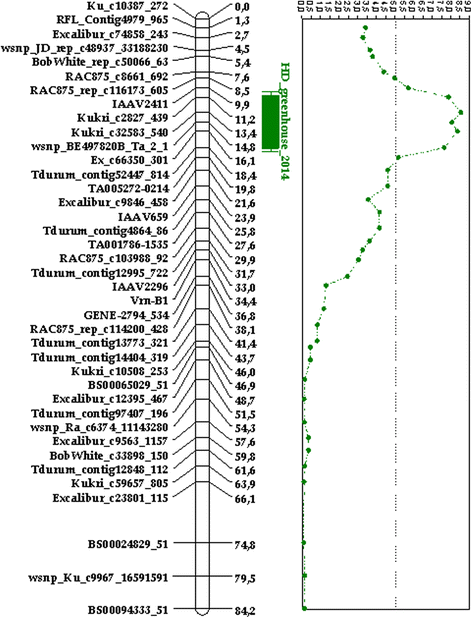
使用116重组近交系的基因分型数据获得遗传联系地图,其中使用多点(版本“Ultradense”)软件,使用116重组近交系的基因分型数据。所得映射的长度等于80.4cm(Kosambi映射函数),总共379个标记(具有82个骨架标记,其中一个代表了一组非重组(共分离)标记)。这些组的尺寸在2至23个标记之间变化。5B染色体的焦化区域的特征在于每个遗传距离的高标记密度。例如,在其他染色体区域中,在焦化区域中具有11个标记的每厘米和2.3个标记。
标记BS00011514_51和wsnp_RFL_Contig2809_2587619与番茄红素相关VRN-B1基因并位于33.4厘米的位置。这些标记包括标记VRN-B1形成了“结合”组(图。3.).
5B染色体遗传连锁图谱。这张地图显示了82个骨骼标记。共隔离标记可以在附加文件中找到1:表S1。以cm表示的距离(Kosambi映射函数)
改进SNP位置
为了确定5BS或5BL染色体臂上标记的位置,我们对无四体系N5BT5D和双体系DT5BL进行了基因分型,并将本研究获得的数据与之前的数据进行了比较。以下11个标记首次在5B染色体上定位:Tdurum_contig31845_322、Kukri_c32583_540、BS00011102_51、TA005272-0214、RFL_Contig3455_700、BS00065265_51、ta00999 -0466(该标记染色体定位有问题,为“附连”标记,未参与稳定骨架图)、BobWhite_c33898_150、Tdurum_contig12848_112、D_GDS7LZN02F4FP5_176、BS00094333_51标记。这些标记均定位于5B染色体长臂上。结合RICLs和N5BT5D、DT5BL的SNP基因分型数据,分析的大多数标记都能准确地归属于5B染色体的短臂或长臂。着丝粒区在11.5 ~ 12.0 cM之间。通过与现有地图的比较,我们可以检测到多个本地化标记。标记位置的数据摘要显示在附加文件中2:表S2。
QTL分析
在材料的表型分析中,我们注意到抽穗期的显著变化只发生在非春化株系和亲本上,而在春化株系中没有发生。利用QTL分析,我们只在非春化群体的实验中检测到显著的lod。QTL位于5BS-5BL中心点区,位于11.2-18.4 cM之间。的VRN-B1基因定位在33.4 cM,与抽穗期无相关性。LOD值的分布在附加文件中3.:表S3。
与抽穗期相关的QTL包括多组共分离标记。由于所使用的阵列的SNP标记是根据编码序列开发的[27我们可以将它们与现有的模式物种数据库进行比较,以检测潜在的标题日期变化的候选基因。
抽穗期变异候选基因的鉴定
分析了位于11.2-18.4cm QTL间隔内的78个SNP,以识别可能涉及航向日期确定的候选基因。与数据库进行比较SNP序列Brachypodium distachyon那栽培稻那Hordeum Vulgare.那山羊草属taushii那T. Uraartu.和T. Aestivum.使用blastn。我们已经鉴定了与SNP序列重叠的编码序列,并检测了编码蛋白的特征、结构域和假定的功能。在检测到的候选基因中,有控制植物抗非生物胁迫的基因,提供损伤保护、植物激素代谢、抗病和一些转录因子(附加文件)4.:表S4)。从这个集合中,我们建议最有可能的前线时间控制候选者是推定转录因子基因怀疑那小块土地/ AP2和FHY3./距离.
讨论
本研究旨在映射座位确定两种小麦线条,品种中国春季和中国春季替代线的标题日期差异,从5B染色体T. dicoccoides..当它们不是vernalized时,这些线在彼此显着分开。解释这些结果的一种方法是研究与标题日期变化相关联的已知基点,例如VRN-1,分析亲本品种及其衍生品系的一级序列和转录水平。第二种方法是开发和研究在单个位点或5B染色体上的某些位点上彼此不同的重组自交系。由于具有无限的品系种质资源和后代中没有进一步分离,RICLs是定位和性状研究的有用遗传模型[47].使用用于构建高分辨率遗传图谱的RILS是有利的,因为重组程度高,并且可以确定紧密连接标记的地图位置[48].
主要结构与表达的比较分析VRN-B1基因
启动子、所有外显子和第1内含子区域的测序VRN-B1结果表明,CS- 5bdic的等位基因与CS的等位基因基本一致,只是在编码区第1内含子上有少量不显著的替换和一个三核苷酸插入。后一种突变似乎是最重要的,因为编码序列的VRN-1与启动子和第1内含子不同,基因即使在不同物种之间也高度保守[34那35].由于其尺寸(〜9 kB),我们没有研究第一个内含子的全序列,然而,大约2kb的上游区域代表了影响的“vernalization关键区域”VRN-1表达和对vernalization的反应[20.那22那33那35那49].
李等人[50表明VRN-A1蛋白c端有一个氨基酸取代,由于该蛋白与TaHOX1蛋白结合的能力降低,使得春化期充分延长T. Aestivum.).与VRN-B1CS和来自数据库的所有其他VRN-B1序列相比,我们发现在CS-5BDIC等位基因的VRN-B1氨基酸序列中插入丝氨酸。与之前的工作一样,此插入也发生在C终端区域内。显然,迟滞着VRN-B1基因T. dicoccoides.可以参与蛋白质 - 蛋白质相互作用,确定小麦的生长习性。然而,这里尚未建立在标题之日起该基因座的效果(图。4.).需要在不同的种植条件下进行额外的实验,以确定这种突变是否涉及上述过程。
影响春化要求和开花时间的另一种方法是调制VRN-1表达式(转录)。以前,直接联想的层次VRN-1建立了小麦抽穗期的转录调控[17那22].在这里,我们没有发现任何的转录水平的差异VRN-1与CS相比,CS- 5bdic基因座(图。1).转录的动力学与以前的数据同意,表明在非春化植物中的表达Vrn-1基因从第三叶期开始,诱导隐性等位基因的转录[17那22].因此,СS-5Bdic和CS都只有一个显性基因那这同样诱导隐性同源性的活动VRN-B1和VRN-B1DIC..
基于CS×CS-5BDIC RICL的遗传联系地图的表征
本研究利用15 K SNP Illumina小麦平台构建了基于CS x CS- 5bdic的5B染色体图谱。短臂上有52个SNP标记,其中骨骼标记13个。CS x CS- 5bdic的5B染色体遗传连锁图上标记总数为379个。这与基于SNP标记的四倍体小麦5B染色体图谱相对应[51].
短臂长度为11.54 cM。5B长臂总长度为68.9,包含327个SNP标记,69个骨骼标记。结果图谱为80.44 cM。一般来说,四倍体小麦5B图谱长度对应206.2 cM [51].在该群体(CS × CS- 5bdic)的重组过程中,5B图谱长度较短。部分品系(20.7%)具有与亲本相同的基因型,其中15.5%的非重组染色体遗传自CS, 5.2%遗传自CS- 5bdic。由于CS-5Bdic具有外来物种的遗传渐渗T. dicoccoides.,可能是CS亲本的等位基因更可取。这一数据表明,来自不同物种的染色体之间的重组过程将受到干扰。
5B染色体的焦化区域的特征在于每个遗传距离的高标记密度。例如,焦化区域中每厘米有11个标记,并且在染色体的远端区域中,每厘米有1.1个标记。这是由于该区域中的低重组率和基因分布的特异性[51].早期的研究表明,重组在5B染色体的中心点周围区域受到抑制,特别是在短臂段[52那53].
除5B染色体长臂远端外,骨骼标记物沿染色体分布有规律。3.).Excalibur_c23801_115与BS00024829_51(约8 cM)、BS00024829_51与wsnp_Ku_c9967_16591591(约4 cM)、Ku_c9967_16591591与D_GDS7LZN02F4FP5_176(约4 cM)之间存在4 ~ 8 cM的间隔,平均距离为0.84 cM。然而,Maccaferri等人在一些地图中报道了这一区域[51在Meridiano x Claudio、Mohawk x Cocorit和Latino x MG5323群体的地图上,标记距离在5BL的远端约4 ~ 10 cM。这些图谱揭示了该染色体区域与CS x CS- 5bdic标记的共性。也许,在这个区域的重组不像在染色体的其他部分那么频繁,在这个区域的基因也更少。然而,利用该阵列获得的基因分型数据构建的图谱可以与90 K Illumina平台获得的更大范围的图谱进行比较。
检测标题日期变化的推定决定因素
小麦标题日期受到春化基因的强烈影响[5.].尽管如此,QTL(图。4.)和表达水平(图。1)分析表明VRN-1与非vernalized CS X CS-5BDIC RICLS中的标题日期变化无关。
5B上另一个对小麦开花很重要的位点是光敏色素C(PHY-Cgene [12].这个基因与VRN-B1[11,但从作图结果和QTL分析中未发现这些位点与所调查品系抽穗期变化相关。
Zanke等[54在5B染色体上显示了标记KUKRI_C10016_369与标题日期相关联。这个基因座与之相关Hd6基因家族的oryza.对小麦抽穗期有重大影响[54].Kukri_C10016_369在我们的调查下的RICLS中是多态的,并且位于地图上的位置为35.6厘米,恰好下方VRN-B1位于33.4厘米的位置。但在该区域未发现显著的QTL。
因此,抽穗期的显著变化仅在非春化发育的情况下观察到,与春化发育无关VRN-B1那PHY-C或Hd6基因。
除了VRN-B1那PHY-C和Hd6位于5B染色体上的其他位点可能影响heading date [55].这些位点被证明与以下标记相关:ACA.CTA13 / CTCG.CAT7.[56],Xwmc745-Xcfa21215B.2[25],Xgwm371[24],wpt - 9814иwpt - 4551[57].5BL上的这些染色体区域均与抽穗期存在关联,大部分与着丝粒不存在关联。虽然每股收益基因座与之相关联Xwmc73[9.]定位于5BL着丝粒区,但未提出候选基因。早熟本身是满足了春化和光周期要求的品种抽穗期的差异[58[我们观察了非春化植物的标题日期变化,表明该特征是通过前线时间路径的变化来确定的。此外,在我们的调查中,日期,QTL在5BL和5BS的焦化区域中定位。
为了检测涉及出头时间控制所涉及的5B的序列,我们开发了5B染色体的遗传联系地图。通过与其他物种的基因组(如紫色假麦片,米饭,大麦,常见的小麦,野生Eckorn和山谷)的比较,鉴定了航向日期的候选基因。QTL区域包括一些转录因子,参与应激适应的基因,植物基族代谢,细胞因子,细胞间转运,蛋白质折叠和光合作用。
影响标题日期的推定基因之一是怀疑转录因子。怀疑转录因子是许多植物过程的关键调控因子,包括对生物和非生物胁迫的响应、衰老、种子休眠和种子萌发、毛状体发育和表皮蜡质加载,介导ABA信号传导和提高抗旱性[59-63].
在大豆怀疑转录因子控制花的起始。GsWRKY20积极调节开花。GsWRKY20通过调控开花相关基因和花分生组织认同基因来加速植物开花[62].同样在miscanthus中MLWRKY12TF建议控制开花[64].
但是,对宪法和功能知之甚少怀疑面包小麦的基因(T. Aestivum.)[65].常见的小麦怀疑基因参与叶片衰老和非生物胁迫[66],冷硬初期的植物抗性[67].Wrky蛋白可能在ABA信号转导途径中发挥重要作用,涉及植物胁迫耐受性和萌发过程中的过程[68].TaWRKY显示基因以ABA依赖方式参与非生物应激反应[65那69].然而,没有关联怀疑在小麦中已经发现了开花基因。
调节出头时间的其他候选基因是一个小块土地/ AP2转录因子。的小块土地/ AP2显示家族基因响应干旱,盐度,温度变化,抗病性和开花控制途径参与涉及[70那71.].拟南芥AP2最典型的基因是AP2 /小块土地家庭。AP2编码参与花分生组织形成的假定转录因子[72.],植物器官特性的说明[73.],花的同源性基因表达调控[74.那75.在拟南芥[76.].
QTL区域与SNP重叠的另一个序列为相关基因红光对于细长HYPOCOTYL3(FHY3.) 和远红障碍的反应1(距离)域名。FHY3.和距离是由古老的转座酶衍生的转录因子。FHY3.和距离是如何调节的凤凰-高等植物的信号[77.),凤凰(光敏色素的)在拟南芥漫长的一天促进开花[78.那79.].其他调查表明FHY3.在昼夜节日和航行日期控制和转向时间的标题中发挥着主要作用ELF4(早期开花4.)[77.].晚些时候FHY3.和距离被证实将光信号整合到昼夜时钟中,并通过直接上调ELF4和叶绿体的积累和复制5(ARC5)[77.那80那81.].在硅分析中栽培稻cis-elements建议FAR1 / FHY3.涉及由于的光周期反应PHY-A规定 [82.].
通过迟钝的延迟开始日期延迟的可能原因T. dicoccoides.可能是由于影响抽穗期的结构基因和调节这些基因活性的转录因子的祖先不同。以前,改变的基因表达被证明在种间杂交中是常见的。例如,在F3h.[83.]和铝耐受基因[84.在小麦-黑麦杂交品种中。在大麦渗入的小麦系中,许多基因的表达量也较低[85.].
结论
研究基因渗入的影响T. dicoccoides.普通小麦的5B染色体T. Aestivum.在抽穗期,我们对开花最有力的调控因子,VRN-B1位于这种染色体上。不同的转录无差异VRN-1在CS-5BDIC替代线和CS之间观察到Homeo并对。存在另外的氨基酸VRN-B1CS-5Bdic蛋白序列提示可能与其他蛋白相互作用,参与调控开花和春化反应。
在该研究中开发的该组RICLS为5B遗传联系地图构建和标志性关联分析提供了良好的基础。当他们未审核时,线条日期不同。标题日期变化的QTL分析证明了5B染色体的终点区域中的基因座与标题日期显着相关。与该区域中的SNP标记相关的一些基因可能是标题日期差异的候选者。怀疑那小块土地/ AP2和FHY3./距离转录因子以前被证明参与了开花时间的调节。我们认为,抽穗期差异的可能原因可能是相互作用的抽穗期途径的起源不同,可能是位于5B上的转录因子的调节。进一步研究这些转录因子及其与已知抽穗期基因的相互作用,有助于我们进一步破译谷类作物抽穗期途径。
参考文献
- 1.
王志强,王志强,王志强,等。Ppd-1对小麦营养和生殖发育阶段开花基因表达的影响。基因工程学报。2012;87:161-8。
- 2.
李福杰,李国华,李国华,等。温带谷物开花时间的控制:基因、驯化和可持续生产力。J Exp Bot. 2007; 58:1231-44。
- 3.
斯内普·j·w,巴特沃斯·K,白教堂·E,沃兰德·j。等待好时机:小麦开花时间的遗传学。Euphytica。2001;119:185 - 90。
- 4.
Seki M,Chono M,Matsunaka H,Fujita M,Oda S,Kubo K,等。光周期不敏感等位基因PPD-B1A和PPD-D1A的分布及其对日本小麦品种前线时间的影响。品种sci。2011; 61:405-12。
- 5.
Worland AJ。开花时间基因对欧洲小麦春化敏感性环境适应性的影响。Euphytica。1996;89:49-57。
- 6.
小麦品种低温需寒性和狭义早熟性的评价方法。日本J品种。1988;38:172-86。
- 7。
Barrett B,Bayram M,Kidwell K.使用往复测绘群体鉴定六倍体小麦中的冬季化响应基因VRN-B1的AFLP和微卫星标记。植物品种。2002; 121:400-6。
- 8。
Leonova I,Pestsova E,Salina E,EFREMOVA T,Roder M,Borner A.使用微卫星标记的Triticum aestivum中的VRN-B1基因的映射。植物品种。2003; 122:209-13。
- 9。
Tóthb,galiba g,fehére,sutka j,snape JW。映射基因影响开花时间和霜抗染色体5B小麦。Al Appl Genet。2003; 107:509-14。
- 10.
Devos Km,Beales J,Ogihara Y,Doust An。Allohexaploid小麦植物中C基因及其上游区域的对比序列分析揭示了其三种组分基因组的演化的新数据。植物mol biol。2005; 58:625-41。
- 11.
Wiebe K, Harris NS, Faris JD, Clarke JM, Knox RE, Taylor GJ,等。针对硬粒小麦(Triticum turgidum L. var durum)籽粒镉浓度调控基因座Cdu1的定位研究Theor Appl Genet. 2010; 121:1047-58。
- 12.
陈安华,李超,胡伟,刘明明,林华,罗克威尔NC,等。在长日照条件下,光敏色素C在小麦开花加速中起主要作用。中国科学技术大学学报(自然科学版)2014;111:10037-44。
- 13.
Murai K,Miyamae M,Kato H,Takumi S,Ogihara Y.WAP1,小麦Apetala1同性恋者在植物转变中起着营业生殖增长起到了核心作用。植物细胞生理。2003; 44:1255-65。
- 14.
陈志强,陈志强,陈志强,等。小麦春化基因VRN1的定位克隆。美国国家科学研究院2003;100:6263-8。
- 15.
Shitsukawa N, Ikari C, Shimada S, Kitagawa S, Sakamoto K, Saito H,等。单粒小麦(Triticum monococcum)突变体,维持营养阶段,是由VRN1基因缺失引起的。基因工程学报2007;82:167-70。
- 16.
Trevaskis B,Hemming Mn,Dennis Es,孔雀WJ。谷物中含葡萄酒诱导的谷物中的分子基础。趋势植物SCI。2007; 12:352-7。
- 17.
Loukoianov A,Yan L,Blechl A,Sanchez A,Dubcovsky J.在正常和转基因多倍体小麦中的VRN-1 vernalization基因的调节。植物理性。2005; 138:2364-73。
- 18。
陈志强,王志强,王志强,等。TaVRT-1转录因子的研究进展[J]。植物杂志。2003;132:1849-60。
- 19。
Trevaskis B, Bagnall DJ, Ellis MH, Peacock WJ, Dennis ES。MADS box基因控制玉米春化诱导的开花。美国国家科学研究院2003;100:13099-104。
- 20。
冯泽维兹,张志强,张志强,等。大麦春化基因的分子和结构特征。中国农业科学(英文版)。
- 21.
Trevaskis B,Hemming Mn,孔雀WJ,Dennis Es。HVVRN2响应DayLength,而HVVRN1受vernalization和发育状态的调节。植物理性。2006; 140:1397-405。
- 22.
schcherban AB, Khlestkina EK, Efremova TT, Salina EA.两个差异表达的小麦VRN-B1等位基因对抽穗期的影响与第一个内含子的结构变化有关。遗传。2013;141:133-41。
- 23.
利用重组自交系对六倍体小麦抽穗性状的分离分析。遗传。2003;90:56 - 63。
- 24.
Hanocq E,Niarquin M,Heumez E,Rousset M,Le Gouis J. QTL在面包小麦重组近交系中QTL对QTL的检测和映射。Al Appl Genet。2004; 110:106-15。
- 25.
Griffiths S,Simmonds J,Leverington M,Wang Y,Fish L,Sayers L等人。ELITE欧洲冬小麦种质中耳出苗遗传控制的META-QTL分析。Al Appl Genet。2009; 119:383-95。
- 26.
Bennett D,Reynolds M,Mullan D,Izanloo A,Kuchel H,Langridge P等。在热,干旱和高产潜在环境下检测面包小麦(Triticum Aestivum L.)中的两个主要谷物产量QTL。Al Appl Genet。2012; 125:1473-85。
- 27.
王胜,黄德华,黄伯仁,等。利用高密度90000单核苷酸多态性阵列分析多倍体小麦基因组多样性。中国生物技术学报(英文版);
- 28.
Avni R,Nave M,EILAM T,SELA H,Alekperov C,Peleg Z等。杜兰姆小麦的超密集遗传地图×野外emmer小麦使用90 k iselect snp基因分型测定产生。mol品种。2014; 34:1549-62。
- 29.
Graingenes:Triticeae和Avena的数据库。http://wheat.pw.usda.gov/GG3/map_summary.2015年5月5日访问。
- 30.
基于Illumina GoldenGate技术的小麦单核苷酸多态性基因分型研究。Theor Appl Genet. 2009; 119:507-17。
- 31。
Zhao K, Aranzana MJ, Kim S, Lister C, Shindo C, Tang C, et al. .一个拟南芥在结构化样本中关联映射的例子。公共科学图书馆麝猫。2007;3:e4。
- 32。
Aranzana MJ, Kim S, Zhao K, Bakker E, Horton M, Jakob K, et al.;拟南芥全基因组关联定位鉴定了已知的开花时间和抗病基因。公共科学图书馆麝猫。2005;1:e60。
- 33.
Shcherban AB, Efremova TT, Salina EA.利用两个不同抽穗期小麦近等基因系鉴定一个新的Vrn-B1等位基因。摩尔品种。2012;29:675 - 85。
- 34.
Yan L,Helguera M,Kato K,Fukuyama S,Sherman J,Dubcovsky J.多倍体小麦VRN-1启动子区的等位基因变异。Al Appl Genet。2004; 109:1677-86。
- 35.
张志强,张志强,张志强,等。VRN-1第一个内含子的大量缺失与大麦和小麦的春季生长习性有关。基因组学杂志。2005;273:54-65。
- 36.
谷子和胚芽鞘颜色基因(R和Rc)上调小麦类黄酮生物合成基因。基因组。2005;48:747-54。
- 37.
TraitGenetics GmbH是一家。http://www.traitgenetics.com/en/.2015年5月1日访问。
- 38.
Ronin Y,Minkov D,Minter D,Akhunov E.在存在基因分型误差和缺失数据的情况下构建超密集的遗传图。横滨:Proc。12实习生。小麦遗传学Symp;2013.
- 39.
建立可靠的遗传图谱:不同的作图策略可能导致不同的图谱。Nat Sci。2010;2:576 - 89。
- 40.
Voorrips RE. MapChart:用于连锁图谱和qtl图形表示的软件。J在这里。2002;93:77-8。
- 41.
SOSNowski O,Charcosset A,joets J. BioMercator V3:遗传地图编译和定量特征锁定算法升级。生物信息学。2012; 28:2082-3。
- 42。
BioMercator V3:QTL Meta-Analyzes的遗传映射编译软件。http://moulon.inra.fr/biomercator.2015年5月29日。
- 43。
黄志强,王志强,王志强,等。小麦亚科遗传分析方法研究进展。在:Feuillet C, Muehlbauer GJ,编辑。小麦科的遗传学和基因组学。我们:施普林格;2009.p . 163 - 99。
- 44。
可从以下提供的TRITITICAE TOOLBOX(T3):https://triticeAetoolbox.org/wheat/.2015年7月10日。
- 45.
Gramene:植物的比较资源(发布46)http://ensembl.gramene.org/Tools/Blast?db=core.2015年7月12日访问。
- 46.
通用蛋白质资源(UniProt)。http://www.uniprot.org/.2015年7月21日。
- 47.
辛格BD,辛格AK。映射的人口。在:标记辅助植物育种:原则和实践。新德里:施普林格印度;2015.125 - 50页。
- 48.
Boopathi NM。遗传定位和标记辅助选择。印度:施普林格;2013.
- 49.
陈志强,王志强,王志强,等。光周期对小麦春化基因VRN1和VRN2调控的影响。植物学报。2006;60:469-80。
- 50。
Li G,Yu M,Fang T,Cao S,Carver BF,Yan L.冬小麦中的vernalization要求持续时间由TA VRN-A1在蛋白质水平控制。工厂J. 2013; 76:742-53。
- 51。
Maccaferri M,Ricci A,Salvi S,Milner SG,Noli E,Martelli Pl等。一种高密度,基于SNP的四倍体小麦的共识图作为整合杜伦姆和面包小麦基因组学和育种的桥梁。植物Biotechnol J. 2014; 13:648-63。
- 52.
Sourdille P,Singh S,Cadalen T,Brown-Guedira GL,Gay G,Qi L等人。基于微卫星的删除箱系统,用于建立小麦遗传物理地图关系(Triticum Aestivum L.)。Funct综合基因组学。2004; 4:12-25。
- 53.
Timonova Em,Dobrovol'skaya ob,Sergeeva em,Bildanova Ll,Sourdille P,Feuillet C等人。小麦染色体5B使用血栓术线的比较遗传和细胞遗传学映射。Russ J Genet。2013; 49:1200-6。
- 54。
Zanke C,Ling J,Plieske J,Kollers S,Ebmeyer E,Korzun v等。欧洲冬小麦QTL主要作用QTL的遗传建筑。前植物SCI。2014; 5:1-12。
- 55。
Milec Z,ValárikM,BartošJ,SafářJ.可以成为早起的鸟儿吗?开花时间调整的工具。Biotechnol adv。2014; 32:200-14。
- 56。
Marza F,Bai GH,Carver BF,周WC。小麦群体产量和相关性状的定量特质基因座Ning7840 x Clark。Al Appl Genet。2006; 112:688-98。
- 57.
legouis J, Bordes J, Ravel C, Heumez E, Faure S, Praud S等。通过全基因组关联分析确定小麦早熟性的染色体区域。Theor Appl Genet. 2012; 124:597-611。
- 58.
陈志强,陈志强。小麦狭感早熟性的遗传分析与筛选。Sci繁殖。1999;49:233-8。
- 59.
等。WRKY转录因子:脱落酸信号转导的关键成分。生物技术学报。2012;10:2-11。
- 60。
拟南芥的毛状体和种皮发育基因GLABRA2编码WRKY转录因子。植物细胞。2002;14:1359 - 75。
- 61。
王浩,郝军,陈旭,郝志,王旭,娄阳,等。WRKY89的过表达增强了水稻植株对紫外线B的耐受性和抗病能力。植物学报。2007;65:799-815。
- 62。
罗X,太阳x,刘b,zhu d,白x,cai h等。从甘氨酸Soja从拟南芥中开花时间改变髋骨同源物的异位表达。Plos一个。2013; 8:E73295。
- 63。
罗X,xi b,太阳x,zhu d,liu b,ji w等。野生大豆包装中的表达在拟南芥中增强了旱粮。J Exp Bot。2013; 64:2155-69。
- 64.
yu y,胡r,王h,cao,he g,fu c等。MLWRKY12,一种新型的MISCanthus转录因子,参与Pith二次细胞壁形成并促进开花。植物SCI。2013; 212:1-9。
- 65.
朱晓霞,刘树华,夏国强,秦磊,孔林,夏国强。小麦WRKY转录因子及其在生物和非生物胁迫下的诱导。植物学报。2013;31:1053-67。
- 66。
李W,林Z,张X.分子标记检测到的亚洲棉花(Gossypium Arboretum L.)内植物中的一种新的分离畸变。j遗传学基因组学。2007; 34:634-40。
- 67。
Talanova vv,Titov Af,Topchieva Lv,Malysheva Ie,Venzhik YV,Frolova Sa。冷硬化和ABA治疗期间小麦植物中毛包转录因子和应激蛋白基因的表达。Russ J植物理体。2009; 56:702-8。
- 68。
徐强,冯文杰,彭海瑞,倪志峰,孙庆新。TaWRKY71是一种小麦wrky转录因子,可以增强转基因拟南芥对非生物胁迫的耐受性。谷物Res Commun. 2014; 42:47-57。
- 69.
王锐,吴华林,张敏,钟福宁,孙启新。小麦(Triticum aestivum L.)的克隆、鉴定及转基因功能分析TaWRKY51。农业生物技术。2013;21:1019-27。
- 70.
细胞对脱水和冷胁迫的反应和耐受性的转录调控网络。植物学报2006;57:781-803。
- 71.
壮吉,彭鲁,程泽,张吉,蔡伯,张Z等。肺炎血管血管血管推定AP2 / ERF家族基因的基因组分析。SCI Hortic。2009; 123:73-81。
- 72。
爱尔兰VF,苏塞克斯。Apetala-1基因在拟南芥中的作用。植物细胞。1990; 2:741-53。
- 73。
Kunst L, Klenz JE, Martinez-Zapater J, hohn GW。AP2基因决定拟南芥花被器官的特性植物细胞。1989;1:1195 - 208。
- 74。
张志强,张志强,张志强,等。拟南芥同源基因AGAMOUS的负调控。细胞。1991;65:991 - 1002。
- 75。
Mandel MA, Bowman JL, Kempin SA, Yanofsky MF。转基因烟草花结构的调控。细胞。1992;71:133-43。
- 76.
王志强,王志强,王志强。aptala2 -like基因家族的系统发育和结构域进化。生物学报2006;23:107-20。
- 77.
Li G,Siddiqui H,Teng Y,Lin R,WAN X,Li J等。拟南芥昼夜昼夜时钟下的协调转录规范。NAT细胞BIOL [Internet]。2011; 13:616-22。
- 78。
王志强,王志强。拟南芥光冠phya突变体的光响应研究。植物杂志。1994;105:141-9。
- 79。
黄志强,王志强,王志强,等。拟南芥光敏色素A、光敏色素B和隐花色素1的遗传相互作用。植物杂志。1998;118:27-35。
- 80。
Limin AE,Fowler DB。小麦(Triticum Aestivum L)的低温耐受性和遗传潜力:对光周期,春化和植物发育的反应。Planta。2006; 224:360-6。
- 81。
欧阳X,李继,李克,李b,陈b,沉h等。远红细长缺口3的基因组宽结合位点分析显示其在拟南芥发育中的新功能。植物细胞。2011; 23:2514-35。
- 82.
关键词:水稻,光周期响应,硅启动子,DNA芯片中国科学(d辑:地球科学)2009;43:164-77。
- 83.
Khlestkina Ek,Tereshchenko Oy,Salina EA。花青素生物合成基因的位置与麦克酸杂交种中的表达。Mol Genet基因组学。2009; 282:475-85。
- 84。
小麦耐铝外源基因表达的调控。基因组。1990;33:9-12。
- 85。
Taketa S,Takeda K.小麦大麦杂交种大麦主导标志基因的表达。基因类型系统。1997年; 72:101-6。
确认
我们感谢A. Korol(Haifa大学进化学院)的教授进行多点和多点培训,使用地图构造和QTL分析。我们也感谢B. S. Gill教授(堪萨斯州立大学,美国)和J. D. Faris(USDA-Ars,Fargo,USA),用于提供CS X CS-5BDIC的F1种子。我们感谢SB RAS Genomics核心设施。该研究得到了俄罗斯科学基金会(项目号14-14-00161)的支持。
声明
本文的出版是由俄罗斯科学基金会资助的(项目号14-14-00161)资助。本文已作为BMC植物生物体积的一部分发布,2015年第1卷:来自Plantgen 2015年会议的所选文章:植物生物学。补充的完整内容可在线提供http://www.biomedcentral.com/bmcplantbiol/补充剂/ 16 / s1。
作者信息
隶属关系
通讯作者
额外的信息
利益争夺
提交人声明他们没有竞争利益。
作者的贡献
AK对小麦15k SNP序列结果进行分析,参与结果解释。AK在ZF辅助下进行了定位和QTL分析。AS提供了分析VRN-1序列和表达,参与结果解释。IL是RICL的发病人群。IL和AK提供了该群体的表型。ES设计并组织了这项工作。AK, AS和ES起草了手稿。所有作者阅读并批准了最终的手稿。
附加文件
附加文件1:表S1。
CS x CS- 5bdic 5B染色体遗传连锁图谱地图是用MultiPoint“UltraDense”软件绘制的。(XLSX 26 kb)
附加文件2:表S2。
由于N5BT5D / DT5BL分析当前调查的标记的定位,并根据与先前报告的5B映射数据的比较。(XLSX 37 KB)
附加文件3:表S3。
标题日期特征间隔分析的价值。(XLSX 10 KB)
附加文件4:表S4。
位于QTL区域的SNP序列BLASTN分析结果。(XLSX 51 KB)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
Kiseleva, a.a., Shcherban, a.b., Leonova, I.N.等等。小麦5B染色体新抽穗期决定因素的鉴定。BMC植物杂志16日,8(2016)。https://doi.org/10.1186/s12870-015-0688-x
关键词
- 标题日期
- VRN-1
- 5B染色体
- 遗传映射
- SNP.
- QTL
- 四倍体
- 小麦属植物