抽象的
背景
鹰嘴豆(中投arietinumL.)是南亚和撒哈拉以南非洲资源贫困农民培养的第二大谷物豆类。为了利用鸡眼的未开发的遗传潜力改进,我们重新测序35个鸡族基因型,代表了16种测绘群体的父母线,用于对非生物(干旱,热,盐度),生物应力(镰刀菌素愿意,ascochyta.枯萎,葡萄孢属灰色模具,Helicoverpa armigera)使用全基因组重新测序方法的营养重要(蛋白质含量)性状。
结果
鹰嘴豆35个基因型共产生192.19 Gb数据,共97313万个reads,平均每行测序深度~10 X。每个基因型与鹰嘴豆参考基因组的平均比对率为92.18%,覆盖率为82.17%。共有2058566个独特的单核苷酸多态性(SNPs)和292588个Indels与参考鸡肝基因组相比,检测到。在Ca4假型中鉴定出最多数量的SNP。此外,拷贝数变异(CNV)诸如基因缺失和重复的蛋白质基因型鉴定,其在PI 489777(1个基因缺失)中最小,最大在JG 74(1,497)中。共有164,856条线特定变化(144,888个SNP和19,968Indels)在ICC 1496(21%)中的编码区中鉴定出最高百分比,然后ICCV 97105(12%)。在539个杂项变化中,339,138和62是染色体间变异(CTX),分别染色体内变化(ITX)和逆转(INV)。
结论
基因组SNP,Indels在不同作图群体中发现的cnv、PAVs和杂项变异是遗传研究的宝贵资源,有助于定位负责经济重要性状的基因/基因组片段。此外,在本研究中发现的全基因组变异可以用于开发用于遗传和育种的高密度SNP阵列。
背景
鹰嘴豆(中投arietinumL.)是最重要的谷物豆类,主要是世界干旱和半干旱地区的残余土壤水分。它是一种自我授粉的作物,交叉授粉是一个罕见的事件(0-1%)[1].鹰嘴豆起源于土耳其东南部,驯化后从一种相近的野生物种进化而来c . reticulatumLadizinsky,在中东,这一作物进一步进一步进一步进一步,印度和埃塞俄比亚[2那3.].它是素食饮食中蛋白质的丰富来源,尤其是在印度。在全球范围内,它的种植面积超过135mha,年产量1310万吨[4.],产量不足1吨/公顷,远低于最佳生长条件下估计的6吨/公顷的潜力。在印度,它的产量为960兆瓦,产量为880万吨,平均产量为920公斤/公顷。印度占全球鹰嘴豆产量的67%,占全球总产量的71%。尽管印度是最大的鹰嘴豆生产国,但印度从澳大利亚、土耳其、墨西哥、美国、加拿大等国家进口鹰嘴豆。
几种生物和非生物胁迫已经影响了鹰嘴豆生产率。然而,由于培养基因库的低遗传多样性,增加生产力的努力无法产生大量成功[5.].这种有限的遗传多样性在培养基池中影响鸡眼的遗传和基因组研究,因为与其他作物相比,父母之间的父母之间的多态性相对非常低。例如,在筛选出在特定于内的鸡斑群体上的超过两千个标记(ICC 4958×ICC 1882和ICC 283×ICC 8261)中,只能为这些群体识别代表〜10%的总多态标记的百分之一。6.].在遗传多样性较低的情况下,亲本间多态标记的鉴定是费时且繁琐的工作[7.].
下一代测序(NGS)技术的最新进展显着降低了测序的成本,并且正在部署以了解基因组结构,基因组的变化,鉴定基因和非生物应激的候选基因,限制作物生产率低于生产潜力的作物生产率[8.].利用全基因组重测序(WGRS)方法对不同作物的多个种质进行了重新测序,以利用一个物种尚未开发的遗传潜力进行作物改良。例如,3000个水稻基因组[9.)、玉米(10],高粱[11]等,已被重新排序。
近年来,单核苷酸多态性(SNP)和Indels在作物改进中被广泛部署。有时,这些小变化不会捕获与特定表型变异相关的所有基因组信息。这可能是由于其他重要种类的大型基因组变化I.E.结构变异(SV)。这些SV包括逆转,易位,横向,拷贝数变异(CNV),插入和缺失,是从50个核苷酸相对于参考基因组到几兆子酶的基因组重排[12那13].在人类的情况下,这些大变化是广泛的研究,并且与重要的复杂疾病表型相关。然而,在植物的情况下,很少有研究探讨了例如在玉米中的大变化的有用性,玉米及其祖先之间研究了SVS [14],而CNVs在大麦中的功能影响和起源机制也有报道[15].
然而,几种作物植物的基因组序列草案的可用性[16包括鹰嘴豆[17,为作物改良策略开辟了新的前景。了解作图群体亲本之间基因组的广泛变异,将有助于对胁迫响应候选基因进行性状作图和鉴定。为了了解鹰嘴豆的全基因组变异,我们对16个定位群体的亲本的35个基因型进行了重新测序,并分离了鹰嘴豆的不同生物和非生物胁迫以及重要的营养性状。
结果与讨论
为了对复杂的生物和非生物胁迫进行解剖,在ICRISAT中使用了几种双重父母绘制群体和多父母先进生成间交互(Magic)人口等下一代制图群体。虽然少数简单的序列重复(SSR)标记可用于Chickpea中的特质映射,但双重父母绘图人口父母线的有限多态性一直阻碍了达到负责兴趣特征的候选基因的特征映射努力[7.].然而,SNPs、CNVs和PAVs等全基因组变异在性状定位和作物改良中非常重要,近年来越来越重要。
为了深入了解可以用于性状分析和鹰嘴豆改良的全基因组变异,本研究对35个不同来源(印度、墨西哥、土耳其、坦桑尼亚、独联体和俄罗斯)和代表两个市场阶层(desi和kabuli)的鹰嘴豆基因型进行了重新测序。这35个鹰嘴豆基因型是16个定位群体的亲本,分别在非生物(干旱、热、盐)、生物胁迫(镰刀菌素愿意,ascochyta.枯萎,葡萄孢属灰色模具,Helicoverpa.POD BORER)和营养重要的(蛋白质含量)特征;魔术人口的父母和标记辅助复发间谍(MARS)群体的父母线(附加档案1).
在Silico.序列数据的映射
共有192.19 GB,在每条线的平均排序深度为10.32倍的平均排序深度为35种Chickpea的35种基因型(附加文件)2).经过数据的裁剪和处理,共产生91122万次高质量读取。使用Bowtie 2将干净数据与CDC Frontier参考基因组进行比对,reads的定位率从90.19% (IG 72953)到95.3% (JG 62)不等。不同基因型间作图率的差异可能是由于本研究所用亲本基因型的差异造成的。平均每个基因型与参考基因组的比对率为92.18%,平均覆盖率为82.17%。从每个基因型到参考基因组的reads数量从12,765,493 (ICC 1496)到87,487,094 (JAKI 9218)不等,而唯一对齐的reads从7,778,952 (ICC 1496)到40,072,407 (JAKI 9218)不等,因此平均53.92%的高质量reads与基因组唯一对齐。平均深度为5.79 ~ 20.04,平均值为~8.6。在ICC 4958和JAKI 9218中观察到更高的平均深度为14.26和20.04,因为这些示例生成的读取量相对较高(附加文件2).
snp及其分布
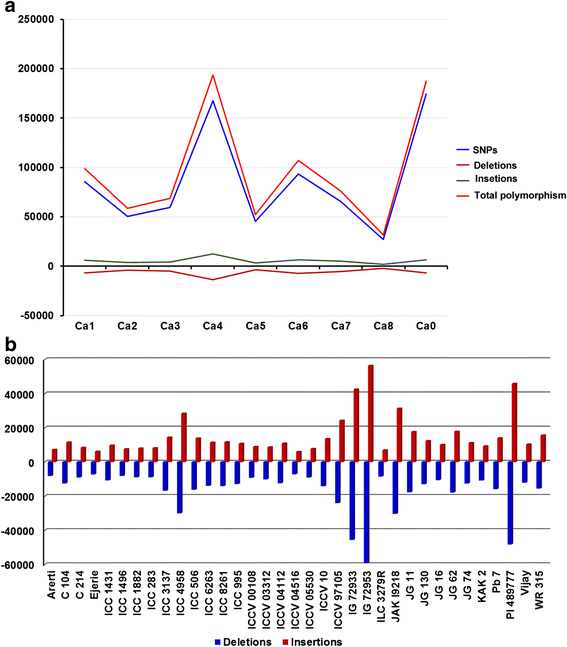
为了确定35只鹰嘴豆基因型之间的序列多样性,清洁读数与鹰嘴豆的参考基因组组装对齐。结果,在重新测序的所有35个基因型中,共鉴定了2,058,566个SNPs(附加文件3.).在本研究之前,通过454个转录组测序鉴定了51,632个SNPs中投arietinum和Cicer网状物基因型[18].此外,还使用索尔岛/ illumina测序,临时正交基因(Togs)的扩增子测序,表达序列标签(EST)的开采和候选基因的测序来报告几百个SNP。和候选基因的测序[19-21].WGRS方法也已经部署在几种作物中,例如大豆[22], 白饭 [23], 胡椒 [24)、玉米(25]和番茄[26].在8个伪分子(Ca1 ~ Ca8)上的SNPs中,Ca4上的SNPs最多(377,491),Ca8上的SNPs最少(79,770),分别占了18.34和3.88%(图2)。1;额外的文件3.).在未定向的支架和Contigs(CA0)上鉴定了361,177个SNP,占确定的17.55%。伪模节之间的SNP密度变化;CA4具有最高密度(每kB每kB的7.67 snps),Ca0具有最低密度(每kB的1.954 snps)(附加文件3.).在伪模粒中,发现CA4具有最大多态性率(8.92 / kB),而CA7具有最低多态性率(3.95 / kB)。本研究进一步重新确认了早些时候报告的结果[17这可能是由于Ca4伪分子中存在较大的重复区域。伪分子中外显子变异密度最小的是Ca7(0.16外显子变异/Kb),最大的是Ca4(0.36外显子变异/Kb)3.).Ca0上最小的外显子密度为0.02个/Kb。这意味着Ca4编码区域的变化最大,与Varshney等[17那31].每个基因型的SNPs数量从97,091 (ICCV 04516)到1,001,744 (IG 72953)不等1).desi基因型中ICC 4958和kabuli基因型中ICC 8261的单核苷酸多态性最多。在IG 72953和IG 72933之间的snp数量较高(1,133,522个),而在CDC-Frontier和ICCV 04516之间的snp数量最少(97,091个)4.).与之前的研究相比,该研究报告的SNPs数量更高[17那27-30.].这可能是由于目前研究中使用的不同父母线和野生基因型。基于每个基因型的称为参考基因组(附加文件,基于所谓的SNPS进一步归类为纯合和杂合子SNPS(附加文件5.和6.).在PI 489777(606,413)的情况下鉴定了最大纯合子SNP的最大数量,并且在ICCV 04516(57,432 SNP)的情况下最低限度。在35个基因型中,在IG 72933(0.84)的情况下观察到最大杂合子率,而在PI 489777(0.08)的情况下观察到至少杂合率。平均杂合子率为35种基因型为0.36(附加文件6.).
插入和删除(Indels)
从1 bp到58 bp的插入和删除被认为是Indels在本研究中。总共有292588Indels通过35个鹰嘴豆基因型进行了鉴定(附加文件3.).最大缺失次数,81,516的长度为1bp,而最小缺失数量(2)为52,53,56和57bp。最大插入数为78,678,长度为1bp长度,而最小数量为1,58bp长度为1(附加文件7.).这些Indels,缺失148,309例,插入144,279例。整个基因组的缺失和插入密度分别为0.28和0.27 / Kb(附加文件3.).更远,indel.针对参考,CDC-Frentier的每个样本的分析显示最大IndelsIG 72953(115,538)和最低限度Indels分别在ICCV 04516(13,146)中(图。1 b).当为每个基因型计算缺失比率时,最大和最小值计算indel在JAKI 9218和ILC 3279的情况下,比率分别为1.03和0.81。在JG 11,JG 62,WR 315和ICCV 97105中indel.比率为〜1(附加文件8.).
拷贝数变异(CNVs)和存在缺失变异(PAVs)
在长于1 kB的基因的情况下确定CNV和PAV。基因本体学分析是使用瑞士 - Prot和Trembl数据库完成的(http://www.ebi.ac.uk/uniprot).在不同的基因型中发现了一组非冗余的9732个重复基因,对9628个基因进行了Uniprot id检索和分配。其中,有4374个基因在其中一个样本中重复,使它们具有特定的重复基因。Ca_27299基因在最多23个样本中被发现重复(附加文件9.).CA_27299具有GO IDS GO:0016021;GO:0005886被发现是存在于用于细胞膜的受体样蛋白12(ATRLP12)。复制基因在不同基因型中的4至1135之间。在JG 74中,发现最多1,135个基因复制,而在JG 62中重复最小基因(4)(附加文件)9.).在耐盐品系ICC 1431中复制了最多的防御相关基因(27个)。
同样,在任何基因型中都没有发现205个非冗余基因。可以为198个基因分配Uniprot id。其中,134个基因在任何基因型中都不存在,这表明系特异性基因缺失(附加文件)10).然而,在八个基因型中不存在基因CA_17015,然而,它是一种不表达蛋白质。CA_13947在7种基因型中并不存在,其推定功能是含有PPR家族的五氢肽重复的蛋白质,PCMP-E亚家族。展示的展示结果表明,ICCV 03312,IG 72953和PI 489777中没有缺失基因(附加文件10).发现最多32个基因在JG 62中删除,然后在ICCV 00108中进行30个基因。
各种各样的变化
除了上述变化之外,还努力识别像染色体间变化(CTX),染色体染色体变异(ITX)和逆转(INV)等杂种变异。539个杂项变异,339,138和62分别为CTX,ITX和INV。为了进一步避免误报,我们使用了严格的截止值99.因此,结果在八个假调子上鉴定了110个杂种变化(表2).CTX在273bp至667bp的范围内,通过Ca 3(22),Ca6(16),Ca4(10),Ca1(7),Ca 5(7)和Ca7(3)。ITX在86bp至3.81mbp的范围内,通过Ca2(11),Ca 4(4),Ca 5(4),Ca7(4),Ca8(3)和Ca 3(2)。虽然INV主要在CA6(8)上的30bp至4.76Mbp的范围内,其次是Ca4(2),Ca7(2)和Ca2(1)。
行具体的变化
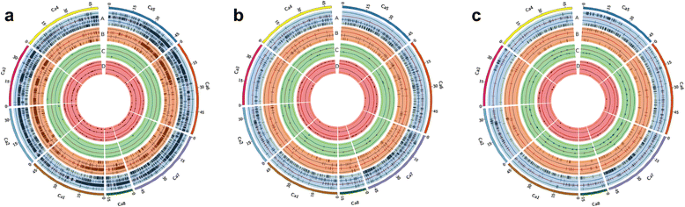
共有164,856个独特的线路特定变体,包括144,888个SNP和19,968Indels在学习的35只鸡族基因型中观察到。在PI 489777中观察到最大线条特定变化,78,320(68,799 SNP和9,521Indels),然后在IG 72953中进行62,808(55,393个SNP和7,415Indels)(图。2;额外的文件11).我们进一步比较了在非生物胁迫(如干旱、盐度)和生物胁迫(如干旱、盐度)下分离的亲本基因型之间的系特异性变异镰刀菌素愿意,ascochyta.枯萎,葡萄孢属灰霉病)。虽然更大的线路特定SNP和Indels被确定为案件Helicoverpa.抗性野生物种基因型Ig 72953,鉴定了没有物种的特异性缺失和基因的重复(图。2).相反,在映射群体的父母基因型的情况下Helicoverpa.电阻(ICC 506×ICC 3137)特定线路的数量和Indels显著不同。同样地,大量的系谱特异变异在亲本间的作图居群中进行分离ascochyta.枯萎(图。2 b),镰刀菌素枯萎(图。2摄氏度),葡萄孢属灰色模具和盐度(附加文件12和13),可用于高密度遗传图谱的构建、性状的定位和标记辅助选择。在35个鹰嘴豆基因型中,ICC 1496、ICCV 00108、Pb 7、JG 130、ICC 4958、JAKI 9218和c214的特异变异均小于100。在35个基因型中,有趣的是在c214中没有观察到系特异性变异。其余26个基因型的株系特异变异数在100 ~ 5000个之间13).在ICC 1496(21%)中的编码区中发现的线路特定变化的最大百分比(21%),后跟ICCV 97105(12%)(附加文件14).找到了编码区域的线路特定变化的平均值6.4%,而在ICC 4958和Jaki 9218的情况下,观察到编码区域的具体变化。当帧移位时,开始丢失,停止获得并停止总结失去的突变,并在编码区的总变化中计算百分比,在Pb 7基因型中观察到最大33.33%,而8个基因型(ICCV 04112,ICCV 04516,C 104,ICCV 00108,JG 11,JG 130,ICCV 10和ICC 1431)(附加文件15).在魔法人群的父母中,线特异性SNP,线特异性缺失和线特异性插入在JG 11的情况下高。此外,基因缺失范围为8(JAKI 9218)至30(ICCV 00108),而基因重复范围从17(JG 130)至1,120(Jaki 9218)(表3.).在魔法人群的父母线的情况下,在基因组水平中,整体大变异是明显的。发展魔法人口的主要目的是创造和利用遗传多样性以进行作物改善。总之,从这些线路开发的魔法线将具有巨大的变化,可用于等位基因挖掘和基因发现。使用Uniprot储存库进一步注释了线特异性变体。注释揭示了这些线特异性变体对许多转录因子及其调节因子的影响,如锌指蛋白,BHLH,WRKY,F-BOX,BZIP,PHD,尖叫和MADS箱等以及TFS,疾病抵抗NB-LRR蛋白,热休克蛋白,DNA损伤修复蛋白,染色信号通路相关蛋白也受到影响(附加文件16).
在牙型基因型中鉴定的基因组范围内抗性或易受重要的生物应激。Circos图表示线路特定SNP,Indels,基因缺失和重复。每个CIRCOS代表八个鹰嘴豆肽,由四个同心环组成,其中表示SNPS,B表示Indels,C表示删除和D表示复制事件。删除事件标有绿环中的三角形,圆圈表示红色的重复。一种差异的分布Helicoverpa.抗性和易感基因型。圆环内的绿色、红色、蓝色和黄色分别代表Vijay、IG 72953、ICC 506和ICC 3137。B.差异的分布镰刀菌素枯萎病抗性和敏感基因型。圆环内部的绿色、红色、蓝色和黄色分别代表c104、jg62、WR 315和ICCV 05530。C差异的分布ascochyta.抗抗性和易感基因型。内部的绿色,红色,蓝色和黄色,分别代表ICCV 04516,JG 62,PB 7和ICCV 05530
注释基因组的变化
通常,过早停止,框架换档和存在/不存在变化通过禁用可能导致近苯累的抑制而导致遗传负载。因此,我们注释了基因组的变化。在2,351,154个变异中,38,078个是无意义的,352个是无意义的,50,290个是静音突变(附加文件17).然而,大变异(1,924,406)在非基因区域中。在基因区域中检测到的419,262个变化,328,171是内血清变化,91,091是偏僻的变化(附加文件18).在这些外显子变异中,有37,875个是非同义替换。snp在基因组区域的分布情况显示,snp在基因间区域最为丰富(81.85%),内含子中snp的比例(13.96%)高于外显子中snp的比例(3.87%)。利用遗传变异注释和效应预测工具对所有35个鹰嘴豆基因型的snp、纯合子SNPs和杂合子SNPs的效应进行预测(附加文件)18那19那和20).在不同基因型中,导致停止获得或无意义突变的SNPs数量从18个(Ejerie)到119个(IG 72953)。的Indels研究了对每个基因型的基因组的影响。影响基因区域的最大插入在IG 72953(21.54%)中观察,而ICCV 00108(12.23%)中最少(附加文件)21).类似地,在IG 72953(21.16%)中观察到遗传区域中发生的最大缺失,在Jaki 9218中最低为12.90%(附加档案22).
我们确定了373个变化“QTL-Hotspot.“CA4的地区,报告前提高了鹰嘴豆的耐旱性[6.那31](附加文件20.).在这些变型中,特别是在CA_04570中发现了两个密码子插入(存在)qtl-hotspot-b),属7S种子贮藏基因家族,据报道可增强种子大小[32].此外,我们还确定了38个变异,其是影响总共17个基因的非同义编码,其功能如热应激转录因子A-6b,EPF样蛋白4和早期光诱导的蛋白质(氯塑料elip)(附加文件23).
结论
本研究中鉴定的基因组宽变化可用于显影用于遗传和育种应用的高密度SNP阵列。此外,野生种类中的大量线特异性变异表明野生鹰嘴豆比栽培的鹰嘴豆更多样化,因此可能含有鸡眼改善的有用遗传资源。
方法
植物材料
在额外文件中介绍了三十五个鹰嘴豆基因型,用于研究,他们的血统,起源,市场类和突出特征1。
DNA隔离
基因组DNA从所有35个鹰嘴豆基因型中分离出来自10天的10天的古代幼苗,如前所述[33].检查0.8%琼脂糖凝胶上的DNA质量。Qubit®2.0荧光计(Life Technologies,Thermo Fisher Scientific Inc. USA)用于量化DNA。
图书馆施工和测序
根据制造商的协议,使用TruSeq DNA样品准备试剂盒LT, (set a) FC-121-2001 (Illumina, San Diego, CA, USA)从每个样品中大约使用1 μg DNA构建MiSeq测序库。简单地说,柱纯化的基因组DNA使用Bioruptor®NGS (Diagenode,比利时)剪切。DNA片段大小在1.2%琼脂糖凝胶上测定,范围在200-1000 bp。剪切后,进行末端修复,将悬垂转化为钝端,然后是腺苷酸3 '端。随后,将有索引的适配器连接到DNA片段的末端,使它们准备在流动细胞上杂交。使用l E-Gel®Size™2%琼脂糖预制凝胶(Invitrogen)进行尺寸选择,获得约400 bp的目标插入块并进行纯化。进行pcr(9个周期),以富集大小选定的DNA片段两端都有Illumina适配器。利用安捷伦高灵敏度DNA芯片,在安捷伦2100生物分析仪上检测扩增DNA库的大小分布。
变性和稀释文库在Illumina MiSeq台式测序仪(Illumina, San Diego, CA, USA)上使用MiSeq Reagent Kit v2(300个循环)进行测序,生成150个碱基对端序列。数据在MiSeq仪器上自动解复用,生成样本压缩的FASTQ文件。
数据过滤和对齐
使用Sickle版本1.200清洁和修剪每条线的原始数据(https://github.com/najoshi/sickle.).清洁的数据对准参考鸡肝基因组[17]使用Bowtie 2 [34].比对数据被进一步过滤,以保留只映射到基因组一个区域的reads。最小映射质量为30的reads被用于进一步分析。基因组分析工具包(GATK, v 3.1-1)的基础质量评分重新校准(BQSR)和InDel重组组件[35],来自Picard的多家公用事业(V1.102)(http://broadinstitute.github.io/picard/)用于bam文件的后处理。
全基因组变异的鉴定
在上面提到的Stringent条件后生成的对齐文件用于使用GATK程序进行变体发现。如果由最小读取深度的PHRED质量得分> 30滤出具有少于5bp侧翼距离的最小读取深度的PHRED质量得分> 30,则将一个位置作为基因型的变体。通过在沿着每种假调子的窗口尺寸为100kb的窗口尺寸的频率下评估DNA多态性的分布。用于识别同义词和非同义SNP的效果Indels, SnpEff程序[36使用了)。内部Perl脚本用于分析变异(SNPs和Indels)在基因组中的分布。只有当变异仅存在于一个基因型,而参考等位基因存在于其他基因型时,才会报道系特异性变异。进一步研究了品系特异性变异对编码序列的影响,并将这些变异与UniProtKB中显示UniProt id匹配的基因信息分配到GO id中。Circos图被用来绘制线的具体变化[37].
利用CNVnator找到e-value截断值为1e-05的CNVs,并以基因长度≥1,000 bp的基因进行注释[38].通过排除通过将读取的CNV映射到CDC Frentier来源的CNV来消除误报。基于测序深度测定蜡,如<10%被认为是不存在变化,> 50%被认为是存在变化)。为了确定ITX,CTX和INV等杂种变化,每个样品的配对末端读取与参考基因组(CDC边界)与Bowtie 2对齐,具有不和谐的标志和端到端映射模式。Picard(v1.102)(http://broadinstitute.github.io/picard/)用于在对齐文件上设置读组信息,并使用samtools(v0.1.19 +)按坐标位置进行排序[39].breakdancer(v1.1.2)[40]用于检测各种变异。显示对照的读数存在的杂项变化被忽略,因为它们被认为是假阳性。杂项变异由等于99分的分数过滤,从而选择一组高度自信的杂项变异。在R (v3.1)中按类型总结了杂项变异的统计。霹雳舞只提供断点坐标,检测到的各种变化驻留在其中。因此,为了找到各种变异的确切核苷酸坐标,我们在比对文件中查找支持每种变异的读取。这是使用c++内部工具完成的。
提供支持数据的可用性
支持本文结果的数据集可用http://ceg.icrisat.org/publicdomain.html.。
缩写
- WGRS:
-
全基因组重新排序
- 德尔:
-
删除
- ins:
-
插入
- CTX:
-
Inter-chromosomal易位
- ITX:
-
Intra-chromosomal易位
- CNV:
-
复制编号变体
参考文献
- 1.
辛格kb。鹰嘴豆育种。在:Saxena MC,Singh KB,编辑。鹰嘴豆。瓦莱德福:CAB INTERINATIONS;1987. p。127-62。
- 2.
Ladizinsky G,Adler A.鹰嘴豆的起源中投arietinuml . Euphytica。1976;25:211-7。
- 3.
范德梅森LJG。加工L.鹰嘴豆的起源,历史和分类。在:Saxena MC,Singh KB,编辑。鹰嘴豆。Aberystwyth:Cab International;1987. p。11-34。
- 4.
FAOSTAT (2013)http://faostat.fao.org/site/339/default.aspx。于2015年7月15日访问。
- 5.
Roorkiwal M,Nayak Sn,Thudi M,Upadhyaya HD,Brunel D,Mournet P等人。基于基于基于基因的SNP标记的鹰嘴豆参考组中非生物应激响应候选基因等位基因。前植物SCI。2014; 5:248。
- 6。
Varshney RK, Thudi M, Nayak SN, Gaur PM, Kashiwagi J, Krishnamurthy L,等。鹰嘴豆抗旱性的遗传分析(中投arietinuml .)。Theor Appl Genet. 2014; 127:445-62。
- 7。
Thudi M, Gaur PM, Krishnamurthy L, Mir RR, Kudapa H, Fikre A,等。基因组学辅助育种抗旱:鹰嘴豆梦想成真!acta botanica botanica(云南植物研究). 2014;41:1178-90。
- 8。
收获基因组学有希望的成果:将基因组测序技术应用于作物育种。公共科学图书馆杂志。2014;12 (6):e1001883。
- 9。
3K RGP。3,000个水稻基因组项目。吉希2014; 3:7。
- 10。
焦勇,赵洪,任磊,宋伟,曾波,郭建军,等。玉米现代育种过程中的全基因组遗传变化。Nat麝猫。2012;44:812-5。
- 11.
Mace ES,Tai S,Gilding Ek,Li Y,Prentis PJ,Bian L等。全基因组测序揭示了非洲土着谷物作物高粱的未开发遗传潜力。NAT Communce。2013; 4:DOI:10.1038 / ncomms3320。
- 12.
Saxena RK,Edwards D,Varshney RK。植物基因组的结构变异。简短的Funct基因组学。2014;DOI:10.1093 / bfgp / elu016。
- 13.
王W,王某,侯c,xing y,cao j,wu k等。通过阵列CGH的各种马品种拷贝数变异的基因组检测。Plos一个。2014; 9(1):E86860。
- 14.
Broohammer A.在当代玉米种质的一组祖细胞中与非生物/生物应力相关的基因拷贝数变异。https://www.ideals.illinois.edu/handle/2142/498032014年。
- 15.
Muñoz-Amatriaín M, Eichten SR, Wicker T, Richmond TA, Mascher M, Steuernagel B,等。大麦基因组拷贝数变异的分布、功能影响及起源机制。基因组医学杂志。2013;14:R58。
- 16。
迈克尔TP,杰克逊S.前50种植物基因组。植物基因组。2013; 6(2)。
- 17。
Varshney RK,Song C,Saxena RK,Azam S,Yu S,Sharpe Ag,等。鸡眼的基因组序列草案(中投arietinum)为特质改进提供资源。NAT BIOTECHNOL。2013; 31:240-6。
- 18。
Deokar AA, Ramsay L, Sharpe AG, Diapari M, Sindhu A, Bett K,等。鹰嘴豆全基因组SNP鉴定用于鹰嘴豆高密度遗传图谱的开发和鹰嘴豆参考基因组组装的改进。BMC基因组学。2014;15:708。
- 19。
Hiremath PJ,农民A,Cannon SB,Woodward J,Kudapa H,Tuteja R等。鹰嘴豆大规模转录组分析(中投arietinum一种生长在亚洲和非洲半干旱热带地区的孤儿豆类作物。生物技术学报2011;9:922-31。
- 20。
Gujaria N, Kumar A, Dauthal P, Dubey A, Hiremath P, Bhanu Prakash A, et al.;鹰嘴豆基因分子标记(GMMs)的开发与应用中投arietinuml .)。Theor app Genet. 2011; 122:1577-89。
- 21。
Varshney RK,Hiremath PJ,Lekha Pt,Kashiwagi J,Balaji J,Deokar Aa,等。对鹰嘴豆基因发现和标记发育的促进和盐度响应EST的综合资源(中投arietinuml .)。BMC基因组学。2009; 10:523。
- 22。
周Z,江y,王z,gou z,lyu j,li w等人。重新排列302野生和栽培的探索鉴定与大豆的驯化和改进有关的基因。NAT。Biotechnol。2014;33:DOI:10.1038 / NBT.3096.。
- 23。
徐X,刘X,GE S,Jensen JD,Hu F,Li X等。重新排列50种培养和野生水稻的含量产生用于识别农艺上重要基因的标志物。NAT BIOTECHNOL。2012; 30:105-11。
- 24.
陈晓东,陈宝林,陈建民,等。对栽培辣椒和野生辣椒的全基因组测序提供了深入了解辣椒驯化和专业化。中国科学院院刊。2014;111:5135-40。
- 25.
徐茹,元y,徐y,张g,guo x,wu f等。玉米全基因组重构耐旱性候选基因的鉴定。BMC植物BIOL。2014; 14:83。
- 26.
100番茄基因组测序联盟,Aflitos S, Schijlen E, de Jong H, de Ridder D, Smit S,等。探索番茄的遗传变异(Solanum.部分Lycopersicon.通过全基因组测序疏水。工厂J. 2014; 80:136-48。
- 27.
Doddamani D,Khan AW,Katta Mavsk,Agarwal G,Thudi M,Ruperao P等人。Cicarvardb:SNP和Indel数据库,用于推进鹰嘴豆植物的遗传研究和育种应用。数据库。2015; 1-7。DOI:10.1093 /数据库/ bav078。
- 28。
Kujur A,Bajaj D,Upadhyaya HD,Das S,Ranjan R,Shree T,等。采用基因组的SNP发现和基因分型策略,以推断鹰嘴豆中的自然等位基因多样性和驯化模式。前植物SCI。2015; 6:162。
- 29。
Hiremath PJ,Kumar A,Penmetsa RV,Farmer A,Schlueter Ja,Chamarthi Sk,等。大规模开发成本有效的SNP标记测定,用于鸡肝的多样性评估和遗传映射和豆类的比较映射。植物Biotechnol J. 2012; 10:716-32。
- 30.
Gaur R,Azam S,Jeena G,Khan AW,Choudhary S,Jain M,Yadav G,Tyagi Ak,Chattopadhyay D,S Bhatia S.高通量SNP发现和基因分型构建鸡豆饱和的联系地图(中投arietinuml .)。DNA Res。2012; 19:357-373。
- 31。
Varshney Rk,Gaur Pm,Chamarthi Sk,Krishnamurthy L,Tripathi S,Kashiwagi J等。快速轨道迟钝“QTL-Hotspot.“对于JG 11中的根特征和其他干旱耐受性状,精英和领先的鹰嘴豆。植物基因组。2013; 6(3)。DOI:10.3835 / Plantgen.Ome2013.07.0022。
- 32。
陈春梅,陈春梅,陈春梅,等。候选基因的优先排序QTL-Hotspot.“鹰嘴豆植物耐受区(中投arietinuml .)。SCI REP 2015; 5:15296。
- 33。
CUC LM,MACE E,Crouch J,Quang VD,Long Td,Varshney RK。新型微卫星标志物的分离与表征及其在耕地中分化评估的应用(arachis hypogaea.).BMC Plant Biol. 2008;8:55。
- 34。
Langmead B,Salzberg SL。与Bowtie 2. NAT方法的快速喷射读取对齐。2012; 9(4):357-9。http://doi.org/10.1038 / nmeth.1923。
- 35。
McKenna A,Hanna M,Banks E,Sivachenko A,Cibulskis K,Kernytsky A等人。基因组分析工具包:MapReduce框架,用于分析下一代DNA测序数据。Genome Res。2010; 20(9):1297-303。
- 36.
Cingolani P,Platts A,Wang Le L,Coon M,Nguyen T,Wang L等。用于注释和预测单核苷酸多态性,SNPEFF:SNP在基因组中的疗程的程序黑腹果蝇应变w1118;iso-2;iso-3。飞(奥斯丁)。2012; 6 2:80 - 92。
- 37.
Krzywinski M,Schein J,Birol I,Connors J,Gascoyne R,Horsman D等人。电讯:对比较基因组学的信息审美。Genome Res。2009; 19:1639-45。
- 38.
李洪波,李洪波,李洪波,等。序列比对/映射(SAM)格式和SAMtools。生物信息学。2009;25:2078-9。
- 39.
CNVnator:一种从家族和群体基因组测序中发现典型和非典型cnv、基因型和特征的方法。基因组研究》2011;21:974 - 84。
- 40.
陈凯-基于对端阅读图谱的基因组结构变异鉴定。生物信息学。2014。DOI:10.1002 / 0471250953.bi1506s45。
确认
这项工作是作为CGIAR研究方案的一部分,是印度ICRISAT粮食植物群体的一部分。ICRISAT是CGIAR联盟的成员。
作者信息
隶属关系
相应的作者
额外的信息
利益争夺
提交人声明他们没有竞争利益。
作者的贡献
rkv-构思了这个想法;MT,VK--进行实验;AWK,AVSK,VG,MT - 分析了数据;PMG,SS-促进了研究的种质线;rkv-研究的试剂;MT,RKV,AWK,Mr-写了稿件;所有作者都阅读了MS并提供了同意。所有作者阅读并认可的终稿。
作者的信息
Rajeev K Varshney目前正作为研究计划总监 - 谷物豆类和主任 - 基因组学卓越中心(http://ceg.icrisat.org.).他在基因组学和标记辅助育种方面有广泛的专业知识。他领导了两种豆科植物(鹰嘴豆和鸽子豆)的基因组测序工作,并对花生基因组测序以及其他几种基因组做出了重大贡献。他与ICRISAT和其他合作机构的育种人员一起,开发了鹰嘴豆和花生的分子育种产品。
Mahendar Thudi-目前作为科学家(应用基因组学和基因分型服务),他正在ICRISAT的基因分型服务实验室,该实验室旨在提高育种计划中的分子工具,以提高更快的遗传收益。CEG向国家合作伙伴提供SSR,DART和SNP基因分型服务,以及全球研究。
Aamir W. Khan正在担任计算基因组学中的科学官员。
Vinay Kumar- ICRISAT的特殊项目科学家,应用基因组学。他正在ICRisat基因组学中的卓越中心照顾下一代测序设施。他在基因组学领域拥有超过5年的博士生经验,特别是NGS应用,DNA测试,分子标记基因分型和多样性分析。
Gaur是主要科学家(鹰嘴豆育种)。他有20年的鹰嘴豆改善经验。
Krishnamohan Katta是计算基因组学的特殊项目科学家。
Vanika Garg是一项在计算基因组学中追求博士学位的研究学者。
Manish Roorkiwal目前正当于科学家 - 基因组学和分子育种。他在分子遗传学和应用基因组学中具有超过10年的研究经验的基本背景。他的研究侧重于利用下一代基因组学和育种方法改善豆类的作物生产力。
Srinivasan Samineni是ICRISAT的鹰嘴豆饲养员。
宣言
我们非常感谢CGIAR研究计划的粮食植物,ICRISAT,印度,发布本条。
本文已作为BMC植物生物学第16卷补充1,2015:来自Plantgen 2015年会议的所选文章:植物生物学。补充的完整内容可在线提供http://www.biomedcentral.com/bmcplantbiol/补充/ 16 / S1。
Mahendar Thudi和Aamir W. Khan对这项工作做出了同样的贡献。
附加文件
附加文件1:
关于研究中使用的35个鹰嘴豆基因型的地理来源,市场类和血统信息的详细信息。(docx 17 kb)
附加文件2:
为35只鹰嘴豆基因型产生的数据摘要并与鹰嘴豆(CDC Frentier)的参考基因组排列。(医生70 kb)
附加文件3:
SNP的分布,Indels及其在鹰嘴豆基因组中的作用(CA1-CA8)。(DocX 14 KB)
额外的文件4:
在本研究中使用的35种鹰嘴豆基因型中鉴定的成对SNP。(XLSX 17 kb)
额外的文件5:
在研究中使用的每种鹰嘴豆基因型中鉴定的纯合和杂合子SNP。在PI 489777中的纯合SNP中,最大杂合子SNP在IG 72933中是显而易见的。(TIF 309 KB)
额外的文件6:
35例鹰嘴豆基因型中鉴定的纯合和杂合子SNP综述。(XLSX 11 KB)
额外的文件7:
的总结indel.长度及其各自的基因组计数。(XLSX 9 KB)
附加文件8:
的总结indel.35个鹰嘴豆基因型的重测序比例。(XLSX 12 KB)
附加文件9:
基因重复在35只鹰嘴豆基因型中观察到。(XLSX 2979 KB)
额外的文件10:
在本研究中使用的35种Chickpea基因型中检测到的基因缺失。(XLSX 67 KB)
附加文件11:
在鹰嘴豆基因型中鉴定的基因组变异抗性或易受影响葡萄孢属灰色模具。Circos图表示直线的特定变化。每个circos代表8个鹰嘴豆伪分子,由4个同心环组成,其中A代表snp, B代表Indels, C表示删除事件,D表示重复事件。删除事件用绿色圆圈中的三角形标记,红色圆圈表示重复。所有圆环内的绿色、红色、蓝色和黄色分别代表ICC 1496(抗)、JG 62(感)、ICCV 10(感)、ICCV 05530(抗)。(PNG 3357 kb)
额外的文件12:
鹰嘴豆耐盐或敏感基因型的全基因组变异。Circos图表示直线的特定变化。每个circos代表8个鹰嘴豆伪分子,由4个同心环组成,其中A代表snp, B代表Indels, C表示删除事件,D表示重复事件。删除事件用绿色圆圈中的三角形标记,红色圆圈表示重复。圆环内绿、红、蓝、黄分别代表ICC 1431(耐受)、jg62(耐受)、ICC 6263(易感)、jg11(耐受)。(PNG 3400 kb)
额外的文件13:
线特定变化摘要。(XLSX 11 KB)
附加文件14:
每种鹰嘴豆基因型中鉴定的编码区的线特异性变异概述。(XLSX 12 KB)
额外的文件15:
线路特定变化摘要导致停止站点。(XLSX 11 KB)
附加文件16:
基因本体论及其对35种基因型的特异性变异的影响。(XLSX 727 KB)
附加文件17:
SNPEff预测的所有变异效应的总结。(XLSX 12 KB)
额外的文件18:
使用SNPeff程序的所有SNP分类。(XLSX 10 kb)
附加文件19:
35例鹰嘴豆基因型中SNPEFF程序杂合SNP的分类。(XLSX 12 KB)
额外的文件20:
35例Chickpea基因型中使用SNPeff程序的纯合SNP分类。(XLSX 12 KB)
附加文件21:
插入效果的总结。(多克斯15 kb)
附加文件22:
删除的效果概括。(多克斯15 kb)
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
图迪,M,可汗,a.w.,库马尔,V。et al。全基因组重新测序揭示了鹰嘴豆中16种测绘群体的父母线之间的基因组变异(中投arietinuml .)。BMC植物杂志16,10(2016)。https://doi.org/10.1186/s12870-015-0690-3
关键字
- 鹰嘴豆
- 重排序
- 复制编号变体
- 映射群体