摘要
背景
MicroRNAs (miRNAs)是基因表达的重要调控元件。与编码基因类似,miRNA基因遵循一种出生和死亡的进化模式,可能反映了功能相关性和分化。例如,miRNA529在进化上与miRNA156(陆生植物中高度保守的miRNA)有亲缘关系,但它却丢失了拟南芥.有趣的是,这两个miRNAs靶序列在某些成员重叠SQUAMOSA启动子结合蛋白样(SPL)家族,提出了关于陆地植物中miR156/ mir529相关调控网络多样化的重要问题。
结果
本研究通过对几个分类学类群的miR156/529靶序列进行系统发育重建,发现尽管miRNA529缺失,但特定的eudicot SPLs保留了相应的靶位点。对miR156/miR529-靶序列的详细分子进化分析表明,在拟南芥等核心eudicots中,miR529的缺失与miRNA529特异性靶元件更宽松的选择相关,而mirna156特异性靶序列则受到更强的选择,表明这两个靶位点可能受到不同的进化约束。重要的是,在拟南芥中过表达MIR529来自单子叶的前体,而不是来自基生单子叶的前体,显示了miR529对AtSPL9而且AtSPL15其中包含miR156和miR529的保守响应元件。
结论
我们的结果表明功能的丧失MIR529研究结果表明,mir529响应元件存在于部分白绒科植物中:仍然有效。我们的数据支持这样的观点,即特定的miRNA156家族成员可能弥补了miR529调控的缺失,这同时也可能有利于eudiicot物种的多样化:.
背景
MicroRNAs (miRNAs)是小rna,对动物、植物和病毒的转录和转录后调控很重要。MiRNAs与目标mRNA序列互补结合,导致翻译抑制、RNA降解或RNA裂解[1].大多数植物miRNAs都是由基因家族编码的,成熟的miRNAs往往有多个靶基因,其mrna中有相似的互补基序。mirna和靶标之间几乎完美的互补促进了计算预测,这可能是由于它们的进化起源。一个被广泛接受的模式米尔基因进化是植物基因组中目标基因序列的反向复制。这些复制的区域米尔基因随着时间的推移通过序列变化,这允许发夹样转录本的生成成为DICER-LIKE酶的底物[2].另一个有趣的模型表明米尔基因可以从整个基因组的各种倒置重复中随机产生,独立于目标基因复制[3.- - - - - -6].例如,最近的证据表明米尔基因可能是转座子活动的结果[7,8].
最近的研究表明,miRNA基因的进化模式(包括谱系特异性的获得或丢失)可能会潜在地影响其靶基因的进化[9].此外,在至少4种植物基因组中,在保守的miRNA靶位点附近发现了目标基因中的同义密码子,这表明同义密码子对miRNA高效结合和miRNA正常功能的选择约束[10].一些miRNAs在从胚胎植物到核心rosids的漫长进化过程中是保守的。然而,一些miRNAs似乎是物种或谱系特异性的[11- - - - - -13].例如,miR156在目前研究的所有被子植物中都是保守的。有趣的是,miR156在核苷酸水平上与miR529相关,共享14-16个nt [12].奇怪的是,尽管这两种miRNAs在胚胎植物中有一个共同的祖先,但miR529似乎在一些分类学类群中丢失了,包括核心eudicots,如拟南芥[12- - - - - -14].这两种miRNAs共享靶基因,靶基因是SQUAMOSA启动子结合蛋白样(SPL)家庭[15].SPL基因是植物特异性转录因子,由76个氨基酸组成的高度保守区域SBP结构域定义[16],以及它们在发展中的重要和广泛作用[17- - - - - -23].SPL基因大致可以分为两大类——长组和短组——后者包含miR156的响应元件,在某些物种中包含miR156和miR529的响应元件。有趣的是,miR156的位点位于靶序列的编码区和非翻译区,而miR529的结合位点主要位于编码区,并与miR156的位点重叠[24,25].
在包含两种miRNA基因的植物谱系中,在营养和生殖器官/组织中miR156和miR529的差异表达可能有利于在其基因组中保留miR156/529序列变体的谱系特异性[26].另一种可能性是,miR156和miR529的联合作用导致了特定谱系中不同靶标的调控,如在Physcomitrella金属盘[27].最近,人们研究了这两个miRNA家族在单子叶植物中的进化差异[25],但miR529缺失在拟南芥等核心eudicots中的后果尚不清楚。更广泛的进化分析MIR156而且MIR529基因及其靶点(包括优迪科特物种)应该为这一问题提供有价值的见解。
在这项研究中,我们扩展了关于miR529和miR156对其保守靶点调控之间的进化和功能差异的知识。通过对目标序列的系统发育重建,我们确认了特异性的eudicotSPL家庭成员保留mir529靶位点,独立存在MIR529它们基因组中的基因。miR529功能的缺失与mirna529特异性靶元件的更宽松选择有关,而mirna156特异性靶序列则受到更强的选择。另外,我们证明了答:芥植物overexpressingMIR529来自单子叶花序的前体,而不是来自基生单子叶花序的前体,表现出与单子叶花序相似的表型spl9; spl15突变的原因是mir156 /529靶向的特异性下调:.基于功能和进化分析,我们提出MIR529基因可能有利于多样化SPLS在尤狄科种。也有可能是新的miR156家族成员取代了eudicots中的miR529功能。
结果与讨论
序列和系统发育分析显示mir529响应元件SPL基因在陆地植物中广泛保守
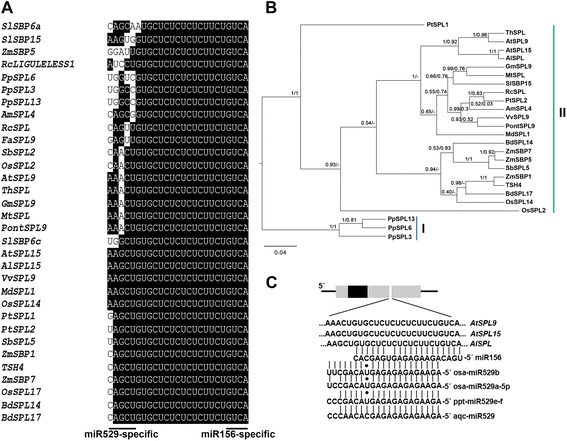
MIR529基因存在于许多陆地植物物种的基因组中。因此,在这样的物种中,转录本的一个子集SPL家族中有miR156和miR529的响应元件[24].为了更好地了解mir529响应元件在陆地植物中的保存程度,我们在植物中寻找mir529响应元件SPL从具有公开基因组序列的物种中获得的基因,包括那些在其基因组中或迄今已识别的转录序列中缺少miRNA529的物种。基于序列互补和进化守恒的psrnatget对miRNA靶点进行高置信度预测[28].我们从三个物种中收集了保守的成熟miR529序列(Physcomitrella金属盘,栽培稻,耧斗菜coerulea),在miRbase v. 21 (http://www.mirbase.org/,如图所示。1.然后,我们对最大期望(E)参数采用更严格的截止阈值(范围为0-2.0;[28])以减少误报目标预测(数据未显示)。令人惊讶的是,我们发现SPL来自几个物种的基因对两种miRNAs(即mir156 /529响应元件)保留高度保守和重叠的响应元件(长度为25 nt);无花果。1和附加文件1),独立于MIR529它们基因组中的基因。这表明,然而MIR529而且MIR156基因经历了不同的进化命运[25],它们的相互靶标(包含mir156 /529响应元件)即使在明显已经消失的物种中也更为保守MIR529基因。例如,eudicot中的mir156 /529响应元件:只存在于编码区,类似于在单目植物和苔藓植物中观察到的情况1;[24])。
含有miR156/ mir529响应元件的SPLs的定位和系统发育。一个25-nt mir156 /529响应元件的比对使用ClustalW (http://www.phytozome.net/).黑色表示保守的核苷酸。b根ML系统发生树描述了代表物种中含有mir156 /529响应元件的SPLs之间的关系。数字表示分支支持。左数,后验概率分支支持。正确的数字,aLRT分支支持(参见方法).“-”表示在贝叶斯或最大似然树中没有观察到特定的分支。蓝线包括苔藓SPLs,绿线包括单子叶植物SPLs和单子叶植物SPLs。cmiR156和miR529的靶位点拟南芥SPL9而且SPL15而且天马草SPL记录。在,拟南芥;艾尔,拟南芥lyrata;Th,Thellungiella halophila;Vv,葡萄;Sl,茄属植物lycopersium;医学博士,马吕斯有明显;足总,草莓属ananassa;Pt,杨树trichocarpa;通用,大豆;太,Medicago truncatula;桥,枳壳trifoliata;点,金鱼草majus;钢筋混凝土,萝藦;Zm评选,玉米;操作系统,栽培稻;双相障碍,Brachypodium distachyon;Aqc,耧斗菜coerulea;页,Physcomitrella金属盘.登记号码在附加文件中给出1
为进一步探讨miR156/529共同靶点的进化关系,系统发育推断SBP-box使用最大似然(ML)和贝叶斯推理方法估计含有mir156 /529响应元件的基因。对齐良好的序列块的成对恒等式的百分比(见方法)为73.3%,替换饱和试验报告对齐不饱和(数据未显示)。我们在共识树中观察到两组SPLs(图。1 b).I组包括已知的miR156/529SPLⅱ组以苔藓植物为目标,以各种单子叶植物和核心单子叶植物为主:含有miR156/529的保守结合位点。这一分析表明:含有miR156/529的靶位点在陆生植物中具有共同的起源(图。1 b).拟南芥SPL9而且SPL15密切相关,极有可能形成一对近亲基因[29,30.].因此,这两个SPL9而且SPL15以及他们的矫形器保留了mir156 /529响应元件(图。1b和c).
它已被提议为单子叶植物和p .金属盘那:相比之下,含有miR156/529位点的基因保守进化速度较慢:只含有mir156响应元件[24].进一步阐明欧迪科特的进化命运:包含mir156 /529响应元件,我们分析了SPL序列中的两个区块:“SBP结构域”区块,其中包含SBP结构域的核苷酸[16]加上上游/下游的几个核苷酸,以及“靶位点”块,该块包含包含mir156 /529响应元件的核苷酸(见方法).对于“SBP域”块,我们估计了代表的非同义替换(Ka)和同义替换(Ks)比率(Ka/Ks):.我们选择拟南芥作为一个代表性的eudicot,是因为即使经过大量的测序工作,miR529的前体或标准成熟序列都没有在其中发现答:芥或者它的近亲答:lyrata[31].中最佳对齐块(249到2061 nt)的成对对齐SPL基因答:芥、monocots和p .金属盘表明“SBP结构域”块在所有比较的纯化选择下,反映了功能限制,这是预期的,因为SBP可能是一个DNA结合结构域(附加文件2;[16])。
考虑到“目标位点”区块很短(25nt),可能会导致不切实际的Ka/Ks比率,我们决定更直接地比较它们的排列(图1)。2).考虑到miR529靶位点上的5个第一碱基,我们发现了包括不同理化特征的氨基酸在内的非同义取代。相比之下,指定miR156靶位点的核苷酸却没有发生如此剧烈的变化(图1)。2).这些观察表明,“目标位点”块的不同区域可能受到不同的进化约束。事实上,在“靶位点”块中分化较多的碱基专门对应于miR529靶位点(图5)。2).因此,eudicots中miR529调控的缺失可能使mirna响应元件区域的选择更加宽松或纯度较低,这反映了其作为miR529靶点的功能的丧失。与这一观察结果相一致的是,在eudicot中,“靶位点”块(包括miR156和miR529反应元件)内的核苷酸识别率较低SPL基因(58.7%)比单子叶植物和苔藓植物的基因(81.3和86.7%)少(图1)。2).在monocots和p .金属盘,“靶位点”区块较为保守,可能是因为它与功能相关,因为miR529仍然存在于这些物种中[24].
miR156和miRNA156/529靶位点的DNA比对。miR156/529靶位点编码的氨基酸如图所示。氨基酸的颜色根据其疏水性而定;红盒子,eudicot序列;绿盒子,单子叶序列;蓝框,苔藓植物序列。5 ' 4个核苷酸的核苷酸识别率显示在每个彩色框的旁边。最上面的标志下面的黑线表示miRNA结合位点。底部的方框显示了基于每个植物组的排列构建的标识。登记号码在附加文件中给出1
基础eudicot microRNA529前体过表达答:芥miR156过表达基因
尽管miR156高度保守,存在于目前评估的所有植物物种中,但miR529似乎仅限于特定的分类组[12].为了看得更清楚MIR156/529基因进化,这些miRNAs的系统发育关系使用最大似然(ML)方法访问。对于系统发育分析,我们包括MIR156而且MIR529的前兆Physcomitrella金属盘,单子叶种(栽培稻,玉米,Brachypodium distachyon,高粱二色的)、一种基本的(耧斗菜coerulea),以及的前身MIR156基因拟南芥.生成了一个一致的ML树,其中很容易观察到两个一般的、广泛的组(附加文件3.A)第一组组成MIR156来自不同物种的前体,而第二类含有MIR529从单花斑和苔藓的前体。单子叶植物MIR529前体被分组到II组的一个不同的子集(附加文件3.A),这表明进化的分歧发生在陆地植物的共同祖先中,这导致了分裂MIR529苔藓和开花植物的基因。
早前有人提出,miRNA进化的一个关键特征是,一旦进化,家庭和家庭成员很少丢失[2].然而,并不是所有的miRNA都是同样保守的,最近的研究表明,在精子植物起源之前进化的科中发生了一些miRNA的丢失[32].我们的数据表明,miR156和miR529家族在胚胎植物中经历了动态复制和丢失,通过这一过程,分支或物种特异性的miRNA基因亚群产生或消失。例如,答:芥至少有10个MIR156位点10个mir156靶向:而大米至少有12个MIR156位点,两个MIR529其中8个靶向miR156, 4个靶向miR156/ mir529:[28].
有趣的是,预测MIR529基部的前体答:coerulea[33)被分到第一组拟南芥MIR156h而且耧斗菜MIR156a而且b前体,表明这些miRNAs的共同起源(附加文件3.此外,A)。耧斗菜MIR529似乎与拟南芥高度保守MIR156h在核苷酸和二级结构水平(附加文件3.B).这些观察结果提出了一个问题米尔的前兆耧斗菜定义为MIR529的确是一个MIR156同族体。为了验证这一假设,我们调查了是否位点侧翼耧斗菜与单子叶种相比,pre-miR529定位于共筋块。我们首先在不同的单子叶植物中寻找这样的同向群,包括z梅斯,o .漂白亚麻纤维卷,b . distachyon,美国二色的(附加文件4A). pre-miR529位点周围的基因共线性相对保守,因此在单性状中定义了保守的共共链块。相反,我们没有检测到包括pre-miR529在内的共通阻滞的任何保守耧斗菜虽然在这个物种中发现了一些同源基因(附加文件4B)。此外,两者之间没有可检测到的同步性MIR156h- eudicots相关区域(答:芥而且诉酿酒用葡萄)及MIR529-基部的相关区域答:coerulea(附加文件4B, C)。这种完全丧失的同步性,以及我们的系统发育和序列分析,表明基础eudicot耧斗菜失去了一个善意的MIR529可能是在miR529的调控作用因miR529序列突变而受到干扰之后,如最近提出的[25].
以确定是否AqcMIR529可以被正确处理并产生功能性miRNAs,我们在答:芥病毒35S启动子控制下的植物,该启动子提供了强烈的和几乎无处不在的表达([34];这种植物以后被称为35 s:: AqcMIR529行;看到方法).我们比较了col0型和野生型的营养表型,35 s:: AqcMIR529超量表达的转基因植物AtMIR156a前体(35 s:: AtMIR156a;[35),以及双突变体spl9; spl15(无花果。3).这个突变体在两个体内都有T-DNA插入AtSPL9而且AtSPL15位点这导致它们的表达和功能明显减少,尽管其他AtSPLs仍然有效。转基因答:芥植物通过耧斗菜-过表达结构的表型类似于35 s:: MIR156a植物,如大量更小,更圆的莲座叶(图。3).此外,过度表达的MIR529的前体Aguilegia在答:芥导致下调的不仅仅是AtSPL9而且AtSPL15基因(包含miR156和miR529反应元件;无花果。1),但也有其他SPL基因(图。3 b).因此,35 s:: AqcMIR529株系表现出较强的表型倾向35 s:: MIR156a植物(图。3).
根据观察到的表型和表达分析,RACE分析SPL15裂解位点显示miR156主要靶向转录本35 s:: AqcMIR529植物(图。3 c).根据这些观察结果AqcMIR529前体比较保守MIR156-like precursors(附加文件3.),我们提出至少两种非排他性的可能性:(1)预测的AqcMIR529前驱者更可能是副手AqcMIR156a而且b基因并产生mirna156样的小RNA;(2)AqcMIR529前体可能如最近提出的那样在miR529序列中积累了突变[25],导致miR529生物生成和/或功能的丧失。由于其他基底瓣似乎积累了mir529样的小rna [36],我们不能排除mir529样小rna仍在特异性积累的可能性答:coerulea组织,因为没有可获得的信息,大规模鉴定小RNA群体在这个基部的eudicot。
MiR156和miR529在水稻营养发育过程中表现出重叠表达模式
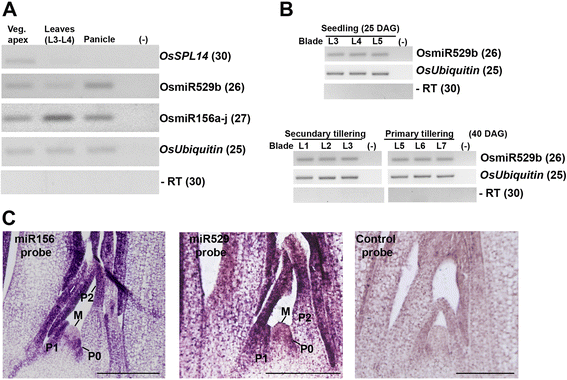
不同于基部的耧斗菜在美国,单子叶植物保留了这两种植物是有据可查的MIR156而且MIR529它们基因组中的前体[12,26,37],提出了一个问题,即miR529调控在单子叶植物中被保留,是因为它是必不可少的,还是仅仅是冗余反映亚功能化的一个经典案例,在亚功能化中,miR529与miR156相比作用有限。赖斯有两份MIR529前体(一个而且b)。已有研究表明,OsmiR529a*(或OsmiR529a-5p)优先表达于穗部,而OsmiR529b则广泛表达于根、茎和穗部[26].然而,作者没有评估OsmiR156或OsmiR529在营养尖和幼叶中的表达:定义内源性开花途径,分别控制叶片成熟和起始[19,38].为了进一步了解OsmiR529可能的作用[26,我们分析了OsmiR529b、OsmiR156a-j及其共同靶标之一(OsSPL14)在营养尖,幼叶(L3-L5)和幼穗。OsmiR529b和OsmiR156a-j在评估的所有组织/器官中均有表达,但表达水平不同(图2)。4).OsmiR156a-j转录本在幼叶组织中大量积累OsSPL14在叶片和幼穗组织中表达下调。4).OsSPL14在幼苗中被两种mirna靶向,而在穗中似乎主要被miR529a-5p靶向[26].同样,OsmiR529b也可能对转录后调控有更显著的影响OsSPL14幼穗的表达。
miR529和miR156的表达谱栽培稻.一个茎环脉冲RT-PCR检测OsmiR529b, OsmiR156a-j和OsSPL14植物尖的转录本(Veg。先端),叶(L),和年轻的圆锥花序组织。无RT(−RT)和无cDNA(−)的反应作为阴性对照。括号内的数字表示PCR周期。DAG,发芽后数天。大米泛素(LOC_Os03g0234200)作为内部控制。b茎环脉冲RT-PCR检测水稻幼苗和分蘖发育阶段叶片(L)组织中OsmiR529b转录本。无RT(−RT)和无cDNA(−)的反应作为阴性对照。括号内的数字表示PCR周期。大米泛素(LOC_Os03g0234200)作为内部控制。cmiR529和miR156在水稻茎尖分生组织(SAM)中的时空表达规律3 '标记的lnna修饰寡核苷酸探针检测miR156和miR529,如前所述[38]与25-DAG水稻幼苗的SAM纵断面杂交。用扰码mirna 3 '标记的LNA探针作为阴性对照。紫色染色显示探针定位。米,分生组织;P,叶原基。棒材:10 μm
OsmiR156在水稻叶片发育过程中是动态表达的,OsmiR156表达的逐渐增加可能是调控靶基因时间表达的必要条件,包括OsSPL14[38].我们评估了OsmiR529b在幼苗(L3-L5)和分蘖期发育相似的叶片中的表达模式。与观察到的OsmiR156表达模式相比[38, OsmiR529b在所有叶片发育阶段都广泛表达(图。4 b),表明该miRNA对的表达的时间控制有微小或可忽略的贡献SPL水稻叶片成熟过程中的基因。然而,miR529也有可能作为一个抑制miRNA来建立正确的平衡SPL单花斑花序颞叶发育过程中的靶点。
考虑到OsmiR529b和OsmiR156a-j转录本在营养顶端积累(图。4),我们决定评估它们在茎尖分生组织(SAM)中的空间表达模式,使用描述的特异性探针进行原位杂交([38];见的方法)。这两种miRNAs都表达在早期(P0)和发育中的叶片原基中,但不在分生组织固有区(即周围区和中心区)中(图。4摄氏度).这种表达模式强化了拟南芥的数据,表明miRNA调控:主要发生在叶原基中,SPL活性可能通过生长素信号通路在SAM非自主抑制新叶的起始[39].我们的原位数据显示,OsmiR529a-b和OsmiR156a-j在叶片原基中具有重叠的空间表达模式,这表明这些miRNAs可以冗余调控或协同调节这些器官的靶蛋白表达调控。然而,基于报道的OsmiR156表达水平高于OsmiR529表达水平[26],为的贡献SPL这两种miRNAs的表达调节不太可能具有可比性,主要是在水稻营养发育的早期阶段。
改进的5 ' -RACE程序可用于访问miRNA靶的切割产物以及miRNA前体的处理[40,41].对来自水稻降解组且仅映射到前mirnas的RNA端(PARE)签名的并行分析可以为miR156和miR529在水稻发育中的作用提供额外的证据。因此,我们收集的PARE签名OsMIR156a-l而且OsMIR529a-b来自公共资源的前体(参见方法).根据现有数据,似乎大米MIR156而且MIR529前体的加工是不同的,这可能导致不同的miRNA积累在水稻组织/器官(图。4和附加文件5).即使是在OsMIR156家庭,特定的成员被区别处理。例如,OsMIR156k而且- l与其余的相比显示更少的PARE签名MIR156体细胞。同样的,OsMIR529a而且b前体与大多数前体相比,具有较少的签名量OsMIR156precursors(附加文件5).前体加工的差异也可能是OsmiR156和OsmiR529在发育过程中功能不同的原因。miR529的调控是否与:对水稻营养发育至关重要。与miR156相比,过表达或下调miR529对转基因/突变单子叶植物的影响还有待研究。
保守的mir529响应元件在AtSPL9而且AtSPL15基因
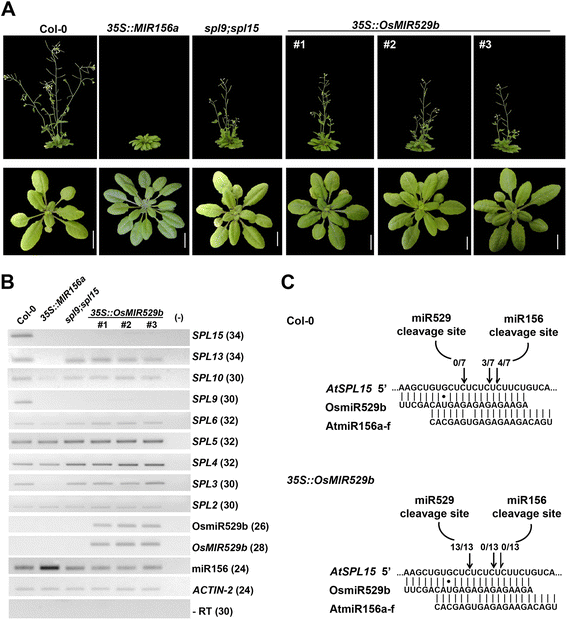
如前所述,miR529在像答:芥,却又密切相关AtSPL9而且AtSPL15基因(29]含有一个保守的mir529响应元件,类似于它们在答:lyrata(无花果。1 c).考虑到miRNA::靶基因相互作用在答:芥[15,17,42,我们询问miR529结合位点是否在这个核心eudicot中起作用。为了验证这一假设,我们培育了转基因拟南芥植株,植株中含有注释良好的水稻MIR529b在病毒35S启动子控制下的前体(植物下文简称35 s:: OsMIR529b行)。作为AtSPL9而且AtSPL15我们比较了Col-0的营养表型和开花表型,35 s:: OsMIR529b超量表达的转基因植物AtMIR156a前体和双突变体spl9; spl15(无花果。5).至少三个独立的35 s:: OsMIR529b株系表现出与双突变体相似的表型spl9; spl15,例如莲座丛叶数量的增加与开花时间的轻微延迟相结合,尽管这些表型比来自35 s:: AtMIR156a植物(图。5).
表型和表达分析35 s:: OsMIR529b拟南芥植物。一个六周大的形态(上面的显示器)和25天(lowerpannel)植物(col0或野生型,35 s:: MIR156a,spl9; spl15突变体,35 s:: OsMIR529b).比例尺代表1厘米。b茎环脉冲RT-PCR检测OsMIR529b前体,OsmiR529b, miR156和AtSPL拟南芥叶片组织中的转录本。无RT(−RT)和无cDNA(−)的反应作为阴性对照。括号内的数字表示PCR周期。Actin-2被用作内部控制。c修正劈裂的5 ' -RACE分析SPL15Col-0和35 s:: OsMIR529b叶组织。5 '端SPL15裂解产物优先映射到miR156(在Col-0中)和miR529b结合位点(在Col-0中)35 s:: OsMIR529b)
以进一步证实表型的相似性35 s:: OsMIR529b而且spl9; spl15植株的嫩叶和莲座叶的平均数量(表1).在我们的长日(LD)生长条件下35 s:: MIR156a株系比Col-0(野生型)产生的幼叶多2.8倍,与先前报道的数据相似[17),而35 s:: OsMIR529b而且spl9; spl15植物的产量,平均只有1.4倍。同样,生产莲座叶的35 s:: OsMIR529b系表现出较强的表型倾向spl9; spl15突变体(表1).
除了估计在第一朵花出现之前叶子形成的数量,我们还确定了35 s:: OsMIR529b线需要螺栓和花(表1).平均而言,过表达OsmiR529b的转基因植物与Col-0(野生型)相比,向开花的过渡略有延迟(3.3天)。根据施瓦茨和同事的观察[29],我们还观察到双突变体spl9; spl15显示了Col-0和miR156过表达之间的中间行为(表1).重要的是,spl9; spl15植物在统计上没有差异35 s:: OsMIR529b在向开花和叶片发育的过渡方面(表1).这些数据加强了我们的观察OsMIR529b过表达蛋白表现出类似的营养和生殖表型spl9; spl15变种人,可能是因为体内的SPL9而且SPL15两种基因型的转录本。
此外,MiR156目标SPL9而且SPL15,仅其他8个SPL家庭成员[15这些基因被显示在AtMIR156b-过度表达的植物[17].相比之下spl9; spl15双突变体和35 s:: OsMIR529b行,35 s:: MIR156a线显示更严重,异常的营养和生殖表型(表1;[35]),这可能是由于额外的mir156靶向SPL基因冗余调节叶片起始和相位变化[43].相反,如在spl9; spl15只有双突变体AtSPL9而且AtSPL15基因可能被抑制35 s:: OsMIR529b使其具有较低的异常表型(图。5).为了证实这一假设,我们评估了几种的表达模式SPL植物叶组织中的家族成员35 s:: OsMIR529b行,spl9; spl15突变体,35 s:: MIR156a和Col-0。我们还评估了来自OsMIR529b前体和其各自成熟miRNA的积累(图。5 b).不出所料,SPL基因下调35 s:: MIR156a植物(图。5 b).然而,根据我们的系统发育(图。1 b)和表型数据(表1),只有转录水平AtSPL9而且AtSPL15显示出严重的减少35 s:: OsMIR529b植物。此外,miR156转录本水平在Col-0和35 s:: OsMIR529b植物(图。5 b),表明在SPL9而且SPL15表达OsMIR529b过表达极有可能是由于水稻miR529b转录本的积累。总之,这些结果证实了单子叶植物MIR529前体在核心eudicot中被正确处理,产生成熟的miR529转录本,并下调特异性SPL基因。
的转录后调节AtSPLmiR529b基因,我们在SPL15记录在35 s:: OsMIR529b采用改良的5 ' -RACE方法的植物[40].RACE分析表明,miR529b的解理位点发生在碱基10和11之间(图5)。5度),类似于对单克隆体的描述[44].这些结果有力地表明SPL15在拟南芥中主要由OsmiR529b调控35 s:: OsMIR529b表明miR529在转录后调节特异性中具有保守功能SPL家庭成员。重要的是,我们的数据表明,mir529响应元件在拟南芥中是保守的和功能的SPL9而且SPL15基因,可能是由于对mir156 /529响应元件周围区域的氨基酸或RNA二级结构的选择性限制。
结论
我们已经证明了这一点MIR529基因在拟南芥中已经丢失,特别是在所有的尤狄科物种中SPL这些物种的基因保留了mir529响应元件,可能是由于保持了同义密码子以有效结合miR156和适当的功能[10].更具体地说,拟南芥SPL9而且SPL15即使没有mir529产生的位点,基因也保留了mir529响应元件的功能。类似于单子叶床:, eudicotSPL含有mir156 /529响应元件的基因似乎与只含有mir156响应元件的基因受进化限制不同。这种趋势可能表明mirna介导的调控抑制了靶点的进化。
这有可能是miR156和miR529之间的相互作用特异调控:调整单花,特别是禾本科植物的花结构发育非常重要[26,37].由于拟南芥没有miR529,可能特定的miR156家族成员(如miR156h。2,在花组织中优先表达;[45)功能上替代miR529。可以想象,其他核心蔷薇属和/或密切相关的物种答:芥miR156/miR529/SPL的进化史相似,但这种确认需要未来的研究。
方法
植物材料和生长条件
拟南芥植物(生态型哥伦比亚-0或Col-0)在21°C(白天)/19°C(夜晚)的长白天条件下(16 h光照/8 h黑暗)生长。转基因植物35 s:: MIR156a而双突变体spl9-1; spl15-2被描述为[35].转基因植株经PCR基因分型证实。
对于转基因拟南芥植物,二元结构35 s:: OsMIR529b而且35 s:: AqcMIR529被送到根癌土壤杆菌用电穿孔法制备GV3101 (pMP90)。用花浸渍法生成转基因植株[46]并用50 mg/mL卡那霉素在半强度MS板上筛选。至少选择6个独立的卡那霉素耐药系进行PCR基因整合,随后检测转基因表达水平(数据未显示)。进一步分析选择的系在T3代。
稻子(栽培稻ssp粳稻)在土壤中发芽,植物在温室条件下生长。
DNA结构
所有结构物的寡核苷酸引物在附加文件中给出6.一个1000 bp的碎片OsMIR529b的基因组DNA扩增出前体o .漂白亚麻纤维卷.PCR产物亚克隆到pGEM (promega)中进行测序。已确认OsMIR529b前体用BamHI而且尚驰随后克隆到CaMV35S启动子后面的二元载体pBI121中。为35 s:: AqcMIR529建筑,一个125 bp的片段,包含注释AqcMIR529前体(33的基因组DNA扩增而成答:coerulea测序后克隆到植物二进制目的载体pK7WG2 (Gateway System;[47)在CaMV35S启动子后面。
RNA提取和茎环脉冲逆转录酶(RT)-PCR
根据制造商说明使用Trizol试剂(Life Technologies, USA)提取拟南芥(叶组织)和水稻(营养尖、叶和穗组织)的总RNA,然后用DNAse I (Life Technologies, USA)处理。根据Varkonyi-Gasic等人的研究,对于miRNA和mRNA检测,DNAse - i处理的RNA (2.0 μg)被反向转录,生成第一链cDNA。[48].在反应中加入Oligo(dT)引物,用于检测靶mrna和内控。PCR反应采用cDNA稀释液:1.0 μL cDNA, 1.5 mM硫酸镁,0.25 mM dNTP, 10 pmol引物,1u Taq DNA聚合酶(Promega, USA)。反应在以下条件下进行:94°C 2分钟,适当的循环数94°C 20 s, 60°C 30 s, 72°C 45 s。用两个生物样品重复所有反应至少两次。引物序列在附加文件中描述6.
5 ' -RACE分析
从拟南芥植物的莲座叶中提取5微克总RNA (Col-0,35 s:: OsMIR529b而且35 s:: AqcMIR529)连接到RNA适配器上,在含有0.5 U/μL T4 RNA连接酶、4 U/μL RNAse抑制剂和1 mM ATP的反应混合物中。后续步骤根据GeneRacer工具包(Invitrogen)的制造商指南执行。第一次PCR是用以下方法完成的AtSPL15特异引物:5 ' -AGCCATTGTAACCTTATCGGAGAATGAG。该PCR反应随后被用作半嵌套PCR的模板AtSPL15-特异性引物(5 ' -TCATCGAGTCGAAACCAGAAGAT)。扩增后对5’-RACE产物进行凝胶纯化克隆,随机选取至少8个独立克隆进行测序。
表型分析
在发芽或DAG后的15、20、25和30天内测量莲座叶的数量。对开花、抽苔时间和幼叶数进行了估计。使用ASSISTAT版本7.6 beta (t-studentP< 0.01)。
原位杂交
非放射性原位杂交按所述方法进行[49].栽培稻种子萌发后25天采集营养尖。所述与miR56和miR529序列互补的锁定核酸探针[38]和阴性对照stir - mir (5 ' -GTGTAACACGTCTATACGCCCA)由Exiqon(美国)合成,地高辛用DIG寡核苷酸3 '端标记试剂盒(美国罗氏应用科学公司)标记。每片用10皮摩尔的探针。杂交和洗涤步骤在55°C进行。
系统发育和序列分析
序列MIR156而且MIR529前体(pre-miRNAs)从miRBase v.21中检索(http://www.mirbase.org/),而SPL序列则从PHYTOZOME v. 9.1中检索(http://www.phytozome.net/)及TAIR (https://www.arabidopsis.org/).对于miRNA前体,检索到的序列使用ClustalW对齐[50使用默认值。系统发育分析在MEGA v. 5.05中进行[51使用默认值。采用最大似然(ML)方法和Bootstrap分析(1000棵树)进行系统发育推断。
DNA序列:使用Muscle算法进行对齐[52对齐好的块使用Gblocks服务器(http://molevol.cmima.csic.es/castresana/Gblocks_server.html)采用最严格的选项。多序列对齐在附加文件中描述7.利用DAMBE5软件实现的Xia检验算法,对对齐进行不变站点比例估计和替代饱和试验[53].使用在jModelTest中实现的Akaike信息准则执行最适合进化模型的选项[54].系统发育重建采用ML和贝叶斯推理方法,使用PhyML v3.0 [55]和Beast v1.8.0 [56],后者正在CIPRES的科学门户(https://www.phylo.org/).近似似然比检验[57]用于ML分析。计算贝叶斯推理的后验概率估计。应用软件Tracer来确定贝叶斯方法生成过程中的老化(使用日志似然分数),以及TreeAnnotator [54]对排除收敛区域外出现的树后的数据进行汇总。估算了固定位点和伽马分布(类别数= 4)的比例,并使用了贝叶斯分析的随机本地时钟模型。
映射的PARE签名OsMIR156而且OsMIR529前体和RNA-seq和sRNA签名从水稻次世代序列db (http://mpss.udel.edu/).通过将所有样本归一化到TP10M(每1000万次阅读的转录本)来估计序列丰度。
非同义(Ka)和同义(Ks)替换计算
选择压力分析(Ka/Ks)使用最佳对齐块对包含SBP结构域和少量上游/下游核苷酸(命名为“SBP结构域”)的核苷酸序列进行。根据图中所示的系统发生树,仔细选择了Eudicot、单子叶植物和苔藓植物的SPL序列。1 b和先前报道的系统发育分析[29].每个编辑对齐的DNA序列的密码子都是使用内部Python脚本选择的。利用模型选择(MS)方法,通过KaKs_Calculator软件比较优子科、单子科和苔藓类群之间和类群内部的序列,估算出Ka/Ks值[58].选定的“目标位点”序列使用Muscle算法进行对齐,并使用genous工具(http://www.geneious.com).
同线性分析
基于pre-miR529位点邻近基因的坐标在O. sativa(OsMIR529a而且OsMIR529b),答:coerulea, pre-miR156h from诉酿酒用葡萄而且答:芥在《genome icus Plants v.16.03》中搜索了单子叶科和双子叶科物种之间和内部的共丝块的保存[59].的共时映射耧斗菜coerulea,我们首先使用Phytozome数据库中pre-miR529位点的坐标(scaffold 4:4,760,784..4,810,783)来绘制周围的侧边基因aqc-MIR529轨迹。在genome icus Plants v.16.03中查询了所选优子花和单子花中这些位点的同源基因。[59].利用Phytozome数据库进行同源物检索答:coeruleapre-miR529和pre-miR156h邻近基因o .漂白亚麻纤维卷,诉酿酒用葡萄,答:芥,分别。
支持数据的可用性
所有在手稿中引用的已发表的数据集都在参考书目中被引用。所有支持数据都包含在附加文件中。
加入数据
的AGI标识符拟南芥基因如下:SPL2At5g43270;SPL3At2g33810;SPL4At1g53160;SPL5At3g15270;SPL6At1g69170;SPL9At2g42200;SPL10At1g27370;SPL13At5g50570;SPL15At3g57920;Actin-2, At3g18780。
参考文献
- 1.
Axtell MJ, Westholm JO, Lai EC。Vive la differénce:植物和动物中microRNAs的生物发生和进化。基因组生物学,2011;2:221。
- 2.
Allen E, Xie Z, Gustafson AM, Sung GH, Spataford JW, Carrington JC。通过反向复制靶基因序列进化的microRNAs基因拟南芥.Nat Genet 2004; 36:1282-90。
- 3.
吕超,Tej SS, Luo S, Haudenschild CD, Meyers BC, Green PJ。转录组小RNA组分的解析。科学。2005;309:1567-9。
- 4.
Kasschau KD, Fahlgren N, Chapman EJ, Sullivan CM, Cumbie JS, Givan SA,等。拟南芥sirna的全基因组分析。生物学报2007;15:e57。
- 5.
felppes FF, Schneeberger K, Dezulian T, Huson DH, Weigel D.进化拟南芥来自随机序列的microRNAs。RNA。2008; 14:2455-9。
- 6.
Piriyapongsa J, Jordan IK。植物转座元件对sirna和miRNAs的双重编码。RNA。2008; 14:814-21。
- 7.
Rajagopalan R, Vaucheret H, Trejo J, Bartel DP。一组多样化的、进化上流动的小核糖核酸拟南芥.基因发展2006;20:3407-25。
- 8.
Creasey KM, Zhai J, Borges F, Van Ex F, Regulski M, Meyers BC等人在拟南芥中从转座子中触发广泛表观遗传激活的sirna。大自然。2014;508:411-5。
- 9.
赵敏,Meyers BC,蔡超,徐伟,马娟。大豆基因组多重复制机制引发的MIRNA基因和靶基因的进化模式及共同进化结果。《植物细胞》2015;27:546-62。
- 10.
顾伟,王旭,翟春,谢曦,周涛。植物miRNA结合位点附近同义位点的选择。中华分子生物学杂志2012;29(10):3037-44。
- 11.
Axtell MJ, Bartel DP。陆生植物中microRNAs的古老及其靶标。植物细胞,2005;17:1658-73。
- 12.
Cuperus JT, Fahlgren N, Carrington C. MIRNA基因的进化和功能多样化。植物细胞,2011;23:431-42。
- 13.
蒙蒂斯,德Fátima Rosas-Cárdenas F,德保利E,阿克比M, Rymarquis LA, Mahalingam G,等。维管植物样品测序显示了广泛的微rna的保守性和发散性。2014;5:3722。
- 14.
ortizs - morea FA, Vicentini R, Silva GF, Silva EM, Carrer H, Rodrigues AP,等。甘蔗微转录组的全局分析揭示了一种与腋芽生长相关的小RNA的特殊组成。J Exp Bot. 2013; 64:2307-20。
- 15.
罗德斯MW,莱因哈特BJ, Lim LP, Burge CB, Barte B, Barte DP。植物microRNA靶点的预测。细胞。2002;110:513-20。
- 16.
一个新的DNA结合蛋白家族包括推测的转录调控因子金鱼草majus花分生组织鉴定基因SQUAMOSA。中华医学杂志1996;250:7-16。
- 17.
Schwab R, Palatnik JF, Riester M, Schommer C, Schmid ME, Weigel D. MicroRNAs对植物转录组的特异性影响。Dev Cell. 2005; 8:517-27。
- 18.
异慢性玉米突变体Corngrass1是由串联microRNA的过表达引起的。Nat Genet 2007; 39:544-9。
- 19.
Wang JW, Czech B, Weigel D. mir156调控的SPL转录因子定义了植物内生开花途径拟南芥.细胞。2009;138:738-49。
- 20.
诺丁MD,巴特尔DP。在植物胚胎发生过程中,MicroRNAs可阻止早熟基因的表达并使模式形成。基因发展2010;24:2678-92。
- 21.
于宁,蔡文杰,王松,单春明,王立军,陈晓燕。microRNA 156靶向SPL基因对毛状体分布的时间控制拟南芥.植物细胞,2010;22:2322-35。
- 22.
苟建勇,felppes FF,刘cj, Weigel D,王建伟。mir156靶向SPL转录因子对拟南芥花青素生物合成的负调控植物细胞,2011;23:1512-22。
- 23.
Silva GFF, Silva EM, Azevedo MS, Guivin MAC, Ramiro DA, Figueiredo CR等。靶向microrna156的SPL/SBP盒转录因子调控番茄子房和果实发育。植物工程学报,2014;38(4):447 - 447。
- 24.
凌丽珍,张士德。探讨miR156和miR529靶向的SBP-box基因在植物中的进化差异。遗传。2012;140:317-24。
- 25.
张德硕,凌丽珍,张秋峰,徐建东,程磊。植物SBP-Box基因MiR156和MiR529组合调控子的进化比较。科学通报,2015;10:e0124621。
- 26.
郑德华,Park S,翟杰,Gurazada SG, De Paoli E, Meyers BC,等。水稻小RNA的大量分析:调控的小RNA和变异的差异目标RNA切割的机制意义。植物学报,2011;23(12):4185-207。
- 27.
Arif MA, Fattash I, MA Z, Cho SH, Beike AK, Reski R,等。像骰子一样的活动Physcomitrellapatens DICER-LIKE4突变体导致严重的发育障碍和不育。植物学报,2012;5:1281-94。
- 28.
戴X,赵PX。psRNATarget:植物小RNA目标分析服务器。核酸Res. 2011;39(Web Server issue): W155-9。
- 29.
Schwarz S, Grande AV, Bujdoso N, Saedler H, Huijser P. microRNA调控SBP-box基因SPL9而且SPL15控制拟南芥芽成熟。植物分子生物学,2008;67(1-2):183-95。doi:10.1007 / s11103 - 008 - 9310 - z.
- 30.
普雷斯顿JC,希勒曼LC。植物SQUAMOSA-PROMOTER BINDING-LIKE (SPL)基因家族的功能进化。植物学报(自然科学版)2013;4(80):1-13。
- 31.
Fahlgren N, Jogdeo S, Kasschau KD, Sullivan CM, Chapman EJ, Laubinger S,等。微基因进化拟南芥lyrata而且拟南芥.植物学报,2010;22(4):1074-89。
- 32.
陈凯。转录因子和microrna调控基因的进化。Nat Rev Genet 2007; 8:93-103。
- 33.
泰勒RS,塔弗JE,希斯科克SJ,多诺霍PCJ。植物微rna的进化史。植物科学进展2014;19:175-82。
- 34.
普泽,克莱默,守恒的识别耧斗菜coruleamicroRNAs和它们的靶标。基因。2009;448:46-56。
- 35.
张国强,张国强,张国强。花菜花叶病毒35s启动子活性所需DNA序列的鉴定大自然。1985;313:810-2。
- 36.
Barakat A, Wall K, Leebens-Mack J, Wang YJ, Carlson JE, Depamphilis CW。基部小叶藻microRNAs的大规模鉴定(Eschscholzia californica)和保护开花植物。植物学报,2007;32(5):571 - 571。
- 37.
郑dh, Szchmidt SA, Rymarquis LA, Park S, Ganssmann M, German MA,等。RNA末端的平行分析增强了对微RNA和靶RNA的全局研究Brachypodium distachyon.中国生物技术学报2013;24:R145。
- 38.
谢凯,沈杰,侯旭,姚杰,李旭,肖杰,等。miR156的逐渐增加调控了水稻叶片发育过程中众多基因的时间表达变化。植物物理学报,2012;158:1382-94。
- 39.
王金伟,Schwab R, Czech B, Mica E, Weigel D. mir156靶向SPL基因和CYP78A5/KLUH对成虫成虫细胞长度和器官大小的双重影响拟南芥.植物细胞。2008;20:1231-43。
- 40.
Addo-Quaye C, Eshoo TW, Bartel DP, Axtell MJ。通过拟南芥降解组测序确定内源性siRNA和miRNA靶点。当代生物学。2008;18:758-62。
- 41.
吴鹏,郭琳,陈东,吴鹏,陈敏。高通量降解组测序可用于深入了解microRNA前体代谢。实验机器人2010;61:3833-7。
- 42.
Jones-Rhoades M, Bartel D.植物microrna及其靶标的计算鉴定,包括胁迫诱导的miRNA。分子细胞。2004;14:787-99。
- 43.
植物营养相变化与芽成熟。Curr Top Dev Biol 2013; 105:125-52。
- 44.
玉米穗鞘4编码的SBP-box转录因子调控苞片发育和分生组织边界的建立。发展。2010;137:1243-50。
- 45.
郑德华,Thatcher SR, Brown RS,翟杰,Park S, Rymarquis LA,等。通过分析序列变异、表达模式、ARGONAUTE负载和目标切割增强microRNAs的全面研究。植物物理学报,2013;162:1225-45。
- 46.
植物浸渍:农杆菌介导的转化的一种简化方法拟南芥.植物学报1998;16:735-43。
- 47.
王晓峰,王晓峰,王晓峰,等。农杆菌介导植物转化的GATEWAY载体。植物学报2002;7:193-5。
- 48.
Varkonyi-Gasic E, Wu R, Wood M, Walton EF, Hellens RP。协议:一种检测和定量microRNAs的高灵敏度RT-PCR方法。植物方法。2007;3:1-12。
- 49.
标枪M, Timmermans MCP。原位利用LNA探针定位植物中的小rna。Nat Protoc 2012; 7:533-41。
- 50.
Larkin MA, Blackshields G, Brown NP, Chenna R, McGettigan PA, McWilliam H,等。Clustal W和Clustal X 2.0版本。生物信息学。2007;23:2947-8。
- 51.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5:利用最大似然、进化距离和最大简约方法进行分子进化遗传学分析。中华分子生物学杂志2011;28(10):2731-9。
- 52.
埃德加钢筋混凝土。MUSCLE:多序列对齐,高精度,高吞吐量。核酸学报,2004;32:1792-7。
- 53.
夏晓。DAMBE5:分子生物学与进化领域的数据分析综合软件包。Mol Biol Evol. 2013; 30:1720-8。
- 54.
Darriba D, Taboada GL, Doallo R, Posada D. jModelTest 2:更多的模型,新的启发式和并行计算。Nat方法。2012;9(8):772。
- 55.
Guindon S, Dufayard JF, Lefort V, Anisimova M, Hordijk W, Gascuel O.估计最大似然系统发育的新算法和方法:评估PhyML 3.0的性能。中华生物学报2010;59(3):307-21。
- 56.
Drummond AJ, Suchard MA, Xie D, Rambaut A.贝叶斯系统发育与BEAUti和BEAST 1.7。中华分子生物学杂志2012;29(4):447 - 447。
- 57.
Anisimova M, Gascuel O.分支的近似似然比检验:一种快速、准确和强大的替代方法。系统生物学杂志2006;55:539-52。
- 58.
张铮,李建林,赵晓强,王杰,王国胜,余杰。KaKs计算器:通过模型选择和模型平均计算Ka和Ks。基因组学蛋白质组学生物信息学2006;4:259-63。
- 59.
Louis A, Nguyen NTT, Muffato M, Crollius HR。2015年基因组更新:核型视图和矩阵视图为多物种比较基因组学提供了全基因组视角。核酸学报2015;43:D682-9。doi:10.1093 / nar / gku1112.
- 60.
Zuker M. Mfold用于核酸折叠和杂交预测的web服务器。核酸Res. 2003; 31:3406-15。
致谢
我们感谢Scott Poethig博士35 s:: AtMIR156a种子;Peter Huijser博士spl9; spl15种子;Renato Vicentini博士进行初步的生物信息分析和有益的讨论;和路易斯·德尔·本博士进行初步的系统发育分析。这项工作得到了巴西圣保罗州研究基金会(资助号:FAPESP)的支持。07/58289-5和12/51146-2)。该中心获得了高等教育人员改进协调中心(巴西cape)的研究金。GFFS(来自Centro de Energia Nuclear na agriculture -CENA /USP)和EMS获得了巴西圣保罗州研究基金会的奖学金。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明他们没有竞争利益。
作者的贡献
EGOM、EMS、GFFS和CHBR进行了分子生物学研究并对数据进行了分析;GTV和MV进行了生物信息学和系统发育分析;FTSN设计和协调了研究,FTSN和MV撰写了手稿。所有作者阅读并批准了最终稿件。
附加文件
附加文件1:
预测SBP / SPL含有mir156 /529响应元件的桉树、单子叶和苔藓植物的靶标。(PDF 11kb)
附加文件2:
估计的Ka/Ks比率SPL来自不同物种的基因。(xls33 kb)
附加文件3:
无根系统发育树和序列比对描述了两者之间的关系MIR156而且MIR529来自代表性物种的前体。(一)可用MIR156而且MIR529前体序列检索自miRbase v. 21 (http://www.mirbase.org/).系统发育分析采用最大似然自举分析(1000棵树)。相关类群聚集在一起的树的百分比(超过50%)显示在分支点的旁边。长沙,拟南芥;aqc,耧斗菜coerulea;bdi,Brachypodium distachyon;阻塞性睡眠呼吸暂停综合症,栽培稻;ppt,Physcomitrella金属盘;印度国家银行,高粱二色的;zma,玉米.(B)序列对齐MIR529而且MIR156体细胞。艾尔,拟南芥lyrata.对齐使用ClustalW [50].哈宾结构预测MIR529而且MIR156前体估计采用MFOLD3.2算法[60].(PDF 569 kb)
附加文件4:
共音块的守恒。(A)含有水稻pre-miR529a/b邻近基因同源物的基因组区域。(B)包含同源物的基因组区域耧斗菜pre-miR529邻近基因。(C)拟南芥和葡萄pre-miR156h邻近基因同源的基因组区。在不同物种中具有相同颜色的箭头表示同源基因。灰色和黑色线的箭头如果它们共享相同的颜色,则表示谬误,尽管不是所有的黑色线箭头都表示正序(请参阅genome icus手册;http://www.dyogen.ens.fr/genomicus).一些箭头上方的描述表明了基因座的名称和在特定基因组中的位置;*, pre-miRNAs区域;": ",位点未显示。这个符号的数量表示基因座的数量;" // ",未显示大基因组块。被方框包围的共线箭头表示保守的共线块。(PDF 458kb)
附加文件5:
削减和RNA-seq水稻数据MIR156而且MIR529体细胞。(PDF 560kb)
附加文件6:
本研究中使用的寡核苷酸序列。(PDF 84kb)
附加文件7:
所选SPL核苷酸序列的多序列比对。(PDF 84kb)
权利与权限
开放获取本文根据创作共用署名4.0国际许可协议发布(http://creativecommons.org/licenses/by/4.0/),该协议允许在任何媒体上不受限制地使用、分发和复制,前提是您适当地注明原作者和来源,提供创作共用许可的链接,并说明是否有更改。创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条提供的资料。
关于本文
引用本文
Morea, E.G.O, da Silva, e.m., e Silva, gfet al。陆生植物miR156和miR529家族的功能和进化分析植物生物学16日,40(2016)。https://doi.org/10.1186/s12870-016-0716-5
收到了:
接受:
发表:
关键字
- miR156
- miR529
- 进化
- 拟南芥
- Eudicots