抽象的
背景
埃拉格罗斯特TEF.为异源四倍体(2n = 4 × = 40),一年生C4草,核基因组大小估计为730 Mbp。它在埃塞俄比亚被广泛种植,为该国一半以上的人口提供基本营养。
虽然草案大会E. TEF.基因组于2014年公布,其重复部分的特性E. TEF.基因组还没有被详细分析过。
在真核生物基因组中,大部分DNA都是重复序列。转座因子通常是植物基因组中最丰富的重复成分。它们有助于基因组大小的变化,导致突变,可以导致染色体重排,并影响基因调控。广泛而深入地描述重复成分对理解基因组的进化和功能至关重要。
结果
利用新的对端序列数据和一种从头重复识别策略,我们识别了中重复次数最多的元素E. TEF.基因组。假定的重复序列是根据与其他草中已知重复组的相似性进行注释的。
我们共鉴定了1389个中/高重复序列,这些序列约占苔草基因组的27%。在一个包括水稻和玉米相似元素的比较框架中,对最重要的te类进行了系统发育分析。最后,我们发现了一个丰富的串联重复序列,占整个基因组的4%以上。
结论
通过对大样本随机剪切读片的分析,得到了重复序列库E. TEF..我们使用的方法旨在避免对重复贡献的低估;这种低估是全基因组组装项目的特点。收集到的数据为进一步分析这种重要的孤儿作物的基因组提供了宝贵的资源。
背景
真核生物基因组显示出尖锐的尺寸变化。变异与生物体的生物复杂性无关;实际上,基因含量在不同的物种上仍然非常相似。这种现象已被描述为“C-Value悖论“其中1C DNA值是配子中DNA的量[1].基因组大小的变化在植物中是非常明显的,跨越至少三个数量级的1C DNA含量基因组Genslisea margaretae(58.68 Mb) [2]和1C DNA含量巴黎japonica(148648 Mb) [3.].有趣的是,多倍性在C价值悖论。”植物基因组大小的大多数变异是基于重复序列内容的差异[4].
重复序列包括:串联排列的卫星序列,端粒序列,微卫星序列,核糖体基因和转换元素(TES)[5].TES,也称为转座子或移动元素,是在几乎所有生物体中发现的DNA序列,并且能够复制和移动到宿主基因组的不同部分[6].根据转座子和/或作为中间体的分子所采用的机制,它们在层次上分为两大类:一类(或RNA转座子或逆转录转座子)和二类(DNA转座子)。I类te使用RNA作为复制的中间分子,并通过“复制和粘贴”机制移动。另一方面,II类元素并不利用RNA中间体,而是使用“剪切和粘贴”机制来移动[7,8].
TEs散布在整个基因组中,在很大程度上影响了植物基因组的大小变化。例如,不同水稻品种的总TE含量从25%到66%不等[9].高粱中TE含量为61% [10,超过85%的玉米[11, 95%在面包小麦中[12].在密切相关的生物体之间,TEs的含量可能有很大的差异。一个显著的例子是选用australiensis由于在不到300万年的进化过程中不断重复扩增,它的基因组大小几乎翻了一番[13].
TES的运动和扩增可能导致突变[14],产生染色体重排[15]影响基因调节[16,17并促进外显子改组[18,19].TE序列可以由宿主基因组共同选择,在一个名为Exactation的过程中,获取新的和潜在的有益职能[20.,21].在系统发育和种群研究中,TEs也是可行的工具[22,它们被用作遗传标记的来源[23- - - - - -25].由于TE扩增会对宿主基因组产生有害影响,这些元件通常处于严格控制之下。事实上,大多数TEs因突变或表观遗传机制(包括DNA和组蛋白甲基化以及小干扰RNA (siRNA)活性)而失活或沉默[26,27].植物主要通过两种导致TE相关序列部分去除的机制来抵消TE扩增导致的基因组扩展:不平等重组和非法重组[28,29].
TEs的存在使基因组组装过程复杂化[30.]并导致基因注释中的困难[31].因此,对重复DNA的识别已成为基因组注释的重要组成部分[22].
我们的研究侧重于Teff重复分数的表征(Eragrostis微软简历Enatite基因组。属Eragrostis是草族Poaceae(禾本科)的一部分[32],共350种,其中约69%为多倍体,从二倍体(2n = 2 ×= 20)到六倍体(2n = 6×= 60) [33].E. TEF.为异源四倍体(2n = 4 × = 40),估计核基因组大小为730 Mbp [34,其大小与二倍体高粱大致相同,比二倍体水稻基因组大60%左右。e .微软目前是一个C4年度草[35]在埃塞俄比亚广泛种植和很好地适应,在那里提供超过一半人口的基本营养[36].然而,有许多限制,例如低生产力和住宿[37,38]仍然影响Teff生产,需要解决以提高总产量。
大会草案E. TEF.《基因组》于2014年发布[36].然而,与其他主要谷物相比,它们的许多基因组特征E. TEF.仍然很差。特别是重复组分迄今为止仅被略微调查。
为了收集tef基因组中中等/高重复部分的代表性样本,采用从头识别策略对随机剪切reads的大数据集进行分析。然后进行相似性和结构特征搜索,以更好地了解重复成分。共分离到1389个不同的中/高重复序列。总的来说,这个文库代表了大约27%的画眉草基因组。利用水稻和玉米的TE同源分析,在一个比较框架中对最重要的TE类进行了系统发育分析。我们鉴定并部分表征了一个丰富的串联重复序列,该序列占整个画眉草基因组的4%以上。
结果
50万对端读取代表0.25×覆盖E. TEF.基因组的分析使用了RepeatScout [39],一项经过验证的程序,在De Novo识别重复上。使用CAP3组装读入共识序列[40],使用cd-hit将一致序列聚类为重复组[41.].总共,两组总计184,986 BP,其对应于估计的〜0.25×覆盖率E. TEF.基因组(即730 MBP)。基因组的这种覆盖率大于几种低通测序分析中使用的那些,这些分析已经用于捕获和表征基因组的培养基/高度重复分数[42.- - - - - -44.].
重复库 - 组成和表征
一组1,389个不同的介质/高度重复序列(库Etef_repeats_V1.4)(附加文件1)被确定在E. TEF.基因组。相似性搜索和结构特征分析用于更好地表征这些序列。重复文库中最代表的TE类是长终端重复逆转录(LTR-RT)占31.82%的条目。特别是Ty1-Copia和Ty3-Gypsy元素分别代表了图书馆的12.17%和16.99%。鉴定的LTR-RT序列的少量(2.66%)并不与两个超级美食中的任何一种令人信服。另一个1.80%的孤立的重复序列与非LTR逆向相似。II类DNA元素序列代表重复文库的9.14%。诸如重复数据集中仅占0.5%的表示。大约1%的序列与其他类别的TES或重复序列相关。最后,55.51%的重复序列在相似性搜索的基础上没有明显与任何TE级相关联(表1).
为了计算不同重复的相对丰度E. TEF.基因组中,25万个随机剪切序列的子集,平均长度为367 bp,使用RepeatMasker [45.],并使用Etef_repeats_V1.4库作为参考。总共,Etef_repeats_V1.4库屏蔽了27.46%的随机剪切序列集。最具代表性的TE类是LTR-RTs,占14.96%。Ty3 -吉普赛superfamily比Ty1-更为丰富copia:11.40%与2.67%。重复类似于LTR-RTS,但不可分类为两个子属中的任何一个屏蔽0.89%的数据集。非LTR回收转换占0.12%,值类似于在许多植物基因组中观察到的值。II类DNA元素,包括螨虫,占基因组的2.33%。单独的单一重复序列似乎存在于TEFF基因组中的大拷贝数中,覆盖4.54%的采样序列集。当使用DOT绘图比较和串联重复发现的结构特征分析了图书馆中该序列的三个拷贝时,串联重复发现[46.,一个串联安排被清楚地识别(附加文件2).为了更好地描述这个序列,我们进一步测试了这个假设(见小节:丰富的卫星序列).
评估图书馆的完整性
将Etef_repeats_V1.4库与随机生成的库进行比较E. TEF.使用RepArk工具读取[47.],tedna [48.]和重复资源管理器[49.].用Etef_repeats_V1.4文库对RepArk分离到的1091个重复序列进行掩码,该文库对候选序列的掩码率为56.54%。通过相似性搜索,剩余43.46%的序列被鉴定为质体、核糖体和细菌污染物。另一方面,RepArk候选者只屏蔽了29.33%的Etef_repeats_V1.4重复库。因此,RepArk似乎错过了大多数重复,而没有捕获任何新的重复。同样,在对TeDNA输出(306个序列)进行相同分析时,Etef_repeats_V1.4掩盖了55.83%的候选TeDNA,其余为质体污染物。TeDNA输出仅掩盖了29.55%的Etef_repeats_V1.4。最终,Etef_repeats_V1.4掩盖了Repeat Explorer中最丰富的200个簇的2722个序列中的87.11%。暴露的候选基因由质体序列、基因家族束和其他污染物代表。RepeatExplorer库屏蔽了78.24%的ettef_repeats_v1.4。 Altogether these data suggest that the library Etef_repeats_V1.4 is highly representative, i.e., RepeatScout was able to collect most repeats from a given dataset (Table1).
系统发育分析
从250,000次随机剪切的子样本检索来自逆转录酶(RT)的逆转录酶(RT)编码结构域的副蛋白酶。E. TEF.序列。来自玉米和米饭中最丰富和研究的LTR-RT元素的ParaLog元素从公共数据库麦德布中开采(http://maizetedb.org/~maize/),Retractza [50.]和repbase [51.].
收集的数据然后对齐(附加文件3.,4和5),并使用邻居连接(neighbor-joining, NJ)方法构建系统发生树,并计算1000个重复的bootstrap值。
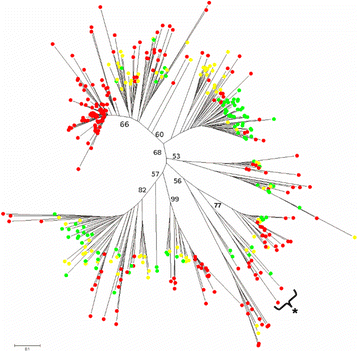
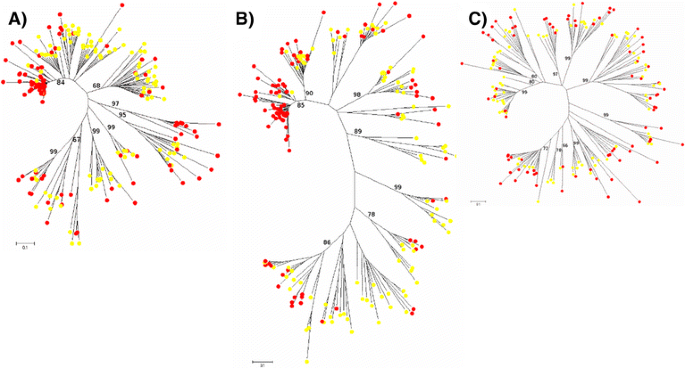
以Ty1-为例copia分析了385个旁边蛋白酶尸体:215从Teff,93来自水稻,77来自玉米(图。1).
假设玉蜀黍属和选用Genera分叉5500万年前(Mya)[52.,53.]系统发育距离分离玉蜀黍属和EragrostisGenera估计为36.47(20.64-50.54)mya [54.].
在大多数Bootstrap支持的图例中,来自三种不同物种的元素混合在一起。然而,有一个具有高自举支持的单个思考,包括85个Teff Paralogs(占所使用的截面总量的39.5%),可能代表Teff特定Ty1-Copia家族。
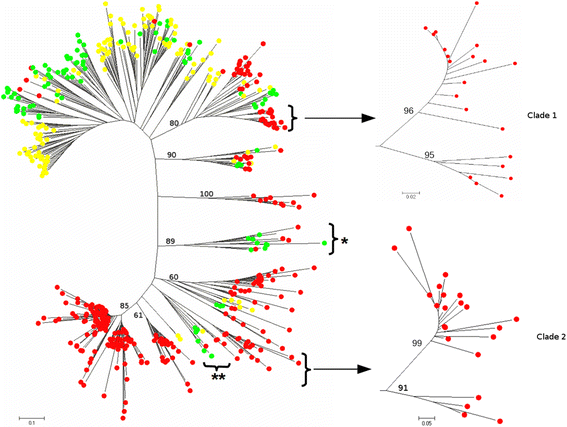
以Ty3-为例吉普赛分析了515种元素的同源性:从画眉草中提取的295种,从水稻中提取的97种,从玉米中提取的123种。这一场景与描述Ty1-copia与大部分teff Ty3-的场景非常不同吉普赛在特定的物种进化枝中,副对数崩溃。一个单独的画眉草分支包含了该物种使用的295个分支中的162个分支(54.9%)。另一方面,混合演化支只包括一小部分的伪演化支。含有高度丰富的栽培稻Ty3-gypsy元素Atlantys [55.]及RIRE2 [56.],以及含有丰富的Ty1-copia家族RIRE1 [13,只包括了数量有限的E. TEF.这表明与这些科相关的元素存在,但在画眉草中并不丰富。在Ty3-gypsy NJ树中,鉴定了两个teff特定分支,每个分支都包含两个独立的亚分支,都具有高bootstrap支持(图2)。2).这是唯一一个在Ty1-copia和Ty3-gypsy树中发现的具有这种特征的分支。
E. TEF.可能从野生的同种异体情况逐渐发展E. Pilosa.[57.].祖先的E. Pilosa.不知道,然而,估计了所有聚宝单化事件发生了4个[36]最高达6.4米亚[54.].人们很容易猜测,在E. TEF.包括来自两个不同群体的副蛋白,从非常相同的LTR-RT系列,殖民化了两个基因组对应物E. Pilosa.基因组。假设是古代LTR-RT家族分别演变为两种贡献基因组E. Pilosa..在Allotetraploid.E. Pilosa.,两个LTR-RT种群继续分别进化。
我们分析了两个演化支的序列资料。进化枝1包括21个分支:在亚进化枝A和亚进化枝B中分别有15和6个分支(附加文件)6A,7).分支2包括22个分支:分支1中有18个分支,分支2中有4个分支6B.,8).在核苷酸水平上比较亚枝A和亚枝B的所有同源序列,分别比较枝1和枝2的同源序列,以估计每对同源序列之间的核苷酸距离。根据San Miguel等人所描述的分子古生物学策略,这些距离被转换成数百万年。58.]使用6.5×10的替代率−8以米计算[29].插入时间估计为9至32个Mya,分别为32 mya和14至26个Mya,用于片状1和2。这种有限的证据似乎支持这两个LTR-RT人群在此之前分裂得很好E. Pilosa.来源。然而,缺乏具体数据的祖先E. Pilosa.,以及它们与共同祖先分离的时间,以及无法获得所有这些物种的广泛基因组序列数据,极大地限制了进一步验证这一假设的可能性。
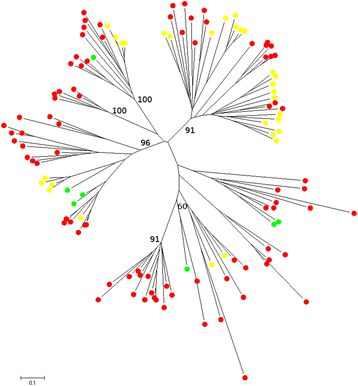
对于非ltr反转录元素,我们鉴定并分析了123个谬误:86个来自E. TEF.其中,大米7块,玉米30块。大约有一半的画眉草与水稻和玉米的混合,反映出这些元素大部分是古老的,并且在三种物种之间共享,尽管在物种形成后出现了一定数量的增殖(图)。3.).
系统发育分析扩展到3个最具代表性的DNA TEs组:CACTA, MuDR和hAT。分析了3种植物中CACTA和MuDR元素的转座酶结构域以及hAT元素的二聚结构域的类似域。(附加文件9,10和11然后用来建立新泽西州的系统发育树。
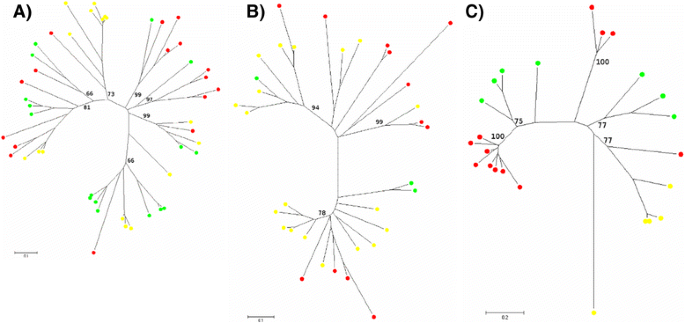
48个CACTA模拟图(画眉草、水稻和玉米各16份)和34个hat样图(画眉草12份、玉米19份和水稻3份)显示出相似的模式(图2)。4A和b)与之前描述的非ltr反转录元素(图。3.).相反,12个中的大部分E. TEF.MuDR类似于单独聚集在物种特异性的高度自举支持的支系中,因此表明了这组te在苔草中最近的活性和分化(图。4C).
我们利用了另一个E. TEF.品种(Tsedy)分析Ty1-Copia,Ty3-Gypsy和非LTR复古 - 在两种品种中的遗传学关系。对于三个TE类中的每一个,从所识别的副病剂rt的总量,我们随机地检索了每个Tsedey和enalite品种的拷贝。序列被对齐(附加文件12,13和14),并用于构建NJ系统发育树。对于Ty1-copia和Ty3-gypsy来说,大部分的拟对数混合在一起,表明导致现有拷贝产生的活性主要发生在两个品种分离之前(图3)。5A和b).然而,鉴定了一些种类的特异性曲线,可能表明近期两种品种的差异TE活性。如果这些特定的曲线代表实际演进的事件,则应假设在品种选择后某些LTR-RT家族的选择性增殖。然而,在这种情况下,Paralogs将表现出极短的分支反映最近和快速放大的分支。由于这种情况似乎没有这种情况,最可能的解释是证据是造成的,并且可能是由于组装序列(即品种Tsedy)中少量LTR-RT亚群的选择性取样。在非LTR逆转带的情况下,几乎所有的曲线都包括来自两个品种的伞菌(图。5C).
丰富的卫星序列
一个串联排列的卫星序列被鉴定为最丰富的重复序列之一E. TEF.基因组。我们从随机剪切读数据集中挖掘出该重复序列的代表性单体。在25万次搜索中,使用RepeatMasker获得了26,595次正命中[45.].其中一千个命中,每个都代表完整的卫星单体,从总量随机提取并用于进一步分析(附加文件15).由Tandem Repeat Finder软件识别的共识单体的长度[46.],是169 bp。单体长度范围为163至177 bp。平均GC含量为:45.21%。当它用于搜索植物卫星序列的综合数据库时,单体的共识序列没有提供任何显着的命中术语59.].1000份随机拷贝之间的总体相似性为79%。然而,超过一半的副本(554份)与随机数据集中至少另一个副本的相似性大于94%。通过分析1000个单体副本来创建一个共识标志(附加文件),研究了单体序列的保守差异16).共识标志是序列的图形表示,其中每个残基的高度反映了其在分析的序列复制的那个位置的守恒[60.].在整个序列中,保护是相当明显的。假设基因组大小为730 Mbp,单体平均长度为169 bp,估计整个基因组的总体丰度(即4.54%)意味着拷贝数大于196,000。
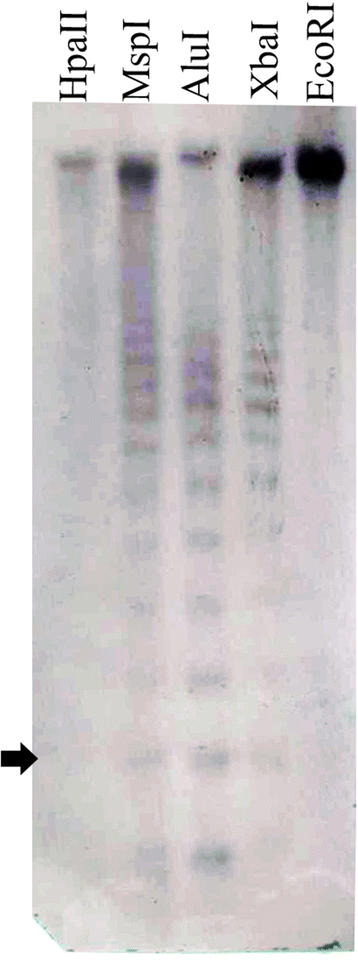
相似性搜索还检测到Teff Teedey的组装支架中的该顺序。由于预期,支架中该序列的总体量非常减少(几百份),因为基因组的卫星富株地区非常难以组装。然而,使用卫星序列的原始Illumina读取的随机样品(来自Teff图书馆Gyn 7,SRR146355)作为查询掩蔽的2.89%核苷酸。该图与计算CV计算的图一致。enalite。为了进一步检查这种卫星序列的特征,以确认从硅分析中获得的证据,并排除由于图书馆建设而导致的任何可能的艺术发现[61.或排序问题,进行了南方印迹杂交实验。使用了五种不同的限制性酶。其中四个(XBA.我,运算器我,MSP.I, HpaII)识别分析序列内的一个限制性位点,一个不:生态RI。杂交产生的信号相当强烈,证实了这个序列是丰富的。此外,所有限制性内切酶(后面讨论的例外)下丘脑-垂体-肾上腺轴的(2)在卫星序列中存在一个限制位点,产生了预期的“梯状”模式,从而确定了该序列的串联式排列(图2)。6).MSP.我和下丘脑-垂体-肾上腺轴的II是两个识别序列5 ' -CCGG-3 '的等裂体。下丘脑-垂体-肾上腺轴的II对两个胞嘧啶中的任何一个的甲基化都很敏感MSP.I只对外部的甲基化敏感。杂化模式MSP.我和下丘脑-垂体-肾上腺轴的二、表现出较大差异。特别是MSP.我消化了一个透明的阶梯,下丘脑-垂体-肾上腺轴的II不表明靶序列内胞嘧啶的甲基化程度较高。然而,两种酶解在高分子量范围内也表现出强烈的信号,表明外部citosine发生了一些甲基化。综上所述,这些结果表明这个重复序列有一定程度的甲基化。
讨论
随机剪切序列的分析假设代表一个无偏样本的基因组是一个很好的实践,用于评估基因组的重复内容。这种方法规避了与全基因组装配中重复的偏置表现相关的大多数限制[49.,62.- - - - - -64.].众所周知,重复序列对基因组组装构成了严重的技术挑战[65.].以及错误装配和基因错误注释[31[最常见和预期的艺术成果之一是最终基因组组件中重复的总体耗尽,从而严重低估了这类序列的总体量。由于这些原因,为了识别,分析和表征基因组成分e .微软我们通过使用重复求解,通过采用DE Novo策略,分析了覆盖整个基因组的约0.25倍的500,000次读取的随机子集[39].因此,我们鉴定了1,389次患者培养基/高度重复序列。我们估计,所有这些都掩盖了超过27%的基因组。这个值远远大于Teff中的上一次估计约14%的重复内容[36基于对现有基因组组装的分析。
随着我们的策略,我们测试了三种利用下一代序列数据的其他工具:Repark,TEDNA和Reptepexplorer。我们采用的策略优于这些工具中的两种(Repark和Tedna),并与重复开发者进行比较。然而,无论使用所用的特定工具如何,DE Novo识别方法需要相当于分离的重复候选的准确表征。特别地,通过性质重复但与TES或卫星重复类似的所有序列,例如基因家族,核糖体序列,低复杂性序列和塑性污染物的卫星重复,需要鉴定和除去并除去并除去。另一个缺点是鉴定的大多数重复不完整,从而导致共有共有序列的严重碎片[47.].
大约三分之一的重复鉴定(442)与LTR-RTS相关的LTR-RT,其代表TEFF基因组中的大部分TE分数如几种植物中的情况[66.].总的来说,LTR-RTs估计约占画眉草基因组的15%。考虑到相似大小的植物基因组,这个值与在猕猴桃对(7.58亿bp中占13.4%;[67.])和vitis Vinifera(4.87亿美元中占14.32%;[68.]但它比在番茄中计算的小得多(460 MB中的62%; [69.]和马铃薯(311 MB的53%; [70])。正如预期的那样,它比玉米等大型基因组的估计要小得多(>在2,300 Mbp中占75%;[11]),大麦(5,100 Mbp的76%; [71.)和挪威云杉(约占20英镑的60%;[72.])。
两种可能的原因,其中之一,明显不足的代表LTR-RTsE. TEF.与类似大小的基因组相比,存在几个高度分化的元件和/或一个丰富的单拷贝或低拷贝LTR-RTs群体。这两种解释并不相互排斥,但在这两种情况下,这些元素都不会被从头搜索发现[73.].Ty3-gypsy超科似乎比Ty1-copia丰富得多(11.40% vs 2.67%)选用属[9],玉米[74.),毛花瓣[75.].我们无法确定这种不平衡的分布是由于属于两个超科的元素的拷贝数不同,还是由于Ty3-gypsy元素的平均长度较长,因为使用的重复库中没有LTR-RTs的完整拷贝,只有部分拷贝。然而,如果用所鉴定的RT束数作为元素丰度的代表,则copia与gypsy的比例仅为1:1.33,远低于用掩蔽碱基量计算的1:4的不平衡值。
这表明,吉普赛元素数量的增加不仅可以用绝对拷贝数来解释,还可以考虑到几种植物基因组中描述的这些元素的较长长度。例如,在水稻中Ty1-copia和Ty3-gypsy元件的平均长度分别为6.2 kb和11.7 kb [76.].在棉花中,TY3-GYPSY平均长度为9.7 kbp,而对于TY-1 Copia元素,它是5.3 kbp [77.,78.].在亚麻(Linus Usatissimum.Ty1-copia元素平均长度为5.3 kb, Ty3-gypsy元素平均长度为8.7 Kbp [79.].尽管当考虑二十多个最丰富的LTR-RT系列时,玉米LTR-RTS没有提供平均值,但TY3-GYPSY元素通常比TY1-Copia更长74.].也有可能是非自主元素的存在导致Ty3-gypsy过量。其他I类te的代表不足:正弦和非ltr逆转录转座体分别只占基因组的0.18%和0.12%。这些结果与在许多植物基因组中收集到的证据一致[80].II类元素总共占画苔草基因组的2.33%,比许多其他谷类作物,如水稻(12.96%,[81.]),毛花瓣(4.77%, (75.]),高粱bicholor(7.46%, (10])和玉米(8.6%,[11])。大部分的重复序列库由“非特征重复序列”(771)组成,这些重复序列可能代表了LTR-RTs中高度分化的te或几乎不保守的区域,如ltr。在相似度搜索中,这些区域显然都没有被发现。无论如何,这一大部分基因库只掩盖了4.44%的基因组。一个以前未被发现的类似卫星的序列被确定并部分描述。它覆盖了总基因组大小的4%以上,它的拷贝数在几十万个量级。单体的平均长度为169 bp,接近于PlantSatDB中最常见的植物卫星序列长度:165 bp [59.].然而,与PlantingAtdB中的任何条目未检测到序列级别的显着相似性。这并不奇怪,因为即使在密切相关的物种之间,这些序列也显示出很大的变化[82.,83.].高拷贝数,单体的长度和该序列的串联布置表明它可能发挥作用作为Centromere组分。然而,这一结论是以迄今为止收集的数据的基础而无法达成。需要进一步的研究和细胞遗传学分析以更好地评估沿着TEFF基因组的卫星序列分布,并推断其结构和功能作用。这种卫星序列虽然在Teff组装的支架中耗尽,但是当分析来自该品种的原始序列时,被证明在Teff Tsedy品种中被大量存在。
我们对不同TE课程之间的系统发育关系进行了广泛的研究E. TEF..采用了一种比较方法,将分析扩展到另外两种草:水稻和玉米。在LTR-RT Ty1-copia元素中,有有趣的证据表明存在各种高度自举支持的枝,包括来自所有三个物种的元素。水平转移(HT)可能是数千万年前彼此分离的物种的类似TE拷贝之间如此密切相关的原因。事实上,在植物王国中,HT已被证明比之前认为的更为普遍[84.].另一种但不是互斥的解释是Ty1的更明显的保护 -copia元素在漫长的进化时间尺度上。事实上,这已被证明适用于各种Ty1-copia家族,如Angela/Martians [85.]和TVV1 [86.]在Agiosperms和Partc中的裸子植物,这个家庭的元素显示出超过2亿年的进化中的引人注目的保护[87.].
Ty3-gypsy parogs主要根据其分离的种类进行分离。这可能反映了这个超级家族的保护程度较低。然而Ty1-copia谬误比Ty3-gypsy谬误表现出更大的异质性。事实上,在Ty3-gypsy超科中,超过一半的相似序列被分析成一个分支。系统发育分析表明,两个LTR-RT超科都存在丰富的teff特异性分支,包括大部分Ty1-copia RT分支和大部分Ty3-gypsy分支。这些发现表明,teff特异的LTR-RT元件的存在,大多数在最近的进化时期增殖,可能是多倍体化后(即在最近4-6.4 mya [36,54.])。这可能是“基因组休克”的效果[88.触发的多倍体化导致苔草物种形成。
LTR-RT相关元素丰富选用LTR-RT家庭atlantys [55.], RIRE2 [56.[reire1 [13]几乎没有代表Teff,因此再次展示了与不同物种中的尖锐不同的速率相比能够引起密切相关的元素[13,78.].
结论
我们的深入分析随机剪切序列数据集从teff cv。Enantite使我们获得了包含1,389个中/高重复序列的综合文库,占该基因组的27%以上。通过利用全基因组猎枪序列数据来识别重复成分,我们的方法克服了从头组装基因组中重复损耗的严重局限性。我们的结果提供了深入了解该物种的TEs动力学和进化历史,以及丰富的卫星序列的详细特征。我们相信,我们的数据为进一步分析这种重要的孤儿作物的基因组提供了有价值的资源。
方法
植物材料和DNA提取
埃拉格罗斯特TEF.var Enatite (accession PI 524439;美国农业部农业研究服务处种质资源信息网络(http://www.ars-grin.gov/npgs/).从生长室生长的五种植物的幼苗在种植两周后收集,并通过使用液氮研磨砂浆和杵。使用基因株植物基因组DNA MiniPREP(Sigma Aldrich)提取基因组DNA。用DEPC水而不是方案洗脱溶液进行最终洗脱。根据标准程序,对分离的DNA进行进一步的酚类纯化和乙醇沉淀。最后,通过在1%的凝胶中使用分光光度计和电泳来检查质量。在调度以进行测序之前,将DNA样品保持在-20℃。
建库、DNA质量检测、测序、组装
文库根据Nextera DNA样品制备指南(Nextera DNA sample Prep Kit 96 sample-ref 15028211)制作,修改如下:
基因组DNA片段后,使用认证的低范围超琼脂酶bio - rad(目录161-3107)进行凝胶提取(片段选择范围在300-700 bp);
使用QIAquick凝胶提取试剂盒(cat.28704) Qiagen对片段DNA进行清理
PCR扩增:进行7个循环而不是5。
使用Agilent Technologies 2100 Bioanalyzer和高灵敏度DNA芯片进行DNA质量控制。
使用MiSeq试剂试剂盒v3(600循环)cat进行测序。ms - 102 - 3003 Illumina公司。试剂试剂盒可在MiSeq系统上进行多达625个循环的测序,包括配对端试剂板(600循环)、MiSeq流动池和洗涤缓冲液。
使用MiSeq平台测序的原始DNA序列对对末端读取的两个文库(每端300bp)合并使用梨[89.].
重复识别
将两组250,000次读出每(XAA和XAB)被随机选择由梨合并的序列总量的总量,并且用于De Novo重复鉴定和表征。使用的策略有三个步骤:
- 一)
RepeatScout [39,使用默认参数分别在两个集合上运行,以识别任何长度超过100 bp、存在于10个以上副本且不低复杂度的重复序列。
- b)
由于RepeatScout是为组装的基因组或至少是长序列量身定制的,因此预计通过分析短读获得的输出将是高度碎片化的。为了进一步组装,如果可能的话,识别出重复的候选序列,并产生更长的一致序列,使用CAP3分别对两个输出进行处理[40在轻松设置下运行(−o 30-p 80-s 500)。
- c)
从b)中获得的重复一致序列,然后使用cd-hit进行分析[41.]将所有序列倒在一起,共享至少80%的相似性。
为了测试该策略在捕获基因组的介质/高度重复部分方面的有效性,将结果与使用RepeatExplorer获得的结果进行了比较[49.], TEDna [48.]及RepArk [47.使用默认设置。
Repeatexplorer被送入1,000,000梨组装读数的数据集。总体结果包括42,045个序列。仅使用含有最代表序列(2,722)的两种簇进行进一步分析(即,排除了低拷贝数重复)。
Repark在500,000次序列上运行,并产生1,019份重复候选人的产出。
TeDNA被用于分析两批25万个读取数据,每批输出包含306个重复的候选数据。
图书馆的特征
在相似度搜索和序列结构特征分析的基础上,对重复序列进行表征。特别是:
- 一)
将推定的重复序列与核苷酸和氨基酸水平进行比较,其中所有植物序列包括在Rebaps中[51.]使用爆炸[90.]并将1E-5的电子值设置为阈值以识别重要的命中。
- b)
然后将没有提供任何显著命中的序列与GenBank的nr分区进行比较[91.]在相同条件下使用BLAST搜索工具)。从数据集中除去具有塑性序列(线粒体和叶绿体)或具有已知基因家族的相似性的序列。相应地注释具有已知TES显着的次数的序列,并且没有命中术语“NHF”即“NHF”的序列。“没有找到命中”。后者是重复序列尚未完全表征的重复序列。
- c)
然后对重复文库进行进一步分析,以确定任何含有长度超过100 nt重复单体的串联排列基序的序列。这项分析使用串联重复查找器[46.].
系统发育分析
来自Ty1-的逆转录酶(RT)结构域的100个氨基酸残基束copiaTy3 -吉普赛以及CACTA和MuDR元件的转座酶结构域和hAT元件的二聚结构域的非ltr反转录元件和100个aa残基长束(Additional file)17),作为TblastN搜索中针对25万次读取数据集xaa的查询。
所有的匹配都带有anE-value低于1e-05并且覆盖查询序列的至少80个aa被保留。从水稻和玉米中鉴定的最丰富和最具代表性的LTR-RTs序列中检索到Repbase [51.],Retratoryza [50.]及MaizeTEDB (http://maizetedb.org/~maize/)并添加到Teff DataSet。然后使用肌肉单独对齐所有副病虫潮[92.].然后使用MEGA版本6的多重对齐来构建NJ树[93.],并计算1000次重复后的bootstrap值。
从teff品种Tsedey的基因组中也发现了LTR-RTs和非ltr反转录元件保守的RT片段[36[然后沿着Teff,CV对齐。enalite鹦鹉以建立NJ树。
使用EMBOSS的“distmat”计算核苷酸距离[94.],应用Kimura 2参数模型[95.].
序列标识
的卫星序列的标志是使用网络标志制作的[60.].
印迹杂交
DNA从E. TEF.在“植物材料和DNA提取”中描述的生长的种子。对于每个酶促反应,分别用以下限制性内切酶酶切5 μg DNA:XBA.我(R0145S;新英格兰Biolabs),生态RI (R0101S;新英格兰生物学实验室),下丘脑-垂体-肾上腺轴的II(R0171S;新英格兰Biolabs),MSP.我(R0106S;新英格兰生物实验室)和运算器我(R0137S;新英格兰生物实验室),遵循制造商的协议。
通过使用PCR反应和向前(5'-CGG-TTA-TTT-CTG-TTT-TTC-TTC-GG-TTC-GG-3')分离靶卫星序列来制备DNA探针,并反向(5'-TGA-CCA-GTC-TGC-AGC-AAA-AC-3')专为此目的而设计。使用奇柱SV凝胶和PCR清理系统(PRomega)提取预期的扩增条带和纯化。然后在1:200中稀释,并使用Dig-11-DUTP标记(Roche)通过聚合酶链反应(PCR)来标记反应。
将消化物在1.5%琼脂糖凝胶上运行2小时,冷却0.5×Tbe缓冲液。然后将凝胶浸泡10分钟以使凝胶在紫外光下可视化凝胶。将DNA转移到带正电荷的尼龙膜(Roche)中。使用NBT / BCIP(挖掘高素DNA标记和roche)比色度检测系统用于可视化膜上的杂交。
数据和材料的可用性
本研究使用的原始序列数据提交至GenBank,生物工程登录号为PRJNA294641。支持本研究结论的系统发育和序列分析相关数据集包括在文章中,并在“附加文件”部分列出。
缩写
- aa:
-
氨基酸
- 英国石油公司:
-
基对
- LTR:
-
长末端重复
- LTR-RT:
-
长末端重复反转录元件
- NJ:
-
neighbor-joining
- NT:
-
核苷酸
- RT:
-
逆转录酶
- te:
-
转座的元素
参考
- 1.
染色体的遗传组织。《Annu Rev Genet》1971;5:237-56。
- 2.
在Lentibulariaceae中发现的最小被子植物基因组,染色体大小为细菌大小。植物医学杂志。2006;8:770-7。
- 3.
Pellicer J,Fay MF,Leitch IJ。他们全部最大的真核基因组?Bot J Linn Soc。2010; 164:10-5。
- 4.
Kidwell毫克。真核生物中转座元件与基因组大小的进化。遗传。2002;115:49 - 63。
- 5。
Mehrotra S,Goyal V.植物核DNA中的重复序列:类型,分布,进化和功能。基因组学蛋白质组学生物信息学。2014; 12:164-71。
- 6。
Miller WJ,Capy P.移动遗传元素作为基因组进化的自然工具。在:Miller WJ,Capy P,编辑。分子生物学的方法,移动遗传元件。第260卷.ToWa,NJ:Humana媒体公司;2004. p。1-20。
- 7。
芬尼根DJ。真核转座元件与基因组进化。趋势麝猫。1989;5:103-7。
- 8。
刘志强,刘志强,刘志强,等。真核生物转座分子的统一分类系统。(1)中国科学(d辑:地球科学)2007;
- 9。
朱科龙,陈志强,陈志强,等。水稻属转座元件的分布、丰度及其在基因组大小变异中的作用。BMC Evol Biol. 2007;7:152。
- 10.
Paterson AH, Bowers JE, Bruggmann R, Dubchak I, Grimwood J, Gundlach H, et al.;高粱双色基因组与禾本科植物的多样性。大自然。2009;457:551-6。
- 11.
Schnable PS,Ware D,Fulton Rs,Stein JC,Wei F,Pasternak S等人。B73玉米基因组:复杂性,多样性和动态。科学。2009; 326:1112-5。
- 12.
Bennetzen杰。转座因子对植物基因和基因组进化的贡献。植物学报。2000;42:251-69。
- 13.
张志强,张志强,张志强,等。在不进行多倍体化的情况下加倍基因组大小:水稻野生亲缘种澳大利亚稻(Oryza australiensis)中逆转录转座驱动基因组扩展的动力学。基因组研究》2006;16:1262-9。
- 14.
视角:转座因子、寄生DNA和基因组进化。进化。2001;55:1-24。
- 15.
灰色YHM。它需要两个转座子:转座子介导的染色体重排。趋势麝猫。2000;16:461-8。
- 16。
葡萄果皮颜色的反转录转座子诱导突变。科学。2004;304:982。
- 17。
Butelli E,Licciardello C,张Y,Liu J,Mackay S,Bailey P等人。转回控制在血液橙子中的果实特异性,冷依赖性积累的花青素。植物细胞。2012; 24(3):1242-55。
- 18。
Morgante M, Brunner S, Pea G, Fengler K, Zuccolo A, Rafalski A.玉米种内多样性的基因复制和外显子改组。Nat麝猫。2005;37:997 - 1002。
- 19。
蒋宁,包志强,张晓霞,Eddy SR, Wessler SR. Pack-MULE转座元件介导植物基因进化。自然。2004;431(7008):569 - 73。
- 20。
GOUDL L.跨选择水平的个性和适应:我们如何命名和概括达尔文主义的单位?Proc Natl Acad Sci U S A. 1999; 96:11904E11909。
- 21。
Hoen DR, TE局。植物中转座因子的定位。在:Grandbastien M-A, Casacuberta JM,编辑。植物转座的元素。德国海德堡:施普林格;2012.219 - 51页。当前遗传学的主题,第24卷。
- 22。
jurka j,宝w,kojima k,kapitonov vv。重复元素:生物信息识别,分类和分析。在:els。Chichester:John Wiley&Sons Ltd;2011年。http://www.els.net.(doi:10.1002/9780470015902. a0005270.pub2].
- 23.
陈志强,陈志强,陈志强,等。利用高通量反转录转座子插入多态性(RBIP)标记研究了豌豆(Pisum)的遗传多样性和进化。BMC Evol Biol. 2010;10:44。
- 24.
Smýkal P, Bačová-Kerteszováč N, Kalendar R, Corander J, Schulman AH, Pavelek M.基于反转录转座子的亚麻种质资源遗传多样性分析。Theor Appl Genet. 2011; 122:1385-97。
- 25.
引用本文:陈志强,陈志强,陈志强。反转录转座子在植物生物学中的应用。植物科学进展,2001;6:127-34。
- 26.
陈志强,王志强,王志强。一种沉默的“千弗伊”:转座因子的表观遗传控制。生物物理学报。2011;18:452 - 8。
- 27.
Bucher E,Reinders J,Mirouze M.拟南芥转运转录和流动性的表观遗传控制。CurrOp植物BIOL。2012; 15:503-10。
- 28.
Devos KM, Brown JKM, Bennetzen JL。通过非法重组减少基因组大小抵消拟南芥基因组扩展。基因组研究》2002;12(7):1075 - 9。
- 29.
Ma J,Devos Km,Bennetzen JL。LTR-RETRONSPOSON结构的分析揭示水稻近期和快速基因组DNA损失。Genome Res。2004; 14:860-9。
- 30.
Claros MG, Bautista R, Guerrero-Fernández D, Benzerki H, Seoane P, Fernández-Pozo N.为什么组装植物基因组序列如此具有挑战性。生物学(巴塞尔)。2012; 1:439-59。
- 31.
刘荣华,马建军,刘荣华。复杂植物基因组中基因数量的一致性高估。植物学报2004;7:732-6。
- 32.
王志强,王志强。绿刺草(Eragrostis tef, T’ef)的数量分类及文献综述。经济学机器人。1979;33:413-24。
- 33.
Brink M, Belay G.谷物和豆类(热带非洲植物资源1)。荷兰莱顿/荷兰瓦赫宁根CTA:荷兰瓦赫宁根PROTA基金会/Backhuys出版社;2006.p。297。
- 34.
Ayele M,Dolezel J,Van Duren M,Brunner H,Zapata-Arias FJ。埃塞俄比亚谷物TEF核基因组的流式细胞统计学分析[鹰岩TEF(ZECC.)托洛特]。genetica。1996年; 98:211-5。
- 35.
Teff (Eragrostis tef)作为无谷蛋白食品和饮料的麦芽酿造、酿造和生产原料的研究进展。食品科学技术。2014;51:2881-95。
- 36.
Cannarozzi G,Plaza-Wütrichs,esfeld K,Larti S,Wilson Ys,Girma D等人。基因组和转录组测序识别孤儿作物TEF(Eragrostis TEF)中的育种靶标。BMC基因组学。2014; 15:581。
- 37.
assefa k,yu J-k,Zeid M,Belay G,Tefera H,Sorrells Me。育种TEF [Eragrostis TEF(ZUCC。)托洛特]:常规和分子方法。植物品种。2011; 130:1-9。
- 38.
朱强,杨磊,杨丽丽,杨丽娟,等。通过下一代测序分析,高通量发现tef半矮化基因突变。遗传学。2012;192:819-29。
- 39。
Price AL, Jones NC, Pevzner PA。大基因组中重复家族的从头鉴定。生物信息学。2005;21 (1):i351-8。
- 40.
黄旭,马丹。CAP3 :DNA序列组装程序。基因组研究》1999;9:868 - 77。
- 41.
黄颖,牛波,高艳,付玲,李伟。CD-HIT Suite:一种用于生物序列聚类和比较的web服务器。生物信息学。2010;26:680-2。
- 42.
在豌豆(Pisum sativum L .)基因组的重复DNA :利用454测序的综合特性,并与大豆和截形苜蓿进行比较。BMC基因组学。2007;8:427。
- 43。
柳条T,拿机械袜,Sabot F,Stein J,VU GTH,Graner A等。大麦基因组的低通霰弹枪测序有助于快速鉴定基因,保守的非编码序列和新型重复。BMC基因组学。2008; 9:518。
- 44。
Macas J,Kejnovskýe,Neumann P,NovákP,Koblížkováa,Vyskot B.中一代基于测序的模型植物硅胶基于重复DNA分析。Plos一个。2011; 6:E27335。
- 45。
Smit AFA, Hubley R, Green P. RepeatMasker Open-4.0。2013 - 2015 <http://www.repeatmasker.org>
- 46。
串联重复序列发现者:一个分析DNA序列的程序。核酸Res. 1999; 27:573-80。
- 47。
Koch P,Platzer M,Downie Br。Repark - 从全基因组NGS读取的重复图书馆创建了Novo。核酸RES。2014; 42:1-12。
- 48.
Zytnicki M,Akhunov E,Quesneville H.Tedna:一个可转换元素De Novo汇编程序。生物信息学。2014; 30(18):1-3。
- 49.
NovàkP,Neumann P,Pech J,Steinhaisl J,Macas J. Repectexplorer:基于星系的Web服务器,用于从下一代序列读取的基因组宽的真核重复元素的基因组特征。生物信息学。2013; 29:792-3。
- 50.
水稻ltr -逆转录转座子的数据库。核酸学报2007;35。
- 51.
jurka j,Kapitonov vv,Pavlicek A,Klonowski P,Kohany O,WalichiewiCz J. Repbase更新,一种真核性重复元素的数据库。CytoOgenet基因组Res。2005; 110:462-7。
- 52.
Wolfe Kh,Gouy M,Yang YW,夏普PM,李WH。单子叶 - DICOT分歧的日期估计来自叶绿体DNA序列数据。Proc Natl Acad Sci U S A. 1989; 86:6201-5。
- 53.
Crepet WL,Feldman Gd。在化石记录中最早的草遗骸。我是J机器人。1991; 78:1010-4。
- 54.
袁勇,王志强,王志强。画眉草5个位点的单倍型分析与连锁不平衡。G3(贝塞斯达)。2(3): 407 - 2012; 19。
- 55.
Zuccolo A, Ammiraju JSS, Kim HR, Sanyal A, Jackson S, Wing RA。Ty3-Gypsy LTR反转录转座子atlys在稻属植物中的快速和差异增殖大米。2008;1(1):85 - 99。
- 56.
大坪夫H,熊川N,大坪夫E. RIRE2,一种新的水稻吉普赛型反转录转座子。基因工程学报1999;74:83-91。
- 57.
英格拉姆·艾尔,多伊尔·JJ。蕨属植物及其相关多倍体的起源和进化:来自核蜡质和质体rps16的证据。[J] .中国生物医学工程学报。2003;90(1):116-22 .]
- 58.
SanMiguel P, Gaut BS, Tikhonov A, Nakajima Y, Bennetzen JL。玉米基因间反转座子的古生物学。Nat麝猫。1998;20(1):43-5。
- 59.
Macas J, Mészáros T, Nouzová M. PlantSat:植物卫星重复的专门数据库。生物信息学。2002;18:28-35。
- 60.
GE, Hon G, Chandonia JM, Brenner SE。WebLogo:一个序列logo生成器。基因组研究》2004;14:1188 - 90。
- 61.
Poptsova MS,Il'icheva Ia,Nechipurenko Dy,Panchenko La,Khodikov MV,Opharina Ny等。下一代测序中的非随机DNA碎片。SCI批准。2014; 4:4532。
- 62.
Rasmussen DA, Noor MAF。用0.1倍的基因组覆盖能做什么?基于大翅蛾基因组调查的个案研究。BMC基因组学。2009;10:382。
- 63.
NovákP,Neumann P,Macas J.基于图形的聚类和表征下一代测序数据中的重复序列。BMC生物信息学。2010; 11:1-12。
- 64.
NovákP,Hřibováe,Neumann P,Koblížkováa,doleželj,玛卡斯J.全基因组分析了家庭山眼的重复多样性。Plos一个。2014; 9(6),E98918。
- 65.
萨斯堡SL,约克JA。小心错误组装的基因组。生物信息学。2005;21:4320-1。
- 66.
王志强,王志强,王志强,等。植物转座因子与基因组学的关系。[j] .地理科学进展,2002;3(5):329-41 .]
- 67。
黄S,Ding J,Deng D,Tang W,Sun H,Liu D等人。Kiwifruit Actinidia Chinensis的基因组草案。NAT Communce。2013; 4:2640。
- 68。
jillon O, Aury JM, Noel B, Policriti A, Clepet C, Casagrande A, et al.;葡萄基因组序列表明主要被子植物门的祖先六倍体化。大自然。2007;449(7161):463 - 7。
- 69。
番茄基因组联盟。番茄基因组序列提供了深入了解肉质水果的进化。大自然。2012;485:635-41。
- 70.
马铃薯基因组测序联盟。块茎作物马铃薯的基因组序列分析。大自然。2011;475:189 - 95。
- 71.
国际大麦基因组测序联盟。大麦基因组的物理,遗传和功能序列组装。自然。2012; 491:711-6。
- 72.
Nystedt B, Street NR, Wetterbom A, Zuccolo A, Lin Y-C, Scofield DG,等。挪威云杉基因组序列和针叶树基因组进化。大自然。2013;497:579 - 84。
- 73.
发现和检测基因组序列中的转座元件。短暂的Bioinform。2007;8(6):382 - 92。
- 74.
Baucom Rs,estill Jc,Chaparro C,uspshaw,Jogi A,Deragon JM等。B73玉米基因组中的卓越多样性,非随机分布和retelement的快速演变。Plos Genet。2009; 5。
- 75.
国际短讲台倡议。模型禾草的基因组测序与分析。大自然。2010;463:763-8。
- 76.
郭立志,刘杰。水稻的长末端重复反转录转座子。基因组医学杂志。2002;3 (10):RESEARCH0053。
- 77.
格罗弗ce,金h,翼ra,帕特森啊,温德尔·杰夫。棉花中局部和全球基因组大小演化的不一致模式。Genome Res。2004; 14(8):1474-82。
- 78.
Hawkins JS, Kim H, Nason JD, Wing RA, Wendel JF。转座元件的差异谱系特异性扩增是基因组大小变化的原因Gossypium..Genome Res。2006; 16(10):1252-61。
- 79.
GonzálezLG,Deyholos MK。亚麻(Linum Usitatissimum L.)基因组中可转换元素的鉴定,表征和分布。BMC基因组学。2012; 21(13):644。
- 80.
植物转座因子与基因组大小的关系。基因组学Inf。2014;12:87 - 97。
- 81.
国际水稻基因组测序项目。基于地图的水稻基因组序列。自然。2005; 436:793-800。
- 82.
Schmidt T, Heslop-Harrison JS。基因组、基因和垃圾:植物染色体的大规模组织。植物科学进展1998;3:195-9。
- 83.
Ugarkovićd,卫星DNA型材的PLOH1 M.变异 - 原因和效果。禁止J. 2002; 21:5955-9。
- 84.
王志强,王志强,王志强,等。转座因子在植物中广泛和频繁的水平转移基因组研究》2014;24:831-8。
- 85.
Smýkal P, Kalendar R, Ford R, Macas J, Griga M.作为全基因组微卫星重复散布剂的angela家族逆转录转座子的进化保守谱系。遗传(Edinb)。2009, 103(2): 157 - 67。
- 86.
Tvv1逆转录转座子家族在分离了1亿多年的植物基因组之间保持保守。Theor Appl Genet. 2014; 127:1223-35。
- 87.
Zuccolo A, Scofield DG, De Paoli E, Morgante M. Ty1-copia LTR retroelement family PARTC在200MY以上的针叶树中高度保守。基因。2015;568:89 - 99。
- 88.
基因组对挑战的响应意义。科学。1984;226:792 - 801。
- 89.
引用本文:张建军,张建军,张建军。PEAR:一种快速、准确的Illumina配对端reAd合并。生物信息学。2014;30:614-20。
- 90.
张建军,张志强,张志强,等。gap BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸学报1997;25(17):3389-402。
- 91.
Benson da,Karsch-Mizrachi我,Lipman Dj,Ostell J,Sayers EW。Genbank。核酸RES。2009; 37:D26-31。
- 92.
埃德加钢筋混凝土。MUSCLE:多序列比对,高精度,高通量。核酸学报2004;32:1792-7。
- 93.
Tamura K,SteCher G,Peterson D,Filipski A,Kumar S. Mega6:分子进化遗传学分析6.0版。mol Biol Evol。2013; 30:2725-9。
- 94.
Rice P, Longden I, Bleasb A, EMBOSS。欧洲分子生物学开放软件套件。趋势麝猫。2000;16(6):276 - 7。
- 95.
Kimura M.通过核苷酸序列的比较研究估计基取代的进化率的简单方法。J Mol Evol。1980; 16:111-20。
确认
该项目由Scuola Superiore Sant'anna,Pisa,意大利(Apomis11az)提供资金,并由Scuola Superiore Sant'anna,Pisa,意大利的生命学院进行。
作者信息
隶属关系
相应的作者
额外的信息
相互竞争的利益
提交人声明他们没有竞争利益。
作者的贡献
YGG进行生物信息学分析,DNA提取和南方杂交并写了稿件。EB促成了数据分析和写作稿件。MEP参加了研究设计和协调。AZ设计了分析,监督实验,协调研究并有助于起草稿件。所有作者都阅读并批准了最终手稿。
附加文件
额外的文件1:
库etef_repeats_v1.4包含本研究中孤立的重复。(FAS 615 kb)
额外的文件2:
a)包含~2.5个串联排列单体副本的重复库入口序列。b)重复库条目的点图自比较(PNG 71 kb)
额外的文件3:
TEFF,米和玉米中鉴定的TY1-Copia RT靶术序列的多次对准。(MSF 110 kb)
额外的文件4:
TEFF,稻米和玉米鉴定的TY3-GYPSYRT靶序列的多次对准。(MSF 135 kb)
额外的文件5:
在画眉草、水稻和玉米中鉴定的非ltr RT类似序列的多重比对。(MSF 33 kb)
额外的文件9:
CACTA转座酶类似序列在苔草、水稻和玉米中的多重比对。(MSF 12 kb)
额外的文件10:
在画眉草、水稻和玉米中发现的hAT二聚结构域相似序列的多重比对。(MSF 7 kb)
额外的文件11:
在苔草、水稻和玉米中鉴定的MuDr转座酶类似序列的多重比对。(MSF 7 kb)
额外的文件12:
对teff cv Enantite和Tsedey中鉴定的Ty1-copia RT相似序列进行多重比对。(MSF 51 kb)
额外的文件13:
TEFF CV enantite和TSEDEY中鉴定的TY3-GYPSYRT靶序列的多次对准。(MSF 49 kb)
附加文件14:
teff cv Enantite和Tsedey中非ltr RT类似序列的多重比对。(MSF 45 kb)
附加文件15:
卫星单体的1000个序列。(FAS 185 kb)
附加文件16:
卫星序列的序列徽标分析。(PNG 93 kb)
附加文件17:
TE编码域的磁盘用作相似性中的查询搜索,以检索TE Paralogs的副本。(FAS 663字节)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Gebre, Y.G, Bertolini, E, Pè, M.E.et al。丰富重复序列的鉴定与表征埃拉格罗斯特TEF.简历。enatite基因组。BMC植物BIOL.16,39(2016)。https://doi.org/10.1186/s12870-016-0725-4
收到了:
接受:
发表:
关键字
- 埃拉格罗斯特TEF.
- 重复序列
- 可转换元素
- 卫星序列