摘要
背景
富亮氨酸重复类受体激酶(LRR-RLKs)是植物中最大的类受体激酶亚家族。许多报告已经证明了那个工厂LRR-RLKs在生长、发育、分化和应激反应中起重要作用。然而,在豆科植物中尚未对该基因家族进行全面分析。
结果
基于序列相似性原则和域守恒原则,共有467个LRR-RLK在大豆基因组中鉴定了相关基因。的gmlrr-rlks.在大豆的所有20个染色体上是非随机分布的,其中约73.3%位于节段重复的区域中。推定寄生基因对的同义替换的分析表明,大多数这些基因对由大豆基因组中的节段性重复产生。此外,在构建的系统发育树中相同基团或亚组的成员之间,外显子/内含子组织,基序组成和布置受到显着保守。大豆之间的密切系统发育关系LRR-RLK基因识别拟南芥同一组的基因还提供了洞察其推定功能。表达分析分析gmlrr-rlks.提示它们在不同的组织中有不同的表达,一些重复的基因表现出不同的表达模式。另外,人工选择gmlrr-rlks.通过比较野生大豆和栽培大豆的SNPs也鉴定出了17个基因,在此前报道的包含驯化相关qtl的区域中检测到了17个基因。
结论
大豆的综合进化分析LRR-RLK基因家族在全基因组水平上进行。这些数据在将来的努力中提供了有价值的工具,以识别这种基因家族的功能分歧和豆类物种中不同基因型之间的基因多样性。
背景
受体样激酶(RLKs)是一组不同的跨膜蛋白,其特征是配体结合结构域接收信号分子,膜跨结构域锚定蛋白质,胞浆蛋白激酶结构域将信号转导到下游[1].在植物和动物中,RLK在细胞表面处介导大量信令消息,并在发育过程中充当关键调节器[2- - - - - -4].高等植物的第一个RLK是从玉米中分离出来的,随后从20多种植物中鉴定出了许多RLK [5].在植物中,RLKs超家族根据受体和激酶结构域的存在或缺失分为三大类[1,6,7].根据胞外结构域的差异,RLKs可进一步分为17个亚群,包括富亮氨酸重复(LRR) RLKs、s结构域RLKs等[8,9].在这些亚群中,LRR-RLK是迄今为止植物中最大的亚群,其成员含有约24个氨基酸串联重复序列,胞外区域有保守的亮氨酸残基[7,10].
遗传和生化研究表明,这种植物LRR-RLKs在生长和发育的不同过程中扮演重要角色[11,12].在拟南芥,LRR-RLKs包括SERK1/2, EMS1,BAM1/2,RPK2和拿来已被证明调节花药开发和施肥的过程[13- - - - - -18].有足够的证据支持CLV和RPK2是茎尖分生组织形成和维持过程中必需的受体样激酶[19,20.].其他一些报道也揭示了这一点LRR-RLK基因如展望和BAK1参与油菜素内酯信号转导,而其他一些LRR-RLK基因与脱落酸的应激反应有关[21- - - - - -23].此外,一些LRR-RLK据报道,由于植物发育和防御过程之间的交叉对话或一个受体对多个配体的识别,基因也具有双重功能[2].例如,拟南芥ERECTA基因的特征不仅是调节胚芽发展[24而且还涉及到对细菌性枯萎病的抗性[25].
快速增加的基因组序列有助于利用生物信息学工具在植物基因组水平上鉴定全基因家族。到目前为止,结构特征和表达式概要LRR-RLK基因已经被描述在植物包括拟南芥[26)、大米(27和白杨树[28].在大多数这些物种中,LRR-RLKs似乎是拥有数百个成员的大家族,并进化到执行不同的功能[28- - - - - -30.].一些报道也透露了这一点LRR-RLK由于基因组中存在大量的基因复制,基因具有冗余功能。例如,虽然是单突变体serk1或serk2显示正常花药形态,serk1 serk2双突变体可以挽救表型练习或ems.突变体由于绒毡层的缺失和胞外产孢细胞的产生而不能形成花粉拟南芥[13,14].SERK1/SERK2与绿色荧光蛋白变体的翻译融合研究也表明,SERK1/SERK2可能作为蛋白质复合物的一部分[13].
大豆(大豆)是动物饲料中最重要的蛋白质来源,也是人类营养的经济植物油来源[31].在进化史上,大豆基因组大约在5900万年前和1300万年前(MYA)经历了两轮全基因组复制(WGD) [32].与其他大部分二倍体不同,近75%的基因由于在相对近期的WGD期间缺乏直接的倍数而在大豆基因组中表现出多份拷贝[33].因此,大豆大部分基因家族的结构特征比大豆复杂拟南芥,米饭或杨树。虽然只有少数成员LRR-RLK大豆基因的功能特征已经得到了充分的证据支持LRR-RLK基因也在植物的各种发育和防御过程中发挥重要作用,包括叶片衰老、细胞伸长和耐冷胁迫[34- - - - - -36].
在目前的研究中,全基因组搜索LRR-RLK在大豆中进行了基因分析,共467个gmlrr-rlks.已经被确认。详细分析了基因组组织、序列系统发育、基因结构、保守结构域、复制状态和表达谱。此外,进化模式LRR-RLK通过基因序列分析和节段复制区分析,研究了大豆基因家族。此外,还探讨了人工选择对大豆的影响LRR-RLK在大豆驯化过程中也发现了基因家族。我们的结果为进一步的进化和功能表征提供了一个框架LRR-RLK大豆的基因家族。
结果和讨论
鉴定和基因组分布LRR-RLK大豆基因家族
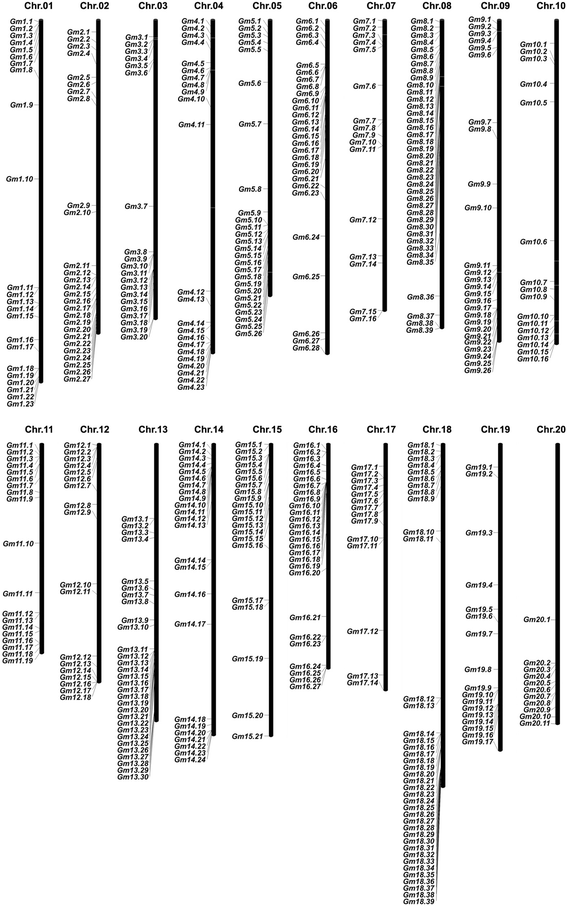
为了鉴定大豆基因组中所有LRR-RLKs的成员,利用大豆蛋白数据库中的氨基酸序列进行BLAST批量搜索拟南芥LRR-RLKs查询。将检索到的所有大豆蛋白提交到SMART和PFAM数据库进行结构域注释。只有包含至少一个LRR结构域和一个激酶结构域的候选LRR- rlk被认为是“真正的”LRR- rlk。人工去除不支持的序列和冗余基因后,共有467个推测的基因LRR-RLK从大豆全基因组中鉴定了相关基因。所确定的大豆LRR-RLK基因编码的多肽长度从423到1563 a.a.不等。每个基因的详细信息,包括登录号和编码蛋白的特征,列在附加文件中1.在所有这些推测的GmLRR-RLKs中,只有3个蛋白(Glyma.03G026800, Glyma.07G047200和Glyma.13G228300)被预测有两个激酶结构域。与LRR-RLK基因识别拟南芥、大米和杨树基因组(分别为213,309及379个成员)[26- - - - - -28)、大豆LRR-RLK本研究发现的基因家族是迄今为止植物中最大的基因家族。的数量gmlrr-rlks.大约是……的2.2倍atlrr-rlks.,这与假定的各大豆同源物的比例是一致的拟南芥基因(32,37].
物理位置的gmlrr-rlks.从Phytozome数据库获得(附加文件1),将其定位到大豆的相应染色体上。结果表明,464种大豆中均含有LRR-RLK从1号染色体到20号染色体的所有染色体上都可以找到基因。1),而其他三个基因只能映射到未组装的基因组序列支架上。尽管每条染色体都含有一定数量的LRR-RLK基因在不同染色体上的分布是不均匀的。每条染色体的分布比例在2.4%(20号染色体上有11个成员)到8.4%(8号和18号染色体上有39个成员)之间。这种分布模式与大豆和其他基因家族的分布模式相似LRR-LRK.其他植物的基因家族[26- - - - - -28,38,39].
大豆LRR-rlks的系统发育分析
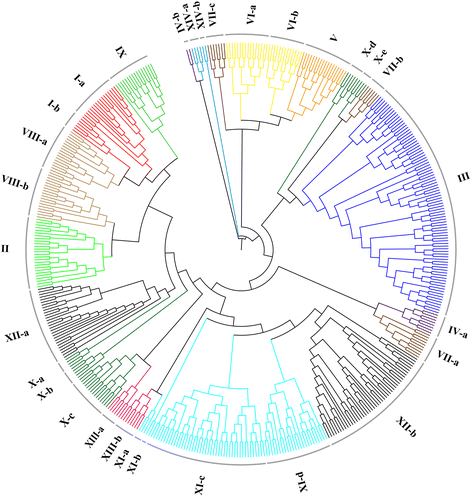
为了研究大豆中LRR-RLK成员的进化关系,我们利用所有GmLRR-RLKs的激酶结构域的氨基酸序列与聚类X进行多重比对,并利用MEGA构建系统发育树(图1)。2).系统发育树显示,所有的GmLRR-RLKs可以根据树的节点分为不同的组或亚组。当所有的GmLRR-RLKs与所有的AtLRR-RLKs聚集在一起时(附加文件2),各大豆LRR-RLKs组的成员根据命名法进行测定拟南芥同一组内的同系物(表1和无花果。2).有趣的是,一些GmLRR-RLKs成员由于基因组的高复制水平而表现出大豆的特异性特征。例如,虽然只有两个成员拟南芥在XII-b亚群中的LRR-RLKs (AT1G35710和AT4G08850)多达45个gmlrr-rlks.被鉴定为这两个的同源基因atlrr-rlks.(附加文件2).GmLRR-RLKs在XII-b亚群中的快速扩增可能是由于16和18号染色体上的两个大基因簇。
因为大多数atlrr-rlks.具有相似功能的大豆有聚集在一起的倾向LRR-RLK同一组或同一亚组的基因可能具有相似的功能拟南芥同源染色体。除IV组和VIII组无拟南芥与已确定的函数正交,所有其他基团都至少有一个AtLRR-RLK功能特点。例如,gmlrr-rlks.I、II、III、VII、XII组为聚类atlrr-rlks.参与器官/组织发育和防御信号[13,14,40- - - - - -43].V组包括拟南芥SCM与根毛形态有关的基因SRF细胞壁生物学中的基因[44,45].此外,Arabidopsis LRR-RLK涉及油菜素内酯和肽信号传导的基因属于X组[46和与细胞命运规范、器官形态发生有关的基因[47,血管发育[48,49,脱落酸信号,防御反应[50被归为十一组。此外,第十三-a小组包括两个范参与细胞壁发育信号通路的基因[51],包括子组XIII-berecta.和ERECTA-LIKE调节气孔发育和器官大小的基因[52].
基因结构和保守基序分析
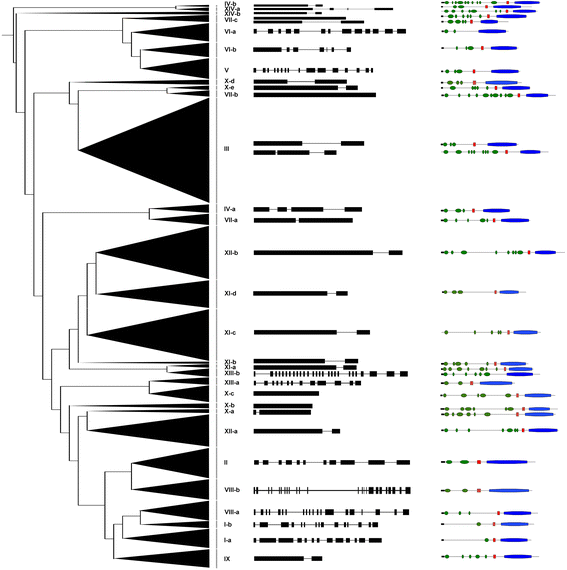
由于基因家族成员的外显子/内含子多样化总是在该基因家族的演变中发挥重要作用[53,大豆个体的外显子/内含子组织LRR-RLK基因也进行了分析。结果表明,近一半的成员gmlrr-rlks.(467个基因中的217个)只有一个内含子,26个基因只有一个外显子。在46、19、4和4个大豆中分别发现了2、3、4和5个内含子LRR-RLK基因。与此同时,共有151个基因含有5个以上的内含子,其中96个基因含有10个以上的内含子1和3.).就内含子的数量和长度而言,绝大多数gmlrr-rlks.在同一组或亚组中有非常保守的外显子/内含子组织(图)。3.).例如,大部分是大豆LRR-RLKVII、X和XI组的基因包含0、1和2个内含子,只有3个成员包含4个内含子。然而,V、VI和XII组的成员在内含子的数量和分布上表现出很大的变异性。最有趣的是,XIII-b亚群的成员包含多达26个内含子,大约是XIII-a亚群成员的两倍。外显子/内含子的结构表明了亚群内的保守性和不同亚群间的差异。
为了进一步理解的势函数LRR-RLK利用程序MEME (Multiple Em for Motif Elicitation, Multiple Em for Motif Elicitation)预测了这些蛋白的所有推定基序。结果表明,类群或亚类群之间的基序组成与系统发育分类一致。组间或亚组间的差异不仅表现在不同的基序类型上,而且还表现在同一蛋白中特定基序的数量上4).此外,利用SignalP在所有大豆LRR-RLKs中搜索可能的信号肽,结果显示有359个成员含有信号肽。同时,利用TMHMM预测了跨膜(TM)结构域,共有442个GmLRR-RLKs含有至少一个TM结构域,25个成员没有TM结构域,其中205个蛋白含有至少两个TM结构域。这些结果也表明,在系统发育树中,大部分密切相关的成员都表现出相似的motif,进一步说明同一亚群的大豆LRR-RLK蛋白之间存在大量的功能冗余(图)。3.和额外的文件5).
大豆的基因复制与同源关系LRR-RLK基因
基因复制一直被认为是基因组进化的主要驱动力之一[54].节段复制、串联复制和转座事件被认为是植物基因家族扩张的三个主要原因[55].在我们的分析中,串联复制簇被定义为包含两个或两个以上大豆的区域LRR-RLK200 KB内的基因。结果表明,该基因家族中约20.3%(464个)基因位于具有串联重复的地区,总共组成33个群集(附加文件6).最大的串联复制集群包含多达10个基因,而最小的只包含两个。进一步分析发现,串联重复簇在14个系统发育类群中分布不均匀。第XII组聚类最多,共8个聚类,包含35个基因,而第III、IV、V、VI、VII、IX、XIV组无聚类。
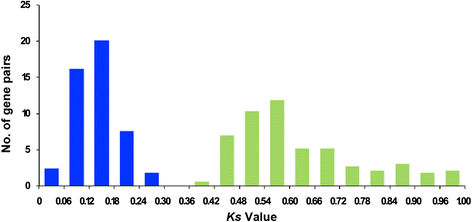
节段性重复通过多倍体产生重复的基因,然后产生染色体重排[56].我们的研究结果表明,共期重复,总共329个推定的副寄生基因对(340个基因或总基因的73.3%)(附加档案7),提示片段复制可能是大豆LRR-RLK基因家族基因扩增的主要机制。为了估计分段复制事件发生的日期,Ks值用于计算每个假定的副同源基因对的分离时间(附加文件7).的分布分析Ks价值观表明所有的Ks值的变化范围为0 - 1.0,有两个峰值分别位于0.12-0.18和0.54-0.6。4).根据大豆中同义替换的钟状速率,大豆的节段复制LRR-RLK基因从0到81.8 MYA,这两个峰值与13和59 MYA左右的全基因组重复事件一致[32].此外,Ka / Ks239对同源基因的比值均小于0.3,其余90对同源基因的比值均大于0.3,说明某些大豆可能存在显著的功能分化LRR-RLK重复事件后的基因。
的分布Ks值在所有段重复gmlrr-rlks..的Ks利用PGDD数据库(http://chibba.agtec.uga.edu/duplication/).0.12-0.18和0.54-0.6的两个峰与大豆的全基因组重复事件约为13和59 mya一致
表达谱的LRR-RLK基因大豆
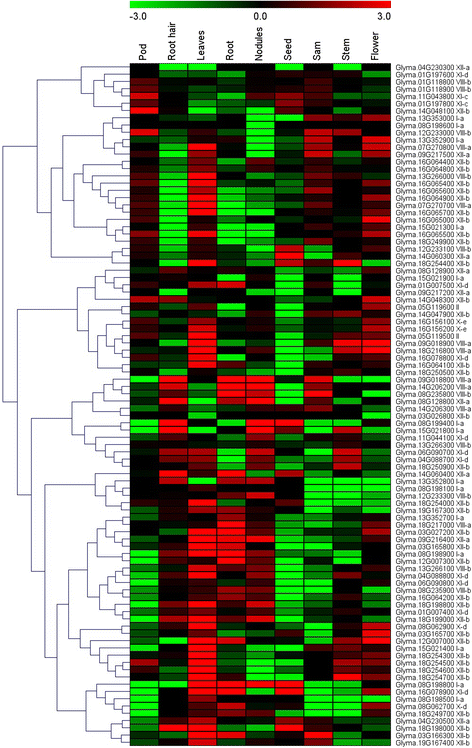
以获得对大豆假定功能的更广泛的理解LRR-RLKs利用不同大豆组织的RNA-Seq数据集分析了这些基因的表达谱。从根、根毛、根瘤、叶、茎、花、SAM、豆荚和种子等组织的RNA-Seq图谱数据中,鉴定了467个LRR-RLK基因的独特转录丰度模式。虽然一些基因表现出低转录丰度,如编码转录因子的基因,但大多数基因表现出不同的组织特异性表达模式(附加文件)8).详细分析结果依次为:53(11.3%)、68(14.6%)、65(13.9%)、53(11.3%)、95(20.3%)、87(18.6%)、75(16.1%)、67(14.3%)、51(10.9%)。gmlrr-rlks.在根、根毛、根瘤、叶、茎、SAM、豆荚、种子和花中分别有特异性的转录本积累,表明这些LRR-RLK基因可能在不同的细胞或器官中作为组织特异性的调节剂发挥作用。
对表达式的详细分析也表明gmlrr-rlks.聚类在同一亚群中有相似的表达模式。例如,所有的LRR-RLK亚群XIII-b的基因主要在种子和SAM中表达,也表明大豆之间存在冗余LRR-RLK这些亚群的基因。然而,也有报道说,超过50%的重复LRR-RLKs在水稻和拟南芥[57,58].我们的结果表明,在大豆中仅在33个串联复制基因中仅有7种簇中表现出类似的表达模式(图。5).为了验证这些重复基因的表达模式,采用qRT-PCR检测随机选择的基因对的表达水平。结果表明,RNA-seq数据集鉴定的这些基因对的相似或不同的表达模式与qRT-PCR结果一致(补充文件)9).329对LRR-RLK同源基因中,只有50对具有相似的表达模式,并可能在功能上相互替代。
人工选择分析LRR-RLKs在大豆驯化
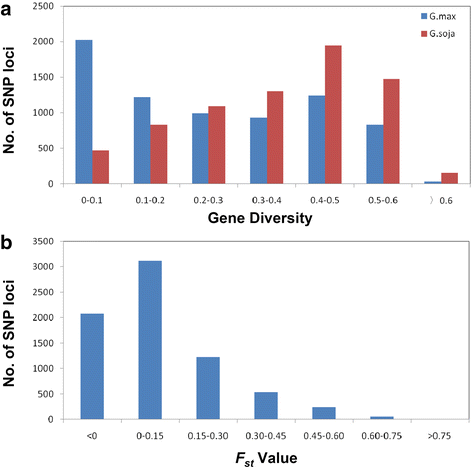
为了分析选择的效果gmlrr-rlks.在大豆驯化期间,使用了野生和栽培大豆的重新排序数据[59,60].在407份大豆基因区共鉴定出7239个SNPsLRR-RLK35种栽培大豆(g.max.)及21种野生大豆(G.soja)(额外的文件10).在这些位点上,栽培群体的基因多样性平均为~0.25,显著低于野生群体(~0.36)。SNP149在Glyma.01G197800是一个典型的例子,在栽培大豆中没有多样性,同时在野生大豆中具有高达0.66的多样性。分布分析还透露,大多数基因群的基因多样性小于0.2g.max.在0.4 - -0.6G.soja(无花果。6),表明它们的基因多样性LRR-LRKs在栽培大豆中,与野生祖先相比减少了。
以确定选择性gmlrr-rlks.在大豆驯化,F圣计算两个种群间每个位点的值(图。6 b和额外的文件10).结果表明,7239个位点中有5182个位点发生了非选择F圣在大豆进化过程中<0.15。然而,在98个大豆品种中总共有302个snpLRR-RLK基因被鉴定为具有F圣value cutoff 0.45(附加文件11).虽然这些SNPs中有一部分(302个中的134个)位于内含子中gmlrr-rlks.,近三分之一(89个SNP)导致了不同义的改变。进一步分析表明所有亚组gmlrr-rlks.除XIV组外,均选择了其他基因。十一组的大豆选择最多LRR-RLK组有21个基因,而IV组只有一个基因。特别是,尽管Glyma.11G214400和Glyma.18G050700所选择的SNPs最多(分别为36个和32个),第一个基因中的大多数SNPs导致了非同义变化,而第二个基因中的大多数SNPs出现在内含子中。此外,选定了一些LRR-RLK在此前报道的包含驯化相关qtl的区域中检测到了野生种群和栽培种群之间的基因(附加文件)11).其中包括6个gmlrr-rlks.定位与荚裂、荚数、荚成熟度等荚果性状相关的qtl [61,62],位于qtl调节双胞胎习惯的五个基因[63- - - - - -65],籽粒重/硬粒性QTL区域有4个基因,倒伏相关QTL区域有2个基因[63,65].这些被选择的基因反映了基因的重要作用gmlrr-rlks.为大豆的驯化做出贡献,以满足人类对大豆的需求。
结论
在这里,我们进行了全面和进化的分析LRR-RLK并提供了其成员的详细信息。总共有467个假设LRR-RLK在大豆基因组中发现了最大的基因LRR-RLK到目前为止在植物中发现的基因家族,在大豆中有一个比较大的基因家族。这些基因在大豆染色体上的分布是非随机的,大部分基因位于节段复制区域而不是串联复制簇。外显子/内含子的组成和基序排列在同一组或亚组的成员之间相当保守。在不同的大豆组织中,许多重复基因的转录谱也很相似,尽管其中一些表现出不同的表达模式。的密切的系统发育关系gmlrr-rlks.和确认AtLRR-RLK同一亚群中的基因提供了它们假定的功能。此外,一些人为的选择gmlrr-rlks.通过比较这些位点在从野生大豆到栽培大豆的进化过程中的基因多样性,也已确定。这些结果为进一步鉴定该家族的特异基因功能和大豆不同基因型间的基因多样性提供了有价值的工具。
方法
拟南芥LRR-RLKs和大豆基因组资源
所有的氨基酸序列拟南芥LRR-RLK从TAIR数据库v10.0获得(http://www.arabidopsis.org/).tlrr - rlks的分类和类群命名基于PlantsP server v.2011 of拟南芥2010项目(http://plantsp.genomics.purdue.edu/projects/lrr/Clouse2010.htm)[66].所有注释的大豆基因的基因组,编码和氨基酸序列根据基因组序列大豆WM82.A2.v1来自植物血统v10(http://phytozome.jgi.doe.gov/pz/portal.html)[67].
鉴定LRR-RLK大豆基因组基因
所有的氨基酸序列拟南芥利用LRR-RLK成员使用Bioedit v7对所有注释大豆基因的蛋白数据库进行局部blast搜索[68]所有蛋白质的蛋白质均为小于10−6被选为推定的大豆LRR-LRKs。通过去除冗余序列和功能注释进一步过滤这些推定的GmLRR-RLKs,然后用SMART (http://smart.embl-heidelberg.de.)[69]及PFAM (http://pfam.xfam.org/)[70以确保LRR和激酶结构域的存在。
多序列比对和系统发育树构建
预测GmLRR-RLK和AtLRR-RLK蛋白的激酶结构域后,提取其激酶结构域的氨基酸序列。使用ClustalX (version 1.83)和默认参数进行多序列比对[71].构建了大豆LRR-RLKs单独和大豆/的非根系统发育树拟南芥与mega 6.0一起[72使用neighbor-joining (NJ)方法。通过1000个重复的bootstrap分析对节点进行测试,并选择可能性最高的树进行进一步分析。
大豆的染色体定位、基因结构及基序分析LRR-RLK基因
的所有成员gmlrr-rlks.基于它们的物理位置映射到大豆染色体上。使用MapInspect软件生产染色体位置的图像(http://mapinspect.software.informer.com).大豆LRR-RLK基因的外显子和内含子的数量和位置是通过与相应的基因组DNA序列进行比较确定的[73].Signalp v4.1预测了信号肽和跨膜结构域的存在(http://www.cbs.dtu.dk/services/signalp//)[74], TMHMM v2.0 (http://www.cbs.dtu.dk/services/TMHMM/)[75]和恐惧(http://phobius.sbc.su.se/)[76分别)。系统发育树、基因和蛋白质结构的组合使用iTOL工具生成(http://itol.embl.de)[77].
重复分析和计算重复事件的日期
串联重复的特征是该基因家族的多个成员发生在邻近的基因间区域。在这项研究中,大豆LRR-RLK在以前的研究中,根据其他植物的标准,200 KB内聚在一起的基因被认为是串联复制基因[28,78].节段重复gmlrr-rlks.根据PGDD数据库的特征(http://chibba.agtec.uga.edu/duplication/).的Ks和卡还通过使用PGDD数据库计算重复基因对的值。的Ks值用于计算重复事件的大约日期,并且同义替换的时钟状速率(λ)设置为6.1x10−9大豆的替代品/同义地点/年[32,79,80].
转录概况分析
大豆根、根毛、根瘤、叶、茎、花、SAM、豆荚和种子的RNA-seq数据来源于Phytozome v10 (http://phytozome.jgi.doe.gov/pz/portal.html)和所有的表情简介gmlrr-rlks.进行进一步分析。大豆LRR-RLK利用MultiExperiment Viewer (Mev) v.4.9.0使用Pearson相关和平均连锁聚类算法对转录数据进行分层聚类[81].
实时定量RT-PCR分析
大豆植株(生态型Williams 82)在25±1°C的长日照条件下(16 h光照/8 h暗循环)在室内土壤中生长。取2周龄幼苗的根、茎、单叶、三小叶、茎尖分生组织(SAM)、花、荚和1周龄幼苗进行总RNA分离。用TRIzol Reagent (Invitrogen公司,美国)提取总RNA,用RNase-free DNase (TaKaRa公司,日本)处理。用日本TOYOBO公司的ReverTra Ace qPCR RT Kit逆转录总RNA 5微克,每反应20 μL。将cDNA稀释50倍作为模板进行定量RT-PCR。PCR在Applied Biosystems 7300 Real-Time PCR系统上进行,使用SYBR Premix Ex Taq试剂盒(TaKaRa,日本)。反应程序根据生产商的方案确定,所用引物的序列见附加文件12.以2计算各基因相对表达量,对应于Actin表达量−ΔΔt方法(82].
选择性的分析gmlrr-rlks.在大豆属
从NCBI网站下载了25个野生大豆和31种栽培大豆的SNP数据(http://www.ncbi.nlm.nih.gov/SNP/snp_viewTable.cgi?handle=NFCRI_MOA_CAAS).SNP位点gmlrr-rlks.根据每个基因的物理位置进行鉴定。由于所有SNPs的物理位置都基于该版本的大豆基因组,所以使用了v1.1版本的大豆基因注释。每个SNP位点的基因多样性g.max.和G.soja,F圣值由Genepop V4.0计算[83].SNP位点F圣>0.45被定义为驯化过程中推定的选择位点。
数据和材料的可用性
支持本文结果的数据包含在文章及其附加文件中。
参考
- 1.
Walker JC。高等植物受体样蛋白激酶的结构和功能。植物mol biol。1994年; 26(5):1599-609。
- 2.
阿夫扎尔·AJ,伍德·AJ,莱特福特地方检察官。植物类受体丝氨酸苏氨酸激酶:在信号传导和植物防御中的作用。植物营养与肥料学报。2008;21(5):507-17。
- 3.
Gish LA, Clark SE。RLK/Pelle激酶家族。植物j . 2011; 66(1): 117 - 27所示。
- 4.
约翰逊KL,英格拉姆GC。发送正确的信号:调节受体激酶活性。植物学报。2005;8(6):648-56。
- 5.
张丽娟,张丽娟。玉米s位点糖蛋白与受体蛋白激酶的关系芸苔属植物.大自然。1990;345(6277):743 - 6。
- 6.
布劳恩DM,沃克JC。植物跨膜受体:信号转导之谜中的新片段。生物化学进展。1996;21(2):70-3。
- 7.
鸟居骨。植物跨膜受体:受体激酶及其配体。Annu Plant Rev. 2008; 33:1-29。
- 8.
植物受体样激酶基因家族的结构、功能和信号转导。Sci抽烟可以。2001, 2001 (113): re22。
- 9.
受体样激酶拟南芥形成一个与动物受体激酶相关的单系基因家族。中国科学院院刊2001;98(19):10763-8。
- 10.
张XR。植物中富含亮氨酸的重复受体样激酶。植物MOL BIOL REP。1998; 16(4):301-11。
- 11.
Dievart A, Clark SE。调控植物发育和防御的lrr受体。发展。2004;131(2):251 - 61。
- 12.
Butenko MA, alen RB。受体配体在发育中。In: Tax F, Kemmerling B, editors。植物的类受体激酶:从发育到防御。柏林:施普林格;2012.p . 195 - 226。
- 13.
Albrecht C, Russinova E, Hecht V, Baaijens E, de Vries s拟南芥体细胞胚胎发生受体样激酶12控制雄性孢子发生。植物细胞。2005;17(12):3337 - 49。
- 14.
Colcombet J, boison - dernier A, Ros-Palau R, Vera CE, Schroeder JI。拟南芥体细胞胚胎发生受体激酶12是绒毡层发育和小孢子成熟所必需的。植物细胞。2005;17(12):3350 - 61。
- 15.
引用本文:杨卫东,杨卫东,杨卫东,等。FERONIA受体样激酶在花粉管接受过程中介导雄性与雌性的相互作用。科学。2007;317(5838):656 - 60。
- 16.
BAM1/BAM2 receptor-like kinase是BAM1/BAM2的重要调控因子拟南芥早期的花药开发。植物细胞。2006; 18(7):1667-80。
- 17.
Mizuno S, Osakabe Y, Maruyama K, Ito T, Osakabe K, Sato T,等。类受体蛋白激酶2 (rpk2)是一种新的调控花药发育的因子拟南芥.植物j . 2007;(5): 751 - 66。
- 18.
赵德志,王光发,Speal B,马海涛多余的小孢子母细胞1基因编码一个假定的丰富亮氨酸重复受体蛋白激酶,控制体细胞和生殖细胞的命运拟南芥花药。基因Dev。2002;16(15):2021 - 31所示。
- 19.
Kinoshita A, Betsuyaku S, Osakabe Y, Mizuno S, Nagawa S, Stahl Y, et al.;RPK2是一种重要的类受体激酶,在细胞内传递CLV3信号拟南芥.发展。2010;137(22):3911 - 20。
- 20.
关键词:受体激酶CORYNE拟南芥独立于CLAVATA1传递干细胞限制信号CLAVATA3。植物细胞。2008;20(4):934 - 46。
- 21.
何凯,郭小平,袁涛,林洪辉,马美,吉田,等。BAK1和BKK1调节芸苔类固醇依赖性生长和芸苔类固醇独立的细胞死亡途径。Curr Biol。2007; 17(13):1109-15。
- 22.
南KH,李建民。BRI1/BAK1,介导油菜素内酯信号的受体激酶对。细胞。2002;110(2):203 - 12所示。
- 23.
Leucine-rich repeat receptor-like kinase 1是一种重要的脱落酸早期信号通路的膜结合调控因子拟南芥.植物细胞。2005;17(4):1105 - 19所示。
- 24.
等。的拟南芥ERECTA基因编码具有细胞外亮氨酸的重复的推定受体蛋白激酶。植物细胞。1996; 8(4):735-46。
- 25.
Godiard L,Sauviac L,Torii Ku,Grenon O,Mangin B,Grimsley Nh等。erecta,一种LRR受体样激酶蛋白控制发育的发育脂肪术地影响对细菌枯萎的抗性。工厂J. 2003; 36(3):353-65。
- 26.
Dievart A, Gilbert N, Droc G, Attard A, Gourgues M, Guiderdoni E, et al.;富亮氨酸重复受体激酶零星分布在真核生物基因组中。BMC Evol Biol. 2011;11:367。
- 27.
关键词:水稻,lrr -激酶,基因组,系统发育PLoS ONE。2011; 6 (3): e16079。
- 28.
Zan YJ,Ji Y,张Y,杨世,歌Yj,王jh。基因组鉴定,表征和表达分析杨树富含亮氨酸重复受体样蛋白激酶基因。BMC基因组学。2013;14:318。
- 29.
受体样激酶/Pelle基因家族的扩增及其受体样蛋白的研究拟南芥.植物杂志。2003;132(2):530 - 43。
- 30.
肖胜,Karlowski WM,潘荣瑞,曾永华,Mayer KFX,李文辉。受体样激酶家族的比较分析拟南芥和米饭。植物细胞。2004;16(5):1220 - 34。
- 31.
哈特曼GL, West ED, Herman TK。养活世界的农作物大豆在世界范围内的生产、使用和受病原体和害虫的限制。食品安全内核。2011;3(1):5。
- 32.
马建新,马建新,米特罗,等。古多倍体大豆的基因组序列。自然。2010;463(7278):178 - 83。
- 33.
关键词:大豆,基因组,动态重排,基因组结构植物杂志。2009;151(3):1066 - 76。
- 34.
Kim S, Kim SJ, Shin YJ, Kang JH, Kim MR, Nam K, et al.;一种非典型的富含亮氨酸重复受体样激酶GmLRK1可能参与了细胞伸长的调控。足底。2009;229(4):811 - 21所示。
- 35.
李雪萍,甘锐,李PL,马媛媛,张丽伟,张锐,等。参与大豆叶片衰老调控的富亮氨酸重复受体样激酶基因的鉴定和功能特征植物学报。2006;61(6):829-44。
- 36.
杨玲,吴克超,高鹏,刘晓军,李桂平,吴志军。GsLRPK是一种新的冷激活的富含亮氨酸重复受体样蛋白激酶甘氨酸大豆,是对冷胁迫耐受性的正向调节因子。植物科学。2014;215:19-28。
- 37.
Kaul S, Koo HL, Jenkins J, Rizzo M, Rooney T, Tallon LJ,等。开花植物基因组序列分析拟南芥.自然。2000;408(6814):796 - 815。
- 38.
大豆嗜环蛋白基因家族的全基因组分析(大豆).BMC Plant Biol. 2014;14:282。
- 39.
王XB,张鹤,高友,孙玉,张武,秋lj。大豆丘比特家族综合分析(大豆).PLoS ONE。2014; 9 (10): e110092。
- 40。
Agusti J,Lichberger R,Schwarz M,Nehlin L,GREB T.在嵌合形成过程中转录组重构的表征鉴定了二次生长的相反调节剂的MOL1和RUL1。Plos Genet。2011; 7(2):E1001312。
- 41。
赵伟林,张志强,张志强,等。MAP激酶信号级联拟南芥先天免疫。自然。2002;415(6875):977 - 83。
- 42.
双生病毒核穿梭蛋白是一种抑制跨膜受体激酶活性的毒力因子。基因Dev。2004;18(20):2545 - 56。
- 43.
Chinchilla D, Zipfel C, Robatzek S, Kemmerling B, Nurnberger T, Jones JDG,等。鞭毛蛋白诱导的受体FLS2和BAK1复合物启动植物防御。自然。2007;448(7152):497 - 500。
- 44.
Dolan L.位置信息和移动转录调节器确定细胞模式拟南芥根表皮。中国生物医学工程学报。2006;57(1):51-4。
- 45.
Eyuboglu B, Pfister K, Haberer G, Chevalier D, Fuchs A, Mayer KFX等。strutbbelg受体家族基因编码推定富亮氨酸重复受体样激酶的分子特征拟南芥.BMC Plant Biol. 2007;7:16。
- 46.
Kinoshita T, Cano-Delgado AC, Seto H, Hiranuma S, Fujioka S, Yoshida S, et al.;油菜素内酯与植物受体激酶BRI1胞外结构域的结合。自然。2005;433(7022):167 - 71。
- 47.
DeYoung BJ, Bickle KL, Schrage KJ, Muskett P, Patel K, Clark SE。与clavata1相关的BAM1、BAM2和BAM3受体激酶样蛋白是细胞分生组织功能所必需的拟南芥.植物j . 2006; 45(1): 1 - 16。
- 48.
PXY-CLE41受体配体对定义了一个控制血管细胞分裂的速率和方向的多功能途径。发展。2010;137(5):767 - 74。
- 49.
PXY是植物维管组织发育过程中维持极性的一种类受体激酶。咕咕叫医学杂志。2007;17(12):1061 - 6。
- 50.
PEPR2是Pep1和Pep2多肽的第二个受体,并参与人体的防御反应拟南芥.植物细胞。2010;22(2):508 - 22所示。
- 51.
Xu SL, Rahman A, Baskin TI, Kieber JJ。两个富含亮氨酸重复受体激酶介导信号,连接细胞壁生物合成和ACC合酶拟南芥.植物细胞。2008;(11):3065 - 79。
- 52.
乌奇达N,Shimada M,Tasaka M. erecta-Family受体激酶通过缓冲其在芽顶部分类中的细胞蛋白响应性来调节干细胞稳态。植物细胞生理。2013; 54(3):343-51。
- 53.
徐光新,郭聪,单海燕,孔洪志。复制基因在外显子-内含子结构上的差异。中国科学院院刊2012;109(4):1187-92。
- 54.
复制基因进化的早期阶段。中国科学(d辑:地球科学)2003;100(26):15682-7。
- 55.
Leister D.植入植物抗病基因演化中的串联和节段性基因复制和重组。趋势类型。2004; 20(3):116-22。
- 56.
于军,王军,林伟,李胜国,李华,周军,等。的基因组栽培稻:复制的历史。公共科学图书馆杂志。2005;3(2):266 - 81。
- 57.
Blanc G, Wolfe KH。多倍性过程中复制基因的功能分化拟南芥进化。植物细胞。2004;16(7):1679 - 91。
- 58.
任WC,李博姆,张世忠。水稻重复基因的表达多样性及其进化动态。中国生物医学工程学报。2009;28(5):483 - 493。
- 59.
Lam HM,Xu X,Liu X,Chen WB,Yang GH,Wong Fl,等。31种野生和培养的大豆基因组的重量识别遗传多样性和选择模式。NAT Genet。2010; 42(12):1053-9。
- 60。
李玉华,赵长生,马建新,李东,严玲,李军,等。全基因组重测序揭示大豆驯化改良的分子足迹。BMC基因组学。2013;14:579。
- 61。
控制大豆荚落的一个主要QTL的简单序列重复标记。植物品种。2006;125(2):195 - 7。
- 62。
张达,程H,王H,张清,刘海,余诗。基因组区域确定大豆中花包数发育的基因组区域(大豆L.)。j遗传学基因组学。2010; 37(8):545-56。
- 63。
刘斌,藤田,严志华,Sakamoto S,徐东,Abe J.大豆驯化相关性状的QTL定位(大豆).安机器人。2007;100(5):1027 - 38。
- 64。
Yang K, Jeong N, Moon JK, Lee YH, Lee SH, Kim HM,等。大豆种皮和花色自然变异控制基因的遗传分析。J在这里。2010;101(6):757 - 68。
- 65。
李玉华,管瑞祥,刘志新,马永山,王丽霞,李丽华,等。栽培大豆的遗传结构与多样性(大豆(l)中国的地方种族。应用计算机学报。2008;117(6):857-71。
- 66.
郭小平,何康,杨华,袁涛,林洪辉,刘志强,等。富亮氨酸重复受体样蛋白激酶基因的全基因组克隆及序列分析拟南芥.BMC基因组学。2010;11:19。
- 67.
许树强,许树强,陈志强,等。植物群落:绿色植物基因组学的比较平台。核酸Res. 2012;40(D1): D1178-86。
- 68.
大厅的助教。BioEdit:一个用户友好的生物序列比对编辑器和分析程序的Windows 95/98/NT。核酸辛普塞尔1999;41:95-8。
- 69.
SMART: 2015年的最新更新、新发展和现状。核酸Res. 2015;43(D1): D257-60。
- 70.
Finn RD, Bateman A, Clements J, Coggill P, Eberhardt RY, Eddy SR,等。蛋白质家族数据库。42(D1): D222-30。
- 71.
Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG。CLUSTAL_X窗口接口:由质量分析工具辅助的多序列比对的灵活策略。核酸学报1997;25(24):4876-82。
- 72.
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析版本6.0。生物医学进展。2013;30(12):2725-9。
- 73.
胡湾,金吉,郭艾,张H,罗杰,高G. GSD 2.0:升级基因特征可视化服务器。生物信息学。2015; 31(8):1296-7。
- 74.
SignalP 4.0:从跨膜区域识别信号肽。Nat方法。2011;8(10):785 - 6。
- 75.
Krogh A, Larsson B, von Heijne G, Sonnhammer ELL。用隐马尔可夫模型预测跨膜蛋白质拓扑结构:在全基因组中的应用。中华医学杂志。2001;305(3):567-80。
- 76.
Kall L, Krogh A, Sonnhammer ELL。结合跨膜拓扑结构和信号肽预测的优势- Phobius web服务器。核酸学报2007;35:W429-32。
- 77.
Letunic I, Bork P.交互式生命树v2:易于在线注释和显示系统发生树。核酸学报2011;39:W475-8。
- 78.
李春华,邹春华,赵淑华。植物类受体激酶/小秋基因的进化历史和胁迫调控。植物杂志。2009;150(1):12-26。
- 79.
Lavin M, Herendeen PS, Wojciechowski MF。进化速率分析豆科表明第三纪时期谱系迅速多样化。54系统杂志。2005;(4):575 - 94。
- 80.
Lynch M, Conery JS。复制基因的进化命运和后果。科学。2000;290(5494):1151 - 5。
- 81.
基因组表达模式的聚类分析和显示。中国科学(d辑:地球科学)1998;95(25):14863-8。
- 82.
Livak KJ, Schmittgen TD。利用实时荧光定量PCR技术分析相关基因表达数据−ΔΔCT方法。方法。2001;25(4):402 - 8。
- 83.
Rousset F. GENEPOP ' 007:对Windows和Linux的GENEPOP软件的完全重新实现。生态学报。2008;8(1):103-6。
确认
国家自然科学基金(No. 31271753);国家高技术研究发展计划(No. 2013AA102602);中国科学院优秀青年基本科研业务费专项资金(No. yg);中国农业科学院农业科技创新计划项目。
作者信息
从属关系
相应的作者
附加信息
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
作者的贡献
FZ进行实验,分析数据,撰写草稿稿。YG设计实验,分析数据,撰写和修改手稿。LJQ构思并监督了这个项目,并对手稿进行了严格的修改。所有作者阅读并批准了最终的手稿。
附加文件
额外的文件1:
确认列表LRR-RLK基因大豆。每个大豆的ID、基因编码、基因长度、染色体物理位置、外显子/内含子/UTR的数目、氨基酸/信号肽的长度、TM的数目和位置、激酶结构域的位置LRR-RLK纳入本研究中注释的基因。(XLSX 66 kb)
额外的文件2:
GmLRR-RLKs和AtLRR-RLKs的非根系统发育树。利用Clustal X 1.8.3软件对467个GmLRR-RLKs和213个AtLRR-RLKs的激酶结构域序列进行比对,并利用MEGA 6.0软件通过1000个bootstrap重复进行邻居连接构建系统发育树。(PDF 1599 kb)
额外的文件3:
所有大豆的外显子/内含子结构LRR-RLK基因。外显子由黑线的黄色盒子和内含子代表。使用蓝色盒子也表示一些基因的UTR区域。外显子,内含子和UTR的相对尺寸可以通过盒子或线的长度估计。(PDF 86 KB)
额外的文件4:
模因预测每个亚群中所有GmLRR-RLKs的假定基序。利用推导出的各组GmLRR-RLKs氨基酸序列,通过MEME软件进行motif鉴定,并给出每个motif的相对位置。(XLSX 3460 kb)
额外的文件5:
所有GmLRR-RLKs的信号肽、lrr、TMs和激酶的模式。信号肽、跨膜结构域和激酶结构域分别用黑色、红色和蓝色方框表示。LRR图案用绿色的椭圆形表示。每个图案的相对大小可以通过长度来估计。(PDF 205 kb)
额外的文件6:
大豆LRR-RLK位于串联复制簇中的基因。包含两个或更多大豆的区域LRR-RLK200 KB以内的基因被定义为串联复制簇。每个基因的基因ID、亚群和染色体GmLRR-RLK位于串联复制簇。(XLSX 11 kb)
额外的文件7:
部分重复事件的日期估计数LRR-RLK大豆的基因家族。(XLSX 21 kb)
额外的文件8:
所有大豆的表达曲线LRR-RLK不同组织的基因。从Phytozome v10中获得了大豆的全基因组RNA-seq数据。的表达式数据gmlrr-rlks.在荚果、根毛、叶、根、根瘤、种子、茎、SAM中,花的基因水平归一化和层次聚类。下面的颜色刻度代表表达值,绿色表示低水平,红色表示高水平的转录本丰度。(PDF 7242 kb)
额外的文件9:
通过qRT-PCR和RNA-seq数据集比较选定的串联复制基因对的表达模式。两个串联重复基因的表达水平对不同器官的分析定量rt - pcr (A和C)是一致的模式识别RNA-seq数据集(B和D)。每个基因的表达水平根设置为1.0,标准和误差三个生物复制的错误。(PDF 140 kb)
额外的文件10:
大豆的SNP位点LRR-RLK通过对25种野生大豆和31种栽培大豆的重测序数据进行分析,确定了相关基因。(XLSX 2377 kb)
额外的文件11:
假定的人工选择gmlrr-rlks.在大豆驯化。(XLSX 15 kb)
额外的文件12:
引物用于实时定量RT-PCR。(PDF 6 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
周飞,郭勇,邱丽娟。大豆富含亮氨酸重复受体样蛋白激酶基因的全基因组鉴定及进化分析。BMC植物杂志16,58(2016)。https://doi.org/10.1186/s12870-016-0744-1
收到了:
接受:
发表:
关键词
- 大豆
- 富亮氨酸重复受体样激酶
- 系统发育分析
- 表达分析
- 进化分析