抽象的
背景
最近被证明CRISPR / CAS9作为改变人,动物,微生物和植物的基因组的有效和流行的基因组编辑工具。这种基因组编辑的成功高度依赖于所编辑的基因组中合适的靶位点的可用性。对于若干年度模型和作物物种来说,CRAP / CAS9的许多特定目标站点已经被计算鉴定出来,但尚未向多年生的木质果实物种报告这些位点。在这项研究中,我们在广泛栽培的葡萄种中识别并表征了五种类型的CRISPR / CAS9靶位点vitis Vinifera并开发了一个用户友好的数据库,用于将来编辑葡萄基因组。
结果
在本研究中的葡萄基因组鉴定了总共35,767,960个潜在的CRISPR / CAS9靶位点。其中,将22,597,817个靶位点映射到特定的基因组位置,并发现7,269,788个是高度特异性的。发现原始扫描仪和PAM在葡萄基因组中均匀地分配。它们存在于基因的所有结构元素中,编码区域具有最高的丰度。观察到五种PAM类型,TGG,AGG,GGG,CGG和NGG。除了ngg型外,它们大量存在于葡萄基因组中。类似基因的同步分析显示,原激晶仪的同时性与同源基因的同时性相匹配。开发了一个用户友好的数据库,并开发了网站的详细信息,可供公共使用葡萄克莱普特网站(http://biodb.sdau.edu.cn/gc/index.html.).
结论
葡萄基因群港口港口潜在的CRISPR / CAS9靶位。这些位点广泛分布在基因的编码区域中具有主要丰富的染色体中。我们开发了一种可公开可访问的葡萄库克劳普斯特数据库,以促进使用CRISPR / CAS9系统作为用于功能研究和葡萄的分子育种的基因组编辑工具。在其他功能中,数据库允许用户识别和选择多个原始空间同时在葡萄基因组中编辑类似序列。
背景
CRISPR(聚类定期间隙的短语重复)/ CAS(CRISPR相关蛋白质)最近被成为一种有效的基因组编辑系统,用于修饰各种生物中的基因,包括人,动物,细菌和植物[1-3.]。该系统有三种类型:I,II和III,通过规范Watson-Crick基础对向靶位点进行高度特异性。II型使用CAS9核酸酶,是到目前为止所展示的最有用的系统[1由于酶的独特特性。Cas9核酸酶可以被CRISPR引导到目标原间隔子区域,位于原间隔子邻近基序(PAM)的上游。然后,Cas9核酸酶可以诱导内源性基因组位点的精确裂解,导致该位点的DNA缺失和其他变化[4.]。此外,CAS9核酸酶可以转化成切口酶,以促进与致突变性的同源性的修复[4.]。CRISPR / CAS9的这些属性使系统成为许多研究应用的有价值和多功能的工具[5.那6.]。
将CRISPR/Cas9系统用于各种基因组编辑目的的成功例子正在快速积累。该系统被用来在基因组中引入精确的突变肺炎链球菌肺炎料和大肠杆菌2013年(2],这证明了细菌基因组工程技术的有效性和多功能性。随后,Crispr / Cas9原核系统系统链球菌Pyogenes作为可编程的RNA引导的内切核酸酶,以靶向的方式裂解DNA,用于在人和小鼠细胞中进行基因组编辑[3.那4.]。现在,已经开发出用于植物中的多重基因编辑的携带PGREEN或PCAMBIA骨干网的各种工具套件,其可以促进CRISPR / CAS9系统的瞬态或稳定表达。5.]。来自许多植物种类的基因,包括拟南芥那Triticum aestivum.那Lycopersicon esculentum那柑橘sinensis.和尼古利亚娜,通过使用CRISPR / CAS9系统成功编辑[7.-11.]。此外,已经建立了几个数据库和网络工具以促进相关研究[12.-14.]。
葡萄是世界上最重要的水果作物之一,其基因组序列草案首先通过组装八折的霰弹枪序列,然后通过将覆盖范围提高到12倍来改善[15.那16.]。由于葡萄的经济性重要性,可以想到,将很快采用CRISPR / CAS9系统来编辑各种研究和应用的葡萄基因组。为了加速在葡萄中采用这种基因组编辑技术,我们分析了葡萄基因组序列,并确定了数百万个潜在的原始化器和基于CRISPr / Cas9基因组编辑的PAM。此外,我们还开发了一个用户友好的葡萄CRISPR数据库,并为公共使用提供。
结果
原种分布的原始分流器和PAMS
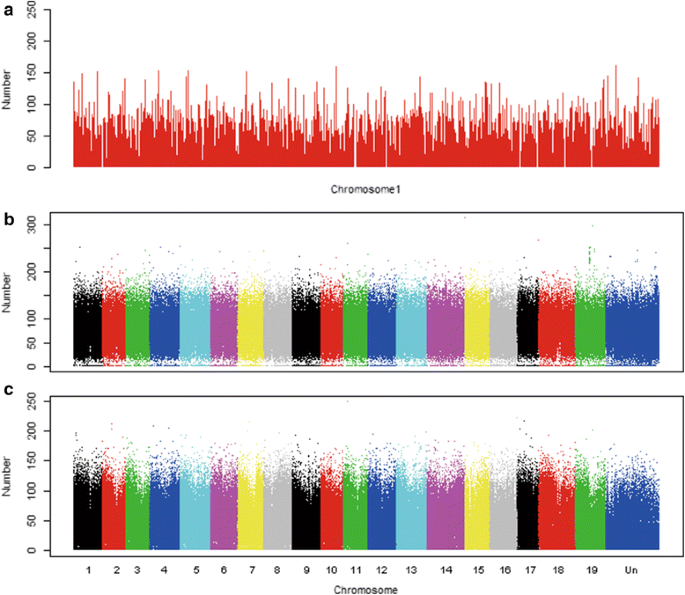
在葡萄基因组草稿中检测到总共35,767,960个改矩器/ PAM,并在特定基因组位置存在6.18%(22,597,817)。平均而言,基因组中的原始填充/ PAM的数量为73.57 / kB,总体特定于46.48 / kB位点(表1).这些原始空间均均匀分布在染色体中。作为图示(图。1A),染色体1上的原始扫描仪或多或少均匀分布,尽管染色体的原始扫描仪/ PAM的丰度范围为0至252 / kB。其他染色体具有相似的分布模式(附加文件1).取决于染色体的长度,20染色体(具有无规标记未映射的可随机标记的19个已知连杆基团和1)之间变化的原始扫描仪的数量。总强化器的范围为1,303,573,在CHR17至2,978,796中,以及独特的原始主流仪(在整个基因组中仅出现一次的原始化器),范围为835,838,在CHR10至1,495,033中。CHR14(附加文件2).当比较总和独特的位点时,不同染色体之间的分布没有显着差异(图。1B.和C(分别)暗示每种染色体具有相似水平的改性器/ PAM丰度,总分布相对均匀。注意,染色体两臂的特异性强化器的丰度高于中央区域(图。1C).此外,总有关的原始主流仪的数量与独特的原始主流仪之间存在显着的相关关系。此外,大约三分之一的独特原始化器(7,269,788)是高度特异性的。
粉斑的构成
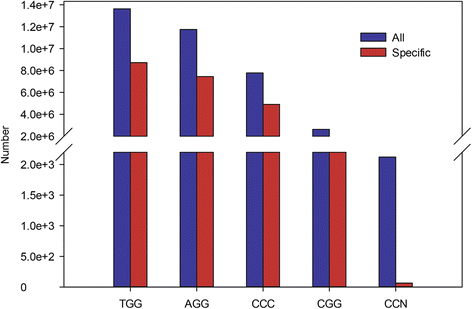
观察到五种PAM类型,包括TGG,AGG,GGG,CGG和NGG。以非常低的频率(0.0029%)观察NGG型,并且可能是由低质量序列引起的。所有染色体上存在另外四种PAM类型。TGG是最丰富的,其次是AGG,GGG和CGG,分别占总PAM的38.10,32.75,21.74和7.40%(图。2和额外的文件1).就PAM类型而言,通过整个葡萄基因组的总PAM和唯一PAM之间没有显著差异(图2)。2).
不同基因组区域的分裂位点
我们调查了葡萄基因组的各个地区的裂解遗址的分布(表1).基因区域的切割位点的裂解位点几乎比基因区域的裂解位点(21,895,244与13,872,716)大。然而,遗传区域中的裂解位点的相对丰度高于总体(81.59 / kb vs.69.25 / kB)和独特(62.41 / kb vs.37.91 / kB)的靶表。在遗传区域中,内含子的裂解位点的数量大于外显子,其具有大约相同数量的裂解位点,如在UTR区域中发现。此外,外显子和UTR中的裂解位点高于内含子的丰度。平均而言,在基因中存在约526.56总切割位点和402.80个独特的。
同步分析类似基因和多强化件
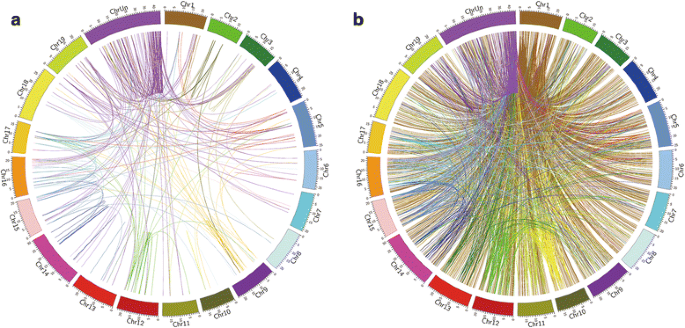
大多数基因具有独特和非独特的原始原始化器,除了141个基因(约0.5%)仅包含非独特的原始扫描仪。将这些141个基因分散在含有4294个强障碍物的所有染色体中,并且每个基因平均平均为30.45个原子晶体。类似基因的同步分析显示,原激晶仪的同时性与同源基因的同时性相匹配。每个单独的基因可能有几个原始扫描仪,其中一些原始扫描仪可以在同一组或家族的所有单个基因成员中找到(图。3A和B.).
葡萄库数据库
为了便于利用CRISPR/Cas9系统识别适合葡萄基因组编辑的目标位点,我们开发了一个可搜索数据库(命名为grape -CRISPR数据库)。数据库包含两个主要部分:搜索和设计。在Search部分,用户可以通过提供位点位置、基因ID或Pfam ID等查询信息来确定合适的原间隔体和PAM位点。数据库将根据每个间隔符的GC内容和PAM (NGG或GGNGG)类型为其提供1-3分的总分。它也将表明一个感兴趣的原间隔子是否可以很容易地合并到具有U6或T7启动子的表达载体中。如果一个间隔者不是唯一的,将提供一个马戏团地图来显示它与其他间隔者的关系。设计部分是原始空间的设计。用户可以使用提供的Perl脚本在感兴趣的序列中检测和设计原始间隔符和PAMs。
讨论
ProTospacers和Pams大量存在于葡萄基因组中。这些原始扫描仪和PAM在染色体和染色体区域之间或多或少地均匀地分布。这些潜在靶位点的丰富存在和均匀分布模式提供了通过使用CRISPR / CAS9系统编辑大多数葡萄基因组区域的可能性。大多数基因含有许多特定/独特的原始主流仪的事实使葡萄研究人员能够通过多种目标网站和巨大特异性来编辑感兴趣的基因。染色体中和染色体中的均匀分布模式表明这些靶位位点显然与葡萄染色体的任何特异性无关。然而,特别是在编码区中的基因区中的裂解位点的相对丰度高于非基因区域中的裂解位点。
在5种PAM类型中,TGG和AGG类型最为丰富。但在所有PAM类型中,除NGG类型的发生率低得多外,其余均无统计学差异。NGG是一种特殊的类型,它包含一个不明确的碱基对。这些NGG PAMs主要存在于基因组序列信息质量较低的区域,可能是由于存在重复序列。实际上,人们可以使用TGG、AGG、GGG和CGG的任何靶点,但不能使用NGG类型的基因编辑葡萄。同源性分析表明,多原间隔人与其同源基因具有同源性。基于序列相似性,可以使用通用原间隔体同时引导CRISPR/Cas9系统同时编辑多个基因组位点。这对于修改同源基因或感兴趣的家族基因尤其有用。因为葡萄是一个高度杂合的物种和基因组单核苷酸多态性丰富,是谨慎的和有用的re-sequence潜在目标网站确认,避免潜在的不匹配造成的SNP (s)和参考基因组之间的葡萄品种或种类的兴趣。
这项研究的重要成果之一是开发了一个grape crispr数据库。与其他类似资料库比较[12.-14.],葡萄克切者数据库是在综合的葡萄基因组序列分析的基础上开发的。我们为特定原创器提供注释,基因ID和PFAM编号信息,使数据库对用户更加丰富。该数据库还包含比其他类似数据库更多的数据,更重要的是,我们提供自定义Perl脚本来扫描和过滤数据库。通过这样做,数据库将允许用户探索各种选项并从中提取相关信息。
结论
葡萄基因组中含有大量的PAM位点和原间隔体,可以利用CRISPR /Cas9系统进行基因组编辑。这些位点广泛且或多或少均匀地分布在染色体之间和染色体内部。葡萄基因组中存在许多潜在的靶位点,且基因区裂解位点相对于基因间区丰度较高,这为利用CRISPR/Cas9系统编辑葡萄基因组提供了一个令人鼓舞的前景。除了对原间隔体和PAM位点的各种性质进行表征外,我们还开发了一个供公众使用的grape crispr数据库。
方法
葡萄基因组序列和注释信息(vitis Vinifera在本研究中使用的12x从植物精血中下载http://phytozome.jgi.doe.gov/pz/portal.html.。
帕姆地点和原创化器的识别和分布
在以往的研究中,发现NGG(互补链上的CCN)序列足以进行靶向[2]。因此,仅被认为是NGG(CCN)作为潜在的PAM网站,并在该研究中设定为20bp的原始空间。通过我们写入的Perl脚本来检测原始化器和PAM站点。考虑所有可能的网站。在序列包含Poly G(GN)或Poly C(CN)的情况下,PAM号将被计为N-1。所有原始扫描仪在其20bp长序列的基础上进行评估,并且鉴定出出现一次的原始扫描仪并指出为“特定的原始扫描仪”。然后,进行进一步的耐受性测试以鉴定“高度特异性”的原始扫描仪。该测试允许原始扫描仪中的大多数不匹配,最后三个基地必须具有高保真度。该测试是通过使用BLASTN完成的。
比较了20条染色体(19个已知连锁群和1个未作图的随机标记)每1kb长序列中所有和特异PAMs的平均丰度,确定了所有和特异PAMs的丰度之间的相关性。
本研究中NGG(CCN)PAM分为五种类型:AGG(CCT),TGG(CCA),GGG(CCC),CGG(CCG)和NGG(CCN),其中n是模糊的基对。如果PAM与特定的原始扫描器相关联,则考虑PAM并将其视为特定的PAM。
Cas9切割位点的鉴定
以前的研究表明,Cas9酶在PAM上游的位点3碱基对的靶序列中切割靶序列,但在互补链上可能有几个从PAM上游的裂解位点3-8个碱基对[1]。在这项研究中,我们专注于乳酪上游的裂解位点3个基础对。基于可用的注释信息确定基因组和基因,基因,外显子,内含子和UTR元素中这些切割位点的分布模式。
同时性评估多种强子间和相应基因
存在含有多个原始空间的基因,因此不能单独地有效地编辑。但是,如果这些基因在其序列和功能中类似,则它们可能被编辑为组或基因家族。我们使用类似的基因作为查询序列,并将它们爆炸在本地CDS数据库中以确定这些基因的相似性。对于那些共享等相似之处的基因高于80%,我们建议他们可能是群体编辑的良好候选人。这些基因的原子化器的基因组位置也位于。同步结果用于分析在一起编辑类似基因的可能性。
数据库架构和Web界面
本研究获得的所有数据均存储在Grape-CRISPR数据库(http://biodb.sdau.edu.cn/gc/index.html.).该数据库包含通过MySQL实现的相关关系数据库和通过HTML和PHP实现的Apache web服务器上运行功能的web界面(附加文件3.).数据库基于Linux服务器,可以通过Internet自由访问。它包含CRISPR / CAS9网站属性的信息,例如基因ID,基因组,原始扫描仪,GC含量和推动者适用性。数据库还包含所有Cas9站点之间的关系信息,其中包含每个PFAM模型的候选基因的PFAM注释数据库。接口是使用HTML和CSS编写的。用户查询将上传到系统并由PHP和MySQL或Perl脚本进行处理。
伦理批准和同意参与
不适用
同意出版物
不适用
可用性数据和材料
本文中的原spacers、注释信息和其他所有细节数据都可以从Grape-Crispr数据库(http://biodb.sdau.edu.cn/gc/).
缩写
- CAS:
-
CRISPR-COSSOCIED蛋白
- CRISPR:
-
集群定期间隔的短文重复
- 帕姆:
-
ProTospacer-相邻的主题
参考文献
- 1。
Jinek M,Chylinski K,Fonfara I,Hauer M,Doudna Ja,Charpentier E.一种可编程的双RNA引导的DNA内切核酸酶,适应性细菌免疫。科学。2012; 337(6096):816-21。
- 2。
蒋伟,毕卡德,考克斯,张飞,马拉菲尼。利用CRISPR-Cas系统对细菌基因组进行rna引导编辑。生物科技Nat》。2013;31(3):233 - 9。
- 3.
Cho SW, Kim S, Kim JM, Kim JS。Cas9 rna引导内切酶在人类细胞中的靶向基因组工程。生物科技Nat》,2013,31(3):230 - 2。
- 4。
丛丽,冉发,Cox D,林森,Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA等。利用CRISPR/Cas系统进行多重基因组工程。科学。2013;339(6121):819 - 23所示。
- 5。
邢HL,东路,王ZP,张海,汉联,刘B,王XC,陈QJ。用于植物中的多重基因组编辑的CRISPR / CAS9工具包。BMC植物BIOL。2014; 14:327。
- 6。
Mali P,Esvelt Km,Church Gm。CAS9作为工程生物学的多功能工具。NAT方法。2013; 10(10):957-63。
- 7。
高JP,王GH,MA SY,谢XD,吴XW,张XT,吴毅,赵P,夏Qy。CRISPR / CAS9介导的尼古利亚纳塔卡宫介导的靶向诱变。植物mol biol。2015; 87(1-2):99-110。
- 8。
贾哈格,王N.使用CAS9 / SGRNA的甜橙色针对性基因组编辑。Plos一个。2014; 9(4):E93806。
- 9。
高y,赵玉。拟南芥的特定和遗传基因编辑。Proc Natl Acad Sci U S A. 2014; 111(12):4357-8。
- 10.
Upadhyay SK,Kumar J,Alok A,Tuli R. RNA引导的小麦靶基因突变的基因组。G3-基因基因类型。2013; 3(12):2233-8。
- 11.
江口,周HB,BI HH,FROMM M,YANG B,周DP。拟南芥,烟草,高粱和稻米的CRISPR / CAS9 / SGRNA介导的靶向基因改性的演示。核酸RES。2013; 41(20):E188。
- 12.
Kaur K,Tandon H,Gupta Ak,Kumar M. Crisprgge:基于CrisPr / CAS的基因组编辑的中心枢纽。数据库。2015; 2015:1-8。
- 13。
关键词:crispr - cas9,基因组编辑,引导RNA间隔区,基因组预测摩尔。2014;7(5):923 - 6。
- 14。
雷勇,陆璐,刘海英,李胜,邢峰,陈丽丽。CRISPR-P:一种用于植物crispr系统单导RNA合成设计的网络工具。摩尔。2014;7(9):1494 - 6。
- 15。
Velasco R, Zharkikh A, Troggio M, Cartwright DA, Cestaro A, Pruss D, Pindo M, FitzGerald LM, Vezzulli S, Reid J等。一个杂合子葡萄品种基因组的高质量共识草案序列。《公共科学图书馆•综合》。2007;2 (12):e1326。
- 16。
jaillon o,yury jm,noel b,policriti a,clepet c,casagrande a,choisne n,aubourg s,vitulo n,jubin c等。葡萄树基因组序列表明祖先六倍化在主要的血症植物中。自然。2007; 449(7161):463-7。
致谢
我们感谢Dr. D.D. Archbold(美国肯塔基大学教授)对手稿的英文改进。本研究由国家自然科学基金项目(31572090)和中国科学院百人计划资助。
作者信息
从属关系
相应的作者
附加信息
利益争夺
提交人声明他们没有竞争利益。
作者的贡献
YW和XJL进行了这个实验。ZCL和SHL设计了这个实验。YW,XJL,RC和LY构建了数据库,SHL和ZCL写道和GYZ修改了手稿。所有作者都已经阅读并赞成最终的手稿。
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
王永强,刘旭东,任超。等等。基于CRISPR/ cas9的基因组编辑的基因组位点鉴定vitis Vinifera基因组。BMC植物BIOL.16,96(2016)。https://doi.org/10.1186/s12870-016-0787-3
收到了:
公认:
发表:
关键词
- CRISPR / CAS9.
- 数据库
- 基因组编辑
- p
- vitis Vinifera