抽象的
背景
它被广泛接受耕种米(栽培稻L.)从普通野生稻中驯养(oryza rufipogon.格里夫。)。与浓缩水稻的其他研究相比,该研究是在整个基因组水平的基因上释放从野生水稻到培养水稻的基本上表型和生理变化。
结果
这项研究而不是比较两种组装的基因组,而是直接比较了Dongxiang野生稻(DXWR)illumina测序与Nipponbare读取(o.苜蓿)完全基因组而无需组装DXWR基因组。基于对比目组科分析的结果,确定DXWR和Nipponbare之间的结构变异(SV)定位在水稻驯化期间Nipponbare可以获得缺失的基因。为了克服SV检测的极限,还测序DXWR转录组,并与Nipponbare转录组进行比较,以发现在驯化期间可能在DXWR中丢失的基因。使用来自多种来源的注释进一步分析了1591个Nipponbare获取的基因和206个DXWR丢失的转录物。NGS数据可在NCBI SRA数据库中使用ID SRP070627。
结论
这些结果有助于更好地理解在全基因组水平的培养水稻中的驯化,并为水稻遗传研究或繁殖提供基因组数据资源。一个发现证实的转产元件对来自野生稻的基因组进化有很大贡献,培养米饭。另一种发现表明培养水稻中的光学磷酸化和氧化磷酸化系统可以在驯化期间同时适应环境变化。
背景
耕种米饭(栽培稻L.)作为最重要的农作物之一,提供世界上一半以上的主要饮食来源[1].尽管人们普遍认为栽培稻是由普通野生稻(oryza rufipogon.格里夫)几千年前[2[培养米的起源和驯化过程已经通过不同的研究已经讨论了几十年[3.-5.].直到最近,人们才发现o.苜蓿L. SSP..japonica.是最先被驯化的O. Rufipogon.在中国南方的珠江中部地区,以及o.苜蓿L. SSP..indic随后从十字架之间发展japonica.当地野生大米作为初始品种蔓延到东南和南亚[6.].随着大米来源的众多研究,需要进一步的工作来遗传阐明从野生水稻到整个基因组水平的栽培水稻的基本上表型和生理变化。
本研究在相比之下进行了比较基因组学分析o.苜蓿L. SPP。japonica.var。Nipponbare和Dongxiang野生稻(DXWR),中国常见的野生稻(O. Rufipogon.).1978年在中国江西省东乡县首次发现了DXWR [7.它被认为是世界上发现的最北端(28°14′n)的普通野生水稻种群。在过去的30年里,DXWR作为水稻栽培改良或遗传多样性基础研究的宝贵遗传资源得到了广泛的研究[8.那9.],杂种优势[10.,细胞质雄性不育[11.],生育恢复[12.],生物量[13.,高产量[14.-16.]和对生物和非生物胁迫的抵抗力[17.-21.].
为了进行比较基因组学分析,我们使用下一代测序(NGS)技术测序DXWR的全基因组。在这项研究中,比较基因组学分析的策略是直接比较DXWR NGS与Nipponbare完全基因组的读数,而无需组装DXWR基因组。该策略避免了高度耗时的工作和大量的错误德诺维使用NGS短读取的DXWR基因组组装。该比较策略中的基本作品正在使用软件SVDETECT和管道SVFILTER来检测结构变异(SV),这对其角色越来越欣赏作为表型变化的原因[22.-24.].使用检测到的缺失(一种重要类型的SVS),我们找到了在米驯化期间Nipponbare获得的基因。为了克服SV检测的极限,还测序DXWR转录组,并与Nipponbare转录组进行比较,以发现在驯化期间可能在DXWR中丢失的基因。Both Nipponbare-acquired genes and DXWR-lost transcripts were further analyzed using annotations from multiple sources (e.g. QTLs for traits and KEGG pathways) to reach two research goals: 1) to help better understanding the domestication from wild rice to cultivated rice at the whole genome level; 2) to provide a genomic data resource for rice genetic research or breeding.
结果与讨论
东乡野生稻全基因组测序
利用Illumina测序技术对东湘野生稻(DXWR)基因组进行测序,共获得282,383,842对端90 bp清洁reads (25.4 Gb data),覆盖参考基因组的68倍(o.苜蓿L. SPP。japonica.var。nipponbare)尺寸为373,245,519 bp。该下一代测序(NGS)数据的高深度满足了可靠地检测结构变化(SV)的要求。然后,我们将所有清洁的读数映射到九个完全稻米基因组(方法).使用正序读取的单端对准,映射读取的速度达到总读数达到74.19,56.98,47.11,36.29,32.02,29.30,19.76,4.20和1.18%o.苜蓿L. SPP。japonica.var。nipponbare,o.苜蓿L. SPP。indic那O. Nivara.那O. Glaberrima.那o . barthii那o. glumaepatula.那O. Meridionalis.那O. Punctata.和O. Brachyantha., 分别。由于两个原因,Nipponbare达到了最高的映射率。第一个原因是o.苜蓿被认为是从中国常见的野生稻中驯化[2].第二种原因是使用克隆克隆方法用Sanger测序技术进行测序,并被排名为最佳组装和注释所有水稻基因组。因此,我们使用NipponBare Genome作为检测SVS的参考。
东乡野生稻和Nipponbare基因组之间的结构变化
我们使用SVDetect软件检测DXWR配对端reads与Nipponbare基因组之间的svv,而不进行DXWR基因组的组装。SVDetect的基本理论是利用来自配对端(本研究为500 bp)的顺序、方向和插入大小等先验信息,将映射读分为正常和异常配对读。在删除正常映射的对端读取时,SVDetect使用异常映射的对端读取来产生svv。由于SVDetect会产生大量的假阳性svv,我们开发了一种名为SVFilter的管道来减少假阳性。SVFilter使用五个独立的程序(比率过滤器、间隙过滤器、SNVfilter、覆盖率过滤器和深度过滤器)依次过滤假阳性(方法).在该研究中,SVDETECT产生了13,767个电位SV,并且RATIOFILTER在很大程度上将SV编号减少至3946(28.66%的13,767%)。然后,GapFilter,SNVFilter,CoverageFilter和DeptryFilter缩小到SV编号到3945,3524,2570和2539(附加文件1), 分别。
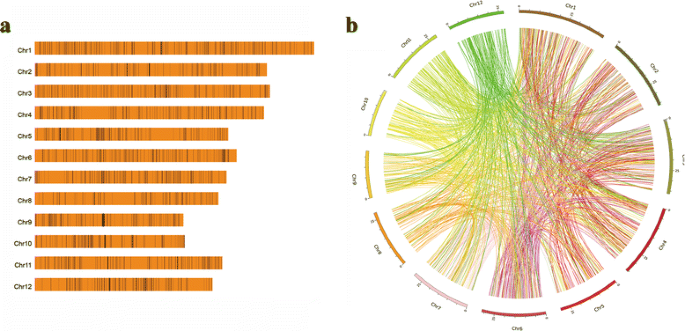
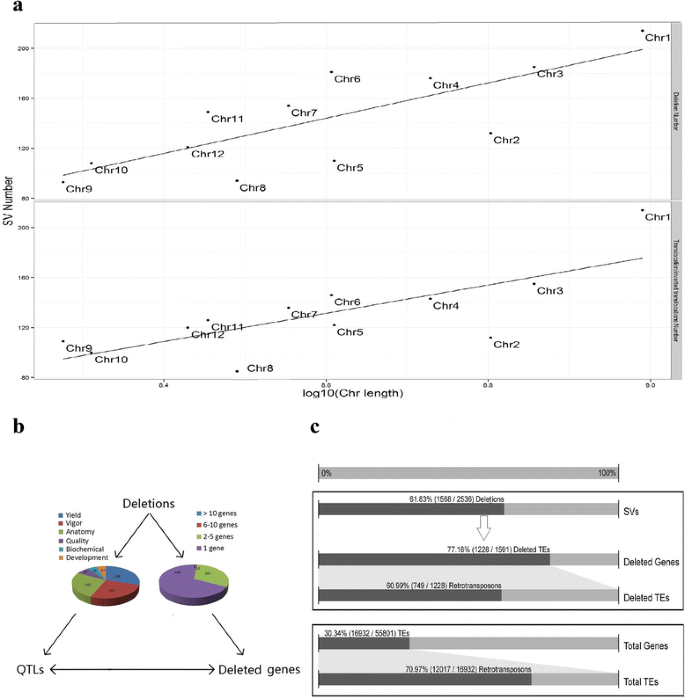
在删除了与内部小缺失重叠的较大缺失后,1571个缺失中有1568个被留作进一步分析。最终鉴定出2536个svv,其中1568个缺失,437个易位,423个倒置易位,88个倒置重复,6个倒置重复,3个倒置片段,1个插入片段和10个未定义的svv。在8种SVs中,缺失占总SVs的61.83%(1568/2536)。1A),其次是易位/倒置易位,占总SVs的33.91%(860/2536)(图。1B.).一般而言,删除号和易位/反相易位数分别具有分别与染色体长度的线性关系(图。2A).除9号染色体外,日本晴基因组的所有染色体均以相同的模式存在缺失和易位/倒位。此外,与其他11条染色体相比,1号染色体包含更多的缺失和易位/倒位。
在米驯化期间发现Nipponbare获得基因
SV检测和过滤过程确定了1568次删除。这些缺失的区域可以含有假设的基因,该基因被Nipponbare在米饭驯化期间被收购。将1568次缺失缩放到Nipponbare Genome中的水稻基因,总缺失的61.47%(964/1568)与1591个基因相关联(附加档案2).在964个缺失中,67.32%(649/964)只包含一个基因。含有十多个基因的前八个缺失从10790002bp到10920963 bp上的基因组区域(CHR10:10790002-10920963),CHR9:10545593-10732410,CHR8:9242722-9284595,CHR2:14239399-14298646,CHR6:23563294-23592857,CHR6:29241098-29330521,CHR2:17681691-17753826和CHR11:11280103-11340819(图。2B.).在日本龙获得的1591个基因中,77.18%(1228/1591)为转座元件(transposable elements, TEs),其中60.99%(749/1228)为逆转录转座子(图1)。2C).在总55,801个Nipponbare基因中,30.34%(16,932 / 55,801)是TES,其中70.97%(12,017 / 16,932),其中是回析组织。TE百分比的两倍以上(77.18 vs.30.34%)差异与先前发现TES对基因组进化从野生稻到栽培稻米作用的贡献一致[25.].
数量性状位点(QTLs)将基因组上的特定区域与农艺性状联系起来。在水稻中,已经鉴定了许多重要农艺性状的QTL,并纳入了Gramene QTL数据库(方法).将删除映射到Nipponbare Genome中的注释米QTL,位于Nipponbare Genome的539个独特地区的731 QTL与937删除相关联(附加文件3.).这731个qtl的表型属于6个性状类别。分别是产量(216个,731个)、活力(197个)、解剖学(192个)、品质(51个)、生化(38个)和发育(37个)。2B.).结合前一步的结果,我们在受删除影响的同一区域构建了QTL和基因之间的关系(附加文件4.).最后,构建了937次缺失,1547个缺失基因和731 QTL的关系进行进一步研究(图。2B.).总结了使用这种关系信息,总结了农艺性状,QTL和相关基因,以帮助更好地了解从野生稻到栽培米的驯化。在该驯化过程中,培养的水稻可以获得涉及植物高度,小穗数,穗数,叶片衰老和穗长的基因,然后是生物质产量,幼苗活力,叶宽,分蘖数和各种其他特征(桌子1).
使用DXWR转录组发现DXWR丢失的基因
在这项研究中,我们只使用500bp配对末端从DXWR基因组进行SV检测。此单个库大小限制了DXWR基因组上检测到的插入的大小,不超过300bp。然后,我们无法检测到大型插入(> 300bp)以在米驯化期间定位DXWR丢失的基因。因此,我们测序并组装了DXWR转录组以发现DXWR丢失的基因。从幼苗叶片和幼苗根中提取总RNA,以构建两个单独的RNA-SEQ文库,其在Illumina Hiseq 2000系统上测序。在数据清洗和质量控制后,总共86,340,332个配对100bp原料读取(8.6 GBP数据)被加工到85,813,832张清洁读数,Q20百分比为99.3%。这些清洁的读数是德诺维组装到DXWR转录组中,过滤掉短于200bp的CONTIG。
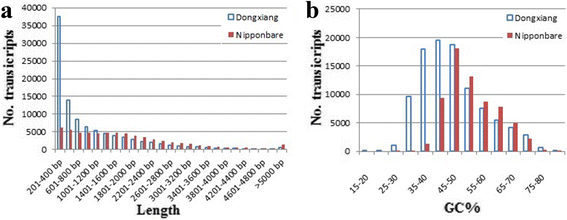
DXWR转录组含有70,747个基因和99,092个转录物,平均长度为968bp和N50长度1655 bp,而Nipponbare转录组含有55,204个基因和65,556转录物,平均长度为1722bp和N50长度2295bp,滤除转录物越短超过200 bp。将组装的DXWR转录组组的长度分布与Nipponbare转录组的长度分布进行比较(图。3A).从图中3A,可以看出,DXWR和Nipponbare转录物的数量以类似的模式的转录长度减少,其成绩称短于1000bp。特别是,DXWR具有比Nipponbare更短的转录物(<500 bp)。这种差异主要是由于这些DXWR转录物的不完全组装使用NGS短读数。虽然45,737(99,092%的46.16%)DxWr转录物的长度小于500bp,但具有长度至少500bp的53,355(99,092%的99,092%的53.84%)仍然提供进一步研究的丰富信息。此外,Nipponbare具有比DXWR的高GC转录物(图。3B.).前一项研究表明,水稻和其他谷物中的高GC基因具有更大的偏差,密码子使用偏向于高表达基因中优选的密码子[26.].
使用Program Blastx,60,247(60.8%的99,092%)转录物通过其前20个类似序列(命中)的功能描述来注释,如果它们存在,来自NCBI非冗余蛋白(NR)数据库(附加文件5.).使用软件BLAST2GO,35,596(35.92%的99,092%)转录物通过基因本体(GO)植物的SLIM术语在三个结构域,分子功能,细胞组分和生物过程中注释。通过将GO SLIM术语进行映射到酶代码和基因和基因组(KEGG)数据库的京都百科全书(KEGG)数据库(4.59%的99,092%)转录物被分配到137 kegg途径(附加档案6.).利用blastn程序,将99092份DXWR转录本定位到日本龙的转录组中,将409份未定位的转录本定位到日本龙的线粒体cDNA、叶绿体cDNA和核基因组序列中。最后,从205个基因中鉴定出206个转录本为dxwl在驯化过程中丢失的转录本。
进一步分析Nipponbare获取和DXWR丢失的基因
为了进一步分析日本晴获得的1591个基因,将其编码的1652个推测蛋白分别定位到KEGG通路、已知转录因子(TFs)和蛋白激酶(PKs)上。有意义的结果包括13个涉及光合作用途径的基因(图。4.),11个涉及氧化磷酸化途径的基因(图。5.),三个由两个基因编码的推定蛋白质(loc_os08g33488和LOC_Os12g31748)属于转录因子MIKC,由三个基因(LOC_Os09g29540那LOC_Os09g29560和loc_os09g29584)属于壁相关激酶(WAK)或壁相关激酶样激酶家族,由该基因编码的7种推定蛋白loc_os01g20900.属于细胞质激酶VIII系列等受体和由基因编码的五种推定蛋白质loc_os01g27020属于CDC2,如激酶家族。进一步的分析显示,1591个Nipponbare获取的基因中的18个只有一种副本在基因组中。这些18个基因编码来自HNH内切核酸酶的蛋白质,海藻糖磷酸酶,MYB系列转录因子,泛素 - 蛋白质连接酶1和肽转运蛋白PTR2,et al。(附加文件7.).进一步分析206例DXWR丢失的转录物显示出18个具有功能注释,其中三种富含氧化磷酸化途径。这三个转录物编码三种蛋白质,即细胞色素B,细胞色素氧化酶亚基I和NAD依赖性醛脱氢酶(附加文件7.).
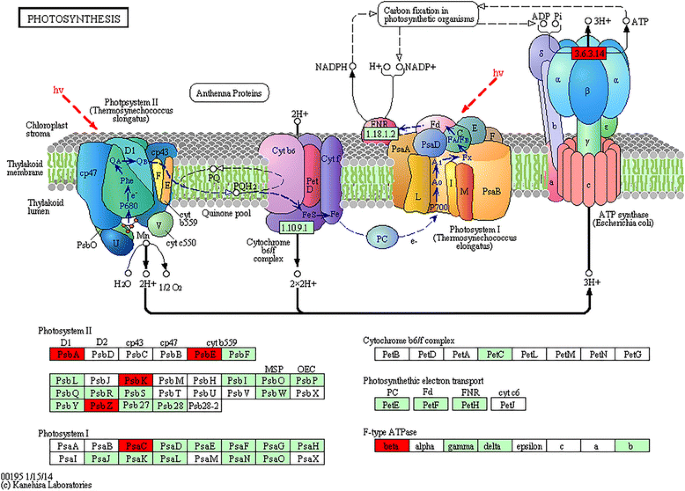
具有Nipponbare获取基因的光合作用途径。此数字(KEGG:MAP00195)从Kegg网站下载,具有复制权限权限。在1591个Nipponbare获得的基因中,将13个光合作用相关基因映射到六种蛋白质(附加文件7.:表S2)。它们是光电系统II PSBA,照片II PSBE,Photosystem II PSBK,Photosystem II PSBK,Photosystem II PSBZ,Photosystem I PSAC和F型ATPase Beta(红色的)
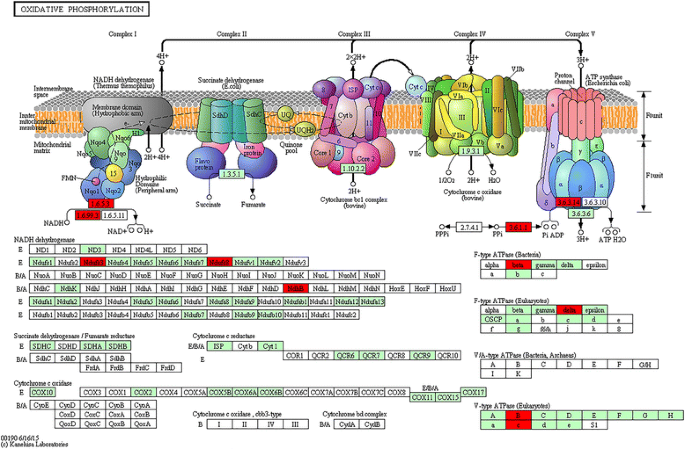
氧化磷酸化途径与Nipponbare获取基因。此数字(kegg:map00190)从Kegg网站下载,具有复制权限权限。在1591个Nipponbare获得的基因中,将11个氧化磷酸化相关基因映射到八个蛋白质(附加文件7.:表S3)。它们是NADH脱氢酶E Ndufs3, NADH脱氢酶E Ndufs8, NADH脱氢酶B/A NdhB, f型atp酶(细菌)beta, f型atp酶(真核生物)delta, v型atp酶(真核生物)B, v型atp酶(真核生物)c和无机焦磷酸酶[EC:3.6.1.1] (红色的)
MIKC作为MADS-box转录因子的一种,参与了植物生长发育的多个方面。对75个MADS-box基因进行了全基因组鉴定和系统发育分析,结果表明:1)日本晴获得的MADS-box基因loc_os08g33488(OsMADS23) 和LOC_Os12g31748(OsMADS20)分别分别分为AgL17样和Squa样组;2)这两个疯狂的箱体基因遗传地远离其群体内的其他基因[27.].WAK基因家族在细胞膨胀,病原体抵抗和重金属胁迫耐受性中起重要作用拟南芥.由于单像的家庭的谱系特异性扩张,含有125个成员的奥波瓦基基因族含有125个成员的植物基因组[28.].nipponbare获得的基因LOC_Os09g29540(OsWAK82),LOC_Os09g29560(OsWAK83) 和loc_os09g29584(OsWAK84)分为OsWAK- rlck型、OsWAK伪基因型和OsWAK- rlp型[28.], 分别。
总共,在光合作用途径中富集了13种Nipponbare获取的基因,富含11个Nipponbare获取的基因和三个DXWR丢失的转录物,富含氧化磷酸化途径(附加文件7.).光磷酸化和氧化磷酸化都是质膜ATP酶的潜在来源,它驱动磷酸化反应释放能量。先前的研究表明,这两种能源系统合作提供能源。一个例子是线粒体氧化磷酸化作用似乎在光合作用中为细胞质提供ATP起着重要作用[29.].另一个例子是在黑暗供应能量中的光和氧化磷酸化中的光学磷酸化,用于在表皮皮下的表皮皮上开口Commelina Communis.土地维亚比亚法瓦L. [30.].以前的研究还表明,栽培的水稻具有比野生大米更高的光合效率,产生高收益率[31.那32.].这些发现在一起表明米饭中的光学磷酸化和氧化磷酸化系统可以在野生稻的驯化期间同时适应环境变化。
结论
在这项研究中,我们表现出东乡野生稻(DXWR)的全基因组测序,中国常见的野生稻。使用SVDETECT和管道SVFILTER,在DXWR和参考基因组Nipponbare之间确定了2536个结构变体(SV)。在2536秒的SV中,使用1568次删除来定位1591个删除基因,这些基因被假设,以在其驯化期间被Nipponbare获得。为了克服DNA文库尺寸限制,测序DXWR转录组,与Nipponbare转录组进行比较,得到在米驯化期间获得206例DXWR丢失的转录物。进一步分析Nipponbare获取的基因和DXWR丢失的转录物显示出在光合途径中富集了13种Nipponbare获得的基因。此外,在氧化磷酸化途径中富集了11个Nipponbare获得的基因和三种DXWR丢失的转录物。米饭中的光学磷酸化和氧化磷酸化系统可以在野生稻驯化中同时适应环境变化。
在该研究中,建立了937次缺失,1547个缺失基因和731 QTL之间的关系,以提供有价值的进一步研究资源。使用这种关系信息,总结了农艺性状,QTL和相关基因,以帮助更好地了解野生水稻的基本上表型和生理变化,在整个基因组水平上培养水稻。该信息还可用于指导未来的水稻遗传研究或繁殖的实验。
方法
东乡野生稻DNA和RNA测序
东乡野生稻(DXWR)是前原因江西省农业科学院,江西南昌(http://www.jxaas.com/index.html),DXWR的种子可免费获得科学研究。为了避免外来花粉的干扰并降低杂合子,DXWR通过装袋栽培超过十年进行自授粉。本研究中使用的种子是通过来自江西农业科学院的物质转移协议(MTA)获得。首先,DXWR的种子在实验站的稻田中的自然条件下生长。然后,从稻田的DXWR种子植入生长室(28±2℃,14h /日和10h /夜)以获得用于DNA和RNA提取的幼苗。在此报道的实验研究符合有关植物基因库库的制度,国家和国际指南。
DNA从幼苗叶片中提取,总RNA分别从四叶期幼苗叶片和幼苗根中提取。使用Illumina TruSeq DNA Sample Prep Kit构建500 bp插入大小的DNA-seq文库,并在Illumina HiSeq 2000系统上使用90 bp对端技术进行测序。使用Illumina TruSeq RNA Sample Prep Kit构建了两个插入长度为200 bp的非链特异性RNA-seq文库,并在Illumina HiSeq 2000系统上使用100 bp对端技术进行测序。使用Fastq_clean管道对DNA-seq和RNA-seq数据进行清洗和质量控制[33.],它被优化以清除来自Illumina平台的原始读取[34.-41.].从DNA-SEQ数据中删除了低质量( 从Ensembl数据库V22下载九个完整的基因组数据(ftp://ftp.ensemblinomes.org/pub/3-22/plants.).具有参考基因组的注释的序列(o.苜蓿L. SPP。japonica.var。NipponBare)在Ensembl V22中与IRGSP v1.0和MSU_OSSR v7.0中的数据相同(MSU RIES GEAMOME ANNORATION项目版本7)。基因和蛋白质注释使用MSU_OSSR v7.0(http://rice.plantbiology.msu.edu/).米QTL数据从Gramene数据库V40下载(http://www.grachene.org.)[42.].去除冗余后,QTL记录数量由8216条减少到5165条。 软件Trinity r20140717 [43.]组装DXWR转录组。软件领结v0.12.7 [44.用于将清洁的DNA对准九种完全稻米基因组,允许两个不匹配。BWA v0.5.7-R1310软件用于将清理的DXWR DNA对准,以进行SV检测的Nipponbare基因组。使用SVDETECT V0.8b进行SV检测[45.].管道Svfilter是在康奈尔大学飞行的实验室开发的,可以从中下载http://bioinfo.bti.cornell.edu/tool/SVFilter/.使用SVFilter需要引用本文。命令行中bwa、SVDetect和SVFilter的参数在附加文件中提供7..使用内部Perl程序进行对基因或QTL的映射删除。基因组图(例如图。1)用Circos v0.66软件绘制[46.].使用软件R v2.15.3进行统计和绘图,与包装gpplot2 [47.]. NGS数据可在NCBI SRA数据库中使用ID SRP070627。所有支持数据都包含在附加文件中。 不适用。 不适用。水稻基因组、MSU注释和QTL数据
软件和计划
数据和材料的可用性
伦理
同意发布
缩写
- 装备:
-
染色体
- DXWR:
-
东乡野生稻
- 去:
-
基因本体论
- Kegg:
-
Kyoto基因和基因组的百科全书
- ngs:
-
下一代测序
- NR:
-
非冗余蛋白质
- PK:
-
蛋白质激酶
- QTLS:
-
定量特质基因座
- SV:
-
结构变异
- TE:
-
可转换元素
- TF:
-
转录因子
- WAK:
-
墙相关的激酶
参考文献
- 1。
赵克,桐树c-w,eizenga gc,wright mh,ali ml,价格ah等。基因组 - 范围的协会映射揭示了奥里佐苜蓿中复杂性状的丰富遗传建筑。NAT Communce。2011; 2:467。
- 2。
oka h-i。栽培米的起源。费城:elestvier;2012年。
- 3。
隆多太平绅士,蒋耀中,洪国辉,蒋廷耀,BA。亚洲野生稻(Oryza rufipogon)的系统地理学揭示了栽培稻(Oryza sativa)的多个独立驯化。中国科学院院刊。2006;103(25):9578-83。
- 4。
Molina J,Sikora M,Garud N,Flowers JM,Rubinstein S,Reynolds A等人。单一进化驯化稻米进化起源的分子证据。PROC NATL ACAD SCI。2011; 108(20):8351-6。
- 5。
何志强,翟伟,文华,唐涛,王颖,陆晓霞,等。水稻基因组的两个进化史:驯化基因的作用。公共科学图书馆麝猫。2011;7 (6):e1002100。
- 6。
黄X,kurata n,wei x,wang z-x,王a,zhao q等。水稻基因组变异的地图揭示了栽培水稻的起源。自然。2012; 490(7421):497-501。
- 7。
高路,陈某,江W,葛,洪D,王X.北部边际人口常见野生稻田紫外线的遗传侵蚀。及其保护,通过群体遗传结构的变化揭示。秘密人。2000; 133(1):47-53。
- 8。
董烨,李F,Pei XW,王F,袁Qh,吴Hj等。基因组多层多点分析奥里扎Rufipogon Griff中的内部分化。来自中国的影响与O. Sativa L. Genet Mol Res的影响。2013; 12(4):6103-19。
- 9。
宋志平,徐旭,王斌,陈家宽,卢斌斌。利用SSR标记估算北缘稻属居群的遗传多样性。应用计算机学报。2003;107(8):1492-9。
- 10。
罗晓军,辛晓燕,杨建生。东乡普通野生稻杂种优势位点的遗传基础分析。麝猫研究》2012;94(2):57 - 61。
- 11.
X-H S,Song Y,Huang R-L,Shan Z,Xiong H-L,Shen L-J。东乡野生稻(Oryza Rufipogon)的新型细胞质雄性无菌源的发展。米科。2013; 20(5):379-82。
- 12.
陈晓荣,杨克生,傅建荣,朱长林,彭晓生,何晓平,等。东乡野生稻育性恢复能力鉴定及遗传分析。水稻科学。2008;15(1):8。
- 13。
董旭,王旭,张磊,杨志新,辛旭,吴胜,等。水稻生物量和小穗数增加相关基因OsEBS的鉴定和特性分析。植物生态学报;2013;11(9):1044-57。
- 14。
李罗他G, X,田F, K,朱Z,苏W, et al。水稻数量性状位点相关基因簇的结构和表达的单倍型变异。基因组研究》2006;16(5):618 - 26所示。
- 15.
田F,李德杰,傅Q,朱紫玉,傅YC,Wang XK等。携带野生稻(Oryza Rufipogon Griff.)栽培水稻(Oryza Sativa L.)背景的凝集线的构建及其与产量相关性状相关的狭窄区段的段。标签护理苹果苹果类型。2006; 112(3):570-80。
- 16。
Zha X,Luo X,钱X,He G,Yang M,Li Y等。水稻LRK1基因的过表达改善了定量产量组分。植物Biotechnol J. 2009; 7(7):611-20。
- 17。
刘F,Sun C,Tan L,Fu Y,Li D,Wang X.识别和定位定量特质基因座控制中国普通野生稻(O. Rufipogon Griff。)在开花阶段启动。Chin Sci Bull。2003; 48(19):2068-71。
- 18。
李F,Guo S,Zhao Y,Chen D,Chong K,Xu Y.来自Dongxiang野生稻的含常肽重复的BHLH蛋白基因(OrbHlH001)的过度表达赋予转基因拟南芥中的冻结和耐盐耐受性。植物细胞代表2010; 29(9):977-86。
- 19。
萧,黄某,李啊,gao y,li yh,pan ch,等。QLOP2和QPSR2-1基因座的精细映射与野生米幼苗的冷胁迫耐受性相关。标签护理苹果苹果类型。2015; 128(1):173-85。
- 20.
肖宁,黄卫宁,张晓霞,高艳,李阿,戴勇,等。水稻幼苗期和成熟期根系耐冷性数量性状位点qRC10-2的精细定位《公共科学图书馆•综合》。2014;9 (5):e96046。
- 21.
关键词:东乡普通野生稻,抗旱性,渐渗系植物学报。2006;62(1-2):247-59。
- 22。
Springer NM,Ying K,Fu Y,Ji T,Yeh C-T,Jia Y等。玉米自扰会在基因组含量中表现出高水平的拷贝数变异(CNV)和存在/不存在变化(PAV)。Plos Genet。2009; 5(11):E1000734。
- 23。
迈克海尔路,胡安WJ徐WW, Bhaskar PB,安德森我,海顿耐DL, et al。大豆基因组的结构变异定位于生物胁迫反应基因簇。植物杂志。2012;159(4):1295 - 308。
- 24。
Feuk L,Carson AR,Scherer SW。人类基因组的结构变异。NAT Rev Genet。2006; 7(2):85-97。
- 25。
Bennetzen JL。转换元素对植物基因和基因组进化的贡献。植物mol biol。2000; 42(1):251-69。
- 26。
郭晓霞,包建军,范丽丽。水稻基因密码子选择性驱动的研究进展。2月。2007;581(5):1015 - 21所示。
- 27。
Arora R,Agarwal P,Ray S,Singh Ak,Singh VP,Tyagi Ak等。稻米的疯狂箱体基因家族:生殖发展和压力期间的基因组识别,组织和表达分析。BMC基因组学。2007; 8(1):242。
- 28。
张树华,陈晨,李丽,陈晨,姜宁,等。水稻壁相关激酶基因家族的进化扩展、基因结构和表达。植物杂志。2005;139(3):1107 - 24。
- 29。
Krömers,stitt m,holdt hw。参与叶细胞光合代谢的线粒体氧化磷酸化。费用。1988; 226(2):352-6。
- 30.
王志强,王志强,王志强。气孔开启的代谢能。光磷酸化和氧化磷酸化的作用。足底。1984;161(2):129 - 36。
- 31.
朱X-G,Long SP,Ort Dr。提高光合效率提高产量。Annu Rev植物Biol。2010; 61:235-61。
- 32.
水稻和小麦的光合作用、产量和氮素利用。植物杂志。2011;155(1):125 - 9。
- 33.
张M,Sun H,Fei Z,Zhan F,Gong X,Gao S. FastQ_CLean:优化的管道,用质量控制清洁Illumina测序数据。在:生物信息学和生物医学(BIBM),2014年IEEE国际会议:2014年纽约:IEEE;2014. p。44-8。
- 34。
周D,Gao S,Wang H,Lei T,Shen J,Gao J等。De Novo测序特生物龙莲属straminea(GentiaCeae)的转录组,以鉴定参与活性成分生物合成的基因。基因。2016; 575(1):160-70。
- 35。
HOLL HM,GAO S,FEI Z,ANDREWS C,Brooks SA。从马拉微组织产生DE Novo转录组。BMC基因组学。2015; 16(1):1。
- 36。
Feder A,Burger J,Gao S,Lewinsohn E,Katzir N,Schaffer Aa,等。含有Kelch结构域的F盒编码基因负调节Cucumis Melo L.植物理性的黄酮覆抗力。2015; 169:1714-26。
- 37。
陈y-r,zhong s,fei z,gao s,张s,李z等。杆状病毒综述Californica多核多核病毒的宿主Trichoplusia Ni感染的转录组响应。J病毒。2014; 88(23):13781-97。
- 38。
徐y,gao s,yang y,huang m,cheng l,wei q等。脱水应力下菊花的转录组测序和全基因组表达分析。BMC基因组学。2013; 14(1):662。
- 39。
郭胜,张建军,孙浩,Salse J, Lucas WJ,张华,郑勇,毛磊,任勇,王铮,等。西瓜(Citrullus lanatus)基因组草案和20份不同种质的重测序。Nat麝猫。2013;45(1):51-8。
- 40。
李锐,高胜,Hernandez AG, Wechter WP, Fei Z, Ling K-S。番茄小rna的深度测序用于病毒和类病毒鉴定和菌株分化。《公共科学图书馆•综合》。2012;7 (5):e37127。
- 41。
Consortium TTG。番茄基因组序列提供了肉质果实演化的见解。自然。2012; 485(7400):635-41。
- 42.
Youens-Clark K,Buckler E,Casstevens T,Chen C,Deplerck G,Derwent P,Dharmawardhana P,Jaiswal P,Kersy P,Kerthikeyan A. Gramene数据库于2010年:更新和扩展。核酸RES。2010; 39(S2):GKQ1148。
- 43.
Friedman N,Grabherr Mg,Haas Bj,Yassour M,Levin JZ,Thompson Da,Amit I,Adiconis X,Fan L,Raychowdhury R等人。来自RNA-SEQ数据的全长转录组件没有参考基因组。NAT BIOTECHNOL。2011; 29(7):644-52。
- 44.
Langmead B,Trapnell C,Pop M,Salzberg SL。超快和记忆高DNA序列对人类基因组的对准。基因组Biol。2009; 10(3):R25。
- 45.
Zeitouni B, Boeva V, Janoueix-Lerosey I, Loeillet S, Legoix-Né P, Nicolas A, Delattre O, Barillot E. SVDetect:从配对端和配对测序数据中识别基因组结构变化的工具。生物信息学,2010,26(15):1895 - 6。
- 46。
kzywinski M, Schein J, Birol I, Connors J, Gascoyne R, Horsman D, Jones SJ, Marra MA。Circos:比较基因组学的信息美学。基因组研究》2009;19(9):1639 - 45。
- 47。
Wickham H. ggplot2:用于数据分析的优雅图形。柏林施普林格Science & Business Media;2009.
致谢
我们感谢Zhangjun Fei副教授的帮助在Boyce Thompson植物研究所,康奈尔大学和南开大学生命科学学院文俊布教授。本研究中的数据分析得到了人口和健康翻译癌症医学特价的国家科学数据共享平台。
资金
国家自然科学基金项目(no . 31201191, no . 31360327);江西省自然科学基金项目(no . 20132BAB214009, no . 20142BAB204012);公益性行业(农业)科研专项(no . 201103007);江西省教育厅重点项目(KJLD12059)和中央高校基本科研业务费专项资金资助。
作者信息
从属关系
相应的作者
附加信息
利益争夺
提交人声明他们没有竞争利益。
作者的贡献
SG和JX构思并监督了该项目。FZ, SY和RC进行了实验。LM、ZW、XL对数据进行了分析。SG起草了主要的手稿。TX和XC对手稿进行了修改,并准备了所有的数字、表格和附加文件。所有作者均已阅读并批准本稿件。
附加文件
额外的文件1:
本文件报告东乡野生稻和Nipponbare之间检测到的2539 SVS(=链接)作为参考。这个文件是以制表符分隔的格式,可以使用Excel软件打开。(TXT 1496 KB)
附加文件2:
该文件报告将1568缺失映射到参考基因组中的水稻基因。这个文件是以制表符分隔的格式,可以使用Excel软件打开。(TXT 268 KB)
额外的文件3:
该文件报告将1568个删除映射到qtl。这个文件是以制表符分隔的格式,可以使用Excel软件打开。(TXT 1015 kb)
附加文件4:
该文件记录参考染色体上同一区域QTL和水稻基因之间的关系。这个文件是以制表符分隔的格式,可以使用Excel软件打开。(TXT 2342 KB)
附加文件5:
该文件使用blast2go软件对NCBI NR数据库中的99,092份DXWR转录本进行了功能注释。这个文件是以制表符分隔的格式,可以使用Excel软件打开。(TXT 12649 kb)
额外的文件6:
此文件报告137 kegg路径为4548 DXWR成绩单。这个文件是以制表符分隔的格式,可以使用Excel软件打开。(TXT 275 KB)
额外的文件7:
表S1。1591个日本人获得的基因中有18个。表S2。13个Nipponbare获取的基因涉及光合作用途径。表S3。11个涉及氧化磷酸化途径的Nipponbare获取基因。表S4。18个dxr在驯化过程中丢失的转录本。最后一节记录了bwa、SVDetect和SVFilter中使用的所有参数。(医生126 kb)
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
张芳,徐涛,毛莉。et al。东乡野生稻的基因组分析(oryza rufipogon.研究水稻驯化过程中丢失/获得的基因。BMC植物BIOL.16,103(2016)。https://doi.org/10.1186/s12870-016-0788.2.
收到了:
公认:
发表:
关键字
- 东乡野生稻
- 全基因组测序
- 转录组
- 比较基因组学分析
- 结构变异