摘要
背景
小热休克蛋白(sHSP)是一个广泛的蛋白家族。SHSP存在于所有王国,它们在植物抗逆性机制中发挥关键作用(以及在病原微生物中,并与人类疾病有关)。
结果
sHSPdb(小型热休克蛋白数据库)是一个集成的资源,包含非冗余的,全长的和精心策划的热休克蛋白序列,根据氨基酸基序和物理化学性质进行分类。sHSPdb收集各种数据库(Uniprot, PFAM, CDD, InterPro)定义的sHSP数据。它提供了一个用于从整个数据库检索信息的浏览器界面,以及一个使用各种标准检索条目精细化子集的搜索界面。物理化学性质,氨基酸组成和组合计算为每个条目。sHSPdb提供所有sHSP属性的自动统计分析。在各种可能性中,sHSPdb允许BLAST搜索,选定序列的对齐和序列提交。
结论
sHSPdb是一个包含所有王国sHSP信息的新数据库。sHSPdb提供了sHSP的分类,以及分析sHSP结构-功能关系的工具和数据。数据主要与sHSP氨基酸序列的各种理化性质有关。sHSPdb可以在http://forge.info.univ-angers.fr/~gh/Shspdb/index.php。
背景
热休克蛋白(HSP)是当细胞暴露在高温或其他压力下时表达增加的蛋白质。在HSP中,sHSP属于蛋白伴侣蛋白超家族。它们抵消了错误折叠蛋白质的不可逆聚集。sHSP是一种普遍存在于所有生物体内的蛋白质,从细菌到哺乳动物,尤其是植物[1- - - - - -4]。此外,sHSP表现出多种亚细胞定位和/或组织分布[3.,5]。
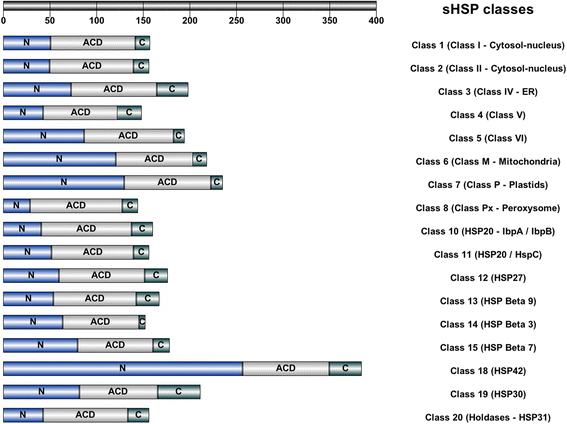
sHSP包含:(i)高度可变的(长度和序列)n端结构域;(ii)一个非常保守的中心结构域,称为α结晶蛋白结构域(ACD),参与二聚化,导致高阶sHSP结构的构建块;(iii)连接二聚体内单体的短c端结构域[6- - - - - -8]。对所有合并的sHSP的研究表明,ACD的平均长度为90±10个残基[9这个长度比N端和c端结构域更稳定。但是,我们将sHSP划分为21个类别,在按类别考虑sHSP时,ACD平均长度的这个值不再适用。ACD的n端部分可能不是二聚化或伴侣活性所必需的,但似乎是高阶聚集体形成所必需的[10]。
作为伴侣网络的一员,sHSP在高温和其他胁迫下的细胞中发挥重要作用,但其功能分子机制尚未完全阐明。sHSP单体(12-42 kDa)通常结合成主要由24个亚基组成的大的同型低聚物[9,11]。由于在不同的细胞条件下其寡聚态的变化或其亚基的交换,sHSP四元结构具有高度的可塑性。亚基组织的松动导致更多的动态特性,从而增强了客户蛋白的可用伴侣位点。例如,研究表明序列RLFDQ(发现于HSP27 - 12类的ACD的n端部分)有助于其亚基的高阶组装和结构稳定性[12]。许多sHSP的结构研究带来了单体,低聚态转变的细节[13- - - - - -15]和sHSP伴侣机制[12,16]。
但是,这种方法不允许一次比较多个sHSP。相反,sHSPdb提供了对sHSP的大规模分析。此外,由于sHSP是一个非常大的蛋白质家族,因此在注释、定义和术语方面存在很大差异:sHSPdb根据选择性基序、理化性质和先前的分类对sHSP进行了分类[17- - - - - -19]。sHSPdb包含多个函数来搜索和描述不同的sHSP。sHSPdb提供了sHSP物理化学性质、氨基酸使用、文献以及用户建立的任何sHSP数据集的统计分析的全局视图。这样的资源对于sHSP结构-功能关系的计算分析是非常宝贵的,特别是关于这三个域中每个域的具体作用。sHSPdb用于收集来自所有领域的shsp的信息。sHSPdb提供了最大的sHSP序列集,一个新的分类和每一类sHSP的精确描述。sHSPdb提供的理化性质和统计分析为实际加号。
结构与内容
数据集的数据源和特征
为了填充sHSPdb,我们使用了一个两阶段的过程。第一步使用PHP和perl脚本自动填充数据库表的字段,从以下数据库中的文本和XML文件中获取信息:NCBI/Proteins, NCBI/CDD, NCBI/Taxonomy, EBI/Picr, UNIPROT, Interpro, SANGER/Pfam, AMIGO。从NCBI加入号开始,我们检索了«GenPept导出地理标志编号、最终的PFAM、CDD和Interpro [8,20.,21标识符和文本信息。然后,利用EBI/Picr的交叉引用,获得Uniprot的登录号和名称,完成并复核数据。填充sHSPdb的主要来源是包含在«中的信息GenPept*文件和相应的Uniprot文件。首要请求得到«GenPept来自公共数据库NCBI的文件包含布尔和关键字的构造。这允许检索≈17,300«GenPept*存放在sHSPdb中的文件。然而,对于许多生物体来说,序列的冗余度非常高。第二步是手动检查,以确保所有信息和链接都与sHSP问题相关。使用以下方法检查文件注释和序列的相关性:(i) sHSPdb«搜索»选项(见下文);(ii) BLAST同源性分析和多重比对软件;(iii) MOCAR (http://forge.info.univ-angers.fr/~gh/wstat/Mocar/),一个专门为我们基于任何Fasta序列数据集的模式基序识别的sHSP分类开发的程序;(iv)«分析和«统计分析在sHSPdb中实现的工具;(v)最后,将确定的每一类sHSP的motif进一步用于PHI-Blast,以获得额外的序列。
序列冲突(特别是在AGI条目的情况下)拟南芥)使用Uniprot文件中的注释进行检查。使用为sHSPdb进料开发的程序去除相同的序列。这允许检测截断的,模糊的(例如,注释的“未命名的蛋白质”,“假设的”,“未知的产品”…,尽管一些少数填充的sHSP类可能包含这样的注释文件)或错误的sHSP序列。包含未定义氨基酸符号(X)的序列被删除。所有文件都存储在sHSPdb中,但只有非冗余的、全长的和带注释的文件是可访问的。
两个持有伴侣蛋白家族,即HSP31和HSP33,不能被正式认为是sHSP,因为它们不含ACD结构域,并通过Cys和二硫键形成与客户蛋白相互作用[22]。然而,这些受氧化还原调控的分子伴侣具有与sHSP相近的物理化学性质,也有助于应激条件下的蛋白质稳态。此外,它们是氧化还原调节的分子伴侣:它们保护热未折叠和氧化损伤的蛋白质免受不可逆聚集,并在细菌防御氧化应激系统中发挥重要作用。因此,将HSP31和HSP33这两个类纳入到sHSPdb中。
sHSPdb的主要目标是分析sHSP的结构-功能关系。这就是为什么只执行氨基酸序列。今天,用户仍然可以BLASTX任何核苷酸序列针对sHSPdb来检查类似或同源sHSP的存在。
最后,sHSPdb包含约4800个经过整理的非冗余全长sHSP序列。3200多个序列(67%)根据其对应的独特序列基序被划分为21类。根据各种标准、眼睛检查和我们的专业知识,大约有400个额外的序列被分类。其余未分类的sHSP序列被归为99类,其中约530个序列被标记为Hsp20。即使我们期望99类的序列属于哪个类,它们也还没有被分类,因为它们不完全匹配任何已定义的基序的正则表达式。由于没有程序可以自动确定任何一组序列的复杂判别基序,我们目前正在开发适当的方法来解决这个问题。特别是,我们正在使用最近的约束规划方法,该方法基于一个两阶段规划,其最后阶段简化为经典的最小集覆盖问题,尽管到目前为止,尚未实现所有类的完整和“完美”表征。部分结果已经公布[23,24]。
序列主要来源于≈2355种细菌,≈2259种真核生物(其中≈1050种病毒植物)和≈129种古细菌(表1)1)。
sHSPdb还包含约14,240个不可访问条目(即,在我们的策展后未保留)。这个数据集在更新数据库和进一步分析sHSPdb时很重要,因为它可以用于统计研究,并禁止在策展集中出现错误的条目。
sHSPdb的组织结构和主要特点
在主页上提供了一个导览链接,以了解如何使用sHSPdb的界面。这个导游提出了各种搜索场景。用户可以通过各种参数选择任何条目,并使用实现的工具进行进一步分析。为此,sHSPdb有三个主要特性:(i)浏览模式,允许用户查询全部或部分数据库;(ii)基于多个搜索条件的搜索方式;(iii)以不同格式检索序列的导出模式。
浏览模式
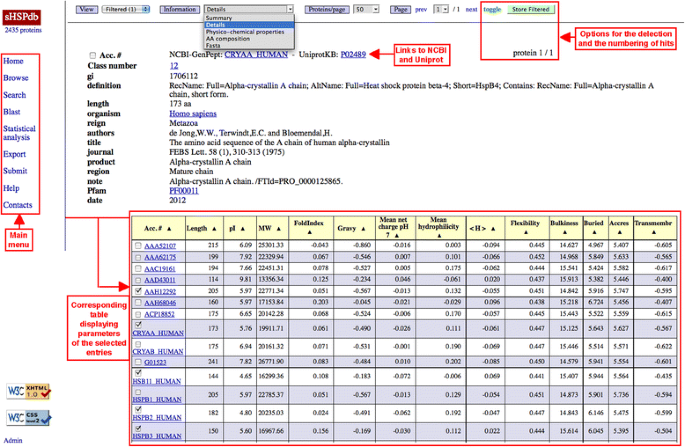
它允许查询整个数据库(图2)。1)。«总结»选项提供NCBI-GenPept加入号和Uniprot加入号,序列和生物体的名称,sHSP的假定功能(如果有的话)。«细节选项提供更多信息(来自GenPept或Uniprot文件的最多20个字段)。加入号,生物体的名称,PFAM, CDD和InterPro号提供了到相关网站的链接。通过选择,得到了一系列的物理化学性质(长度/pI/MW/ foldinindex /GRAVY/ pH7下的平均净电荷/平均亲水性/平均疏水性< H>/柔韧性/体积/残基的可及性/残基的跨膜倾向)物理化学性质»选项。«AA排版»选项显示sHSP的氨基酸组成。最后,«Fasta»选项提供Fasta格式的所有序列。«对齐序列»选项允许对齐选定的序列。
搜索模式(多个搜索功能)
它对应于具有多个参数的高级搜索,并允许检索非常精细的数据子集(搜索示例:来自拟南芥,包含与关键词“染色体”相关的第一类基序,由一位名为Walker的作者于2012年发表,长度由150至200个氨基酸组成。搜索可以通过使用接受通配符和布尔值的复杂文本表达式进行-此搜索适用于整个数据库的所有字段;通过加入号(接受通配符)-一个或多个加入号(s)由一个空格分隔可以一次搜索;通过生物;按PFAM或CDD编号;按日期;氨基酸序列的长度范围。用户也可以检索序列通过输入氨基酸基序精确或退化使用正则表达式与复杂的语法,允许指定复杂的基序。已知的母题(表1)2)在sHSPdb中实现。
用户可以使用sHSPdb中实现的BLASTP进行相似度搜索。这允许获得额外的信息,并通过经典BLAST输出接口检索最佳得分序列。也可以对sHSPdb的核苷酸序列进行BLASTX,以检查是否存在类似或同源的sHSP,例如,从EST构建推定的sHSP cDNA序列。
(i)当检索到条目时,信息可以在浏览模式的任何视图中显示,并且根据在NCBI找到的相关信息,将诸如加入号、分类法、PFAM、CDD和InterPro号码等字段链接到它们的原始网站;(ii)用户可以选择或取消选择从他的搜索(«切换»选项),最后的选择可以被存储。这适用于所有显示(浏览模式,BLAST输出,…);(iii)一个原始点是将表内显示的数据重新排序(升序或降序排序),以便更好地比较和分析数据,使用任何显示的信息作为排序标准;(iv)用户可以自动对齐所选序列。
导出模式
SHSPdb中的所有序列都可以通过«导出出口»接口。«视图»菜单按用户最后操作的顺序显示条目(全部,过滤,选择或爆炸)。«格式菜单显示三种导出格式:FASTA, XML和Excel (CSV)。
分析工具
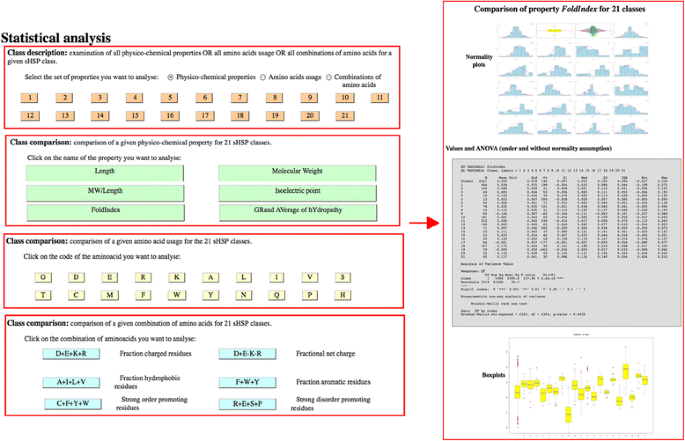
sHSPdb的一个原始特性是可以使用左侧面板获取任何自建数据集的大量信息。右边的面板允许选择所需的信息。对于分类数据,结果是计数表,分别按照字母顺序和计数值排序;对于定量数据,提供了经典的描述性统计和图形(如平均值、中位数、直方图等)。
统计分析工具
sHSPdb的另一个原始功能是可以使用不同的标准动态生成sHSP的物理化学性质或氨基酸使用情况图。因此,人们可以立即对自己的数据集进行多次比较。用户可以通过菜单«访问统计数据统计分析»。此选项显示«类描述(检查特定sHSP类别的所有物理化学性质或所有氨基酸的使用或所有氨基酸的组合)或类比较(sHSP类的物理化学性质比较)。绘图是自动生成的,可点击调整大小(图2)。2)。统计分析包括描述性(平均值,中位数…)和推断性(参数或非参数方差分析)计算和图形(具有密度和正态近似的直方图,箱形图,准确时的豆形图…)。
提交表格
用户可以通过提交表单提交序列。在验证它们的相关性后,它们将被整合到sHSPdb中。
数据库实现
sHSPdb是一个快速、交互式、独立于平台的基于web的数据库,具有用PHP编写的用户友好界面。有些选项需要激活Javascript。信息从MySQL关系数据库提供给用户。统计计算是使用PHP脚本调用的R软件执行的。
效用与讨论
sHSPdb与其他sHSP数据集
Poulain等。[9]报道了一个包含3787个sHSP序列的数据集。然而,该数据集包含一些冗余:该数据集中的774个序列是对齐的,70个序列是相同的,对应于9%的冗余。由此可以估计,整个数据集大致对应3400个不同的sHSP,即少于sHSPdb中包含的sHSP的数量。此外,该数据集仅限于具有加入号的平面PDF文件,因此不提供任何细节或功能。我们的选择非常严格,因为我们排除了大多数带有“推定”、“可能”、“未知”、“假设”、“未命名的蛋白质产物”、“合成结构”等注释的文件,以便在保留较少序列的情况下提高注释的质量。我们没有考虑片段,即注释为“partial”的文件。保留了带有上述注释但附加指示和/或基于基序可分类的序列。
据我们所知,只有一个专用于HSP的数据库(HSPIR):http://pdslab.biochem.iisc.ernet.in/hspir/shsp.php),收集热休克蛋白序列的加入编号和FASTA档案,并提供不同热休克蛋白家族的结构和功能的一般资料[25]。然而,HSPIR仅包含约1300个sHSP序列,并且仅提供基本的搜索和BLAST工具。因此,sHSPdb是一个独特的、全面的、高度实现的数据库,用于sHSP的计算和统计分析。
sHSP的分类
sHSP构成了一个非常大的蛋白质亚分类家族。然而,在注释、定义和术语方面存在着很大的差异。sHSPdb提供了一个统一的、同构的sHSP分类。sHSP分类最重要的参数之一是将每个类别与另一个类别区分开来的共识基序(表1)2)。建议的分类基于sHSPdb带来的专业知识、文献数据和以往的分类[1,2,9,17,19,26- - - - - -30.]。
相当简单的c端基序[ADHKNQ]G。[AILV]匹配约89%的序列和更复杂的中心基序[APS][EGM]。{4,5}[ADENQSK]匹配≈99%的序列。因此,这些基序几乎描述了所有的sHSP,因此与歧视无关。由于大多数sHSP类都含有ACD(除了sHSP类9、16、17和21),我们在n端或c端区域选择性地搜索基序,如果有的话(表1)3.)。
这个主题搜索强调了注释的困难。例如,基序[AFLSTVWY]P[AGPS][FHY][DN][ILV]匹配约540个序列,这些序列的注释主要是“热休克蛋白HSP20”、“HSP20家族蛋白”或“HSP20 / α晶体蛋白家族蛋白”。因此,我们的基序的高选择性有助于改善全局注释,如通用的«HSP20»。
先前的分类首先基于sHSP的细胞内定位:细胞质-核室(CI类、CII类和CIII类)、质体(P类)、内质网(ER类)、线粒体(M类)、过氧化物酶体(Po类或Px类)。这七个植物sHSP类是迄今为止最具特征的植物sHSP类(它们由14个基因编码)拟南芥)。的完整分析拟南芥基因组导致了另外四个细胞质核sHSP和第二个针对线粒体的独特sHSP家族。因此,高等植物可能包含至少12个保守的sHSP亚科。到目前为止,细胞器靶向的sHSP是特定于植物的,有一些例外(线粒体靶向的sHSP来自黑腹果蝇从刚地弓形虫) [31,32]。然而,确定蛋白质的亚细胞定位仍然很困难,特别是在大量蛋白质上,因为预测因素不够可靠。最后,各类间的生物分类是同质的,说明我们对sHSP的分类是准确的(表1)2)。
因此,这种分类以及自动生成的统计数据是有用的。例如,sHSPdb有助于分析表征sHSP的三个结构域在寡聚化和底物结合中的各自作用。一个保守的基序[ILV]。位于c端结构域的[ILV]对于通过与ACD中的疏水斑块接触形成低聚物至关重要[9,13,33]。在文献中,ACD结构域是sHSP的标志性特征[19,28],在sHSPdb的21个类中,有17个类确实存在。表格3.和无花果。3.为n端结构域、ACD和c端结构域大小的平均值的比较。
n端结构域、ACD和c端结构域大小平均值的比较。只表示包含ACD的HSP类(见表)3.)。上面的刻度表示氨基酸序列的大小。ER:内质网
然而,在我们看来,一些缺乏ACD序列的热休克蛋白应该被认为是sHSP,并定义为“与α结晶蛋白无关的sHSP”。因此,我们实现了4类不带ACD的sHSP。这类没有ACD结构域的sHSP的分子量完全符合sHSP的预期。它们的实现提供了额外的信息,并允许对sHSP进行更多的比较和分析。
使用sHSPdb的简短示例:线粒体sHSP的分析
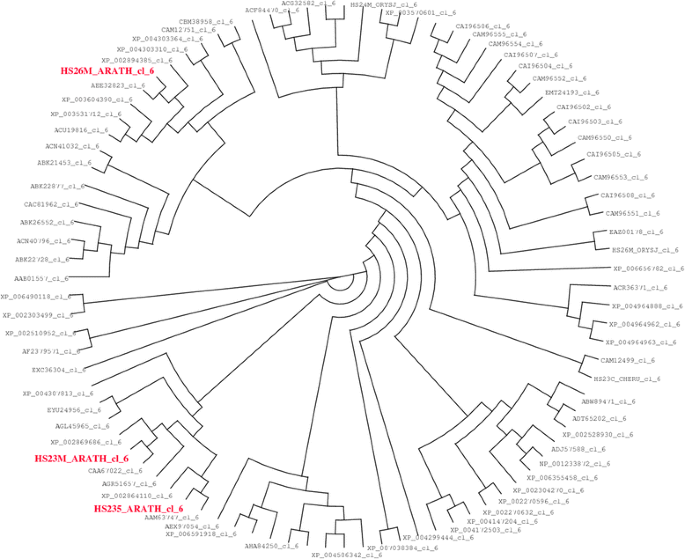
建立了植物线粒体sHSP(6类)的系统发育树。4)。5条序列对应拟南芥线粒体sHSP (AAM63747、CAA67022、HS235_ARATH、HS23M_ARATH和HS26M_ARATH,均来自6类),AAM63747与HS235_ARATH的差异为[S/A]31和[D/H]69, CAA67022与HS23M_ARATH的差异为[D/H]143。ACD和c端结构域的序列非常相似(图2)。5)。HS235_ARATH和HS23M_ARATH的n端结构域序列与HS26M_ARATH不同,具有更强的内在无序性(HS26M_ARATH为负foldinindex)。这可能对二聚体的形成有影响[34,35]。
植物线粒体sHSP系统发育树。对47种植物的91个线粒体sHSP序列进行了分析。该序列的特征是:长度在187 ~ 248个氨基酸之间;从4.620到10.15的等电点;一个pH值为7的净电荷−0.041 ~ +0.031;a folindex from−0.044 ~ 0.138;(肉汁)来自−0.908−0.274。使用Clustal-W进行对准,使用Dendroscope绘制图像[36]
结论
sHSPdb为sHSP提供了一个全面的数据集,以及为他们的在线分析设计的工具。据我们所知,目前还没有相应的sHSP数据库。sHSP可以根据不同的参数进行分类,特别是根据氨基酸基序进行分类。因此,sHSPdb构成了一个有效的工具:(i)用于编译和组织有关sHSP的不断增长的数据;(ii)小水蚤各亚科的分类;(iii)设计实验以阐明这些重要蛋白质的功能;(iv)帮助分析sHSP的结构-功能关系。
未来发展和展望:(i)统计分析了所有sHSP类sHSP的理化性质和氨基酸使用情况。因此,我们将能够比较三个结构域(即n端,ACD和c端),从而为已经由结构方法确定的结构域提供额外的信息。(ii)我们目前正在开发软件,分析用户提交的序列,以预测该序列是否属于任何sHSP类别。(iii)由于破译sHSP的分子功能是一个主要问题,我们将提供词汇工具(按字母顺序或出现或同义词排列的字典……),以便更好地对描述sHSP功能已知元素的单词进行语义分析。如前所述,在一些预测值和正在开发的约束编程软件的帮助下,正在研究未完全分类的保留蛋白质。
可用性和需求
sHSPdb是一个免费的数据库和可视化工具,对所有用户开放,无需登录要求,可以通过以下URL访问:http://forge.info.univ-angers.fr/~gh/Shspdb/index.php。该网络工具适用于所有现代网络浏览环境,包括Mozilla Firefox, Safari和Google Chrome。
缩写
ACD, α结晶蛋白结构域;内质网;NA,不适用;小热休克蛋白;sHSPdb,小型热休克蛋白数据库
参考文献
王晓东,王晓东,王晓东,王晓东,等。植物sHSP超家族:拟南芥中五个具有意想不到特性的新成员。细胞应激伴侣。2008;13:183-97。
[3]李建军,李建军,李建军,等。水稻sHsp基因的克隆与表达分析。生物医学工程学报。2009;10:393。
Kampinga HH, Hageman J, Vos MJ, Kubota H, tanaway RM, Bruford EA, Cheetham ME, Chen B, Hightower LE。人热休克蛋白的命名指南。细胞应激伴侣。2009;14:105-11。
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军。小麦热休克因子HvHsfB2c对干旱胁迫下小麦种子发育的影响。科学通报,2014;9:e89125。
Waters ER, Aevermann BD, Sanders-Reed Z.三个被子植物基因组中小热休克蛋白的比较分析发现了新的亚家族并揭示了不同的进化模式。细胞应激伴侣。2008;13:27 - 42。
Kriehuber T, Rattei T, Weinmaier T, Bepperling A, Haslbeck M, Buchner J.小热休克蛋白核心结构域及其侧翼序列的独立进化。中国生物医学工程学报,2010;24(4):344 - 344。
李建军,李建军,李建军,等。晶体蛋白结构域折叠的研究进展。中华生物医学杂志。2005;33(3):391 - 391。
Finn RD, Mistry J, Schuster-Böckler B, Griffiths-Jones S, Hollich V, Lassmann T, Moxon S, Marshall M, Khanna A, Durbin R, Eddy SR, Sonnhammer EL, Bateman A. Pfam:部落,web工具和服务。中国生物医学工程学报,2006;33(4):557 - 557。
张建军,张建军,张建军,等。热休克小分子蛋白的检测与结构分析。科学通报,2010;5:990。
Feil IK, Malfois M, Hendle J, van Der Zandt H, Svergun DI。具有伴侣蛋白样活性的二聚体α -结晶蛋白结构域的新型四元结构。生物化学学报,2001;26(6):1145 - 1145。
范孟福,陈建军,陈建军,陈建军,等。一种热休克蛋白的克隆与合成。生物工程学报。2001;8(10):1025 - 1030。
Pasta SY, Raman B, Ramakrishna T, Rao CM。小热休克蛋白α-晶体蛋白SRLFDQFFG区域的保守作用。对寡聚物大小、亚基交换和伴侣活性的影响。生物化学学报,2003;28(2):591 - 591。
王志刚,王志刚,王志刚,王志刚,王志刚,王志刚,王志刚,王志刚,王志刚。一种来自嗜酸嗜热古菌的小热休克蛋白。菌株7;蛋白质。2008;71:771 - 82。
bagnsamris C, Bateman OA, Naylor CE, Cronin N, Boelens WC, Keep NH, Slingsby C. α-晶体蛋白域二聚体与Hsp20的晶体结构。中华生物医学杂志。2009;32(2):442 - 442。
希利EF。热冲击响应中异齐聚物形成的模型大肠杆菌。中国生物医学工程学报,2012;22(4):639 - 639。
贾亚东,李建军,李建军,等。热休克蛋白分子伴侣蛋白的分子结合位点灵活性研究。科学通报,2009;26(6):591 - 591。
马建军,李建军,李建军,等。植物过氧化物酶体热激蛋白的克隆与鉴定。植物生理学报。2006;41(1):47 - 60。
de Jong WW, Leunissen JA, Voorter CE。α -结晶蛋白/小热休克蛋白家族的进化。生物医学进展。1993;10:103-26。
张建军,张建军,张建军。热休克蛋白与α-晶体蛋白的研究进展。生物化学进展,2012;37(1):106 - 17。
Marchler- bauer A, Anderson JB, Derbyshire MK, dewese - scott C, Gonzales NR, Gwadz M, Hao L, He S, Hurwitz DI, Jackson JD, Ke Z, Krylov D, Lanczycki CJ, Liebert CA, Liu C, Lu F, Lu S, Marchler GH, Mullokandov M, Song JS, Thanki N, Yamashita RA, Yin JJ, Zhang D, Bryant SH. CDD:基于交互域族分析的域域数据库。中国生物医学工程学报,2009;33(2):444 - 444。
猎人,Apweiler R, Attwood TK, Bairoch,贝特曼,宾斯D,博克P, Das U,多尔蒂L, Duquenne L,芬恩RD,高夫J,把手D, Hulo N,卡恩D,凯利E, Laugraud, Letunic我,朗斯代尔D,洛佩兹R,ㄧM,套装J, McAnulla C, McDowall J, Mistry J,米切尔,穆德N,纳塔尔D, Orengo C,奎因房颤,Selengut JD, Sigrist CJ, Thimma M,托马斯•PD Valentin F,威尔逊D,吴CH,叶芝C InterPro:整合蛋白签名数据库。核酸学报,2009;37:D224-228。
陈晓华。分子伴侣。2008.http://faculty.washington.edu/baneyx/Chaperones/Chaperones.html#Hsp33。
李建军,李建军,李建军,等。基于约束优化的蛋白质家族预分类软模式发现。第九届基于约束的生物信息学(WCB)方法研讨会,乌普萨拉,2013。
李建军,李建军,李建军,等。基于多模态分解的基因序列识别方法。第21届欧洲人工智能会议(ECAI), IOS出版社,布拉格:2014。
Ratheesh KR, Nagarajan NS, Arunraj SP, Sinha D, Veedin Rajan VB, Esthaki VK, D 'Silva P. HSPIR:人工注释热休克蛋白信息资源。生物信息学。2012;28:2853-5。
Scharf KD, Siddique M, Vierling E.拟南芥小热应激蛋白家族的扩展和α-晶体蛋白结构域ACD蛋白的新家族。细胞应激伴侣。2001;6:225-37。
陶鹏,郭伟,李碧,王文辉,岳正成,雷建林,钟晓明。白菜sHSP基因的全基因组鉴定、分类及表达分析中华医学杂志,2015;14(1):75-93。
刘建军,张建军,张建军,等。植物热休克蛋白/α-晶体蛋白家族的进化与功能分化。足底。2012;235:1299 - 313。
水域。植物sHSPs的进化、功能、结构和表达。[J] .中国生物医学工程学报,2013;33(4):391 - 391。
刘张J, B,李江,张L,王Y,郑H,鲁米,陈J . Hsf和Hsp在杨树基因家族:全基因组鉴定、组织和在开发过程中相关的表达式和应激反应。BMC Genomics. 2015;16:181。
莫罗G,伊纳古马Y,加藤K,唐伟RM。黑腹果蝇的小热休克蛋白Hsp22是一种具有低聚结构的线粒体蛋白。生物化学学报,2000;27(5):398 - 398。
de Miguel N, Echeverria PC, Angel SO。刚地弓形虫小热休克蛋白家族成员的差异亚细胞定位。真核细胞。2005;4:1990-7。
孙旸,麦克雷·特。小热休克蛋白:分子结构与伴侣蛋白功能。细胞与生命科学,2005;62:2460-76。
Chen J, Feige MJ, Franzmann TM, Bepperling A, Buchner J.小热休克蛋白Hsp26的二聚化需要α -结晶蛋白结构域外的区域。中国生物医学工程学报,2010;38(1):1 - 3。
Sudnitsyna MV, Mymrikov EV, Seit-Nebi AS, Gusev NB。内在无序区域在小热休克蛋白结构和功能中的作用。中国生物医学工程学报,2012;13(3):776 - 785。
Huson DH, Richter DC, Rausch C, Dezulian T, Franz M, Rupp R.树窥镜:大型系统发育树的交互式观察器。生物医学工程学报,2007;22:460。
Corpet F.基于层次聚类的多序列比对。核酸学报。1988;16:10881-90。
李建军,李建军,李建军,等。基于分子结构的蛋白质三维信息提取方法研究。核酸学报。2003;31:3320-3。
ᅟ
ᅟ
资金
这项研究得到了ANR Blanc MITOZEN (ANR-12- bsv8 -0021-01)和昂热大学(ACS13LES)的资助。
数据和材料的可用性
支持本文结论的数据集可以在sHSPdb存储库中免费访问http://forge.info.univ-angers.fr/~gh/Shspdb/index.php。
作者的贡献
EJ担任项目顾问。GH实现了MySQL数据库,PHP和Perl脚本,以及界面设计。EJ收集数据,检查数据完整性,并将其输入数据库。GH和EJ撰写稿件,检查数据库和web界面的准确性,阅读并批复最终稿件。
相互竞争的利益
作者宣称他们没有竞争利益。
发表同意书
不适用。
伦理批准并同意参与
不适用。
作者信息
从属关系
相应的作者
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
Jaspard, E., Hunault, G. sHSPdb:用于分析小热休克蛋白的数据库。BMC Plant Biol16日,135(2016)。https://doi.org/10.1186/s12870-016-0820-6
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-016-0820-6
关键字
- 小热休克蛋白
- 氨基酸序列
- 物理化学性质