摘要
背景
SBP-BOX.基因属于最大的转录因子家族之一。虽然这个家族的成员已经被描述为不同生物过程的重要调节者,信息SBP-BOX.第三种最重要的油料作物的基因芸苔栗鸟在很大程度上是未定义的。
结果
在本研究中,通过全基因组生物信息学分析和转录谱分析,58个可能的成员SBP-BOX.油菜的基因家族(芸苔栗鸟鉴定了L.)并分析了不同组织中的表达模式以及与miRNA的可能相互作用。此外,B. Napus.采用具有不同分支角的线来研究分支角的影响SBP-BOX.植物建筑规范中的基因。系统地表征了详细的基因信息,包括基因组组织,结构特征,保守结构域和系统发育关系。通过系统发育分析,将BNASBP蛋白分为八个不同的群体,其代表与拟南芥和稻米的家庭成员的透明正交关系。12个组织中的表达分析,包括植物和生殖器官在内的表达分析表明了不同的表达模式SBP-BOX.基因和一些基因表现出组织特异性表达,表明它们在发育过程中的不同功能。44SBP-BOX.确定基因以含有调用的miR156结合位点,分别在编码和3'URR区域的MiR156靶向的30和14。miR156的相对表达水平在组织中变化。一些不同的表达模式BnaSBPmiR156与靶基因的转录水平呈负相关BnaSBP以不同分支角度的线观察到基因。
结论
携带,本研究代表了第一次系统分析SBP-BOX.基因家族在芸苔栗鸟.这里提供的数据为理解的关键角色提供了基础BnaSBP植物发育和其他生物过程中的基因。
背景
转录因子通过激活或抑制不同靶基因的表达,在植物的生命周期中发挥着关键作用[1].Squamosa启动子结合蛋白(SBP)盒系列是由高度保守的SBP结构域,其长度的76个氨基酸的转录因子家族之一[2- - - - - -4].自从第一次SBP-BOX.基因被鉴定在Antirrhinum Majus.,许多这样的基因已经用不同的植物物种表征,因此鉴定了中等大小的基因家族。十六SBP-BOX.基因已在模式植物拟南芥中被鉴定,许多基因也已在全球重要的农业作物中被鉴定,如水稻(奥雅萨苜蓿)和玉米(玉米) [5- - - - - -7].的SBP-BOX.已被证明基因影响了许多发展的发展,包括叶和培养的发育,植物和生殖相转变,植物激素信号转导等生理过程[8- - - - - -15].
在确定SBP-BOX.许多基因被证明在不同的发育过程中起着重要的作用。构建表达拟南芥基因的转基因植物SPL3表现出非常早的开花和频繁的形态变化[16].拟南芥spl8突变体花粉囊发育改变,花粉囊过表达SPL8通过介导GA依赖的信号通路影响植物的育性[9,17].此外,SPL8和其他SPL.基因通过干扰生长素稳态来控制雌蕊模式[18].AtSBP7是拟南芥铜稳态的中央调控因子[19].AtSPL2,AtSPL10和AtSPL11在拟南芥中已被证实控制与生殖阶段芽成熟相关的形态变化[20.].braspl9-2是MicroRNA BRA-MIR156的目标,并控制大白菜的前线时间[21].除了在双子叶植物中有重要作用外,SBP-BOX.单子叶植物的基因,如水稻和玉米,也被证明调节必要的发育过程。高的表达OsSPL14在生殖阶段促进水稻中穗分枝和更高的谷物产量,表明重要作用SPL.植物建筑监管中的基因[22,23].玉米转录因素unbranched2和unbranched3编码SBP-box蛋白还可以通过调节侧原基起始率来改变植物结构和影响产量性状[24].
MiRNA是小的非编码20-24NTRNA,可以通过转化抑制或转录裂解和降解来互补地与其靶mRNA结合并降低蛋白质水平[25,26].许多开发过程由SBP-BOX.基因与mir156密切相关。计算分析表明了很多SBP-BOX.基因由拟南芥MiR156家族调节[27].一些重要的发育过程似乎都是由miR156及其靶标介导的SBP-BOX.由于MiR156过表达的基因导致各种表型,包括叶片数量增加,延迟开花并降低了顶端主导地位[28].拟南芥mir156互补地绑定到3'UTRSPL3并通过翻译抑制和转录下调调控其表达[16,29].米米米米的过度表达也导致表达下降SPL.靶基因,表明相关的相互作用SPL.单子植物植物中的mir156 [6].拟南芥miR156调节对反复出现的热胁迫的耐受性SPL.基因在热应激后受到miR156的转录后调控[30.].最近有报道称miR156/SPLs调控拟南芥侧根发育[31].除了miR156的调控作用,SBP-BOX.基因也显示在草中的miR529调节[32].有趣的是,miR156和miR529在核苷酸水平上相互关联,共享一个14-16 nt的结合位点[33].然而,没有miR529候选基因调控SBP-BOX.在核心eudicots中发现基因,例如拟南芥和杨树[34,35].
尽管有重要的作用SBP-BOX.拟南芥或米饭的基因,信息SBP-BOX.油菜籽中的基因(B. Napus.)在很大程度上没有定义。全基因组分析SBP-BOX.基因已在几种物种中进行[36- - - - - -40].然而,尚未进行该基因家族的分析芸苔属植物物种。同时,二者之间的相互作用BnaSBP基因和BnaMiR156没有被清楚地理解。根据最近的调查结果SBP-BOX.拟南芥、水稻等生物基因功能分析SBP-BOX.基因B. Napus.肯定会加速利用这些基因。在这里,我们报告系统地分析SBP-BOX.基因B. Napus.研究其基因结构、系统发育、基序组成、miRNA靶位点、染色体定位和在各组织器官中的表达模式。的相对转录水平Bnamir156.在不同的组织中也进行了功能关系的研究SBP.和mir156.基因。
方法
识别和注释SBP-BOX.基因的B. Napus.基因组
首先,PFAM数据库中的SBP域(PF03110)的HMM配置文件(http://pfam.xfam.org/)下载并用于搜索的基因组数据库B. Napus.(http://www.genoscope.cns.fr/brassicanapus/)使用HHMER搜索程序。所有非冗余序列都提交给Interpro(http://www.ebi.ac.uk/interpro)确认SBP域的存在。没有完整SBP域的序列被排除在结果之外。我们还执行了HHMER搜索芸苔属植物拉伯和Brassica oleracea基因组数据库以识别收缩压蛋白。其次,拟南芥SBP蛋白序列从TAIR (http://www。拟南芥.org/)作为查询来执行BLASTPB. Napus.基因组。SBP-BOX.基因登录数B. Napus.提取基因组数据库。假定的命名法SBP-BOX.基因B. Napus.符合拟南芥中的同源基因ID。对于一个人SBP-BOX.拟南芥的同源基因SBP-BOX.油菜油的基因按字母顺序制定。作为序列AtSBP1和AtSBP12显示高度相似,只是BNASBP1.基因被在油菜中命名。SBP-BOX.水稻中的基因从水稻基因组项目下载(http://rice.plantbiology.msu.edu/).
基因结构,染色体位置,重复和系统发育分析BnaSBP基因
所有的BnaSBP基因被绘制到B. Napus.根据近似位置信息基因组染色体。每个外显子/内含子结构BnaSBP基因结构显示基因结构显示服务器程序(http://gsds.cbi.pku.edu.cn/index.php.),通过比较编码序列和基因组序列。MCScanX软件(http://chibba.pgml.uga.edu/mcscan2/)的复制模式分析BnaSBP油菜基因组中的基因。采用local blast +软件进行BLASTP分析B. Napus.在1E-5下的电子值。职位SBP-BOX.基因和爆炸输出导入MCSCanx软件,以在默认标准下生成圆形绘图。SBP-Box蛋白序列的多序列对准奥雅萨苜蓿,拟南芥蒂利亚纳和Brasscia显著使用Clustalx2.0执行默认参数[41].使用邻近加入(NJ)方法和具有1000个引导复制的最大似然(ML)方法,在Mega6.0软件中构建了系统发育树。
保守motif鉴定和miR156靶点预测
使用MEME在线工具(http://meme-suite.org/),参数设置如下:最大图案数量,20;重复次数,任意;motif宽度范围为6 ~ 80。在InterPro数据库(http://www.ebi.ac.uk/Interpro/)使用WebLogo Online软件创建序列徽标(http://weblogo.threeplusone.com/).预测miR156的推测靶点,全长BnaSBP基因包括外显子、内含子和UTR序列,使用psRNATarget工具(http://plantgrn.noble.org/psrnatarget/?function.).通过Genedoc软件修改保守的靶序列。
植物材料及生长条件
用于表达模式分析和RNA-SEQ的植物样品B. Napus.中国农业科学院油料作物研究所(OCRI-CAAS) var. zhongshu11。RNA-seq数据来源于12个不同的组织(根、叶、芽、角果、雄蕊、新生花瓣、开花花瓣、凋萎花瓣、茎、萼片、胚珠和果皮)。高分辨率RNA-seq数据BnaSBP曾刘刘教授从ocri-caas(未公布的数据)恳求基因。详细的FPKM值(每百万百万映射读取的每千级型号的碎片)是补充数据列表(附加文件3.:表S2)。FPKM值为LOG2转化,并计算所有基因的欧几里德距离。通过“完整链接聚类”的分层群集方法构造和显示聚类树R包中。
分析miR156和BnaSBP基因,在与RNA-SEQ的样品相同的发育阶段,也从相同的组织部位收集12个组织样品。将所有样品快速收集并在液氮中冷冻并储存在-80℃。B. Napus.分别用于表达分析的线麦体和6098B分别覆盖大型和小分支角。不同年份的结果表明,6098b的分支角度比成熟阶段的麦体大30-32°[42].在RNA-SEQ分析的螺栓和早开花阶段收集分支位点处的组织样品。如中顺11所述分析RNA-SEQ数据。从6098B和幻灯中的其他组织样品作为来自中顺11的其他组织样品进行RT-PCR以验证RNA-SEQ结果。所有植物材料都在中国武汉Ocri-Caas的田野上生长。
RNA提取和实时定量RT-PCR分析
不同生长阶段的不同组织中的总RNA用Trizol试剂(Invitrogen,America)提取。在逆转录之前,用RNase-Prese I(Promega,America)处理总RNA 15分钟以降低基因组DNA。茎环RT-PCR用于在先前报告的过程后检查不同组织中的miR156表达水平[43].miRNA序列在B. Napus.已从miRBase序列数据库下载[44].根据Zhao等人设计用于茎环RT的引物。(2012)[45].同时加入U6特异性引物作为参考,在每次反应中精确归一化。由于miR156家族的成熟序列在5 '区存在差异,我们设计了5个不同的正向引物进行实时qPCR。qRT-PCR在CFX96 Real Time System (Bio-Rad, Hercules, California, USA)中按照说明书使用SYBR Green (Tiangen, China)进行。简单地说,每个反应分别加入12.5 μl SYBR混合物、1 μl通用反向引物和1 μl特异性引物。以U6反应为对照,采用特异性引物进行反应。每个样品按以下程序进行3次重复反应:95℃10 min, 95℃5 s, 60℃30 s, 40个循环。每对引物扩增的特异性通过熔融曲线分析得到验证。对于RT-PCR,使用转录本第一链cDNA合成试剂盒(天根,中国),根据制造商说明使用2 μg RNA进行第一链cDNA合成。反应程序如下:95°C 5 min, 95°C 30 s, 54°C 40 s, 72°C 1 min,循环31-37次。 Primers used in the qPCR and RT-PCR were listed in Additional file1S1:表。的U6和肌动蛋白如前所述选择基因作为内部参考基因[45].
结果
SBP基因的鉴定B. Napus.
所有拟南芥SBP蛋白序列都用作Tblastn的疑问。结果,五十八个推定SBP-BOX.最初鉴定基因。通过Interpro工具检查所有子序列以搜索SBP域。不包括三种没有SBP结构域或不完全SBP域的蛋白质。HHMER搜索也违反了B. Napus.蛋白数据库,以sbp结构域PF03110为查询对象。获得了10个额外的蛋白质序列;然而,其中只有3个包含Interpro扫描检查的完整SBP域。最终鉴定出58个SBP蛋白。6个SBP蛋白不能被分配到任何位置B. Napus.染色体准确。所有SBP-BOX.基因B. Napus.是指定为BnaSBP并根据拟南芥最近的正交学命令命名。加入数,染色体分布,蛋白质分子量和长度BnaSBP基因列于表1.hhmer搜索芸苔属植物拉伯和Brassica oleracea基因组中分别有26个和19个SBP蛋白。此前的研究结果表明,拟南芥中存在16个SBP蛋白。通过比较三个密切相关物种的基因数量,SBP-BOX.基因家族成员B. Napus.在基因数量上表现出明显的扩展。
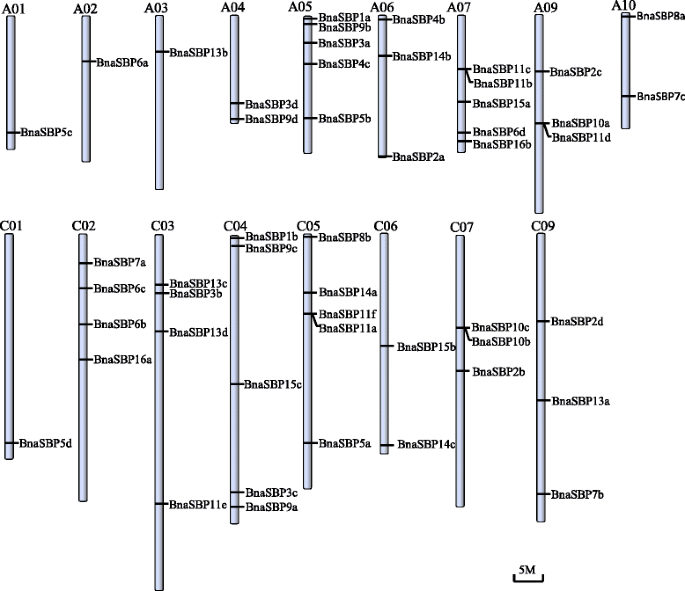
染色体定位和基因复制分析
确定染色体分布和基因重复SBP.基因B. Napus.,所有的SBP.除了一个位于未定向支架上的四个外,映射到近似染色体位置(图。1).这五十四岁SBP.基因分布不均芸苔属植物染色体。除A8和C8除外,所有染色体都含有至少一个SBP.基因。在A1, A3和C1染色体上,只有一个SBP.发现基因。四条染色体包含最大数量SBP.基因,即A5,A7,C4和C5各自有五个SBP.基因。四个集群各有两个SBP.基因的鉴定以相邻距离为标准SBP.基因小于50 kb。26、32SBP.发现基因分别位于A基因组和C基因组。发现的数量很有意思SBP.位于A基因组的基因B. Napus.等于多少SBP.基因b·拉伯基因组。然而,只有19SBP.确定基因b . oleracea基因组,它是C基因组的祖先B. Napus.,表明SBP.基因扩增可能发生在多倍体C基因组中。
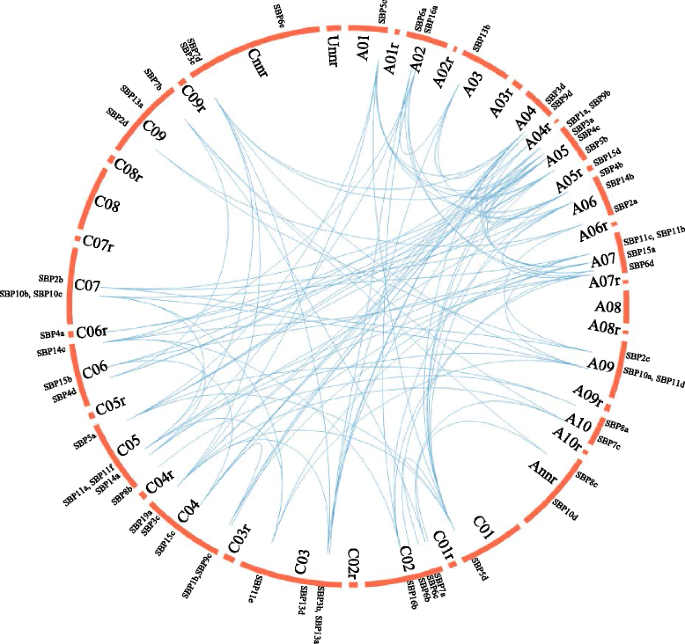
的串联和分段复制芸苔属植物SBP基因也进行了分析。在所有的SBP.8个成员(13.8%)显示串联重复,其中包括4个串联重复基因簇(图4)。1).58名中49名(84.5%)BnaSBP发现基因是分段重复的基因。这些基因位于十七个不同的染色体(图。2).
结构组织和保守域识别
了解SBP蛋白的进化关系B. Napus.在mega6.0中,我们基于SBP全长蛋白序列的序列比对,采用neighbor-joining (NJ)方法构建了无根树。其中的58个SBP蛋白B. Napus.分为8个不同的组(从Ito VII开始),组I由最多的14个BnaSBPs组成,而组v仅包含3个BnaSBPs。根据其他物种(如番茄)的研究结果,整个串联复制的BnaSBPs被分配到一组,杨树trichocarpa[40,46].基因组序列BnaSBP基因范围从510 bp到约5 kb。为了获得进一步的基因结构信息,我们将编码序列与所有基因的基因组序列进行了比较BnaSBP基因(图。3).不同的内含子(0 ~ 10个)BnaSBP基因。除了BnaSBP6d,所有BnaSBP基因含有至少一个内含子。该基因具有最大数量的内含子是IV组和VII组。的BnaSBP分为同一组的基因簇表现出类似的结构。在SBP蛋白中鉴定了几个基序B. Napus.(无花果。3 b).在所有BNASBP蛋白中检测到含有SBP结构域的一个基序,除BNASBP8B,含有类似的SBP结构域,其由于缺少少数氨基酸而无法检测到。同一组中的BNASBP蛋白表现出类似的基序组合物。
所有BnaSBP蛋白用ClustalX 2.0进行比对,保守的SBP结构域用Weblogo在线工具构建。58个BnaSBP蛋白包含完整的SBP结构域,具有两个锌基序和一个核定位信号(图)。4).除BnaSBP5组外,SBP蛋白中第一锌指motif均为C3H型。所有的SBP蛋白都含有第二CCHC型锌基序。由于SBP蛋白具有转录因子的特性,所有的SBP蛋白都含有保守的核定位信号。
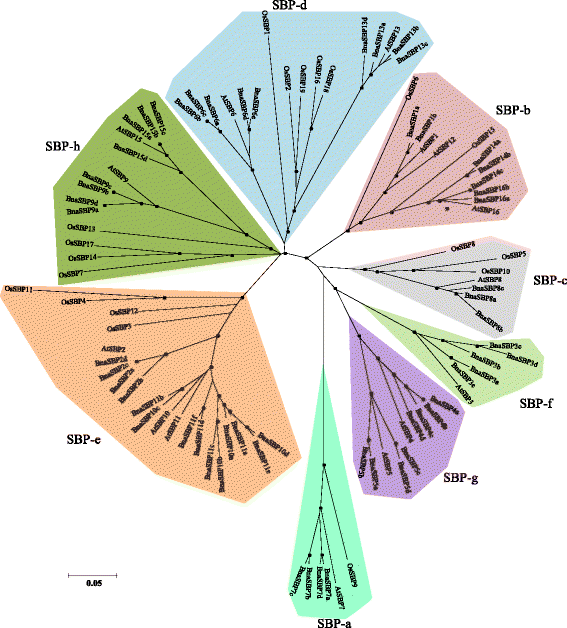
系统发育分析SBP.基因B. Napus.,拟南芥和水稻
之间的系统发育关系BnaSBP基因和其他SBP.具有其他物种已知功能的基因有助于预测其在油菜发育中的作用。十六SBP.基因来自拟南芥和19个SBP.从公共基因库中提取来自水稻的基因,这些基因分别为DiCot和单焦度物种。五十八SBP.来自油菜油的基因与拟南芥和水稻基因一起用于构建一个无发电的系统发育树(图。5额外的文件2:图S2)。根据系统发育分析,SBP.来自这三种植物物种的基因可以分为七组(SBP-A至SBP-H)。最大的组(SBP-E)包含21名成员,占总SBPS的23%,而组SBP-A组是只有五名成员的最小组。如图1所示。5在美国,SBP-a组的基因比其他组的基因更分散。BnaSBP基因与拟南芥同源基因具有高度的相似性,并被归为同一类群。在系统发育分析发现的类群中,类群SBP-f仅包含拟南芥和油菜的sbp,表明拟南芥的多样性SBP.单子叶和双子叶植物之间的基因。
mir156家庭B. Napus.他们的目标网站BnaSBP基因
通过对miRBase数据库的查询,发现油菜miR156 (BnaMiR156a-g)中有7个可能的成员。最近,研究人员预测了32个推测的miR156的早熟结构B. Napus.通过高吞吐量小RNA深度测序[47].前期研究结果表明miR156可以互补结合SBP.在编码或3'UTR区域的基因,并通过翻译抑制或裂解降低基因活性[27,29].结果显示,44个SBP蛋白具有miR156结合位点,其中30个位于编码区,14个位于3'UTR区(图2)。6).根据之前的调查结果,17人中有11人SBP.miR156可以靶向拟南芥中的基因。油菜中的同源基因也被预测为miR156的靶点。这些结果表明miR156与SBP.基因在物种间是保守的。然而,三个BnaSBPmiR156靶向的基因与其他基因不同。BnaSBP5c具有编码区域内的结合位点,而其他三个BNASBP5miR156靶向3'UTR的基因。MiR156预测结合到3'UTR序列BnaSBP6d和BnaSBP10a,虽然拟南芥中的相对同源基因在编码区的MiR156中结合。同源基因之间的不同调控模式B. Napus.拟南芥揭示了油菜中SBP箱基因的分歧。
表达档案BnaSBP
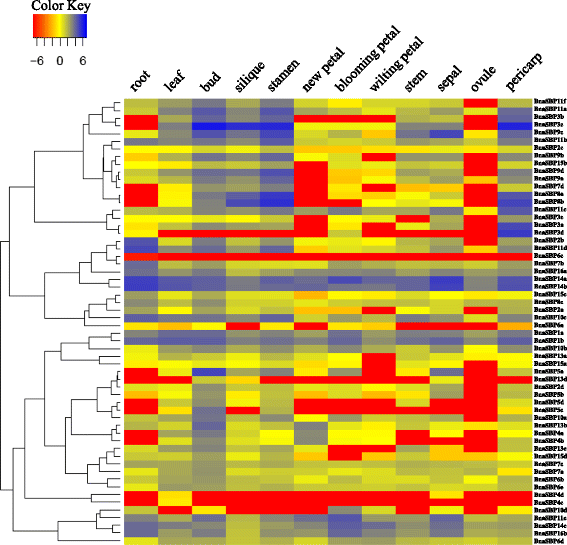
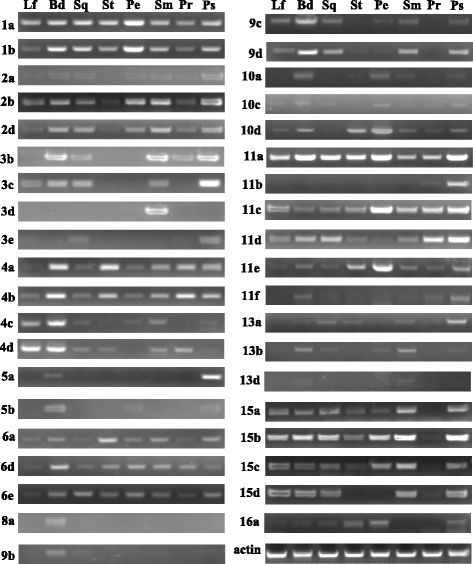
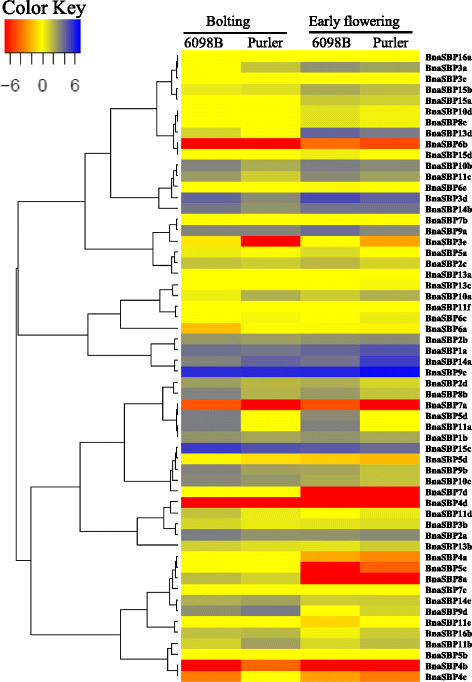
广泛的SBP.基因在植物发育过程中起着重要作用。缺少SBP.基因突变体,表达模式可以提供线索以阐明不同的潜在作用SBP.基因B. Napus..的表达水平BnaSBP通过热图表示表示12个组织中的基因(图。7额外的文件3.:表S2)。文字记录的BnaSBP6c在所有12个组织样本中为零,而BnaSBP4c在叶中被检测到。基于分层聚类分析,BnaSBP基因可以分为八个类别。大量的抄写BnaSBP基因富含芽,雄蕊和果皮。相比之下,大多数BnaSBP基因在胚珠和花瓣中表达量低。八个BnaSBP的基因,BnaSBP1a,1 b,11 e,14个,14 b,14摄氏度,16一个和16 b似乎是由根到果皮的组成型表达。应该指出的是,所有这些基因,不包括bnasbp11e.,不预测由MIR156定位。BnaSBP4c,4 d,5度,5 d,10 d和13 d大多数组织持续低表达。的表达水平BnaSBP3a和3 d在大多数组织样品中未检测到,但在Pericarp中达到了更高的水平。相对更高的表达水平BnaSBP2b和11 d也能在根组织中识别出来。相比SBP.不受miRNA的基因,即BnaSBP具有靶位点的基因表现出更多样化的表达模式。我们还进行了RT-PCR,以确定部分表达水平BnaSBP8种不同的组织(图。8).三十九BnaSBP以验证RNA-seq数据的结果。结果表明,RT-PCR数据与RNA-seq数据基本一致BnaSBP在大多数组织中。例如,表达水平BnaSBP1a,1 b和11 e可在大多数组织中检测到(图。8).虽然BnaSBPS至少在其中一种组织中表达,在基因组上观察到表达模式的区别。一些人BnaSBPS属于同一组,表现出相似的表达模式,如BnaSBP1a和1 b在第四组,bnasbp15a.和15 b在第三组中,表明冗余角色BnaSBP我们在同一组。因此,油菜SBP转录因子的表达模式多样,其生物学功能可能是冗余的,每个个体负责某些生理过程。
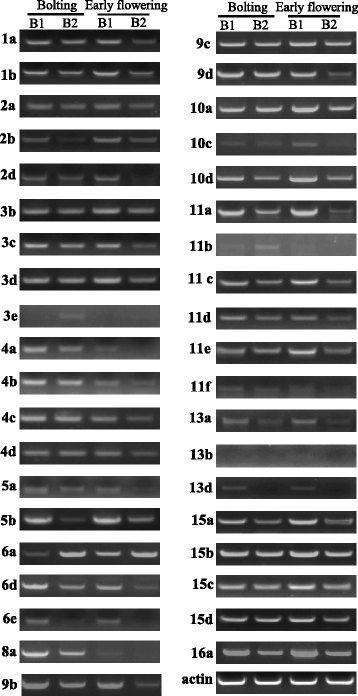
研究涉及分支角度调节的推定基因,两个表达曲线B. Napus.进行具有不同分支角的材料(6098b和麦板)(附加文件4:图S1)。在抽薹期和开花期两种材料的分枝点取样,进行DEGs (Different Expression Genes)测定。所有的转录水平SBP.从表达谱系中提取基因(附加文件5:表S3)。图中显示了两个发育阶段的表达水平。8.许多BnaSBP在两个发育阶段,两种品系的基因表现出不同的表达模式。BnaSBP5c,8A和7D.抽薹期表达量高,开花期表达量少或无表达。十、十三BnaSBP发现基因分别在两条发育阶段的两条线之间差异表达。其中,六BnaSBP基因在两个发育阶段有差异表达(图。9).进一步的研究可以关注这些基因对分支角度调节的作用。进行RT-PCR以确认表达水平BnaSBP在同样的组织中进行rna测序。大量的BnaSBP粉丝在比6098b中的更高水平表达的s(图。10).该RT-PCR结果通常与RNA-SEQ数据一致。
miR156的表达谱
几个BnaSBP基因将互补序列携带至miR156。因此,预期MiR156预计将成为表达这些的重要决定因素BnaSBP基因。miR156的表达水平在不同发育阶段的中子11的芽和石灰中大多数丰富(图。11个).在叶片样品中发现了相对低水平。同时,还确定了6098B和麦克风中miR156的表达水平。结果表明,与螺栓连接时间相比,早期开花时间的MiR156的丰度显着下降(图。11 b).除了两种材料的茎样品之外,MiR156的转录在蜡烛中比其他组织的6098b更强。
miR156在不同组织样本中的表达模式。采用qRT-PCR分析成熟miR156在不同组织中的相对表达水平。一个miR156在中双11不同组织样本中的表达水平。将该值归一化到苗期的根部。R,根;圣,阀杆;L,叶;B,芽;Sl1,开花后15天呈角形;Sl2,开花后18天呈角果状;Sl3,花期20天后角果;Sl4,开花后23天呈角果状。bMIR156分别在脉冲和6098b的五个组织样本中的miR156的表达水平。该值在螺栓阶段标准化为6098b。L,叶;B,芽;圣,阀杆;BS1,螺栓阶段的分支站点;BS2,早期开花阶段的分支网站。星号表示在相同组织样品中麦克风和6098B之间检测到显着差异学习任务在*P < 0.01
讨论
SBP-BOX.基因玉萝卜岛和他们的演变
SBP盒蛋白质的特征在于保守的SBP结构域,具有76个氨基酸,并构成植物中的一大族转录因子。植物特异性SBP箱转录因子仅在绿色植物中检测到,这表明它可能来自预测绿藻和土地植物的祖先的分歧[5,48].不同数量的SBP-BOX.基因已在各种土地植物中表征[39,40,49].在目前的研究中,58SBP-BOX.基因B. Napus.确定基因组,这是拟南芥数量的约四倍SBP-BOX.基因。B. Napus.包含13SBP-BOX.基因的总和b·拉伯(AA,2n = 20)和b . oleracea(CC, 2n = 18),是两种直接祖先种B. Napus.(AACC, 2n = 38) [50].对于一种基因家族,串联和节段性重复事件是基因扩张的主要原因。SBP-BOX.基因在19条染色体中的17条上分布不均匀B. Napus.,四个集群,每个集群有两个BnaSBPs被鉴定(图。1).不均匀和集群分配SBP-BOX.在水稻和桃子中也发现基因家族基因[6].7个和49个BnaSBP发现是串联和节段性重复的基因。多样化BnaSBP从系统发育、基因组结构、miR156靶位点定位等多个方面对miR156基因进行观察。这种多样性的SBP-BOX.基因复制后内含子和外显子的缺失可能引发基因结构的变化。
的功能差异SBP-BOX.基因
随着SBP-BOX.基因具有转录因子的特征,预计它们的表达模式与其在植物发育的功能相关。表达剖面BnaSBP-box基因在不同组织中显示出明显的表达模式。在拟南芥中,一些SPL.基因组成型表达,而其他的转录水平正在发生发展控制[5].表达式的分析SBP-BOX.其他生物中的基因也呈现了不同的时尚表达模式[39,40,49,51].收缩压转录因子B. Napus.在不同的组织中表现出不同的表达模式,表明它们可能在不同的生物过程中发挥作用。大量的抄写BnaSBP花蕾、雄蕊和果皮中基因富集,表明花蕾、雄蕊和果皮中大部分基因富集SBP-BOX.油菜的基因可能与生殖器官的发育有关。
SBP-BOX.许多物种的基因,特别是水稻和拟南芥,已经被证明在不同的发育过程中发挥重要作用。微控制SBP-BOX.基因SPL9和SPL15,是拟南芥中最接近的同源基因,已被证明可以控制茎成熟[52].进一步支持可能的角色BnaSBP在开发中来自水稻基因SPL14水稻穗发育与理想株型调控[22,23].我们确定了四个BnaSBP油菜基因组中的9个基因。虽然这是BNASBP9.基因具有相似的基因结构,观察到不同的表达模式。应该指出的是表达的bnasbp9d.在致密材料中Purler高于在疏松材料中6098B(图。9和10).的表达bnasbp9d.从螺栓到早开明显减少。应进行进一步的研究以验证是否bnasbp9d.可能对油菜的枝角有调节作用。
拟南芥基因SPL8影响花粉囊发育,并控制古尼亚百年图案[18].三BnaSBP的基因,BnaSBP8a,8B.和8C.显示最相似于AtSBP8,通过系统发育分析加入同一类群。BnaSBP8a和BnaSBP8b在雄蕊中高度表达。进一步的研究可以关注潜在的作用BNASBP8.在花的发育。
组成型表达的AtSPL3导致提早开花[53].的SPL3同源基因Antirrhinum Majus.和黄桦还通过与疯箱基因结合来调节花发育[16,54].番茄LESPL-CNR.,哪个与之相似AtSPL3基因,对正常水果发育和成熟是至关重要的[55].在拟南芥中,miR156-SPL3模块控制FT表达以调节环境温度响应开花[56].在同源的五种基因中AtSPL3确定B. Napus.在我们的研究中,BnaSBP3c在芽,雄蕊,硅藻土和果皮中显示出更高的表达水平,表明在繁殖阶段中的起源中可能作用。拟南芥基因AtSPL2,AtSPL10和AtSPL11在决定叶片形状和胚胎形态发生方面起重要作用[20.,57].所有的BNASBP2.,10和11基因被归为同一组SBP-e。探索这些群体的确切作用将是很有趣的SBP-BOX.基因的功能特性。
miR156靶位点的保存SBP-BOX.基因
更多的mirna靶点是转录因子,如SBP、MYB、NAC、ARF、GRAS、AP2 [27].miRNA在调节靶基因的转录方面发挥重要作用。前述结果表明,MiR164,miR159a和mir319的过度表达分别影响了NaC,MyB和TCP系列的NAC,MYB和TCP系列的成员[58- - - - - -60].在目前的研究中,目标预测显示58中的44个BnaSBP基因受到miR156的调控。miR156的互补位点位于30的编码区BnaSBP基因,以及其他14个基因的3 ' UTRBnaSBP基因。拟南芥中,10 (AtSBP2, 3,4,5,6,9,10,11,13,15)在17个中SBP.预测或验证基因以由miR156靶向。另外六个SBP.基因包括(在sbp1, 7,8,12,14,16)不是miR156的靶标。AtSPL7已被证明可直接与含有GTAC核心序列的Cu反应元件(CuRE)结合并调节Cu稳态[3.].44BnaSBP预测由miR156靶向的基因是拟南芥中的同源基因,其也形成了10个基因簇。因此,SBP-Box基因中的miR156靶位位点在植物物种中保守。
miR156在拟南芥中过表达显著抑制SPL.从而降低顶端优势,导致矮化,增加总叶数和植物生物量[28].目标的转录物SBP.其他miR156过表达植物中的基因也受到抑制[29,56].在目前的研究中,miR156的转录物水平在芽和石渣中丰富(图。11).相比之下,大多数假定的目标SBP.miR156靶位点预测的基因在这些组织中表达水平较低(图1)。9和10).在花器官中,最BnaSBP基因在花瓣和胚珠中表达量较低,而在角果的主要成分果皮中转录量较高。这些结果提示miR156的转录与大部分的表达呈负相关BnaSBP基因。miR156水平下降,同时在SPL.在拟南芥衰老期间的水平[61].SPL9和SPL10.通过直接激活miR172的表达,介导了miR156从高水平向高水平的miR172的转变,从而促进了青少年向成人阶段的转变[57,62].我们的结果显示,miR156在分支角较大的6098B中的表达量低于分支角较小的Purler(图。11)与多种表达差异呈负相关SBP-BOX.基因,如。BNASBP2A,2D,3D,3E,5D,8B,9A,9B,10B,11A,11C,13D和15摄氏度(图。9和10)表示SBP / MIR156模块可能参与调节工厂架构B. Napus..
结论
通过全基因组分析SBP-BOX.油菜籽中的基因(B. Napus L.), 58SBP-BOX.在中发现基因B. Napus.基因组。将BNASBP蛋白分为八个不同的群体,并显示出来自水稻和拟南芥的SBP成员的明确正交关系。我们的结果表明很多SBP-BOX.预测由MiR156靶向的基因具有组织特异性表达模式,并且在基因重复后发散的表达模式。miR156s的表达水平在根,花和硅样本中丰富。miR156与mir156之间的不同表达模式SBP-BOX.提示SBP/miR156模块可能在发育过程中发挥重要作用。11SBP-BOX.类似于拟南芥的基因组预计将被MiR156瞄准,这意味着SBP / MIR156模块调节模式的保护。一些人的参与BnaSBP基因以及植物架构调节中的SBP / MIR156模块也涉及结果。携带我们的数据,提供了有价值的信息,以进一步研究功能SBP-BOX.in.B. Napus..
缩写
治愈,Cu-response元素;Mw:分子量;SBP,鳞状细胞启动子结合蛋白
参考资料
- 1。
锣,沉yp,ma lg,潘y,du yl,王dh,yang jy,hu ld,刘xf,董cx,ma l,chen yh,yang xy,gao y,zhu d,tan x,mu Jy,张某,刘友,dinesh-kumar sp,li y,wang xp,gu hy,qu lj,bai sn,lu yt,李jy,zhao jd,zuo j,huang h,邓xw,zhu yx。拟南芥转录因子基因的基因组宽的污声与分析。植物理性。2004; 135:773-82。
- 2.
Klein J,Saedler H,Huijser P.新的DNA结合蛋白质包括推定的转录调节因子Antirrhinum Majus.花分生组织同源基因SQUAMOSA。Mol Gen Genet 1996; 250:7-16。
- 3.
Yamasaki K, Kigawa T, Inoue M, Tateno M, Yamasaki T, Yabuki T, Aoki M, Seki E, Matsuda T, Nunokawa E.拟南芥SBP-family转录因子的dna结合域的解决结构揭示了一个新的锌结合基序。中华医学杂志。2004;37:49 - 63。
- 4.
Birkenbihl Rp,Jach G,Saedler H,Huijser P.植物特异性SBP结构域的功能解剖:DNA结合和核定位域的重叠。J Mol Biol。2005; 352:585-96。
- 5.
Cardon G,Hohmann S,Klein J,Nettesheim K,Saedler H,Huijser P.拟南芥SBP-Box基因的分子表征。基因。1999年; 237:91-104。
- 6.
谢凯,吴超,熊磊。水稻SQUAMOSA启动子结合转录因子与microRNA156的基因组结构、差异表达及相互作用。植物杂志。2006;142:280 - 93。
- 7.
Hultquist JF, Dorweiler JE。玉米mop1和ts1雌穗突变体表现出miR156和特异性SBP-box基因水平的改变。足底。2008;229:99 - 113。
- 8.
Moreno Ma,Harper LC,Krueger Rw,Dellaporta SL,Freeling M.Ligulless1编码含有核局部化蛋白质所需的核局部化蛋白,玉米叶片器官发生在玉米叶片器官期间。基因开发。1997; 11:616-28。
- 9。
张Y,Schwarz S,Saedler H,Huijser P.Spl8,拟拟拟拟拟拟拟征白的发育过程中的局部调节剂。植物mol biol。2007; 63:429-39。
- 10。
王建伟,王志强,王志强,等。mir156靶向SPL基因和CYP78A5/KLUH对水稻叶绿体长度和器官大小的双重影响拟南芥蒂利亚纳.植物细胞。2008;20:1231-43。
- 11.
Nodine MD, Bartel DP。在植物胚胎发生过程中,microrna可以防止早熟的基因表达并使模式形成成为可能。基因Dev。2010;24:2678 - 92。
- 12.
Martín-Trillo M, cuba P. TCP基因:十年后的家庭快照。植物学报。2010;15:31-9。
- 13.
关键词:拟南芥,mir156 -靶向SBP-box转录因子,雄性育性植物细胞。2010;22:3935-50。
- 14.
余宁,蔡伟,王绍平,单超,王磊,陈晓霞拟南芥蒂利亚纳.植物细胞。2010;22:2322-35。
- 15.
李志刚,李志刚。植物发育阶段转变的调控。植物学报。发展。2011;138:4117-29。
- 16.
拟南芥SBP box基因SPL3的3'UTR miRNA156/157识别元件通过翻译抑制抑制幼苗早期开花。植物j . 2007; 49:683 - 93。
- 17.
SPL8是一种影响拟南芥花粉囊发育的SBP-box基因。植物细胞。2003;15:1009-19。
- 18.
关键词:拟南芥,SPL8, mir156, SPL基因,分化模式植物j . 2013; 75:566 - 77。
- 19。
在拟南芥中,SQUAMOSA启动子结合蛋白- like7是铜稳态的中心调控因子。植物细胞。2009;21:347 - 61。
- 20。
Shikata M,Koyama T,Mitsuda N,Ohme-Takagi M.拟南芥SBP-Box基因SPL10,SPL11和SPL2对生殖阶段芽成熟的形态变化。植物细胞生理。2009; 50:2133-45。
- 21。
王T,潘H,王继,杨W,程T,张Q.新型和保守微润罗斯的识别与剖析李春万通过深度测序。Mol Genet基因组学。2014; 289:169-83。
- 22。
焦勇,王勇,薛东,王杰,严明,刘国栋,董刚,曾东,卢志杰,朱旭,钱强,李军。OsSPL14对水稻理想植株构型的调控。Nat麝猫。2010;42:541-4。
- 23。
Miura K,Ikeda M,Matsubara A,Song X,Ito M,Asano K,Matsuoka M,Kitano H,Ashikari M. Osspl14促进了水稻中的穗分支和更高的谷物生产力。NAT Genet。2010; 42:545-9。
- 24.
玉米SBP-box转录因子unbranched2和unbranched3通过调控侧原基起始率来影响产量性状。中国科学院院刊。2014;111:18775-80。
- 25.
植物microrna的起源、生物起源和活性。细胞。2009;136:669 - 87。
- 26.
陈晓东,陈晓东。植物microrna的形成、转化和作用机制。植物细胞。2013;25:2383 - 99。
- 27.
Rhoades MW, Reinhart BJ, Lim LP, Burge CB, Bartel B, Bartel DP。植物microRNA靶标的预测。细胞。2002;110:513-20。
- 28.
Schwab R, Palatnik JF, Riester M, Schommer C, Schmid M, Weigel D. microrna对植物转录组的特异作用。Dev细胞。2005;8:517-27。
- 29.
吴刚,吴国华。植物生长发育的时间调控拟南芥蒂利亚纳通过mir156及其目标spl3。发展。2006; 133:3539-47。
- 30。
拟南芥miR156通过SPL转录因子调控对反复环境胁迫的耐受性。植物细胞。2014;26:1792 - 807。
- 31。
yu n,niu qw,ng kh,chua nh。MIR156 / SPLS模块在拟南芥中的作用的作用。工厂J. 2015; 83:673-85。
- 32。
山东省,公园,翟杰,古佐达,德波利e,迈耶斯,绿色pj。大米小RNA的大规模分析:鉴别靶RNA裂解调节微小RNA的机械效力。植物细胞。2011; 23:4185-207。
- 33。
Cuperus JT,Fahlgren N,Carrington JC。miRNA基因的进化与功能多样化。植物细胞。2011; 23:431-42。
- 34。
张SD,凌光,易Ts。陆地植物中SBP箱基因的演变与分歧。BMC基因组学。2015; 16:787。
- 35。
Morea EG, da Silva EM, E Silva GF, Valente GT, Barrera Rojas CH, Vincentz M, Nogueira FT.陆地植物miR156和miR529家族的功能和进化分析。BMC Plant Biol. 2016;16:40。
- 36.
Riese M, Höhmann S, Saedler H, Münster T, Huijser P.种子植物中SBP-box基因家族的比较分析。基因。2007;401:28-37。
- 37.
杨Z,王X,顾S,Hu Z,徐H,徐C.拟南芥SBP-Box基因家族的比较研究。基因。2008; 407:1-11。
- 38.
侯H,李继,高M,歌手SD,王H,毛L,Fei Z,王X.基因组组织,系统发育比较和SBP-Box家族基因葡萄的差异表达。Plos一个。2013; 8:E59358。
- 39.
李军,侯华,李旭,向杰,殷旭,高华,郑勇,Bassett CL .,王旭。苹果SBP-box家族基因的全基因组鉴定与分析。植物生理学报。2013;70:100-14。
- 40.
李春华,卢树华SPL.基因家族在杨树trichocarpa.BMC植物BIOL。2014; 14:131。
- 41.
Larkin Ma,Blackshields G,Brown NP,Chenna R,McGettigan Pa,McWilliam H,Valentin F,Wallace Im,Wilm A,Lopez R,Thompson JD,Gibson TJ,Higgins DG。Clustal W和Clustal X 2.0版。生物信息学。2007; 23:2947-8。
- 42。
刘杰,王W,梅D,王H,傅L,刘D,Li Y,Hu Q.在油菜籽中分支角度和基因组关联映射的特征芸苔栗鸟L.)。前植物SCI。2016; 7:21。
- 43。
沉J,谢克,熊L.单管茎环逆转录量化PCR的水稻微卷曲的全局表达分析揭示了MicroRNA在非生物应激反应中的重要作用。Mol Genet基因组学。2010; 284:477-88。
- 44。
Kozomara A,Griffiths-Jones S. Mirbase:使用深度测序数据注释高置信微大乐队。核酸RES。2014; 42:68-73。
- 45。
赵永涛,王敏,付士祥,杨伟伟,祁昌科,王学军。两个甘蓝型油菜品种的小RNA分析鉴定了与油料生产和发育相关表达的小RNA和新的小RNA类。植物杂志。2012;158:813-23。
- 46。
Salinas M,兴S,HöhmannS,伯恩特·克,番石司机P.基因组组织,系统发育比较,番茄转录因子的转录因子族的差异表达。Planta。2012; 235:1171-84。
- 47。
沉娥,邹茹,休伯特州eprens f,chen l,ye c,dai s,li r,ni m,jiang x,秋j,liu y,王w,zhu qh,chalhoub b,bancroft i,蒙友D,AlloPyploid中MiRNA的鉴定,演化和表达分区芸苔栗鸟.J Exp Bot。2015;ERV420。[epub领先]
- 48.
郭艾,朱庆华,顾旭,葛旭,杨静,罗静。植物特异性SBP-box转录因子家族的全基因组鉴定及进化分析。基因。2008;418:1-8。
- 49.
张S,Ling L. SBP-Box基因家族的基因组鉴定和进化分析蓖麻.《公共科学图书馆•综合》。2014;9:e86688。
- 50。
联合国。基因组分析芸苔属植物特别参照实验形成B. Napus.和特殊的施肥方式。JPN J BOT。1935; 7:389-452。
- 51。
徐Z,Sun L,周Y,杨W,程T,王J,张Q.施氏氏氏氏菌鳞片启动子结合蛋白(SBP)箱基因家族的鉴定及表达分析。Mol Genet基因组学。2015; 290:1701-15。
- 52。
Schwarz S,Grande Av,Bujdoso N,Saedler H,Huijser P. MicroRNA调节的SBP-Box基因SPL9和SPL15对照拟南芥中的测量成熟。植物mol biol。2008; 67:183-95。
- 53。
Cardon G, Höhmann S, Nettesheim K, Saedler H, Huijser P.功能分析拟南芥蒂利亚纳SBP-box基因SPL3:一个参与花的转变的新基因。植物j . 1997; 12:367 - 77。
- 54。
Lännpääm,jänöneni,hölttävuorim,Gardemeister M,Porali I,Sopanen T.一种新的SBP-Box基因BPSPL1in.黄桦(Betula Pendula).杂志杆菌。2004;120:491 - 500。
- 55。
Manning K,Törm,普尔M,Hong Y,Thompson Aj,King GJ,Giovannoni JJ,Seymour GB。在编码SBP箱转录因子的基因中的天然存在的表观遗传突变抑制了番茄果实成熟。NAT Genet。2006; 38:948-52。
- 56。
Kim JJ, Lee JH, Kim W, Jung HS, Huijser P, Ahn JH。在拟南芥中,microRNA156-SQUAMOSA启动子结合蛋白- like3模块通过开花位点T调控环境温度响应的开花。植物杂志。2012;159:461 - 78。
- 57。
6 .吴刚,王金文,王金文,魏格尔。miR156和miR172对拟南芥发育时间的调控作用。细胞。2009;138:750-9。
- 58。
作者:陈志强,陈志强,陈志强,陈志强。用赤霉素调控的microRNA调控花的发育。发展。2004;131:3357 - 65。
- 59。
Laufs P,Beucelle A,Morin H,CUC基因的Traas J. microRNA调节是拟南芥分生商品中的边界大小控制所必需的。发展。2004; 131:4311-22。
- 60.
吴晓霞,王志刚,王志刚,王志刚。微rna对水稻叶片形态发生的调控作用。华北农学报。大自然。2003;425:257 - 63。
- 61.
Wang JW, Czech B, Weigel D. mir156调控SPL转录因子的内源性开花途径拟南芥蒂利亚纳.细胞。2009; 138:738-49。
- 62.
Fornara F, Coupland G.植物的相变引起了轰动。细胞。2009;138:625-7。
致谢
我们要感谢约翰英纳斯中心的雷切尔威尔斯博士对手稿的修订和评论。
资金
中国农业科学院科技创新项目(组号:118);中国农业科研体系专项资金(CARS-13);湖北省农业科技创新中心资助项目;
可获得的支持数据
支持本文结果的数据集包含在文章及其附加文件中。本文中描述的材料将根据要求自由地提供,要求任何希望为非商业用途使用它们的科学家。从Datead数据库中可以实现系统发育和基因组数据(http://dx.doi.org/10.5061/dryad.3rk33).
作者的贡献
HTC和QH设计调研;HTC、WXW进行生物信息学分析,HTC、MYH进行qRT-PCR实验和miRNA分析,DSM、JL进行RNA-seq分析,CBT进行表达模式分析,HW、LF提供植物材料并制备RNA样本,HTC和QH撰写论文。所有作者都已阅读并批准了本手稿的版本。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
同意出版
不适用。
伦理批准和同意参与
不适用。
作者信息
隶属关系
通讯作者
附加文件
附加文件1:表S1。
基因表达分析中用于定量聚合酶链反应(QPCR)的引物。(Doc 36 KB)
附加文件2:图S2。
BnaSBP蛋白系统发育分析。拟南芥(AtSBP)、水稻(OsSBP)和水稻(OsSBP)编码的保守SBP结构域序列B. Napus.使用ClustalW对SBP-box蛋白进行比对。系统发育树采用最大似然法,复制1000次。条形图表示每个残基0.1 aa替换。(PPTX 73 kb)
附加文件3:表S2。
十二个组织样品中的绝对基因表达值。(XLSX 18 KB)
附加文件4:图S1。
两条线具有不同分支角的表型。(a)在中间开花阶段生长的6098B和幻灯线。酒吧= 25厘米。(b)6098b的分支角度大于麦体的分支角度。箭头表示两条线的不同分支角度。酒吧= 2厘米。(PPTX 145 KB)
附加文件5:表S3。
两个样品中的绝对基因表达值。(XLSX 15 KB)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
程华,郝敏,王伟。et al。基因组鉴定、鉴定及差异表达分析SBP-BOX.基因家族在芸苔栗鸟.BMC植物杂志16,196(2016)。https://doi.org/10.1186/s12870-016-0852-y
收到:
接受:
发表:
关键词
- SBP-BOX.
- Squamosa启动子结合蛋白
- 转录因子
- 芸苔栗鸟