摘要
背景
属brasiliense被强调为卡兰内酯的重要资源,卡兰内酯是双吡喃香豆素,可抑制人类免疫缺陷病毒1型(HIV-1 RT)的逆转录酶。尽管具有重要的医学意义,但参与卡兰内酯、生物合成和途径本身的酶在很大程度上仍然未知。此外,没有这种植物的基因组资源存在。
结果
在这项工作中,我们首先分析了转录组c . brasiliense叶子,茎和根使用RNA-seq策略,这为功能基因挖掘提供了数据集。根据该化合物的结构,提出了可能的生物合成途径。最后,通过基于同源性的BLAST和系统发育分析,筛选出转录组数据集中可能参与伞形虫酮和角型pyranocoumarin生物合成途径的候选unigenes。
结论
本研究生成的单基因数据集为进一步的分子研究提供了重要的资源c . brasiliense特别是用于线性和角型吡喃香豆素生物合成途径中候选基因的功能分析。
背景
属sp.(卡茶树科)是热带树木的一大群,有180-200多种[1].目前,该属的一些物种由于其有前途的植物化学方面引起了科学界的极大兴趣。在墨西哥,在美国发现的八种中分布最广的一种是属brasiliense综眼(2],生长在巴西至墨西哥西北部的热带雨林中[3.].本种含有大量多样的次生代谢产物,包括黄酮类化合物、三萜、香豆素类、色酮类和山酮类[4],其中一些具有有趣的抗利什曼原虫、抗菌、抗癌、抗寄生虫和抗病毒特性[4,5].两种化学类型已根据其地理来源进行了分类。Chemotype 1 (CTP 1),生长在墨西哥韦拉克鲁斯州的Sierra de Santa Marta,生产mammea型香豆素在体外对人肿瘤细胞的细胞毒活性及抑菌特性Staphyloccoccus球菌,美国epidermidis而且枯草芽孢杆菌[6].与此同时,化学型2 (CTP 2)生长在墨西哥韦拉克鲁斯州的San Andres Tuxtla,并产生calanolides,这是一系列四环双吡喃香豆素,对人类免疫缺陷病毒1型(HIV-1 RT)的逆转录酶具有抑制作用[2,7].有三种不同的卡兰内酯(A, B和C)已被发现c . brasiliense并表现出对HIV-1病毒复制的显著抑制。有趣的是,这些生物活性化合物对MT2人类淋巴细胞没有毒性[7].更多的研究表明,高剂量的B和C卡兰内酯可导致脾脏巨核细胞数量增加,而肝细胞没有改变[8].Calanolide A具有最高的病毒复制抑制作用,已被合成,据报道具有与天然产物相似的作用[9,10].事实上,这种分子正在临床开发中,作为一种对抗HIV-1感染的新型治疗剂[11,12].在植物中,卡兰内酯主要可以在叶片中检测到(从CTP2中),即使它们来自于植物的幼苗c . brasiliense从种子开始生长在温室里[13].在植物愈伤组织中也可检测到Calanolides [13],细胞悬浮培养物,以及12个月大的植物叶子在体外的幼节茎再生的c . brasiliense植物(14].
卡拉内酯生物合成的代谢途径涉及一系列复杂的酶促反应,其中l -苯丙氨酸和反式-肉桂酸可以被认为是主要前体。重要的是要强调一些中间体,如7-羟基香豆素(伞形花序酮)具有抗真菌等有趣的特性[15,16]及杀虫剂[17]的活性,而且对人膀胱癌E-J细胞系的增殖也有很强的抑制活性([18]引于[15])。
尽管有很大的药理学意义c . brasiliense代谢化合物如卡兰内酯的合成和功能的基因组基础仍然知之甚少。在这里,我们提出了一个完整的转录组分析的第一份报告C.brasiliense.本研究的主要目标是描述C.brasiliense(CT2),为未来该物种的基因鉴定和功能基因组学研究提供参考。我们进行了新创转录组测序和RNA文库的组装从末端叶,茎和根在体外再生c . brasiliense幼苗。我们为公共数据库提供注释,并将转录本分类为生物学功能和通路。此外,我们还提出了几种甘兰内酯的生物合成途径,基于部分基因的同源性,我们认为其中一些基因有望成为未来甘兰内酯生物合成途径分析的候选基因。
结果和讨论
测序与装配
共生成16,842,368对端读(2x150)(叶为5,276,841,茎为5,000,558,根为6,240,602)。在组装过程之前,使用SeqPrep管道对成对的读取进行修剪和/或合并(有关详细信息,请参阅方法)。一个新创使用oasis [19],一个基于Bruijn图的汇编器,被设计为Velvet的扩展[20.]主要用于组装来自转录组数据的短读序列。Velvet/Oases一共生产了61620个contigs,长度从0.1到10 kb不等,平均长度为547.28 bp1).contig集的GC含量约为44.7%,与Malpighiales目其他物种的编码区GC含量相近(见[21])。对这些contigs的N50进行了估算,N50值较高,为867 bp。在200 bp ~ 500 bp长度范围内,有40,727个组合片段(40,727个,占总数的66.01%),表明存在组合片段。出于实际目的,在本文中,数据集中的所有contigs都被称为unigenes。BLASTx算法[22],基于传统的top-BLAST-hit标注方法对unigenes进行标注。作为参考,一个蛋白质数据库的集合,包括拟南芥(拟南芥)和植物RefSeq数据库用于此目的。有效值(e值)为10−5作为BLASTx相似性搜索的阈值(附加文件2:表S1)。
功能注释
基于拟南芥的top hits, Gene Ontology (GO)进行了注释c . brasiliense得到Unigenes。WEGO软件[23]对GO进行功能分类,分为生物过程、分子功能和细胞成分三大类。在拟南芥命中的unigenes中,42090个(68.30%)被分配到不同的基因本体类别中,共有367994个功能项,其中103865个是唯一的。生物过程占功能术语的大多数(178,629个;48.54%),其次是细胞成分(95,428;25.93%)和分子功能(93,937;25.52%)(附加文件3.:图S1;参见附加文件4:表S2)。氧化石墨烯生物过程中排名最高的类别是对应于细胞(27,090个unigenes)和代谢(24,653个unigenes)过程的子类别。有趣的是,刺激反应(14,101个unigenes)和生物调节(12,646个unigenes)在GO生物过程类别中也有显著表现。除了基于GOs的功能注释,c . brasilienseunigenes根据可用的代谢途径进行分类,并在京都基因和基因组百科全书(KEGG)中进行了描述。KEGG自动注释服务器(KAAS;[24)被分配给C.brasilienseunigenes KEGG Orthology (KO)编码和酶委员会(EC)编号。KO编码被分配给4881个unigenes,其中1733个可能与226个不同代谢途径相关的特定EC编号相关(附加文件)2:表S1)。
基因表达谱c . brasiliense器官
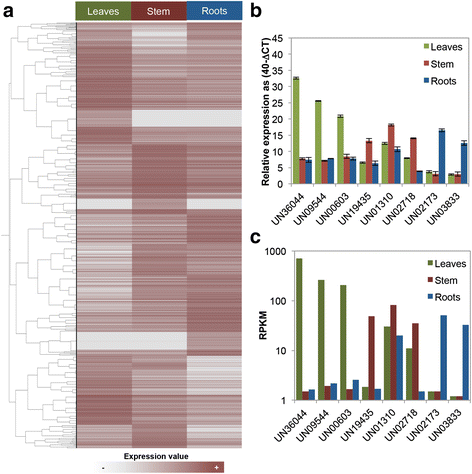
根、茎和叶的基因表达谱通过配对reads到转录组组装进行分析。在每个转录组样本(列)中创建一个包含contigs(行)和映射读数的表达谱矩阵(附加文件)4:表S3)。为了使样本之间的读取计数具有可比性,进行了每千碱基每百万(RPKM)读取的归一化。为了从背景样本中区分至少一个器官中的表达基因,使用RPKM≥3的阈值[25].根据严格度,在三种样本组织中均表达了26,299个unigenes(占总数的59.26%)(泛在基因)。其余的unigenes在单个组织中表达(器官特异性)或分布在不同的组织组合中。似然比统计量(r统计量)[26],并用于识别样本器官间表达水平有显著差异的基因。r值越高表示差异表达的概率越大,而r值接近零表示本构表达。在这种情况下,它被认为是unigenes的优先表达基因R-值≥8(真阳性率~ 98%)[26].共计3173人(7.15%)c . brasilienseUnigenes被鉴定为优先表达基因,在叶片中有1274个,在茎中有965个,在根中有934个,其中一些(分别为247个、143个和231个)可被认为是器官特异性的。(无花果。1和附加文件4:表S4)。
一个层次聚类c . brasiliense基于RPKM值优先表达unigenes。日志2RPKM值用不同的颜色表示。深棕色表示较高的表达值,而热图中的灰色表示最低的表达值。b基于rt - qpcr的八选表达c . brasiliense根据各器官(叶、茎和根)的表达水平优先表达unigenes。表达水平为40-ΔCT,其中ΔCT为对应基因与参考基因UN18770(同源于拟南芥上编码UBQ11的AT4G05050)的阈值周期数之差;选择数字40是因为PCR运行在40个循环后停止,并且需要一个恒定值作为校准器(见示例[76])。结果显示为三个生物重复的平均值±SEcRNA-Seq表达水平测量为每kb每百万reads (RPKM)
为了深入了解器官功能之间的联系,我们调查了排名前10位的器官特异性unigenes。叶单位是3c . brasiliense与拟南芥甘氨酸-天冬氨酸-丝氨酸-亮氨酸基序脂肪酶/水解酶(GDSL脂肪酶)家族成员同源的UN36044、UN28345和UN34582,以及3-酮酰基辅酶a合成酶(KSC)蛋白的一些同源物(UN09544和UN13106)高表达。一直以来,GDSL脂肪酶基因家族的成员,如AT5G33370和AT3G04290,都与参与角质层生物合成的基因如CER6/KCS6/CUT1 (AT1G68530)共同调控,后者在角质层蜡合成的超长链脂肪酸伸长中起主导作用[27- - - - - -29].脂氧合酶/过氧合酶途径也参与角质单体的生物合成[30.];这可以解释UN00603的表达谱,该基因被鉴定为叶片特异性的单基因,并注释为与叶绿体脂氧合酶LOX2 (AT3G45140)同源,后者是拟南芥损伤诱导茉莉酸积累所需的酶[31].考虑到表皮蜡质要么只在叶片发育和扩张过程中产生,要么在叶片的整个生命周期中产生,所有这些基因在叶片转录组中的存在是意料之中的[18].
对于茎,束素样阿拉伯半乳聚糖蛋白的同源物FLA12 (AT5G60490;unigene UN35075)作为茎特异性基因表现出最高的表达水平,这与之前的报道一致,这些报道表明FLA基因家族的一些成员的表达与拟南芥茎中次生壁纤维素合成的开始有关,并且与树木茎和树枝中的木材形成有关。这一数据提示UN35075单基因可能在该疾病中发挥生物学作用c . brasiliense茎发展[32].此外,编码与单木酚生物合成相关的酶的基因,如苯丙氨酸解氨酶(PAL1;UN01310),咖啡酰辅酶a 3-O甲基转移酶(CCoAOMT;UN21637和UN12250)和4-香豆酸:CoA连接酶(4CL;UN01988)被鉴定为优先在茎中表达,尽管在所有三个器官样本中都能检测到转录本。木质素在植物茎的组织传导中起着至关重要的作用,它是由单木质素的氧化偶联合成的[33].此外,拟南芥IRX蛋白同源的16个unigenes也被分类为在茎中特异性表达或优先表达;考虑到不规则的木质部(irx)突变体的特征是茎组织中的纤维素减少[34].最后,两个c . barsilienseUN02173和UN03226与高亲和磷转运蛋白(PHT1)家族成员同源,仅在根中高表达,UN03833单基因与ARSK (AT2G26290)同源,ARSK是一种编码根特异性激酶的基因[35].
为了验证归一化读计数获得的表达谱,使用9个选择的基因进行RT-qPCR。所有被评价基因的RT-qPCR表达谱与读计数分析得出的谱完全一致(图2)。1).
优先表达基因的功能注释
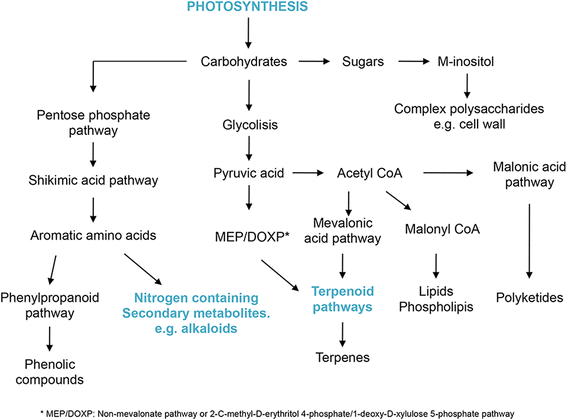
对GO术语进行富集分析,以确定在生物过程类别中,那些在所分析的至少一个器官中过度代表的子类别(附加文件4:表S5)。为了比较某些优先表达基因与特定生物过程的丰度,对优先表达基因使用了相同方法的修改(在器官优先表达基因中标注给定GO项的unigenes的百分比与在完整的基因集中标注相同GO项的基因的百分比进行比较c . brasiliense转录组;额外的文件4:表S6)。将GO项分配给3173个的子集c . brasiliense在生物过程类别中优先表达unigenes,一些过度代表的生物过程是光合作用,生物碱和萜类代谢相关的(附加文件3.:图S2;参见附加文件4:表S7)。这与所有次生植物产物的碳骨架来源于光合作用合成的碳水化合物的概念是一致的,其中一些碳水化合物的合成依赖于初级代谢产物(图2)。2).值得注意的是,当比较每个器官中每个过程的unigenes分组数量时,某些生物过程没有显着差异。苯丙烷生物合成途径(GO: 0009699)的情况就是如此,它显示在每个器官样本中平均有39个基因分组在这一类别中(附加文件)4:表S6)。
导致次生代谢产物合成的主要生物合成途径(由[77])。以青色显示的代谢途径代表了在叶片优先表达基因中鉴定的过度代表的生物过程
伞形花序酮的生物合成,伞形花序酮是卡兰内酯形成的关键前体
香豆素在植物中是通过海草酸途径合成的,其中苯丙氨酸是最终产物,还产生芳香族氨基酸酪氨酸和色氨酸以及其他小分子,如黄酮类化合物和羟基肉桂酸缀合物[36].连续的帕拉- - -昊图公司-羟基化反式-肉桂酸盐(的共轭碱反式-肉桂酸)通过2-香豆酸形成香豆素,或通过4-香豆酸形成羟基香豆素,如伞形花序酮(7-羟基香豆素)。在C-7缺乏氧合的其他羟基香豆素也共享反式-肉桂酸为前驱体。根据EC编号分配给c . brasiliensE单基因,只有一个例外(谷氨酸-预苯酸转氨酶;EC: 2.6.1.79),通过莽草酸途径形成l -苯丙氨酸所需的所有酶的同源物被鉴定出来(附加文件3.:图S3)。
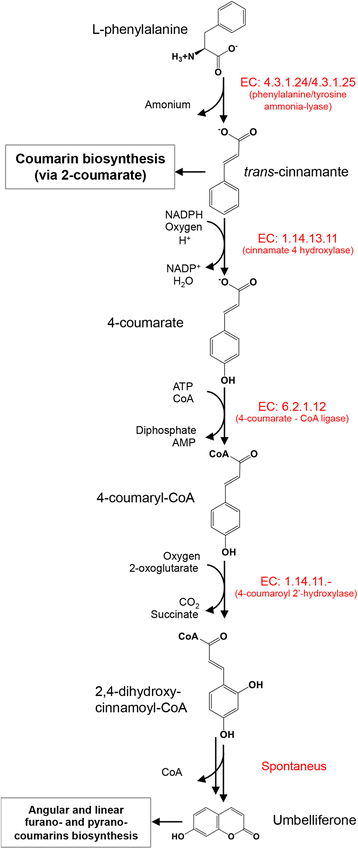
伞形花序酮生物合成途径(通过4-香豆酸酯)涉及的关键酶是:a)苯丙氨酸解氨酶[PAL;EC: 4.4.1.24], b)肉桂酸4羟化酶[C4H;EC:1.14.13.11], c) 4-香豆酸酯:CoA连接酶[4CL;d) 4-香豆油基2 ' -羟化酶[EC:1.14.11]。-)(无花果。3.).这些参与伞形花序酮生物合成的酶已在太阳之graveolens物种(39].
伞形花序酮的生物合成途径始于l -苯丙氨酸。该图是从MetaCyc (http://metacyc.org/)寻找伞形花序素的生物合成
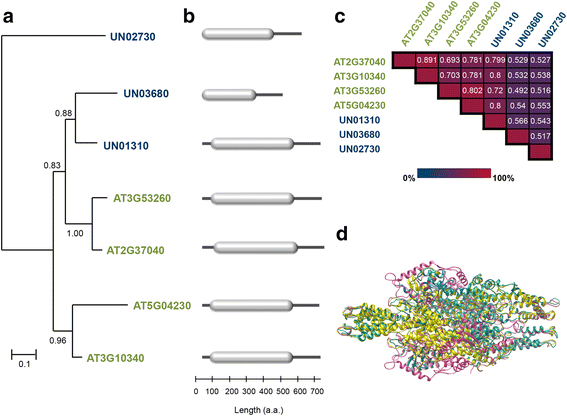
PAL基因家族成员编码的蛋白可以催化l -苯丙氨酸向氨和氨的转化反式-肉桂酸,并在被子植物和裸子植物中发现,也在一些苔藓、藻类、真菌和细菌中发现[37].根据KEGG中提取的注释,UN01310被鉴定为PAL的同源基因(K10775, EC:4.3.1.24)。WISE2程序[38) (http://www.ebi.ac.uk/Tools/psa/genewise/)和拟南芥PAL1蛋白(AT2G37040)被用于识别其正确的开放阅读框中的编码序列(该程序使用蛋白质序列作为模板来预测分析的DNA序列中的相关蛋白质序列),然后使用SeaView程序[39],蛋白质编码的核苷酸序列根据其相应的氨基酸翻译进行对齐,以计算核苷酸和氨基酸水平上的同一性百分比(附加文件5).两者的同一性c . brasiliense/拟南芥朋友在核苷酸水平(CDS)和氨基酸水平上分别为69.05%和79.9%。通过基序/结构域搜索,在Pfam数据库中鉴定了独特的芳香族氨基酸裂解酶保守结构域(PF00221) [40) (http://pfam.xfam.org/).通过基于PF00221域的BLAST搜索,共有18个c . brasilienseUnigenes被鉴定为同源于朋友所有这些基因都被注释为现有四种拟南芥中的一些同源基因朋友基因([41] PAL1;PAL2 AT2G37040);AT3G53260 PAL3;AT5G04230和PAL4;AT3G10340)在顶部爆炸命中注释过程(附加文件2:表S1)。一种系统发育树c . brasiliense生成PAL蛋白。只有来自其相应编码序列的蛋白质至少占其同源物长度的65%的unigenes被纳入(UN01310, UN03680和UN02730)。的朋友拟南芥的家族成员也被纳入系统发育分析。多个序列对齐(附加文件6)表明c . brasiliense这些蛋白与拟南芥中的PAL家族蛋白具有高度的同源性(从49.2%到80.0%),三维结构分析表明,这些蛋白也含有与先前报道的其他PAL植物蛋白高度相似的推测结构(图2)。4).蛋白质序列的高度相似性c . brasiliense拟南芥PAL家族的同源物和3D模型表明它们可能发挥类似的功能作用。
一个最大似然系统发生树描述不同的关系c . brasiliense(蓝色)和拟南芥(绿色)PAL家族成员。蛋白质序列与MUSCLE [69],利用SeaView v4.0软件在最大似然框架下进行系统发育分析[39].b其中芳香族氨基酸裂解酶保守结构域(PF00221)的初级蛋白质结构用灰色框表示。c来自PAL家族成员的对齐蛋白序列之间的百分比身份矩阵c . brasiliense和拟南芥。dPAL蛋白的结构模型叠加c . brasiliense[UN01310(粉色),UN02730(黄色)和UN03680(青色)]。PAL蛋白的可用结构Petroselinum crispum在蛋白质数据库(PDB条目1w27.1)中作为模板。同源性建模使用SWISS-MODEL程序和SWISS-PDB viewer v4.1.0 ([73,75];http://swissmodel.expasy.org/)被用来叠加c . brasiliense朋友的蛋白质
使用与上述类似的方法来确定其余的c . brasiliense伞形花序素生物合成相关基因。共有3个unigenes被鉴定为唯一的同源基因反式-肉桂酸4-羟化酶(AT2G30490;拟南芥基因组中的C4H)(附加文件3.:图S4和附加文件7).C4H是一种植物特异性细胞色素P450 (PF00067),可催化多分支苯丙烷途径的第二步[42].关于4-香豆酸CoA连接酶[4CL;EC:6.2.1.12],共检测到6个与这些蛋白同源的unigenes;然而,其中只有三个(UN01603, UN01988和UN01725)是完整的开放阅读帧(附加文件)8).motif/domain搜索结果显示,在与之对应的翻译序列中,amp结合的c端结构域(PF13193)和共同的amp结合中心结构域(PF00501)都存在c . brasilienseunigenes(附加文件3.:图S5)。通常,大多数被子植物编码为4CL的小家族(例如,拟南芥为7个成员)。这些酶参与一般苯丙酸途径的最后一步,除了以4-香豆酸为底物外,它们还以不同的催化效率转化对香豆酸、阿魏酸、咖啡酸和5- oh -阿魏酸[43,44].
最后,采用双向最佳击中法(BBH)鉴定了4-香豆酚基2 ' -羟化酶的同源物[EC:1.14.11]。——从太阳之graveolens.分离得到的4-香豆酚基2 ' -羟化酶r . graveolens(编号JF799117.1)是唯一一种专门用于香豆素合成的酶,在较小程度上,该酶也接受4-香豆素- coa来产生伞形花序酮[45].的c . brasilienseUN02124基因中存在细胞色素P450保守结构域PF00067,与该基因的同源性为87.2%r . graveolens(附加文件3.:图S6和附加文件9).最后一步在活的有机体内伞形花序酮的合成途径涉及一个trans-cis异构化,然后是2,4 -二羟基肉桂酰- coa的内酯化,关闭侧链,这个反应是自发发生的(图。3.).
考虑到我们能够通过4-香豆酸盐识别伞形酮生物合成途径中涉及的所有基因的同源物,并且由于缺乏几个可能编码酶的转录本,如肉桂酸2-羟基化酶(EC: 1.14.13.14), 2-香豆酸盐O-β-葡萄糖苷转移酶(EC: 2.4.1.114)、2-香豆酸β- d -葡萄糖苷异构酶(EC: 5.2.1.-)和香豆酸葡萄糖苷β-葡萄糖苷酶(EC:3.2.1.21),这些酶首次在Melilotus阿尔巴[46,47]如果它们都通过2-香豆酸酯参与香豆素生物合成途径,我们认为在c . brasiliense,伞形花序酮是通过4-香豆酸途径合成的。这是在预料之中的,因为与哺乳动物相比,只有少数植物物种(例如。Catharanthus roseus也叫而且毒参属植物欧)是否有人认为存在能够使C-7中的香豆素羟基化以产生伞形花序素的酶(综述于[48])。
calanolide(角吡喃香豆素)生物合成途径的候选基因分析
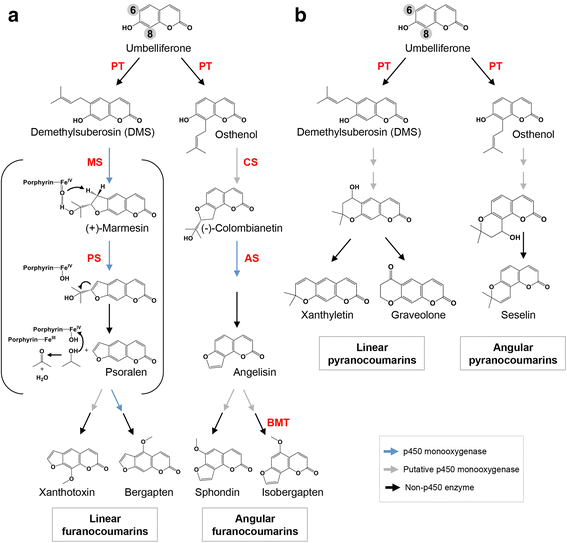
蒽醌内酯为吡喃香豆素,是香豆素衍生物的一类。吡喃香豆素的生物合成仍然知之甚少,但据报道,呋喃香豆素和吡喃香豆素之间存在着显著的结构相似性。两类香豆素(呋喃香豆素和吡喃香豆素)可分为线性型和角型。类型的确定,无论是线形还是角形,是基于共同前体(伞形酮)的烯酰化位置。在线性型中,吡喃(或呋喃)环附着在C-6和C-7上,而在角型中,取代发生在C-7和C-8 [49].这两种类型的吡喃香豆素都来自于相同的前体,分别产生线性和角状呋喃香豆素(分别为去甲基亚麦皂苷和邻苯二酚)(图。5).
生物合成途径,线性和角,呋喃-一个和pyrano-coumarinsb.已鉴定/特征酶的缩写用红色表示:PS,伞形虫烯酰基转移酶;MS, marmesin synthase;PS,补骨脂素合成酶;CS,耧斗菜素合成酶;AS,当归素合成酶;BMT,佛手酚O甲基转移酶。补骨脂素合成酶催化反应的细节在括号中。这个数字由[56]
先前在Pimpinella麦格纳而且Pastinaca漂白亚麻纤维卷植物可以被认为是第一个实验证据,证明线性呋喃香豆素和角呋喃香豆素来源于伞形花序酮,丙烯化在C-6(导致形成去甲基亚eronosin;DMS)或C-8位(osthenol), [50].在后来的几年里,进一步的研究表明,去甲基亚脂蛋白的环化导致(+)-marmesin的形成,这是通过NADPH和分子氧存在时发生的酶促反应发生的[51].这些'乳蛋白合成酶'已被鉴定为细胞色素P450单加氧酶Petroselinum crispum而且Ammi maju植物种类(在[52])。P450s催化的反应范围包括通过插入“公牛”来环氧化烯烃[53],该反应的反应产物往往通过假体的烷基化使酶失活亚铁血红素集团(54].然而,在mamersin合成酶反应中没有释放中间体,可能是去甲基亚铁红蛋白的7-羟基使双键电子离域,有利于二氢呋喃香豆素的瞬时环化(图2)。5).已经提出了P450酶催化作用介导的反应的模型机制,其中一种机制包括催化P450氧衍生物的主要相互作用,由铁-过氧化氢物种中的氧-氧键与脂肪族双键的杂溶裂解形成[52,54].这种反应机制与去甲基亚脂蛋白环化成(+)-marmesin相容,避免了中间环氧化合物的形成(图2)。5).以类似的方式催化从茴香醇生成(-)-耧耧素,并且当归素和补骨脂素合酶的后续活性已得到实验支持[55,56].线性和角形吡香豆素的合成似乎是可行的,如黄叶素[57], graveolone [58],或seselin [59]可与呋喃香豆素同时产生,其方式与分别从去甲基亚铁红素或洋樟醇产生非常相似(图。5 b).
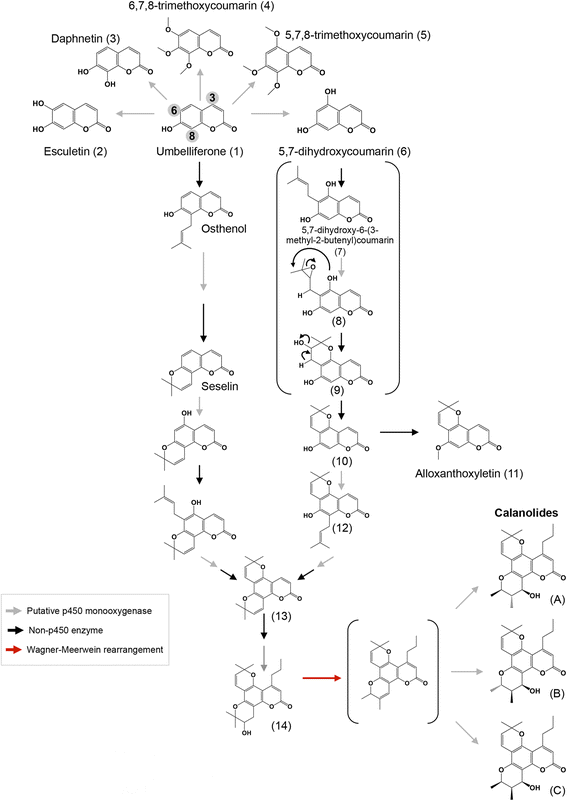
在这里,我们提出了两个密切相关的卡拉内酯生物合成途径(图。6).这些代谢途径意味着在先前在其他植物物种中发现的一些相关化合物的基础上进行生物合成的一些步骤。从伞形酮开始(1),在属于芸香科和菊科的几种植物物种中发现的高度丰富的代谢物[60],它很可能是第一个中间产物之一,之前被认为是角吡喃香豆素生物合成的前体C-8戊烯化衍生物(洋樟醇)。然而,考虑到一些伞形花序素相关的代谢物,如牛皮素(2)、达芙素(3)、6,7,8-三甲氧基香豆素(4)和5,7,8-三甲氧基香豆素(5),已在一些不同的植物物种中被发现(在[49,61]),这可能表明,5,7-二羟基香豆素(6)的形成可能是在c伞形花序酮在C-6的戊烯化反应,生成树甾醇。卡兰内酯生物合成的其他步骤包括5,7-二羟基-6-(3-甲基-2-丁烯基)香豆素(7)的环氧化到(8),然后是(8)的环化到(9),经过脱水步骤后得到(10)。(10)可能是异氧西素的前体(11)[61],或者以类似的方式,第二个c在C-8(12)上的-戊烯酰化可导致相应的环氧环化(13)。从(13)开始,依次经过C-3烷基化和吡喃环的单羟基化反应生成(14),再经过Wagner-Meerwein重排(1,2-甲基移位),再经过羟基化反应生成卡兰内酯A、B和C(图13)。6).Wagner-Meerwein重排的细节可能导致卡拉内酯的形成(可能有利于其中一种的生物合成)显示在附加文件中3.,图S7。第二个建议的途径可能以非常相似的方式发生,从c伞形花序酮在C-8位点的戊烯化,这将导致seselin的产生[59,接着是第二声c-烯丙基化在C-4位置(图。6).显然,与呋喃香豆素的生物合成不同,在呋喃香豆素的生物合成过程中避免了中间物环氧化物的形成,吡喃香豆素生物合成途径中的吡喃环的形成恰恰依赖于中间物。
考虑到呋喃香豆素和吡喃香豆素(线性和角形)之间显著的结构相似性,以及这两类化合物来源于相同的前体(DMS和邻苯二酚)的事实,有可能假设参与这些生物合成途径的酶可能具有共同的祖先。这一假设与呋喃香豆素和吡喃香豆素在同一物种中通常不共存的观察是一致的。即使在许多关于呋喃香豆素的生物合成和功能的未知问题仍未解决的情况下,很明显,我们对密切相关的化合物(如吡喃香豆素)的了解似乎更少。
关于呋喃香豆素,据报道,它们是由各种各样的植物产生的,以应对病原体或食草动物的攻击。它们被紫外线激活,对某些脊椎动物和无脊椎食草动物具有剧毒,因为它们与DNA结合,有助于细胞快速死亡[49,62].有人认为线性呋喃香豆素和角呋喃香豆素是植物和昆虫共同进化过程的结果。植物-昆虫相互作用研究表明,线形呋喃香豆素比角形呋喃香豆素毒性更大;而角结构与线性结构结合时,明显产生协同效应[62].当线性呋喃香豆素和角呋喃香豆素混合时,会形成一种组合,使昆虫更难解毒[62].显然,在进化过程中,角型呋喃香豆素出现的时间比线性型的晚,这一假设得到了基于观察的支持,即角型呋喃香豆素总是与线性结构同时出现,而线性类型则可以单独发现(在[49,63])。
近年来,许多参与呋喃香豆素生物合成的酶已在分子水平上被描述,包括三种P450单加氧酶(补骨脂素-、angelicin-和(+)-marmesin-合成酶[55,56]),佛手酚O甲基转移酶(64],以及关键酶戊烯基转移酶[63])参与了导致前体合成线性呋喃香豆素和角状呋喃香豆素的关键步骤(分别为DMS和osthenol;参见无花果。5而且6).其他依赖p450的酶步骤仍未解决,但这类酶的参与已被认为在呋喃香豆素生物合成的许多步骤。(无花果。5而且6,参见[49]的扩展修订)。呋喃香豆素是由多种植物在应对病原体或食草动物攻击时产生的。
吡喃香豆素的生物合成尚未得到研究。然而,考虑到补骨脂素合成酶与angelisin合成酶具有70%的同源性,并且其参与线性和角度呋喃香豆素生物合成已被证实[55,56],基于参与吡喃香豆素生物合成的酶可能有一个未知功能的共同祖先的假设(可能是基因复制和随后的分子进化的结果),我们使用这些和其他参与呋喃香豆素生物合成的酶作为序列参考,以识别同源性c . brasilienseunigenes集。
首先,在欧芹中发现的戊烯基转移酶(PT)Petroselinum crispum) [63], angelicin合成酶(CYP71AJ4)从Pastinaca漂白亚麻纤维卷,补骨脂素合成酶(CYP71AJ1)从Ammi majus及其同源物(CYP71AJ2和CYP71AJ3)芹菜graveolens而且Pastinaca漂白亚麻纤维卷,分别)[55,56],作为tBLASTn相似度搜索的参考(e值为10−6)对c . brasilienseunigene数据集。此外,CYP82H1特征不明显Ammi majus在植物-真菌相互作用和伴随呋喃香豆素生物合成中增加其表达水平,也被包括在内[65].在翻译时,在代表完整编码序列(CDS)的驻留窗口中显示至少20%相同的unigenes序列,或至少70%的同源蛋白,被保留以供将来分析。
共鉴定出6个pt样序列。随后与PFAM数据库的比较证实了无论在哪里戊烯基转移酶结构域(PF01040)。单基因UN01964(有35%的共享身份)与欧芹烯基转移酶在同一分支中被解析(附加文件)3.:图S8和附加文件10).考虑到这些结果,我们认为UN01964应该被认为是编码一种可能参与前体生物合成的戊烯基转移酶的主要候选者,这些前体导致了线性和角型吡喃香豆素的合成。
在吡喃香豆素生物合成的后续步骤中,共鉴定出34个unigenes与先前鉴定的参与呋喃香豆素生物合成途径的P450单加氧酶同源(严格程度:覆盖率≥70%,同源性≥20%)。在系统发育树上发现了三个主要的进化支,其中一个只包括三个进化支c . brasilienseunigenes。第二个分支汇集了系统发育分析中包含的所有CYP71AJ蛋白,共14个c . brasilienseunigenes。最后,第三个分支包括CYP82H1 P450单加氧酶和其余的c . brasiliense已识别的cyp类序列(附加文件3.:图S9,附加文件11).cyp71相关的分支包括两个姐妹分支,分别被命名为I类和II类。翻译的I类unigenes与CYP71AJ蛋白的相似性在31%到46%之间,而II类成员的相似性平均低10%(附加文件)4:表S8)。考虑到呋喃香豆素的存在,在蛋白质水平上的相似性较低是预期的c . brasiliense,它能够在幼苗(主要在叶片上)和愈伤组织培养中产生吡喃香豆素(线性和角状,包括卡龙内酯化合物)[13].根据这些基因的表达谱,44%被鉴定为优先表达在一些器官样本。UN02363、UN03063、UN04124和UN02841在叶片中优先表达。除了一个例外(UN02841),这些基因被归类为cyp71相关基因;其中两个(UN02363和UN03063)归为II类,另一个归为I类(UN04124)。此外,UN04124与补骨脂素合成酶和当归素合成酶的同源性分别为35.6%和35.8%Pastinaca漂白亚麻纤维卷.总之,这些数据表明UN04124可以被认为是参与角状和/或线性吡喃香豆素生物合成的主要候选者。
结论
本研究生成的单基因数据集为进一步的分子研究提供了重要的资源c . brasiliense特别是用于表征线性和角型吡喃香豆素生物合成途径中的候选基因。采用适当的方法,鉴定出一系列候选基因。进行连续分析,以确定其相应的表达模式和系统发育关系。候选基因在c . brasiliense转录组被认为可能参与角香豆素的生物合成,可以克隆和进一步研究。为了减少可能在特定步骤催化特定反应的候选蛋白质的数量,可以进行额外的生物信息学分析。我们认为,至少对于P450单加氧酶,可以进行对接和分子动力学分析,以减少参与calanolide生物合成途径的候选基因的数量。
方法
材料计划
此前,Bernabé等,[13],已表明愈伤组织培养或幼苗的c . brasiliense在温室中生长的能够产生卡兰内酯。为了保证卡拉内酯的生产,在体外从Bernabé等先前使用的相同植物(化学型2)的年轻节茎中再生出幼苗。这些植物是从韦拉克鲁斯州San Andrés Tuxtla收集的种子中发芽的。这些植物在温室里生长了大约5-6个月。从植株上采集幼节茎作为外植体来源。叶子,茎和根从在体外再生12个月的植物。根据制造商的说明,用TRIzol®试剂(Life technologies)分离总rna。从三个器官样本中分离出的rna,用RNeasy试剂盒(Qiagen)重新纯化,并用无rnase - DNase I (Invitrogen)处理,以去除DNA残留。使用NanoDrop 2000(赛默飞世尔),以OD260/230比值评估rna的质量和纯度。使用Agilent 2100生物分析仪(Agilent Technologies)通过RNA完整性数(RIN)评估RNA完整性。只有RIN值≥8.5的rna被用于文库生成。
cDNA文库制备及测序(RNA-seq)
cDNA制备、文库构建和测序在墨西哥LANGEBIO-CINVESTAV基因组服务实验室使用MiSeq™平台,根据制造商的说明(Illumina, San Diego, CA)进行。简单地说,利用Sera-mag Magnetic Oligo (dT) Beads (Illumina)从20 μg总RNA中分离出poly (A) RNA。为了避免在合成cDNA时引物偏误,首先将纯化的mRNA片段化成小片段。然后使用SuperScript双链cDNA合成试剂盒(Invitrogen, Camarillo, CA)和随机六聚体(N6)引物(Illumina)合成双链cDNA。利用T4 DNA聚合酶、Klenow DNA聚合酶和T4 PNK对合成的cDNA进行末端修复和磷酸化。这些修复的cDNA片段用Klenow Exo-(3 '到5 ' Exo-, Illumina)进行3 '腺苷化。Illumina配对端适配器被连接到这些3 ' -腺苷化cDNA片段的末端,对每个样本(根、茎和叶)使用特定的条形码。利用Phusion DNA聚合酶PCR引物PE 1.0和PE 2.0 (Illumina)进行15轮PCR扩增,扩增得到的cDNA模板。构建cDNA文库,片段长度范围为278 bp(±0.5 SD)。最后,在安捷伦技术2100生物分析仪上使用安捷伦DNA 1000芯片试剂盒进行验证后,使用Illumina MiSeq测序仪在一对端(2x150)流式细胞上对cDNA文库进行测序。 Files containing sequence reads and quality scores were deposited in the Short Read Archive of the National Center for Biotechnology Information (NCBI) [Accession number SRP079249].
数据过滤和新创组装
正向和反向读对(由Illumina-MiSeq生成)使用SeqPrep管道(https://github.com/jstjohn/SeqPrep),使用默认参数(phred 33的质量分数截断值,最小合并读长度为15 bp,重叠区域没有错配)。使用滑动窗口方法(窗口大小为10个基底,移位1个基底)对没有重叠的成对末端读取进行修剪。FASTX-Toolkit (http://hannonlab.cshl.edu/fastx_toolkit/index.html)被用于这个目的。如果在修剪后,读取的碱基小于30个,则会被丢弃,孤儿读也会被删除,以便只保留成对的读取。使用Oases模块的天鹅绒汇编器[19]用于序列组装。一个unigenes集合c . brasiliense只考虑最小尺寸为100bp的contigs。
注释的c . brasilienseunigenes
注释获得的unigenes新创我们使用BLASTx算法(e-value 10−5)拟南芥(TAIR v11)和其他植物蛋白(NCBI;ftp://ftp.ncbi.nlm.nih.gov/refseq/release/plant/)数据库。来自拟南芥或其他植物蛋白的顶部蛋白匹配被分配到每一个c . brasilienseunigenes。每个基因本体(GO)的功能类和路径c . brasiliense基于拟南芥GO SLIM基因和通路注释(ftp://ftp.arabidopsis.org/home/tair/Ontologies/).数据采用WEGO软件进行统计分析[23它是绘制GO注释结果的有用工具。此外,还使用KEGG自动注释服务器(KAAS;http://www.genome.jp/tools/kaas/)提供KEGG Orthology (KO)代码的注释。采用双向最佳命中(BBH)方法。根据京都基因和基因组百科全书(KEGG)中提取的注释分配酶委员会(EC)编号,并与植物代谢网络(PMN;http://www.plantcyc.org/),以及相应的ec格式MetaCyc [66]添加了交叉引用。
表达谱分析c . brasiliense转录组
组装完毕后c . brasiliense每个RNA-seq文库使用Bowtie方法分别与生成的转录组组装对齐[67].校准计数使用RSEM [68].差异表达统计分析使用FPKM(每千碱基的contigs每百万次映射读取的片段数)值和Sketel [26].简而言之,所有聚类都提交了一个对数似然比统计数据,该统计数据渐近于Stekel所描述的χ2分布。它基于单一的统计检验来描述基因在不同文库之间表达差异的程度。这种方法允许在任意数量的文库中鉴定差异表达基因。
RT-qPCR
为了在正确的开放阅读框中识别编码序列,8个unigenesc . brasiliense被鉴定为差异表达的基因与拟南芥同源基因对齐。使用SeaView程序,蛋白质编码核苷酸序列根据其相应的氨基酸翻译进行对齐(附加文件)12).基因特异性引物对(附加文件4:表S9),使用Primer3 v.0.4.0 web工具(http://bioinfo.ut.ee/primer3-0.4.0/primer3/),然后用于RT-qPCR检测。
根据制造商的说明,使用SuperScript®III逆转录酶(Life Technologies)逆转录总共10ug的总RNA。所选基因通过SYBR绿色化学(Applied Biosystem)在实时热循环仪(AB7500, Applied Biosystem)上进行qPCR。采用UBQ11 (UN18770)作为内部控制。热循环程序设置为95°C持续5分钟,95°C持续30秒,60°C持续30秒,72°C持续1分钟,循环40次。所有反应在3个生物重复中重复进行。
系统发育分析
系统发育分析采用最大似然框架,使用SeaView v2.4软件进行[39].该比对和系统发育分析由SeaView使用Muscle [69]和PhyML [70]程序。拓扑结构、分支长度和平衡频率进行了优化,并在LG (Le和Gascuel)模型下使用PhyML选项[71].使用BioNJ确定起始树,并使用最近邻交换(NNI)和子树修剪和重新嫁接(SPR)算法进行树搜索。采用近似似然比检验(aLRT)分析分支鲁棒性[72].
蛋白质建模
的c . brasiliense的三维蛋白质结构采用刚体分组方法,使用瑞士模型工作空间(http://swissmodel.expasy.org/) [73,74].该服务器用于校准蛋白质数据库(PDB)中可用的目标序列和模板结构。一旦选择了模板,目标序列的三维结构就可以建模。苯丙氨酸解氨酶(PAL)模板为1w27.1,苯丙氨酸解氨酶模板为3a9u。4-香豆酸1:辅酶a连接酶(4CL)。每个生成的模型都检查了各种参数,包括Z、GMQE(全球模型质量估计)和QMEAN(定性模型能量分析)得分,以评估模型的准确性。建模c . brasiliense使用SWISS-PDB viewer v4.1.0程序将蛋白质叠加到其相应的同源结构上[75].
参考文献
- 1.
旧大陆种的修订属(藤黄科)。阿诺德植物园学报。1980;61:117-699。
- 2.
Huerta-Reyes M, Basualdo Mdel C, Lozada L, Jimenez-Estrada M, Soler C, Reyes-Chilpa R.墨西哥Clusiaceae植物提取物对HIV-1的抑制作用。生物医药通报2004;27(6):916-20。
- 3.
潘宁顿TD, Sarukhán J. Arboles tropicales de México:手册para la identificación de capo de los principales。墨西哥:国家林业研究所;1968.
- 4.
Bernabé-Antonio A, Álvarez-Berber LP, Cruz-Sosa F.植物化学物质的生物学重要性属brasilienseCambess。生物科学进展。2014;4(10):1502-17。
- 5.
Brenzan MA, Santos AO,中村CV, Filho BP, Ueda-Nakamura T, Young MC, Correa AG, Junior JA, Morgado-Diaz JA, Cortez DA。(−)mammea A/BB分离的影响属brasiliense叶及其在线粒体膜上的衍生物利什曼虫amazonensis.植物医学:国际植物疗法和植物药理学杂志。2012; 19(3 - 4): 223 - 30。
- 6.
re耶斯- chilpa R, Estrada-Muniz E, Apan TR, Amekraz B, Aumelas A, Jankowski CK, Vazquez-Torres M.来自mammea型香豆素的细胞毒性作用属brasiliense.生命科学学报,2004;29(3):344 - 344。
- 7.
Huerta-Reyes M, Basualdo Mdel C, Abe F, Jimenez-Estrada M, Soler C, Reyes-Chilpa R. HIV-1抑制化合物属brasiliense叶子。生物医药通报2004;27(9):1471-5。
- 8.
García-Zebadúa JC, Magos-Guerrero GA, Mumbrú-Massip M, Estrada-Muñoz E, Contreras-Barrios MA, Huerta-Reyes M, Campos-Lara MG, Jiménez-Estrada M, Reyes-Chilpa R. HIV-1逆转录酶的抑制作用,毒理学和化学特征属brasiliense墨西哥恰帕斯的提取物。Fitoterapia。2011;82(7):1027 - 34。
- 9.
黄素芬MT, Rizzo JD, Khilevich A, Kucherenko A, Sheinkman AK, Vilaychack V, Lin L, Chen W, Greenwood EM, Pengsuparp T,等。(+/−)-卡兰内酯A及其对映体的合成、色谱解析及抗人类免疫缺陷病毒活性中华医学化学杂志,1996;39(6):1303-13。
- 10.
Flavin MT,徐志强,Rizzo JD, Kucherenko A, Khilevich A, Sheinkman AK, Wilaychack V, Lin L, Chen W, Boulanger WA。(±)-卡兰内酯a及其中间体的制备方法。在。谷歌的专利;1996.
- 11.
Cragg GM, Newman DJ。植物是抗癌和抗hiv药物的来源。中国生物医学工程学报,2003;43(2):344 - 344。
- 12.
Kashman Y, Gustafson KR, Fuller RW, Cardellina 2nd JH, McMahon JB, currenstmj, Buckheit Jr RW, Hughes SH, Cragg GM, Boyd先生。属lanigerum.中华医学化学杂志,1992;35(15):2735-43。
- 13.
Bernabé-Antonio A, Estrada-Zúñiga ME, Buendía-González L, rees - chilpa R, Chávez-Ávila VM, Cruz-Sosa F.抗hiv -1卡兰内酯在植物愈伤组织培养中的产生属brasiliense(综眼)。植物科学进展。2010;30(1):344 - 344。
- 14.
西斯内罗斯-托雷斯·D. Análisis活动计算和栽培方法células en suspensión de属brasiliense康贝斯evaluación德拉活动性消炎。México: Universidad Autónoma metropolis - Iztapalapa;2015.
- 15.
白晓宁,梁伟,程杰,马立强,刘永波,史桂林,王艳娜,顾建超。伞形花序酮对植物病原真菌的抑制作用及其抑菌机制。在:朱娥,Sambath S,编辑。信息技术与农业工程“,”柏林海德堡:施普林格;2012.p . 693 - 702。
- 16.
Montagner C, De Souza SM, Groposoa C, Delle Monache F, Smania EF, Smania Jr A.香豆素类抗真菌活性。生物科学杂志。2008; 63(1 - 2): 8。
- 17.
张刚,王勇,徐辉,吴刚,赵松狼毒种群.安徽农学学报。2000;27(4):345-7。
- 18.
杨晓文,徐斌,冉晓霞,王仁强,吴娟,崔俊杰。11种香豆素类化合物对人膀胱癌E-J细胞生长的抑制作用在体外.中西医结合杂志。2007; 5(1): 56 -。
- 19.
舒尔茨MH,泽比诺DR,维格隆M,伯尼e新创RNA-seq组装跨越表达水平的动态范围。生物信息学。2012;28(8):1086 - 92。
- 20.
Zerbino DR, Birney E. Velvet:使用de Bruijn图的从头短读组装算法。基因组学报,2008;18(5):821-9。
- 21.
Ibarra-Laclette E, Albert VA, Herrera-Estrella A, Herrera-Estrella L.是食肉植物核基因组的GC偏倚Utricularia由基于ros的突变和偏倚基因转换驱动?植物信号行为研究。2011;6(11):1631-4。
- 22.
Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ。gapping BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。中国生物医学工程学报,1997;25(17):3389-402。
- 23.
叶静,方磊,郑辉,张勇,陈杰,张震,王杰,李松,李锐,Bolund L,等。WEGO:用于绘制GO注释的网络工具。核酸研究,2006;34:W293-7。
- 24.
后藤凯格:京都基因基因组百科全书。核酸研究,2000;28(1):27-30。
- 25.
Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B.用RNA-Seq绘制和量化哺乳动物转录组。光子学报。2008;5(7):621-8。
- 26.
李文杰,李文杰,李文杰,等。cDNA文库中基因表达的比较。基因组学报2000;10(12):2055-61。
- 27.
Fiebig A, Mayfield JA, Miley NL, Chau S, Fischer RL, Preuss D.与CUT1相同的CER6基因的改变对花粉和茎表面长链脂质含量的影响存在差异。《植物科学》2000;12(10):2001-8。
- 28.
Joubes J, Raffaele S, Bourdenx B, Garcia C, Laroche-Traineau J, Moreau P, Domergue F, Lessire R. VLCFA延长酶基因家族拟南芥:系统发育分析、三维建模和表达分析。植物分子生物学杂志,2008;37(5):344 - 344。
- 29.
Kannangara R, Branigan C, Liu Y, Penfield T, Rao V, Mouille G, Hofte H, Pauly M, Riechmann JL, Broun P.转录因子WIN1/SHN1调控Cutin生物合成拟南芥.植物学报。2007;19(4):1278-94。
- 30.
Blée E, Schuber F.角质单体的生物合成:脂氧合酶/过氧合酶途径的参与。植物学报,1993;4(1):113-23。
- 31.
Bell E, Creelman RA, Mullet JE。一个叶绿体脂氧合酶是必需的创伤诱导茉莉酸积累拟南芥.中国科学院学报(自然科学版),2001;26(3):344 - 344。
- 32.
麦克米伦CP,曼斯菲尔德SD,斯塔彻斯基ZH,埃文斯R,南顿SG。束素样阿拉伯半乳聚糖蛋白:茎生物力学和细胞壁结构的专门化拟南芥而且桉树.植物学报,2010;36(4):369 - 369。
- 33.
范霍梅,李志强,李志强,李志强。木质素的生物合成与结构研究。植物科学进展。2010;29(3):344 - 344。
- 34.
特纳SR,萨默维尔CR.崩溃木质部表型拟南芥鉴定次级细胞壁中缺乏纤维素沉积的突变体。植物学报。1997;9(5):689-701。
- 35.
黄奕,古德曼HM。一个拟南芥根特异性激酶同源物由脱水、ABA和NaCl诱导。植物学报,1995;8(1):37-43。
- 36.
Dewick点。莽草酸途径:芳香族氨基酸和苯丙酸。在:药用天然产品。奇切斯特:约翰·威利父子有限公司;2009.p . 137 - 86。
- 37.
玄文伟,尹永华,金志勇,金绍生。真菌与植物苯丙氨酸解氨酶。Mycobiology。2011;(4):257 - 65。
- 38.
伯尼E,卡普M,德宾r。基因组学报,2004;14(5):988-95。
- 39.
Gouy M, Guindon S, Gascuel O. SeaView版本4:用于序列比对和系统发育树构建的多平台图形用户界面。分子生物学杂志,2010;27(2):221-4。
- 40.
Bateman A, Coin L, Durbin R, Finn RD, Hollich V, griffith - jones S, Khanna A, Marshall M, Moxon S, Sonnhammer EL,等。Pfam蛋白家族数据库。Nucleic Acids Res. 2004;32(数据库issue): D138-41。
- 41.
李国强,李国强,李国强,等。植物苯丙氨酸解氨酶基因家族的研究进展拟南芥.植物分子生物学,1995;27(2):327-38。
- 42.
水谷,高田,佐藤,等。肉桂酸4-羟基化酶cDNA和克隆的分离拟南芥及其在植物中的表达方式。植物科学进展。1997;29(3):344 - 344。
- 43.
王倩,王志刚,王志刚,王志刚。4-香豆酸辅酶A的研究进展拟南芥代表被子植物中两个进化上不同的类。植物学报,1999;19(1):9-20。
- 44.
Hamberger B, Hahlbrock K. 4-香豆酸:CoA连接酶基因家族拟南芥包括一种罕见的、活化酸盐的同工酶和三种常见的同工酶。中国科学(d辑),2004;26(3):344 - 344。
- 45.
Vialart G, Hehn A, Olry A, Ito K, Krieger C, Larbat R, Paris C, Shimizu B,杉本Y, Mizutani M,等。一种2-羟戊二酸依赖的双加氧酶太阳之graveolensL.具有对香豆油酰辅酶a2’-羟化酶活性(c2’h):植物伞形花序酮合成过程中缺失的一步。植物学报,2012;29(3):344 - 344。
- 46.
Gestetner B, Conn EE。通过叶绿体对反式肉桂酸的2-羟基化反应Melilotus阿尔巴白玉兰。生物化学学报。2004;26(2):344 - 344。
- 47.
波尔顿JE,麦克雷DE,康EE。参与香豆素生物合成的两种酶的细胞内定位Melilotus阿尔巴.植物科学进展。1980;26(2):379 - 379。
- 48.
罗宾斯议员。植物次生代谢的生物化学。植物年度评论,第2卷。迈克尔·温克编辑。中国植物病虫害杂志,2000;26(5):487。
- 49.
Bourgaud F, Hehn A, Larbat R, Doerper S, Gontier E, Kellner S, Matern U.植物中香豆素的生物合成:细胞色素P450酶的主要途径仍未阐明。植物化学,2006;5(2):293-308。
- 50.
Harborne简森-巴顿。天然香豆素:发生,化学和生物化学(书)。植物细胞环境。1982;5(6):435 - 36。
- 51.
马特恩U,施特拉瑟H,温多夫H,哈默斯基D.第一章-香豆素和呋喃香豆素。入:Vasil FCK,编辑。植物细胞培养中的植物化学物质。牛津大学:学术;1988.3-21页。
- 52.
引言:次生代谢产物的生物化学、生理和生态功能。见:植物年刊第40卷:植物次生代谢的生物化学。奇切斯特:著名;2010.p -。
- 53.
王志强,王志强,王志强,等。植物细胞色素P450。植物化学,1994;37(6):1491 - 506。
- 54.
Halkier英航。细胞色素P450酶的催化反应及结构/功能关系。植物化学,1996;43(1):21。
- 55.
Larbat R, Kellner S, Specker S, Hehn A, Gontier E, Hans J, Bourgaud F, Matern U.补骨脂素合成酶的分子克隆和功能表征,第一个参与呋喃香豆素生物合成的单加氧酶。中国生物化学杂志,2007;28(1):542-54。
- 56.
杨晓明,刘志刚,杨晓明,杨晓明,杨晓明,Jugdé H .角呋喃香豆素生物合成中P450单加氧酶CYP71AJ4的分离与功能研究。中国生物医学工程学报,2009;26(4):457 - 457。
- 57.
Khan A, Kunesch G, Chuilon S, Ravisé A.一种新的植物抗毒素——柑橘黄叶素的结构和生物活性。水果。1985;40(12):807 - 11。
- 58.
贝尔RC,艾维GW,欧特利EH。从普通草药欧芹中提取的线性呋喃香豆素和呋喃香豆素。植物化学。1994;(4):869 - 72。
- 59.
李志强,李志强,李志强,等。柑桔根中芝麻素的分离与鉴定。植物化学,1969;8(7):1315 - 6。
- 60.
Borges F, Roleira F, Milhazes N, Santana L, Uriarte E.药物化学中的简单香豆素及其类似物:发生,合成和生物活性。中国生物医学工程学报,2005;12(8):887-916。
- 61.
Poulsen S-A, Davis R.抑制碳酸酐酶的天然产物。进:Frost SC, McKenna R,编辑。碳酸酐酶:机制,调控,与疾病的联系和工业应用。荷兰:施普林格;2014.325 - 47页。
- 62.
李志刚,李志刚。植物与植食性昆虫的化学表型匹配。中国科学院学报(自然科学版),1998;26(3):344 - 344。
- 63.
Karamat F, Olry A, Munakata R, Koeduka T, Sugiyama A, Paris C, Hehn A, Bourgaud F, Yazaki K.香豆素特异性戊烯基转移酶催化香菜中呋喃香豆素形成的关键生物合成反应。植物学报,2014;26(4):344 - 344。
- 64.
黄志刚,张志刚,张志刚。呋喃香豆素的生物合成Ammi majusL.柏嘉酚的克隆O甲基转移酶。欧洲生化杂志/ FEBS。2004年,271(5):932 - 40。
- 65.
Larbat R.贡献à l ' étude des P450 impliqués dans la biosynthèse des furrocoumarines。法国:Unité Mixte de Recherche Agronomie et environment (UMR INPL-INRA);2006.
- 66.
Caspi R, Foerster H, Fulcher CA, Kaipa P, Krummenacker M, Latendresse M, Paley S, Rhee SY, Shearer AG, Tissier C,等。代谢途径和酶的MetaCyc数据库和途径/基因组数据库的BioCyc集合。核酸Res. 2008;36附录1:D623-31。
- 67.
Langmead B, Trapnell C, Pop M, Salzberg SL.人类基因组短DNA序列的超快和内存高效对齐。中国生物医学工程学报。2009;10(3):R25。
- 68.
Li B, Dewey C. RSEM:从RNA-Seq数据中精确的转录物定量,有或没有参考基因组。BMC生物信息学。2011; 12(1): 323。
- 69.
埃德加钢筋混凝土。MUSCLE:多序列比对,精度高,通量大。中国生物工程学报,2004;32(5):1792-7。
- 70.
Guindon S, Gascuel O.一种通过最大似然估计大系统发育的简单、快速和准确的算法。中国生物医学工程学报。2003;52(5):696-704。
- 71.
Le SQ, Gascuel O.一种改进的通用氨基酸替代基质。分子生物学杂志,2008;25(7):1307-20。
- 72.
Anisimova M, Gascuel O.分支的近似似然比检验:一个快速,准确和强大的替代方案。中国生物医学工程学报。2006;55(4):539-52。
- 73.
郭文杰,李文杰,李文杰,等。基于网络的蛋白质结构同源性建模环境。生物信息学。2006;22(2):195 - 201。
- 74.
Kiefer F, Arnold K, Kunzli M, Bordoli L, Schwede T.瑞士模型资源库和相关资源。核酸决议2009;37(数据库issue): D387-92。
- 75.
Guex N, Peitsch MC. SWISS-MODEL和Swiss-PdbViewer:用于比较蛋白质建模的环境。电泳。1997;18(15):2714 - 23所示。
- 76.
Nischal L, Mohsin M, Khan I, Kardam H, Wadhwa A, Abrol YP, Iqbal M, Ahmad A.水稻耐低氮基因型相关microrna的鉴定与比较分析。PLoS ONE。2012; 7 (12), e50261。
- 77.
王志强,王志强,王志强。药用植物生物活性分子的化学多样性及其治疗潜力。入:Ramawat KG,编辑。草药:民族医学到现代医学。柏林海德堡:施普林格;2009.7-32页。
致谢
我们要感谢Victor A. Albert的积极和相关的评论,这些评论提高了本手稿的质量。我们也感谢Victor对语言编辑的支持。最后,我们感谢Aldo Segura-Cabrera、Ana-Luisa Kiel-Martínez和Ofelia Ferrera-Rodríguez的积极讨论和宝贵建议。
资金
这项工作得到了CONACyT (CONACyT)的支持,资助223323 (EIL)。
数据和材料的可用性
所有支持性数据都包含在文章中或附加文件中。
作者的贡献
HBG-R撰写论文,EI-L、HBG-R、FC-S、AB-A、AG-A、JLO-R、AA-S、EV和EI-L收集的数据做出了重要贡献。HBG-R、AG-A、JLO-R、AA-S、EV、EI-L分析数据。eil构思并领导了这项研究。所有作者都阅读并批准了最终的手稿。
相互竞争的利益
作者宣称他们之间没有利益冲突。
发表同意书
不适用。
伦理批准并同意参与
不适用。
作者信息
从属关系
相应的作者
附加文件
附加文件1:
c . brasilienseunigenes集。(TXT 6810kb)
附加文件2:表S1。
注释的c . brasilienseunigenes。(xlsx20582 kb)
附加文件3:图S1-S9。
图S1:基因本体分类c . brasilienseunigenes。图S2:差异表达基因的生物学过程。图S3:重建c . brasiliense代谢网络从葡萄糖到反式肉桂酸。图S4:系统发育关系,主要蛋白质结构和同源性百分比反式-肉桂酸4-羟化酶蛋白。图S5: 4-香豆酸辅酶a连接酶(4CL)的结构和系统发育分析c . brasiliense.图S6: 4-香豆酚基2 ' -羟基化酶的排列c . brasiliense.图S7: Wagner-Meerwein重排示意图。图S8:c . brasilienseprenyltransferases。图S9:所选的系统发育树c . brasiliense细胞色素P450。(PPTX 1219 kb)
附加文件4:表S2-S9。
表S2:功能分类c . brasiliense基于拟南芥GO注释的unigenes集。表S3:的表达式轮廓矩阵c . brasilienseunigenes。表S4:植物器官样本(叶、茎、根)中优先表达的基因c . brasiliense.表S5:优先表达基因的功能分类。表S6:与特定GO术语相关的优先表达基因数量。表S7:特定器官中生物过程的比例过高。表S8:排列的细胞色素P450之间的百分比恒等式矩阵。表S9: RT-qPCR检测中使用的引物。(xlsx16223 kb)
附加文件5:
编码序列在pal样之间对齐c . brasilienseunigenes和拟南芥PAL1蛋白。(TXT 8kb)
附加文件6:
编码序列对齐c . brasiliense和拟南芥PAL蛋白。(TXT 16kb)
附加文件7:
编码序列之间对齐c4h样c . brasiliense和拟南芥C4H蛋白。(TXT 6kb)
附加文件8:
编码序列对齐c . brasiliense拟南芥4CL蛋白。(TXT 18kb)
附加文件9:
单基因UN02124之间的编码序列比对c . brasiliense而且太阳之graveolensCYP98A22蛋白质。(TXT 3kb)
附加文件10:
来自pt样unigenes之间的编码序列比对c . brasiliense而且Petroselinum crispumPT蛋白质。(TXT 9kb)
附加文件11:
编码序列在cyplike unigenes之间的对齐c . brasiliense来自不同植物种类的CYP蛋白参与呋喃香豆素的生物合成。(TXT 74kb)
附加文件12:
这些unigenes之间的编码序列对齐c . brasiliense及其对应的拟南芥同源物用于RT-qPCR分析。(TXT 29 kb)
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
Gomez-Robledo, HB。,Cruz-Sosa, F., Bernabé-Antonio, A.et al。通过转录组测序鉴定甘兰内酯生物合成相关的候选基因属brasiliense(Calophyllaceae)。BMC植物生物学16日,177(2016)。https://doi.org/10.1186/s12870-016-0862-9
收到了:
接受:
发表:
关键字
- 基因本体论
- 苯丙氨酸解氨酶
- 酶委员会
- Umbelliferone
- KEGG Orthology