摘要
背景
栽培亚麻(亚麻属植物usitatissimumL.)广泛用于纺织、食品、化工和医药产品的生产。然而,各种胁迫会降低亚麻产量。寻找与胁迫反应有关的基因,是选育适应性品种的必要条件。土壤中营养元素浓度的不平衡降低了亚麻产量,并导致一些亚麻品系的遗传变化。Linum插入序列1 (LIS-1)的出现是目前研究最多的修饰。然而,LIS-1的功能尚不清楚。
结果
利用Illumina平台对正常(N)、缺磷(P)和养分过剩(NPK)条件下生长的亚麻植株转录组进行了高通量测序。进行转录组组装,N、P和NPK测序文库分别鉴定出34924、33797和33698个唯一转录本。在我们的测序数据中,我们没有发现任何来自LIS-1的mRNA。高通量测序数据的分析使我们能够识别在营养不平衡下具有潜在差异表达的基因。为了进一步研究,我们选择了15个基因,并在31株亚麻植物的扩展样本中进行了表达水平的评估。在P和NPK条件下,编码WRKY和JAZ蛋白家族的基因表达发生了显著变化。此外,WRKY家族基因的改变因LIS-1在亚麻植物基因组中的存在而不同。此外,我们发现轻微的和LIS-1独立的mRNA水平变化KRP2而且ING1与LIS-1相邻的基因在营养压力下。
结论
基于高通量测序和qPCR数据,在缺磷和营养过剩条件下生长的亚麻植株中鉴定出差异表达基因。结果表明,WRKY和JAS基因家族参与了亚麻对土壤养分不平衡的响应。此外,在我们的转录组测序数据中,我们还没有发现任何可能来源于LIS-1的mRNA。LIS-1侧翼基因表达,ING1而且KRP2,建议不是营养应激引起的。研究结果为研究亚麻土壤胁迫响应及LIS-1在胁迫响应过程中的作用提供了新的思路。
背景
栽培亚麻(亚麻属植物usitatissimumL.)广泛用于纺织、化工、食品和医药产品的生产。然而,各种生物和非生物胁迫会降低亚麻油和纤维的产量。因此,栽培对不利环境有抵抗力l . usitatissimum品种是获得高产稳定产量所必需的。了解亚麻的抗逆性机理是培育耐胁迫品种的必要条件。研究与胁迫反应相关的基因对亚麻耐受性遗传和基因组学辅助育种具有重要意义。土壤胁迫,包括营养不足或过度,极大地限制了亚麻的生产[1],尤其值得研究。此外,土壤养分元素浓度的不平衡导致一些亚麻品系的遗传表型和基因型发生变化,这些品系被称为塑性[2- - - - - -9].可塑系的后代稳定地继承了这些变化,称为基因营养体[7].最著名的亚麻塑料系是“Stormont cirrus”,研究最多的基因修饰是Linum插入序列1 (LIS-1)。LIS-1是一个5.7 kb的序列,在营养缺乏的情况下出现在亚麻基因组的一个特定位点,但其功能尚不清楚[2- - - - - -4,10,11].
在本研究中,我们研究了土壤养分胁迫对亚麻植物的影响,并比较了有和没有LIS-1和非可塑性品种“白求恩”的“Stormont cirrus”对养分缺乏或过剩的反应。我们利用高通量测序和定量RCR (qPCR)技术对亚麻植物的基因表达进行了评价,从而鉴定出可能与土壤养分不良反应有关的基因。
方法
植物材料
l . usitatissimum如前所述,在正常(N)、缺磷(P)和营养过剩(NPK)条件下,种植含有或不含LIS-1的“白求恩”品种和“Stormont Cirrus”品系的植株[11].植物材料来自9株“白求恩”(3株在N、3 - P和3 - NPK条件下生长),10株稳定存在LIS-1的“斯托蒙特卷云”(3 - N、3 - P和4 - NPK), 12株稳定不存在LIS-1的“斯托蒙特卷云”(4 - N、4 - P、4 - NPK)。收集生长6周后的单株上部叶片,立即在液氮中冷冻。植物样品储存在−70°C。总DNA采用氯仿:异戊醇提取,如前所述[12].使用RNA MicroPrep试剂盒(Zymo Research, USA)提取总RNA。采用Chen等引物PCR检测单株的LIS-1状态[3.]和琼脂糖凝胶电泳,如前所述[11].
转录组测序
CDNA文库制备使用TruSeq RNA SamplePrep (Illumina, USA)。用聚t寡聚磁珠分离含聚a的mRNA片段。将混合样本用于在N、P和NPK条件下生长的“Stormont Cirrus”品系的三个文库。使用Agilent 2100 Bioanalyzer (Agilent Technologies, USA)对文库质量进行评估,然后在Illumina GAIIx平台上对文库进行测序。使用trimmomatic对原始读取进行修剪和过滤[13].转录组的组装使用SOAPdenovo-Trans转录组组装程序进行。将组装好的转录本映射到l . usitatissimum“参考”JGI基因组门户转录组数据库与blastn (PhytozomeV11: lusitatissimum_200_v1 .0. transcriptome .fa.gz;[14])。如果几个组合的转录本被映射到一个“参考”转录本,那么我们将它们合并为一个转录本,并填补“参考”序列的空白。接下来,我们将注释从“参考”数据集转移到组装的文本。为了进一步分析,只使用了使组装更可靠的映射转录本。
为了评估差异表达,用bowti2 [15],每个转录本值的读计数用rsem计算[16].使用读取总数执行库规范化。比较N, P和NPK文库的mRNA水平褶皱变化(NPK中的归一化读计数或P/ N中的归一化读计数)和假定值(χ2测试)参数[17].
利用基因本体数据,使用Goseq对折叠变化最高的基因进行基因集富集分析(http://bioconductor.org/packages/release/bioc/html/goseq.html).
为了鉴定来自is -1或其5 '和3 '侧翼序列的mrna,使用blast方法将获得的Illumina转录组reads与GenBank的AF104351.1、AF300798.1和AF300797.1进行比对。18].
QPCR分析
采用7500 Real-Time PCR系统(Applied Biosystems, USA),在20 μl反应混合物中进行PCR反应,反应混合物中含有1× 2- frt PCR混合物(Amplisens, Russia)、250 nM dNTPs混合物(Fermentas, Lithuania)、350 nM正向和反向引物、2u TaqF聚合酶(Amplisens)、1× EvaGreen (Biotium, USA)和cDNA。采用以下扩增程序:95°C 15 min;50个循环,95°C持续15秒,60 - 62°C持续60秒。在N(10株)、P(10株)和NPK(11株)条件下,研究了9株白鹤亚麻品种(Bethune)、10株带LIS-1的“Stormont Cirrus”和12株不带LIS-1的亚麻。用NCBI Primer Blast筛选的引物对15个基因的表达进行评价[19]最好是用扩增子跨外显子-外显子结(见附加文件1:表S1)。qPCR数据分析,参考基因ETIF3H而且ETIF3E[20.]被使用。所有计算均使用程序ATG (一个分析的T转录的Gen) [21].的ΔCteff数值与表达量成正比,根据公式[11]:
在哪里Ct,重复平均阈值周期;E,每对引物的反应效率。每个qPCR反应重复3次。扩增效率为95%或更高,所有引物对的阈值周期从23到34不等(附加文件)1:表S1)。
采用Kruskal-Wallis和Mann-Whitney检验评估表达改变的统计学意义。P≤0.05为有统计学意义。
结果
亚麻植物N、P和NPK文库的高通量测序
从正常(N)、缺磷(P)和养分过剩(NPK)条件下生长的亚麻植株构建了3个转录组文库。使用GAIIx Illumina平台的高通量测序在N、P和NPK条件下分别产生了106、13.6和1860万个读取(75个核苷酸长度)(序列读取档案- SRP083007)。这三个文库的统计数据非常相似:GC含量为47 - 48%,平均每次阅读质量约为38,PCR序列重复水平在15 - 17%之间。映射到引用之后l . usitatissimumN、P和NPK文库分别鉴定出34924、33797和33698个独特转录本。最大转录长度为14619个核苷酸。
基于高通量测序数据的基因表达分析
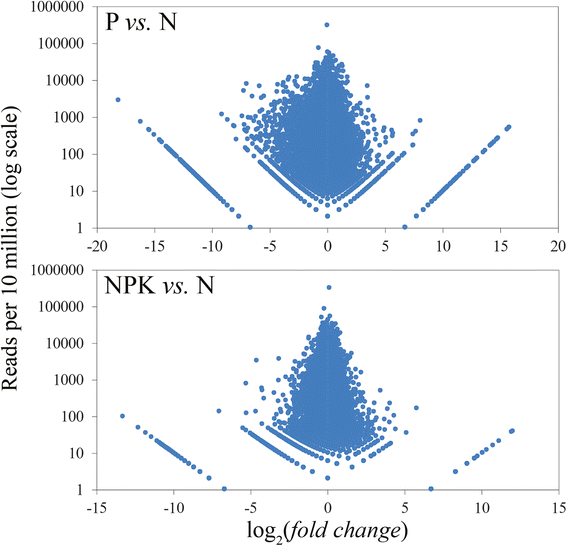
确定每1000万次阅读中与每个汇编的转录本对齐的阅读数并进行归一化。为了表达分析结果的可视化,我们构建了图表(图1)。1).对差异表达前30位的基因进行基因本体富集分析。P和NPK条件下,前30个基因的大部分表达量均下降。在缺磷条件下,亚麻植株的生长主要是转录因子(p= 2.4 * 10−7).因此,转录因子可能在亚麻适应营养胁迫中发挥重要作用,这与转录因子在植物耐受非生物胁迫中的作用的数据是一致的[22].在P条件下,参与组蛋白乙酰转移酶活性的基因表达改变(p= 2.9 * 10−5)、序列特异性DNA结合(p= 8.0 * 10−5)、天冬酰胺生物合成过程(p= 2.1 * 10−3)、蛋白质结合过程(p= 4.8 * 10−3)、锌离子结合(p= 0.03),甲基转移酶活性(p= 0.07)。在NPK条件下,参与组蛋白乙酰转移酶活性的基因(p= 4.6 * 10−4)、铁离子结合(p= 4.6 * 10−4),转录辅因子活性(p= 4.6 * 10−4)、氧化还原酶活性(p= 2.6 * 10−3)、蛋白质结合(p= 3.4 * 10−3)、离子通道活性(p= 0.02),等.
LIS-1衍生的rna
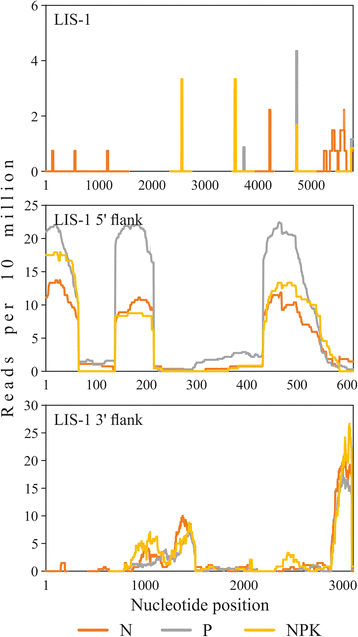
为了鉴定来自LIS-1的mrna,其5 '和3 '侧翼序列的reads分别与GenBank上的AF104351.1、AF300797.1和AF300798.1进行比对。在LIS-1的5’和3’侧翼序列中,发现了区域的一致性,这些区域对应于生长抑制子1 (ING1)和kip相关周期蛋白依赖性激酶抑制剂2 (KRP2)基因(图;2).基于我们的高通量测序数据的表达分析显示,在表达水平上没有差异KRP2N、P和NPK条件下的基因。的表达略有增加ING1P条件下的基因。对于LIS-1,我们只鉴定出少数读k-mers,它们与序列的短区域重合。此外,在有LIS-1和没有LIS-1的样本中都发现了匹配的k-mers。因此,我们得出的结论是,没有从LIS-1中得到read。
营养不平衡条件下亚麻基因表达的QPCR分析
我们使用高通量测序数据对三个在不同营养条件下生长的植物样本进行了初步评估。为了可靠地评估表达变化,我们在扩展样本(10株亚麻植物在N、11 - P和10 - NPK条件下生长)中进行了qPCR分析。在高通量测序数据的基础上,选择了20个在营养胁迫下上调或下调的基因。设计并测试了20对qPCR引物,其中只有13对引物具有高效和特异性。采用QPCR方法对带有和不带有LIS-1的“Stormont cirrus”株系和品种“Bethune”进行分析,分析编码WRKY dna结合蛋白70 (WRKY70)的基因的表达情况;WRKY dna结合蛋白33 (WRKY33);WRKY dna结合蛋白40 (WRKY40);JAZ8/TIFY5A茉莉酸津结构域蛋白8;JAS1, JAZ10, TIFY9茉莉酸zimm结构域蛋白10 (JAZ10);推定核酸酶HARBI1 (HARBI1); S-adenosyl-L-methionine-dependent methyltransferase (SAM MTase); Neutral invertase (A/N-Invs); CCR4-associated factor 1 homolog 11 (CAF1-11); BTB and TAZ domain protein 4 (BT4); MLP-like protein 423 (MLP423); Ethylene-responsive transcription factor ERF (ERF); Myb domain protein 41 (MYB41). Besides, expression ofING1而且KRP2对位于LIS-1侧翼序列的基因进行了评估。
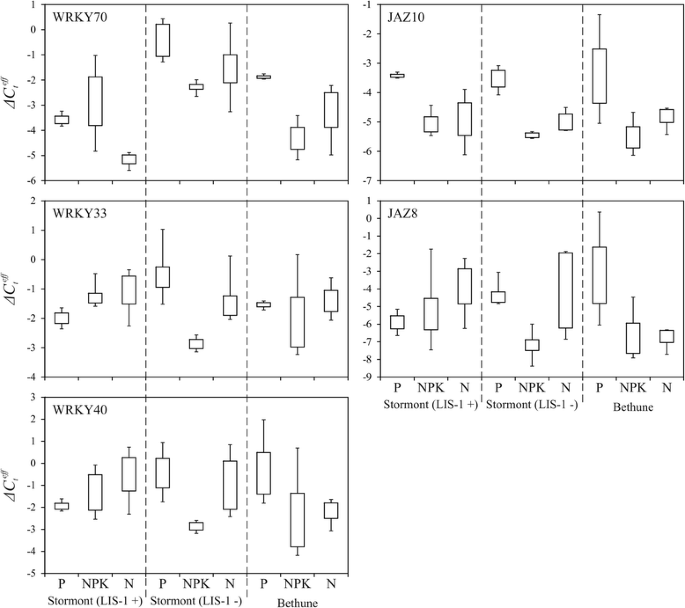
在添加和不添加LIS-1的“Stormont cirrus”植物中,以及在不同营养条件下生长的“Bethune”植物中,编码WRKY dna结合蛋白家族和编码JAZ茉莉酸- zimm -结构域蛋白家族的基因表达发生了显著变化。3.).
JAZ基因家族在不同营养条件下的反应在大多数组样品中是相似的——P条件下比NPK条件下表达增加。中值的\(\varDelta {C}_t^{eff} \)为JAZ10“Stormont卷云”与LIS-1的P和NPK分别为−3.4和−5.1;无LIS-1的“Stormont卷云”P和NPK分别为- 3.5和- 5.5;−《白求恩》中P为3.7,NPK为5.6。中值的\(\varDelta {C}_t^{eff} \)为JAZ8is -1的“Stormont卷云”P和NPK分别为- 5.9和- 5.7;无LIS-1的“Stormont卷云”P和NPK分别为4.6和7.2;−《白求恩》中P为3.7,NPK为7.4。JAZ家族蛋白是茉莉酸的抑制因子,调控植物生长发育和胁迫反应的许多方面[23].我们的结果表明,JAZ家族蛋白参与了亚麻营养缺乏和过度的反应。
与NPK相比,P处理也增加了WRKY基因家族的表达,但仅在不含LIS-1的基因型中。中值的\(\varDelta {C}_t^{eff} \)为WRKY70在没有LIS-1的‘Stormont卷云’中P和NPK分别为- 0.4和- 2.3;在《白求恩》中P为1.9,NPK为4.3。中值的\(\varDelta {C}_t^{eff} \)为WRKY33在没有LIS-1的‘Stormont卷云’中P和NPK分别为- 0.7和- 2.9;《白求恩》中P为1.5,NPK为2.7。中值的\(\varDelta {C}_t^{eff} \)为WRKY40在没有LIS-1的‘Stormont卷云’中P和NPK分别为- 0.5和- 2.9;在《白求恩》中P为1.0,NPK为3.4。对于带有LIS-1的“斯托蒙特卷云”,趋势相反:中位数为\(\varDelta {C}_t^{eff} \)为WRKY70P处理- 3.6,NPK处理- 2.8;为WRKY33 -P处理-2.0,NPK处理- 1.4;为WRKY40,P为2.0,NPK为1.3。WRKY家族的蛋白质是转录因子,参与植物的许多过程,包括胁迫反应[24].根据我们的数据,我们可以推测该家族的基因参与了亚麻对营养不平衡的反应。值得注意的是,WRKY家族基因的表达改变因LIS-1在亚麻植物基因组中的存在而不同。
对于其他被检查的基因,仅在特定的样本组中显示了统计学上显著的表达改变(附加文件)2:图S1)。在“Stormont cirrus”中,LIS-1改变了编码假定核酸酶HARBI1的基因的表达(中位数为\(\varDelta {C}_t^{eff} \)P为−5.2,NPK为−3.8,N为−2.8),MLP423 (P为−4.2,NPK为−2.9,N为−4.8),ING1 (P为−3.4,NPK为−2.4),A/N- invs (P为−1.5,NPK为0.2),BT4 (P为−4.2,NPK为−3.0);在没有LIS-1 -的‘Stormont cirrus’中,基因编码ING1 (P下−4.2,N下−3.2)、BT4 (P下−4.6,N下−3.1)、KRP2 (P下−1.8,NPK下−2.4,N下−2.0)和ERF (P下−3.4,NPK下−7.2,N下−5.7);在‘Bethune’中,只有MYB41 (P处理下为−1.8,NPK处理下为−3.7,N处理下为−2.6)。编码CAF1-11和SAM MTase的基因,未发现显著的表达改变。由此可见,上述基因在营养不平衡条件下的大部分表达变化都发生在塑性品系“Stormont cirrus”上,而非塑性品系“Bethune”中只有1个基因表达差异。
讨论
LIS-1在应对营养不平衡中的作用
对不同营养条件下生长的亚麻植物的三个cDNA文库进行了高通量测序。转录组组装后,分别鉴定出34924、33797和33698个N、P和NPK文库的唯一转录本。对于LIS-1衍生reads的识别,我们选择了Blast,因为它对错配和长插入/缺失的耐受性,因为不能排除基因型之间的LIS-1高多态性。因此,在我们的测序数据中,我们没有发现任何来自LIS-1的mRNA。此前,有报道称LIS-1序列不包含大的开放阅读帧[4].在我们之前的研究中,我们也没有发现任何LIS-1转录的microRNA [11].在目前的工作中,我们揭示了统计学上显著的表达改变KRP2而且ING1在营养不平衡的条件下,与LIS-1相邻的基因。但变化程度不高,且未观察到LIS-1状态与表达改变之间的相关性。为ING1在P条件下,“Stormont cirrus”(is -1 -中位)基因的表达均略有增加\(\varDelta {C}_t^{eff} \)P = 3.4, N = 2.8;没有LIS-1中位数的“斯托蒙特卷云”\(\varDelta {C}_t^{eff} \)−P为4.2,N为3.2;白求恩——中位数\(\varDelta {C}_t^{eff} \)P为−3.8,n为−2.9KRP2基因在不同营养条件下略有差异(中位数\(\varDelta {C}_t^{eff} \)在所有组样本中均在1.4至2.4之间)。因此,我们可以得出结论,LIS-1的存在很可能不会导致其侧翼基因在营养胁迫下的表达改变。
P和NPK条件下基因表达变化的评价
在我们的研究中,我们使用了从具有稳定的LIS-1状态的植物中获得的塑料系“Stormont cirrus”种子:有LIS-1和没有LIS-1。此外,我们使用了非塑性品种“白求恩”,其中从未出现过LIS-1。在N、P和NPK条件下种植亚麻植株,通过qPCR分析,评估了31株扩展样本中基于高通量测序数据选择的基因的表达。JAZ家族基因也有相似的表达改变(JAZ8而且JAZ10大多数组在N、P和NPK条件下表达升高,NPK条件下表达降低(只有is -1的‘Stormont cirrus’表达升高)JAZ8P和NPK条件下基本相同)。JAZ家族蛋白是植物激素茉莉酸酯的抑制因子,在植物生长发育和抗逆性中起重要作用[23,25- - - - - -27].此外,泛素化过程与茉莉酸信号的相互作用也被揭示[28].然而,关于JAZ蛋白在应对营养胁迫中的作用知之甚少。在缺磷条件下,芸豆生长25 d后,TIFY家族基因表达存在差异,部分表达被诱导,部分表达被抑制。TIFY家族基因参与菜豆假设对磷饥饿的反应和适应[29].在水稻和鹰嘴豆中,JAZ蛋白参与了营养缺乏反应:在宏微量营养缺乏下表达差异显著;大部分是鹰嘴豆和米饭JAZ基因在缺磷7 d后上调,15 d后下调[30.].在目前的研究中,在缺磷条件下亚麻植株生长6周后,JAZ基因上调,而在营养过剩的情况下,JAZ基因被下调。结果表明,JAZ蛋白参与了亚麻对营养失衡的反应。我们的数据为了解JAZ蛋白在植物非生物胁迫反应中的作用做出了贡献。
在营养不平衡条件下生长的亚麻植株中,WRKY家族基因的表达发生了显著变化(WRKY33,WRKY40,WRKY70).WRKY基因家族是植物中最大的转录因子家族之一,参与多种生物过程,包括对生物和非生物胁迫的响应[31].许多关于WRKY蛋白在不同植物物种中的作用的研究[31,32].土壤营养元素的缺乏或营养元素的不平衡是农业生产的一个严重问题,因此,植物对这种胁迫的反应被广泛研究。结果表明,WRKY家族基因参与了水稻抗磷饥饿的抗性:WRKY家族基因的过表达WRKY74基因对磷饥饿耐受性增加,而品系对磷饥饿耐受性降低WRKY74水平对这种压力很敏感[33].在拟南芥, WRKY42调节磷酸盐稳态:WRKY42过表达系对磷酸盐缺乏更敏感[34].然而,过度表达WRKY45在拟南芥磷酸盐摄取量增加[35].对于亚麻,已知WRKY家族基因仅参与盐碱胁迫响应[36,37].在目前的工作中,我们发现了显着的表达减少WRKY33,WRKY40,WRKY70与P条件下无LIS-1的亚麻基因型(无LIS-1的“Stormont cirrus”和“Bethune”)比较。然而,对于带有LIS-1的“Stormont cirrus”,与P条件相比,NPK条件下WRKY家族基因表达增加或保留。已知具有LIS-1的亚麻小基因营养系比没有LIS-1的亚麻小基因营养系更适应营养缺乏[2].在本研究中,我们发现,在对P条件更适应的“Stormont cirrus”基因型(带有LIS-1的“Stormont cirrus”)中,WRKY家族基因的表达水平高于不适应P条件的“Stormont cirrus”基因型(没有LIS-1的“Stormont cirrus”)。在白求恩品种中,WRKY家族基因的表达变化与不含LIS-1的Stormont cirrus相似。我们认为WRKY家族基因参与了亚麻营养胁迫反应,其过表达是对缺磷品系和品种不适应的一个特征。
结论
寻找基因,这涉及亚麻对营养不平衡的反应,进行了。基于高通量测序和qPCR数据,在缺磷和营养过剩条件下生长的亚麻植株中鉴定出差异表达基因。WRKY和JAS基因家族在所有亚麻植物组中的表达都发生了变化,这使我们可以认为这些基因在亚麻对营养胁迫的反应中发挥了重要作用。此外,WRKY家族基因在对磷酸盐缺乏反应和适应的基因型中表达水平不同,这因LIS-1的存在而不同。此外,在我们的转录组测序数据中,我们还没有发现任何可能来源于LIS-1的mRNA。LIS-1侧翼基因的表达变化ING1而且KRP2与亚麻植物基因组中LIS-1的存在无关,表明这些基因不是营养胁迫诱导的。这些数据为研究亚麻土壤胁迫反应以及LIS-1在这一过程中的作用提供了新的见解。
参考文献
- 1.
Muir AD, Westcott ND。亚麻:亚麻属。伦敦:Taylor & Francis;2003.
- 2.
李志刚,李志刚,李志刚。亚麻植株可塑性基因座的研究。Res Rep Biol, 2012; 3:1-11。
- 3.
陈勇,刘志刚,李志刚。环境诱导的亚麻自适应插入事件。中华分子生物学杂志,2009;1(3):38-47。
- 4.
陈勇,陈志强,李志强,等。环境对亚麻基因营养体插入序列的影响。植物科学进展。2005;26(1):1 - 8。
- 5.
卡利斯CA,查尔顿L.核糖体DNA变化的诱导亚麻。植物科学,1981;20:213-7。
- 6.
亚麻的环境调节。大自然。1958;181:928-9。
- 7.
林楠属植物遗传变化的环境诱导。遗传。1962;17:27 - 61。
- 8.
李志强,李志强,李志强。亚麻遗传营养体诱导的相关核变化。大自然。1966;212:697-9。
- 9.
泰森H,菲尔德斯MA。L和S亚麻基因营养系F1杂种过氧化物酶同工酶的分子量和净电荷。生物化学,1982;20:19 - 27。
- 10.
巴斯塔基NK, Cullis CA.花浸转化亚麻(Linum usitatissimum)以产生高转化率的转基因后代。科学通报,2014(94)。doi:10.3791/52189.
- 11.
Melnikova NV, Dmitriev AA, Belenikin MS, Koroban NV, Speranskaya AS, Krinitsina AA,等。正常和营养胁迫条件下亚麻基因滋养体microrna的鉴定、表达分析和靶标预测中国植物科学,2016;7:399。
- 12.
Melnikova NV, Kudryavtseva AV, Zelenin AV, Lakunina VA, Yurkevich OY, Speranskaya AS等。基于反转录转座子的分子标记在亚麻属遗传多样性分析。生物医学学报(英文版)。2014;
- 13.
Bolger AM, Lohse M, Usadel B. Trimmomatic:一种用于Illumina序列数据的灵活修剪器。生物信息学。2014;30(15):2114 - 20。
- 14.
王震,Hobson N, Galindo L,朱松,石东,McDill J,等。利用短霰弹枪序列reads,对亚麻(Linum usitatissimum)基因组进行了从头组装。植物学报,2012;32(3):461 - 466。
- 15.
朗米德B,萨尔茨堡SL。快速间隙读取对齐领结2。光子学报。2012;9(4):357-9。
- 16.
李B,杜威CN。RSEM:从RNA-Seq数据中精确的转录物定量,有或没有参考基因组。生物信息学。2011;12:323。
- 17.
Melnikova NV, Dmitriev AA, Belenikin MS, Speranskaya AS, Krinitsina AA, Rachinskaia OA,等。中国植物学报,2015;39(2):344 - 344。
- 18.
张志强,张志强,张志强。一种基于贪心算法的DNA序列匹配算法。中国生物医学工程学报。2000;7(1):203-14。
- 19.
叶j, Coulouris G, Zaretskaya I, Cutcutache I, Rozen S, Madden TL. Primer-BLAST:一种设计聚合酶链反应靶特异性引物的工具。BMC生物信息学。2012;13:134。
- 20.
胡晓明,李志强,李志强,等。亚麻定量基因表达规范化内参基因的筛选。植物学报。2010;10:71。
- 21.
Krasnov GS, Oparina NY, Dmitriev AA, Kudryavtseva AV, Anedchenko EA, Kondrat 'eva TT,等。RPN1是肺癌和肾癌定量数据规范化的新参考基因。Mol biology, 2011;45(2): 211-20。
- 22.
王辉,王辉,邵辉,唐霞。利用转录因子提高植物非生物胁迫耐受性的转基因技术研究进展。前沿植物科学,2016;7:67。
- 23.
Santner A, Estelle M. JAZ蛋白将茉莉酸盐感知与转录变化联系起来。植物学报。2007;19(12):3839-42。
- 24.
张艳,王磊。WRKY转录因子超家族:真核生物的起源及在植物中的扩增。BMC进化生物学,2005;5:1。
- 25.
Pauwels L, Goossens a . JAZ蛋白:茉莉酸盐信号级联的关键界面。植物学报。2011;23(9):3089-100。
- 26.
茉莉酸盐和乙烯在非生物胁迫耐受中的不同作用。植物科学进展,2015;20(4):219-29。
- 27.
杨晓东,陈志伟,陈志伟,陈志伟,等。环境胁迫下茉莉酸类植物的信号转导及其在植物体内的作用。植物分子生物学杂志,2016;
- 28.
刘国强,刘国强,刘国强,刘国强,等。JAZ抑制蛋白是茉莉酸信号通路中SCF(COI1)复合物的靶标。大自然。2007;448(7154):661 - 5。
- 29.
Aparicio-Fabre R, Guillen G, Loredo M, Arellano J, Valdes-Lopez O, Ramirez M,等。菜豆(菜豆)PvTIFY在茉莉酸盐和磷缺乏的转录谱反应中编排全局变化。BMC植物生物学2013;13:26。
- 30.
杨晓明,杨晓明,杨晓明,杨晓明,杨晓明。水稻和鹰嘴豆JAZ抑制因子在水稻和鹰嘴豆营养缺乏反应中的作用。前沿植物科学,2015;6:975。
- 31.
WRKY转录因子:植物中的多面手。植物信号与行为研究。2014;29(2):374 - 379。
- 32.
陈玲,宋勇,李松,张玲,邹超,于东。WRKY转录因子在植物非生物胁迫中的作用。生物化学学报,2012;1819(2):120-8。
- 33.
戴旭,王勇,张文辉。WRKY转录因子OsWRKY74调控水稻抗磷饥饿能力。中国生物医学工程学报,2016;36(3):344 - 344。
- 34.
苏涛,徐强,张富峰,陈勇,李丽强,吴文华,等。WRKY42通过调节拟南芥中磷酸盐的转运和获取来调节磷酸盐稳态。中国生物医学工程学报。2015;37(4):379 - 379。
- 35.
王辉,徐强,孔玉华,陈勇,段建勇,吴文华,等。拟南芥WRKY45转录因子激活磷酸盐TRANSPORTER1;1的表达以响应磷酸盐饥饿。中国生物医学工程学报,2014;29(4):344 - 344。
- 36.
于勇,吴刚,袁浩,程林,赵东,黄伟,等。生理盐水、碱性和盐碱胁迫下亚麻(Linum usitatissimum) mirna和靶标的鉴定和鉴定。中国生物医学工程学报。2016;16(1):124。
- 37.
于勇,黄伟,陈宏,吴刚,袁宏,宋旭,等。亚麻(Linum usitatissimum L.)盐碱胁迫差异表达基因的数字表达鉴定。基因。2014;549(1):113 - 22所示。
致谢
作者感谢罗蒙诺索夫莫斯科国立大学在植物种植方面的帮助,以及全俄亚麻研究所在种子选择和提供方面的帮助。这项工作是利用恩格尔哈特分子生物学研究所“基因组”中心的设备进行的。
声明
本文已作为BMC植物生物学第16卷增刊3的一部分发表,2016:选自BGRS\SB-2016:植物生物学。该补充的全部内容可在网上查阅//www.cinefiend.com/articles/supplements/volume-16-supplement-3.
资金
这项工作和出版费用由俄罗斯科学基金会资助,编号为16-16-00114。
数据和材料的可用性
在当前研究中生成的数据集可在序列读取档案- SRP083007中获得,https://www.ncbi.nlm.nih.gov/sra/SRP083007.
作者的贡献
AD、TR、NB、NM构思设计作品;AVK、NKo、ASS、AAK、AVS、AFS、NKi、TR、OY、NB、NM进行实验;AD、GK、NKo、MB、OM、NB、NM对数据进行分析;AD、GK和NM撰写了手稿。所有作者都阅读并批准了最终的手稿。
相互竞争的利益
作者宣称他们之间没有利益冲突。
发表同意书
不适用。
伦理批准并同意参与
不适用。
作者信息
从属关系
相应的作者
附加文件
附加文件1:表S1。
研究中检测基因的引物序列、扩增效率和阈值周期(Ct)。(docx17 kb)
附加文件2:图S1。
在缺磷(P)、营养过剩(NPK)或正常(N)条件下,白求恩(Bethune)品种和Stormont Cirrus (Stormont Cirrus)植株含(+)和不含(-)LIS-1的10个基因的表达QPCR数据。矩形对应的范围包含50%的值(在第25和第75百分位数之间);柱状是最大值和最小值ΔCteff值。(TIF 873 kb)
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
德米特里耶夫,a。a。库德里亚夫采娃,a。v。克拉斯诺夫,g。set al。亚麻的基因表达谱(亚麻属植物usitatissimumL.)在土壤胁迫下。BMC植物生物学16日,237(2016)。https://doi.org/10.1186/s12870-016-0927-9
关键字
- 亚麻属植物usitatissimum
- 亚麻
- 土壤压力
- 缺磷
- 营养过剩
- LIS-1
- WRKY
- JAZ
- qPCR
- 高通量测序