抽象的
背景
大多数植物的叶绿体负责光合作用,含有保守的约110个基因,可编码家务基因表达机械和光合作用相关功能的组件。从其他生物中获得营养素的异养植物及其塑性基因组代表了用于研究轻松选择性压力对光合功能的影响的模型系统。最明显的是降低塑料的大小和基因含量,其与编码不需要的光合作机的基因的丧失相关。预计将在核基因组中放松对非光合生活方式的非光合生活方式的选择性压力,然而,相应的变化较少。
结果
在这里我们报道了质体基因组的完整序列单调的低估,真菌异养植物属于真菌异养植物科的一种专性叶绿素植物ericaceae..质体系的M.低估大小(35,336 bp)大大降低,缺乏具有两个单拷贝区域和倒置重复的典型的四极结构。只有45个基因仍然是完整的 - 编码核糖体蛋白,核糖体和转移RNA和家务基因的那些inf那MATK,ACCD.和CLPP..这CLPP.和ACCD.虽然它们的序列高度分散,但基因可能保持稳定。对于核糖体蛋白和转移RNA的基因相对于光合植物的叶绿体不完全。两种亚种级分离株的体积基因组的比较M.低估揭示了与重复驱动的重组相关的主要结构重排和分离物特异性TRNA基因的存在。的分析M.低估通过RNA-SEQ的转录组显示,没有表达照相I和II反应中心蛋白的核编码组分,细胞色素的组分B6F.复合物,ATP合成酶,核苷酸二磷酸羧酸酶组分,以及来自原生骨蛋白IX生物合成途径的叶绿素。
结论
随着与光合作用,NADH脱氢酶,体塑性编码的RNA聚合酶和ATP合酶有关的基因的完全丧失M.低估质体基因组是专性非光合寄生物种中功能最退化的基因组之一。的分析M.低估转录组揭示了核和塑性基因组的协调演化以及在两个基因组中的光合作用相关功能的丧失。
背景
异养植物从其他生物中获得营养素,直接来自其他植物(直接寄生虫)或通过它们助理的菌根真菌(MycoheteroRrophs),通常会显示形态和基因组特征的降低。一些异养植物保留光合能力是偏瘫,而其他植物完全失去了进行光合作用的能力(即Holoparasites)。寄生植物的塑性基因组代表了研究宽松选择性压力对光合作用的影响的模型系统。最明显的是塑体基因组的大小和基因含量的降低,其与编码光合作机的基因的丧失相关,这变得不必要的[1-3.].几乎所有的异养植物都证明了与光合亲属相比的塑料的减少,在一些“早期”寄生虫中的最小值范围内Corallorhiza Striata.[4.] 和五角sp。[5.,在一些内寄生物种中达到极端,比如艾萨比卡的潜灵质[6.]但甚至可能造成塑料的完全损失大花草属lagascae[7.].
预计向非光合生活方式过渡将在不仅在叶绿体中的光合作用机械上放松选择性压力,也可以在核基因组中放松光合机械的选择性压力。然而,核基因组和寄生植物的转录组的相应变化较少并且限于少数研究(例如[8.那9.])。例如,Holoparasitic植物的转录组分析phelipanche aegyptiaca家庭的Orobanchaceae.揭示了光合作用相关基因表达的预期丧失和叶绿素生物合成途径的令人惊讶的保守和转录[10.].
MyCoheteroTorophy在至少50种植物谱系中独立进化,包含大约400种[11.].大多数完整的真菌异养体是单子体,主要代表兰科西家庭 [12那13].相对较少少量的MyCoherTotoRophic植物的体积基因组已经测序,包括兰花种类纹珊瑚,蔷薇花冠那Epigogium Aphyllum,Neottia nidus-Avis和Rhizanthella Gardneri.[4.那14-16],Petrosavia stellaris[17],Sciadphila densiflora.[18]和利物革Aneura mirabilis.[19].在别善之中,在家庭中发现了MycoheteroTrophsericaceae.[11.],属于订单杜鹃花目分子[20.].许多种类的Ericaeae.与菌根真菌形成关联,从中获得营养素。假设MyCoheter促型植物在低光的条件下从光合菌根谱系发生,其中它们损失光合能力和与真菌的紧密关联,这渗透了绿树根部,并为其肌萎缩伴侣提供了足够量的碳和营养素[12那21].
单调低价(Pinesap)是一个富人的家庭肌肤营养植物ericaceae.,亚科单调薄膜(审核[22])。地上的部分M.低估植物是5-35厘米的壮大的不分支偶然的reneme花序,顶部有2到11个花朵,覆盖大部分花序(附加文件1:图S1)。短根由蚊虫的蚊虫投资,形成用于附着真菌合作伙伴的样品样结构。最近我们测序了塑性基因组M.低估孤立Mon2-Kalr [23,首次报道真菌异养双二体质体。质体基因组只有34800-bp长;它相对于被子植物中大多数其他质体的基因顺序高度重排,缺乏具有两个反向重复区域的四分结构。大小的减少与编码NADH脱氢酶、光合作用相关蛋白、质体编码RNA聚合酶和一些其他功能的基因的丢失有关[23].在肌营养营养学中失去光合相关基因的丧失ericaceae.此前还由Braukmann和Stefanovic显示[24]使用插槽污迹南部杂交方法。
我们以前的研究揭示了大量的遗传多样性M.低估并且可能存在两个不同的亚种或品种[25那26].考虑到寄生植物中叶绿体基因组的加速演变,存在这种密切但不同的亚种性的存在提供了对分子变化进行比较分析,其分子变化标记了塑性基因组降解的进展。在这里,我们报告了第二个亚种隔离的全塑性序列M.低估,Mon-1Volr并提出了两种亚种的塑料的比较分析。此外,在这项研究中,我们分析了转录组M.低估评估核基因组的变化与过渡到MyCoheterootrophy的过渡和丧失进行光合作用的能力。
方法
塑性基因组测序和注释
一个单一的单调低价从俄罗斯(58°35'14“,37°59'20”E)中收集了植物。标本存储在登录号Mon-1Volr下。从单个单独的新鲜叶片中提取总基因组DNA,并使用钛XL +方案的罗氏基因组序列仪GS FLX(Roche,瑞士)对霰弹枪基因组文库进行测序。产生约86 MB的平均读取长度为356bp的序列。De Novo组件是用新手组合者v.2.9(454人生科学,美国Branford,USA)进行,其中含有18倍的覆盖率。基于相关物种的叶绿体基因组的序列相似度鉴定这些葡萄片(杜鹃花目)和高覆盖范围。在发射覆盖Contigs与其在ABI棱镜3730(应用生物系统)上的Sanger方法之间的适当PCR片段的产生时,获得完整的体积基因组序列。为了验证重建的体积基因组的正确组装,将原始读取映射到与GS参考映射器(454寿命,美国Branford)的重建序列映射。使用教条进行体积基因组注释[27,并进行进一步的人工修正。利用OrganellarGenomeDRAW工具绘制了质体的圆形图[28].
M的体积基因组的序列低估Mon-1Volr在加入号码KU640957下提交给Genbank。
转录组测序和组装
叶子(花苞片)和两个人的花朵单调低价收集植物(不是用于叶绿体基因组测序的同一样本)和两个带有吸器的根集合样本用于转录组分析。使用RNeasy Plant Mini试剂盒(Qiagen, Valencia, CA)从每6个样本的~0.3 g组织中提取总RNA。根据制造商说明书(Illumina Inc., USA),使用Illumina HiSeq2500平台(100 bp reads)对RNA样本进行测序。RNA-seq读取数据已存储在NCBI SRA数据库中,登录SRP069226。
M.低估转录组测序导致总共10300万高质量的测序读取,并用CENCADATT进行修剪[29镰刀(https://github.com/najoshi/sickle.), 分别。通过三位一体平台进行从组合的六个RNA-SEQ数据集中组装转录组组合[30.].将转录组组装成98350 unigenes,其长度为201至12993bp,N50为1342bp。使用TransDecoder(http://transdecoder.github.io.)。Trinotate(https://trinotate.github.io/)用于从Trembl和Swiss-Prot数据库分配命中(http://www.uniprot.org/uniprot/),并分配GO条款和PFAM域(访问15/02/2016)。使用预测的蛋白质序列在Trembl蛋白质数据库中注释了37977 unigenes,并且使用Blastx在Swiss-Prot数据库中注释了38419个unigenes。使用基于Web的KAAS服务器将蛋白质编码基因分配给Kegg纠正识别因子[31].
多样性基因组中蛋白质编码基因的转录水平被定量采用RSEM [32]和Bowtie 2计划[33].转录水平表达为每百万片段的每千碱基映射(FPKM)值的每千碱基碎片。
结果和讨论
塑料大小,基因含量和表达
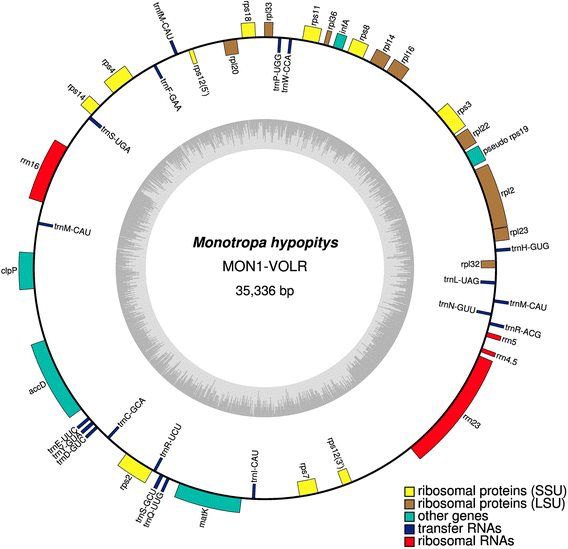
的质体染色体单调低价MON1-VOLR的长度仅为35,336英尺,缺乏具有两个单拷贝区域和倒置重复的典型的四极结构(图。1)。这是在那些之后从非水性高血管植物中获得的最高度减少的塑性基因组之一皮原士sp(11-15 kB,[6.]),Sciadphila densiflora.(21 KB,[18]), 和Epigogium.sp。(19-31 KB,[16])。这M.低估预计Mon1塑料含有45个可能是完整的基因(表1)。大多数这些基因涉及蛋白质合成:四种核糖体RNA基因,19转移RNA基因,小亚基的九个基因编码和大亚基核糖体蛋白的9个。剩余的基因是inf(翻译开始因子),mat(用于剪接组IIA内含子的熟酶),ACCD.和CLPP..
的功能ACCD.编码乙酰-CoA羧酸酯的β-亚基的基因尚不清楚,因为它被推导的蛋白质产物在N-末端区域非常分散并且相对于典型的ACCD蛋白具有长的重复的内部插入(例如,尼科尼亚塔哈瓦姆, 无花果。2)。但是,插入不会中断开放阅读框架。ACCD参与脂肪酸合成和叶片发展[34]应该对塑料维护至关重要[35].寄生植物的大多数塑料含有功能副本ACCD.基因;但是,在菲律宾人它也高度分化,含有长凹凸,似乎缺乏标准起始密码子[36].
喜欢ACCD., 这CLPP.基因应该对塑性维持至关重要,并且即使在寄生植物的强烈减少塑料中也存在[14].CLPP.编码Clp蛋白酶的蛋白水解亚基,参与蛋白质进入质体[35].M.低估Mon1塑料塑料CLPP.基因缺乏两个内含子,并且具有具有N末端延伸的非常分散的蛋白质序列,但是存在整个功能域(PFAM00574)(附加文件2:图S2)。该蛋白质的最接近的相对于来自eudicot的叶绿体的CLPPAsclepias Nivea.只有33%的氨基酸序列同一性。应该注意的是,在几种寄生和光合的高管谱系中观察到CLPP和内含子损失的加速演变(例如,[36那37])。
这RPS19可能是从它的5'-终端截断的伪果酱和缺乏与该地区短串联重复的累积相关联的起始密码子(附加文件3.:图S3)。功能的功能rpl22由于C末端截短(130A.A.VS154-159A.A.),因此该基因的预测蛋白质产物比其相关物种中的同源物短于其同源物,因此保留全RPL22保守域(PFAM00237 / CHL00034)。用于核糖体蛋白RPS15和RPS16的基因缺失,寄生植物的许多其他塑料中缺失[28那36].
始终如一的纯粹的肌肤萎缩M.低估,其质体缺乏类囊体nadh -脱氢酶复合物、光合作用相关蛋白(光系统I和II、细胞色素)的所有编码基因B.6.F复杂,鲁米斯科大亚基和ATP合成酶)。塑性编码RNA聚合酶的基因,光合作用相关基因YCF1[38保护基因YCF2,YCF3和YCF4.也不见了。
减少尺寸和基因含量的M.低估塑料也反映在内含子的损失中。只发现了一个内含子rpl2.基因,而内含子rpl16.和CLPP.缺失。这RPS12.是在大多数其他高血管植物中的跨剪接基因,但它仅由两个而不是三个外显子组成,表明缺乏一个内含子。存在mat尽管塑性基因组的含量强烈降低,但可以通过在IIA组内含子的保留来确定塑料基因组rpl2.需要治疗的熟酶活性的基因[39].
我们分析了预测蛋白编码基因的表达M.低估利用单株植物地上部分的RNA-seq数据进行质体分析。将reads映射到质体基因组中,计算蛋白质编码基因(外显子)对应的reads数量,并按基因长度的千碱基归一化。共绘制了33659个reads到蛋白编码基因。所有基因均有表达(见表)2)有趣的是,最表达的基因是ACCD.和CLPP.FPKM值> 100,000。相比之下,mat和rpl32.显示出较低的表达(FPKM值<10,000)。
两种塑性基因组的比较M.低估透露了广泛的重新排列
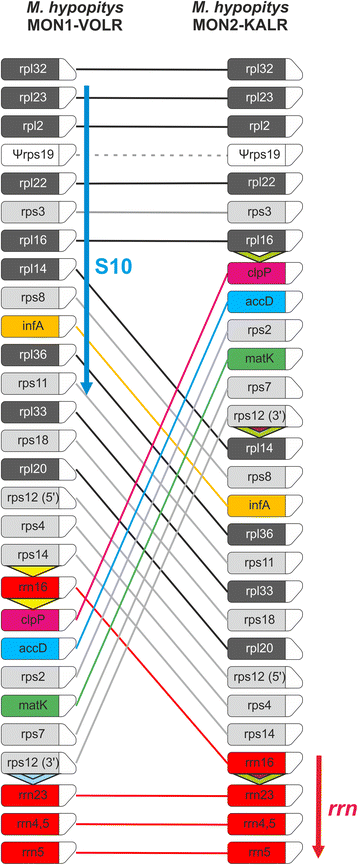
第二个完全塑性基因组的可用性M.低估从另一个亚种范围样本获得,称为Mon2-Kalr [23[允许我们通过比较分析在短时间内研究进化事件。令人惊讶的是,对于单个物种,这两种塑性基因组不仅略微不同(35,336bp的Mon1与mon2基因组的34,800bp),而且表明基因阶序列和含量的差异(图。3.)。
血浆基因组之间的示意性基因命令比较映射M.低估Mon1-Volr和M.低估MON2-KALR。利用圆形映射基因组的线性表示rpl32.基因作为起点不反映编码区之间的实际基因尺寸或间距。盒子代表蛋白质编码和RRNA基因,具有倒角顶角所示的转录方向。三角形M.低估地图表示重复序列。S10和rrn操纵子由箭头显示。请注意,塑体基因组结构的比较M.低估和尼科尼亚塔哈瓦姆显示在附加文件中4.:图S4
基因组在单个主要重排和几个局部次要诱导中不同。主要差异是根据该界限的基因组区域的位置交换M.低估MON1质体图,基因RPS14-RRN16和CLPP-RPS12(5 '外显子)。在两个基因组中,基因顺序明显偏离光合被子植物中保存的顺序(附加文件)4.:图S4)和无可以被视为“祖传”形式。第一个基因组Mon1保留了高度保守的S10操纵子的完整结构(RPS11,RPL36,INFA,RPS8,RPL14,RPL16,RPS3,RPL22,RPS19,RPL2,RPL23)通过重新排列在第二个基因组中被破坏。相比之下,M.低估mon2塑料包含RRN16-RRN23-RRN4.5-RRN5基因簇,其在mon1基因组中分开(图。3.)。S10和核糖体RNA操纵子的结构在叶绿体基因组中高度保守,因为它确保了组装核糖体所需的组分的共表达。尽管在过渡到非光合生活方式的过程中有许多重排,但S10的基因令rrn即使在如此高度减少的基因组中,操作犬也被保守sciadphila.和Epigogium.sp。[16那18].
重新排列的区域侧翼,重复序列可以促进重组事件[40那41].在标记重排区域的“结”点处,在Mon2塑料中发现三种这样的重复(图。3.);位于第一和第二重复之间的片段的碎片与蒙2塑料中的第二和第三之间的第二重复将产生观察到的Mon1基因塑性结构。
蛋白质编码基因的含量在两者中都是相同的M.低估塑料,虽然一些功能是值得怀疑的一些基因非常有变化。预测的蛋白质产品rpl22长度不同,两者都短于相关杜鹃花目物种。推导的产品ACCD.两者都具有n末端延伸和不同尺寸和低相似性的内部插入(图。2)。预测的CLPP.基因在MON2的5 '区包含连续72-bp长的不完全重复(附加文件2:图S2),但编码蛋白的常见部分高度相似(87%的身份),表明功能守恒。虽然本文正在审查,但是一份描述了描述塑性基因组的报告Monotropa Unvlora.另一个孤立的孤立M.低估发表了[42].它确认了我们以前关于减少的结果M.低估plastid基因组[23],并提供证据CLPP.和ACCD.在这些物种中完整。
令人惊讶的是,两种塑性基因组编码类似但不相同的TrNAS库存。Mon1塑料编码19种氨基酸的19种TRNA物种,第二塑料编码16个氨基酸的16个TRNA。两个TRNA基因仅存在于MON1和仅在MON2塑料中的一个(表1)。有趣的是,第二种塑料编码Trnf-AAA,对于植物叶绿体基因组是不寻常的,并且已经在寄生植物中报道E.乌克兰[16].
最近Barrett和Davis提出了在过渡到异营养素的叶绿体基因组降解模型[4.].根据这种模型,体液基因损失以下顺序:NADH脱氢酶,对光合作用的基因(PSA,PSB,PET,RBCL),质体编码的RNA聚合酶和ATP合酶基因。编码核糖体rna和核糖体蛋白质、转移rna和其他几个基本基因的基因(accD, clpP, matK, ycf1, ycf2)最后一个要丢失。的基因含量M.低估塑性基因组表明,这种塑料近于减少过程的结束,因为它甚至缺乏一些内政基因(YCF1,YCF2,一些TrNAS和核糖体蛋白)。值得注意的例外是CLPP.和ACCD.虽然它们的序列高度分散,但可能仍有功能的基因。塑料中这些基因的高水平转录也支持其功能。ACCD和CLPP是必须在叶绿体内组装的多亚基蛋白复合物的组分,因此CLPP.和ACCD.基因保留在塑料中[14].编码CLP蛋白酶其他亚基的基因的存在M.低估转录组(在其他地方发表)进一步支持这一假设。
核光合作用相关基因的丧失,如转录组分析所揭示
结构和基因含量M.低估塑性基因组清楚地显示出与光合作用有关的完全丧失。因此,我们分析了光合作用函数是否伴随着在核基因组中编码的相应基因的丧失。为了回答这个问题,我们分析了与光合作用相关基因的存在M.低估转录组使用这兆拟南芥作为查询的参考路径[10.].在kegg途径的63个基因中,在转录组中只检测到三个(额外文件)5.:表S1)。光照I和II反应中心蛋白,细胞色素组分B.6.F复合物,ATP合酶和核苷酸二磷酸羧基酶组分,在转录组中没有检测到,表明基因损失和/或不存在表达。核基因组的测序与分析M.低估将回答这些基因是否丢失或仅沉默的问题。
与光合反应中心的组成不同,叶绿素生物合成基因的表达在非光合寄生植物中已被检测到P. Aegyptiaca[10.].但是,分析了M.低估转录组显示,在叶绿素生物合成途径部件中[43仅在低水平且仅在花中表达单个基因,二乙烯基 - 还原酶,仅在花中表达,而原子卟啉IX mg-chelatase,原子卟啉IX甲基转移酶,Mg-原激霉素IX单甲酯氧化环化酶,蛋白氯化物氧化酶和叶绿素从转录组中不存在合成酶。这些发现与缺乏叶绿素相一致M.低估[44].从AminoLevicic酸产生的上游生物合成途径产生并表达,因为预期,因为这些步骤对于血红素和叶绿素合成常见。
结论
随着与光合作用有关的基因NAD(P)H脱氢酶、质体编码的RNA聚合酶和ATP合酶的完全丢失M.低估塑性基因组是在寄生非光合寄生物种中最具功能上减少的基因组。基因丧失和基因组减少与重排相关,损失典型的塑体基因组的典型四胞胎结构以及内含子的损失。质塑性基因组降解的先进过程也涉及一些管家功能,这是通过一些核糖体蛋白和转移RNA基因的丧失所证明的。两种亚种级分离株的体积基因组的比较M.低估揭示了与重复驱动的重组相关的主要结构重排和分离物特异性TRNA基因的存在。的分析M.低估转录组揭示了核和塑性基因组的协调演变和在两个基因组中的光合作用功能丧失。
参考
- 1。
depamphilis cw,palmer jd。寄生开花植物塑性基因组的光合作用和氯化血管基因的丧失。自然。1990年; 348:337-9。
- 2.
Wolfe Kh,Morden CW,Palmer JD。非分水性寄生植物最小血浆基因组的功能和演变。Proc Natl Acad Sci U S A. 1992; 89:10648-52。
- 3.
Barrett CF,Freudenstein JV,Li J,Mayfield-Jones博士,Perez L,Periz JC,Santos C.研究了在异养兰花早期过渡型植物中的塑性基因组降解的程度,以及对异养心血管植物的影响。mol Biol Evol。2014; 31:3095-112。
- 4.
Barrett CF,Davis Ji。肌雌性养殖的塑性基因组Corallorhiza Striata.(兰科)处于相对较早的降解阶段。我是J机器人。2012; 99:1513-23。
- 5.
Funk HT,Berg S,Krupinska K,Maier UG,Krause K.两种寄生花植物种类的塑性基因组的完整DNA序列,Cuscuta Reflexa.和Cuscuta Gronovii..BMC植物BIOL。2007; 7:45。
- 6。
内寄生属两种植物的质体皮原士(Apodanthaceae)每次保留只有五个或六种可能的功能基因。基因组Biol Evol。2015; 8(1):189-201。
- 7。
Molina J,Hazzouri Km,Nickrent D,Geisler M,Meyer Rs,Pentony MM,Flowers JM,Pelser P,Barcelona J,Inovejas SA等。寄生开花植物中叶绿体基因组的可能丧失大花草属lagascae(rafflesiaceae)。mol Biol Evol。2014; 31:793-803。
- 8。
Torres MJ,Tomilov Aa,Tomilova N,Regan RL,Yoder Ji。pscroph,寄生植物EST数据库富集寄生虫相关的转录物。BMC植物BIOL。2005; 5:24。
- 9。
Yoshida S,Ishida JK,Kamal Nm,Ali Am,Namba S,Shirasu K.一种全长富集的cDNA文库,并表达了寄生杂草的序列标签分析,Striga Hermonthica.BMC植物BIOL。2010; 10:55。
- 10。
wickt NJ, Honaas LA, Wafula EK, Das M, Huang K, Wu BA, Landherr L, Timko MP, Yoder J, Westwood JH,等。寄生植物科的转录组Orobanchaceae.揭示胃痛合成的令人惊讶的保护。Curr Biol。2011; 21:2098-104。
- 11.
Merckx V,Freudenstein JV。植物中肌萎缩的演变:一种系统发育观点。新植物。2010; 185:605-9。
- 12.
真菌共异养的进化生态学。新植醇。2005;167(2):335 - 52。
- 13.
Westwood Jh,Yoder Ji,Timko MP,Depamphilis CW。植物中寄生派的演变。趋势植物SCI。2010; 15:227-35。
- 14.
Delannoy E,Fujii S,Des Francs-Small CC,Brundrett M,小I.地下兰花中的猖獗基因流失Rhizanthella Gardneri.突出塑性基因组的进化约束。mol Biol Evol。2011; 28:2077-86。
- 15.
Logacheva MD,Schelkunov MI,Penin AA。肌营养型兰花中塑性基因组的测序与分析Neottia nidus-Avis.基因组Biol Evol。2011; 3:1296-303。
- 16.
Schelkunov Mi,Shtratnikova Vy,Nuraliev MS,Selosse Ma,Penin AA,Logacheva MD。探索塑性基因组减少的限制:肌营养植物的案例研究Epigogium aphyllum和Epigogium乌兰木.基因组生物学进展。2015;7:1179-91。
- 17。
Logacheva MD,Schelkunov MI,Nuraliev MS,Samigullin Th,Penin AA。Mycoheterotocrophic Monocot的塑性基因组Petrosavia stellaris表现出基因丢失和多重重排。基因组生物学进展。2014;6:238。
- 18。
Lam VK,Soto Gomez M,Graham SW。高度减少的肌营养营养型塑料sciadphila.(三尿基)是具有绿色亲属的Colinear,并在强烈的净化选择下。基因组Biol Evol。2015; 7(8):2220-36。
- 19。
Wickett NJ,张Y,汉森斯SK,roper JM,Kuehl JV,Plock SA,狼PG,Depamphilis CW,Boore JL,Goffinet B.发生寄生利物族血浆基因组的最小尺寸减少Aneura mirabilis..mol Biol Evol。2008; 25:393-401。
- 20。
Anderberg AA,Rydin C,Kallersjo M.在Oricales S.L订单中的系统发育关系:来自塑性和线粒体基因组的五种基因的分子数据分析。我是J机器人。2002; 89:677-87。
- 21。
Motomura H,Selosse Ma,Martos F,Kagawa A,Yukawa T. Mycoheretotrophy从混合营养祖先演变出来:证据兰花(兰科西)。安。机器人。2010; 106(4):573-81。
- 22。
Kron KA, Judd WS, Stevens PF, Crayn DM, Anderberg AA, Gadek PA, Quinn CJ, Luteyn JL。杜鹃花科的系统发育分类:分子和形态学证据。机器人启2002;68(3):335 - 423。
- 23。
Gruzdev Ev,Mardanov Av,Beletsky Av,Kochieva EZ,Ravin NV,Skryabin KG。寄生开花植物的完整叶绿体基因组单调的低估:广泛的基因损失和尺寸减少。线粒体DNA部分B:资源。2016; 1(1):212-3。
- 24.
Braukmann T,斯特凡诺维斯氏菌血液化基因组在肌营养性中的进化ericaceae..植物mol biol。2012; 79:5-20。
- 25.
Filyushin Ma,Reshetnikova NM,Kochieva EZ,Skryabin KG。寄生植物中序列的内部内部变异性单调低价L.来自欧洲俄罗斯人口。Genetika。2015; 51(11):1330-4。
- 26.
Filyushin Ma,Reshetnikova NM,Kochieva EZ,Skryabin KG。序列序列多态性和线粒体基因NAD1中的B / C内含子的二次结构,单调低价和相关煤矿族物种。Biol Bull。2016; 43(3):271-5。
- 27.
Wyman Sk,Jansen Rk,Boore JL。用教条自动注释细胞细胞基因组。生物信息学。2004; 20:3252-5。
- 28.
Lohse M,Drechsel O,Kahlau S,Bock R. Consterellargenomedraw - 一种用于生成塑体和线粒体基因组的物理图和可视化表达数据集的工具套件。核酸RES。2013; 41(Web服务器问题):W575-81。
- 29。
Martin M.Cutadapt从高吞吐量测序读取中删除适配器序列。嵌入期刊。2011; 17(1):10-2。
- 30.
Haas BJ,Papanicolaou A,Yassour M,Grabherr M,血液PD,Bowden J,等人。从RNA-SEQ使用Trinity平台进行参考生成和分析的DE Novo转录序列重建。NAT PROTOC。2013; 8(8):1494-512。
- 31。
Moriya Y,Itoh M,Okuda S,Yoshizawa Ac,Kanehisa M. Kaas:自动基因组注释和路径重建服务器。核酸RES。2007; 35(Web服务器问题):W182-5。
- 32。
李斌,杜威出版社。RSEM:从有或没有参考基因组的RNA-Seq数据中准确的转录本定量。BMC生物信息学。2011;12:323。
- 33。
Langmead B,Salzberg S.与Bowtie 2. NAT方法的快速喷射读取对齐。2012; 9:357-9。
- 34。
烟草质体accD基因是烟草叶片发育所必需的基因。植物j . 2005; 44:237-44。
- 35。
Krause K.寄生植物的塑性基因组:减少和损失的痕迹。在:Bullerwell CE,编辑。细胞器遗传学。柏林,海德堡:斯普林克;2012. p。79-103。
- 36.
Wicke S,MüllerKF,De Pamphilis CW,Quandt D,Wickett NJ,Zhangne,Renner SS,Schneeweiss Gm。武装系列光合作用和非水性寄生植物的功能性和物理基因组的机制。植物细胞。2013; 25(10):3711-25。
- 37.
Sloan DB,Triant Da,Forrester NJ,Bergner LM,Wu M,Taylor Dr。Angiosperm部落中加速塑性基因组进化的反复综合征硅藻素(蛛网曲霉)。mol phylocyet evol。2014; 72:82-9。
- 38.
Kikuchi S,BédardJ,Hirano M,Hirabayashi Y,Oishi M,Imai M,Takase M,IDE T,Nakai M.在叶绿体内包络膜中揭开蛋白质转运蛋白。科学。2013; 339:571-4。
- 39.
Zoschke R,Nakamura M,Liere K,Sugiura M,BörnerT,Schmitz-Linneweber C.一个细胞细胞成熟酶与多组II内含子相关联。Proc Natl Acad Sci U S A. 2010; 107:3245-50。
- 40。
Ogihara Y,Terachi T,Sasakuma T.叶绿体基因组的分子内重组在小麦物种中的短切片序列介导。Proc Natl Acad Sci U S A. 1988; 85:8573-7。
- 41。
MaréchalA,Brisson N.重组和植物细胞器基因组稳定性的维持。新植物。2010; 186:299-317。
- 42。
Logacheva MD,Schelkunov Mi,ShtraTnikova Vy,Matveeva MV,Penin AA。非光合作用塑性基因组的比较分析ericaceae.和他们的光合亲戚。SCI批准。2016; 6:30042。
- 43。
Beale Si。叶绿素生物合成的酶。Photosynth Res。1999; 60:43-73。
- 44。
Leake Jr。岩白型植物植物植物的生物学(“皂苷”)。新植物。1994年; 127:171-216。
确认
我们感谢两个匿名审查员进行其建设性评论,这帮助我们改进了稿件。这项工作是使用科学设备的核心研究设施的“生物工程”进行。
宣言
本文已作为BMC植物生物体积16补充3,3,2016的一部分发布:来自BGRS \ SB-2016的所选文章:植物生物学。补充的完整内容可在线提供//www.cinefiend.com/articles/supplements/volume-16- supplement-3.
资金
这项工作得到了俄罗斯科学基金会授予14-24-00175(转录组和MON2-KALR塑料测序)和俄罗斯基础研究授予的基础资金14-14-00749(Mon1-Volr塑性测序)。出版成本由俄罗斯科学基金资助。
作者的贡献
NVR构思了该研究,分析了数据并起草了稿件。EVG,AVB和VVK分析了基因组和转录组数据。AMM,EBP和AVM进行测序。MAF和EZK收集了植物样品和分离的核酸。KGS构思了这项研究,参与了其设计和协调并帮助起草了稿件。所有作者阅读并认可的终稿。
利益争夺
提交人声明他们没有竞争利益。
同意出版物
不适用。
伦理批准和同意参与
不适用。
作者信息
隶属关系
通讯作者
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
Ravin,N.v.,Gruzdev,E.v.,Beletsky,A.v.等等。非光合肌营养营养不良癖的塑性和核基因组中的光合途径丧失单调低价.BMC植物杂志16日,238(2016)。https://doi.org/10.1186/s12870-016-0929-7.
关键词
- 叶绿体基因组
- 寄生厂
- Mycoheterotrophy
- 光合作用
- 基因损失
- 转录组