摘要
背景
亚麻木脂素,俗称secoisolariciresinol (SECO)二葡萄糖苷(SDG),最近被报道具有促进健康的活动,包括其对代谢性疾病的积极影响。然而,关于SDG及其单葡萄糖苷(SMG)的生物合成,直到最近才有报道。最近报道了亚麻UGT74S1在体外将葡萄糖酸酯SECO依次转化为SMG和SDG。然而,该基因是否是亚麻中唯一实现SECO糖基化的UGT基因尚不清楚。
结果
进行了亚麻UGTs全基因组挖掘。通过系统发育分析、基因复制分析、外源基因表达分析和酶学分析等方法,鉴定出与之密切相关的家系成员UGT74S1并确定它们在SECO糖基化中的作用。共鉴定出299个不同的ugt,其中241个(81%)重复。亚麻UGTs分化为2.4 ~ 153.6 MYA, 71%处于纯化选择压力下。UGT74S1一个位于7号染色体上的单一拷贝基因,没有显示出复制的证据,被认为处于正选择压力下。系统发育分析确定了4个主要聚类,其中聚类4包括UGT74S1,是最多样化的。复制UGT74S4而且UGT74S3分别位于8号染色体和14号染色体上,它们的亲缘关系最为密切UGT74S1并在不同组织中有差异表达。UGT74S1、UGT74S4和UGT74S3蛋白的外源表达量相似,但UGT74S4和UGT74S3对SECO的糖基化活性比UGT74S1低7倍。此外,他们都未能实现可持续发展目标,这表明他们的分歧导致了新功能化UGT74S1.
结论
研究表明,UGT74S1与两个重复的基因UGT74S4和UGT74S3密切相关,与UGT74S1不同,UGT74S3未能将SMG糖甙转化为SDG。研究表明,UGT74S1可能是控制亚麻SECO糖基化转化为SDG的关键基因,但其密切相关的基因也可能在向UGT74S1供应SMG前体方面发挥了较小的作用.
背景
据报道,亚麻木脂素有广泛的健康益处[1- - - - - -3.].不同种类的植物已被证明产生不同类型的木脂素,包括被称为亚麻(亚麻)中主要木脂素的secoisolariciresinol diglucoside (SDG)。亚麻属植物usitatissimuml .)种子4,5].亚麻籽中的木脂素通常以糖基化形式存在,SDG和酯内衬在寡聚链中[6制造大分子[7,8];其单体苷元(SECO)和中间体单糖苷(SMG)在种子中不积累而形成。糖基化导致了植物化学物质结构的复杂性和多样性[9].确保植物天然产物的稳定性和水溶性,降低其反应性或毒性[10],便于细胞的运输和储存[11].糖基化是通过包括糖基转移酶(GTs)超家族的CAZymes实现的。GTs被分为94个家族,家族1被称为尿苷糖基转移酶(UGTs) [12,13].在植物中,ugt有一个由44个氨基酸组成的特征标记盒,俗称植物次生产物糖基转移酶(PSPG)盒[13,14]以及植物中配体结合和催化的作用[10].UGTs将udp活化的糖(如udp -葡萄糖)转移到受体的减法上,形成糖苷[15,16].在拟南芥,已有超过120个ugt被报道,并根据同源性分为30个亚家族[13].
在亚麻方面,Barvkar等[17]最近报告了来自亚麻草图基因组的137个UGTs [18],并将其分为14组(命名为A ~ N)。但没有提供功能表征。同时进行Barvkar的亚麻基因组数据挖掘研究[17],克隆并鉴定了5个家族1 UGT基因(JX011632, JX011633, JX011634, JX011635, JX011636),结果表明UGT74S1 (JX011632)是唯一一个能够通过SMG和SDG形成葡萄糖酸SECO的基因[19].通过对UGT74S1蛋白进行三维建模,然后进行配体对接、靶向位点诱变、外源表达和酶分析,得到Gln337和爵士357SMG的糖基化转化为SDG是必需的,而Trp355和他的352UGT74S1对SECO的糖基化活性是至关重要的[20.].然而,UGT74S1是否是亚麻基因组中唯一实现SECO糖基化转化为SDG的UGT尚不清楚。
基因复制是许多植物基因组的共同特征。有证据表明古代亚麻的全基因组复制事件[18],这一发现得到脂肪酸生物合成基因重复的深入研究的支持[21- - - - - -23].类似的UGT家族研究尚未在亚麻中进行,尽管已报道UGT家族存在多样性[17].更具体地说,命运UGT74S1通过亚麻基因组进化和它与其他ugt的关系,以及它的近亲的功能作用是未知的。本研究的目的是进一步研究亚麻UGT基因的多样性和进化,鉴定与UGT密切相关的基因UGT74S1,并通过对SECO生成SDG的糖基化活性测定来评估它们的功能。
方法
植物材料
亚麻(亚麻属植物usitatissimumL.) cv AC McDuff种子[24]于2008-2011年生长季节在AAFC哈林顿农场(哈林顿,PEI,加拿大)种植,并按之前描述的方法取样组织[19].
RNA和DNA分离
使用Trizol RNA试剂盒(Thermo Fisher Scientific, Burlington, ON, Canada)从发育中的亚麻种子、叶子和茎组织中提取总RNA,如前所述[25].提取的RNA样品使用PureLink™RNA Mini试剂盒(Thermo Fisher Scientific)进一步纯化,使用Qubit RNA HS分析试剂盒(Thermo Fisher Scientific)进行定量,质量由琼脂糖凝胶电泳和Experion RNA分析仪(BioRad, Missisauga, ON, Canada)进行检测,如前所述略有修改[19].采用Qiagen DNeasy Plant Mini Kit (Qiagen, Mississauga, Ontario)从亚麻叶片中提取基因组DNA,使用Qubit dsDNA BR分析试剂盒(Thermo Fisher Scientific)进行定量,并通过琼脂糖凝胶电泳验证质量。
UGTs的全基因组挖掘和系统发育分析
UGT74S1(JX011632),UGT74T1(JX011633),UGT89B3(JX011634),UGT94H1(JX011635),UGT712B1(JX011636) [19]及137种曾被鉴定的亚麻UGTs [17]作为查询,从亚麻基因组组合的43,471个注释基因中识别假定的ugt [18使用BLAST,其e值为1e-10。对已鉴定的UGT候选基因进行基因结构和序列相似性分析。
利用MEGA 6.0 [26].299个ugt中有68个的蛋白质序列与其他ugt相比非常短,可能是被截断的基因,因此被排除在树的构建之外。为了从尽可能多的UGT中获得树状拓扑结构的总体概况,利用ClustalW [27],采用Neighbor-Joining (NJ)算法构建系统发育树,自举重复500次。
因为ugt的特征是一个44个氨基酸的PSPG基序签名框[13,14已知参与底物识别和催化作用的[10,11], UGT候选基因列表被缩小到仅携带PSPG基序的基因。从192个ugt中提取PSPG基序的44个保守氨基酸及其50 bp上下游序列。这些氨基酸序列与肌肉对齐[28],采用最大简约分析法构建系统发育树,自举重复500次。
基因复制分析与UGT分化
利用整个假定的UGT数据库的self-BLAST进行基因复制分析,阈值e-30为[23].成对的UGT基因相互返回对等的顶部命中,并具有相同或高度相似的基因结构被宣布为重复拷贝。根据整理的亚麻基因组序列,将鉴定到的UGT基因分配到15个亚麻连锁组中,每个连锁组代表一条染色体[29].
重复和发散时间由计算得到的非同义替换比(卡)在计算出的同义替换(Ks)值29].简单地说,在Mega v7.0中对192条全长cDNA序列(不包括所有部分序列)进行比对[26)和Ka / Ks比率计算[30.].Ks根据Sveinson等人的建议,将低于0.001的值从数据集中删除[31].此外,Ks排除大于2的值以减少饱和度影响[32].遗传算法确定了基因对间的进化距离Ks用Nei-Gojobori核苷酸进化模型校正,该模型解释了每个位点的多个取代26]。散度(k一对重复基因转化为重复或分化的时间(t(MY)按照这个等式t=k/ (2r) / 106,在那里r替代率是6.5 × 10吗−9每年每个同义词站点的替换量[33].
的功能分析UGT74S1以及与其密切相关的ugtLus10006353而且Lus10014148
来评估两种ugt最密切相关的功能UGT74S1,对基因组DNA进行基因剪接,恢复其全长编码序列(CDS)Lus10006353而且Lus10014148含有短的单内含子[17,18].同时进行了这两个基因的组织特异性和外源基因表达研究UGT74S1.
Lus10006353而且Lus10014148基因组DNA的基因剪接
PCR扩增重叠基因片段是一种简单的基因剪接技术1) ((34),http://www.methods.info/Methods/Mutagenesis/PCR_splicing.html).将两个外显子连接在一起,将内含子从基因组DNA中删除,对应的基因组序列Lus10006353(JN088326.1)和Lus10014148(JN088327.1)设计基因特异性引物(附加文件1而且2).对于每个序列,从CDS的5’和3’端设计一个外侧翼引物对,称为外显子正向引物和反向引物。设计了2个30核苷酸的内引物。第一个内部引物称为外显子I- r,由外显子II意义链的前15个核苷酸的反向补体(内含子下游+15 bp)和外显子I意义链的后15个核苷酸(内含子下游- 15 bp)组成。第二个内部引物称为外显子II- f,由外显子I感觉链的最后15个核苷酸(内含子下游15个bp)和外显子II感觉链的前15个核苷酸(内含子下游+15个bp)组成,如前所述(http://www.methods.info/Methods/Mutagenesis/PCR_splicing.html)(额外的文件1).
基因剪接分三步进行。第一步是在第一次反应中使用5 '外显子I正向和I- r外显子扩增两个外显子,在第二次反应中使用II- f外显子和3 '外显子II反向扩增。将DNA稀释,以3 μL (10 ng/μL)为模板进行后续PCR反应。PCR循环为:94°C初始变性2min, 94°C 30s, 55°C 45s, 72°C 90s,最后延长72°C 10min。6 μL PCR产物在1%琼脂糖凝胶上用溴化乙锭染色。用QIAquick PCR纯化试剂盒(Qiagen)纯化扩增的I和II外显子片段,将每个纯化产物稀释50倍,每4 μL作为模板,在第二个基因剪接步骤中生成每个基因的全长模板。在PCR过程中,两个中间产物的重叠链形成双链,提供Taq有一个自由的3 '端进行延伸和一个单链进行聚合,从而产生一个完整的产品。PCR反应共10个循环,95℃30 s, 25℃45 s, 72℃90 s。结果PCR产品稀释50倍和3μL整除被用作模板在第三个和最后一个PCR反应在94°C 3分钟紧随其后30 94°C的周期30年代,55°C 45和72°C的90年代,紧随其后的是最后一个在72°C扩展10分钟。融合全长片段与QIAquick凝胶萃取净化设备(试剂盒),克隆到威尼斯平底渔船TA向量和转换大肠杆菌(Thermo Fisher Scientific)类似于之前的描述[19].
UGT74S1, Lus10006353而且Lus10014148不同亚麻组织的基因表达
量化基因表达水平Lus10006353而且Lus10014148在不同的亚麻组织(发育亚麻种子、根、叶、茎)中,从3′末端设计实时PCR引物LuS10014148、LuS10006353 UGT74S1,和一个核糖体RNA (EU307117),后者用于数据归一化(附加文件2)如上一份报告所述[19].第一链cDNA的合成和实时PCR条件如前所述[19].输出基因表达数据使用2——∆∆CT方法(35],结果以相对于花后0天(DAA)的折变表达[19].
克隆和外源表达
该TOPO TA克隆的全长度片段为Lus10006353而且Lus10014148测序确认。携带限制性内切酶位点的侧翼引物(附加文件2)被设计成全程穿梭Lus10006353而且Lus10014148放入酵母(酿酒酵母)表达载体pYES2/NT C和B (Thermo Fisher Scientific)。pYES2/NT C构建了携带的全长cDNAUGT74S1曾被描述过[19].所有三个结构体都在酵母中转化(酿酒酵母)按照制造商的说明(Thermo Fisher Scientific)培养INVSc1菌株,并按照之前报道的方法培养、诱导、收获和裂解单个转化菌落[19,20.].分别用等量蛋白和稀释后的抗Xpress抗体,western blot检测3个ugt的蛋白表达,其中UGT74S1为对照TM如前所述,6×组氨酸标签和多个克隆位点之间存在的表位[19,20.].
酶分析和反应产物的测定和定量
确定两种ugt (LuS10014148或LuS10006353)是否能像之前报道的UGT74S1一样将SECO葡萄糖酸酯转化为SDG [19,20.],从表达UGT74S1、LuS10014148或LuS10006353的酵母培养物中纯化的天然蛋白与SECO (Chromadex, Irvine, CA, USA)在udp -葡萄糖存在下反应,SECO是与本研究相关的唯一底物[19,20.)或UDP-galactose。100 μL反应混合物组成和反应孵育条件与我们之前的报告中描述的完全一致[20.].
对于反应物及其产物的分离、鉴定和定量,采用Waters H-Class Acquity UPLC系统(Waters, Milford, MA, USA),配备TQD串联质谱计(Waters)和Waters CSH C18柱(100 mm × 2.1 mm, 1.8 μm粒径),如前所述[19,20.,尽管有轻微的修改。除MS2扫描模式外,采集选定的离子记录(SIR)光谱,以提高SECO、SMG、SDG和SECO单半乳糖苷(SMGal)的检测灵敏度。毛细管电压设为3 kV,萃取器电压设为3 V,射频透镜电压设为0.1 V。色谱参数采用3%甲酸水溶液(a)和乙腈(B)组成的二元梯度体系,t0, a = 98%;t1 = 4.4 min, A = 0%;t2 = 6 min, A = 0%等分度;t3 = 7 min, A = 98%;t4 = 8 min, A = 98%等度。在280 nm处检测到的峰,表明含有酚类化合物,并使用从Chromadex(美国加州尔湾市Chromadex)购买的正宗标准(SECO和SDG)进行验证,如[19].使用上述SDG标准创建了SDG的标准曲线。如前所述,纯化SMG标准是内部制备的[19,36].
所有反应进行三次,数据以平均值±标准差表示。称为单侧学生的t-检验UGT74S1、Lus10014148和Lus10006353的代谢物产生水平的统计学意义[20.].
结果
亚麻基因组中UGT基因的复制和多样性
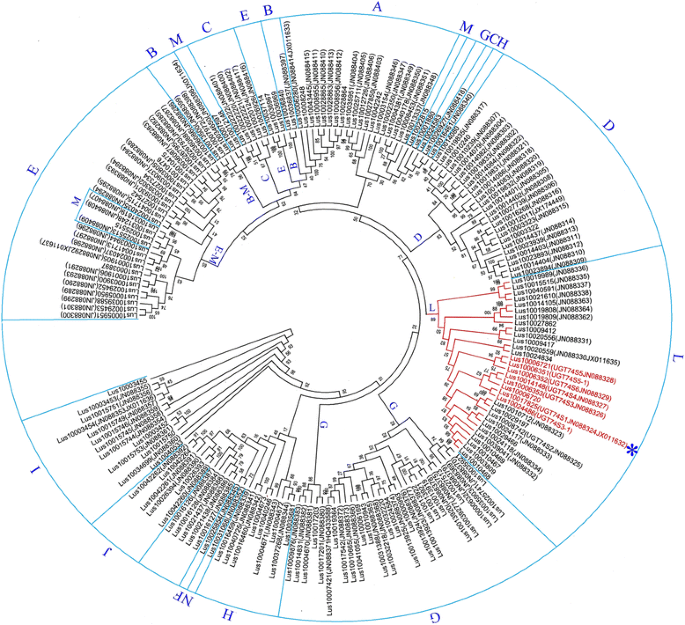
对UGT基因的全基因组挖掘发现了299条分散在所有15条亚麻染色体上的非冗余序列(附加文件3.而且4).12号染色体上UGT基因最少,只有10个序列,而1号染色体上UGT基因最多,有35个3.而且5).共检测到241个(81%)的UGT基因重复,其中56个(23%)对应于染色体内重复(附加文件3.).其中14例(25%)发生在1号染色体上,而在2、7、10、11和14号染色体上未观察到染色体内重复3.).UGT74S1位于7号染色体上,没有基因重复的迹象。
对299个ugt中的231个(不包括68个截断序列)进行系统发育分析,发现了4个主要的集群(附加文件6)及15个子组别。聚类4是最多样化的,占UGT基因的53% (122/231 UGTs),包括此前报道的14个组中的8个(F、G、H、I、J、K、L和N) [17].本研究发现的第15个新组命名为“O”(附加文件6).UGT74S1聚在L组内,属于5个ugt小基因家族,包括Lus10017825(JN88324 / JX011632),Lus10006353(JN088326), Lus10014148(JN088327)Lus10024486,而且Lus10008742(JN088325),由53%的自举值支持,以后称为Lus10017825子支系。这个分支的两个成员,Lus10014148而且Lus10006353,分别位于8号和14号染色体上的重复基因,与UGT74S1(附加文件3.而且6).该小基因家族与一个姐妹子支聚在一起,这里称为Lus10006352/Lus10006351子支,该子支的bootstrap值为51%,其中包括Lus10006352(JN088329), Lus10006721(JN088328),Lus10006351而且Lus10006720(附加文件6).
为了进一步评估观察到的分组的一致性,我们利用192个UGT序列中的保守区域构建了一棵新的树。与第一个大型系统发育树一致,这个简化树显示,相对较高的bootstrap值(10-100%),与之前报道的相同配置和分组[17].然而,A、B、C、E和M组没有像在前一棵树中那样被很好地分解。在这第二棵系统发育树中,组“L”得到了89%的bootstrap值,包括Lus10017825子枝Lus10017825(JN88324 / JX011632),Lus10006353(JN088326), Lus10014148(JN088327), Lus10024486而且Lus10006720而Lus10006352/Lus10006351子序列包括在内Lus10006352(JN088329), Lus10006721(JN088328)和Lus10006351(无花果。1),从而为我们推断ugt的进化历史提供更多的系统发育的信心。UDP-glucosyltransferase命名后,路10006353,路10014148,路10024486,路10008742,Lus100Lus10003520352, Lus10006721而且Lus10006351被注释为UGT74S3,UGT74S4,UGT74S3-1,UGT74S2,UGT74S6,UGT74S5,UGT74S5-1分别sub-families。因此,Lus10014148而且Lus10006353将于下文以UGT命名:UGT74S3而且UGT74S4,与同UGT74S1(Lus10017825).
UGT基因差异,
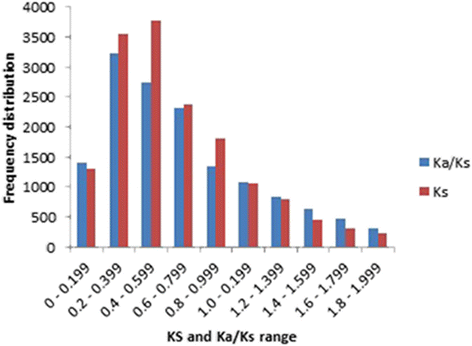
我们通过测定UGT序列散度来检测UGT序列散度Ka / Ks192条UGT序列中PSPG基序的比例。后Ks计算和数据过滤,15616对(85%)UGT基因与Ks范围在0.031到1.997之间。总的来说,82%的基因对有Ks值<1,包括45%与Ks值< 0.5。的Ks平均值为0.642,峰值为0.493。2).同样,47和71%的UGT基因对有Ka / Ks< 0.6和Ka / Ks< 1,分别。只有不到1%的基因对有Ka / Ks= 1(附加文件7).个体的分布频率Ka / Ks值在0.306处达到峰值。2).
涉及SDG木聚糖形成基因的基因对UGT74S1(Lus10017825)Ks< 1,Ka / Ks< 1的比例分别为85和56%。在Lus10017825和Lus10006352/Lus10006353子枝内,所有Ks由涉及的基因对衍生而来UGT74S1范围从0.208到0.264。相比之下,8对基因中只有2对(25%)有Ka / Ks< 1,其余75%的基因对显示Ka / ks> 1。
192个UGT基因对间的变异时间在2.4 ~ 153.6 MYA之间7).聚在L组的31个基因间的差异时间从7.4 ~ 148个MYA(附加文件8).在Lus10017825和Lus10006352/Lus10006351子枝中,Lus10024486(UGT74S3-1),Lus10006352(UGT74S2)似乎是最古老的,在大约42年玛雅时期彼此分离。UGT74S1(Lus10018725)可能与聚集在这两个子枝上的其他7个ugt在16 - 20mya之间发生了分化。根据我们的推断,UGT74S3(Lus10006353)可能重复的UGT74S4(Lus10014148)~ 15米娅。它们之间的进化关系UGT74S5(路10006721),路10006720,UGT74S6(路10006352)而且UGT74S5-1 (Lus10006351)被发现是复杂的。重复研究确定了两者UGT74S6(对应8)UGT74S5-1(第8条)作为UGT74S5(Chr 5)一方面,而且UGT74S6(第8条)作为Lus10006720(Chr 5)的重复/发散时间分别约为16和15.5年UGT74S5 / UGT74S5-1而且Lus10006720 / UGT74S6,分别。不过,散度的时间为UGT74S5 / UGT74S6pair不能使用过滤标准集(表1).
结构基因组织UGT74S1和它最近的亲戚
UGT74S1是一个1407 bp的基因,含有一个739 bp的内含子[19].评估两种ugt的功能,这是最密切相关的UGT74S1,也就是说,UGT74S3而且UGT74S4,我们通过基因剪接得到它们的全部编码区。UGT74S3将1492 bp的基因组DNA片段的678和738 bp的外显子连接,得到1416 bp的剪接融合CDS。同样,将1464 bp基因组DNA的643 bp和728 bp的外显子连接,得到1371 bp的剪接融合CDSUGT74S4(附加文件9).从NCBI数据库(JN088326.1;JN088327.1)和Phytozome (Lus10006353。g, Lus10014148.g)。在这项研究中,UGT74S3而且UGT74S4分别有76和93 bp的内含子,它们的融合剪接编码序列被证实与已经报道的完全相同(附加文件10).值得一提的是UGT74S4,对应NCBI加入JN088327.1,由Barvkar等人报道[17在它的5 '末端有一个27bp的截断(附加文件10).通过比较扩增的基因组和剪接的融合序列,检测到这种截断UGT74S4与JN088327.1 [17]和Lus10014148。G来自亚麻草图基因组(http://phytozome.jgi.doe.gov/pz/portal.html#!gene?search=1&detail=1&method=3127&searchText=transcriptid:23152916).在UGT74S3,内含子位于679 ~ 756核苷酸位置之间,而位于643 ~ 737核苷酸位置之间UGT74S4(附加文件10).因此,全基因组序列的大小为UGT74S4是1464 bp,而不是先前报道的1437 bp [17].拼接的全长cdna为UGT74S3(1416个基点)UGT74S4(1371 bp)预测分别编码471和456个氨基酸,本研究也观察到了同样的结果(附加文件11).两者都显示了近62%的身份UGT74S1并在氨基酸水平上显示出80%的同一性。保守的44个氨基酸PSPG基序(附加文件11)位于UGT74S3的331 ~ 374氨基酸之间,UGT74S4的343 ~ 386氨基酸之间。UGT74S3和UGT74S4的PSPG motif与UGT74S1的身份分别为90和80%11).UGT74S3和UGT74S4 PSPG基序中保守的4个氨基酸被替换到UGT74S1中:E339Q、A345K、M362L和W374C。UGT74S3有3种独特的氨基酸变化(L350V、R373E和T375G), UGT74S4有2种独特的氨基酸变化(Q373E和N375G;额外的文件11).
UGT74S1,UGT74S3而且UGT74S4不同亚麻组织的基因表达
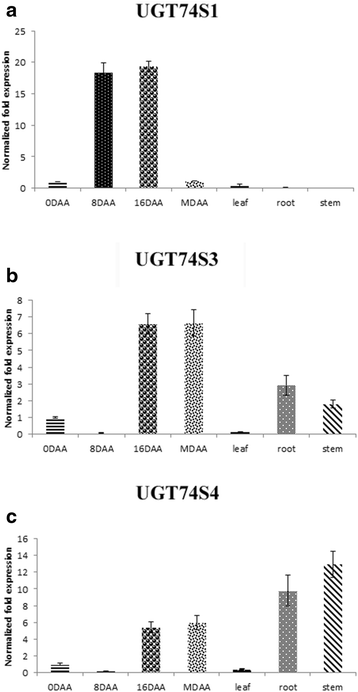
的实时基因表达水平UGT74S4而且UGT74S3在种子和叶子的发育上相似,但在茎和根上明显不同。此外,他们的表达模式也不同于UGT74S1在所有组织(图。3.).在发展中种子,UGT74S1表达模拟钟形曲线模式,峰值在16 DAA(图。3)如前所述[19].在8岁和16岁的时候,UGT74S4而且UGT74S3表达量比对照组低3 ~ 260倍UGT74S1然而,它们的表达水平却高于(5-6倍)UGT74S1到期(无花果。3 c).相比之下,UGT74S1在根和茎组织中几乎不表达在哪里UGT74S4而且UGT74S3高度表达。三种UGT基因在叶片中的表达量都很低(图4)。3.).
外源蛋白表达及酶活性
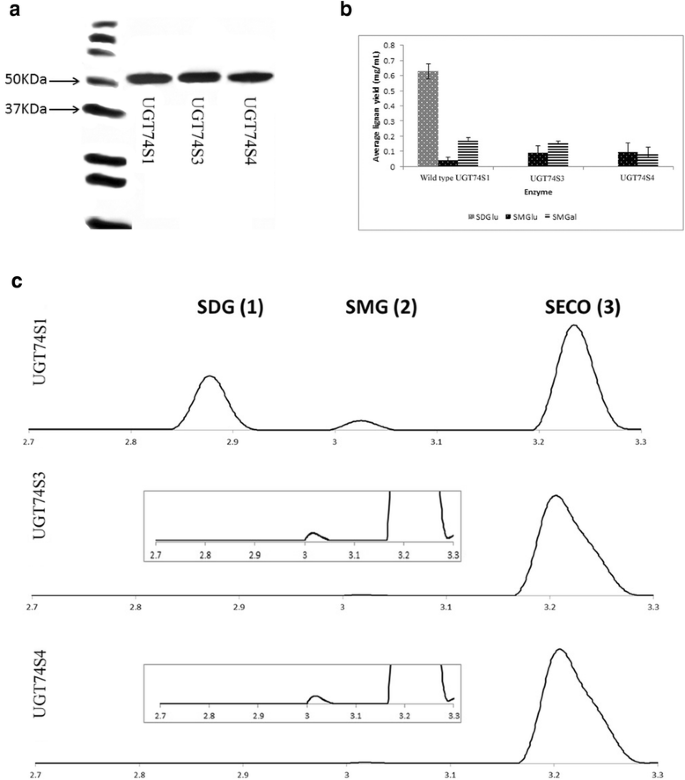
在这项研究中,我们想要评估是否UGT74S4而且UGT74S3据报道,UGT74S1的酶具有糖基化SECO的能力[19,20.].结果表明,UGT74S4和UGT74S3的SECO蛋白表达量和糖基化活性均高于UGT74S1。的全长cdnaUGT74S4而且UGT74S3如前所述在酵母中表达UGT74S1.western blot分析显示,在2%半乳糖诱导8小时后,三种蛋白均高表达(图2)。4).UGT74S4和UGT74S3显示出较强的信号强度,与之前报道的UGT74S1的56.4 kDa相当[19,20.],分别产生了54.5和56.1 kDa的蛋白质,与他们的预测序列一致(图。4).
用纯化的天然酶对UGT74S1、UGT7 4S3 (Lus10006353)和UGT74S4 (Lus10014148)及反应产物进行Western blot检测。一个,使用AntiXpress对histag纯化的UGT74S1、UGT74S3和UGT74S4进行Western blot检测TM抗体与Western C精密加蛋白标记物混合偶联物(BioRad)相对应的37和50 kDa蛋白分子标记物被精确定位。b, UGT74S1、UGT74S3和UGT74S4分别以SECO为底物,以udp -葡萄糖或udp -半乳糖为糖供体酶促反应后形成的反应产物。西班牙secoisolarciresinol二糖苷,SMGsecoisolariciresinol monoglucoside,SMGalsecoisolariciresinol monogalactoside。c。以SECO为底物,udp -葡萄糖为糖供体的UPLC色谱(280 nm)显示了50 μg纯化的三种ugt蛋白的反应产物。Insets10倍放大显示UGT74S3和UGT74S4反应产物中SMG的痕量峰。只有UGT74S1的SDG(峰1)和SMG(峰2)有明显的积累。未反应的SECO(峰3)存在于所有三种色谱图中
为了测定UGT74S4和UGT74S3的糖基化活性,我们使用纯化的蛋白进行酶分析,以UGT74S1为阳性对照(图4)。4 c).虽然UGT74S4和UGT74S3的蛋白表达与UGT74S1相似(图;4),它们对SECO的葡糖基化活性减少了7倍,在我们的实验条件下都不能产生SDG(图。4 c).没有显著差异(P= 0.46)的糖基化活性在UGT74S4和UGT74S3之间。
利用udp -半乳糖作为SECO糖苷生产的替代糖供体,三种蛋白都形成了微量的SECO单半乳糖苷(SMGal)。但三种酶均未检测到SECO二半乳糖苷(图。4 c).当udp -半乳糖作为替代糖供体时,三种蛋白质的SMGal水平没有差异。
讨论
ugt是一个庞大而复杂的酶家族,用于催化糖苷键的形成[19].最近有报道称,亚麻UGT74S1是一种酶,在酵母中表达时,将SECO葡萄糖化成单糖甙SMG和双糖甙SDG [19,20.].为了进一步了解UGT74S1在亚麻中实现SECO糖基化转化SDG的专一性,我们进行了全基因组挖掘、组织特异性和外源基因表达以及酶活性测定。这项研究证实了我们之前的发现[19,20.],进一步表明UGT74S1可能是控制亚麻SECO糖基化的关键酶,而UGT74S4和UGT74S3也可能在一定程度上影响SMG作为底物供给UGT74S1进行第二糖基化.
共发现299个ugt分布在373 Mb的亚麻基因组中[18],对应的基因密度为0.80 UGT/Mb,与拟南芥(0.79)高于水稻(0.46)、苹果(0.32)、葡萄藤(0.37)、杨树(0.37)、大豆(0.16)和高粱(0.25)。UGTs在亚麻总预测基因中的比例为0.69%,高于双子叶(0.32-0.54%),与单子叶(0.65-0.72%)相差不大[14].
基因复制是真核生物中常见的现象,如植物[37,38].它导致基因的新颖性和多样性,进化和功能多样化[39- - - - - -41],指维管植物为适应和可塑性所必需的现象[13].包括全基因组重复的古倍体事件在该属中已被描述亚麻属植物(31],并被认为是基因复制的关键驱动因子[42- - - - - -44].以往的研究预测了亚麻基因组中的43,484个基因,其中9920个基因被预测为重复[18].在本研究鉴定的299个ugt中,有重复基因241个(81%),占亚麻基因组重复基因的2.4%。
Ks以前被用作重复事件的时间指标,其频率分布被报道反映基因组重复的相对时间[33,45,46].因此,Ks由UGT基因对计算得到的最大值为0.493,范围为0.4 - 0.6,表明存在古老的重复[46在UGT进化过程中。使用Ks结果表明,192对基因之间的差异在2.4 ~ 153.6倍之间。聚在L组的31个基因的差异时间为7.4 ~ 148个MYA。这些结果表明,一些亚麻UGT重复事件可能发生在最近估计在5-9 MYA的全基因组重复之前[31].我们的Ka / Ks分析显示71%的UGT基因对具有Ka / Ks< 1,说明亚麻的大部分ugt是在净化(负)选择下进化而来的[47].大约四分之一(28%)的基因对有Ka / Ks> 1,因此处于正选择压力下,而0.75%的UGT基因处于中性选择[46].在我们的研究中,我们使用携带PSPG基序的全蛋白序列来计算卡而且Ks。类似地,全长基因序列已被用来确定Ks /卡小麦和水稻同源基因的值[48].无论潜在的偏差是否与保守区域自身的发散率有关,我们可以合理地假设,如果存在这种偏差,那么这种偏差应该是均匀分布的,并且最小的,因为考虑的所有序列都是PSPG motif,因此不会影响得出的结论。
尽管UGT在亚麻中的复制率很高,UGT74S1该基因为单拷贝基因,位于连锁群7(7号染色体)上。根据本研究设定的标准,共发现160对基因涉及该基因,其中56%的基因涉及该基因Ka / Ks< 1, 44%与Ka / Ks> 1,消极选择和积极选择的比例几乎是50/50。当一个基因家族的一个成员进行正向选择时,它显示为高Ka / Ks与所有家族成员配对时的比率[49].这一事实在Lus10017825和Lus10006352/Lus10006353亚支中得到证实,其中75%的基因对涉及UGT74S1有Ka / ks> 1,表明UGT74S1正处于积极选择之中,因此,它正在经历选择约束的减少[50保持单例[51].阳性选择已被报道UGT73C10而且UGT73C11,在皂素介导的昆虫抗性中发挥作用[52]和木脂素,也有报道称木脂素参与植物防御昆虫[53,54].有可能UGT74S1在一个或两个被提议的古代复制事件中被复制,并且如先前报道的那样,它的类似物随后通过基因丢失被删除[47].单拷贝基因通过大分子复合体的形成在维持植物必要代谢过程中发挥关键作用[51].UGT74S1在适应性分化期间,SECO糖基化转化为SDG木脂素的过程中,可能作为一个单一拷贝基因在亚麻中保持不变UDP-glucose焦磷酸化酶在高粱(52].
此前对UGT的系统发育研究已经描述了14和17个不同的组,使用了107个UGT氨基酸序列拟南芥(15]及来自九种高等植物的1520 ugt [14),分别。在亚麻方面,Barvkar等[17]描述了14组,基于137 ugt。目前研究中观察到的15个亚群包括以前报道的14个亚麻类群[17并确定了一个名为“O”的新组。这里观察到的分组与先前报道的14-17组相当[14,15,17]并强调了亚麻中ugt的多样性,这可能是通过协同进化实现的,这一现象在其他高等植物中也存在[14].
重复的基因可能面临三种不同的命运:非功能化,其中一个副本变得沉默;新功能化,即一份拷贝获得新的有益功能,而另一份拷贝保留原有功能;或子功能化,即两个副本都因突变的积累而部分受损[39].UGT74S4而且UGT74S3,它起源于重复事件,是最接近于将SECO葡糖化成SDG的UGT74S1 [19,20.].因为高序列相似性并不一定转化为表达和功能上的相似性,UGT74S4而且UGT74S3研究了SECO的组织特异性表达和蛋白功能。相反对UGT74S1,UGT74S4而且UGT74S3在8-16 DAA发育阶段,SDG木脂素合成迅速增加[19,55].但是,这2个重复的基因在营养组织和成熟种子(SDG合成已经完成的阶段)中表达更多[56)比UGT74S1,并表现出不同的表达模式,可能是因为它们的组织特异性启动子或复制事件后的新功能化[57].报道的数据表明,这两个基因可能在其他尚未阐明的生物过程中发挥重要作用,而不是SECO糖基化进入SDG。在外源酵母表达系统中获得了高水平的蛋白表达,但在本研究条件下,UGT74S4和UGT74S3无法将SECO转化为SDG。然而,它们能够产生少量的SMG,其对SECO的总体糖基化活性比UGT74S1低7倍,这表明UGT74S4和UGT74S3蛋白可能偏好不同的底物。相比之下,UGT74S1持续产生更高数量的SDG和少量的SMG,并且它的基因在SDG木脂素积累的发育种子中表达更高,如先前报道的[19,20.].这些观察结果表明,UGT74S1可能是SECO糖基化转化为SDG的关键功能角色,因为它能够实现两个必需的糖基化步骤;首先将SECO糖化成SMG,再将SMG糖化成SDG ([19和这项研究)。此外,突变UGT74S1导致减少或取消体外可持续发展目标的产生[20.,进一步展示了它的基本功能。虽然在这项研究中,并不是所有的299个ugt都被生物化学评估了它们对葡萄糖酸酯SECO的能力,但研究发现UGT74S4而且UGT74S3(UGT基因与UGT74S1)从SECO中以很低的速率产生SMG,并且没有形成任何SDG,这表明这两个基因不像UGT74S1那样重要,它完成了所有需要的步骤[19].虽然还没有完全排除SECO糖基化过程中涉及其他隐性ugt导致SDG,但人们可能会质疑为相同的生化功能维持冗余基因和重复通路的进化优势。此外,最近在我们实验室进行的一项后续研究的证据支持了这一观点,该研究聚焦于乙基甲烷磺酸盐(EMS)亚麻突变系的TILLINGUGT74S1在控制SECO糖基化转化为SDG方面是独一无二的(数据未显示)。在本研究中,UGT74S4和UGT74S3能够在描述的条件下以较低的效率与udp -葡萄糖或udp -半乳糖进行SECO糖基化,分别形成SMG或SMGal,但不能像UGT74S1那样对SDG进行糖基化,因此没有对这两种酶进行进一步的生化表征或优化。事实上,如果UGT74S4和UGT74S3能够以较低的速率将SMG糖基化转化为SDG,那么即使在标准条件下,也可以使用非常敏感的MS检测方法观察到微量SDG峰,这将为进一步的酶反应生化优化提供依据。然而,在我们的实验条件下,情况并非如此,这表明这两种蛋白质可能不具备进行第二步糖基化的能力。这些酶活性数据与发育中种子基因表达差异时间数据的结论一致,并进一步证实了UGT74S3和UGT74S4可能在SDG木聚糖糖基化之外的不同代谢过程中发挥其他作用。PSPG基序在配体结合和催化中的作用已经在植物中得到了充分的证明[10,58].UGT74S1及其两个密切相关的UGT基因在PSPG内观察到的差异可能是它们底物特异性差异的原因[20.],可以解释观察到的微分功能。
结论
UGTs形成了一个大的基因家族,其中大多数成员在亚麻基因组中是重复的,并受到负选择压力或净化选择压力,而UGT74S1是在正选择压力下进化的单例基因。亚麻UGT74S4(Lus10014148),UGT74S3(Lus10006353)的关系最为密切UGT74S1但它们编码的蛋白无法将SMG糖基化成SDG。虽然在本研究中并没有对基因组中所有UGT进行生物化学测试,但其他与UGT74S1在SMG糖基化过程中具有相同作用的冗余UGT基因不太可能在没有特定进化优势的情况下得到维持。然而,不能完全排除神秘的UGT功能的可能性。尽管如此,目前的研究为亚麻中UGT基因复制事件提供了额外的见解,可能对植物进化生物学家、育种家和植物天然产物化学家有兴趣。
缩写
- cd:
-
编码序列
- DAA:
-
天开花
- EMS:
-
乙甲烷磺酸盐
- 卡:
-
非同义替换
- Ks:
-
同义替换
- 女士:
-
质谱分析
- 米娅:
-
几百万年以前
- NCBI:
-
国家生物技术信息中心
- PSPG:
-
植物次生产物糖基转移酶
- 西班牙:
-
Secoisolariciresinol二糖苷
- 入股事宜:
-
Secoisolariciresinol
- SMG:
-
Secoisolariciresinol monoglucoside
- SMGal:
-
Secoisolariciresinol monogalactoside
- 耕作:
-
基因组的靶向诱导局部病变
- UDP:
-
尿苷二磷酸
- UGT:,
-
尿苷糖基转移酶
- UPLC:
-
超高效液相色谱法
参考文献
刘志强,刘志强,刘志强,等。植物雌激素作为预防癌症的天然前药:膳食类黄酮。Phytochem启8:375 2009;86年。
张志勇,张志勇,张志勇,张志勇,张志勇,等。估计富含肠木脂素的食物和纤维与绝经后乳腺癌生存率的关系。Brit J Cancer. 2011; 105:1151-7。
王勇,傅凡娜,Roy M, Ghose K,姚X-H, Nixon M- s,等。亚麻籽木脂素癸二醇二葡萄糖苷通过上调GLUT4表达改善肥胖小鼠胰岛素敏感性。J功能性食品。2015;18:1-9。
潘建勇,陈淑林,杨明华,吴军,辛科宁J,邹凯。木脂素的天然产物及其合成研究进展。Nat Prod rep 2009; 26:1251-92。
张志勇,张志勇,张志勇,张志勇,等。呋喃木脂素(+)-芝麻醇的有序糖基化胡麻属indicumUGT71A9和UGT94D1葡萄糖基转移酶。植物j . 2008; 54:415-27。
Touré A,薛明X.亚麻籽木脂素:来源、生物合成、代谢、抗氧化活性、生物活性成分、健康益处。食品科学与食品安全。2010;9:261-9。
Struijs K, Vincken JP, Doeswijk DG, Voragen AG, Gruppen H.亚麻籽壳木脂素大分子链长度通过香豆酸苷和阿魏酸苷的加入来确定。植物化学。2009;70:262-9。
Kosińska A, Penkacik K, Wiczkowski W, Amarowicz R.亚麻籽木聚糖大分子中咖啡酸的存在。植物营养杂志。2011;66:270-4。
黄晓明,王晓燕,王晓燕,等。植物次生代谢糖基转移酶的功能分析。植物科学进展。2005;
Osmani SA, Bak S, Møller BL.利用晶体结构和同源性模型预测植物udp依赖的糖基转移酶的底物特异性。植物化学。2009;70:325-47。
李志强,李志强,李志强,等。糖基转移酶的研究进展。植物学报,2005;8(4):393 - 393。
王杰,侯斌。糖基转移酶:参与植物次生代谢产物修饰的关键分子。医学杂志。2009;4:39-46。
黄晓燕,王晓燕。糖基转移酶1家族功能多样性的进化研究。植物j . 2011; 66:182 - 93。
Caputi L, Malnoy M, Goremykin V, Nikiforova S, Martens S.对family 1 udp -糖基转移酶的全基因组系统发育重建揭示了该家族在植物适应陆地生活过程中的扩张。植物j . 2012; 69:1030-42。
Ross J, Li Y, Lim EK, Bowles DJ。高等植物糖基转移酶。基因组医学杂志。2001;2:30041-6。
黄晓燕,刘志强,刘志强,等。黄酮类糖基转移酶基因的重组表达及功能研究Hieracium pilosellal .足底。2009;229:1135-46。
巴夫卡,Pardeshi VC, Kale SM, Kadoo NY, Gupta等亚麻属植物usitatissimum发现具有不同表达模式的基因。BMC基因组学。2012;13:175。
王铮,N Hobson, L Galindo,朱松,史丹,J McDill,等。亚麻(亚麻属植物usitatissimum)组装新创从短霰弹枪序列读取。植物j . 2012; 72:461 - 73。
Ghose K, Selvaraj K, McCallum J, Kirby CW, Sweeney-Nixon M, Cloutier SJ,等。亚麻udp -糖基转移酶将secoisolariciresinol (SECO)糖化成secoisolariciresinol单葡萄糖苷(SMG)和二葡萄糖苷(SDG)的鉴定和功能表征。植物学报。2014;14:82。
Ghose K, McCallum JL, Sweeney-Nixon M, Fofana B. Histidine 352 (His352)和色氨酸355(色氨酸355)是亚麻UGT74S1对secoisolariciresinol糖基化活性的基础。《公共科学图书馆•综合》。2015;10 (2):e116248。
Pan X, Siloto RMP, Wickramarathna AD, Mietkiewska E, Weselake RJ。亚麻发育过程中产生的双酰基甘油酰基转移酶的鉴定(亚麻属植物usitatissimumL.)种子催化三聚氰菊酯的选择性生产。中国生物化学杂志。2013;28(3):433 - 436。
张志强,张志强,张志强,张志强,张志强,等。亚麻6个去饱和酶基因的遗传变异及其对脂肪酸组成的影响。中国科学:地球科学。2013;
李鹏,傅亚斌,李泽恩,王志勇,等。亚麻脂肪酸生物合成控制基因家族的全基因组鉴定与特性研究(亚麻属植物usitatissimumL).生物信息学。2014;7:310-26。
Kenaschuk EO, Rashid KY。AC McDuff亚麻。中国植物科学。1994;21(4):393 - 393。
Fofana B, Cloutier S, Duguid S, Ching J, Rampitsch C.硬脂酰- acp去饱和酶(SAD)和12脂肪酸去饱和酶2 (FAD2)的基因表达在亚麻种子发育过程中受到调控,并影响亚麻种子脂肪酸组成。亚麻属植物usitatissimum).脂质。2006;41:705-12。
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析6.0版本。《生物学报》2013;30:2725-9。
汤普森·JD,希金斯·DG,吉布森·TJ。CLUSTAL W:通过序列加权、位置间隔惩罚和权重矩阵选择,提高渐进式多序列比对的灵敏度。核酸杂志1994;22:4673-80。
埃德加钢筋混凝土。肌肉:多序列比对,精度高,吞吐量高。中国生物医学工程学报。2004;32(3):393 - 393。
李鹏飞,李志强,李志强,李志强,等。亚麻精细化基因组及其进化与应用。2015;中国生物医学工程学报,2015,31(1):1 - 6。
估计同义和非同义核苷酸取代数的简单方法。Mol Biol Evol 1986; 3:18 - 26。
黄国强,李俊,李晓欣,陈晓明,等。亚麻属内一个古多倍体事件的系统发育定位(亚麻属植物利用转录组)。安机器人。2014;113:753 - 61。
陈文敏,陈文敏。基于年龄分布的基因组重复推断。《生物学报》2013;30:177-90。
王海燕,刘志强,刘志强,等。禾草和棕榈树取代率的比较:核基因上的同义取代率差异抗利尿激素质体基因上的平行率差异:.中国科学:地球科学(英文版)1996;
Heckman KL, Pease LR。pcr驱动重叠延伸的基因剪接和突变。Nat协议。2007;2:924-32。
Livak KJ, Schmittgen TD。利用实时荧光定量PCR和2——ΔΔCT.方法。2001;25:402-8。
李霞,袁建平,徐世平,王建辉,刘旭。高效液相色谱法分离测定亚麻籽提取物中secoisoriciresol二葡萄糖苷低聚物及其水解物。J .色谱。2008;1185:223-32。
张国刚,黄淑珍,Pin A-L, Adams KL.基因和基因组重复后的剪接模式在进化历史中的广泛差异拟南芥.生物学报。2010;27:1686-97。
木特,竹林N, Barker MS, Mayrose I, Greenspoon PB, Rieseberg LH。维管植物多倍体形成的频率。中国科学(d辑)2009;
Lynch M, Conery JS。重复基因的进化命运和结果。科学。2000;290:1151-5。
Jordan IK, Wolf YI, Koonin EV。重复的基因比单基因进化得慢,尽管最初的速率增加了。BMC Evol biool . 2004;4:22。
王勇,冯玲,朱勇,李勇,颜慧,向勇。WRKY III基因家族的比较基因组分析杨树、葡萄。拟南芥(Arabidopsis and Rice Biol Direct) . 2015;
吴旭,杨华,曲川,徐智,李伟,郝波,等。杨树AMT基因家族的序列与表达分析。植物学报,2015;6:337.54。
王欣,王晓峰,王晓峰。植物基因组序列比较的研究进展。植物学报,2010;31(3):349 - 356。
王勇,谭欣,陈晓明。不同的基因结构模式随着基因的重复而分化拟南芥.BMC基因组学。2013;14:652。
杨胜,张晓华,岳建新,田德昌,陈建强。在两种木本植物中,nbs编码基因扩增主要是近期重复。浙江大学学报(自然科学版)2008;
钟艳,李艳,黄凯,程志明。板栗nbs编码基因的种特异性重复(齿栗叶mollissima).Sci众议员2015;5:16638。
杨丽娟,王晓燕,王晓燕。杨柳近缘性基因的序列分析与表达进化。Sci众议员2015;5:18662。
K kwaura, Mochida K, Enju A, Totoki Y, Toyoda A, Sakaki Y,等。基于普通小麦全长cDNA序列数据和表达模式的小麦和水稻适应性进化评估BMC基因组学。2009;10:271。
塔克AE。基因变异水蚤Pulex基因组。博士论文,新罕布什尔大学,2009。可用:https://books.google.ca/books?isbn=1109233620
花青素途径基因的进化速率变异。生物学报2003;20:184-1853。
韩峰,彭勇,徐玲,肖鹏,29个被子植物基因组单拷贝基因的鉴定、特性和利用。BMC基因组学。2014;15:504。
蒋思远,池玉华,王建中,周建新,程玉生,张宝林,等。蔗糖代谢基因家族及其生物学功能。Sci众议员2015;5:17583。
麦克雷WD,塔GHN。木脂素的生物活性。植物化学。1984;23:1207-20。
李志强,李志强。木脂素和二苯乙烯类化合物在植物-昆虫化学相互作用中的生物活性。植物化学启2003;2:321-30。
Ramsay A, Fliniaux O, Quéro A, Molinié R, Demailly H, Hano C,等。亚麻籽发育过程中木脂素大分子中主要酚类化合物的掺入动力学。食品化学。2017;217:1-8。
李志强,李志强,李志强,李志强,等。激光显微解剖和时空松脂醇-松脂醇还原酶基因表达揭示了亚麻仁皮中松脂醇二葡萄糖苷的细胞层特异性积累。植物学报。2016;21(4):433 - 436。
张志强,张志强,张志强,等。大麦HD-Zip I复制转录因子表达模式差异导致其新功能化。新植醇。2013;197:939-48。
Masada S, Terasaka K, Mizukami H. PSPG-box中有一个单独的氨基酸在caUGT2 (Cucurmin glucosytransferase, Cucurmin glucosytransferase)的催化功能中起重要作用Catharantus roseus也叫.2月。2007;581:2605-10。
确认
作者感谢David Main, Colin Stetson, Ashok Somalraju和所有在夏洛特敦研究和发展中心(夏洛特敦,PEI)的暑期学生,感谢他们在这个项目过程中的帮助。
资金
这项研究是由加拿大基因组/基因组草原资助的亚麻总利用基因组(TUFGEN)项目的一部分,加拿大亚麻理事会向BF提供了资金支持。资助者在研究设计、数据收集和分析、决定发表或准备手稿中没有任何作用。
数据和材料的可用性
支持这篇文章结果的数据包括在手稿和附加文件中。
作者的贡献
BF:稿件的构思、协调、设计、实验、数据分析、解读和撰写;KG:进行实验、数据分析、解读、起草、修改稿件;JM:酶反应产物UPLC和质谱数据采集与分析,稿件起草与修订;FY:数据分析、解读、起草、修改稿件;SC:稿件的协调、管理和修改。所有作者阅读、评论、审稿。
相互竞争的利益
作者声明他们之间没有利益冲突。
同意出版
不适用。
伦理认可和同意参与
不适用。
作者信息
从属关系
相应的作者
附加文件
附加文件1:图S1。
基因剪接PCR示意图(改编自Gratchevhttp://www.methods.info/Methods/Mutagenesis/PCR_splicing.html).(TIF 90 kb)
附加文件2:表S1。
采用引物进行PCR介导剪接、克隆和实时表达Lus10006353和Lus10014148基因。(多克斯16 kb)
附加文件3:图S2。
15条亚麻染色体299个ugt的Circos图谱。映射中心的线连接副本。每条染色体的大小以Mb表示。UGT74S1,UGT74S4(LuS10014148),UGT74S3(LuS10006353)均以星号标示。(TIF 4003 kb)
附加文件4:表S2。
亚麻基因组中发现的299个ugt的列表、位置和坐标。对应于UGT74S1的UGT位点用粗体红色突出显示。(XLSX 25 kb)
附加文件5:图S3。
UGT基因在亚麻基因组15条染色体上的分布。“第一副本和第二副本”一词是任意的,仅用于图表表示目的。亚麻基因组中的15条染色体由Lu1 ~ Lu15标记。(TIF 40 kb)
附加文件6:图S4。
231份亚麻UGTs的15个类群的系统发育关系。68个序列相对较短的ugt被排除。四个主要的集群由alpha数字1-4表示,不同的组由字母A-O表示,在圆形树周围和内部。UGT74S1属于L组中的一个小基因家族,用红色标出,并用星号标记。(TIF 8558 kb)
附加文件7:表S3。
卡,公里,Ka /公里15616对UGT基因过滤后的分化时间。(XLSX 641 kb)
附加文件8:表S4。
系统发育树“L”组31个基因在MYA中UGT分化时间的成对矩阵。在Lus10017825和Lus10006351/Lus1006352子枝中发现的ugt用绿色突出显示。蓝色和黄色分别强调了最近和古代的分歧时间。(XLSX 15 kb)
附加文件9:图S5。
通过基因融合PCR法比对Lus10006353 (UGT74S3)和Lus10014148 (UGT74S4)的基因组DNA和全长编码区。A,将Lus10006353扩增的I外显子(678 bp)与II外显子(738 bp)融合,得到基因融合产物,并与基因组DNA扩增子进行比较;B,将Lus10014148扩增出的I外显子(643 bp)和II外显子(728 bp)融合,与其基因组DNA扩增子比较相似。分子阶梯的大小用箭头表示。(TIF 83 kb)
附加文件10:图S6。
熔接CDS之间的序列对齐Lus10006353 (UGT74S3)而且Lus10014148 (UGT74S4)NCBI和Phytozome报道了它们各自对应的基因组和转录序列。A、熔接接头的顺序对准Lus10006353 (UGT74S3)剪接前基因组区扩增的CDS (genomic Lus10006353),其相应的基因组序列在NCBI和Phytozome (JN088326.1和Lus10006353。g,分别)和Phytozome (Lus10006353)转录本序列。基因组序列显示76 bp的内含子位于核苷酸位置679和756之间,而该内含子在已发表的转录序列和本研究报道的融合剪接CDS中缺失;B、熔接接头的顺序对中Lus10014148 (UGT74S4)剪接前的扩增区域CDS (genomic Lus10014148),其相应的基因组序列在NCBI和Phytozome (JN088327.1和Lus10014148。g,分别)和Phytozome (Lus10014148)转录本序列。基因组序列显示,该内含子长93 bp,位于核苷酸位置643和737之间,而该内含子在已发表的转录序列和本研究报道的融合剪接CDS中缺失。(TIF 1168 kb)
附加文件11:图S7。
UGT74S1、Lus10006353 (UGT74S3)和Lus10014148 (UGT74S4)的蛋白序列比对。PSPG区域用红色框表示,三个蛋白质之间的氨基酸差异用黑色框中的白色压痕表示一致序列。所有氨基酸的位置根据其在UGT74S1中的位置编号。(TIF 1731 kb)
权利和权限
开放获取本文根据知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许不受限制地在任何媒体上使用、分发和复制,前提是您要适当地注明原作者和来源,提供创作共用许可的链接,并说明是否进行了更改。知识共享公共领域转让豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
福法娜,B.,古斯,K.,麦卡勒姆,J.。et al。UGT74S1是控制亚麻中secoisolariciresinol diglucoside (SDG)形成的关键角色。BMC植物杂志17日,35(2017)。https://doi.org/10.1186/s12870-017-0982-x
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-017-0982-x
关键字
- 亚麻
- Secoisolariciresinol
- 西班牙木酚素
- 基因组挖掘
- 基因复制
- ugt
- Neofunctionalization