摘要
背景
抗叶锈病(Lr)引起的柄锈菌triticina在小麦(小麦在过去的几十年里,L.)已经得到了很好的研究,超过70Lr基因被定位到不同的染色体上,大量的qtl(数量性状位点)被检测或利用DNA标记定位。这种耐药性通常分为种族特异性和种族非特异性两种。小种非特异性耐药可进一步分为对同一病原体的大多数或所有小种的耐药和对多种病原体的耐药。在分子水平上,这三种类型的抗性可能覆盖了小麦中由编码不同蛋白质家族的基因控制的整个病原体特异性谱。本研究的目的是预测和分析三个家族的基因:NBS-LRR(核苷酸结合位点和富含亮氨酸的重复序列或NLR), START(甾体急性调节蛋白[STaR]相关脂质转移)和ABC (atp结合盒)转运蛋白。分析的重点是这些蛋白编码基因在基因家族中的关系模式和检测到的叶锈病抗性qtl。
结果
我们预测是526美国广播公司, 1117年NLR和144年开始通过小麦蛋白质组结构域分析小麦六倍体基因组中的基因。在小麦幼苗期和成虫期抗叶锈病qtl的1809个snp中,有126个snp位于这些基因的编码区或邻近区域(转录起始位点(TSS)上游5 Kb或转录终止位点(TTS)下游5 Kb)。其中43个用于成虫抗性的snp和18个用于幼苗抗性的snp位于编码区或邻近区域美国广播公司而成虫抗性的14个snp和幼苗抗性的29个snp位于编码区或邻近区域NLR基因。此外,我们还发现了17个成虫抗性非同义snp和5个幼苗抗性snp美国广播公司5个非同义snp用于成虫抗性,6个snp用于幼苗抗性NLR基因。大多数这些编码snp被预测会改变编码的氨基酸,这些信息可以作为对指定的更彻底的分子和功能表征的起点Lr基因。利用来自叶锈病抗性qtl的99个已知非snp标记的引物序列,我们发现了与这些标记密切相关的候选基因,包括Lr34与它的两个基因特异性标记的距离为1212个碱基(到cssfr1)和2189个基地(到cssfr2).
结论
本研究是对……的综合分析美国广播公司,NLR和开始小麦六倍体基因组基因及其与苗期和成虫期抗叶锈病qtl的物理关系。我们的分析表明美国广播公司(和开始)基因更有可能与非种族特异性的成虫抗性qtl共定位NLR基因更有可能与通常在苗期表达的种族特异性抗性qtl共定位。虽然我们的分析受到目前可用的复杂六倍体小麦基因组不完整组装的许多qtl的不准确或未知物理位置的阻碍,但观察到的(i)种族特异性抗性qtl与NLR(ii)非特异性抗性qtl和美国广播公司这些基因将有助于发现幼苗期和成虫期抗叶锈病的SNP变异。含有非同义snp的基因是小麦抗叶锈病新来源的潜在候选基因,可以在未来的研究中进行研究。
背景
叶锈病是由真菌引起的一种疾病柄锈菌triticina(=p . recondita罗伯吉前德斯马兹。f . sp。tritici)已严重威胁到世界面包小麦的生产(小麦在过去的几十年里,[1,2,3.]。叶锈病是一种具有许多生理小种的生物营养性寄生虫,通常对具有亲和性抗性基因的小麦品种具有高度特异性。迄今为止,至少有75种叶锈病(Lr)抗性基因已被鉴定出来,其中大多数在苗期就具有特定的抗性[4]。然而,少数种族非特异性Lr基因包括Lr34和Lr67还发现,特别是在成虫阶段,对多种病原体具有抗性[5,6,7,8,9]。
根据Krattinger等人的研究。[10),Lr小麦和其他谷物中的基因根据其特异性和持久性可分为三类。第一组含有赋予对同一病原体物种的一个而不是其他种族特定抗性的基因。如上所述,大多数的Lr基因就在这个群体中。这些Lr基因通常编码细胞内具有核苷酸结合位点和富含亮氨酸重复序列(NLR,也称为NBS-LRR或NLR)的免疫受体蛋白R基因)。由R基因直接或间接感知病原体衍生的毒力效应物,这些效应物分泌到宿主细胞的细胞质中,以抑制基础免疫。第二组含有同时对多种真菌病原体具有非种族特异性抗性的基因。这组中被充分研究的例子是Lr34.该位点最早由Dyck [11并在小麦染色体7D上进行了定位。后来人们知道它具有抵抗多种疾病的能力,包括叶锈病的抗性基因Lr34、茎锈蚀(引起的)茎杆),抗性基因命名为Sr57、条纹(黄)锈(造成)p . striiformis),抗性基因命名为Yr18、白粉病(引起的)Blumeria茎),抗性基因命名为Pm38并将大麦黄矮病毒的抗性基因命名为击穿电压1 (11,12,13,14]。因此,Lr34其他名称包括Lr34/Yr18, Lr34/Yr18 / Sr57 / Pm38和Lr34/Yr18 / Sr57 Pm38 /击穿电压1在文献中。Lr34它编码一种假定的ABC转运蛋白,包含跨膜(TM)和核苷酸结合(NB)结构域[6,15]。与第二组一样,第三组具有种族非特异性抗性,但与第二组不同的是,这种抗性针对同一病原体物种中的所有种族。这一组中一个已知的例子是小麦中一种抗黄锈病的基因。Yr36),该组中的耐药基因被称为START基因,因为它们编码START(甾体急性调节[StAR]蛋白相关脂质转移域)蛋白[16]。的其他示例开始其他谷物品种的基因包括隐性稻瘟病抗性基因pi21编码富含脯氨酸的小蛋白质[17],以及大麦抗白粉病的隐性基因枣疯病膜锚定蛋白的编码[18]。
上述NLR、ABC和START蛋白的一个共同特征是在每个家族中存在不同的结构域。在NLR组中,大量潜在的r-基因或抗性基因类似物(RGAs)编码r蛋白或称为细胞内免疫受体的效应识别受体,大多数属于核苷酸结合位点- lrr (NBS-LRR或NLR)类[19]包括7个结构域或基序:Toll/白介素-1受体(TIR-NBS-LRR或TNL)、卷曲卷曲(CC- NBS-LRR或CNL)、亮氨酸拉链(LZ)、NBS、LRR、TM和丝氨酸苏氨酸激酶(STK) [20.]。在ABC组中,唯一特征明确的基因(Lr34)编码全尺寸atp结合盒(ABC)转运蛋白,该蛋白是ABCG亚家族的成员,也称为PDR(多效耐药)亚家族[6,10,12]这种蛋白质阻止相容的病原体在小麦和其他谷物中的入侵和传播[21]。的功能Lr34是组成型而不是诱导型,因为基因的功能与病原体是否存在无关。目前还不知道Lr这种基因在START组中是可用的。Yr36是目前已知的唯一具有抗小麦条锈病广谱性的抗锈病基因,这种成体抗性在高温(25-35°C)下高度表达[16]。一般来说,开始基因还没有得到很好的研究,但已知它们在植物中编码许多功能的蛋白质。当然,START蛋白的第一个功能是对植物病原体的抗性(例如,上面描述的三个START基因:Yr36,pi21[17),枣疯病[18])。第二个功能是对非生物胁迫的响应,例如,鹰嘴豆在干旱、盐、伤口和热胁迫下增加跨膜START (TM-START)基因的表达[22]。第三个功能是调节拟南芥转录因子活性的能力[23]。
RNA-Seq是一种比以前的表达分析技术(如微阵列)更准确的定量基因表达水平的方法[24]。由于RNA-Seq无需基因组序列即可工作,因此可以对宿主和病原体转录组进行联合分析,从而深入了解病原体如何调节其基因表达以促进疾病进展,以及它们如何在防御反应期间影响宿主植物的电路[25,26,27]。最近的一项研究[27]报道了一个敏感小麦品种(Vuka)和一个抗性品系(Avocet- 2)的详细RNA-Seq时间过程Yr5)接种了小麦黄锈病菌柄锈菌striiformisf . sp。tritici(太平洋标准时间)接种后不同天数(dpi)。这些作者能够在小麦植株和小麦中鉴定出差异表达的基因簇太平洋标准时间.例如,他们共鉴定出7个具有相似表达谱的基因簇,这些基因簇在小麦寄主的GO(基因本体)术语注释和KEGG(京都基因与基因组百科全书)途径成员关系中富集。其中,宿主基因簇III在11 dpi处表达高峰,其中膜转运、ABC转运蛋白和几丁质酶基因显著富集。因此,我们希望鉴定出属于宿主基因的表达谱美国广播公司,NLR和开始基因家族。
最近的QTL定位研究已经报道了数百个小麦群体抗锈病的QTL [28,29,30.,31]。此外,在一些全基因组关联研究(GWAS)中已经报道了与小麦抗锈病相关的新型qtl [32,33,34],使用9k或90k单核苷酸多态性(SNP)芯片[35,36]。大部分幼苗期和成虫期的叶锈病抗性qtl现在都存储在T3数据库中[37]。然而,人们对这些qtl与基因之间的物理关系知之甚少美国广播公司,NLR和开始基因。例如,可以预期[10那……美国广播公司(和开始)基因更有可能与非种族特异性成虫抗性的qtl共享基因组区域NLR基因更有可能与易于在幼苗中表达的种族特异性抗性qtl共享基因组区域。本研究的目的是进行生物信息学预测和基因注释美国广播公司,NLR和开始与T3数据库中获得的小麦苗期和成虫期抗叶锈病qtl共享基因组区域的基因家族[37]。具体来说,我们首先预测了假设美国广播公司,NLR和开始小麦基因组中的基因。然后我们试图在这些假定的基因和指定的基因之间建立物理关系Lr从叶锈病qtl两侧的SNP标记序列中提取基因。本研究为进一步研究小麦基因库中现有和新的抗锈病基因的分子功能提供了重要的框架,从而为小麦抗锈病改良品种的开发提供依据。
结果
小麦中可能存在的ABC、NLR和START蛋白
我们总共预测了526个美国广播公司小麦基因组中的基因。这些ABC蛋白被分为8个亚家族,其中G亚家族又分为Gwbc和Gpdr(表1)1).结果表明,小麦的G (Gwbc和Gpdr)、C、B和I亚家族是ABC家族中最大的4个亚家族,分别占526个亚家族的30.4%、23.0%、18.8%和16.2%美国广播公司基因,分别。值得注意的是,在小麦基因组中预测了78个ABC Gpdr转运蛋白,略高于之前根据拟南芥和水稻的全尺寸ABCG基因数量估计的60个全尺寸ABCG转运蛋白[15,38]。相比之下,我们还对拟南芥进行了平行分析,以验证预测管道。我们的结果显示,在拟南芥中发现的131个ABC蛋白中,G(33.5%)、B(20.8%)、I(16.1%)、C(12.8%)是4个最大的亚家族,这与Andolfo等人最近报道的拟南芥中ABC蛋白的结果一致。39]。这些作者还报道了大米中的146种ABC蛋白[39),包括7(4.8%)、29日(19.9%)、18(12.3%),3例(2.1%),3例(2.1%),7(4.8%),41(28.1%)、22日(15.1%)和16例(11.0%),B, C, D, E, F, Gwbc,分别Gpdr和我的家庭。
对于NLR家族,我们在小麦中预测了1117个NLR蛋白(表2)1).这一数字略高于之前对小麦nlr编码基因全基因组分析的研究[40,41但略低于厄尔勒姆研究所(Earlham Institute)利用小麦基因组组装新基因模型预测的1185个NLR蛋白[42]。在一项同步分析中,我们在拟南芥中预测了249个NLR蛋白,其中大多数是TNL (Tir-NBS-LRR),占近69%(171个),而CNL (coil- coil- nbs - lrr)蛋白不到21%(52个)。这一较高的TNL/CNL比率与先前对双科植物NLR蛋白的研究一致[40,43,44]。相比之下,近88%(979)的小麦NLR蛋白被归为CNL类。然而,使用NLR-Parser检测到9个(0.8%)小麦TNL蛋白,这与普遍认为TNL基因可能在单子房中完全丢失的观点相反[19,40,45]。用PfamScan进一步分析这9种蛋白[46],分析结果表明,它们中没有一个含有预测的TIR结构域(PF01582)。分析还表明,其中8个含有截断的NB-ARC结构域(PF00931),其长度在63 ~ 271个氨基酸之间(Pfam中NB-ARC结构域的全长为288个),其余1个(Traes_3B_706A56165)被PfamScan注释为神经酰胺酶家族蛋白。因此,在小麦中预测到的有限数量的TNL蛋白可能是由于NLR-Parser注释错误导致的截断NB-ARC结构域的存在。此外,在拟南芥和小麦中,分别有10.4%和11.6%的NLR蛋白不能被分类为CNL和TNL, NLR- parser将其指定为“N/A”。
的开始使用InterProscan对小麦蛋白质组进行结构域分析后,仅根据START (IPR002913和IPR005031)和/或START样结构域(IPR023393)的存在来预测家族基因。47]。从这个分析中,我们检测到139和144开始拟南芥和小麦的基因。5个类群中,拟南芥和小麦的START类群最多,前者有75个(54%),后者有54个(37.5%)。HD-START在拟南芥中是第二大类,有21个成员(15.1%),而MINIMAL-START在小麦中是第二大类,有47个成员(32.6%)。HD-START-MEKHLA最小,各有7个成员,在拟南芥和小麦中分别占5.0%和4.9%。在早期的研究中,只有35种拟南芥开始基因被报道[48],但这些作者只使用了5种起始结构域蛋白(拟南芥GL2和ATML1,水稻ROC1和AAP54082,以及人类PCTP)作为BLAST搜索的查询。最近,satheth和同事[22]报告的开始鹰嘴豆(中投arietinumL.),利用拟南芥基因编码的START结构域(约200个氨基酸)AT5G54170(在我们的拟南芥中开始在鹰嘴豆基因组序列数据库的TBLASTN检索中,以基因列表(gene list)作为查询序列。据我们所知,目前还没有关于小麦START蛋白基因组规模分析的报道。
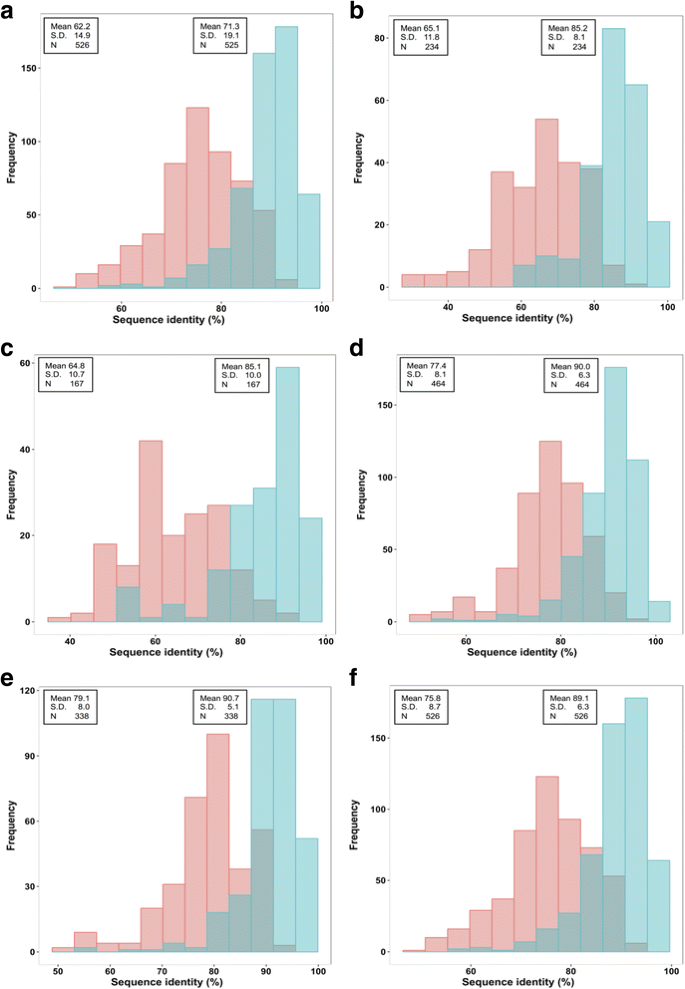
我们的预测主要是基于领域分析,他们的领域在附加文件中总结1.然而,我们将小麦中假定的ABC蛋白划分为不同亚科是基于与拟南芥和水稻中ABC蛋白的同源性(栽培稻).因此,我们分析了小麦、拟南芥和水稻之间ABC蛋白及其两种主要结构域(跨膜结构域或TMDs,以及胞质核苷酸结合结构域或NBDs)的序列相似性(图2)。1).该蛋白序列在小麦和水稻之间的同源性高于小麦和拟南芥之间。平均而言,这些ABC蛋白在小麦和拟南芥之间具有62.2%(标准差±14.9%)的序列一致性,在小麦和水稻之间具有71.3%(标准差±19.1%)的序列一致性。1).正如预期的那样,ABC结构域在小麦-拟南芥和小麦-水稻的比较中显示出更大的同源性,特别是NBDs,包括ABC转运蛋白样结构域(IPR003439), AAA+ atp酶结构域(IPR003593)和p环NTPase折叠(IPR027417)。例如,ABC转运蛋白1型跨膜结构域(IPR011527)在小麦和拟南芥之间的平均序列一致性为65.1%,在小麦和水稻之间的平均序列一致性为85.2%(图2)。1 b).同样,ABC-2转运体结构域(IPR013525)在小麦和拟南芥之间的平均同一性为64.8%,在小麦和水稻之间的平均同一性为85.1%。1 c).对于ABC转运蛋白样结构域(IPR003439),小麦和拟南芥的平均同源性增加到77.4%,小麦和水稻的平均同源性增加到90.0%。1 d).AAA+ atp酶结构域(IPR003593)的同源性在这些物种之间甚至更高,小麦与拟南南的平均同源性为79.1%,小麦与水稻的平均同源性为90.7%(图2)。1 e).最后,p环NTPase折叠(IPR027417)的同源性稍低,小麦与拟南芥的平均同源性为75.8%,小麦与水稻的平均同源性为89.1%(图2)。1 f).虽然小麦和水稻之间的ABC蛋白序列一致性大多高于小麦和拟南芥之间,但部分小麦ABC蛋白的顶击来自拟南芥。因此,我们使用拟南芥和水稻的ABC来对小麦的ABC蛋白进行分类。
小麦注释美国广播公司,NLR和开始基因
几乎所有人美国广播公司,NLR和开始基因在Ensembl Plants数据库中没有信息性的注释(只有模糊的注释,如未表征的蛋白质或假设的蛋白质)[49],我们使用BLAST的序列分析增强了它们的注释[50]。我们首先对GenBank和PRGdb中已知的植物抗病基因进行BLAST搜索[51,52]。145个ABC蛋白与已知的LR34蛋白具有不同程度的序列相似性,但只有2个具有97%以上的同源性(附加文件)2),表明各自的Lr34和Lr34-B基因是Traes_7DS_25B02BA46和Traes_4AL_603B6DC64在运用。Traes_7DS_25B02BA46是部分的,全长LR34编码只有799个氨基酸(aa)而不是1401个氨基酸(附加文件2).在Ensembl release 33中,我们找到了完整的长度Lr34基因(TRIAE_CS42_7DS_TGACv1_621754_AA2025300)编码1401 aa的蛋白。我们的注释Lr34和Lr34-B通过对Ensembl中LR34和LR34- b蛋白的多序列比对(MSA)以及GenBank中已知的抗性(accession: ACN41354)和易感(ADK62371) LR34蛋白的多序列比对(MSA)证实了这一结论。2).有趣的是,MSA表明,在Ensembl中推定的LR34蛋白(第31版中的Traes_7DS_25B02BA46和第33版中的TRIAE_CS42_7DS_TGACv1_621754_AA2025300)代表了LR34的抗性版本。此外,推测的LR34- b (Traes_4AL_603B6DC64)蛋白与LR34的序列一致性约为97%2),与先前的研究一致[15]。相比之下,在NLR和START家族中,没有基因与已知锈病相关蛋白的序列一致性超过90%,因此不太可能代表相同的基因。例如,NLR家族的Traes_1DS_5FF8D9E2D与GenBank中已知的抗条锈病蛋白YR10的同源性为85% (accession: AAG42168)。在START家族中,只有一个激酶-START结构域蛋白的序列来自t . dicoccoides与Traes_7DL_A330C0F90在蛋白序列水平上的最高同源性为~63%(附加文件2).相对较低的序列一致性可能是由于(i)使用中国春(CS)进行基因组测序和不同的小麦品种进行基因分离;(ii)增加了小麦的等位基因多样性R小麦品种间的基因。例如,Lr1克隆自Glenlea [7),Lr10从撒切尔夫人Lr10[8]。相反,当使用相同的小麦基因型进行基因组测序和基因克隆时,有可能找到完美的序列匹配。到目前为止,我们找到的唯一例子是Lr34其测序和克隆基于CS [6],其蛋白序列与我们在本研究中标注的推定LR34完全一致(图2)。2;额外的文件2).
Genbank中抗性和易感Lr34-D和Ensembl中推测的Lr34-D和Lr34-B的蛋白序列比对ACN41354:抗性版本GenBank LR34-D;ADK62371: GenBank LR34-D敏感性版本;Traes_7DS_25B02BA46: LR34-D in Ensembl release 31 (partial);TRIAE_CS42_7DS_TGACv1_621754_AA2025300: LR34-D在Ensembl发布33(全长)。Traes_4AL_603B6DC64: LR34-B在Ensembl发布31。蛋白质长度显示在每个标识符的斜杠(' / ')之后。箭头LR34res-D中546位的苯丙氨酸残基(Phe/F)缺失,634位的酪氨酸(Y)被组氨酸(His/H)取代。为了清晰起见,删除了序列比对的开始和结束区域
由于许多拟南芥基因已被表征,特别是ABC家族,其主要功能已广为人知[21,38,53,54,55(摘要见附加文件)3.表S1),然后我们根据这些基因在拟南芥基因组中的最佳位置对它们进行了注释(附加文件)2).例如,enmebl中假定的LR34被注释为ATPDR5 (Pleiotropic Drug Resistance 5, AT2G37280,也称为ABCG33或ABC转运体G家族成员33)。同样,LR34-B (Traes_4AL_603B6DC64)被注释为ATPDR9 (Pleiotropic Drug Resistance 9, AT3G53480,也称为ABCG37或ABC转运体G家族成员37)。因此,该注释可以利用模式植物中的知识来帮助选择小麦分子表征的候选基因。
的分布美国广播公司,NLR和开始小麦3个基因组中的基因及其与抗叶锈病qtl的物理接近性
的美国广播公司我们在本研究中预测的基因中,分别有165、189和172个来自A、B和D基因组(表2)1).为NLR其中370个(CNL: 320, TNL: 3, N/A: 47), 395个(CNL: 346, TNL: 3, N/A: 46)和352个(CNL: 313, TNL: 3, N/A: 36)来自A、B和D基因组。为开始family: 45 (START: 15, HD-START: 3, HD-START- mekhla: 2, MINIMAL-START: 16, OTHER: 9), 51 (START: 20, HD-START: 4, HD-START- mekhla: 2, MINIMAL-START: 16, OTHER: 9), 48 (START: 19, HD-START: 4, HD-START- mekhla: 3, MINIMAL-START: 15, OTHER: 7)分别来自A, B和D基因组。的美国广播公司,NLR和开始基因均匀分布在三个小麦基因组(A、B和D)上,从不显著的卡方(χ2)测试单个子家族类。
从T3数据库下载的两个数据集中,每个数据集有2500个抗叶锈病qtl [37],我们保留了1090和1542有效(P幼苗和成虫抗性qtl分别≤0.01)。在这两个数据集中,共使用了1809个独特的SNP标记(附加文件4),其中451个位点存在于幼苗和成虫的qtl中(附加文件)3.图S1A)。因为共有570个用于幼苗和成虫抗性的snp(代表391个独特标记)有未知(UNK)染色体(附加文件)3.(图S1B和C),我们将它们的侧翼序列映射到小麦基因组中,以确定这些snp是否位于本研究分析的ABC、NLR和START基因或其邻近区域(5 '转录起始位点[TSS]上游5 Kb或3 '转录终止位点[TTS]下游5 Kb至预测基因)。
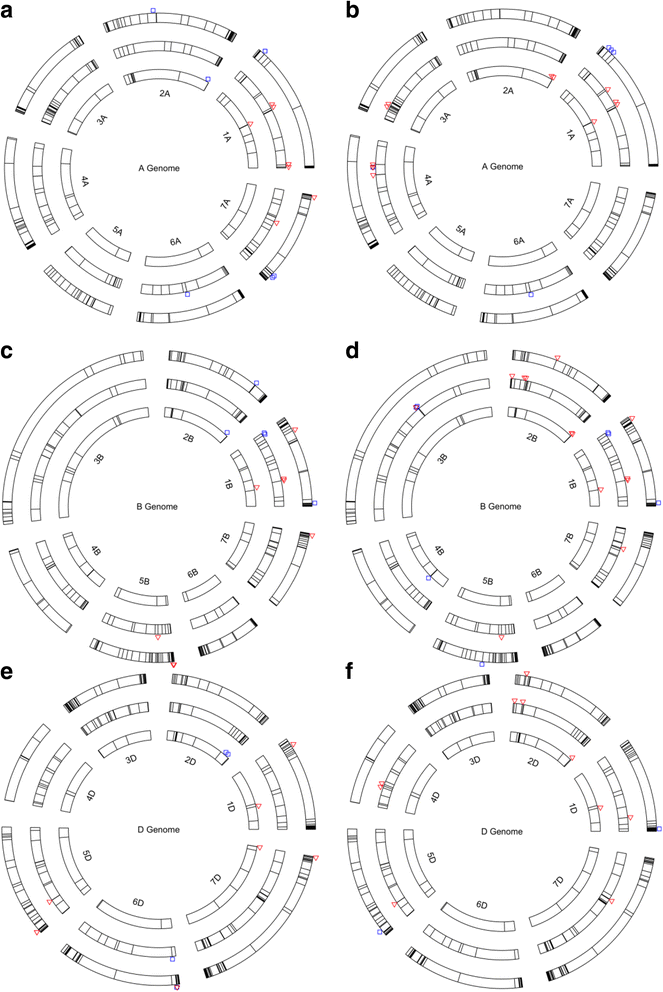
预测的美国广播公司,NLR和开始在预测基因的编码区或邻近区域(上游或下游5kb)内的基因和幼苗和成体抗性snp被定位到三个小麦基因组(A、B和D)中的单个染色体上(图2)。3.).指定的Lr以前在每条染色体上绘制的基因也显示在图3的外层。3.以表明它们与抗叶锈病候选基因和snp的物理亲和力。不幸的是,由于缺乏映射位置,这种亲和性非常粗糙Lr单个染色体上的基因。一些具有叶锈病snp的候选基因只在支架上有位置(一组有序序列组合,它们之间有间隙),因此无法在图中描述。3.(附加文件5).因此,我们将这些snp分为两类:染色体位置基因(WCP)和支架位置基因(WSP)。编码区存在的苗期和成虫抗性snp数目美国广播公司,NLR和开始基因及其邻域(5′端上游或3′端下游或5 Kb以内)表现出不同的模式(表2)2).例如,我们在基因或邻近区域发现了18个snp美国广播公司在幼苗中有39个基因,在成年植物中有39个。相反,在苗期和成虫抗性的基因区或邻近区域分别发现29个和14个snpNLR基因,分别。相比之下,START基因或它们的邻域包含的幼苗和成虫抗性的QTL snp数量大致相同。
的分布美国广播公司,NLR和开始六倍体小麦基因组A、B和D基因组抗叶锈病基因和SNP标记。圆圈按照每个家庭的大小排列:内圈、中圈和外圈开始,美国广播公司和NLR基因。一个:幼苗A基因组;b:成人A基因组;c:幼苗B基因组d:成人B基因组;e:幼苗D基因组;f:成人D基因组。红色的△表示基因内的标记;蓝色的□表示基因上游5kb(转录起始位点或TSS)或下游5kb(转录终止位点或TTS)的标记。7个snp重叠,因为它们在同一染色体上的物理距离很近,见附加文件5获取详细信息。我们没有显示指定的叶锈(Lr)染色体图谱上的幼苗抗性和成虫抗性基因,列示如下:Lr59;2:Lr11,Lr17a,Lr17b,Lr37,Lr38,Lr45,Lr49;3:Lr63;4:Lr28;5: -;6:Lr56,Lr62,Lr64;7:Lr20,Lr47;1 b:Lr33,Lr44,Lr46, Lr51, Lr55;2B: Lr13、Lr16、Lr23、Lr35、Lr50、Lr58;3 b: Lr27;4B: Lr12, Lr25, Lr30,Lr31,Lr48;5 b:Lr18,Lr52;6 b:Lr3a,Lr3bg,Lr3ka,Lr9,Lr36,Lr53,Lr61;7 b:Lr14a,Lr14b;1 d:Lr21,Lr42,Lr60;2 d:Lr2a,Lr2b,Lr2c,Lr15,Lr22a,Lr22b,Lr39,Lr54;3 d:Lr24,Lr32;4 d: -;5 d:Lr1,Lr57;6 d: -;7 d:Lr19,Lr29,Lr34
的非编码和邻近区域的snp被排除在外美国广播公司和NLR基因如表所示2,我们获得了位于编码区并导致这些基因家族氨基酸变化的SNPs子集(表2)3.).这些编码snp是上述小麦幼苗和成体抗叶锈病qtl的标记。的编码区非同义snp的数目美国广播公司和NLR在幼苗和成株中,基因表现出不同的模式。在美国广播公司在成虫抗性基因中发现17个错义snp,在幼苗抗性基因中发现5个错义snp。相反,在NLR我们只预测了5个非同义snp对成虫抗性和6个snp对幼苗抗性(表2)3.;额外的文件6).这些候选基因在QTL数据中具有非同义snp,可以用于进一步的研究,以研究它们在疾病中的功能作用Lr小麦的基因。
美国广播公司,NLR和开始与抗锈病非snp分子标记相关的基因
利用99个与指定锈病基因连锁的非snp分子标记的引物序列,鉴定出相应的候选锈病基因美国广播公司,NLR和开始小麦的基因。我们总共在这些标记附近发现了75个rust和Ensembl基因组合,平均物理距离约为1.7 Mb7).其中,8个基因位于距离锈病相关标记不到100 Kb的位置,包括2个、5个和1个位于锈病相关标记的基因美国广播公司,NLR和开始家庭,分别(表4).例如,我们发现gwm350标记只有293 bp到Traes_4AL_7AFBA6334.因此,这个基因可能是SrND643一种暂时指定的茎锈病基因,赋予对几种Ug99品种的抗性页面表[56]。此外,Lr34(Yr18 / Bdv1 Pm38 / Ltn1)与Traes_7DS_25B02BA46,距离为1212bpcssfr12189bp到cssfr2,两个已知的标记Lr34[13,57]。还有图。2(上述MSA),该结果进一步支持Traes_7DS_25B02BA46作为Lr34,展示了生物信息学分析在发现抗锈病基因功能表征中的有用性。
基因表达
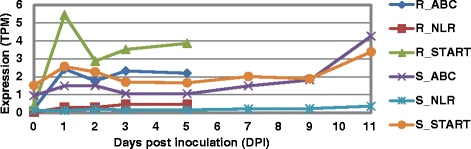
假定者的表达差异美国广播公司,NLR和开始抗病和易感小麦植株在不同侵染时间点(接种后几天或dpi)的基因见附加文件8.我们的分析显示NLR在所有时间点上,抗性和易感植物的基因都接近于零,在相同的dpi下,抗性植物的表达量仅略高于易感植物(图2)。4).相比之下,我们观察到在感染早期(≤5 dpi),抗性小麦植株中ABC和START基因的表达水平高于易感植株,但在感染后期(11 dpi),易感植株的表达量相似。这与图1中的发现是一致的。3.Dobon等。[27],其中鉴定出7个宿主基因簇,其中包括编码ABC转运蛋白的基因的一个簇(簇III)在11 dpi时达到峰值表达。
然而,这个结果需要小心解释。抗性和易感植物可能具有非常不同的遗传背景(即与其他抗锈病基因相比)Yr5因为它们是不同的品种,但不是等基因系。在携带抗叶锈病基因的小麦品种撒切尔(Thatcher)及其近等基因系(NIL)中进行了类似的基因表达分析Lr9(撒切尔Lr9)或Lr10(撒切尔Lr10)(抵抗)[58,59],但两者都使用基于EST(表达序列标签)的方法。这些分析表明,一些基因在抗性品种中表达水平较高,包括一个美国广播公司转运基因,而几个NLR在感染的易感和抗性样品中均有基因表达Ptr.此外,抗锈基因诱导的基因表达也发生了变化Lr34 / Yr18使用Affymetrix小麦基因芯片芯片在小麦NILs中进行了报道[60],该研究发现了与耐药性相关的上调和下调基因。
讨论
本研究采用小麦蛋白质组结构域分析,共预测了1787个基因,其中526个编码ABC蛋白,1117个编码NLR蛋白,144个编码START蛋白1,无花果。3.).我们能够识别出一些位于蛋白质编码区域的snp,并导致预测的氨基酸变化美国广播公司,NLR和开始基因或它们的邻域(5 '端上游5kb或3 '端下游5kb)。共有59个snp被鉴定为苗期叶锈病抗性qtl的标记,67个snp被鉴定为成虫期叶锈病抗性qtl的标记2;无花果。3.).存在的成虫抗性snp的数量美国广播公司在成虫期,基因或邻近基因的抗苗SNP数量增加了一倍以上,而与抗苗基因相关的SNP变异则相反NLR基因(表2).这似乎与预期一致[10]表明,非种族特异性、成虫抗锈病的qtl更有可能与候选基因共享基因组区域美国广播公司(和开始)家族,而幼苗中特定种族的抗锈病基因更有可能与候选基因共享基因组区域NLR家庭。
这一研究代表了对三个基因家族中候选基因的分类以及它们与小麦叶锈病抗性snp(因此qtl)的染色体物理亲和力的重要一步。这些信息将为进一步在分子水平上表征qtl及其与基因的功能关系提供有价值的信息美国广播公司,NLR和开始基因。我们已经能够记录我们预测的氨基酸变化,这是由于编码区单snp(点突变)的改变美国广播公司,NLR和开始基因(图。2;表格3.).我们的分析是基于最近的报道,两个氨基酸的变化导致了由抗性和敏感性等位基因编码的蛋白质的差异Lr34 (6,15),Lr67年(61]。然而,我们基于点突变的预测必须被视为初步结果,因为抗性基因的拷贝数变异(CNV)等结构变异可能对疾病和其他生物胁迫具有更大的影响[62]。进一步的复杂性来自于假定的表达的时间变化美国广播公司,NLR和开始亲和性(易感)和不亲和性(抗性)小麦锈病相互作用中的基因在我们的RNA-Seq分析中得到了证明。4).因此,基于点突变的预测,例如我们的预测,只能作为一个起点,为抗性基因的最终克隆提供更彻底的分子和功能表征。
我们的预测表明,种族特异性的数量NLR基因(R几乎是非种族特异性基因(美国广播公司和开始基因)。这似乎与大多数人的现象一致Lr目前在小麦中发现的基因仅对单一品种的锈病有效。正如Krattinger等人所言。[10]指出,这一现象也适用于小麦和其他谷物对其他疾病的抗性。使用这种种族歧视的问题Lr在小麦品种中部署的基因是,当叶锈病病原体中出现新的、毒性更强的小种时,它们很快就失效了[63]。因此,小麦育种家和病理学家一直致力于发现、鉴定和利用非种族特异性基因来获得持久抗性。然而,几十年的遗传和育种研究只能在小麦中识别出有限数量的具有持久和广谱抗锈病能力的基因,包括Lr34 / Yr18 Sr57 / Pm38,Lr46 / Yr29 / Pm39,Lr67 / Yr46 Sr55 / Pm46,Lr68,Lr75,和Yr36[5,6,15,16,61,64,65]。到目前为止,Lr34 / Yr18 Sr57 / Pm38(或者只是Lr34(以下简称)是位于小麦染色体7D上最具特征的多病原体抗性基因[11]。不幸的是,我们在T3数据集中没有发现任何已知的抗叶锈病snp [34的编码区域Lr34基因或其邻域在上下游5kb范围内。当然,小麦9k和90k SNP芯片几乎不足以覆盖大而复杂的小麦基因组,特别是D基因组(参见图2)。3.).随着国际上对小麦基因组多样性测序的不断努力,这些SNP变异在小麦基因组中的确切物理位置和功能美国广播公司基因(表2)将进一步澄清和表征。
也许有人会问为什么Lr67 / Yr46 Sr55 / Pm46(或者只是Lr67)不包括在本研究中,因为它是另一个在分子水平上被充分表征的种族非特异性基因Lr34.原因是它不编码ABC转运蛋白,而是编码己糖转运蛋白,并对所有三种小麦锈病病原体和白粉病具有类似的,但略有降低的部分抗性[62]。就像在Lr34抗性蛋白(LR67res)与敏感性蛋白(LR67sus)仅通过两个氨基酸替换(Arg144Gly和Leu387Val)进行区分[62]。LR67的己糖转运活性已在酵母(酿酒酵母)缺乏葡萄糖摄取能力的突变体[62,66]。的5个蛋白质序列的初步分析Lr67[62],包括A、B、D染色体上的三个同源物,以及GenBank的抗性/敏感性版本(收录:ALL26327、ALL26328、ALL26329、ALL26330和ALL26331)显示,它们与本研究分析的三个家族蛋白(ABC、NLR和START)的同源性都不高(e值>1 × 10)−5序列一致性<36%)。由于LR67属于糖转运蛋白(STP)家族,使用主要促进剂糖转运结构域(InterPro加入IPR005828,上述5种LR67蛋白均存在该结构域)对STP蛋白进行全基因组搜索,发现了261种STP蛋白(e值<1 × 10)−5小麦。该STP数据集以及ABC和其他可能的转运蛋白家族可以在未来的研究中进一步研究它们与小麦和其他谷物中对多种病原体部分抗性的qtl的物理关系。
本研究的重点是注释和表征之间的物理关系美国广播公司,NLR和开始抗叶锈病基因和qtl。茎锈病和条纹锈病(黄色)也可以采取类似的措施。截至2017年2月这项工作完成时,T3数据库中只有少量的茎和条锈病抗性QTL数据可用[37],可用于此类注释和表征;特别是,幼苗和成虫抗性qtl的可用性不平衡,使得有效的比较评估更加困难。随着茎锈病和条锈病的QTL数据越来越多,可以想象,基因家族和QTL之间的物理关系模式将被确定,就像我们在叶锈病中观察到的那样。
我们的序列分析在很大程度上受到小麦基因组组装草图片段状态的影响[3.]。在本研究使用的小麦基因组的Ensembl release 31中,21条小麦染色体共有317,977个组装支架序列。支架平均长度为20389 bp (sd±222 7384 bp),中位数为3800 bp,最大值为774,434,471 bp。由于对组装质量的类似担忧,我们选择不使用集成了短rna序列和全长cDNA序列的Ensembl release 33(2016年10月的最新版本)[42但根据我们的评估,它似乎比第31版更加分散。例如,第33版小麦基因组包含735,943个超序列序列(第31版为317,977个),平均超序列长度为18,245 bp (sd±41,822 bp),中位数为2431 bp,最大值为823,974 bp。小麦基因组的片段组装也可能影响了我们使用引物序列对小麦基因组序列进行BLAST搜索,这可能是为什么只有7个引物序列的主要原因美国广播公司,NLR和开始基因在100Kb内鉴定为抗锈病qtl标记(表2)4、附加文件7).其他基于类似片段组合的研究甚至不能确定候选基因,只能确定抗病和其他农艺性状的一些基因组区域。其中两项研究是:(i)利用计算机绘制DArT标记序列,以鉴定对三种锈病具有抗性的基因组区域[67]和(ii)用于鉴定小麦收获前发芽(PHS)抗性候选基因和区域的小麦90k芯片的单核苷酸多态性侧翼序列[68],但候选基因是从短柄草(Brachypodium distachyon)和水稻基因组通过比较作图。
除了美国广播公司,NLR和开始在本研究中分析的基因中,其他家族的基因也可能与抗锈病有关。我们已经提到过STP转运蛋白家族中的基因,比如Lr67.其他基因包括编码异常激酶、受体样激酶(RLKs)和受体样蛋白(rlp)的基因[69],以及涉及细胞壁生物合成和代谢的基因,如半胱氨酸蛋白酶、苯丙氨酸解氨酶、质膜三磷酸腺苷酶和查尔酮合成酶[58]。当病原体攻击宿主时,宿主的第一道防线是通过细胞表面模式识别受体(PRRs)的分泌,这种防御诱导的蛋白质可能被特定的病原体效应物识别和克服。大多数有特征的PRRs是RLKs或rlp [70,71,72]。最近开发的一种名为rgauury的工具[73]重点研究了这些蛋白和NLR蛋白家族的分类。然而,我们的研究对小麦叶锈病抗性相关的三个主要蛋白质家族有不同的关注,这些家族含有适合大规模分析的不同结构域。
当然,随着国际上对更深入的小麦基因组测序的不断努力,我们为三个基因家族确定的候选基因最终将在单个染色体上的准确位置进行物理映射。换句话说,图。3.将进行重大澄清和更新。这种更新将使小麦育种家和病理学家能够使用具有良好特征的小麦美国广播公司,NLR和开始基因进行指定的分子表征Lr抗锈病育种中的基因。而美国广播公司和开始基因最好按上述理由使用,单独使用时,它们的抗性只是部分的,在高疾病压力下是不足的。通过将这些非种族特异性基因与种族特异性基因结合,获得了强大而持久的抗性NLR基因,比如“老两个复”(74]。然而,对其他组合基因及其在复合体中的相互作用知之甚少。因此,我们希望未来的研究能够探讨在人类中非种族特异性基因是如何形成的美国广播公司或开始基因家族与弱者和强者相互作用R基因NLR在分子水平上利用候选基因对三个基因家族进行了鉴定和表征。
结论
在这项研究中,我们预测了假定的美国广播公司,NLR和开始对小麦六倍体基因组中的基因进行了综合分析,并与现有遗传资源进行了相关的抗锈病性分析,包括绘制了图谱Lr基因、snp和其他分子标记。我们的分析表明美国广播公司(和开始)基因更有可能与非种族特异性的成虫抗性qtl共享基因组区域,而NLR基因更有可能与通常在苗期表达的种族特异性抗性qtl共享基因组区域。候选基因,如含有一个或多个错义编码snp的基因,可以在未来的研究中进行测试。随着对小麦基因组组装和注释的不断改进,我们的生物信息学分析可以帮助鉴定小麦抗锈病育种的新基因。
方法
小麦基因组中ABC、NLR和START基因的预测
小麦蛋白质组(第31版)从FTP存档站点(ftp://ftp.ensemblgenomes.org/pub/release-31/plants/fasta/triticum_aestivum/pep/Triticum_aestivum.IWGSC1+popseq.31.pep.all.fa)的综合植物[49]。该数据库共包含100,344个预测蛋白序列。使用这些蛋白质序列,我们使用独立软件InterProScan(版本5.21-60.0,64位)预测每个蛋白质的结构域[47],安装在CentOS(发行7.2)Linux系统上,具有8核CPU (1600 MHz)和32 Gb RAM。InterProscan e值截止值设置为1 × 10−5[47]。从InterProscan输出文件中,蛋白家族数据库Pfam中存在PF00005 (ABC转运蛋白的atp结合域)[46]用于选择候选ABC蛋白。然后根据这些候选物对拟南芥和拟南芥中已知ABC蛋白的最佳BLASTP命中,将其分为8个家族(A, B, C, D, E, F, G和I)栽培稻(大米),这些都是从文献中收集来的[54,75]。G家族进一步分为Gwbc (White Brown Box)和Gpdr (Pleiotropic Drug Resistance)。对于START家族,我们使用START结构域(InterPro: IPR002913和IPR005031)或START-like结构域(IPR023393)作为特征,并将假定的START蛋白分为五类:MINIMAL-START, START, HD-START, HD-START- mekhla和OTHER。该分类方案与以往研究报告的分类方案相似[22,48]。一个MINIMAL-START蛋白只包含一个START或START-like结构域,而一个START蛋白包含多个至少有一个START或START-like结构域的结构域。此外,多结构域START蛋白如果包含同源结构域样结构域(IPR009057)则被命名为HD-START,如果还包含MEKHLA结构域(IPR013978)则被命名为HD-START-MEKHLA。使用NLR-Parser工具预测NBS-LRR抗性蛋白[76这个过程分为两步。首先,使用MEME(4.9.1版)套件中的Motif Alignment and Scanning Tool (Motif Alignment and Scanning Tool, Motif Alignment and Scanning Tool)对整个小麦蛋白质组进行了MEME格式的Motif (MEME .xml)扫描[77]。其次,对MAST输出(MAST .xml)进行解析,利用NLR-Parser将NBSLRR基因分为CNL (COIL-NBS-LRR)、TNL (TIR-NBS-LRR)和N/A三类。
QTL SNP标记的计算机定位美国广播公司,NLR和开始小麦染色体上的基因
从Triticeae Toolbox (T3)数据库的“GWAS Results”(GWAS Results)中提取两个数据集,每个数据集含有2500个幼苗期和成虫期抗叶锈病数量性状位点(qtl)。https://triticeaetoolbox.org/wheat/qtl/qtl_report.php) [37]。通过在“类别”栏中选择“生物胁迫”,在“性状”栏中点击相应的成年期或幼苗期叶锈病性状,T3将显示标记(包括Infinium 9 K、Infinium 90 K和GBS酶切位点)与T3数据库中单个试验性状(单个位置或接种类型)之间关联的GWAS分析结果。使用rrBLUP GWAS软件包进行GWAS分析[78]用于单个试验或所有试验的组合分析,通过将占环境关系矩阵方差5%以上的主成分作为固定效应纳入混合模型分析,对基因型-环境相互作用效应进行调整。为了更好地控制这些初步QTL数据集中的假阳性率,我们应用了显著性水平为P≤0.01,允许在幼苗中保留1090个qtl,在成株中保留1542个qtl用于后续分析(另附文件)4).
上述GWAS结果的SNP标记序列在T3和CerealsDB中得到[79]。为了预测它们的功能后果,这些snp首先被转换成VCF(变体调用格式),并用VEP注释[80]。然后,为了更好地估计每个SNP的基因组位置及其与预测基因的接近程度美国广播公司,NLR和开始通过与小麦基因组组装(Triticum_aestivum)比对,我们绘制了每个SNP侧面的序列(去除' / ',B等位基因和周围的方括号)。IWGSC1 + popseq.31.dna_sm. topplevel .fa)在BLAST+包中使用BLASTN(2.5.0+版本)[50]。这个组装是由Ensembl Plants从全基因组霰弹枪测序数据中生成的[2],以染色体为基础的草图序列[3.]和POPSEQ(种群测序)数据[81]。这些基因的染色体位置基于Ensembl注释文件(Triticum_aestivum)。IWGSC1 + popseq.31.gff3), GFF3 (General Feature Format)格式。这两个文件都是从Ensembl FTP存档网站(ftp://ftp.ensemblgenomes.org/pub/release-31/plants/).由于每个侧翼序列通常匹配多个基因组区域,我们使用e值、比对身份和查询覆盖率作为标准来保留前三个候选序列,这些候选序列通常代表三个小麦基因组中的一个命中。
对于位于基因组区域覆盖的snp美国广播公司,NLR和开始通过将编码区基因的侧翼序列与从Ensembl下载的编码序列(CDS)比对来识别编码区基因。然后,我们将每个SNP标记的两个等位基因与Ensembl CDS相同位置的等位基因进行比较,并确定Ensembl等位基因(作为参考)是否与SNP位点的A或B等位基因相同,或者在某些情况下与两个等位基因都不同。为了预测氨基酸的变化,我们用A或B等位基因替换了Ensembl的CDS,使用标准密码子表将新生成的CDS翻译成蛋白质序列。因此,如果Ensembl参考等位基因与SNP位点的A或B等位基因匹配,则生成两个蛋白质序列,否则每个SNP标记翻译三个蛋白质序列。为了验证这一过程,我们利用了dna中已知的SNP(和缺失)Lr34以及耐药和易感的LR34蛋白[6,15]。然后,我们发现每个含有不同SNP等位基因的CDS翻译的每个蛋白质对应位置的氨基酸差异。
基于小麦非snp标记序列的候选基因预测
我们从Liu等人那里收集了99个非snp标记,这些标记与小麦抗锈病表现出显著的相关性(LOD评分≥3.0)。57]和其他来源。这些标记的引物序列在GrainGenes [82], MASWheat (http://maswheat.ucdavis.edu/protocols/stemrust/)和文献(附加文件7).与上述SNP侧翼序列分析类似,我们检索了小麦基因组序列组合(Triticum_aestivum)的引物序列。IWGSC1 + popseq.31.dna_sm.top - level.fa)使用BLASTN [50来识别这些标记所覆盖的基因组区域。每个引物查询通过将正向引物和反向引物连接起来生成,并在它们之间插入5个“N”字母作为间隙。由于每个引物对的长度较短(通常小于40 bp), BLASTN程序针对短于50个碱基的序列进行优化[50],单词大小设置为7,宽松的e值为100。为了比较标记和候选基因的染色体位置,我们还使用了Ensembl注释文件(Triticum_aestivum)。IWGSC1 + popseq.31.gff3)。
转录分析
Dobon等。[27最近发表了抗性品系的RNA-seq时间过程数据集(Avocet-Yr5)和一个接种了黄锈病菌的敏感小麦品种(Vuka)柄锈菌striiformisF. sptritici (太平洋标准时间).菌株的锈病接种量为87/66太平洋标准时间对耐药菌株(牛油果)是无毒的Yr5),但对敏感品种(Vuka)有毒性。我们分析了123,532个小麦转录本的表达值(转录本Per Million reads或TPM)(基于Ensembl Plants [49]),摘自Dobon等人的补充表S20和S21。[27]。我们认为美国广播公司,NLR和开始从这些寄主转录本中提取基因,并分析了它们在接种后不同时间在小麦抗感植株中的表达谱。表达谱上的原始数据包含Avocet-的5个时间点(0、1、2、3和5 dpi)Yr5, Vuka为8(0、1、2、3、5、7、9和11 dpi)。然后,我们提取假定的TPM值美国广播公司,NLR和开始基因,剔除所有dpi为零的基因。对于这三个基因家族中的每一个假定基因,我们计算了一个倍数变化,即抗性宿主中五个表达值的总和除以易感宿主中的总和。此外,我们确定美国广播公司,NLR和开始仅在抗性宿主或易感宿主中表达的基因。
缩写
- 美国广播公司(ABC):
-
磷酸腺苷磁带
- cd:
-
编码序列
- DPI:
-
接种后天数
- NLR:
-
核苷酸结合位点/富亮氨酸重复序列(NBS-LRR)
- Pdr:
-
多效耐药
- QTL:
-
数量性状位点
- SNP:
-
单核苷酸多态性
- 开始:
-
类固醇急性调节(StAR)蛋白相关脂质转移域
- STP:
-
糖转运蛋白
- UTR:
-
翻译区
- 白细胞:
-
白棕复合体
参考文献
- 1.
张建军,张建军,张建军,等。面包小麦3B染色体的结构和功能分配。科学。2014;345(6194):1249721。
- 2.
Brenchley R, Spannagl M, Pfeifer M, Barker GLA, D'more R, Allen AM,等。面包小麦全基因组霰弹枪测序分析。大自然。2012;491(7426):705 - 10。
- 3.
国际小麦基因组测序联盟。基于染色体的六倍体面包小麦(小麦)基因组。科学。2014;345(6194):1251788。
- 4.
李建军,李建军,李建军,李建军,李建军,等。小麦种质资源的研究进展。2013.
- 5.
李建军,李建军,李建军,等。小麦种质Lr34/Yr18抗锈病区分析。作物科学,2008,48(5):1841-52。
- 6.
Krattinger SG, Lagudah ES, Spielmeyer W, Singh RP, Huerta-Espino J, McFadden H,等。一种假定的ABC转运体赋予小麦对多种真菌病原体的持久抗性。科学。2009;323(5919):1360 - 3。
- 7.
刘建军,刘建军,刘建军,等。面包小麦抗叶锈病基因Lr1 (小麦L.)是psr567基因家族的成员。植物化学学报,2009;32(1):391 - 391。
- 8.
Feuillet C, Travella S, Stein N, Albar L, Nublat A, Keller B.六倍体小麦抗叶锈病基因Lr10的定位分离(英文)小麦l .)基因组。科学通报,2003;30(5):563 - 563。
- 9.
黄磊,李伟林,李永祥,李永祥,李永祥,李永祥。从面包小麦大、多倍体基因组中克隆抗叶锈病基因Lr21。遗传学。2003;164(2):655 - 64。
- 10.
Krattinger SG, Sucher J, Selter LL, Chauhan H,周波,唐明志,等。小麦耐多病基因Lr34在水稻中具有部分稻瘟病抗性。植物学报,2016;14(5):1261-8。
- 11.
李建平。普通小麦叶片抗锈病基因与茎秆抗锈病染色体7D抑制基因的关联。基因组。1987;29(3):467 - 9。
- 12.
李建军,李建军,李建军,等。小麦Lr34基因提供了对大麦多种真菌病原菌的抗性。植物学报,2013;11(7):847-54。
- 13.
Lagudah ES, Krattinger SG, Herrera-Foessel S, Singh RP, Huerta-Espino J, Spielmeyer W,等。小麦抗多种真菌病原菌基因Lr34/Yr18/Pm38的基因特异性标记理论与应用,2009;19(5):889-98。
- 14.
杨建军,杨建军,李建军,等。小麦抗白粉病基因与抗叶锈病和条锈病耐久基因Lr34/Yr18在7D染色体短臂上共聚。理论与应用,2005;31(4):731 - 731。
- 15.
Krattinger SG, Lagudah ES, Wicker T, Risk JM, Ashton AR, Selter LL等。Lr34多病原体抗性ABC转运体:六倍体小麦和其他禾本科植物同源和同源基因的分子分析。植物学报,2011;36(3):394 - 394。
- 16.
傅德龙,uway C, Distelfeld A, Blechl A, Epstein L,陈晓明,等。一种激酶- start基因赋予小麦条锈病的温度依赖抗性。科学。2009;323(5919):1357 - 60。
- 17.
福冈S, Saka N,古贺H, Ono K, Shimizu T, Ebana K,等。一种含有脯氨酸的蛋白质的功能丧失赋予了水稻持久的抗病能力。科学。2009;325(5943):998 - 1001。
- 18.
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军等大麦mlo基因:一种新的植物抗病控制元件。细胞。1997;88(5):695 - 705。
- 19.
金俊,林俊杰,李宝伟,崔太平,吴淑琪,Ahmad R,等。植物界NB-LRR型抗性基因类似物(RGA)的全基因组比较生物化学学报,2012;33(4):385-92。
- 20.
van Ooijen G, van den Burg HA, Cornelissen BJC, Takken FLW。茄属植物抗性蛋白的结构与功能。植物化学学报,2007;45(5):463 - 467。
- 21.
姜杰,朴杰,崔海,Burla B, Kretzschmar T, Lee Y,等。植物ABC转运蛋白。《拟南芥学报》/《美国植物生物学学会》2011;9:e0153。
- 22.
Satheesh V, Chidambaranathan P, Jagannadham PT, Kumar V, Jain PK, Chinnusamy V, Bhat SR, Srinivasan R:鹰嘴豆跨膜START结构域蛋白的筛选、鉴定和表达分析[j]。中投arietinuml .)。植物信号行为2016年,11 (2):e992698。
- 23.
Schrick K, Bruno M, Khosla A, Cox PN, Marlatt SA, Roque RA等。植物和哺乳动物star相关脂质转移(START)结构域在调节转录因子活性中的共同功能中华生物医学杂志,2014;12:70。
- 24.
王忠,Gerstein M, Snyder M. RNA-Seq:一种革命性的转录组学工具。学报,2009;10(1):57-63。
- 25.
许桂荣,强MJ, Lacey MR, Baribault C, Flemington EK, Taylor CM: RNA罗盘:一种用于病原体和宿主转录组分析的RNA- seq数据集。PLoS One 2014,9(2).
- 26.
杨建军,杨建军,张建军,等。中国血吸虫病原与宿主双rna序列分析。微生物学报,2012;10(9):618-30。
- 27.
Dobon A, hunting DCE, Cabrera-Quio LE, way C, Saunders DGO。小麦与黄锈病的寄主-病原菌互作诱导了基因表达的时间协调波。BMC Genomics. 2016;17:380。
- 28.
苏利亚诺,罗耀,李建平,等。小麦抗叶锈病遗传结构的QTL meta分析。植物病理学。2015;105(12):1585 - 93。
- 29.
鲍嘉文P,劳斯MN, Bhavani S,安德森JA。春小麦群体RB07/MN06113-8成株抗Ug99茎锈病的QTL定位动物学报,2015;35(8):170。
- 30.
罗世文,何志华,何志强,何志强,何志强,何志强,何志强,何志强。小麦抗条锈病数量性状位点。理论与应用,2013;26(10):2427-49。
- 31.
刘建军,刘建军,刘建军,刘建军。小麦品种‘Pavon 76’耐久成虫抗Ug99的QTL鉴定Euphytica。2013;190(1):33-44。
- 32.
Zegeye H, Rasheed A, Makdis F, Badebo A, Ogbonnaya FC。合成六倍体小麦幼苗和成株抗条锈病的全基因组关联图谱。科学通报,2014;9(8):e105593。
- 33.
高丽丽,王晓明,赵晓明,高晓明。春小麦优良选育系幼苗与成株叶锈病抗性的全基因组关联研究。科学通报,2016;11(2):e0148671。
- 34.
Maccaferri M,张建林,Bulli P, Abate Z, Chao SM, Cantu D, Bossolini E,陈晓明,Pumphrey M, Dubcovsky J:全球六倍体春小麦对条锈病抗性的全基因组关联研究(英文)小麦l .)。G3-Genes genome Genet2015年,5(3): 449 - 465。
- 35.
Cavanagh CR, Chao S, Wang S, Huang BE, Stephen S, Kiani S,等。全基因组比较多样性揭示了六倍体小麦地方品种和栽培品种改良的多个选择目标。中国科学:自然科学版,2013;39(5):557 - 562。
- 36.
王思聪,王德宝,Forrest K, Allen A, Chao SM,黄be,等。利用高密度90000单核苷酸多态性阵列分析小麦多倍体基因组多样性。植物学报,2014;12(6):787-96。
- 37.
Blake VC, Birkett C, Matthews DE, Hane DL, Bradbury P, Jannink JL等。植物Genome-Us。2016:9(2)。
- 38.
王晓明,王晓明,王晓明,等。植物多效性耐药ABC转运蛋白家族的组成和功能。生物化学学报。2006;58(4):1123-30。
- 39.
Andolfo G, Ruocco M, Di Donato A, Frusciante L, Lorito M, Scala F,等。植物膜ABC转运体的遗传变异和进化多样化。植物学报,2015;15
- 40.
李鹏,林毅,王晓霞,刘志强,尤福明。植物抗病基因类似物(RGAs)。中华医学杂志,2015;16(8):19248-90。
- 41.
boktila D, Khalfallah Y, habachii - houimli Y, Mezghani-Khemakhem M, Makni M, Makni H.小麦NBS结构域编码抗性基因类似物的大规模分析。中国生物医学工程学报,2014;37(3):598-610。
- 42.
克拉维jo BJ, Venturini L, Schudoma C, Accinelli GG, Kaithakottil G, Wright J等。改进的异源六倍体小麦基因组的组装和注释鉴定了完整的农艺基因家族,并为染色体易位提供了基因组证据。基因工程学报。2017;27:885-96。
- 43.
于静,特林森,张峰,童超,黄健,程旭,等。芸苔属植物nbs编码基因的全基因组比较分析拟南芥.BMC Genomics. 2014;15:3。
- 44.
Meyers BC, Kozik A, Griego A, Kuang HH, Michelmore RW。拟南芥nbs - lrr编码基因的全基因组分析。植物学报。2003;15(4):809-34。
- 45.
塔尔·德克,亚历山大·HM。TIR-NBS-LRR基因在单子房中很少见:来自不同单子房目的证据。生物医学工程学报。2009;2:197。
- 46.
Finn RD, Coggill P, Eberhardt RY, Eddy SR, Mistry J, Mitchell AL,等。Pfam蛋白质家族数据库:迈向更可持续的未来。中国生物医学工程学报,2016;44(1):379 - 379。
- 47.
Jones P, Binns D, Chang HY, Fraser M,李文忠,mccanulla C,等。InterProScan 5:基因组尺度的蛋白质功能分类。生物信息学。2014;30(9):1236 - 40。
- 48.
施里克K,阮D, Karlowski WM, Mayer KFX。START脂质/固醇结合结构域在植物中被扩增,主要与同源结构域转录因子相关。中国生物医学工程学报,2004;5(6):441。
- 49.
Cunningham F, Amode MR, Barrell D, Beal K, Billis K, Brent S,等。运用2015年。核酸学报,2015;43(D1): D662-9。
- 50.
李建军,李建军,李建军,等。BLAST plus:架构和应用程序。生物信息学报,2009;10:421。
- 51.
李建军,李建军,李建军,李建军。基因库。核酸学报,2016;44(D1): D67-72。
- 52.
张建军,张建军,张建军,等。PRGdb 2.0:基于社区的植物r基因分析数据库模型核酸学报,2013;41(数据库版):D1167-71。
- 53.
意图。植物atp结合盒转运体。植物学报,2007;38(4):357 - 357。
- 54.
李建军,李建军,李建军,等。植物ABC蛋白-统一的命名法和更新的目录。植物科学进展,2008;13(4):151-9。
- 55.
李建军,李建军,李建军,等。植物ABC转运蛋白的研究进展。生物化学学报,2011;50:145-60。
- 56.
Basnet BR, Singh S, Lopez-Vera EE, Huerta-Espino J, Bhavani S, Jin Y,等。小麦抗茎锈病Ug99新基因SrND643的分子定位与验证植物病理学。2015;105(4):470 - 6。
- 57.
刘世义,陆家杰,白光华,Haley SD, Ibrahim AMH,薛群伟,等。硬冬小麦重要基因的分子标记。作物科学,2014,54(4):1304-21。
- 58.
Manickavelu A, Kawaura K, Oishi K, Shin IT, Kohara Y, Yahiaoui N,等。小麦锈病感、抗近等基因系基因表达比较分析。DNA学报,2010;17(4):211-22。
- 59.
Dmochowska-Boguta M, Alaba S, Yanushevska Y, Piechota U, Lasota E, Nadolska-Orczyk A,等。小麦等基因系抗褐锈病差异的病原调控基因。生物医学工程学报,2015;16:742。
- 60.
胡尔伯特,白杰,费勒斯JP,帕切科MG,鲍登RL。小麦抗锈病基因Lr34/Yr18近等基因系的基因表达模式植物病理学。2007;97(9):1083 - 93。
- 61.
Moore JW, Herrera-Foessel S, Lan C, Schnippenkoetter W, Ayliffe M, Huerta-Espino J,等。最近进化的己糖转运体变体赋予小麦对多种病原体的抗性。地理学报,2015;47(12):1494-8。
- 62.
李建军,李建军,李建军,等。植物基因组拷贝数多态性研究进展。理论与应用,2014;127(1):1 - 18。
- 63.
谷物基因组进化——乐高基因组的田园追求。当前意见和技术发展。1995;5(6):717-24。
- 64.
Herrera-Foessel SA, Singh RP, Huerta-Espino J, Rosewarne GM, Periyannan SK, Viccars L,等。Lr68:小麦抗叶锈病缓慢锈蚀的新基因。理论与应用,2012;24(8):1475-86。
- 65.
李建军,李建军,李建军,李建军。小麦抗叶锈病基因Lr75的克隆与鉴定。理论与应用,2017;130(1):1 - 12。
- 66.
怀特FF, Frommer W.破译持久抗性的一个R基因。地理学报,2015;47(12):1376-7。
- 67.
李洪辉,辛格S, Bhavani S,辛格RP, Sehgal D, Basnet BR,等。南亚流行小麦品种PBW343成株抗性的基因组关联鉴定植物科学,2016;7:1674。
- 68.
Cabral AL, Jordan MC, McCartney CA, You FM, Humphreys DG, MacLachlan R等。小麦收获前发芽抗性候选基因、区域和标记的鉴定(小麦l .)。植物学报,2014;14(4):334。
- 69.
陈建军,陈建军。水稻抗病性的分子遗传与进化。植物化学学报,2016;32(2):332 - 332。
- 70.
植物模式识别受体。免疫学杂志,2014;35(7):345-51。
- 71.
张建军,张建军,张建军,等。植物模式识别受体复合物的研究进展。植物学报,2012;15(4):349-57。
- 72.
李建军,李建军,李建军,等。植物细胞表面免疫受体复合物的研究进展。植物学报,2014;20(2):447 - 454。
- 73.
李鹏平,权晓东,贾国锋,肖军,Cloutier S,游调频。rgaury:植物抗性基因类似物(RGAs)全基因组预测的管道。生物医学工程学报,2016;17(1):852。
- 74.
Singh RP, Hodson DP, Jin Y, Lagudah ES, Ayliffe MA, Bhavani S,等。小麦茎锈菌新品种的出现和传播:对粮食安全的持续威胁和遗传控制的前景。植物病理学。2015;105(7):872 - 84。
- 75.
Andolfo G, Ruocco M, Di Donato A, Frusciante L, Lorito M, Scala F,等。植物膜ABC转运体的遗传变异和进化多样化。植物学报,2015;15:51。
- 76.
刘建军,刘建军,刘建军,刘建军。NLR解析器:快速标注植物NLR补语。生物信息学,2015,31(10):1665 - 7。
- 77.
贝利TL, Boden M, Buske FA, Frith M, Grant CE, Clementi L,等。模因套件:模因发现和搜索工具。中国生物医学工程学报,2009;37(6):591 - 591。
- 78.
Endelman简森-巴顿。R包rrBLUP基因组选择的岭回归和其他核方法。植物Genome-Us。2011; 4(3): 250 - 5。
- 79.
Wilkinson PA, Winfield MO, Barker GLA, Tyrrell S, Bian X, Allen AM,等。CerealsDB 3.0:资源扩展和数据整合。生物信息学报,2016;17:256。
- 80.
李建军,李建军,李建军,等。Ensembl变异效应预测器。中国生物医学工程学报,2016;17(1):122。
- 81.
Chapman JA, Mascher M, Buluc A, Barry K, Georganas E, Session A等。全基因组鸟枪法组装和固定面包小麦六倍体基因组。基因组生物学。2015;16:26。
- 82.
Carollo V, Matthews DE, Lazo GR, Blake TK, Hummel DD, Lui N,等。GrainGenes 2.0。小谷物社区的改良资源。植物生理学报,2005;39(2):643 - 651。
致谢
我们要感谢三位审稿人对早期初稿提出的批判性和建设性意见,以及胡志秋博士的有益讨论和他对图2的准备工作的帮助。3..
资金
这项研究是由日益增长的向前2 .艾伯塔省农林研究机遇与创新内部倡议
数据和材料的可用性
所有支持数据都可以在手稿及其附加文件中找到。
作者的贡献
构思和设计分析:RCY和YFP;进行分析:FYP和RCY;稿件写作:YFP、RCY;两位作者都阅读并批准了手稿的最终版本。
相互竞争的利益
作者宣称他们没有竞争利益。
发表同意书
不适用。
伦理批准并同意参与
不适用。
出版商的注意
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
作者信息
从属关系
相应的作者
附加文件
附加文件1:
综述了小麦ABC、NLR和START蛋白的家族分类及其对InterPro结构域的预测,以及各InterPro标识符的描述。(xlsx144kb)
附加文件2:
假定的注释美国广播公司,NLR和开始利用GenBank和PRDdb中的植物抗性基因序列对小麦的抗性基因进行分析。(xlsx228kb)
附加文件3:表S1。
植物ABC转运蛋白家族、结构域及主要功能综述。表S2。对T3数据库中1809个与小麦幼苗和成体叶片锈病抗性相关的独特snp进行VEP注释。图S1。T3数据库中幼苗和成体抗叶锈病qtl SNP标记的维恩图(A)及其在小麦不同染色体上的分布(B:幼苗;C:成虫)。(PDF 544kb)
附加文件4:
抗叶锈病qtl (P≤0.01),其SNP标记的侧翼序列分别来自CerealsDB和T3数据库。(XLSX 335kb)
附加文件5:
在同一染色体上位于基因起点和基因末端之间的SNP标记;ABC、NLR和START基因分别位于它们的上游(转录起始位点或TSS)和下游(转录终止位点或TTS) 5kb处。(xlsx78kb)
附加文件6:
预测锈菌snp对ABC、NLR和START基因编码蛋白氨基酸变化的影响。具有多个snp的基因被突出显示。(xlsx50kb)
附加文件7:
这些分子标记及其引物序列在graines和这些maker附近的候选基因中发现。(xlsx22kb)
附加文件8:
的RNA-seq表达值(单位为TPM或转录本/百万)美国广播公司,NLR和开始5个时间点(接种后0、1、2、3、5 dpi或d)的抗性和感感小麦寄主基因,抗性和感感小麦中表达的基因,以及仅在抗性或感感小麦中表达的基因的折叠变化。(XLSX 112kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
彭凤云,杨荣荣。叶锈病相关基因家族的预测与分析(柄锈菌triticina小麦的抗性(小麦l .)。BMC Plant Biol17,108(2017)。https://doi.org/10.1186/s12870-017-1056-9
收到了:
接受:
发表:
关键字
- ABC转运蛋白
- NLR (NBS-LRR)
- 开始
- 抗锈病基因
- 分子标记
- 单核苷酸多态性(SNP)
- 面包小麦
- 小麦
- 基因组分析