摘要
背景
基因组预测是一种基因组学辅助育种方法,可以通过加速育种周期和潜在地提高育种值的准确性来增加遗传收益。在本研究中,我们使用了41,304个信息性snp基因型桉树育种人口涉及90E.Grandis.和78年E.urophylla父母和他们的949 F1杂交种制定八种表型特性的基因组预测模型 - 基本密度和纸浆产量,乳房高度和高度和树木的周长,年龄三到六年。我们评估了不同基因组预测方法的影响,训练和验证集的组成和大小以及SNP的数量和基因组定位对预测能力(PA)。
结果
利用已实现的基因组关系矩阵(GRM)估计的遗传力远高于基于预期系谱的估计,这主要是由于预期系谱的不一致性很容易被GRM校正。此外,GRM更精确地捕捉相关个体之间的孟德尔抽样,因此遗传协方差是基于个体之间共享的基因组的真实比例。当增加训练集的大小和增强与验证集的相关性时,PA得到了相当大的改善。纯亲本训练的预测模型不能很好地预测F1混合动力车,表明必须在混合群体中进行模型培训,如果一个是在混合选择候选者中预测。不同的基因组预测方法为所有特征提供了类似的结果,因此GBLUP或RRBLUP表示计算时间和预测效率之间更好地妥协。当所有特征使用超过5000个SNP时,PA中只观察到轻微改善。在代亚基地区使用SNP提供稍微更好的PA,而不是在基因区域中使用的SNP。
结论
与统计方法和SNPs的基因组位置相比,训练集的大小和组成以及所用SNPs的数量是模型预测的两个最重要因素。此外,基于纯亲本种训练的预测模型对种间杂交种性状的预测能力有限。我们的结果为基因组预测的实施提供了额外的前景桉树通过种间杂交选择的育种程序。
背景
桉树物种及其杂种是热带,亚热带和温带地区种植的硬木,由于它们的快速增长,旋转时间短,环境适应性,商业纸浆和造纸生产的适宜性[1那2].种间杂交的E.Grandis.和E.urophylla特别是,通常优于其生长,木质品质和生物和非生物胁迫的父母,通过继承快速生长和良好的生根能力E.Grandis.虽然通过维持疾病耐受性和广泛适应性E.urophylla[3.].在杂交种群中走向克隆选择的传统育种周期包括交配、子代试验、小规模克隆试验和第二次扩大克隆试验,这通常需要12至18年[1那4.].为了加速单位时间内的遗传增益,需要新的方法来帮助缩短育种周期。
基因组预测或基因组选择(GS)是基因组学辅助方法的最新发展之一,其旨在提高育种效率和遗传收益。基因组预测提供了一种用于标记辅助选择的基因组范式(MAS)[5.那6.].在GS中,所有基因组 - 宽标记都在依赖于联动不平衡(LD)原理的模型中,以捕获整个基因组的大多数相关变异,而MAS专注于先前已有的离散定量特质基因座(QTLS)。检测到,通常在动力量的实验中,因此留下大部分表型变异因[7.].GS通常分为三个步骤:(1)基因分型和对“参考”或“训练人群”进行表型分型,并结合发展基因组预测模型,允许从基因型预测表型;(2)在“验证群体”中验证预测模型,即一组没有参加模型培训的个体;(3)应用该模型预测非表现型个体的基因组估计育种值(GEBVs),然后根据其GEBVs进行选择[6.].GS已成功应用于牲畜的饲养[7.那8.[农作物[9.那10最近的几篇论文还列举了森林树育种的巨大潜力[11那12].
基因组预测模型的准确性取决于所采用的统计方法。目前已发展了几种方法,包括脊回归最佳线性无偏预测(rrBLUP)、基因组最佳线性无偏预测(GBLUP)、BayesA、BayesB、Bayesian LASSO、BayesR和再生核希尔伯特空间(RKHS)回归[7.那13].这些方法的差异主要体现在对标记效应分布和方差的假设上。对于rrBLUP,所有位点都是先验假设,以解释等量的方差,因此假设标记效应遵循正态分布,所有效应都缩减到类似的较小规模。[6.那14]在贝叶斯方法(Bayesa,Bayesb,Bayesian Lasso和Bayesr)中解释的遗传方差一世轨迹,V.胃肠道,假设它们遵循先验分布,p(V.胃肠道).因此,方差可以在基因座中各不相同,并将信息与数据的现有分布相结合,产生数据产生的估计V.胃肠道[6.那15].例如,BayesA假设遗传方差遵循倒卡方分布,而Bayesian LASSO假设遗传方差遵循双指数分布。GBLUP方法从基因组关系矩阵计算可加性遗传优点,在实践中普遍满足的条件下相当于rrBLUP [16].RKHS回归模型是再现内核提供的基本功能的线性组合[17].最近的研究表明,选择合适的统计方法依赖于手头的实际数据和感兴趣性状的表型变异模式,并使用参考群体[9.那18].
除了统计方法外,已知的其他因素也会影响基因组预测模型的准确性,如训练群体的规模、使用的标记数量、训练和验证群体之间的相关性,以及延伸到未来的选择候选者。Hayes等人[19]发现,对于给定的有效人口大小(NE.),增加参考种群的规模可以提高基因组预测的准确性。训练群体和选择候选群体之间更密切的关系也被报道导致更高的基因组预测准确性,而扩大训练群体的遗传多样性导致更低的准确性[20.].许多仿真和经验研究表明,增加标记数量可以提高预测性精度NE.也增加了(9.那21那22那23].然而,增加的标记数量在小NE.人口数量对预测准确性的改善微乎其微或根本没有改善[24那25].
从以前的森林树上进行进一步迈出一步,其中分配了相同育种生成的个体,用于评估基因组预测模型的训练和验证集,在这项研究中我们使用了父母和后代大肠茅那大肠urophylla及其f1杂交建立预测模型,使用不同的子集的父母和后代的训练和验证集。包含60,904个SNP的多物种单核苷酸多态性(SNP)芯片[26]来提供两代的高密度基因分型。基于这些数据,我们开发了身高、胸围(CBH)、体积、木材基本密度和纸浆产量的基因组预测模型,并使用一些统计方法与传统的基于系谱的预测进行了比较。此外,我们评估了不同snp数量、训练和验证集的组成和大小对基因组预测能力(PA)的影响。
方法
繁殖种群
本研究的育种群体是通过86个品种的对照杂交获得的大肠urophylla和95大肠茅结果表明:G1群体有16660个子代个体,包括476个全同胞家系,每个家系35个个体。2009年,在Veracel Celulose S.A. (Eunápolis, BA, Brazil) Belmonte(巴西,海拔210 m, 39.19 W, 16.06 S)采用单树随机完全区组设计进行田间试验,每科35个重复。我们的实验群体由168名父母组成(78名E.urophylla和90年的E.Grandis.) (G0),因为在研究时并非所有父母都还活着,并通过避免表现较差的树,在338个全同胞家庭中抽样了958个后代个体(G1)。每个全同胞家庭的人数从1人到13人不等,平均每个家庭2.8人。
表型
对于乳房高度(CBH)的958个G1样品,高度,体积和周长分别测量,分别测量了木质性状(基本密度和纸浆产量)5岁。对于168 G0父母,相同的特征已经在七年龄龄阶段大肠茅五岁的时候大肠urophylla.简单地说,高度是用Suunto hypometer /height meter (PM-5/1520系列)测量的,CBH是在离地面130厘米处用一厘米长的胶带测量的。利用FOSS NIRSystem 5000-M在乳房高度采集的锯末样品的近红外反射光谱,并应用早前由Veracel S.A开发的校准模型,估计木材性质。
应用混合线性模型,以最大限度地减少对每个特征的环境和年龄差异的影响。
在哪里y是观察到的单一特征的向量;β是一个固定效应矢量,包括总体平均值、实验地点和年龄差异;u为正态分布个体的随机加性遗传效应向量,u ~ N(0,一种\({\σ}_u ^ 2 \)),一种是个体间加性遗传关系的矩阵;B.为各实验站点嵌套的随机不完全块效应向量;和E.为各实验点的异质随机残留效应。X那Z.和W.的关联矩阵β那u和B.,分别。然后通过减去所有个体的位点、年龄和块效应的变异来校正每个性状的表型,并将其作为调整后的表型。利用调整后的表型性状计算性状的遗传力,并建立基因组预测模型。
基因分型和质量控制
使用Illumina Infinium Euchip60K进行168 G0和958 G1种群进行基因分型[24包含60904个snp的探针。EUChip60K强度数据(。一世D.一种t files) were obtained through GENESEEK (Lincoln, NE, USA). SNP genotypes were called using GenomeStudio (Illumina Inc., San Diego, CA, USA) following standard genotyping and quality control procedures with no manual editing of clusters as described earlier [26].使用PLINK对基因型样本进行进一步的质量控制[27].去除9个样本呼叫率小于70%或近交系系数大于1的G1个体进行进一步分析。10240个SNPs由于低呼叫率(小于70%)而被排除,9243个SNPs由于单型性或小等位基因频率(MAF)小于0.01而被过滤掉。最后去除117个snp,因为它们与Hardy-Weinberg平衡(p-value <1 × 10-6).
质量控制后,对剩余个体缺失的基因型进行归并。我们首先通过人工去除一部分基因型中的SNPs,测试了一系列缺失数据(2% - 30%)中输入方法的准确性。在现有的基于家庭和基于人群的方法中,我们评估了以下程序的准确性:BEAGLE [28],fastphase [29],孟德尔[30.,随机森林,SVD Impute, k-最近邻[31, BLUP A矩阵,贝叶斯PCA, NIPALS,概率PCA [32].BEAGLE为所有丢失的数据百分比提供了最佳的准确性,在所有情况下的准确性都超过95%(附加文件1).因此,我们使用比格犬在上述过滤步骤后保留的41,304个SNP的缺失基因型,遍布所有168 G0和949 G1个体。随后在所有基因组预测分析中使用抵抗基因型数据。SNP对之间的LD使用平方相关系数测量(R.2)对于位于同一染色体上的SNP。追随雷丁顿等。[33,然后使用非线性回归方法模拟LD随物理距离的衰减。
我们进一步通过主成分分析(PCA)估算了1117个个体之间的种群结构和配对基因组关系[34]并通过计算个人之间的基因组关系[14使用10213个独立的SNPs (LD-pruned) (r2< 0.2)在PLINK中计算[27].在ASReml中使用ABLUP估计基于谱系的遗传关系(详见下文)。
基因组预测的统计方法
评估了四种统计方法对eq. (1)和预测gebv。这些方法包括基因组最佳线性无偏见预测因子(GBLUP) [5.],岭回归BLUP(rrblup)[6.],贝叶斯套索(提单)35),而重新生成核希尔伯特空间再生核希尔伯特空间理论()回归17].选择这些方法以表示基因组预测的各种方法。GBLUP表示不依赖标记效应估计的方法;RRBLUP使用线性和惩罚参数估计标记效应;BL表示用于标记效应估计的线性,参数和贝叶斯方法;虽然RKH表示非线性半参数方法。将四种基因组预测方法的性能与常用的血统的增殖(ABLUP)进行了比较[36].
gebv的估计采用以下混合线性模型:
在哪里y是单性状调整表型的载体,β为作为固定效应拟合的总体均值向量,一种是随机效果的矢量,和E.为随机残留效应向量。1和Z.为关联矩阵β和一种,分别。
ABLUP
ABLUP是基于系谱信息利用个体间的预期亲缘关系预测育种值的标准方法[36].对于ablup,随机添加剂效应的载体(一种在Eq. (1假设)遵循正态分布\ (\ boldsymbol{一}\ sim N \离开(0 \ boldsymbol{一}{\σ}_a ^ 2 \) \), 在哪里一种可加性分子关系矩阵是由系谱信息估计的吗\({\ sigma} _a ^ 2 \)为加性遗传方差。残余向量E.假设是假设的\ (\ boldsymbol {e} \ sim N \离开(0 \ boldsymbol{我}{\σ}_e ^ 2 \) \), 在哪里一世是单位矩阵。在这些假设下,Eq. (1)可以重写为:
在哪里\({\ sigma} _e ^ 2 \)和\({\ sigma} _a ^ 2 \)使用受限制的最大似然方法估计。估计的繁殖价值({\ \(\帽子mathbf{一}}\))及固定效果(\(\ hat {\ boldsymbol {\ beta}} \))可以直接由式(2).使用ASReml 3.0进行ABLUP计算[37].
GBLUP.
GBLUP方法是由ABLUP派生而来的,但与ABLUP方法的矩阵不同一种在情商。2)用基因组关系矩阵替换(G)由使用基因型数据计算\(\ mathbf {g} = \ frac {\ left(\ mathbf {m} - \ mathbf {p}右){\ left(\ mathbf {m} - \ mathbf {p} \ revaly)} ^ {\mathrm {t}} {2 {\ sum} _ {j = 1} ^ p {p} _j \ left(1- {p} _j \右)} \), 在哪里m是SNP的样本矩阵,SNP编码为0,1,2(即次要等位基因的数量),P.是等位基因频率的矩阵j-第2列(pj- 0.5),在哪里pj为样本观察到的等位基因频率[5.].在GBLUP中,随机添加效果(一种)。(1)的假设\ (\ boldsymbol{一}\ sim N \离开(0 \ mathbf {G}{\σ}_g ^ 2 \) \), 在哪里\({\σ}_g ^ 2 \)是基于基因组的遗传变异和GEBVs ({\ \(\帽子mathbf{一}}\))同样由式(2),但与一种-1取而代之的是G-1和\({\ sigma} _a ^ 2 \)取而代之的是\({\σ}_g ^ 2 \).使用ASREML 3.0进行GBLUP计算[37]使用来自r中的rrblup包的“a.mat”函数估计G矩阵[14].
rrBLUP
与前面两个方法不同,rrBLUP改变参数的表示法一种和Z.在方程。(1), 在哪里Z.现在指的是SNP效应的设计矩阵,而不是事件矩阵和一种是指假设遵循的SNP效果\ (\ boldsymbol{一}\ sim N \离开(0 \ mathbf{我}{\σ}_m ^ 2 \) \), 在哪里\({\σ}_m ^ 2 \)表示由每个SNP贡献的遗传变异的比例[6.].有了这些变化,Eq. (2)就变成:
在哪里{{sigma}_e^2/{{sigma}_u^2}是残差和标记差异之间的比率。计算每个单独的GEBV的预测\({\帽子{g}} _i = {\ mathbf {Z}} _ {\ mathbf{我}}^ {\ mathbf {T}} \帽子{\ mathbf{一}}\)从等式(3.), 在哪里\ ({\ mathbf {Z}} _ {\ mathbf{我}}^ {\ mathbf {T}} \)是个人的SNP矢量一世和{\ \(\帽子mathbf{一}}\)是估计SNP效应的载体。所有计算都使用“混合”。在R [14].
贝叶斯套索
Bayesian LASSO (BL)方法是Legarra等人提出的LASSO回归的贝叶斯处理方法[34].在BL的SNP效果的载体中,一种在方程(1),假设遵循层次先验分布\ (\ boldsymbol{一}\ sim N \离开(0 \ mathbf {T}{\σ}_m ^ 2 \) \), 在哪里\(\ mathbf {t} = \ operatorname {diag} \ left({\ tau} _1 ^ 2,\ dots,{\ tau} _p ^ 2 \右)\).\({\τ}_j ^ 2 \)被分配为\({\ tau} _j ^ 2 \ sim exp \ left({\ lambda} ^ 2 \右)\)那j = 1,…,p.λ2被分配为λ2 ~ γ(r,δ).残差方差\({\ sigma} _e ^ 2 \)被分配为\({\ sigma} _e ^ 2 \ sim {\ chi} ^ { - 2} \ left(d {f} _e,{s} _e \右)\).
我们使用来自r中的BLR包的“BLR”功能实现了BL方法[38].本文采用蒙特卡罗马尔可夫链采样器,先验参数(dfE.,S.E.,r,δ,λ2)的定义遵循de los Campos等人提出的指导方针[39].链长为20000次迭代,前2000次被排除为老化,随后的细化间隔为100次。
rkhs.
RKHS假设方程式中的随机添加效应。(1)\(\ boldsymbol {a} \ sim n \ left(0,\ mathbf {k} {\ sigma} _g ^ 2 \右)\), 在哪里K.通过k给出的高斯内核来计算ij= exp(−高清ij)[17].H是一个半参数,可以控制先前的协方差函数随着遗传距离增加和的快速下降的速度D.ij两个样本之间的遗传距离是否计算为\({d} _ {ij} = {\ sum} _ {k = 1} ^ p {\ left({x} _ {ik} - {x} _ {jk} \ otive)} ^ 2 \), 在哪里x本土知识和xjk是kth snps(k = 1,…,p)一世他和jth样本,分别。我们通过R中的BGLR包中的“BGLR”函数实现RKHS方法[40],它使用Gibbs采样器为贝叶斯框架分配了先前分配\({\σ}_g ^ 2 \)和\({\ sigma} _e ^ 2 \)作为\({\ sigma} _g ^ 2 \ sim {\ chi} ^ { - 2} \ left(d {f} _g,{s} _g \右)\)和\({\ sigma} _e ^ 2 \ sim {\ chi} ^ { - 2} \ left(d {f} _e,{s} _e \右)\),分别。在这里,我们选择了佩雷斯建议的多核模型[40),三H值被定义为\({h} _1 = 2 /左({5} ^ {\ ist} \ surnst { - } {d} \右)\)那\({h} _2 = 2 / \ versst { - } {d} \)那\({h}_3={2}^{\ast}5/\overset{-}{d} \)那\(\ verset { - } {d} \)是中位数D.ij.GIBBS链长度为20,000次迭代,前者丢弃为烧坏和更薄的间隔设置为100。
遗传性估计
我们估计了基于系谱的狭义遗传率(\ ({h} _a ^ 2 \)),利用ABLUP方法得到的关系矩阵和狭义基因组遗传率(\ ({h} _g ^ 2 \))使用GBLUP的基因组关系矩阵([41])。各自的遗传率计算为:
在哪里\({\ sigma} _a ^ 2 \)是从ABLUP估计的添加方差,而\({\σ}_g ^ 2 \)为GBLUP估计的基于标记的遗传方差。\({\σ}_y吗^ 2 \)是种群的表型变异。
培训和验证集的尺寸和遗传构成
我们同时评估了G0和G1个体在基因组预测模型的训练集(TS)和验证集(VS)中的G0和G1个体的大小和遗传参与的影响。关于相对TS / VS尺寸,我们将所有1117(G0和G1)个体分成五个不同大小的组,其中TS:Vs比为1:1,2:1,3:1,4:1或9:1。TS / VS的相应大小分别为558/559,743/374,836 / 281,892 / 225和1003/114个体。在这些预先建立的尺寸组合物中,采用了四种情况,其中评估了G0和G1个体的参与以评估改变TS和VS之间的关系和多样性的影响。在第一场景中(简历1)将个人分配给Ts或Vs是随机的。对于第二种情况(简历2)所有G0家长都被分配给TS并补充到随机选择G1个体,到集合中所需的数字,而VS由剩余的G1个人组成。第三(简历3.第四个(简历4.)基于最小化和最大化TS和VS之间的相关性建立的场景使用Spindel等人描述的程序确定了基于相关的个体分配。[9.].简单地说,根据1117个个体的基因型,使用r中fpc包的“pamk”函数实现的k-means聚类算法,将其分配到182个聚类中。这种方法试图最小化聚类中个体与聚类中心的距离。使用相关性估计,CV3.然后通过将个体分配给TS和VS基于不相似性来构建,使得来自同一群集群的个体不允许在同一TS或VS中兼而有之适用于简历4.来自同一群集的个人被迫在TS或VS中增加TS和VS中的相关性[9.].
基因预测模型
我们评估了五种统计方法的影响(GBLUP,RRBLUP,BL,RKHS和ABLUP),五个TS / VS尺寸和四个TS / VS组成方案(总共5 * 5 * 4 = 100型号)在预测能力(PA)基因组预测。对于100个型号中的每一个,为每个特征进行200个复制运行,并在其PA(R.)评估模型的性能(R.y,\(\帽子{g} \)),其定义为调整后的表型与VS样本的GEBV之间的Pearson相关性。Anova进行了在经过测试的100款(排除20个Ablup模型)中固定的所有申请的所有效果,以将总方差分为不同源(基因组预测方法,TS / VS尺寸和遗传组合物)。我们发现的显着差异通过配对的T测试(α= 5%)进一步评估,通过Bonferroni校正调整。如上所述的80种型号用于评估TS / VS组成和TS / VS尺寸的影响,而所有100种模型用于评估ABLUP的统计方法。所有可用的SNP都用于这些模型的所有分析中。
snp亚群的数量和基因组定位
最后,我们评估了snp的数量及其位置(基因与基因间区域)对基因组预测模型PA的影响。随机选取10、20、50、100、200、500、1000、2000、5000、10000、20000和41,304个SNPs,生成12个不同数量的SNPs子集。在SNP定位方面,通过包含4个不同区域的SNPs,建立了位于基因组不同区域的SNPs亚群:(i)仅编码序列(CDS)(11786个SNPs);(ii)整个基因区域,包括CDS、utr、内含子和基因上下2kb的序列(30,405个SNPs);(iii)基因间区域(10,899个SNPs)和(iv)全部41,304个SNPs。每个SNP的位置和分类通过将SNP映射到E.Grandis.基因组使用snpeff [42].我们仅使用两种预测性能最好的统计方法(GBLUP和RKHS)对所有四种TS/VS组合建立基因组预测模型,使用TS/VS大小比4:1(892/224)。
结果
表型性状的相关性
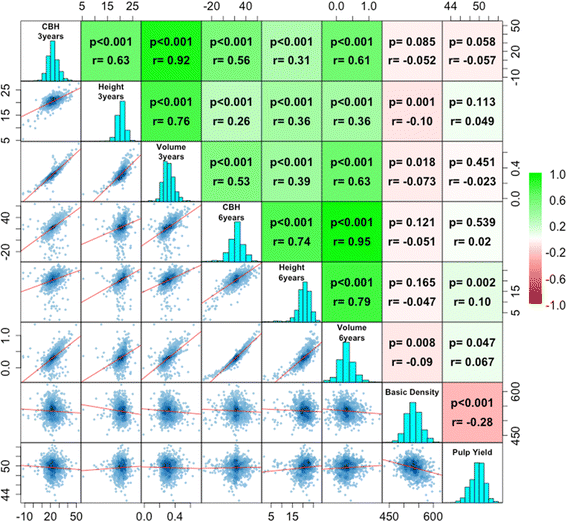
测量所有168 G0和949 G1个体测量生长(高度,体积和CBH)和木质性质(基本密度和纸浆产量)。使用混合线性模型进行调整原料表型数据,以最大限度地减少环境和年龄差异的影响。通过计算Pearson相关系数来描述调整的特征之间的成对相关性(图。1).生长性状之间相互关联。然而,有趣的是,3岁和6岁时CBH与体积高度相关(r = 0.92 and 0.95 respectively), height at age three was only weakly correlated with height at age 6 (r= 0.36)。在木材特性性状方面,基本密度与纸浆产量呈负相关,但相关性较弱(r=−0.28)。生长性状与木材性状无相关性(r=−0.1到0.1)。
种群结构和亲缘关系
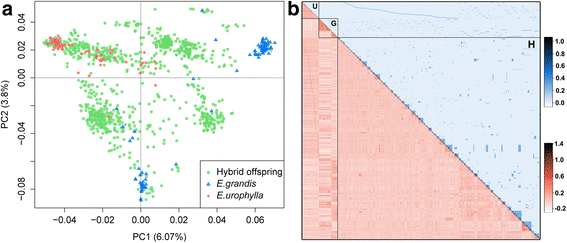
基于10213个ld -剪枝的独立SNPs (r2<0.2)。前两台PC解释了总遗传方差的6.07%和3.8%(图。2),并明确区分出两个物种的G0个体,E.Grandis.和E.urophylla,与E.Grandis.个人进一步细分为两个可能代表巴西育种计划中使用的两个主要种植的亚组。G1个体通常被投射到父母所定义的空间中,而是有几个异常值。使用热插拔(图1中的蓝色和红色)可视化G0和G1个体中的预期基于基于基于基于基于基因组的基因组关系。2 b分别)。基因组关系分析结果证实了主成分分析的结果大肠urophylla都集中在一个组里,然而大肠茅形成了两个亚组。被认为是来自血统数据的全部SIB,半SIB和无关个体的实现基因组关系的平均值通常低于预期的关系值(0.309,0.5,0.131,0.25和0.0056 Vs.0,分别为0)(表1).这一结果表明,这种人口中可能存在血统误差。这些推定的血统误差反过来影响了我们基于血统信息估计特征的可遗传性的能力,这比使用基于基于基于基于基因组的实现遗传关系的估计值(表2).
不同统计方法的预测能力
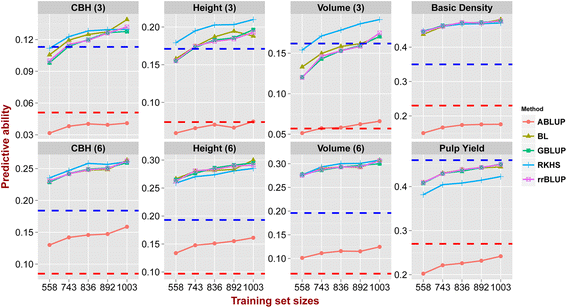
利用不同的统计方法、TS/VS的组成和大小估算了每个性状的PAs(附加文件)2).方差分析显示,所有这些因素对PA有显著影响(P.-value <0.005)(附加文件3.).在使用的四种基因组预测方法(GBLUP、rrBLUP、BL和RKHS)中,平均PA在0.27到0.274之间变化4.).所有四种方法在8个性状上都比基于系谱的ABLUP预测(平均PA = 0.121)平均高出80%-200%(图)。3.).RKHs在八个特征中产生了略微更好的PAS,并且该方法特别适用于预测涉及近秘伤性的特性,例如CBH和高度。另外三种方法通常在所有特征上产生类似的结果,尽管具有比纸浆产率的RKH略微更好的性能(图。3.).
TS / VS组成和相对尺寸对预测能力的影响
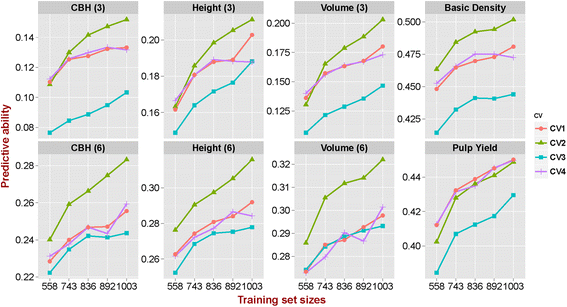
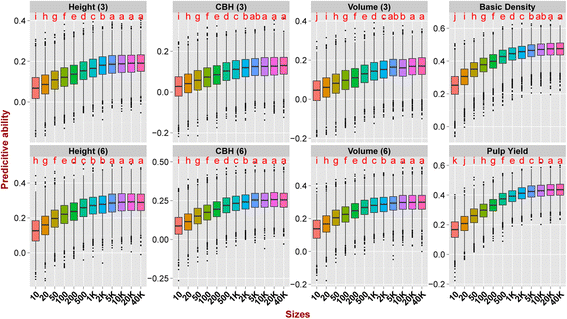
平均PAS对于测试的不同TS / VS组合物,从0.253变为0.286(附加文件5.).利用CV建立基因组预测模型2(TS中所有G0亲本)除果肉产量外,所有性状的PAs值均最高3.(TS和VS之间的最小相关性)给出了最差的预测。基于CV的模型1(随机分配)和简历4.(TS和VS之间的最大相关性显示PA没有显着差异(图。4.额外的文件5.).随着TS/VS比率从1:1(558/559)增加到9:1(1003/113),平均PA从0.251显著提高到0.2856.),与预测方法无关(图。3.)或TS/VS的遗传组成(图。4.),清楚地表明了培训大小的重要性,培训设置为构建预测模型。此外,当TS / VS比率从1:1(558/559)增加到2:1(743/374)时,PA陡峭增加,比2:1(743/374)至9:1(1003 /114)对于所有特征(图。3.和4.).
snp的数量及其基因组位置对预测能力的影响
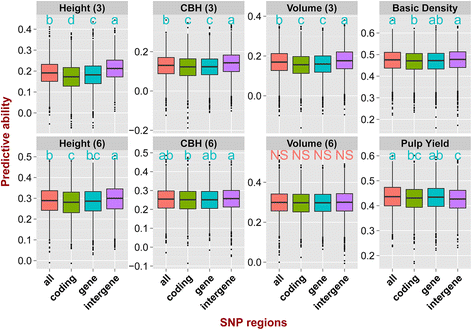
使用不同数量的SNP估计PA(附加文件7.)和不同基因组位置的snp子集(附加文件8.),采用TS/VS为892/225和4种不同TS/VS组成的预测方法。方差分析显示,单核苷酸多态性的数量和它们的基因组位置显著影响两种预测方法(GBLUP和RKHS)的PA (P.-Value <0.005),并且SNP的数量比其基因组位置更大的影响(附加文件9.).当预测模型中使用的SNP的数量从41,304降至仅10时,所有特征跨越0.278到0.113的平均PAS减少,并且当SNP的数量低于5000时,减少尤其强劲(附加文件10).另一方面,当使用超过5000个snp时,PA的平均值一般没有显著改善(附加文件10,无花果。5.).不同性状的结果表明,遗传力较低的性状对SNPs数量的减少更为敏感(图。5.).例如,PA为基本密度(h2 = 0.35) went from 0.47 to 0.24 (a 50% decrease) when the number of SNPs dropped from 40,000 to 10, whereas CBH of age three (h2= 0.113)从0.128降至0.03(下降77%)。总体而言,通过使用位于不同基因组区域的SNP集,PAs有轻微显著差异(图)。6.),平均PAS范围为0.270至0.284(附加文件11).使用位于基因间区域的SNPs的预测比使用位于基因区域的SNPs或所有SNPs的预测略好,除了纸浆产量,基于来自编码和基因区域的SNPs的模型可以更好地预测(图)。6.).当使用SNP进行编码与整个基因区域的SNP进行比较时,后者具有稍微更好的性能,最有可能是由于使用的较大的SNP(30,504与11,786)而不是由于基因组定位的任何特定效果。当我们评估成对ld(r2)在测试的四个区域中的SNP中,LD的程度在于它们的程度,LD显示了编码区域中最快速的衰减和基于代理区域中最慢的衰减(附加文件12).
讨论
本研究提出了对基因组预测的准确性对生长和木材品质性状的准确性的实证评价结果桉树使用来自高密度SNP阵列的数据。我们的结果基于来自两代育种人口的数据,并在利用基因组预测加速育种的前景提供额外的令人鼓舞的结果。我们评估了一系列因素,包括用于估计预测能力的统计方法,训练和验证集的规模和组成以及预测模型中使用的SNP的数量和基因组位置。以下,我们将讨论这些因素如何影响预测准确性。
基因组数据校正家系的不一致性
所有四种基因组预测方法对所有复杂性状的评估都明显优于基于系谱的评估(图)。3.).而类似的结果在动物身上也有报道[18那43]和作物品种[9.那36],在森林树木中,使用基因组数据预测的准确性通常与使用系谱估计育种值获得的准确性相似,或低10-30%,包括桉树[4.]、火炬松(Pinus Taeda.)[44]、白云杉(Picea glauca.)[45那46]、室内云杉(Picea Engelmannii.×葡萄糖)[47那48]和海洋松(Pinus Pinaster.)[49].基因组预测的准确性低于基于家谱的预测,这可能是由于标记密度不足造成的,因此不是所有偶然的变异都能在基因组估计中捕捉到[41],或由于无法确定同父异母兄弟家庭的真实遗传关系而高估了基于系谱的预测[47].然而,我们的结果与森林树木中以前的研究不同,因为由于基于基于血统信息的预期,个体之间的遗传关系的平均对遗传关系的平均成对估计基本上降低了(表1),这清楚地表明,预期的系谱以及由此产生的两两关系存在相当大的不一致性,这些不一致性已被SNP数据修正。我们推测,这些不一致可能来自花粉污染和/或在产生完整和半同胞家庭的过程中的错误标记。除了纠正潜在的血统错误,使用相对密集的SNP的数据在我们的研究中也能够准确地捕捉孟德尔抽样家庭中的变异,遗传方差估计是基于基因组的真实比例相同的血统(IBD)或状态(IBS)之间的一半——或者full-sib个体,结果改进了性状遗传率的估计(表2).
基因组预测显示,性状完全符合无穷小模型
总体而言,不同的基因组预测方法对所有性状的预测结果相似,RKHS仅略占优势,对低遗传力的生长性状表现出更好的PAs(图)。3.).然而,对于纸浆产率,RKHS是最糟糕的表演方法,并且核的定义可能根本不适合这种特定特征[17].我们的结果证实了之前从作物和动物身上得到的报告[18那50那51,以及森林树木。以火龙松为例,在17个具有明显遗传力的性状中,rrBLUP和三种贝叶斯方法的性能仅略有不同,而BayesA仅在抗梭状体锈病方面有小幅改善,在这些性状中,已经描述了影响相对较大的位点[44].其他森林树木的生长和木材性状也得到了类似的结果,表明rrBLUP和贝叶斯方法之间没有性能差异[46那48那49].尽管模拟研究表明,像BL这样的贝叶斯方法应该比rrBLUP和GBLUP这样的单变量方法表现更好,但这种情况还是发生了[6.那52那53].模拟数据和经验数据集之间存在明显分歧的一个可能原因是,大多数性状的真正QTL效应相对较小,分布也没有模拟数据中那么极端[54].因此,我们的结果支持RRBLUP或GBLUP在计算时间和预测效率之间提供最佳折衷方面的提议[55我们研究中评估的数量性状充分符合无穷小模型的假设。
训练集的大小、组成和相关性对预测能力有很大影响
我们的结果表明,无论使用的分析方法如何,训练和验证集的尺寸和组合物对PA对PA产生了最大的影响(图。4.).平均PA随TS大小的增加而迅速增加,且未表现出任何平稳的迹象。早期的模拟桉树繁殖方案实际上显示了N = 1000 individuals in the TS, the accuracy would rapidly increase, and additional gains were seen up toN = 2000 individuals for traits with low heritabilities, for larger numbers of QTLs involved in traits and for larger effective population size (NE.).后N= 2000的预测精度将趋于稳定,而不管NE.基因分型密度[22].模拟(19那56]及概念验证研究[57]在作物品种中也表现出较大的TS尺寸改善PA。随着世代选择的推进,更大的训练种群减少了从育种种群中失去稀有有利等位基因的可能性。此外,通过取样更多的个体进行训练,可以捕获更大的多样性,并获得更好的标记效应估计,这反过来会对交叉验证和未来基因组选择候选者的预测产生积极的影响。
正如预期的那样,TS和VS之间的相关性对所有特征的PA有很大影响。根据场景CV构建的预测模型3.(最小化TS和VS之间的相关性导致预测明显差,而不是方案CV4.当亲缘关系最大化时。我们的结果与之前关于森林树木的报告一致,例如白云杉[45那46),桉树[4.,其中为一个群体开发的模型预测不相关群体的表型的能力有限或没有,这表明预测模型在很大程度上是特定群体的。TS与VS的关系较低,群体中远亲个体的LD程度较短且不稳定,因此降低了基因组预测模型的预测能力。最近的模拟表明,基因组预测模型的准确性随着训练人群和预测人群之间遗传距离的增加而近似线性下降[58].增加的亲缘性减少了独立分离的染色体片段的数量,因此增加了在训练群体中取样的IBD染色体片段也在选择候选中表示的概率。我们的结果提供了额外的实验证据,为成功实施GS,选择候选人必须显示出与训练人群密切的遗传关系。
当所有G0亲本都保持在TS(情景CV2).这结果可能是由于两个原因。一方面,通过将所有G0父母保持在TS中,我们确保为模型培训提供了大的遗传多样性,这可以解释G0纳入预测的正面影响。另一方面,通过将所有G0个体分配给TS,我们观察到的正效应是严格的,这是严格的,因为增加了预测力,而是因为我们避免在组合验证中具有纯种父母的潜在负面影响与g1后代主要是f1混合动力车。为了评估这个,我们估计PA基因预测模型利用再生核希尔伯特空间理论GBLUP和,只有TS 168 G0父母和随机选择168年与G1个人控制的效果强烈降低TS大小,我们将这个设置与随机分配个人TS或VS但保持每一个的大小N= 168。结果显示,当仅使用纯种亲本预测G1杂种后代表型时,PAs相当低(甚至为零或负)13).这一观察结果,以及场景CV的pa4.(TS和VS之间的最大相关性也通常低于CV2,表明我们在情景CV中观察到较高的pa2主要是为了避免在VS中有纯物种亲本的负面影响。
迄今为止,杂交育种中的基因组预测问题只在物种内部和家畜中进行了研究,更具体地说,牛和猪育种是在纯品种中进行选择,但目的是提高杂交性能[43那59].模拟结果表明,通过选择纯种个体进行杂交,可以提供良好的pa,尽管pa随着品种间距离的增加而下降[60].当不可用帧交叉数据时,单独的纯种训练群体可以单独使用或组合使用,这取决于LD相位之间的LD相位之间的相关性[61,而这在一定程度上又取决于人口之间分化的时间。与属于同一物种但最近已分化的牛品种相比(<300KYA) [62],两者之间的估计发散时间桉树我们研究中使用的物种要古老得多,估计有2-5个MYA [63].因此,我们不期望两种物种之间的LD相位阶段的相关性并不令人惊讶地训练组合的纯种在F中验证1杂交导致PA差。我们所知,我们的结果是第一个提供一个初始基因预测的问题从纯物种种间杂交和我们的研究结果表明,与理论预期一致,模型必须被训练使用混合动力车如果预测表型混合选择候选人。
SNP的数量比SNP基因组定位更重要
在所有特征上,在使用超过5000个SNP时,PA中没有检测到重大改进(附加文件10,无花果。5.),但使用GBLUP对2万个单核苷酸多态性进行分析时,3岁时的身高、基本密度和纸浆产量均有轻微增加。先前的几项研究表明,相对于使用所有可用SNPs所观察到的,提供PAs的SNPs数量要低得多[24那64].基因组预测模型所需的SNPs数量取决于LD的程度,LD的程度严格依赖于NE..我们的结果是,我们使用全部或仅10-20%的基因型标记获得了等价的PAs,这表明它代表了一个相对温和的封闭育种群体NE..这在家畜中是一种常见的方法,目的是开发低密度基因分型芯片,以降低基因分型成本[8.].使用减小的SNP面板的主要优点是成本效益,尽管预计由于重组和选择对LD图案的综合效果,使用更高密度的标记将使PA的衰减减轻PAS衰减。65.].对于特定群体或完整的SNP芯片具有更具针对性的低密度SNP芯片,可以在若干组织的繁殖群体中使用的针对特定群体或全SNP芯片更具成本效益。通过拥有一个SNP芯片,该芯片将容纳几个群体的成本效益和分配规模的经济性许多待基因分型的样品可能会大于通过在每个特定上使用较少数量的SNP观察到的成本减少人口。
SNP位置也有助于基因组预测模型的预测能力,尽管效果相当谦虚。除了在基因区域中使用SNP或使用所有SNP,除了使用SNP,除了纸浆产量外,PAS略微好得多,除了在编码和基因区域中的SNP略微预测(图。6.).这可能代表了一种随机抽样效应,而不是该性状的功能变异的任何特定富集。然而,基因间snp的LD下降速度要慢于基因和/或编码区域的snp(附加文件)12), LD范围略长,这可能有助于解释为什么在基因间区域使用SNPs可以提供更好的PAs。由于LD衰减较慢,基因间区域的SNPs可能比编码区域的SNPs更能捕获更长基因组片段的qtl,而编码区域的LD衰减较快。
影响模型预测准确性的进一步问题
为了在桉树育种中成功地采用基因组预测,还需要研究几个问题。首先,由于重组的影响,基因组预测的准确性如何在连续几代选择中下降?模拟研究表明,在早期的几代中,预测精度迅速下降,但这种下降在后代中放缓[6.那16].因此,在下一代个体的表型可用后,应更新GS模型。其次,多种环境中的基因组预测模型是多么稳定,并且在模型中的环境互动考虑基因型的重要性是多么重要?基因组预测和环境影响之间的相互作用将基本上遵循传统的G X E策略。预期预测模型预计在同一育种区内的网站是准确的(可以在不担心不担心不担心的不担心不担心的情况下种植单一种植的树木),但不一定跨越不同的繁殖区[12].此外,在基因组预测中,个体的评估不是基于他们自己的表型表现,而是基于其他个体、年份和环境的基因组信息,这给了一个机会来评估在多个环境中个体之间共享的特定基因组片段的影响。Burgueno等人[66.[展示了从多个环境中的小麦线上的模型和标记数据的模型可以基本上增强相对于仅基于谱系的预测或相对于来自单个环境的基因组学预测模型的预测精度。最后,我们只考虑了在我们的桉树种群中建立基因组预测模型的添加剂遗传方差,但也可能甚至可能是,非添加性遗传效应在许多繁殖人群中发挥着重要作用,并且特别是在包括的人群中起着重要作用早期的杂种。最近基因组预测的模拟研究桉树育种报告称,包括显性效应在内的基因组预测在克隆选择中表现更好,而非加性效应并没有提高亲本选择育种价值的估计[67.].据我们所知,在森林树木中没有关于GS预测个体基因型值的能力的实验数据,包括加性和非加性效应。
结论
我们的实验结果为实施基因组预测提供了进一步的有希望的观点桉树育种计划。基因组预测在我们的实验中基于基于血统的血统预测,主要是由于我们的预期血统具有重大不一致的事实,导致所有基于血统的估计的低估。这种相当意外的结果说明了在育种程序中使用SNP数据和基因组预测的额外优点。虽然基因组预测在桉树育种中的主要优点可能是育种周期长度的减少[4.[允许我们使用基因组关系矩阵获得精确的遗传关系估计,否则我们无法访问的遗传关系和遗产。此外,我们的结果证实了相关性作为PA驱动器的关键作用,使用较低密度SNP面板的潜力,以及生长和木质特征充分适合无穷大的模型,使得GBLUP或RRBLUP表示计算之间的良好折衷时间和预测效率。与之前的研究相比桉树,我们已经访问了两个纯物种的父母(大肠茅和大肠urophylla)他们的f1后代。我们表明,在纯物种亲本上训练的模型不允许对F的准确预测1杂交种,可能是由于两种物种之间强的遗传分歧和两种物种与其杂种之间的LD缺乏一致的模式。
缩写
- BL:
-
贝叶斯套索
- CBH:
-
胸高围
- cd:
-
编码序列
- GBLUP:
-
基因组最佳线性无偏预测器
- GEBV:
-
基因组育种价值估算
- 克:
-
基因组的关系矩阵
- g:
-
基因组选择
- 炎症性肠病:
-
身份的血统
- 肠易激综合症:
-
身份由国家
- LD:
-
连锁不平衡
- 马斯:
-
分子标记辅助选择
- NE.:
-
有效的人口规模
- PA:
-
预测能力
- PCA:
-
主要成分分析
- QTLS:
-
数量性状位点
- RKHS:
-
重新生成核希尔伯特空间
- rrBLUP:
-
岭回归最佳线性无偏预测
- SNP:
-
单核苷酸多态性
- TS:
-
训练集
- 与:
-
验证设置
参考文献
- 1.
Rezende GDSP, Resende MDV, Assis TF。桉树无性系林业育种。出自:芬宁·T,编辑。21世纪世界森林面临的挑战和机遇。荷兰多德雷赫特:施普林格;2014.p . 393 - 424。
- 2.
Myburg Aa,Potts Bm,Marques Cm,Kirst M,Gion JM,Grattapaglia D,Grima-Pettenati J.桉树。基因组测绘和植物分子育种。第7卷由:KOLE CR编辑纽约:林林树木;2007.第115-160页。
- 3.
Bison o,Ramalho M,Rezende G,Aguiar A,De Resende M.开放式授粉后的大豆和杂种性能之间的比较桉树祖母和桉树尿尿.Silvae麝猫。2006;55(4 - 5):192 - 6。
- 4.
Resende MD,Resende MF JR,Sansaloni CP,Petroli CD,Missiaggia AA,Aguiar Am,等。生长和木材质量的基因组选择桉树:森林树木中复杂性状的遗产和加速遗传性和加速育种。新植物。2012; 194(1):116-28。
- 5.
戈达德我,海耶斯BJ,莫维森。利用基因组关系矩阵预测基因组选择的准确性。畜牧兽医学报。2011;128(6):409-21。
- 6.
Meuwissen The,Hayes Bj,Goddard Me。基因组致密标记图预测总遗传价值。遗传学。2001; 157(4):1819-29。
- 7。
利用基因组选择技术加速畜禽改良。生物科学学报。2013;1:221-37。
- 8。
Van Eenennaam Al,Weigel Ka,Young Ae,克利夫兰马,Dekkers JCM。应用的动物基因组学:田间的结果。Annu Revian Biosci。2014; 2:105-39。
- 9。
主轴J,Begum H,Akdemir D,Virk P,Collard B,Redona E,等。水稻基因组选择和关联映射(栽培稻):性状遗传构型、训练群体组成、标记数量和统计模型对热带粳稻优良品系基因组选择精度的影响。公共科学图书馆麝猫。2015;11 (2):e1004982。
- 10.
温斯豪森,阿特林,希基,克罗斯,索雷尔斯等。不同群体和环境下玉米杂种性能基因组预测的有效性中国生物医学工程学报。2012;2(11):1427-36。
- 11.
森林树木育种中的基因组选择:概念和未来展望。新森林。2014;45(3):379 - 401。
- 12.
通过基因组选择培育森林树木:当前的进展和前进的道路。见:植物遗传资源基因组学:第1卷遗传资源管理、测序和挖掘。编辑:Tuberosa R, Graner A, Frison E. Dordrecht:施普林格荷兰;2014.651 - 82页。
- 13.
引用本文:王志强,王志强,王志强。全基因组回归与预测方法在植物和动物育种中的应用。遗传学。2013;193(2):327 - 45。
- 14.
Endelman简森-巴顿。岭回归和其他核的基因组选择与R包rrBLUP。植物基因组。2011;4(3):250 - 5。
- 15.
混合模型在作物品种数量遗传中的贝叶斯推理。acta photonica sinica . 2013;126(7): 1749-61。
- 16.
Habier D,Fernando RL,Dekkers JCM。遗传关系信息对基因组辅助育种价值的影响。遗传学。2007; 177(4):2389-97。
- 17。
De Los Campos G,Gianola D,Rosa GJ,Weigel Ka,Crossa J.使用再现核Hilbert空间方法的遗传值的半导体基因组的预测。Genet Res。2010; 92(4):295-308。
- 18。
Neves HH, Carvalheiro R, Queiroz SA。小鼠群体基因组选择统计方法的比较。BMC麝猫。2012;13(1):100。
- 19。
基因组选择的准确性:理论和结果的比较。见:第18次会议论文集:促进动物育种和遗传学协会,Barossa Valley,澳大利亚;2009.34-37页。
- 20。
吴X,隆康MS,Sun D,张Q,苏G.测试与训练动物与训练动物之间的影响对基因组预测的可靠性。j anim繁殖遗传群。2015; 132(5):366-75。
- 21。
Zhong S, Dekkers JC, Fernando RL, Jannink JL。影响多自交系衍生群体基因组选择准确性的因素:一个大麦案例研究。遗传学。2009;182(1):355 - 64。
- 22。
Grattapaglia D, Resende MDV。林木育种中的基因组选择。植物基因组学。2011;7(2):241-55。
- 23.
Moser G, Khatkar MS, Hayes BJ, Raadsma HW。利用SNP标记亚群对荷斯坦牛和奶牛直接基因组值的准确性Genet Sel Evol 2010;42
- 24.
Su G, Brondum RF, Ma P, Guldbrandtsen B, Aamand GR, Lund MS.利用中密度(类似于54000)和高密度(类似于777000)单核苷酸多态性标记板预测北欧荷斯坦奶牛和红奶牛群体的基因组。中国乳业科学。2012;95(8):4657-65。
- 25.
麦克劳德IM,海耶斯BJ,戈达德ME。人口统计学和长期选择对序列数据基因组预测准确性的影响。遗传学。2014;198(4):1671 - 84。
- 26.
Silva-Junior Ob,Faria Da,Grattapaglia D.一种柔性多种全面的60K SNP芯片,从汇总重置240桉树树基因组跨越12种。新植物。2015; 206(4):1527-40。
- 27.
Purcell S,Neals B,Todd-Brown K,Thomas L,Ferreira Ma,Bender D等人。PLINK:用于全基因组协会和基于人口的联系分析的工具。我是j嗡嗡声的遗传。2007; 81(3):559-75。
- 28.
褐变SR,布朗宁BL。通过使用局部单倍型聚类,可以快速准确的单倍型分阶段对全基因组关联研究的缺失数据推断。我是j嗡嗡声的遗传。2007; 81(5):1084-97。
- 29.
Stephens M,Scheet P.核算单倍型推理和缺失数据归档中的联动不平衡衰退。我是j嗡嗡声的遗传。2005; 76(3):449-62。
- 30.
CANDES EJ,RECHT B.通过凸优化完成精确的矩阵完成。找到了计算数学。2009; 9(6):717-72。
- 31.
rutkoski je,波兰j,jannink jl,Sorrells Me。无序标记的归责和对基因组选择精度的影响。G3-基因基因类型。2013; 3(3):427-39。
- 32.
pcammethods -生物导体包,提供不完整数据的PCA方法。生物信息学。2007;23(9):1164 - 7。
- 33.
引用本文:雷明顿·达林,索恩斯伯里·J·m,松冈·Y, Wilson LM, Whitt SR, Doeblay J,等。玉米基因组连锁不平衡结构和表型关联。美国国立科学院。2001; 98(20): 11479 - 84。
- 34.
王志强,王志强。种群结构与特征分析。公共科学图书馆麝猫。2006;2(12):2074 - 93。
- 35.
引用本文:陈志强,陈志强,陈志强。基因组选择的改进套索。麝猫研究》2011;93(1):77 - 87。
- 36.
交叉学报,Campos Gde L, Perez P, Gianola D, Burgueno J, Araus JL,等。利用系谱和分子标记预测植物育种中数量性状的遗传价值。遗传学。2010;186(2):713 - 24。
- 37.
Gilmour AR,Gogel B,Cullis B,Thompson R,Butler D. Asreml用户指南版本3.0。英国https://www.vsni.co.uk/: VSN International Ltd, Hemel Hempstead;2009.
- 38.
王志强,王志强,王志强。基于贝叶斯线性回归包的植物基因组预测。植物遗传资源学报。2010;3(2):106-16。
- 39.
los Campos G, Pérez P, Vazquez AI, Crossa J.使用BLR(贝叶斯线性回归)R-package进行基因组预测。适用于:全基因组关联研究和基因组预测.由Gondro C编辑,Van der Werf J,Hayes B. Totowa,NJ:Humana媒体;2013:299-320。
- 40。
利用BGLR统计软件包进行全基因组回归和预测。遗传学。2014;198(2):483 - 95。
- 41。
de los cg,sorensen d,gianola d.基因组遗传性:它是什么?Plos Genet。2015; 11(5):E1005048。
- 42。
Cingolani P,Platts A,Wang LL,Coon M,Nguyen T,Wang L等。注释和预测单核苷酸多态性效应的程序。snpeff飞。2012; 6(2):80-92。
- 43。
Hidalgo Am,Bastiaansen JWM,Lopes Ms,Harlizius B,Groenen Mam,De Koning Dj。纯种和杂交猪中预测基因组育种值的准确性。G3-基因基因类型。2015; 5(8):1575-83。
- 44。
reende MF Jr, Munoz P, reende MD, Garrick DJ, Fernando RL, Davis JM等。火炬松标准数据集基因组选择方法的准确性(Pinus Taeda.l .)。遗传学。2012; 190(4):1503-10。
- 45。
Beaulieu J,Doerksen T,Clement S,Mackay J,Bousquet J.白色云杉大量开放式授粉家族中基因组选择模型的准确性。遗传。2014; 113(4):343-52。
- 46。
关键词:白云杉,基因组选择,环境,小群体BMC基因组学。2014;15:1048。
- 47.
El-Dien OG, Ratcliffe B, Klapste J, Chen C, Porth I, El-Kassaby YA。利用基因分型对空间云杉内部生长和木材属性的预测精度。BMC基因组学。2015;16:370。
- 48.
陈志强,陈志强,陈志强,等。云杉内部基因组选择模型的时间比较(Picea Engelmannii.x葡萄糖)使用无序的SNP估算方法。遗传。2015; 115(6):547-55。
- 49.
Isik F, Bartholome J, Farjat A, Chancerel E, Raffin A, Sanchez L, et al.;海松的基因组选择。植物科学。2016;242:108-19。
- 50.
Crossa J, Perez P, Hickey J, Burgueno J, Ornella L, Ceron-Rojas J,等。CIMMYT玉米和小麦育种计划中的基因组预测。遗传。2014;112(1):48-60。
- 51.
小野木,井田,井下,江花,吉冈,山崎,等。亚洲水稻全基因组预测方法的应用领域(栽培稻l .)。acta photonica sinica . 2015;128(1): 41-53。
- 52.
Clark SA,Hickey JM,Van der Werf JHJ。不同模型的遗传变异及其对基因组评价的影响。Genet Sel Evol。2011; 43(1):1-9。
- 53.
Honarvar M,Rostami M.使用RR-Blup和Bayesian Lasso的基因组预测的准确性。EUR J EXP BIOL。2013; 3:42-7。
- 54.
Daetwyler HD, Pong-Wong R, Villanueva B, Woolliams JA。遗传结构对全基因组评价方法的影响。遗传学。2010;185(3):1021 - 31所示。
- 55.
赵志强,赵志强,赵志强,等。植物育种中的基因组选择:知识与展望。阿阿格龙。2011;110年
- 56.
洛伦茨AJ。植物育种中基因组选择预测精度和遗传增益最大化的资源配置:模拟实验。acta Genom Genet. 2013;3(3): 481-91。
- 57.
Riedelsheimer C, Endelman JB, Stange M, Sorrells ME, Jannink JL, Melchinger AE。相互联系的双亲本玉米群体的基因组可预测性。遗传学。2013;194(2):493 - 503。
- 58.
利用遗传距离推断基因组预测的准确性。公共科学图书馆麝猫。2016;12 (9):e1006288。
- 59.
基于优势模型的纯种长白和约克郡杂交性能基因组预测。2016;48(1):1 - 9。
- 60.
Ibánẽz-Escriche N, Fernando RL, Toosi A, Dekkers JC。杂交性能纯种的基因组选择。植物学报。2009;41(1):1 - 10。
- 61.
通过纯种基因组选择最大化杂交性能。中国生物医学工程学报。2015;47(1):1 - 16。
- 62。
Murray C, Huerta-Sanchez E, Casey F, Bradley DG。从常染色体序列变异建立的牛人口统计历史模型。李志刚;2010;3(1):1 - 5。
- 63。
从聚合重测序和单核苷酸多态性基因分型中揭示了重组、连锁不平衡和核苷酸多样性的全基因组模式桉树祖母.新植醇。2015;208(3):830 - 45。
- 64。
张Z,丁X,刘继,张Q,de koning dj。利用低密度标记面板的基因组预测的准确性。j乳制品sci。2011; 94(7):3642-50。
- 65。
Solberg TR, Sonesson AK, Woolliams JA, Meuwissen THE。使用不同标记类型和密度的基因组选择。动物科学学报。2008;86(10):2447-54。
- 66。
利用系谱和密集分子标记建立基因型与环境互作模型时的育种值预测。作物科学。2012;52(2):707 - 19所示。
- 67。
Denis M, Bouvet J-M。在桉树育种背景下利用显性效应模型进行基因组选择的效率。植物基因组学。2012;9(1):37-51。
确认
我们要感谢Michelle Bayerl Fernandes对育种群体表型的贡献。计算是在瑞典国家计算基础设施(SNIC)在upmax和HPC2N提供的资源上进行的。
资金
这项研究的部分资金来自Vetenskapsrådet和Kempestiftelserna对PKI的拨款。BT感谢Umeå植物科学中心(UPSC)“森林遗传、生物技术和育种研究学院”的财政支持。
数据和材料的可用性
支持这项研究结果的数据可从Veracel获得,但这些数据的可用性受到限制,这些数据是在许可下用于当前研究的,因此不能公开获得。在合理的要求和Veracel的许可下,作者可以提供数据。
作者的贡献
BT、BS和PKI对实验进行了构思和设计;GSM表型数据;GSM和KZF采集样本进行基因分型;DG负责基因分型;电讯管理局在总监及公匙基建的指引下分析有关资料;BT起草了手稿的第一版,BT, DG, BS和PKI对手稿的最终版本做出了重要贡献。所有作者阅读并批准了最终的手稿。
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
提交人声明他们没有竞争利益。
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
作者信息
隶属关系
相应的作者
附加文件
额外的文件1:
SNP估算方法的平均精度随着缺失数据的增加。染色体6和8上的SNP从数据集中随机删除,以产生特定的缺失数据比例。随后用不同方法计算算术和真正的SNP基因型之间的准确性。(docx 1714 KB)
额外的文件2:
基因组选择模型的预测能力,该模型包括统计方法、遗传成分和每个性状的训练集/验证集的相对大小。(XLSX 16 kb)
额外的文件3:
影响预测能力的变异源的ANOVA分析。(docx 48 kb)
额外的文件4:
5种预测方法对8个性状的预测能力的平均值和标准差。多克斯(96 kb)
额外的文件5:
用四个训练集/验证集组合估计的预测能力的平均值和标准差。多克斯(84 kb)
额外的文件6:
预测能力的平均值和标准偏差,以比例和数量表示的训练集/验证集的五种相对尺寸。(docx 89 kb)
额外的文件7:
在越来越多的SNPs、统计方法(RKHS和GBLUP)、8个性状的4个训练集/验证集组成中预测能力的平均值和标准偏差。(XLSX 61 kb)
额外的文件8:
在四个基因组位置中的SNP估计预测能力的平均值和标准偏差,具有两个统计方法(RKHS和GBLUP),四个训练设置/验证组组合物中的每一个特征。(XLSX 58 KB)
额外的文件9:
以SNP基因组位置和SNP数量作为变异来源的预测能力的方差分析。多克斯(62 kb)
额外的文件10:
用不同数量的SNPs拟合模型估计平均预测能力。多克斯(136 kb)
额外的文件11:
使用位于不同基因组区域的SNP集估计的平均预测能力。(DocX 82 KB)
额外的文件12:
连锁不平衡衰减(LD)与物理距离估计的SNPs在不同的基因组位置。(a)分别位于编码区、基因区、基因间区和所有区域的四类SNPs的LD随物理距离衰减的比较。分别位于所有区域(b)、编码区域(c)、基因区域(d)和基因间区域(e)的snp的LD对物理距离和LD衰减的成对点。多克斯(1375 kb)
额外的文件13:
纯种桉树父母的培训预测能力,并在其f中预测1混合动力车。在三个训练/验证集(TS/VS)场景下,用两种方法(GBLUP和RKHS)估计每个特质的预测能力。PO168(红框):全部168大肠茅和大肠urophylla纯种G0亲本进行训练,168个G1随机选择杂交后代进行验证;random168(绿色):从所有1117名患者中随机选择168名TS患者,也随机选择168名VS患者;random558(蓝色):将所有1117个个体随机分为相同大小的TS和VS(558/558)。离群值估计用黑点表示。多克斯(174 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Tan,B.,Grattapaglia,D.,Martins,G.S.et al。评价了两个桉树种生长和木材性状基因组预测的准确性1混合动力车。BMC植物杂志17,110(2017)。https://doi.org/10.1186/s12870-017-1059-6
收到了:
公认:
发表:
关键字
- 基因的关系
- 基因遗传
- 大多数
- 基因组注释
- 高密度SNP-chip
- 贝叶斯套索
- GBLUP.
- rrBLUP