摘要
背景
在CESA类似的基因超家族中,纤维素合酶样D(CSLD)基因最类似于纤维素合成酶基因,并且已据报道涉及尖端的细胞和干发炎。然而,在棉花上没有这种基因亚家族的基因组。因此,我们试图基于完全测序的棉花基因组分析CSLD蛋白的进化和功能表征。
结果
共鉴定到23个CSLD全长蛋白Gossypium raimondii就,木本棉和陆地棉。系统发育树将CSLD蛋白分为五个枝条,具有强大的支持:CSLD1,CSLD2 / 3,CSLD4,CSLD5和CSLD6。的总表达式GhCSLD基因在雄蕊雌蕊和最高(主要贡献的CSLD1和CSLD4)与其他相比CSL基因。CSLD1和CSLD4在Androecium&Gynoecium(A&G)中仅表达,并显示出组织特异性表达。的总表达式CSLD2/3, 5和6在特定组织中最高。这些结果表明CSLD基因显示出不同的表达模式。棉CSLD蛋白质受到不同的进化压力,CSLD1和CSLD4蛋白表现出扩展和长期变化阳性选择。GRCSLD1的预测三维结构表明GRCSLD1属于糖基转移酶22. CSLD1谱系中的阳性选择的氨基酸残基位于与特定于类别的区域(CSR),β1股和跨膜螺旋相邻的区域中(TMHS)在GRCSLD1结构中。
结论
我们的研究结果通过包含系统发育、转录分析和3D建模的综合方法对CSLD蛋白进行了表征。本研究进一步了解了CSLD家族的重要性,并为棉花细胞壁生物合成候选基因的选择及其关联提供了有益的参考。
背景
植物细胞壁在植物发育中起着中心作用,主要由三种多糖组成:纤维素、半纤维素和果胶[1,2,3.].木质素是二级细胞壁的主要聚合物[4].纤维素由β-1,4-连接葡萄糖单元的不支链均聚物组成,是植物细胞壁的核心结构成分[5].纤维素的生物合成引起了极大的兴趣,因为纤维素微纤维是细胞壁物理特性的关键决定因素[6];为生物燃料提供可再生资源[7,8].纤维素合酶(CESA)合成纤维素,其属于糖基转移酶27(GT2)[9].CSLs和CESA组成CESA超科,CSLA, B, C, D, E, F, G, H, J,和K 10科[10,11,12].然而,水稻有6个科(CSLA, C, D, E, F和H) [13,14].CSL也是GT2的成员[9].在植物中,中国极限运动协会基于与细菌的序列同源性,首次在棉纤维中鉴定出基因中国极限运动协会基因[15].几乎完全的基因组序列拟南芥蒂利亚纳透露10.中国极限运动协会基因[2,12],分类为初级(CESA1,2,3.,5,6,9)和次要(CESA4,7,8)细胞壁合成[16,17,18].CESA1, CESA3和CESA6被认为是主壁CESA复合体的一部分,CESA5和CESA2与CESA6在不同生长阶段存在部分功能冗余[16].CESA6相关的CESA9与CESA6表现出功能冗余[17].二次壁CESA复合物包含CESA4,CESA7和CESA8,如所识别的irx(不规则的木耳突变体的A. Thaliana.[18].与主要墙壁CESA复合物相比,这三个中国极限运动协会在次级细胞壁中,基因对纤维素的合成似乎同样重要,这表明它们不是相互冗余的[18].在棉花中,CESA1, 2, 7, 8(同源A. Thaliana.CESA8,4,7和7分别在纤维素生物合成二次细胞壁中相关,而CESA3,5,6,9和10(矫形器)A. Thaliana.CESA3分别为2/5/6/9、1/10、2/5/6/9和3)参与棉纤维原代细胞壁合成[19,20.,21,22].此外,CESA8 (ATCESA7)在棉纤维发育过程中对二次细胞壁纤维素快速大量积累起促进作用,这与其他禾草品种有很大的不同[19].最近,据报道,有一个“接力赛”模式的纤维发展涉及中国极限运动协会基因海岛棉[23].
CSL编码糖基转移酶进行性基因已经在植物墙的非纤维素多糖的生物合成被指示。例如,里昂证券基因编码甘露聚糖合酶[24,25],CSLC介导木葡聚糖生物合成基因编码β-1,4-葡聚糖合酶[26],CSLF和CSLH蛋白参与(1,3; 1,4)-β-D-葡聚糖生物合成[27,28].之间CSL基因家庭,CSLD基因家族与CESA家族最为相似,拥有最古老的内含子/外显子结构[12].csld在纤维素和甘露聚糖合成中有牵连[29,30.,31].在A. Thaliana.,五分之一的突变体CSLD基因已被描述为引起不同的表型。在CSLD3突变体,根毛在起始后不久形成隆起[32,33]并且CSLD3参与在根毛细胞的顶端血浆膜中的β-1,4-葡聚糖多糖的合成[29].CSLD2突变体生长根部毛发[34],其中可能存在部分发散和冗余CSLD2和CSLD3根毛和雌配子体发育过程中的基因功能[34,35.].CSLD5突变体显著减少茎和根的生长[36.].最近的报道表明,CSLD5参与了新形成细胞板的构建,并且是在完成细胞分裂时降解的不稳定蛋白质[37.].此外,合作活动CSLD2,CSLD3和CSLD5对于正常发展是必要的[31].CSLD1和CSLD4突变体的花粉管上纤维素沉积显著减少,花粉管壁层明显紊乱,表明突变体的生长发育不正常CSLD1和CSLD4正常花粉管生长需要基因[30.,34].在水稻中,突变OsCSLD1和OsCSLD4,orthologsA. Thaliana CSLD2 / 3和CSLD5分别对叶片形态发生和工厂建筑具有重要影响[38.,39.,40.,41.].OsCSLD1突变体的根毛异常[38.].玉米CSLD1(与A. Thaliana.CSLD5)所需的细胞分裂,膨胀和叶生长[42.].以上主要研究表明,CSLD蛋白可能参与顶端生长细胞(花粉管和根毛)的纤维素合成和茎的生长。
棉花是最重要的经济作物之一,其纤维是纺织工业的主要天然来源[43.].棉也是研究多倍体,细胞壁生物合成和细胞伸长的优秀模型系统[44.,45.,46.].尽管取得了突出的进展A. Thaliana.和米饭,对棉花的CSLD蛋白很少。棉花CSL基因参与了围绕棉花纤维素微纤维的细胞壁基质多糖的合成[20.].基因CSLD2/3和CSLD6但不是CSLD1和CSLD4在纤维发育过程中表达强烈[21,45.].的CSLD2/3基因也被认为参与了棉纤维细胞发育过程中甘露聚糖的合成[47.].
最近组装和公布的基因组序列Gossypium raimondii就[20.],Gossypium植物园[48.]和gossypium hirsutum[45.提供一个机会来识别和分析CSL全基因组水平的基因家族。为了深入了解棉花基因组CSLD蛋白的进化和功能特征,我们对CSLD蛋白进行了鉴定,并构建了最大似然(ML)和贝叶斯系统发育树,重建了CSLD蛋白的进化起源CSLD基因。然后分析基因表达,QRT-PCR和阳性选择。最后,我们生成了CSLD1的三维结构模型,以阐明CSLD1的功能。我们展示了1)23个全长CSLD蛋白分为五个枝条;2)CSLD与CESA等基因的表达模式不同CSL基因;3)CSLD1和CSLD4 CLADES表现出集体和长期换档阳性选择;4)GRCSLD1蛋白属于糖基转移酶系列2,并且可能参与纤维素,甘露甘露糖或其他多糖的生物合成。这些结果提供了纯棉CSLD蛋白的进化和生物学和分子功能的彻底了解。
结果
CSLD蛋白在棉花基因组中的分布
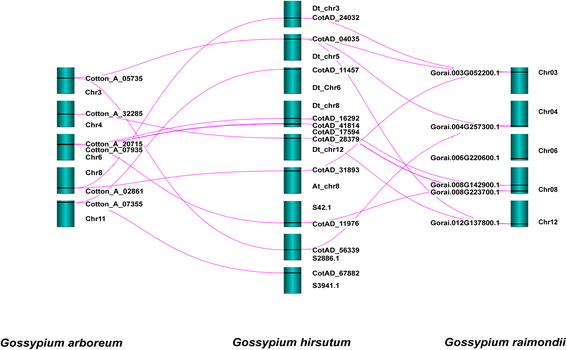
棉花全基因组序列的获得为进一步鉴定和分析CSLD蛋白的进化和功能提供了机会。其中1923 Mb(88.5%)、1532 Mb(90.4%)和761.4 Mb(99.95%)被锚定并定向到26条假染色体G. Hirsutum[45.,到13个假染色体G.植物园[48.]和13个假瘤组织g . raimondii就[20.),分别。通过同源性蛋白搜索,我们鉴定了23个CSLD全长蛋白G.植物园(六),G. Hirsutum(11)和g . raimondii就(六)(表1)和86个CSLD蛋白,来自15种其他植物物种,如预期(附加档案1:表S1,附加文件2).G. Hirsutum大约是许多CLSD蛋白的两倍G. Arboreum.或g . raimondii就.在棉花中,一些CSLD蛋白有一个或两个cellulose_synt (PF03552)结构域和一个zf-RING_4 (PF14570)结构域,但其他CSLD蛋白只有一个或两个cellulose_synt (PF03552)结构域(图)。1).的CSLD基因分布在6条染色体(Dt_chr3, 5, 6, 8, 12和At_chr8)和3个支架(S42.1, S2886.1, S3941.1)上G. Hirsutum,五种染色体(Chr3,4,6,8和11)G. Arboreum., 5条染色体(Chr03, 04, 06, 08, 12)g . raimondii就.大多数染色体分布CSLD基因包含一个单一的位点的CSLD基因,除染色体6中G. Arboreum.,染色体Dt_Chr8在G. Hirsutum染色体08g . raimondii就,包括2、3和2CSLD基因位点,分别(图2).同期位置G. Arboreum.和g . raimondii就与G. Hirsutum(图。2).鉴定了一到两个的同步关系G. Arboreum.或g . raimondii就和G. Hirsutum除了CHR4(两对一的同时性关系G. Arboreum和G. Hirsutum), Chr06(两者之间没有共序关系雷蒙地葛和陆地葛)和Chr03(一到三个之间的关系同线雷蒙地葛和陆地葛).
使用IBS软件说明的领域体系结构[112].GRCSLD,CSLD蛋白质g . raimondii就根据图1中的CSLD蛋白的系统发育树。3.;zf-RING_4, zf-RING域(蓝色);cellose_synt, cellose_synt域(绿色)。GrCSLD2/3、GrCSLD5和GrCSLD6具有一个cellulose - synt结构域和zf-RING_4结构域。GrCSLD1只包含一个cellulose_synt结构域,而GrCSLD4包含一个zf-RING_4和两个cellulose_synt结构域。图中显示了两个GrCSLD2/3 s。1因为GORAI.004G257300.1,GORAI.003G052200.1,AT_CSLD2和AT_CSLD3形成单晶群(图。3.).这些域的功能域和位置通过序列搜索与在线程序Smart,Interpro和NCBI保守域数据库进行序列搜索
棉CSLD蛋白的进化
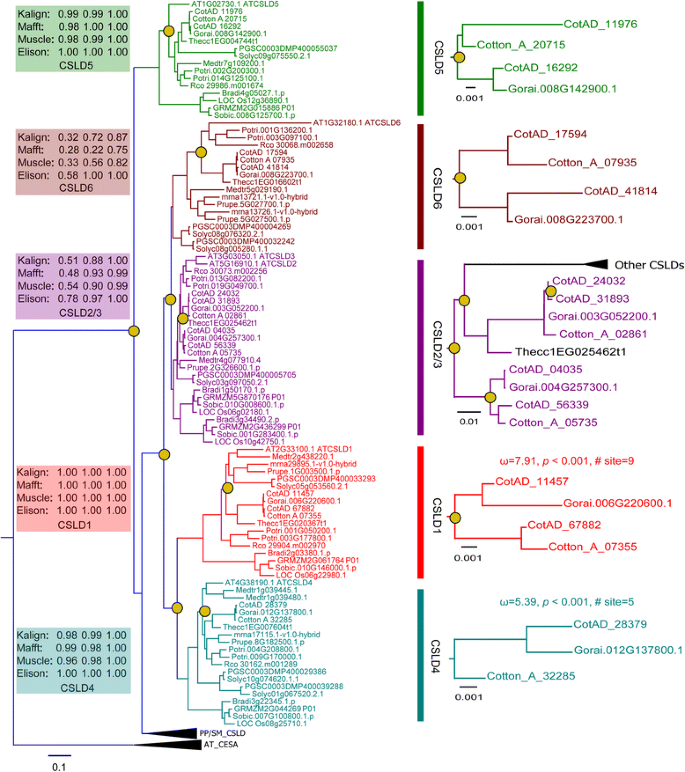
重建系统发育树,我们使用了不同的对准方法,进化模型和多种统计支持措施(见方法详细信息部分)。用PROTTEST3.2分析每个对准,选择最合适的氨基酸替代模型,用于推断最大似然(mL)系统发育树。根据AIC,AICC和BIC标准选择LG + I + G + F型号作为最佳模型(附加文件3.:表S2)。为了进一步验证PhyML重建的系统发生树的稳健性,我们使用MrBayes在混合模型下推断贝叶斯系统发生树,该模型整合了所有可用的替代模型,而不是指定一个氨基酸替代模型[49.].在这种方法中,每个多个氨基酸替代模型对结果的贡献都与其后验概率成比例。使用Ktreedist从ML和贝叶斯方法获得的系统发育树的比较显示在附加文件中4:表S3。基于K-score和对称差异(Robinson-Foulds距离),基于省略和Muscle排列的ML树和贝叶斯树具有几乎相同的拓扑和分支长度(K-score, 0.09和0.13;然而,基于两种对齐方式(Kalign和Mafft)的ML和贝叶斯树表现出比省略和肌肉对齐更大的拓扑差异。
基于elision序列的贝叶斯树将CSLD蛋白划分为5个强支持分支:CSLD1、CSLD2/3、CSLD4、CSLD5和CSLD6A. Thaliana.[12,35.].为关键节点的支持增加时,我们使用了省音策略,串接的多重比对,和MrBayes的混合模型方法,这表明最可靠的对准位置一致地支持在其中CSLD蛋白被分类成五个分支系统发育(图.3.).与对整个CSLD系统发育树的分析一样,棉花CSLD系统发育被稳健地划分为5个分支,当我们使用省略策略时,几乎所有节点的支持值也增加了5:图S1)。从ML和贝叶斯方法推断出的棉花CSLD树之间基于Kalign、Mafft和Muscle三种对齐方式的拓扑差异显示在附加文件中5:图S1。
CSLD蛋白的系统发育分析Gossypium.并使用其他15种植物A. Thaliana.CESA基因作为外群(附加文件1:表S1)。利用极大似然法和贝叶斯方法推断系统发生树。关键节点的支持值显示为bootstrap比例/类似sh的aLRT分数/贝叶斯后验概率。CSLD蛋白分支用不同的颜色表示。复制事件被注释为黄铜圆。PP/SM_CSLD表示中CSLD蛋白p .金属盘和美国moellendorffii.AT_CESA礼物A. Thaliana.CESA蛋白作为外群。ω表示dN:dS值。的P值用Bonferroni校正
系统进化树的拓扑结构表明CSLD祖先基因复制发生在苔藓谱系从维管植物分化之前。祖先基因的两个副本各自进化,导致CSLD5思克和另一个CSLD片状(图。3.).棉花的拓扑CSLD树与整体相同CSLD系统发育树,表明棉花CSLD祖先基因分裂为CSLD5思工和其他CSLD片状,后来分为了CSLD1,CSLD2/3,CSLD4,和CSLD6通过基因重复亚克士片。来自三个的CSLD蛋白Gossypium.种形成5个单系群,每个群由3或4个CSLD蛋白组成。CSLD1和CSLD2/3形成姐妹团体CSLD4和CSLD6,分别。的GhCSLD基因,除了CSLD4,最近再次被复制(图。3.)通过约150万年前两个祖先种的杂交(MYA)45.].两者g . raimondii就和G. Arboreum.经历了一个古老的六倍体化事件,在115-146 MYA的双二体中共享,然后在13-20 MYA经历了一个棉花特异性全基因组复制[43.,48.].这些结论支持多重CSLD三种棉花的基因复制。
棉花的表达概况CSLD基因
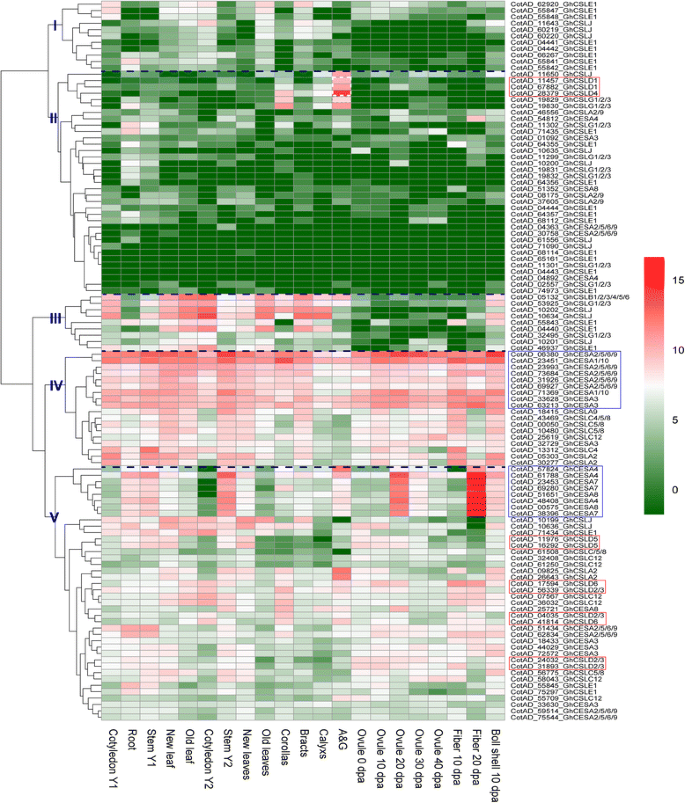
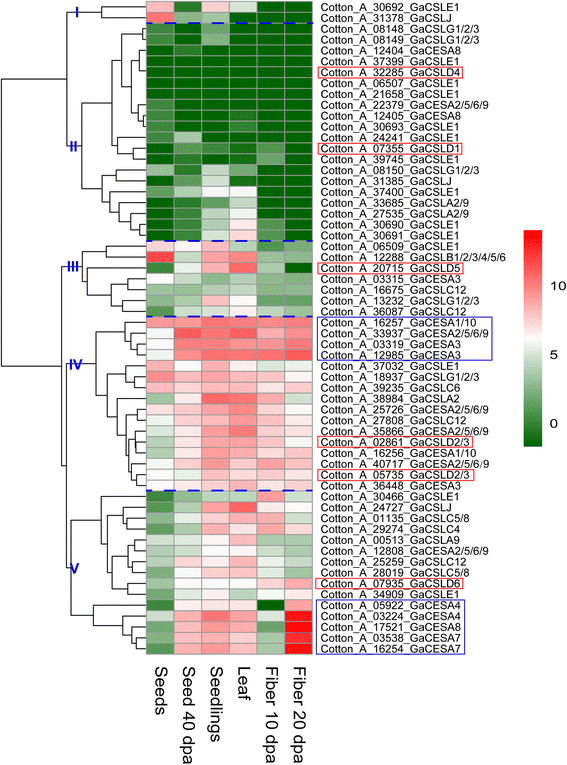
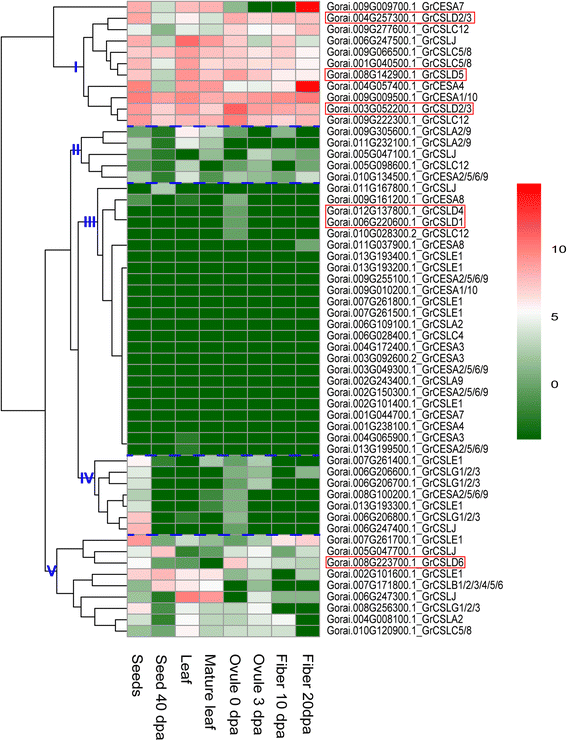
基因表达谱分析可以为了解基因功能提供有用的信息。表明是否CSLD基因有中是独一无二的功能中国极限运动协会/ CSL超家族,我们进行了基因表达和qRT-PCR分析。之前的报告已经表明OSCESA基因在大多数被检测的组织中高度表达OSCSL.基因有相当多变的表达[13].基于层次聚类分析中国极限运动协会/ CSL基因超家族可分成五大组G. Hirsutum,G. Arboreum.和g . raimondii就(图。4,5和6).CSLD1和CSLD4在显示出高表达水平的基团的II雄蕊及雌蕊(A&G)G. Hirsutum.然而,成绩单CSLD1和CSLD4在其他组织中是零或非常小的分数G. Hirsutum,G. Arboreum.和g . raimondii就(图。4,5和6额外的文件6:表S10,S11和S12)。CSLA2.,CSLBCSLG,CSLJCESA1/10,CESA3和CESA4在A&G的表达G. Hirsutum,但是这些基因,除了CotAD_11650_GhCSLJ,在其他组织中也有表达(图。4额外的文件6:表S10)。这一结果表明CSLD1和CSLD4似乎有很强的特异性,这与报告相似OSCSLD3和5(直接同源答:芥CSLD1和CSLD4)在水稻的雄蕊(花粉)中表达较强A. Thaliana.[13].CSLD5主要表达于幼苗、根(胚根)、茎、叶和胚珠等CSL在这些组织中也表达基因(图。4,5和6额外的文件6:表S10,S11和S12)。GHCSLD6在所有组织中均有表达,在老叶、苞片、胚珠30dpa和40dpa表达较低。4额外的文件6:表S10)。CSLD 6在纤维中表达强烈,与之前的报告一致[21,45.].然而,GrCSLD6在胚珠中仅表现出强烈的表达(图。6).GhCSLD2/3不同的是,基因分为三个不同的组(V字)ghcsld1.,4和5,分为一组(图。4).CSLD2/3显示种子,幼苗,子叶,根,茎,叶,花冠,卵子,纤维和棉铃壳中的主要表达(图。4,5和6额外的文件6:表S10,S11和S12)。CSLD2/3三个不同群体的基因(两个不同的群体G. Arboreum.和g . raimondii就)显示不同的表达模式(图。4,5和6).这些结果表明CSLD2/3在不同发育阶段的细胞壁合成中具有多种功能。
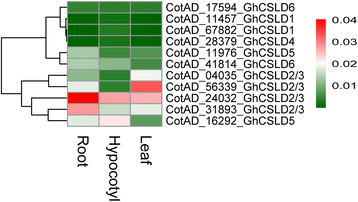
一般来说,中国极限运动协会基因在检查的所有组织中显示出广泛的高表达(图。4,5和6).CESA2/5/6/9 Gh (a),1/10和3.在所有组织的原发性细胞壁生物合成中表达。GH(a)CESA4,7和8在组织的次级细胞壁中强烈表达,例如,纤维20dPa。表达模式中国极限运动协会基因与那些相似OSCESA和艾尔斯卡[13].全部CSL基因表现出相对的组织特异性表达中国极限运动协会,这是组成的。的总表达式GhCSLA基因在子叶Y1中表达量最高,其次是铃壳,在子叶Y2中表达量最低。与此形成鲜明对比的是,总表达GhCSLB子叶Y2中基因含量最高。的总表达式GhCSLC,D,E,G和J在茎Y1、A&G中基因含量最高(主要由CSLD1和CSLD4),根,子叶y1和子叶y2,分别(附加文件6:表S10)。的总表达式GaCSLA和E苗期基因含量最高。的总表达式Gacslb.和G种子中基因含量最高。的总表达式Gacslc.种子40 DPA中最高。的总表达式Gacsld.(主要是贡献CSLD5)在叶片中最高(附加文件6:表S11)。GrCSLA基因在所有组织中都表现出几乎无法检测的表达。的总表达式GrCSLB种子40 DPA中最高。的总表达式GrCSLC和J成熟叶中最高。的总表达式GrCSLD(主要是贡献CSLD2/3)胚珠0 dap最高。的总表达式GrCSLE和G种子中最高(附加文件6:表S12)。这些结果表明CSL整个家族的基因经常在一个或多个组织中积累到高水平CSL成员展示了偏好,类似于稻米的前一份报告[13].以获得更多的洞察是否表达CSLD基因的差异,我们用特异性引物进行qRT-PCR实验G. Hirsutum(附加文件7:表S13)。在所有11分析的情况下GhCSLD基因,一份副本GhCSLD2/3(Cotad_24032)在所有组织中具有最突出的表达水平(图。7),其次是GhCSLD2/3(Cotad_56339),GhCSLD2/3(CotAD_31893)和GHCSLD5.(CotAD_16292)。ghcsld1.和GHCSLD4.表明在所有的组织中几乎检测不到表达。GHCSLD6以中等水平表达。总的来说,qRT-PCR表达数据的结果与RNA-seq的结果基本一致(图1)。4).结果表明CSLD基因表现出与其他基因不同的表达模式CSL基因,以及所有的CSLD在正常生长条件下,棉花不同组织中基因的表达存在差异,说明了棉花的功能分化CSLD基因在棉花。
GrCSLD1蛋白阳性选择鉴定
阳性选择增加了赋予携带这些突变的个体的新健身优势的突变突变频率[50.].对于蛋白质编码的DNA序列,阳性选择是通过非同义/同义突变率的比率表示(ω=牛顿/ DS)大于一[51.].如果该基因与植物-病原体竞争有关,则可能发生正向选择[52.]如果在重复点出现新的和有益的功能[53.,以回应压力[54.等等。
如果它在大多数或所有分枝的系统发生的情况下,可以定义一个现场的长期阳性选择[54.,55.].我们仅专注于定义棉花大型棉质的分支,表示CSLD1至CSLD6(图。3.).我们使用了Notung方法[56.]通过在疏水板水平上应用分支站点模型(在每个指定的分支机构中的所有分支机构)来推断基因复制并确定重复后的长期正选择。两个分支(CSLD1和CSLD4)在Bonferroni校正后表现出焦点正面选择。九次和五个地点被确定为分别经过重复的经历阳性选择,分别经历经验贝叶斯分析(表2).CSLD1和CSLD4还显示了棉花谱系各个分支的积极选择中的长期转变。在CSLD1和CSLD4片状中检测到十三个具有显着选择阳性选择的迹象。阳性位点的氨基酸残基显示在表中2.
表中积极选择的地点2位于GRCSLD1蛋白的预测三级结构上。K116在空间上靠近特定于类区域(CSR),A244位于β1股上(附加文件8:图S3和附加文件9:图S4)。C157,C795,Q894,A912,C917和C947位于跨膜螺旋(TMH)中。此外,C795,A912和C917位于Memsat预测的跨膜孔隙中(附加文件8:图S3和附加文件9:图S4)。
GrCSLD1蛋白的结构
在A. Thaliana.,CSLD1和CSLD4在花粉管和成熟花粉粒中均有高水平表达,而在CSLD1和CSLD4突变体(30.].在萌发之前,CSLD1和CSLD4蛋白质在GOLGI装置中定位,然后在花粉管尖端的血浆膜中运输到血浆膜中[30.,34].这些结果表明,所述CSLD1和CSLD4蛋白可能存在不同的纤维素合成在花粉管细胞尖端生长期间根尖质膜活动。为了深入了解棉花CSLD1蛋白质的功能,结构模型,通过基于模板的和无模板模型建造。
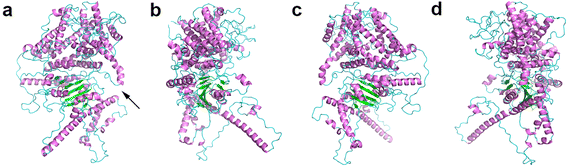
i-tasser,phyre2和robetta(具有比较造型的域的预测,见方法)均与BcsA一起使用[57.]作为主要模板。全长GRCSLD1蛋白的模型验证评分如附加文件所示10:表S15。我们确定了Robetta预测的得分最高的模型。通过与BcsA的最高分模型的结构对齐,得出的TM-score为0.65,表明GrCSLD1与BcsA具有相同的fold [58.].
所有已知的CSLD蛋白在CAZy数据库中被归类为GT2家族[9].预测GT2蛋白为转化酶,即在转移反应中,异位糖碳的构型发生了倒置[59.].GT2家族包括纤维素合酶,β-1,4-甘露合酶和甲壳素合酶。GT结构域的GT-A折叠由七个α-螺旋组成,三个两亲界面(IF1-3)连接到跨膜区域,以及类似于Rossmann折叠的七链β-纸张[57.,60.].然而,棉花CSLD蛋白的三维结构尚未得到解决。
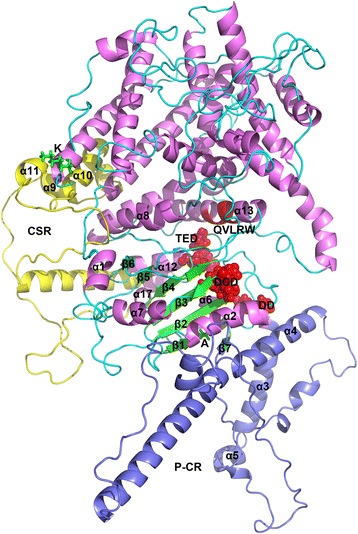
我们的结果表明,预测的GrCSLD1结构包含31个α-螺旋和9个β链(图1)。8和9额外的文件9:图S4)。GrCSLD1核域与GhCESA1叠加[61.]及BcsA [57.]在UCSF-Chimera中使用MatchMaker [62.].叠加的GrCSLD1 BcsA和GhCESA1结构匹配的域包括8螺旋线(α2α4α,α,α,α,α,α17)和7β链组成的β片(β1、β2、β3β,β,β,β7),用一个总均方根偏差(RMSD), 1.84和6螺旋线(α,α,α,α,α12日α13)和6条β链(β1, β2, β3, β4, β5, β6),总RMSD分别为2.74 Å。9和10额外的文件9:图。S4)。因此,GRCSLD1的核心结构域包含8α-螺旋(α2,α4,α6,α7,α8,α12,α13,α17)和形成Rossmann折叠的七链β-片材(图。9额外的文件9:图S4)。GRCSLD1和GHESA1的核心域表明了结构一致性,即使GHESA1结构未用于预测GRCSLD1模型。通过对GHESA1进行类比,GRCSLD1的催化袋包括紧密排列,保守的DD,DCD,TED和QVLRW图案。α13螺旋定位在与IF2相对应的这些保守的基序附近,其与纤维素受体衬底相互作用(图。10)在BCSA [57.].GRCSLD1仅包含Cellulose_synt域(图。1),CSLD2/3基因也被认为参与了棉纤维细胞发育过程中甘露聚糖的合成[47.].这些结果表明GRCSLD1属于GT2,并且可能参与纤维素,甘露甘露糖或其他多糖的生物合成。
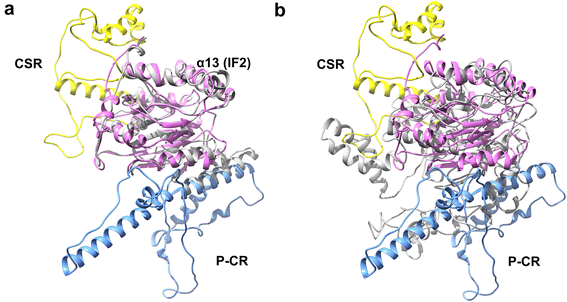
GrCSLD1的结构模型,显示了阳性选择下氨基酸残基的位置、植物特异性区域和活性位点基序。GrCSLD1、P-CR和CSR的结构分别为紫色、淡蓝色和黄色。核心结构域包含8个α-螺旋(α2, α4, α6, α7, α8, α12, α13, α17)和7股β-薄片。α-螺旋和β-股的编号是基于它们在GhCESA1二级结构中的顺序(附加文件9:图。S4)。红色亮点DD,DCD,TED(球)和QVLRW。受到正选择作用的位点(K和A)显示为青枝
讨论
CSLD蛋白具有保守的D, D, D, QXXRW基序,属于古老的纤维素合成酶超家族[10,12].除了CESA之外,CSLD蛋白质还是具有ZF环的超家族的唯一成员(图。1),该结构域被认为在蛋白质-蛋白质相互作用中起作用[12].尽管其重要性对尖端的细胞和茎生长至关重要,但CSLD蛋白仍然明显差。
守恒的同线性的CSLD分布在棉花基因组上的基因
我们确定了23个全长CSLD蛋白:六,六到11个亚洲棉,陆地棉和g . raimondii就分别(表1).的CSLD基因分布在几个染色体上。所有的保守的同音CSLD观察到基因G. Arboreum.和G. Hirsutum.这些是一对决的同义关系,除了CSLD4CHR4上的基因,它们之间具有一对一的同步关系G. Arboreum和G. Hirsutum(图。2).一对一的同步关系CSLD4基因存在,因为CSLD4是单拷贝的基因G. Hirsutum.然而,保守的同音CSLD1基因之间没有检测到g . raimondii就和G. Hirsutum(图。2).有一对到一对的同向关系CSLD4,CSLD5,CSLD6还有一份CSLD2/3.未鉴定出同宗关系CSLD1,可能是因为SyMAP[注释字符串搜索]隐藏了synteny命中。63.].的一个副本中的一到三的同步关系CSLD2/3在CHR03之间g . raimondii就和G. Hirsutum可能是由两个密切相关的亚型(ATCSLD2/3)引起的A. Thaliana..1 - 2同向关系多见于G. Arboreum.或g . raimondii就和G. Hirsutum因为G. Hirsutum基因组来源于A的杂交2和D.5基因组祖先[45.].同源图与CSLD系统发育关系比较表明,同源图与CSLD系统发育关系最为密切CSLD同生块中的基因形成了一个单系分支,表明CSLD基因在相当长的时间内得到了保守,而植物中的基因已经进化。
最近的研究表明,目前的异源四倍体G. Hirsutum是由A杂化而来的2和D.5基因组祖先约1.5 MYA [45.].的G. Arboreum.和g . raimondii就基因组经历了两轮全基因组复制(WGD),估计分别发生了约13-20和115-146 MYA [43.,48.].古代复制事件对应于逃避者之间共享的古代六翻来事件[64.].G. Arboreum.和g . raimondii就大致相同的CSLD数量为A. Thaliana.,栽培稻和玉米,和G. Hirsutum大约是?的两倍G. Arboreum.或g . raimondii就.此外,棉花的CSLD蛋白和那些Physcomitrella金属盘和卷柏moellendorffii与CSLD5形成姐妹组。这些结果也表明CSLD棉花基因是保守的,有一个古老的基因家族,并不断扩大CSLD基因与WGD相关。
重建CSLD蛋白的进化树
多序列对准(MSA)的质量对给定的系统发育树的鲁棒性具有深远的影响[65.].因为基因以不同的速度进化,一个序列中的一些区域非常保守,适合进行系统发育分析,而另一些区域则充满了缺口,差异很大。这些不同的区域不能精确地对齐,因此必须在系统发育分析之前将其移除[66.].系统发育重建通过检查替代树木,以及量化序列数据支持或抑制不同系统发育结果的程度,产生真实历史的估计。最大可能性[67.和贝叶斯推理[68.]是构建系统发育树最常用的方法。因此,在本研究中,我们使用了多种对齐策略(Kalign、Mafft和Muscle)、支持测度(sh类近似似然比检验、非参数自举比例和贝叶斯后验概率)和对齐修剪(Gblocks)。
结果表明,基于ML和贝叶斯方法推断的CSLD系统发育树可以分为5个强支持支系。CSLD蛋白的系统发生树分为五个分支,与其他已知影响系统发生树准确性的因素(包括统计支持措施和进化模型)相比,也是稳健的。然而,基于两种对齐方式(Kalign, Mafft)和省略策略,从ML和贝叶斯推断出的棉花CSLD5和CSLD2/3树的拓扑结构存在一些差异(Additional file5:图S1)。CSLD5和CSLD2 / 3树的贝叶斯树基于两个对齐(Kalign和Mafft)具有多重性。我们考虑这些柔软的多元质,因为来自其他方法的树是完全二进制的。多瘤的外观可能是由于矛盾的数据矛盾的结果和缺乏关于蛋白质的真正分叉模式的信息[69.].基于Muscle和省略排列的ML和贝叶斯树显示了几乎相同的拓扑结构,并最好地估计了CSLD蛋白的真实进化。我们使用基于Muscle (bootstrap分支支持)的ML树来推断重复和评估积极选择。棉花CSL基因参与了棉花中围绕着纤维素微纤维的细胞壁基质多糖的合成[20.].CSLD2/3和CSLD6但不是CSLD1和CSLD4基因在纤维发育过程中表达强烈[21,45.].CSLD2 / 3蛋白还建议参与棉纤维细胞发育过程中的人类合成[47.].这些结果表明,CSLD蛋白可能参与了纤维素、甘露聚糖或其他多糖的生物合成。
描述的CSLD基因家族
A. Thaliana.水稻提供了理解棉CSLD蛋白的功能的参考点。CSLD蛋白A. Thaliana.可能参与尖端生长细胞(花粉管和根毛)中的纤维素合成,茎生长和甘露合成,这表明CSLD蛋白已获得不同的功能。
展示功能表征CSLD进行基因表达和qRT-PCR分析。之前的报告已经表明OSCESA基因高表达,OSCSL.基因有相当多变的表达[13].几乎所有的中国极限运动协会棉花基因在检查的所有组织中表现出高表达,这意味着它们在纤维素生物合成中的主要作用,细胞壁的核心结构组分。CESAS.(2/5/6/9,1/10和3.)和CESAS.(4、7和8)强烈联合表达(在IV和V组),暗示CESAS.(2/5/6/9,1/10和3.)和中国极限运动协会(4、7和8)可能形成两个合成复合物,参与初级和次级细胞壁合成,如在模式植物中观察到的A. Thaliana.[2,12]和米[13].结果也与CESA1, 2, 7, 8(同源性)的报道一致A. Thaliana.CESA8,4,7和7分别在纤维素生物合成二次细胞壁中相关,而CESA3,5,6,9和10(矫形器)A. Thaliana.分别为2/5/6/9,1/10,2/5/6/9和3)参与棉花初级细胞壁合成[19,20.,21,22].
一份GRCESA1 / 10.基因在所有组织中均有高表达GrCESA4和7在纤维20dPa中强烈表达,这表明涉及细胞壁的生物合成的GRCENA蛋白质较少(图。6额外的文件6:表S12)。相比之下,中国极限运动协会的基因,CSLD基因在一个或多个组织中表达。
在CSL基因超家族,总表达CSLD基因与其他基因不同CSL基因。此外,不同的拷贝CSL基因显示出不同的表达模式。CSLD1和CSLD4在A&G中强烈共表达,并表现出组织特异性表达(图。4和7),建议CSLD1和CSLD4可能形成与多糖有关的合成复合物。的整体表达CSLD2/3和CSLD5基因分别在根和叶中最高(附加文件6:表S10)。这些结果与之前的报道一致A. Thaliana.和大米[13,30.,34].CSLD 6在纤维中表达强烈,与之前的报告一致[21,45.].GrCSLD6仅在胚珠上表现出表达(图。6);但是,ATCSLD6似乎是一个伪基因[31].这些结果表明CSLD基因表明了相对组织特异性的表达,表明它们在多糖的生物合成中的潜在不同功能。
GRCSLD1阳性选择下氨基酸的空间分布
Branch-site模型分析表明,主要进化支的选择压力存在差异,说明棉花CSLD蛋白的某些位点在进化过程中受到了不同的约束。这些限制是由CSLD蛋白的不同功能和进化起源所决定的。CSLD2/3、CSLD5和CSLD6进行了松散的纯化筛选。然而,CSLD1和CSLD4基因复制后,棉系各分支均表现出间歇性正向选择和长期转移正向选择。CSLD1(ATCSLD4),CSLD2和CSLD4(ATCSLD5)在草地上显示出强烈的正选择信号[54.].棉花和草可能有不同的进化压力。的CSLD1和CSLD4花粉管的正常生长需要基因A. Thaliana.,可能参与花粉管纤维素合成[30.,34].基因表达和QRT-PCR分析表明CSLD1和CSLD4基因仅在A&G中表现出强烈的表达,并具有来自其他的不同表达模式CSLD与其他CSLD蛋白相比,基因暗示CSLD1和CSLD4在多糖的生物合成中具有潜在不同的功能。我们的结果表明CSLD1和CSLD4基因可能在基因复制通过积极选择中的长期班次复制后进化新功能。
最近报道的一个细菌纤维素合成酶复合物的A和B亚基的三维结构Rhodobacter sphaeroides[57.]和棉花GhCESA1的计算模型[61.]提供一个机会,用于定义GRCSLD1中肯定选定的站点的三维分布。然而,CSLD蛋白的不同功能仍然是未知的。有些报道表明,CSLD蛋白质与纤维素和甘露植物生物合成相关[29,30.,31,47.].没有直接的证据表明,GrCSLD1蛋白具有独特的催化功能。基于GrCSLD1蛋白的预测的三维结构功能表征是非常困难的。GrCSLD1含有保守cellulose_synt域(图1)与其他植物中的其他功能已知的CSLD蛋白质和BCSA和GHESA1表现出与其他功能已知的CSLD蛋白的系统发育关系。预测GRCSLD1的三维结构含有Rossmann折叠,并且具有保守的D,D,D,QXXRW基序(图。9).这些结果表明,GrCSLD1属于GT2;然而,GrCSLD1的具体作用尚不清楚。我们仅认为GrCSLD1可能参与了纤维素、甘露聚糖或其他多糖的生物合成。
根据CSLD1谱系的阳性选择鉴定的氨基酸残基位于GRCSLD1结构中的CSR,β1-股和TMH附近的区域上(图。9额外的文件8:图S3和附加文件9:图S4)。GRCSLD1的结构显示CSR和P-CR折叠成细胞溶质区域内的不同亚域。CSR区域可能通过非共价互动有助于稳定CESA组装[61.].K116是一个在空间上与CSR区域相邻的正选择残基,它可能有助于稳定CSLD1组装成与其他csld的复合物(CSLD1和CSLD4基因在A&g)中强烈表达,类似于CESAS。有趣的是,A244位于GRCSLD1的核心结构域内的β1-股上,这表明该残余物具有影响GRCSLD1活性的潜力。C157,C795,Q894,A912,C917和C947位于预测的TMHS中,并在跨膜孔内涉及挤出细胞膜的新生多糖的挤出(附加文件8:图S3和附加文件9:图S4)。我们的分析表明,GrCSLD1在正压下的残基对酶的活性或功能以及酶合成的多糖的精细结构都有较为显著的影响。这些位点在GrCSLD1阳性选择下的具体作用尚不清楚,值得进一步研究。
最近的报道显示CSLD蛋白不包括在水稻细胞壁聚合物生物合成模块中[70]并且CSLD蛋白质不与棉花中的纤维素合成酶复合物相互作用[71.].CSLD1和CSLD4基因可以特别参与生长花粉管尖端的纤维素的生物合成,并且在成熟的花粉颗粒和花粉管中高度表达A. Thaliana.[30.].这些结果还表明CSLD1和CSLD4可能用作纤维素生物合成中的复合物。
结论
CSLD系列在社区内仍然相对不协调,以及关于其进化历史和功能的许多问题。在这项研究中,我们进行了严格的系统发育分析,具有最大可能性和贝叶斯方法来解决棉花中CSLD蛋白的系统发育拓扑。在确定表征的背景下进行阳性选择,基因表达分析和QRT-PCR分析的测试CSLD基因,与...相比中国极限运动协会和其他CSL基因。这些分析补充了GRCSLD1同源性建模,以提供CSLD蛋白的进化和功能表征的结构背景。这些数据为了解CSLD蛋白在棉花中的进化历史和3D建模提供了依据。
方法
鉴定CSLD蛋白
我们使用已确认的CSLD功能蛋白序列A. Thaliana.作为查询从棉花完全测序的基因组中鉴定新的CSLD蛋白质同源物(亚洲棉,陆地棉和雷蒙德氏G.)及15种其他植物(附加档案1表S1)使用BLASTP (E-value≤1E-5) [72.].的G. Arboreum.和G. Hirsutum从CGP (http://cgp.genomics.org.cn).g . raimondii就从Phytozome (V11)中检索到其他15个全序列植物基因组[73.].为了区分CSLD与CESA和其他CSLD,我们使用候选CSLD的蛋白组进行搜索A. Thaliana.利用BLASTP从Phytozome中提取。每个真正的CSLD都应该标识一个CSLDA. Thaliana.作为顶根据纤维素的命名法命中超家族合酶A. Thaliana.[12].中国极限运动协会外显子比CSLD.通过与在线程序智能的序列搜索验证所有获得的序列的保守结构域[74.],Interpro [75.]和NCBI保守的域数据库[76.].同线性块之间G. Hirsutum和G. Arboreum.或g . raimondii就默认使用SyMAP检测[63.]和果馅奶酪一起观察[77.].
多个序列对齐
所述CSLD蛋白序列使用Kalign V2.04使用默认参数[对齐78.],来自Mafft v7.215的电子Ins-i方法[79.],肌肉v3.8.31 [80].使用Gblocks v0.91b对结果对齐中的发散和模糊对齐区域进行修剪[81.在系统发育分析之前。我们还通过连接所有三个单独的GBLOCKS处理对齐来制作精选对齐[82.].
系统发育分析
使用PHYML V3.0重建最大似然性系统发育树木[83.],采用抗议型V3.2选择的氨基酸替代型氨基酸替代模型[84.].使用sh类近似似然比检验估计分支支持[85.]和非参数自举比例(500次重复)。使用MrBayes v3.2.5重建贝叶斯系统发育[49.].我们集成了氨基酸替代模型(PRESET AAMODELPR =混合),并假设跨站点的离散伽马分布式速率变化模型。每那项第100代采样马尔可夫链,初始化25%的样品被抛弃为烧伤,其余的样本用于产生共识树。我们通过运行两个同时,独立的分析并终止分析来评估链接趋同,当两次运行之间的分流频率的平均标准偏差低于0.01时。比较来自ML和贝叶斯重建的系统发育树,用于使用Ktreedist的拓扑和分支长度进行比较[86.].
基因表达和QRT-PCR分析
高通量RNA测序数据从国家生物技术信息中心的简短阅读档案下载(http://www.ncbi.nlm.nih.gov/sra.额外的文件11:表S4,S5和S6)。将RNA-SEQ读数映射到与TOPHAT2的参考棉花基因组[87.].包含来自TopHat2的映射读的文件集使用samtools进行排序和索引[88.].使用HTSEQ计数计算与基因的读数重叠[89.].对基因计数进行归一化和离散估计,并用DESeq(附加文件)转化为方差稳定数据12:表S7,S8和S9)90.,91.].我们根据方差稳定转换数据制作了热图中国极限运动协会/ CSL使用phatmap package (pheatmap: Pretty Heatmaps, R package version 1.0.8,https://CRAN.R-project.org/package=pheatmap).我们使用PF03552 (Cellulose_synt)和PF00535 (glycos_trans_2)作为查询,从棉花全序列基因组中鉴定新的CESA/CSL同源蛋白(G. Arboreum.,G. Hirsutum和g . raimondii就)使用HMMER 3.1b2包[92.].CESA / CSL蛋白序列使用肌肉V3.8.31对齐[80].使用Gblocks v0.91b对结果对齐中的发散和模糊对齐区域进行修剪[81.在系统发育分析之前。使用PhyML v3.0重建最大似然系统发育树[83.],由Protest V3.2选择的最佳拟合模型(JTT + G + F)[84.].CESA/CSL蛋白序列A. Thaliana.从Tair 10数据库下载(https://www.arabidopsis.org.) [93.].所有确定的中国极限运动协会/ CSL棉花的基因(G. Arboreum.,G. Hirsutum和g . raimondii就)基于外向性序列提供特定名称A. Thaliana.(附加文件13:图S2)。G. Hirsutum(中国棉花品种银山2号,河南秋乐种业科技有限公司)在28°C的生长室中生长,光照14 h,暗10 h。当3片完全展开的叶片出现时,分别采集根、下胚轴和叶片,立即在液氮中冷冻,在−80℃保存至RNA提取。每个样品经过3个生物重复。根据制造商说明(TaKaRa),使用Trizol试剂从根、下胚轴和叶中提取总RNA,并使用RNase-free DNase i进行广泛处理。1 μg总RNA用第一链cDNA Synthesis Kit (Invitrogen)合成cDNA。的引物CSLD基因G. Hirsutum专为QRT-PCR分析而设计的额外文件7:表S13。如前所述进行QRT-PCR [94.,95.].比较2——ΔΔCT方法用于计算相对表达水平CSLD基因(额外的文件14:表S14) [96.].QRT-PCR分析的热线映射由Pheatmap封装(Pheatmap:漂亮的热手套,R包1.0.8)产生,https://CRAN.R-project.org/package=pheatmap).
积极的选择
在slicodeml中实现了棉花CSLD蛋白编码基因的分支位点模型在整个系统发生中的阳性选择检测[97.,98.].在这个模型中,我们测试正选择的分支称为前景分支,而树上的所有其他分支称为背景分支。假设密码子的ω比随密码子位点的变化而变化,将密码子序列分为4类。场地类别0(与比例p0)包括高度保守的密码子或在所有分支上经过净化选择而进化的密码子,其中0 <ω0<1.网站1(按比例p1)包括中性的密码子ω1= 1。位点2a和2b的密码子(按比例1- P.0- p1)在积极选择下进化,与ω2> 1,但背景分支是保守的或中立的[99.].我们使用分支点模型A(模型= 2,NSsites = 2)与相应的零模型计算了沿着棉花枝条的每个位点的正选择的可能性。为了防止codeml陷入局部极大值,在初始dN:dS变化的情况下进行了三次分析[54.,One hundred.].P使用具有一定自由度的Chi-Square分布估计值。贝叶斯经验贝叶斯(BEB)可用于计算每个网站的后验概率[99.].阳性选择下考虑BEB后验概率>0.95的位点。为了验证复制后选择是否代表了选择压力的长期转变或功能分化的进化,我们在棉花分支分支位点水平进行了分支位点模型(模型= 2,NSsites = 2)分析,将复制事件之后的所有分支视为前台,其余分支视为后台。我们还使用Bonferroni校正对多个测试进行了校正。
结构建模
使用Pymol(Pymol分子图形系统,版本1.7,Schrodinger,LLC)和Memsat的DSS算法预测GRCSLD1的二级结构和TMHS。One hundred.,101),分别。
预测三维蛋白质结构的计算方法一般可分为两类,基于模板(比较和线程建模)和无模板建模(从头计算建模),一些复合协议结合了这两方面[102,103].为了获得GRCSLD1的精制三维结构,使用I-Tasser预测预测[104,105], Phyre2 [106]和罗贝塔[107,108].由于Robetta使用Ginzu方法将输入蛋白质序列解析为域,因此使用比较建模的PDB蛋白质与PDB蛋白的域构建模型,以及使用Rosetta AB Initio协议的没有可检测的PDB同性恋,GRCSLD1的模型被分成了两个推定域,分别建模。使用Modeler的掺杂功能进行评估Rosetta的域模型[109],verfity3d [110], ProSA [111].使用RosettacM的杂交态效果重新组合两个域的顶级计量型号。使用Modeler,Verfity3D和ProSa的掺杂功能再次评估全长GRCSLD1的候选模型。我们使用TM-Signal结构对齐程序将彩色评分模型与BCSA的结构匹配[57.].TM-score的值在(0,1],大于0.5表示SCOP/CATH中两个结构共享同一折叠[58.].
缩写
- A&G:
-
AndroeCium&gynoecium.
- BEB:
-
贝叶斯经验贝叶斯
- CESA:
-
纤维素合成酶
- CSL:
-
纤维素合酶样
- CSLD:
-
纤维素synthase-like D
- CSR:
-
特定类地区
- GT2:
-
糖基转移酶家族2
- 如果:
-
接口
- irx:
-
不规则的木耳
- ML:
-
最大可能性
- MSA:
-
多个序列对齐
- MYA:
-
百万年前
- P-CR:
-
植物保守区
- RMSD:
-
根均线偏差
- TMH:
-
跨膜螺旋
- WGD:
-
全基因组复制
参考资料
- 1.
泰勒NG。高等植物中纤维素的生物合成和沉积。新植醇。2008;178(2):239 - 52。doi:10.1111 / J.1469-8137.2008.02385.x.
- 2.
麦克法兰他,DöringA,Persson S.纤维素合成的细胞生物学。Annu Rev植物Biol。2014; 65(1):69-94。doi:10.1146 / annurev - arplant - 050213 - 040240.
- 3.
Somerville C,Bauer S,Brininstool G,Facette M,Hamann T,Milne J等。朝向理解植物细胞壁的系统方法。科学。2004; 306(5705):2206-11。doi:10.1126 / Science.1102765.
- 4。
王志强,王志强,王志强,等。木质素生物合成与结构研究进展。植物杂志。2010;153(3):895 - 905。doi:10.1104 / pp.110.155119.
- 5。
植物纤维素的合成。生物医学进展。2006;22(1):53-78。doi:10.1146 / annurev.cellbio.22.022206.160206.
- 6。
Cosgrove DJ。重新构建我们的纤维素和主细胞壁组件的模型。CurrOp植物BIOL。2014; 22:122-31。doi:10.1016 / J.PBI.2014.11.001.
- 7。
王勇,范超,胡海涛,孙东,王勇,等。转基因植物细胞壁,以提高生物质产量和生物燃料生产的生物能源作物。Biotechnol放置2016;34(5):997 - 1017。doi:10.1016 / j.biotechadv.2016.06.001.
- 8。
Burton RA, Fincher GB。植物细胞壁工程:在生物燃料生产和改善人类健康中的应用。《生物技术》2014;26:79-84。doi:10.1016 / J.Copbio.2013.10.007.
- 9。
Cantarel BL,Coutinho PM,rancurel C,Bernard T,Lombard V,Henrissat B.碳水化合物活性酶数据库(Cazy):糖蛋白酶体的专家资源。核酸RES。2009; 37(4):D233-D38。doi:10.1093 / nar / gkn663.
- 10.
Yin Y,Johns Ma,Cao H,Rupani M.植物和藻类基因组和转录om的调查显示出纤维素合酶超家族的进化和功能的新见解。BMC基因组学。2014; 15(1):1-15。doi:10.1186 / 1471-2164-15-260.
- 11.
尹勇,黄建平,徐勇。植物和藻类纤维素合成酶超家族的全序列分析。BMC Plant Biol. 2009; 9:1-14。doi:10.1186 / 1471-2229-9-99.
- 12.
里士满TA,萨默维尔CR。纤维素酶家族。植物理性。2000; 124(2):495-8。doi:10.1104 / pp.124.2.495.
- 13.
王L,Guo K,Li Y,Tu Y,Hu H,Wang B等人。表达分析和整合分析中国极限运动协会/ CSL超家庭在米饭中。BMC植物BIOL。2010; 10(1):1-16。doi:10.1186 / 1471-2229-10-282.
- 14。
黑曾SP,斯科特-克雷格JS,沃顿JD。水稻纤维素合成酶类基因。植物杂志。2002;128(2):336 - 40。doi:10.1104 / pp.010875.
- 15.
梨JR川越Y,Schreckengost WE,德尔默DP,潜行者DM。高等植物含有编码纤维素合成酶的催化亚基的细菌酶celA基因的同源物。国家科学院院刊美A. 1996; 93(22):12637-42。
- 16。
desprez t,juraniec m,crowell ef,jouy h,pochylova z,parcy f等。组织纤维素合成酶复合物,参与原发性细胞壁合成拟南芥蒂利亚纳.国家科学院院刊美A. 2007; 104(39):15572-7。doi:10.1073 / pnas.0706569104.
- 17。
Persson S, Paredez A, Carroll A, Palsdottir H, Doblin M, Poindexter P, et al.;初级细胞壁纤维素合酶复合物中三种独特成分的遗传证据拟南芥.中国科学(d辑:地球科学)2007;104(39):15566-71。doi:10.1073 / pnas.0706592104.
- 18。
Taylor Ng,Howells RM,Huttly Ak,Vickers K,Turner SR。三种不同CESA蛋白质中的相互作用对纤维素合成必不可少的。Proc Natl Acad Sci U S A. 2003; 100(3):1450-5。doi:10.1073 / PNAS.0337628100.
- 19。
李安,夏涛,徐伟,陈涛,李旭,范军,等。纤维素生产中四种CESA亚型的综合分析gossypium hirsutum和海岛棉.Planta。2013; 237(6):1585-97。doi:10.1007 / S00425-013-1868-2..
- 20。
帕特森啊,温德尔JF,Gundlach H,Guo H,Jenkins J,Jin D等。反复多倍化Gossypium.基因组与可纺棉纤维的进化。大自然。2012;492(7429):423 - 7。doi:10.1038 / nature11798.
- 21。
塔特尔JR,罗G,杜克MV,亚历山大DC,关X,宋之问,等。代谢组学和转录组学分析上市公司棉纤维如何转变到次生壁合成,阻遏木质化,延长的伸长率。BMC基因组学。2015; 16(1):1-28。doi:10.1186 / s12864-015-1708-9.
- 22。
张特,胡y,江w,方l,guan x,chen j等。分子素棉花的测序(陆地棉TM-1)提供改良纤维的资源。Nat生物技术。2015;33(5):531 - 7。doi:10.1038 / NBT.3207.
- 23.
袁东,唐志,王明,高伟,涂磊,金旭,等。海岛棉(海岛棉)提供对各种可纺纱纤维的全多聚纤维化和开发的见解。SCI REP。2015; 5:17662。doi:10.1038 / srep17662.
- 24.
等。瓜尔种子ß-甘露聚糖合酶是纤维素合酶超基因家族的成员之一。科学。2004;303(5656):363 - 6。doi:10.1126 / science.1090908.
- 25.
纤维素合成酶样蛋白的表达CSL.)基因揭示了这一点CSLA.家族成员编码甘露聚糖合酶。中国科学院院刊2005;102(6):2221-6。doi:10.1073 / pnas.0409179102.
- 26.
Cocuron J-C, Lerouxel O, Drakakaki G, Alonso AP, Liepman AH, Keegstra K,等。一个来自纤维素合成酶样C家族的基因编码β-1,4葡聚糖合成酶。中国科学院院刊2007;104(20):855 - 5。doi:10.1073 / pnas.0703133104.
- 27。
Doblin MS, Pettolino FA, Wilson SM, Campbell R, Burton RA, Fincher GB,等。大麦纤维素合成酶CSLH基因介导(1,3;1,4)-β-d-葡聚糖的合成拟南芥.Proc Natl Acad Sci U S A. 2009; 106(14):5996-6001。doi:10.1073 / pnas.09019106.
- 28。
Burton Ra,Wilson Sm,Hrmova M,Harvey Aj,Shirley NJ,Medhurst A等。纤维素合酶样CSLF.基因介导细胞壁的合成(1,3; 1,4)-Beta-D-葡聚糖。科学。2006; 311(5769):1940-2。doi:10.1126 / science.1122975.
- 29。
陈志强,郭峰,陈志强,等。CSLD3在根尖毛细胞顶质膜细胞壁合成中的作用。细胞生物学杂志。2011;13(8):973-80。doi:10.1038 / ncb2294.
- 30。
王伟,王林,陈超,熊刚,谭晓燕,杨克忠,等。拟南芥CSLD1和CSLD4是纤维素沉积和花粉管正常生长所必需的。J Exp Bot, 2011。doi:10.1093 / jxb / err221.
- 31。
Yin L,Verhertbruggen Y,Oikawa A,Manisseri C,Knierim B,Prak L等。CSLD2,CSLD3和CSLD5的合作活动是正常的拟南芥发展。摩尔。2011;4(6):1024 - 37。http://dx.doi.org/10.1093/mp/ssr026
- 32。
Favery B, Ryan E, Foreman J, Linstead P, Boudonck K, Steer M, et al.;侦探科杰克编码根毛细胞形态发生所需的纤维素合成酶样蛋白拟南芥.基因Dev。2001;15(1):79 - 89。doi:10.1101 / gad.1888801.
- 33.
Wang X, Cnops G, Vanderhaeghen R, De Block S, Van Montagu M, Van Lijsebettens M。ATCSLD3.,纤维素合酶样基因对于根发生长很重要拟南芥.植物理性。2001; 126(2):575-86。doi:10.1104 / pp.126.2.575.
- 34.
刘志强,刘志强,刘志强,等。纤维素合成酶样基因的功能分析CSLD1,CSLD2,和CSLD4在尖端生长拟南芥细胞。植物杂志。2008;148(3):1238 - 53年。doi:10.1104 / pp.108.121939.
- 35.
Yoo C-M,Quan L,Blancaflor EB。发散和冗余CSLD2和CSLD3功能期间拟南芥蒂利亚纳根发和雌性配子体发育。前植物SCI。2012;3. DOI:10.3389 / fpls.2012.00111.
- 36.
等。中断的ATCSLD5导致生长率降低,Xylan和同级肌病合酶活性减少,改变了木聚糖的发生拟南芥.工厂J. 2007; 52(5):791-802。doi:10.1111 / j.1365 - 313 x.2007.03281.x.
- 37.
顾峰,Bringmann M, Combs JR, Yang J, Bergmann DC, Nielsen E。拟南芥CSLD5以细胞周期依赖的方式参与细胞板形成。植物细胞。2016;28(7):1722 - 37。doi:10.1105 / tpc.16.00203.
- 38。
Kim Cm,Park Sh,Je Bi,Park Sh,Park SJ,Piao Hl等。OsCSLD1,纤维素合酶样D1基因是水稻中的根毛形态发生所必需的。植物理性。2007; 143(3):1220-30。doi:10.1104 / pp.106.091546.
- 39。
栾W,刘y,张f,宋y,王z,peng y等。OSCD1.编码纤维素合酶样D子家族的推定构件,对水稻植物建筑和生长至关重要。植物Biotechnol J.1111; 9(4):513-24。doi:10.1111 / j.1467-7652.2010.00570.x.
- 40.
吴C,傅y,hu g,si h,cheng s,刘W.窄卷叶片的稻米突变体的分离与表征。Planta。2010; 232(2):313-24。doi:10.1007 / S00425-010-1180-3.
- 41.
胡j,zhu l,zeng d,gao z,guo l,fang y等。鉴定和表征狭窄和滚动的叶子1,一种新型基因调节叶片形态和植物建筑在水稻中。植物mol biol。2010; 73(3):283-92。doi:10.1007 / s11103 - 010 - 9614 - 7.
- 42。
Hunter CT, Kirienko DH, Sylvester AW, Peter GF, McCarty DR, Koch KE。纤维素synthase-like D1是玉米正常的细胞分裂、扩张和叶片发育的组成部分。植物杂志。2012;158(2):708 - 24。doi:10.1104 / pp.111.188466.
- 43。
王k,王z,李f,ye w,王j,歌g等。二倍体棉花草案Gossypium raimondii就.Nat麝猫。2012;44(10):1098 - 103。doi:10.1038 / ng.2371.
- 44。
基因与基因组:棉花作为研究多倍体的模型。在:Soltis SP, Soltis ED,编辑。多倍性和基因组进化。柏林:施普林格柏林海德堡;2012.p . 181 - 207。doi:10.1007 / 978-3-642-31442-1_10.
- 45。
李F,风扇G,Lu C,Xiao G,Zou C,Kohel RJ等。栽培高地棉花的基因组序列(陆地棉TM-1)提供对基因组进化的见解。NAT BIOTECH。2015; 33(5):524-30。doi:10.1038 / NBT.3208.
- 46。
秦永明,朱永祥。棉纤维如何伸长:一个线性细胞生长模式的故事。植物学报。2011;14(1):106-11。doi:10.1016 / j.pbi.2010.09.010.
- 47.
Hernandez-Gomez MC,Runavot J-L,Guo X,Bourot S,Benians Tas,Willats WGT等。异甘油和杂氧基细胞壁多糖在棉纤维细胞发育的伸长和次级细胞壁沉积相期间显示不同动力学。植物细胞生理。2015; 56(9):1786-97。doi:10.1093 / PCP / PCV101.
- 48.
李F,风扇G,王k,Sun F,Yuan Y,Song G等人。栽培棉的基因组序列木本棉.Nat麝猫。2014;46(6):567 - 72。doi:10.1038 / ng.2987.
- 49.
Ronquist F,Teslenko M,Van der Mark P,Ayres DL,Darling A,HöhnaS等人。MRBAYES 3.2:高效的贝叶斯系统发育推理和跨大型模型空间的模型选择。系统中的生物学。2012; 61(3):539-42。doi:10.1093 / sysbio / sys029.
- 50.
Kelley JL,Swanson WJ。人类基因组中的阳性选择:从基因组扫描到生物学意义。Annu Rev Genomics Hum Genet。2008; 9(1):143-60。doi:10.1146 / annurev.genom.9.081307.164411.
- 51。
杨Z,Bielawski JP。检测分子适应的统计方法。趋势ECOL EVOL。2000; 15(12):496-503。doi:10.1016 / s0169 - 5347 (00) 01994 - 7.
- 52。
Bishop JG, Ripoll博士,Bashir S, Damasceno CMB, Seeds JD, Rose JKC。甘氨酸β-1,3-内切葡聚糖酶基因的选择疫霉葡聚糖酶抑制剂蛋白。遗传学。2005; 169(2):1009-19。doi:10.1534 / genetics.103.025098..
- 53。
基因复制的进化:模型的分类和区分。中国科学(d辑:地球科学)2010;doi:10.1038 / nrg2689.
- 54。
Schwerdt JG,Mackenzie K,Wright F,Oehme D,Wagner JM,Harvey AJ等人。纤维素合成酶基因在草丛中的进化动态。植物理性。2015; 168(3):968-83。doi:10.1104 / pp.15.00140.
- 55。
Garvin Mr,Bielawski JP,Gharrett AJ。在活塞的阳性达尔文选择,在太平洋三文治局线粒体的复杂I中为质子泵提供了质子泵。Plos一个。2011; 6(9):E24127。doi:10.1371 / journal.pone.0024127.
- 56。
Chen K,Durand D,Farach-Colton M. Notung ::约会基因重复和优化基因家族树的计划。j计算biol 2000;7(3-4):429-447。doi:10.1089 / 106652700750050871.
- 57.
纤维素合成与膜转运的晶体学研究。大自然。2013;493(7431):181 - 6。doi:10.1038 / nature11744.
- 58.
基于TM-score的蛋白质结构比对算法。核酸学报2005;33(7):2302-9。doi:10.1093 / nar / gki524.
- 59.
Slabaugh E,Davis JK,Haigler Ch,Yingling Yg,Zimmer J.纤维素合成酶:晶体学和建模的新见解。趋势植物SCI。2014; 19(2):99-106。doi:10.1016 / j.tplants.2013.09.009.
- 60.
Lairson LL,Henrissat B,戴维斯GJ,威瑟斯SG。糖基转移酶:结构,功能和机制。annu rev biochem。2008; 77(1):521-55。doi:10.1146 / annurev.biochem.76.061005.092322.
- 61.
SethaPong L,Haigler Ch,Kubicki JD,Zimmer J,Bonetta D,Debolt S等人。植物纤维素合成酶的三级模型。Proc Natl Acad Sci U S A. 2013; 110(18):7512-7。doi:10.1073 / pnas.1301027110.
- 62.
黄建中,黄建中,黄建中,黄建中,黄建中,黄建中。UCSF chimera—一个用于探索性研究和分析的可视化系统。化学学报。2004;25(13):1605-12。doi:10.1002 / jcc.20084.
- 63.
Soderlund C,Bomhoff M,Nelson WM。Symap V3.4:具有施用于植物基因组的交钥匙同轴系统。核酸RES。2011; 39(10):E68。doi:10.1093 / nar / gkr123.
- 64.
唐华,王旭,Bowers JE, Ming R, Alam M, Paterson AH。通过多重排列的被子植物基因图谱揭示古代的六倍性。基因组研究》2008;18(12):1944 - 54。doi:10.1101 / gr.080978.108.
- 65.
ogden th,罗森伯格ms。多序列对准精度和系统发育推理。系统中的生物学。2006; 55(2):314-28。doi:10.1080 / 10635150500541730..
- 66。
Castresana J.从多次对准中选择保守块,用于其在系统发育分析中的使用。mol Biol Evol。2000; 17(4):540-52。doi:10.1093 / oxfordjournals.molbev.a026334.
- 67。
基于DNA序列的进化树:最大似然方法。中国科学(d辑:地球科学)1981;17(6):368-76。doi:10.1007 / bf01734359.
- 68。
Huelsenbeck JP,Ronquist F,Nielsen R,Bollback JP。Phylogyy的贝叶斯推断及其对进化生物学的影响。科学。2001; 294(5550):2310-4。doi:10.1126 / science.1065889.
- 69。
MADDISON W.重建多种子薄膜的角色演变。克莱斯。1989; 5(4):365-77。doi:10.1111 / J.1096-0031.1989.TB00569.X..
- 70.
郭k,zou w,feng y,zhang m,zhang j,tu f等。水稻中细胞壁生物学的综合基因组和代谢组织框架。BMC基因组学。2014; 15(1):596。doi:10.1186 / 1471-2164-15-596.
- 71.
李安,王锐,李旭,刘敏,樊军,郭轲,等。纤维素酶辅助提取膜蛋白的蛋白质组学分析用于棉纤维中纤维素合酶复合物及其潜在相关成分的功能鉴定。Sci众议员2016;6:26356。doi:10.1038 / srep26356.
- 72.
张建军,张志强,张志强,等。gap BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸学报1997;25(17):3389-402。doi:10.1093 / nar / 25.17.3389.
- 73.
Goodstein DM,Shu S,Howson R,Neupane R,Hayes Rd,Fazo J等人。植物血统:绿色植物基因组学的比较平台。核酸RES。2012; 40(D1):D1178-D86。doi:10.1093 / NAR / GKR944.
- 74.
Leatunic I,Deerks T,Bork P. Smart:2015年最近的更新,新的发展和地位。核酸Res。2015; 43(D1):D257-D60。doi:10.1093 / nar / gku949.
- 75。
Mitchell A,Chang H-Y,Daugherty L,Fraser M,Hunter S,Lopez R等人。TheRofto蛋白质家庭数据库:15年后的分类资源。核酸RES。2015; 43(D1):D213-D21。doi:10.1093 / nar / gku1243.
- 76。
Marchler-Bauer A, Derbyshire MK, Gonzales NR, Lu S, Chitsaz F, Geer LY等。CDD: NCBI的保守域数据库。核酸学报2015;43(D1): D222-D26。doi:10.1093 / nar / gku1221.
- 77。
拜耳男,Milne的I,斯蒂芬G,肖P,Cardle L,赖特F等。用点心遗传和物理图谱的比较可视化。生物信息学。2011; 27(9):1307-8。doi:10.1093 /生物信息学/ btr111.
- 78。
Lassmann T,Frings O,Sonnhammer ell。Kalign2:高性能多次对准蛋白质和核苷酸序列,允许外部特征。核酸RES。2009; 37(3):858-65。doi:10.1093 / NAR / gkn1006.
- 79。
kaoh K, Standley DM. MAFFT多序列比对软件版本7:性能和可用性的改进。生物医学进展。2013;30(4):772-80。doi:10.1093 / molbev / mst010.
- 80.
埃德加钢筋混凝土。MUSCLE:多序列比对,高精度,高通量。核酸学报2004;32(5):1792-7。doi:10.1093 / NAR / gkh340.
- 81.
Talavera G,Castresana J.从蛋白质序列比对去除发散和模棱两可的嵌段后改善文学发生。系统中的生物学。2007; 56(4):564-77。doi:10.1080 / 10635150701472164.
- 82.
惠勒WC,GatesyĴ,DeSalle R.省音:一个用于容纳多分子序列比对具有对准歧义位点的方法。摩尔Phylogen EVOL。1995; 4(1):1-9。doi:10.1006 / MPEV.1995.1001.
- 83.
Guindon S,Dufayard J-F,Lefort V,Anisimova M,Hordijk W,Gascuel O.新算法和估计最大似然性的方法:评估PHYML 3.0的性能。系统中的生物学。2010; 59(3):307-21。doi:10.1093 / SYSBIO / SYQ010.
- 84.
Darriba D,Taboada GL,Doallo R,Posada D. Prottest 3:快速选择蛋白质进化最佳型号.BioInformatics。2011; 27(8):1164-5。doi:10.1093 /生物信息学/ btr088.
- 85.
Anisimova M, Gil M, Dufayard J-F, Dessimoz C, Gascuel O.分支支持方法的研究证明了快速似然近似方案的准确性、有效性和鲁棒性。系统医学杂志。2011;60(5):685 - 99。doi:10.1093 / sysbio / syr041.
- 86.
索里亚卡斯科诉塔拉维拉G,Igea J,Castresana J.K树评分:系统发育树的相对分支长度和拓扑的定量。生物信息学。2007; 23(21):2954-6。doi:10.1093 / Bioinformatics / BTM466.
- 87.
Kim D, Pertea G, Trapnell C, Pimentel H, Kelley R, Salzberg SL. TopHat2:在插入、缺失和基因融合中转录组的精确比对。基因组医学杂志。2013;14 (4):R36。doi:10.1186 / GB-2013-14-4-R36.
- 88.
李H,手机B,Wysoker A,Fennell T,Ruan J,Homer N等人。序列对齐/地图格式和samtools。生物信息学。2009年; 25(16):2078-9。doi:10.1093 /生物信息学/ btp352.
- 89.
Anders S,Pyl Pt,Huber W. htseq - 使用高吞吐量排序数据的Python框架。生物信息学。2015; 31(2):166-9。doi:10.1093 / Bioinformatics / BTU638.
- 90.
Anders S,Huber W.序列计数数据的差异表达分析。基因组Biol。2010; 11(10):R106。doi:10.1186 / GB-2010-11-10-R106.
- 91.
Anders S, McCarthy DJ, Chen Y, Okoniewski M, Smyth GK, Huber W,等。利用R和bioconductor对RNA测序数据进行基于计数的差异表达分析。Nat协议。2013;8(9):1765 - 86。doi:10.1038 / nprot.2013.099.
- 92.
涡流SR。加速的Pofile HMM搜索。PLOS COMP BIOL。2011; 7(10):E1002195。doi:10.1371 / journal.pcbi.1002195.
- 93.
李志强,李志强,李志强,等。的拟南芥信息资源(TAIR):改进的基因注释和新工具。核酸RES。2012; 40(D1):D1202-D10。doi:10.1093 / NAR / GKR1090.
- 94.
富W,沉Y,郝Ĵ,吴Ĵ,柯L,吴C,等人。酰基辅酶A的N-影响酰基转移酶通过调节脂质代谢和茉莉酸的生物合成在棉花生育力。SCI代表2015年; 5:11790。doi:10.1038 / srep11790.
- 95.
李伟,赵发,方伟,谢东,侯军,杨旭,等。陆地棉幼苗根系早期盐胁迫响应蛋白的鉴定(gossypium hirsutum l。)采用基于ITRAQ的蛋白质组学技术。前植物SCI。2015; 6(732)。doi:10.3389 / FPLS.2015.00732.
- 96.
Livak KJ,Schmittgen TD。使用实时定量PCR和2分析相对基因表达数据−ΔΔCT方法。方法。2001;25(4):402 - 8。doi:10.1006 / meth.2001.1262.
- 97.
Schabauer H,Valle M,Piher C,Stockinger H,Stamatakis A,Robinson-Rechavi M,等。SLIMCODEML:分支站点模型的Rodeml的优化版本。在:平行和分布式处理研讨会厂房和博士论坛(IPDPSW),2012年IEEE 26国际。2012; 706-14。doi:10.1109 / IPDPSW.2012.88.
- 98.
杨Z.PAML 4:通过最大可能性进行系统发育分析。mol Biol Evol。2007; 24(8):1586-91。doi:10.1093 / molbev / msm088.
- 99。
张j,尼尔森r,杨z.评估改进的分支部位似然方法,用于检测分子水平的阳性选择。mol Biol Evol。2005; 22(12):2472-9。doi:10.1093 / molbev / msi237.
- One hundred.
Fletcher W,杨Z.插入,缺失和对准误差对正选择的分支部位测试的影响。mol Biol Evol。2010; 27(10):2257-67。doi:10.1093 / molbev / msq115.
- 101.
努特,琼斯·琼斯。使用支持向量机的跨膜蛋白拓扑预测。BMC生物信息学。2009; 10(1):1-11。doi:10.1186 / 1471-2105-10-159.
- 102.
琼斯dt。使用进化信息提高跨膜蛋白拓扑预测的准确性。生物信息学。2007; 23(5):538-44。doi:10.1093 /生物信息学/ btl677.
- 103.
Källbergm,王H,王S,彭俊,王Z,Lu H等。基于模板的蛋白质结构建模使用Raptorx Web服务器。NAT PROTOC。2012; 7(8):1511-22。doi:10.1038 / nprot.2012.085.
- 104.
张玉华:一种蛋白质结构和功能预测的统一平台。Nat Protoc。2010;5(4):725 - 38。doi:10.1038 / nprot.2010.5.
- 105.
Zhang Y. i-Tasser服务器用于蛋白质3D结构预测。BMC生物信息学。2008; 9(1):1-8。doi:10.1186 / 1471-2105-9-40.
- 106.
Kelley LA, Mezulis S, Yates CM, Wass MN, Sternberg MJE。用于蛋白质建模、预测和分析的Phyre2网站门户。Nat Protoc。2015;10(6):845 - 58。doi:10.1038 / nprot.2015.053.
- 107.
宋y,dimaio f,王雷y-r,金d,miles c,brunell tj,等。高分辨率比较玫瑰花苜蓿。结构。2013; 21(10):1735-42。doi:10.1016 / j.str.2013.08.005.
- 108.
拉曼,弗农,汤普森,泰卡,萨德雷耶夫,裴俊,等。CASP8的结构预测与全原子细化使用Rosetta。蛋白质。2009;77 (S9): 89 - 99。doi:10.1002 / prot.22540.
- 109.
Šalia,blundell tl。空间束缚满足的比较蛋白质模型。J Mol Biol。1993; 234(3):779-815。doi:10.1006 / jmbi.1993.1626.
- 110.
关键词:蛋白质模型,三维模型,蛋白质模型大自然。1992;356(6364):83 - 5。doi:10.1038 / 356083 a0.
- 111.
Wiederstein M, Sippl MJ。ProSA-web:用于蛋白质三维结构错误识别的交互式web服务。核酸学报2007;35(增刊2):W407-W10。doi:10.1093 / nar / gkm290.
- 112.
刘W,谢y,ma j,luo x,nie p,zuo z等。IBS:用于呈现和可视化生物序列的插画者。生物信息学。2015; 31(20):3359-61。doi:10.1093 /生物信息学/ btv362.
致谢
作者感谢河南省农业科学院经济作物研究所李武博士和傅文峰女士在qRT-PCR分析方面的帮助。
资金
河南省科技计划项目(No. 152102110130)。关键词:边坡,边坡稳定性,边坡稳定性
数据和材料的可用性
支持本文结果的数据集包含在本文及其附加文件中。文章中描述的材料可以免费提供给任何希望将其用于非商业目的的科学家。
作者信息
隶属关系
贡献
YPL和TGY构思和设计了这项研究。YPL进行了生物信息学实验并写了稿件。DDD和YH收集了序列数据并进行了QRT-PCR分析。XYG和HXG收集组织并进行了RNA提取。所有作者均阅读并批准最终手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
附加文件1:表S1。
其他植物中CSLD蛋白的染色体轨迹ID和长度。(XLSX 16 KB)
附加文件2:
所有植物中CSLD蛋白的序列。(FASTA 134 KB)
附加文件3:表S2。
使用ProtTest选择模型。(多克斯23 kb)
附加文件4:表S3。
比较ML和贝叶斯树基于三种对齐(Kalign, Mafft和Muscle)使用Ktreedist。(多克斯33 kb)
附加文件5:图S1。
基于三种排列和省略策略,利用ML和贝叶斯方法重建了棉花CSLD树的不同拓扑结构。支持值显示为A. Thaliana.-cotton和cotton CSLD节点使用不同颜色的圆圈作为bootstrap比例/类似sh的aLRT分数/贝叶斯后验概率。棉花CSLD蛋白分支以不同的颜色表示。“Other CSLD”表示来自其他植物的CSLD蛋白。(TIFF 2007 kb)
附加文件6:表S10、11和12。
的中国极限运动协会/ CSL棉花HTseq-count基因计数数据。(XLSX 31 kb)
附加文件7:表S13。
底漆的底漆GhCSLD用于qRT-PCR分析的基因(XLSX 9 kb)
附加文件8:图S3。
跨膜螺旋(TMHS)和GRCSLD1中的细胞溶质环图。细胞骨环和TMHS(1-8)内的标记显示了阳性选择(红色)下的四个保守基序(黑色),P-CR(紫色),CSR(蓝色)和氨基酸残基的近似位置。(TIFF 6688 KB)
附加文件9:图S4。
GRCSLD1,GHESA1,BCSA和ATCSLD1的多个序列对齐。使用Pymol的DSS算法计算GRCSLD1的二次结构。紫圆柱,黄色箭头和黑线表示GRCSLD1的α-螺旋,β股和线圈;红色矩形和黄色矩形表示Ghcesa1的α-螺旋和β-股,红线和黄线表示BCSA的α-螺旋和β-纤维。植物保守区(P-CR)和特定类别区域(CSR)用蓝色和绿线突出显示。大红色字母表示GRCSLD1中的集体阳性选择的位点。(TIFF 4834 KB)
附加文件10:表S15。
全长GRCSLD1蛋白的模型验证分数。(DOCX 22 KB)
附加文件11:表S4,5和6。
转录组数据的来源G. Hirsutum,G. Arboreum.和g . raimondii就.(XLSX 12 KB)
附加文件12:表S7、8和9。
的详细概况中国极限运动协会/ CSL基因超级植物具有正常化和棉花中DESEQ稳定转化的规范化。(XLSX 49 KB)
附加文件13:FIGRUE S2。
棉花中CESA / CSL蛋白的系统发育分析A. Thaliana..系统发育树采用最大似然法。关键节点的支持值显示为引导比例。(TIFF 2921 kb)
附加文件14:表S14。
的相对表达水平CSLD基因G. Hirsutum通过比较2——ΔΔCT方法使用中存在。(XLSX 10 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
李,Y.,Yang,T.,Dai,D。et al。棉花CSLD蛋白的进化、基因表达谱分析及三维建模BMC植物BIOL.17,119(2017)。https://doi.org/10.1186/s12870-017-1063-x
收到:
接受:
发表:
关键字
- 棉花
- CSLD
- 种系发生树
- 积极的选择
- CSL超级家族
- 结构建模
- 纤维素合成酶
- 细胞壁