摘要
背景
RNA定向的DNA甲基化(RDDM)是一种植物特异性的表观遗传方法,其依赖于用于产生24个核苷酸小干扰RNA(siRNA)的RNA聚合酶IV(POL IV),所述胞嘧啶甲基化和沉默基因和转座子的沉默。玉蜀黍RPD1 / RMR6基因编码Pol IV的最大亚基,是正常植物发育,载气,某些转移元素(TES)的转录抑制和特异性等位基因的转录调节所必需的。
结果
在本研究中,我们采用全RNA-Seq方法比较B73和RPd1时/ rmr6叶子转录组。虽然之前的研究表明,在RdDM突变体中,sirna的产生缺失会引起CHH DNA甲基化的强烈缺失,但在拟南芥和玉米中,并没有大量的基因或TEs转录激活RPd1时/ rmr6转录表明,波尔IV活性的丧失导致在玉米基因组的转录的部分全球增长。我们的研究结果指向基因与附近TE插入,是最强烈影响波尔IV介导的基因沉默。附近的基因表达的转座因子的调制被链接到替代甲基化谱上基因的侧翼区,以及这些配置文件是严格依赖于插入TE构件的具体特征。虽然波尔IV是必不可少的siRNA的生物发生,具有相关联的siRNA位点的基因是较少受波尔四世突变。
结论
这项对基因表达、TEs分布、smallRNA靶向和DNA甲基化水平的深入和综合分析表明,Pol IV活性的丧失在全球范围内影响基因组调控,指出TEs是附近基因表达的调节剂,并表明存在多个水平的表观遗传沉默机制。我们的研究结果还表明,Pol iv介导的RdDM途径在玉米基因组显性调控和亚基因组稳定性和进化中发挥着重要作用。
背景
基因表达和转座元件(TE)活性的调控是植物正常发育和基因组完整性保护的基础。在植物中,24个核苷酸小干扰rna (24nt sirna)在TEs、重复序列、转基因和内源性基因的转录基因沉默(TGS)中发挥关键作用,引导rna导向的DNA甲基化途径(RdDM;[1,2,3.,4])。这些24 nt sirna整合到ARGONAUTE (AGO)蛋白(主要是AGO4,但也有AGO6和AGO9 [5,6]),引导相应DNA序列的胞嘧啶甲基化和组蛋白修饰,导致染色质状态向转录折射。
在规范的RdDM路径中[7] 2个植物特异性核RNA聚合酶,聚合酶IV和极化电压[8,9,10.,11.],以及依赖RNA的RNA聚合酶2 (RDR2 [12.)是合成小而长的非编码rna所必需的,这些rna会在同源序列上触发rna依赖的DNA从头甲基化。
Pol IV引发非编码RNA的转录,然后通过RDR2转化为DSRNA:两种酶对于24 nt siRNA的生物发生至关重要,并且它们在拟南芥和玉米中物理上助理[13.,14,15].Pol IV转录本很短(大多为26-45 nt,其大小分布的峰值出现在约30 bp),且表达水平较低,这一特征阻止了它们在经典RNA-Seq实验中的识别[16,17].
大多数位点的siRNA生物发生并不直接需要Pol V [18],但产生长的非编码rna,作为与ago结合的sirna碱基配对的支架[19,20.,允许重新甲基转移酶DRM2 (DOMAINS rearrange METHYLASE 2)和染色质修饰成分募集到靶向DNA甲基化和TGS的位点。在CG、CHG和CHH序列中,RdDM引导胞嘧啶甲基化,但CHH甲基化是RdDM的最佳指标,因为CG和CHG甲基化可以通过其他DNA甲基化途径维持(综述于[1])。
RdDM以重复序列(如te)为靶点,并影响植物发育、防御和等位基因串扰的许多过程。拟南芥中约4000-8000个位点和玉米中约244000个位点产生24个nt sirna簇[18,21,22,23],它们在两种物种的RdDM突变体中的积累显著减少[22,23,24,25].缺乏Pol IV功能的突变植物在sirna生产中存在缺陷[18,22,23,26]在表型水平下呈现脂肪缺陷[8,9,27].拟南芥核RNA聚合酶d1a(NRPD1A)功能丧失的突变体是晚开花的[11.],而玉米rna聚合酶d1(rpd1)与非突变体同胞相比,突变体具有多种发育缺陷和植物质量跨代退化[27,28].单一多样性对玉米和拟南芥siRNA位点数量的影响rpd1 / NRPD1A在两个物种的表型的突变可能与他们的不同的基因组TE的内容(在玉米它们占相比,在拟南芥〜10%基因组的〜85%29,30.,31])。
最近的研究报告称,功能突变体RdDM中sirna产生的缺失会导致RdDM位点CHH甲基化的强烈减少。然而,在所谓的mCHH岛甲基化的损失并不会导致大量的TEs释放,即使在TEs富集的基因组中,如玉米[25,32,33,预测Pol iv依赖的胞嘧啶甲基化并不是维持TE沉默所必需的。此外,在全基因组水平上,CHH甲基化倾向于在玉米基因转录开始的上游发现,并且只与某些类型的转座子相关,而CG和CHG甲基化普遍存在于所有玉米转座子中,但在基因开始和停止附近相对较低[34].
作为移动元素,可以在基因组周围移动并通过植物基因组中的周期性转脉爆发,TES长时间被认为是寄生垃圾DNA和最佳“砂浆”元素的基因组结构变化。但是,当Barbara McClintock发现时,[35, TEs最初被描述为“控制元素”,这是基于它们影响附近基因表达的能力。它们可以通过中断基因编码序列或改变其调控元件来影响基因功能,已经报道了几个TE影响附近基因表达的具体例子[36,37,38].经进一步鉴定,te可参与植物对应激环境的反应和适应[39].
在以前的著作中[23,40我们分析了B73野生型和B73野生型玉米的胁迫响应转录组调控和小RNA群体rpd1Pol IV突变体(也称为需要保持压抑-rmr6[27])。首次重新注释了玉米转录组,包括非聚腺苷酸转录本,并鉴定了其非编码部分,揭示了数百个基因和lncrna在长期胁迫作用下的差异表达。有趣的是,B73野生型和B73野生型之间的胁迫调节基因的幅值有很大的不同RPd1时/ rmr6突变植物,由于表面调节剂突变本身引起的基因组调控的压力效应,这似乎激活了许多与压力相关的基因,甚至在控制生长条件下[40].24个nt siRNA被在显着地减少RPd1时/ rmr6突变植物但突变基因上调没有显著具有侧翼的siRNA [损失相关23].同样,最近的一些研究报道转录组、核和甲基化分析玉米RdDM突变体,强调,尽管小干扰rna基因座24元的大幅下降以及随后失去mCHH岛观察,发现差异表达的基因数量相对较低,大多数都是上调(25,32,33,41,42,43].这些工作清楚地证明了RdDM活性对于在沉默的近基因插入转座子和基因本身之间创建染色质边界是至关重要的,但也支持了RdDM损伤引起的siRNA和甲基化谱的整体改变对转座子和基因转录的影响较小的论点[1,44].
在本研究中,我们从玉米的第一个全RNA-Seq分析开始,深入研究了玉米的全RNA-SeqRPd1时/ rmr6突变体转录识别和特征RdDM目标。完整基因组的基因组中并表达谱比较的转录部分的全局分析显示在玉米基因组的转录的总体增加RPd1时/ rmr6突变体与野生型相比。我们还提供了有关错误调控基因的进一步细节,并对Pol iv介导的玉米转录调控的新见解。通过广泛的基因表达和综合分析,测试工程师分布,smallRNA瞄准和DNA甲基化水平,我们的研究结果指出,基因与附近TE插入作为最强烈影响波尔IV-mediated基因沉默,但也表明,尽管波尔IV的生源论siRNAs至关重要,具有相关siRNA位点的基因受Pol IV功能丧失的影响较小。此外,我们的研究结果表明,Pol iv介导的RdDM途径参与了基因组显性调控和亚基因组稳定性和进化。
结果
玉米基因组转录程度的定量分析
分析的RNA-Seq数据集由16个链特异性和16个非定向、总转录组、Illumina单端文库组成,文库取自B73野生型最年轻的包裹叶RPd1时/ rmr6-1突变体植物。玉米植株在正常(C)、干旱胁迫(D)、盐胁迫(S)或干旱+盐胁迫(D + S)条件下生长,处理10天后或处理后恢复7天后采集样品,如[40].经过质量修剪和rRNA污染过滤后,高质量的读取(每个文库从2300万到5400万读取;额外的文件1)被定位到玉米B73参考基因组(RefGen ZmB73 Assembly AGPv3)。多映射的reads(主要由于副同源基因和重复序列,在两个或两个以上基因组位置映射同样好的reads)约占我们reads的50-60%(附加文件)1).由于这些多映射reads包含重要的生物学信息,但同时也代表了RNA-Seq分析的一个重要挑战,我们决定丢弃10多个不同位置的映射,将多映射reads减少到约30-40%的映射reads(附加文件1).为了克服不同的文库尺寸,最初组合了来自每个基因型的16个过滤的BAM文件,获取438,275,238和458,429,084读取B73和RPd1时/ rmr6,分别对应714,514,067和775,422,668个基因组序列。然后通过向下抽样进行归一化,随机选择7亿对B73和rpd1/的比对rmr6(分别来自430 M和410 M reads)来评估两个基因型中基因组的转录部分。
在至少两个RNA-Seq reads的阈值下,我们观察到reads映射到10.94%和10.32%的基因组rmr6分别和B73。在玉米基因组的20亿个碱基对中,这些百分比导致了大约1300万个碱基对的额外转录RPd1时/ rmr6而B73 (226,194,451 bp)表达于RPd1时/ rmr6VS在B73,指示的转录的部分为6%的净增加值213448475碱基;额外的文件2).相似的比例是单独考虑每个基因型的2.9亿个独特的图谱reads(明确分配到参考文献中的一个位置),8.90%的基因组(184,118,131 bp)被至少两个reads in覆盖RPd1时/ rmr6而在B73中为8.45% (174,6354,415 bp)2 b).
To exclude any bias introduced by down-sampling of the entirety set of reads (i.e. selection of different ratios of reads derived from stressed and control conditions in the two genotypes), the transcribed fraction of the genome was calculated also for each individual sample (after down-sampling at level of individual RNA-Seq libraries). The lower number of reads analyzed resulted in the missing of many low-expressed regions: for example, in control condition at T0, 140,831,603 bp and 141,845,343 bp resulted covered by at least 2 reads in B73 andrpd1/rmr6与之前从池读中观察到的2.13 - 2.26亿相比。尽管覆盖率较低,但独立分析(考虑到唯一mapping或多重mapping reads)证实了转录部分的增加rpd1/rmr6与B73相比,在所有分析的样品中,无论应力处理或时间点(附加文件3.).按照这种方法,增加的部分基因组转录rpd1/rmr6平均由200万个碱基对组成。当单个下采样库被池时(从B73和rpd1/rmr6从每个生长条件)在基因组中的转录的增加由约1280万个碱基对(考虑多映射读取)或900万个碱基对(只考虑唯一映射读取;其他文件3.).
总之,这些结果表明,在波尔四世突变体并不局限于重复序列(RdDM的主要目标),而是对独特的基因组区域产生强烈的兴趣,而且,更多的是由少数reads覆盖的区域所代表,它很可能是由许多位点上的微小增加而不是少数基因的强烈激活引起的。为了确定这种转录获得的基因(以及可能的共同特征,如果有的话,使它们成为Pol IV靶标),我们研究了全部玉米基因的表达谱,并将它们进行了比较RPd1时/ rmr6和野生型。
玉米基因的全球表达变异rmr6突变体叶
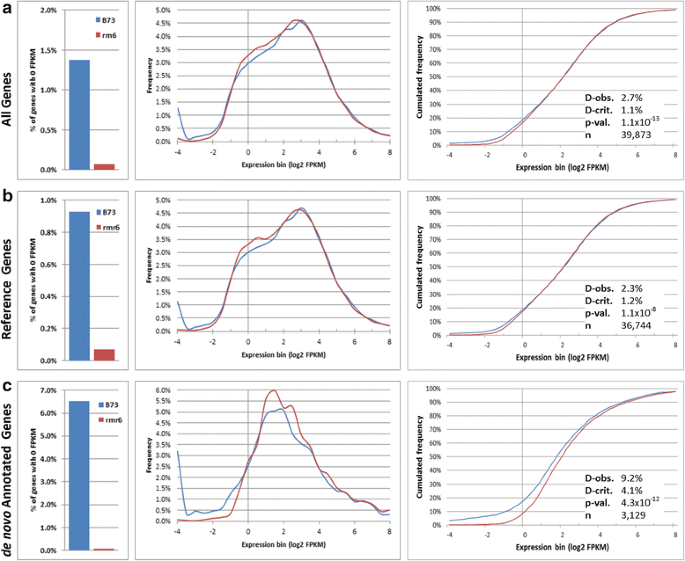
所有参考注释基因(AGPv3.20)和新注释基因的表达值(见方法和[40]用于对基因组引导的转录组件的详细描述和新玉米表达的基因座的注释)进行了计算,并在铆钉和袖口之间的样品之间标准化[45在基因和转录水平。同一基因型同时考虑不同的生长条件和时间点。筛选掉两种基因型中不表达或表达过低的基因和转录本(见方法;附加文件4和5),并比较了B73和B73之间表达基因的表达分布RPd1时/ rmr6如[46].在筛选的40,457个基因中,B73中单基因型(0 FPKM)无表达基因的频率显著高于单基因型rmr6(P.= 0,通过Fisher精确检验;无花果。1)和基因表达分布(仅包括FPKM > 0基因)在两种基因型之间有统计学差异(P.= 1.1 × 10−13,采用Kolmogorov-Smirnov检验;无花果。1).当RPd1时/ rmr6突变似乎导致大量基因的基础表达的整体增加,频率值较高,从0.5到4 FPKM(表达箱从图中的−1到2)。1),而B73中表达值低于0.5 FPKM的频率较高。将参考注释基因与新鉴定的基因分离,我们观察到FPKM为0的基因的频率明显较低(P = 0, Fisher’s exact test)RPd1时/ rmr6两组的突变体,在新识别基因水平上有更显著的差异(图。1 c).Kolmogorov-Smirnov(K-S)对基因表达分布的测试也揭示了B73与B73之间的显着差异RPd1时/ rmr6供两者参考(P.= 1.1 × 10-7)和de novo注释(P.= 4.3 × 10−12)基因。
基因表达的分布在B73和RPd1时/ rmr6突变体。报道了所有表达基因的FPKM为0的基因频率(左),>0 FPKM基因的表达分布直方图(中)和累积频率(右)(A.),并进一步细分为参考注释(B.)和全新注释位点(C.).在这三种比较中,一个基因型中无表达基因的频率(0 FPKM)在B73中显著高于在RPd1时/ rmr6(P.= 0,通过Fisher的确切试验)和基因表达的分布(包括仅具有FPKM> 0的基因)在两个基因型之间存在统计学意义(P.Kolmogorov-Smirnov检验< 0.01)。x轴为基因表达量的log2转换后的FPKM值(0对应FPKM = 1,2, 2对应FPKM = 4,4对应FPKM = 16,以此类推)。D-obs。为D-crit时,两条曲线间观察到的最大垂直距离。为试验的临界距离值。当两个d-OBS> d-STAT和P.< 0.01表示表达式分布在统计学上不同
这些比较表明,在B73中无表达或低表达(FPKM <1)的基因频率高于在RPd1时/ rmr6,而该突变体在较高表达值下的表达频率较高,证实了Pol IV活性丧失导致基因表达量上升。这种转录的增加,并不完全是由重新注释的位点引起或限制的,似乎涉及大部分玉米基因。
Pol IV控制非编码te相关转录本的基础表达
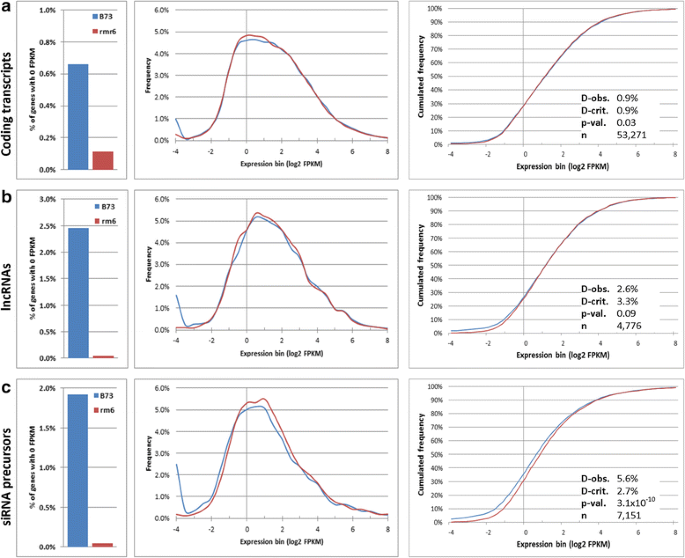
来确定直接导致RPd1时/ rmr6总体转录增加,我们深入分析了特定基因亚群(根据鉴别特征细分)的表达谱RPd1时/ rmr6相比于B73野生型。我们最初集中在编码潜力显着特点,调查编码,非编码和TE相关的转录物的表达谱。整套玉米转录物以前归类为蛋白质编码和非编码转录本(即比200bp的更长,并且不编码蛋白质,或如果一个开放读码框是本,这应该是短于120个氨基酸和预测的蛋白质不能匹配公共数据库的任何蛋白质)。该组非编码转录物的进一步细分成非编码smallRNA前体(如果它们具有针对玉米smallRNAs的已知序列的匹配)和长的非编码RNA [40].66153的表达过滤了转录本(见方法;附加文件4和5)因此细分为蛋白质编码(53,654转录本)、长链非编码rna (lncRNAs, 4898转录本)和非编码小rna前体(sRNA前体,7292转录本)。在这三种类型中,B73中无表达转录本的频率(0 FPKM)显著高于对照组rmr6(P.= 0,通过Fisher精确检验;无花果。2ncRNA组中B73零表达转录本的比例(2.45-1.92%)高于编码组(0.66%),而在ncRNA组中B73零表达转录本的比例(2.45-1.92%)高于编码组(0.66%)RPd1时/ rmr6该趋势与趋势相反(编码转录物的0.11%与NC类的0.04%)。令人惊讶的是,转录表达的分布谱在两个基因型之间显着分歧,其仅为全球更为表达的非编码SRNA前体RPd1时/ rmr6突变体比B73 (P.= 3.1 × 10−10,用K-S试验;无花果。2摄氏度).
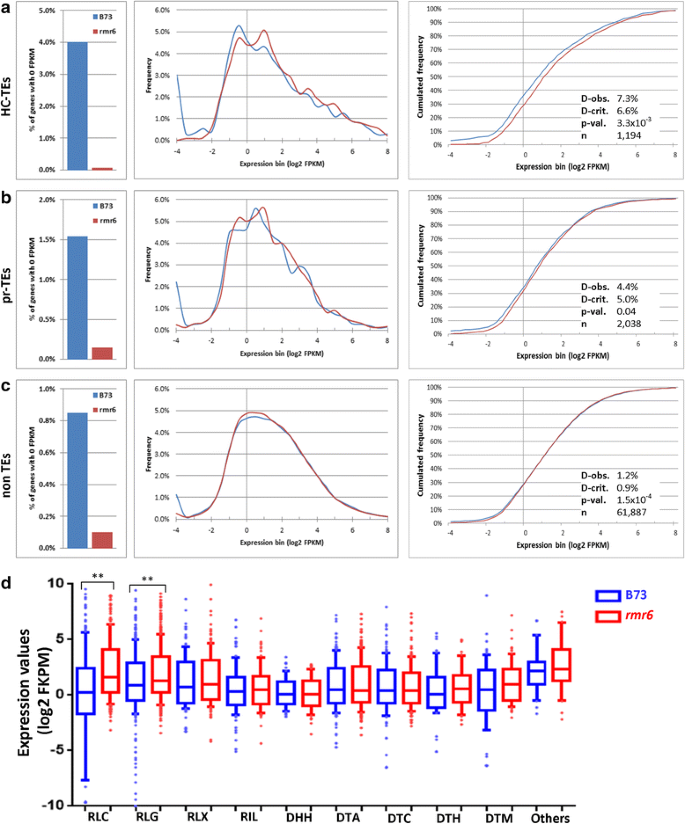
然后,我们通过Blast-search方法对玉米TE数据库中鉴定的TE相关转录本的表达行为进行了分析,并将它们归类为高置信TE (HC-TEs;9766个转录本,其中1245个通过了表达过滤器)或推定/遗迹- tes (pr-TEs;9013转录本,2073表达;见的方法)。e相关转录本表达0 FPKM的比例在B73中高于在rmr6(P.= 0,通过Fisher精确检验;无花果。3得了),沉默分数与TE相似度直接相关(HC-TEs为4.02%,pr-TEs为1.54%,no-TEs为0.85%)。同样地,它们的总体表达分布在HC-TEs的两种基因型之间有显著但不显著的差异(P.= 3.3×10-3由钴测试;无花果。3.).在HC-TE转录本中,Copia (RLC)和Gypsy (RLG) LTR元件对应的转录本均显著高表达RPd1时/ rmr6与B73相比,而与其他TE超家族(I类反转录元件的RLX和RIL, II类DNA TEs的DHH、DTA、DTC、DTH和DTM)匹配的转录本表达谱显示了两种基因型之间的相似表达谱(图)。3 d额外的文件6).这一证据似乎指向TE相关转录本是pol - iv介导的基因沉默的重要靶点,也证实了RdDM突变体中特定TE亚家族的不同转录调控[41,43,47].
Te相关但非TE转录表达的分布在B73和B73之间不同RPd1时/ rmr6突变体。对于之前分类的高可信度- tes (HC-TEs;A.),假定的/ relics-TEs (pr-TEs;B.)及非te成绩单(C.).对于所有三组,在一个基因型中无表达转录本的频率(0 FPKM)在B73中显著高于在RPd1时/ rmr6(P = 0,通过Fisher的确切测试)。转录物表达的分布在HC-TES和非TE转录物的两个基因型之间具有统计学意义(P <0.01通过K-S试验)。超级家庭细分的HC-TE成绩单的表达值图(D.)表示中较高的表达式RPd1时/ rmr6与B73相比,RLC-Copia和RLG-Gypsy相关转录本(**:p < 0.01;Mann-Whitney U),而其余的超级家族在两种基因型中表现出相似的表达水平。这些框表示第一个四分位数(底部范围)和第三个四分位数(上一行),并由一个与数据集的中位数相对应的横条相交。胡须代表的数据是在第10 - 90个百分点和单点描述的异常值
位于基因边界的te负责突变体的转录促进
虽然te相关的转录本似乎是Pol iv介导的基因沉默的目标,但我们认为,这数千个转录本不能解释观察到的整体转录增加rmr6突变叶,还给出了转录增压也全局影响非TE转录物(P.= 1.5×10-4由钴测试;无花果。3 c).考虑到玉米基因的66%位于1Kb的带注释的转座子[内30.,39, te可能驱动在全基因组中观察到的表达变异rmr6突变体。
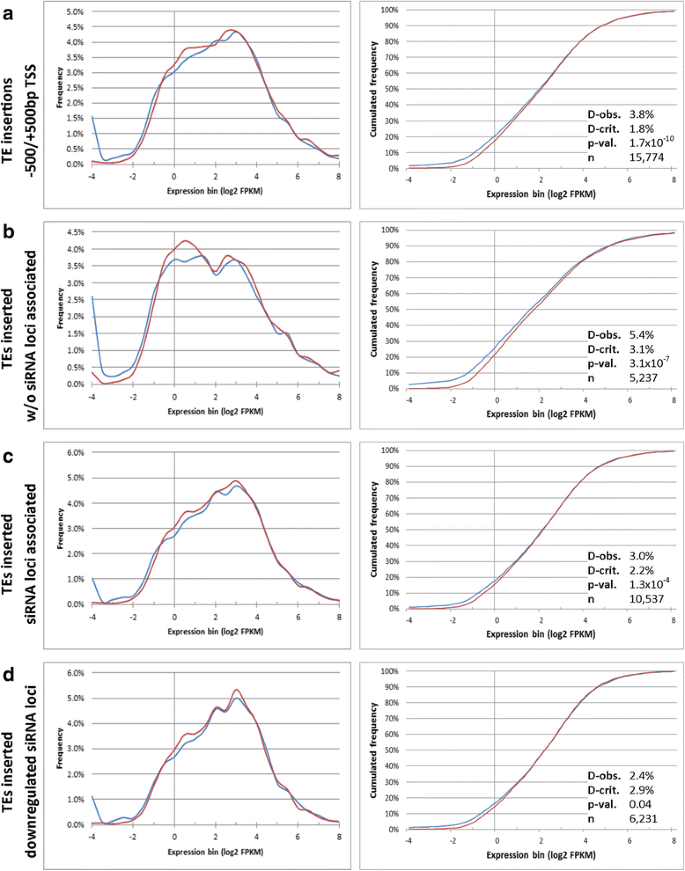
为了验证这一假设,我们将TE插入作为一种独特的特征,将TE插入到转录起始位点上游1Kb内(−1Kb TSS)、基因体内或转录终止位点下游1Kb内(+1Kb TTS;见附加文件7以获得基因如何根据te分类的示意图),并分析它们在B73和B73中的表达谱RPd1时/ rmr6突变体。可以预测,在所有三个基因类中,B73中表达0 FPKM的基因百分比(上游1.52%,下游1.53%,内部1.06%)都高于在B73中RPd1时/ rmr6(分别为0.08%、0.08%和0.04%)。此外,两种基因型的基因表达分布也有统计学差异(附加文件)8 a - c),尤其对于由te插入TSS上游的基因(P.= 3.3×10−11,通过K-S测试)或TTS下游(P.= 1.0 × 10−11)与在基因体内的TE插入的基因相比(P.= 8.6×10-9).表达分布和累积频率的直方图显示出与附近的TE插入(以及在基因体内的较小程度上的基因表达更高,用于表达值为0.5至10 fpkm(在附加文件中表达箱-1至3。8 a - c)在RPd1时/ rmr6突变体与B73相比。相反,没有TE插入(内部或附近)的基因在基因型之间的基因表达分布相似(P.= 0.08,用K-S试验;额外的文件8D.).此外,平均基因表达水平和RPd1时/ rmr6对邻近基因的沉默效果与距离最近TE的距离直接相关(附加文件8E).在B73中,有近端te(上或下)的基因的平均表达水平低于没有近端te和的基因波尔四世功能丧失导致其在更高的表达(附加文件8E).
通过1 Kb滑动窗口扫描TE插入考虑的基因列表坐标,重新定义TSS的最大TE效应区间(-500 bp ~ +500 bp),改进了TE对基因表达的影响分析。这个基因集,带有跨越TSS的TE插入(参见附加文件中的示意图表示)7),在B73中表达0 FPKM的比例(1.9%)高于rmr6(0.11%;P = 0 / Fisher精确检验)。他们的表达谱分布在统计学上也有很大的差异(P.= 1.7 × 10−10,用K-S试验;无花果。4),两个基因型的累计频率之间的最大距离为3.8%(插入TSS上游、TTS下游和基因体内的基因的累计频率分别为3.4%、3.5%和3.3%)。te的影响跨越TSS插入基因的表达非常明显:减去这些位点插入上游的TSS或基因体内导致这两个基因的表达谱差异的扁平化组织和任何的损失统计差异基因型(数据没有显示)。
TE插入跨越基因TSS影响邻近基因的表达RPd1时/ rmr6突变体,独立的siRNA位点。表达分布(左)和累积频率(右)的基因具有> 0的FPKM直方图,揭示之间的表达分布的高方差RPd1时/ rmr6和B73基因的TE插入跨越TSS(−500/+500 bp TSS;A.;经K-S检验,P < 0.01)。与插入到转录起始位点上游1Kb内(−1Kb TSS)、转录终止位点下游1Kb内(+1Kb TTS)或基因体中的基因相比,这些基因型的累积频率之间的距离最大(3.8%)8).附近的TES插入(-500 / + 500bp TSS)的基因进一步细分:没有相关的siRNA基因座(B.),siRNA基因座在其上游区域相关联(-1kb tss;C.)和先前被鉴定为下调的siRNA位点RPd1时/ rmr6突变体(D.).基因表达的分布在两种基因型之间有很强的统计学差异(D-obs = 5.4%;无sirna相关位点的基因(K-S检验P < 0.01)B.),较小程度上(D-obs = 3%)具有相关siRNA位点的基因(C.).具有sirna相关位点的基因在RPd1时/ rmr6突变体在两种基因型之间显示出非不同的特征(P.= 0.04,经K-S检验)
总之,这些结果表明,位于附近基因(特别是在- 500/+500 bp的TSS区间)的te是Pol iv介导的基因沉默释放的主要调控因子rmr6突变体。
te介导的基因表达变化与体内sirna的缺失没有直接联系rmr6突变体
Pol IV需要产生24nt siRNA,其进入触发DNA甲基化和在同源DNA序列中的转录沉默的RDDM途径(在[1])。我们的研究小组最近在B73和RPd1时/ rmr6共鉴定出244000个产生23- 24nt sirna的基因组位点(主要位于基因侧翼区),其中1 / 4的sirna显著下调RPd1时/ rmr6而B73。不管怎样,sirna的丢失是由RPd1时/ rmr6突变一般不能预测突变体中上调的809个基因的表达诱导[23].
为了进一步解决波尔四世我们将之前鉴定的24个nt siRNA位点的基因组坐标与覆盖TSS(−500/+500 bp TSS)的16,091个TE插入基因的基因组坐标进行交叉,并比较了不同亚群的表达分布。在TSS 1 Kb范围内,5425个没有sirna相关位点的基因表达谱差异较大可和B73(P.= 3.1 × 10-7由钴测试;无花果。4 b).在这个基因中,表达频率较高RPd1时/ rmr6与B73相比,在1 - 8 FPKM(表达箱0 - 3)范围内,两个基因型的累积频率之间的最大距离为5.4%,而与至少一个siRNA位点相关的10,666个剩余基因的表达谱差异较小(最大距离为3.0%;P.= 1.3 × 10-4;无花果。4摄氏度).出乎意料的是,具有sirna相关位点的6289个基因,先前发现的显著下调RPd1时/ rmr6突变体(23]显示两种基因型间无差异(最大距离2.4%;P.= 0.04;无花果。4 d).
相同的结果分析的21447个基因内TE插入1 Kb TSS和21333个基因的上游+ 1 Kb TTS TE插入有关,其中14952年和13215年有一个核轨迹关联与抑制核位点(8699和6111),分别(数据没有显示)。
虽然波尔IV需要siRNAs生源论,这些结果表明,与相关的核基因位点被波尔IV功能丧失的影响较小,比那些没有他们,确认还在全基因组水平,失去siRNAs并不直接相关转录激活在这些位点。
特定基因的识别失调rmr6-1突变体叶
将邻近TE插入作为Pol IV介导的基因表达沉默的主要特征,我们进行了差异表达分析,以进一步了解Pol IV靶点。与以往应用的表达谱比较不同,差异表达分析倾向于鉴定高表达基因;换句话说,在标准的转录组分析中,高表达基因被检测为差异表达的概率比低计数基因的概率大,而低计数基因是主要原因RPd1时/ rmr6转录上升(在[48])。无论如何,这差异表达分析方法具有限制的目标的数量的优点,特别是着眼于主要和统计学显著的。鉴于大量样品进行分析(4个生长条件中,两个时间点对于每个基因型),我们进行了三个独立的成对比较分组以不同的组合被测序的样品:i)对照试验组(B73:C_T0 + B73:C_T7 VSrmr6: C_T0 +rmr6: C_T7);ii)压力测试集(B73:D_T0 + B73:S_T0 + B73:S + D_T0 vsrmr6: D_T0 +rmr6:S_T0 +rmr6: S + D_T0);iii)所有测试集(所有B73样本vs所有rmr6样本)。除了典型的TopHat/Cuffdiff管道[49],也分析了这最后一组与RSEM软件工具[50],使用一种替代策略来有效处理模糊/多重定位读取,从大量短的单端读取开始准确估计基因水平丰度。RSEM定量后,用EBSeq计算差异表达[51].RSEM + EBSeq鉴定出更多的差异表达基因(log2FC > |2|,对应4倍的转录变异,且FDR < 0.05)1).
在所有四种分析中,有更多的基因上调(是沉默基因的7-9倍),证实了RPD1/RMR6负调控基因表达。的诊断型RPd1时/ rmr6-1纯合个体是表观遗传沉默的释放b1(booster1)基因,该基因编码一种基本的螺旋-环-螺旋蛋白,该蛋白是花青素生物合成酶的转录激活物[52].的一个标志波尔四世因此,功能的丧失是B1,在所有进行的差异表达分析中发现其高度上调(附加文件9).
我们选择了四种独立表达分析中至少三种表达差异的基因进行进一步研究,因为它们可能代表RPD1/RMR6的特异性靶标。基因位于30 Mb的区域周围RMR6-1基因座(21个上调和12个下调)被排除在下游分析之外:它们的错误调控可能是由于基因导入之间的序列多态性(增加基因表达或损害读图)RPd1时/ rmr6突变体及B73 [41].由此产生的880个位点通常在RPd1时/ rmr6而B73中只有71个基因表达上调,说明该基因的保护程度较高波尔四世去抑制的目标与沉默的目标形成对比,沉默的目标可能代表间接的、不共享的目标(附加文件10.).
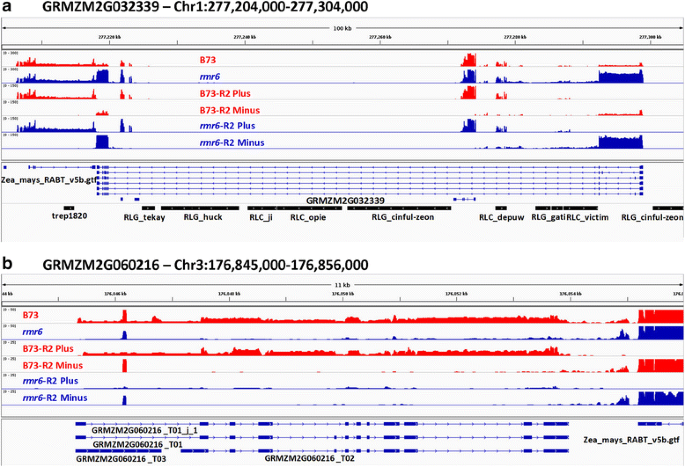
在共享差异表达基因中,未富集GO项;而DNA整合(GO:0015074)这一项在两者之间最具代表性RPd1时/ rmr6上调基因,提示在这一组中te相关基因过多。其中约三分之一是由从头注释的基因间位点(附加文件10.;参见[40]);许多特征明确的蛋白编码基因也在RPd1时/ rmr6叶片,如先前在幼苗中所示[41].te控制的同源结构域亮氨酸拉链IV的过表达(HD-ZIP IV)ocl2在叶片中也得到了证实,同时还鉴定出了两个受Pol IV功能缺失强烈上调的MADS-box转录因子:ZmMADS4 (GRMZM2G032339;无花果。5A)和ZmMADS52 (GRMZM2G446426)。前者被认为是花诱导和花序发育的基本调节剂[53],而后者虽然在茎尖和花序原基(http://www.qteller.com/qteller4/)表明它参与了营养到生殖阶段的转变和花的发育。
基因组浏览器的RNA-Seq读取映射在RPd1时/ rmr6misregulated转录因子。基因组浏览器(IGV -整合基因组浏览器;http://software.broadinstitute.org/software/igv/) B73(红色)及RPd1时/ rmr6-1突变体(蓝色)RNA测序读取(归一化至总的映射读取)在ZmMADS4(A.)及LG2 (B.)转录因子。报告总映射读(重复1和2)和特定链映射读(重复2)。在(A.)只是在长中心内含子中注释的30多个te中的一小部分
在下调的基因中,我们发现了另一个叶片发育的调控因子:基本区域/亮氨酸拉链基序(bZIP)转录因子Ligulless2.(LG2./ GRMZM2G060216;无花果。5B.)在缩小Ligule和耳廓在玉米叶中发育的区域,以及调节生殖相转变的植物[54,55].
突变体分析显示ZmMADS4过度表达导致早期开花表型[53,而在没有的情况下lg2基因功能正常的营养向花序阶段的转变可能被延迟[55]:其结果是显著推迟了开花时间RPd1时/ rmr6突变体植物(28].
RdDM直接目标的预测rmr6-1和mop1-1misregulated基因整合
Pol IV转录本被Pol IV相关RNA依赖RNA聚合酶2 (RDR2 [14,16]),加工成双链RNA (dsRNA)进入RdDM沉默通路。找出那些可能是RdDM直接靶标的基因RPd1时/ rmr6-1在两项独立的研究中发现的失调基因MOP1-1 / RDR2.玉米突变体42,43].
考虑到两种不同的组织mop1-1突变 - 射击顶端营业[43和未成熟的耳朵[42,我们发现在两种基因型中有27个基因表达上调,7个基因表达下调11.和12.):代表最可能直接受Pol IV/RDR2表观遗传调控途径调控的基因。
第一个共享的RdDM上调基因是MFP1附着因子1 (GRMZM2G479245),该基因包含正确的蛋白靶向和细胞分裂刺激所必需的核膜靶向结构域(WPP)。第二个候选基因是GRMZM2G012160,它是半胱氨酸蛋白酶抑制剂cystatin2的编码基因。它在拟南芥中过表达增加了对非生物胁迫(如盐、渗透、冷胁迫;[56,57].该观察结果进一步证实了先前假设的应力状上造成的epiregulator突变[基因组调节作用40].先前描述的上调ZmMADS4(GRMZM2G032339)基因强调了RdDM在控制植物开花和繁殖等重要方面的作用。
其中一个共享的下调基因是GRMZM2G131756,推测为沉默抑制因子1 (ROS1)/ de米特- like1 (DML1)的同源基因,一个拟南芥DNA糖基化酶蛋白,可以主动去甲基化DNA [58,59].ROS1在几个拟南芥RdDM突变体中下调[60],正如之前的假设,去甲基化活性的降低可能导致RdDM突变体中几个二级靶标的下调[42].
Pol IV沉默位点在I类te中富集
尽管出现在控制缺乏功能性波尔IV的基因表达的跨国企业的重要作用,总体TE转录出现温和。与至少一个转录先前分类为HC-TE或PR-TE的轨迹分别表示有关上调和下调的基因,为20%(176个基因)和15.5%(11个基因),而这些的TE代表16%全基因注释。打破差异表达TE相关基因组到不同的家庭(图6A额外的文件13.), RLC Copia和RLG吉普赛class I retroelement超级家族尤其丰富RPd1时/ rmr6上调基因,而DTC-仙人掌和其他II类DNA转座子包括在11个下调的TE相关基因中。为了确定与野生型突变叶转录组中不同的TE系列在突变叶转录组中表达,我们重新映射了对代表从玉米转换元素数据库检索的1526个全长TE家族的基因组序列的所有测序读数(http://maizetedb.org/~maize/).在此分析中,我们选择了唯一映射的reads,使用edgeR进行差异表达分析[61,共鉴定出43个表达强烈的转座子家族和13个表达强烈的转座子家族RPd1时/ rmr6突变体(附加文件14).此外,该分析还证实了许多LTR反转录转座子(RLG和RLC超家族)的去抑制作用,以及许多II型Mutator元件,其中RLX和CACTA元件在沉默的TEs中最为丰富。
POL IV沉默基因座在I类转座元件富集和优先地共定位。上调和下调TE超家族的相对百分比相比,他们丰富的基因注释(A.),表明RLC-的Copia和RLG-吉普赛I类retroelement超级家庭特别是中富集RPd1时/ rmr6上调的基因,而DTC-CACTA和其他II类DNA转座子超家族矿下调的基因中过表达。该分布图(B.),表明沿1号染色体的差异表达基因优先共定位RPd1时/ rmr6去抑制的基因。染色体中的1.2Kbp 100不重叠的窗口,并为每个窗口的基因的百分比划分的(相对于总的染色体上的基因;蓝色条)和过表达的基因(相对于该窗口基因含量)的报告。黄条和红条分别表示在至少三到两个独立比较中共享的上调基因的窗口百分比。置换试验采用regioneRř包(C.)显示,RPd1时/ rmr6强烈上调基因(bin 3)比中度或轻度上调基因(bin 2和1)更接近te:每个bin与最接近te的平均距离分别为1500 bp、1547 bp和1605 bp(整个基因集的平均距离为2017 bp;灰色分布)。同样,稍微和适度下调基因(垃圾箱−1和−2)位于逐渐远离te(分别为2461个基点和2514个基点),而基因的本−3(那些表现最强的下调)没有显示任何位置偏离整个基因集(2008 bp平均距离)。用Cuffdiff从所有测序样本出发,得到用于排列分析的差异表达基因(log2FC > |1|, FDR < 0.05)
其中RLG Ahoru过表达量最高RPd1时/ rmr6突变体,也是新注释的基因间差异表达位点中最具代表性的一个。如果它的RMR6沉默是不可否认的,那么对读比对文件的人工检查显示,所有唯一映射的读都被限制在GAG编码区域的上游300 bp长的区域内,而所有其他DE元素的读均分布在整个TE序列上。这个300 bp长的区域在B73基因组中高度重复(通过blast分析至少有100个近乎完美的匹配),可能与玉米基因组中注释的385个Ahoru部分片段中的一部分相对应[31].这些作者报告了AHORU仅一个单个全长插入和385个局部碎片,384nt平均长度,这可能会受到RDDM沉默。
RPd1时/ rmr6过表达的LTR家族是低拷贝数家族(附加文件14),这些基因以前被证明是最常插入或靠近基因的[31,证实了最近提出的RdDM在控制近TE插入基因中的作用。
Pol IV沉默位点是共定位的,更接近te
分布分析rmr6玉米基因组中上调的基因显示约有三分之一处于聚类位置。我们鉴定了100多个100 Kbp基因组窗口,包含2到10个RPd1时/ rmr6de-repressed基因(图。6B和附加文件15).而这71个下调的基因却均匀地分布在玉米染色体上,被包含在67个独立的基因组窗口中16).作为对照,将上调基因沿玉米染色体的分布与100个独立数据集的平均分布进行比较,每个数据集随机从40457个表达基因中选取880个基因。这两种分布在统计上有很大的不同(P.= 1 × 10−16通过Wilcoxon检验):随机基因导致更均匀地沿基因组中(附加文件分布15),平均而言,它们被包含在876个基因组100 Kb窗口中,而包含上调基因的737个基因组中(143个上调基因- 16.25%-随后在其他上调基因的同一窗口中共定位)。
人工检测聚类上调基因发现TEs插入每个聚类或附近,明显占优势的是LTR逆转录转座子的RLC和RLG超家族(Additional file)17).此外,我们还发现,在许多共同表达的簇中,上调基因仅在前面描述的一到两个独立比较中显著上调(图)。6B额外的文件15).
进一步研究差异表达基因与邻近TE插入之间的关系发现,上调基因比基因组中基因的平均值更接近TE (P.< 0.005):他们与TEs的平均距离为1550 bp,而基因组中整个基因集的平均距离为2017 bp,通过排列检验获得(附加文件18).来自的下调的基因的TE相反平均距离较长(2374碱基对; P <0.005)比在基因组中的基因的(附加文件18).
当差异表达基因根据它们的折叠变化进行细分时(附加文件)19),转录变异与距离最近的te的距离之间的直接关系更为明显,特别是对于上调基因。RPd1时/ rmr6高上调基因(bin 3)比中上调基因(bin 2)和轻度上调基因(bin 1)更接近TE,分别与TE距离近1500 bp、1547 bp和1605 bp(图1)。6C).同样,轻微下调(bin - 1)和适度下调(bin - 2)的基因位于离TEs越来越远的地方(分别为2461 bp和2514 bp)。相反,排列测试显示,沉默最强的基因RPd1时/ rmr6突变体(bin−3)与TEs的距离与玉米基因(2008 bp;无花果。6C).
波尔四世错误调节的基因座位于与B73不同DNA甲基化谱区域
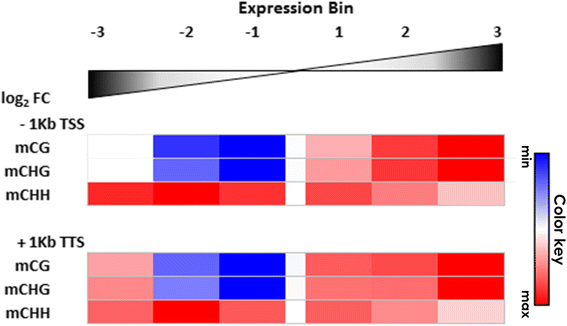
上述结果进一步证明了TES和基因误解之间的相互作用。它们还表明了基因调节的多个互连机制:如果接近TES与基因过度表达的大小直接相关RPd1时/ rmr6突变时,强沉默基因不显示任何特定的位置偏差。由于在整个RdDM通路中,Pol IV对于DNA甲基化的建立和维持是必需的,特别是在mCHH岛水平上,我们确定了在已识别的错误调控位点上的位点特异性DNA甲基化状态。B73胞嘧啶甲基化全基因组图来自已发表的对幼苗的亚硫酸氢盐全基因组测序研究[62来创建DNA甲基化水平的快照。每一种情况下(CG, CHG和CHH)的DNA甲基化谱都在调控错误的基因的侧边区域进行了评估,如前所述,在表达箱中细分(见方法;额外的文件19).甲基化图和排列分析表明RPd1时/ rmr6差异基因在CG和CHG基因高度甲基化区域与整个组(浓缩似乎直接相关的超表达的程度),而理气的,属于垃圾箱和−−1 2 CG和两侧翼CHG甲基化区域较低(图7额外的文件20.和21).需要特别注意的是那些在基因中表现出最强下调的基因RPd1时/ rmr6(bin−3):在它们的侧翼区域,这些基因的CG和CHG甲基化水平与整个基因组的平均水平相当,高于−2和−1的水平。7额外的文件20.和21).
差异表达基因侧翼区域的DNA甲基化概况。在每个上下文甲基化水平(CG,CHG和CHH)计算了侧翼区(2 KB为CG和CHG,1 KB的CHH;参见方法)的差异表达的基因和相比,平均的基因在基因组中使用regioneR排列方法。对于每个区间,在各侧翼区的平均甲基化水平是通过热图的方式报告。红色和蓝色渐变表明甲基化值统计学高于或低于平均值的整个基因组的。平均甲基化值和相关P.-values在附加文件中报告21
相反,在RPd1时/ rmr6(bin 3)与其他上调(bin 1和bin 2)和下调(Fig. 2)相比,其侧面有轻微的CHH甲基化区域。7额外的文件21),其中mCHH岛可明显检测为尖锐的甲基化峰(附加文件20.).
Pol IV活性与重复基因调控的关系
玉米基因组与其他草类物种基因组的共线比较显示,玉米在500万至1200万年前经历了整个基因组复制[63,64,这就产生了两个截然不同的亚基因组,每个亚基因组最初都包含一套完整的基因和调控序列[65].在许多情况下,一个基因的两个副本中的一个在基因组中丢失,这个过程被称为分馏;然而,现代玉米基因组中保留了3000到5000对基因[65].因此,玉米基因补料可以分为三类:两种子组织(两种基因已保留的两种基因的重复基因,只有一个亚基因的单拷贝基因,也称为Singletons(相应的职业从四倍化以来已经丢失)),并且不能分配给亚基因组的基因。亚基因段分区显示,玉米隐性亚基因2的特征在于较高的基因损失而不是优势亚基因1.此外,在保留对中,隐性亚基因组中的基因往往比其优势职业更低的表达[65].与之相反,第三类的特征是在其他草类物种的基因组中缺乏同源基因,这表明大多数这些非同源基因是由单基因复制机制产生的,很可能是在玉米全基因组复制之后[66].
为了探讨Pol IV在重复的基因调节和基因组占优势中的作用,我们分析了B73和B73中的ParaLog表达动态RPd1时/ rmr6突变的叶子。从该组验证基因模型(FGS:过滤基因组; 39656个基因)的开始,我们确定了23895个基因传递前面所述的表达滤波器。这其中,我们制定了两套基因列表:在我)基因显著上调rmr6突变体(log2FC> 1,FDR <0.05)或ii)的基因显著下调(B73过表达时,log2FC <-1,FDR <0.05)。用Cuffdiff在先前所描述的“全测试集”而获得该分析表达的倍数变化。各基因的频率集合分裂成两个subgenomes(http://www.skraelingmountain.com/datasets/maize_indexed_by_subgenome.csv),并在附加文件中报告五个不同的类22.需要注意的是上调的总数和下调的基因是在FGS,进行基于序列相似性的其他物种和推定的全长编码序列,以排除转座子,假基因和其他低的存在所选择之间非常相似-confidence结构。
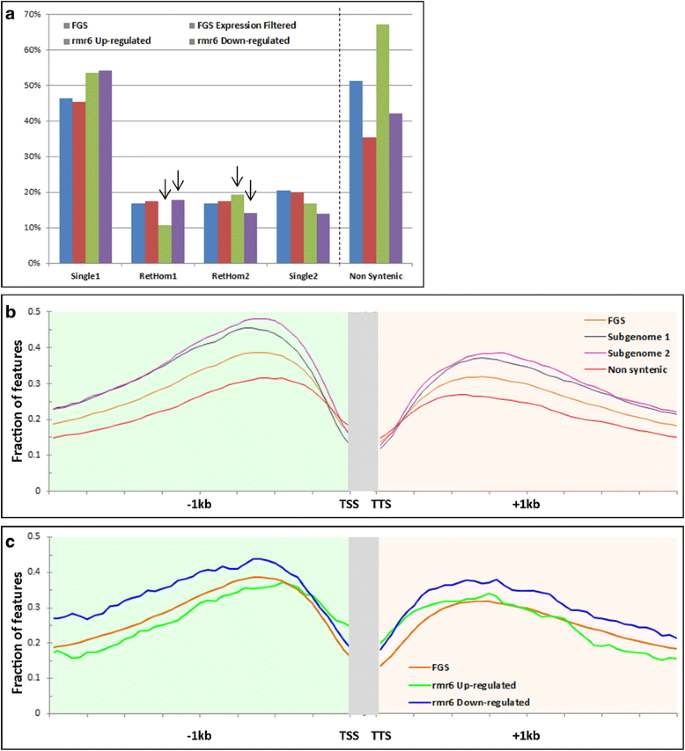
与“参考”FGS表达过滤基因集相比,rmr6上调和下调基因在保留同源基因中呈现两种截然相反的富集RPd1时/ rmr6亚基因组2中上调基因所占比例明显高于表达基因列表,而亚基因组1中下调基因所占比例高于亚基因组2(图2)。8A;额外的文件22).如果亚基因组2的主要组成部分在RPd1时/ rmr6突变体过表达基因与保留在亚因子中保留的重复基因的特殊行为有关,更深入的分析显示了POL IV上调和下调基因中的亚群单身百分比的变化(图。8A).在未分配给任何亚基因的基因水平上观察到进一步的致命偏差,即使表达过滤基因的表达较少,也高度超过RPd1时/ rmr6上调(67%),高于B73 (42%;无花果。8A和附加文件22)诱导的基因。
基因组显性、Pol iv介导的基因沉默与siRNA位点占位的相关性将差异表达基因模型(log2FC > |1|, FDR < 0.05)细分为单个、两个亚基因组中的重复或非同染色体基因,显示亚基因组2同源基因和非同染色体基因在亚基因组中富集RPd1时/ rmr6向上调节目标(A.).在分配到亚基因组的基因总数中计算了单重和重复的百分比,而非同合基因的百分比则是在FGS基因总数中计算的,并在一个独立的尺度上进行了报道。siRNA位点在不同亚基因组侧翼区域的分布图(B.)揭示了亚基因组2基因比亚基因组1基因更可能有侧翼的24个nt-siRNA,而非同位基因的上游区域被24个nt-siRNA覆盖的比同位基因少。同样,24sRNA位点沿侧翼区域分布RPd1时/ rmr6上调或下调基因(C.)显示,突变的上调基因的侧翼siRNA往往比下调基因的少
通过对DE基因(至少有一个相似物差异表达的保留同源基因对)的鉴定,获得了两种基因型之间的相似物对差异表达的分析。62.5%的rmr6基因表达上调对,差异表达由隐性类似物引起,而基因表达下调对,差异表达率降至38.7%(表1)2).
24个nt- sirna优先靶向隐性亚基因组2,当它们从非同合基因中耗尽时
多倍体的最新研究芸苔属植物拉伯发现24 nt sirna定位于隐性亚基因组时优先靶向并覆盖保留基因的上游区域[67.,假设RdDM在基因组显性调控中的作用。
为了证实这一发现,并分析Pol iv介导的sirna在基因组优势中的作用,我们分析了已鉴定的24sRNA位点沿FGS基因集侧边区域和不同亚基因组类别的分布(我们的参考文献)。
正如在芸苔属中报道的,在玉米中观察到基因组显性和siRNA覆盖之间的间接相关性:隐性亚基因组2的基因优先在上游区域富集siRNA,保留同源基因和单拷贝基因之间没有差异(图)。8B.额外的文件23),而这些被分析的基因组之间的差异在下游区域不那么明显。此外,我们的分析指出,在非同位基因的上游区域,siRNA位点的频率非常低。
值得注意的是,与同序基因相比,非同序基因在其上游区域TE插入丰富(见表)3.),证实了它们起源于现有基因转座子改组的假设[68.,69.]但也再次确认TES沉默和siRNA基因座存在和表达之间的弱关系。
最后,沿FGS基因侧面沿着FGS基因侧的24SRNA基因座的分布分析及RPd1时/ rmr6上调或下调的基因进一步证实了siRNA位点丰度与基因多态性之间缺乏直接联系RPd1时/ rmr6去阻遏,与突变体上调基因趋向于有较少的siRNA在其侧翼高于那些下调(图8C.).
讨论
Pol IV活性的丧失影响基因的调控和植物的发育
中差异表达分析RPd1时/ rmr6突变体从1到4000个差异表达基因的鉴定,根据不同的应用生物信息学工具。在绝大多数的这些基因都上调波尔四世突变体,证实RDDM对基因转录的抑制作用。差异表达的基因包括直接POL IV抑制基因和间接靶标,由基因组的变化间接引发通过POL IV活性的损失。常规表达基因的一部分通常被解压RPd1时/ rmr6而在MOP1 / RDR2.突变体,甚至在分析的不同组织中,已经被确定为RdDM可能的直接靶标。
根据以前的研究[41,42,43],一个功能性RdDM通路的缺失会导致许多对植物发育重要的基因表达的失调和改变,包括miRNAs表达的差异[24这可能导致观察到的多性发育缺陷和突变表型RPd1时/ rmr6和mop1植物。虽然我们没有检测到任何差异表达的miRNARPd1时/ rmr6开发叶(23分生组织中多种调控途径的改变可能被认为是导致叶片组织基因表达变化的重要原因。因此,在RPd1时/ rmr6突变体(28[所产生的细胞类型在叶组织中的不同分布可以解释特定发育调控基因的间接计性。
DNA去甲基化酶ROS1/DML1的下调可能与mCHH水平的降低有关rmr6和mop1事实上,拟南芥ROS1的表达是由启动子区域的DNA甲基化促进的。这种甲基化诱导的表达导致去甲基化酶活性的微调,以响应甲基化改变,这使得基因表达和基因组防御之间的精细甲基化稳态得以实现[60].
Pol IV活性的丧失在全球范围内影响基因组调控
除了相对较低数量的差异表达基因外,对我们总RNA-SEQ数据的深度分析表明,POL IV活性的损失引起了玉米基因组的转录部分的全局增加。在差异表达分析中,由于呼叫显着差异表达的统计功率低,低计数基因确实不太可能被发现与高表达的差异表达。转录基因组的级分的升高,其不限于其重复部分,而是由许多基因座的小增加引起,并且伴随着野生型和突变植物之间的不同基因表达分布谱。RPd1时/ rmr6突变导致在新注释位点的水平,广大相比,B73,具有较大的差异基因的基础表达一个全球性的增加,先前确定的非编码斯尔纳前体[40].
这些位点应该是由Pol IV/Pol V RNA聚合酶正常转录进入规范RdDM通路,因此其表达量增加RPd1时/ rmr6突变体似乎是矛盾的。Pol iv转录本长度减少(25-45 bp)、低表达和寿命短[16,17,33]阻止它们在经典RNA-Seq实验中被识别。这些特征排除了在波尔四世突变:Pol IV转录本在野生型和野生型中均未被成功检测和分析RPd1时/ rmr6突变体。因此,缺乏波尔四世rna在RPd1时/ RPd1时与野生型B73相比,突变体不会导致人工过表达Pol II转录本,即由于阅读正常化偏差。观察到的表达增加smallRNA前体应该归因于RNA聚合酶II或活动的增加波尔诉在缺乏功能性RdDM通路,非规范的参与这些repeats-related成绩单波尔II-RDR6-dependent RdDM通路已经证明(70,71.].这个沉默途径已经被关联到TAS基因座和新集成的转座子,并且依赖于由RNA依赖性RNA聚合酶复制6(RDR6)和处理被Dicer样家族蛋白(DCL2和DCL4)插入21-22-聚合酶II转录NT的siRNA。当转座子沉默逸出,并且通过聚合酶II转录,它们的mRNA可被识别和降解,产生21-22个nt小RNA也可以指导初始DNA甲基化,在波尔V-依赖性方式与AGO4或AGO6相互作用以引发RdDM[1,72.].有趣的是,22 nt小rna在RPd1时/ rmr6以及发现的少数上调的小rna位点rpd1 / rmr6相对于野生型,大多数由22种nt rna主导[23].
总之,这些研究结果表明Pol II-RDR6非规范性RDDM沉默途径克服玉米中缺乏功能性Pol IV的累积。
TEs在RdDM中的阴暗角色
即使TE的整体转录水平适中,差异表达基因的比例也很低(上调基因占20%,下调基因占15.5%;相比16%的te相关基因注释),大多数非编码siRNA前体转录本都与te相关。表达谱的比较也表明,TEs可以直接影响邻近基因的表达,清楚地表明转座子是Pol iv介导沉默的重要靶点。
拟南芥的一些研究表明,TEs可以通过不同的机制影响近端基因的表达[73.,74.,75.].而据报道RdDM以转座子为靶点,主要在更小、更年轻的转座子水平上重复[76.,77.,78.,79.,这些元素在拟南芥RdDM缺陷突变体中一般不被调动。我们的结果证实了玉米中RdDM对年轻te的优先定位,也加强了转座子的传统调节作用。
我们确实发现,位于邻近基因的TEs(特别是在TSS的−500/+500 bp区间,但也在TTS的下游)是Pol iv介导的基因沉默释放的主要效应因子RPd1时/ rmr6突变体。这些结果证实了TES在玉米基因调控的“控制元素”中的作用。此外,与拟南芥中的TE缺乏基因相比,我们确认先前观察到靠近TES的基因的低表达[74.,80,这种定位沉默是由功能性RdDM通路介导的。最近证实了TEs对玉米基因响应非生物胁迫的调控作用[39,我们可以推测,在整个玉米发育过程中,te与宿主基因组的转录调控之间存在高度复杂的表观遗传和调控相互作用,以应对环境挑战和适应。
POL IV沉默基因座是独立的siRNA位点的
Pol IV在sirna的生物发生中是必不可少的,24nt sirna在细胞中显著减少波尔四世和其他RdDM突变体。相反,我们的结果表明,具有相关siRNA位点的基因较少受到基因沉默释放的影响波尔四世功能缺失突变体。这些明显相互矛盾的结果加强了对Pol iv介导的基因沉默在植物特别是玉米中的实际机制的讨论。在拟南芥中,转座子DNA甲基化从Pol IV/Pol V/RDR2自增强RdDM进展到RdDM独立沉默状态,涉及MET1-和CMT3/ cmt2介导的CG和CHG甲基化,以及抑制组蛋白标记H3K9me2的沉积[7].类似地,Pikaard和同事证明了RdDM位点上存在双锁机制[47].这些位点的沉默通过组蛋白修饰和CG DNA甲基化稳定和记忆,不受Pol IV功能丧失的影响。因此,可以假设绝大多数转座子的沉默仍然维持在两种玉米中(其中CG和CHG甲基化普遍存在,而在RdDM突变体中仅部分丢失[32,34)和拟南芥在缺乏功能的RdDM通路的情况下。此外,在这些稳定的沉默位点上,Pol IV/RDR2通路产生的sirna可以通过反式作用来沉默同源te、重复序列和附近插入年轻te的基因。用于siRNA位点分析的方法确实是旨在识别小rna转录起源的位点,而不是所有可能的靶标[23,81.].sirna的反式作用,以前与转基因沉默有关[82.[杂交种中的等位基因调控[83.,因此可能参与维持基因的抑制和缺乏siRNA直接转录的TEs,在缺乏功能Pol IV的情况下,沉默将失败。
POL IV活性的丧失导致染色质特征从基因传播到TES,反之亦然
与CG和CHG环境下的甲基化不同,CHH甲基化包围着基因,并被发现仅与某些类型的转座子有关[25,34,84.].独立分析(TE-related鉴定差异表达基因和读取重新映射的策划序列1526户长篇TE)透露,失去RdDM活动优惠转录激活的结果类我LTR反转位子活动属于RLG-Gypsy RLC-Copia总科。此外,许多RPd1时/ rmr6过表达的ltr由低拷贝数家族组成,这些家族以前被证明是最常插入或邻近基因的[31],确认先前讨论的结果和最近提出的RDDM在抑制在基因附近的可进入的活性染色质环境中的转座子[25,33].
RLC和RLG超家族的成员优先插入基因的侧翼区域,表现出强烈的上调RPd1时/ rmr6(它们与侧翼TE的66%的Bin 3基因相关),并且先前被证明,与II类DNA TES相比,较低水平的CG和CHG甲基化和较低水平的MCHH [25,84.].现有甲基数据的重新分析证实了高水平的MCG和mCHG的和mCHH相对低水平在上调的基因:在这些区域mCHH的松动,可预测在RPd1时/ rmr6突变体,可能伴随着转座子内CG和CHG甲基化的额外损失和te和基因表达的增加,如前所述mop1-1突变体(33].因此,功能性POL IV活性的丧失和连接的表观遗传调节导致可获得的染色质状态的蔓延与广泛染色体区的转录脱落(高达100 kbp)RPd1时/ rmr6,有几个位点一般错误调控。
相反,我们发现CACTA II类转座子大多包含在波尔四世下调TE范例,如先前所观察到mop1-1突变体(43].II类DNA TEs已被更多地描述为与24个nt srna和mCHH岛相关[23,33],也优先发现于下调基因的侧翼区域(主要是CACTA, Mutator和hAT超家族)。综上所述,mCHH岛作为开放染色质和异染色质边界的作用,不能排除这些基因在RPd1时/ rmr6与野生型相比都受到从异色侧翼区域到相邻基因的扩频沉默痕引起的。即使这种现象不是由在先前的研究中证实mop1-1突变体(33],它进一步通过相对高的扩散LTR元件(LTR回收器的频率频率(将沉默标记的扩展散布)进一步证实,例如DNA甲基化和H3K9ME2到相邻序列的沉积标记[85.)发现更接近于强沉默基因(箱2和箱3)。
TEs附近,这些证据表明基因(基因,或者附近的TE插入)在玉米RdDM的优先目标,同意的建议角色RdDM开放染色质拥有之间的边界地区和访问异色的区域与高架CG / CHG甲基化(25,33].
RDDM指导玉米基因组的进化和稳定性吗?
结果还表明存在额外的表观遗传TE沉默机制:在没有Pol iv衍生的24 nt- sirna的情况下,Pol ii - rdr6 -依赖的21-22 nt sinas可以有效地维持TE沉默,至少在第一代(RPd1时/ rmr6与非突变体同胞相比,突变体在植物质量方面呈现渐进性跨代退化;[27,28])。此外,在其他生物中,如果没有额外的RNA聚合酶和专门的转录机制,转座子就会沉默。最近的研究表明,在开花植物中,RdDM途径的Pol V分支已经达到了最大的复杂性:这可能表明,RdDM具有一种尚未明确的功能,而这种功能对这一特定的植物群尤其重要。因此,对RdDM在开花植物中的作用的推测,提出了该途径在加速多倍体基因组二倍体化中的新作用[44].
在波尔四世突变后,差异表达基因在两个玉米亚基因组中出现了意想不到的表现。玉米基因组由来自古代异源四倍体化事件的两个亚基因组组成:这两个亚基因组是可区分的,因为优势亚基因组1比亚基因组2保守更多的基因,这种现象被称为“基因组优势”。此外,在保留基因对中,显性亚基因组上的基因往往比隐性同源基因表达更多[65,66].更大比例的基因上调RPd1时/ rmr6突变体属于亚基因组2,代表重复对的隐性同源。在成熟花粉的转录组中仅观察到隐性亚基因组2成员的类似表达富集[86.和一些基因特异性地去抑制RPd1时/ rmr6突变体以前被认为是花粉特异的。玉米25个组织的RNA-Seq数据来源于6个独立的研究(http://www.qteller.com/qteller4/)显示表达的表达RMR6Gene在花粉细胞中不可检测到(http://qteller.com/bar_chart.php?name=zm00001d031459&info=.).同样,在拟南芥花粉发育过程中发现RdDM成分下调[87.,88.,89.,并随之耗尽CHH甲基化花粉转座子。这一现象可能代表了一种古老的转座子识别机制,以下是拟南芥胚胎受精后CHH甲基化的恢复[88.,或为使通常在营养组织中被RdDM沉默的花粉特异基因得以表达所必需。
高百分比RPd1时/ rmr6未分配到亚基因组1或亚基因组2的上调基因(67%的上调基因)是相当罕见的,因为它们代表50%的FGS基因,而且在全球范围内比同基因的表达更少。非同同基因被认为比同同基因“年轻”,因为它们是在最后一次全基因组复制之后进化的(例如,通过蛋白质编码域的模块化重排[90.],藉由现有基因的转位[68.],或通过从陆心胶和其他转座子的外显子切开,导致融合基因[69.])。最近,非同步基因已与杂种优势和杂化活力表现有关[91.].
值得注意的是,与同序基因相比,非同序基因在其上游区域TE插入丰富,这证实了之前的假设,即它们是由现有基因的“近期”转座子改组产生的。此外,非同同基因在其上游区域被24nt siRNA覆盖的程度低于同同基因,再次证实了TEs附近基因与siRNA位点占位之间的弱关联。这种较低的siRNA丰度与它们全球较低的表达量是一致的:无论是编码基因还是非编码基因,都证明了基因表达水平与上游siRNA位点的占据率呈正相关[23,34].另外,24个nt-siRNA位点在其侧翼区域的低丰度也可以解释,考虑到它们最近的进化,并假设需要更长的时间来设置和建立功能性siRNA位点。
相反,在玉米中发现了基因组优势度和siRNA覆盖度之间的负相关:隐性亚基因组2的基因(总体表达水平低于亚基因组1的基因)在上游区域富集siRNA,保留同源基因和单拷贝基因之间没有差异。所有这些数据都与Pol iv介导的RdDM途径参与(并可能继续指导)基因组优势调控和亚基因组稳定性和进化的想法一致,可能通过24个nt- sirna的产生平衡TEs活性,如之前的报道芸苔属植物拉伯[67.,92.].
结论
在本研究中,我们从玉米的第一个全RNA-Seq分析开始,广泛地研究了RPd1时/ rmr6这表明,RdDM活性的丧失导致玉米基因组转录部分的整体增加。深入转录组分析与TEs分布、smallRNA靶向和DNA甲基化水平的调查相结合,证实TEs插入邻近基因广泛影响邻近位点的表达。TE对邻近基因表达的影响与基因侧翼区域的甲基化谱有关,而这些谱严格依赖于插入的TE成员的特定特征。
该研究还表明,Pol iv介导的RdDM途径参与了基因组显性调控和亚基因组稳定性和进化。由于缺少MET1同源性的玉米突变体(负责CG甲基化维持),如果可能的话,只能在CHG和CHH甲基化途径之间创建可行的双突变体(到目前为止不成功;[32[]),最终探讨RdDM和DNA甲基化在玉米精细平衡te沉默中的作用,通过基因表达调控和基因组世代稳定性。
方法
植物材料、生长条件和总RNA-Seq
当Zea Mays.B73自交系和rpd1-1(也称为rmr6-1)空的突变体[27],用于RNA-Seq和sRNA-Seq分析。B73近交系种子取自中北地区植物引种站(NCRPIS;USDA-ARS),而rdp1-1 / rmr6-1变种种子由加州大学伯克利分校根据材料转让协议提供。在2011年和2012年的春夏生长季,植物在温室的盆栽中生长,并遭受干旱(D)、盐(S)和干旱+盐(D + S)胁迫,如[23,40,93.].3个生物复制(R1, R2, R3)分别采集16个叶片样品。
根据制造商的说明,使用光谱植物总RNA试剂盒(SIGMA)从冷冻组织中提取总RNA,并进行柱上dna酶切(SIGMA)。使用epicter - illumina公司的Ribo-Zero™rRNA Removal kit (Plant Leaf)进行rRNA清除。使用TruSeq RNA Library Prep Kit进行第一次复制(非定向测序),使用TruSeq strand RNA Library Prep Kit(定向测序)进行生物复制2和3,并将其合并测序[40].Illumina对32个RNA-Seq文库的测序是在意大利的Istituto di Genomica Applicata (Udine, Italy)平台上进行的,多重水平为4,平均每个文库产生3700万个50 bp的单端读取(附加文件)1).
总RNA测序读取对准和基因组引导转录组件
Forestan等人详细描述了生物信息学分析[40].简而言之,使用Cutadapt 1.2.1对序列读取进行预处理,以便进行适配器剪切[94.]然后修剪质量评分并从RRNA污染物读取过滤(RRNA参考序列被从中检索http://www.arb-silva.de/)与ERNE-FILTER 1.2 [95.].由此产生的高质量读取(附加文件1)被定位到玉米B73参考基因组(RefGen ZmB73 Assembly AGPv3和Zea_mays.AGPv3.20。gtf transcript annotation)与Tophat 2.0.9 [96.].大约50-60%的比对结果为多重图谱(同样可以很好地映射到两个或更多的基因组位置):为了保存它们的生物信息(即来自副同源基因和重复序列的转录),避免对下游分析产生负面影响,我们决定放弃对10多个不同基因组位置的mapping。因此,使用Samtools过滤掉MAPping Quality (MAPQ)小于1的Reads和PCR重复[97.,将多映射读减少到大约30-40%的映射读(附加文件1).如[40,从R2 + R3测序中读取,以链特异性模式对齐,使用Cufflinks 2.2.1 - RABT模式- [45,49结果发现3410个新的转录位点和21,400个已知位点的潜在新亚型。
利用BLAST Best Hits对转座因子相关转录本序列进行鉴定和分类。所有160488份转录本模型(参考加上新注释的转录本[40玉米TE数据库中包含1526条非冗余玉米TE (http://maizetedb.org/~maize/).采用严格的标准对比特评分和覆盖率进行评分,以识别与te相关的转录本,并将其分为两个亚组:9766高置信- tes (HC-TEs:比特评分> 500和覆盖率>50%)和9013假设/残留- tes (PR-TEs:比特评分> 250或覆盖率>30%)。然后将每个te相关转录本与其特定的te家族和超家族关联,分析突变样本中特定te类的优先转录。
基因组转录部分的定量分析
从16个测序的读取对齐(MAPQ≥1)从16个测序的每个基因型的读取对齐(MAPQ≥1)组合产生两个BAM文件,其中包含438,275,238和458,429,084的B73和458,429,084RPd1时/ rmr6,分别对应714,514,067和775,422,668条线。这两个BAM文件通过随机向下采样归一化到7亿对齐(由4.3亿和4.1亿读取B73和RPd1时/ rmr6,分别),然后计算每个基因组碱基位置的覆盖率。类似地,唯一映射的读取(MAPQ = 50:只分配到引用中的一个位置,并使用Samtools [97.),在基因组转录估计前,每个BAM文件随机取样至2.9亿reads。然后,利用BEDTools功能“genomeCoverageBed”计算出两种基因型玉米基因组的转录部分[98.]从下缩放的BAM文件开始。在三次重复中进行了缩小和覆盖率计算,并报告了平均覆盖率。
对每个个体样本(在个体RNA-Seq文库水平上进行降采样后;额外的文件3.).
表达量化和差异表达分析
基因及转录本表达值(包括已知及新注释)[40]为每种基因型量化(将不同的生长条件和时间点考虑在一起)与铆接并用袖口归一化[45生成所有基因和转录本的标准化FPKM(每千碱基每百万映射片段的片段)值的制表符分隔的矩阵(附加文件24).
使用Cuffdiff进行两两差异表达分析[45选择以下选项:——multi-read-correct,−-compatible-hit -norm,−-dispersion-method per-condition和——library-norm-method quartile。在B73和RPd1时/ rmr6在控制条件下生长的植物中,初步试验显示没有差异表达所述两个时间点(T0和T7,数据未示出)之间的基因(或转录物),这表明在两个收集点不同的植物发育阶段没有引起表达的变化。
为了鉴定Pol IV调控基因,采用3个两两差异表达分析,将测序样品按不同的生长条件进行分组:a)对照试验集(B73:C_T0 + B73:C_T7 vsrmr6: C_T0 +rmr6: C_T7);b)中的应力 - 测试组(B73:D_T0 + B73:S_T0 + B73:S + D_T0 VSrmr6: D_T0 +rmr6:S_T0 +rmr6: S + D_T0);c)所有测试集(所有样品B73 vs所有样品rmr6).除了典型的TopHat/Cuffdiff管道[49,使用RSEM v1.2.25软件在“All-test-set”上进行差异表达分析[50]和EBSeq [51].在这种情况下,每个库的高质量读数都用Bowtie2单独映射[99.]来推断归一化基因丰度,EBSeq使用这些归一化基因丰度来检测差异表达基因。滤除Cuffdiff和EBSeq输出的log2倍变化率≥|2|和FDR调整P.值≤0.05,识别有统计学差异表达的基因。
基因表达分布及统计分析
在使用Cuffdiff(大致对应于所有条件下FPKM < 1)进行的所有三种表达分析中,检测状态为NOTEST或LOWDATA的基因和转录本被过滤掉,作为未表达的基因和转录本,在表达频率比较和图中不考虑(73,925个低数据位点,40,457个表达位点;94335份低数据,66163份表达)。我们决定对编码和ncrna应用同样严格的过滤器,即使这导致超过60%的非编码转录本的丢失,这些转录本通常以低于编码转录本的水平表达。不同基因和转录组的表达谱进行了调查和统计分析,如[46]:采用Kolmogorov-Smirnov非参数检验比较两种基因型之间的基因表达分布。在每次测试中,两条曲线之间观察到的最大垂直距离(D-obs)、测试的临界距离值(D-crit)P.- 值和用于其表达进行了比较报告基因的数目。当两个d-OBS> d-STAT和P.< 0.01表示表达式分布在统计学上不同。
基因边界的转置元素鉴定和注释
从RefGen ZmB73 repeatmask Assembly AGPv3中提取重复区域和TEs注释。这个冗余注释文件包含超过500万个屏蔽区域,范围从几个碱基对到几个千碱基长,所以在过滤了低复杂度区域(“dust”)和串联重复区域(“trf”)后,只有超过1 Kb的I类反转录元件注释和超过50 bp的II类DNA TEs被纳入注释位点的基因组分类。然后使用“intersectBed”工具识别转座子位于转录起始位点(−1 Kb TSS)上游1 Kb内、基因体中或转录终止位点下游1 Kb内(+1 Kb TTS)的位点[98.和自定义带有修改坐标的loci注释文件。经te插入的基因跨越TSS(−500/+500 TSS)以同样的方式被鉴定。附加文件中报道了基因如何与te相关分类的示意图7.
差异表达基因沿玉米基因组分布图如下:利用Bedtools“制造窗口”将基因组划分为100 Kbp的不重叠窗口。对于每个窗口,使用BEDTools的“intersectbed”和“groupby”功能计算基因总数和上调或下调基因数量[98.].每个窗口的基因含量除以对应染色体上的基因数量归一化,每个窗口的差异表达基因数量除以该窗口的基因含量。作为对照,将上调基因的分布与从表达基因列表中获得的880个随机基因数据集进行比较。更具体地说,bash函数“shuffle”获得了100组880个表达基因,每组不包含重复的条目。然后将每组基因的坐标与100 Kbp窗口相交,得到每个窗口的基因数量。然后在100次重复中计算每个窗口的平均基因数,以得到“随机”基因分布的统计表示。
差异表达基因与TE之间的关系通过使用regioneR R package v1.2.0进行排列试验评估[100.].将上调和下调基因分离,每组TE与基因组中其他基因的平均距离进行排列检验(N= 200),以“resampleRegions”作为随机函数,以“meanDistance”作为评价函数,将整套基因作为“universe”。为了有足够数量的基因(特别是下调基因)保证该分析的统计效力,使用Cuffdiff在前面描述的“all -test set”(log2FC > |1|, FDR < 0.05;额外的文件19).
此外,根据基因日志的分布情况,将上调和下调基因分为表达箱2FC值。对于每一组基因,我们创建了三个容器:1)log2FC小于第一个四分位数,2)log2FC包括在四分位范围和3)日志2FC高于第三个四分位数(附加文件19).如前所述,对每个箱子进行排列测试,以评估它们与测试仪器的距离。
为了检查周围的差异表达的基因的DNA甲基化,为的CpG,CHG和CHH甲基化值的数据集B73是从全基因组亚硫酸盐测序研究检索[62].利用BEDOPS将两个方向的基因坐标扩展到2Kbp (CHH上下文为1Kbp) [101.],并与甲基化数据一起导入为regioneR [100.)变量。如前所述,将调控错误的基因再次分为三个表达箱,并对每个箱进行排列测试(n = 200),以比较箱中基因相对于基因组其余部分的平均甲基化水平。使用“resamplregions”作为随机函数,“meanInRegions”作为评价函数,并给出完整的基因集作为“universe”。通过BEDTools获得了差异表达基因的甲基化水平图,将2Kbp上游和下游区域在100 bp窗口内进行了划分[98.并将它们与包含整个基因组CG/CHG/CHH甲基化值的文件进行交叉。最后,使用自定义脚本计算每个窗口的平均甲基化值。同样的程序被用于提取基因组中所有基因的平均甲基化值。
差异表达TE家族的鉴定
由于TE在玉米基因组和转录组中占非常高的比例,Pol iv介导的TE调控也被评估在特定TE家族的水平上。高质量的reads被映射到Bowtie2 [99.]针对1526的全长的TE共有序列(http://maizetedb.org/~maize/).使用“multiBamCov”工具获得的每个TE登录的惟一映射读的计数表[98.],与edgeR包一起使用[61用于微分表达式分析。56个log2倍变化率≥|2|和FDR调整的TE家族P.值≤0.01视为差异表达。
小RNA分析
玉米smallRNA位点以前确定和分析[23].简言之,高品质的sRNA读取从相同样品获得的(B73和RPd1时/ rmr6从受欢迎的植物,盐度和干旱和盐度应力的植物中的幼叶用于使用Shortstack版本3.3注释SRNA生成基因座[102.].多映射读取被分配到他们最有可能的位置上的概率方式,由在每个可能的多个位置计数的唯一映射读取引导[81.].
以这种方式注释的244,050个24 nt siRNA簇与基因的1kb侧边区域之间的重叠用“intersectBed”BEDTools功能计算[98.].所述“coverageBed”功能代替用于计算基因的侧翼区的每个单独的位置以产生侧翼属于不同亚基因组类别的基因的siRNA的区域位点的分布图24 nt的siRNA的位点的存在。
缩写
- RdDM:
-
RNA定向DNA甲基化
- 核:
-
小干扰rna
- TES:
-
转座的元素
参考文献
- 1。
Matzke Ma,Mosher Ra。RNA定向的DNA甲基化:一种增加复杂性的表观遗传途径。NAT Rev Genet。2014; 15(6):394-408。
- 2.
Pikaard CS, Haag JR, Pontes OM, bllevins T, Cocklin R.一个Pol IV和Pol v依赖的rna导向DNA甲基化的转录叉模型。Cold Spring Harb Symp Quant Biol. 2012; 77:205-12。
- 3.
Wierzbicki AT。长非编码RNA的转录后基因沉默的作用。CurrOp植物BIOL。2012; 15(5):517-22。
- 4.
张浩,朱家宽。RNA-directed DNA甲基化。植物学报。2011;14(2):142-7。
- 5.
Zilberman D,Cao X,Jacobsen SE。Argonaute4控制轨迹特异的siRNA积累和DNA和组蛋白甲基化。科学。2003; 299(5607):716-9。
- 6.
Argonaute蛋白:功能见解和新兴角色。中国科学(d辑:地球科学)2013;14(7):447-59
- 7。
作为植物基因组免疫系统的DNA甲基化。植物科学进展。2014;19(5):320-6。
- 8。
Herr AJ, Jensen MB, Dalmay T, Baulcombe DC。RNA聚合酶IV调控内源性DNA的沉默。科学。2005;308(5718):118 - 20。
- 9。
Onodera Y, Haag JR, Ream T, Costa Nunes P, Pontes O, Pikaard CS。植物核RNA聚合酶IV介导siRNA和DNA甲基化依赖的异染色质形成。细胞。2005;120(5):613 - 22所示。
- 10。
Kanno T,Huettel B,Mette MF,Aufsatz W,Jaligot E,Daxinger L,Kreil DP,Matzke M,Matzke AJ。RNA定向DNA甲基化所需的非典型RNA聚合酶亚基。NAT Genet。2005; 37(7):761-5。
- 11.
Pontier D, Yahubyan G, Vega D, Bulski A, sarez - vasquez J, Hakimi MA, lebers - mache S, Colot V, Lagrange T.在拟南芥中,转座子和高度重复序列的沉默需要两种不同的RNA聚合酶的协同作用。基因Dev。2005;19(17):2030 - 40。
- 12.
Xie Z, Johansen LK, Gustafson AM, Kasschau KD, Lellis AD, Zilberman D, Jacobsen SE, Carrington JC。植物小RNA通路的遗传和功能多样化。公共科学图书馆杂志。2004;2 (5):E104。
- 13.
Haag JR, Brower-Toland B, Krieger EK, Sidorenko L, Nicora CD, Norbeck AD, Irsigler A, LaRue H, Brzeski J, McGinnis K, Ivashuta S, Pasa-Tolic L, Chandler VL, Pikaard CS。玉米RNA聚合酶IV和V亚型通过替代催化亚基的功能多样化。细胞众议员2014;9(1):378 - 90。
- 14.
Haag JR,REAR TS,Marasco M,Nicora CD,Norbeck AD,Pasa-Tolic L,Pikaard CS。Pol IV,POL V和RDR2的体外转录活性POL IV和RDR2的偶联在植物RNA沉默中进行DSRNA合成。Mol细胞。2012; 48(5):811-8。
- 15.
Law JA, Vashisht AA, Wohlschlegel JA, Jacobsen SE。SHH1是DNA甲基化所需的同源结构域蛋白,RDR2、RDM4和染色质重塑因子与RNA聚合酶IV相关。PLoS Genet. 2011;7(7):e1002195。
- 16.
Blevins T,Podicheti R,米什拉V,马拉斯科男,汤H,Pikaard CS。波尔IV和的24点核苷酸的siRNA在拟南芥引导从头DNA甲基化RDR2依赖性前体的鉴定。网上生活。2015; 4https://doi.org/10.7554/eLife.09591.
- 17。
Zhai J, Bischof S, Wang H, Feng S, Lee TF, Teng C, Chen X, Park SY, Liu L, Gallego-Bartolome J, Liu W, Henderson IR, Meyers BC, Ausin I, Jacobsen SE。Pol iv依赖的siRNA生物发生的一前体一siRNA模型。细胞。2015;163(2):445 - 55。
- 18。
Mosher RA, Schwach F, Studholme D, Baulcombe DC。PolIVb影响rna导向的DNA甲基化,独立于其在siRNA生物发生中的作用。中国科学院院刊2008;105(8):3145-50。
- 19。
Wierzbicki AT, Haag JR, Pikaard CS。RNA聚合酶Pol IVb/Pol V的非编码转录介导了重叠和邻近基因的转录沉默。细胞。2008;135(4):635 - 48。
- 20.
Wierzbicki AT, Ream TS, Haag JR, Pikaard CS。RNA聚合酶V转录引导ARGONAUTE4到染色质。Nat麝猫。2009;41(5):630 - 4。
- 21.
法JA,杜Ĵ,海尔CJ,丰S,KRAJEWSKI K,帕兰卡AM,施特拉尔BD,帕特尔DJ,雅各布森SE。在RNA指导的DNA甲基化位点的聚合酶IV占用要求SHH1。自然。2013; 498(7454):385-9。
- 22。
张旭,韩德胜,陆超,王志强,张志强。RNA聚合酶IV在植物小RNA代谢中的作用。中国科学院院刊2007;104(11):4536-41。
- 23。
玉米小RNA位点的全基因组特性及其在维持抑制6-1 (rmr6-1)突变体和长期非生物胁迫中的调控植物杂志。2016;170(3):1535 - 48。
- 24.
Nobuta K, Lu C, Shrivastava R, Pillay M, De Paoli E, Accerbi M, Arteaga-Vazquez M, Sidorenko L, Jeong DH, Yen Y, Green PJ, Chandler VL, Meyers BC。玉米中内源性sirna的不同大小分布:来自mop1-1突变体的深度测序证据。中国科学(d辑:地球科学)2008;51(39):14958-63。
- 25.
JI先生,Madzima TF, Bader R, Kent MR, Zhang X, Stam M, McGinnis KM, Dawe RK。玉米基因座H3K9me2基因的接近DNA和相对耗竭进行rna定向DNA甲基化。植物细胞,2014,26(12):4903 - 17所示。
- 26.
Huang Y, Kendall T, Mosher RA。油菜中Pol iv依赖的siRNA产量减少。生物学(巴塞尔)。2013; 2(4): 1210 - 23所示。
- 27.
Erhard KF, Stonaker JL, Parkinson SE, Lim JP, Hale CJ, Hollick JB。RNA聚合酶IV在玉米副反应中的作用。科学。2009;323(5918):1201 - 5。
- 28.
帕金森SE,大SM,霍利克JB。玉米的性别决定和叶片背面的命运是由一个维持被抑制的表观遗传状态的因素决定的。Dev杂志。2007;308(2):462 - 73。
- 29.
拟南芥基因组计划。分析开花植物拟南芥的基因组序列。自然。2000; 408(6814):796-815。
- 30.
Schnable PS,器皿D,富尔顿RS,斯坦JC,魏F,帕斯捷尔纳克,梁C,张J,富尔顿L,坟墓助教,风骚女子P,雷伊广告,考特尼L, Kruchowski党卫军,汤姆林森C, C, Delehaunty K, Fronick C,考特尼B,摇滚SM,嘹亮的歌E, F Du,金正日K,雅培RM,棉花,征收,Marchetto P,奥乔亚K,杰克逊SM Gillam B,陈W,燕L, Higginbotham J,Cardenas M, Waligorski J, Applebaum E,菲尔普斯L,要求J,凯蒂K, T领主,Scimone,领主N,亨特J,王T, Ruppert J,沙N, K无赖,霍奇斯J, Ingenthron E, Cordes M, Kohlberg年代,Sgro J, Delgado B,米德K, Chinwalla,伦纳德,克劳斯K, Collura K, Kudrna D,库里J R, Angelova, Rajasekar年代,穆勒T, Lomeli R, Scara G, Ko,德莱尼K, Wissotski M,洛佩兹G,坎波斯D, Braidotti M,阿什利·E, Golser W,金正日H,李年代,林J, Dujmic Z,金正日W, Talag J, Zuccolo,风扇C,塞巴斯蒂安,克雷默M,明镜周刊L, L Nascimento, Zutavern T, B,米勒Ambroise C,穆勒,斯普纳W, Narechania,任L,魏年代,Kumari年代,丝腰带B,利维MJ,麦克马汉L,范布伦P,沃恩MW,应K,叶CT,Emrich SJ,贾Y, Kalyanaraman,夏朝美联社,Barbazuk WB, Baucom RS, Brutnell TP, Carpita数控,查C,贾JM, Deragon JM, Estill JC,傅Y, Jeddeloh是的,汉Y,李,李P, Lisch博士,刘,刘Z,内格尔DH,麦肯MC, SanMiguel P,迈尔斯,Nettleton D,阮J,彭宁BW, Ponnala L,施耐德KL,施瓦茨,沙玛,C,索德伦德施普林格NM, Sun Q, Wang H, Waterman M, Westerman R, Wolfgruber TK, Yang L, Yu Y, Zhang L, Zhou S, Zhu Q, Bennetzen JL, Dawe RK, Jiang J, Jiang N, Presting GG, Wessler SR, Aluru S, Martienssen RA, Clifton SW, McCombie WR, Wing RA, Wilson RK。B73玉米基因组:复杂性、多样性和动态。科学。2009;326(5956):1112 - 5。
- 31.
Baucom RS, Estill JC, Chaparro C, Upshaw N, Jogi A, Deragon JM, Westerman RP, Sanmiguel PJ, Bennetzen JL。B73玉米基因组中反转录因子的异常多样性、非随机分布和快速进化。公共科学图书馆麝猫。2009;5 (11):e1000732。
- 32。
李强,Eichten SR, Hermanson PJ, Zaunbrecher VM, Song J, Wendt J, Rosenbaum H, Madzima TF, Sloan AE, Huang J, Burgess DL, Richmond TA, McGinnis KM, Meeley RB, Danilevskaya ON, Vaughn MW, Kaeppler SM, Jeddeloh JA,施普林格NM。玉米甲基组的遗传扰动。植物细胞,2014,26(12):4602 - 16。
- 33。
李Q,绅士,Zynda G,Song J,Makarevitch I,Hirsch CD,Hirsch CN,Dawe RK,Madzima TF,McGinnis Km,Lisch D,Schmitz RJ,Vaughn MW,Springer NM。RNA定向的DNA甲基化在玉米基因组中的异铬酰脲和欧氏素素之间强制义。Proc Natl Acad Sci U S A. 2015; 112(47):14728-33。
- 34。
纪根,郭玲,Harkess AE,姚颖,张旭,Dawe RK。CHH岛:玉米近基因染色质调控中的DNA甲基化。基因组研究》2013;23(4):628 - 37。
- 35。
McClintock B.控制元素和基因。冷泉Harb Symp量子Biol。1956年; 21:197-216。
- 36.
转座元件与基因组的表观遗传调控。(1)中国科学(d辑:地球科学)2007;
- 37.
转座子对植物进化有多重要?中国科学(d辑:地球科学)2013;14(1):49-61。
- 38.
考利,奥基RJ。转座子重新连接并微调转录组。公共科学图书馆麝猫。2013;9 (1):e1003234。
- 39.
Makarevitch I, Waters AJ, West PT, Stitzer M, Hirsch CN, Ross-Ibarra J,施普林格NM。转座因子有助于玉米基因在非生物胁迫下的激活。公共科学图书馆麝猫。2015;11 (1):e1004915。
- 40.
通过转录组重注释和差异表达分析,研究胁迫诱导和表观遗传介导的玉米转录组调控。Sci众议员2016;6:30446。
- 41.
Erhard KF Jr, Talbot JE, Deans NC, AE MC, Hollick JB。RNA聚合酶IV对玉米初生转录的影响。遗传学。2015;199(4):1107 - 25所示。
- 42.
Madzima TF, Huang J, McGinnis KM。玉米中染色质结构和基因表达的变化与MOP1活性的丧失相关。表观遗传学。2014;9(7):1047 - 59。
- 43.
佳Y,Lisch DR,大津K,斯坎隆MJ,内特尔顿d,钳夹PS。RNA依赖的RNA的损失聚合酶2(RDR2)函数导致在转座子,基因的表达和广泛的意外变化,和24-nt的小RNA。Plos Genet。2009; 5(11):e1000737。
- 44.
Matzke MA, Kanno T, Matzke AJ。rna导向的DNA甲基化:开花植物复杂表观遗传途径的进化。植物学报2015;66:243-67。
- 45.
基于RNA-seq的转录解译基因调控的差异分析。生物科技Nat》。2013,(1):31日46-53。
- 46。
邓x, Hiatt JB, Nguyen DK, Ercan S, Sturgill D, Hillier LW, Schlesinger F, Davis CA, Reinke VJ, Gingeras TR, Shendure J, Waterston RH, Oliver B, Lieb JD, Disteche CM。哺乳动物,秀丽隐杆线虫和黑腹果蝇表达的x -连锁基因代偿性上调的证据。Nat麝猫。2011;43(12):1179 - 85。
- 47。
Blevins T, Pontvianne F, Cocklin R, Podicheti R, Chandrasekhara C, yereni S, Braun C, Lee B, Rusch D, Mockaitis K, Tang H, Pikaard CS。拟南芥表观遗传的两步过程。摩尔细胞。2014;54(1):30-42。
- 48.
Conesa A, Madrigal P, Tarazona S, Gomez-Cabrero D, Cervera A, McPherson A, Szczesniak MW, Gaffney DJ, Elo LL, Zhang X, Mortazavi A. RNA-seq数据分析的最佳实践调查。基因组医学杂志。2016;十七13 - 016 - 0881 - 8。
- 49.
Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR, Pimentel H, Salzberg SL, Rinn JL, Pachter L. TopHat和袖扣RNA-seq实验的差异基因和转录表达分析。Nat Protoc。2012;7(3):562 - 78。
- 50.
李斌,杜威出版社。RSEM:从有或没有参考基因组的RNA-Seq数据中准确的转录本定量。BMC生物信息学。2011;12:323 - 2105 - 12 - 323。
- 51.
Leng N, Dawson JA, Thomson JA, Ruotti V, Rissman AI, Smits BM, Haag JD, Gould MN, Stewart RM, Kendziorski C. EBSeq:用于RNA-seq实验推理的经验Bayes层次模型。生物信息学。2013;29(8):1035 - 43。
- 52.
霍利克·杰比,科米克尔·j·l,帕金森·SE。Rmr6在玉米中维持副变状态的减数分裂遗传。遗传学。2005;171(2):725 - 40。
- 53.
Danilevskaya ON,孟X, Selinger DA, Deschamps S, Hermon P, Vansant G, Gupta R, Ananiev EV, Muszynski MG。MADS-box基因ZMM4在玉米花诱导和花序发育中的作用。植物杂志。2008;147(4):2054 - 69。
- 54.
玉米基因ligulless 2编码一种参与建立叶片鞘边界的基本亮氨酸拉链蛋白。基因Dev。1998;12(2):208 - 18。
- 55.
玉米营养茎尖向生殖茎尖转化过程中叶舌缺失2基因的功能。植物j . 1999; 19(4): 489 - 95。
- 56。
Zhang X,刘S,Takano T.来自拟南芥,Atcysa和Atcysb的两种半胱氨酸蛋白酶抑制剂,增加了盐,干旱,氧化和耐寒性。植物mol biol。2008; 68(1-2):131-43。
- 57。
关键词:拟南芥,植物胱抑素,AtCYS1, AtCYS2,非生物胁迫,表达模式植物学报2010;29(8):905-15。
- 58。
Morales-Ruiz T, ortega - galsteo AP, Ponferrada-Marin MI, Martinez-Macias MI, Ariza RR, roldan - arjoa T. DEMETER and suppressor OF SILENCING 1编码5-甲基胞嘧啶DNA糖基酶。中国科学(d辑:地球科学)2006;103(18):6853-8。
- 59。
Penterman J, Zilberman D, Huh JH, Ballinger T, Henikoff S, Fischer RL。拟南芥基因组的DNA去甲基化。中国科学(d辑:地球科学)2007;104(16):6752-7。
- 60.
DNA去甲基化酶基因的甲基敏感表达可作为表观遗传变阻器。公共科学图书馆麝猫。2015;11 (3):e1005142。
- 61.
罗宾逊医学博士,麦卡锡DJ,史密斯GK。edgeR:用于数字基因表达数据差异表达分析的生物导体包。生物信息学,2010,26(1):139 - 40。
- 62.
Regulski M, Lu Z, Kendall J, Donoghue MT, Reinders J, Llaca V, Deschamps S, Smith A, Levy D, McCombie WR, Tingey S, Rafalski A, Hicks J, Ware D, Martienssen RA。玉米甲基体影响mRNA的剪接位点,并揭示由小RNA引导的广泛的paramutslike开关。基因组研究》2013;23(10):1651 - 62。
- 63.
Blanc G, Wolfe KH。从重复基因的年龄分布推断模式植物广泛存在的古多倍性。植物细胞。2004;16(7):1667 - 78。
- 64.
Swigonova Z,Lai J,MA J,Ramakrishna W,Llaca V,Bennetzen JL,杂乱J.近距离分裂高粱和玉米基因组祖细胞。Genome Res。2004; 14(10A):1916-23。
- 65.
钳夹JC,施普林格NM,通过基因组的主导地位和古老而又持续的基因丢失玉米subgenomes的弗里M.分化。国家科学院院刊美A. 2011; 108(10):4069-74。
- 66.
玉米的四倍性之后,一种短缺失机制优先从两个同源基因中的一个删除基因。公共科学图书馆杂志。2010;8 (6):e1000409。
- 67.
王雪峰,陈峰,王学军。多倍体基因组显性基因的起源、遗传和调控。中国科学(d辑:地球科学)2014;
- 68.
Freeling M,Lyons E,Pedersen B,Alam M,Ming R,Lisch D.拟南芥中的许多或大多数基因在订单黄铜的起源之后转换。Genome Res。2008; 18(12):1924-37。
- 69.
Barbaglia AM,Klusman KM,希金斯Ĵ,肖JR,汉娜LC,拉尔SK。通过Helitron转座子基因捕获改组玉米的转录。遗传学。2012; 190(3):965-75。
- 70.
Nuthikattu S, McCue AD, Panda K, Fultz D, DeFraia C, Thomas EN, Slotkin RK。活性转座元件的表观遗传沉默是由RDR6和21-22核苷酸小干扰rna触发的。植物杂志。2013;162(1):116 - 31所示。
- 71.
Panda K, Slotkin RK: rdr6导向的DNA甲基化途径启动转座元件沉默的机制。植物信号学报2013,8(8);https://doi.org/10.4161/psb.25206.6月5日。
- 72.
McCue AD, Panda K, Nuthikattu S, Choudury SG, Thomas EN, Slotkin RK。ARGONAUTE 6桥转座元件mrna衍生的sirna,以建立DNA甲基化。EMBO j . 2015; 34(1): 20 - 35。
- 73.
Lippman Z, Gendrel AV, Black M, Vaughn MW, Dedhia N, McCombie WR, Lavine K, Mittal V, May B, Kasschau KD, Carrington JC, Doerge RW, Colot V, Martienssen R.转座元件在异染色质和表观遗传控制中的作用。大自然。2004;430(6998):471 - 6。
- 74.
霍利斯特·JD,高特·BS。转座元件的表观遗传沉默:减少转座和对邻近基因表达的有害影响之间的权衡。基因组研究》2009;19(8):1419 - 28。
- 75.
张X,肇SH,加州A,Borevitz JO。使用全基因组瓦片阵列在拟南芥基因,后生和转录多态性的全局分析。Plos Genet。2008; 4(3):e1000032。
- 76.
关键词:拟南芥,rna,甲基化,Pol IVEMBO j . 2006; 25(12): 2828 - 36。
- 77.
在拟南芥中,染色质和siRNA通路共同维持小转位元件的DNA甲基化。基因组医学杂志。2005;6 (11):R90。
- 78.
钟X,海尔CJ,法律JA,约翰逊LM,冯S,涂某,雅各布森SE。DDR复杂功能有助于RNA的全球性协会聚合酶V至发起人和进化的年轻转座子。纳特结构分子生物学。2012; 19(9):870-5。
- 79.
李婷婷,李淑娟,陈旭,李淑娟。在拟南芥中,RNA聚合酶v依赖的小RNA来源于小的基因间位点,包括大多数的SINE重复序列。表观遗传学。2012;7(7):781 - 95。
- 80.
王X,Weigel D,史密斯LM。转座子变体及其对拟南芥基因表达的影响。Plos Genet。2013; 9(2):E1003255。
- 81。
约翰逊、杨家强、柯鲁杰、艾克斯特尔。改进多映射小rna的位置。G3(贝塞斯达)。2016; 6(7): 2103 - 11所示。
- 82。
在拟南芥低拷贝转基因位点RNA定向DNA甲基化中,RNA聚合酶II、IV和V的相互作用。acta botanica sinica(云南植物学报). 2013;42(1):85-96。
- 83.
Greaves IK, Groszmann M, Ying H, Taylor JM, Peacock WJ, Dennis ES。拟南芥杂种的反式染色体甲基化。中国科学院院刊2012;109(9):3570-5。
- 84.
李强,季龙,李强,宋杰,Vaughn MW, Schmitz RJ,施普林格NM。玉米基因组中H3K9me2的基因组分布和DNA甲基化。《公共科学图书馆•综合》。2014;9 (8):e105267。
- 85.
Eichten SR, Ellis NA, Makarevitch I, Yeh CT, Gent JI, Guo L, McGinnis KM, Zhang X, Schnable PS, Vaughn MW, Dawe RK,施普林格NM。异染色质的扩散仅限于特定家族的玉米反转录转座子。公共科学图书馆麝猫。2012;8 (12):e1003127。
- 86.
Chettoor AM, Givan SA, Cole RA, Coker CT, Unger-Wallace E, Vejlupkova Z, Vollbrecht E, Fowler JE, Evans MM.通过玉米配子体转录组的RNA-seq分析发现新的转录本和配子体功能。基因组医学杂志。2014;15(7):414-014-0414-2。
- 87.
Slotkin RK, Vaughn M, Borges F, Tanurdzic M, Becker JD, Feijo JA, Martienssen RA。花粉转座因子的表观遗传重编程和小RNA沉默。细胞。2009;136(3):461 - 72。
- 88.
Calarco JP, Borges F, Donoghue MT, Van Ex F, Jullien PE, Lopes T, Gardner R, Berger F, Feijo JA, Becker JD, Martienssen RA。花粉中DNA甲基化的重编程通过小RNA引导表观遗传。细胞。2012;151(1):194 - 205。
- 89.
何华,杨涛,吴伟,郑斌。花粉中小rna的研究。中国生命科学。2015;58(3):246-52。
- 90.
植物基因组进化过程中蛋白质结构域出现和排列的动力学和适应效益。生物医学进展。2012;4(3):316-29。
- 91.
Paschold A,Larson NB,Marcon C,Schnable JC,Yeh CT,Lanz C,Nettleton D,Piepho HP,Schnable PS,Hochsheger F.不合因基因驱动玉米杂交种中基因表达的高度动态互补。植物细胞。2014; 26(10):3939-48。
- 92.
弗里男,徐军,豪斯男,Lisch D.溶液到C值悖论和垃圾DNA的功能:基因组平衡假说。莫尔植物。2015; 8(6):899-910。
- 93.
Morari楼Meggio楼Lunardon A,Scudiero E,Forestan C,Farinati S,Varotto S.时间过程的生化,生理的,并在两个玉米品系干旱,盐度,和恢复的场模仿条件分子反应。前植物SCI。2015; 6:314。
- 94.
Martin M. Cutadapt从高通量测序读取中移除适配器序列。EMBnet杂志。2011;17(1):10 - 2。
- 95.
Del Fabbro C, Scalabrin S, Morgante M, Giorgi FM。对Illumina NGS数据分析中读取微调效果的广泛评估。《公共科学图书馆•综合》。2013;8 (12):e85024。
- 96.
Kim D, Pertea G, Trapnell C, Pimentel H, Kelley R, Salzberg SL. TopHat2:在插入、缺失和基因融合中转录组的精确比对。基因组医学杂志。2013;14 (4):r36 - 2013 - 14 - 4 - r36。
- 97.
Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, Marth G, Abecasis G, Durbin R. 1000基因组项目数据处理子组:序列比对/地图格式和SAMtools。生物信息学。2009;25(16):2078 - 9。
- 98.
昆兰AR, Hall IM。BEDTools:用于比较基因组特征的一套灵活的工具。生物信息学,2010,26(6):841 - 2。
- 99.
Langmead B, Salzberg SL.快速间隙读对齐与领结2。Nat方法。2012;9(4):357 - 9。
- 100。
Gel B, Diez-Villanueva A, Serra E, Buschbeck M, Peinado MA。Malinverni R: regioneR:用于基于排列试验的基因组区域关联分析的R/bioconductor包。生物信息学。2016;32(2):289 - 91。
- 101。
Neph S, Reynolds AP, Kuehn MS, Stamatoyannopoulos JA。使用BEDOPS操作基因组范围。方法:生物医学杂志2016;1418:267-81。
- 102。
Axtell乔丹。ShortStack:小RNA基因的全面注释和定量。RNA。2013; 19(6): 740 - 51。
确认
笔者想感谢教授杰伊B.霍利克,朱利奥Pavesi集团教授,文森佐·罗西博士和的Massimiliano劳里亚博士的手稿和有益的讨论的批判性阅读。我们还要感谢A. Garside的英语版本。
资金
本研究获得了欧盟委员会FP7项目KBBE 2009 226,477 -“AENEAS”:获得性环境表观遗传学进展:从拟南芥到玉米)和意大利CNR旗舰项目EPIGEN的资助。
数据和材料的可用性
支撑本文(RNA-SEQ数据,转录和注释RPKM值的所有注释的基因和转录物的不同样品中的摘要)的结论的数据集可保藏在地理位置与登录号GSE71046(http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE71046).
作者信息
从属关系
贡献
CF、SF和SV设计了实验。CF、SF和AL进行实验并采集样品。CF、RAC和WS分析RNA-Seq数据。CF和SV写的手稿,所有作者的贡献。所有作者阅读并批准了最终的手稿。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
附加文件1:
RNA-Seq读取每个样本和映射统计的摘要。每个分析样本的原始序列读、高质量读和映射读的数量。过滤掉10个以上不同位置(MAPQ = 0)的多映射读,并报告过滤后的库统计信息。R1和R2分别表示复制1和复制2。(XLSX 16 kb)
额外的文件2:
RPd1时/ rmr6突变导致基因组转录部分的增加。直方图总结了B73野生型和玉米基因组的RNA-Seq覆盖RPd1时/ rmr6突变体。在两个过滤的映射RNA-SEQ读数的阈值下,226,168,609 BP导致转录RPd1时/ rmr6与B73的213,466,972 bp相比,增加了6% (A),而单独考虑unique mapping reads (B),转录增加了5.4% (184,098,461 bp in)RPd1时/ rmr6, 174,661,954 bp)。在三次重复中进行了缩小和覆盖率计算,并报告了平均覆盖率。(TIFF 642 kb)
额外的文件3:
分析各种生长条件水平的基因组转录分数。表表总结了转录的基因组分数(至少两个映射读数)的增加RPd1时/ rmr6突变体与B73相比,独立计算每个个体样本。在单个RNA-Seq文库水平下采样后,考虑多映射reads (MAPQ≥1)或唯一映射reads (MAPQ = 50),计算转录的基因组比例。也报告了至少两个reads覆盖的基因组部分的平均值和标准偏差。(XLSX 13 kb)
额外的文件4:
注释基因和转录物上丰富过滤器的结果。本研究中使用的玉米转录物注释包括160,488个转录物,其在114,382个锁上[40],其最初基于袖手扣测试状态(参见方法)来排除未表达或过低表达的基因/转录物(大致不包括所有这些FPKM <1在所有分析的样本中)。(多克斯16 kb)
额外的文件5:
不同编码电位的转录本的丰度过滤结果。丰度滤波器(基于Cuffdiff测试状态;(见方法)用于筛选未表达或过低表达的转录本(大致排除所有分析样本中FPKM < 1的转录本)。(多克斯16 kb)
额外的文件6:
转录本在B73和RPd1时/ rmr6te相关的HC-TE转录本突变。据报道,基于Blastn结果,HC-TE转录本(>0 FPKM的转录本)在超级家族中细分的累积频率(见方法)。RLC - Copia和RLG - Gypsy I类反转录转座子的两种基因型的表达分布有统计学差异(P.经K-S检验< 0.01)。(TIFF 1163 kb)
额外的文件7:
基因如何根据te分类的示意图。“intersectBed”工具[98]鉴定了转座子位于转录起始位点上游1 Kb内的玉米基因(A;−1 Kb TSS)、基因体(B)或转录终止位点下游1 Kb (C;+ 1 Kb TTS)。当te插入不同的基因位点时,同一位点可被分为两类或三类。在−500/+500 bp范围内插入跨越TSS的TEs在A和b中以灰色表示(TIFF 71 kb)
额外的文件8:
插入到基因边界的te影响邻近基因的表达RPd1时/ rmr6突变体。在转录起始位点上游1 kb内插入te的基因中,>0 FPKM的基因的表达分布直方图(左)和累积频率(右)均有报道(−1 kb TSS;A),转录终止位点下游1kb内(+ 1kb TTS;体内基因B), (C)或没有TE插入(D)。三组基因与附近的TE插入基因表达的分布(包括只有基因FPKM > 0)在统计上两者之间的不同基因型(P < 0.01 Kolmogorov-Smirnov测试),高表达RPd1时/ rmr6相反,没有TE插入(内部或附近)的基因在基因型之间的基因表达分布相似。图(E)代替了作为两个基因型到最近TE距离的函数的平均基因表达水平。距离被划分为500 bp窗口,距离为0表示基因体中含有TE。标准误差显示。(TIFF 1251 kb)
额外的文件9:
b1基因超表达RPd1时/ rmr6突变的叶子。当booster1(b1,编码基本螺旋环螺旋蛋白的GrmZM2G172795)基因在所有进行的差异表达分析中产生高度上调,代表了表观遗传沉默释放的标志RPd1时/ rmr6与野生型相比。(多克斯16 kb)
额外的文件10:
RPd1时/ rmr6至少有三个四个独立分析的具体目标,通常去调节。(XLSX 108 KB)
附加文件11:
对导致这两种疾病失调的基因的总结RPd1时/ rmr6和rdr2 / mop1玉米突变体。维恩图表示两种玉米RdDM突变体中普遍上调和下调的基因。基因表达差异rdr2 / mop1突变体取自[42,43]。(TIFF 257 KB)
额外的文件12:
基因的列表和注释导致了两者的失调RPd1时/ rmr6和rdr2 / mop1玉米突变体。(docx 19 kb)
额外的文件13:
在差异表达的基因中鉴定的TE SuperfamiliesRPd1时/ rmr6突变体。归类为TES的上调基因的数量和相对百分比(LOCI与至少一个先前分类为HC-TE或PR-TE的转录物,参见方法),属于超科。本文还报道了每个TEs超家族在114 382个注释基因中的相对丰度。(多克斯18 kb)
额外的文件14:
TE家族差异转录在RPd1时/ rmr6突变体。差异表达TE家族(log2FC > |2|;FDR < 0.01);对于I类te,全长元素和部分片段的数量从[31]中得到。(多克斯22 kb)
额外的文件15:
Pol IV沉默位点沿玉米基因组分布。差异表达基因沿10条玉米染色体的分布曲线表明,玉米染色体具有优先共定位的特性RPd1时/ rmr6去抑制基因(左图)。染色体按100Kbp不重叠窗口划分,每个窗口的基因(相对于染色体总基因;蓝条)和过表达基因(相对于窗口基因含量)。黄条和红条分别表示在至少三到两个独立比较中共享的上调基因的窗口百分比。从40457个表达基因中随机选取880个基因作为对照,用绿色条形图(右图)表示。这两种分布在统计上有很大的不同(P.= 1 × 10−16通过Wilcoxon检验),随机基因沿基因组均匀分布(结果包括876个基因组100 Kb窗口,而包含上调基因的737个基因组)。(PDF 1141 kb)
其他文件16:
rpd1/rmr6突变体叶片中下调基因沿玉米基因组分布图。71的分布图RPd1时/ rmr6下调基因显示,它们包含在沿着玉米染色体均匀分布的67个独立的基因组窗口中。染色体按100Kbp不重叠窗口划分,每个窗口的基因(相对于染色体总基因;蓝色条)和下调基因(就窗口基因含量而言;红色条)。(TIFF 2873 kb)
其他文件17:
基因组浏览器的RNA-Seq读取映射在RPd1时/ rmr6misregulated基因簇。基因组浏览器(IGV -整合基因组浏览器;http://software.broadinstitute.org/software/igv/) B73(红色)及RPd1时/ rmr6-1突变体(蓝色)RNA测序读取(归一化至总的映射读取)在大的染色体区域的三个例子在DE-压抑RPd1时/ rmr6突变体。Pol IV的转录释放可能引起一个(A, C)或两个链(B)的兴趣,并引起富含te区域的几个基因的兴趣。报告总映射读(重复1和2)和特定链映射读(重复2)。(TIFF 1222 kb)
额外的文件18:
阐述了差异表达基因与睾酮酶相关性的排列试验的原始结果。使用regioneR R包的排列试验[100]显示,上调基因与TEs(平均距离:1550 bp)的距离显著接近于基因组中基因的平均距离(排列评估的平均距离:2017 bp;P.< 0.005),而下调基因导致的距离较远(2374 bp;P < 0.005)。(A)中的图是结合(B)和(C)中报道的单图而得到的,分别是上调和下调基因的独立分析。该分析的差异表达基因(log2FC > |1|, FDR < 0.05)由Cuffdiff从所有测序样本中获得。(TIFF 600 kb)
附加文件19:
RPd1时/ rmr6Cuffdiff获得的差异表达基因(log2FC > |1|, FDR < 0.05),从所有测序样本开始,根据其fold change variation细分到bins。(XLSX 357 kb)
额外的文件20:
差异表达基因侧翼区域的DNA甲基化概况。计算侧翼区域(CG、CHG和CHH)的甲基化水平(CG和CHG为2 Kb, CHH为1 Kb;参见差异表达基因的方法)。将基因按fold change variation分为bins,并将每个bins的甲基化水平与基因组中基因的平均值进行比较。(TIFF 1402 kb)
附加文件22:
亚基因组1和亚基因组2指定基因的表达RPd1时/ rmr6突变体和B73野生型植株。利用Cuffdiff从所有测序样品中获得了玉米亚基因组或非同基因中由单重子和重复子组成的差异表达基因模型(log2FC > |1|, FDR < 0.05)。(多克斯17 kb)
附加文件23:
siRNA位点分布图。亚基因组基因侧翼区域的siRNA位点覆盖图,进一步在每个亚基因组的同源基因和单拷贝基因之间进行了分裂,证实了隐性亚基因组2的基因在上游区域优先富集siRNA,保留的同源基因和单拷贝基因没有差异。(TIFF 395 kb)
附加文件24:
所有注释基因和转录本的归一化FPKM(每千碱基每百万映射片段)值矩阵。(XLSX 11865 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Forestan, C. Farinati, S. Aiese Cigliano, R.et al。玉米RNA PolIV影响邻近TE插入基因的表达,并对转录具有全基因组的抑制作用。BMC植物杂志17,161(2017)。https://doi.org/10.1186/s12870-017-1108-1
收到:
接受:
发表:
关键字
- Zea Mays.
- 转录组分析
- RdDM
- siRNAs
- 转座的元素