- 研究gydF4y2Ba
- 开放获取gydF4y2Ba
- 发布:gydF4y2Ba
面包小麦5B染色体的5S rDNA位点的基因组区域精细组织gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba17gydF4y2Ba文章编号:gydF4y2Ba183.gydF4y2Ba(gydF4y2Ba2017gydF4y2Ba)gydF4y2Ba
抽象的gydF4y2Ba
背景gydF4y2Ba
编码5S rRNA的多基因家族是大核糖体亚基中最重要的结构功能部分之一,是所有真核生物基因组的专有性组成部分。5S rDNA由于其结构结构的固有特性,如重复单元的串联排列和高的种间差异,长期以来一直是细胞学和系统发育研究的首选靶点。面包小麦基因组复杂的多倍体特性,gydF4y2Ba小麦gydF4y2Ba并且串联重复测序簇的技术上困难的任务意味着含有5S rRNA基因的扩展基因组区域的详细组织仍不清楚。尽管最近在小麦基因组测序中取得了进展。使用BAC克隆的焦点测序,在这项工作中,我们研究了两个不同的5秒rdna标记的面包小麦染色体的两个不同5s的组织。gydF4y2Ba
结果gydF4y2Ba
在5BS染色体特异性bac文库中鉴定了3个含5S rDNA的bac克隆gydF4y2Ba小麦gydF4y2Ba.利用焦磷酸测序和组装的结果,我们获得了6个包含5S rDNA的contigs,总长度为140,417 bp,以及两组独立的、位于5BS染色体上的基因组区域的5S rDNA序列(池)。这两个区域的特征是存在大约70-80个5S rDNA拷贝,但它们的结构结构完全不同。第一个区域包含高度分化的短型5S rDNA单元,被多次插入的转座元件破坏。第二个区域包含更保守的长型5S rDNA,组织成单个串联序列。利用特异于两种5S rDNA单位类型的探针进行FISH,发现多倍体小麦及其二倍体祖先染色体上的信号分布和强度存在差异。gydF4y2Ba
结论gydF4y2Ba
已经建立了在5BS染色体上的两种紧密地位的5BS rDNA标记的基因组区域的详细结构组织已经建立。这两个区域在组织5S RDNA和由转换元件组成的组织中不同,暗示这些区域的不同演化模式。gydF4y2Ba
背景gydF4y2Ba
真核生物中编码5S rRNA基因的多基因家族通常排列在一个或几个染色体上,与编码45S rRNA的基因分开[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba].5S RDNA的重复单元包含120bp编码区域和未转录的间隔序列(NTS)。前者在结构中高度保守,而NTS在长度和核苷酸序列中是多态性。在面包小麦的六倍体基因组中gydF4y2Ba小麦gydF4y2BaL.(2N = 6×= 42,BBAADD,A,D和B基因组的潜在供体是gydF4y2BaT. Uraartu.gydF4y2Ba,gydF4y2BaAe。tauschiigydF4y2Ba和gydF4y2BaAe。speltoidesgydF4y2Ba根据NTS的结构来区分两个主要的单位类:长约500 bp的长单位包含380 bp的NTS,短约400 bp的短单位包含280 bp的NTS [gydF4y2Ba2gydF4y2Ba,gydF4y2Ba4gydF4y2Ba].5S rDNA的短单位优先定位于同源群1(臂1AS, 1BS, 1DS)的染色体上,而长单位定位于同源群5(臂5AS, 5BS, 5DS) [gydF4y2Ba2gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba].基于5S RDNA NTS序列的三分特异性,区分了许多不同的单位类型,并分配给饲料中的常规单体(基因组类型)[gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba].gydF4y2Ba
5S rDNA组织成重复簇及其NTS变异使其成为细胞学和系统发育研究的流行工具。对于小麦及其亲缘,已经进行了一些研究:使用单个克隆的5S rDNA副本进行系统发育分析[gydF4y2Ba10gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,以及对5S rDNA染色体定位的多项细胞学研究[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba].非常兴趣的是与其二倍体祖先相比,对所有多利多倍性瘤的RRNA基因组织的研究。使用综合allopolyploid.gydF4y2Ba小子gydF4y2BaxgydF4y2BaAegilops.gydF4y2Ba,发现5S rDNA重组发生在异源多倍体形成后不久[gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba].这种重组在具有相似基因组结构的合成异源多倍体和天然小麦异源多倍体中都具有可复制性。gydF4y2Ba
尽管在总的基因组测序和对面包小麦品种中国春(IWGSC以RefSeq V1.0)的参考序列的第一个版本的外观进度(gydF4y2Bahttp://www.wheatgenome.org/gydF4y2Ba),一些串联重复阵列,包括5S rDNA,仍需要额外的分析[gydF4y2Ba22gydF4y2Ba].gydF4y2Ba
在这项工作中,我们描述了属于5BS染色体的两个包含5S rdna的基因组区域的详细结构。利用454/GS FLX平台、BAC-End-和IonTorrent测序技术研究了5S rDNA阵列及其邻近区域的组织结构。结合荧光原位杂交(FISH),这种分析使我们能够建立不同单位的5S rDNA及其周围“景观”的染色体位置。应用不同小麦多倍体及其二倍体前体中期染色体5S rDNA NTS序列FISH,研究所研究的5S rDNA位点从二倍体前体向六倍体小麦进化过程中染色体组织的变化。gydF4y2Ba
方法gydF4y2Ba
植物材料gydF4y2Ba
我们使用gydF4y2Ba小麦gydF4y2Ba(2n = 6× = 42, AABBDD), var. Chinese Spring;gydF4y2BaT. Timopheevii.gydF4y2Ba(2n = 4×= 28,AAGG)gydF4y2Ba,gydF4y2Bak - 38555;gydF4y2BaAe。speltoidesgydF4y2Ba(2n = 2× = SS), K-389;gydF4y2BaAe。tauschiigydF4y2Ba(2n = 2×= DD),К-1662;和gydF4y2BaT. Uraartu.gydF4y2Ba(2n = 2× = AA), IG45298*。这些材料来自俄罗斯n.i.a Vavilov全俄罗斯植物遗传资源研究所RAN (St Petersburg, Russia)和ICARDA (Syria)基因库的小麦种质资源,由细胞学和遗传学研究所保存。gydF4y2Ba
bac文库的筛选和单个bac克隆的DNA分离gydF4y2Ba
5BS特异性BAC-LIBRITERYgydF4y2Bat . aestivumgydF4y2Ba(43,776个Bac-Clones,平均插入尺寸122 kbp,15倍的覆盖率为280 mbp长度的染色体臂),从实验植物学研究所(奥洛姆,捷克共和国),由J.Doleëel教授提供。图书馆的副本在细胞学研究所和遗传学SB ras的维持在-80°C。对于BAC文库筛选,我们跑用引物特异于5S rDNA的编码序列的2D BAC-池的PCR分析(5SrDNA_F:5'-GAGAGTAGTACTAGGATGGG-3'; 5SrDNA_R:5'-GGAGTTCTGACGGGATCCGG-3')。在含有0.5μl细胞培养的20μl反应混合物中以0.5μl细胞培养物作为模板进行的,特异性前向和反向引物的0.25μm,2μlPCR缓冲液(65mM Tris-HCl,pH 8.9; 1.5mm MgClgydF4y2Ba2gydF4y2Ba;16毫米(NHgydF4y2Ba4gydF4y2Ba)gydF4y2Ba2gydF4y2Ba所以gydF4y2Ba4gydF4y2Ba;0.05% Tween 20), 0.2 mM的每个dNTP和1单位的gydF4y2BaTaqgydF4y2BaDNA聚合酶。94°C初始变性4 min后,94°C 30 s, 55°C 30 s, 72°C 30 s运行35个循环。PCR片段在1%琼脂糖凝胶上电泳分离。gydF4y2Ba
使用NucleoSpin 96 Flash试剂盒(machery - nagel, Germany)分离选定BAC克隆的DNA。gydF4y2Ba
Sanger bac端和IonTorrent测序gydF4y2Ba
选用通用M13反向(5 ' -CAGGAAACAGCTATGAC -3 ')和T7正向(5 ' -TAATACGACTCACTATAGGG-3 ')引物,使用BigDye3.1终结者试剂盒(Applied Biosystems, USA)获得选定bac克隆的bac端序列。每20 μl反应含有200 μg BAC-DNA、1.5 μl BigDye 3.1、0.25 pM的正向或反向引物、4 μl的5x缓冲液和去离子水。在95°C初步变性5分钟后,在95°C 30s, 55°C 15 s, 60°C 4分钟运行80个循环。使用乙醇沉淀反应产物,并在3730XL DNA分析仪(Perkin Elmer Cetus, USA)中分离。gydF4y2Ba
由5S rDNA标记的BAC克隆包含在属于染色体5Bs不同位置的134个BAC克隆中,并在离子转子平台(Thermo Fisher Scientific)上被共同称为一个样品。测序和组装在Nesterov等人中描述。[gydF4y2Ba23gydF4y2Ba].gydF4y2Ba
BAC克隆的焦磷酸测序和组装gydF4y2Ba
5BS染色体的bac克隆被测序为两个库。BAC库52包括6个克隆,包括5S rdna标记克隆TaaCsp5BS010O13和TaaCsp5BS025F09。10个5BS染色体重叠克隆(gydF4y2Bahttp://www.wheatgenome.orggydF4y2Ba/),包括BAC TAACSP5BS096G09,具有5S rDNA序列,为89个池。使用GS FLX / 454焦塞序列平台(Roche)捕获Bac池霰弹枪。克隆TaacSP5BS096G09的序列数据已提供(如CTG4)已提供(gydF4y2Bahttps://urgi.versailles.inra.fr/gb2/gbrowse/wheat_phys_5BS_v1/gydF4y2Ba,gydF4y2Bahttps://wheat-urgi.versailles.inra.fr/Seq-Repository/AssembliesgydF4y2Ba).gydF4y2Ba
对于两个游泳池,创建了一个随机BAC片段,范围为400到1000bp的尺寸。此外,对于两个池,构建了6-10 kBp的配对终端库。根据测序方法手册GS FLX +系列与钛L +试剂盒(Roche)进行DNA测序。读,属于gydF4y2Ba大肠杆菌gydF4y2Ba去除DH10B和pindigobac5载体。使用GS DeNovo Assembler V 2.9将选中的散弹读取和对端读取从头组装成contigs(散弹读取)和scaffold(对端读取)。gydF4y2Ba
选定bac克隆中5S rdna标记序列的鉴定和后续分析gydF4y2Ba
首先,我们搜索超过700 bp的454-contigs (shotgun reads)和带有5S rDNA标记的支架(paired-end reads),方法如下:(1)使用完整的5S rDNA单元序列(包括编码和非转录序列)搜索BLASTn;(2)利用得到的BES (bac端序列)进行BLASTn搜索[gydF4y2Ba24gydF4y2Ba].此外,为了验证和延伸一些contigs,我们使用了IonTorrent bac克隆测序数据和PCR产品序列获得的特定引物(附加文件gydF4y2Ba1gydF4y2Ba:表S1)。选取的6个含5S rdna的contigs和scaffold(以下命名为“5SrDNA_fragments”)通过在TREP (gydF4y2Bahttp://botserv2.uzh.ch/kelldata/trep-db/index.html.gydF4y2Ba)和NCBI非冗余核苷酸数据库(gydF4y2Bahttps://blast.ncbi.nlm.nih.gov/blast.cgi.gydF4y2Ba).5 s rDNA片段的核苷酸序列分别以登录号MF467437(5个无序片段)和MF467438 (pool_89)保存在GenBank中。gydF4y2Ba
用Primer3程序开发引物[gydF4y2Ba25gydF4y2Ba].为了测试Bac-Clone重叠程度,在各个BAC-DNA模板上设计并测试了3种基于型的多态性(ISBP)底漆对(附加文件gydF4y2Ba2gydF4y2Ba:图S1)。ISBP利用TE侧翼序列的知识,以pcr扩增横跨TE和侧翼序列连接处的片段[gydF4y2Ba26gydF4y2Ba].gydF4y2Ba
迄今为止,自主转座元件的ltr -逆转录转座子插入事件,我们分析了当两个ltr同时存在于元件结构时,两个ltr之间的核苷酸差异率。为了确定LTR边界,使用Blast2seq (gydF4y2Bahttps://blast.ncbi.nlm.nih.gov/blast.cgi.gydF4y2Ba).另外,考虑了每个LTR的开始和结束的特征基序,5'-TG-3'和5'-CA-3'的存在。每对LTR使用Mega4程序中的Clustalw算法对齐[gydF4y2Ba27gydF4y2Ba].使用Kimura双参数方法计算序列分歧[gydF4y2Ba28gydF4y2Ba],带有完全删除选项。为了将这一项转换为插入日期,我们使用了以下公式:gydF4y2BaTgydF4y2Ba=gydF4y2BaDgydF4y2Ba/2gydF4y2BargydF4y2Ba, 在哪里gydF4y2BaTgydF4y2Ba自插入以来经过的时间;gydF4y2BaDgydF4y2Ba,估计LTR散度;和gydF4y2BargydF4y2Ba,每年的每个网站的替代率[gydF4y2Ba29gydF4y2Ba].我们采用了1.3 × 10的替代率gydF4y2Ba−8gydF4y2Ba植物LTR反转录转座子每年每个位点的突变[gydF4y2Ba30.gydF4y2Ba].gydF4y2Ba
5S RDNA序列对准和聚类分析gydF4y2Ba
在contigs和scaffold中进行5S rDNA序列的搜索,在primary reads中使用BLASTn搜索5S rDNA编码序列作为查询。为了找到5S rDNA编码区之间的间隔序列,我们对所有包含两个或两个以上5S rDNA拷贝的reads进行了识别,并提取了位于5S rDNA拷贝之间的序列。分别对编码序列和间隔序列进行分析。用MUSCLE程序对5S rDNA间隔序列进行比对[gydF4y2Ba31gydF4y2Ba]包含在Mega 4软件中。使用成对缺失选项和Kimura-2参数模型来完成5S rDNA编码序列分歧的计算。5S rDNA编码序列和间隔物的聚类分析是用CD-BIT程序进行的[gydF4y2Ba32gydF4y2Ba].利用MEGA4程序,使用双缺失选项,bootstrap重复500次,获得了邻居连接系统发生树。gydF4y2Ba
二级5S结构分析gydF4y2Ba
二级结构建模在RNAFOLD Web服务器上使用在线工具(gydF4y2Bahttp://rna.tbi.univie.ac.atgydF4y2Ba/)。二次结构是基于最小自由能(MFE)计算,使用基于环路的能量模型和Zuker和Stiegler引入的动态规划算法[gydF4y2Ba33gydF4y2Ba].对两组5S rDNA (pool_52和pool_89分别代表短单元和长单元)构建二级结构的一致模型。程序设置如下:避免分离出的核苷酸;在任何情况下,都应该投票给螺旋两侧悬挂着的能量。gydF4y2Ba
荧光原位杂交(鱼)gydF4y2Ba
中期染色体制备、FISH和染色体鉴定方法参照Salina等[gydF4y2Ba34gydF4y2Ba]微小修改。每个探针中各自植物的分析的中源性的总数为15-30。对于鱼类,我们使用PCR扩增序列短路和LONG5(94bp和131bp)。通过PCR从Bac-Clones Taacsp5BS0101013和TaacSP5BS096G09扩增探针,PCR具有特异性引物短5S-F(5'-GCGTGCACTGGTGCGGGGGGAG-3')和SHORT5S-R(5'- GacgattgcacattGCTTGGC-3');Long5S-F(5'-GGAAAAAACCGTGTGTGTGCTGC -3')和LONG5S-R(5'-CTCactaccattacaAccGTTC-3')使用以下程序:35次循环在94°C,45°C,55°C,45℃,72°C 30秒。将引物设计成短5S和LONG5S类型的代表性间隔序列(通过阈值确定的阈值98%同一性),通过测序验证每种类型的单带PCR产物。对于短5S和LONG5S 5S rDNA序列(从454焦点测定)的可推测扩增区域计算的平均整体序列分歧,两种探针对应于0.8-0.9%。它们的完整长度的短路和LON5s探针之间的分歧水平为45%,其中62bp的小区域在探针之间具有84%的同一性,并且剩余序列没有身份。gydF4y2Ba
用生物素或石榴素标记PCR衍生的探针。用荧光素抗生物素蛋白D(矢量实验室,美国)检测生物素化探针。使用荧光素抗逃亡者(矢量实验室,美国)增强了杂交信号。用抗Digoxigenin-rhodamine(Fab碎片,Sigma-Aldrich,美国)的抗体检测到Digoxigenin标记的探针。将制剂嵌入Vectashield安装介质(载体实验室)中,含有0.5μg/ ml DAPI(4',6-二脒基-2-苯基吲哚,Sigma-Aldrich,美国)用于染色体染色。用Axioskop 2加(Zeiss)显微镜检查染色体,并用VC-44(PCO)CCD相机记录。gydF4y2Ba
为了识别携带信号的染色体,我们使用了探针pSc119.2 [gydF4y2Ba35gydF4y2Ba]和pas1 [gydF4y2Ba36gydF4y2Ba].gydF4y2Ba
这项工作在生物物体显微分析集体中心(新西伯利亚SB RAS)进行。gydF4y2Ba
结果gydF4y2Ba
5S RDNA标记的BAC克隆的鉴定,测序和组装gydF4y2Ba
从5BS特异性BAC文库中分离出含有三种5S rDNA的BAC-CLONES TAACSP5BS010O13,TAACSP5BS025F09和TAACSP5BS096G09gydF4y2Bat . aestivumgydF4y2Bavar。中国春天。如该方法中所述,进行这些BAC-克隆的焦磷酸盐和组装。表中介绍了排序和组装数据的摘要gydF4y2Ba1gydF4y2Ba.gydF4y2Ba
大致计算BAC池中的5S rRNA基因数。对于池52,将5S rDNA序列覆盖范围建立为3600,而COLIG覆盖率为454读数为42,从而评估为86.池89的5S rDNA序列覆盖率为1500,并且CONTIG覆盖率24,使5S RDNA拷贝数为63。gydF4y2Ba
为了增加池52的组装质量,执行配对结束454排序(附加文件gydF4y2Ba3.gydF4y2Ba:表S2)。将测序结果排列成11个长度为2159 ~ 164054 bp的支架。值得注意的是,单个支架的长度与bac克隆的长度相当。gydF4y2Ba
使用散弹枪和对端焦磷酸测序数据分析面包小麦5BS染色体上的5S rdna标记基因组片段(附加文件)gydF4y2Ba1gydF4y2Ba:表S1)。gydF4y2Ba
5BS染色体5S rdna标记序列的结构gydF4y2Ba
为了鉴定52池和89池中带有5S rdna标记的长基因组序列,我们使用大于700 bp的contigs作为查询,进行BLASTn搜索。结果,我们获得了6个含5S rDNA的contigs(以下简称“5S rDNA片段”),长度为2503 ~ 52840 bp(见表)gydF4y2Ba2gydF4y2Ba;附加文件gydF4y2Ba1gydF4y2Ba:表S1)。gydF4y2Ba
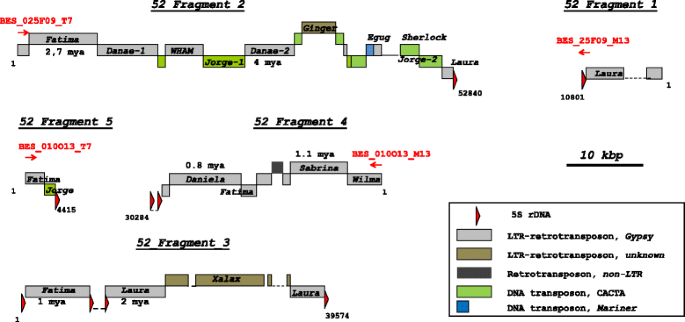
对于池52,鉴定了五个5S rDNA片段,仅发现池89的5S rdNA片段。使用Blastn针对妥塞可转换元素数据库(TREP)和NCBI核苷酸数据库进行注释5S rDNA片段(表gydF4y2Ba2gydF4y2Ba).所有的5S rDNA片段由转座因子(TE)和5S rDNA组成。未发现代表潜在基因或伪基因的独特序列。在5S rDNA片段中,TE以单个元件(或其组成部分)的形式分布在5S rDNA中,或以多个嵌套TE插入(主要是ltr反转录转座子)的形式出现。5S rDNA序列主要位于5S rDNA片段的末端,代表从92 bp到完整单元的部分序列。在5S rDNA片段3中,将5S rDNA引入片段序列内(图3)。gydF4y2Ba1gydF4y2Ba).仅在一种情况下(5S rDNA片段4)ltr逆转录转座子gydF4y2Ba法蒂玛gydF4y2Ba插入到5S rDNA编码序列中,而在其他所有情况下,te插入到间隔序列中。gydF4y2Ba
采用PCR方法检测52株bac克隆TaaCsp5BS010O13与TaaCsp5BS025F09的重叠性,并设计特异性引物进行TE插入(isbp标记)(附加文件)gydF4y2Ba2gydF4y2Ba:图S1)。我们发现,BAC克隆TaaCsp5BS025F09与TaaCsp5BS010O13完全重叠,明显较长。由于池52和池89由不同的重叠BAC克隆组组成,我们可以得出,池52和池89归因于5BS染色体上的不同基因组位置gydF4y2Bat . aestivumgydF4y2Ba.gydF4y2Ba
对52池和89池序列的分析表明它们的结构结构不同。在52池中,5S rDNA序列被多次TE插入打断,而在89池中,仅在一个侧翼区域观察到一次TE插入(表)gydF4y2Ba2gydF4y2Ba).池89的重复5S rDNA单元在组装过程中显然合并,表明5S rRNA基因的单一串联簇相邻的Nusif LTR-Retrotranposon。这是通过池89对5Bs Illumina假调子的对应(IWGSC Refseq V1.0,gydF4y2Bahttp://www.wheatgenome.orggydF4y2Ba/)。同时,52池的bac克隆TaaCsp5BS010O13和TaaCsp5BS025F09与Illumina数据没有对应关系。gydF4y2Ba
5S RDNA编码和间隔序列分析gydF4y2Ba
从池52和池89的霰弹枪454读取5S rdNA编码和间隔序列。将5S rdNA序列组装成NT的两个文件,以及分别分析的每个池的编码序列。gydF4y2Ba
在BAC池52和89的reads中分别鉴定出了1511和615个5S rDNA序列。为了进行分析,我们删除了发生次数少于5次的5S rDNA序列,因为这些序列可能是测序错误的结果。聚类分析得出pool_52的完整5S rDNA编码序列为30种,pool_89的完整5S rDNA编码序列为21种。序列已对齐(附加文件gydF4y2Ba4gydF4y2Ba),并计算平均总体序列发散度:池52的值为2.8%,池89-1.1%。因此,池52的编码序列比池89的编码序列具有更大的序列异质性。gydF4y2Ba
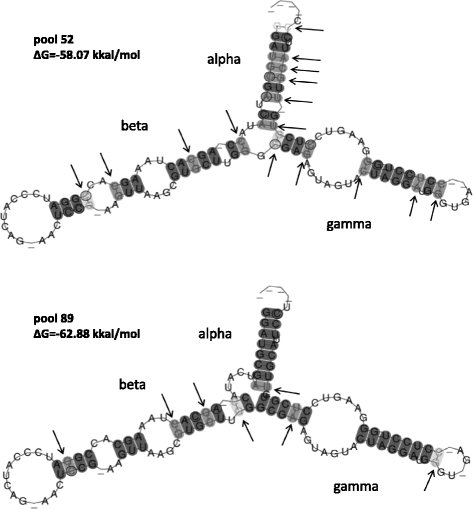
为了评估5BS染色体的不同基因位的RRNA基因的功能,我们使用了一个特殊程序,该特殊程序预测了每个RNA序列的共有二次结构,并伴随着其热力学稳定性的份额(参见方法)。实际上,两个游泳池都产生了由三个功能域组成的相同分子形状(图。gydF4y2Ba2gydF4y2Ba).89库的5S RNA的热力学稳定性略高于52库,尽管在这两种情况下,它与功能基因的典型特征(~ 50千卡/ mol)相比相当高[gydF4y2Ba37gydF4y2Ba].然而,两个游泳池在不相容的对数量(池52和7的15个用于池89)的数量中有显着不同。换句话说,池52中的基因序列大致两倍于折叠后产生具有未配对核苷酸的RNA结构。gydF4y2Ba
推测为5S rRNA二级结构。箭头表示在排列的某些序列中不能形成碱基对。5S rRNA的结构域遵循[gydF4y2Ba65.gydF4y2Ba]gydF4y2Ba
池52和池89的序列在NTS类型的结构中不同。在Pool 89中,我们发现27个完整的NTS序列。进行368bp和低序列异质性的平均序列,以及部分间隔序列进行聚类分析(附加文件gydF4y2Ba5gydF4y2Ba).在98%的序列一致性阈值下获得三个聚类。为了对齐,我们取了这三个集群的代表性序列(附加文件gydF4y2Ba6gydF4y2Ba).所有池89 NTS序列都归因于LONGS1类型(由BAUM和Bailey指定[gydF4y2Ba10gydF4y2Ba]).gydF4y2Ba
在池52中,我们发现144个完整的NTS序列,其分为两个88和10个序列的两个主要簇(在98%同一性的阈值下),其平均间隔长度为295bp,以及39个少量簇,含有一至四个序列207到390 BP(附加文件gydF4y2Ba5gydF4y2Ba).来源于小簇的22个NTS有30 ~ 95 bp的大量插入,不可能是测序错误。gydF4y2Ba
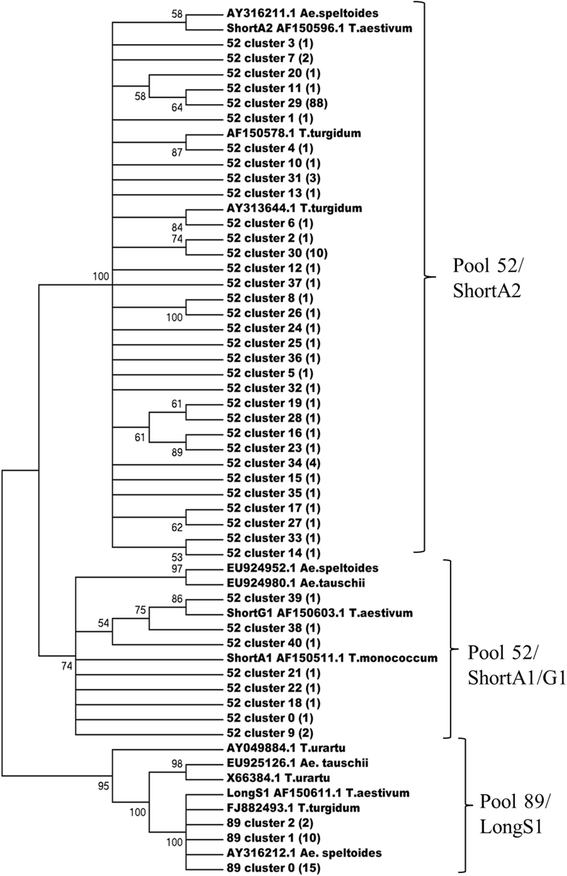
邻居连接树的构建表明,池52(133)中的大多数NTS序列对应于ShortA2型(与Baum和Bailey的分类一致[gydF4y2Ba10gydF4y2Ba]),而8个小NTS簇更接近于ShortA1和ShortG1单元。89号库的27条NTS序列均为LongS1型(图1)。gydF4y2Ba3.gydF4y2Ba).gydF4y2Ba
从池52和池89测序的5S rDNA的代表性簇的邻近的文献簇。使用CLUSTALW对5S rDNA间隔核苷酸序列的多次对准构建系统发育树(邻接方法,成对删除选项,500引导repactate).对应分支显示Bootstrap支持超过50%。括号中统一的间隔序列数量在括号中表示。还显示了使用的Shorta1,ShortG1,Shorta2和Longs1单元类型[gydF4y2Ba10gydF4y2Ba].根据是否存在相应的5S rDNA类型,将分支机构指定为ShortA1、ShortA2和LongS1gydF4y2Ba
与树枝序列之间的成对缺失选项的总体平均序列分歧是在短片2和Shorta1之间的21%,短达1和LONGS1之间的24%,29%之间,短达2和LONGS1之间的29%。在树枝中,平均整体发散值为:Shorta2的8%,对于Longs1的Shorta1和12%的11%。代表性池52群集序列的对齐来自树的Shake2和Shorta1分支(附加文件gydF4y2Ba7gydF4y2Ba88个间隔序列的主要代表性聚类与ShortA2型完全相同;而来自ShortA1的两个小集群与ShortA1和ShortG1相比,差异为19%。gydF4y2Ba
检查池52的亲和力和池89nts序列向推定的小麦祖细胞,gydF4y2BaT. Uraartu.gydF4y2Ba,gydF4y2BaAE Spottoides.gydF4y2Ba和gydF4y2BaAe。tauschiigydF4y2Ba,我们首先以89_cluster_0和52_cluster_29为查询序列执行GenBank BLAST搜索。在二倍体的5S rDNA序列中gydF4y2BaAe。speltoidesgydF4y2Ba序列AY316211.1是一个与ShortA2型高度同源的单一序列(同一性99%),而其余29gydF4y2BaAe。speltoidesgydF4y2Ba来自Genbank的序列仅显示了这种类型的75-76%的身份。与Shorta2类型类似的同源程度是5S RDNA的特征gydF4y2BaT. Uraartu.gydF4y2Ba,gydF4y2Bat . monococcumgydF4y2Ba和gydF4y2BaAe。tauschiigydF4y2Ba.gydF4y2Ba
Longs1型(池89)对来自二倍体的5s RDNA的相应序列具有更大的亲和力:对于gydF4y2BaAe。speltoidesgydF4y2Ba5S rDNA有18个GenBank序列,同源性在91 ~ 99%之间gydF4y2BaAe。Tauschii -gydF4y2Ba19条序列同源性为82-87%,30条序列同源性为82-84%gydF4y2BaT. Uraartu.gydF4y2Ba和gydF4y2Bat . monococcumgydF4y2Ba.值得注意的是,LongS1和ShortA2对来自四倍体的大量5S rDNA序列具有较高的同源性gydF4y2Bat . turgidumgydF4y2Ba.我们添加了最佳匹配的Genbank 5s RDNA序列gydF4y2BaT. Uraartu.gydF4y2Ba,gydF4y2Bat . monococcumgydF4y2Ba,gydF4y2BaAe。speltoidesgydF4y2Ba,gydF4y2BaAe。tauschiigydF4y2Ba和gydF4y2Bat . turgidumgydF4y2Ba到系统发育树(图。gydF4y2Ba3.gydF4y2Ba).gydF4y2Ba
5S rDNA NTS探针FISHgydF4y2Ba
对于FISH,我们使用两个探针代表52和89池的主要类型5S rDNA间隔序列。在此之前,完整的5S rDNA单元序列通常被用作FISH探针,因此Mukai和合作者[gydF4y2Ba7gydF4y2Ba采用黑麦5S rDNA探针pScT7, Badaeva等人[gydF4y2Ba15gydF4y2Ba,gydF4y2Ba38gydF4y2Ba]使用克隆pta794 [gydF4y2Ba39gydF4y2Ba].BAUM和COAUTHORS [gydF4y2Ba11gydF4y2Ba]评估是否有所不同gydF4y2BaTriticeaegydF4y2Ba归属于haploes的5S rDNA单元可作为FISH-探针,用于追踪多倍体小麦的二倍体起源,或在物种形成过程中染色体重塑。但是,由于不同单元之间,特别是在编码区域和NTS的5 ' -和3 ' -末端之间存在高度的交叉同源性,因此在这些情况下解释结果相当困难。因此,为了使FISH信号具有最大的特异性,我们设计了特异性引物,允许在NTS内扩增一个短序列作为探针。探针Short5S长94 bp,来自于主簇(52_cluster_29),探针Long5S长- 131 bp,来自于89池的5S rDNA的代表性副本(附加文件gydF4y2Ba6gydF4y2Ba,附加文件gydF4y2Ba7gydF4y2Ba).gydF4y2Ba
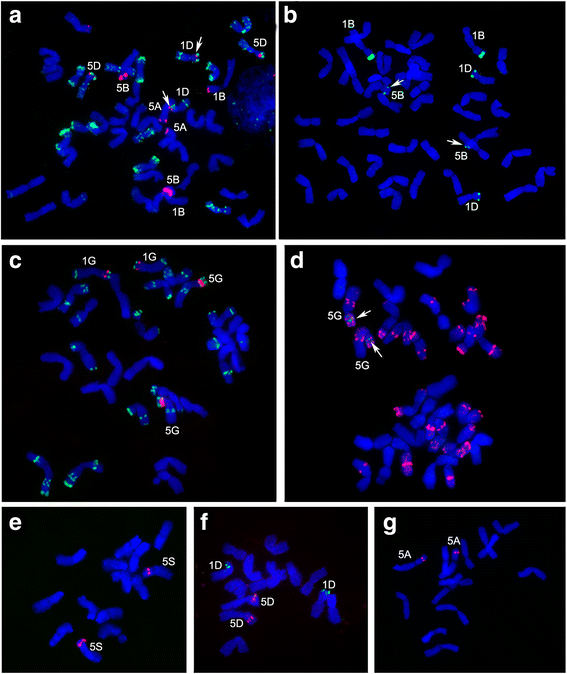
数字gydF4y2Ba4gydF4y2Ba表示两个探针在上的分布gydF4y2Bat . aestivumgydF4y2Ba染色体。探针Short5S和Long5S的染色体位置不同:Long5S在5BS、1BS、5DS、5AS、1DS染色体远端有10个杂交位点,信号最强的位置在5BS(表)gydF4y2Ba3.gydF4y2Ba).Short5s在1DS和1DS和5BS上显示了1BS和弱信号的最强杂交信号。四倍体小麦gydF4y2BaT. Timopheevii.gydF4y2Ba在染色体5GS上也有强大的Long5s和弱的短5S信号,并在染色体5as上弱的Long5s信号。短5s和long5s的鱼类到二倍体物种的染色体gydF4y2BaT. Uraartu.gydF4y2Ba,gydF4y2Bat . monococcumgydF4y2Ba, 和gydF4y2BaAe。speltoidesgydF4y2Ba未发现Short5S杂交位点gydF4y2Ba.Ae。tauschiigydF4y2Ba有5S rDNA单元的两个位点,Short5S在1DS上,Long5S在5DS上。值得注意的是,在多倍体小麦中,短5s单元的定位位点比长5s少,但在大多数情况下,染色体上的定位位点接近长5s。拷贝数变异或5S rDNA单位差异水平可能是导致某一染色体座短5S和长5S信号强度差异的原因。本研究显示,5B染色体的Short5S和Long5S信号水平的差异与Short5S相对于Long5S的发散程度较高有关(图5)。gydF4y2Ba2gydF4y2Ba),尽管这些基因在5BS位点的拷贝数大致相同(84和75,表gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
鱼到有丝分裂中期染色体。gydF4y2Baa、bgydF4y2Bat . aestivumgydF4y2Ba中文简历(gydF4y2Bac, dgydF4y2Ba)gydF4y2BaT. Timopheevii.gydF4y2Ba,(gydF4y2BaegydF4y2Ba)gydF4y2BaAe。speltoidesgydF4y2Ba,(gydF4y2BafgydF4y2Ba)gydF4y2BaAe。tauschiigydF4y2Ba, 和 (gydF4y2BaggydF4y2Ba)gydF4y2BaT. Uraartu.gydF4y2Ba.探针组合为:(a) Long5S(红色)和pAs1(绿色),(b) Short5S(绿色),(c) Long5S(红色)和pSc119.2(绿色),d) Short5S(绿色)和pSc119.2(红色),(e, f, g) Long5S(红色)和Short5S(绿色)。箭头表示信号强度较弱的地点gydF4y2Ba
讨论gydF4y2Ba
根据Nieto Feliner等人回顾的“协同进化”模型[gydF4y2Ba40gydF4y2Ba, rRNA基因的突变会迅速扩散到整个阵列,导致rDNA单位的基因组内同质性。rRNA基因在不同阵列中的保守和协同进化已经在一些研究中得到证实[gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba].另一方面,积累的数据也显示了种内和种间rDNA位点的位置和数量的变化[gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba].然而,由于与串联重复序列长簇测序相关的技术困难[gydF4y2Ba22gydF4y2Ba,关于单独rDNA位点的结构组织的信息很少。由于植物基因组的多倍体特性和重复成分的高发生率,在植物中获取这些信息要比在动物中困难得多。gydF4y2Ba
如这里所示,5BS染色体gydF4y2Bat . aestivumgydF4y2Ba包含5S rDNA的两个区域,显示出不同类型的结构组织。第一个区域(池89)似乎有一个“经典”组织,形成一个串联重复的5S rDNA长单元的单一簇,在一边与代表移动元素的区域相邻。第二个区域(池52)由至少5簇5S rDNA短单元组成(图52)。gydF4y2Ba1gydF4y2Ba),被由移动元素组成的插入块打断。在5S rDNA本身的重复单元的区域之间也存在显著差异。每个区域都有自己的单元类型,例如,池89包含大约63个在编码和NTS部分都高度相似的单元,并与LongS1类型的单元相似[gydF4y2Ba10gydF4y2Ba].池52含有约86个单位的5S rDNA,其特征在于编码(总体平均分歧为2.8%)和NTS零件中的较高程度的异质性。池52中的大多数间隔物都是型短轴2,约8%的序列更接近型短件1。gydF4y2Ba
后一区域TE的高度饱和表明它们参与了从基因组不同位置到该区域的rDNA单位的转座。支持这一观点的是gydF4y2BaAegilops Spottoides.gydF4y2BaTausch。除en / SPM转座簇相结合,减少了5S rdNA位点在Meiosis期间出现[gydF4y2Ba17gydF4y2Ba].CACTA组的转座子元件有时包含类似于5S rDNA基因的序列[gydF4y2Ba47gydF4y2Ba].另一类丰富的移动元件,LTR反转录转座子,经常与rDNA簇,包括5S rDNA [gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba].gydF4y2Ba
可以提出几种机制来解释RDNA基因座中明显的变化:gydF4y2Ba
- 1.gydF4y2Ba
这些位点的变异性(缺失、扩增、易位)可能是同源重组、转换和不相等交叉的结果[gydF4y2Ba50gydF4y2Ba,gydF4y2Ba51gydF4y2Ba].在这种情况下,移动元素可以作为重组热点。转座子介导的干扰和染色体重组在异源四倍体rDNA重组中发挥了重要作用gydF4y2Ba答:suecicagydF4y2Ba[gydF4y2Ba52gydF4y2Ba].gydF4y2Ba
- 2.gydF4y2Ba
5S rRNA基因的增殖和它们整合到基因组的其他区域可能被一种类似于逆转录转位的机制所解释。这些基因的转录(RNA聚合酶III,启动子)与一些反转录因子(如SINE)使用相同的机制[gydF4y2Ba53.gydF4y2Ba].此外,一些作者发现了一种独特的逆压织,使用插入的5S rdNA采用的启动子进行自己的传播[gydF4y2Ba54.gydF4y2Ba].gydF4y2Ba
- 3.gydF4y2Ba
5S基因可能通过染色体外复制传播到基因组的新位置。rDNA的染色体外环已经在分化的类群中发现,如人类,gydF4y2Ba非洲爪蟾蜍光滑的gydF4y2Ba和一些植物[gydF4y2Ba55.gydF4y2Ba,gydF4y2Ba56.gydF4y2Ba,gydF4y2Ba57.gydF4y2Ba].gydF4y2Ba
为了进行5S rRNA基因的细胞学分析,我们使用了NTS中心部位的Long5S和Short5S探针(图)。gydF4y2Ba4gydF4y2Ba;表格gydF4y2Ba3.gydF4y2Ba).Long5S探针的定位与之前通过pScT7和pTa794探针获得的小麦二倍体和多倍体基因组中5S rDNA单元分布数据具有良好的相关性[gydF4y2Ba7gydF4y2Ba,gydF4y2Ba38gydF4y2Ba和Baum等[gydF4y2Ba11gydF4y2Ba].Short5S探头显示了非常明显的定位。最有趣的观察是在染色体上没有一个短5s的信号gydF4y2BaT. Uraartu.gydF4y2Ba和gydF4y2BaAe。speltoidesgydF4y2Ba(图。gydF4y2Ba4 e, ggydF4y2Ba),分别是A-和B-基因组最有可能的供体[gydF4y2Ba58.gydF4y2Ba,gydF4y2Ba59.gydF4y2Ba],它在1ds染色体上的存在gydF4y2BaAe。tauschiigydF4y2Ba(图。gydF4y2Ba4F.gydF4y2Ba), d基因组的供体。对于多倍体物种,在1BS, 1DS, 5BS, 5GS染色体上发现了长和短5S rDNA单元的共定位(图2)。gydF4y2Ba4模拟gydF4y2Ba).所研究的多倍体小麦Сhromosomes 5DS和5AS仅显示了Long5S探针的信号,但这些染色体是否存在Short5S单元还需要通过其他方法来验证。根据我们的结果,Short5S探针在面包小麦的1BS染色体上表现出强烈的信号,而在5BS -染色体上表现出微弱的信号,很可能对应于库52。这种二倍体和相应的多倍体中5S rDNA组织结构的变化,提示二倍体小麦祖细胞中同时含有短5S基因家族和长5S基因家族,但它们的丰度、多样性和染色体位置存在显著差异,尤其是短5S单元。因此,在异源多倍体化时,现代的二倍体可能与真正的基因组供体包含不同的5S rRNA基因谱和拷贝数。gydF4y2Ba
rDNA位点的变异通常通过多倍性和种间杂交增强[gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba60.gydF4y2Ba,gydF4y2Ba61.gydF4y2Ba,gydF4y2Ba62.gydF4y2Ba].然而,新合成的异源多倍体中并没有发现新的5S rDNA位点的出现或亲本位点的完全消失,只有定量变化可能与单独位点的5S rDNA拷贝数减少有关[gydF4y2Ba20.gydF4y2Ba].这些在多倍体形成的早期阶段建立起来的变化往往会在接下来的进化阶段持续下去。具有相似基因组结构的合成和天然异源多倍体的rDNA模式相似,这一事实支持了这一观点[gydF4y2Ba20.gydF4y2Ba].在这里,我们估计了5BB的研究区域中的移动元件插入的时间,并透露了这些事件发生了2.7-4.0 mya(百万年前)。在这方面,我们可以假设二倍体祖先的5S rdna网站gydF4y2Ba小子gydF4y2Ba在小麦多倍体进化过程中,不同位点的结构逐渐发生差异,包括NTS的缺失导致出现了小麦多倍体特异的短5S rDNA单元库。在多倍体小麦物种中,ShortA2家族的高丰度支持了这一假设,该家族构成了库52的大部分。gydF4y2Ba
这里研究的5S rRNA基因的两种池具有相同的共有二次RNA结构(图。gydF4y2Ba2gydF4y2Ba)但是,它们显示出不同的保护级别。由于基因序列中的突变,池52可能产生更多的非功能性RNA分子。与池89相比,池52的NTS序列的变化远高得多。因此,后者在较强的选择约束下或在跨域重组中具有降低的效率。这提出了关于各种RDNA基因座的演变率的原因的重要问题,以及其差异表达和转录调节的相关问题。gydF4y2Ba
众所周知,大多数rRNA基因在植物和动物基因组中是不活跃的,或仅在某些发育阶段才被激活[gydF4y2Ba63.gydF4y2Ba].基因的非活性副本可以按照中性进化机制进化,并随着重组事件、移动元件的插入等而逐渐被破坏,这是我们在这里观察到的强分化短单元位点。但是,为了区分表达副本和非表达副本,需要对单个副本的转录进行分析,由于编码5S rDNA区域的长度较短,同源性高,现阶段很难实现。gydF4y2Ba
因此,我们首先利用焦磷酸测序和计算机分析3个含5S rRNA基因的BAC克隆,在面包小麦5BS染色体上重建了含有这些基因的两个延伸区域,揭示了它们不同的结构结构模式。gydF4y2Ba
结论gydF4y2Ba
本文首次描述了面包小麦5BS染色体上含有5S rRNA基因的延伸区。对这些区域的测序和后续分析表明,它们的结构组织存在差异,其中一个区域呈现高度分化型组织,多个TE插入打断了5S rDNA阵列,而另一个区域则是单个单元簇。此外,这些区域在单位异质性的来源和水平上也有所不同:第一个区域包括短单位,它们的间隔是可变的,与ShortA1和ShortA2 haples有关,而第二个区域由LongS1 haples的均匀长单位组成。另一个有趣的发现是A-和B-二倍体前体的染色体短单位探针缺乏FISH信号gydF4y2Bat . aestivumgydF4y2Ba表明自多倍体小麦形成以来,这些单位的分化程度很高。gydF4y2Ba
缩写gydF4y2Ba
- BAC:gydF4y2Ba
-
细菌人工染色体gydF4y2Ba
- 贝斯:gydF4y2Ba
-
BAC-end序列gydF4y2Ba
- 爆破:gydF4y2Ba
-
基本的局部对齐搜索工具gydF4y2Ba
- DTC:gydF4y2Ba
-
CACTA DNA-transposongydF4y2Ba
- 鱼:gydF4y2Ba
-
荧光原位杂交gydF4y2Ba
- ISBP:gydF4y2Ba
-
基于插入大小的多态性gydF4y2Ba
- LTR:gydF4y2Ba
-
长末端重复gydF4y2Ba
- NTS:gydF4y2Ba
-
非转录的垫片gydF4y2Ba
- rDNA:gydF4y2Ba
-
核糖体DNAgydF4y2Ba
- RLG:gydF4y2Ba
-
吉普赛LTR-retrotransposongydF4y2Ba
- TE:gydF4y2Ba
-
转座因子gydF4y2Ba
参考gydF4y2Ba
- 1.gydF4y2Ba
真核生物中的重复基因。《生物化学学报》1980;9:727-64。gydF4y2Ba
- 2.gydF4y2Ba
Apples R,Gerlach Wl,Dennis Es,Swift H,孔雀WJ。谷物中核糖体RNA的DNA序列的分子和染色体组织。染色体瘤。1980; 78:293-311。gydF4y2Ba
- 3.gydF4y2Ba
Ellis TN, Lee D, Thomas CM, Simpson PR, Cleary WG, Newman MA, Burcham KWG。5S rRNA基因gydF4y2BapisumgydF4y2Ba:序列,长程和染色体组织。孟根,1988;14:33 - 42。gydF4y2Ba
- 4.gydF4y2Ba
DVORAK J,Zhang HB,Kota Rs,Lassner M.组织和5S核糖体RNA基因家族的组织和相关物种。基因组。1989; 32:1003-16。gydF4y2Ba
- 5.gydF4y2Ba
关键词:染色体配对基因,5SrRNA,基因定位gydF4y2Ba小麦gydF4y2Ba在染色体臂5Bp的自发缺失。美国基因杂志1986;28:266-71。gydF4y2Ba
- 6。gydF4y2Ba
Secale L中5S多基因家族的第二个位点:该家族两个谱系的序列差异。基因组。1989;32(3):457 - 67。gydF4y2Ba
- 7。gydF4y2Ba
Mukai Y, Endo TR, Gill BS。普通小麦5S rDNA多基因家族的物理定位。J在这里。1990;81:290-5。gydF4y2Ba
- 8。gydF4y2Ba
鲍姆BR,贝利LG。5S rRNA基因的分子多样性gydF4y2BaKengyilia alatavicagydF4y2Ba杨金龙,杨金龙。小麦科不同rDNA单元的潜在基因组分配。基因组。1997;40:215-28。gydF4y2Ba
- 9。gydF4y2Ba
鲍姆BR,约翰逊DA,贝利LG。小麦科5S rDNA单元的分析:将序列单元分配给单链酶的潜力。在:Jaradat AA,编辑。Triticeae III,科学出版社。新罕布什尔州:恩菲尔德;1998.p . 85 - 96。gydF4y2Ba
- 10.gydF4y2Ba
鲍姆BR,贝利LG。小麦和多倍体小麦祖细胞中5S rRNA基因序列变异及其传递的基因组构成信息gydF4y2Ba小子gydF4y2BaL.(Poaceae:Triticeae)。遗传资源作物Evol。2001; 48:35-51。gydF4y2Ba
- 11.gydF4y2Ba
5S rDNA单元的非转录间隔被分成单元类分配到haplomes -在栽培小麦和小麦祖先上的试验。基因组。2004;47:590-9。gydF4y2Ba
- 12.gydF4y2Ba
Allaby Rg,Brown Ta。网络分析提供了在Triticum和Aegilops中5S RDNA阵列的演变的见解。遗传学。2001; 157(3):1331-41。gydF4y2Ba
- 13.gydF4y2Ba
Baum Br,Edwards T,Johnson Da。二倍体中的系统发育关系gydF4y2BaAegilops.gydF4y2Ba从5S rRNA单位推断的物种。分子系统学学报2009;53:34-44。gydF4y2Ba
- 14.gydF4y2Ba
Baum Br,Edwards T,Johnson Da。基因组中重复序列类别的共同依赖性:5S rDNA家族的系统发育分析gydF4y2Ba大麦gydF4y2Ba(Triticeae:禾本科)。基因组。2010;53:180 - 202。gydF4y2Ba
- 15.gydF4y2Ba
Badaeva ED, Friebe B, Gill BS。山羊草的基因组分化。二倍体物种中5S和18S-26S核糖体RNA基因家族的物理定位基因组。1996;(6):1150 - 8。gydF4y2Ba
- 16。gydF4y2Ba
Badaeva ED, Friebe B, Gill BS。基因组分化gydF4y2BaAegilops。gydF4y2Ba1.二倍体物种染色体上高度重复DNA序列的分布。基因组。1996;(2):293 - 306。gydF4y2Ba
- 17。gydF4y2Ba
acta physica sinica, 2017, 36 (4): 569 - 572 . acta physica sinica, 2017, 36(4): 569 - 572。中国科学(d辑:地球科学)2004;101(41):14818-23。gydF4y2Ba
- 18。gydF4y2Ba
Baum BR, Edwards T, Mamuti M, Johnson DA。从核5S rDNA序列推断多倍体和二倍体羊蹄草属植物之间的系统发育关系(禾科:麦科)。55基因组。2012;(3):177 - 93。gydF4y2Ba
- 19。gydF4y2Ba
大鳞羊齿草边缘种群的染色体进化:原因和后果。安机器人。2013;111(4):531 - 8。gydF4y2Ba
- 20。gydF4y2Ba
Shcherban AB, Sergeeva EM, Badaeva ЕD, Salina EA.合成异源多倍体的5S rDNA变化分析Triticum×Aegilops。分子生物学(俄国人)。42 2008;(4): 604 - 11所示。gydF4y2Ba
- 21.gydF4y2Ba
Baum Br,Feldman M.在新形成的Alegilops和Triticum的新形成的Allopolyploids中消除5S DNA单位课程。基因组。2010; 53(6):430-8。gydF4y2Ba
- 22.gydF4y2Ba
Staśkováh,陈克斯,陈,春,佛罗里达州,Tulpováz,kubalákovám,Visendi P,Hayashi S,Luo M,Batley J,Edwards D,DoleëelJ,Šimkováh。伊索米诺·基因组映射的单个染色体支持物理映射和序列组装在复杂的植物基因组中。植物Biotechnol J. 2016; 14(7):1523-31。gydF4y2Ba
- 23.gydF4y2Ba
Nesterov MA, Afonnikov DA, Sergeeva EM, Miroshnichenko LA, Bragina MK, Bragin AO, Vasiliev GV, Salina EA.基于BAC测序数据的微卫星位点鉴定及其在软质小麦5B染色体上的物理定位。中国科学(d辑:地球科学)2016;6(8):825-37。gydF4y2Ba
- 24.gydF4y2Ba
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ。基本的本地木质素搜索工具。中国生物医学杂志。1990;215:403-10。gydF4y2Ba
- 25.gydF4y2Ba
Untergasser A, cutcuttache I, Koressaar T, Ye J, Faircloth BC, Remm M, Rozen SG。Primer3 -新的功能和接口。核酸学报2012;40(15):e115。gydF4y2Ba
- 26.gydF4y2Ba
Paux E, Faure S, Choulet F, Roger D, Gauthier V, Martinant JP, Sourdille P, Balfourier F, Le Paslier MC, Chauveau A, Cakir M, Gandon B, Feuillet C.插入位点多态性标记为小麦基因组饱和和标记辅助选择开辟了新的前景。植物生态学报;2010;8(2):196-210。gydF4y2Ba
- 27.gydF4y2Ba
Tamura K, Dudley J, Nei M, Kumar S. MEGA4:分子进化遗传学分析(MEGA)软件4.0。生物化学学报2007;24:1596-9。gydF4y2Ba
- 28。gydF4y2Ba
通过核苷酸序列的比较研究估计碱基取代进化速率的一种简单方法。J Mol Evol. 1980; 16:111-20。gydF4y2Ba
- 29。gydF4y2Ba
SanMiguel P, Gaut BS, Tikhonov A, Nakajima Y, Bennetzen JL。玉米基因间反转座子的古生物学。Nat麝猫。1998;20:43-5。gydF4y2Ba
- 30.gydF4y2Ba
MA J,Bennetzen JL。近期水稻核基因组的快速增长和分歧。Proc Natl Acad Sci U S A. 2004; 101:12404-10。gydF4y2Ba
- 31。gydF4y2Ba
Edgar Rc。肌肉:具有减少时间和空间复杂性的多序列对准方法。BMC生物信息学。2004; 5:113。gydF4y2Ba
- 32。gydF4y2Ba
富L,NIU B,Zhu Z,Wu S,Li W.CD-HIT:加速用于聚类下一代测序数据。生物信息学。2012年; 28(23):3150-2。gydF4y2Ba
- 33.gydF4y2Ba
基于热力学和辅助信息的大RNA序列的最优计算机折叠。核酸Res. 1981;9(1): 133-48。gydF4y2Ba
- 34.gydF4y2Ba
Salina EA, Lim YK, Badaeva ED, Shcherban AB, Adonina IG, Amosova AV, Samatadze TE, Vatolina TY, Zoshchuk SA, Leitch AR.系统发育重建gydF4y2BaAegilops.gydF4y2Ba小麦二倍体和衍生多倍体的串列重复序列的进化。基因组。2006;49(8):1023 - 35。gydF4y2Ba
- 35.gydF4y2Ba
Bedbrook JR, Jones J, O 'Dell M, Thompson RD, Flavell RB。端粒异染色质的分子描述gydF4y2Ba秘书长gydF4y2Ba物种。细胞。1980;19:545-60。gydF4y2Ba
- 36.gydF4y2Ba
Rayburn AL, Gill BS。d -基因组特异性重复DNA序列的分离gydF4y2BaAegilops squarrosa.gydF4y2Ba.Plant Mol Biol rep 1986; 4:102-9。gydF4y2Ba
- 37.gydF4y2Ba
Smirnov AV, Entelis NS, Krasheninnikov IA, Martin R, Tarassov IA。5S rRNA结构的特殊特征——它与大分子的相互作用和可能的功能。生物化学(Mosc)。2008; 73:1418-37。gydF4y2Ba
- 38.gydF4y2Ba
关键词:细胞遗传学,分子生物学,细胞遗传学gydF4y2Ba小麦属植物timopheeviigydF4y2Ba染色体为人类基因组进化提供了新的视角gydF4y2Bat . zhukovskyigydF4y2Ba.植物生态学报。2016;42(2):943 - 56。gydF4y2Ba
- 39.gydF4y2Ba
Gerlach WL,Dyer Ta。含有5S-RRNA基因的小麦核中的重复单元的序列组织。核酸RES。1980; 8:4851-65。gydF4y2Ba
- 40.gydF4y2Ba
Nieto Feliner G,Rossello Ja。多烯家族和职业重组的协调演变。在植物基因组多样性,Wien,Springer。2012; 1:171-94。gydF4y2Ba
- 41.gydF4y2Ba
多佛。分子驱动:物种进化的一种内聚模式。大自然。1982;299:111-7。gydF4y2Ba
- 42。gydF4y2Ba
Eickbush TH, Eickbush DG。精心安排的运动:核糖体RNA基因的进化。遗传学。2007;175:477 - 85。gydF4y2Ba
- 43。gydF4y2Ba
Dubcovsky J,Dvorak J.核糖体RNA多烯烯基因烯基因组。遗传学。1995年; 140:1367-77。gydF4y2Ba
- 44。gydF4y2Ba
Shishido R,Sano Y,Fukui K.核糖体DNA:水稻基因组中基因令保护的例外。Mol Gen Genet。2000; 263:586-91。gydF4y2Ba
- 45.gydF4y2Ba
李志刚,李志刚,李志刚,等。拟南芥DNA中异色重复序列的大规模多态性分析。BMC Plant Biol. 2007;7:44。gydF4y2Ba
- 46.gydF4y2Ba
Britton-Davidian J,Cazaux B,加泰罗尼亚J.核心组织区域(NORS)的染色体动力学在鼠标中:微型进化见解。遗传。2012; 108:68-74。gydF4y2Ba
- 47.gydF4y2Ba
麦类植物中CACTA转座子的研究。一个多样化的高复制重复元素家族。植物杂志。2003;132:52 - 63。gydF4y2Ba
- 48.gydF4y2Ba
Kalendar R, Tanskanen J, Chang W, Antonius K, Sela H, Peleg O, Schulman AH。Cassandra反转录转座子携带独立转录的5S rRNA。美国国家科学学院学报2008;105:5833-8。gydF4y2Ba
- 49.gydF4y2Ba
Cioffi MB, Martins C, Bertollo LA。赤藓鱼相关转座子和核糖体DNA的染色体扩散。对鱼类基因组变化和核进化的影响。BMC Evol Biol. 2010;10:271。gydF4y2Ba
- 50.gydF4y2Ba
林志强,李志强,李志强。烟草毛囊部分的分子细胞遗传学分析和系统发育研究。Chromosoma。2000;109:245-58。gydF4y2Ba
- 51.gydF4y2Ba
Kovarik A,Dadejova M,Lim K-Y,Chase MW,Clarkson JJ,Knapp S,Leitch AR。芽孢杆菌菌rDNA的演变 - rDNA均质化与表观遗传学之间的潜在联系。Ann Bot。2008; 101:815-23。gydF4y2Ba
- 52.gydF4y2Ba
Pontes O, Neves N, Silva M, Lewis MS, Madlung A, Comai L, Viegas W, Pikaard CS。染色体位点重排是对异源四倍体拟南芥基因组形成的快速反应。中国科学院院刊2004;101:18240-5。gydF4y2Ba
- 53.gydF4y2Ba
Paule Mr,White RJ。调查和概述:RNA聚合酶I和III的转录。核酸RES。2000; 28:1283-98。gydF4y2Ba
- 54。gydF4y2Ba
Kapitonov vv,jurka J.一种新颖的一类来自5s rrna的正弦元素。mol Biol Evol。2003; 20:694-702。gydF4y2Ba
- 55。gydF4y2Ba
Roomcade D,Dressler D,Wolfson J.核糖体RNA基因的扩增涉及滚动圆形中间体。Proc Natl Acad Sci U S A. 1973; 70:2926-30。gydF4y2Ba
- 56。gydF4y2Ba
人类细胞中卫星重复和5S核糖体DNA的染色体外环。暴徒的DNA。2010; 1:11。gydF4y2Ba
- 57.gydF4y2Ba
Navratilova A,Koblizkova A,Macas J.来自植物卫星重复的果实圆形DNA的调查。BMC植物BIOL。2008; 8:90。gydF4y2Ba
- 58.gydF4y2Ba
张海波,张海波。小麦多倍体的进化:基因组供体物种的鉴定。基因组。1993;36:21-31。gydF4y2Ba
- 59.gydF4y2Ba
Dvorak J,张赫伯。反复核苷酸序列的变异脱落在小麦B和G基因组的系统中的光。Proc Natl Acad SCI U S A. 1990; 87:9640-4。gydF4y2Ba
- 60。gydF4y2Ba
关键词:烟草,异源四倍体,18-5.8-26S rDNA,重复序列中国科学(d辑:地球科学)2004;gydF4y2Ba
- 61。gydF4y2Ba
Shcherban AB, Badaeva ED, Amosova AV, Adonina IG, Salina EA.合成异源四倍体沙龙羊盾草rDNA的遗传和表观遗传变化。Umbellulata。51基因组。2008;(4):261 - 71。gydF4y2Ba
- 62.gydF4y2Ba
郭旭,韩峰。小麦rDNA序列多倍体化的非对称表观遗传修饰和消除。植物细胞。2014;26:4311-27。gydF4y2Ba
- 63.gydF4y2Ba
Grummt I,Pikaard CS。RNA聚合酶I转录的表观遗传沉默。NAT Rev Mol Cell Biol。2003; 4:641-9。gydF4y2Ba
- 64.gydF4y2Ba
Wicker T, Sabot F, Hua-Van A, Bennetzen JL, Capy P, Chalhoub B, Flavell A, Leroy P, Morgante M, Panaud O, paaux E, SanMiguel P, Schulman AH。真核生物转座分子的统一分类系统。Nat Rev Genet, 2007; 8:973-82。gydF4y2Ba
- 65.gydF4y2Ba
Wicke S,Costa A,Munoz J,Quandt D.躁动5S:陆地植物中的核核糖瘤DNA的重新安排和演变。mol phylocyet evol。2011; 61:321-32。gydF4y2Ba
确认gydF4y2Ba
新西伯利亚SB RAS生物对象显微分析集体中心的工作得到了IC&G预算项目(0324-2016-0001)的支持。我们感谢SB RAS基因组学核心设施和基因组研究中心ICG SB RAS。利用“生物工程”核心研究设施的科学设备对BAC克隆进行焦磷酸测序。我们非常感谢Carly Schramm对手稿的评论和英语润色。gydF4y2Ba
资金gydF4y2Ba
本研究和出版成本得到俄罗斯科学基金会(gydF4y2Bahttp://rscf.rugydF4y2Ba/)补助№14-14-00161。gydF4y2Ba
可用性数据和材料gydF4y2Ba
本研究过程中产生或分析的所有数据均包含在本发表的文章及其补充信息文件中。5S rDNA片段核苷酸序列以MF467437和MF467438的登录号保存在NCBI GenBank数据库中。gydF4y2Ba
关于这个补充gydF4y2Ba
本文已作为gydF4y2BaBMC植物生物学gydF4y2Ba2017年第17卷增编1:PlantGen 2017节选文章。该补充的全部内容可在网上找到gydF4y2Ba//www.cinefiend.com/articles/supplements/volume-17- supplement1.gydF4y2Ba.gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
概念:EMS、ABS、EAS。EMS - DNA序列的注释和分析。参与bac克隆的pcr分析。IGA原位杂交。MAN - BAC克隆管理、AVM和ALR对BAC克隆进行焦磷酸测序。AVB对焦磷酸测序数据进行生物信息学分析,NVR对数据进行分析,参与研究协调。项目管理、监理:EAS。写作-初稿准备:EMS, ABS,写作-审查和编辑:EMS, ABS, NVR, EAS。所有作者阅读并批准了最终的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准和同意参与gydF4y2Ba
不适用。gydF4y2Ba
同意出版gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
提交人声明他们没有竞争利益。gydF4y2Ba
出版商的注意gydF4y2Ba
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。gydF4y2Ba
附加文件gydF4y2Ba
附加文件1:表S1。gydF4y2Ba
面包小麦5BS染色体上5S rdna标记基因组片段的描述。这些片段作为散弹枪454测序结果的contigs(用于池52和89)和一个支架,由对端454测序结果(pool_52)。与bac端序列具有完全同源性。此外,通过IonTorrent BAC-sequencing和PCR测序(J2f2 5 ' - AGGTGTTACCAGCTAGATCGATGTGACATC-3 '和010 L1 5 ' - agaggcccttatctattccagaattgctg -3 ')得到的数据进一步确认和延长了部分片段。(DOC 39 kb)gydF4y2Ba
附加文件2:图S1。gydF4y2Ba
PCR检测pool_52 (TaaCsp5BS025F09和TaaCsp5BS010O13) bac克隆重叠情况。红色箭头表示TaaCsp5BS025F09中两个片段的BAC端序列位置。将3对ISBP引物设计为TE插入物(Danae-1/Fatima: 5 ' - gacaaaaatggccaacatcc -3 '和5 ' - GACCCCCTAATCCAGGACTC-3 ';Fatima/Danae-1: 5 ' - tgtccccagcctcttgttac -3 ' and 5 ' - GTGAAGGTGCCAACGAACTC-3 ';5SrDNA/Laura: 5 ' - accctagttggtttcagagg -3 '和5 ' - TGGGTGCTCACGATTCAC-3 ')用黑色箭头表示。引物在单个BAC-DNA模板TaaCsp5BS025F09和TaaCsp5BS010O13上进行检测。每对引物的PCR扩增结果显示在相应的TE连接上,TaaCsp5BS025F09与TaaCsp5BS010O13重叠。多克斯(152 kb)gydF4y2Ba
附加文件3:表S2。gydF4y2Ba
从霰弹枪+配对读取池52的数据集。(docx 12 kb)gydF4y2Ba
额外的文件4:gydF4y2Ba
pool_52 5S rDNA编码序列(a)和pool_89 5S rDNA编码序列(b)的MUSCLE比对。每个有代表性的序列对应的读号表示。(多克斯20 kb)gydF4y2Ba
额外的文件5:gydF4y2Ba
用于Pool_89(A)和Pool_52(B)序列的5S RDNA的非转录间隔物的聚类分析。(docx 17 kb)gydF4y2Ba
附加文件6:gydF4y2Ba
pool_89和LongS1单位类型各簇中代表性5S rDNA间隔位点的肌肉比对[gydF4y2Ba10gydF4y2Ba].用括号表示的簇中间隔序列的数目。保存地点用星号标示。Long89_F和Long89_R引物的位置用于获得灰色指示的鱼探针。(docx 13 kb)gydF4y2Ba
附加文件7:gydF4y2Ba
代表性5S的肌肉对齐来自池中的每棵树早午间的rop_52的rdna痉挛,短动物组织,shorta2和shortg1类型的5s rdna单位[gydF4y2Ba10gydF4y2Ba].用括号表示的簇中间隔序列的数目。保存地点用星号标示。用于获取FISH探针的Short52_F和Short52_R引物的位置用灰色表示。(docx 13 kb)gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文根据创意公约署署署的条款分发了4.0国际许可证(gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)除非另有说明,否则适用于本文中提供的数据。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
瑟吉娃,e.m.,施切班,a.b.,阿多妮娜,I.G.gydF4y2Ba等等。gydF4y2Ba基因组区域的精细组织标记为面包小麦5B染色体的5S rDNA轨迹。gydF4y2BaBMC植物杂志gydF4y2Ba17,gydF4y2Ba183(2017)。https://doi.org/10.1186/s12870-017-1120-5gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-017-1120-5gydF4y2Ba
关键词gydF4y2Ba
- 5 s rDNAgydF4y2Ba
- bac克隆gydF4y2Ba
- 多倍体小麦gydF4y2Ba
- 5 b染色体gydF4y2Ba
- 鱼gydF4y2Ba