摘要
背景
在自然种群中,有大麦不同颜色(大麦L.)。大麦颗粒的颜色与果冻层,卵巢层和引理的不同颜料的积累直接相关。蓝色颗粒颜色是由于阿仑酮层中的花青素的积累,这取决于五个存在BLX未测序的基因(BLX1.,BLX3.和BLX4.基因聚类在染色体4HL和BLX2.和BLX5.在7HL)。由于花青素的健康益处,蓝色颗粒大麦可被视为膳食食品的来源。目前研究的目的是鉴定和表征大麦中的果酮层的花青素合成调节网络的组分。
结果
调节综合体MBW组分的候选基因(由转录因子组成米YB,b通过/ MYC和WD40),用于大麦糊粉中花青素的合成。这些基因被指定HVMYC2.4 (hl),HVMPC2.(4HL),和HvWD40(6 hl)。HVMYC2.仅在含菌细胞中表达。函数损失(帧移位)突变HVMYC2.与蓝粒大麦相比,没有颜色。不像aleurone-specificHVMYC2.,HVMPC2.基因在不同组织中表达;然而,与有色糊粉相比,在非有色糊粉中未检测到其活性,并且在其启动子区域发现了等位基因特异性突变。单拷贝基因HvWD40编码调控MBW复合物的所需组分,在着色和非彩色组织中表达了调节MBW复合物所需的组分,并且没有等位基因差异。HVMYC2.和HVMPC2.使用特定于等位基因的开发帽标记进行了遗传映射。HVMYC2.映射到SSR基因座之间的位置XGBS0875-4H(3.4 cM的远端)和XGBM1048-4H(3.4厘米的近端)与区域染色体染色体4HLBlx -群集被发现。在这个位置,一种花青素生物合成结构基因之一(hvf3'5'h.)也使用当前研究中开发的等位基因特异性帽标记进行映射。
结论
在大麦糊粉层参与花色素苷合成的基因进行鉴定和表征,包括调控复合MBW的部件,从该MYC编码基因(HVMYC2.)似乎是由糊粉彩色底层大麦的变化的主要因素。
背景
黄酮类化合物是由植物产生的天然生物活性化合物。黄酮类色素含有花青素,以其植物保护功能而闻名[1,2]和人类健康的好处[3.,4].
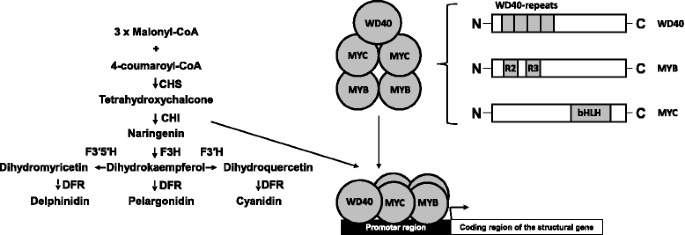
植物中的各种着色模式是通过种类的各种调控因素来实现,所述对类黄酮颜料的生物合成的各种调节因素。在MBW复合物的帮助下,借助于三种类型的转录因子组成,伴有类黄酮生物合成的活化。米YB,b通过/ MYC和WD40 [5,6,7](图。1).这些调节元件激活编码涉及类黄酮类化合物生物合成的酶的结构基因,提供色素的颜料积聚。
花青素生物合成的方案(从[42.])。MYB,MYC,WD40 - 转录因子。WD40重复,R2R3,BHLH - 它们的功能域。Chi - Chalcone-Flavanone异构酶;CHS - Chalcone合成酶;DFR - 二氢萘酚4-还原酶;F3H - 黄烷酮3-羟化酶;F3'H - 黄酮3'-羟化酶;F3'5'H - 黄酮类化合物3',5'-羟化酶
大麦(大麦L.)是一种重要的农作物。自然群体中,与不同类型的谷物着色的大麦植物中有描述。紫色,黄色和蓝色类型的色素沉着与类黄酮色素的在颗粒的不同层中的积累相关联。粮食果皮的紫色依赖于HvAnt2基因。位于2H (2HL)染色体长臂上,编码bHLH/MYC蛋白[8,9,10.],其与MYB因子一起(推定由编码HVMPC1./HvAnt1基因(7 h) (11.,12.)激活结构基因。大麦种皮中原花青素色素沉着的出现与原花青素的表达有关HvAnt28基因(3HL),编码myb型因子[13.,14.].糊粉层的蓝色取决于5个尚未测序的互补基因的存在:BLX1.,BLX2.,BLX3.,BLX4.和BLX5..这些基因中的三个(BLX1.,BLX3.,BLX4.)与彼此紧密相连,并映射到染色体4HL。BLX2.和BLX5.位于7HL染色体[15.].当互补显性等位基因出现时,糊粉颜色由蓝色变为粉红色(红色)BLX1.,BLX2.,BLX3.,和BLX5.Loci,而不是atBLX4.[15.].
MYB和MYC因子调节花青素合成糊粉层和他们的关系BLX基因尚不清楚。尚未确定和研究大麦MBW调节综合体的大麦MBW调节综合体的WD40分量。
在目前的研究中,我们检查了尚未注释的大麦序列的数据库,以查找和分析编码转录因子Myc,MyB和Wd40的基因,这些序列是与阿仑酮层中的花青素合成有关的,以及F3'5'H.- 一个假定的候选基因BLX4..
结果
在大麦甲酮中调节花青素合成的基因结构组织的鉴定,测序和研究
bhlh / myc.
我们找到了一个Myc-edoding的一份副本HvAnt2基因(GenBank:KX035100)位于染色体4H长臂(表1).所指定的基因的预测编码序列(1683bp长度)HVMYC2.股与70.8%的一致性HvAnt2。
完整的编码序列HVMYC2.获得了。它由八个外显子组成。第六和第7个外显子含有基因活化所需的保守Myc型BHLH结构域,通过与DNA和蛋白质结合(图。2).比较HVMYC2.BW和BA近代的序列英航基因等位基因态显示了几个同义单一核苷酸取代和一种单一核苷酸缺失(58bp上游BHLH编码基序),导致unoloured bw中的框架换档(图。3.).序列的序列HVMYC2.绘图人口(DOM和REC)的父母中的基因显示出非结块型官方父母中相同的功能突变。我们开发了一个针对功能的帽标记HvMyc2-BA并将其用于扩增具有蓝色或未着色糊粉颜色的不同大麦品种的DNA。的HvMyc2-BA存在于所有蓝色的麦芽虫和两个基因型中,含有非洛克葡萄酒(OWB_03和OWB_28)(附加文件1).的存在HvMyc2-BA等位基因在缺乏糊粉花青素合成在一些样品中可以通过在其它突变进行说明HVMYC2.或者是其他花青素生物合成基因的功能缺失突变。
MYB
全长基因序列(861碱基对)用69.0%同一性的编码MYB-R2R3基因HvAnt1(GenBank登录号:KP265977)中的溶液在4HL染色体上发现的(表1).该基因被命名为HVMPC2。的HVMPC2.基因在BW,BA,DOM和REC中重新测序。基因由两个外显子组成(图。2)和携带多酚生物合成所需的R2和R3基序。着色(BA,DOM)和未着色的(BW,REC)基因型的编码序列仅包含同义的单核苷酸取代。在启动子区域中揭示了一些契佩(附加档案2).
WD40.
爆炸分析后,使用玉米的WD40编码基因的序列(Genbank:Ay115485 [16.)和高粱(GenBank: JX122967 [17.]),我们发现大麦染色体上的正交基因(全长序列1071bp)6hl(与玉米相比,身份水平87.0%ZmPAC1高粱占87.8%SbTan1)(表1).该基因被命名为HvWD40.HvWD40在BW,BA,DOM和REC中重新测序。该基因缺乏内含子,并含有四种Wd40在C-末端的TRP-ASP(WD)双细胞残基重复(图。2).序列的四种基因型仅由同义单核苷酸取代彼此不同。
F3'5'H
我们在数据库中的搜索是基于序列F3'5'H双子叶植物的基因:葡萄(GenBank登录号:NM_001281235),枸杞(GenBank登录号:KC161969),大豆(基因库:EF174665),黑加仑(GenBank登录号:KC493688),桔梗(GenBank登录号:JQ403611),仙客来(GenBank登录号:GQ891056)(用于多个序列BLAST搜索被采取交叉验证)。具有最高同一性的序列(全长1689碱基对的长度)是在染色体4HL,其中发现BLX4.位于(表1).该基因被指定为hvf3'5'h..编码区hvf3'5'h.被分离成三个外显子(图。2).它含有细胞色素P450主题(Interpro:IPR002401)。BW,BA,DOM和REC仅由同义单核苷酸取代彼此不同。
所研究的基因的转录活性的RT-PCR分析
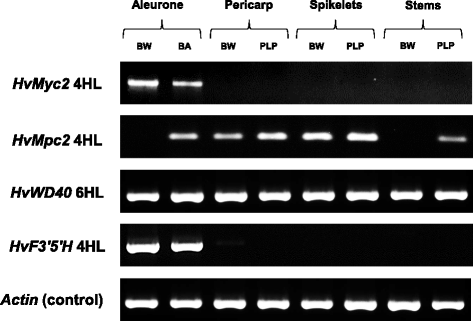
组织特异性表达HVMYC2.和hvf3'5'h.在糊粉层中观察到的基因,无论是在着色和未着色的近等基因系(图4).它们不转录在果皮或外稃(发育的小穗)和茎(有叶鞘)。不像aleurone-specificHVMYC2.和hvf3'5'h.,HVMPC2.基因在上述所有组织中均有表达;与有色糊粉相比,非有色糊粉中未检测到其活性。HvWD40在所有分析的cDNA样本中都观察到了转录本(图)。4).
分子映射
对于分子映射,我们选择了俄勒冈沃尔夫大麦(OWB)人口。对于花青素晶粒着色和用于花青素的粒度和占地带HvMyc2-BA特定的CAPS标记(表2).携带四十四条线HvMyc2-BA,而48线缺乏它。1(χ:匹配预期1所述的分离比率2= 0.17;P> 0.50)。我们使用过HvMyc2-BA基因分型数据以及可用的SSR和RFLP-LOCI数据,用于连接分析。基因HVMYC2.与SSR基因座密切相关XBMAC186-4H..同样,对于制图HVMPC2.和HvF3 5是什么,CAPS标记物被开发(基于DOM和REC之间的单核苷酸多态性)(图3.).分离率为HVMPC2.(44:48)和hvf3'5'h.(44:48)匹配预期的1:1(χ2= 0,17;p> 0.50HVMPC2.;χ2= 0,17;p> 0.50hvf3'5'h.).的hvf3'5'h.基因密切映射到XBMAC186-4H.和HVMYC2.,而且HVMPC2.SSR位点之间的图谱XGBS0875-4H(3.4 cM的远端)和XGBM1048-4H(3.4 cM的近端)(图5).
讨论
重复性是基因组中新基因的主要来源 - 约90%的真核生物中发现的基因是重复的结果[18.,19.].基因复制后转录因子的保留表明转录因子(TF)家族的扩展可能提供适应性益处[20.].MYB家族中属于最大量的类蛋白,和bHLH蛋白是第二最大的一类植物分类中的转录因子家族。蛋白的bHLH和MYB相互作用与蛋白质WD40,形成高动态复数MYB /的bHLH / WD40(MBW)。这些复合物调节各种细胞过程例如响应生物和非生物应力,形成根毛和毛状体,和酚类化合物包括类黄酮的合成[5,6,7].在大麦,并非所有涉及类黄酮生物合成的MYB和MYC因子都已识别和描述。缺少了关于大麦WD40编码基因的结构和功能组织和染色体定位的数据。在目前的研究中,我们在大麦籽粒的阿仑酮细胞中鉴定和表征MBW综合体的组分。
目前研究中确定的BHLH / MYC编码基因(HVMYC2.染色体4HL)似乎是的一个旁系同源副本HvAnt2紫色果皮颜色基因,位于2H染色体上。早期的一项研究旨在识别该基因的副本TaMyc1,调节小麦果皮中的花青素合成。该研究发现了11份同源群2和同源族第4族染色体[21.].结论是第一次重复的Myc基因发生在Triticeae部落的共同祖先中[21.].HVMYC2.,这可能源于重复的基因Myc基因在祖先的4号染色体上,很可能与一些Tamyc1.拷贝定位在染色体4Al,4bl和4dl上。小麦物种中,英航发现了蓝色的葡萄牙酮基因野生一粒小麦只(22.].面包小麦(t . aestivum)只有因染色体4Ag替换一个同源群4染色体,只能具有蓝色的葡萄酮颜色Agropyron[22.].在大麦中,已知染色体4h是对蓝色菌丝控制的负责[15.,23.]和携带的直向同源物英航基因T. Beeoticum.和Agropyron.我们的研究结果表明,Myc编码基因(HVMYC2.)是调控网络通过糊粉色大麦变化底层的主要成分。首先,我们观察到这种基因只在糊粉层中特异性表达,而其他候选基因没有表现出这样的特异性(图4).第二,我们发现了功能的缺失(阅读帧移位;无花果。3.)大多数大麦样品中的突变,具有unmolourta alleurone(附加文件1).此外,比较了HVMYC2.染色体4HL的精确位置BLX1./BLX3./BLX4.群集[15.,23.,24.它们是关于它们的分层化的结论。我们建议HVMYC2.候选基因是什么BLX1.或者BLX3.,而且hvf3'5'h.被证明是BLX4..众所周知,编码F3'5'H酶的基因中的功能突变失去缺乏花青素的“蓝色”分数[25.]和该突变BLX4.导致糊粉颜色由蓝色变为粉红色(红色)[15.].此外,hvf3'5'h.共定位是与BLX4.基因(图。5,[24.])。
就像HVMYC2.,hvf3'5'h.有糊粉特异性表达(图。4).结构基因的组织特异性表达可能表明存在潜在的复制副本。例如,类黄酮生物合成的两个平行副本F3 ^ h基因发生在一些细节植物物种中,一种副本在根中特别表达,第二份复制在植物的其他部分中[26.].我们认为大麦基因组应该包含其他基因F3'5'H.由于Delphinidin衍生累积而复制,不仅在阿列酮层中,而且还在例如终食层和茎中[8,12.].两者的存在F3'5'H.拷贝在植物物种中并不罕见。
MYB编码基因HVMPC2.在目前关于染色体4HL的研究中鉴定出似乎位于远端BLX1./BLX3./BLX4.集群。这个基因可能是一些小麦的直系同源tapl1.4BL和4DL染色体上的基因拷贝[27.].HVMPC2.在大麦植株的几个部位表达,对糊粉没有特异性。然而,产品的HVMPC2.基因被认为是糊粉中调控花青素合成BMW复合体的一部分,因为HVMPC2.编码一种myb样转录因子,与HvMpc1(调节紫色叶鞘中花青素的生物合成)高度相似[12.])和因为HVMPC2.表达相关用糊粉色(图4).地点HVMPC1.(7HS)是从不同的BLX2.和BLX5.hl 7日(BLX2.和BLX5.产品仍然未知;可能是编码花青素分子修饰所必需的酶)。因此,在大麦中编码了染月状的花青素调节基因(HVMPC1.和HVMPC2.)不要与BLX基因并与大麦的蓝色菌丝颜色的变异无关。植物不同部位的花青素色素沉着的变异通常是由MyB编码基因的突变引起的,而Myc编码伴侣更加保守。在小麦粥,茎,叶护套,叶片和花药中观察到在表型变异中的MyB编码基因变异性的主要作用[28.,29.]和大麦叶鞘[12.].在终食着色的情况下,MYB和MYC编码的调节基因都有助于表型变异[30.,31.]但在含有葡萄酮颜色的情况下,Myc-基因贡献至关重要(目前的研究)。
此外,我们还鉴定了wd40的编码基因HvWD40(6 hl)。单拷贝基因HvWD40编码监管MBW复杂的所需组件不断表达了彩色和非彩色的组织,并且没有等位基因差异。该类蛋白参与多种细胞过程的,这可能是为什么他们被高保守性中指定的原因[32.].WD40.其他植物物种(例如马铃薯)的基因有相似的表达谱,这与组织特异性或组织颜色的强度无关。
我们假设HvWD40一起HVMPC2.和HVMYC2.形成MBW调节综合合物,这对于激活Aleurone的结构花青素生物合成基因是必要的,同时HvAnt1和HvAnt2,它形成MBW复合物,这是治疗Pericarps中的花青素合成所必需的。
结论
鉴定参与大麦葡萄酮层中的花青素合成的基因,并表征,包括MBW调节综合体的组分,来自编码Myc编码基因(HVMYC2.)似乎是大麦糊粉颜色的主要因素底层的变化。
方法
植物材料
两个亲本系(DOM和REC)和92周的植物从大麦作图群体俄勒冈沃尔夫大麦(OWB)33.],三鲍曼近等基因系(近等基因系)(表3.),来自ICG系列“Genagro”(NovoSibirsk,俄罗斯)和来自IPK Genbank(Gaterslebendeclar,德国Gaterslebendeclar,德国)的九种品种被筛选出来HvMyc2-BA等位基因(附加文件1).三个近等基因系被利用基因表达分析。DOM,REC,BW和BA也用于测序。将植物在温室ICG核心设施(新西伯利亚,俄罗斯),12小时的光周期下,在20〜25℃下生长。
基因鉴定和硅分析
同源序列的数据库检索,进行存放于IPK大麦BLAST服务器没有被标注大麦序列(http://weblast.ipk-gatersleben.de/barley_ibsc/)使用本服务器提供的软件[34.].使用Fgenesh +程序进行检测到的序列的注释[35.]和cDNA的测序证实。核苷酸和氨基酸序列的比对使用的MULTALIN V5.4.1程序[由36.].大麦基因HvAnt2(Genbank:KX035100)和HvAnt1(GenBank: KP265977)进行鉴定Myc样和Myb例如序列,分别。利用玉米进行wd40编码基因的筛选ZmPAC1(Genbank:Ay115485)和高粱SbTan1(Genbank:JX122967)基因。为了识别编码F3'5'H的基因,使用已知的搜索完成F3'5'H.双子叶植物物种的基因序列:VvF3的5是什么(Genbank:NM_001281235),lrf3'5'h.(Genbank:KC161969),GMW1(基因库:EF174665),RNF35H.(Genbank:KC493688),PgF3'5'H(基因库:JQ403611)cpf3'5'h.(GenBank登录号:GQ891056)。氨基酸序列使用的InterPro [预测37.].FGENESH+软件预测基因的外显子-内含子结构[35.]使用同源基因的多肽序列HvAnt2,HvAnt1,ZmPAC1和VvF3'5'H。
DNA和RNA提取,cDNA合成
总基因组DNA从以下的程序先前描述的植物的新鲜叶中提取[38.].果壳和糊粉用于RNA提取从早期阶段的面团成熟(BBCH代码83),用于BW和BA线晶粒scalpeled。从果皮以及来自外稃的RNA(与显影小穗;在开花结束时收集; BBCH代码69)和茎(与叶鞘;在同一阶段收集的)中提取施加ZR植物RNA MiniPrep试剂™(ZYMO RESEARCH,USA)。从糊粉层的样品的RNA使用RNeasy Mini试剂盒(QIAGEN,德国)提取。所有分离RNA用无RNA酶的DNA酶组(QIAGEN,德国)处理。总RNA转化成单链cDNA在选自0.2微克使用的RevertAid第一链cDNA合成试剂盒(赛默飞世尔科技公司,美国)的总RNA的模板的20-μL反应。
PCR,限制性和序列分析
在20μLPCR中制备GDNA和cDNA的扩增。反应混合物含有50-100ng的基因组模板DNA,每种引物的1ng,每个DNTP的0.25mm,1×反应缓冲液(67mm Trishcl,pH 8.8; 2mM MgCl2;18毫米(NH4)2所以4;0.01%Tween 20)和1u Taq聚合酶。在94℃下在94℃下初始变性扩增DNA模板2分钟,35个循环在94℃下在94℃下运行1分钟,50-60℃,1分钟,72℃,0.5-2分钟,然后是最终的在72℃下延伸5分钟。使用Oligo7程序进行引物设计。PCR产物在琼脂糖凝胶上分离,用溴化乙锭染色并在UV光下可视化。使用DNA清洁试剂盒(Cytokine,St. Petersburg,俄罗斯)从琼脂糖凝胶中纯化扩增的片段。辨别不同的等位基因HVMYC2.,HVMPC2.和hvf3'5'h.位点,我们开发了CAPS标记。相应的PCR产物(表2)用限制性内切酶消化Bse1我,无形体病我和EcoR我分别分别分离2-5%高分辨率琼脂糖凝胶中的DNA片段(Radagene Co.,China)(附加档案3.).使用SB RAS基因组核心设施(新西伯利亚,俄罗斯)进行DNA测序。由一系列重叠扩增子(表的全长基因序列进行了重新构建1).所有获得的序列都沉积在Genbank(NCBI)中。
遗传映射
识别位点相对于俄勒冈沃尔夫大麦(OWBs)作图群体的RFLP和SSR位点被映射[33.,39.].基因HVMYC2.,hvf3'5'h.和HVMPC2.使用目前研究中开发的基因特异性帽标记映射。用MapMaker 2.0构建联系地图[40]使用Kosambi功能[41.].
参考资料
- 1。
黄酮类化合物在植物中的氧化作用:从生化特性到生理功能。植物科学进展。2007;12(1):29-36。doi:10.1016 / J.Plants.2006.11.006.
- 2。
Landi M,Tattini M,Gould Ks。花青素在植物环境相互作用中的多功能作用。Environ Exp Bot。2015; 199:4-17。https://doi.org/10.1016/j.envexpbot.2015.05.012.
- 3.
李d,王P,罗Y,赵敏,花青素和分子机制的陈F.健康益处:从最近十年的更新。CRIT REV FOOD SCI NUTR。2017年; 57:1729年至1741年。doi:10.1080 / 10408398.2015.1030064.
- 4.
优素福B,古尔K,迩AA,花青素辛格P.生益在粮食系统的应用潜力封装:审查。CRIT REV FOOD SCI NUTR。2016; 56(3):2223-30。doi:10.1080 / 10408398.2013.805316.
- 5。
Feller A, Machemer K, Braun EL, Grotewold E. MYB和bHLH植物转录因子的进化和比较分析。植物j . 2011; 66(1): 94 - 116。doi:10.1111 / j.1365-313X.2010.04459.x.
- 6。
徐W,Dubos C,Lepiniec L. myB-BHLH-WDR复合物对类黄酮生物合成的转录控制。趋势植物SCI。2015; 20(3):176-85。doi:10.1016 / j.tplants.2014.12.001.
- 7。
霍勒斯基娜,苏耶娃,葛德娃EI。小麦类黄酮生物合成基因。中国生物医学工程学报2015;5(3):268-78。doi:10.1134 / S2079059715030077.
- 8。
jende-strid b.大麦类黄酮类生物合成的遗传控制。秘密人。1993; 3(5):187-204。doi:10.1111 / j.1601-5223.1993.00187.x.
- 9。
Cockram J,White J,Zuluaga DL,史密斯D,Comadran J,Macaulay M,Luo Z,Kearsey MJ,Werner P,Harrap D,Tapsell C,Liu H,Hedley Pe,Stein N,Schulte D,Steuernagel B,Marshall DF,WTB T, Ramsay L, Mackay I, Balding DJ, AGOUEB Consortium, Waugh R, O’Sullivan DM. Genome-wide association mapping to candidate polymorphism resolution in the unsequenced barley genome. Proc Natl Acad Sci U S A. 2010;107(50):21611–6. doi:10.1073 / pnas.1010179107.
- 10。
Shoeva OY,模拟H-P,Kukoeva电视,伯尔纳A,Khlestkina EK。在紫色和黑色谷物类黄酮生物合成途径的基因的调控大麦.Plos一个。2016; 11(10):E0163782。doi:10.1371 /杂志。pone.0163782.
- 11.
大麦候选基因的分离Ant1和小麦rc.不同营养组织中控制花青素色素沉着的基因。中国生物医学工程学报。2015;29(4):1287-98。doi:10.1007 / S00438-015-0991-0.
- 12.
Shoeva Oy,Kukoeva TV,BörnerA,khlestkina ek。大麦Ant1是玉米的同系物吗C1其产品是控制叶子鞘中的植物素合成的监管机械的一部分。植物品种。2015; 134(4):400-5。doi:10.1111 / PBR.12277.
- 13。
冰见E,山下Y,春山N,柳泽T,前川男,竹田S.Ant28原花青素合成基因编码R2R3 MYB结构域蛋白(Hvmyb10)高度影响大麦的籽粒休眠。Euphytica。2012;188(1):141 - 51。doi:10.1007 / s10681-011-0552-5.
- 14。
Garvin DF,Miller-Garvin JE,VICCARS EA,Jacobsen JV,Brown Ahd。鉴定链接的分子标记ANT28-484这种突变可以消除大麦种子中原花青素的产生。作物科学。1998;38(5):1250 - 5。doi:10.2135 / cropsci1998.0011183x003800050023x.
- 15.
大麦中新的颜色和互补色基因。Z Pflanzenzucht。1978年,81(1):40-53。
- 16。
CARY CC,Strahle JT,Selinger DA,Chandler VL。突变在苍白糊粉COLOR1调节基因玉米花青素途径具有不同的表型相对于功能相似透明Testa Glabra1.基因拟南芥蒂利亚纳.植物细胞。2004; 16(2):450-64。doi:10.1105 / TPC.018796.
- 17。
吴y,李x,xiang w,zhu c,林z,吴y,李j,潘德拉达岛s,ridder dd,白克,王ml,trick r,totso tt,yu J.存在高粱谷物中的单宁是由不同的自然等位基因调节Tannin1.中国科学(d辑:地球科学)2012;doi:10.1073 / pnas.1201700109.
- 18。
Ohno S.基因复制的进化。纽约:施普林格Verlag;1970.
- 19。
布伦纳SE,哈伯德T,Murzin A,在流感嗜血杆菌Chothia的C.基因重复。自然。1995; 378(6553):140。doi:10.1038 / 378140 a0.
- 20。
Lang D,Weiche B,Timmerhaus G,Richardt S,Riaño-PachónDM,CorrêaLGG,Reski R,Mueller-roeber B,Rensing SA。植物转录规例的基因组系统介绍比较分析:损失,增益,扩张和复杂性相关的时间表。基因组Biol Evol。2010; 2:488-503。doi:10.1093 / gbe / evq032.
- 21。
Strygina kv,khleskina ek。谷物中的Myc基因家族:转型在六倍面包小麦及其亲属的演变过程中。mol Biol。2017; 51(5):572-9。doi:10.7868 / S0026898417050032.
- 22。
Zeven AC。紫色和蓝色谷物的小麦:审查。Euphytica。1991年; 56(3):243-58。doi:10.1007 / BF00042371.
- 23。
Mullick DB,Faris DG,Brind VC,Acheson RM。花青素和大麦果皮和阿仑酮组织的花青素。可以j植物sci。1958年; 38(4):445-56。doi:10.4141 / CJPS58-071.
- 24。
O 'Sullivan H. gringenes:麦类和燕麦的基因组数据库。In: Edwards D, editor。植物生物信息学:方法与协议。分子生物学方法;2007.301 - 14页。doi:10.1007 / 978-1-59745-535-0_14.
- 25。
植物色素的遗传与生物化学。植物学报2006;57:761-80。doi:10.1146 / annurev.arplant.57.032905.105248.
- 26。
Khlestkina EK,Dobrovolskaya OB,Leonova IN,萨利纳EA。重复的多元化F3H在Triticeae的基因。J Mol Evol。2013; 76(4):261-6。doi:10.1007 / s00239-013-9554-3.
- 27。
Shin DH,Choi Mg,Kang CS,Park CS,Choi SB,Park Yi。小麦R2R3-MYB蛋白紫色植物1(TAPL1)用作花青素生物合成的正调节剂。Biochem Biophys Res Communce。2016; 469(3):686-91。doi:10.1016 / J.BBRC.2015.12.001.
- 28。
Khlestkina EK。基因决定小麦不同器官的着色。拉斯Ĵ遗传学申请RES。2013; 3(1):54-65。https://doi.org/10.1134/S2079059713010085
- 29。
Adzhieva VF, Babak OG, Shoeva OY, Kilchevsky AV, Khlestkina EK。植物果实和种子着色发育的分子遗传机制。中国生物医学工程学报2016;6(5):537-52。doi:10.1134 / S2079059716050026.
- 30.
Khlestkina Ek,RöderS,BörnerA.在四倍体小麦上控制花洒和Pericarp上的花青素色素沉着的映射基因(小麦属植物硬质L.)。Euphytica。2010; 171(1):65-9。doi:10.1007 / s10681-009-9994-4.
- 31。
Shoeva Oy,Gordeeva ei,khlestkina ek。小麦果皮中花青素合成的调节。分子。2014; 19(12):20266-79。doi:10.3390 /分子191220266.
- 32。
Neer EJ, Schmidt CJ, Nambudripad R, Smith TF。WD-repeat蛋白古老的调控蛋白家族。自然。1994;371(6495):297 - 300。doi:10.1038 / 371297A0.
- 33。
Costa JM, Corey A, Hayes PM, Jobet C, Kleinhofs A, Kopisch-Obusch A, Kramer SF, Kudrna D, Li M, Riera-Lizarazu O, Sato K, Szucs P, Toojinda T, Vales MI, Wolfe RI。俄勒冈州沃尔夫大麦的分子定位:表型多态双单倍体群体。应用电子学报。2001;103(2-3):415-24。doi:10.1007 / s001220100622.
- 34。
邓炜,尼克斯直流,学习GH,玛斯特B,穆林斯吉。Viroblast:一个独立的Blast Web服务器,用于灵活查询多个数据库和用户数据集。生物信息学。2007; 23(17):2334-6。doi:10.1093 /生物信息学/ btm331.
- 35。
索洛维约夫五,统计在真核基因预测方法。在:D. J.秃头,M.毕晓普,统计遗传学的C.坎宁斯编手册。John Wiley和Sons,2008年公司;doi:10.1002 / 9780470061619.ch4..
- 36。
与聚类Corpet F.多序列比对。核酸研究。1988; 6(22):10881-90。doi:10.1093 / NAR / 16.22.10881.
- 37。
Finn RD, Attwood TK, Babbitt PC, Bateman A, Bork P, Bridge AJ, Chang H-Y, Dosztányi Z, El-Gebali S, Fraser M, Gough J, Haft D, Holliday GL, Huang H, Huang X, Letunic I, Lopez R, Lu S, Marchler-Bauer A, Mi H, Mistry J, Natale DA, Necci M, Nuka G, Orengo CA, Park Y, Pesseat S, Piovesan D, Potter SC, Rawlings ND, Redaschi N, Richardson L, Rivoire C, Sangrador-Vegas A, Sigrist C, Sillitoe I, Smithers B, Squizzato S, Sutton G, Thanki N, Thomas PD, Tosatto SCE, Wu CH, Xenarios I, Yeh LS, Young SY, Mitchell AL. InterPro in 2017—beyond protein family and domain annotations.Nucl Acids Res. 2017;45(D1):D190-D199; doi:10.1093 / nar / gkw1107.
- 38。
Plaschke J,Ganal MW,Röders。使用微卫星标志物密切相关的面包小麦遗传多样性的检测。Al Appl Genet。1995; 91(6):1001-7。doi:10.1007 / BF00223912..
- 39。
关键词:大麦,基因组,基因组学,锚点,基因工程,基因工程引用本文:应用计算机学报。2007;114(5):823-39。doi:10.1007 / s00122-006-0480-2.
- 40。
MAPMAKER:一种交互式计算机软件包,用于构建实验群体和自然群体的初级遗传连锁图。基因组学。1987;1(2):174 - 81。doi:10.1016 / 0888-7543(87)90010-3.
- 41。
Kosambi DD。从重组值估计地图距离。安参。1944; 12(1):172-5。doi:10.1111 / j.1469-1809.1943.tb02321.x.
- 42。
Kuhn N,Guan L,Dai Zw,Wu Bh,Lauvergeat v,Gomèse,李世国,Godoy F,Arce-Johnson P,Delrot S. Berry成熟:最近听到了葡萄葡萄酒。J Exp Bot。2013; 65(16):4543-59。doi:10.1093 / jxb / ert395.
致谢
我们感谢Galina Generalova女士和Tatiana Kukoeva(ICG,Novosibirsk,俄罗斯)的技术援助。
资金
这项工作由俄罗斯科学基金会资助(№16-14-00086)。ICG项目0324-2016-0001支持ICG工厂生长核心设施中大麦植物的生长。
可用性数据和材料
本研究获得的序列可在NCBI查询:MF679149-MF679162。
关于这个补充剂
本文已作为一部分发布BMC植物生物学第17卷补充1,2017:来自Plantgen 2017的选定文章。补充剂的全部内容可在线获得//www.cinefiend.com/articles/supplements/volume-17-supplement-1.
作者信息
隶属关系
贡献
KVS执行的所有分子遗传实验,在硅片和统计分析进行,并参与起草的手稿。AB提供的植物材料,促成了数据的解释和审慎修改草稿。EKK促成了研究的构思和设计,数据的解读和批判性地修改稿件。所有作者均阅读并批准最终手稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
附加文件1:
的存在HvMyc2-BA大麦Bowman NILs等位基因(1-2)、定位群体亲本(3-4)、重组DH系(5-96)、IPK Genbank(97-98)和ICG收集GenAgro(99-107)的材料和品种。(PDF 149 kb)
附加文件2:
大麦的启动子区的多次对准HVMPC2.基因.(PDF 196 KB)
附加文件3:
对于大麦基因的分子映射作图群体(俄勒冈沃尔夫大麦,OWB)的基因分型的结果:A -HVMYC2.B -HVMPC2.C -hvf3'5'h..用限制性内切核酸酶消化映射群体的映射群体的扩增片段Bse1我,无形体病我和EcoR我分别(CAPS-analysis)。DOM和REC是亲本。(PDF 327 kb)
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
Strygina,K.V.,伯尔纳,A&Khlestkina,E.K.识别与大麦糊粉层花青素合成的调控网络组件的特性。BMC植物BIOL.17,184(2017)。https://doi.org/10.1186/s12870-017-1122-3
关键词
- bHLH
- 细胞色素P450
- 类黄酮合成
- 基因重复
- 大麦
- MBW
- MYB
- MYC
- 转录因子
- WD40.