摘要
背景
近年来,大豆已成为哈萨克斯坦最重要的油料作物之一。仅在过去十年(2006 - 2016年),大豆种植面积就从2006年的4.5万公顷(公顷)扩大到2016年的12万公顷。大豆扩张的总趋势是由东南部向平均气温较低、生长季节较短的东部和北部地区。这些新的大豆种植区在不同的大豆品种样品中对生产力水平的一般影响方面没有得到很好的检验。本研究收集了哈萨克斯坦三个独立地区的表型数据,并对整个大豆样品进行基因分型,以进行标记-性状关联(MTA)鉴定。
结果
在这项研究中,2015-2016年在哈萨克斯坦的北部、东部和东南部地区种植了代表世界5个不同地区的113种植物。据观察,北美品种在六次试验中有四次表现出最高产量,特别是在哈萨克斯坦北部两年内。采用6k SNP Illumina序列对整个样本进行基因分型。4442个snp被发现具有多态性,并被用于全基因组基因分型目的。获得的SNP标记数据和现场数据用于GWAS(全基因组关联研究)。在6个研究环境中的42个mta中,30个SNPs似乎非常显著。
结论
这项研究证实GWAS的分子标记物标记重要农艺性状的鉴定效率。共鉴定了30个与开花和成熟时间、株高、可育节数、单株种子和产量相关的SNP标记。42名总的MTA鉴定出32的物理位置与已知的类似的QTL的位置一致良好。这一结果表明揭示的mta对哈萨克斯坦大豆种植区的重要性。得到的结果将作为所需的必备形成和具体的育种计划实现有效的适应实现和哈萨克斯坦的三个不同区域增加了大豆的生产效率。
背景
大豆是全球最重要的油菜和蛋白质源作物之一。在哈萨克斯坦,大豆种植区已从2006年的45千公顷增加到2016年的120万人[1].这一大规模扩张是在KZ政府采取的作物多样化政策下进行的,最终目标是到2020年,每年商业大豆占用面积达到40万公顷。扩建的指定区域是KZ东南部(SEK)、东部(EK)和北部(NK)地区用于农业的土地[2,3.].
大豆是哈萨克斯坦的一种新作物。它说明了进行初步研究的必要性,如对在新环境中生长的多种大豆品种进行评估,以及它们作为特定常规育种目的受体种质的潜在用途。在这些目标区域生长的大豆株系中,开花基因的某些等位基因与产量之间的非随机关联的最新发现[1]是在这个方向一个良好的开端。在这项研究中,时间跨度开花在所有三个地区进行了评估在伴随变化E基因与产量表现。四种特定的等位基因组合E基因和开花和成熟时间相应的最佳范围被确定每个实验的网站[1].然而,要想在KZ成功开发和实施特定的常规大豆育种项目,还需要更全面的研究支持。
近年来,在大豆群落中产生了一些基因组导向工具,以促进世界范围内的育种计划(Soybase.org.).这些基因组工具包括组装Williams 82基因组序列(http://www.soybase.org/ sequenceintroduction .php), Affymetrix SoyChip注释[4],搜索引擎大豆和甘氨酸大豆序列(http://plants.ensembl.org/Glycine_max/Info/Index),来自Illumina的Soysnp50k Iselect Beanchip [5),等。研究发现,50,000 (50 K) SNP iSelect阵列在复杂农艺性状的QTL(数量性状位点)的遗传定位中尤其有用,包括非生物[6,7]和生物胁迫抗性[8]及种子品质[9,10].
全基因组关联研究(GWAS)被认为是最有前途的农艺性状QTL鉴定方法之一[11,12].在大豆中,GWAS实验表明MTA发现对环境因素有强烈的偏向[13,14,15].例如,从三个不同的GWAS研究中获得了与加拿大产量表现qtl鉴定相关的结果[16], 美国 [13,15,巴西[11]表现出不同的响应,并在基因组的不同部分鉴定了产量组成的qtl。据推测,为了为区域性的育种项目提供可靠的数据,需要单独的MTA实验。本研究的主要目的是在KZ三种不同环境下的大豆田间试验中鉴定非随机mta。这是基于关联作图方法在哈萨克斯坦首次尝试用相关qtl鉴定大豆产量。
结果
表型变异的收集和GEI模式
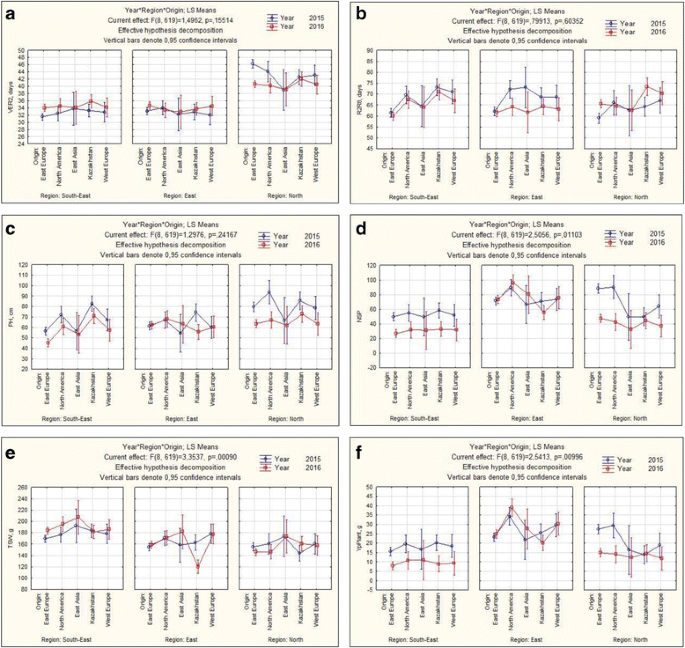
在两年多的研究中,113份供试材料的总体平均产量最高的是EK(26.53 + 1.09),其次是NK(18.19 + 0.72)和SEK(12.88 + 0.39)。对比评估2015-2016年3个地区5组大豆产地的开花时间(VER2)和种子成熟时间(R2R8)、株高(PH)、单株种子数(NSP)、千粒重(TSW)和单株产量(YP)的差异(图2)。1 f).
在大多数田间试验中,北美培育的大豆品种表现出最高的产量潜力。这一趋势在北部和东部地区尤其明显,2015年EK产量最高(图)。1 f).在东南和东部地区进行了两年的Pearson相关试验,结果表明,所有测定的产量成分(PH、NFN、NP、NSP和TSW)的产量均具有高度的可靠性。然而,在东南部地区,开花时间和种子成熟时间与产量显著相关,而在东部地区,产量与种子成熟时间高度相关(P<0.0001),但不与开花时间。在北面2年相关结果并不完全相同,如开花时间(P< 0.001)和株高(P<0.02)均显著相关的产量NK2015,但两者都性状无关在NK2016的产量的影响。收集的选育原点在六个时空环境(O×R个X Y)和地方生长(O X R)(表的表现出与选择的产量构成一个显著相互作用1).
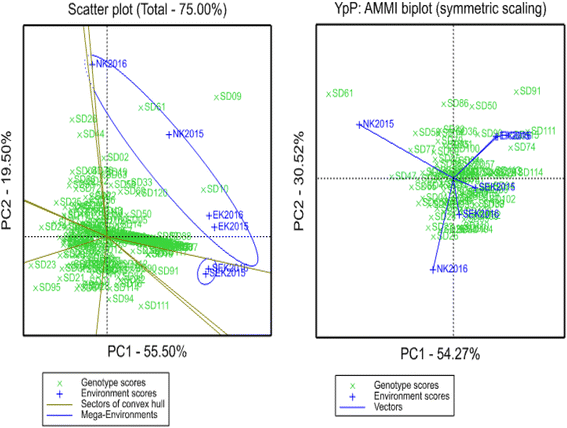
6个试验之间的Pearson相关性表明NK试验与EK和SEK位点无关。这一结果与GGE双标图和AMMI结果吻合较好。2).特别是,作为PC1的AMMI对称缩放试验将两个NK位点从EK和SEK的现场研究中分离出来(图1)。2 b).在三个区域的产量性能的ANOVA测试表明,环境(e)显着影响了基因型X环境互动,其中e贡献了81.9%,而G和GE仅在一起提供18.1%。
基于SNP标记的大豆遗传变异
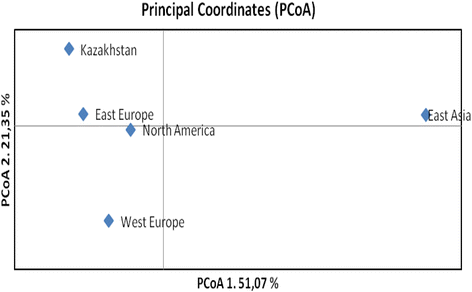
利用Illumina iSelect SNP阵列对大豆进行基因分型,共发现4442个多态SNPs(74.03%成功),其中77.98%的变异为转化,22.01%的变异为转化。主坐标分析(PCoA)允许所有113份材料根据其育种来源进行分组。这些资源被划分为5个地理上不同的组。每一组的加入人数参差不齐。东亚组只有3个样本,东欧组有67个样本(表)2).所述PCoA1图表显示,来自东亚基因型在遗传上从其它四组(图更远。3.).PCOA2有效地分开了剩下的四组,因为东欧和北美洲的进程似乎是最密切的团体,而哈萨克斯坦的加入物与来自东欧的品种最近的相似性(图。3.).
113种大豆种的主坐标分析根据其繁殖来源分为5组(附加文件6),基于4442个snp
关联映射
基因分型数据包括20条染色体上的4442个SNPs,平均长度为47.4 Mb,平均每条染色体上的SNPs数为222.1个。每条染色体的标记数在Gm11中为163个,在Gm13中为286个,在Gm16中为37.3 Mb,在Gm18中为62.2 Mb。SNP图谱的平均密度为每213kb一个标记。LD衰减曲线(附加文件1)在门槛r2= 0.1为20 Kbp。在GWAS中应用K + Q矩阵的MLM(混合线性模型),共鉴定了64个MTAs中的46个snp(附加文件)2).获得的结果进一步统计验证使用T-Test.用于识别假阳性mta。应用t检验后,在6个研究环境中发现了30个snp用于鉴定42个真mta(表1)3.).所揭示的42个mta与开花时间(VER2)和成熟时间(VER8)、株高(PH)、第一次分枝高度(HFB)、可育节数(NFN)、单株种子数(NSP)、千粒重(TSW)和单株重量(YPP)有关3.).结果表明,22只MTA与与适应相关性状相关的性状有关,20只MTA与产量组分有关。发现在两个或更多个地区中只发现了四种MTA,鉴定出在两种或更多个地区中具有重要意义,并且所有这些都与植物生长特征有关。在区域分布方面,为东部地区确定了9个MTA,在东南部和北部地区都有33个MTA(表3.).
对相关SNP标记的基因组物理位置分析表明,在全部鉴定的30个SNP中,有10个是CDS(编码DNA序列)的一部分,其余20个SNP位于基因间区域(附加文件)3.).根据邻近定位基因的实际邻近程度,考虑基因间位置的每个SNP作为潜在的功能注释。此外,每个关键SNP标记的物理位置叠加在已知qtl的位置上(https://soybase.org/search/qtllist_by_symbol.php).有趣的是,42个mta中有32个恰好位于类似QTL在大豆基因组中的位置4).例如,在TSW的3个MTA中,有2个与种子重量有关的QTL位于相同的位置,在NSP的4个MTA中有4个与种子重量有关的QTL,在PH的4个MTA中有4个与节间长度有关的QTL。(表4)。根据Soybase的数据,在9个mta中有8个SNPs未发现qtl,尽管8个SNPs中有4个位于CDS区域。
讨论
对哈萨克斯坦3个大豆产区5个不同育种来源的113份大豆材料的产量性能进行了比较。在大多数试验中,北美品种表现出最高的产量(图。1),支持这种遗传物质对KZ环境的优异适应性。值得注意的是,本研究中还包括一些当地高性能品种的血统基于美国繁殖大豆背景。另一方面,主坐标分析(PCA)表明,众多局部品种遗传更接近东欧种质,虽然北美和东欧加入彼此不远(图。3.).
研究的遗传图谱中遗传变异的大小和水平似乎对基于GWAS的项目的积极成果至关重要。实验结果表明,样本量小于384份的实验[17]及大型楼宇[18,19]可能导致识别错误的积极协会。另一方面,在Turner等人的研究中。2016年,显示较小的面板可以允许检测在较大的面板中未检测到的假阴性关联[20.].这些研究结果在相对较小的大豆样本量(n事实上证实了上述发现。首先,利用TASSEL MLM模型应用K + Q矩阵检测了64个mta。然而,进一步验证显示的关联使用T-Test.建议,其中只有42个是真正的协会。GWAS导致识别涉及42名确认的MTA的30个SNP标记。具有先前映射QTL的位置的识别SNP的物理位置的比较(https://soybase.org/search/qtllist_by_symbol.php)表明,在42个已鉴定的mta中,有32个与已知qtl位于同一位点。本研究发现的其余10个mta可能被认为是新的qtl。此外,对已鉴定的SNPs和已知qtl的物理位置进行高比例的阳性匹配,支持GWAS作为哈萨克斯坦大豆非随机mta搜索的有用研究工具。
也许在这项研究中的QTL新颖的鉴定有关的其他致病因素是特定的环境条件的其他文件中列出的影响5.在基因型x环境交互作用(GEI)评估中,通过方差分析检测出显著的环境影响(81.9%)对产量绩效的影响。4个mta在两次或两次以上的田间试验中表现显著,这显然可以解释为较大的环境影响导致鉴定的位点相对较低的稳定性。
本研究鉴定了24个与株高等植物适应性状相关的mta。在开花时间方面,在Gm06和Gm19染色体上发现了2个mta(表2)3.).GM06_20,370,075位于附近的近距离GmPhyA1基因,将其标注为对应于开花E1基因座的光敏色素受体21].Gm19_49,964,637已定位大约2.5 Mb从GmPhyA3基因,另一种与成熟E3位点相对应的光敏色素受体[22].四个鉴定的SNP,包括染色体GM10和GM19的SNP与成熟时间有关,其地图位置与遗传位置一致E2和E3成熟位点(21,22].Gm10染色体的Gm10_48,586,134距离约3.9 MbGmGIa(Glyma10g36600),一个涉及调节大豆成熟和开花时间的基因[23].染色体GM19的GM19_48,168,077,如开花MTA的情况下,距离开花大约1.5 MBGmPhyA3基因(Glyma19G41210)。值得注意的是,另一个SNPS GM07_10009107的位置邻近光敏色素乙(glyma07g11790)基因,通常编码对植物萌发的抑制作用[24].与PH的MTA相关的Gm09_42,241,644位点的SNP位点与之前定位的节间长度QTL (Soybase.org.)(附加文件3.).
第二组mta属于产量构成性状,包括NFN、NSP、TSW和YPP。总的来说,GWAS允许鉴定20个mta的产量组分分布在10个不同的染色体上(表)3.).
结论
共鉴定了30个与开花和成熟时间、株高、可育节数、单株种子和产量相关的SNP标记。42个mta中鉴定出的32个与已知类似qtl的物理位置吻合良好(www.soybase.org).这一结果表明揭示的mta对哈萨克斯坦大豆种植区的重要性。在获得额外证据之前,其他10个mta可能会提出一些新颖性要求。得到的结果将作为所需的必备形成和具体的育种计划实现有效的适应实现和哈萨克斯坦的三个不同区域增加了大豆的生产效率。
方法
植物材料
大豆样品由113份材料组成,包括18个已发布的品种和来自哈萨克斯坦的候选育种系(附加文件)6)在本研究中使用[1].植物研究材料来自5个地理区域的12个国家,包括西欧和东欧、北美、东亚和哈萨克斯坦。2015-2016年,在KZ的3个农业区进行了3次随机复制,所有113个品种都是在2015-2016年种植的。位于哈萨克斯坦东南部、东部和北部地区的作物育种站提供了专门的研究地块。此外,与EK和NK站点不同,这些地区的农田以灌溉农业为代表,有着明显的环境差异。具体位置,各自的气象数据和现场情况显示在附加文件5.植物以1米长的行中生长相邻的行和列内的植物之间5厘米空间30之间厘米的距离。
表型分析研究
统计分析采用GraphPad Prism 7.0 (https://www.graphpad.com/scientific-software/prism)及statisticka 13.2 (http://software.dell.com/products/statistica) 电脑程序。基因型 - 环境相互作用,包括AMMI(添加剂主要相互作用)和GGE双批准方法,通过Genstat软件包进行分析(https://www.vsni.co.uk/software/genstat/).两种方法的对称比例选择和所有三个站点的可用现场数据都用于估计。GGE双图的关键特性是它是基于Tester-Centered数据,去掉测试者(环境)的主效应(E),保留并合并入口主效应(G)和测试者交互入口(GE)。
DNA基因分型和遗传变异研究
DNA样品中提取并使用商业试剂盒(Qiagene,CA,USA)个别品种的单粒种子进行纯化。每个样品的DNA浓度调节至50ng /μl。所有样品均使用大豆5403 Illumina的I选择SNP阵列[基因型5tritgenetics GmbH (Gatersleben, Germany)。Illumina Infinium程序是根据制造商的协议进行的。采用Illumina Genome Studio软件(GS V2011.1)进行SNP基因型分析。采用GenAlEx 6.5进行群体遗传分析和主坐标分析[25].
映射研究协会
该SNP数据集用10%截断值与最小等位基因频率≥0.10被认为是用于GWAS丢失的数据和标志物过滤。假设组范围从k的数= 1〜10,使用50000老化迭代随后100000记录马尔可夫链迭代评估。估计总体结构推理的采样方差,五个独立的运行进行了针对每个k.对STRUCTURE HARVESTER中的delta K值(ΔK)进行了输出分析[26].在最后的基础k开发了三个识别的集群的值,Q矩阵。使用4442过滤的SNP对次要等位基因进行研究,研究了用于植物生长和产量组分的定量性状的GWA。基于MLM + Q + K型模型的基因组 - 宽关联映射使用流苏5进行[27].
参考文献
- 1.
Abugalieva S, Didorenko S, Anuarbek S, Volkova L, Gerasimova Y, Sidorik I, Turuspekov Y.不同纬度地区哈萨克斯坦大豆开花和种子成熟时间的评价。《公共科学图书馆•综合》。2016;11(12):1 - 11。
- 2.
Didorenko SV, Zakieva AA, Sidorik IV, Abugalieva AI, Kudaibergenov MS, Iskakov AR.作物生产多样化是大豆向哈萨克斯坦共和国北部地区推广的手段。生物科学生物技术研究。亚洲。2016;13(1):23-30。
- 3.
关键词:大豆,遗传-环境交互作用,基因型,遗传-环境交互作用1圣2 .“植物遗传学与基因组学与粮食安全”国际研讨会(PGGFS-2016),俄罗斯新西伯利亚,2016;
- 4.
David Grant D, Nelson RT, Cannon SB, Shoemaker RC。SoyBase,美国农业部大豆遗传学和基因组学数据库。核酸Res. 2010;38: 836 - 836。
- 5。
宋Q,Hyten DL,贾G,Quigley CV,Fickus Ew,Nelson Rl等。大豆,大豆高密度基因分型阵列的发展与评价。Plos一个。2013;8(1);E54985 DOI:ORG /https://doi.org/10.1371/journal.pone.0054985.
- 6。
阚刚,张伟,杨伟,马东,张东,郝东,胡震,于东。盐胁迫下大豆种子萌发的定位。基因组学。2015;290:2147-62。
- 7。
曾安华,陈鹏,史志强,陈鹏。大豆品种耐盐性的全基因组关联研究(GWAS)。摩尔繁殖。2017;37:30。https://doi.org/10.1007/S11032-017-0634-8。
- 8。
郝东,程华,尹震,崔胜,张东,王辉,等。大豆(Glycine max)地方品种产量和产量构成因素单核苷酸多态性和单倍型的鉴定Theor Appl Genet. 2012; 124:447-58。
- 9。
黄安英,宋强,贾刚,Specht JE, Hyten DL, Costa JM,等。大豆籽粒蛋白质与籽粒含油量的全基因组关联研究。BMC基因组学,2014;15 (1)https://doi.org/10.1186/1471-2164-15-1.
- 10.
张捷,宋之问,Cregan PB,江GL。全基因组关联分析,基因组预测和大豆为种子重量的标记辅助选择(大豆)。理论应用遗传学。2016; 129:117-30。
- 11.
基于单倍型分析和SNP标记的大豆农艺性状全基因组关联研究。《公共科学图书馆•综合》。2017;12 (2)https://doi.org/10.1371/journal.pone.0171105.
- 12.
Wallace GJ, Zhang X, Beyene Y, Semagn K, Olsen M, Prasanna BM, Buckler ES。撒哈拉以南非洲15个热带玉米种群在管理干旱胁迫和充足水分条件下的株高和开花时间全基因组协会作物科学。2016;56(5):2365 - 78。
- 13.
Bandillo N,Jarquin d,宋Q,纳尔逊R,Cregan P,施佩希特Ĵ,洛伦茨AA。人口结构和全基因组关联分析,美国农业部大豆种质资源收集。植物基因组中。2015; 8(3)
- 14.
牛勇,徐勇,刘雪峰,杨淑霞,魏素萍,谢峰涛,张玉梅。大豆品种种子大小和形状性状的关联分析。摩尔繁殖。2013;31:785 - 94。
- 15.
Wen Z, Boyse JF, Song Q, Cregan PB, Wang D.大豆基因组选择结果和全基因组关联作图。BMC基因组学。2015;16:671。https://doi.org/10.1186/s12864 - 015 - 1872 - y.
- 16。
基于GBS-GWAS方法的大豆8个农艺性状位点鉴定及QTL定位验证植物生态学报。2015;13:211-21。
- 17。
王H,史密斯kp,combs e,blake t,horsley rd,muehlbauer gj。群体大小和不平衡数据集对大麦繁殖种质基因组关联映射QTL检测的影响。2012年Ampl Genet; 124:111-124。
- 18。
王静,楚S,张辉,朱Y,程H,得月Y.发展和新的全基因组SNP阵列的应用揭示了大豆的驯化历史。SCI代表2016; 6(20728):1-10。
- 19。
张志刚,张志刚,张志刚,张志刚。关联定位:从基因分型到实验设计的关键考量。植物细胞。2009;21(8):2194 - 202。
- 20.
特纳MK,KOLMER JA,Pumphrey Mo,Bulli P,Chao S,安德森JA。春小麦核心集合中叶锈蚀位点的关联映射。理论应用遗传学。2016; DOI.https://doi.org/10.1007/s00122-016-2815-y.
- 21.
等。大豆成熟位点E1、E2、E3和E4基因的自然变异。安机器人。2014;113:429-41。
- 22.
渡边S,Hideshima R,夏Z,坪仓Y,佐藤S,中本Y,等人。与大豆基因E3相关的基因的克隆基于地图的。遗传学。2009; 182:1251年至1262年。
- 23.
渡边S,夏Z,Hideshima R,坪仓Y,佐藤S,山中N,等人。采用剩余杂合体的地图为基础的克隆策略揭示了GIGANTEA基因参与大豆的成熟和开花。遗传学。2011; 188(2):395-407。
- 24.
施穆茨Ĵ,佳能SB,Schlueter的Ĵ,马静,Mitros T,纳尔逊W,等人。在palaeopolyploid大豆的基因组序列。自然。2010; 463:178-83。
- 25.
Peakall R, mouse PE。GenAlEx 6.5: excel中的遗传分析。用于教学和研究的人口遗传软件更新。生物信息学。2012;28:2537-9。
- 26.
利用结构化软件检测个体聚类数量:一项仿真研究。摩尔生态。2005;14:2611-20。
- 27.
Bradbury PJ, Zhang Z, Kroon DE, Casstevens TM, Ramdoss Y, Buckler ESTASSEL。用于不同样本中复杂性状关联映射的软件。生物信息学。2007;23:2633-5。
确认
作者愿意承认哈萨克斯坦共和国教育部的资金,为项目1108 / GF4。
资金
本文的发表得到了哈萨克斯坦共和国教育和科学部支持的项目1108/GF4的资助。
数据和材料的可用性
支持本文结论的数据集包含在本文中。大豆收集的材料存放在植物生物学和生物技术研究所(哈萨克斯坦)。
关于这个补充
本文已作为BMC Plant Biology Volume 17 Supplement 1, 2017: Selected articles from PlantGen 2017的一部分发表。该补充的全部内容可在网上找到//www.cinefiend.com/articles/supplements/volume-17-supplement-1.
作者信息
从属关系
贡献
SAb和YT进行了试验设计,SD、EG和IS在哈萨克斯坦三个不同地区进行了田间试验,AZ和SAn分析了SNP数据并进行了统计分析,AZ进行了GWAS、YT、SAb和AZ参与了手稿的编写。所有作者都审阅了手稿的初稿。所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
本研究不包含任何需要伦理同意或批准的研究。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Zatybekov, A., Abugalieva, S., Didorenko, S.等等。哈萨克斯坦育种库收集的大豆农艺性状的GWAS。BMC植物BIOL.17,179(2017)。https://doi.org/10.1186/s12870-017-1125-0
关键词
- 大豆
- GWAS
- 单核苷酸多态性
- 产量构成